- 研究文章
- 开放获取
- 发表:
一种越南特有物种的从头组装和转录组特征,人参vietnamensisHa Grushv。,including the development of EST-SSR markers for population genetics
BMC植物生物学体积20.文章编号:358(2020)
摘要
背景
了解森林残余物中濒危物种的遗传多样性,对制定有效的物种保护、恢复和管理策略具有重要意义。人参vietnamensisHa Grushv。是越南重要的药用、特有和濒危物种。然而,由于缺乏有效的分子标记,种群的遗传多样性和结构尚不清楚。
结果
在本研究中,我们使用Illumina HiSeq™4000测序技术分析p . vietnamensis(根,叶和茎)。共获得23741783个Raw reads并进行组装,其中生成的unigenes为89271个(平均长度为598.3191 nt)。31686个unigenes分别在Gene Ontology、Kyoto Encyclopedia of Genes and Genomes、Nucleotide Collection (NR/NT)和Swiss-Prot数据库中进行功能注释。此外,检测到11343个EST-SSRs。从7774对引物中筛选出101对进行多态性验证,其中;20对引物成功扩增到DNA片段,在群体内观察到大量多态性。利用9个多态微卫星位点进行种群结构和多样性分析。结果表明,群体间遗传多样性较高,平均观察杂合度和期望杂合度均为HO= 0.422, HE分别为= 0.479。在使用TPM和SMM模型进行瓶颈分析时(p< 0.01)表明目标群体存在明显的杂合子缺陷。这表明在所有种群中都有瓶颈的迹象。居群间遗传分化适中(F圣= 0.133),表明基因流动水平略高(纳米= 1.63)。分子变异分析(AMOVA)显示个体间变异率为63.17%,居群间变异率为12.45%。我们的研究结果显示了两个与地理距离相关的遗传簇。
结论
我们的研究将有助于保护人员在未来的保护管理、繁殖、生产和生境恢复。
背景
人参种(五棱科)是北美洲和东亚的重要药用植物[1,2].在19种人参属(3.,4,三个已知物种,人参vietnamensis,p . stipuleanatus而且p . bipinatifidus与越南的高山有关[5,6].人参品种的特征是存在人参皂苷,指一系列达玛烷[7].p . vietnamensis首次发现于今都省的Ngoc Linh山[8].p . vietnamensis(越南人参)是越南特有的一种人参,富含皂苷类化合物[1,8,9].它是一种多年生植物,高可达1米,在腐殖质林冠下直径可达4-8毫米。它的叶子呈椭圆形,边缘呈锯齿状。花为花序,成熟时果实变红,有1-2颗白色种子。p . vietnamensis通常用于治疗许多严重的疾病和增强身体耐力的山地旅行的色当族[1].过去几十年的过度开发和增长速度缓慢,再生能力差p . vietnamensis自然种群急剧减少,使该物种濒临灭绝。10].因此,它被列入2007年越南红皮书(EN A1a,c,d, B1 + 2b,c,e) [11].目前它已被列入越南中央和地方政府的保护名单。它急需保护和恢复,但主要的障碍是尚未开发的结构和遗传多样性p . vietnamensis野生群体由于缺乏信息和可靠的分子标记p . vietnamensis。
简单序列重复序列(SSRs)标记是植物遗传学研究、育种、个体、物种和品种鉴定以及生成遗传地图的有用工具,因为它们广泛分布在基因组中,具有高水平的多态性、共显性遗传、丰度、最大可重复性、多等位基因变异和良好的基因组覆盖率[12,13,14,15,16,17,18].表达序列标记(EST)的有效性,增强了部分木本植物SSR鉴定的可能性[19].由表达序列标记(EST-SSRs)产生的SSRs作为一种功能分子标记,由于其与控制表型的功能基因的紧密联系,可以研究环境异质性和局部适应的影响[20.,21,22].迄今为止,许多EST-SSRs被开发出来,并在许多物种中进行多态性检测,如红薯[23),胡麻属indicum[24],萝卜[25),兰花sinense[26,杨梅[27,银杉[28),柳树,杨树,桉树[29];罗莎roxburghii[30.),Neottopteris病灶[16,漆树[31,面包小麦[32, Proso Millet [33)、杏仁(34和人参[35].有限的基因组资源已被开发用于人参迄今为止的物种,例如,p . vietnamensisvar。fuscidiscus[2),p .人参[35,36和其他作物相比。
Illumina HiSeq™4000是一种允许对基因表达谱进行全面分析的新一代方法,为生命科学提供了迷人的机会,并促进了转录组测序,以低成本和快速识别EST-SSRs [16,22,25,26,34,35,37,38,39].转录组从头组装对于非模式植物研究中的功能基因组学或标记挖掘是必不可少的,特别是在基因组序列不可用时[16,23,30.,37,38,39].到目前为止,只有核苷酸序列p . vietnamensisvar。fuscidiscus而且p .人参在国家生物技术信息中心(NCBI)数据库可用(2019年8月),而在GenBank中没有ESTsp . vietnamensis.前人对其遗传变异进行了研究,证实了其分类学地位人参分子层面的物种[40,41,42,43,44,45,46,47,48,49].然而,很少有研究者对此进行研究p . vietnamensis在越南50,51,52].
在目前的研究中,(i)我们从p . vietnamensis使用Illumina HiSeq™4000分析得到的转录本的功能、分类和代谢途径。(ii)然后我们开发了一套EST-SSRs用于p . vietnamensis(iii)通过对3个野生居群的遗传结构和多样性的研究,证实了这些标记的有效性p . vietnamensis.(iv)最后检验了地理距离对野生种群内基因流动的影响。
结果
从头组装和Illumina测序p . vietnamensis转录组
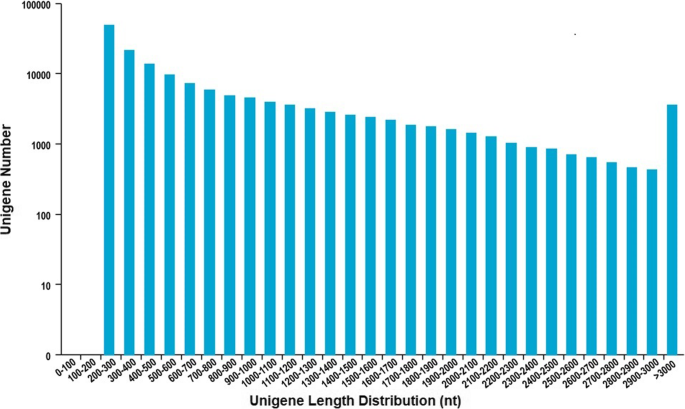
转录组测序的p . vietnamensis共产生7,083,775,547个碱基,经过严格的质量检查,获得了23,741,783对端高质量、干净的reads,其中Q20和Q30碱基分别为97.52%和93.5%,GC含量为51.43%。通过Trinity对De novo assembly进行进一步检测,共检测到153074个转录本,长度为117,954,630 bp, N50值为1,268 bp,平均长度为770.572 bp。在转录本总数中,200 ~ 300 bp的转录本有48314个(31.56%);35174个(22.98%)转录本范围在301 ~ 500 bp之间;32031(20.93%)转录本范围在501 ~ 1000 bp之间;25800份(16.85%)转录本在1001 - 2000 bp之间,11755份(7.68%)大于2000 bp。同时,该组合共产生89,271个unigenes, N50长度为942 bp(平均长度为598.319 bp)。在200 ~ 300 bp之间的unigenes有39,947个(44.75%);22049人(24.70%)在301 ~ 500 bp之间;13669人(15.31%)在501 - 1000 bp之间; 9048 (10.14%) ranged from 1001 to 2000 bp and 4558 (5.11%) were larger than 2000 bp (Fig.1、表1).
装配unigenes的功能注释
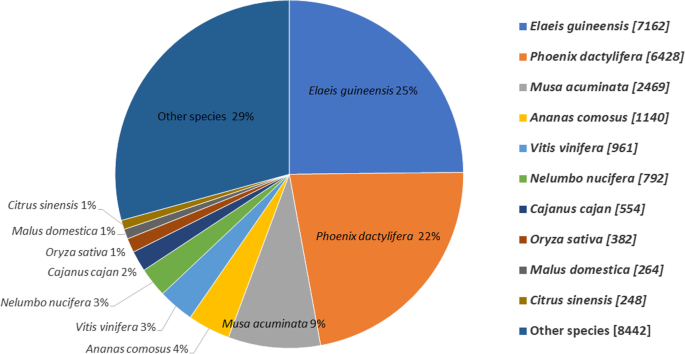
对7个数据库(COG、GO、KEGG、KOG、Pfam、Swissprot和NR)的unigenes进行功能注释分析,共发现31686个匹配序列(表1)2).在89,271个unigenes中,成功注释7647个(8.57%)齿轮数据库,14568个(16.32%)去数据库,5838 (5.42%)KEGG数据库,16,860 (18.89%)KOGPfam蛋白数据库18,600(20.845)个,Swiss-Prot蛋白数据库19,228(21.54%)个,NR蛋白数据库16,659(18.66%)个unigenes2).对于物种分布BLASTx是用来对Nr数据库进行搜索的p . vietnamensis转录组与Elaeis guineensis(25%)紧随其后凤凰dactylifera(22%)和穆萨acuminata(9%)(图。2).
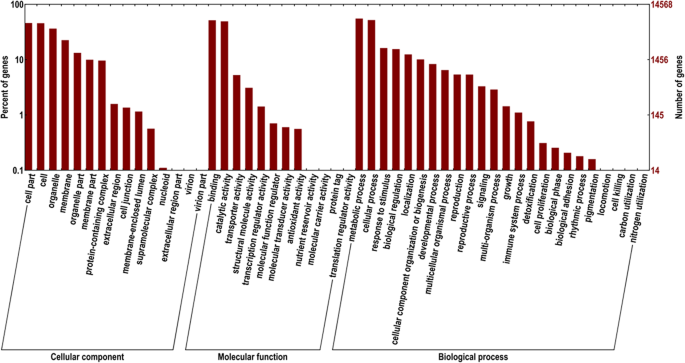
基于Nr注解,我们用了去系统对unigenes的可能功能进行分类。共72,183个(80.86%)unigenes被成功分为3类(生物过程、分子功能和细胞成分)和51个亚类(图1)。3.).生物过程是最主要的一类(28,653;39.69%),而亚类别为“代谢过程”(8016;27.98%)“细胞过程”(7528;26.27%)和“对刺激的反应”(2347;8.19%)。细胞组分unigenes为27232个(37.72%),分类为“细胞部分”(6645个;24.40%)“细胞”(6596;24.22%)和“细胞器”(5269;19日,35%)。 The 16,298 (22.58%) unigenes were related to “molecular function” in which prominent subcategories are “binding” (7459; 45.77%) and “catalytic activity” (7130; 43.75%). It was also observed that the few genes are enriched in the terms of “nutrient reservoir activity”, “molecular carrier activity”, “protein tag” and “translation regulator activity”.
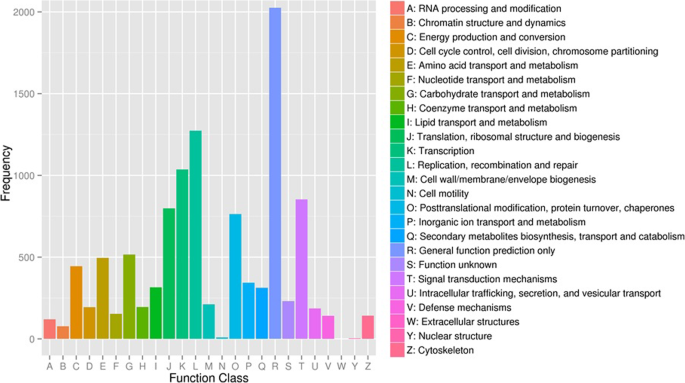
共7647个unigenes被分配到cluster of Orthologous Groups (COG),以检查转录组文库的可靠性和注释过程的有效性,用于功能预测和分类(图1)。4).cog注释的推定蛋白在功能上分为25类。排名靠前的组分别是“一般功能预测”(9089)、“翻译、核糖体结构和生物发生”(3388)和“转录”(977)。然而,只有少数unigenes被注释为“细胞外结构”和“核结构”。
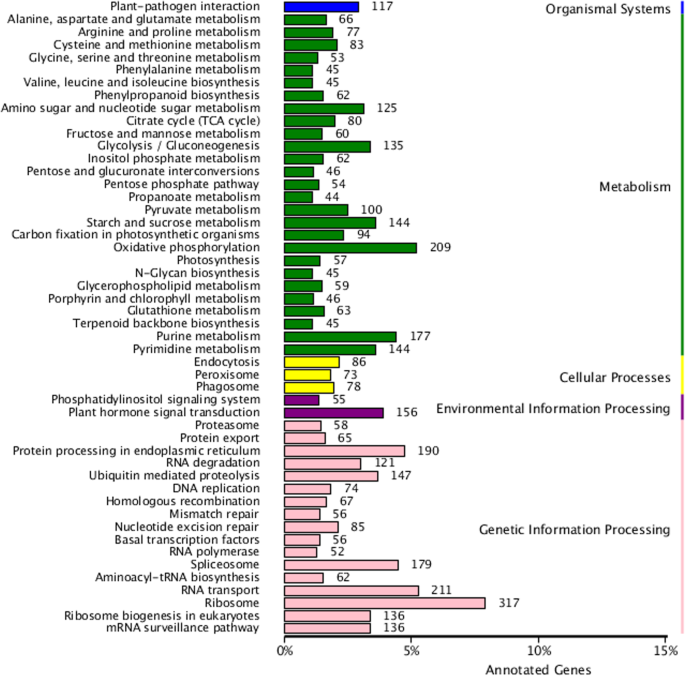
采用KEGG通路分析方法,探讨其生物学通路p . vietnamensis当E值截止值< 10时,它可能是活跃的−5.5838个unigenes在KEGG数据库,分配给118KEGG功能通路(图。5).具体途径包括植物激素信号转导、嘌呤代谢、核糖体、RNA转运剪接体等多种途径。另外,有45个unigenes位于萜类主干生物合成通路中。
EST-SSR标记的开发和鉴定p . vietnamensis转录组
为了开发新的分子标记和检查组装质量,89,271个unigenes被用于微卫星挖掘,这些unigenes被定义为二核苷酸到六核苷酸基序。使用SSRIT鉴定了11343个EST-SSRs。6949个序列包含1个SSR, 2763个序列包含1个以上SSR。EST-SSRs频率为12.71%,单个EST-SSRs分布密度为5.98千碱基(kb)。
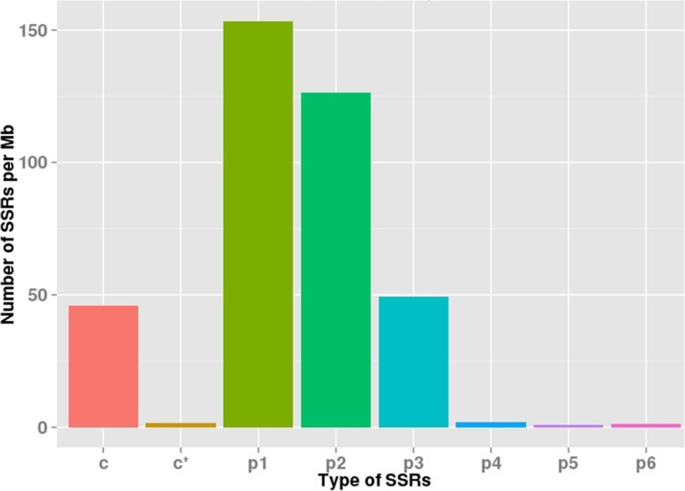
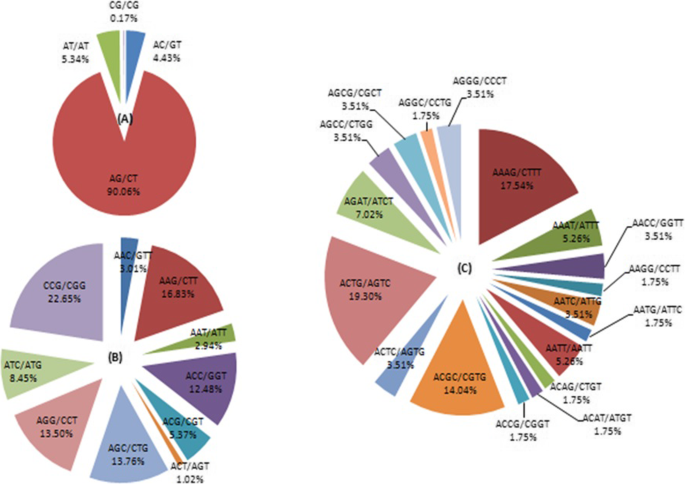
分析了潜在的EST-SSRs的频率、类型和分布。最常见的重复基序是单核苷酸(5004;44.12%),其次是双核苷酸(4648;40.98%), tri-nucleotide (1563;13.78%), tetra-nucleotide (66;0.58%), hexa-nucleotide (29;0.26%)和五核苷酸(32;0.28%)重复(无花果。6;表格3.).带有十个重复基序的EST-SSRs (2040;20.50%), 6个重复基序(1363;12.02%), 5个重复基序(925;8.15%), 7个重复母题(862个;7.6%), 8个重复基序(594个;5.24%), 9个重复基序(428个;3.77%)最为常见。双核苷酸重复序列中以AG/TC(90.06%)为主,其次为AT/TA(5.34%)和AC/TG(4.43%)。在第10型三核苷酸重复序列中,基序分布最高的是CCG/GGC(22.65%),四核苷酸重复序列中常见的基序是ACTG/TGAC(19.30%)(图4)。7).此外,EST-SSRs分别检测到16和17个不同类型的五核苷酸和六核苷酸重复序列。
群体的遗传结构与多样性
一共98个人从三个p . vietnamensis群体产生27个不同的等位基因,在9个位点上从120到265 bp不等(表4).在本研究中,多态性信息含量(PIC)值范围为0.325 (L111) ~ 0.493 (L145),平均值为0.361。在98个个体中,每个位点(A)检测到的等位基因数从两个位点(L119和L145)的2个到两个位点(L37和L111)的4个不等,平均值为3。最低检测杂合度(HO)在L73位点最多,为0.178,L111位点最高,平均为0.422。同样,最低期望杂合度(HE)为L73位点(0.208),L37位点最高(0.65),平均0.479。各位点在种群中的固定指数(F)平均值为0.14,范围为- 0.185 (L111) ~ 0.386 (L115)。
在种群水平上,遗传多样性值见表5,包括等位基因平均数(A = 2.6),有效等位基因数(A = 2.6)E= 2.2)、多态位点比例(92.59%)、观察到的杂合度(HO= 0.422)和期望杂合度(HE= 0.479)。固定指数(F)在所有种群中均为阳性(F = 0.13)。因此,这些结果显示了杂合子缺陷和显著的近交系(p< 0.05)。9个基因座中有7个基因座阳性固定,提示高纯合度和近交。但有5个位点发生了显著近交(p< 0.05)。两个位点为负值。
在采用逐步变异模型(SMM)和两阶段模型(TPM)进行瓶颈分析时(p< 0.01)表明目标人群存在明显的杂合子缺陷5).这表明在所有人口中存在瓶颈的迹象。
分子方差分析(AMOVA)显示总变异高度显著(p< 0.001),即63.17%6).的F圣是重要的(p< 0.05),取值范围为0.072 ~ 0.182(平均值为0.133),基因流量为1.63。低遗传分化值(F圣= 0.072),而高值(F圣= 0.182)7).
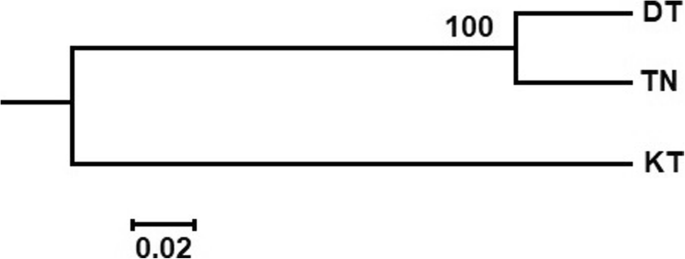
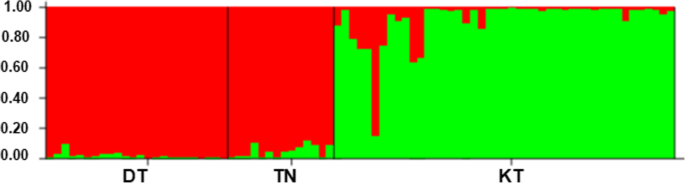
遗传关系p . vietnamensis居群如图所示。8.DT和TN种群被分组在一起,并以100%的自举值固定为一个聚类。在结构分析中,最大∆K值为2032.81(图1)。S1结果表明,K = 2是遗传簇的最佳数目,表明所有研究植物均表现为两个遗传簇的混合。两个遗传群体中每个群体和个体的血统百分比表明,一个群体(红色)在两个群体(DT和TN)中占优势,第二个群体(绿色)在一个群体(KT)中占优势(图。9).
讨论
转录组测序/分析是基因鉴定非常有效的工具[53,54,55,56]及识别细胞在不同发育阶段或生理条件下的基因表达[47].Illumina HiSeq™4000技术是一种高效、持久、经济、可靠的工具,可用于非模式植物的转录组描述和基因检测。以前的研究表明,无害环境技术的数量产生于p .人参叶子(48,57),三七根(58)和西洋参(p . quinquefoliusL.)花、叶和根[44].到目前为止,许多研究人员已经研究了用于遗传分析的分子标记人参即物种。p .人参[40,41,42,46,48,49),三七[45].然而,由于没有参考基因组p . vietnamensis,使用Illumina HiSeq™4000生产的读数通过从头组装器Trinity组装。我们首次报道了EST-SSR标记发育的完整转录信息,并对现存的天然植物的多样性和遗传结构进行了探讨p . vietnamensis人群。
Illumina配对测序技术产生了23,741,783个清洁高质量reads, Q30碱基93.5%,GC含量51.43%。目前的结果高于之前的研究[2)p . vietnamensisvar。fuscidiscus(43.25%),说明测序质量更好。在序列组合中记录到89271个unigenes(平均长度为598.32 bp, N50为942 bp),比Cao等的结果短。[47在p .人参(平均长度= 690-698 bp, N50 = 1130-1161 bp)和Zhang等。[2在p . vietnamensisvar。fuscidiscus(平均长度= 1304 bp, N50 = 2018 bp)。我们使用了同样的技术;这可能是测序的深度,组装的方法和植物的自然特征。转录组测序数据p . vietnamensis进一步对遗传多样性、群体结构和标记开发进行了探讨。72,183个(80.86%)unigenes被注释为51个GO分类。在生物活性、分子功能和细胞成分中,“代谢活性”、“结合活性”和“细胞部分”分别居首位。结果与GO的功能分类一致p . vietnamensisvar。fuscidiscus[2),p .人参[47].通过KEGG通路预测的unigenes 5838(5.42%)被映射到118条生物学通路中,其中大部分通路与代谢有关。具体途径包括核糖体、RNA转运剪接体、嘌呤代谢和植物激素信号转导等。这些数据揭示了活跃的代谢过程以及各种代谢产物的合成。在p . vietnamensis和其他人参物种,叶子有高价值的醛,酯和萜类化合物,这些化合物有助于抵抗生物和环境压力,如寒冷,干旱和害虫。在目前的研究中,我们记录了植物激素对植物环境条件作出反应的信号转导的unigenes。
微卫星分布在植物基因组中,参与调控其表达和功能[13,59].对SSR在物种中的分布、变异机制和比较的研究是阐明其功能的第一步[59,60].SSR标记种类繁多,在植物基因组中广泛分布[43,61].在8927个unigenes中,通过RNA测序鉴定出11343个EST-SSR分子标记,其中2763个unigenes具有多个EST-SSR位点。张等人。[2也鉴定出21,320个SSRsp . vietnamensisvar。fuscidiscus其中2918个包含多个SSR。在Um等人之前的研究中[49)p .人参和张等人。[2)p . vietnamensisvar。fuscidiscusEST-SSRs中双核苷酸重复序列最多,分别为60.1和52.25%。我们已经鉴定出SSR标记(11343)p . vietnamensis,单核苷酸(5004;重复次数最多的是二核苷酸(4648,40.98%)和三核苷酸(1563,13.78%)。以二核苷酸、三核苷酸、四核苷酸重复基序为主p . vietnamensisAG/TC(90.06%)、CCG/GGC(22.65%)、ACTG/TGAC(19.30%)。这证实了Um等人的研究。[49)p .人参,但三核苷酸重复基序不同p . vietnamensis比其他植物,如Myricarubra[27),柳树,杨树而且桉树[29].CG/CG基序(0.17%)不规则检出p . vietnamensis, Wu等人也观察到了这一点。[44],证实了CG/CG重复基序在许多双子叶植物中并不常见[62,63,64,65].
遗传多样性是种质改良的重要特征,广泛应用于各种药用植物[65,66,67,68].许多植物的遗传多样性程度与基因座和种群数量有关[31,69],地理范围大小[70]和基因交换[71].在本研究中,9个SSR标记表现出较高的遗传多样性p . vietnamensis群体和期望杂合度(HO= 0.422, HE= 0.479)人参物种,如p . stipuleanatus[50),p .人参[72,73然而,我们的结果与reuova等人的研究一致。[46)p .人参俄罗斯的天然物种(HO= 0.453, HE= 0.393), Liu等。[74)三七(HE= 0.350)和reuova等人。[75)p . vietnamensis(HE= 0.55)使用微卫星标记。有三个基因多样性高p . vietnamensis居群、TN、DT和KT均显示该种以异交为主。高基因流(N米> 1)可能是三个种群高异交率的结果。昆虫传播花粉可能被认为是这个物种的一个主要因素。所有病例均检出阳性固定指数p . vietnamensis由于近交系繁殖,杂合度不足。这表明邻里面积小,种群中兄弟姐妹之间的关系也不融洽。我们的研究结果还显示,在所有三个研究人群中都出现了瓶颈迹象(p< 0.005)。在TPM和SMM条件下,TN、DT和KT三个居群均存在显著的杂合度缺陷(p< 0.005)模型。这些模型表明目标种群的数量会减少。
F圣是测量群体中基因流动和遗传变异的清晰方法[76].基因变异p . vietnamensis人口是温和的。低F圣两个居群(DT和TN)之间的值可以促进居群内强的基因流动(N米= 3.2)。然而,TN和DT两个居群之间的遗传变异水平较低(F圣= 0.072)可能与地理距离有关。这两个种群位于广南的同一个省。AMOVA分析结果显示,63.17%的变异分布在个体内部,12.45%的变异分布在群体内部。这些结果显示出适度的遗传结构p . vietnamensis.群体间的遗传变异受遗传漂变、基因流动、突变、选择和长期进化的影响很大[77].长寿和异交物种在群体中保持高度的遗传变异和低的遗传分化,反映了最大水平的基因流动。以前的研究报告了种群之间的低分化,反映了强烈的基因流动p .人参[46,72),p . stipuleanatus[50].群体间强烈的基因流动可能是由于群体内的高异交率。因此,花粉粒通过昆虫的传播可以被认为是影响种群结构的一个主要因素。贝叶斯分析和UPGMA树显示了两组不同的遗传混合个体p . vietnamensis.在当前的研究中,我们通过地理距离将不同省份的人口隔离。DT和TN两个居群在广南省内闭合形成遗传聚类,而今都省KT居群因地理距离分离形成聚类,两居群之间的基因交换受到限制。的p . vietnamensis面临着严重的生存威胁。基于我们的研究结果,所有研究的种群都可以考虑就地和迁地保护策略。
结论
De-novo转录组测序p . vietnamensis由Illumina测序平台完成。我们产生了大量的ESTs,并确定了在p . vietnamensis.共鉴定出11343个EST-SSRs。从数据中可以明显看出p . vietnamensis保持高水平的遗传多样性。大量的SSR标记被鉴定出来,将有助于标记辅助育种p . vietnamensis.本研究不仅为p . vietnamensis育种也是其保护的平台,保持遗传多样性。
方法
植物材料
我们在液氮中采集了植物的根、叶和茎p . vietnamensis(无花果。10a)从野生种群(广南省)提取RNA,储存在−80°C。p . vietnamensis利用广南省(10种植物)野生群体进行EST-SSRs的开发,以检验合成的EST-SSR引物的扩增相关性(表2)8).三种不同的野生居群(98种植物)p . vietnamensis以评估结构和遗传多样性(图。10b,表8).野生种群p . vietnamensis分别于2019年春季和夏季采样。为了提取DNA,新鲜的叶子在硅胶中干燥。这个物种被阮氏芳庄博士鉴定为人参vietnamensisHa et Grushv(百分比鉴定:100%),并进一步通过核基因序列数据(ITS-rDNA), Genbank登录号为MH238443。在广南省和今都省采集样本的许可(2019年2月20日第123号/QĐ-STTNSV函和2019年5月10日第819号/QĐ-STTNSV函)由生态和生物资源研究所(IEBR)批准,并由广南省和今都省人民委员会进一步确认。该物种的凭证标本保存于越南科学技术研究院生态与生物资源研究所分子系统学与保护遗传学研究室。
RNA提取
用OmniPlant RNA Kit (DNase I)从每个样本(根、叶和茎)中提取总RNA,用于Illumina测序。RNA的质量和数量通过Nanodrop和1.2%琼脂糖凝胶电泳验证[18].将RNA(每个样本等量)汇集在一起,送往育种生物技术有限公司,使用Illumina HiSeq™4000进行转录组测序。
转录组测序和De novo组装
纯化后的mRNA用Oligo (dT)从200 μg总RNA中提取cDNA文库。以mRNA为模板,从随机六聚体中提取cDNA第1链,从缓冲液、dNTPs、RNase H和DNA聚合酶I中提取cDNA第1链,用AMPure XP珠清洗。将清洗后的双链cDNA进行末端修复,结扎测序连接子,用AMPure XP珠筛选片段大小。通过PCR富集获得cDNA文库。在生物分析仪(Agilent 2100)上进行文库验证后,育种生物技术有限公司在MiSeq (Illumina HiSeq™4000)上对cDNA文库进行测序。
Trimmomatic v3.0 [78]用于原始reads过滤。将显示适配器污染、长度< 36 bp和低质量值(质量< 20)高于15%的读数剔除。三一(79]和默认参数用于清洗后的读的从头组装。然后TIGR基因指数聚类工具(TGICL) v2.1 [80]用于聚类和清除多余的转录本,并识别unigenes用于进一步分析。
注释和功能分类
在功能注释方面,所有unigene序列都与NCBI非冗余(NR)蛋白序列进行比较[81], Swiss-Prot [82,基因本体论(GO) [83],同源组簇(COG) [84], KOG [85和KEGG [86]数据库使用BLAST软件[87来预测氨基酸。然后使用HMMER软件对序列进行排列[88]与蛋白质家族(Pfam) [89数据库获取Unigene注释信息。
EST-SSR标记的开发、检测和引物设计
聚集在一起p . vietnamensis利用MISA(微卫星识别工具)挖掘转录组标记。根据Jurka和Pethiyagoda的说法,候选SSRs从2到6个核苷酸范围定义为二核苷酸6个重复序列,所有高阶基序5个重复序列[90].端到端EST-SSRs(中断< 100 bp)被认为是复合EST-SSRs。分析了unigenes中utr和orf中不同核苷酸重复序列的分布。对注释的富含ssr的unigenes进行GO分析,以评估其意义。引物5.0 [91]用于ETS-SSR引物设计,以生成100-300 bp大小的PCR产物。引物长度为18 ~ 24 bp,最优为20 bp,退火温度为55 ~ 65℃,最优为60℃。多态最大化准则用于多态位点的选择。为了使多态性最大化,对于二核苷酸、三核苷酸和四核苷酸,分别选择重复最少10个、7个和5个的SSR位点。反应在25 μl的体积,2 μl基因组DNA(共10 ng), 12.5 μl Master Mix 2X,每个10 μM引物1 μl和9.5 μl H中进行2去离子的。循环条件为95°C 3 min,然后35次循环,94°C 45 s, 60°C 45 s, 72°C 45 s, 72°C最后延伸10 min。PCR产物按照Vu等的方法进行分离、测定和分析。18].
数量遗传分析
目的:评价小檗种群结构和遗传多样性p . vietnamensis筛选了101个多态SSR标记,成功扩增了20对引物。在所选SSR标记中,有9个标记具有清晰、可重复的图谱,被选为研究对象2).DNA分离试剂盒(Norgenbiotek,加拿大)用于基因组DNA提取。样品用混合磨MM 400 (Retsch)在液氮中粉碎。根据Vu等人的方法验证DNA质量和浓度。[18].将浓度稀释至10 ng/μl。
PCR量为25 μl,其中基因组DNA 2 μl(共10 ng), Master Mix 2X 12.5 μl, 10 μM引物各1 μl, H 9.5 μl2去离子的。反应在热循环器条件下被放大:在94°C初始变性3分钟,在94°C下循环40次,在54-56°C退火温度下30秒,引物对(每个)在72°C下延伸1分钟,在72°C下最后循环10分钟,然后在4°C保存样品直到分析。使用Sequi-Gen®GT DNA电泳系统,用8%聚丙烯酰胺凝胶在1 x TAE缓冲液中分离扩增产物,然后用GelRed™核酸凝胶染色仪进行可视化。采用gengens1850 (Clinx Science Instruments Co., Ltd)的Gel-Analyzer软件,用20 bp的DNA ladder (Invitrogen)检测等位基因大小。
在GenAlEx上分析遗传参数[92],用多态位点的比例(P)、有效等位基因(一个E),每个位点的等位基因数(一个).观察到的杂合性(HO),期望杂合度(HE),固定指数(F),两两居群间的分化指数(F圣),的矩阵F圣在不同种群之间和基因流动之间(纳米)由公式计算纳米= [(1/Fst) - 1]/4 [93多态信息含量(PIC)值根据Botstein等计算。[94Genepop v.4.6对每个群体中每个基因座成对组合的Hardy-Weinberg平衡的偏离和连锁不平衡进行了检验[95].使用Botteneck v.1.2测试通过SSM和TPM的种群(每个)的最近瓶颈事件[96].这些检验的显著性用单尾Wilcoxon符号秩检验来衡量。日志含义默认情况下,SSM占比设置为70%。利用GenAlEx计算种群间的遗传距离。F的意义圣通过应用顺序Bonferroni校正来检验所有位点上每个群体对的值。
采用Arlequin 3.1对数据进行AMOVA分析,并在10,000个排列基础上进行显著性检验[97].UPGMA方法通过poptre2确定种群间的遗传关联[98].STRUCTUREv.2.3.4采用贝叶斯聚类方法探索种群结构[99].使用相关的等位基因频率设置混合模型,即在数据集(K)中,在不同的组中,在50万次马尔可夫链蒙特卡罗(MCMC)重复和10万次磨合周期下,对K在1和15之间进行10次单独的运行。收割机结构(One hundred.]用于根据Evanno等人的研究,在基于∆K的数据集中进行最适合的组(数)检测。[101].
数据和材料的可用性
支持结果和结论的数据图表包含在文章和附加文件中。部分SSR序列数据存放在NCBI,登录号为MK802095 ~ MK802103 (https://www.ncbi.nlm.nih.gov/).
缩写
- AMOVA:
-
分子方差分析
- 齿轮:
-
同源基团的簇
- EST-SSRs:
-
表达的序列标记-简单序列重复
- 走:
-
基因本体论类别
- KEGG:
-
京都基因和基因组途径百科全书
- UPGMA:
-
算术平均的非加权对组法
- 舰导弹:
-
逐步突变模型;TPM:两相模型
参考文献
- 1.
Nhut DT, Hai NT, Huy NP, Chien HX, Nam NB。新成就人参vietnamensis研究。生物技术被忽视的未充分利用作物。2013:43-57。
- 2.
张光华,马春春,张建军,陈金伟,唐启勇,何明明,徐晓忠,蒋乃华,杨淑琴人参vietnamensisvarfuscidicus发现推测的ocotillol型人参皂苷生物合成基因和遗传标记。BMC基因组学。2015;16:159。
- 3.
潘迪AK,阿里MA。种内变异人参assamicus禁令。基于nrDNA内部转录间隔(ITS)序列的群体。印度生物技术杂志,2012;11:30-8。
- 4.
Momang TM, Das AP, Tag H.人参L.(五棱科)产自印度**。Pleione。2018;12(2):315 - 21所示。
- 5.
越南植物图集。普二世。分册。2002;2:640-1。
- 6.
的种类人参在越南l。中华医学杂志2005;10:71-6。
- 7.
金DH。化学的多样性人参,西洋参,田七.人参杂志2012;36(1):1 - 15。
- 8.
哈TD, Grushvitzky IV.新物种人参在越南(五茄科)。J植物学。1985;70:518-22。
- 9.
Tran LQ, Adnyana IK, Tezuka Y, Harimaya Y, Saiki I, Kurashige Y, Tran KQ, Kadota s越南人参主要皂苷马菊皂苷R2的肝保护作用(人参vietnamensis).足底医学。2002;68:402-6。
- 10.
朱德华、黎慧玲、阮维康、黎德明、杜迈、黄婷婷、杜忠忠。人参vietnamensis:越南珍贵的国药植物。越南科学技术杂志2018;1:33-5(越南文)。
- 11.
最巨大的。越南红皮书,第二部分。植物。酒吧科技,2007。(越南)。
- 12.
guro - arie R, Cohen CJ, Eitan Y, shelf L, Hallerman EM, Kashi Y.简单序列重复大肠杆菌丰度、分布、组成和多态性。基因组研究》2000;10:62 - 71。
- 13.
李永春,Korol AB, Fahima T, Nevo E.基因中的微卫星:结构、功能和进化。Mol Biol Evol 2004; 21:991-1007。
- 14.
Kalia RK, Rai MK, Kalia S, Singh R, Dhawan AK。植物微卫星标记的研究进展。Euphytica。2011;177:309-34。
- 15.
微卫星标记的开发樟树(樟科)。中华医学杂志,2012;99:e1-3。
- 16.
贾鑫,邓勇,孙鑫,梁亮,苏娟Neottopteris病灶利用Illumina对端测序和EST-SSR标记的开发。摩尔品种。2016;36:94。
- 17.
维埃拉MLC, Santini L, Diniz AL, Munhoz CF.微卫星标记:它们的意义和为什么它们如此有用。基因分子生物学杂志2016;39:312-28。
- 18.
Vu DD, Bui TTX, Nguyen MT, Vu DG, Nguyen MD, Bui VT,黄旭,张颖。越南两种濒危物种的遗传多样性:红豆杉而且水松wallichiana.中国农业科学,2017;28(2):265-72。
- 19.
考尔S, Pembleton LW, Cogan NOI, Savin KW, Leonforte T, paul J, Materne M, Forster JW。对豌豆和蚕豆的转录组测序,以发现和验证SSR遗传标记。BMC基因组学。2012;13:104。
- 20.
Kumari K, Muthamilarasan M, Misra G, Gupta S, Subramanian A, Parida SK, Chattopadhyay D, Prasad MSetaria italica在研究谷子和非谷子物种的遗传多样性、交叉转移性和比较定位等方面具有一定的应用价值。《公共科学图书馆•综合》。2013;8:e67742。
- 21.
陈丽丽,曹燕,袁宁,中村康,王根刚,邱玉玺。基于新一代测序技术的EST-SSR转录组的表征和新型制造者的开发Neolitsea sericea(樟科)东亚陆桥岛屿特有的。摩尔品种。2015;35:1-15。
- 22.
郭锐,Landis JB, Moore MJ,孟A, Jian S,姚旭,王宏。转录组衍生微卫星的研究与应用猕猴桃eriantha(猕猴桃科)。植物科学进展2017;8:1383。
- 23.
王志英,方沛波,陈建勇,张旭军,罗志新,黄磊,陈旭,李艳。甘薯根转录组的从头组装与Illumina配对测序分析及ssr标记的开发(番薯甘薯).BMC基因组学。2010;11:726。
- 24.
魏伟,齐旭,王磊,张颖,华伟,李东,吕辉,张旭。芝麻的表征(胡麻属indicumL.)使用Illumina配对末端测序和开发EST-SSR标记的全球转录组。BMC基因组学。2011;12:451。
- 25.
王松,何强,刘旭,徐伟,李亮,高洁,王峰。萝卜幼苗早期和晚期根系转录组分析及EST-SSR标记的开发。植物细胞杂志2012;31:1437-47。
- 26.
张军,吴坤,曾松,Silva JAT,赵旭,田灿,夏红,段杰兰花sinense及其在花发育相关基因鉴定中的应用。BMC基因组学。2013;14:279。
- 27.
焦宇,贾艳梅,李晓伟,柴明民,贾海军,陈铮,王光宇,柴春春,Weg EVD,高志生。杨梅基因组调查中SSR标记的开发(英文)杨梅).BMC基因组学。2012;13:201。
- 28.
Postolache D, Piotti A, Spanu I, Vendramin GG.转录组与基因组微卫星标记:基因分型的高信息复用冷杉属阿尔巴轧机。和同类的物种。Plant Mol Biol journal 2014; 32:750-60。
- 29.
何小东,郑建伟,周杰,何凯,石淑贞,王斌。EST-SSRs的表征与比较柳树,杨树,桉树.树Genet genomics 2015;11:820。
- 30.
颜旭,张旭,陆明,何莹,安宏罗莎roxburghii果实转录组揭示了推测的抗坏血酸生物合成基因和EST-SSR标记。基因。2015;561:54 - 62。
- 31.
吴德东,裴廷祥,阮顺宁,朱永华,张亮,张勇,黄晓华。多态微卫星标记的分离与表征漆树科vernicifluumf·a·巴克利。捷克植物育种杂志,2018;54(1):17-25。
- 32.
褚铮,陈娟,孙娟,董铮,杨旭,王艳,徐辉,张旭,陈峰,崔东。面包小麦胚性愈伤组织形成的De Novo组装及转录组比较分析(小麦l .)。BMC Plant Biol 2017; 17:44。
- 33.
侯松,孙铮,李艳,王艳,凌红,邢刚,韩艳,李慧。小米转录组分析、遗传SSR的建立及遗传多样性(黍miliaceum;禾本科)。应用植物学报2017;5(7):1600137。
- 34.
张磊,杨旭,祁霞,郭晨,景震。杏仁转录组和微卫星标记的研究(英文)菊花普通的L.)使用Illumina测序平台。Hereditas。2018;155:14。
- 35.
杨bw,咸依特。利用新生RNA-seq进行转录组分析,比较不同环境下栽培的人参根。植物生长学报2018;84(1):149-57。
- 36.
Nguyen VD, Ramchiary N, Choi SR, Uhm TS, Yang TJ, Ahn IO, Lim YP。新型微卫星标记的开发与表征人参(C.a. Meyer)从BAC端序列。Conserv麝猫。2010;11:1223-5。
- 37.
胡铮,张涛,高XX,王颖,张强,周海军,赵广发,王明丽,Woeste KE,赵鹏。易损树叶、芽和果实转录组的重新组装与表征胡桃效果用于利用Illumina测序开发20种新的微卫星标记。Mol Genet Genomics. 2016; 291:849-62。
- 38.
Rajesh MK, Fayas TP, Naganeeswaran S, Rachana KE, Bhavyashree U, Sajini KK, Karun A.椰子全球转录组的De novo组装与表征(椰子L.)用Illumina配对端测序法培养胚性愈伤组织。原生质。2016;253:913-28。
- 39.
闫丽萍,刘春玲,吴金东,李丽,舒娟,孙晨,夏勇,赵丽娟。的从头转录组分析Fraxinus velutina利用Illumina平台,开发EST-SSR标记。生物植物。2017;61(2):210 - 8。
- 40.
金俊,赵洪波,李建林,尹世宇,刘海华,郑克伟。中国植物中微卫星标记的新鉴定人参.摩尔细胞。2007;24(1):60-8。
- 41.
马赫,Dixit A, Kim YC, Lee DY, Kim TS, Cho EG, Park YJ。人参微卫星新标记的开发与鉴定(人参c·a·迈耶)。Conserv麝猫。2007;8:1507-9。
- 42.
赵洪波,徐道德,赵恩恩,金杰,柳海华,钟文华。栽培和野生植物微卫星多态位点的特征分析人参.麝猫染色体组。2009;2:119-27。
- 43.
赵ih, Kim YC, Kim DH, Kim KH, Hyun TL, Ryu H, Bang KH。分子标记在植物鉴别中的应用人参种及韩国人参品种(人参).人参杂志2017;41:444e449。
- 44.
吴强,宋杰,孙颖,索凡,李超,罗红,刘颖,李颖,张旭,姚辉,李霞,胡姝,孙聪人参quinquefolius从花、叶和根三个方面对人参皂苷生物合成和转录调控相关基因有了新的认识。杂志。2010;138:134-49。
- 45.
刘辉,夏涛,左玉军,陈志军,周少林。微卫星标记的研究与表征田七(五茄科)。Am J Bot. 2011: e274-6。
- 46.
reuova GD, Koren OG, Muzarok TI, Zhuravlev YN。微卫星分析人参俄罗斯的自然种群。下巴医学。2014;5:231-43。
- 47.
曹辉,Nuruzzaman M,修辉,黄洁,吴凯,陈霞,李娟,王磊,郑建华,朴世杰,杨芳,罗杰,罗卓。茉莉酸甲酯诱导的转录组分析人参不定根发现人参皂苷生物合成和转运基因。国际分子生物学杂志2015;16:3035-57。
- 48.
蒋鹏,石凤仙,李永利,刘斌,李丽芙。高度可转移微型卫星的发展人参(五加科)的全基因组数据。应用植物学报2016;4(11):1600075。
- 49.
严燕、金明良、金OT、金YC、金SC、车文伟、钟KW、金S、钟CM、李颖韩国人参的鉴定(人参)品种使用简单序列重复标记。植物育种技术,2016;4(1):71-8。
- 50.
Le NT, Nguyen TM, Tran VT, Nguyen VK, Nong VD。遗传多样性的人参stipuleanatus利用ISSR标记检测北越地区蔡氏菌。Biotec Eq. 2016;30(3): 506-11。
- 51.
庄NTP,麦n,朱拉夫列夫YN。DNA条形码技术在真品鉴定中的应用人参vietnamensis.中国工程技术科学,2017;29(1):60-7。
- 52.
Manzanilla V, Kool A, Nguyen LN, Nong VH, Le TTH, Boer HJ。系统基因组学和条形码人参:对人参品种的鉴定。BMC Evol . 2018;18:44。
- 53.
张燕,张旭,王玉华,沈sk。新创EST-SSR新标记的开发和转录组的组装杜鹃雷克斯Lévl通过Illumina测序。植物科学进展2017;8:1664。
- 54.
李伟,张晨,蒋旭,刘强,刘强,王凯。EST-SSRs的从头转录组分析与构建苏合香对虾.森林。2018;9:748。
- 55.
Park S, Son S, Shin M, Fujii N, Hoshino T, Park S剪秋罗属kiusiana(石竹科)。BMC Plant Biol 2019;19:14。
- 56.
Taheri S, Abdullah TL, Rafi MY, Harikrishna JA, werbrook SPO, Teo CH, Mahbod SM, Azizi P. De novo转录组的组装、EST-SSR新标记的挖掘和开发姜黄alismatifolia(姜科)通过Illumina测序。Sci众议员2019;9:3047。
- 57.
刘胜,王胜,刘明,杨芳,张宏,刘胜,王强,赵颖。新创的转录组测序和分析人参在叶展开期。Mol Med rep 2016; 14:1404-12。
- 58.
罗红,孙超,孙颖,吴强,李颖,宋娟,牛颖,程欣,徐辉,李超,刘杰,Steinmetz A,陈松田七根揭示了推测的三萜皂苷生物合成基因和遗传标记。BMC基因组学,2011;12(增刊5):S5。
- 59.
李永春,Korol AB, Fahima T, Beiles A, Nevo E.微卫星:基因组分布、功能和突变机制的研究进展。摩尔生态。2002;11(12):2453 - 65。
- 60.
高春,唐志林,尹建明,安志生,付德华,李坚。芸苔属植物基于基因的简单序列重复序列的表征与比较。中国生物医学工程学报,2011;28(2):381 - 381。
- 61.
黄亮,吴斌,赵军,李辉,陈伟,郑莹,任晓,陈勇,周晓,雷勇,廖斌,蒋慧。野生和栽培植物微卫星标记的表征及转化利用落花生物种。《公共科学图书馆•综合》。2016;11:e0156633。
- 62.
Kumpatla SP . Mukhopadhyay S.双子叶植物表达序列标签中简单序列重复的挖掘与研究。基因组。2005;48:985 - 98。
- 63.
黄丹,张永强,金德明,李泓,宋志平,王毅刚,陈杰。黄丹花转录组微卫星标记的特征及其跨种转移性叶兰saxicola(Asparagaceae)。中国生态学报,2014;14:569-77。
- 64.
岳晓燕,刘国强,宗燕,滕玉伟,蔡元元。梨花蕾转录组测序的基因SSR标记开发。浙江大学学报(自然科学版)2014;15(4):303-12。
- 65.
李霞,李敏,侯琳,张铮,庞旭,李艳。中国一种濒危地方病的从头转录组组装与群体遗传分析宏碁miaotaiense(槭树科)。基因。2018 b; 9:378。
- 66.
El-Domyati FM, Younis RAA, Edris S, Mansour A, Sabir J, Bahieldin A.西奈半岛部分药用植物遗传多样性的分子标记。医学植物学报,2011;5(10):1918-29。
- 67.
冯松,何锐,陆杰,蒋明,沈旭,蒋勇,王铮,王辉。药用植物SSR标记的开发与遗传多样性评价菊花品种。麝猫。2016;7:113。
- 68.
Bakatoushi R, Ahmed DGA。野生居群遗传多样性评价Peganum harmala一种药用植物。生物工程学报。2018;16:143-51。
- 69.
Vu DD, Bui TTX, Nguyen MD, Shah SNM, Vu DG,张勇,Nguyen MT,黄晓华。越南东南部两种受威胁的龙舌兰属植物的遗传多样性与保护。J为res 2019; 30:1823-31。
- 70.
Tam NM, Duy VD, Duc NM, Thanh TTV, Hien DP, Trang NTP, Hong NPL, Thanh NT.热带濒危物种的遗传变异与异交率Dipterocarpus dyeri.热带科学2019;31(2):259-67。
- 71.
王晓燕,王晓燕。气候变化对适应的约束与强化:遗传相关性状的选择。Conserv杂志。2007;140:599 - 609。
- 72.
朱拉夫列夫YN, Koren OG, reuova GD, Muzarok TI, Gorpenchenko TY, Kats IL, Khrolenko YA。人参自然种群:它们的过去、现状和前景。药理学学报。2008;29(9):1127-36。
- 73.
Artyukova EV, Kozyrenko MM, Koren OG, Kholina AB, Nakonechnaya OV, Zhuravlev YN。生存在边缘:一些远东物种在范围边缘的各种生存模式。编辑者:加里斯坎·M。植物的遗传多样性。InTech里耶卡:的哲理;2012.p . 349 - 74。
- 74.
刘辉,夏涛,左玉军,陈志军,周少林。微卫星标记的研究与表征田七(五加科)中国传统草药。机械工程学报。2011;38 (8):e218-20。
- 75.
reuova GD, Kats IL, Muzarok TI, Nguyen TTP, Dang TT, Brenner EV, Zhuravlev YN。微卫星位点的多样性人参vietnamensisHa Grushv。(五茄科)的人口。中国生物工程学报2011;441(1):408-11。
- 76.
彭丽萍,蔡春芳,钟燕,徐xx,鲜海林,程飞,毛建峰。遗传分析揭示了这种新兴油料作物的独立驯化起源芍药属ostii是一种具有长期栽培历史的牡丹。Sci众议员2017;7:5340。
- 77.
Schaal BA, Hayworth DA, Olsen KM, Rauscher JT, Smith WA。植物系统地理学研究:问题与展望。摩尔生态。1998;7:465 - 74。
- 78.
Bolger AM, Lohse M, Usadel B. Trimmomatic:一种用于Illumina序列数据的灵活微调器。生物信息学。2014;30:2114-20。
- 79.
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng O, Chen Z, Mauceli E, haacohen N, Gnirke A, Rhind N, Palma FD, Birren BW, Nusbaum C, lindbladal - toh K, Friedman N, Regev A.无参考基因组RNA Seq数据的完整转录组集合。生物科技Nat》。2011;29:644-52。
- 80.
Pertea G, Huang X, Liang F, Antonescu V, Sultana R, Karamycheva S, Lee Y, White J,张F, Parvizi B, Tsai J, Quackenbush J. TIGR基因指数聚类工具(TGICL):一种用于大型EST数据集快速聚类的软件系统。生物信息学。2003;19(5):651 - 2。
- 81.
邓媛媛,李建强,吴顺生,朱一平,陈玉伟,何方锋。蛋白质注释系统中nr数据库的集成及其定位。第一版Eng。2006;32(5):71 - 4。
- 82.
Apweiler R, Bairoch A, Wu CH, Barker WC, Boeckmann B, Ferro S, Gasteiger E, Huang H, Lopez R, Magrane M, Martin MJ, Natale DA, O'Donovan C, Redaschi N, Yeh LS。UniProt:通用的蛋白质知识库。核酸学报2004;32:D115-9。
- 83.
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Tarver IL, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G.基因本体论:统一生物学的工具。Nat麝猫。2000;得分上以25 - 9胜过25(1):。
- 84.
Tatusov RL, Galperin MY, Natale DA。COG数据库:用于蛋白质功能和进化基因组尺度分析的工具。核酸决议2000;28(1):33-6。
- 85.
Koonin EV, Fedorova ND, Jackson JD, Jacobs AR, Krylov DM, Makarova KS, Mazumder R, Mekhedov SL, Nikolskaya AN, Rao BS, Rogozin IB, Smirnov S, Sorokin AV, Sverdlov AV, Vasudevan S, Wolf YI, Yin JJ, Natale DA。编码在完整真核生物基因组中的蛋白质的全面进化分类。基因组医学杂志。2004;5 (2):R7。
- 86.
Kanehisa M, Goto S, Kawashima S, Okuno Y, Hattori M.解码基因组的KEGG资源。《核酸学报》2004;32:D277-80。
- 87.
阿尔特舒尔SF,麦登TL, Schäffer AA,张杰,张卓,米勒W,李普曼DJ。gap BLAST和PSI BLAST:新一代蛋白质数据库搜索程序。核酸学报,1997;25(17):3389-402。
- 88.
Eddy SR. Profile隐藏马尔可夫模型。生物信息学。1998;14(9):755 - 63。
- 89.
Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer EL, Tate J, Punta M. Pfam:蛋白质家族数据库。核酸学报,2014;42:D222-30。
- 90.
王晓燕,王晓燕。灵长类动物重复DNA序列的编译与分析。中华分子生物学杂志1995;40:120-6。
- 91.
Clark KR, Gorley注册护士。Primer 5.0(多元生态研究中的普利茅斯例程)。发行,普利茅斯,英国:Primer-E Ltd;2001.
- 92.
Peakall R, Smouse PE。Genalex 6.5: excel中的遗传分析。群体遗传软件的教学与研究更新。生物信息学。2012;28:2537-9。
- 93.
自然种群的基因流动。安Rev生态系统1985;16:393-430。
- 94.
伯特斯坦D,怀特RL,斯科尔尼克M,戴维斯RW。利用限制性片段长度多态性构建人类遗传连锁图谱。胡姆吉内。1980;32:314-31。
- 95.
雷蒙M,罗塞f。1.2):用于精确测试和普世主义的群体遗传学软件。J在这里。1995;86:248-9。
- 96.
皮雷S, Luikart G, Cornnet JM。瓶颈:一种计算机程序,用于检测有效种群规模频率数据最近的减少。J在这里。1999;90:502-3。
- 97.
Excoffier L, Laval G, Schneider S. Arlequin。3.0 v。群体遗传数据分析综合软件包。另一个星球Bioinforma。2005;1:47-50。
- 98.
竹崎N,内明,田村k。用windows界面从等位基因频率数据构建种群树和计算其他种群统计的软件。摩尔另一个星球。2010;27(4):747 - 52。
- 99.
王晓燕,王晓燕,王晓燕,等。基于多位点基因型数据的群体结构推断。遗传学。2000;155:945-59。
- One hundred.
Earl DA, Von-Holdt BM。结构收割机:一个用于可视化结构输出和实现Evanno方法的网站和程序。生态学报,2012;4:359-61。
- 101.
王晓燕,王晓燕,王晓燕,等。利用软件结构检测个体集群数量的模拟研究。摩尔生态。2005;14:2611-20。
确认
感谢越南-俄罗斯热带中心热带生态研究所、越南科学技术研究院生态与生物资源研究所、今都省Ngoc Linh人参股份公司;广南省南特美区Ngoc Linh人参中心,感谢他们支持我们的实地工作并颁发相关许可证。我们感谢Bui Thi Tuyet Xuan博士(越南科学技术学院生态与生物资源研究所)、Altaf Hussain Lahori教授(信德省伊斯兰大学,巴基斯坦卡拉奇74000)对本研究的支持。
资金
这项工作得到了越南科学技术学院(VAST)在生物多样性和生物活性化合物(VAST04.07/19-20)项目下的资助。资助者在研究的设计、收集、分析和数据的解释以及撰写手稿中没有任何作用。
作者信息
从属关系
贡献
DDV、TPTN:设计研究;TPTN、MTN、MPP、VTB、DDV:采集样品;MPP、VTB、DDV:进行实验;VTB、MTN、DDV、TPTN、SNMS:分析数据;DDV, TPTN, MTN, SNMS:起草和修改稿件。所有作者已阅读并认可最终稿。
相应的作者
道德声明
伦理批准和同意参与
已获越南科学技术学院生态及生物资源研究所所长批准/同意。#123/QĐ-STTNSV日期为2019年2月20日,编号为#829/QĐ-STTNSV,日期2019年5月10日)和广南省南Tra My区人民委员会Ngoc Linh人参中心主任(附同意信),然后再研究项目下的目标物种(VAST04.07/19-20).
同意出版
不适用。
相互竞争的利益
作者声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1:图S1。
K分布图。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
伍,民主党,沙阿,新党,范,国会议员et al。一种越南特有物种的从头组装和转录组特征,人参vietnamensisHa Grushv。,including the development of EST-SSR markers for population genetics.BMC植物杂志20.358(2020)。https://doi.org/10.1186/s12870-020-02571-5
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-020-02571-5
关键字
- 保护,EST-SSRs
- 转录组
- 人参vietnamensis
- 群体遗传学