摘要
背景
植物特异性的成员SPL基因家族(Squamosa启动子结合蛋白 - 样)含有SBP保守域,并参与植物生长和发育的调节,包括植物花和植物表皮发,植物应激反应和次级代谢物的合成。.这个家庭已经在各种植物中识别出来。但是,没有系统分析SPL小麦全基因组水平的基因家族。
结果
在这项研究中,有56个假定TASPL.采用比较基因组学方法进行基因鉴定;我们重命名TaSPL001-TaSPL056他们的染色体分布。根据非根邻居连接系统发生树、基因结构和基序分析,对56TASPL.基因分为8个亚组。共81TASPL.基因对被指定为从复制事件中产生的,并且鉴定了64个相互作用的蛋白质分支,如蛋白质相互作用网络涉及。21的表达式模式随机选择TASPL.通过定量的实时聚合酶链反应(QRT-PCR)检测不同组织(根,茎,叶和花序)和4个处理(脱离酸,赤霉素,干旱和盐)的基因。
结论
小麦基因组含有56个TASPL.基因和同一亚家族的基因具有相似的基因结构和基序。TASPL.基因扩展通过节段性重复事件发生。结合转录和QRT-PCR分析的结果,大部分TASPL.发现基因调节花序和尖峰发育。此外,我们发现13TaSPLs通过脱钙酸上调,表明这一点TASPL.基因在幼苗阶段的脱落酸介导的途径中发挥着积极作用。本研究提供了关于的全面信息SPL为阐明小麦的生物学功能奠定了坚实的基础TaSPLs并提高小麦产量。
背景
基因家族,由股票和函数的多种基因组成,对给定生物体的重要作用。现在,在真核生物中发现了各种基因系列,例如bHLH[1],TCP[2),而PRX.[3.].在所有基因家族中,SPL(Squamosa启动子结合蛋白,SBP)是一种植物特异性家族,广泛分布在绿色植物中。植物SPL蛋白与MADS箱基因的Squamosa启动子结合,首先在CDNA文库中鉴定金鱼草majus花序(4].这些蛋白质包含一个特定的SBP结构域,包含大约70个氨基酸,并具有两个锌指位点(Cys-Cys-His-Cys和Cys-Cys-Cys-His) [5,6].在这个域内,4个氨基酸残基配位一个锌离子,对维持蛋白质构型的稳定性起着重要的作用。此外,一个位于SBP结构域c端的保守核定位信号与第二锌指结构重叠[7并将蛋白质引导到细胞核,以调节下游基因的转录。
近年来,大量的SPL基因已经成功克隆到各种植物中,这些SPL基因分布从c . reinhardtii来p .金属盘高等植物[8].目前,鉴定和演变SPL基因家族已经研究过答:芥[9,10)、大米(5,大豆11],杨树[12],佩妮[13],Gossypium[14,以及烟草[15].基于序列同源性和系统发育的分析,这个家庭通常被纳入6-9个亚组。郭[8]报道120SPL9个物种的基因全部分为3组SPL陆地植物的基因被分为两个不同的组和7个亚组。杨(5)分35SPLS.来自米饭和答:芥分为三类:A、B、CSPL基因分为9个亚属。在杨树[12), 28ptspls.分为8个亚组。在丹参,15SPL基因被分为6个亚组[16].不幸的是,已经进行了很少的研究以展示了分类SPL小麦的基因家庭。
的表达水平SPL基因可受miR156/157控制。罗迪斯[17和嘉信理财[18]发现11个中的17个SPLS.在答:芥包含miR156/157识别位点。施瓦布(18报道,过表达MIR156答:芥下调多重表达SPLS.然而,SPLS.没有这个识别网站就不受影响了。突变miR156/157的识别位点后atspl3.的转录水平atspl3.显著增加[19].Salinas观察到雄蕊和尖部的互补表达方式SlySPL基因与miR156/157,揭示SlySPL基因受MiR156 / 157调节[20.].
的SPL这些基因的表达水平受miR156/157调控,在植物胚胎、组织和营养相变化、赤霉素和光信号转导、干旱和盐胁迫响应等植物生长和生理的各个方面发挥着重要作用。例如,抑制AtSPL3蛋白的翻译后修饰可导致花期提前拟南芥[19].atspl3.,−4,−5调节关键下游基因的表达atap1.,areful.,atlfy.参与花分生组织的发育[10].张证明了SPL8.该基因在花中对GA信号通路起正向调控作用,而在幼苗中起负向调控作用atspl8.在植物发育中调节GA信号[21].王传说,OsSPL16通过增加细胞分裂和谷物填充,可以有效提高晶粒质量和产量,这意味着该基因对大米籽粒发育具有阳性后果[22].OsSPL14抑制水稻中的分蘖数,但促进穗分枝以增加粒度,较强的茎[23].在低温(5°C)时,两者VvSBP3和VvSBP5调节,但VvSBP4和VvSBP7被下调了,这说明了什么VvSBP3和VvSBP5与低温反应有关吗vitis Vinifera L.[24].的BpSPL9对盐和干旱胁迫的反应,过度表达BdSPL9通过清除活性氧来提高对盐和干旱胁迫的耐受性[25].关于……的功能我们知道得很多SPL基因在答:芥然而,小麦的这种信息是有限的。
的植物SPL家庭在成长和发展中起着重要作用。随着基因组测序技术的进展,已经确定了植物中的各种转录因子或基因家族。虽然很多植物SPL基因家庭成员已被确定,这不是小麦的情况,以及知识SPL这个物种的基因功能是有限的。在本研究中,我们使用生物信息学方法来鉴定SPL我们分析了小麦的基因TASPL.基于基因结构,基序,顺式元素,系统发育关系,基因重复,GO注释,蛋白质 - 蛋白质相互作用和基因表达模式的特征,并预测其功能。我们的结果提供了有关功能阐释和演变的信息SPL小麦的基因。
结果
全基因组鉴定和染色体定位TASPL.基因
获得TASPL.采用HMM和BLASTP两种方法对小麦全基因组基因进行鉴定,采用pam和CDD两种网站进行鉴定。最终,56个基因被指定TASPL.基因;一切都不均匀地分布在小麦染色体上。56推出的推定TASPL.基因重命名TaSPL001来TaSPL056基于染色体位置(附加文件1:表S1)。其中,34%(19个)分别定位于A和D基因组;32%(18)位于B基因组。TaChr7A和TaChr7D中含有大量的SPL基因,但没有TASPL.基因位于染色体4B和4D上。
将56个SPL蛋白提交至大提琴网站,预测其亚细胞定位;如表S1,所有SPL蛋白均定位于细胞核,3个SPL (TaSPL034、TaSPL049、TaSPL052)也分布于质膜,说明56个TASPL.蛋白质在细胞核中进行它们的功能。该56预测的促使蛋白质在氨基酸长度和蛋白质分子量(MWS)中表现出多样性。TASPL蛋白的MWS范围为20.11至123.69kDa。TASPL蛋白的长度在192至1129AA之间变化,最长的蛋白质是TASPL043,最短的是TASPL004,TASPL007和TASPL010。
基因结构和主题组合物SPL小麦基因家族
基因结构和motif多样性是促进基因家族进化的一种机制。我们构建了一个系统发育树TASPL.并分析了其外显子、内含子和保守基序(图1)。1).结果表明,尽管在外显子位置有所不同,TASPL.来自同一亚家族的基因分享类似的遗传结构,以及大多数外显子的数量TASPL.基因是守恒的。约59%(33)的基因包含3个外显子,但在VIII亚群和X亚群中有12个基因拥有最多的外显子,在8 ~ 11个。例如,TaSPL001在X亚家族中包含10个外显子TaSPL042在亚家族中,VIII含有11个外显子。
我们将TaSPL的全部蛋白序列提交给MEME软件,检测motif组成,探索motif多样性。20个特定的母题被定义并命名为母题1到20。其中,除TaSPL036外,其余的TaSPL蛋白均含有motif 1和motif 2,表明这两个motif是TaSPL蛋白序列的重要组成部分。所有SPL蛋白中不仅有共同的基序,而且在不同的子群中也有各自特定的基序。例如,母序18存在于所有SPL亚家族VII和亚家族IV成员中;motif 20在亚科VII中。总之,同一亚家族的成员具有相似的基因结构和基序组成,而不同亚家族的成员具有特定的结构,这意味着不同亚家族的成员具有相同的基因结构和基序组成TASPL.基因家族在进化过程中表现出功能保护和多样性。
多序列比对,系统发育分析TaSPLs
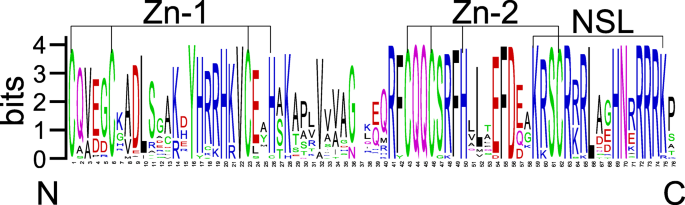
进行全长TASPL蛋白的多序列对准。结果表明,特定的SBP结构域在56个促型蛋白之间被保守(图。2).大约76个aa包含一个SBP结构域,包含两个锌指基序,结构为Cys-Cys-Cys-His和Cys-Cys-His-Cys,在c端有一个核定位信号(NSL)。TaSBP域中有三个保守序列CQQC、SCR和RRR,但TaSPL036不具有CQQC序列。
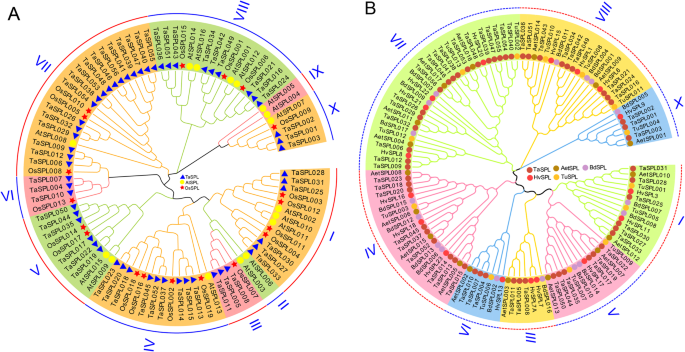
评估进化关系SPL小麦,米饭和答:芥,利用全长SPL蛋白构建邻居连接系统发育树(图1)。3).九十一年SPL以上3个物种的基因被分为10个亚科(I ~ X)SPL小麦家族更接近SPL米饭中的基因答:芥.除II、III、VI、IX亚科外,其余亚科共3种SPL基因。例如,子家族我包含3atspls.,4OsSPLs、6TaSPLs.2atspls.,2OsSPLs、6TaSPLs包括子家族V.这表明了TASPL.基因没有随着单圈和双旋代岩的特征而发展,并且是SPL基因家族在这两类植物分化之前就已经形成。在nj树中发现22对同源基因对,其中4对来自nj树答:芥来自小麦的18个基因,表明了TASPL.基因以特异性的方式扩张。
用同样的方法鉴别了17、13和17SPL基因b . distachyon,T.urartu,答:tauschii基因组,下载了17个SPL大麦基因序列[26].然后构建系统发育树(图1)。3 b).120年SPL5个物种的基因仍分为8个亚群。我们检测到13个同源的SPL来自小麦和答:tauschii, 3来自小麦和T.urartu和1个来自小麦和大麦。相反,在小麦和小麦中没有发现完全基因对b . distachyon.这意味着TASPL.基因在进化上与AetSPLs和TuSPLs.
基因复制和同步分析TASPL.基因
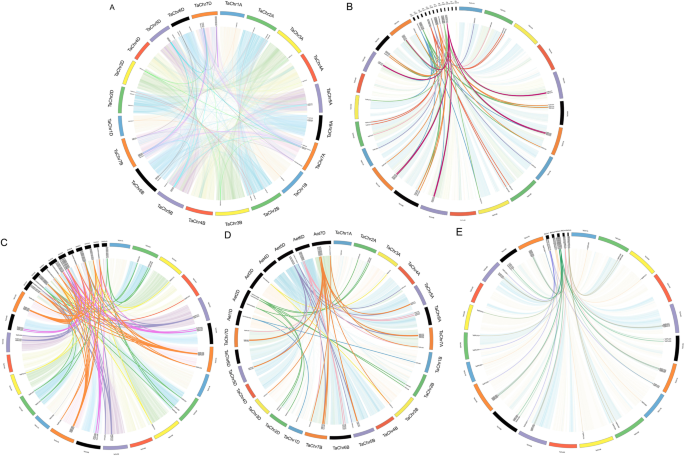
基因复制,一种不可或缺的机制,可以扩展享有类似或不同功能的新基因;因此,我们分析了发生的重复事件TASPL.基因家族。八十一SPL检测到小麦的基因对为重复基因对(图1)。4,附加文件1:表S3),其中8个是串联重复,73个与节段重复事件相关。中的分段复制事件的数量TASPL.基因家族被发现高于串联重复,这表明前者是扩展的主要途径SPL小麦的基因和不同小麦染色体上的许多同源基因支持对家庭的高度保守。
非同义替代比Ka/Ks决定了重复基因的选择压力。对每个重复基因对的蛋白质序列和CDS序列进行比较,计算Ka/Ks比和发散次数。根据结果(附加文件1:表S3),所有的KA / KS比TASPL.基因对< 1,表示进化TASPL.基因伴随着强烈的净化选择。并对散度时间进行了预测TASPL.基因对大约是27米亚。
我们在基因组水平下用其他物种检测到小麦的离岸性,以更好地理解TASPL.基因家族。大多数TASPL.基因在水稻,玉米,答:tauschii,b . distachyon(无花果。4 b-4 e,附加文件1:表S4-S7)。共检测到67对、110对、52对和56对基因。小麦与水稻、玉米、答:tauschii,b . distachyon分别为0.38、0.39、0.29、0.31。所有共线基因对的值均小于1,证实了该基因的进化SPL小麦基因家族经历了强烈的纯化选择.水稻共线基因对的分化时间与玉米相同,约为52米亚,且早于玉米答:tauschii(39米娅)b . distachyon(41Mya),表明这一点TASPL.基因家族与水稻、玉米、答:tauschii,b . distachyon.
的GO注释分析和蛋白-蛋白相互作用网络TaSPLs
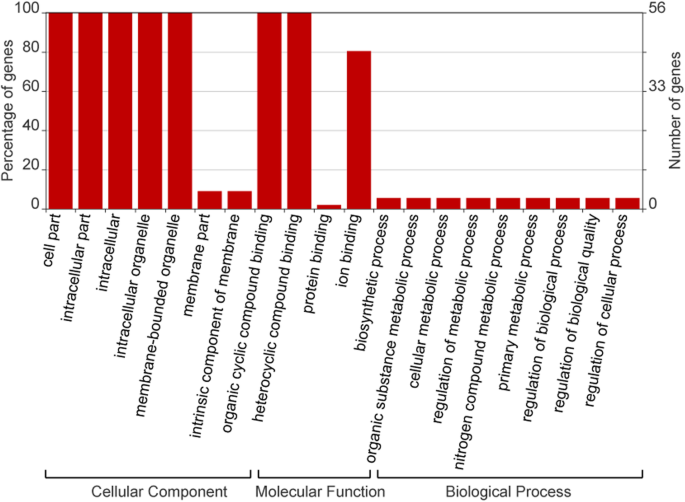
我们对56个蛋白进行了GO注释分析,揭示了它们可以参与一系列细胞组分,分子函数和生物过程(图。5,附加文件1:表S8)。对56个TaSPL蛋白共分配20个GO项;虽然只有少数的氧化石墨烯术语属于细胞成分,但许多蛋白质富集在这一范畴。在细胞成分类别下,富集程度最高的类别是细胞部分、细胞内部分和细胞器。所有的TaSPLs都可以参与这三个过程,而只有不到30%的TaSPLs参与膜的形成。在分子功能下,所有蛋白质都具有与其他分子结合的能力,如有机环化合物结合和离子结合。其中,100%的TaSPLs能与杂环化合物结合,80%能与离子结合。在生物过程方面,只有不到20%的TaSPLs参与氮代谢、细胞过程调控等生物过程。
为了了解小麦中TaSPLs与其他蛋白质之间的蛋白-蛋白相互作用,我们构建了一个蛋白-蛋白相互作用网络(图1)。6,附加文件1:表S9)。检测到三十两种促吐仑蛋白和总共64个相互作用的蛋白质分支。其中,10%的促使蛋白可以与至少6个蛋白质相互作用,例如TASPL001,TASPL002和TASPR003,表明这3个TASPL蛋白在蛋白质网络的调节中发挥着重要作用。将与TaspL蛋白相互作用的额外小麦蛋白的序列被提交至CDD数据库以预测其保守的结构域,我们揭示了各种转录因子家族,例如AP2,Cu-Zn_superoxide_disticutase和Mads_mef2_like。其中,将32个相互作用分支鉴定为TASPLS与AP2家族的相互作用。因此,我们表明,TASPL蛋白可能与AP2家族的蛋白质相互作用以调节小麦发育。
启动子的分析TASPL.基因
启动子区包含的特异顺式作用元件可调控基因的表达。我们分析了56株的上游1.5 kb DNA序列TASPL.基因和识别潜在的顺式作用元件(图S1,附加文件1:表S10)。的启动子区中多种顺式元件TASPL.聚集成三个细分的基因:激素和应力响应元素,轻响应元件和植物生长和发育元素。三种类型中,大多数TASPL.基因中含有光响应元件,如ACE、Sp1和G_box。特别是,在60%(34)的肿瘤中发现了G_boxTASPL.基因,这表明TASPL.表达可能受光调控,G_box在小麦发育中起着重要作用。此外,与脱落酸响应性相关的ABRE元件广泛分布在80%的样品中TASPL.基因,这表明TaSPLs与ABA代谢途径的调节有关。同时,有一些与植物生长和开发有关的元素,如CAT_BOX,O2_SITE和HD_ZIP 1,TASPL.基因,表明TASPL.基因家族在调节生理过程中起作用。
表达式模式TASPL.不同小麦组织中的基因
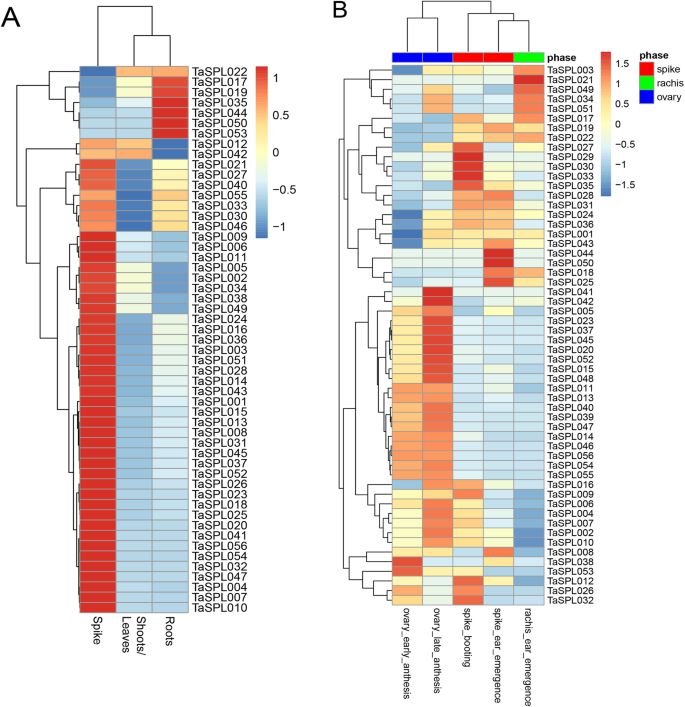
转录数据从小麦表达浏览器网站下载,以确定组织特异性的表达模式TASPL.基因(图。7一个).结果表明,53TaSPLs在芽/叶子,根和小麦尖峰中表现出不同的表达水平。例如,46TaSPLs结果表明,7个基因在根中均有高表达TaSPLs主要表现在尖峰中,参与尖峰的发展。此外,同一亚组的基因具有相似的表达模式。所有TASPL.V亚家族的基因在根中表达量高,VIII亚家族成员在芽/叶中表达量低,而根中表达量低TASPL.在同一族共享守恒函数。
表达式TASPL.还分析了五种器官中的基因(Ovary_early_anthesis,Ovary_late_anthesis,Spike_booting,Spike_ear_emergence和Rachis_emercence)(图。7 b).56个基因的表达情况如下:75%(42)在子房花中高表达,55%(31)在穗中高表达,14%(8)在穗轴穗突中高表达。这些发现表明TASPL.基因主要在与花发育相关的器官中表达。其中,55%(32)主要发生在子房早花期。另外,68%(38)在子房后期表达量高,特别是12TASPL.基因,如TaSPL005,−038,−052,在卵巢_anthesis的较高水平表达,表达了这一点TASPL.基因参与小麦花的发展和12TASPL.基因对花的发育是必不可少的。此外,46%(26)的TaSPLs在Spike_booting中显示出高的表达,29%(16)在尖峰_ear_emercence中表达了更高的水平。因此,我们推断出来TASPL.基因与花和穗发育相关,通过参与花药和穗的发育来影响花和穗的形成。
21TASPL.基因
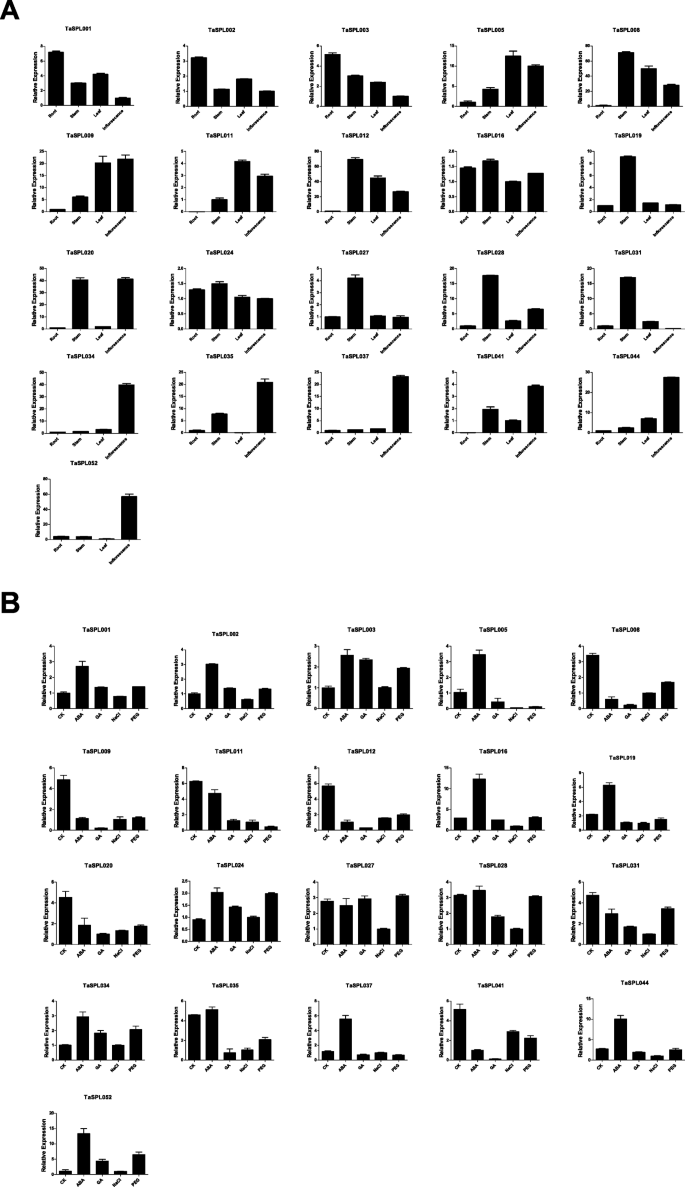
进一步调查可能的功能TaSPLs,采用qRT-PCR方法检测21个基因的表达模式TASPL.四种组织(根、叶、茎和花序)中的基因(图。8).21TaSPLs在四种组织中均有表达,具有较强的组织特异性表达模式。的数量TASPL.在花序和茎中表达量较高的基因在根和叶中表达量较高。例如,8个基因(TaSPL009,−020,−034,−035,−037,−041,−044−和−052)在花序中表达量高,8个基因(TaSPL008,−012,−016,−019,−024,−027,−028,−031)用茎表达;但是,只有3(TaSPL001,−002,−003)主要分布于根中,2 (TaSPL005,−011)在叶子中显示出高表达。这表明了TASPL.基因主要在花序和茎中表达,并在小麦花和茎的发展中起着关键作用。
作为SPL基因可能与植物应激,激素响应和其他信号过程相关联,表达水平为21TaSPLs分析4以下处理(图。8 b).与ck,13个基因相比,如TaSPL001,−002和 - 019.通过ABA治疗上调,建议TASPL.基因参与了ABA途径。超过一半基因的表达水平在其他3个处理下逐渐降低。例如,14个基因,例如TaSPL009,−005,−008GA处理下调;19个基因,比如TaSPL011,−012,−016,−019被盐处理下调;通过PEG处理下调14个基因。这些结果表明TASPL.基因在盐,GA和PEG处理下发挥负面角色。
讨论
特点SPL小麦的家庭基因
的SPL编码植物特异锌指蛋白的基因在调节植物生长发育中起着重要作用。这些基因已经在许多绿色植物中被发现,例如水稻[27],答:芥[10],玉米[28]和荞麦[29].小麦是一种主要作物,中国春季完全基因组测序将提供学习的详细信息SPL基因家庭。在这项研究中,我们使用比较基因组学方法来检查56TaSPLs, 13TuSPLs,17AetSPLs, 17BdSPLs.的数量SPL基因在T.urartu,答:tauschii和b . distachyon和那个相似吗答:芥(16)10,大米(19)[27但低于玉米(31)[28荞麦(24)[29].但是,数量TaSPLs高于上述物种;特别是,有三倍多TaSPLs比TuSPLs和AetSPLs.这种关系的TASPL.和TuSPL和AetSPL与以前的研究一致PRX.和PK小麦的基因家庭,T.urartu,答:tauschii[30.].这是因为小麦是异源六倍体,其起源涉及两个多倍化事件,而且似乎在小麦基因组中发生了许多基因复制事件,允许数量TaSPLs与其他物种相比增加。所有TaSPL蛋白均定位于细胞核;然而,在演变过程中TASPL.基因家族、外显子的插入或丢失以及不同亚家族中SPL蛋白的功能多样性和亚功能化导致了氨基酸长度和类型的巨大差异。因此,TaSPL蛋白的等电点和mw存在显著差异。这一结果与其他禾草类作物的结果一致SPL基因在物种之间被保守。
的进化SPL小麦的家庭基因
有人建议SPL基因仅存在于绿色植物中并达到绿藻的分歧[8].祖先SPL最初形成了两种不同的血统,名为Clade I和Clade II。CLADE I具有保守的结构特征,具有更多的外显子和更长的蛋白质序列[31].我们的结果显示SPL来自小麦的基因包含1-11个外显子。大多数外显子的数量SPL只有两个亚群(VIII和X)具有超过10个外显子。此外,第VIII亚组和第X亚组的TaSPL蛋白序列比其余亚组的TaSPL蛋白序列大。我们推断,TASPL.X亚群和VIII亚群的基因属于第I支,其余基因属于第I支TASPL.基因属于支支II。根据张的[32研究,我们发现,亚组X和VIII的Ka和Ks的手段低于其他亚组的手段。进一步的分析显示,与其他重复基因相比,亚组VIII和X基因具有较低的Ka / Ks比(0.25)。我们认为这是一种进化机制TASPL.基因家族类似于其他陆地植物可以聚集成两种片状,亚组VIII和X基因已经发展慢于其他基因。
探索进化和功能分歧SPL对小麦基因家族的系统发育关系进行了鉴定。17对同源基因TASPL.在系统发育树中检测到13对来自小麦答:tauschii, 3对同源基因T.urartu, 1对与大麦同源。没有与水稻同源的基因对,答:芥或b . distachyon,建议TaSPLs有密切的关系SPL基因在答:tauschii和T.urartu.来自基因重复结果,81TASPL.基因对是重复的基因,并且在米,玉米中观察到高水平的共线基因对,答:tauschii,b . distachyon.节段重复事件的数量大于串联重复事件的数量,说明节段重复是导致重复发生的原因之一TASPL.基因的扩张。基因复制和共系分析证实,在基因中未发生阳性选择TaSPLs,此外,TASPL.基因经历了强烈的净化选择。我们的研究结果与SPL荞麦基因[29[米[31],表明进化TASPL.基因与其他植物的相似。
简而言之,我们推断出进化的SPL小麦的基因家族与其他陆生植物相似。相比之下,答:芥和b . distachyon,TASPL.基因与之共享与之相关的关系AetSPLs和TuSPLs.
的表达模式和潜在功能SPL小麦基因家族
通常,基因基于它们在生物体中的表达进行功能,并且基因的表达模式反映了基因功能[13].在本研究中,我们检测了21TASPL.基因,38%显示茎和花序的高表达,意味着TASPL.基因主要在小麦的花序和茎中发挥作用。在答:芥,曹国伟(33报道了ATSPL1和ATSPL012这两个基因在花序中表达量较高,过表达增强了花序的耐热性。徐(10)发现,atspl2.,−9,- 10.,- 11.,- 13.,- 15.在植物从营养阶段到生殖阶段的转化中起主导作用。除了atspl10,其余5个基因既促进花分生组织的特性,又促进花的诱导。同源基因可能具有相似的功能。进化分析结果表明ATSPL1和atspl12是高度同源的TaSPLs在VIII子组,包括TaSPL034;atspl9.,- 15.与...分享密切关系TaSPL044和−035.ATSPL13是同源TASPL.属于亚家族IV的基因,如TaSPL020,−037和TaSPL052.这6TASPL.基因在花序中表达量较高。我们推断这些基因参与了花序的发育。三个基因(TaSPL001,−002,−003)中10个外显子在小麦根中的表达量高于其他3个组织。然而,其余的基因,包含较少或多于10个外显子,在根中表达较低。因此,我们推断这3TASPL.基因对根的发育是必不可少的,外显子的插入也会产生影响SPL小麦组织中的基因表达。
功能分析发现SPL基因家庭治理植物生长和环境压力发展的许多方面[32].在这项研究中,我们发现SPL在4个处理下,幼苗阶段的基因低。吴[34,35王[36据报道,MIR156呈现出苗期的最高表达水平。随着植物从幼苗进展到更成熟的阶段,其表达逐渐减少。相反,靶基因的表达水平atspl3.和atspl9.在苗期较低,在营养生长期逐渐升高。因此,我们推断miR156抑制了TASPL.苗期基因。在我们的研究中,我们发现了21个TASPL.4种不同处理使基因表达上调或下调,其中13种TASPL.由ABA上调的基因,其中大多数都含有其启动子的ABRE元素,表明在ABA介导的植物生长中作用。十四TASPL.这14个基因在GA介导的植物生长中起负调控作用。崔(37)报道,atspl9.被盐和干旱治疗下调,调节答:芥开花,这意味着atspl9.响应这两个应力处理。我们发现了TaSPL035和−044,由NaCl和PEG治疗下调,分享高同源性atspl9.,表明这两个基因可以对盐和干旱胁迫做出反应。简而言之,我们推断TASPL.基因家族在小麦发育过程中起着至关重要的作用,该家族成员参与了小麦茎秆和花的发育,并参与了非生物胁迫。
结论
系统发育和多样化SPL从基因结构、进化关系、共时分析、GO注释、蛋白-蛋白相互作用和表达模式等不同层面对小麦基因进行研究。的SPL在进化期间,小麦中的基因家族膨胀并纯化选择。所有56TASPL.基因被分成8个亚组,同一亚组的基因份额相似的进化特征和表达模式,意味着可能的相似函数TASPL.基因。检测到21个基因表达TASPL.基因在不同组织中具有组织特异性的表达模式,这些基因在茎和花药、花序等与穗发育相关的组织中表达量较高TASPL.基因调节茎的发育并影响花和小穗的形成。在这里,我们首先报道了鉴定,系统发育和表达分析TASPL.基因家族,这将为进一步阐明其潜在的生物学功能奠定基础SPLS.在植物。
方法
鉴定SPL基因在小麦
小麦全基因组数据来源于植物数据库(http://plants.ensembl.org/info/website/ftp/index.html),并从PFAM数据库下载SBP域(PF03110)(https://pfam.xfam.org/).然后,以SBP结构域作为查询序列,使用HMMER 3.0程序寻找含有SBP结构域的小麦蛋白,阈值e < 1e−5[38].水稻SBP蛋白序列[27] 和答:芥[10]作为查询序列,通过BLASTP程序对小麦蛋白数据集进行搜索,阈值设置为e < 1e−5和50%的身份。假定的小麦SPL通过分析HMM和BLASTP的结果预先鉴定基因。然后,ncbi-cdd web服务器(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi),并利用PFAM数据库进一步确定候选SPL小麦的基因。蛋白质序列TaSPLs在ExPASy服务器(https://web.expasy.org/compute_pi/)[39],得到理论等电点(PI)和分子量(MW),以及大提琴web服务器(http://cello.life.nctu.edu.tw/)[40]来预测这些蛋白的亚细胞定位。
染色体位置,基因结构和保守的基序分析TASPL.基因
物理位置的TASPL.基因是从小麦的基因组注释信息(GFF3)获得的基因,TASPL.基因映射到小麦染色体上。基因和编码序列(CDS)的结构TaSPLs分析外显子-内含子的组织,结果显示在基因结构显示服务(http://gsds.cbi.pku.edu.cn/)[41].保守的主题TASPL.使用MEME V4.9.0鉴定蛋白质(http://meme-suite.org/tools/meme),最佳基序集为≥10和≤200个氨基酸,最大基序集数为20 [42].
系统发育分析
小麦的SPL蛋白序列,b . distachyon,答:tauschii,T.urartu、大米和答:芥进行系统发育分析。ClustalX 2.0软件[43使用默认参数用于进行多个序列对齐。使用Mega 6.0构建未生根邻居加入(NJ)树[44具有1000个引导复制的软件。
小麦的基因复制与稻米,玉米,答:tauschii,b . distachyon
McScanx [45]软件用于检测之间的共线区域TASPL.基因,以及共线块TASPL.4种其他物种的基因(b . distachyon,大米,答:tauschii和玉米)。基因复制事件TaSPLs使用电池0.67工具可视化上述物种之间的同时关系[46].比较CRInear基因对的CDS和蛋白质序列,使用KAKS_CALCULATOR软件计算KAKS比率[47].最后,利用Wang [T = Ks/(2λ × 10)]方法计算了共线基因对的发散时间−6λ =6.5× 10−9)] [3.].
TaSPLs的GO注释及蛋白-蛋白相互作用网络分析
小麦SPL蛋白的GO注释可从PLAZE数据库(https://bioinformatics.psb.ugent.be/plaza/versions/plaza_v4_monocots/)[48]及植物转录调控图谱数据库(http://plantregmap.gao-lab.org/).GO注释结果使用Wego可视化(http://wego.genomics.org.cn/)在线工具[49].
基于小麦和小麦之间的正交基因答:芥ArenaNet V2工具[50]和字符串数据库(https://string-db.org/cgi/input.pl)[1]构建了TaSPL蛋白与其他小麦蛋白之间的相互作用网络。使用String数据库中的可信值> = 0.8来确定交互网络,并由Cytoscape软件图形化显示[51].
TaSPLs的启动子分析
上游1.5 kb的DNA序列TASPL.从Ensembl植物数据库中下载基因,然后提交到PLACE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测启动子区顺式调控元件[40].
基因表达,材料和QRT-PCR分析TaSPLs
的转录数据TASPL.小麦3个组织(根、芽/叶、穗)和5个器官(子房早花、子房晚花、穗孕穗期、穗穗出穗和穗穗出穗)的基因来自小麦表达浏览网站(http://www.wheat-expression.com/download)检测表达式剖面[52].R用于在热图中显示表达式模式。
对于qRT-PCR,中国春小麦品种于2019年在温室中种植。抽穗期采集根、叶、茎和花序。将3周龄幼苗置于200 mM NaCl、100 μM赤霉素酸(GA)、100 μM脱落酸(ABA)或20% PEG-600处理2 h后采集样品。所有样本保存在−80℃,使用RNAiso试剂(TaKaRa,北京,中国)进行RNA提取,有3个生物重复。使用RT Master Mix Perfect Real-Time试剂盒(TaKaRa,中国北京)合成cDNA。采用QuantStudio™Real-Time PCR软件进行qRT-PCR, 2(-ΔΔct)采用分析方法,随机选择21株,测定相对表达量TaSPLs.我们选择TraesCS6B02G243700作为参考基因,以规范化表达水平TASPL.基因。使用的引物列于其他文件中1S2:表。
数据和材料的可用性
本试验所用中国春季品种由西北农林大学马令健教授提供。支持本文结果的数据集包含在文章和附加文件中。
缩写
- TaSPL:
-
小麦SPL
- 阿坝:
-
脱盐酸
- GA:
-
赤霉酸
- cd:
-
编码序列
- 嗯:
-
隐马尔可夫模型
- 存在:
-
定量实时聚合酶链反应
参考
- 1.
关键词:小麦,bHLH,转录因子,全局鉴定,结构分析,表达中国生物医学工程学报。2017;17(1):90。
- 2.
关键词:柳枝稷(Panicum virgatum L.), TCP基因家族,全基因组鉴定,表达分析基因。2019;702:89 - 98。
- 3.
王Y,王Q,赵y,汉族,朱S.玉米III级过氧化物酶基因家族的系统分析揭示了一种保守的亚家族涉及非生物应激反应。基因。2015; 566(1):95-108。
- 4.
Huijser P,Klein J,Lonnig We,Meijer H,Saedler H,Sommer H.Bractebomia,花序异常,是由疯狂箱基因Squamosa在Antirrhinum Majus的功能丧失引起的。Embo J. 1992; 11(4):1239-49。
- 5.
关键词:拟南芥,SBP-box,基因家族,水稻基因。2008;407(1 - 2):1 - 11。
- 6.
Yamasaki K,Kigawa T,Inoue M,Tateno M,Yamasaki T,Yabuki T,Aoki M,Seki E,Matsuda T,Nunokawa E等。通过拟南芥SBP系列转录因子的DNA结合结构域溶液结构揭示了一种新的锌结合基序。J Mol Biol。2004; 337(1):49-63。
- 7.
植物sbp结构域的功能解剖:dna结合和核定位结构域的重叠。中国生物医学工程学报。2005;352(3):585-96。
- 8.
关键词:SBP-box,转录因子家族,全基因组鉴定,进化分析基因。2008;418(1 - 2):1 - 8。
- 9.
Cardon G,Hohmann S,Klein J,Nettesheim K,Saedler H,Huijser P.拟南芥SBP-Box基因的分子表征。基因。1999; 237(1):91-104。
- 10.
关键词:拟南芥,mir156,启动子结合蛋白样(SPL),发育功能公共科学图书馆麝猫。2016;12 (8):e1006263。
- 11.
关键词:大豆,SPL,家族基因,系统发育,表达谱基因进化研究。2017;227(2):101-19
- 12.
[15]李超,陆胜。毛叶杨SPL基因家族的分子特征分析。中国生物医学工程学报,2014;
- 13.
周Q,张某,陈F,刘B,吴L,李F,张继,宝M,刘G.蚕豆SBP-Box基因家族的基因组综合征与表征。BMC基因组学。2018; 19(1):193。
- 14.
张X,窦,庞C,宋m,魏H,范思,王C,yu S.基因组组织,差异表达及SPL型Hirsutum中SPL基因家族的功能分析。Mol Genet基因组。2015; 290(1):115-26。
- 15.
韩云云,马永强,李德志,姚建文,徐志强。烟草15个NtabSPL基因的特征及系统发育分析。Qinyan95。基因进化研究。2016;226(1):1 - 14。
- 16.
关键词:丹参,SPL,基因家族,全基因组分析,分子解剖中国生物医学工程学报(英文版),2014;
- 17.
Rhoades MW,Reinhart Bj,Lim LP,Burge CB,Bartel B,Bartel DP。植物微稻草靶的预测。细胞。2002; 110(4):513-20。
- 18.
Schwab R,Palatnik JF,Riester M,Schommer C,Schmid M,Weigel D. MicroRNA对植物转录组的特异性效果。DEV细胞。2005; 8(4):517-27。
- 19.
拟南芥SBP box基因SPL3的3 ' UTR中miRNA156/157识别元件通过转录抑制抑制幼苗的早期开花。植物j . 2007; 49(4): 683 - 93。
- 20.
关键词:番茄,SBP-box,转录因子,系统发育,序列分析足底。2012;235(6):1171 - 84。
- 21.
张Y,Schwarz S,Saedler H,Huijser P.Spl8,拟拟拟拟拟拟拟征白的发育过程中的局部调节剂。植物mol biol。2007; 63(3):429-39。
- 22.
王松,吴凯,袁强,刘鑫,刘志林,曾锐,朱华,董刚,钱谦,等。OsSPL16调控水稻籽粒大小、形状和品质。Nat麝猫。2012;44(8):950 - 4。
- 23.
王L,张Q.通过微调SPL基因表达提高水稻产量。趋势植物SCI。2017; 22(8):643-6。
- 24。
关键词:SBP-box,葡萄,基因组组织,系统发育,差异表达《公共科学图书馆•综合》。2013;8 (3):e59358。
- 25。
关键词:SPL, BpSPL9,基因家族,非生物胁迫,分子生物学桦木属platyphyllaSuk。植物细胞组织器官培养物(PCTOC)。2017; 130(3):469-81。
- 26。
Tripathi RK,Bregitzer P,Singh J. Genome-Wide A分析SPL / MIR156模块及其与大麦AP2 / MIR172单元的交互。SCI批准。2018; 8(1):7085。
- 27。
谢克,吴C,熊L.基因组组织,差异表达和水稻毒素促进剂结合等转录因子和微稻草中的相互作用。植物理性。2006; 142(1):280-93。
- 28。
关键词:玉米,SPL,基因家族,比较基因组分析,进化特征中国生物医学工程学报。2019;42(2):380-94。
- 29。
刘M,Sun W,MA Z,Huang L,Wu Q,唐Z,Bu T,Li C,陈H.S SPL基因家族在水果发育阶段的表达分析和表达分析中的SPL基因家族的基因组覆盖.BMC植物BIOL。2019; 19(1):299。
- 30.
关键词:小麦,过氧化物酶,过氧化物酶基因,过氧化物酶基因,过氧化物酶基因BMC基因组学。2019;20(1):666。
- 31.
关键词:SQUAMOSA,启动子结合蛋白样(SPL),基因家族,进化分析中国植物科学,2019;
- 32.
陆生植物SBP-box基因的进化与分化BMC基因组学。2015;16:787。
- 33.
超利民,刘永强,陈东元,薛晓燕,毛义波,陈晓燕。拟南芥转录因子SPL1和SPL12赋予植物生殖阶段的耐热性。摩尔。2017;10(5):735 - 48。
- 34.
吴克,公园我,康威,王家,伟莱D,Poithig卢比。miR156和miR172的顺序作用调节拟南芥的发育时间。细胞。2009; 138(4):750-9。
- 35.
吴刚,蒲秀珍。植物茎部发育的时间调控。植物学报,2003,54 (5):574 - 578拟南芥miR156和它的目标SPL3。开发(英国剑桥)。2006; 133(18): 3539 - 47。
- 36.
拟南芥(Arabidopsis thaliana) mir156调控的SPL转录因子定义了一个内源开花通路。细胞。2009;138(4):738 - 49。
- 37.
崔LG,山爵士,石米,高JP,林HX。MiR156-SPL9-DFR途径坐标植物中发育和非生物胁迫耐受性之间的关系。植物J. 2014; 80(6):1108-17。
- 38.
关键词:二穗短柄草,AP2/ERF转录因子,全基因组鉴定,系统发育BMC基因组学。2016;17(1):636。
- 39.
Artimo P, Jonnalagedda M, Arnold K, Baratin D, Csardi G, de Castro E, Duvaud S, Flegel V, Fortier A, Gasteiger E,等。ExPASy: SIB生物信息资源门户。核酸研究2012;40(Web服务器版本):W597-603。
- 40。
朱T,Xin F,Wei S,Liu Y,Han Y,Xie J,Ding Q,MA L.基因组鉴定,III级过氧化物酶基因族基因系列的基因发生和表达谱。基因。2019; 700:149-62。
- 41。
胡b,金吉,郭艾,张h,罗j,高g. gsds 2.0:升级基因特征可视化服务器。生物信息学(英国牛津大学)。2015; 31(8):1296-7。
- 42.
关键词:番茄,黄卷叶病毒,bHLH,转录因子,全基因组分析BMC基因组学。2015;16:39。
- 43.
黄志明,黄志明,黄志明,黄志明,黄志明,黄志明,黄志明等。Clustal W和Clustal X版本2.0。生物信息学(英国牛津大学)。2007; 23(21): 2947 - 8。
- 44.
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析6.0版。中国生物医学工程学报。2013;30(12):2725-9。
- 45.
王颖,唐浩,Debarry JD,谭鑫,李俊,王鑫,Lee TH, Jin H, Marler B, Guo H,等。MCScanX:一个用于基因共线性和共线性检测和进化分析的工具包。2012;40(7):e49。
- 46.
Krzywinski M, Schein J, Birol I, Connors J, Gascoyne R, Horsman D, Jones SJ, Marra MA。Circos:比较基因组学的信息美学。基因组研究》2009;19(9):1639 - 45。
- 47.
王D,张Y,张Z,朱家,余J.Kaks_Calculator 2.0:一种工具包,包括伽马系列方法和滑动窗策略。基因组学蛋白质组学生物信息学。2010; 8(1):77-80。
- 48。
关键词:植物,转录因子,中心枢纽,转录因子,转录因子核酸研究2017;45(D1): D1040-d1045。
- 49。
叶杰,张勇,崔红,刘杰,吴勇,程勇,徐辉,黄鑫,李硕,周安,等。WEGO 2.0:一个用于分析和绘制GO注释的web工具,2018年更新。核酸图谱2018;46(W1): W71-w75。
- 50.
Lee T,Yang S,Kim E,KO Y,Hwang S,Shin J,Shim Je,Shim H,Kim H,Kim C等人。Aranet V2:用于研究拟南芥和27种其他非典范植物物种的共同官能基因网络的改进数据库。核酸RES。2015; 43(数据库问题):D996-1002。
- 51.
Shannon P,Markiel A,Ozier O,Baliga NS,Wang JT,Ramage D,Amin N,Schwikowski B,Ideker T.Cytoscape:用于生物分子交互网络的集成模型的软件环境。Genome Res。2003; 13(11):2498-504。
- 52.
Ramirez-Gonzalez RH, Borrill P, Lang D, Harrington SA, Brinton J, Venturini L, Davey M, Jacobs J, van Ex F, Pasha A等。多倍体小麦的转录景观。科学(纽约,纽约)。2018; 361:6403。
致谢
非常感谢马教授和丁教授的指导。感谢西北农林大学仪器共享平台和实验室成员对本次研究的协助。我们感谢自然研究编辑服务的英文编辑。
资金
该研究得到了中国国家重点研究和发展方案(2016年美国专利号0101602)。资金机构没有参与研究,分析或解释数据或写作稿件的设计。
作者信息
从属关系
贡献
TZ整理设计研究,分析数据,撰写稿件。YL和LTM完成了数据的可视化。XYW、DZZ和YCH进行了实验。QD和LJM指导了这项研究。所有作者均已阅读并批准稿件。
相应的作者
道德声明
伦理批准并同意参与
本试验所用中国春季品种由西北农林大学马令健教授提供。这些植物材料在全球广泛使用。本文不包含任何人类或动物参与的研究,也不涉及任何濒危或受保护物种。
同意出版
不适用。
相互竞争的利益
作者们宣称他们没有相互竞争的利益。
附加信息
出版商的注意
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充信息
附加文件1:表S1。
特征的特征SPL小麦的基因家族。表S2。qRT-PCR引物。表S3。KA / KS比率和重复的估计分歧时间TASPL.基因对。表S4。Ka / Ks比率和估计的直向性发散时间TASPL.小麦和水稻之间的基因。表S5。Ka / Ks比率和估计的直向性发散时间TASPL.小麦和玉米之间的基因。表S6。Ka / Ks比率和估计的直向性发散时间TASPL.小麦和小麦之间的基因答:tauschii.表S7。Ka / Ks比率和估计的直向性发散时间TASPL.小麦和小麦之间的基因b . distachyon.表S8。TaSPL蛋白的GO注释。表S9。小麦蛋白质 - 蛋白质与其他蛋白质之间的相互作用。表S10。中包含的顺式元素TASPL.基因启动子区域。图S1。启动子地区的CIS-MOVINACATION元素TASPL.基因。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
朱涛,刘洋,马磊。等等。全基因组鉴定,系统发育和表达分析SPL小麦的基因家庭。BMC植物杂志20.420(2020)。https://doi.org/10.1186/s12870-020-02576-0
收到了:
接受:
发表:
关键词
- SPL基因家族
- 系统发育分析
- 表达模式分析
- 小麦