摘要
背景
绿豆(豇豆属辐射动物(L.)R. Wilczek或Green Gram)是重要的热带和亚热带豆类和丰富的膳食蛋白质和微量营养素来源。在这项研究中,我们使用GWA来检查Mungbean在Mungbean中几种重要特征的变异遗传基础,使用世界蔬菜中心建立的迷你核心收集,其中包括296个代表主要市场班级的加入。这一系列已在2018年俄罗斯南部南部部分的共同场地情节中成长。
结果
通过对293份遗传多样性、连锁不平衡、群体结构和GWAS分析的严格筛选,我们使用了5041个SNPs。多态性分布在所有染色体之间,但分布密度不同。连锁不平衡衰减约105 kb。293份供试材料中鉴定出4个不同的亚群,其中70%的供试材料来自4个群体之一。通过对微核心种质进行GWAS分析,我们发现了几个与两个重要农艺性状显著相关的位点。2018年在俄罗斯南部库班地区发现了4个与成熟可能性相关的snp,这些snp位于一个强连锁区域,包含编码锌指A20和AN1域胁迫相关蛋白的基因。
结论
世界蔬菜中心建立的绿豆核心种质是绿豆育种的宝贵资源。2018年,在异常炎热的天气和不同的光周期造成的偶然压力下,该系列在俄罗斯的南欧部分种植。我们发现了几个与下胚轴颜色和在这些胁迫条件下成熟的可能性显著相关的位点。与成熟可能性相关的SNP定位于2号染色体上具有强连锁的区域,其中编码锌指A20和AN1结构域应激相关蛋白(SAP)的基因位于该区域。由于物候学仍然是绿豆的一个关键育种目标,因此,在温带气候地区对WorldVeg采集的成熟性状进行表型分析非常重要。随着绿豆需求的增加,生长季节较短的温带地区的生产对于满足需求至关重要。发现物候性状的SNP将加速育种工作。
背景
绿豆(豇豆属辐射动物(l)是一种重要但未得到充分利用的热带和亚热带豆类植物,是膳食蛋白质和微量营养素的丰富来源,在各种作物轮作系统中恢复土壤肥力发挥着重要作用。在印度以及中国、印度尼西亚、泰国、缅甸和菲律宾等其他热带国家,绿豆具有最重要的农学和文化意义,在全球600万公顷的绿豆产量中,印度占大多数[1].但是,它在温带地区的温带地区增加了澳大利亚南欧的干旱地区,美国南部大平原,零星的俄罗斯联邦,在那里可以补充冬季年度,如小麦和大麦等持续时间短,允许在休耕季节以前常见的地方每年进行两次作物周期。目前,Mungbean占谷物豆科作物世界生产区的8.5%(不计算油籽豆科大豆)[2].
虽然历史上看了一个孤儿豆类[3.[世界上35个基因库中积累了相当大的遗传资源[4.,其中一些是很好的表型。n.i.瓦维洛夫全俄罗斯遗传资源研究所(VIR)最古老的表型数据已被用于追溯过去100年的作物育种趋势[5.].此外,近年来绿豆基因组资源的开发,使分子解剖重要农艺和驯化性状。种植绿豆的基因组于2014年完成,它的野生亲缘种的基因组进行了补充诉放射虫纲var。sublobata.和诉reflexo-pilosa[6.].高密度分子标记已经开发出来,允许对多样性面板(如核心收藏品)进行表征。世界蔬菜中心(worldvec)发展了应用最广泛的核心收集和微型核心收集[7.],这对Mungbean育种和改进有国际授权。
鉴于生产莫尔奇的广泛的地理范围,以及诸如缺乏避免居住地(干旱,冬季感冒或更经济上有价值的旋转伴侣,如小麦或米),评估的疾病允许它完成其生命周期的重要性开花时间和相关农艺性状的遗传基础对于寻找持续的育种努力来寻找有用的遗传变异至关重要。基因组 - 范围协会研究(GWAS)已成为寻找种质遗传变异的强大方法。这种方法已被用于来自大豆的一系列豆类[8.和模范豆科植物Medicago Truncatula.[9.那10那11,以及其他以前被认为是“孤儿”的豆类,如鹰嘴豆[12那13豇豆[例如,[14]和鸽子豌豆[15].
在这里,我们使用GWA来检查Mungbean中几种重要特征的遗传基础,使用Worldveg建立的Mungbean的迷你核心收集,其中包括296种代表Mungbean的主要市场类和所有的微卫星等位基因1400种附加的集合[7.].这一系列已在2018年俄罗斯南部南部的共同场地情节中成长。
结果
标记多态性分析
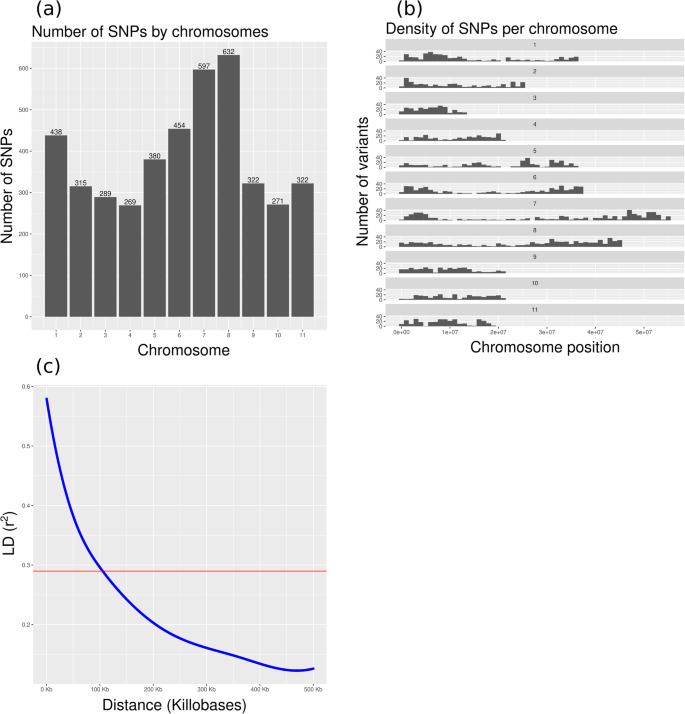
采用基因分型法(GBS)对293份材料的基因组内多态性进行了调查。SNPs被过滤以保留至少90%的基因型中存在的多态性,次要等位基因频率至少为3%。4288个多态性分布在所有染色体上,但其密度不同(从第4染色体上的269个SNPs到第8染色体上的632个SNPs),其中752个SNPs在支架上(图)。1a、b)。
估计联动不平衡
估计连锁不平衡估计在5041的SNP和R的一半之间2最大值(右2= 0.29)为临界值。染色体内r2值与该阈值相交于大约105 kb的物理距离(图。1c) 。
Mungbean迷你核心系列人口结构
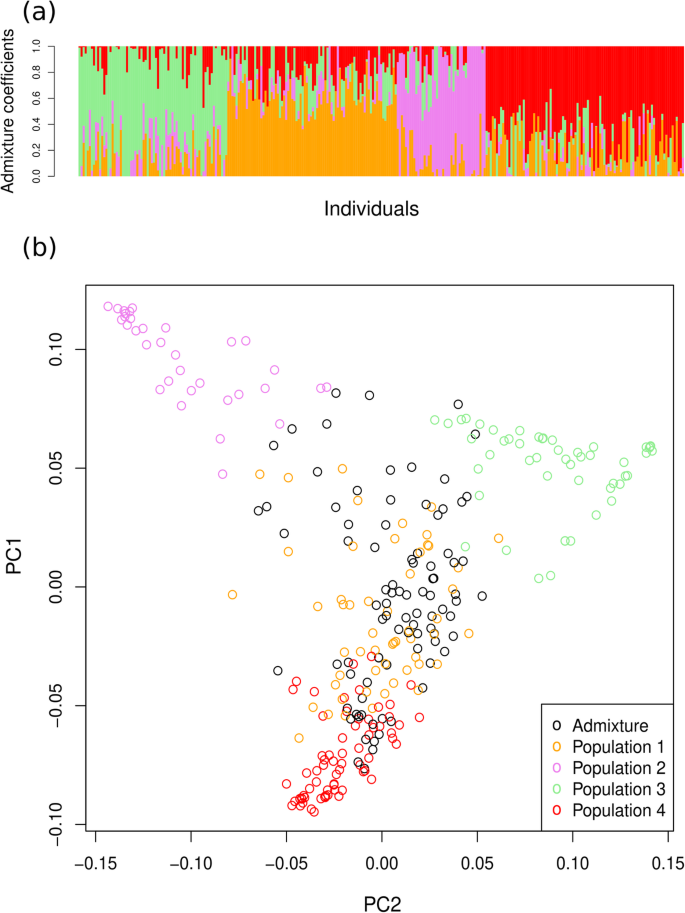
R包Lea的结构状群体遗传分析[16]用于分析来自绿豆微型核心种质的293份材料的群体结构,并根据最小交叉熵标准(附加文件)确定K值4,以最佳捕获群体结构1:图S1)。使用0.55作为4个群体中每个加入的聚类可能性,204份(70%)加入归于4个群体中的一个(图)。2一种)。第一个群体由54名加入组成,第二个包括33名加入,第三个包括50个载体,第四组由67名加入组成。293名中剩余的89个被陈述(附加文件2:表S1;附加文件3.:表S2)。
利用主成分分析法(PCA)对种群分化模式进行了分析。图形2b描绘了第一与第二组件的遗传数据和附加文件的PCA图4.:图。S2描绘了归因于前五个原理组分的变化和协变的概述,其解释了所有标记的差异的32%。
固定索引(f英石)使用VCFTOOLS计算四个子群[17].种群1和种群4亲缘关系最密切(F英石= 0.08),种群2、3和4分化程度相当高(F英石= 0.42, 0.43, 0.37)(附加文件5.:表S3)。
单一特征关联
遗传和表型数据(表型列表和测量单位在附加文件6.:表S4)是弱一致性的,如附加文件中所述7.:表S5,显示了在库班中测量的基因型解释的表型方差的比例。表型特征之间没有观察到强烈的正和负相关(附加文件8.:表S6)。
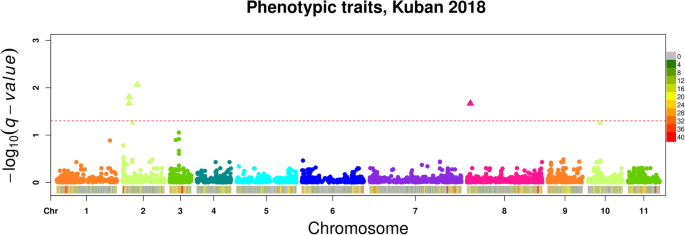
GWAS分析由使用的前五个PCA轴分数用于所有表型数据的协变量。基于基于基因组对照参数分别为每个特征选择最佳类型的分析(λGC).分析显示,这与Kuban(图2)的两个表型性状(成熟的可能性和下胚轴颜色)有显著的相关性。3.;附加文件9.:图。S3;附加文件10:图。S4;附加文件11:表S7)。
为了注释基因组中重要SNPs所在的区域,基于连锁不平衡将基因组划分为单倍型块(haplblock)(附加文件)12:表S8)。
四个与俄罗斯南部库班地区成熟可能性相关的SNP定位于250个区域 2号染色体上的kb具有强连锁。在该区域内确定的基因Vradi02g04380编码锌指A20和AN1结构域应激相关蛋白。胁迫相关蛋白(SAPs),如含A20/AN1锌指结构域的蛋白质,在植物非生物胁迫信号传导中起着重要作用。SAP是防止因暴露于环境压力而导致产量损失的重要候选材料[18].
讨论
绿豆,长期以来是热带世界的一种孤儿豆类作物,正在成为一种适合多种农业生态的多方面作物。作为一种丰富的蛋白质和微量营养素来源,可通过多种方式(达尔、汤、豆芽、面粉等)食用,其种子可快速烹调,在亚洲的许多传统菜肴中以及在日益多样化的全球菜色中占有一席之地。它是新素食蛋(仅蛋)的关键成分,显示出它作为蛋白质来源的适应性。此外,它的生命周期短,适合一系列作物轮作。А使品种适应从热带到温带的一系列条件是满足不断增长的全球需求所必需的。
WorldVeg建立的绿豆核心种质是绿豆育种的宝贵资源。来自世界各地17个国家的293份样本通过了所有筛选,供我们分析。在结构分析中,70%的种质归属于4个群体之一。种群1和种群4由来自印度次大陆(印度和巴基斯坦)的物种组成,在PCA图中可以看到它们聚在一起(图2)。2b) 。人口2代表来自中东的品种,人口3代表来自东南亚和大洋洲的品种。尽管混合程度很高,但在PCA图上,将材料分成四组是非常清楚的(图。2b).根据F的计算英石在4个亚种群中,2个种群(代表印度次大陆的种群1和种群4)关系密切,而其他种群则表现出相当程度的分化。
LD估计在5041个SNP之间,并跌至最大r的一半2物理距离约为105 kB的值。LD的程度反映了人口的有效规模和人口混合物的程度。它在天然群体中通常较小,在面包品种中强烈延伸,以及体重的中间体[19].
候选将是Mungbean的关键育种目标。随着需求升起的摩尔邦,在增长季节较短的温带地区的生产可以帮助需求。此外,随着气候变化,半干旱热带地区的季节,如南亚和东非的大部分,也可能变短。在2018年的库班地区,通过开花和开始成熟阶段的异常炎热的炎热天气,以及一些进入的阶段的开始,以及换档的发展非常不均匀。通过植物暴露于不同的光周期,与干旱结合不同光周期引发这种不均匀性,并且代表对这些偶然应力的植物反应。
通过在俄罗斯南部种植的Mungbean微型核心系列上进行GWA,我们发现了几个与幼杆子成熟和颜色显着相关的基因座。我们发现,具有在库曼的可能性相关的SNP,其具有染色体2上的区域,具有强烈的连锁,其中编码锌指A20和AN1域SAP的基因。已知SAP,以防止由环境压力引起的产量丧失[18].揭示这些特征的SNP将换药繁殖锻炼,适用于温带地区的培养。此外,将有必要了解与种子尺寸或叶形等其他特征的相关性诸如种子尺寸或叶片形状的候选,以适应新品种以改变农生病病症。
结论
在这里,我们使用Worldveg建立的迷你核心集合来执行Mungbean的加瓦斯。该系列于2018年在俄罗斯南部的南部部分种植,在异常炎热的天气和不同的光周期引起的偶然的压力下。我们发现了几个与次髓基质成熟和颜色显着相关的基因座。我们发现,具有在施克曼在施施染色体2上的区域的可能性相关的SNP,其具有强的连杆,其中编码锌指A20和AN1结构域SAP的基因。已知SAP,以防止由环境应力引起的产量丧失。温带气候位置的成熟特性的该系列的表型对于未来的育种应用很重要。
方法
植物材料
绿豆(豇豆属辐射动物)迷你核收集收集自世界蔬菜中心基因库在多边体系下托管的品种,详情见[7.].该系列的种子是从世界蔬菜中心收到的标准材料转移协议。该集合的种子可供公众使用(http://seed.worldveg.org/)(见附加文件2:表S1)。
表型
在2018年在古兰经施工实验站进行了293种进程的表型。
Kuban实验站Vir酒店位于Prikubanskaya平原的草原区45°22 n,40°37e,距离高加索山脊的马刺队的开头80公里。土壤是一个弱浸出的切屑。土壤表面视野中的腐殖质含量为3.6-4.6%。
Kuban附近的气候为温带大陆,夏季的夏季,其特点是水分不足,极端不稳定的所有气候元素。1月份最冷的月份的平均月度温度为2.6°C,最温暖,7月,23°C。年平均降雨量为565毫米。
播种于五月五日进行。夏天雨量不足。平均气温为24.4℃,比正常气温高2.2℃。夏季气温的绝对最高温度为39.9°C,出现在6月-植物幼体生长期间。6月,高温和降水不足导致土壤和大气干旱。异常炎热的天气持续了整个7月-在开花和开始成熟的一些材料。28天有干风,其中8月18天。夏季的降水量为73毫米,是正常值的40%。
加入的发展极不均匀。第一次选择性收割是在7月的第二个十年开始的,目的是防止豆子破裂。最后一次采收时间为10月前10个月,刈割后116天,夜间温度降至0.3℃,低于绿豆植株生长的最适温度。其中128份未进入成熟期。
以下特征在现场记录:缺点颜色,植物习性,天数至50%开花,天至第一成熟豆荚,天数到成熟,植物身高,每个植物荚数,豆荚长度,每个豆荚数,100种子重量,植物重量和产量。只有35个样本成熟,因此我们只接受了六个特征,用于进一步分析和转化日期,以便到期到分类性状的可能性成熟的可能性(是否成熟或未成熟)。表型列表和测量单位是额外的文件6.:表S4。
表型数据分析
Shapiro-Wilk常态测试[20.对表型性状进行定量分析。利用“Hmisc”R库中的“rcorr”函数计算性状的Spearman相关系数[21].
测序(GBS)和SNP呼叫的基因分型
总基因组DNA从三种单株植物的碎片幼叶中提取。在多样性阵列技术P / L(DART)下进行GBS。然后将产生的序列数据与Mungbean参考基因组序列(Vradi_ver6)对齐[6.来识别单核苷酸多态性(SNPs)。
使用VCFtools进一步筛选snp [17,需要少量等位基因频率(MAF) > 3%和基因型调用率> 90%,因此293份材料中有5041个snp通过了所有筛选,有待进一步分析。
遗传数据分析
使用“SNPRelate”R库进行主成分分析(PCA)[22].R中的自定义脚本[23]用这些方法绘制染色体上SNP的深度和分布。
估计联动不平衡(LD)
使用平方相关系数估计LD(R.2基因型之间)。VCFtools [17用来计算染色体内r2值。只有r2标记间物理距离小于500 kb的snp值绘制LD衰减图。通过拟合染色体内r的光滑样条来评估LD衰变2r中标记之间的物理距离(bp)的值[23].r的一半2最大值被视为临界值。估计LD被接受的阈值估计是r2= 0.29。染色体内r的封闭样条的交点2具有此阈值的值被认为是LD范围的估计。
估计所有基因组SNP所解释的表型方差比例
GCTA计划[24用来估计所有基因组SNP所解释的表型的差异比例。首先,表型数据被标准化。然后,使用GCTA-GRM分析计算来自基因组SNP的个体之间的遗传关系。最后,进行GCTA-GREML分析以估计所有GWAS SNP(即基于SNP的可遗传性)解释的表型中的变化比例。
人口结构分析
R包Lea的结构状群体遗传分析[16用于分析来自Mungbean的迷你核心收集的293种载体的人口的结构。选择亚步骤的数量是基于交叉熵标准。该标准基于对掩蔽基因型(矩阵完成)的一小部分的预测,以及交叉验证方法。跨熵标准的较小值通常意味着更好的运行。对每个模拟值的k的10个独立运行,范围为1至10. k值,其中跨熵曲线表现出平台(k = 4)。从单个亚群体中获得身份超过55%的个体加入被归类为该亚群体的代表。固定索引(f英石)使用VCFTOOLS计算四个子群[17].
映射方法
GWAS分析使用单位点线性混合模型,在FaST-LMM工具集(因子光谱变换线性混合模型)中实现[25].5041 SNP的主成分分析(PCA)显示,前五个重要的主要成分(PC)解释了所有标记的差异的32%。LMM模型在没有使用作为所有表型数据的协变量的前五个PCA轴分数的情况下实现。基于基于基因组对照参数分别为每个特征选择最佳类型的分析(λGC).我们使用了虚假的发现率(FDR)[26] 0.05为每个特征分别确定重要的性状相关基因座。使用“cmplot”库构建曼哈顿图[27]在R.使用豆类信息系统(LIS)进行显着相关标记的注释[28.]数据库。
估计Haploblocks.
将基因组分成单倍型块(HaploObls)基于连杆不平衡,Haploview Tools [29.应用于位于染色体上的4289个SNP。使用默认的HAPLOVIEW参数鉴定具有强烈连接的染色体区域(LD的置信区间[0.7,0.98])。每个HPPOOBLOCK被认为是位于给定的HAPLOOCK内的一组SNP,并用于注释显着的相关标记。
可用性数据和材料
在Bioproject Prjna645721下的国家生物技术数据库中获得序列数据。
改变历史记录
2021年1月15日
这篇文章的修订本已经发表,可以通过原始文章访问。
缩写
- GWAS:
-
基因组协会研究
- 树液:
-
压力相关的蛋白质
- Worldveg:
-
世界蔬菜中心
- SNP:
-
单核苷酸多态性
- vir:
-
N. I. Vavilov全俄遗传资源研究所
- GBS:
-
基因分型结果进行排序
- PCA:
-
主要成分分析
- F英石:
-
固定指数
- LD:
-
连锁不平衡
- 省:
-
分集阵列技术
- MAF:
-
轻微的等位基因频率
- FAST-LMM:
-
分解谱变换线性混合模型
- 罗斯福:
-
错误发现率
参考
- 1.
粮农组织。2018年粮食和农业状况。罗马:移民、农业和农村发展;2018年。
- 2.
建立国际绿豆改良网络。2016。https://avrdc.org/intl-mungbean-network.访问2019年6月29日。
- 3.
Chivenge P, Mabhaudhi T, Modi AT, Mafongoya P.在撒哈拉以南非洲缺水条件下,被忽视和未充分利用的作物物种作为未来作物的潜在作用。国际环境与公共卫生杂志2015;12(6):5685-711。https://doi.org/10.3390/ijerph120605685.
- 4。
比什特是,辛格M。亚洲豇豆:谷类豆类改良的遗传和基因组资源。Singh M,Bisht IS,编辑:伦敦:Elsevier Insights;2013年,第页。237–67.https://www.sciencedirect.com/science/article/pii/B9780123979353000104?via%3Dihub.
- 5。
Burlyaeva M, Vishnyakova M, Gurkina M, Kozlov K, Lee CR, Ting CT, Schafleitner R, Nuzhdin S, Samsonova M, von Wettberg E.绿豆系列[Vigna radiata (L.)]urdbean [V.]曼戈(l)瓦维洛夫研究所(VIR)的性状多样性和育种过程在过去100年的趋势。植物资源学报。2019;66(4):767-81。
- 6。
姜玉杰,Kim SK, Kim MY, Lestari P, Kim KH, Ha B-K, Jun TH, Hwang WJ, Lee T, Lee J.绿豆基因组序列和对Vigna物种进化的认识。Nat Commun。2014;5:5443。
- 7。
王永强,林春华,朱绍峰,等。AVRDC -世界蔬菜中心绿豆(Vigna radiata)核心和迷你核心收藏。BMC基因组学。2015;16(1):344。https://doi.org/10.1186/s12864-015-1556-7.
- 8.
黄玉宇,宋琪,贾刚,Specht JE, Hyten DL, Costa GL, Cregan PB。大豆种子蛋白质和含油量的全基因组关联研究。BMC基因组学。2014;15:1。
- 9.
Bonhomme M, André O, Badis Y, Ronfort J, Burgarella C, Chantret N, Prosperi JM, Briskine R, Mudge J, Debellé F, Navier h。新植醇。2014;201(4):1328 - 42。
- 10.
Michno J-M,Burghardt Lt,Liu J,Jeffers Jr,Tiffin P,Stupar R,Myers Cl。鉴定候选基因底层特异性表型表型Medicago Truncatula.通过整合全基因组关联研究和共表达网络。BioRxiv。2018年。https://doi.org/10.1101/392779.
- 11.
康永强,张志强,张志强,张志强,崔永强,黄志强。短梗紫花苜蓿盐胁迫响应性状的全基因组关联分析。植物学报。2019;42(5):1513-31。https://doi.org/10.1111/pce.13508.
- 12.
Plekhanova E,Vishnyakova Ma,Bulyntsev S,Chang Pl,Carrasquilla-Garcia N,Negash K,等。Vavilov的历史体育群体基因组和表型分析揭示了农艺性状环境和基因组岛的影响。SCI REP。2017; 7:4816。
- 13。
瓦尔沙斯rk,thudi m,roorkiwal m,他w,upadhyaya hd,yang w等人。429个国家的429鸡眼的重组提供了对基因组多样性,驯化和农艺性状的洞察。NAT Genet。2019; 51:857-64。https://doi.org/10.1038/s41588-019-0401-3.
- 14。
徐鹏,吴晓霞,Muñoz-Amatriaín M,王波,吴晓霞,胡颖,Huynh BL, Close TJ, Roberts PA, Zhou W, Lu Z.豇豆荚果长度变化的基因组区系、细胞组成和基因调控基础。生物技术学报2017;15(5):547-57。
- 15.
Varshney Rk,Saxena RK,Upadhyaya HD,Khan Aw,Yu Y,Kim C等人。全基因组重量为292个Pigeonpea附加物,识别与驯化和农艺性状相关的基因组区域。NAT Genet。2017; 49:1082-8。https://doi.org/10.1038/ng.3872.
- 16。
Falush D,Stephens M,Pritchard JK。使用多点基因型数据推理人口结构:主导标记和零等位基因。Mol Ecol Notes。2007; 7:574-8。
- 17。
DaneCek P,Auton A,Abecasis G,Albers Ca,Banks E,Depisto Ma,等。变体呼叫格式和vcftools。生物信息学。2011; 27:2156-8。
- 18.
Ben Saad R, Safi H, Ben hsoua A, Brini F, Ben RW。LmSAP蛋白的功能域分析揭示了锌指A20结构域在非生物胁迫耐受中的重要作用。原生质。2019;256(5):1333 - 44。https://doi.org/10.1007/s00709-019-01390-2.
- 19.
Von Wettberg EJB,Chang Pl,Başdemirf,Carasquila-garcia n,Korbu Lb,Moenga Sm,等。一种重要作物野生的生态学和基因组学相对农业创新的前奏。NAT Communce。2018; 9(1):649。https://doi.org/10.1038/S41467-018-02867-018-02867-0.
- 20。
Shapiro SS,Wilk MB。正常性差异试验分析(完整样品)。Biometrika。1965年; 52(3-4):591-611。https://doi.org/10.1093/biomet/52.3-4.591.
- 21.
Harrell JR Fe。HMISC:Harrell杂项。R包版本4.1-1。2018年。https://CRAN.R-project.org/package=Hmisc.2019年4月20日。
- 22.
郑晓东,沈建军,李志强,李志强。基于主成分分析的SNP数据相关性分析方法。生物信息学,2012,28(24):3326 - 8。
- 23.
r核心团队。R:统计计算的语言和环境。R基金会统计计算,维也纳,奥地利。2018年。https://www.R-project.org/.2019年3月15日访问。
- 24.
杨杰,李舍,戈达德我,visscher pm。GCTA:一种用于基因组复杂性状分析的工具。我是j嗡嗡声的遗传。2011; 88:76-82。
- 25。
Lippert C,Listgarten J,Liu Y,Kadie Cm,Davidson Ri,Heckerman D.基因组关联研究的快速线性混合模型。NAT方法。2011; 8:833-5。
- 26。
楼层jd。正面虚假发现率:贝叶斯解释和Q值。安统计。2003; 31:2013-35。
- 27。
CMplot:环绕曼哈顿地块。https://github.com/YinLiLin/R-CMplot. 2019年6月10日查阅。
- 28。
Dash S, Campbell JD, Cannon EK, Cleary AM, Huang W, Kalberer SR,等。豆科信息系统(LegumeInfo。Org):豆科植物的一组联邦数据资源的关键组件。诊断。酸> 2016;44:D1181-8。
- 29。
巴雷特JC,弗莱B,马勒J,戴利MJ。Haploview: LD和单体型图的分析和可视化。生物信息学。2005;21:263-5。
致谢
我们感谢Joyce Yen了解说明Mungbean迷你核心集合的GBS如何进行。EVW的庄稼驯化课程为稿件提供了有用的评论。
关于这个补充
本文已作为《BMC植物生物学》第20卷补编1(2020年):第五届国际科学会议“植物遗传学、基因组学、生物信息学和生物技术”(PlantGen2019)选文的一部分发表。补编的全部内容可在以下网址查阅://www.cinefiend.com/articles/supplements/volume-20- supplement-1..
资金
俄罗斯科学基金会授予18-46-08001支持数据的研究,分析和解释(植物生长和表型,遗传多样性分析和GWA)的设计,以及本文的写作和出版。EVW也支持通过佛蒙特州实验站的USDA舱口资金。CRL和CTT由台湾科技部107-2923-B-002-004-MY3支持。基因型数据的集合由澳大利亚国际农业研究中心(ACIAR) - 废弃的国际Mungbean改善网络(CIM-2014-079)和其他核心捐助者以及世界蔬菜中心的其他核心捐助者:中华民国(台湾),联合国国际发展局(USAID),澳大利亚国际农业研究中心(ACIAR),泰国,菲律宾,韩国和日本。
作者信息
从属关系
贡献
AS进行GWAS分析。MB、TV、MV进行数据集表型分析。RMN建立了绿豆迷你核心收集物并对其进行基因分型。RS放大并提供了迷你核心集合。MG、MV、SN、CRL、CTT、RS和EvW负责项目开发,监督分析和项目人员,并制定手稿大纲和分析框架。AS在所有作者的帮助下完成了手稿。所有作者都对手稿的修订和编辑做出了贡献。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
提交人声明他们没有竞争利益。
补充资料
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1。
293份绿豆材料的交叉熵图。X轴表示祖先种群的数量,Y轴表示最小交叉熵。对K的每个模拟值进行十次独立运行,范围从1到10。选择交叉熵曲线呈现平台的K值(K = 4).
附加文件2:表S1。
目录的加入,收集地点和加入的团体。利用R package LEA的类结构群体遗传分析鉴定了4个群体。从单个亚群体中获得身份超过55%的个体加入被归类为该亚群体的代表。其余的加入物被认为是外加剂。
附加文件3:表S2。
17个原产国的人口中绿豆的加入数。
附加文件4:图S2。
基于5041 SNP的PCA分析前五个主要成分的散点图。每个点表示加入。颜色编码根据会员(基于> 55%的身份)到从结构分析中识别的群体。
附加文件5:表S3。
固定索引(f英石)在四个亚种群中。
附加文件6:表S4。
2018年在库班测量的表现型列表。
附加文件7:表S5。
所有GWAS SNPS(即SNP的遗传性)解释的表型中的差异比例为在库班测量的特征。
附加文件8:表S6。
表型性状之间的相关系数。
附加文件9:图S3。
在库班测量的表型性状成熟可能性的GWAS分析总结(a) SNP QQ图(b) SNP曼哈顿图(不同的颜色对应不同的染色体)。q值单核苷酸多态性< 每个染色体显示0.05,标记为三角形。
附加文件10:图S4。
库仑术术分析Gwas分析综述。(a)SNP QQ-PLOT。(b)SNP曼哈顿图(不同颜色对应于不同的染色体)。每条染色体显示带Q值<0.05的SNP,标记为三角形。
附加文件11:表S7。
在库曼测量的表型特征的重要SNP。在施克曼,2018中测量的表型特征的GWAS分析结果。使用豆类信息系统(LIS)数据库进行了重要的相关标记的注释。
附加文件12:表S8。
由Haploview工具推断的haploblock。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Sokolkova,A.,Burlyaeva,M.,Valiannikova,T.等等。绿豆微型核心种质资源全基因组关联研究(豇豆属辐射动物)来自世界蔬菜基因库(台湾)。BMC植物BIOL.20,363(2020)。https://doi.org/10.1186/s12870-020-02579-020-02579-0.
已收到:
公认:
发表:
关键词
- 绿豆
- 核心
- 表型
- GWAS
- 人口结构
- 候选性状