摘要
背景
杜鹃molle是的(Ericaceae)是一种传统的中国药用植物,其花和根在中国已有几千年的历史,被广泛用于治疗风湿病和缓解疼痛。化学研究已经揭示了这一点r . molle是的含有丰富的次生代谢物,如萜类、类黄酮和木酚素,其中一些表现出各种生物活性,包括抗氧化、降压和镇痛活性。尽管次级代谢物具有巨大的药学重要性,但其生物合成机制仍然未知,基因组信息也无法获得。
结果
为了深入了解这种植物的分子结构,特别是包括灰烷二萜在内的重要次生代谢产物的信息,我们对其进行了深度转录组测序r . molle是的花和根使用Illumina Hiseq平台。通过de novo组装共生成了100603个unigenes,平均长度为778 bp,其中57.1%的unigenes在公共数据库中有注释,其中17906个unigenes在KEGG数据库中有显著匹配。注释了参与次生代谢物生物合成的Unigenes,包括可能负责灰色类化合物生物合成的TPSs和CYPs。此外,还鉴定出3376个转录因子和10828个简单序列重复序列(SSRs)。此外,我们进一步对花和根转录组文库进行了差异基因表达(DEG)分析,发现了许多在花中特异性表达或上调的基因。
结论
据我们所知,这是第一次生成并彻底分析两者的转录组数据r . molle是的花和根。该研究为阐明植物次生代谢物的生物合成途径提供了重要的遗传资源r . molle是的特别是对那些具有药用价值和允许药物开发的植物。
背景
杜鹃molle是的是一种多年生开花灌木,原产于中国的长江以南,如广东和广西。这种植物多生长在海拔1000米左右的丘陵地区的灌木或乔木下[1].的药用特性r . molle是的已经被确认的历史可以追溯到几千年前[2,3.].这种植物在最早的中国药典中有记载和描述神农不同经(《神农本草经》,编撰于东汉初年(公元25年至220年)。作为中国最著名的传统药用植物之一,r . molle是的自古以来被广泛用作止痛药和麻醉剂[4].据古代医书记载,干花和根都可入药,治疗风湿、外伤、偏头痛和神经痛[1,4].在一些农村地区,这种植物还被用作杀虫剂[5].现代化学研究已经证明了这一点r . molle是的产生丰富的次生代谢物,主要包括萜类、类黄酮和木酚素[6,7,8,9],在这些化合物中,灰烷二萜是该植物最重要的特征成分,这是该植物最显著的生物活性,即镇痛、抗痛觉作用[7,10,以及低血压活动[11].药理学研究证明,类灰色化合物对急性、炎症性和神经性疼痛具有显著的镇痛活性[7].到目前为止,从菊科植物的花和根中分离出了许多类灰岩r . molle是的而且这个数字还在不断增加。黄酮类化合物是天然植物中常见的次生代谢物,具有多种药理活性,目前已从黄酮类化合物中分离到大量的黄酮类化合物r . molle是的,如槲皮素、山奈酚、皮黄素、牡荆素[3.,12,13].此外,Zhi等人从根中分离出一些有趣的ligansr . molle是的[14].近年来,基于NGS(下一代测序)技术的RNA-Seq方法得到了发展,并被广泛用于快速和低成本的转录组表征许多重要的药用植物,如百合[15),乌拉尔甘草[16),尤金尼亚uniflora[17),Carthamus tinctorius[18].它还提供了一种有效的方法来加速发现参与特定代谢途径的新酶[19,20.].近年来,RNA-Seq方法也被用于分析植物的开花和花色形成机制r . molle是的[21].参与其他次生代谢途径的基因尚不清楚。在本研究中,对花和根进行了深度从头转录组测序r . molle是的使用Illumina Hiseq平台进行。共获得unigenes 100603条,平均长度为778 bp,其中长度大于1 kb的unigenes 20886条。57.1%的未基因通过序列相似度搜索和蛋白结构域扫描与公开数据库进行了注释。此外,生物信息学分析表明,两种植物的转录组中都存在编码萜类主干生物合成相关酶的基因r . molle是的花和根。共鉴定出9个萜类合成酶(TPS),其中3个二萜合成酶。此外,筛选可能负责进一步主干修饰的候选基因,得到包含61个细胞色素P450 (CYP)序列的基因库。在我们的转录组数据集中,也发现了黄酮和木脂素生物合成的假定成员。我们分别分析了花和根的转录组,并进行了比较分析,以丰富次生代谢产物的生物信息学。我们的转录组数据为发现与各种代谢途径相关的功能基因提供了宝贵的资源,特别是与具有重要药用价值的次生代谢物的生物合成途径相关的假设基因,将为通过合成生物学方法增强具有药用价值的次生代谢物的生物合成铺平道路。
方法
材料及RNA提取
杜鹃molle是的采自中国广西桂林广西植物研究所,经广西植物研究所李光昭教授鉴定。凭证标本ID-24757保存于中国医学科学院药物研究所药用植物研究室植物标本室。这种植物是在中国科学院药用植物发展研究所的温室里种植的。采收根和花,先后用自来水和超纯水冲洗,在滤纸上晒干。树根被切成小块。所有样品立即在液氮中冷冻,并在- 80°C保存,然后进行进一步处理。用Trizol试剂从根和花中提取总RNA,用RNase-free DNase I处理去除基因组DNA。用分光光度计测定RNA的质量和纯度。RNA完整性数(RIN)用安捷伦生物分析仪2100系统进行检测。选择RNA完整性值(RIN)大于8.0的样品进行进一步研究。
cDNA文库构建及测序
根据制造商的说明,使用用于Illumina®(NEB, USA)的NEBNext®Ultra™RNA文库准备试剂盒(NEB, USA)收集一个mirgram RNA样本构建cDNA文库。简单地说,用磁Oligo (dT)珠纯化mRNA分子,破碎并合成cDNA,然后通过PCR生成cDNA文库。使用安捷伦生物分析仪2100系统评估每个样本库的质量。最终,每个组织的转录组文库通过Illumina HiSeq 2000平台(生物标记技术公司,北京,中国)进行测序,并生成配对的端reads。
转录组从头组装和注释
通过cDNA文库测序获得的原始reads首先通过修剪适配器和低质量reads进行处理,以产生干净的reads。使用Trinity软件(2.5.1版本)组装干净的读取以生成转录本[22].然后进行转录本分析,使用TGICL软件(2.1版本)去除冗余,获得无冗余的unigenes [23].所有组装的unigene序列在主要的公共数据库中进行相似性搜索,包括NCBI非冗余蛋白(NR) (https://www.ncbi.nlm.nih.gov/protein/) [24,瑞士Prot数据库(https://www.uniprot.org/uniprot/) [25]、同源基团簇(COG) (http://www.ncbi.nlm.nih.gov/COG/) [26), eggNOG4 (http://eggnogdb.embl.de/) [27),包含了(https://pfam.xfam.org) [28].blast算法用于识别截断值小于10的同源序列−5.最好的歌曲的注释被记录下来。基因本体(GO) (http://www.geneontology.org/)进一步使用Blast2GO软件(2.5版本)对unigenes的功能进行分类,默认参数[29,30.].利用TransDecoder软件(5.0.0版本),根据Pfam数据库中氨基酸序列与蛋白结构域序列的比对,预测unigenes的编码区序列(CDS)和相应的氨基酸序列。
使用KEGG进行功能表征
所有组装的unigenes被映射到京都基因和基因组百科全书(KEGG)数据库(http://www.genome.jp/kegg/) [31使用BLASTX和阈值E-value < 10−5.KEGG矫形(KO)作业是通过KOBAS软件(2.0版本)进行的[32使用默认参数。
差异表达分析
通过片段FPKM计算基因表达量[33](每千基每百万片段映射)方法,使用RSEM软件(版本1.2.19)[34].采用EBSeq软件(版本为1.6.0)对两个样本进行差异表达分析。使用qvalue调整Pvalue [35],将qvalue< 0.005阈值和log2(倍数变化)| > 1设置为表达差异显著的两个标准。
转录因子分析
为了鉴定转录因子(TF)家族,将组装好的unigenes与植物转录因子数据库(PlnTFDB)中的TF蛋白结构域进行比对[36](植物转录因子数据库)E价值截止1 e−06.
简单序列重复序列的识别
为了鉴定SSR基序,利用MISA (Microsatellite searching Tool)(1.0版本)对花和根组织转录组序列产生的unigenes进行搜索。在本研究中,我们检测了从单核苷酸到六核苷酸的微卫星,并识别了完美重复序列(包含一个重复基序)和复合重复序列(包含两个或多个重复基序,间隔100个碱基对)。
系统发育分析
利用该菌株中TPSs和CYPs的氨基酸序列构建了系统发育树r . molle是的转录组,以及来自其他植物的具有代表性的功能特征的蛋白质。来自GenBank和swissprot的蛋白序列的登录号列在附加文件中1S1:表。在ClustalX 2.1中对氨基酸序列进行比对。手动调整了对中,去掉了终端间隙。使用MEGA7软件构建TPS和CYP系统发育树[37]的邻居连接(NJ)方法。系统发生树的显著性水平通过1000个重复的自举检验进行评估。
萜类相关基因的鉴定
根据公开的蛋白序列建立了TPSs和cyp的定制数据库。从GenBank检索到的序列在附加文件中给出2S2:表。使用tBLASTn程序从生成的集合中挖掘候选基因E-值阈值1.0 × 10−50最小读取长度为500个碱基对。所有鉴定的unigenes在NCBI数据库中使用BLAST检索进行验证。
实时聚合酶链反应
RNA样本从根和花组织中分离出来。使用TransScript®One-Step gDNA Removal and cDNA Synthesis Super Mix kit (TransGene, Beijing, China)按照制造商说明执行反转录。反应在42°C 15 min, 80°C 5 s。采用UltraSYBR mix (CWBIO, Beijing, China)和LightCycler480 II (Roche, Switzerland) Real-Time PCR System进行实时定量。反应液(20 μL)中含有2 × UltraSYBR混合液10 μL,正、反引物各0.5 μL,模板cDNA 1 μL (150 ng/ul)。PCR扩增过程如下:95°C 10 min, 95°C 15 s, 60°C 1 min, 40个循环。基因特异性引物采用Primer 5.0软件设计,并在附加文件中列出3.:表S3。以GAPDH基因为内标,基于每个样本的3个生物重复确定Ct值,并使用2- - - - - -△△Ct相对定量法[38].
结果
转录组测序和从头组装
从总RNA构建了两个cDNA文库r . molle是的分别为花和根。使用Illumina HiSeq 2000平台对文库进行测序,产生了大约26.58 Gb的干净数据(8900万次读取)。质量检查显示,92%的读数基本质量高于Q30,原始读数在装配前被修剪。适配器和低质量读数被修剪,短读数(< 50 bp)也被删除。从花库和根库中分别获得47,559,180和41,387,924个高质量reads用于进一步分析。采用Trinity软件和TGI聚类工具(TGICL)进行从头组装,去除冗余簇,共生成unigenes 100603个,平均长度为778 bp, N50长度为1384 bp, GC含量为47.7%。40.88%(41,129)的组装unigenes长度大于500 bp, 20.96%(20,886)的组装unigenes长度大于1000 bp。如附加文件所示,大部分unigenes的长度在200bp到2000bp之间4:图S1a。此外,共预测76,198个编码序列(CDS),平均长度515 bp,其中20,213个(26.5%)完整的CDS。在所有预测的CDSs中,18,106例(23.8%)的长度超过200 bp4:图印地)。高质量的测序结果将有利于后续的函数注释。
功能注释
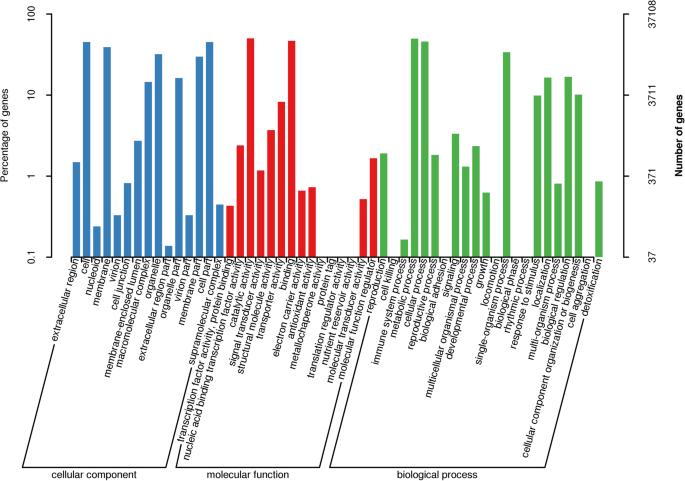
为了对组合unigenes进行综合注释,对8个公共数据库进行序列相似性搜索。结果显示,共有57,416个(57.1%)unigenes在这些公共数据库中具有显著匹配,而其他的则没有信息(如“未知的”、“未命名的”或“假设的蛋白质”)。NR数据库的注释最多(56.2%),COG数据库的unigenes注释最少(16%)。此外,50.2、36.9、32.8、27%、26.8和17.8%的unigenes分别在eggNOG、GO(基因本体)、Pfam(蛋白质家族)、KOG(真核同源组)、swissprot和KEGG中获得了显著的hit(表2)1).此外,E-值和恒等分布,进一步分析BLAST结果。统计分析显示,45.71%的序列具有明显的同源性(< 1.0E−50),而其余的unigenes具有E-取值范围为1.0 × 10−50到1.0 × 10−11(附加文件5:图S2a)。此外,同源分布显示,大部分(73.64%)的unigenes相似性为> 60,37.84%的unigenes相似性介于60 ~ 80%之间,13.83%的unigenes相似性介于50 ~ 60%之间,只有12.54%的unigenes相似性< 50%5:图开通)。更高的身份伴随着高质量E-value证明了本研究生成的从头组装的可靠性。根据Nr注释结果,最佳点数最多的前两种为Quercus木栓(8.88%匹配unigenes)和葡萄(匹配unigenes 5.95%)。(附加文件5:图S2c)。为了方便unigenes的功能分类,我们进行了GO注释,提供了代表基因产物属性的定义术语本体。GO注释进一步分为生物过程、细胞成分和分子功能三类。根据序列同源性,共鉴定出37108条序列,可分为52个官能团(图1)。1).细胞组分类分为15个纲,其中以细胞(16736个unigenes, 45.10%)和细胞部分(16728个unigenes, 45.07%)为主,其次为膜(14451个unigenes, 38.9%)、细胞器(11873个unigenes, 31.9%)和膜部分(11098个unigenes, 29.9%)。在分子功能类中,排名前两位的是催化活性(18589个,50.09%)和结合活性(17291个,46.59%),远远超过转运蛋白活性(3066个,8.26%)和结构分子活性(1373个,3.7%)对应的unigenes。在22组生物过程中,代谢过程的unigenes最多(18425个,49.65%),说明次生代谢物积累丰富r . molle是的其次是参与细胞过程(17,024个unigenes,48.65%)和单生物过程(12,502个unigenes, 32.73%)。
京都基因和基因组百科(KEGG)的路径分析
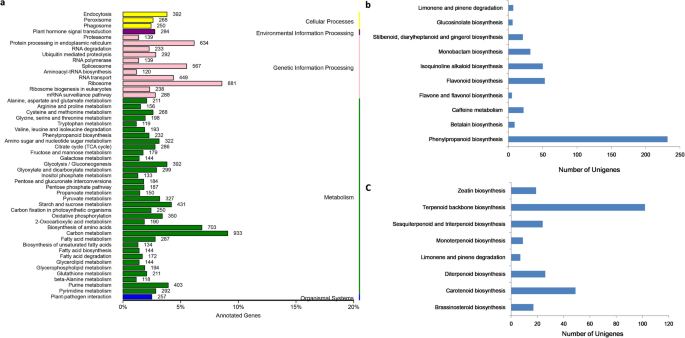
同一通路内的基因通常相互合作,以行使其生物学功能。基于途径的分析有助于理解这些功能和识别参与各种生物合成途径的unigenes。本研究采用阈值进行KEGG通路分析E-value < 10−5.KEGG数据库中有17906个(17.8%)unigenes显著匹配,主要分为五大类,包括细胞过程、环境信息处理、遗传信息处理、代谢和包含130个通路的有机体系统(图1)。2a).在我们的数据集中,最多的unigenes被分为“碳水化合物代谢(993个unigenes)”,其次是“核糖体(881个unigenes)”和“氨基酸生物合成(703个unigenes)”。我们进一步探索了与次生代谢相关的unigenes,共发现了11条通路,包括437个unigenes参与“其他次生代谢产物的生物合成”,其中富集在β -丙氨酸代谢的unigenes最多(118个)(图2)。2b),其次是“苯丙氨酸生物合成”(100个)和“类黄酮生物合成”(53个)。此外,“萜类和多酮类化合物的代谢”亚类包含8个通路,包括253个unigenes,“萜类主干生物合成”是最大的一组(102个),其次是类胡萝卜素生物合成(49个)和二萜生物合成(26个)(图)。2c)。
从差异表达基因的角度来看
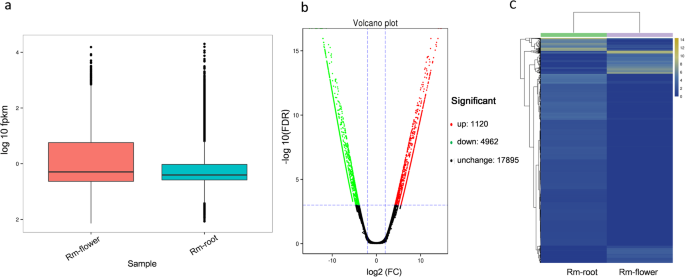
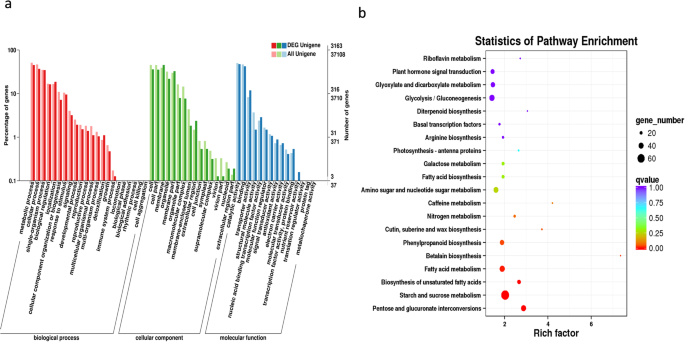
我们对两个转录组文库进行了DEGs分析,以发现表达有显著差异的unigenes。FPKM值用于测定unigenes的表达水平。花unigenes的整体表达水平高于根unigenes(图。3.a).进一步分析发现,在花和根转录组组合产生的100603个unigenes中,有6082个unigenes在花和根中差异表达,其中根和花中上调的unigenes有1120个,下调的unigenes有4962个(图4)。3.B),其中507个unigenes在花中唯一表达。6082个DEGs的分级聚类显示两个组织聚类相对紧密(图。3.c),表明某些DEGs可能参与同一代谢途径。在6082个unigenes中,5314个使用不同的数据库进行了注释。在GO富集分析中,基于序列同源性将花特异性上调unigenes分配到多个本体中,包括细胞成分1254个,生物过程716个,分子功能1133个。在生物过程类别中,GO术语“代谢过程”(GO:0008152)最显著富集(304 unigenes),表明花中存在重要的代谢活动(图1)。4a).为了进一步了解DEGs的代谢途径,我们进行了KEGG富集分析。共鉴定出115个KEGG通路1433个unigenes,其中DEGs富集最多的前3个通路分别是碳代谢(78个,5.4%)、淀粉和蔗糖代谢(66个,4.6%)和氨基酸生物合成(61个,4.3%),均与一级代谢相关(图4)。4b).此外,在次生代谢产物的生物合成通路中也富集有DEGs,共鉴定出222个DEGs,涉及20个生物合成通路,其中32个DEGs集中在类黄酮和苯丙类生物合成通路中,6个DEGs集中在苯丙类生物合成通路中。此外,“萜类和多酮类代谢”亚类包含8个通路,包括27个DEGs,其中最多的DEGs(7个)集中在萜类生物合成主干中。此外,单萜生物合成中有1个DEG,倍半萜和三萜生物合成中有4个DEG,二萜生物合成中有6个DEG6表S4)对这些基因的进一步研究有助于更好地理解萜类生物合成途径。
次生代谢途径分析
萜类主干生物合成相关基因的鉴定
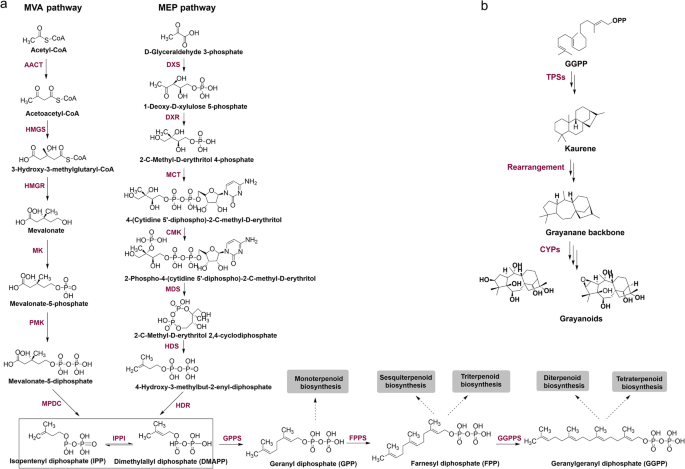
萜类化合物是主要的次生代谢物R.molle特别是属于四环二萜的类灰色化合物。萜类的生物合成过程可分为两个阶段,即萜类主链的合成和特异性萜类的形成和修饰。萜类主链是由二甲基烯丙基二磷酸(DMAPP)和异戊基二磷酸(IPP)合成的,它们是C-5的一般组成单元[39,40].在植物中,胞质甲戊二酸(MVA)和质体甲基赤藓糖醇磷酸(MEP)途径都有助于通过交叉流向DMAPP和IPP提供[41,42].然后,DMAPP与丙烯酰基转移酶催化的IPP进行顺序凝聚,形成不同种类萜的起始前体,即单萜为香叶酰二磷酸(GPP, C-10),倍半萜为法尼基二磷酸(FPP, C-15),二萜为香叶酰香叶酰二磷酸(GGPP, C-20) [39(图。5a).基于KEGG路径分配,共注释了17个与萜类主干生物合成相关的关键酶的102个unigenes,占组装的有路径注释的unigenes总数的0.57%。这些unigenes主要分布在MVA(46个unigenes, 6个酶)和MEP(17个unigenes, 6个酶)通路上,可能参与萜类化合物的共同构建模块IPP的生物合成。下游分布有多个基因(28个unigenes, 3个酶)。在大多数情况下,不止一个unigenes被注释为同一种酶,这表明这些unigenes可能代表同一基因家族的不同成员或单个转录本的不同片段。对应的unigenes列于表中2.其中,发现7个DEGs, 3个unigenes上调与MEP通路相关,包括DXS的1个,2- c -甲基- d -erythitol 2,4环二磷酸(MDS)的1个,异戊二烯合成酶的1个。此外,我们还发现了三个参与MVA通路的下调unigenes,包括两个HMGS基因,一个PMK基因(Table2).这些结果表明,MEP途径主要负责花中萜类化合物的合成。
参与类灰岩生物合成的酶
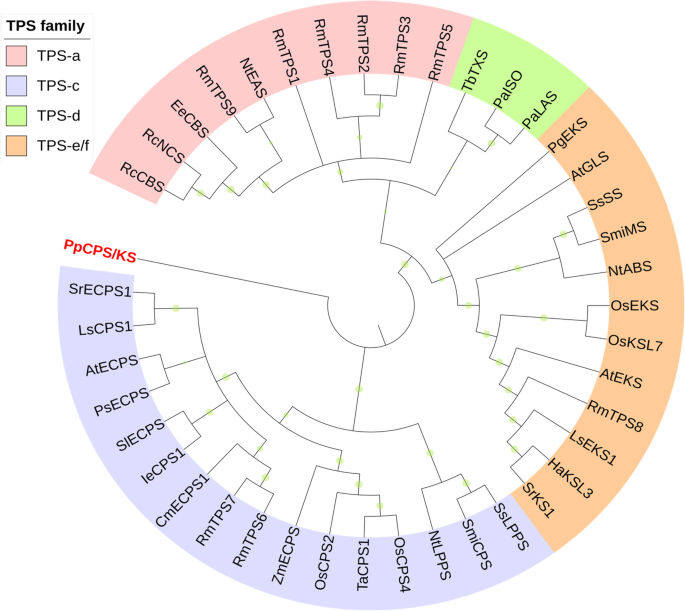
以往的研究报道,从根和花中分离出大量的灰烷二萜r . molle是的[6,7,10这些化合物被认为是这种植物的特有代谢物,具有显著的镇痛活性。提出的灰色阳极生物合成途径是由共同前体GGPP(20-碳)通过萜烯合成酶(TPSs)转化为kaurene,最终生成灰色阳极。从kaurene到grayanane的生物合成过程可能涉及氧化重排(图。5b).主要通过细胞色素P450 (CYP)酶的活性进行修饰。为了识别TPS和CYP候选蛋白,基于公开的蛋白质序列构建了定制数据库,这些序列代表了人口最少且无冗余的序列集。根据与NCBI NR数据库的序列同源性,从转录组数据中鉴定出9个萜类合成酶(附加文件)7:表S5),其中3个unigenene被注释为参与单萜生物合成的芳樟醇合成酶,1个unigenene被注释为germacrene D合成酶。另外,还鉴定出了2个共聚二磷酸合酶副本和1个ent-kaurene合成酶副本,具体情况见表3..根据系统发育关系,这些酶可以分为四个科。6).其中6个候选TPS (RmTPS1-5、RmTPS9)属于TPS-a家族,RmTPS8属于TPS-e/f家族。上述TPSs均具有I类萜类合成酶的特征,在我们的数据集中只发现了两种II类萜类合成酶,分别为TPS-c家族的RmTPS6和RmTPS7。在被子植物中,二萜骨架的形成需要I类和II类萜类合成酶,TPSs的具体功能有待进一步验证。
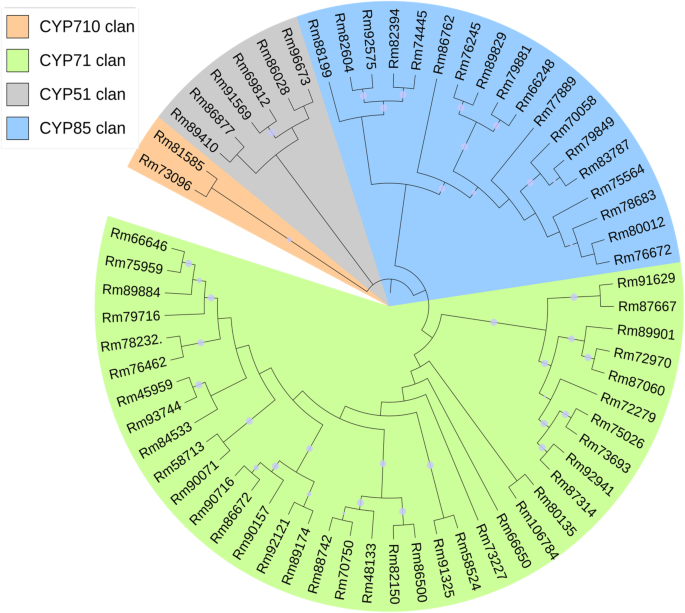
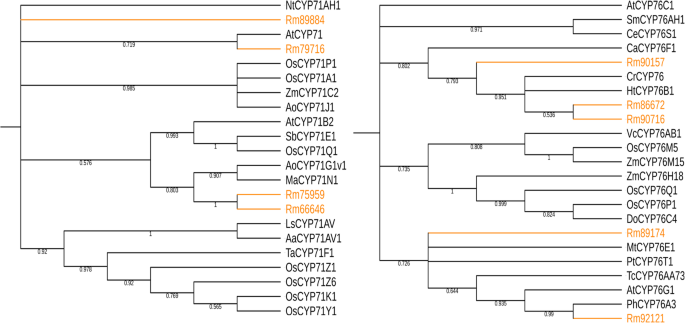
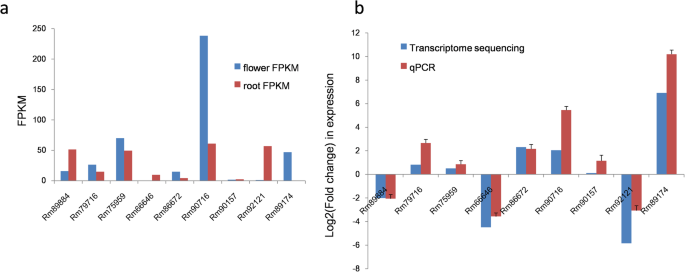
细胞色素P450单加氧酶(CYP)是植物中最大的酶超家族(约占已测序植物基因组的1%),但只有少数参与萜类代谢的CYP被鉴定出来,这使得在植物萜类生物合成途径中鉴定CYP具有挑战性r . molle是的。在这项研究中,R.molle根据p450特异性蛋白数据库挖掘花和根的转录组,共确定了61个候选基因(附加文件7:表S5)。系统发育分析将这些候选CYPs分为4个氏族,其中CYP71氏族成员最具代表性(图1)。7),该族包含了大部分参与植物次生代谢的CYP家族[43,44].植物的萜类代谢主要由少数CYP家族主导,其中CYP71和CYP76家族是主要贡献者[44,45].我们使用来自这两个CYP家族的蛋白质作为探针来研究我们的转录组,共鉴定出了来自CYP71和CYP76家族的9种CYP酶(图1)。8).为了进一步筛选极有可能的CYP候选基因,根据FPKM值评估基因表达水平。结果表明,大部分基因在花和根中均有表达,但表达水平存在差异。其中,Rm89174在花组织中显著上调,Rm66646和Rm92121在根组织中高表达r . molle是的(无花果。9a).通过qRT-PCR分析进一步验证FPKM分析中检测到的unigenes的表达水平(图5)。9b).一般情况下,qRT-PCR检测的表达水平与RNA-Seq数据一致。CYP unigenes在qRT-PCR和FPKM分析中表达一致,证实了测序结果的可靠性。这些结果为进一步研究萜类合成中TPS和CYP候选化合物的功能特性提供了参考r . molle是的.然而,进一步的研究有必要揭示这些unigenes的真正功能。
木酚素生物合成的基因
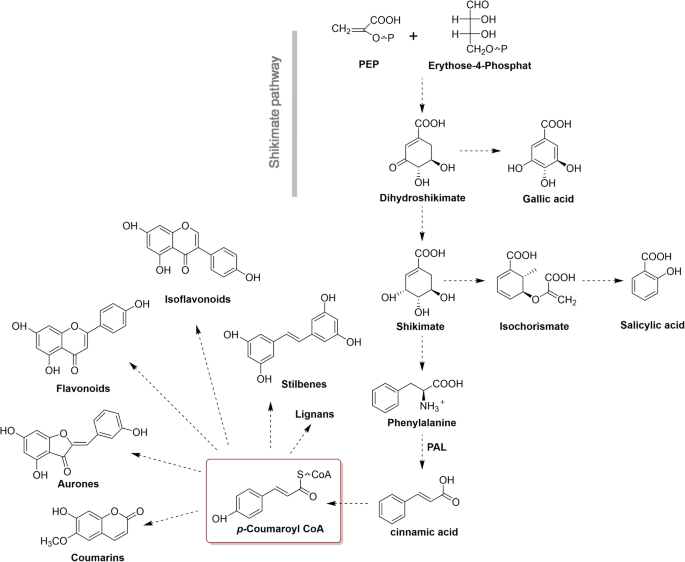
苯丙素是由苯丙氨酸衍生而来的一大批植物天然产物,具有广泛的保护肝脏、抗氧化等生物活性。这些化合物参与植物对生物和非生物刺激的所有方面的反应[46].一般的苯丙类代谢利用石草酯途径的少数中间产物作为基本前体,产生大量的次生代谢物。生物合成途径从苯丙氨酸生成肉桂酸开始,生成肉桂酰-辅酶a和对香豆酰-辅酶a。这些辅酶a激活的化合物是合成木酚素、类黄酮、黄酮醇以及许多其他次生代谢产物的前体。10).在本研究中,我们对两者进行了KEGG分析r . molle是的花和根的转录组和结果显示,共有232个unigenes参与苯丙类生物合成途径。对苯丙氨酸解氨酶(PAL)进行了10个ungenes的注释,对反式肉桂酸4-单加氧酶进行了3个ungenes的注释。这两种酶在重要中间体电影酸的形成中都起着重要作用。18个unigenes被注释为4-香豆酸辅酶a连接酶(4CL), 6个unigenes被注释为肉桂酰辅酶a还原酶(CCR)。此外,还对分支点的酶进行了鉴定,代表性酶列于表中4.
类黄酮合成相关基因的鉴定
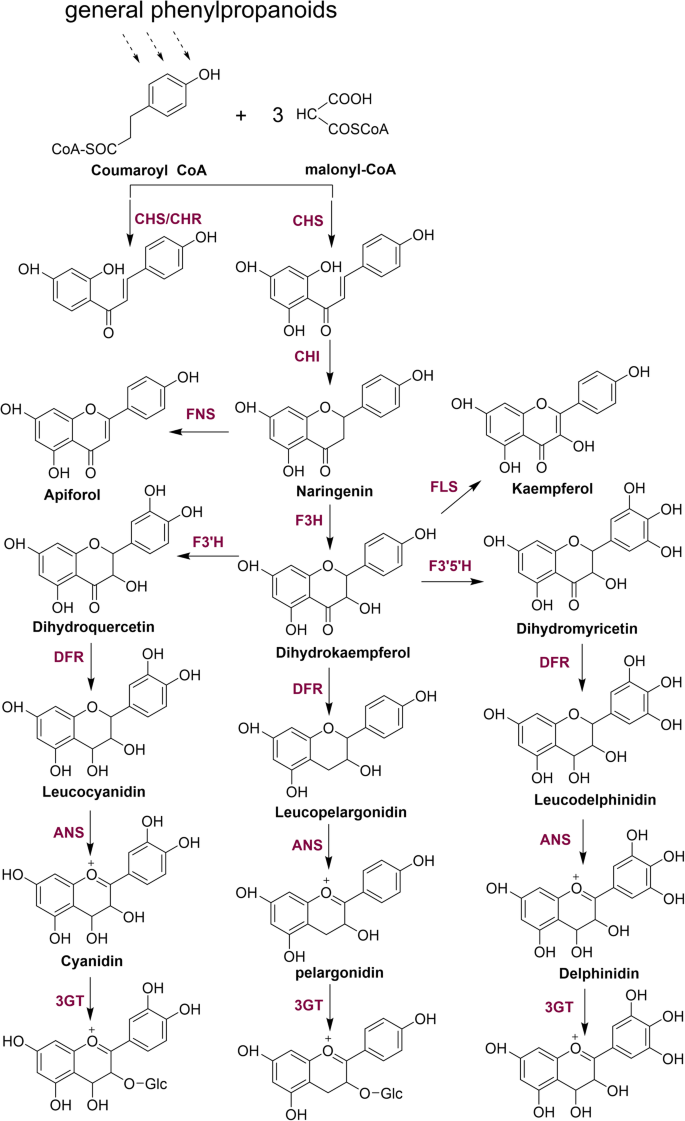
类黄酮是重要的多酚类植物次生代谢物,可分为黄酮、黄酮、黄酮、异黄酮、儿茶素和查尔酮[47].适当摄入类黄酮可减少癌症、心血管疾病、脂质过氧化和骨质疏松症的发生[48].以往的植物化学研究已经揭示了许多类黄酮存在于花r . molle是的[3.,12,13].考虑到黄酮类化合物的多种有益作用,本研究还探索了与黄酮类化合物生物合成相关的转录组unigenesr . molle是的.香马酰-辅酶a和丙二酰-辅酶a是生物合成类黄酮的常见前体,分别来自苯丙类途径和碳水化合物代谢。类黄酮的生物合成是由查尔酮合成酶(CHS)启动的,它生成查尔酮作为重要的中间体,该途径经过几个酶法步骤,形成其他类黄酮,如黄酮和二氢黄酮醇。此外,类黄酮途径的侧分支导致其他类黄酮类的合成,包括异黄酮、黄酮和黄酮醇(图。11).共注释了53个与类黄酮生物合成途径相关的unigenes。从黄酮类化合物生物合成的最初作用酶出发,鉴定了查尔酮合成酶(CHS)、查尔酮异构酶(CHI)和黄酮羟化酶(F3H),它们都能连续催化对香豆油酰辅酶a和丙二酰辅酶a生成重要的中间产物二氢-异丙酚。此外,还鉴定出了将二氢山柰酚转化为二氢槲皮素和二氢杨梅素所必需的黄酮-3 ' -羟基酶和黄酮-3 ',5 ' -羟基酶。参与类黄酮生物合成的主要酶列于表中5.
转录因子的鉴定
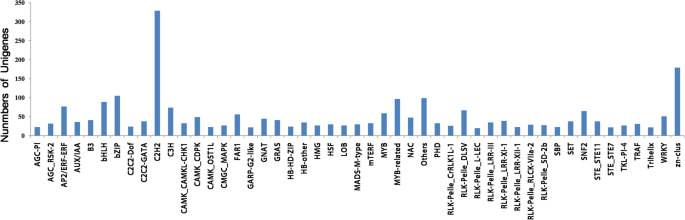
在植物中,转录因子(转录因子)往往在转录水平上调节基因表达,并通过与基因启动子区域的相互作用影响代谢通量。根据我们对已知植物转录因子数据库的Blast X搜索,假设3376r . molle是的至少鉴定出分布在49个TF家族中的转录因子,占组装的unigenes总数的3.35%(图1)。12).其中,C2H2是最丰富的TF家族(329个unigenes, 9.7%),其次是zn-clus(179个,5.3%)和bZIP(105个,3.1%)。C2H2家族成员在植物发育过程中起着至关重要的作用,包括花的器官发生、叶片和侧枝的起始以及种子的发育[49].bZIP调节包括病原体防御和应激信号传递在内的过程[50].本研究还分别鉴定出59个和97个编码MYB和MYB相关tf的unigenes。MYB转录因子在几种植物中调节苯丙类化合物的生物合成,其中主要包括R2R3-MYB转录因子,它们也被证明调节苯丙类化合物生物合成的主要分支即类黄酮代谢途径,包括拟南芥[51),碧桃[52),淫羊藿sagittatum[52].另外,与bHLH、AP2-ERF和WRKY相关的unigenes分别为89、83和51个。这些转录因子在植物的整个生命周期中发挥着不同的作用,从调节几个发育过程到对环境胁迫的响应[53,54,55].此外,它们对植物的次生代谢尤为重要。例如,bHLH tf调节植物中的类黄酮生物合成途径[56,57].AP2/ERF TF家族成员调节萜类吲哚类生物碱的生物合成基因Catharanthus roseus也叫[58].对这些转录因子的进一步研究可能会为植物次生代谢物生物合成的调控网络提供一个清晰的轮廓r . molle是的.
SSRs的识别
简单序列重复序列(SSRs)也被称为微卫星,是一到六个碱基对的短DNA基序的串联重复序列。它们广泛分布于真核生物(例如植物、动物和真菌)以及一些原核生物中[59].SSRs通常与表型变异相关,表型变异已成为应用最广泛的信息性分子标记,有利于各种应用,包括植物的遗传育种,基因定位和遗传标记辅助选择[60].为了识别SSRs,需要将两者的所有unigenes组装起来r . molle是的花和根采用MISA分析。从7799个单基因中共鉴定出10,828个SSRs,其中单核苷酸重复序列(4868,44.95%)最多,其次是二核苷酸重复序列(3872,35.75%)和三核苷酸重复序列(1292,11.93%),六核苷酸重复序列(6,0.055%)最少。另外,有66个(6.1%)四核苷酸SSR和14个(0.13%)五核苷酸SSR8:图S3)。一些SSR基序与编码萜类生物合成酶的unigenes (unigenes)有关。HMGR, dx)(表6).这些ssr位点可为进一步分析植物遗传多样性提供依据r . molle是的以及相关物种。
讨论
r . molle是的是一种著名的中国传统药用植物,过去几年对其天然产物进行了广泛的研究[5,6,7].它含有丰富的次生代谢产物,包括萜类、黄酮类、木酚素等,具有镇痛、抗氧化、钠通道调节等广泛的生物活性[7,61].特别是灰烷二萜因其独特的镇痛活性而引起了研究者的广泛关注。然而,包括灰烷二萜在内的许多参与这些复杂化合物生物合成途径的假定基因还不清楚。最近,肖等人。21]对分别从花的四个发育阶段提取的混合RNA进行了转录组测序分析r . molle是的.共生成约2000万个clean reads,并组装成66026个unigenes,平均长度为698 bp,其中31542个unigenes在公共数据库中被注释,共预测了55456个CDSs。然而,作者主要研究了类胡萝卜素的开花和花色形成机制,特别关注了类胡萝卜素生物合成途径中涉及的关键酶。在本研究中,我们对植物的根和花进行了高通量的转录组测序r . molle是的.共组装了100603个unigenes,平均长度为778 bp, N50为1384 bp,表明组装完整性合格,可用于后续分析。其中,有57,416个unigenes在公共数据库中被注释。然而,42.9%的unigenes仍未注释,这表明已发表的植物转录组和基因组数据由于相对种数较少而有限r . molle是的测序。这些unigenes可能与特定次生代谢产物的生物合成有关r . molle是的.此外,预测的CDSs共76,198个,占unigenes总数的75.7%,这有利于进一步分析unigene在蛋白质水平上的功能,促进关键基因的研究。与Xiao等人的研究相比,我们的研究提供了更多高质量的clean reads、注释unigenes和预测CDSs。
利用基因本体赋值程序对标注的unigenes进行功能分类,共获得37108个unigenes GO术语。在很多情况下,同一个unigene被分配了几个术语,因此,25,634个unigenes被分配给细胞成分,27,218个unigenes被分配给生物过程,29,126个unigenes被分配给分子功能。其余的unigenes仍然没有注释,这可能是由于相对较短的序列无法覆盖保守的蛋白质结构域。在分子功能范畴内,绝大多数unigenes与“代谢过程”有关,这与在产生的丰富次生代谢物相一致r . molle是的.通过将unigenes定位到KEGG通路上,发现有232个unigenes富集在苯丙烷类生物合成通路上,这是所有次生代谢通路中最大的聚类。这一结果表明,苯丙烷类生物合成过程是一个蓄藏库,为其他次生代谢产物的生物合成提供中间体r . molle是的.我们的研究还发现了53个与类黄酮生物合成相关的unigenes。这些注释和分类为研究特定的途径提供了资源r . molle是的.
许多unigenes参与萜类主干生物合成,包括编码众所周知的酶AACT、HMGR、DXR、MCS、IPPI、FPPS和GPPS的unigenes。对其他unigenes的进一步表征将提高我们对萜类生物合成的分子机制的理解。TPS被认为是萜类生物合成生物过程中第一个参与的酶,它可以将常见的二磷酸异戊烯基前体(GPP, C-10 FPP, C-15和GGPP, C-20)初始环化形成萜类支架。我们挖掘了转录组r . molle是的花、根和一组TPSs,包括三二萜合成酶(diTPSs)。在这3个ditps中,RmTPS6和RmTPS7被注释为共聚二磷酸合成酶,RmTPS8被注释为kaurene合成酶,这可能与kaurene的生物合成有关,而kaurene被认为是grayanane二萜的生物遗传学前体。进一步分析发现,RmTPS7只在根中表达,而RmTPS6和RmTPS8在花和根中都表达较多。这表明RmTPS6和RmTPS8更可能参与了灰色素的生物合成途径,这与这些萜类化合物在花和根样品中的分布一致。这些酶的功能有待进一步研究。此外,由于灰烷二萜的分布广泛,可能很难区分两种组织中差异表达的生物合成基因。因此,可能需要组织培养结合茉莉酸甲酯诱导法,结合多层次组学进一步分析grayanoids的生物合成途径。
从其结构的复杂性来看,其中大部分都具有较高的氧合性,极有可能被CYPs催化。为了探索萜烯骨架修饰的分子基础,从转录组中鉴定出61个可能编码CYPs的unigenes,其中4个和5个unigenes分别属于CYP71和CYP76家族。这两个家族被认为是植物二萜多样性的主要驱动力,这使得来自这两个家族的unigenes成为灰色烷类生物合成的首选候选基因。然而,由于TPS和CYP家族内部的功能可塑性,序列相似性不能保证特定的功能。因此,对这些候选TPS和CYP进行准确的功能注释仍然需要生物化学方法。
此外,我们的研究还基于FPKM值检测了花和根之间差异表达的unigenes。结果表明,许多unigenes分别在花和根组织中表达,且表达水平不同。进一步研究这些DEGs与代谢物的结合将使我们更清楚地了解次生代谢物的生物合成过程。此外,转录组也是发现SSRs的宝贵资源。在本研究中,共鉴定出10,828个SSRs,略多于Xiao的研究中发现的SSRs(8266个)[21].两项研究的分类SSRs的分布和频率也不同。在我们的研究中,最丰富的SSRs是单核苷酸重复序列(44.95%),其次是双核苷酸重复序列(35.75%)。相比之下,Xiao等人报道双核苷酸重复基序(56.15%)是最常见的重复基序r . molle是的转录组(21].这两项研究的结合将有助于栽培品种指纹图谱的鉴定和理想基因型的选择r . molle是的育种和基因操纵r . molle是的.转录因子通过调节基因表达来影响代谢通量。在这项工作中,共鉴定了3376个tf,包括bHLH(89)、AP2/ERF(83)、MYB(59)、MYB相关(97)和WRKY(51)家族。tf的数量r . molle是的是否高于著名的生产二萜的药用植物美国miltiorrhiza(1948 TFs)和模范工厂答:芥(2357 TFs),表明其存在复杂的代谢调节网络r . molle是的.这些转录因子可能在调节次生代谢产物的生物合成中发挥重要作用。在青蒿,bHLH转录因子AabHLH1有效调控青蒿素的生物合成[62),在Catharanthus roseus也叫, ORCA2和ORCA3的AP2/ERF成员可与狭窄苷合成酶(STR)的启动子结合,调节萜类吲哚生物碱代谢[58].利用基因工程方法控制转录因子对调控萜类生物合成具有重要意义r . molle是的.对这些代谢因子的进一步研究将有助于调控代谢途径,最终提高具有药用价值的次生代谢产物的产量r . molle是的.
结论
在本研究中,我们提出了从头转录组测序R.molle花和根。共生成100603个unigenes,公共数据库注释了57,416个unigenes。KEGG数据库鉴定出17960个unigenes,涉及133种不同的植物代谢途径。我们专注于寻找参与灰色类生物合成的候选基因,在17906个unigenes中,102个unigenes参与萜类骨干生物合成。通过BLAST相似性搜索,鉴定出9个萜类合成酶,包括3个二萜合成酶。共发现61种CYP酶,其中9种CYP来自CYP71和CYP76家族。本研究的转录组信息还揭示了木酚素、类黄酮的生物合成途径中涉及的各种基因。此外,我们的研究还发现了一些与次生代谢产物生物合成相关的转录因子,并从我们的转录组数据库中发现了10,828个SSRs,这些SSRs有可能用于遗传操作r . molle是的.综上所述,本研究获得的转录组数据将有助于发现参与特定次生代谢途径的新基因,并为通过代谢工程提高植物或微生物宿主体内有价值代谢物的产量提供依据。此外,这也为未来该药用植物的功能和比较基因组研究铺平了道路。
数据和材料的可用性
所有样本测序结果的原始数据可在NCBI生物项目数据库中获得,登录号为PRJNA565490 (https://www.ncbi.nlm.nih.gov/bioproject/?词= prjna565490).该转录组霰弹枪组装项目已存放在GenBank,注册号为GIKT00000000。本文描述的版本是第一个版本GIKT01000000 (https://www.ncbi.nlm.nih.gov/ nuccore / GIKT00000000).所有支持数据都包含在附加文件中。
缩写
- AACT:
-
乙酰辅酶a乙酰转移酶
- CCR:
-
Cinnamoyl-CoA还原酶
- cd:
-
编码序列
- 气:
-
查耳酮异构酶
- CHS:
-
查耳酮合酶
- 肤色线:
-
4-coumarate——辅酶a连接酶
- CMS:
-
2-C-methyl-D-erythritol 4-phosphate cytidylyltransferase
- 齿轮:
-
正交基团的簇
- CYP:
-
细胞色素P450酶
- 度:
-
不同表达基因
- DMAPP:
-
Dimethylallyl焦磷酸
- DXR:
-
1-deoxy-D-xylulose-5-phosphate reductoisomerase
- dx:
-
1-deoxy-D-xylulose-5-phosphate合酶
- FPKM:
-
每千碱基转录片段每百万映射读取
- FPP:
-
通过二磷酸
- FPPS:
-
通过二磷酸合酶
- GGPP:
-
Geranylgeranyl二磷酸
- GGPPS:
-
Geranylgeranyl二磷酸合酶
- 走:
-
基因本体论
- GPP:
-
香叶二磷酸
- HDR:
-
4-hydroxy-3-methylbut-2-enyl二磷酸还原酶
- HDS:
-
4-hydroxy-3-methylbut-2-enyl二磷酸合酶
- HMGR:
-
Hydroxymethylglutaryl-CoA还原酶
- 邮政编码:
-
Hydroxymethylglutaryl-CoA合酶
- IPP:
-
Isopentenyl二磷酸
- IPPI:
-
Isopentenyl二磷酸异构酶
- KEGG:
-
京都基因和基因组百科全书
- KOG:
-
真核直接同源组
- 主持人:
-
2, 4-cyclodiphosphate合酶
- 议员:
-
Methylerythritol磷酸
- 可:
-
甲羟戊酸激酶
- MVA:
-
甲羟戊酸
- NJ:
-
Neighbor-Joining
- 门店:
-
新一代测序
- Nr:
-
NCBI冗余蛋白质
- 朋友:
-
苯丙氨酸氨裂解酶
- 包含了:
-
蛋白家族
- PlnTFDB:
-
植物转录因子数据库
- 两家公司:
-
Phosphomevalonate激酶
- TFs:
-
转录因子
- TPS:
-
萜烯合成酶
- 苏维埃社会主义共和国:
-
简单序列重复
参考文献
- 1.
朱镕基y p。中药材:化学、药理学及应用。博卡拉顿:CRC印刷机;1998.
- 2.
陈刚,金辉,李霞。杜鹃花科植物的化学和药理研究进展[J]。中国药学实践杂志2008;26(4):255-7。
- 3.
杜鹃花(Ericaceae):植物化学、药理和毒理学研究。中华化学杂志2018;16(6):401-10。
- 4.
福特C,克罗WE。《中药注释》;2006.
- 5.
郭家佳,胡爱梅,赵顺峰,久保一。杜鹃花中灰状二萜类昆虫抗食剂及杀虫剂的研究。植物化学。1991;30(6):1797 - 800。
- 6.
周淑珍,姚硕,唐超,柯超,李亮,林刚,等。杜鹃花中的二萜。中国生物医学工程学报(英文版);2014;
- 7.
李勇,刘亚波,张建军,刘勇,马韶光,曲静,等。从根中提取的抗感受器杜鹃molle是的.中国生物医学工程学报,2015;38(12):1391 - 1391。
- 8.
钟刚,胡敏,魏旭,翁强,谢军,刘杰,等。杜鹃花中的格雷烷二萜类化合物对一种夜蛾细胞系具有细胞毒活性。中国生物医学工程学报2005;68(6):924-6。
- 9.
李勇,刘永波,张建军,李永华,蒋建东,余少生,等。从杜鹃花(Rhododendron molle)根中分离出具有1,10,2,3 -异生态灰烷骨架的二萜。Org。2013;15(12):3074 - 7。
- 10.
张志荣,钟建东,李红梅,李红志,李荣涛,邓学林。从花中提取的两个新的灰烷二萜杜鹃molle是的.中国植物学报,2012;14(8):764-8。
- 11.
从“疯狂的蜂蜜”到降压药,灰色二萜。Sci学监。2016;99(3):327 - 34。
- 12.
王松,杨勇,石娟。杜鹃花蕾的二氢查尔酮[J]。中草药。2005;36(1):21-3。
- 13.
王旭,胡勇,袁东,付华。杜鹃花的化学成分。中国药学杂志2014;23(2):94-8。
- 14.
支旭,肖亮,梁山,易峰,阮克峰。化学成分的杜鹃molle是的.化学通报,2013;49(3):454-6。
- 15.
Shahin A, van Kaauwen M, Esselink D, Bargsten JW, van Tuyl JM, Visser RG,等。百合和郁金香超大型基因组表达序列标签的生成与分析。BMC基因组学。2012;13(1):640。
- 16.
李燕,罗红梅,孙超,宋金燕,孙永忠,吴强,等。EST分析揭示了可能参与甘草酸苷生物合成的基因。BMC基因组学。2010;11(1):268。
- 17.
Guzman F, Kulcheski FR, Turchetto-Zolet AC, Margis R. De novo单叶Eugenia L.转录组的组装和萜类生物合成途径基因的鉴定。植物科学。2014;229:238-46。
- 18.
吕林,肖燕,裴松,温婷,尚勤。基于illumina的红花花从头转录组测序与分析。《公共科学图书馆•综合》。2012;7 (6):e38653。
- 19.
肖明,张颖,陈旭,李e - j, Barber CJ, Chakrabarty R,等。基于下一代测序的非模式植物的转录组分析,产生具有生物技术意义的专门代谢产物。生物科技J》。2013;166(3):122 - 34。
- 20.
哈格尔JM,莫里斯JS,李e - j, Desgagné-Penix I,布罗斯CD,张磊,等。20种分类相关的苄基异喹啉生物碱生产植物的转录组分析。植物生物学杂志,2015;15(1):227。
- 21.
肖铮,苏静,孙旭,李超,何亮,程硕,等。利用Illumina测序技术对杜鹃花的转录组进行从头分析。基因组的基因。40 2018;(6): 591 - 601。
- 22.
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I,等。没有参考基因组的RNA-Seq数据的全长转录组组装。生物科技Nat》。2011;29(7):644。
- 23.
Pertea G,黄X,梁峰,Antonescu V, Sultana R, Karamycheva S,等。TIGR基因指数聚类工具(TGICL):一种用于快速聚类大型EST数据集的软件系统。生物信息学。2003;19(5):651 - 2。
- 24.
李伟,Jaroszewski L, Godzik A.容忍一些冗余显著加速大型蛋白质数据库的聚类。生物信息学,2002;18(1):77 - 82。
- 25.
2000年SWISS-PROT蛋白序列数据库及其补充数据库TrEMBL。核酸Res. 2000;28(1): 45-8。
- 26.
Tatusov RL, Fedorova ND, Jackson JD, Jacobs AR, Kiryutin B, Koonin EV等。COG数据库:更新版本包括真核生物。BMC生物信息学。2003;4(1):41。
- 27.
Huerta-Cepas J, Szklarczyk D, Forslund K, Cook H, Heller D, Walter MC等。eggNOG 4.5:一个具有改进的真核、原核和病毒序列功能注释的分级orthology框架。核酸学报2015;44(D1): D286-D93。
- 28.
贝特曼A,科因L,德宾R,芬恩RD,霍利希V,格里菲斯-琼斯S,等。Pfam蛋白家族数据库。核酸学报2004;32(suppl_1): D138-D41。
- 29.
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM,等。基因本体论:统一生物学的工具。Nat麝猫。2000;25(1):25。
- 30.
Conesa A, Götz S, García-Gómez JM, Terol J, Talón M, Robles M. Blast2GO:功能基因组学研究中注释、可视化和分析的通用工具。生物信息学。2005;21(18):3674 - 6。
- 31.
Kanehisa M, Goto S, Kawashima S, Okuno Y, Hattori M.解码基因组的KEGG资源。核酸学报2004;32(suppl_1): D277-D80。
- 32.
谢超,毛旭,黄娟,丁勇,吴俊,董松,等。KOBAS 2.0:用于注释和识别丰富途径和疾病的web服务器。核酸学报,2011;39(suppl_2): W316-W22。
- 33.
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ,等。RNA-Seq的转录本组装和丰度估计揭示了数千个新的转录本和异构体之间的转换。生物科技Nat》。2010;28(5):511。
- 34.
杜威中国出版社。RSEM:从有或没有参考基因组的RNA-Seq数据中精确的转录物定量。BMC生物信息学。2011;12(1):323。
- 35.
层JD。阳性错误发现率:贝叶斯解释和q值。安Stat。2003;31(6):2013 - 35。
- 36.
Riaño-Pachón DM, Ruzicic S, Dreyer I, muller - roeber B. PlnTFDB:植物综合转录因子数据库。BMC生物信息学。2007;8(1):42。
- 37.
Kumar S, Stecher G, Tamura K. MEGA7:用于更大数据集的分子进化遗传学分析7.0版本。Mol Biol evl 2016;33(7): 1870-4。
- 38.
Livak KJ, Schmittgen TD。使用实时定量PCR和2−ΔΔCT方法分析相关基因表达数据。方法。2001;25(4):402 - 8。
- 39.
Vranová E, Coman D, Gruissem W.合成类异戊二烯的MVA和MEP途径的网络分析。植物生物学,2013;64:665-700。
- 40.
程亚安,娄毅刚,毛亚波,吕松,王立军,陈晓燕。植物萜类:生物合成与生态功能。植物生物学杂志,2007;49(2):179-86。
- 41.
Dudareva N, Andersson S, Orlova I, Gatto N, Reichelt M, Rhodes D,等。非甲戊酸途径同时支持金鱼花中单萜和倍半萜的形成。中国生物医学工程学报,2005;32(3):447 - 447。
- 42.
Hemmerlin A, Hoeffler J-F, Meyer O, Tritsch D, Kagan IA, Grosdemange-Billiard C,等。烟草亮黄-2细胞中胞质甲戊酸盐与质体甲基赤藓糖醇磷酸通路的交联。生物化学杂志,2003;278(29):26666-76。
- 43.
pataki I, Heskes AM, Hamberger B.用于萜烯功能化和代谢工程的细胞色素P450。见:类异戊二烯生物技术。可汗:施普林格;2015.107 - 39页。
- 44.
植物P450s作为种特异性化学多样性进化的万能驱动因子。中国生物医学工程学报(英文版)2013;
- 45.
细胞色素P450酶:植物二萜多样性的驱动力。植物化学,2019;161:149 - 62。
- 46.
苯丙烷生物合成。摩尔。2010;3(1):2 - 20。
- 47.
Schijlen EG, De Vos CR, van Tunen AJ, Bovy AG。作物类黄酮生物合成的修饰。植物化学。2004;65(19):2631 - 48。
- 48.
Nijveldt RJ, Van Nood E, Van Hoorn DE, Boelens PG, Van Norren K, Van Leeuwen PA。类黄酮:可能的作用机制和潜在的应用综述。中华儿科杂志2001;74(4):418-25。
- 49.
吉田宁,柳井勇,陈磊,加藤勇,平冢佳,美和田,等。胚胎花2是一个新的多梳族蛋白同源体,在拟南芥中介导芽的发育和开花。植物细胞。2001;13(11):2471 - 81。
- 50.
Jakoby M, Weisshaar B, Dröge-Laser W, Vicente-Carbajosa J, Tiedemann J, Kroj T,等。拟南芥bZIP转录因子。植物科学进展2002;7(3):106-11。
- 51.
Mehrtens F, Kranz H, Bednarek P, Weisshaar B.拟南芥转录因子MYB12是一种黄酮醇特异性苯丙类生物合成调控因子。植物杂志。2005;138(2):1083 - 96。
- 52.
黄伟,孙伟,吕辉,罗敏,曾森,Pattanaik S,等。淫羊藿R2R3-MYB转录因子调控类黄酮生物合成途径。《公共科学图书馆•综合》。2013;8 (8):e70778。
- 53.
米井勇,王晓燕,王晓燕。AP2/ERF家族转录因子在植物非生物胁迫应答中的作用。生物化学生物物理学报2012;1819(2):86-96。
- 54.
王晓东,陈晓东,陈晓东,等。AP2/ERF转录因子对植物抗病途径的调控。植物生物学杂志2004;7(4):465-71。
- 55.
沈启军,沈启军。WRKY转录因子。植物学报,2010;15(5):247-58。
- 56.
戴维斯KM,施文KE。次生代谢的转录调控。植物功能生物学,2003;30(9):913-25。
- 57.
徐伟,杜波。MYB-bHLH-WDR复合物对黄酮类生物合成的转录控制。植物学报,2015;20(3):176-85。
- 58.
茉莉酸诱导的AP2/ erf域转录因子ORCA3通过与茉莉酸响应启动子元件的相互作用激活基因表达。植物j . 2001; 25(1):利润率达到。
- 59.
Morgante M, Hanafey M, Powell W.微卫星在植物基因组中优先与非重复性DNA相关。Nat麝猫。2002;30(2):194。
- 60.
微卫星标记在面包小麦遗传分析和植物育种中的开发和应用。Euphytica。2000;113(3):163 - 85。
- 61.
强艳,周波,高凯。杜鹃属植物的化学成分。化学Biodivers。2011;8(5):792 - 815。
- 62.
季勇,肖杰,沈勇,马东,李铮,蒲刚,等。黄花蒿中正调控青蒿素合成的bHLH转录因子AabHLH1的克隆与鉴定。植物细胞学报,2014;55(9):1592-604。
确认
我们感谢博士们。李勇和卢志彪,感谢他们的帮助杜鹃molle是的.我们还要感谢宋敬远教授为我们提供了栽培的温室r . molle是的.
资金
本研究得到国家自然科学基金项目(No.81874299)资助;药物创新重大项目(批准号:2018ZX09711001-006-001);中国科学院医学科学创新基金(CIFMS)(批准号:2017-I2M-2-004;国家重点研发计划项目(批准号:2018YFA0901900)。
资助机构没有参与研究的设计或数据的收集、分析和解释,也没有参与撰写手稿。
作者信息
从属关系
贡献
GL Z设计并实施了本研究并起草了手稿。P Z构思、设计和监督了这项研究并修改了手稿。所有作者都认可了最终稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1:表S1。
从GenBank和swissprot提取的用于系统发育分析的蛋白质序列的登录数。
附加文件2:表S2。
TPS和CYP序列来源于GenBank,用于建立自定义数据库。
附加文件3:表S3。
qPCR引物。
附加文件4:图S1。
(a)组合unigenes的长度分布。(b)预测cds的长度分布。
附加文件5:图S2。
组合unigenes对NR数据库的同源搜索特征。(a) Blast点击率最高的e值分布。(b) unigenes的相似度分布。(c)爆炸命中的种类分布。
补充文件6:表S4。
DEGs参与次生代谢物的生物合成途径。
附加文件7:表S5。
中TPS和CYP候选体的RNA-Seq数据r . molle是的.
附加文件8:图S3。
SSR密度分布图。x轴为SSR类型,y轴为每Mb序列的SSR数量。SSR类型(p1:单核苷酸重复基序p2:二核苷酸重复基序p3:三核苷酸重复基序p4:四核苷酸重复基序p5:五核苷酸重复基序p6:六核苷酸重复基序c:复合体重复基序c*:两个共有部分重复的SSR序列。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
周桂华,朱培平,周桂华,等杜鹃molle是的并鉴定参与次生代谢产物生物合成的基因。BMC植物杂志20.414(2020)。https://doi.org/10.1186/s12870-020-02586-y
收到了:
接受:
发表:
关键字
- 杜鹃molle是的转录组新生组合次生代谢物生物合成