- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
大豆种子重数量性状核苷酸和候选基因的全基因组关联研究gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba20.gydF4y2Ba、物品编号:gydF4y2Ba404gydF4y2Ba(gydF4y2Ba2020gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
种子重是一种复杂的产量相关性状,通过连锁作图研究报道了大量的数量性状位点。由于诸多限制,将QTL从连锁定位整合到育种计划中具有挑战性,因此,全基因组关联研究(GWAS)由于在非相关个体中具有更高的分辨率和多样化的遗传多样性,提供了更精确的QTL定位。gydF4y2Ba
结果gydF4y2Ba
本研究利用573个选育群体的61166个单核苷酸多态性(snp),鉴定了中国夏播大豆的数量性状核苷酸(QTNs)和种子重候选基因。GWAS采用2个单位点模型(SLMs)和6个多位点模型(MLMs)。2个SLMs检测到39个snp, 6个MLMs检测到209个snp。在YHSBLP中,共发现231个qtn与种子重量相关,并具有不同的影响。其中,70个snp在8条染色体上同时被SLMs和MLMs检测到。通过连锁/关联图谱研究,94个QTN与先前报道的QTL/QTN共定位。共预测了36个候选基因。在这些候选基因中,有四个中心基因(gydF4y2BaGlyma06g44510, Glyma08g06420, Glyma12g33280gydF4y2Ba和gydF4y2BaGlyma19g28070gydF4y2Ba)通过共表达网络的整合来识别。其中,3种高HSW基因型在R5期的表达量相对高于低HSW基因型gydF4y2BaGlyma12g33280gydF4y2Ba。结果表明,利用更多的模型,特别是MLMs,可以有效地发现重要的QTNs,鉴定出的HSW QTNs/基因可用于大豆种子质量和产量的分子育种工作。gydF4y2Ba
结论gydF4y2Ba
应用2个单位点加6个多位点GWAS模型鉴定出231个qtn。四个中心基因(gydF4y2BaGlyma06g44510gydF4y2Ba,gydF4y2BaGlyma08g06420gydF4y2Ba,gydF4y2BaGlyma12g33280gydF4y2Ba&gydF4y2BaGlyma19g28070gydF4y2Ba)通过整合预测候选基因之间的共表达网络来检测。gydF4y2Ba
背景gydF4y2Ba
据估计,到30年后,世界人口将达到1000万。gydF4y2Ba1gydF4y2Ba]随着非生物和生物压力的增加以及用于农业活动的可耕地的减少[gydF4y2Ba2gydF4y2Ba]。这意味着粮食和营养安全受到威胁。豆科作物,包括大豆(gydF4y2Ba大豆gydF4y2BaL. Merr.)在确保全球粮食和营养安全方面发挥着重要作用,此外它们还具有通过固氮改善土壤质量的能力[gydF4y2Ba3.gydF4y2Ba]。食用豆类作物对健康和生理有益,如预防心血管疾病、肥胖、糖尿病、癌症和缓解更年期症状[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba]。然而,中国和许多欠发达国家的大豆产量不足是一个巨大的挑战,大豆单产需要迅速提高。gydF4y2Ba
大豆产量、种子利用和进化适应性的主要决定因素之一是种子重量[gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba]。例如,大种子品种用于煮熟大豆(gydF4y2BanimamegydF4y2Ba)、青豆(gydF4y2Ba毛豆gydF4y2Ba)、豆浆及豆腐(gydF4y2Ba豆腐gydF4y2Ba),而小粒品种则适合制作发酵大豆(gydF4y2Ba奈特ōgydF4y2Ba)及萌芽生产[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba]。大豆育种者需要创造较大的种子重量变化,以选择具有不同最终用途的品种。种子重也是大豆驯化过程中一个重要的目标性状[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba], 100粒重的范围从小于1粒不等gydF4y2BaggydF4y2Ba在野生大豆(gydF4y2Ba甘氨酸大豆gydF4y2Ba摘要。(et Zucc.)加入的公司超过60家gydF4y2BaggydF4y2Ba具体来说gydF4y2Ba毛豆gydF4y2Ba品种。gydF4y2Ba
作为一种复杂的数量性状,种子重量被认为是由几个主要基因/基因座和许多影响较小的无法检测的基因座(即多基因)控制的,它们与环境相互作用。大豆种子质量的数量性状位点/核苷酸(QTL/QTNs)在SoyBase (gydF4y2Bawww.soybase.orggydF4y2Ba),通过联动映射。然而,由于较高的置信区间和较少的遗传变异,将连锁图谱的结果整合到育种计划中是具有挑战性的[gydF4y2Ba17gydF4y2Ba]。因此,近年来,标记-性状关联被用于利用自然种群进化历史中基于连锁不平衡(LD)的所有重组事件[gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba]。标记-性状关联使研究人员能够利用自然多样性并在基因组中定位有价值的基因[gydF4y2Ba18gydF4y2Ba]。例如,Miao等人。[gydF4y2Ba20.gydF4y2Ba]最近应用了种子油的区域关联制图,并确定了gydF4y2BaGmSWEET39gydF4y2Ba(gydF4y2BaGlyma.15 g049200 / Glyma15g05470)gydF4y2Ba随后在拟南芥中过度表达,导致种子油含量至少增加10%。gydF4y2Ba
通过20条染色体的全基因组关联研究(GWAS)已经检测和报道了数十个qtn [gydF4y2Ba13gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba]。然而,由于种群类型、规模和GWAS方法的不同,绘制结果会有所不同。单标记全基因组扫描模型如混合线性模型(MLM)和一般线性模型(GLM)是大豆种子重遗传分析中最常用的模型。这些模型存在一定的局限性,包括对显著性阈值的多次检验校正问题,以及映射能力问题[gydF4y2Ba31gydF4y2Ba]。近年来,许多多基因座模型已被开发并应用于包括大豆在内的几种作物的GWAS中。六个这样的模型(mrMLM, FASTmrMLM, FASTmrEMMA, pLARmEB, pKWmEB和ISIS EM-BLASSO)在R中与mrMLM一起实现。GUI包[gydF4y2Ba32gydF4y2Ba]。由于其检测能力和鲁棒性,这些模型已成为识别具有复杂特征的qtn的最先进程序[gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba]。gydF4y2Ba
生物信息学工具提高了鉴定目标QTL潜在基因的方便性。其中一种策略是利用共表达网络,对功能相关基因进行优先排序。它已成功地用于几种作物,如玉米[gydF4y2Ba38gydF4y2Ba],大米[gydF4y2Ba39gydF4y2Ba],花生[gydF4y2Ba40gydF4y2Ba]、拟南芥[gydF4y2Ba41gydF4y2Ba],大豆[gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba]等。通过整合共表达网络分析,鉴定出一类在葡萄发育过程中诱导主要转录组重编程的枢纽基因(hub genes)。[Palumbo等]gydF4y2Ba44gydF4y2Ba]。枢纽基因(高度连接的基因)可能为这些基因在网络中的作用提供线索[gydF4y2Ba45gydF4y2Ba]。gydF4y2Ba
在大多数早期报道的种子重GWAS中,群体大小大多小于500 [gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba]。据报道,群体大小、遗传多样性以及基因组覆盖/ snp数量、连锁不平衡和使用的统计方法都会影响GWAS的能力[gydF4y2Ba17gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba]。因此,本研究利用573个育种品系61166个snp进行标记-性状关联,即2个单位点模型(SLMs)和6个多位点模型(MLMs)来识别显著snp。预测了潜在的候选基因,并通过功能共表达网络的整合鉴定出了其中的枢纽基因。应用多种GWAS模型检测到231个QTNs,其中94个与先前报道的QTL/QTNs共定位。这表明使用多种GWAS模型来解开我们最近开发的多种育种系种子重量的复杂结构。gydF4y2Ba
结果gydF4y2Ba
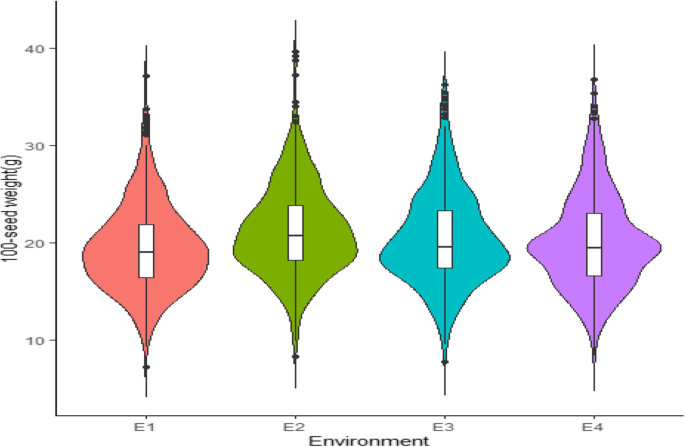
HSW在YHSBLP中的表型变异gydF4y2Ba
在4种环境(E1、E2、E3和E4)中,YHSBLP中HSW的表型变化服从正态分布,具有典型的数量性状特征(图2)。gydF4y2Ba1gydF4y2Ba).E1的HSW范围为7.24 ~ 37.19 g,平均值为19.40±4.47 g, E2、E3和E4的HSW范围(平均值±标准差)分别为8.23 ~ 39.70 g(21.22±4.62 g)、7.71 ~ 36.32 g(20.43±4.80 g)和8.38 ~ 36.78 g(20.09±4.78 g)gydF4y2Ba1gydF4y2Ba:表1)。HSW显著(gydF4y2BaPgydF4y2Ba< 0.001)受基因型、环境和基因型受环境相互作用的影响(表2)gydF4y2Ba1gydF4y2Ba).广义遗传力(gydF4y2BahgydF4y2Ba2gydF4y2Ba)为98.53%。由此可见,YHSBLP夏播时的高通量主要受遗传因素影响,环境因素影响较小(表2)gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
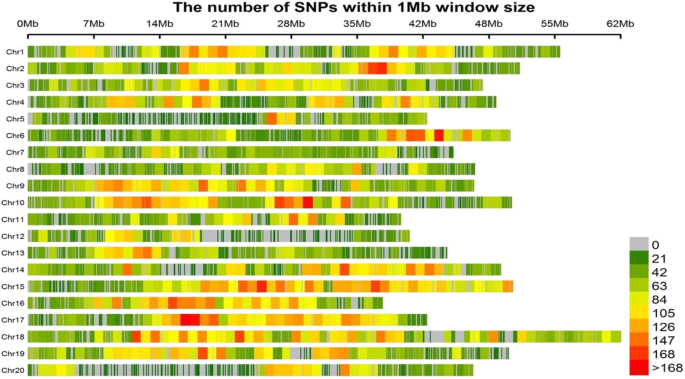
YHSBLP的snp分布与种群结构gydF4y2Ba
采用RAD-seq技术对573份材料进行基因分型。剔除单态标记、MAF < 5%、缺失和杂合等位基因率> 30%的标记后,共使用了61166个snp。该基因组全长9500,068,807 bp (950.07 Mb),占大豆基因组的85.21%。每条染色体的snp数范围为1467 ~ 4844,其中第5号染色体最少,第18号染色体最多。最高SNP密度为91.9 SNP /Mb,最低SNP密度为35 SNP /Mb,分别出现在染色体(Chr.) 15和Chr.05上,最长的染色体为Chr.18。gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
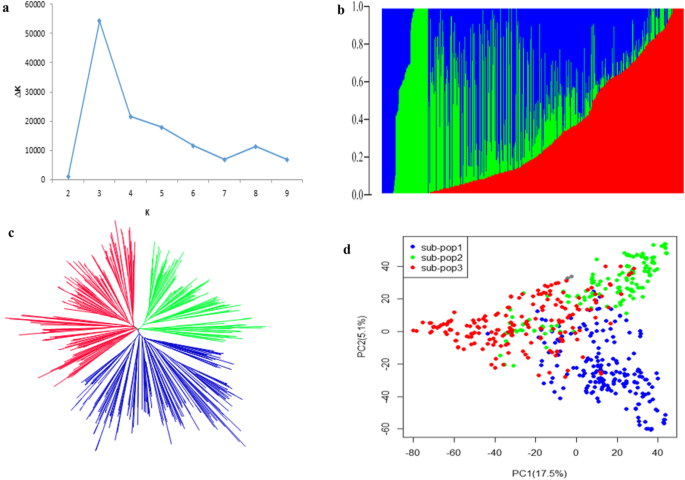
从admix软件获得的种群结构可以看出,573个物种被分为三个亚种群(图3)。gydF4y2Ba3.gydF4y2Baa和b),系统发育分析(图2)。gydF4y2Ba3.gydF4y2Bac)和PCA(图3)。gydF4y2Ba3.gydF4y2Bad)。前两个PC占22.60%的变化(图2)。gydF4y2Ba3.gydF4y2Bad).概率(Q)大于0.70分的线视为纯线,Q≤0.7分的线视为混杂线。亚群1由107个纯种系组成,平均高重为17.68 g。亚群2和亚群3分别由101个和92个纯种系组成,同一序列的平均HSW分别为22.95 g和19.73 g。不同亚群的HSW差异显著。其余273种外加剂的平均HSW为20.52 g。gydF4y2Ba
SNP-trait关联映射gydF4y2Ba
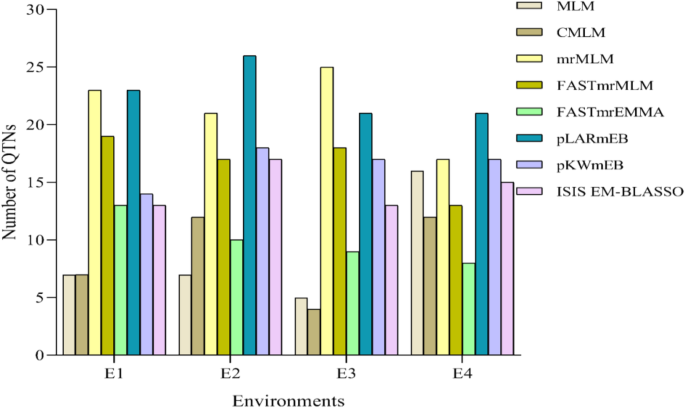
通过MLM (K + Q)和CMLM (K + PCA)分别检测到至少一种环境中与HSW显著相关的39个snp,分别为−gydF4y2Ba日志gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba) = 4.00-12.25(附加文件gydF4y2Ba2gydF4y2Ba:表S2;额外的文件gydF4y2Ba3.gydF4y2Ba:图S1;额外的文件gydF4y2Ba4gydF4y2Ba:图S2)。39个基因座中,24个同时被MLM和CMLM模型检测到(附加文件)gydF4y2Ba2gydF4y2Ba:表S2;额外的文件gydF4y2Ba5gydF4y2Ba:图S3A)。这些qtn不均匀地分布在11条染色体上。每个模型(MLM和CMLM)分别在Chr.02和Chr.13上检测到最多的6个qtn。在这些QTNs中,有7个在两种环境中被5条染色体上的两个slm检测到。14号染色体包含3个qtn (gydF4y2BaqHSW-14-5, qHSW-14-10gydF4y2Ba&gydF4y2BaqHSW-14-11gydF4y2Ba),而在2008年三月(gydF4y2BaqHSW-8-8gydF4y2Ba),第13段(gydF4y2BaqHSW-13-26gydF4y2Ba),第15段(gydF4y2BaqHSW-15-4gydF4y2Ba)及Chr.16 (gydF4y2BaqHSW-16-5gydF4y2Ba)(附加文件gydF4y2Ba2gydF4y2Ba表2)。MLM在环境(E4)中最多检测到16个QTNs,而CMLM在E2或E4中均检测到12个QTNs(图2)。gydF4y2Ba4gydF4y2Ba).大多数检测到的snp与先前发表在SoyBase上的连锁图谱研究和一些关联图谱研究中检测到的QTL重叠gydF4y2Ba2gydF4y2Ba表2)。gydF4y2Ba
6种MLMs即mrMLM、FASTmrMLM、FASTEMMA、pLARmEB、pKWmEB和ISIS EM-BLASSO在所有20条染色体上共检测到209个QTNs,其奇数(gydF4y2BaLODgydF4y2Ba) = 3.01-17.31(附加文件gydF4y2Ba2gydF4y2Ba表2)。mrMLM在Chr.13上检测到最多的13个qtn,而pLARmEB在E2上检测到25个qtn(图2)。gydF4y2Ba4gydF4y2Ba;额外的文件gydF4y2Ba2gydF4y2Ba表2)。6个MLMs检测到的qtn数量为32-75个,其中11-44个qtn被至少两个MLMs同时检测到(附加文件)gydF4y2Ba2gydF4y2Ba表S3)。在6个传销检测到的总共209个qtn中,有39个是在至少两个环境中同时检测到的,其中至少两个模型包括gydF4y2BaqHSW-13-26gydF4y2Ba&gydF4y2BaqHSW-14-10gydF4y2Ba(附加文件gydF4y2Ba2gydF4y2Ba表2)。gydF4y2Ba
2个slm和6个mlm共识别出231个qtn,其中17个qtn被两类模型同时检测到(附加文件)gydF4y2Ba2gydF4y2Ba:表S2;额外的文件gydF4y2Ba5gydF4y2Ba:图S3B)。gydF4y2BaqHSW-2-1gydF4y2Ba,gydF4y2BaqHSW-2-8gydF4y2Ba,gydF4y2BaqHSW-2-10gydF4y2Ba,gydF4y2BaqHSW-9-12gydF4y2Ba,gydF4y2BaqHSW-13-8gydF4y2Ba,gydF4y2BaqHSW-13-26gydF4y2Ba,gydF4y2BaqHSW-14-10gydF4y2Ba,gydF4y2BaqHSW-14-12gydF4y2Ba&gydF4y2BaqHSW-17-4gydF4y2Ba在至少三个环境中被两个以上的传销检测到,以及在一个环境中被至少一个传销检测到(附加文件gydF4y2Ba2gydF4y2Ba表2)。8个模型的两两比较显示QTNs(2-44)同时检测的范围不同(附加文件)gydF4y2Ba5gydF4y2Ba:图S3)。gydF4y2Ba
QTNs对种子重的等位基因效应gydF4y2Ba
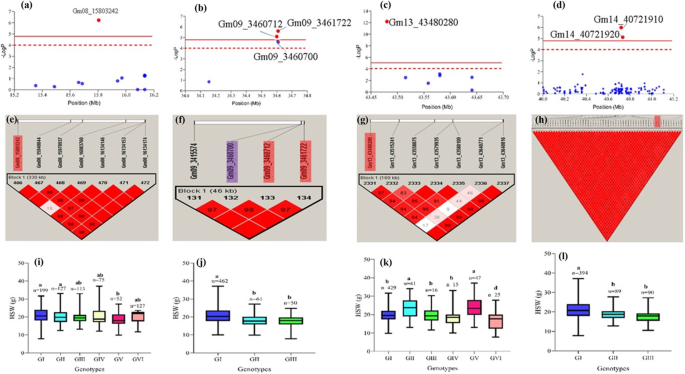
进行单倍型块分析gydF4y2BaqHSW-8-8gydF4y2Ba(Gm08_15803242)gydF4y2Ba, qHSW-9-4gydF4y2Ba(Gm09_3461722gydF4y2Ba), qHSW-13-26gydF4y2Ba(Gm13_43480280)和两个紧密相连的SNPs, Gm14_40721910 (gydF4y2BaqHSW-14-10gydF4y2Ba) & Gm14_40721920 (gydF4y2BaqHSW-14-11gydF4y2Ba)在Haploview软件中采用四配子规则方法(图2)。gydF4y2Ba5gydF4y2Bal)。每个区块内的距离为46 ~ 490 kb,范围为4 ~ 97个snp。573份材料被分成3 ~ 6类,每个区种子重差异显著。gydF4y2Ba
曼哈顿图,所选qtn的单倍型块区分析和等位基因对种子重的影响(箱线图)。gydF4y2BaqHSW-8-8gydF4y2Ba在gydF4y2Ba一个gydF4y2Ba,gydF4y2BaegydF4y2Ba&gydF4y2Ba我gydF4y2Ba。gydF4y2BaqHSW-9-4gydF4y2Ba在gydF4y2BabgydF4y2Ba,gydF4y2BafgydF4y2Ba&gydF4y2Baj。gydF4y2BaqHSW-13-26gydF4y2Ba在gydF4y2BacgydF4y2Ba,gydF4y2BaggydF4y2Ba&gydF4y2BakgydF4y2Ba。gydF4y2BaqHSW-14-10 & qHSW-14-11gydF4y2Ba在gydF4y2BadgydF4y2Ba,gydF4y2BahgydF4y2Ba&gydF4y2BalgydF4y2Ba。在曼哈顿图中显示了每个块中检测到的显著snp (gydF4y2Ba一个gydF4y2Ba,gydF4y2BabgydF4y2Ba&gydF4y2BacgydF4y2Ba),虚线表示简化阈值(4),补全的红线表示Bonferroni校正阈值(4.79)。在每个块中检测到的SNPs分别为红色和紫色,分别超过4.79和4。通过4个环境(E1, E2, E3和E4) (I, J, K和L)的平均种子重得到每个块的箱线图。采用Duncan’s Multiple Range Test对各块进行分组和两两比较gydF4y2Bap < 0.05。gydF4y2Ba具有相同字母的盒子表明种子重量没有显著差异。每个子类的加入数(n)表示在每个框的顶部。GI-GVI表示每个块中的573个条目的若干组gydF4y2Ba
候选基因预测及进一步分析gydF4y2Ba
从至少在两种环境中检测到的显著snp的上游和下游500 kb中挖掘出潜在的候选基因。在至少一个环境中检测到的区域内的snp,只有一个被用来鉴定候选基因。利用拟南芥同源物共鉴定出36个候选基因,其中14个和22个分别位于snp位点的上游和下游gydF4y2Ba6gydF4y2Ba:表S4)。五个SNP各有两个潜在的候选基因,例如,在Gm08_15803242 (gydF4y2BaqHSW-8-8gydF4y2Ba)gydF4y2BaGlyma08g20770gydF4y2Ba和gydF4y2BaGlyma08g20780gydF4y2Ba在38.65和27.86 kb的下游。这两个基因编码ATP结合盒(ABC)转运蛋白,具有转运的生物学功能;跨膜转运已被证明在调节种子大小/重量和影响种子产量方面发挥重要作用[gydF4y2Ba51gydF4y2Ba]。这两个基因一起gydF4y2BaGlyma06g17520, Glyma06g44510, Glyma09g04840, Glyma11g27070, Glyma12g33280, Glyma13g18280gydF4y2Ba和gydF4y2BaGlyma13g17890gydF4y2Ba被标注为参与糖/蔗糖/单糖运输。其余25个基因参与两个或两个以上的生物过程,如细胞增殖、细胞大小调节、细胞壁修饰、花/其部分发育、种子发育、种皮发育和其他在调节种子大小/重量中起关键作用的生物过程gydF4y2Ba6gydF4y2Ba:表S4)。来自Severin等人开发的RNA-Seq图谱。[gydF4y2Ba52gydF4y2Ba],结果发现,所有预测的候选基因除了在种子相关组织和种子发育阶段高度表达外,都在种子相关组织和种子发育阶段高度表达gydF4y2BaGlyma09g24020gydF4y2Ba(附加文件gydF4y2Ba6gydF4y2Ba表5)。gydF4y2Ba
在每个候选基因上游1.5 kb处寻找与种子相关的调控元件。三个seed-relatedgydF4y2Ba独联体gydF4y2Ba-元件(GCN4_motif, MSA-like和RY-element)在13个预测候选基因的启动子区域被鉴定出来(附加文件gydF4y2Ba6gydF4y2Ba表6)。参与胚乳表达的GCN4_motif [TGAGTCA]在gydF4y2BaGlyma01g38450, Glyma04g06760, Glyma13g17890, Glyma15g17040, Glyma15g39730gydF4y2Ba和gydF4y2BaGlyma19g28070gydF4y2Ba[gydF4y2Ba53gydF4y2Ba]。发现参与细胞周期调控的MSA-like [(T/C)C(T/C)AACGG(T/C)(T/C)A]元件gydF4y2BaGlyma02g07240, Glyma09g04840gydF4y2Ba和gydF4y2BaGlyma14g11930gydF4y2Ba[gydF4y2Ba54gydF4y2Ba]。RY-element [CATGCATG]参与种子特异性调控gydF4y2BaGlyma03g06420, Glyma03g06600, Glyma06g17520gydF4y2Ba和gydF4y2BaGlyma12g33280gydF4y2Ba[gydF4y2Ba55gydF4y2Ba]。植物的生长发育受昼夜节律相关基因的调控,尤其是花期调控。此外,在gydF4y2BaGlyma01g38450, Glyma04g06760, Glyma06g44510, Glyma07g11550, Glyma08g20780gydF4y2Ba和gydF4y2BaGlyma13g17890gydF4y2Ba。Hudson [gydF4y2Ba56gydF4y2Ba证明了生物钟控制着大豆种子发育的转录组。预测的候选基因中有20个含有ABRE元件,该元件参与脱落酸响应,据报道,脱落酸响应在种子成熟中起主要作用[gydF4y2Ba57gydF4y2Ba]。gydF4y2Ba
为了了解36个候选基因之间可能的相互作用,同时挖掘其他基因,预测的候选基因受到SoyNet的影响,该网站拥有40,182个大豆基因(占编码基因组的73%),在大豆中有200万个功能网络[gydF4y2Ba58gydF4y2Ba]。在分布在20条染色体上的213个基因之间发现了一个密集的相互作用网络,其中包括本研究预测的15个基因(附加文件)gydF4y2Ba7gydF4y2Ba:图s4a和b)。四个中心基因(gydF4y2BaGlyma06g44510, Glyma08g06420, Glyma12g33280gydF4y2Ba和gydF4y2BaGlyma19g28070gydF4y2Ba)是预测的候选基因。通过qRT-PCR鉴定了7个极端基因型,分别为5个高HSW基因型(P048、P130、P227、P589和P602)和2个低HSW基因型(P415和P579)。除了…gydF4y2BaGlyma12g33280,gydF4y2Ba剩下的三个中心基因(gydF4y2BaGlyma06g44510, Glyma08g06420gydF4y2Ba和gydF4y2BaGlyma19g28070gydF4y2Ba)在R5期高HSW基因型中相对于低HSW基因型表达较高(图2)。gydF4y2Ba6gydF4y2Ba).但在R7期表达不一致。gydF4y2Ba
通过qRT-PCR分析选择基因型的种子重量和4-hub基因的相对表达量。gydF4y2Ba一个gydF4y2Ba&gydF4y2BabgydF4y2Ba所选基因型的qRT-PCR表型分析。的相对表达gydF4y2BaGlyma06g44510gydF4y2Ba(gydF4y2BacgydF4y2Ba)gydF4y2Ba, Glyma08g06420gydF4y2Ba(gydF4y2BadgydF4y2Ba)gydF4y2Ba, Glyma12g33280gydF4y2Ba(gydF4y2BaegydF4y2Ba),gydF4y2BaGlyma19g28070gydF4y2Ba(gydF4y2BafgydF4y2Ba),在种子发育的R5和R7阶段进行qRT-PCR。(LHSW和HHSW分别代表低HSW和高HSW。误差条表示均值的标准误差)gydF4y2Ba
讨论gydF4y2Ba
YHSBLP种子重的表型变异及遗传基础gydF4y2Ba
鉴定与目标性状相关的分子标记是分子育种的先决条件之一。大豆种子重作为产量组成部分,是直接影响产量的最关键性状之一,作为品质属性及其对种子利用的影响具有重要意义[j]。gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba]。种子重(HSW)最终决定了大豆生产的经济回报。然而,该性状是一个复杂的性状,由许多多基因控制,具有主要和次要效应,也受环境和基因型的显著影响。这使得仅根据表型进行筛选非常困难和低效。因此,确定QTL/ qtn用于种子重的标记辅助育种将是有益的。gydF4y2Ba
大豆的高分子量是一种典型的易受基因型、环境或基因型与环境相互作用(G × E)影响的数量性状,这与早期的一些研究结果一致[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba46gydF4y2Ba]。YHSBLP有很大的变异性(7.24-39.70)gydF4y2BaggydF4y2Ba)(附加文件gydF4y2Ba1gydF4y2Ba:表S1),加上更大的群体规模和高snp,通过snp -性状关联提高了QTN检测的有效性和效率[gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,gydF4y2Ba59gydF4y2Ba]。HSW在各环境(E1、E2、E3和E4)中呈正态分布(图2)。gydF4y2Ba1gydF4y2Ba)加上高gydF4y2BahgydF4y2Ba2gydF4y2Ba这表明HSW的变异是由多个遗传位点控制的,影响有大有小(表gydF4y2Ba1gydF4y2Ba).本研究利用了573个新开发的育种系群体(YHSBLP),而大多数早期的研究使用了野生加入、地方品种和优良品种[gydF4y2Ba24gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba46gydF4y2Ba]。高遗传变异性表明,YHSBLP具有针对种子重量进行遗传改良的潜力[gydF4y2Ba60gydF4y2Ba]。gydF4y2Ba
单位点和多位点模型检测的qtn及其比较gydF4y2Ba
大多数先前报道的QTL/ qtn是在< 500个加入和snp < 60,000个的情况下进行的[gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba]。GWAS的检测能力受到群体规模、遗传多样性、基因组覆盖率/ snp数量、连锁不平衡和统计模型的限制[gydF4y2Ba17gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba]。因此,本研究使用两个slm (MLM和CMLM)和六个MLM (mrMLM、FASTmrMLM、FASTmrEMMA、pLARmEB、pKWmEB和ISIS EM-BLASSO)来鉴定与种子重量相关的基因组区域。通过两种单位点模型(MLM和CMLM),共有39个snp与HSW显著相关,涉及20条染色体中的10条,即chr01、chr02、chr04、chr08、chr09、chr11、chr13、chr14、chr15、chr16和chr17(额外文件)gydF4y2Ba2gydF4y2Ba:表S2;额外的文件gydF4y2Ba3.gydF4y2Ba:图S1;额外的文件gydF4y2Ba4gydF4y2Ba:图S1)。其中,24个snp被MLM和CMLM共同检测到,而6个和9个snp分别被CMLM和MLM单独检测到gydF4y2Ba5gydF4y2Ba图5gydF4y2Ba3.gydF4y2Ba另一方面,209个snp通过20条染色体上的6个MLMs与HSW相关(附加文件)gydF4y2Ba2gydF4y2Ba表2)。6个模型检测到的snp数量各不相同:pLARmEB (82) > mrMLM (75) > pKWmEB (61) > FASTmrMLM (56) > ISIS EM-BLASSO (47) > FASTmrEMMA(32)。这表明每个模型的检测是不同的。此外,6个模型的两两比较表明,每个模型都具有同时检测彼此SNP的能力,尽管所有6个模型都没有同时检测到SNP。例如,mrMLM和FASTmrMLM同时检测到44个snp,其次是mrMLM和pKWmEB(22),最小的是ISIS EM-BLASSO与FASTmrMLM(11)和FASTmrEMMA(11)(附加文件)gydF4y2Ba2gydF4y2Ba表S3)。多种多位点关联分析方法不仅可以提高qtn检测的可靠性,而且可以相互补充,检测出更多的qtn。在本研究中,两个重要的qtn:gydF4y2BaqHSW-8-1gydF4y2Ba和gydF4y2BaqHSW-19-4gydF4y2Ba/gydF4y2BaqHSW-19-5gydF4y2Ba所鉴定的4个枢纽基因中有2个仅被MLMs检测到。这也支持了传销在GWAS中的作用。gydF4y2Ba
在2个SLMs和6个MLMs的比较中,在6条染色体(chr02, chr08, chr09, chr11, chr13和chr14)上检测到17个共同snp(额外文件)gydF4y2Ba5gydF4y2Ba:图S3B;额外的文件gydF4y2Ba4gydF4y2Ba:图S2)。尽管将SLMs的显著性阈值调整为4而不是4.79 (Bonferroni校正),但SLMs检测到的显著性snp数量低于MLMs检测到的显著性snp数量,这证实了后期模型的鲁棒性和检测能力。SLMs和MLMs的联合使用增强了主要和次要qtn的检出率。在特定环境中检测到多个snp,与方差分析结果一致,说明夏播大豆种子重也受环境调控。两个slm和六个mlm的组合相互补充,确定了231个qtn,这些qtn可能在任何一个模型中都丢失了。然而,MLMs在检测更多snp方面比SLMs更强大。大豆中也有类似的趋势。gydF4y2Ba34gydF4y2Ba],棉花[gydF4y2Ba35gydF4y2Ba],玉米[gydF4y2Ba61gydF4y2Ba]和亚麻[gydF4y2Ba62gydF4y2Ba],其中MLMs比SLMs检测到更多的显著snp。因此,利用多位点模型及其检测能力可以促进育种中的基因组选择。gydF4y2Ba
QTL/ qtn的稳定性对育种计划的使用至关重要。本研究中发现的94个QTNs与先前报道的几个QTL/QTNs共定位,可以利用并整合到育种中gydF4y2Ba2gydF4y2Ba表2)。然而,基于SoyBase上记录的QTL/QTNs,首次报道了137个新的QTNs,因为最近发表的一些报告需要进一步验证。这可能归因于我们最近开发的育种系的多样化背景,指出了它们在育种计划中的潜力。在137个新基因座中,有22个在至少两种环境中被至少两种模型检测到gydF4y2Ba2gydF4y2Ba表2)。此外,利用单倍型块分析的等位基因对种子重的影响,可以开展基于单倍型的育种方案,开发具有理想种子重的基因型,同时探索YHSBLP巨大的遗传基础。gydF4y2Ba
预测候选基因并进一步分析gydF4y2Ba
候选基因的鉴定和利用是GWAS的关键目标之一。到目前为止,很少有基因被证实可以调节大豆种子的重量/大小[gydF4y2Ba63gydF4y2Ba,gydF4y2Ba64gydF4y2Ba,gydF4y2Ba65gydF4y2Ba]与拟南芥和水稻相比[gydF4y2Ba66gydF4y2Ba,gydF4y2Ba67gydF4y2Ba,gydF4y2Ba68gydF4y2Ba,gydF4y2Ba69gydF4y2Ba,gydF4y2Ba70gydF4y2Ba,gydF4y2Ba71gydF4y2Ba]。成熟种子分别由胚、胚乳和种皮、受精卵中心细胞和母被组成[gydF4y2Ba67gydF4y2Ba,gydF4y2Ba72gydF4y2Ba]。种子的重量/大小也取决于细胞大小及其增殖、开花发育、蔗糖运输和其他相关活动,这些活动受几种信号通路的调节[gydF4y2Ba66gydF4y2Ba,gydF4y2Ba67gydF4y2Ba,gydF4y2Ba68gydF4y2Ba,gydF4y2Ba73gydF4y2Ba,gydF4y2Ba74gydF4y2Ba,gydF4y2Ba75gydF4y2Ba]。蔗糖是主要的糖成分,在种子发育过程中占胚胎的97% [gydF4y2Ba76gydF4y2Ba]。在我们的研究中,36个候选基因中有5个与蔗糖转运有关gydF4y2Ba6gydF4y2Ba:表S4)。例如,在单倍型块中,Gm06_13796257-13926598在13909376 bp处有一个显著的SNP (gydF4y2BaqHSW-6-5gydF4y2Ba),含有三个蔗糖转运基因(gydF4y2BaGlyma06g17520, Glyma06g17530gydF4y2Ba和gydF4y2BaGlyma06g17540gydF4y2Ba),分别位于SNP上游40.04 kb、21.72 kb和7.62 kb(附加文件gydF4y2Ba6gydF4y2Ba:表S4)。这些基因99.68%,99.62%和99.61%相似gydF4y2BaAtSWEET12, AtSWEET13gydF4y2Ba和gydF4y2BaAtSWEET10gydF4y2Ba,分别。这是另一个蔗糖转运基因gydF4y2BaGlyma11g27070gydF4y2Ba哪个是同源的gydF4y2BaAtSWEET15gydF4y2Ba在SNP位点27075467 (Gm11_26861064-27075467)下游450.05 kb处发现。的作用所知甚少gydF4y2BaGmSWEETgydF4y2Ba大豆种子发育及其相关性状的基因研究[j]。gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba77gydF4y2Ba]与其他作物相比[gydF4y2Ba78gydF4y2Ba,gydF4y2Ba79gydF4y2Ba]。gydF4y2Ba
从SoyNet获得的功能网络揭示了4个中心基因,即。gydF4y2BaGlyma06g44510, Glyma08g06420, Glyma12g33280gydF4y2Ba和gydF4y2BaGlyma19g28070gydF4y2Ba在我们预测的候选基因(附加文件gydF4y2Ba7gydF4y2Ba:图S4)。虽然共表达网络描述的功能关系并没有包括本研究预测的全部36个候选基因,但它对预测基因中的15个基因以及本研究未捕获的其他基因提供了较好的线索。关于核糖体蛋白和运动蛋白在调节胚胎/种子大小和胚根生长中的相互作用,有大量的文献[gydF4y2Ba80gydF4y2Ba]。这种相互作用在本研究构建的网络中是明显的。一些驱动蛋白的成员已被证明可以调节水稻胚/种子的大小[gydF4y2Ba81gydF4y2Ba,gydF4y2Ba82gydF4y2Ba]。该网络捕获了两个基因(gydF4y2BaGlyma17g17850gydF4y2Ba和gydF4y2BaGlyma17g18360gydF4y2Ba),位于Gm17_15178123-15400615区块内(SNP为15346512)。这两个基因分别属于Subtilase家族蛋白和CYCLIN D3;2,分别参与种皮和种子重调节的其他重要过程[gydF4y2Ba73gydF4y2Ba,gydF4y2Ba83gydF4y2Ba]。同样的,gydF4y2BaGlyma10g38580gydF4y2Ba这是一个K-box区域,MADS-box转录因子在网络中被捕获。据报道,MADS-box基因是所有性状的杰克[gydF4y2Ba84gydF4y2Ba]。该网络还覆盖了多药耐药相关蛋白6的两个基因;ABC转运蛋白跨膜区;最近证明能提高鹰嘴豆种子产量和品质的ABC转运蛋白[gydF4y2Ba51gydF4y2Ba]。功能共表达网络捕获了一些具有同源物的关键基因,这些基因被证明可以调节种子发育,这意味着整合共表达可能是识别作物主要农艺性状关键的策略之一。gydF4y2Ba
独联体gydF4y2Ba-作用调控元件(CAREs)是基因表达动态网络转录调控的主要开关,调控不同的生物过程,如非生物应激反应、激素反应和发育过程[gydF4y2Ba85gydF4y2Ba]。进一步的生物信息学分析显示,四个中心基因中的三个(gydF4y2BaGlyma06g44510, Glyma12g33280, Glyma19g28070gydF4y2Ba)和其他人一起拥有与种子有关的gydF4y2Ba独联体gydF4y2Ba-元件,因此,GCN4_motif, MSA-like, ry元件,circadian clock和ABRE元件(附加文件)gydF4y2Ba6gydF4y2Ba:表S6) [gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba,gydF4y2Ba86gydF4y2Ba]。我们将在未来的工作中验证预测的候选基因,特别是中心基因,通过过表达、CRISPR/Cas9等功能方法确定其在种子重调节中的实际作用。gydF4y2Ba
结论gydF4y2Ba
夏季种植的YHSBLP遗传结构受不同QTNs分布不均的影响。同时检测到17个QTNs,分别占2个slm和6个mlm QTNs总数的43.54%和8.13%。在6种传销中,mrMLM和pLARmEB在检测更多qtn方面的鲁棒性最强。包括Gm08_15803242、Gm09_3461722、Gm13_43480280和Gm14_40721910/ Gm14_40721920在内的多个模型在至少两种环境下检测到的snp可以进一步验证并用于标记辅助育种(MAB)。总共鉴定出36个可能调控大豆种子重的候选基因。这将有助于比较基因组学旨在揭示大豆种子发育/重量的分子机制。四个中心基因,即。gydF4y2Ba, Glyma06g44510, Glyma08g06420, Glyma12g33280gydF4y2Ba和gydF4y2BaGlyma19g28070gydF4y2Ba,通过共表达网络的整合来识别。在R5期,高HSW基因型种子的中心基因表达量均高于低HSW基因型gydF4y2BaGlyma12g33280gydF4y2Ba因此,可以克隆它们来研究它们在种子发育中的调节作用。本研究结果将为通过单抗和单倍型育种方案获得理想的种子重量提供有价值的育种。gydF4y2Ba
材料与方法gydF4y2Ba
种质、田间评价和表型分析gydF4y2Ba
被命名为YHSBLP的试验组共包括573个适合中国长江-淮河地区粮食和菜用大豆的育种品系。所有品系均来自南京农业大学国家大豆改良中心。该群体主要来自育种计划中的核心亲本(南农86-4、南农88-48、玉初4号和南农彩豆5号)和其他国内外优良品种。采用杂交选育法选育豆荚gydF4y2Ba2gydF4y2Ba- fgydF4y2Ba4gydF4y2Ba各组合代,选择高产、优良株型gydF4y2Ba5gydF4y2Ba- fgydF4y2Ba6gydF4y2Ba的一代。本研究选育的品系包括高产稳产品系gydF4y2Ba8gydF4y2Ba- fgydF4y2Ba14gydF4y2Ba的一代。gydF4y2Ba
在江浦地区(北纬33°03′N;经度118°63’E), 2013年、2014年、2017年和2018年夏季,NAU实验站编码分别为E1、E2、E3和E4(附加文件)gydF4y2Ba9gydF4y2Ba表S8,或可在美国国家大豆改良中心网站查阅。gydF4y2Bahttp://ncsi.njau.edu.cn/info/1150/2069.htmgydF4y2Ba).株系按随机完全区组设计种植,每组50株gydF4y2Ba厘米gydF4y2Ba×50gydF4y2Ba厘米gydF4y2Ba3个重复的Hill图。所有推荐的农艺和文化做法都得到了遵循。每个重复100粒重(HSW),在13%水分条件下通过电子天平进行2次技术重复测定。计算两次技术重复中每个基因型的平均值。gydF4y2Ba
百粒重的统计分析gydF4y2Ba
收集的数据在SAS中进行方差分析(SAS Institute, 2010)。SAS/STAT软件版本9.2。SAS Institute Inc., Cary, NC)以下统计模型:gydF4y2Ba
在哪里gydF4y2BaygydF4y2Ba枣疯病gydF4y2Ba表示个人对…的观察gydF4y2Ba枣疯病gydF4y2BathgydF4y2Ba实验装置,gydF4y2BaμgydF4y2Ba为总平均HSW,gydF4y2BaGgydF4y2Ba米gydF4y2Ba效果如何gydF4y2Ba米gydF4y2BathgydF4y2Ba基因型,gydF4y2BaEgydF4y2BalgydF4y2Ba效果如何gydF4y2BalgydF4y2BathgydF4y2Ba环境中,gydF4y2Ba通用电气gydF4y2Ba毫升gydF4y2Ba是相互作用的效果吗gydF4y2Ba米gydF4y2BathgydF4y2Ba基因型和gydF4y2BalgydF4y2BathgydF4y2Ba环境中,gydF4y2BaRgydF4y2BaogydF4y2Ba(gydF4y2BalgydF4y2Ba)gydF4y2Ba效果如何gydF4y2BaogydF4y2BathgydF4y2Ba内的块gydF4y2BalgydF4y2BathgydF4y2Ba环境和εgydF4y2Ba枣疯病gydF4y2Ba为残差。所有因素都被认为是随机的。gydF4y2Ba
使用OriginPro 8统计软件(Origin Corporation, Northampton, MA, USA)在每个环境中计算描述性统计数据,如平均值、平均值标准误差、峰度和偏度,而使用小提琴图(Violin plot)可视化HSW在基因型之间的变化gydF4y2Baggplot2gydF4y2BaR [gydF4y2Ba87gydF4y2Ba]。广义遗传力(gydF4y2BahgydF4y2Ba2gydF4y2Ba)为以下组合环境计算gydF4y2Ba\ ({h} ^ 2 ={\σ}_g ^ 2 / \离开({\σ}_g ^ 2 +{\σ}_{通用电气}^ 2 / n +{\σ}_e ^ 2 / nr \) \)gydF4y2Ba在哪里gydF4y2Ba\({\sigma}_g^2 \)gydF4y2Ba是基因型方差,gydF4y2Ba\({\sigma}_{ge}^2 \)gydF4y2Ba是基因型受环境相互作用的变异,gydF4y2Ba\({\sigma}_e^2 \)gydF4y2Ba是误差方差,gydF4y2BangydF4y2Ba是环境的数量,和gydF4y2BargydF4y2Ba为复制次数[gydF4y2Ba88gydF4y2Ba]。gydF4y2Ba
基因分型gydF4y2Ba
每个加入的DNA样本通过限制性内切位点相关DNA测序(RAD-seq)技术进行基因分型,以产生高通量的snp。简单地说,用CTAB法从幼叶中提取了573份材料的基因组DNA [gydF4y2Ba89gydF4y2Ba]。所有400 ~ 600 bp的DNA片段均通过TaqI酶切得到。利用Illumina HiSeq 2000测序仪对片段进行测序,测序结果显示,片段对端reads长度为90 bp(含6 bp索引),reads长度为[gydF4y2Ba90gydF4y2Ba]。所有的序列读取都与参考Glyma.Wm82.a1.v1.1 [gydF4y2Ba91gydF4y2Ba使用SOAP2软件[gydF4y2Ba92gydF4y2Ba],通过RealSFS软件进行SNP调用[gydF4y2Ba93gydF4y2Ba]。573份材料的snp筛选标准为等位基因缺失率和杂合子召唤率≤30%,次要等位基因频率(MAF)≥5%。fastPHASE软件[gydF4y2Ba94gydF4y2Ba]将杂合等位基因转化为缺失等位基因后,对SNP进行基因分型,得到61166个高质量SNP标记(可在NCBI数据库查询:PRJNA648781,或在国家大豆改良中心网站查询)。gydF4y2Bahttp://ncsi.njau.edu.cn/info/1149/2070.htmgydF4y2Ba).SNP密度图采用gydF4y2BaCMPlotgydF4y2BaR [gydF4y2Ba95gydF4y2Ba]。gydF4y2Ba
遗传多样性、群体结构与单倍型块gydF4y2Ba
使用pLINK的-indep-pairwise命令选项进一步修剪过滤的snp。然后通过ADMIXTURE V1.3.0软件使用修剪后的snp来估计群体结构[gydF4y2Ba96gydF4y2Ba]。在admix设置中,簇数(K)初始设置为1 ~ 10;然后,每个Q和相关gydF4y2BaPgydF4y2Ba-value被估计。最可能的亚种群数量是使用Evanno等人描述的方法确定的。[gydF4y2Ba97gydF4y2Ba]。性状分析采用TASSEL (aSSociation, Evolution and Linkage)软件5.2版进行主成分分析(PCA) [gydF4y2Ba98gydF4y2Ba]。在TASSEL中计算一对Nei’s遗传距离矩阵,用于构建邻居连接树。gydF4y2Ba
关联映射和单倍型块分析gydF4y2Ba
在TASSEL 5.2中进行了GWAS的两种slm,即MLM (Q + K)和压缩MLM (CMLM)(PCA + K) [gydF4y2Ba98gydF4y2Ba]和基因组全关联预测工具(GAPIT)环境[gydF4y2Ba99gydF4y2Ba],其中Q矩阵由ADMIXTURE V1.3.0软件计算的种群结构得到[gydF4y2Ba96gydF4y2Ba],在每个软件中估计亲属矩阵(K),并在CMLM中使用3个pc进行主成分分析。阈值(−gydF4y2Ba日志gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba)≥4.00),表明snp与种子重有显著相关性。gydF4y2Ba
六种传销(即mrMLM) [gydF4y2BaOne hundred.gydF4y2Ba], FASTmrMLM [gydF4y2Ba101gydF4y2Ba], fastemma [gydF4y2Ba31gydF4y2Ba], pLARmEB [gydF4y2Ba102gydF4y2Ba], pKWmEB [gydF4y2Ba103gydF4y2Ba]和ISIS EM-BLASSO [gydF4y2Ba104gydF4y2Ba]与包装一起以R计算gydF4y2BamrMLM。GUIgydF4y2Ba(gydF4y2Bahttps://cran.r-project.org/web/packages/mrMLM.GUIgydF4y2Ba/ index . html)。在这些模型中,使用Q矩阵来解释群体结构,而亲属矩阵(K)在mrMLM中计算。GUI环境。临界LOD值设置为3。在至少一种环境中,至少2种模型检测到的snp被认为是相对稳定的snp。QTL命名遵循McCouch等人的命名法。[gydF4y2Ba105gydF4y2Ba],因此以“q”开头,然后是性状名称的缩写(HSW,百粒重)和染色体名称,最后是在同一染色体上检测到的QTL数量。gydF4y2Ba
在Haploview软件中,采用Haploview软件4.2版本中默认参数的四配子规则方法,对单位点和至少2个多位点模型中2个相对稳定的snp进行单倍型块分析[gydF4y2Ba106gydF4y2Ba,gydF4y2Ba107gydF4y2Ba]。采用Duncan Range多重检验(两两比较),在显著水平上评价各单倍型区各加入组间种子重的差异gydF4y2BaP≤gydF4y2Ba0.05.gydF4y2Ba
候选基因预测与分析gydF4y2Ba
通过单位点模型或多位点模型,在至少2种环境中检测到的显著snp的500 kb范围内检索到潜在的候选基因gydF4y2Bag·马克斯gydF4y2BaWilliam 82参考基因模型1.0在SoyBase [j]gydF4y2Ba108gydF4y2Ba]。从SoyBase下载人工筛选的模型基因功能注释。利用豆科植物基因功能和基因组进化综合研究平台(LegumePI) version 2 (gydF4y2Bahttp://plantgrn.noble.org/LegumeIPgydF4y2Ba) [gydF4y2Ba109gydF4y2Ba]及豆类资讯系统(LIS) (gydF4y2Bahttps://legumeinfo.org/gydF4y2Ba) [gydF4y2Ba110gydF4y2Ba]。gydF4y2Ba
每个基因的上游1.5 kb (ATG前)序列从Phytozome数据库(gydF4y2Bahttps://phytozome.jgi.doe.govgydF4y2Ba).然后将获得的序列提交到PlantCare数据库gydF4y2Bahttp://bioinformatics.psb.ugent.be/webtools/plantcare/htmlgydF4y2Ba识别gydF4y2Ba独联体gydF4y2Ba-每个基因启动子区域与种子相关功能相关的元件[gydF4y2Ba111gydF4y2Ba]。gydF4y2Ba
预测的候选基因和其他相关基因之间的蛋白-蛋白相互作用的功能网络通过SoyNet (gydF4y2Bahttps://www.inebio.org/soynet/serach.phpgydF4y2Ba) [gydF4y2Ba58gydF4y2Ba]。然后在独立版本的Cytoscape软件中可视化从SoyNet导出的功能网络[gydF4y2Ba112gydF4y2Ba]和网络分析师[gydF4y2Ba113gydF4y2Ba]。gydF4y2Ba
实时定量聚合酶链反应(qRT-PCR)验证枢纽基因gydF4y2Ba
在种子发育的R5和R7阶段,选取了7个HSW差异极大的YHSBLP基因型(分别包括2个低HSW和5个高HSW的基因型)[gydF4y2Ba114gydF4y2Ba],将于2019年夏季在江浦实验站采集。总RNA采用Plant RNA Extract Kit (TIANGEN Co. Ltd.)进行分离。利用HiScript II QRT SuperMix for qPCR (+gDNA wiper) (Vazyme Biotech,南京,中国)合成的cDNA。酶2x ChamQ™SYBR qPCR Master Mix Kit (Vazyme Biotech,南京,中国)在Light Cycler 480系统(Roche, Roche Diagnostic, Basel, Switzerland)中按照标准方案和程序使用。采用3个生物重复和3个技术重复。的gydF4y2BaGmActin 11gydF4y2Ba(gydF4y2BaGlyma18g52780gydF4y2Ba)作为管家基因,使所选基因型种子在R5和R7期的相对表达水平正常化。用于qRT-PCR的引物见附加文件gydF4y2Ba8gydF4y2Ba表S7。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
除SNP数据集外,本研究过程中产生或分析的所有数据均包含在本文及其补充信息文件中。本研究中使用的SNP数据集可在NCBI (SRA登录:PRJNA648781)库的序列读取档案(SRA)和国家大豆改良中心的网站上获得。gydF4y2Bahttp://ncsi.njau.edu.cn/zygx.htmgydF4y2Ba。gydF4y2Ba
缩写gydF4y2Ba
- 关心:gydF4y2Ba
-
独联体gydF4y2Ba-作用调控要素gydF4y2Ba
- GWAS:gydF4y2Ba
-
全基因组关联研究gydF4y2Ba
- 传销:gydF4y2Ba
-
多位点模型gydF4y2Ba
- QTL:gydF4y2Ba
-
数量性状位点gydF4y2Ba
- 本考察团:gydF4y2Ba
-
数量性状核苷酸gydF4y2Ba
- RAD-seq:gydF4y2Ba
-
限制性位点相关DNA测序gydF4y2Ba
- SLMs:gydF4y2Ba
-
Single-locus模型gydF4y2Ba
- 单核苷酸多态性:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
Hickey LT, Hafeez AN, Robinson H, Jackson SA, Leal-Bertioli SC, Tester M, Gao C, Godwin ID, Hayes BJ, Wulff BB。培育农作物养活100亿人。生物工程学报。2019;37:744-54。gydF4y2Ba
- 2.gydF4y2Ba
Dita MA, Rispail N, Prats E, Rubiales D, Singh KB。克服豆科植物生物和非生物胁迫限制的生物技术方法。Euphytica。2006;147(1 - 2):24。gydF4y2Ba
- 3.gydF4y2Ba
Pandey MK, Roorkiwal M, Singh VK, Ramalingam A, Kudapa H, Thudi M, Chitikineni A, Rathore A, Varshney RK。新兴的豆类育种基因组工具:现状与未来展望。植物学报,2016;7:455。gydF4y2Ba
- 4.gydF4y2Ba
Sparvoli F, Bollini R. Cominelli E. In: DeRon AM,编辑。谷物豆类。柏林:施普林格;2015.p . 291 - 318。gydF4y2Ba
- 5.gydF4y2Ba
大豆与健康更新:临床和流行病学文献的评价。营养。2016;8(12):754。gydF4y2Ba
- 6.gydF4y2Ba
墨西拿乔丹。豆类和大豆:它们的营养概况和对健康的影响。中华临床医学杂志。1999;20(3):349 - 349。gydF4y2Ba
- 7.gydF4y2Ba
崔忠,James A, Miyazaki S, Wilson RF, Carter TE Jr.。传统和新型大豆食品的特色大豆育种。编辑:刘凯。大豆作为功能性食品和配料:AOCS出版社;2004.p . 74 - 332。gydF4y2Ba
- 8.gydF4y2Ba
甘地A.大豆及其食品的质量。粮食学报,2009;16(1):11-9。gydF4y2Ba
- 9.gydF4y2Ba
李宁,徐锐,李勇。植物种子大小调控的分子网络。植物学报,2019;70(1):1 - 30。gydF4y2Ba
- 10.gydF4y2Ba
梁H, Lanjie徐,永亮,杨H,魏D,张H .与主要识别法,上位性QTL和环境交互作用在大豆种子形状和重量hundred-seed跨年。[J] .生物医学工程学报,2016;95(2):475。gydF4y2Ba
- 11.gydF4y2Ba
滕伟,冯丽,李伟,吴东,赵霞,韩勇,李伟。大豆种子重的多环境遗传结构解析。作物学报,2017;68(4):358-65。gydF4y2Ba
- 12.gydF4y2Ba
吴东,詹艳,孙强,徐磊,连明,赵旭,韩勇,李伟。大豆数量性状位点的鉴定gydF4y2Ba大豆gydF4y2Ba(L。[Merr.])种子重,包括主要效应、上位效应和QTL ×环境效应。植物学报,2018;137(2):194-202。gydF4y2Ba
- 13.gydF4y2Ba
王健,褚生,张辉,朱勇,程华,于东。一种新的全基因组SNP阵列的开发与应用揭示了大豆驯化历史。科学通报,2016;6:20728。gydF4y2Ba
- 14.gydF4y2Ba
韩艳,赵霞,刘东,李艳,Lightfoot DA,杨志,赵玲,周刚,王志,黄磊。大豆驯化足迹锚定基因组区在农艺上的重要性。植物化学学报,2016;29(2):871-84。gydF4y2Ba
- 15.gydF4y2Ba
李国安,Crawford GW,李玲,Yuka S,薛祥c。gydF4y2Ba大豆gydF4y2Ba在东亚:规模重要吗?科学通报,2011;6(11):e26720。gydF4y2Ba
- 16.gydF4y2Ba
周忠,于军,郑伟,郭志恒,李军,李卫宇,杨彦军,孙丽萍,张颖军,m彦明。302份大豆野生和栽培材料驯化改良相关基因重测序。生物工程学报,2015;33(4):408-14。gydF4y2Ba
- 17.gydF4y2Ba
古普塔PK, Rustgi S, Kulwal PL.高等植物连锁不平衡与关联研究的现状与展望。植物学报,2005;37(4):461 - 461。gydF4y2Ba
- 18.gydF4y2Ba
A.单核苷酸多态性在作物遗传中的应用。植物学报,2002;5(2):94-100。gydF4y2Ba
- 19.gydF4y2Ba
正如乔丹。植物育种中数量性状位点分析的现状与展望。植物育种,2002;121(4):281-91。gydF4y2Ba
- 20.gydF4y2Ba
苗林,杨生,张凯,何军,吴超,任勇,盖军,李勇gydF4y2BaGmSWEET39gydF4y2Ba影响大豆种子含油量。中国生物医学工程学报,2019;22(4):661 - 661。gydF4y2Ba
- 21.gydF4y2Ba
张宏,郝东,司陶明,尹忠,胡忠,张刚,于东。大豆植株结构与产量组成性状关系的遗传解析(英文)gydF4y2Ba大豆gydF4y2Ba),通过跨多个环境的关联分析。植物育种,2015;134(5):564-72。gydF4y2Ba
- 22.gydF4y2Ba
张军,宋强,葛立平,蒋国良。大豆种子重的全基因组关联研究、基因组预测和标记辅助选择(gydF4y2Ba大豆gydF4y2Ba).理论与应用,2016;129(1):117-30。gydF4y2Ba
- 23.gydF4y2Ba
方超,马勇,吴森,刘忠,王忠,杨锐,胡刚,周忠,于海,张明。大豆农艺性状遗传网络的全基因组关联研究。中国生物医学工程学报,2017;18(1):161。gydF4y2Ba
- 24.gydF4y2Ba
Copley TR, Duceppe MO, O 'Donoughue LS。早熟大豆植株引种系成熟及产量性状相关新位点的鉴定。生物医学工程学报,2018;19(1):167。gydF4y2Ba
- 25.gydF4y2Ba
李霞,张霞,朱丽,卜勇,王霞,张霞,周莹,王霞,郭宁,邱玲。大豆R6期4个产量相关性状的全基因组关联研究。生物医学工程学报,2019;20(1):39。gydF4y2Ba
- 26.gydF4y2Ba
Chaudhary J, Patil GB, Sonah H, Deshmukh RK, Vuong TD, Valliyodan B, Nguyen HT。扩大组学资源改善大豆种子组成性状。前沿植物科学,2015;6(31)。gydF4y2Ba
- 27.gydF4y2Ba
赵鑫,董辉,常辉,赵健,滕伟,邱磊,李伟,韩勇。大豆百粒重全基因组关联定位及候选基因分析[J]。gydF4y2Ba大豆gydF4y2Ba(l)美林]。生物医学工程学报,2019;20(1):648。gydF4y2Ba
- 28.gydF4y2Ba
孙勇,潘杰比,石晓玲,杜学祥,吴强,齐志明,蒋宏伟,辛大伟,刘云云,胡海华。大豆百粒重的多环境定位与meta分析。中国生物医学工程学报,2012;39(10):935 - 943。gydF4y2Ba
- 29.gydF4y2Ba
引用本文:陈建军,陈建军,陈建军,陈建军。大豆节间数、株高、籽粒重和籽粒产量的全基因组关联及互作互作。生物医学工程学报,2019;20(1):52。gydF4y2Ba
- 30.gydF4y2Ba
闫丽,Hofmann N,李松,Ferreira ME,宋波,姜刚,任松,Quigley C, Fickus E, Cregan P.大豆选择群体中对种子质量影响较大的QTL的全基因组关联和固定指数分析。生物医学工程学报,2017;18(1):529。gydF4y2Ba
- 31.gydF4y2Ba
温永杰,张辉,倪玉玲,黄斌,张军,冯建勇,王世斌,Dunwell JM,张永明,吴瑞。多基因座全基因组关联研究中混合线性模型的方法实现。生物通报,2017;19(4):700-12。gydF4y2Ba
- 32.gydF4y2Ba
张玉明,贾志,Dunwell JM。新的多位点GWAS方法在复杂性状遗传解剖中的应用。植物科学,2019;10:100。gydF4y2Ba
- 33.gydF4y2Ba
常敏,何玲,蔡玲。全基因组关联研究综述。编辑:黄婷。计算系统生物学:方法和协议。纽约:Springer New York;2018.p . 97 - 108.8。gydF4y2Ba
- 34.gydF4y2Ba
常峰,郭超,孙峰,张健,王铮,孔健,何青,夏敏拉,赵涛。夏播大豆主茎动态株高和节数的全基因组关联研究。植物科学,2018;9:1184。gydF4y2Ba
- 35.gydF4y2Ba
陈丽,冯远志,王润润,王媛媛,王青莲。陆地棉纤维品质性状的单位点与多位点全基因组关联研究(gydF4y2Ba陆地棉gydF4y2Bal .)。植物科学,2018;9(10):1083。gydF4y2Ba
- 36.gydF4y2Ba
张凯,刘松,李伟,刘松,李旭,方勇,张健,王勇,徐松,张健,宋健。利用多位点全基因组关联研究鉴定大豆种子蛋白含量控制QTNs。植物科学,2018;9(9):1690。gydF4y2Ba
- 37.gydF4y2Ba
张超,李丽,刘琴,顾玲,黄娟,魏慧,王慧,于森。陆地棉纤维长度基因位点及候选基因的鉴定(gydF4y2Ba陆地棉gydF4y2BaL.)通过关联映射和联动分析。前面。植物科学。2019;10:53。gydF4y2Ba
- 38.gydF4y2Ba
Schaefer R, Michno J- m, Jeffers J, Hoekenga OA, Dilkes BP, Baxter IR, Myers C.整合共表达网络的玉米致病基因排序。植物学报,2018;30(3):922 - 942。gydF4y2Ba
- 39.gydF4y2Ba
Sarkar NK, Kim Y-K, Grover A.水稻幼苗遇热胁迫呼叫的共表达网络分析。植物学报,2014,34(1):444 - 444。gydF4y2Ba
- 40.gydF4y2Ba
张辉,王明丽,Schaefer R,党鹏,姜涛,陈晨。GWAS与共表达网络揭示栽培花生的基因组变异。中国农业科学学报,2019;37(6):1226 - 1236。gydF4y2Ba
- 41.gydF4y2Ba
杨建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,等。植物生理学报,2017;17(1):872-86。gydF4y2Ba
- 42.gydF4y2Ba
杨松,苗丽,何军,张凯,李勇,盖军。大豆种子发育中油脂积累相关的动态转录组变化。光子学报,2019;20(9):2202。gydF4y2Ba
- 43.gydF4y2Ba
吴忠,王敏,杨松,陈生,常霞。大豆基因全球共表达网络为非豆科植物和豆科植物结瘤进化提供了新的视角。新植物学报,2019;23(3):2104 - 19。gydF4y2Ba
- 44.gydF4y2Ba
Palumbo MC, Zenoni S, Fasoli M, Massonnet M, Farina L, Castiglione F, Pezzotti M, Paci P.综合网络分析确定了搏击俱乐部节点是一类包含关键开关基因的枢纽,这些基因在葡萄生长过程中诱导主要转录组重编程。植物学报,2014,26(12):4617-35。gydF4y2Ba
- 45.gydF4y2Ba
Das S, Meher PK, Rai A, Bhar LM, Mandal BN。基因共表达网络分析中基因选择、枢纽基因鉴定和模块相互作用的统计方法:在大豆铝胁迫中的应用(gydF4y2Ba大豆gydF4y2Bal .)。科学通报,2017;12(1):e0169605。gydF4y2Ba
- 46.gydF4y2Ba
李军,赵军,李勇,高勇,华森,Nadeem M,孙刚,张伟,侯军,王旭。一个新的种子大小相关基因位点的鉴定gydF4y2BaSW9-1gydF4y2Ba在大豆。作物学报;2019;7(4):548-59。gydF4y2Ba
- 47.gydF4y2Ba
张雪,李文静,张晓月,王金阳,李志阳,韩勇,李伟。大豆种子形态全基因组关联定位及候选基因分析[j]。gydF4y2Ba大豆gydF4y2Ba(l)Merri]。作物学报,2019,30(8):684-93。gydF4y2Ba
- 48.gydF4y2Ba
孔treas - soto RI, Mora F, Oliveira MARD, Higashi W, Scapim CA, Schuster I.大豆农艺性状的全基因组关联研究。科学通报,2017;12(2)。gydF4y2Ba
- 49.gydF4y2Ba
Josephs EB, Stinchcombe JR, Wright SI。关于维持数量性状遗传变异的进化力量,全基因组关联研究能告诉我们什么?植物学报,2017;21(1):21-33。gydF4y2Ba
- 50.gydF4y2Ba
徐勇,刘霞,傅健,王辉,王健,黄晨,Prasanna BM, Olsen MS,王刚,张安。通过基因组选择提高遗传增益:从家畜到植物。植物学报,2019;1:100 . 05。gydF4y2Ba
- 51.gydF4y2Ba
Basu U, Upadhyaya HD, Srivastava R, Daware A, Malik N, Sharma A, Bajaj D, Narnoliya L, Thakro V, Kujur A. ABC转运蛋白介导的谷胱甘肽结合物转运提高鹰嘴豆种子产量和品质。植物生理学报,2019;18(1):253-75。gydF4y2Ba
- 52.gydF4y2Ba
Severin AJ, Woody JL, Bolon YT, Joseph B, Diers BW, Farmer AD, Muehlbauer GJ, Nelson RT, Grant D, Specht JE。RNA-Seq图谱gydF4y2Ba大豆gydF4y2Ba大豆转录组指南。植物学报,2010;10(1):160。gydF4y2Ba
- 53.gydF4y2Ba
Onodera, Suzuki A, Cy WH, Takaiwa F.水稻功能转录激活因子RISBZ1通过GCN4基序负责储存蛋白基因的胚乳特异性表达。生物化学学报,2001;27(6):1249 - 1249。gydF4y2Ba
- 54.gydF4y2Ba
伊藤M,岩濑M,玉玉H,拉维斯P,小明A,西滨R,町田Y,渡边A.小说gydF4y2Ba独联体gydF4y2Ba植物b型周期蛋白基因启动子中的-作用元件激活M期特异性转录。植物学报。1998;10(3):331。gydF4y2Ba
- 55.gydF4y2Ba
Gea G, Nathalie M, Anna G, Sundström JF, Lars R, Ines E. RY/Sph元件介导成熟基因的转录抑制。植物学报,2010;18(3):552 - 565。gydF4y2Ba
- 56.gydF4y2Ba
发育中的大豆种子的生物钟控制转录组。植物基因组,2010;3(1):3 - 13。gydF4y2Ba
- 57.gydF4y2Ba
王晓明,王晓明,王晓明,等。拟南芥种子萌发过程中mRNA的表达及转录调控。植物学报,2010;41(5):697-709。gydF4y2Ba
- 58.gydF4y2Ba
金恩,黄松,李一。大豆协同功能网络数据库SoyNetgydF4y2Ba大豆gydF4y2Ba。中国生物医学工程学报,2016;45(1):1082 - 991。gydF4y2Ba
- 59.gydF4y2Ba
杨军,张强,杨军,杨军,杨军,杨建军,杨建军,杨建军,杨建军,杨建军,杨建军。[J] .中国生物医学工程学报,2017;31(1):5-22。gydF4y2Ba
- 60.gydF4y2Ba
Varshney RK, Sinha P, Singh VK, Kumar A,张强,Bennetzen JL。5亿美元用于作物基因改良。植物学报,2020;19(3):1190 - 1190。gydF4y2Ba
- 61.gydF4y2Ba
徐勇,杨涛,周勇,尹松,李鹏,刘健,徐松,杨震,徐超。基于单位点和多位点模型的玉米淀粉糊化特性全基因组关联定位。植物科学,2018;9:1311。gydF4y2Ba
- 62.gydF4y2Ba
何磊,肖军,Rashid KY,姚忠,李鹏,贾刚,王旭,Cloutier S,游调频。亚麻抗pasmo基因全基因组关联研究(gydF4y2Ba亚麻属植物usitatissimumgydF4y2Bal .)。植物科学,1982;2019:9。gydF4y2Ba
- 63.gydF4y2Ba
郑宁,徐世杰,金明辉,李顺,文建奎,金洪生,郑世昌。gydF4y2BaLngydF4y2Ba是大豆小叶形状和每荚种子数的关键调节剂。植物学报,2012;24(12):4807-18。gydF4y2Ba
- 64.gydF4y2Ba
卢鑫,熊强,程涛,李秋冬,刘晓林,毕彦东,李伟,张文科,马斌,赖永昌,杜文文。一个数量性状位点下的PP2C-1等位基因提高了大豆百粒重。植物学报,2017;10(5):670-68。gydF4y2Ba
- 65.gydF4y2Ba
赵波,戴安,魏华,杨生,王波,姜宁,冯霞gydF4y2BaKLUgydF4y2Ba同系物gydF4y2BaGmCYP78A72gydF4y2Ba调节大豆种子大小。植物学报,2015;39(1):357 - 357。gydF4y2Ba
- 66.gydF4y2Ba
李宁,李艳。泛素介导的植物种子大小调控。植物科学,2014;5:332。gydF4y2Ba
- 67.gydF4y2Ba
李宁,李艳。植物种子大小调控的信号通路。植物学报,2016;33:23-32。gydF4y2Ba
- 68.gydF4y2Ba
朱鑫,梁伟,崔旭,陈敏,尹超,罗忠,朱军,卢卡斯文杰,王忠,张东。油菜素内酯通过触发MYB结构域蛋白碳饥饿花药的表达促进水稻花粉粒和种子的发育。植物学报,2015;32(4):557 - 561。gydF4y2Ba
- 69.gydF4y2Ba
杨任D,王X,杨M, L,他邓XW G。一项全基因组关联研究发现了拟南芥种子大小控制的新调控因子。植物学报,2019;22(2):895-906。gydF4y2Ba
- 70.gydF4y2Ba
苗超,王东,何锐,刘生,朱建军。突变gydF4y2Ba米尔396 egydF4y2Ba和gydF4y2Ba米尔396 fgydF4y2Ba增加水稻籽粒大小,调节水稻茎部结构。植物学报,2010;18(2):491 - 591。gydF4y2Ba
- 71.gydF4y2Ba
刘娥,曾生,朱生,刘毅,吴刚,赵凯,刘霞,刘强,董志,党霞,谢华。灌浆速率有利等位基因ate1提高水稻灌浆速率和产量。植物生理学报,2019;18(1):1267 - 1267。gydF4y2Ba
- 72.gydF4y2Ba
labon - placette C, Kōhler C.胚和胚乳在种子发育中的合作伙伴。植物学报,2014;17(5):669 - 669。gydF4y2Ba
- 73.gydF4y2Ba
李建军,张建军,张建军,等。豆科植物胚乳定位枯草酶对种子大小的调控作用。植物学报,2012;36(3):738-51。gydF4y2Ba
- 74.gydF4y2Ba
朱波,李慧,韦江琪,马淑娟,韦先兵,潘艳喜,宁丽芳,郝丽。AGAMOUS同源物在花器官发育调控中的功能专一化gydF4y2BaMedicago truncatulagydF4y2Ba。植物科学,2018;9:854。gydF4y2Ba
- 75.gydF4y2Ba
袁峰,李军,黄燕,刘玲,李东,陈玲,栾思。FERONIA受体激酶调控种子大小gydF4y2Ba拟南芥gydF4y2Ba。植物学报,2014;5(7):920-2。gydF4y2Ba
- 76.gydF4y2Ba
山乔丹。种子贮藏产物合成的控制。植物学报,2004;7(3):302-8。gydF4y2Ba
- 77.gydF4y2Ba
王松,横阔阔,郭锐,惠兰君,阮玉林,马建峰,寿华。大豆糖转运蛋白的研究进展gydF4y2BaGmSWEET15gydF4y2Ba介导蔗糖从胚乳向早期胚胎的输出。植物生理学报,2019;18(4):2133 - 2141。gydF4y2Ba
- 78.gydF4y2Ba
杨军,罗丹,杨斌,陈文博,严建新。gydF4y2Ba甜蜜的11gydF4y2Ba和gydF4y2Ba15gydF4y2Ba在水稻灌种中起关键作用。植物化学学报,2018;18(2):604-15。gydF4y2Ba
- 79.gydF4y2Ba
陈立强,林毅伟,曲小强,Sosso D, McFarlane HE, Londoño A, Samuels AL, Frommer WB。蔗糖转运蛋白在种皮和胚乳中的序列表达为拟南芥胚提供营养。植物学报,2015,27(3):607-19。gydF4y2Ba
- 80.gydF4y2Ba
田松,吴军,刘勇,黄霞,李飞,王忠,孙明霞。核糖体蛋白NtRPL17与激酶-12家族蛋白NtKRP相互作用,参与调控胚/种子大小和胚根生长。生物学报,2017;68(20):5553-64。gydF4y2Ba
- 81.gydF4y2Ba
Kitagawa K, Kurinami S, Oki K, Abe Y, Ando T, Kono I, Yano M, Kitano H, Iwasaki Y.一个新的Kinesin 13蛋白调控水稻种子长度。植物生理学报,2010;51(8):1315-29。gydF4y2Ba
- 82.gydF4y2Ba
吴涛,沈毅,郑敏,杨超,陈毅,冯志,刘旭,刘松,陈忠,雷成gydF4y2Ba西格里碳素集团gydF4y2Ba该基因编码一种具有反活化活性的运动蛋白样蛋白,与水稻的粒长和株高有关。植物学报,2014;33(2):235-44。gydF4y2Ba
- 83.gydF4y2Ba
Dewitte W, Scofield S, Alcasabas AA, Maughan SC, Menges M, Braun N, Collins C, Nieuwland J, Prinsen E, Sundaresan V, Murray JA。拟南芥CYCD3 d型细胞周期蛋白连接细胞增殖和内环,并限制细胞分裂素反应的速率。科学通报,2007;35(6):537 - 542。gydF4y2Ba
- 84.gydF4y2Ba
李建军,李建军,李建军,等。小麦品种的遗传变异及其对水稻品种驯化的影响。生物学报,2018;69(7):1447-69。gydF4y2Ba
- 85.gydF4y2Ba
Pandey S, Reddy CS, Yaqoob U, Negi YK, Arora S, Kau T。gydF4y2BaIn-silicogydF4y2Ba抗坏血酸谷胱甘肽途径上游基因的顺式调控元件分析gydF4y2Ba栽培稻gydF4y2Ba。生物物理学报,2015;4:159。gydF4y2Ba
- 86.gydF4y2Ba
福特斯F, Strömvik MV。在芸苔科、豆科和豆科植物中,种子贮藏蛋白基因启动子含有保守的DNA基序。植物学报,2009;9(1):126-0。gydF4y2Ba
- 87.gydF4y2Ba
拉琴情节:盒子情节-密度轨迹协同作用。医学杂志。1998;52(2):181-4。gydF4y2Ba
- 88.gydF4y2Ba
奈奎斯特WE,贝克RJ。植物群体遗传力估算与选择响应预测。植物学报,1991;10(3):235-322。gydF4y2Ba
- 89.gydF4y2Ba
Murray MG, Thompson WF。快速分离高分子量植物DNA。核酸学报,1980;8(19):4321 - 322。gydF4y2Ba
- 90.gydF4y2Ba
李丽,郭宁,牛静,王铮,崔旭,孙健,赵涛,邢华。大豆抗疫霉基因位点及候选基因的关联分析[J]。稳定)。生物化学学报,2016;29(3):1095-103。gydF4y2Ba
- 91.gydF4y2Ba
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W, Hyten DL, Song Q, Thelen JJ, Cheng J。古多倍体大豆基因组序列分析。大自然。2010;463(7278):178。gydF4y2Ba
- 92.gydF4y2Ba
李锐,于超,李勇,林涛涛,姚思敏,Kristiansen K,王杰。SOAP2:一种改进的超快速短读比对工具。生物信息学,2009,25(15):1966 - 7。gydF4y2Ba
- 93.gydF4y2Ba
易祥,梁勇,Huerta-Sanchez E,金祥,郭志平,Pool JE,徐祥,姜浩,Vinckenbosch N, Korneliussen TS,等。50个人类外显子组的测序揭示了对高海拔的适应。科学gydF4y2Ba。gydF4y2Ba2010, 329(5987): 75 - 8。gydF4y2Ba
- 94.gydF4y2Ba
李建军,李建军,李建军,等。一种快速、灵活的大规模群体基因型数据统计模型:用于推断缺失基因型和单倍型期的应用。[J] .中国生物医学工程学报。2006;31(4):629 - 629。gydF4y2Ba
- 95.gydF4y2Ba
李林Y. CMplot: Circle Manhattan Plot. 2019。可以在:gydF4y2Bahttps://CRAN.R-project.org/package=CMplotgydF4y2Ba。2019年11月23日通过。gydF4y2Ba
- 96.gydF4y2Ba
Alexander DH, nove J, Lange K.基于快速模型的非亲属个体祖先估计。基因工程学报,2009;19(9):1655-64。gydF4y2Ba
- 97.gydF4y2Ba
李建军,李建军,李建军,等。基于结构的个体聚类数量检测方法研究。中华生物医学工程学报,2010;14(8):2611-20。gydF4y2Ba
- 98.gydF4y2Ba
张志强,张志强,张志强。TASSEL:软件的关联映射复杂性状在不同的样本。Bioinfor。2007;23(19):2633 - 5。gydF4y2Ba
- 99.gydF4y2Ba
李丽娟,冯涛,王岐山,贾森·P,孟莉,Bradbury PJ,戈尔·马,Buckler ES,张志武。基因组关联与预测的集成工具。Bioinfor。2012;28(18):2397 - 9。gydF4y2Ba
- One hundred.gydF4y2Ba
王绍斌,冯建勇,任文龙,黄斌,周磊,温永杰,张杰,Dunwell JM,徐生,张永明。通过多位点混合线性模型方法提高全基因组关联研究的能力和准确性。科学通报,2016;6:1944。gydF4y2Ba
- 101.gydF4y2Ba
Tamba CL .,张玉明。多基因座全基因组关联研究的快速mrMLM算法。BioRxiv。2018年,341784年。gydF4y2Ba
- 102.gydF4y2Ba
张军,冯军,倪莹,温燕,牛燕,谭巴,岳晨,宋强,张勇。pLARmEB:基于最小角回归和经验贝叶斯的多位点全基因组关联研究。遗传。2017;118(6):517。gydF4y2Ba
- 103.gydF4y2Ba
任伟,温彦军,Dunwell JM,张彦明。pKWmEB:将多基因背景控制下的Kruskal-Wallis检验与经验贝叶斯相结合,用于多位点全基因组关联研究。遗传。2018;120(3):208 - 18。gydF4y2Ba
- 104.gydF4y2Ba
坦巴长荣,倪玉玲,张彦明。多基因座全基因组关联研究的迭代确定独立筛选EM-Bayesian LASSO算法。科学通报,2017;13(1):e1005357。gydF4y2Ba
- 105.gydF4y2Ba
mcouch SR,陈霞,Panaud O, Temnykh S,徐勇,Yong GC,黄宁,Ishii T, Blair M.水稻微卫星标记的开发、定位及其在水稻遗传育种中的应用。植物学报。1997;35(1):89-99。gydF4y2Ba
- 106.gydF4y2Ba
张建军,张建军,张建军。Haploview: LD和单倍型图谱的分析和可视化。Bioinfor。2005;21(2):263 - 5。gydF4y2Ba
- 107.gydF4y2Ba
Ning W, Akey JM, Kun Z, Ranajit C, Li j。重组交叉分布和单倍型块的起源:种群历史、重组和突变的相互作用。[J] .中国生物医学工程学报,2002;31(5):1227 - 1234。gydF4y2Ba
- 108.gydF4y2Ba
Grant D, Nelson RT, Cannon SB, Shoemaker RC。SoyBase, USDA-ARS大豆基因和基因组数据库。核酸研究。2010;38(数据库版):D843。gydF4y2Ba
- 109.gydF4y2Ba
李军,戴旭,刘涛,赵培培。LegumeIP:一个比较基因组学和转录组学的综合数据库。中国生物医学工程学报,2011;40(1):1221 - 1221。gydF4y2Ba
- 110.gydF4y2Ba
Gonzales MD, Eric A, Andrew F, Kamal G, David G, Randy S, Beavis WD, Waugh ME。豆科植物信息系统(LIS):比较豆科植物生物学的综合信息资源。核酸Res. 2005;33(数据库问题):D660-5。gydF4y2Ba
- 111.gydF4y2Ba
Lescot M, Patrice D, Gert T, Kathleen M, Yves M, Yves VDP, Pierre R, Stephane R. PlantCARE,植物顺式调控元件数据库和启动子序列分析工具门户。核酸学报,2002;30(1):325-7。gydF4y2Ba
- 112.gydF4y2Ba
刘建军,刘建军,刘建军,刘建军,刘建军。Cytoscape web:一个交互式的基于web的网络浏览器。Bioinfor。2010;26(18):2347 - 8。gydF4y2Ba
- 113.gydF4y2Ba
周刚,吴素凡,夏军,李建军,李建军,李建军,李建军。基于网络分析的基因表达分析平台。核酸学报,2019;47(1):W234-41。gydF4y2Ba
- 114.gydF4y2Ba
Fehr W, Caviness C, Burmood D, Pennington J.大豆发育阶段描述。gydF4y2Ba大豆gydF4y2Ba(l)美林。作物科学,1971;11(6):929-31。gydF4y2Ba
致谢gydF4y2Ba
作者感谢所有在研究项目和撰写论文过程中支持我们的团队和实验室伙伴和朋友。gydF4y2Ba
资金gydF4y2Ba
国家重点研发计划项目(2016YFD0100304, 2018YFD0100800)、国家自然科学基金项目(31871646)、教育部长江学者及高校创新团队计划项目(PCSIRT_17R55)、中央高校基本科研业务费专项资金(KYT201801)、江苏省现代作物生产协同创新中心项目(jsic - mcp)资助。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
tjz构思并设计了这些实验。b.k., z.l.w., Y.L.Z.和W.L.Y.进行了实验。B.K. & J.Y.F分析了数据并起草了手稿。T.J.Z. & J.Y.F修改了论文。作者阅读并批准了最后的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
不适用。gydF4y2Ba
发表同意书gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明无利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1gydF4y2Ba
:gydF4y2Ba表S1。gydF4y2Ba在4种环境中评估YHSBLP中SW的描述性统计。gydF4y2Ba
附加文件2gydF4y2Ba
:gydF4y2Ba表S2。gydF4y2Ba通过单位点模型和多位点模型鉴定出数量性状核苷酸与百粒重显著相关。gydF4y2Ba表S3。gydF4y2Ba8种GWAS模型的两两比较。gydF4y2Ba
附加文件3gydF4y2Ba
:gydF4y2Ba图S1。gydF4y2Ba利用CMLM (PCA + K)对E1 (A)、E2 (B)、E3 (C)和E4 (D) 573条HSW的GWAS进行曼哈顿图(左)和qq图(右)分析。4的阈值在曼哈顿地块中用蓝线表示。x轴表示染色体数目,y轴表示−gydF4y2Ba日志gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba).QQ图中的X轴和Y轴表示期望和观察到的−gydF4y2Ba日志gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba),分别。iq图中带阴影区域的红线表示95%的置信区间。gydF4y2Ba
附加文件4gydF4y2Ba
:gydF4y2Ba图S2。gydF4y2Ba利用MLM (Q + K)对E1 (A)、E2 (B)、E3 (C)和E4 (D)的573个HSW接入的GWAS进行了Manhattan图(左)和qq图(右)。4的阈值在曼哈顿地块中用蓝线表示。x轴表示染色体数目,y轴表示−gydF4y2Ba日志gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba).QQ图中的X轴和Y轴表示期望和观察到的−gydF4y2Ba日志gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba),分别。iq图中带阴影区域的红线表示95%的置信区间。gydF4y2Ba
附加文件5gydF4y2Ba
:gydF4y2Ba图S3。gydF4y2Ba在模型之间检测到的共同显著snp数量。(A)两种单位点模型(mlm -蓝色和cmlm -棕色)检测到的一些常见snp。(B) 2个单位点模型(MLM和cmlm -蓝色)和6个多位点模型(mrMLM、FASTmrMLM、FASTEMMA、pLARmEB、pKWmEB和ISIS em - blasso -棕色)检测到的一些常见snp。gydF4y2Ba
附加文件6gydF4y2Ba
:gydF4y2Ba表S4。gydF4y2Ba稳定snp的潜在候选基因。gydF4y2Ba表S5gydF4y2Ba。从SoyBase获得候选基因在不同组织和种子发育阶段的表达。gydF4y2Ba表S6。gydF4y2Ba与种子发育相关的顺式调控元件。gydF4y2Ba
附加文件7gydF4y2Ba
:gydF4y2Ba无花果。S4gydF4y2Ba。本研究预测的候选基因和从SoyNet获得的其他相关基因的功能基因网络。(A).从Cytoscape软件独立版本获得的密集相互作用网络。(B).在NetworkAnalyst版本3中可视化的4-hub基因和其他基因。节点颜色表示交互程度之间:红色、粉色、紫色和蓝色分别表示非常高、高、中等和低交互水平。gydF4y2Ba
附加文件8gydF4y2Ba
:gydF4y2Ba表S7。gydF4y2BaqRT-PCR引物。gydF4y2Ba
附加文件9gydF4y2Ba
:gydF4y2Ba表S8。gydF4y2Ba本研究中使用的573份材料的表型数据(平均值)。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba。创作共用公共领域免责声明(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非在数据的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
王志强,王志强,周勇。gydF4y2Baet al。gydF4y2Ba大豆种子重数量性状核苷酸和候选基因的全基因组关联研究。gydF4y2BaBMC Plant BiolgydF4y2Ba20.gydF4y2Ba404(2020)。https://doi.org/10.1186/s12870-020-02604-zgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-020-02604-zgydF4y2Ba
关键字gydF4y2Ba
- 关联映射gydF4y2Ba
- 复杂的自然gydF4y2Ba
- 单体型gydF4y2Ba
- 中心的基因gydF4y2Ba
- Co-expression网络gydF4y2Ba