- 研究文章gydF4y2Ba
- 开放访问gydF4y2Ba
- 发布:gydF4y2Ba
全基因组关联研究确定了与大豆籽粒油酸含量相关的候选基因gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba20.gydF4y2Ba文章编号:gydF4y2Ba399.gydF4y2Ba(gydF4y2Ba2020gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
大豆油是五个脂肪酸(棕榈酸,硬脂酸,油酸,亚油酸和亚麻酸)的复杂混合物。具有高油酸含量的大豆油是理想的,因为这种单不饱和脂肪酸改善油的氧化稳定性。探讨大豆种子油酸的遗传结构,从中国东北260个大豆种质收集自然种群。全基因组关联研究(GWAS)中的溶液的260个种质资源一个面板上进行的。gydF4y2Ba
结果gydF4y2Ba
表型鉴定结果表明,油酸含量在8.2 ~ 35.0%之间。共获得单核苷酸多态性(SNP)标记2,311,337个。GWAS分析表明,与油酸含量相关的基因较多,贡献率为7%。候选基因gydF4y2Baglyma.11g229600.1gydF4y2Ba11号染色体上,并gydF4y2BaGlyma.04G102900.1gydF4y2Ba在长达2年的GWAS中检测到4号染色体。候选基因gydF4y2Baglyma.11g229600.1gydF4y2Ba显示出与油酸含量正相关,相关系数为0.980,而gydF4y2BaGlyma.04G102900.1gydF4y2Ba呈负相关,相关系数为−0.964。gydF4y2Ba
结论gydF4y2Ba
Glyma.04G102900.1gydF4y2Ba在4和染色体上gydF4y2Baglyma.11g229600.1gydF4y2Ba在两项分析(2018年和2019年)中都检测到了11号染色体。gydF4y2BaGlyma.04G102900.1gydF4y2Ba和gydF4y2Baglyma.11g229600.1gydF4y2Ba是大豆种子中与油酸相关的新的关键候选基因。这些结果对高油大豆的育种具有一定的指导意义。gydF4y2Ba
背景gydF4y2Ba
大豆[gydF4y2Ba大豆gydF4y2Ba(l)美林]发源于中国,已有3000多年的栽培历史[gydF4y2Ba1gydF4y2Ba].大豆油占全球脂肪和油脂总产量的20-25%,食用植物油总产量的30-35% [gydF4y2Ba2gydF4y2Ba].在中国,大豆油是饮食的重要组成部分,被认为是维持人群健康的主要因素。大豆油是五种脂肪酸(棕榈酸、硬脂酸、油酸、亚油酸和亚麻酸)的复杂混合物,所有这些脂肪酸具有不同的熔点、氧化稳定性和化学功能[gydF4y2Ba3.gydF4y2Ba].gydF4y2Ba
大豆油的脂肪酸组成约为5 ~ 11%的亚麻酸、43 ~ 56%的亚油酸、15 ~ 33%的油酸和11 ~ 26%的饱和酸[gydF4y2Ba4gydF4y2Ba].棕榈酸和硬脂酸的饱和脂肪酸在构成大豆油15%。最近,出现了一个正在运行的争论,主要集中在主流文学,关于棕榈酸和硬脂酸消费对心脏功能的影响,尤其是在冠状动脉疾病的发展[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba].亚麻酸和亚油酸是多不饱和脂肪酸,占大豆油的80%。亚麻酸是人类正常生长和发育所必需的,可以降低血液中的胆固醇含量,但这种酸不耐高温。大气中的氧气和紫外线会氧化亚麻酸,产生大豆油的气味,降低大豆油的营养价值。油酸是一种单不饱和脂肪酸,油酸含量高的大豆种子也表现出减少或消除化学氢化过程,降低了大豆油加工成本[gydF4y2Ba7gydF4y2Ba].大豆品种具有高油酸含量的种植已成为高品质的大豆育种的重要目标[gydF4y2Ba8gydF4y2Ba].gydF4y2Ba
全基因组关联分析(GWAS)提供了一个强有力的工具,将这一性状与潜在的遗传学联系起来。随着新一代测序技术的快速发展,GWAS已成功应用于水稻和水稻等植物gydF4y2Ba拟南芥gydF4y2Ba[gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10.gydF4y2Ba].大量与复杂性状相关的遗传变异已经被GWAS方法鉴定出来[gydF4y2Ba11.gydF4y2Ba].以大豆为研究对象,利用GWAS技术对298份大豆种质材料中籽粒蛋白质和籽粒含油量变化较大的籽粒含油量进行了定量性状位点(qtl)鉴定。gydF4y2Ba12.gydF4y2Ba].建立大豆育种种质群体(279个品系)进行GWAS分析,发现8个qtl可解释6.3 ~ 26.3%的表型变异[gydF4y2Ba13.gydF4y2Ba].这些结果表明,利用GWAS和专门设计的作图群体可以有效地揭示关键农艺性状的基础。gydF4y2Ba
科学家们已经成功地使用GWAS方法获得了大量的候选基因[gydF4y2Ba14.gydF4y2Ba,gydF4y2Ba15.gydF4y2Ba,gydF4y2Ba16.gydF4y2Ba].然而,在发现新的候选基因后,验证其生物学功能成为研究的热点;RNA干扰技术、生物芯片的建立与应用、实时荧光定量PCR技术(qRT-PCR)、基因编辑技术等为候选基因功能验证提供了理论依据。本研究选取了2个关键候选基因,并利用qRT-PCR技术对14个不同大豆品系的根、茎、叶和种子4个不同组织(根、茎、叶和种子)进行了表达检测。gydF4y2Ba
具体而言,来自中国东北(黑龙江省,吉林省和辽宁省)的260种大豆种质被收集为自然人。从2018年到2019年吉林农业大学种植大豆线。大豆种子中的脂肪酸含量,由NIRS DS 2500仪器在收获后确定。使用特异性基因座扩增的片段测序(SLAF-SEQ)技术用于序列260大豆质材料的基因组,并且Gwas用于筛选与大豆油酸含量有关的候选基因。gydF4y2Ba
结果gydF4y2Ba
大豆种子油酸含量的表型鉴定gydF4y2Ba
2018 - 2019年,采用SPSS 22.0软件对各大豆品系油酸含量进行分析。种子的油酸含量接近正态分布。gydF4y2Ba1gydF4y2Baa,b)。大豆种子的油酸含量的标准偏差(SD)是在2018年6.7和在2019 7.5(图gydF4y2Ba1gydF4y2Bac)。正常分布是描述如何分布变量的值的概率函数。它是一个对称的分布,其中大多数观察结果围绕中心峰值,以及概率,用于在两个方向上同样地远离平均逐渐切断的值。结果还表明,不同大豆线中的油酸含量显着不同,大豆种子中油酸含量的分布是连续的,这与定量性状的遗传法一致。大豆线Q070中的油酸含量在2019年为35.32%,2018年的34.84%,这是所有大豆线中最高的。大豆系Q024中的油酸含量为2018年为5.49%,2019年为6.33%,这是所有大豆线中最低的。gydF4y2Ba
大豆脂肪酸相关分析及遗传力计算gydF4y2Ba
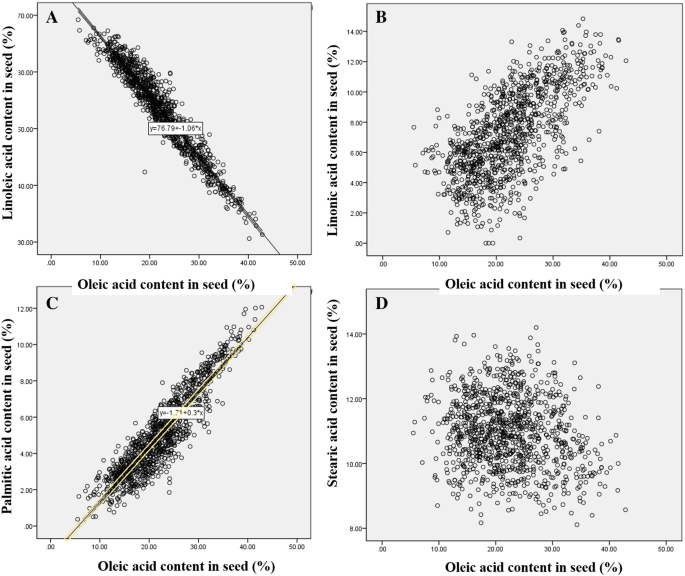
多重线性回归模型来分析不同的脂肪酸之间的关系。根据标准化的多元线性回归模型的回归系数,所述大豆油酸含量显著负与亚油酸含量(图相关。gydF4y2Ba2gydF4y2Ba).油酸与亚油酸的相关系数为−0.660gydF4y2Ba1gydF4y2Ba).有大豆油酸含量和棕榈酸含量之间的显著正相关,具有0.581的相关系数。的在260个大豆品系的五个脂肪酸的遗传力为不同的,并且油酸的遗传力为0.652(附加文件gydF4y2Ba1gydF4y2Ba:表S1)。gydF4y2Ba
SNP基因分型和SNP注释gydF4y2Ba
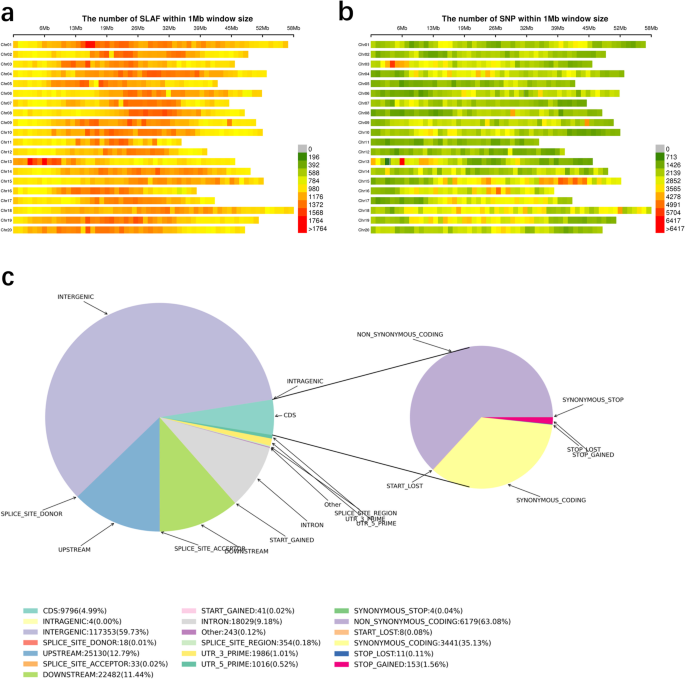
本实验采用SLAF-seq技术对大豆基因组DNA进行测序。共获得1,102,987个SLAF标记和2,311,337个SNP标记(图)。gydF4y2Ba3.gydF4y2Baa, b). SNP在染色体上的分布结果如图所示。gydF4y2Ba3.gydF4y2Bab.根据SNP位点在参考基因组中的位置信息(CDS区、基因区或基因间区),预测突变位点(非同义突变)。超过50%的SNPs位于基因间区域(位于基因之间的DNA序列延伸)。10%的SNP标记位于基因的上游区域,10%的SNP标记位于基因的下游区域。4.99%的SNP位点位于蛋白编码区。9%的SNP标记位于内含子中。gydF4y2Ba3.gydF4y2Bac)。gydF4y2Ba
系统发育分析、遗传结构分析、主成分分析(PCA)gydF4y2Ba
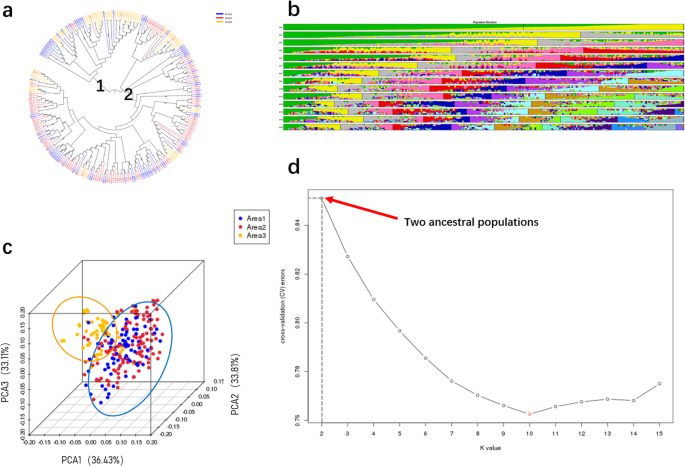
从系统发育树上,可以得出结论,大豆线起源于两个大分支。这结果表明,260个大豆线来自同一个祖先。然而,在进化过程中,它们在两个方向上进化(图。gydF4y2Ba4gydF4y2Baa)。gydF4y2Ba
群体结构分析可以量化研究群体的祖先数量,推断每个样本的来源。它是目前应用广泛的聚类分析方法,有助于了解基于SNPs的材料进化过程。利用本试验对大豆群体结构进行了分析。对于研究群体,本试验预置的亚群体数量为15个(图2)。gydF4y2Ba4gydF4y2Bab)。我们分析了eigenstrat的数据,用于研究260个大豆线。得出结论是,我们收集的样品可以代表为两个祖先群体的混合物(图。gydF4y2Ba4gydF4y2Bad)。gydF4y2Ba
基于SNPs的差异,利用EIGENSOFT软件进行主成分分析,对260份大豆材料进行聚类。主成分分析结果表明,260个大豆品系聚在一起,形成两个亚群gydF4y2Ba2gydF4y2Ba:图S1)。PC1、PC2和PC3分别占36.43、33.82和33.12%。gydF4y2Ba4gydF4y2Bac)。gydF4y2Ba
种子油酸含量的全基因组关联研究gydF4y2Ba
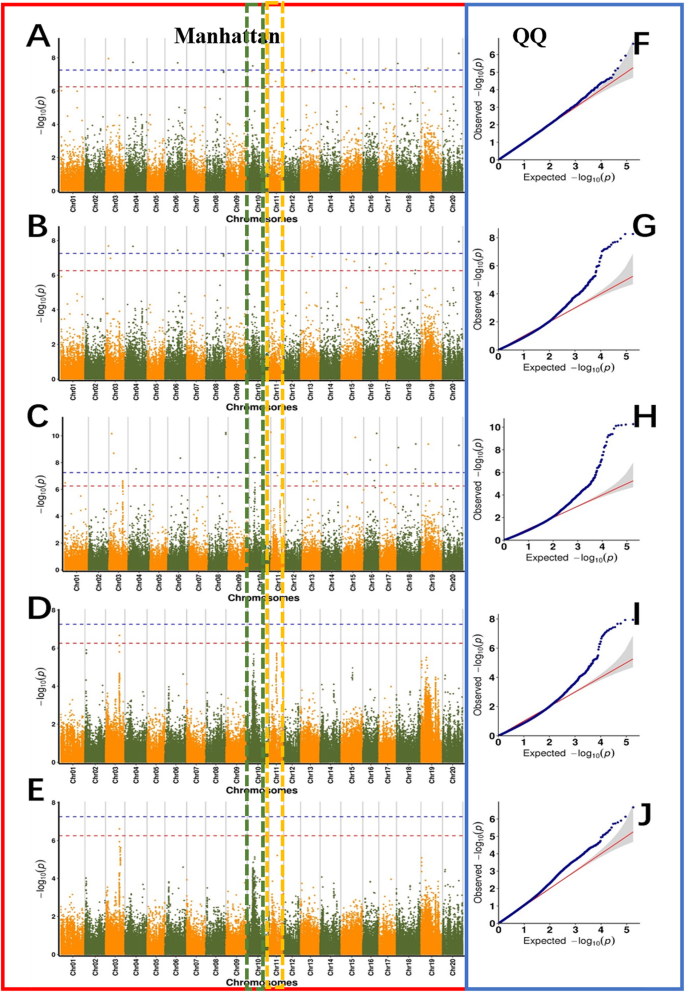
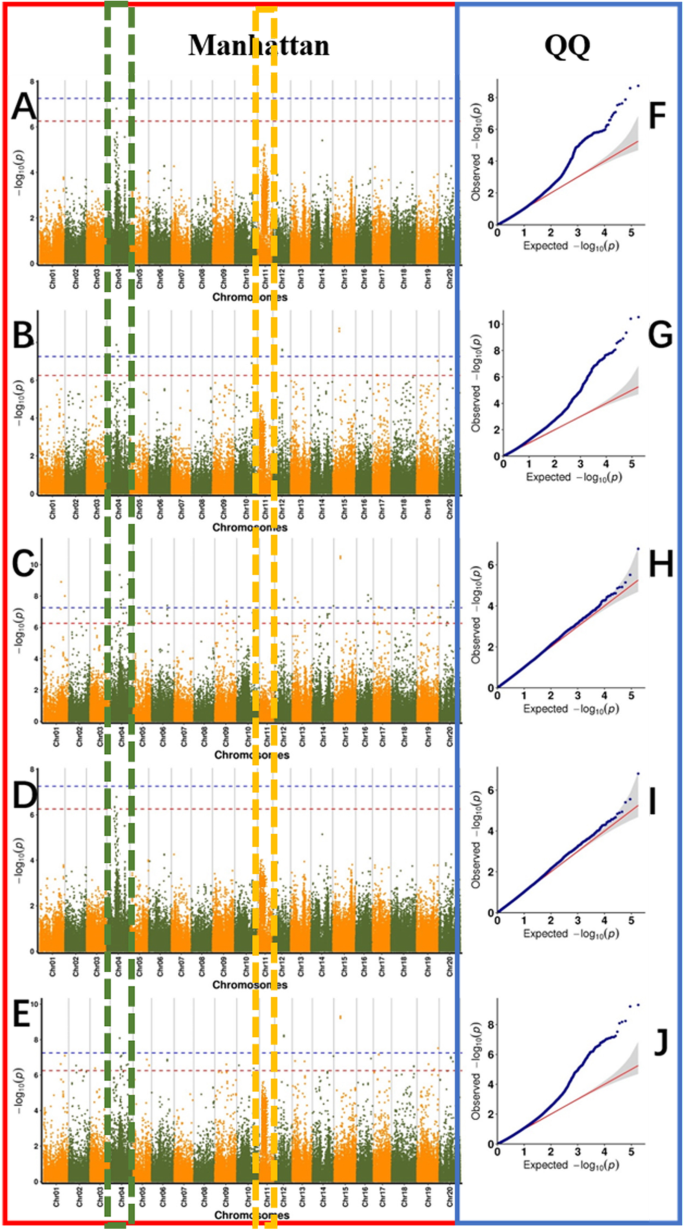
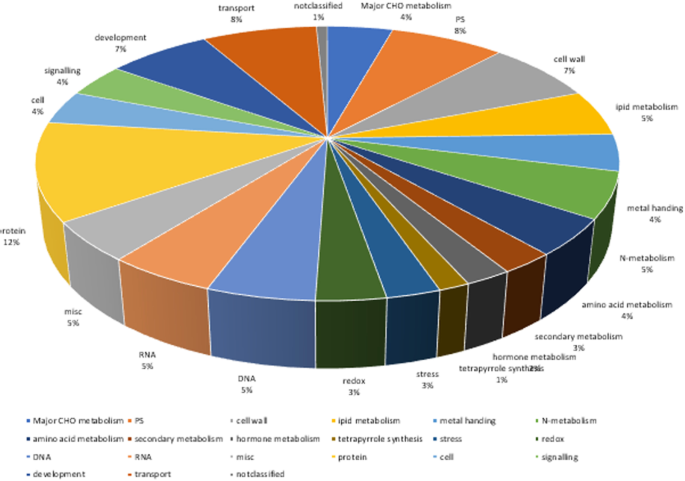
基于260豆系的油酸含量,使用Tassel软件(GLM型号,MLM模型,CMLM型号),FastLMMC软件和EMMAX软件用于GWA。检测与大豆种子的油酸含量显着相关的SNP标记,并将连接不平衡(LD)距离设定为8.9kb。2018年和2019年的油酸含量的曼哈顿和QQ(定量定位率)图如图1所示。gydF4y2Ba5gydF4y2Ba和图。gydF4y2Ba6gydF4y2Ba.对与大豆籽粒油酸含量显著相关的SNP标记进行了搜索gydF4y2Ba3.gydF4y2Ba:表S2)。在这项研究中,我们在260张加入中评估了基因组LD,发现LD(rgydF4y2Ba2gydF4y2Ba)值衰减到9.5 kb内最大值的一半(附加文件gydF4y2Ba4gydF4y2Ba:图S2)。以9.7 kb为连锁不平衡衰减距离,在LD距离内筛选与大豆油酸性状相关的候选基因。2018年,通过全基因组关联分析,筛选出21个与大豆油酸含量相关的候选基因(表1)gydF4y2Ba2gydF4y2Ba).2019年,通过基因组关联分析筛选8个候选基因(表。2)。基于GO术语,基因的功能如下:1。主要CHO新陈代谢(GO:0004527),2.细胞壁(GO:0009058),3.脂质代谢(GO:0005737),4.金属离子结合(GO:0046872),5. n新陈代谢(GO:0006499),氨基酸代谢(GO:0008152),7.次生新陈代谢(GO:0016491),8.压力(GO:0034976),9.氧化还原(ECO:0000313),10. MISC。(GO:0004526),11.蛋白质(GO:0004674),12.单元格(GO:0051510),13.信令(GO:0009862),14.发展(GO:0007275)和15.运输(GO:0003712).候选基因的功能分布如图1所示。gydF4y2Ba7gydF4y2Ba.第一个有希望的候选基因,gydF4y2BaGlyma.11G229600.1,gydF4y2Ba位于11号染色体上,通过GWAS期间2018和2019,其功能是不是在大豆数据库注释的检测。据Swissprot可批注,则gydF4y2Baglyma.11g229600.1gydF4y2Ba基因属于植物BAG蛋白家族。第二个有希望的候选基因,gydF4y2BaGlyma.04G102900.1,gydF4y2Ba位于4染色体上,在2年内也被GWA检测到。同样,根据Swissprot注释,没有关于大豆数据库中该候选基因的功能的报道。类似的基因gydF4y2Ba拟南芥gydF4y2Ba属于植物GRAS蛋白家族。gydF4y2Ba
在2018年的分析中,曼哈顿全基因组图谱与油酸含量相关。在左图中,x轴表示每条染色体上的SNPs;y轴为- log 10 (gydF4y2BaPgydF4y2Ba- 对于关联,阈值设置为-log(gydF4y2BapgydF4y2Ba)> 6.20(红色)和-log(gydF4y2BapgydF4y2Ba) > 7.20(蓝色)。gydF4y2Ba一个gydF4y2Ba到目前为止gydF4y2BaegydF4y2Ba基于CMLM模型,EMMAX模型,FastLMM模型,GLM和MLM分别是F到J的GWAS结果。gydF4y2BaegydF4y2Ba基于传销模型的GWAS结果。cmlm模型油酸的Q-Q图(gydF4y2BafgydF4y2Ba)、EmMax模型(gydF4y2BaggydF4y2Ba)、fastlmm模型(gydF4y2BahgydF4y2Ba),GLM(gydF4y2Ba我gydF4y2Ba),传销(gydF4y2BajgydF4y2Ba).灰色区域表示95%的浓度频带。每个点代表一个SNPgydF4y2Ba
2019年分析的全基因组曼哈顿油酸含量关联图在左图中,x轴表示每条染色体上的SNPs;y轴为关联的−log 10 (P-value),阈值设置为-log (gydF4y2BapgydF4y2Ba)> 6.20(红色)和-log(gydF4y2BapgydF4y2Ba) > 7.20(蓝色)。gydF4y2Ba一个gydF4y2Ba基于CMLM模型的GWAS结果,gydF4y2BabgydF4y2Ba基于EmMax模型的GWAS结果,gydF4y2BacgydF4y2Ba基于FastLMM模型的GWAS结果,gydF4y2BadgydF4y2Ba基于GLM模型的GWAS结果,gydF4y2BaegydF4y2Ba基于传销模型的GWAS结果。cmlm模型油酸的Q-Q图(gydF4y2BafgydF4y2Ba)、EmMax模型(gydF4y2BaggydF4y2Ba)、fastlmm模型(gydF4y2BahgydF4y2Ba),GLM(gydF4y2Ba我gydF4y2Ba),传销(gydF4y2BajgydF4y2Ba).灰色区域表示95%的浓度频带。每个点代表一个SNPgydF4y2Ba
在不同组织的两个候选基因的表达gydF4y2Ba
为了验证其与油酸含量的关系,通过使用QRT-PCR在不同的组织(根,茎,叶和种子)中测量两种选定的候选基因的表达。当gydF4y2Ba植物血凝素gydF4y2Ba基因(GenBank: A5547-127)作为内参基因。结果表明候选基因gydF4y2Baglyma.11g229600.1gydF4y2Ba在不同组织中均有表达,但相对表达量差异显著,叶片为1.23 ~ 4.31,茎为10.21 ~ 39.56,根为16.21 ~ 43.14。gydF4y2Ba8gydF4y2Baa)候选基因gydF4y2Baglyma.11g229600.1gydF4y2Ba大豆品系q001叶片中相对表达量最低,为1.23,油酸含量最低。的相对表达水平gydF4y2Baglyma.11g229600.1gydF4y2Ba大豆品系q001的种子产量最低,为25.26粒。候选基因的相对表达量gydF4y2Baglyma.11g229600.1gydF4y2Ba在大豆线的叶子中,Q353比大豆线Q001叶片高4.3倍,种子的水平比叶子中的水平高10倍(附加文件gydF4y2Ba5gydF4y2Ba:图。S3)。通常,相关系数之间gydF4y2Baglyma.11g229600.1gydF4y2Ba油酸含量为0.980 ~ 0.994 (gydF4y2BaP < 0.01gydF4y2Ba)(附加文件gydF4y2Ba6gydF4y2Ba:表。S3)。这一结果有力地表明候选基因gydF4y2Baglyma.11g229600.1gydF4y2Ba对调节种子中油酸含量有积极作用。gydF4y2Ba
的相对表达gydF4y2BaGlyma.04G102900.1gydF4y2Ba还在大豆叶,茎,根和种子中分析。的相对表达水平gydF4y2BaGlyma.04G102900.1gydF4y2Ba叶的变化范围为9.62 ~ 44.41,茎的变化范围为3.18 ~ 28.11。gydF4y2Ba8gydF4y2Bab).具体而言,在种子中油酸含量最低的大豆品系q001中,有候选基因gydF4y2BaGlyma.04G102900.1gydF4y2Ba显示出最高的表达水平,这是49.01(附加文件gydF4y2Ba7gydF4y2Ba:图。S4)。一般来说,相对表达水平gydF4y2BaGlyma.04G102900.1gydF4y2Ba在不同的组织中差异显著(gydF4y2BaP < 0.05gydF4y2Ba),相关系数之间gydF4y2BaGlyma.04G102900.1gydF4y2Ba油酸含量为−0.964 ~−0.998(附加文件gydF4y2Ba8gydF4y2Ba:表。S4)。这一结果表明gydF4y2BaGlyma.04G102900.1gydF4y2Ba与油酸含量密切相关,特别是对种子中油酸含量的负面影响。gydF4y2Ba
讨论gydF4y2Ba
大豆油是由五个脂肪酸:棕榈酸(16:0),硬脂酸(18:0),油酸(18:1),亚油酸(18:2)和亚麻酸(18:3)[gydF4y2Ba17.gydF4y2Ba[豆油平均值10,4,18,55和13%的豆油平均值10,4,18,55和13%的这五种脂肪酸的百分比。发现不同大豆品种的谷物中的油酸含量变化大大变化[gydF4y2Ba18.gydF4y2Ba].2016年,日本科学家收集了319个日本大豆品种;319份材料籽粒油酸含量为7.66 ~ 15.86%,101份材料籽粒油酸含量为11.5% [gydF4y2Ba19.gydF4y2Ba].2008年,对来自不同地区的栽培大豆和野生大豆种质进行了脂肪酸含量分析。结果表明:栽培大豆的平均脂肪含量为17.21%,比野生大豆高6.22%;栽培大豆油酸含量为23.25%,比野生大豆高7.75%;亚油酸含量为53.53%,比野生大豆低2.57% [gydF4y2Ba20.gydF4y2Ba].在本研究中,油酸含量为13.5 ~ 38.4%。结果还表明,大豆种质平均油酸含量差异较大,大豆籽粒油酸含量在不同地区间存在显著差异。在Kurt的研究中,相关分析清楚地表明油酸与亚油酸存在显著的负相关关系(gydF4y2BargydF4y2Ba = − 0.701,PgydF4y2Ba < 0.0001) and stearic acid (rgydF4y2Ba = − 0.218,PgydF4y2Ba< 0.001),硬脂酸与油酸和花生酸显著正相关,而硬脂酸与亚油酸和亚麻酸均呈负相关[gydF4y2Ba21.gydF4y2Ba].在我们的研究中,油酸含量也与亚油酸(0.454)显着呈正相关(0.454)。结果表明,油酸和亚油酸之间的关系可能有助于评估富含油酸的品种。gydF4y2Ba
Glyma.11G229600.1,gydF4y2Ba位于第11号染色体上,于2018年和2017年被GWAS同时检测到。gydF4y2Baglyma.11g229600.1gydF4y2Ba属于植物BAG蛋白家族。BAG蛋白是一个广泛保守的基因家族,其同源基因跨越了广泛的进化距离,包括酵母、动物和植物[gydF4y2Ba22.gydF4y2Ba].研究表明,袋子蛋白也存在gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba[gydF4y2Ba23.gydF4y2Ba].BAG蛋白家族在植物生长发育中起着重要的作用。过表达BAG7可增加植物对温度的敏感性,而BAG4编码抗凋亡基因,据报道,这些基因可使转基因烟草耐盐和干旱胁迫[gydF4y2Ba24.gydF4y2Ba].不同生长阶段的干旱治疗也有助于脂肪酸的差异[gydF4y2Ba25.gydF4y2Ba].干旱胁迫显著影响脂肪酸组成和氨基酸组成[gydF4y2Ba26.gydF4y2Ba].严重干旱使蛋白质含量增加4.4个百分点,油分含量减少2.9个百分点。随着干旱胁迫的增加,随胁迫天数的增加,蛋白质含量呈线性增加,含油量呈下降趋势[gydF4y2Ba27.gydF4y2Ba].可以推测这一点gydF4y2Baglyma.11g229600.1gydF4y2Ba可能增加大豆的耐旱性,从而影响油酸在大豆种子中的积累。gydF4y2Ba
Glyma.04G102900.1gydF4y2Ba属于植物GRAS蛋白家族。GRAS蛋白是一个重要的植物特异性蛋白家族,以发现的前三个成员命名:赤霉素不敏感蛋白(GAI)、赤霉素抑制蛋白(RGA)和稻草人蛋白(SCR)。至少有33个GRAS基因已被鉴定gydF4y2BaA. Thaliana.gydF4y2Ba和大米(gydF4y2Ba28.gydF4y2Ba].最近在脉冲中发现了两种GRAS结构域蛋白[gydF4y2Ba29.gydF4y2Ba].根瘤菌通过脂质寡糖信号分子Nod因子进入与豆类共生的相互作用,在根中激活各种反应。事实上,一项研究表明,GRAS蛋白在植物中转导钙信号,并可能作为nod因子诱导基因表达的调节器[gydF4y2Ba30.gydF4y2Ba].在该研究中,我们通过QRT-PCR研究了两种候选基因的两种候选基因的表达,发现候选基因的表达在这些线中变化。我们在2018年和2019年分析中发现了两种与大豆种子中的油酸含量相关的两种基因。这是第一次关键基因gydF4y2BaGlyma.04G102900.1gydF4y2Ba和gydF4y2Baglyma.11g229600.1gydF4y2Ba已有报道与油酸含量有关。因此,需要进一步的研究来支持这一发现。本研究结果为阐明大豆脂肪酸组成测定的机理提供了依据。此外,本文鉴定的SNP标记表明,标记辅助选择是鉴定大豆相关基因的一种强有力的策略,可用于优化种子脂肪酸谱的育种计划。gydF4y2Ba
结论gydF4y2Ba
本研究采用全基因组关联研究(GWAS)技术寻找与油酸含量相关的SNP标记。2018年共检测到20个与油酸含量相关的新候选基因,2018年共检测到8个与油酸含量相关的新候选基因。gydF4y2BaGlyma.04G102900.1gydF4y2Ba在4和染色体上gydF4y2Baglyma.11g229600.1gydF4y2Ba在两项分析(2018年和2019年)中都检测到了11号染色体。gydF4y2BaGlyma.04G102900.1gydF4y2Ba和gydF4y2Baglyma.11g229600.1gydF4y2Ba是大豆种子中与油酸相关的新的关键候选基因。gydF4y2Ba
方法gydF4y2Ba
植物材料gydF4y2Ba
将由吉林农业大学生物技术中心提供的260份大豆材料于2018 - 2019年(共2年)在吉林农业大学(中国长春)试验田进行种植。采用随机完全区组设计。每个大豆品系采用3个生物学重复进行检验。该油田被划分为三个260米的区块gydF4y2Ba2gydF4y2Ba(26 × 10 m),每个区块又细分为8个区段。每个节又细分为260个小节。自然干燥(阳光)发生,然后,种子脱粒,以确定油酸。选择油酸水平差异显著的14个大豆品种,检测候选基因的表达。大豆品系名称及脂肪酸含量见表gydF4y2Ba3.gydF4y2Ba.gydF4y2Ba
脂肪酸水平的大豆种子中的测定gydF4y2Ba
采用NIRSTM DS 2500仪器(FOSS, Hillerod, Denmark)测定大豆种子收获后油酸和硬脂酸、软脂酸、亚油酸和亚麻酸的含量。采用SPSS 22.0软件(SPSS Inc., Chicago, IL, USA)计算大豆种子中脂肪酸的相关系数。gydF4y2Ba
大豆种质资源的基因分型gydF4y2Ba
根据Murray & Thompson(1980)的研究,采用CTAB法从每个大豆品系的叶片中提取总基因组DNA [gydF4y2Ba31.gydF4y2Ba].通过特异性轨迹扩增的片段测序(SLAF-SEQ)进行260种大豆材料,并开发SNP分子标记。DNA提取是测序的第一步。SNP分子标记用于系统发育分析和遗传进化相关分析。限制性内切核酸酶组合是gydF4y2BaRSAI-HaeIIIgydF4y2Ba.北京生物标志物生物科技有限公司提供测序服务,PR中国。gydF4y2Ba
人口结构评价gydF4y2Ba
利用EIGENSOFT软件包,采用主成分分析(PCA)对种群结构进行评估。基于邻域连接方法,利用MEGA5软件构建包含各样本的系统发育树。gydF4y2Ba
全基因组关联研究gydF4y2Ba
以SLAF-Seq技术获得的SNP标记为基础,利用TASSEL软件中的5个模型(glm、mlm、cmlm、fastlmm和emmax),得到SNP标记与油酸含量的相关值。TASSEL软件可以根据样本总体结构计算Q矩阵gydF4y2BaKgydF4y2Ba矩阵最终获得每个SNP标记的相关值。每个特征的每个模型的结果基于0.000001的显着性注释。在该实验中,使用HAPLOVIEW软件构建曼哈顿地图和QQ地图。曼哈顿地图用于表示基因型数据和表型数据之间的相关性。QQ MAP用于表示观察和预测值之间的差异。在该研究中,通过使用SWISS-PROD和NR数据库来预测候选基因。gydF4y2Ba
定量逆转录PCRgydF4y2Ba
使用Bio-Rad CFX系统(Amersham Biosciences,Little Chalfont,Buckinghamshire,UK)进行QRT-PCR分析。使用Eastep®超级RNA提取套件(Takara公司,Kusatsu,Shiga,日本)提取总RNA。扩增反应条件如下:在95℃下预先饱和10分钟,在95℃下变性10 s,然后在53℃下退火20s,并在72℃下延伸15秒。扩增反应条件gydF4y2Baglyma.11g229600.1gydF4y2Ba基因分别为:95℃处理10 min;95°C变性30 s, 67°C退火30 s, 72°C延伸30 s共35个循环;72°C拉伸10分钟。以上反应均包含40个循环。扩增后,用2gydF4y2Ba——ΔΔCtgydF4y2Ba方法[gydF4y2Ba32.gydF4y2Ba].基因扩增反应条件gydF4y2BaGlyma.04G102900.1gydF4y2Ba95℃预变性10 min;95°C变性30 s, 59°C退火30 s, 72°C延伸35 s共35个循环;72°C拉伸10分钟。每个基因使用3个生物重复。gydF4y2Ba
数据分析gydF4y2Ba
采用Microsoft Excel 2010软件测量并记录表型数据。采用SPSS 19.0 (IBM Corp, Armonk, NY, USA)软件进行差异显著性分析、方差分析、相关分析和描述性分析[gydF4y2Ba33.gydF4y2Ba].在正面和负面的地图和直方图被使用GraphPad Prism软件(GraphPad公司,圣地亚哥,CA)构成。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
支持这项研究结果的原始序列信息可从[北京生物标志物生物科技有限公司,北京,北京,中国北京,但限制适用于这些数据的可用性,这些数据是根据当前研究的许可使用的,所以不公开。但是,在合理的请求和[北京生物标志物生物科技有限公司,北京,中国]的许可中获得数据。支持本文结论的其他数据集包含在文章及其附加文件中。gydF4y2Ba
缩写gydF4y2Ba
- SNP:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
- SLAF-SEQ:gydF4y2Ba
-
特异性位点扩增片段测序gydF4y2Ba
- GWAS:gydF4y2Ba
-
全基因组关联研究gydF4y2Ba
- LD:gydF4y2Ba
-
连锁不平衡gydF4y2Ba
- 存在:gydF4y2Ba
-
实时定量聚合酶链反应gydF4y2Ba
- 米德:gydF4y2Ba
-
厘摩gydF4y2Ba
- QTL:gydF4y2Ba
-
数量性状位点gydF4y2Ba
- PCA:gydF4y2Ba
-
主要成分分析gydF4y2Ba
- QQ:gydF4y2Ba
-
Quantile-quantilegydF4y2Ba
参考资料gydF4y2Ba
- 1。gydF4y2Ba
关键词:大豆,种植行为,影响因素,黑龙江省农业。2019;9(9):188。gydF4y2Ba
- 2。gydF4y2Ba
Tunde-Akintunde T,Olajide J,Akintunde B.大众体积区与水分和品种的函数相关的大豆和机械性能。int J Food Prop。2005; 8(3):449-56。gydF4y2Ba
- 3.gydF4y2Ba
赵晓霞,常洪,冯丽丽,景勇,滕伟,邱林,等。大豆种子饱和脂肪酸全基因组关联定位及候选基因分析。植物品种。2019;138(5):588 - 98。gydF4y2Ba
- 4。gydF4y2Ba
柯林斯F,Sedgwick V.脂肪酸组成的几种大豆。Ĵ上午石油化学会志。1959; 36(12):641-4。gydF4y2Ba
- 5。gydF4y2Ba
Patil S,Balu D,Melrose J,Chan C.脑区特异性与阿尔茨海默病相关的棕榈酸诱导的异常。BMC RES笔记。2008; 1(1):20。gydF4y2Ba
- 6。gydF4y2Ba
门辛克RP,Zock PL,凯斯特AD,卡坦MB。60个对照试验的荟萃分析:的膳食脂肪酸和碳水化合物对血清总的以HDL胆固醇和血脂和载脂蛋白的比率的影响。牛J临床营养学。2003; 77(5):1146至1155年。gydF4y2Ba
- 7。gydF4y2Ba
宣威E.氧化和油脂加工的抗氧化剂。Ĵ上午石油化学会志。1978; 55(11):809-14。gydF4y2Ba
- 8。gydF4y2Ba
Combs R,B内利KD。FAD2-1A的新等位基因在大豆油中诱导高水平的油酸。mol繁殖。2019; 39(6):1-11。gydF4y2Ba
- 9.gydF4y2Ba
王旭,庞勇,张建军,吴志,陈凯,叶光,徐建军,李正忠。水稻食性和蒸煮特性的全基因组关联分析。Sci众议员2017;7(1):1 - 10。gydF4y2Ba
- 10.gydF4y2Ba
Verslues PE, Lasky JR, Juenger TE, Liu TW, Kumar MN。全基因组关联定位结合反向遗传学鉴定了低水势诱导的拟南芥脯氨酸积累的新效应。植物杂志。2014;164(1):144 - 59。gydF4y2Ba
- 11.gydF4y2Ba
Brachi B, Morris GP, Borevitz JO。植物的全基因组关联研究:缺失的遗传力正在田间进行。基因组医学杂志。2011;12(10):232。gydF4y2Ba
- 12.gydF4y2Ba
Hwang Ey,Song Q,Jia G,Specht Je,Hyten DL,Costa J等。大豆种子蛋白和油含量的基因组型缔合研究。BMC基因组学。2014; 15(1):1。gydF4y2Ba
- 13。gydF4y2Ba
Cao Y,Li S,Wang Z,Chang F,Kong J,Gai J等。通过组合联系和基因组关联映射来识别大豆种子油含量的主要定量特性基因座。前植物SCI。2017; 8:1222。gydF4y2Ba
- 14。gydF4y2Ba
巴斯蒂安男,SONAH H,菌核病在大豆抗性与基因分型逐测序方法的Belzile F.全基因组关联映射。植物基因组。2014; 7:1。gydF4y2Ba
- 15.gydF4y2Ba
Kaler AS, Ray JD, Schapaugh WT, King CA, Purcell LC。不同基因型大豆冠层萎蔫的全基因组关联定位。中国海洋大学学报。2017;130(10):2203-17。gydF4y2Ba
- 16。gydF4y2Ba
张建军,王旭,卢勇,宋强,Cregan PB,等。对种子组成的全基因组扫描为大豆品质改良和驯化育种的影响提供了深刻的见解。摩尔。2018;11(3):460 - 72。gydF4y2Ba
- 17。gydF4y2Ba
Priolli Rh,Campos JB,Stabellini NS,Pinheiro JB,Vello Na。大豆中油含量和脂肪酸成分的关联映射。Euphytica。2015; 203(1):83-96。gydF4y2Ba
- 18。gydF4y2Ba
Diers BW, Shoemaker R.大豆脂肪酸含量限制性片段长度多态性分析。石油化学学报1992;69(12):1242-4。gydF4y2Ba
- 19.gydF4y2Ba
wei CD, Hashiguchi M, Anai T, Suzuki A, Akashi R.日本野生大豆(Glycine soja)种子脂肪酸组成及分布。中国植物科学(英文版);2017;gydF4y2Ba
- 20。gydF4y2Ba
彭德龙VR,Rebetzke GJ,威尔逊RF,伯顿JW。种子质量和亚麻酸在栽培和野生大豆之间杂交的后代的关系。Ĵ上午石油化学会志。1997; 74(5):563-8。gydF4y2Ba
- 21.gydF4y2Ba
陈志强。不同产地芝麻籽粒油脂含量和脂肪酸组成的变化。它含有或氢化。2018;69(1):241。gydF4y2Ba
- 22.gydF4y2Ba
AtBAG7是一种拟南芥bcl -2相关的athanogene,位于内质网中,参与蛋白质的折叠反应。中国科学(d辑:地球科学)2010;gydF4y2Ba
- 23.gydF4y2Ba
关键词:拟南芥,bag家族,蛋白质植物科学。2003;165(1):1 - 7。gydF4y2Ba
- 24。gydF4y2Ba
Hoang Tm,Moghaddam L,Williams B,Khanna H,Dale J,Mundree SG。通过涉及编程细胞死亡调控的基因组成型过表达的盐度耐受性的发展。前植物SCI。2015; 6:175。gydF4y2Ba
- 25。gydF4y2Ba
Sánchez-MartínJ,Canales FJ,Tweed JK,Lee Mr,Rubiales D,Gómez-Cadenas A等。燕麦逐渐土壤水耗水过程中脂肪酸谱改变表明茉莉酸盐在应对干旱方面的作用。前植物SCI。2018; 9:1077。gydF4y2Ba
- 26。gydF4y2Ba
Dwivedi S,Nigam S,Rao RN,Singh U,Rao K.干旱对油脂,脂肪酸和蛋白质含量的影响(Arachis Hypogaea L.)种子的影响。现场作物RES。1996; 48(2-3):125-33。gydF4y2Ba
- 27。gydF4y2Ba
Dornbos D,Mullen R.豆豆种子蛋白和油含量和脂肪酸组成通过干旱和温度调节。Ĵ上午石油化学会志。1992年; 69(3):228-31。gydF4y2Ba
- 28。gydF4y2Ba
美国好施集团(Hirsch S), Oldroyd GE。调控植物发育的草结构域转录因子。植物学报。2009;4(8):698-700。gydF4y2Ba
- 29。gydF4y2Ba
海克曼AB, Lombardo F, Miwa H, Perry JA, Bunnewell S, Parniske M,等。荷花结瘤需要两个GRAS域调控因子,其中一个在非豆科植物中具有功能保守性。植物杂志。2006;142(4):1739 - 50。gydF4y2Ba
- 30。gydF4y2Ba
斯密特P,RaedtsĴ,Portyanko V,Debellé楼高夫C,Bisseling T,等人。该GRAS蛋白家族的NSP1是根瘤菌结瘤因子诱导的转录至关重要。科学。2005; 308(5729):1789至1791年。gydF4y2Ba
- 31。gydF4y2Ba
默里M,汤普森WF。高分子量植物DNA的快速分离。核酸RES。1980; 8(19):4321-6。gydF4y2Ba
- 32。gydF4y2Ba
Livak KJ,Schmittgen TD。使用实时定量PCR和2相对基因表达数据的分析gydF4y2Ba- ΔΔctgydF4y2Ba方法。方法。2001; 25(4):402-8。gydF4y2Ba
- 33.gydF4y2Ba
verma jp。SPSS软件管理中的数据分析。Springer。柏林:科学与商业媒体;2013年。gydF4y2Ba
致谢gydF4y2Ba
我们感谢吉林农业大学生物技术中心和吉林省农业科学院的慷慨资助,使这项工作成为可能。gydF4y2Ba
资金gydF4y2Ba
国家作物育种重点研发计划项目(2016YFD0100201)资助。资助者参与了研究的设计;数据的收集、分析和解释;以及手稿的写作。gydF4y2Ba
作者信息gydF4y2Ba
隶属关系gydF4y2Ba
贡献gydF4y2Ba
WP和QD设计了实验。QD和XYL计划并执行了实验。CM、AP和QD编辑了手稿。所有作者都对结果进行了讨论,并对手稿进行了评论。所有作者均已阅读并批准本稿件。gydF4y2Ba
相应的作者gydF4y2Ba
伦理宣言gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用。gydF4y2Ba
同意出版gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
提交人声明他们没有竞争利益。gydF4y2Ba
附加信息gydF4y2Ba
出版商的注意事项gydF4y2Ba
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1表S1。gydF4y2Ba
大豆种子脂肪酸含量的遗传力。gydF4y2Ba
附加文件2图S1。gydF4y2Ba
大豆种质资源的群体结构。(PPTX 182 kb)gydF4y2Ba
附加文件3表S2。gydF4y2Ba
在2年内鉴定出与油酸含量相关的snp (gydF4y2BaPgydF4y2Ba < 0.000001).
附加文件4图S2。gydF4y2Ba
全基因组连杆不平衡(LD)腐烂,适用于所有260种载体。(PPTX 223 KB)gydF4y2Ba
附加文件5图S3。gydF4y2Ba
的表达gydF4y2Baglyma.11g229600.1gydF4y2Ba在大豆不同的组织中。(PPTX 354 kb)gydF4y2Ba
附加文件6表S3。gydF4y2Ba
之间的相关性gydF4y2Baglyma.11g229600.1gydF4y2Ba表达量和油酸含量。gydF4y2Ba
附加文件7gydF4y2Ba
的表达gydF4y2BaGlyma.04G102900.11gydF4y2Ba在大豆不同的组织中。(PPTX 233 kb)gydF4y2Ba
附加文件8gydF4y2Ba
之间的相关性gydF4y2BaGlyma.04G102900.1gydF4y2Ba表达量和油酸含量。gydF4y2Ba
权利和权限gydF4y2Ba
开放访问gydF4y2Ba本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.Creative Commons公共领域奉献豁免(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非另有用入数据的信用额度。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用本文gydF4y2Ba
刘旭东,秦德成,杨晓东。gydF4y2Baet al。gydF4y2Ba与在大豆种子的油酸含量全基因组关联研究确定候选基因。gydF4y2BaBMC植物BIOL.gydF4y2Ba20,gydF4y2Ba399(2020)。https://doi.org/10.1186/s12870-020-02607-wgydF4y2Ba
收到gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
迪伊gydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-020-02607-wgydF4y2Ba
关键字gydF4y2Ba
- 大豆gydF4y2Ba
- 油酸gydF4y2Ba
- 单核苷酸多态性gydF4y2Ba
- 全基因组关联研究gydF4y2Ba
- 油酸相关基因gydF4y2Ba