摘要
背景
我国的植甲科植物不仅是观赏植物,而且是与人类健康密切相关的多年生草本植物。然而,植物科成员的大规模全长cDNA测序和内参基因验证仍然缺乏。因此,我们采用单分子实时测序技术在入侵中生成全长转录组商美国和非侵入性外来p . icosandra.基于转录组数据,使用RT-QPCR来评估两种植物物种和另一种土着同胞的基因表达稳定性p . acinosa.
结果
共19.96英镑和19.75 GB清洁阅读p .美国和p . icosandra,其中非嵌合(FLNC)全长分别为200,857和208,865条。FLNC reads的转录本聚类分析鉴定出89,082和98,448个一致亚型,其中86,989和96,764个优质亚型。去除冗余reads后,获得46369和50220份转录本。通过结构分析,共检测到1675个和1908个可变剪接变异,25641个和31800个SSR, 34971个和36841个完整编码序列。预测了3574个和3833个lncRNA,注释了41676个和45,050个转录本。随后,在两种植物和一种本地植物中分别发现7个内参基因p . acinosa采用RT-qPCR进行基因表达分析。在不同的组织(叶、茎、根和花)中进行测试时,18s rrna.表现出最高的稳定性p .美国,是否受感染spodoptera litura与否。EF2有最稳定的表达p . icosandra, 尽管EF1 -α攻击时是最合适的美国litura.EF1 -α表现出最高的稳定性P.acinosa,而GAPDH.被推荐的时候美国litura.而且,EF1 -α仅考虑萌发的种子或花朵时,为三种植物中最稳定的一种。
结论
全长转录组p .美国和p . icosandra单独生产。基于转录组数据,评价7个候选内参基因在不同实验条件下的表达稳定性。这些结果将为进一步探索植物的功能和比较基因组学研究提供参考,并为植物的入侵成功提供依据p .美国.
背景
商美国是Phytolaccaceae家族的成员,是美国原产于东北美国。由于其装饰性和药用应用,它于1935年被引入中国[1].不幸的是,它已经演变为一种入侵物种,并扩散到中国的大部分地区,特别是中国的中部和南部。与非入侵的外来同类比较p . icosandra和原生同胞p . acinosa,p .美国是兴趣的,因为它表现出多种生物学活性,例如植物农药,抗菌性质,重金属积累能力[2,3.,4].
为了研究黄酮的多种生物活性机制p .美国此外,全转录组研究是必要的,以促进。有报道表明茉莉酸诱导和镉处理的转录组数据p .美国是由Illumina Hiseq 2500和Illumina Hiseq 2000平台获得的[5,6].然而,这些数据都是通过第二代测序(SGS)获得的,不能产生全长转录本。基因组数据的p .美国在项目Prjna544344下的SRA上可用,但它是未经编码序列预测和功能注释的原始读取[7].
第三代测序(TGS)是千碱基长读取的,并且是更好地理解RNA处理的优异策略。例如,它可用于分析通过替代剪接调节的不同的转录同种型,这大大增加了蛋白质的曲目,导致遗传和功能性多样性,并且在大多数真核生物中普遍存在[8].长读取还可以提供关于基因编码区的序列信息,用于在转录水平处的功能分析,因此可以应用于细化组装的基因组以获得更好的注释[9].然而,TGS目前还不能量化基因表达,故障率高于SGS。TGS和SGS的结合能够解决这一问题,被大多数研究者强烈推荐[10.].
随着转录组和基因组数据的可用,功能基因组学的研究正在进行,这严重依赖于基因表达分析。逆转录实时定量PCR (RT-qPCR)是一种非常敏感和准确的分析基因表达水平的技术,但它需要合适的内参基因作为内对照,使不同样本之间的mRNA水平正常化,以便准确地比较基因表达[11.,12.].应在各种实验条件下以恒定水平表达理想的参考基因。然而,研究表明,对于所有实验条件,没有单一参考基因是普遍的[13.,14.,15.].因此,在使用内参基因进行基因表达分析之前,有必要对内参基因在特定实验条件下的稳定性进行评估。
在本研究中,为了提供高质量和更完整的组装在基因组和植物植物的转录组研究中,进行了组合SGS和TGS技术的混合测序方法。第一,全长转录物的侵入性植物种类P.ameracana和一种非入侵的外来同类p . icosandra通过单分子实时测序(SMRT)生成。在转录水平上分别分析了可变剪接事件、简单重复序列(SSR)、编码序列、蛋白质注释和长非编码序列。此外,还对上述两种植物和一种本地同属植物的内参基因的稳定性进行了评价p . acinosa为进一步研究功能基因表达提供参考。
结果
为了对植物进行分类,这三个植物科成员,美洲P. americana, icosandra和P.acinosa经PCR鉴定后,根据第二内转录间隔区(ITS2)和叶绿体基因组光系统II蛋白D1基因和tRNA-His基因的基因间隔区(psba-trn.) (桌子S1).的序列ITS2和psba-trn.在p .美国我们所雇用的序列具有100%的身份,陈报告的序列[16.].在p . icosandra,序列ITS2有99.5%的同一性和序列psba-trn.有95.7%的身份,陈某的结果[16.].在P.acinosa, 100%相似度ITS299.4%的识别psba-trn.被发现 [16.].
SMRT测序数据输出
使用Pacific Biosciences的SMRT测序协议,19.96 Gb的入侵物种的干净读取p .美国预处理后得到。在> = 3和序列质量> 0.9的基础上,共获得235,097条环状一致序列(CCS),全长率85.44%,其中非嵌合(FLNC)全长200,857条,高质量一致异构体86,989条。在剔除冗余序列后,共获得46,369个转录本,1675个选择性剪接事件,25,641个SSR, 34,971个完整编码序列,3574个lnc rna和41,676个注释转录本p .美国实现了。同样,19.75 GB清洁读入p . icosandra共筛选到223203个CCS,全长率89.56%,208865个FLNC序列和96764个高质量一致异构体。随后,获得了50220个转录本和1908个选择性剪接事件。鉴定出31,800个SSRs, 36,841个完整的编码序列,3833个lnc rna和45,050个注释转录本p . icosandra(表1).
转录组分析
基于实现的转录物的结构,鉴定了1675和1908年的替代剪接事件p .美国和p . icosandra, 分别。记录为43,942(总共100,469,039英镑)p .美国在序列长度大于500 bp的基础上,采用SSR方法进行分析,包括25641条SSR和17231条含SSR的序列。类似地,共有48,070个转录本(总计117,904,499 bp)p . icosandra用于SSR分析,鉴定了31,800个SSR,鉴定了与20,155SSR含序列。关于包含多个SSR的序列数的详细信息,复合地层中存在的SSR数量以及不同类型的SSR数量2.此外,总共34,971个完整的编码序列(CD)p .美国和36,841个CDp . icosandra通过使用TransDecoder来识别。预测蛋白的长度分布如图2所示。S1.
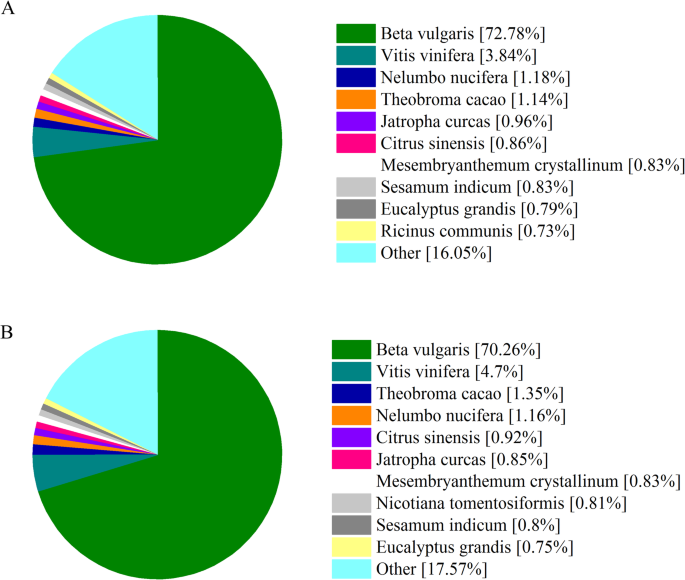
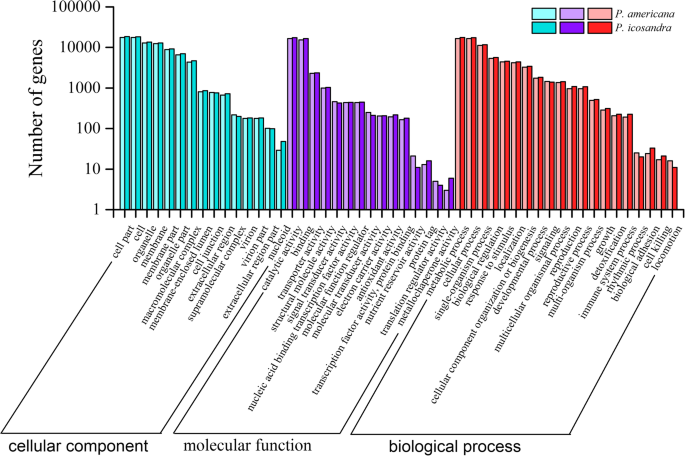
利用八种蛋白质数据库,进行序列比对以注释预测的蛋白质。总共有41,676名成绩单p .美国和45,050名成绩单p . icosandra分开注释(表3.).中注释蛋白序列的数量p .美国是类似于p . icosandra在特定数据库下。具体地,NR(NCBI非冗余蛋白)分析显示,大约30,000个转录物p .美国还有31000份成绩单p . icosandra显示了最高的序列相似之处Beta寻常魅力(图。1).GO (Gene Ontology)赋值也表明,两种植物中序列数量相近的序列属于同一术语。其中主要分为细胞组分的细胞部分和细胞项、分子功能的催化活性和结合、生物过程的代谢过程和细胞过程(图1)。2).COG(蛋白质群体的簇)注释表明,两种植物物种中的大量预测蛋白与功能类R(仅限通用函数预测),J(转换,核糖体结构和生物发生),T(信号转导机制),g(碳水化合物运输和代谢)和o(蛋白质转化,蛋白质周转,伴侣)(图。S2).Eggnog的结果(基因的进化族学术语:未经监督的正非群体)注释表明,两种植物物种中的大多数注释蛋白属于功能类S(功能未知)(图。S3.).Kog(真核ortholog组)功能分类表明,R(一般功能预测)和O(后期改性,蛋白质周转和伴侣)是两种植物物种中最丰富的功能类别(图。S4).这些结果表明,所获得的大多数序列是真正的功能性蛋白质,并且具有类似的功能分类p .美国及其同意p.icosandra.尽管还需要更多的工作来识别调控或参与入侵成功的序列p .美国,预测蛋白的注释为进一步研究提供了必要的信息。
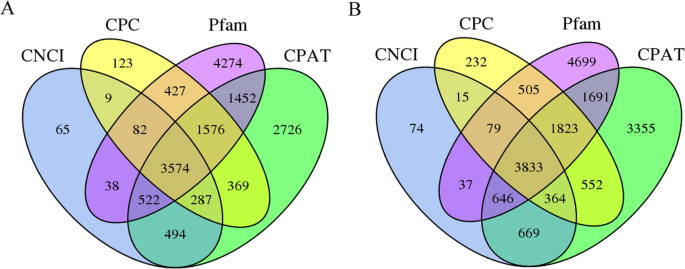
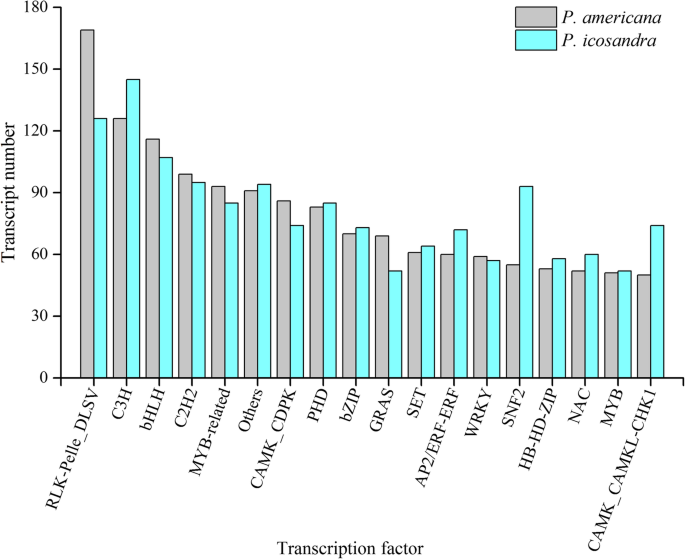
除了编码蛋白质的转录物之外,实现了长期的非编码RNA(LNCRNA)。据报道,LNCRNA是植物生物过程中的关键调节因子。lncrna的数量P.ameracana和p.icosandra通过CPC(编码潜在计算器),CNCI(编码 - 非编码指数),PFAM和CPAT(编码潜在评估工具)预测。总共3574磅P.americana和3833 lncrnap.icosandra所有这四种方法都预测(图。3.).随后,对转录调控系统中关键的转录因子(transcription factors, TFs)进行了预测。在p .美国,过滤了204个类型的3991个TFs。而在p . icosandra,预测了201个类型的4022个TFs。这两种植物共有前10种TF,但每种TF的数量并不相似,尤其是rlk - pelle_dllsv、C3H、SNF2和CAMK_CAMKL-CHK1,说明它们在转录调控方面具有特殊的功能(图1)。4).
RT-qPCR的扩增性能
专为RT-QPCR设计的引物首先通过PCR评估。选择在没有引物二聚体的单一扩增子制备单个扩增子的引物用于熔化曲线分析。仅选择制备单个片段的引物进行QPCR扩增效率评估。每个引物对的QPCR效率由汇集的cDNA的10倍连续稀释产生,并显示在表中4.
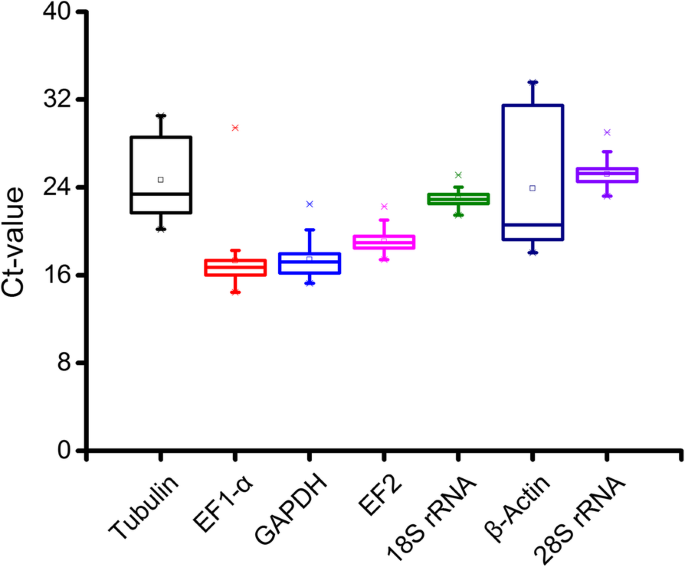
使用每个参考基因的阈值循环(CT)值在不同的实验条件下评估表达水平(图。5).所有七个候选参考基因的平均CT值范围从17.3到25.2,其中EF1 -α表达最高的表达水平和28s rrna.有最低的表达水平。还有人建议CT值β-肌动蛋白和管蛋白在所有实验样本中波动显著。
候选参考基因的稳定性
为了确定不同实验条件下适合归一化的内参基因,分别采用geNorm、NormFinder和BestKeeper对表达数据进行分析(表1)S2).
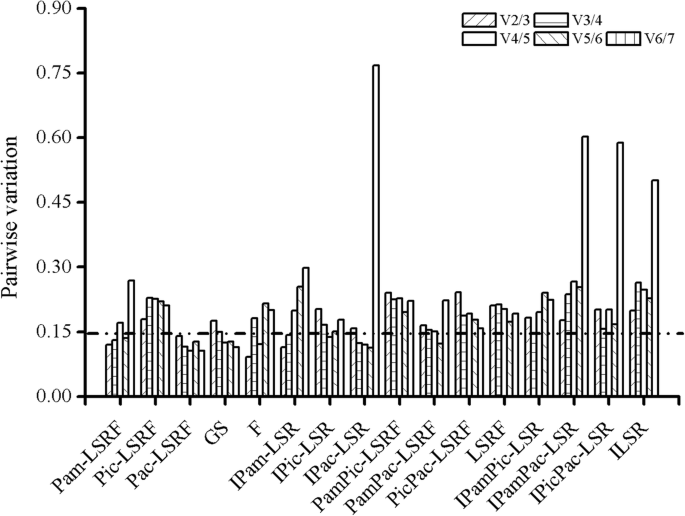
当分析参考基因的表达稳定性(在不同的组织(叶,茎,根和花)中分析p .美国,18s rrna.和EF2的P.americana被Genorm和Normfinder鉴定为最合适的参考基因,以及18s rrna.也是BestKeeper的建议。V的两两变异值2/3低于0.15的截断值,说明两个内参基因组合最适合基因表达归一化(图5)。6).当测试p.icosandra,EF1 -α不仅geNorm和NormFinder推荐用于基因表达分析的规范化。EF2也是geNorm和BestKeeper的建议。在P.acinosa,EF1 -α是改变和最好的人建议的最佳参考基因,但是18s rrna.由诺维师推荐。使用两种参考基因是合适的,因为v的成对变化值2/3低于0.15。当收集不同组织的数据时P.americana和p.icosandra在一起,EF2被证明是所有三种方法最稳定的基因。当研究不同组织中参考基因的表达稳定性P.americana和P.acinosa,18s rrna.通过Genorm和Normfinder显示出最佳的表达稳定性,而EF2被称为最稳定的最好的人。然而,对于V的Genorm建议使用五种参考基因的组合5/6小于0。15。当数据来自不同的组织p.icosandra和P.acinosa被置在一起,EF1 -α被统一作为最佳的Genorm和Normfinder,而EF2BestKeeper建议为最佳的稳定性内参基因。当将这三种植物的数据设为一个池时,EF1 -α被geNorm和NormFinder认为是最稳定的,而EF2还由Genorm和Bestkeeper推荐。根据这些结果,在分析植物物种中的基因表达水平时,选择适当的参考基因是非常重要的。
当分析发芽种子之间的数据时,28s rrna.和EF1 -α被geNorm鉴定为最佳内参基因,18s rrna.是NormFinder推荐的,那么GAPDH.是BestKeeper建议的。3个内参基因足以使V的基因表达正常化3/4低于0.15。在这三种植物中,EF1 -α三种方法均证实。基因型分析表明,V4/5低于0.15。因此,建议4个内参基因组合。这些结果表明,当关注不同植物物种的特定组织时,内参基因的选择也是非常必要的。
当植物被侵染时美国litura,18s rrna.在不同组织中,geNorm和NormFinder的表达稳定性最高P.americana, 尽管EF1 -α由bestkeeper透露。由于V的值,通过v的增值表明两种参考基因的组合2/3小于0.15。18s rrna.也被改变了美国litura- 漂白p.icosandra,EF1 -α被NormFinder和BestKeeper证明是最稳定的。geNorm推荐了4个内参基因组合。18s rrna.也被植物物种中的植物蛋白鉴定为最佳参考基因P.acinosa, 尽管微管蛋白是由NormFinder和28s rrna.由Bestkeeper推荐。三种参考基因的组合是通过Genorm的合适。在将两种植物物种的数据分析在一起时,18s rrna.geNorm和NormFinder的表达稳定性最好EF1 -α是由bestkeeper建议的P.americana和p.icosandra.18s rrna.通过所有三种算法识别为最佳参考基因P.americana和P.acinosa.18s rrna.和β-肌动蛋白被改变建议p.icosandra和P.acinosa, 尽管GAPDH.和EF2分别由NormFinder和BestKeeper推荐。当取所有数据时美国litura考虑受感染的植物,18s rrna.表现出最稳定的geNorm和NormFinder,而EF2是BestKeeper发现的表达最稳定的基因。
讨论
全长成绩单是结构,功能和比较基因组学研究的基础资源[9,17.].通过能够产生多酮硼梭序列来改善基因组和转录组组件来确认SMRT测序[18.].生成的全长cDNA序列能够表征转录后的过程,如选择性剪接、lncRNA预测和编码序列,用于进一步的基因功能研究。根据生成的全长转录组数据,得到约20gb的干净数据P.americana和p.icosandra分别为(表1).因此,分析了CCS,FLNC,共有同种型,高质量同种型,转录物,替代剪接事件,SSR,完全编码序列,LNCRNA和注释转录物的数量,提供了进一步研究的基本转录组信息。
有报道表明,全长转录组Zea Mays.对完善基因注释和揭示玉米基因表达的复杂性有很大帮助[9].类似的分析也在高粱双色[17.].更重要的是,世界扩张能力Cydia pomonella.已根据其基因组信息通知[19.].入侵种快速生长和入侵适应的分子机制Mikania Micrantha.也根据它的参考基因组进行了研究[20.].因此,全长转录组数据P.americana和p.icosandra将有助于基因组研究,并向侵入机制提供见解P.americana通过比较基因组学研究植物的种类。
用于进一步基因表达分析的RT-qPCR的准确相对定量依赖于稳定表达的内参基因的稳健归一化,以减少实验过程中的误差[21.].因此,适用于三种植物溶解物种中相对基因表达数据的标准化的合适参考基因,P.americana,p.icosandra和P.acinosa,被寻求在各种各样的条件下。这些结果证明了在相关实验条件下验证参考基因的重要性。例如,在不同的组织(叶,茎,根和花)中P.americana,18s rrna.和EF2建议是最适合最佳的参考基因,并发现类似的结果美国litura- 漂白P.americana.然而,即使适当的内参基因p.icosandra根据三种方法的分析结果排序,所有成对变化值高于0.15的截止值,而组合18s rrna.,β-肌动蛋白,EF1α.和EF2最适合美国litura- 漂白p.icosandra.EF2和EF1α.已被认为是理想的参考基因P.acinosa,而结合18s rrna.,β-肌动蛋白和GAPDH.建议后被推荐美国litura侵扰。研究也表明,没有一个内参基因在生物体的不同组织中稳定表达,例如内参基因的选择Amygdalus Persica,茄属植物lycopersicum和甘氨酸最大[11.,13.,14.].更重要的是,我们的结果还表明,应通过实验证据确认基于转录组数据鉴定的参考基因。在JA诱导的转录组中p .美国,28s rrna.表现出外源JA处理和对照植物之间的稳定表达[5].鳞翅目食草动物侵染可诱导植物JA信号通路[22.].然而,18s rrna.和EF2被鉴定为最稳定的表达参考基因p .美国后美国litura侵扰。
为了对三种植物进行基因表达分析,我们还将三种植物的数据进行了比较。在比较三种植物种子萌发数据时,三种方法推荐了不同的基因。其他试验条件下的植物组合显示,除组合外,几乎所有组合的两两值都高于0.15的截断值P.americana和P.acinosa其中5个内参基因被推荐用于数据标准化以及组合美国litura- 漂白P.americana和美国litura- 漂白p.icosandra提出了三个内参基因。这些结果表明,即使这些植物是同种植物,也没有特定的基因在不同的植物物种中持续表达。因此,在相关实验条件下,应适当使用内参基因。
结论
该研究提供了转录组宽全长同种型P.americana和p.icosandra,提供侵入性成功的见解P.americana。此外,还提出了在同一种植物的不同组织下或不同植物之间选择合适内参基因的指南。在不同的实验条件下,没有特定的基因持续表达,说明内参基因鉴定的必要性。这些结果将有助于探索植物的功能和比较基因组学研究,更好地了解植物生物学。
方法
植物及昆虫材料
植物的p .美国(24°49镑,102°52说的),p . icosandra(25°08年’,102°44说的)p . acinosa(25°26′n, 104°19′e),首先命名为M、K和Q,采集于中国云南。在符合当地法规的情况下,允许进行抽样。据介绍,这些标本的正式鉴定是由云南大学生物资源保护与利用国家重点实验室生态与进化生物学实验室植物学专业的陈超进行的中国植物志(Vol.5:435-436,2013),北美植物志(Vol.4:3-11,2003),中国虚拟植物标目(http://www.cvh.ac.cn/)JSTOR上的全球植物(http://plants.jstor.org/).DNA鉴定也是根据ITS2核核糖体DNA区域,是植物分子系统学中属和种水平上应用最广泛的DNA片段之一,也是叶绿体psba-trn.基因间区域(16.].所有凭证标本都是在云南大学云南省生物资源保护和利用国家重点实验室的生态学和进化生物学实验室实验室的实验领域。
单株植物的叶、茎、根和花的组织p .美国或者p . icosandra均是在云南省野外单独采集的,这些样本的采集不需要获得许可。每个样品在液氮中快速冷冻,保存在−80°C,以备进一步实验。
第三次幼虫spodoptera litura购自中国河南济源白云实业有限公司(shop101732681.taobao.com.),然后在气候室(27°C有光14小时,25°C无光10小时)人工饲养,供进一步使用。
用于参考基因评估,种子p .美国,p . icosandra和p . acinosa首先从云南省的野外收集,不需要许可。将种子分别在1%琼脂平板中播种并在气候室中栽培。5天后,将五种发芽种子作为一个样品一起收集,用于后续实验。每种植物物种都有三种复制。两周后,将每个物种的其他发芽种子移植到塑料罐(15厘米直径和12.5厘米高度),土壤(江苏培氏赛矩阵技术开发有限公司,中国),并在人造室中用足够的水栽培如上所述的条件相同。四个月后,每种植物物种的叶,茎,根和花朵被单独收集。同时,六只幼虫美国litura三龄幼虫的幼虫p .美国,p . icosandra或者p . acinosa每片叶子有一个昆虫。对照治疗是不含食草动物。24小时后,单独收获这三种植物物种的叶,茎和根。收集收集的所有样品在液氮中冷冻并储存在-80℃,以进行后续测定,每次处理进行三种重复。
核酸提取和测定
根据DNAquick植物系统(天根生物技术有限公司,北京)提供的方案,从不同植物种类的叶片中分离出基因组DNA。然后将其作为PCR模板用于植物种类鉴定。
采用RNAprep Pure Plant Kit(多糖多酚;天根生物技术有限公司,中国北京)根据制造商的说明。使用纳米光度计N60(德国Implen)和安捷伦生物分析仪2100系统(美国安捷伦科技公司,CA)测定RNA质量和纯度。我们选择的样本只有280/260比值为1.9 ~ 2.1,比值为2.0 ~ 2.4,比值为230/260,且RIN值大于7.0。将来自同一种植物的4个不同组织的相同数量的总rna混合为一个样本,进行全长转录组测序。
如上所述,还单独提取来自收集的用于参考基因评价的样品的总RNA。对于每个样品,通过使用1.5μg总RNA制备CDNA,这是安全RT试剂盒的推荐说明(用GDNASE;天根生物技术有限公司,北京,中国)。
PacBio cDNA文库制备及SMRT测序
使用Smarter TM PCR cDNA合成试剂盒(Clontech,CA,USA)合成全长cDNA。然后使用PCR重新扩增生成的cDNA。在终止之后,将带有发夹环结构的SMRT适配器连接到cDNA中。通过外切核酸酶消化,构建了cDNA文库。在CDNA文库的质量测量后,使用Pacific Bioscience eSearel平台进行SMRT测序后,在提供的方案之后进行。
Illumina cDNA文库构建及二代测序
提取的mRNA用寡聚物(dT)粘附的磁珠纯化。片段在NEBNext第一链合成反应缓冲液中进行。根据随机六聚体获得第一链cDNA,然后用dNTPs、RNase H和PrimeSTAR GXL DNA聚合酶合成第二链cDNA。用AMPure XP珠纯化合成的cDNA。修复结束后,加入poly A和适配器连接,使用AMPure XP珠选择尺寸。扩增产生的cDNA,构建cDNA文库。在Illumina nova平台上对符合条件的库进行配对测序。
长读取的质量过滤和纠错
通过除去小于50bp的聚合酶和序列精度小于0.9的聚合酶进行过滤RAW SMRT测序读数。去除适配器后,获得了底亚。使用超过50bp的底次生产清洁数据。CCSS由Clean数据生产,具有完整通行证的参数> = 3,精度超过0.9。在检查5'和3'适配器和Poly(A)尾部的共存后,选择全长转录物。在图书馆制剂的过程中,由于浓度低浓度的适配器或SMRTBELL而被两种cDNA模板链的直接连接形成的嵌合序列称为人造嵌合序列。全长转录物中的非嵌合序列是全长非嵌合(FLNC)序列。
由于SMRT测序的错误率较高,因此需要进行纠错。首先采用迭代聚类方法获得一致异构体,然后利用Quiver [9,10.].此外,滤除了原始illumina SGS读取以除去适配器序列,低质量读取,并且使用SGS与软件Provread读取进行低质量同种型的误差校正[23.].简而言之,Illumina RNA-Seq数据的短reads被映射到低质量亚型,然后低质量亚型中的碱基被映射过程中出现次数最多的特定碱基取代。校正后准确性为> 99%(高质量共识亚型)的全长共识亚型被生成用于进一步分析。使用CD-HIT去除高质量全长序列中的任何冗余[24.].
转录组分析
替代剪接检测
RNA替代剪接发生在预mRNA转录物中,这导致编码多种蛋白质的单个基因,因此增加转录组和蛋白质组多样性,并在调节基因表达和蛋白质功能方面发挥关键作用[8].在该研究中,如SMRT测序中所述筛选替代剪接同种型Agasicles hygrophila[25.].
SSR检测
SSRs,也称为微卫星,是特定DNA基序的串联重复序列(通常是5-50次),长度从2 bp到13 bp [26.].根据转录组数据,选择500 bp以上的序列进行SSR分析。利用MIcroSAtellite鉴定工具(MISA)分析了单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸和化合物7种SSR类型;http://pgrc.ipk-gatersleben.de/misa/http://pgrc.ipk=gatersleben.de/misa/).研究结果可用于植物遗传研究,特别是入侵植物种群遗传学和入侵历史的研究p .美国.
编码序列预测
为了预测所有蛋白质编码序列,TransDecoder (http://github.com/transdecoder/transdecoder/release.)受雇[27.].然后计算不同长度蛋白质的数量。
转录本的功能注释
为了确定假定的基因功能,获得的非冗余转录本通过BLASTx与8个蛋白数据库进行比对,e值< 10- 5,将检索到的与给定转录本序列相似度最高的蛋白质用于蛋白质功能注释。这些数据库包括Nr、KEGG(京都基因和基因组百科全书)、SwissProt、COG、GO、KOG、Pfam和eggNOG [25.].
长期非编码RNA(LNCRNA)预测
LNCRNA是生物学中有趣的话题,已被发现是在广谱的生物过程中是基因表达调控,对翻译或印迹的控制[28.].因此,我们利用CPC、CNCI、CPAT和Pfam蛋白结构域分析筛选转录本的编码潜力,筛选lncrna。
候选参考基因选择,QPCR和数据分析
对于内参基因评估,七个常用的候选基因(Tublin;延长因子1 -α,EF1 -α;甘油醛-3-磷酸脱氢酶,GAPDH.;延长因子2,EF2;18s rrna.;β-肌动蛋白;28s rrna.)基于全长转录组数据的注释选择。七个参考基因的引物由信标设计器7.9(Premier Biosoft International,CA)单独设计。表格中显示了引物,扩增子长度,PCR效率和线性回归系数的序列4.
在ABI QuantStudio 7 Flex PCR系统(应用系统,Carlsbad,CA)上进行QPCR测量。根据制造商的指导,使用超良预混料加套件(Sybr Green;北京,北京,北京),以20μL反应体积进行测定所有样品。RT-QPCR反应的热分布如下:在95℃下15分钟,然后在95℃和32℃下在60℃下进行40个循环。通过每种PCR反应结束熔化曲线分析(60-95℃)以验证引物特异性。熔融曲线显示所有基因的单一扩增产物。所有样品均在技术上以三份测试。
表达水平由CT值的默认设置确定。使用比较CT法计算不同样品之间基因表达的相对折叠变化。在使用五个稀释点上使用10倍稀释系列汇集的cDNA产生的标准曲线计算引物扩增效率和标准偏差。通过Genorm评估七种选定参考基因的表达稳定性[29.],bestkeeper [30.]和rangfinder [31.].通过geNorm [14.,29.].
可用性数据和材料
本研究中产生的所有数据均包含在本文及其补充信息文件中。所有转录组数据均保存在NCBI Sequence Read Archive (SRA)中,登录号分别为PRJNA613739和PRJNA613892。由于进一步研究的原因,转录组数据将不会在2021年12月31日之前公布,但可在合理的要求下从通讯作者处获得。
缩写
- SMRT:
-
单分子实时
- RT-qPCR:
-
逆转录定量实时PCR
- TGS:
-
第三代测序
- SGS:
-
第二代测序
- ITS2:
-
第二个内部转录间隔
- CCSS:
-
圈子共识序列
- FLNC:
-
全身non-chimeric
- SSR:
-
简单的序列重复
- CD:
-
编码序列
- ORF:
-
开放阅读框
- NR:
-
NCBI冗余蛋白质
- KEGG:
-
Kyoto基因和基因组的百科全书
- COG:
-
蛋白质的同源基团簇
- 走:
-
基因本体论
- KOG:
-
真核直接同源组
- 蛋酒:
-
基因的进化族学系:非监督正非群体
- lncrana:
-
长期非编码RNA
- 中国共产党:
-
编码潜在计算器
- CNCI:
-
编码 - 非编码索引
- CPAT:
-
编码潜在评估工具
- EF1 -α:
-
延长因子1 -α
- GAPDH.:
-
甘油醛-3-磷酸脱氢酶
- EF2:
-
延长因子2
- CT:
-
阈值周期
参考
- 1。
肖丽,Hervé MR, Carrillo J,丁建强,黄伟。入侵植物生长繁殖的纬向趋势及防御。植物学报,2006,54(5):754 - 759。生物入侵杂志。2019;21(1):189 - 201。
- 2。
李杰尔,Gurajala HK,Wu LH,Van der Ent A,Qiu Rl,Baker AJM等。来自中国的超成本植物:当前知识状态的合成。环境科技。2018; 52(21):11980-94。
- 3.
Pizzo E,Pane K,Bosso A,Landi N,Ragucci S,Russo R等。来自PD-L1 / 2的新型生物活性肽,1型核糖体灭活蛋白商dioica评价其抗菌性能和抗生物膜活性。BBA-Biomembranes。2018年,1860(7):1425 - 35。
- 4.
Sisay B,Tefera T,Wakgari M,Ayalew G,Mendesil E.选定的合成杀虫剂和植物对阵秋季植物的疗效,Spodoptera Frugiperda.,在玉米中。昆虫。2019; 10(2):45。
- 5。
纳勒K,Klenov A,Hudak Ka。淘汰叶mRNA转录组及其茉莉酸的调控。前植物SCI。2016; 7:283。
- 6。
珍妮卡,克伦诺夫A,Guzman Jc,Hudak Ka。戳性miRNA和mRNA转录om的整合揭示了茉莉酸反应基因的靶向。前植物SCI。2018; 9:589。
- 7。
珍妮特KCM,Diaz Ca,Platts Ae,Hudak Ka。淘汰基因组的De Novo组装提供了对炭抗病毒蛋白(PAP)基因表达的洞察力。前植物SCI。2019; 10:1002。
- 8。
罗杰斯MF,Thomas J,Reddy Asn,Ben-Hur A.脾脏选手:在基因模型和EST数据的背景下检测从RNA-SEQ数据的替代拼接模式。基因组Biol。2012; 13(1):R4。
- 9.
王B,Tseng E,Regulski M,Clark Ta,Hon T,jiao yp等。通过单分子长读取测序揭示玉米转录组的复杂性。NAT Communce。2016; 7(1):11708。
- 10。
Xu ZC, Peters RJ, Weirather J, Luo HM, Liao BS, Zhang X,等。通过应用于不同根组织的测序平台组合获得的全长转录组序列和剪接变体丹参和tanshinone生物合成。工厂j . 2015, 82(6): 951 - 61。
- 11.
桐ZG,高Zh,王F,周J,张Z.使用实时PCR选择桃中基因表达研究的可靠参考基因。BMC MOL BIOL。2009; 10(1):71。
- 12.
万洪军,赵志刚,钱传庭,隋永红,马立祺,陈建峰。黄瓜实时荧光定量聚合酶链反应基因表达研究中内参基因的选择。肛门。2010;399(2):257 - 61。
- 13。
Libault M, Thibivilliers S, Bilgin DD, Radwan O, Benitez M, Clough SJ等。四个大豆内参基因的鉴定及基因表达标准化。植物基因组。2008;1(1):44-54。
- 14。
Løvdal T, Lillo C.番茄氮、冷、光胁迫实时荧光定量PCR归一化的内参基因选择。学生物化学肛门。2009;387(2):238 - 42。
- 15。
X,Hou M,Liu Y.参考基因选择和基因表达研究的评价使用QRT-PCR在白背垫锅中的QRT-PCR,Sogatella furcifera(半翅类:Delphacidae)。中国寄生虫学与寄生虫病杂志。2015;
- 16。
陈双林,姚浩,韩建平,刘超,宋建勇,史丽玲,等。ITS2区域作为一种新的药用植物DNA条形码的验证。《公共科学图书馆•综合》。2010;5 (1):e8613。
- 17。
Abdel-Ghany Se,Hamilton M,Jacobi JL,Ngam P,Devitt N,Schilkey F等人。使用单分子长读取高粱转录组的调查。NAT Communce。2016; 7:11706。
- 18。
Koren S,Schatz MC,Walenz BP,Martin J,Howard JT,Ganapathy G等人。单分子测序读取的混合误差校正和de Novo组装。NAT BIOTECHNOL。2012; 30(7):693。
- 19.
万FH,Yin Cl,Tang R,Chen Mh,Wu Q,Huang C等人。染色体级基因组组装Cydia pomonella.提供对化学生态和杀虫剂抗性的见解。NAT Communce。2019; 10(1):1-14。
- 20.
刘波,严军,李文华,尹丽娟,李平,于海霞,等。Mikania Micrantha.基因组提供了快速生长的分子机制。Nat Commun。2020;11(1):340。
- 21。
关键词:荧光定量PCR,内参基因选择,荧光定量PCR生物化学有限公司2004;313(4):856-62。
- 22。
刘莹莹,杜美玲,邓玲,沈建峰,方明,陈强,等。MYC2通过自调节负反馈回路调节茉莉酸信号的终止。植物细胞,2019,31(1):106 - 27所示。
- 23。
验证:通过迭代短读共识的大规模高精度PacBio校正。生物信息学。2014;30(21):3004 - 11。
- 24。
Li W,Godzik A. CD-his:用于聚类和比较大套蛋白质或核苷酸序列的快速计划。生物信息学。2006; 22(13):1658-9。
- 25。
贾德,王yx,刘毅,胡j,guo yq,gao ll等。SMRT测序跳蚤甲虫全长转录组Agasicles hygrophila(Selman和Vogt)。SCI Rep-UK。2018; 8(1):2197。
- 26。
Guichoux E,Lagache L,Wagner S,Chaumeil P,LégerP,Lepais O,等。微卫星基因分型的目前趋势。Mol Ecol Resour。2011; 11(4):591-611。
- 27。
哈斯(Haas)等。利用trinity平台进行RNA-seq从头转录本序列的构建,进行参考生成和分析。Nat Protoc。2013;8(8):1494。
- 28。
李军,马伟,曾平,王继勇,耿斌,杨继军,等。LncTar:用于预测长链非编码RNA的RNA靶标的工具。短暂的Bioinform。2015;16(5):806 - 12。
- 29。
VandeSompele J,De Preter K,Pattyn F,Poppe B,Van Roy N,De Paepe A等。通过多个内部对照基因的几何平均来精确正常化实时定量RT-PCR数据。基因组Biol。2002; 3(7):Research0034。
- 30.
Pfaffl MW, Tichopad A, Prgomet C, Neuvians TP。稳定管家基因、差异调控靶基因和样本完整性的测定:使用成对相关性的bestkeeper -excel工具。Biotechnol。2004;26(6):509 - 15所示。
- 31.
Andersen CL, Jensen JL, Ørntoft TF。实时定量逆转录- pcr数据的归一化:一种基于模型的方差估计方法,用于识别适合归一化的基因,应用于膀胱癌和结肠癌数据集。实用癌症杂志2004;64(15):5245 - 50。
致谢
我们感谢生物标记技术公司(中国北京)在转录组测序方面的帮助。
陈述
在户外收集植物材料时,我们遵守国家准则和濒危野生动物群和植物群的贸易公约。
资金
该工作得到了国家重点研究和发展计划(2017年FFC1200100)和中国国家自然科学基金(31870362)的支持。资助机构在设计和撰写稿件的研究和收集,分析和解释方面没有发挥作用。
作者信息
隶属关系
贡献
DFL和YW策划并设计了研究。研究对象进行实验。LC, CC, XKA辅助进行实验。XKA提供基因表达评估软件。DFL分析数据并撰写论文。YJZ, YW, QJL对稿件进行了修改。所有作者均已阅读并批准稿件。
通讯作者
伦理宣言
伦理批准并同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充信息
附加文件1:
表S1。鉴定三种使用的Phytolacceae物种。表S2。根据三种软件对内参基因进行验证排序。图S1。从编码序列预测蛋白质序列的长度分布。图S2。由同源蛋白组簇(COG)注释的转录本分类。图S3。由基因进化系谱注释的转录本分类:非监督同源组(eggNOG)。图S4。由真核外科群(Kog)注释的转录物的分类。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
刘,D.,陈,L.,陈,C.等等。全长转录组分析商美国及其同意p . icosandra三种植物的基因表达正常化。BMC植物BIOL.20.396(2020)。https://doi.org/10.1186/s12870-020-02608-9
收到了:
接受:
发表:
关键词
- phytolaccaceae.
- SMRT测序
- 全长转录组分析
- 参考基因评价
- RT-QPCR.