摘要
背景
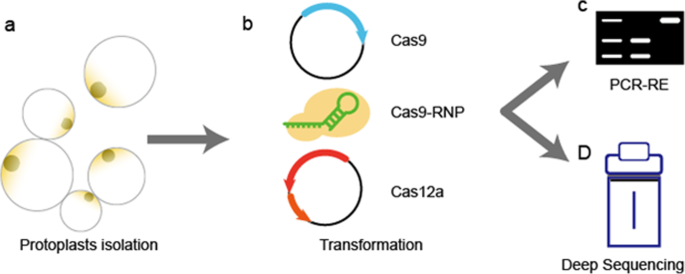
迄今为止,CRISPR/Cas9 RNP编辑工具尚未应用于香蕉的基因改造。本研究通过peg介导的香蕉原生质体转化体系的建立,为建立高效的无dna位点诱变体系提供了可能。
结果
原生质体在植物科学中是一个多功能的瞬时表达平台。在本研究中,我们建立了peg介导的香蕉原生质体转化体系。该系统进一步优化,成功递送CRISPR/Cas9和CRISPR/Cas12a质粒和CRISPR/Cas9核糖核蛋白(RNPs)靶向递送PDS基因进入香蕉原生质体。pcr -限制性内切酶切(PCR-RE)检测中观察到特异性条带,单克隆Sanger测序进一步证实了目标位点存在indel。深度扩增子测序结果显示,CRISPR/Cas9系统的编辑效率高于其他两个系统。
结论
peg介导的香蕉原生质体转化系统可作为快速有效的香蕉sgRNA瞬时表达分析和验证工具。CRISPR/Cas9 RNP系统的应用使无dna基因编辑工程香蕉植株得以产生。
背景
原生质体是植物、真菌、细菌或古菌的细胞,其细胞壁通过质分解而被除去,留下原生质和质膜。早在1892年,Klercker就通过机械方法获得原生质体[1],导致收率低,操作困难,适用性差。1960年,Cocking首次利用酶解方法成功分离出番茄根尖原生质体[2]。该方法收率高、活性高、操作简便、适应性广,得到了广泛应用。植物原生质体的瞬时转化系统没有独特的细胞壁特征,在基因功能鉴定、亚细胞定位和基因编辑等遗传学研究中被广泛应用。用于植物原生质体转化的常用方法包括peg介导的转化[3.,4]、电穿孔介导转化[5,6,7,8,9]和显微注射转化[10]。其中,peg介导法因其操作简单、成本低、对特定设备不要求、结果产生稳定等优点被广泛应用。到目前为止,已经建立了成熟稳定的原生质体瞬时表达遗传转化体系拟南芥[11,12,13,14],小麦[15],大米[16],玉米[17和其他物种。
目前,关于peg介导的DNA原生质体转化的报道较多,但关于peg介导的核糖核蛋白(RNP)原生质体转化的报道较少。2015年,Woo等人将RNP直接转移到拟南芥第一次种植烟草和大米。由原生质体再生的基因组编辑突变体不含转基因成分[18]。2016年Malnoy等人成功将RNP转移到葡萄和苹果的原生质体中,突变效率高达6.9% [19]。迄今为止,已有几项研究报道了CRISPR-Cas9基因编辑技术在香蕉中的应用[20.,21,22,23],但在这些CRISPR/Cas9基因编辑案例中,都产生了含有T-DNA的转基因植株,而香蕉的无dna基因组编辑方法尚未开发出来。本研究建立了peg介导的转化体系,为检测gRNA活性提供了一种有效的方法。该系统成功将RNP传递到香蕉原生质体中,并通过深度扩增子测序检测到RNP系统有效,为香蕉基因组编辑的进一步研究奠定了良好的基础。
结果
peg介导香蕉原生质体转化体系的建立
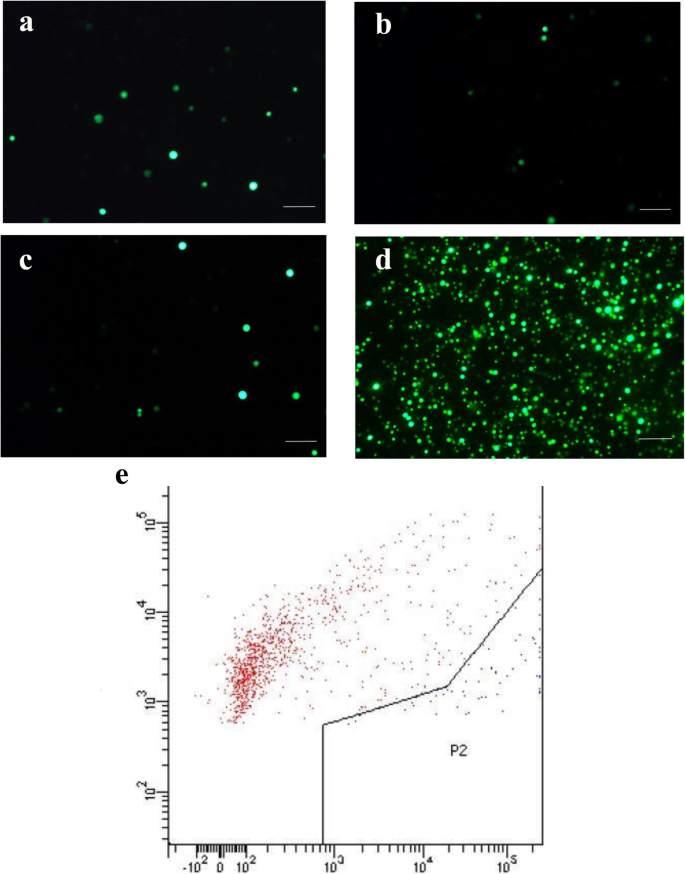
为了建立peg介导的香蕉原生质体转化体系,我们在水稻和小麦转化方案的基础上对转化方法进行了优化。通过流式细胞术检测,水稻和小麦的原生质体转化效率分别达到58.4%和64.5%。24]。该方案在香蕉中的转化效率远低于水稻和小麦;在黑暗中孵育5天后,仅出现少量GFP荧光点(图2)。1a).随后,在水稻和小麦的基础上,利用pUbi-GFP质粒对香蕉进行转化,优化PEG的浓度和培养时间。当PEG浓度增加到50%,诱导时间高达30min时,转化率最高(图2)。1d)。通过流式细胞术检测,转化效率为5.6%。1e).确定不同编辑方法对香蕉、CRISPR/Cas9-的编辑效率PDS质粒,CRISPR / Cas12a -PDS质粒和CRISPR/Cas9 RNP-PDS利用优化后的原生质体转化方案,按照图2所示的流程图检测复合物(RNPs)。2。
peg介导的质粒DNA转化香蕉原生质体基因编辑的PCR-RE检测
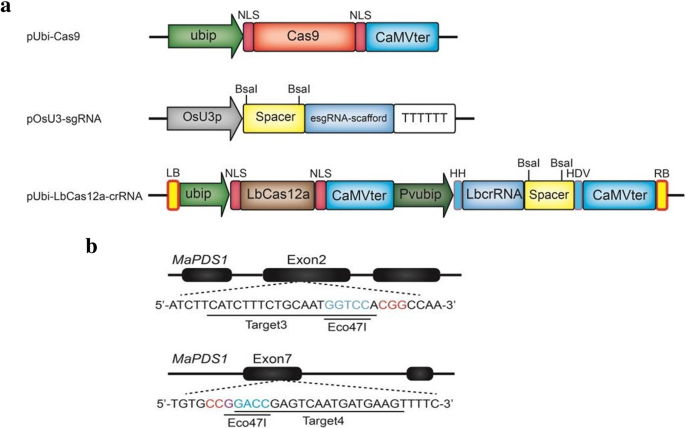
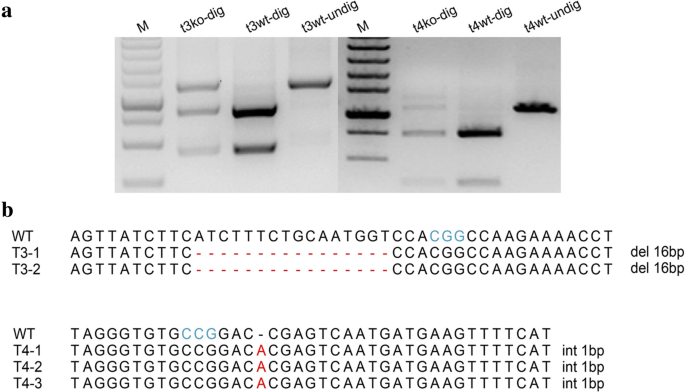
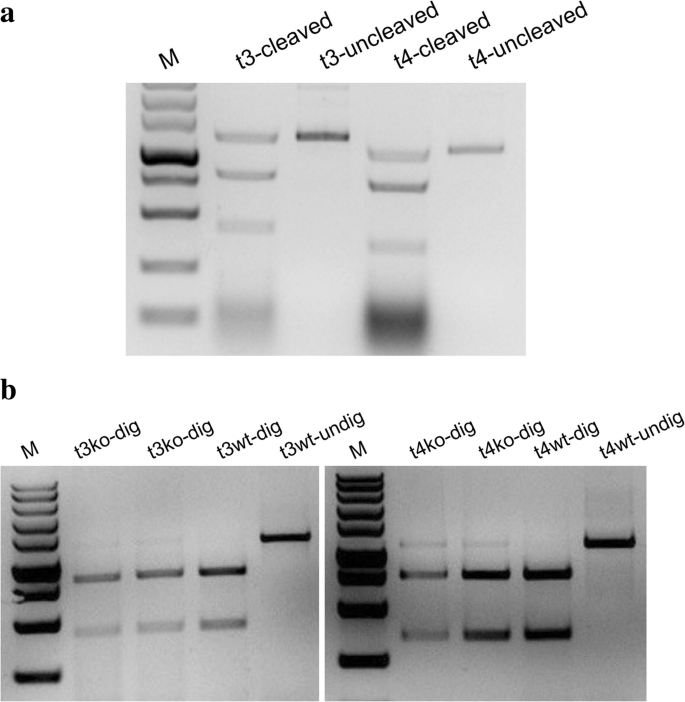
为了探索我们的原生质体转化系统是否可以通过转移质粒dna进行基因组编辑,我们设计了9个sgRNAs,其中两个sgRNAs靶向香蕉的第2外显子(靶标3:osu3d - pdst3)和第7外显子(靶标4:osu3d - pdst4)PDS基因,分别。这两种sgrna都含有生态47I可用于PCR-RE测定(图2)。3.).这9种引导RNA中的每一种都与增强的支架RNA融合(图2)。3.a).利用上述方法将这些质粒dna和Cas9质粒转化到香蕉原生质体中。从转化和未转化(对照)原生质体中分离的DNA用于PCR-RE分析和测序分析。PCR- re结果显示,对照样品的PCR产物完全被酶切成两个条带(t3wt-dig, t4wt-dig),而质粒转化样品的PCR产物在相同条件下仅被部分酶切(t3ko-dig, t4ko-dig),提示限制性内切酶位点被基因编辑盒突变(图2)。4a).为了进一步表征由Cas9产生的突变类型(即插入、缺失等),对未被生态47I被回收并克隆到T-blunt载体上进行Sanger测序。选取10个OsU3p- PDSt3单克隆和3个OsU3p- PDSt4单克隆进行Sanger测序。结果表明,OsU3p- PDSt3的两个克隆片段存在16 bp的缺失,OsU3p- PDSt4的三个克隆片段均存在1 bp的插入。插入和删除从PAM序列的第4个核苷酸开始(图2)。4b).我们还设计了香蕉的11个sgrnaPDSLbCas12a基因,但我们没有通过PCR-RE检测结果进行验证。
质粒DNA转入香蕉原生质体后单克隆的PCR-RE测定和Sanger测序。将编辑后的靶标3 (t3ko)或其野生型(t3wt)和编辑后的靶标4 (t4ko)或其野生型(t4wt)进行酶切生态我(挖)或未消化(挖)。基因编辑片段用转化原生质体的DNA扩增,野生型目标片段用未转化原生质体的DNA扩增。M表示DNA标记(一个).图3未消化片段单克隆Sanger测序。4一个(b).蓝色字母表示PAM序列;红色字母表示有碱基插入的突变;红色的“-”表示有碱基缺失的突变
RNP转化香蕉原生质体基因编辑的PCR-RE分析
为了检测RNP复合物进入原生质体后靶位点是否发生突变,我们按照Liang [25,26]。在使用RNPs转化原生质体之前,我们首先测试了RNPs是否可以在体外有效地编辑靶标。结果如图2所示。5a, RNPs能够将PCR产物中包含的靶标切割成两个片段。接下来,我们从转化和未转化的原生质体中分离DNA进行PCR扩增。含有目标序列的扩增片段用生态47我。结果表明,在rnp转化的样品中,大量扩增片段不能被消化,而对照样品的扩增子则可以被完全消化,这表明rnp基因被破坏了生态47 . RNP变换后样本中RNP识别位点(图2)。5b)。
香蕉原生质体中PEG介导的DNA转化的深度扩增子测序
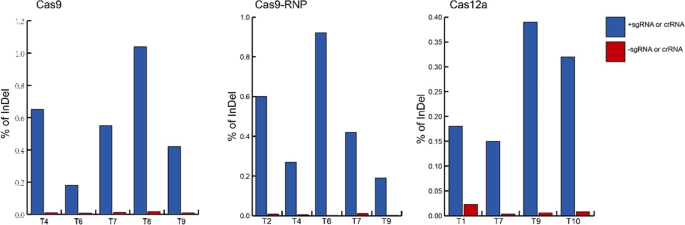
用pUbi - Cas9质粒与9种sgRNA质粒混合分别转化卡文迪什香蕉原生质体PDS11个Cas12a - PDS质粒(Cas12a - PDSt1至Cas12a - PDSt11)。为了检验编辑效率,从每个sgRNA加Cas9或LbCas12a质粒转化的原生质体中分离基因组dna,进行深度扩增子测序。测序结果显示,在用sgRNAs和Cas9质粒转化的原生质体中,共编辑了5个靶点(PDSt4、PDSt6、PDSt7、PDSt8和PDSt9)。在所编辑的目标位点中,目标位点PDSt8的编辑效率最高,为1.04%,在107,568个测序reads中观察到320个插入和800个删除。目标位点PDSt6的编辑效率最低,为0.18%。在80384个测序reads中观察到突变,147个测序reads包含各种缺失(附加文件)1:表S1)。
对于LbCas12a系统,成功编辑了四个靶点(Cas12a- PDSt1、Cas12a- PDSt7、Cas12a- PDSt9和Cas12a- PDSt10)。靶位点Cas12a- PDSt9的编辑效率最高,为0.39%,Cas12a- PDSt9的29008个测序reads中有114个被发现缺失型编辑。相比之下,52,965个测序读数中只有78个(3个插入和75个删除)包含在目标位点Cas12a-PDSt7上产生的突变,导致该目标位点的编辑效率最低(0.15%)(附加文件)2:表S2)。
9个Cas9和sgrna复合物在peg介导的香蕉原生质体转化中的深度扩增子测序
将Cas9蛋白分别与9个sgrna结合,转化为香蕉原生质体。为了检验转化RNPs的编辑效率,我们还对Cas9和sgRNAs的9个复合物靶向9个不同位点转化的原生质体中分离的基因组DNA进行了深度扩增子测序PDS网站。我们在PDS- sgrnat2、PDS -sgRNAt4、PDS -sgRNAt6、PDS -sgRNAt7和PDS -sgRNAt9五个靶点检测到该RNP系统的编辑作用。目标位点PDS -sgRNAt9的编辑效率最低(0.19%),在102,866个reads中包含166个插入reads和26个删除reads。目标位点PDS -sgRNAt6表现出最高的编辑效率(0.92%),在138,085个reads中包括169个插入reads和1104个删除reads (Additional file3.:表S3)。
非目标检测
分析CRISPR/Cas9-的脱靶效应PDS质粒和CRISPR/Cas9 RNPPDS复合体(RNPs)基因编辑系统,一个潜在的脱靶位点(AGCTTCGTGTACCGCAGTAGTGG), GSMUA_Achr6G21680_001,通过CRISPR-P 2.0网站预测。脱靶位点与sgRNA序列之间存在4个碱基错配。通过深度扩增子测序,在CRISPR/Cas9-中只检测到一个位点PDSRNPs基因编辑系统,脱靶效率为0.01%。(附加文件4表S4,附加文件5表S5,附加文件6:表S6)。
讨论
香蕉是热带和亚热带的一种多年生单子叶草本植物。大多数栽培品种是三倍体,肥力高。由于食用香蕉没有种子,通过传统的杂交栽培很难获得品质优良、抗病能力强的优良品种。然而,通过诱变育种和突变体筛选选育新品种存在一定的盲目性和较长的发育周期要求。用于稳定的CRISPR/Cas9基因编辑农杆菌属在主要香蕉品种中,花粉的不育性使其难以像二倍体植物那样通过杂交去除外源整合DNA。由于CRISPR/Cas9 RNP复合物在基因编辑中没有外源DNA整合到基因组中,因此该方法为香蕉基因编辑分子育种提供了一种有效的选择。
Woo等人(2015)报道CRISPR/Cas9 RNP复合物可以被递送到拟南芥在烟草、生菜和水稻中进行基因编辑,靶基因在烟草中的突变效率高达44% [18]。Malnoy等人(2016)通过PEG将RNPs导入葡萄和苹果原生质体,通过深度扩增子测序测量,苹果的突变效率达到6.9% [19]。2016年,Svitashev等人通过生物轰击将RNP复合物直接传递到玉米未成熟胚中,实现了靶向基因编辑,突变效率为0.69% [27]。同样,Liang等人(2017)利用颗粒轰击Cas9 RNP复合物,开发了一种高效的面包小麦无dna基因组编辑方法,突变效率为0.56% [25]。这些结果表明,利用peg介导的原生质体转化RNP复合物的突变效率远高于生物轰击法。
自2013年首次报道CRSPR/Cas9基因编辑技术以来,得到了广泛的应用和快速的发展。原生质体瞬时转化系统可以有效检测靶基因位点的活性,为筛选gRNA提供了有效手段,是应用CRSPR/Cas9基因编辑技术的非常好的辅助工具。本研究利用Cas9、OsU3p- PDS、RNPs和Cas12a-PDS对香蕉原生质体系统进行转化。通过深度扩增子测序,发现Cas9系统的突变效率高于相同靶点的RNP;例如Cas9和OsU3p- PDSt4 (0.65%) > PDSt4- rnp (0.27%), Cas9和OsU3p- PDSt7 (0.55%) > PDSt7- rnp (0.42%), Cas9和OsU3p- PDSt9 (0.42%) > PDSt9- rnp(0.19%)。在9个靶位点中,Cas9和OsU3p - PDSt8对检测到的Cas9系统的编辑效率最高,突变效率为1.04%;检测到的RNP系统突变效率最高的是PDSt6-RNP,突变效率为0.92%;检测到的Cas12a系统编辑效率最高的是Cas12a- PDSt9,其突变效率为0.39%,与预测结果基本一致(图2)。6).
最近,CRISPR/Cas9技术被应用于敲除PDS香蕉基因实现白化突变体20.,21,22并破坏MaGA20ox2基因获得半矮秆香蕉材料[23]。然而,目前还没有关于RNPs转化香蕉原生质体或将RNPs传递到香蕉胚性细胞悬液(ECS)中的报道。虽然有报道称香蕉原生质的护理培养可以获得再生植株[28,29],该方法再生率低,重现性差,再生困难。因此,进一步研究利用Cas9 RNP复合物转化原生质体提高植株再生效率对该技术在香蕉育种中的应用具有重要意义。原生质体转化RNP的突变效率远高于生物轰击将RNP传递到ECS中的突变效率。只要能够提高香蕉原生质体的再生效率,通过peg介导的方法将RNP转化成无外源DNA的再生植株就有很大的潜力。这对香蕉的遗传改良和非转基因新种质的产生具有重要意义。
结论
本研究在水稻和小麦原生质体转化的基础上,对香蕉原生质体转化方法进行了优化和建立,为gRNA活性验证奠定了基础。此外,CRISPR/Cas9、CRISPR/Cas12a和Cas9 RNP复合物可以通过优化的协议成功编辑内源基因。CRISPR/Cas9系统的效率高于其他两种系统。此外,利用peg介导的方法将Cas9-RNP复合物传递到原生质体中,与生物轰击ECS相比具有明显的优势,进一步研究原生质体的植物再生对于成功建立香蕉无dna基因编辑系统至关重要。
方法
植物材料
本研究使用的植物材料是卡文迪什香蕉(穆萨spp。卡文迪什;AAA集团简历“巴喜”);八喜香蕉是一种在中国种植多年的香蕉品种,是中国主要的香蕉品种之一。我们从广东省农业科学院果树研究所获得了“八溪”香蕉的雄花蕾。ECS由广东省农业科学院果树研究所实验室诱导。
香蕉原生质体制备
选择小而均匀分布的ECS传代培养,培养时间约为10 d;取出M2培养基,10 ml酶解液(3.0%纤维素R)−101%分离酶R-10, 0.2%果胶酶Y-23, 15.2 g/L KCl, 7.8 g/L CaCl2,添加MES 100 mg/L, 10%甘露醇,pH 5.7);细胞在摇床中以50 rpm/min孵育6-8 h。显微镜下观察原生质体产量。如果酶解足够,用10ml W5溶液稀释水解液,振荡10s以分离原生质体。采用75 μm膜将酶解溶液过滤到圆底离心管中。100 g离心3 min,用移液管除去上清。原生质体悬浮于15ml W5溶液中,冰上孵育30min,丢弃上清。
gRNA设计与向量构建
共有9个载体针对PDS利用SnapGene软件设计香蕉基因。用Bsa1酶切OsU3p载体,从琼脂糖凝胶中回收片段。串行联合引物(附加文件7:表S7)通过T4连接酶与回收的OsU3p载体连接,生成OsU3p- PDSt1到OsU3p- PDSt9,然后进行转化大肠杆菌DH5α主管细胞。37℃培养过夜后,测序证实的单菌落接种于含有氨苄西林的LB液体培养基中。在37°C 220 rpm下培养过夜后,提取质粒。
CRISPR/Cas12a载体构建
共有11种针对PDS利用SnapGene软件设计香蕉基因。Cas12a载体经Bsa1酶切,从琼脂糖凝胶中回收片段。串行联合引物(附加文件8:表S8)通过T4连接酶与回收的Cas12a载体连接,生成Cas12a- PDSt1到Cas12a- PDSt11,然后进行转化大肠杆菌DH5α主管细胞。37℃过夜培养后,将测序证实的单菌落接种于加卡那霉素的LB液体培养基中。37℃、220 RPM培养过夜后,提取质粒。
sgRNAs在体外的转录
目标位点的特定引物(附加文件)9以OsU3p- PDSt1至OsU3p- pdst9质粒为模板,用高保真酶FastPfu扩增,用EasyPure PCR Kit纯化。纯化的PCR产物通过NEB HiScribe™T7体外转录试剂盒进行转录。体外转录产物用天根RNA纯化试剂盒纯化。
用质粒或Cas9 RNP复合物转化原生质体
原生质体浓度调整为2 × 106-2 × 107用MMG,原生质在冰上孵育。将20个μg质粒加入2 ml离心管中,离心沉淀至管底。向试管中加入200 μl原生质体,并进行轻度混合。加入250 μl 50% PEG 4000,在黑暗中诱导转化30 min。加入900 μl W5溶液停止转染。样品在100 g下离心3 min,丢弃上清。原生质体用1ml W5重悬,26-28℃黑暗培养。
按照上述方法,将10 μg的pUbi-Cas9质粒分别与10 μg的质粒OsU3p- PDSt1 ~ OsU3p- PDSt9、20 μg的质粒Cas12a- PDSt1 ~ Cas12a- PDSt11、Cas9蛋白(20 μg)和sgRNA (20 μg)混合。用PEG法分别转化香蕉原生质体,然后进行暗培养。以pUbi-GFP质粒作为对照进行转化。
PCR-RE检测及单克隆的sanger测序
DNA和RNP转化后,选择具有特异性内切酶位点的靶位点进行PCR - RE检测。提取原生质体基因组DNA。用PFU酶扩增出长度约为1000 bp的含靶点序列。特异PCR产物经Eco471酶切,37℃下酶切2 h,在2%琼脂糖凝胶上电泳分析。为确定是否存在碱基突变的特异性片段,电泳后回收特异性片段,连接到T-blunt载体上进行转化大肠杆菌然后选择DH5α能态细胞进行单克隆Sanger测序。
体外RNP切割
用PCR扩增含有靶标3和靶标4的DNA片段,用EasyPure PCR纯化试剂盒纯化,用无rnase水洗脱。体外裂解反应体系为:Cas9蛋白(1 μg)、sgRNA (1 μg)、靶片段(100 ng)、10 × Cas9反应缓冲液(20 mM HEPES、pH 7.5、150 mM KCl、10 mM MgCl)2, 0.5 mM DTT) 2 μl, RNase-free水可达总容积20 μl。样品在37℃下孵育1 h,然后在65℃下孵育10 min。最后,在2%琼脂糖凝胶上进行电泳检测。
深度扩增子测序
用制备好的DNA或RNP样品转化香蕉原生质体。暗培养4-5天后,12000 RPM离心收集香蕉原生质体,用天根DNA提取试剂盒提取基因组DNA。深度扩增子测序引物(附加文件10表S10,附加文件11:表S11),采用巢式PCR扩增长度约为200 bp的片段。凝胶纯化后送上海盛工生物有限公司进行深度扩增子测序,确定靶序列中是否存在碱基突变及突变类型。
数据和材料的可用性
深度扩增子测序数据可在BioProject id PRJNA637446 (https://www.ncbi.nlm.nih.gov/sra/PRJNA637446), prjna637703 (https://www.ncbi.nlm.nih.gov/sra/PRJNA637703)和PRJNA637699(https://www.ncbi.nlm.nih.gov/sra/PRJNA637699).
缩写
- 挂钩:
-
聚乙二醇
- CRISPR:
-
有规则间隔的短回文重复
- RNP:
-
核糖核蛋白
- PCR-RE:
-
PCR-Restriction酶
- PDS:
-
八氢番茄红素脱氢酶
- 绿色荧光蛋白:
-
绿色荧光蛋白
- ECS:
-
胚性细胞悬液
- sgRNA:
-
Single-guide RNA
参考文献
klerck于JAF。用线性方法分离弯曲原生质体[J]。股票报价。1892;49:463-75。
翘起欧共体。植物原生质体和液泡的分离方法研究[J]。大自然。1960;187:962-3。
王晓明,王晓明,王晓明,等。水稻基因直接转移的研究进展[J]。植物化学通报。1985;3:117-28。
刘建军,刘建军,李建军,等。植物原生质体ti -质粒DNA的离体转化[J]。大自然。1982;296:72-4。
张晓明,李晓明,王晓明,等。植物细胞电穿孔基因的克隆及表达分析[J]。自然科学进展,1985;32(1):1 - 8。
王晓明,王晓明,王晓明,等。烟草花叶病毒RNA对黄瓜花叶病毒的影响[J]。Plant Cell, 1986; 5:57-60。
王晓明,王晓明,王晓明,等。水稻、小麦、高粱等原生质体的电孔效应研究[J]。科学通报,2006;30(5):389 - 389。
Hauptmann RM, Ozisa-Akins P, Vasil V, Tabaeizadeh Z, Rogers SG, Horsch B,等。单子叶植物和双子叶植物电穿孔DNA的瞬时表达[J]。植物学报,1987;6:265-70。
琼斯H,琼斯G,琼斯MGK。电穿孔茄原生质体瞬时基因表达分析[J]。植物化学学报。1989;13(3):591 - 591。
希尔默S,吉尔罗伊S,琼斯RL。观察单个大麦的酶分泌(大麦芽)糊粉质原生质体[J]。植物生理学报。1992;42(2):279 - 86。
邹俊杰,魏凤杰,王超,吴俊杰,Disna R,刘文霞,等。拟南芥钙依赖蛋白激酶CPK10在干旱胁迫下的表达及调控[J]。植物生理学报,2010;32(3):444 - 444。
王晓明,王晓明,王晓明,等。玉米叶肉原生质体信号转导的研究进展[J]。植物生理学报,2001;27(4):444 - 444。
刘绍军,赵玉华,王晓明,等。拟南芥叶肉原生质体的基因表达分析[J]。地理学报,2007;2(7):1565-72。
马提浩,孔祥华,孔祥华,刘建平,等。拟南芥叶肉原生质体的miRNA通路分析[J]。植物学报,2015;8(2):261-75。
李建军,李建军,李建军,等。大麦和小麦颖果糊粉质原生质体瞬时基因表达分析[J]。植物学报。1989;13(1):21-9。
杨建伟,付建新,李军,程磊,李峰,董建峰,等。基于原生质体瞬时基因表达的共免疫沉淀技术在水稻蛋白互作中的应用[J]。植物化学通报,2014,32(1):153-61。
曹建民,姚德明,林峰,蒋梅斯。peg介导的玉米叶肉原生质体瞬时基因表达与沉默系统的研究[J]。植物物理学报,2014,36(5):1271-81。
吴建伟,金俊,权思,Corvalan C,赵世文,金辉,等。CRISPR-Cas9核糖核蛋白在植物基因组编辑中的应用[J]。生物工程学报。2015;33:1164 - 4。https://doi.org/10.1038/nbt.3389。
M michael, Roberto V, J Min-Hee, K ok jae, K Seokjoong, K Jin-Soo,等。利用CRISPR/Cas9核糖核蛋白对葡萄和苹果原生质体进行无dna编辑[J]。植物科学,2016;7:1904。
胡超,邓国光,孙晓明,左超武,李云云,邝瑞麟,等。CRISPR/ cas9介导的香蕉高效基因编辑系统的建立[J]。中国农业科学,2017;50(7):1294-301。
Kaur N, Alok A, Shivani KN, Pandey P, Awasthi P,等。CRISPR/ cas9介导的植物烯去饱和酶(PDS)高效编辑在香蕉cv中的精确操作Rasthali基因组。函数集成基因组学,2018;18(1):89-99。
Fatima N, Benjamin D, Jennifer K, Anthony B, Kylie S, Peter W,等。利用CRISPR/Cas9基因编辑卡文迪什香蕉植物烯去饱和酶等位基因[J]。转基因学报,2018;27:451-60。
邵晓华,吴斯平,窦天天,朱慧慧,胡超,霍海琦,等。利用CRISPR/Cas9基因组编辑系统进行创建MaGA20ox2转基因香蕉[J]。植物学报,2019;18(1):17-9。
宗勇,王玉平,李超,张锐,陈克林,冉勇,等。Cas9-胞苷脱氨酶融合在水稻、小麦和玉米中的精确碱基编辑[J]。生物工程学报,2017;35(5):438-41。
梁震,陈克林,李道德,王玉平,赵强,刘建新,等。利用CRISPR/Cas9核糖核蛋白复合物对面包小麦进行无dna基因组编辑[J]。生态学报,2017;8:1461。
梁震,陈克林,张勇,刘建新,尹克强,邱建林,等。利用CRISPR/Cas9基因的体外转录本或核糖核蛋白进行面包小麦基因组编辑[J]。地理学报,2018;13(3):413-30。
李建军,李建军,李建军。CRISPR-Cas9核糖核蛋白复合物在玉米基因组编辑中的应用[J]。中文信息学报,2016;7:13274。
王晓明,王晓明,王晓明,等。香蕉原生质体直接体细胞胚再生技术的研究进展[J]。植物学报,1993;12:403 - 407。
张建军,张建军,张建军,等。香蕉种质资源的克隆与利用[J]。植物学报。1998;49:455 - 62。
致谢
作者感谢高彩霞的技术支持和提供Cas9、gRNA和Cas12a载体。
资金
国家重点研发计划项目(2018YFD1000302和2019YFD1000901)、国家自然科学基金项目(31872939)、广东省现代农业创新团队项目(2018LM2150和2019KJ106)、广东省重点领域研发计划项目(2018B020202005)、广东省科技计划项目(2015B070701011)、广州市科研计划项目(201904020033)。农业科学院高水平科研创新战略建设专项资金(R2017PY-JX001)。研究的设计、数据的收集、分析、解释和撰写稿件均由所有作者完成。资助机构只是提供财政支持。
作者信息
从属关系
贡献
G.Y, K.C.设计研究;白雪,H.Z。学生论文,Q.Y,临睡时,f . B。刘昀,进行研究;s.w., h.z.分析数据;白雪,H.Z得出,Q.Y. wrote the paper. The authors have read and approved the manuscript.
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
所有列出的共同作者都对稿件做出了贡献,审阅了稿件并同意其出版BMC植物生物学。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1:表S1。
Cas9系统的深度扩增子测序结果。
附加文件2:表S2。
Cas12a系统的深度扩增子测序结果。
附加文件3:表S3。
RNP系统的深度扩增子测序结果。
附加文件4:表S4。
Cas9系统的脱靶效应。
附加文件5:表S5。
RNP系统的脱靶效应。
附加文件6:表S6。
脱靶检测引物对。
附加文件7:表S7。
用于构建OsU3p-PDS的引物对。
附加文件8:表S8。
构建Cas12a-PDS的引物对。
附加文件9:表S9。
用于sgRNA体外转录的引物对。
附加文件10:表S10。
用于Cas9和RNP系统捕获测序的引物对。
附加文件11:表S11。
用于Cas12a系统捕获测序的引物对。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
吴生,朱华,刘杰,et al。基于DNA和CRISPR/Cas9核糖核蛋白复合物的香蕉peg介导原生质体转化体系的建立BMC Plant Biol20.425(2020)。https://doi.org/10.1186/s12870-020-02609-8
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-020-02609-8
关键字
- PEG-mediated
- 原生质体转化
- 深度扩增子测序
- 基因组编辑
- DNA-free