摘要
背景
纤维品质是棉花的重要经济性状,纤维品质的改善是棉花育种的主要目标。为了更好地理解纤维品质性状的遗传机制,我们进行了全基因组关联研究,以识别和挖掘纤维品质相关的数量性状位点(qtl)和基因。
结果
共鉴定出42个单核苷酸多态性(SNPs)和31个与5个纤维品质性状显著相关的qtl。在前期研究中共鉴定出25个qtl,本研究首次鉴定出6个新的qtl。在QTL区域,共鉴定出822个基因,并根据其表达谱将其划分为4个簇。我们还发现了两个多效snp。SNP位点i52359Gb与纤维伸长、强度、长度和均匀度相关,而i11316Gh与纤维强度和长度相关。而且,这两个snp是不同义的,位于基因中Gh_D09G2376而且Gh_D06G1908,分别。RT-qPCR分析表明,这两个基因在棉纤维发育的一个或多个阶段优先表达,这与RNA-seq数据一致。因此,Gh_D09G2376而且Gh_D06G1908可能与纤维发育过程有关。
结论
本研究结果为研究棉花品质性状的遗传基础提供了依据,所鉴定的qtl或基因可用于棉花品质改良育种。
背景
棉(Gossypium是一种重要的经济作物,也是世界各地纺织工业天然纤维的主要来源[1].陆地棉(陆地棉属异源四倍体植物,具有适应性广、产量高的优点。因此,它被用来生产世界上大约95%的棉纤维[2].在过去的几十年里,棉花育种者主要集中在提高棉花产量上。目前,随着消费者生活品质的提高和纺织技术的进步,对高品质纤维的需求不断增加[3.,4].因此,提高纤维品质已成为棉花育种的新目标。然而,通过常规育种来提高陆地棉的纤维品质已经被证明是低效的[5].因此,明确陆地棉纤维品质性状的遗传基础具有重要意义,分子标记在优质棉花育种中发挥重要作用。
棉花的纤维品质性状主要包括纤维伸长(FE)、纤维马克隆(FM)、纤维强度(FS)、纤维长度(FL)和纤维均匀度(FU)。这些都是受环境因素影响的复杂数量性状,尽管它们主要受遗传因素控制[5].在棉花基因组发表之前,棉花纤维品质性状研究主要采用双亲本分离群体连锁分析方法。迄今为止,利用该作图方法已报道了分布在棉花26条染色体上的近1000个与纤维品质性状相关的qtl [6,7].这些研究为棉花纤维相关遗传研究提供了重要信息,并促进了棉花纤维品质育种的发展。
近年来,随着棉花基因组序列的释放[8,9,10]以及分子标记技术的迅速发展[11,12],已在棉花的全基因组水平上鉴定出大量的单核苷酸多态性(SNP)标记。此外,关联作图已被广泛用于发现棉花复杂性状的遗传基础[13,14,15,16].与传统的连锁作图相比,关联作图可以利用自然群体揭示基因型与表型之间的关联,并在一项研究中同时检测到许多自然等位变异[17].利用这种作图方法,已鉴定出许多与纤维品质相关的qtl和候选基因[18].例如,Sun等人(2017)对719份陆地棉不同种质和10511个使用棉花snp63k阵列识别的多态snp进行了纤维品质性状的全基因组关联研究(GWAS)。他们总共确定了46个与5个纤维品质性状相关的显著snp。此外,结合GWAS和转录组分析发现了19个与FL和FS相关的有前途的基因[19].黄等人(2017)使用含有503的自然群体检测到79个与纤维品质性状相关的显著snpg .分子棉花snp63k基因分型[20.].此外,基因GhXI-K,GhFL1而且GhFL2使用关联映射法也确定与纤维品质性状相关[13,21].
在我们之前的研究中,利用cotton snp63k阵列对276份陆地棉材料进行了基因分型,鉴定出10660个高品质snp [22].在此,我们利用276份不同种质和10660份高质量单核苷酸多态性(SNPs)进行了GWAS分析,以确定遗传位点与纤维品质性状之间的相关性。此外,我们还挖掘了潜在的纤维品质性状的候选基因,并通过RNA-seq和RT-qPCR分析对它们进行了评估。本研究结果对棉花纤维品质性状的改良育种具有一定的指导意义。
结果
纤维品质性状的表型表征
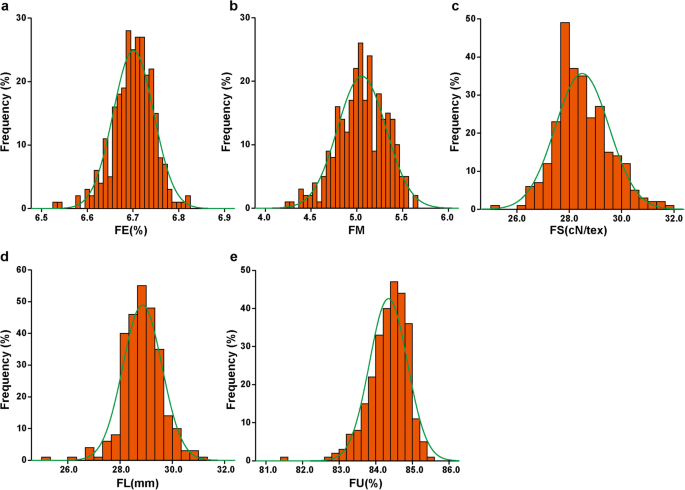
276份来自不同生态区的陆地棉种质资源,表型数据的最佳线性无偏预测(BLUPs)显示出丰富的多样性。FE、FM、FS、FL和FU的取值范围分别为6.53% ~ 6.82%、4.24 ~ 5.63、25.08 ~ 31.69 cN/tex、25.17 ~ 31.02 mm和81.55% ~ 85.41%,平均值分别为6.70%、5.05、28.49 cN/tex、28.85 mm和84.35%1).各纤维品质性状均呈正态分布(图1)。1),说明这些性状是由多基因控制的数量性状。FE、FM、FS、FL和FU的变异系数cv分别为0.66%、5.24%、3.61%、2.60%和0.61%(见表1)1).
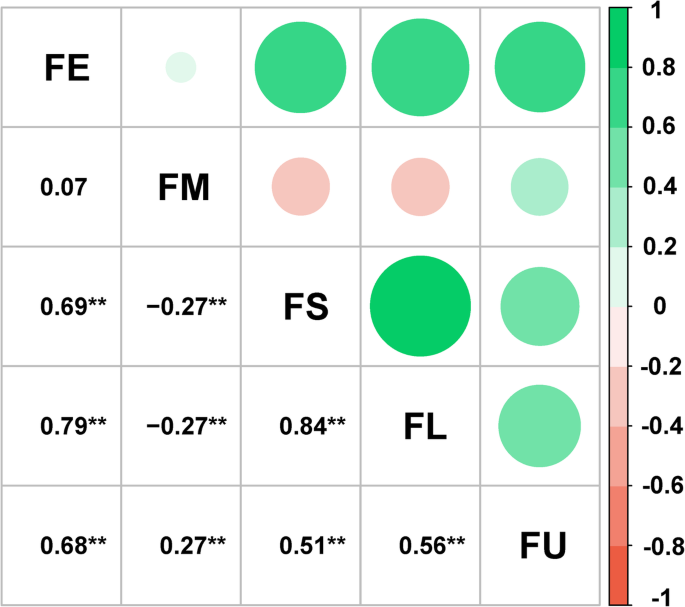
5个纤维品质性状的相关分析表明,5个纤维品质性状之间存在极显著的相关关系(图;2).FE、FS、FL和FU 4个性状间呈显著正相关(0.51 ~ 0.84),FM与FS和FL呈显著负相关(0.51 ~ 0.84)。此外,基因型(G)、环境(E)以及基因型与环境(G × E)的交互作用(P< 0.001)的纤维性状(附加文件1:表S1)。这表明纤维品质性状受基因型和环境的双重影响。广义遗传力(H2)的FE、FM、FS、FL和FU分别为73.31%、91.38%、84.78%、84.54%和72.06%(表1),说明纤维品质性状主要受遗传效应控制。
纤维品质性状GWAS
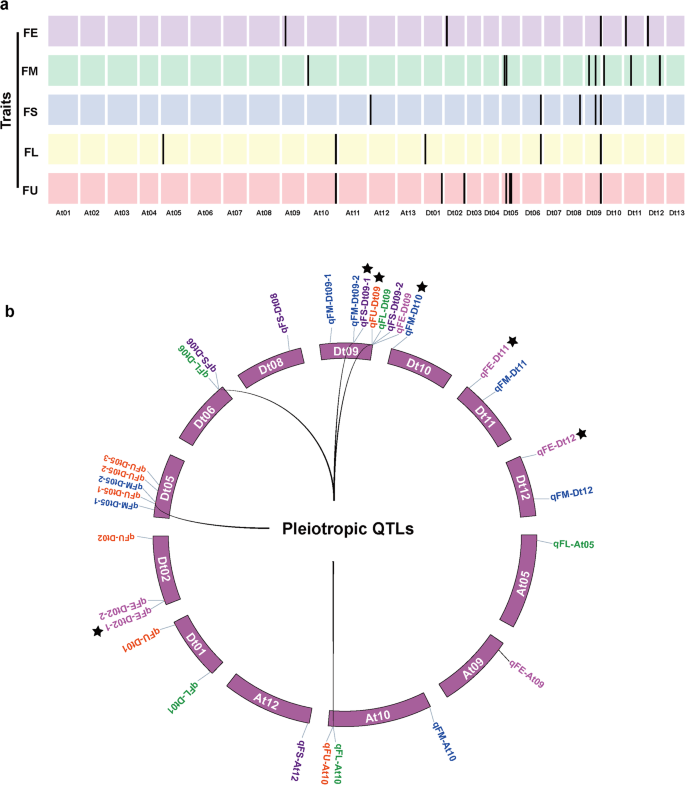
为了确定影响纤维品质性状的遗传因素,我们结合CottonSNP63K阵列识别的10660个高质量SNP标记和来自多个环境的表型数据,对5个纤维品质性状进行了GWAS分析。利用BLUP值共检测到42个与5个纤维品质性状显著相关的snp(图3)。3.一个额外的文件2:图S1和附加文件1:表S2)。它们分布在13条染色体上,包括At05、At09、At10、At12、Dt01、Dt02、Dt05、Dt06、Dt08、Dt09、Dt10、Dt11和Dt12。此外,9个和33个显著snp分别位于At和Dt亚基因组。
FE在At09、Dt02、Dt09、Dt11和Dt12染色体上发现了10个显著的单核苷酸多态性。各SNP解释的表型变异(PVE)范围为3.00% ~ 4.41%1:表S2)。其中,i52264Gb位点对FE有正向影响,−log最高10(P)值(4.18),i39571Gh对FE有负面影响,以−log最低10(P)值(3.02)。
在FM中,在At10、Dt05、Dt09、Dt10、Dt11和Dt12染色体上检测到11个显著的单核苷酸多态性,解释了3.06% ~ 3.98%的表型变异1:表S2)。在这些snp中,基因座i35898Gh对FM的负面影响最高,为−log10(P)值(3.73),i09491Gh对FM有正向影响,−log影响最小10(P)值(3.00)。
FS在12、Dt06、Dt08和Dt09位点发现了8个显著的snp位点。他们解释了3.33%-4.65%的表型变异(补充文件1:表S2)。基因座i52359Gb对FS有负面影响,最高为−log10(P)值(4.03),而i30183Gh对FS有正向影响,最低的为−log10(P)值(3.04)。
FL在At05、At10、Dt01、Dt06和Dt09染色体上观察到6个显著的snp。他们解释了3.31%-4.16%的表型变异(补充文件1:表S2)。基因座i52359Gb对FL有负性影响,最高为- log10(P)值(3.66),i33845Gh对FL有正向影响,最低为−log10(P)值(3.02)。
FU在At10、Dt01、Dt02、Dt05和Dt09染色体上发现11个显著的snp位点,占表型变异的2.64% ~ 4.19%(附加文件)1:表S2)。基因座i09849Gh对FU的负向影响最高,为−log10(P)值(4.42),i65397Gm对FU有正向影响,最低为−log10(P)值(3.00)。
qtl的鉴定与多效性
根据前人对QTL的定义[19,22],共检测到31个qtl,其中FE 6个,FM 8个,FS 5个,FL 5个,FU 7个(图3)。3.b).这些qtl分布在不同的染色体上,包含显著相关的snp。在这些qtl中,有6个(qFE-Dt02-1,qFE-Dt11,qFE-Dt12,qFM-Dt10,qFS-Dt09-1而且qFU-Dt09)为新发现的qtl,剩余的qtl与之前发现的qtl重叠(图1)。3.b、附加文件1:表S3)。
在复杂农艺性状中,基因连锁和多效效应是常见现象[23].在我们的研究中,两个显著的SNPs (i52359Gb和i11316Gh)表现出多效性,分别与四个和两个纤维性状相关(附加文件1:表S3)。同时,含有这些显著snp的qtl被锚定在相同的基因组区域。例如,有4个qtl (qFE-Dt09,qFS-Dt09-2,qFL-Dt09而且qFU-Dt09),与不同纤维性状相关,同时定位于Dt09基因组区50.51-50.91 Mb。在Dt06上,两个qtl (qFS-Dt06而且qFL-Dt06),具有相同的SNP位点,均位于相同的基因组区间(59.8-60.2 Mb)。此外,一些qtl在物理位置上与其他qtl重叠或相邻,如qFL-At10/qFU-At10,qFM-Dt05-2/qFU-Dt05-1而且qFM-Dt09-2/qFS-Dt09-1(无花果。3.b)这些结果表明,这些纤维性状可能受一个多效应QTL网络控制。
QTL区基因表达谱分析
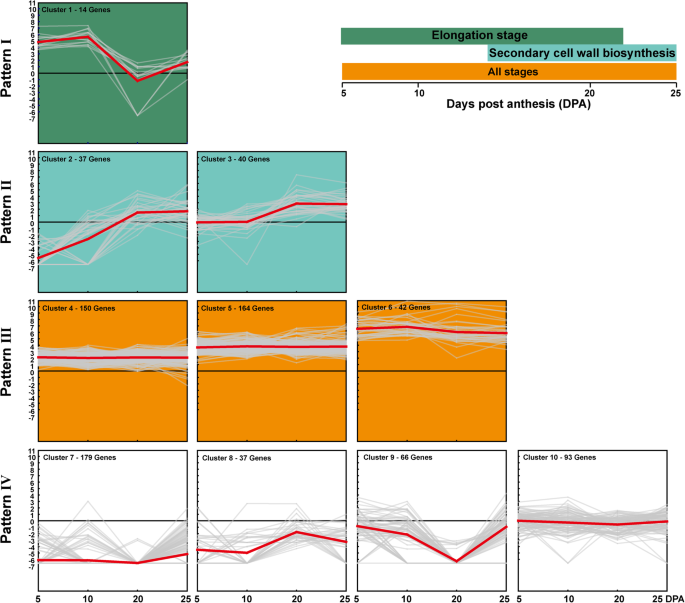
基于g .分子TM-1参考基因组序列[10],在这些QTL区域共检测到822个基因(补充文件1:表S4)。这些基因在整个基因组中分布不均匀,At和Dt亚基因组中分别有154和668个基因(附加文件3.:图S2)。此外,不同染色体的基因数量也不同。基因数最多的是在Dt05上,共有188个基因,而在At09上只有14个基因。此外,我们利用NCBI Sequence Read Archive (SRA)数据库中获得的纤维发育的转录组数据研究了这些基因的表达模式。结果显示,这些基因可以分为四种模式,I-IV(图;4).模式一包含14个在花期后5 ~ 10天(DPA)高表达的基因。模式II包括两个簇(簇2和3),包含77个优先表达于次级细胞壁合成阶段的基因(20-25 DPA)。模式III包含356个基因,在所有纤维发育阶段保持高表达水平。模式IV包含375个在所有纤维发育阶段低表达水平的基因。这表明这些基因可能参与了纤维的发育,并在纤维发育的不同阶段发挥重要作用。
与纤维品质相关的多基因多态性分析
在植物中,许多性状是由多个基因控制的,有些基因对产量性状或其他性状具有多效作用[20.].基因多效性是指一个基因决定或影响多种性状的形成[24].在本研究中,我们在两条染色体Dt09和Dt06上发现了具有多个纤维品质性状的多性状关联的单基因组区域。
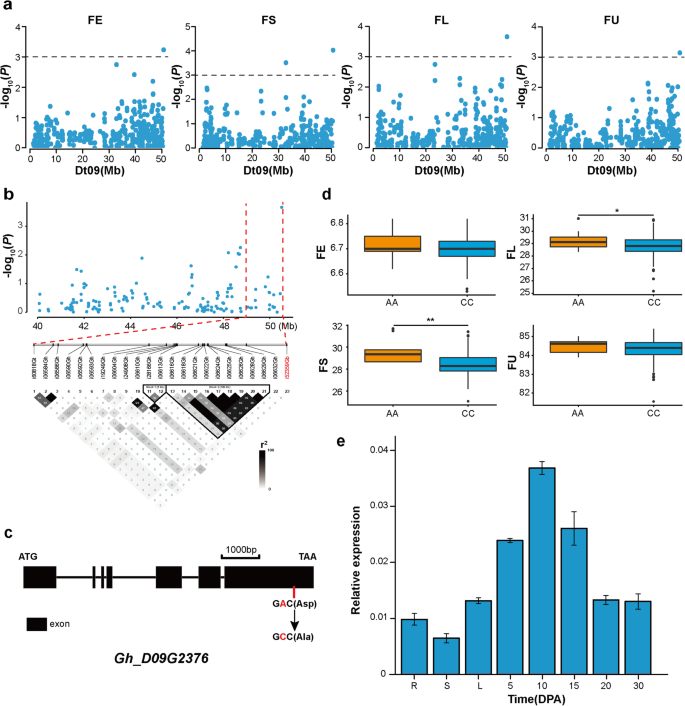
在Dt09上,关联信号位于49.0-50.7 Mb(图;5).在该候选区域,一个SNP与4个纤维品质性状(FE、FS、FL和FU)显著相关(图3)。5A)和位于基因内Gh_D09G2376,该蛋白编码一个Jumonji N/C和锌指结构域蛋白(图。5c).连锁不平衡(LD)区块分析显示,该区域存在两个区块,但显著相关的SNP不属于任何区块(图2)。5b).而且,i52359Gb是一个非同义SNP,在基因编码区7215 bp的位置从a突变到CGh_D09G2376,导致氨基酸由天冬氨酸(Asp)转变为丙氨酸(Ala)(图1)。5c).有趣的是,该SNP位点产生的两个基因型与这些纤维性状的表型表现相关。AA基因型材料的FE、FS、FL和FU值均高于CC基因型材料。特别是FS和FL在这两种基因型之间存在显著差异(图。5d). RT-qPCR分析显示该基因在纤维伸长阶段(10 DPA)高表达(图1)。5e),该基因在优质陆地棉品种新陆早30的10 DPA表达量显著高于低质陆地棉品种苏康191(附加文件)4图S3a和b).在棉花中,其功能Gh_D09G2376基因是未知的。然而,其同源基因在拟南芥,JMJ12(At3g48430)编码一个含有Jumonji N/C和锌指结构域的蛋白质,并在细胞伸长中发挥重要作用5:图S4) [25,26].因此,我们推测这是一个控制FE、FS、FL和FU的多效基因。
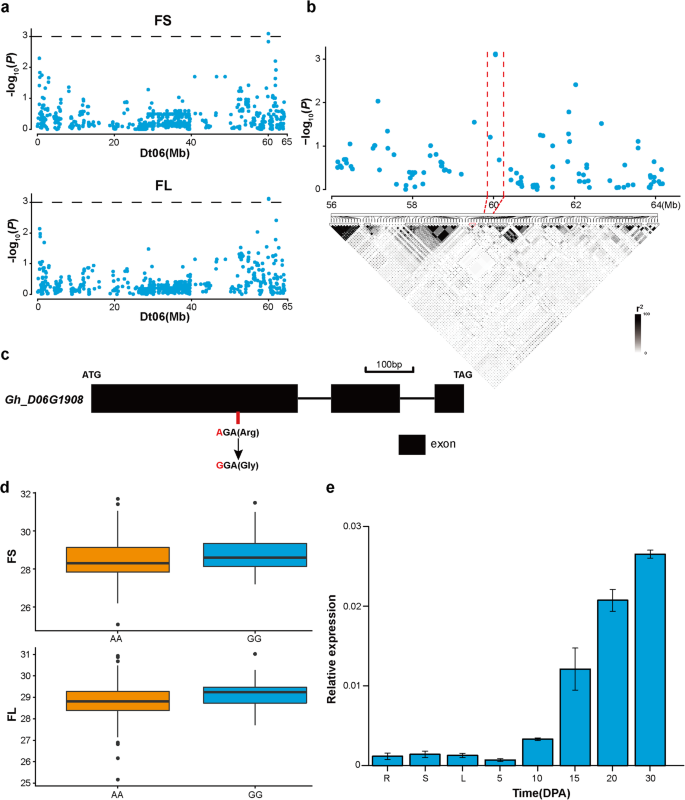
此外,在Dt06上59.8-60.2 Mb范围内,还有另一个与FS和FL相关的关联信号(图。6).在这个候选区域有4个snp,其中2个位于一个LD块内(图;6在这个区块中,只有SNP i11316Gh与这些性状显著相关,并且位于该基因中Gh_D06G1908.该SNP导致该基因310 bp处的氨基酸由精氨酸(Arg)转变为甘氨酸(Gly)(图3)。6c).根据该SNP的等位基因,将这些材料分为AA和GG两种基因型,其中GG基因型比AA基因型具有更大的FS和更长的FL(图2)。6d).该基因在纤维发育阶段优先表达,在纤维发育过程中表达量增加,在30 DPA时达到峰值(图3)。6e)。我们还发现该基因在优质陆地棉品种(新陆早30)中10、15和20 DPA的表达量高于低质陆地棉品种(苏康191)(附加文件)4:图S3a和c)拟南芥,CYSb(At3g12490的同义词Gh_D06G1908,并编码具有半胱氨酸蛋白酶抑制剂活性的蛋白质(附加文件6:图S5)的过度表达CYSb能刺激植物生长[27].这些结果表明Gh_D06G1908可能参与纤维发育过程。
讨论
纤维品质性状的表型多样性与遗传力
遗传多样性,包括基因型和表型多样性,在关联作图中起着至关重要的作用[28].本研究的基因型多样性程度与以往研究测量的相似[19,20.],它适合GWAS。此外,为了提高表型数据的可靠性,减少环境对GWAS结果的影响,本研究在2年时间内收集了7个环境的表型数据,并估算了5个纤维品质性状的blup值。表型统计结果显示,各性状表型变异幅度在0.61% ~ 5.24%之间(表1)1),这与以往的研究结果一致[19,20.,23].
遗传力是影响关联映射准确性的另一个主要因素[29].一般来说,H2用于判断遗传性状的稳定程度,而H2值大于50%被认为是高[30.].陆地棉纤维品质性状具有较高的遗传力水平。例如,H2Nie等(2016)、Huang等(2017)和Dong等(2018)报告的这些性状的值分别在86% ~ 93%、84% ~ 92%和69.54% ~ 91.05%之间[5,20.,23].在我们的研究中H2各纤维品质性状的值也较高,介于72.06% (FU) ~ 91.38% (FM)之间(见表1)1),这与以往的研究结果一致[5,20.,23].此外,方差分析表明,基因型、环境及其互作对这些性状的方差均显著(补充文件)1:表S1),表明这些性状受环境的影响。然而,基因型效应起主导作用。因此,棉纤维品质性状主要受遗传因素控制,适合进行关联作图分析。
本研究发现了多效snp和qtl
纤维品质性状通常是复杂的数量性状,由复杂的多基因网络控制[31].部分qtl或基因同时调控多种纤维品质性状[13,19].在本研究中,我们利用关联映射方法鉴定了与这5个纤维品质性状相关的42个显著snp和31个qtl1:表S3、表S4,图3.).有趣的是,其中一些显著的snp或qtl(在相同的基因组区间内)与多个性状相关。显著SNP i52359Gb同时与FE、FS、FL和FU相关,另一个SNP i11316Gh同时与FS和FL相关。qFL-At10,qFM-Dt05-2而且qFM-Dt09-2与之重叠或相邻的qFU-At10,qFU-Dt05-1,qFS-Dt09-1,分别。通过对纤维品质性状的相关分析,FE、FS、FL和FU呈显著正相关,FM与FL和FS呈显著负相关。因此,纤维品质性状之间存在较强的相关性,这与以往报道的结果一致[5,15,19].此外,棉纤维品质性状间的相关系数普遍较好[32,33].因此,利用不同纤维品质性状所涉及的基因,可以通过棉花纤维的改良育种,有效地获得所需的纤维品质。
比较我们的GWAS结果与QTL或GWAS结果从以前的研究
近几十年来,利用不同的遗传群体或不同的作图方法,已经发现了大量与棉花纤维品质性状相关的qtl或GWAS信号[18,34].在本研究检测到的与这5个纤维品质性状相关的31个QTL中,有25个QTL位于或邻近于前人研究中确定的QTL区间或GWAS信号范围内(附加文件1:表S3)。对铁、法qFE-At09,qFE-Dt02-2而且qFE-Dt09与DPL0679a重叠[5, nau3308−nau5467 [35),qFE23.1[36,37),分别。FM的六个qtl (qFM-At10,qFM-Dt05-1,qFM-Dt05-2,qFM-Dt09-2,qFM-Dt11而且qFM-Dt12)与之前研究报道的10个qtl相对应,其中一些qtl也定位在TM33781邻近区域[38],qFM-Chr19-1[39], NAU1004 [40], qFM23.2 [37和TM79085 [38].对于FS, qtlqFS-At12,qFS-Dt06而且qFS-Dt08是相关的qFS-A12-1[41], i38606Gh [19和D08_54727428 [42),分别。此外,qFS-Dt09-2是相邻的qFS23.2[37].对于FL, qtlqFL-At05,qFL-At10而且qFL-Dt01重叠的qFL-c10-1[43, TM10319和TM47849 [38]和qtlqFL-Dt06而且qFL-Dt09接近GH185 [44]及TM72969 [38),分别。FU的qtlqFU-At10,qFU-Dt02,qFU-Dt05-1,qFU-Dt05-2而且qFU-Dt05-3重叠的qFU-D5-2[45],qFU14.2[36], HAU1384 [46, TM57244和TM36632 [38]及一个QTL (qFU-Dt01)位于GWAS信号D01:60914905附近[13].因此,本研究的GWAS结果是可靠的,这些通过不同遗传背景的分离群体或不同作图方法同时检测到的稳定遗传qtl /基因在未来棉花品质育种中具有很大的应用潜力。此外,我们发现更多的qtl位于Dt亚基因组中而不是At亚基因组中,这与之前发现Dt亚基因组中含有的纤维质量qtl比At亚基因组多25%的结果一致,这一数据支持了Dt亚基因组在纤维调控机制中发挥更重要作用的猜测[21,30.,47,48].
潜在的候选纤维品质基因
迄今为止,许多与纤维发育阶段相关的候选基因已经通过不同的方法被鉴定出来,例如GhHD1而且GhHOX3通过同源基因克隆[49,50],GhMML3_A12而且GhMML4_D12基于地图的克隆[51,52),GhFL1而且GhFL2GWAS [21].此外,棉纤维的发育是一个复杂的动态过程,包括起始、伸长、二次细胞壁合成和成熟阶段。相应的,纤维发育的调控机制也是复杂的,包括保守的转录因子、植物激素、表观遗传修饰和代谢途径[31].
本研究共鉴定出822个基因在QTL区域(附加文件)1:表S4)。基因表达谱分析表明,许多基因在纤维发育的特定阶段或所有发育阶段优先表达。在这些基因中,有两个候选基因,Gh_D09G2376而且Gh_D06G1908(无花果。5而且6).这两个基因都包含一个显著相关的、引起氨基酸变化的非同义SNP。根据这些非同义snp的两个等位基因,将材料分为两种类型,分别在i52359Gb和i11316Gh携带AA/CC和AA/GG等位基因。基因表达分析显示,这两个基因在纤维发育阶段的表达水平高于其他组织,表明它们可能参与了纤维的发育。
我们重点研究了上的同源注释信息Gh_D09G2376而且Gh_D06G1908.的JMJ12Gene的同义词Gh_D09G2376在拟南芥,编码一个Jumonji N/C结构域蛋白。在拟南芥,BES1新兵JMJ12油菜素甾醇(BR)反应基因的活性和突变JMJ12基因导致细胞伸长受损[25,26].在棉花中,BR是纤维伸长的重要调节剂[53),而BES1基因影响纤维的发育[54].Gh_D06G1908是的同系词拟南芥CYSb编码抑制蛋白酶催化活性的蛋白质的基因。在拟南芥,CYSb-过表达转基因株系比野生型植株表现出更大的鲜重[27].因此,我们推测这两个基因可能是棉花纤维发育的基础。这些候选基因的功能研究需要进一步验证它们在棉纤维发育中的作用。
结论
本研究采用关联作图法对陆地棉纤维品质性状的遗传结构进行了研究。共鉴定出与这些纤维品质性状显著相关的42个snp和31个qtl。共定位到822个基因。此外,Gh_D09G2376而且Gh_D06G1908通过RNA-seq和RT-qPCR分析,筛选出控制纤维品质性状的候选基因。综上所述,本研究发现的snp、qtl和候选基因可用于纤维品质性状的标记辅助选择(marker-assisted selection, MAS),有助于加深对纤维品质分子机制的认识。
方法
植物材料和田间试验
一组276种陆地棉品种被用于GWAS,关于这些品种的详细信息以前已经发表过[22].采用2个重复的完全随机区组设计,分别种植于河南安阳(2016年和2017年)、湖北荆州(2016年和2017年)、新疆阿拉尔(2016年和2017年)、江西九江(2016年)和湖北黄冈(2017年)8个环境。在所有环境中,每个小区长6.0 m,行间距0.8 m,种植20-25株。田间管理遵循当地的农艺做法。
表型数据收集与统计分析
每个小区人工采收25个自然开铃。棉纤维轧净后,每个样品10 - 15g纤维送到中国安阳农业部棉纤维质量检验检测中心,用HVI1000自动纤维检测系统(http://www.uster.cn/en/instruments/fiber-testing-1/uster-hvi-3/),包括FE(%)、FM、FS (cn - tex)、FL (upper-half mean FL, mm)和FU(%)。
为减少环境误差,在R软件中使用lme4软件包计算纤维品质性状的blps [55].BLUP值用于后续的关联映射。在R软件中进行统计分析、不同纤维品质性状间的Pearson线性相关系数和方差分析[56].此外,H2每个性状的值按照之前报道的方法计算[22].
纤维品质性状的关联作图
使用CottonSNP63K阵列进行SNP基因分型,共鉴定出10660个高质量SNP并用于关联图谱。该人群的群体结构、亲缘关系和LD分析以前有报道[22].在这里,我们使用基因组关联和预测集成工具(GAPIT)和混合线性模型(PCA + K)进行了GWAS [57].为了获得更显著相关的SNP,阈值为P= 1.0 × 10−3被选中。使用R软件中的qqman包绘制曼哈顿图[58].使用Haploview 4.2软件绘制了峰值snp附近的LD热图[59].
qtl鉴定及基因表达分析
按照先前报告的方法[19,22],我们选择显著性状相关snp的上下行200 kb区域作为qtlg .分子TM-1参考基因组序列[10].我们的GWAS结果和先前报告的结果的共定位分析是通过先前研究中描述的以下步骤实现的[60:(1)之前报道的QTL和GWAS信号均来自棉花QTL数据库(CottonQTLdb,http://www2.cottonqtldb.org/) [6],这些标记的引物序列从Cottongen数据库(https://www.cottongen.org/) [61];(2)利用电子PCR (e-PCR)方法获得这些qtl的物理位置g .分子TM-1参考基因组序列[10];(3)将上述qtl与本研究中发现的qtl在物理图谱上进行比较。
QTL区域的基因从基因注释中提取g .分子TM-1参考基因组[10].为了研究这些基因的表达谱,利用RNA-seq数据g .分子TM-1组织取自NCBI Sequence Read Archive (SRA)数据库,该数据库已接入PRJNA248163 [10].RNA-seq数据使用TopHat和Cufflinks软件进行处理[62],并使用标准化片段per kilobase per million map read (FPKM)值来表示基因表达水平。
RNA提取及RT-qPCR验证
在5、10、15、20和30个DPA的根、茎、叶和纤维样品g .分子选用优质陆地棉品种新陆棉30和低质陆地棉品种苏康191的TM-1和10、15、20 DPA纤维样品进行总RNA的分离。按照之前的研究进行总RNA提取、cDNA合成和RT-qPCR [22].每个样本进行3次独立的生物重复。GhHis3选择内参基因。采用对比Ct法计算相对基因表达值[63].本研究中使用的所有引物都列在附加文件中1:表S5。
数据和材料的可用性
原始RNA-seq数据可从NCBI序列读取存档(SRA)数据库中获得,该数据库已接入PRJNA248163。本研究过程中产生和分析的所有数据都包含在这篇发表的文章及其补充信息文件中。在当前的研究中产生和分析的数据集可从通信作者在合理的要求。
缩写
- 菲:
-
纤维伸长
- FM:
-
纤维麦克隆
- FS:
-
纤维强度
- FL:
-
纤维长度
- 傅:
-
纤维的均匀性
- GWAS:
-
全基因组关联研究
- 经济价值:
-
数量性状位点
- SNP:
-
单核苷酸多态性
- 通过:
-
表型变异解释
- BLUPs:
-
最佳线性无偏预测
- H2:
-
广义遗传
- 分区:
-
开花后的天数
- LD:
-
连锁不平衡
参考文献
陈志军,张涛,郭伟,陈欣,Stelly DM, Rabinowicz PD, Town CD,等。棉花测序(Gossypium)的基因组。植物杂志。2007;145(4):1303 - 10。
张海斌,李勇,王波,徐培文。棉花基因组学研究进展。植物基因组学杂志。2008;
李志强,于军,李志强,等。棉花纤维品质控制性状的分子定位与鉴定。Euphytica。2001;121(2):163 - 72。
苏健,李丽,庞超,魏华,王超,宋明,王华,赵松,张超,毛刚,等。中国陆地棉与纤维品质性状相关的两个基因组区。Sci众议员2016;6:38496。
聂新,黄超,尤超,李伟,赵伟,沈超,张波,王华,颜智,戴波,等。基于ssr的中国陆地棉自交系品种纤维质量全基因组关联定位BMC基因组学。2016;17:352。
季军,Knapka JA,宋明,张军说:棉花QTLdb:用于棉花QTL分析、可视化和比较的QTL数据库陆地棉而且g .分子xg .取得人群。浙江大学学报(自然科学版),2015;
纪、林珍、张欣、宋敏、张军介绍了四倍体棉花纤维品质、产量、产量相关性状和形态性状、耐旱性和抗病性的综合QTL分析。BMC基因组学。2013;14:776。
李锋,范刚,王凯,孙锋,袁勇,宋刚,李强,马智,陆晨,邹晨,等。栽培棉花的基因组序列木本棉.Nat麝猫。2014;46(6):567 - 72。
刘志强,刘志强,刘志强,刘志强,刘志强,等。重复多倍化Gossypium可纺棉纤维的基因组与进化。大自然。2012;492(7429):423 - 7。
张涛,胡艳,蒋伟,方磊,关旭,陈杰,张杰,Saski CA, Scheffler BE, Stelly DM,等。异源四倍体棉花测序(陆地棉l . acc。TM-1)为纤维改良提供了一种资源。Nat生物技术。2015;33(5):531 - 7。
胡尔思-坎普,兰姆J, Plieske J, Ashrafi H, Buyyarapu R,方DD, Frelichowski J, Giband M, Hague S, Hinze LL,等。棉花63K单核苷酸多态性阵列的建立及棉花种内和种间群体的高密度定位Gossypiumspp。G3(贝塞斯达)。2015; 5(6): 1187 - 209。
蔡超,朱刚,张涛,郭伟。高密度80k SNP芯片是基因分型的有力工具g .分子材料和基因组分析。BMC基因组学。2017;18(1):654。
方磊,王强,胡勇,贾勇,陈健,刘波,张智,关旭,陈松,周波,等。棉花基因组分析鉴定了与棉花品质和产量性状相关的选择特征和位点。Nat麝猫。2017;49(7):1089 - 98。
李涛,马旭,李宁,周丽,韩慧,韩华,桂宇,鲍勇,陈健,戴旭。全基因组关联研究发现的候选基因轮枝菌属陆地棉抗枯萎病(陆地棉l .)。植物学报,2017;15(12):1520-32。
林林王M,你L, M, Z,王P,杨问,你们Z,沈C,李J,张L,等。棉花驯化过程中的不对称亚基因组选择和顺调控分化。Nat麝猫。2017;49(4):579 - 87。
孙智,李华,张勇,李智,柯华,吴玲,张刚,王旭,马智。棉花幼苗期耐盐相关snp及候选基因的鉴定(英文)陆地棉l .)。植物学报(英文版). 2018;
黄欣,韩斌。作物植物的自然变异与全基因组关联研究。中国植物学报。2014;35(5):531 - 532。
Yu JZ, Gervers KA。陆地棉标记相关纤维发育基因的基因组分析(陆地棉L)。Euphytica。2019;215(4):74。
孙智,王旭,刘智,顾强,张勇,李智,柯华,杨健,吴健,吴玲,等。陆地棉的全基因组关联研究发现了陆地棉纤维品质性状的遗传变异和候选基因。Plant biotechnology . 2017;15(8): 982-96。
黄超,聂新,沈超,游超,李伟,赵伟,张欣,林志。高密度snp关联分析中国陆地棉群体结构及农艺性状的遗传基础。植物学报,2017;15(11):1374-86。
马智,何松,王旭,孙健,张勇,张刚,吴丽,李智,刘智,孙刚,等。对陆地棉核心种质进行重测序,确定了影响棉花品质和产量的基因组变异和位点。Nat麝猫。2018;(6):803 - 13。
宋超,李伟,裴旭,刘勇,任智,何凯,张峰,孙凯,周旭,马旭,等。陆地棉的遗传变异和衣分候选基因的全基因组关联研究中国科学:地球科学。2019;32(7):991 - 998。
董超,王杰,余宇,鞠丽,周旭,马旭,梅刚,韩智,思智,李波,等。识别功能基因陆地棉纤维质量。植物学报,2018;9:68。
杨超,李超,王强,钟东,赵华。生物医学大数据挖掘的机遇与挑战——生物医学大数据挖掘的新视角。麝猫。2015;6:229。
于欣,李玲,李玲,郭敏,张建军,尹勇。油菜素甾醇类激素调控蛋白的研究进展ELF6而且REF6在拟南芥.中国科学:地球科学(英文版)2008;
吕芳,崔欣,张松,张健,曹鑫。拟南芥REF6是一个组蛋白H3赖氨酸27去甲基化酶。Nat麝猫。2011;43(7):715 - 9。
张欣,刘松,高野涛。两种半胱氨酸蛋白酶抑制剂拟南芥,AtCYSa而且AtCYSb,增加耐盐、耐旱、抗氧化和耐寒性。植物化学学报。2008;24(3):393 - 393。
李晨,王艳,艾娜,李艳,宋杰。基于棉花snp80k序列的陆地棉早熟性状全基因组关联研究。中国生物医学工程学报,2018,30(3):393 - 393。
在植物中已声明的QTL中虚假QTL的比例是多少?中国生物医学工程学报。2004;10(2):419 - 424。
张超,李丽,刘强,顾玲,黄健,魏华,王慧,于松,陆棉纤维长度相关基因座及候选基因的鉴定(陆地棉L.)通过关联映射和连锁分析。前面。植物科学。2019;10:53。
王智,杨智,李峰。水稻分枝毛状体发育的分子机制研究进展拟南芥不分枝的棉花。植物学报,2019;17(9):1706-22。
陆棉品质参数与铃内产量组成的关系。作物科学。1997;37:1775-9。
mcall LL, Verhalea LM, McNew RW。陆地棉纤维强度的多向选择。作物科学。1986;26:744-50。
季敏,宋明,王辉,林志,张欣,方德东,张军。种内QTL的比较meta分析陆地棉和种间g .分子xg .取得人群。浙江大学学报(自然科学版),2015;
王杰,郭伟,张涛。棉花品种豫棉1号纤维品质性状的QTL定位。作物学报。2007;33(12):1915-21。
谭智,张智,孙旭,李强,孙勇,杨鹏,王伟,刘旭,陈超,刘东,等。陆地棉snp80k序列遗传图谱构建及纤维品质QTL定位植物学报。2018;9:225。
刘旭,滕智,王健,吴涛,张智,邓旭,方旭,谭智,阿里I,刘丹,等。丰富陆地棉种内遗传图谱并确定不同环境下纤维品质和产量组成性状的QTL (陆地棉l .)。基因组学杂志。2017;292(6):1281-306
李晨,付艳,孙锐,王艳,王强。陆地棉纤维品质性状的单位点和多位点全基因组关联研究(英文)陆地棉l .)。前面。植物科学。2018;9:1083。
尚玲,王勇,王旭,刘峰,阿卜都威利,蔡松,李勇,马玲,王凯,华杰。陆地棉重组自交系及其回交群体纤维性状的遗传分析与QTL检测。G3(贝塞斯达)。2016; 6(9): 2717 - 24。
蔡超,叶伟,张涛,郭伟。陆地棉品种/材料纤维品质性状关联分析及优良等位基因的探索(陆地棉l .)。中国生物医学工程学报,2014,29(1):357 - 362。
司智,陈慧,朱鑫,曹智,张涛。皮棉产量与纤维品质性状的遗传解剖g .分子在g .取得背景。摩尔品种。2017;37(1):9。
苏娟,马琴,李敏,郝峰,王超。中国早熟陆地棉纤维品质相关性状的多位点全基因组关联研究。植物学报(英文版). 2018;
王慧,黄超,赵伟,戴斌,沈超,张斌,李丹,林贞。陆地棉两个永生回交群体纤维品质和产量性状QTL的鉴定。《公共科学图书馆•综合》。2016;11 (12):e0166970。
聂X,你J,王B,林周X, z人口来自基本脉冲电平g .分子而且g .取得为棉花遗传育种提供资源。PloS one。2015; 10 (10): e0141064。
胡伟,张旭,张涛,郭伟。陆地棉优良品质QTL的分子标记及来源分析。作物学报,2008;34(4):578-86。
董清源,王杰,于宇,李宝斌,陈启军。陆地棉纤维品质性状的关联定位和有利QTL等位基因(陆地棉l .)。J麝猫。2018;97 (1):e1-e12。
J荣,Feltus FA, waghare VN, Pierce GJ, Chee PW, Draye X, Saranga Y, Wright RJ, Wilkins TA, May OL,等。对棉花多倍体QTL的荟萃分析表明,棉纤维发育过程中各亚基因组对一个复杂的基因网络和基因簇的贡献是不等的。遗传学。2007;176(4):2577 - 88。
方德东,邓德东,王志强,李萍,吴军。陆地棉纤维品质性状的数量性状位点分析(英文)陆地棉l .)。BMC基因组学。2014;15:397。
Walford SA, Wu Y, Llewellyn DJ, Dennis ES。同源结构域亮氨酸拉链基因介导的棉花表皮细胞分化GhHD-1.植物j . 2012; 71(3): 464 - 78。
单春梅,上官XX,赵波,张晓峰,晁丽玲,杨春青,王丽娟,朱海燕,曾永德,郭文智,等。同源结构域转录因子对棉纤维伸长的控制GhHOX3.Nat Commun。2014;5:5519。
万强,关旭,杨宁,吴华,潘明,刘波,方磊,杨松,胡勇,叶伟,等。来自双向转录的小干扰rnaGhMML3_A12调节棉纤维发育。新植醇。2016;210(4):1298 - 310。
吴华,田勇,万强,方磊,关旭,陈健,胡勇,叶伟,张慧,郭伟,等。遗传与进化MIXTA调控棉绒纤维发育的基因。新植醇。2018;217(2):883 - 95。
杨智,张超,杨旭,刘凯,吴智,张旭,郑伟,荀强,刘超,陆磊,等。PAG1它是一种棉花油菜素内酯分解代谢基因,调节纤维伸长。新植醇。2014;203(2):437 - 48。
刘智,甘甘伯,陆璐,秦伟,刘军,李军,马胜,杨智,杨震BES1基因在Gossypium揭示了它们在油菜素内酯信号转导中的进化保守作用。中国科学:生命科学。2018;31(12):1566 - 1572。
刘志刚,刘志刚。线性混合效应模型的拟合方法。J统计软件。2015;67(1):1 - 48。
团队RDC。R:统计计算的语言和环境。R统计计算基金会,维也纳,奥地利。计算。2014;14:12-21。
张志强,王琪,李敏,张志强,张志强,张志强,张志强,张志强。基因组关联与预测的研究进展。生物信息学。2012;28(18):2397 - 9。
特纳SD。用Q-Q和曼哈顿图将GWAS结果可视化的R程序包。bioRxiv。2014;(5): 005165。https://doi.org/10.1101/00516.
Barrett JC, Fry B, Maller J, Daly MJ。Haploview: LD和单体型图的分析和可视化。生物信息学。2005;21(2):263 - 5。
苏娟,范松,李丽,魏华,王超,王华,宋明,张超,顾磊,赵松,等。GWAS法检测中国陆地棉优良QTL等位基因及衣分候选基因。植物学报。2016;21(4):433 - 436。
于军,郑绍荣,程昌昌,李涛,郑平,Jones D, Percy RG, Main D. D.棉花基因组学与育种数据库。中国生物医学工程学报(英文版)2014;
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, Pimentel H, Salzberg SL, Rinn JL, Pachter L.用TopHat和袖扣进行RNA-seq实验的差异基因和转录本表达分析。Nat Protoc。2012;7(3):562 - 78。
Schmittgen TD, Livak KJ。采用比较C(T)法分析实时荧光定量PCR数据。Nat Protoc。2008;3(6):1101 - 8。
确认
感谢彭火(中国农业科学院棉花研究所郑州研究中心,中国郑州)的技术支持。
资金
国家自然科学基金项目(31901580)和河南省科技重点项目(192102110032和182102110306)资助。这些资助机构在研究的设计、数据的收集、分析和解释以及手稿的写作中没有任何作用。
作者信息
从属关系
贡献
WL (Li Wei)、DGY和XXW构思和设计了这项研究。WL(刘伟)、CXS、ZYR、ZQZ、JJZ和JZ进行了实验。XYP、YGL、KLH和FZ制备了该材料。WL (Wei Liu)和CXS对数据进行分析,撰写论文。WL(魏力)、DGY和XXW对手稿进行了修改。WL(李伟)指导实验和指导研究。所有作者已阅读并批准最终稿。
相应的作者
道德声明
伦理认可和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格《自然》对出版的地图和机构附属关系中的管辖权要求保持中立。
补充信息
附加文件1:表S1。
棉纤维品质性状的方差分析。表S2。GWAS检测到棉纤维品质性状的显著单核苷酸多态性。表S3。将本研究中发现的qtl与以往研究中发现的qtl进行比较。表S4。这些基因在QTL区域的注释信息。表S5。本研究中RT-qPCR引物序列。
附加文件2:图S1。
曼哈顿图以及FE、FM、FS、FL和FU的分位数图。虚线表示显著性阈值(P< 10−3).
附加文件3:图S2。
这些QTL区域的基因分布。
附加文件4:图S3。
表达水平的比较Gh_D09G2376而且Gh_D06G1908在新路勉30号和素康191号之间。一个新陆棉30和苏康191纤维品质性状的箱型图。b的表达Gh_D09G2376通过RT-qPCR检测新路棉30和苏康191的基因表达。c的表达Gh_D06G1908通过RT-qPCR检测新路棉30和苏康191的基因表达。GhHis3被用作管家基因。误差条表示三个独立的生物重复实验的标准差。采用双尾t检验方法,**表示显著性水平为0.01。
附加文件5:图S4。
同源性分析Gh_D09G2376.一个系统发育树Gh_D09G2376而且拟南芥JMJ基因家族。b蛋白质的结构Gh_D09G2376而且At3g48430 / JMJ12.
附加文件6:图S5。
同源性分析Gh_D06G1908.一个系统发育树Gh_D06G1908而且拟南芥半胱氨酸基因家族。b蛋白质的结构Gh_D06G1908而且At3g12490 / CYSb.
权利和权限
开放获取本文根据知识共享署名4.0国际许可协议授权,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和源代码,提供知识共享许可协议的链接,并说明是否进行了修改。本文中的图像或其他第三方材料均包含在本文的知识共享许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的知识共享许可中,并且您的预期使用不被法定法规允许或超过允许的使用,您将需要直接从版权所有者获得许可。如欲查阅本牌照副本,请浏览http://creativecommons.org/licenses/by/4.0/.知识共享公共领域转让豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在对数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
刘伟,宋超,任正。et al。全基因组关联研究揭示了陆地棉纤维品质性状的遗传基础(陆地棉l .)。BMC植物杂志20.395(2020)。https://doi.org/10.1186/s12870-020-02611-0
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-020-02611-0
关键字
- 陆地棉
- 纤维质量
- 全基因组关联研究
- 单核苷酸多态性
- 数量性状位点