- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
利用RTM-GWAS方法,研究了陆地棉3个产量相关性状的QTL等位基因和候选基因gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba20.gydF4y2Ba,文章号:gydF4y2Ba416gydF4y2Ba(gydF4y2Ba2020gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
棉(gydF4y2BaGossypiumgydF4y2Ba纤维产量是植物的主要目标性状之一,提高纤维产量一直被认为是育种和生产的重要目标。虽然已有一些研究报道了棉花产量相关性状的遗传基础,但所检测到的数量性状位点(QTL)仍然非常有限。为探明陆地棉3个产量相关性状的全基因组QTL (gydF4y2Ba陆地棉gydF4y2BaL.),在4种种植环境下进行表型性状分析,在315份亲本的亲缘组合中筛选出9244个单核苷酸多态性连锁不平衡区(SNPLDB)标记。gydF4y2Ba
结果gydF4y2Ba
通过限制性两阶段多位点多等位基因全基因组关联研究(RTM-GWAS),在多种环境下分别检测到53、70和68个与铃数(BN)、铃重(BW)和皮分(LP)相关的SNPLDB显著位点。估计了重要SNPLDB位点的单倍型/等位基因效应,组织了qtl -等位基因矩阵,以提供该群体的简短遗传组成。在显著的SNPLDB位点中,有6个位点同时存在于两个或多个单一种植环境中,被认为是稳定的SNPLDB位点。此外,在6个SNPLDB稳定位点的附近区域共标注了115个基因,并通过2个RNA-seq数据预测了16个共同的潜在控制目标性状的候选基因。16种基因之一(gydF4y2BaGH_D06G2161gydF4y2Ba)主要在胚珠发育早期表达,并在其启动子区定位到稳定的SNPLDB位点(LDB_19_62926589)。gydF4y2Ba
结论gydF4y2Ba
本研究鉴定了陆地棉产量相关性状的QTL等位基因和候选基因,为棉花高产品种的选育提供了重要的遗传基础。gydF4y2Ba
背景gydF4y2Ba
作为主要的工业原料,棉花(gydF4y2BaGossypiumgydF4y2Ba(Spp .)纤维在日常生活和世界纺织工业中发挥着重要作用[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba].在种植棉花中,陆地棉(gydF4y2Ba陆地棉gydF4y2BaL.)是最大的栽培品种,占世界棉花产量的90%以上[gydF4y2Ba3.gydF4y2Ba].棉花纤维产量是棉花的关键目标性状之一,提高纤维产量一直被认为是育种和生产的重要目标[gydF4y2Ba4gydF4y2Ba].植物纤维产量的组成因子包括铃数(BN)、铃重(BW)和衣分(LP),这三个性状由数量性状位点(QTL)序列控制。近几十年来,通过连锁作图法检测了大量棉花产量相关性状的QTL,其QTL作图结果在棉花[gydF4y2Ba5gydF4y2Ba].在过去5年里,利用全基因组关联研究(GWAS)方法,在陆地棉中发现了许多与纤维产量组成性状相关的显著单核苷酸多态性(SNPs) [gydF4y2Ba1gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba].这些结果为棉花产量相关性状的遗传基础的解析奠定了良好的基础。然而,通过传统的GWAS方法检测到的主要QTL仍然有限。gydF4y2Ba
早期的GWAS方法在降低假阳性率方面存在很大困难,影响了相关位点的识别精度[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba].为了提高真实QTL的检测效率,关联分析中广泛应用了结构化关联分析(SA)、主成分分析(PCA)和混合线性模型(MLM)三种GWAS方法[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba].其中,MLM-GWAS手术一直是最受欢迎的手术,在临床上得到了广泛的应用gydF4y2Ba拟南芥gydF4y2Ba、水稻、玉米、高粱和棉花[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba].统计学家进一步得出结论,上述基于每次检测一个标记的全基因组扫描的GWAS方法(SA、PCA和MLM)被归类为单位点模型[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].然而,由于Bonferroni校正的选择标准非常严格,在单位点GWAS模型中,一些与客观性状相关的重要位点往往没有被检测到[gydF4y2Ba21gydF4y2Ba].此外,传统的基于单位点模型的GWAS程序主要关注植物中少数主要QTL的探索,难以解析全基因组QTL等位基因[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].然而,通过分子育种鉴定种质资源中qtl等位基因的组成是必要的。gydF4y2Ba
幸运的是,多位点GWAS (ML-GWAS)模型使探索全基因组QTL等位基因成为可能。最近,统计学家开发了一种新的限制性两阶段、多位点、多等位基因GWAS (RTM-GWAS)程序[gydF4y2Ba20.gydF4y2Ba,以揭示控制植物目标性状的全基因组QTL等位基因。采用RTM-GWAS方法检测大豆种子异黄酮含量和百粒重的相对全基因组qtl -等位基因系统[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba22gydF4y2Ba].然而,利用RTM-GWAS方法检测棉花育种目标性状全基因组QTL等位基因的研究较少。gydF4y2Ba
因此,为探索与棉花产量相关性状显著相关的全基因组QTL等位基因,采用RTM-GWAS方法对3个棉花产量相关性状在不同种植环境下进行关联分析。为了达到这一目的,我们使用了一个由315个陆地棉花资源组成的自然群体,并开发了13391个高质量单核苷酸多态性(SNPs),这些SNP被组织成9244个SNP连锁不平衡块(snpldb)。本研究鉴定了3个产量相关性状的显著snpldb和稳定QTL,并通过RTM-GWAS方法建立了表征群体多样性的QTL-等位基因矩阵;通过两条RNA-seq数据预测潜在候选基因。研究结果不仅为研究棉纤维产量性状的遗传结构提供了重要依据,而且为棉花高产品种的选育提供了重要依据。gydF4y2Ba
结果gydF4y2Ba
3个产量相关性状的表型变异gydF4y2Ba
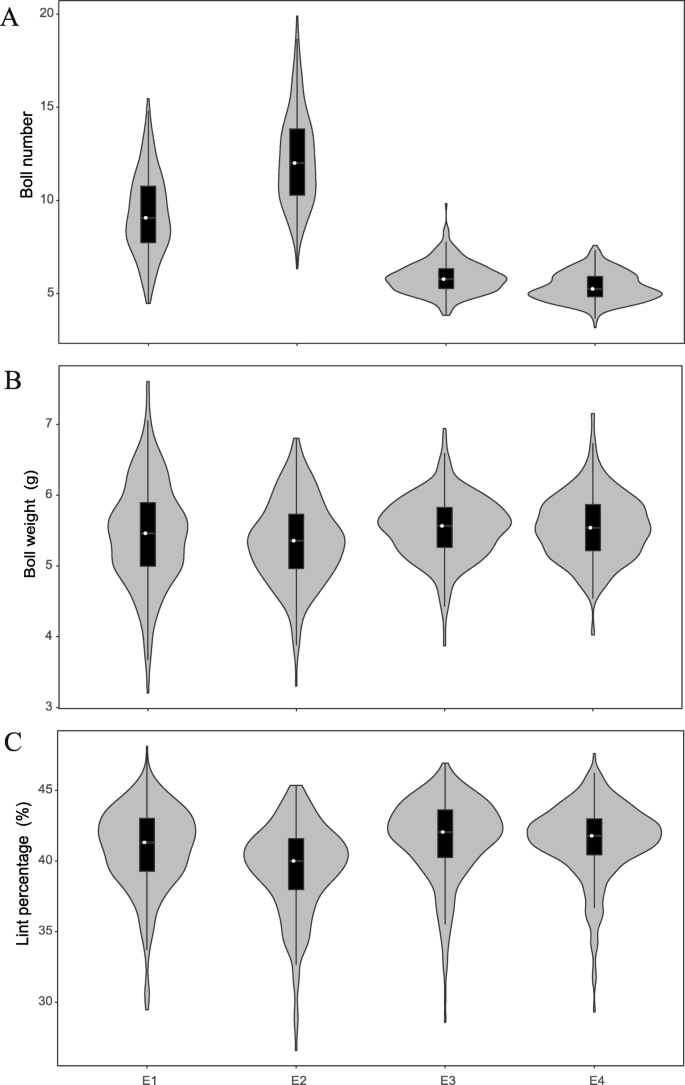
2014 - 2015年,在4种种植环境(E1-4)中,对自然居群的3个产量相关性状(BN、BW和LP)进行了评价。我们观察了3个产量相关性状在4种环境中平均值的变化范围。BN、BW和LP分别为4.45 ~ 13.19、3.60 ~ 7.13 g和28.49 ~ 47.01%,平均分别为8.18、5.47 g和40.89%(图2)。gydF4y2Ba1gydF4y2Ba;gydF4y2Ba表S1gydF4y2Ba)。我们观察到安阳(E1和E2)的BNs明显大于石河子(E3和E4)。此外,3个产量相关性状间也呈显著正相关,但各性状间相关性不显著(gydF4y2Ba表S2gydF4y2Ba)。连续而广泛的表型分布表明它们具有数量性状的分离特征,适合于GWAS。gydF4y2Ba
虽然这些性状表现出较大的表型变异,但LP的变异系数(CV)在4种环境中仅相对一致。E1-2组BN和BW的CV值大于e1 - 4组(gydF4y2Ba表S1gydF4y2Ba)。方差分析(ANOVA)表明,3个性状的遗传、环境和遗传-环境互作之间存在极显著差异(p < 0.05)。gydF4y2Ba表S3gydF4y2Ba)。BN、BW和LP的广义遗传力(BSH)分别为17.76、40.80和66.67% (gydF4y2Ba表S3gydF4y2Ba),表明LP和BW性状相对稳定遗传,而BN性状受种植环境因子影响较大。上述结果表明,种植环境对3个产量相关性状均有显著影响。gydF4y2Ba
snpldb标记的构建与种群结构gydF4y2Ba
利用slf -seq方法对315份陆地棉种质资源组成的关联组进行测序。测序结果与新的陆地棉TM-1参考基因组v2.1比对[gydF4y2Ba23gydF4y2Ba]和1,236,418个SNP标记。以召唤率> 0.90和小等位基因频率(MAF) > 0.05为对照,共保留了13391个高质量SNPs。这些snp可定位于陆地棉基因组全部26条染色体上,其中At亚基因组和Dt亚基因组分别有8491个和4900个snp,共组成9244个snpldb。以snpldb为基础,通过主成分分析和层次系统发育树将关联面板分为两组,并估计了群体中约500 kb的连锁不平衡(LD)衰减距离。关于协会小组的PCA和LD的详细结果将在另一项研究中报告(即将发表)。gydF4y2Ba
通过RTM-GWAS程序检测QTL等位基因gydF4y2Ba
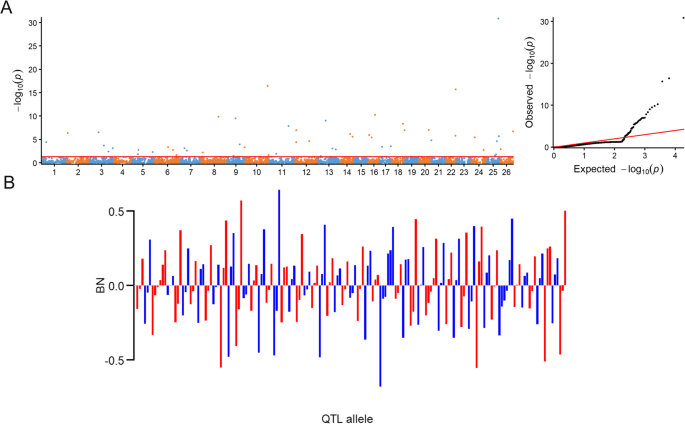
本研究采用基于多基因座模型的RTM-GWAS方法,对3个棉花产量相关性状进行全基因组QTL等位基因的检测。由于两个位点在BN性状上存在显著差异,本研究分别对多环境和单环境进行了RTM-GWAS程序。能够在多个环境和两个或多个单一环境中同时检测到的显著SNPLDB位点被认为是稳定的SNPLDB位点。利用多种环境下的RTM-GWAS程序,我们分别鉴定出53、70和68个与BN、BW和LP相关的SNPLDB显著位点(gydF4y2BaPgydF4y2Ba< 0.05;无花果。gydF4y2Ba2gydF4y2Ba;gydF4y2Ba表S4gydF4y2Ba)。gydF4y2Ba
在BN的多个环境的重要SNPLDB中,有一个稳定的高对数SNPLDB位点gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba尽管8个snpldb在单一种植环境中均有表现(图2),但在关联中未发现表型变异解释。gydF4y2Ba2gydF4y2Ba一个;表格gydF4y2Ba1gydF4y2Ba)。在53个与BN相关的显著位点中,有40个SNPLDB单位点和13个SNPLDB多位点。在逐步回归分析的基础上,我们在这些SNPLDB位点中鉴定出170个等位基因,其中阳性等位基因84个,阴性等位基因86个。正向等位基因效应的范围为0.0039 ~ 0.64,而负向等位基因效应的范围为−0.00034 ~−0.68。gydF4y2Ba2gydF4y2Bab).此外,SNPLDB显著位点的等位基因效应可以进一步组织成一个53 × 315(位点×接入)矩阵,该矩阵真实地反映了315个陆地棉品系BN性状的遗传变异构成(gydF4y2Ba图S1gydF4y2Ba一个)。gydF4y2Ba
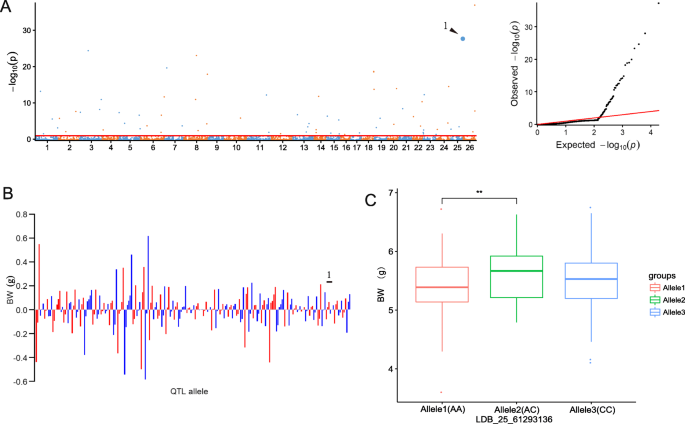
在BW多环境显著SNPLDB中,在3个单一种植环境中检测到1个稳定的SNPLDB位点(LDB_25_61293136),在单一种植环境中也出现了11个SNPLDB位点(图。gydF4y2Ba3.gydF4y2Ba一个;表格gydF4y2Ba1gydF4y2Ba)。位于第25染色体(D12)上的稳定SNPLDB位点LDB_25_61293136在三个单一种植环境(E1、E2和E4)中同时被鉴定出来,且−对数较大gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba)值(28.02)和表型变异解释(1.05%)。在70个与BW相关的SNPLDB位点中,15个位点包含多个SNP,其余55个位点包含单个SNP。115个阳性等位基因的等位基因效应值范围为0.0011 ~ 0.62 g,平均为0.47 g; 113个阴性等位基因的等位基因效应值范围为−0.000070 ~−0.58 g,平均为−0.10 g。gydF4y2Ba3.gydF4y2Bab).稳定的SNPLDB位点LDB_25_61293136存在AA、AC和CC三种等位基因类型,效应值分别为−0.12、0.045和0.073(图。gydF4y2Ba3.gydF4y2Bab);阴性等位基因(AA)的BW值显著低于杂合子等位基因(AC),而不显著低于阳性等位基因(CC)的BW值(图5)。gydF4y2Ba3.gydF4y2Bac).此外,对于70个具有147个单倍型/等位基因的SNPLDB显著位点,等位基因效应被组织成一个70 × 315(位点×接入)矩阵,包含关联面板中BW性状改善的核心遗传信息(gydF4y2Ba图S1gydF4y2BaB)。gydF4y2Ba
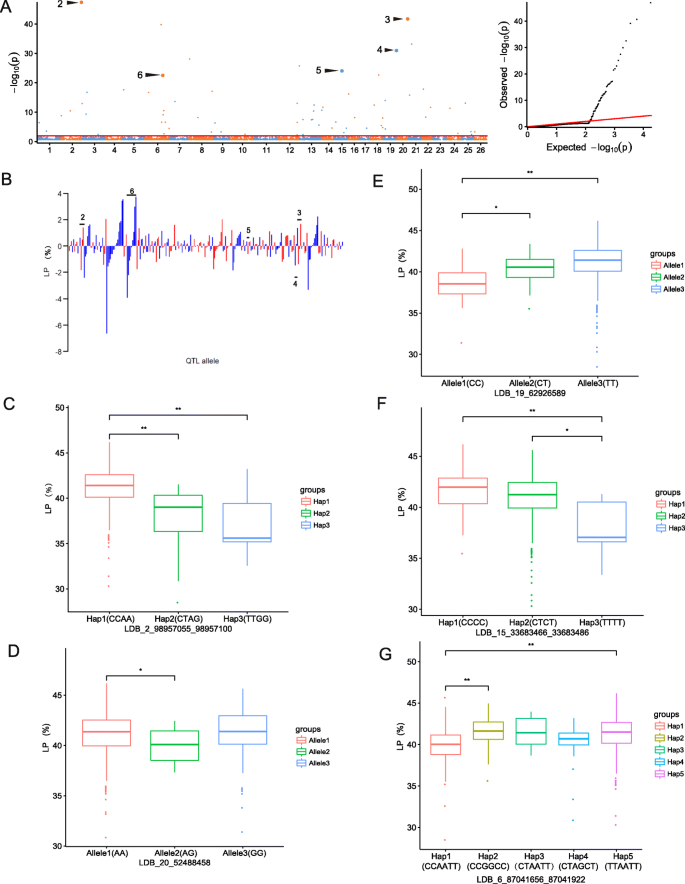
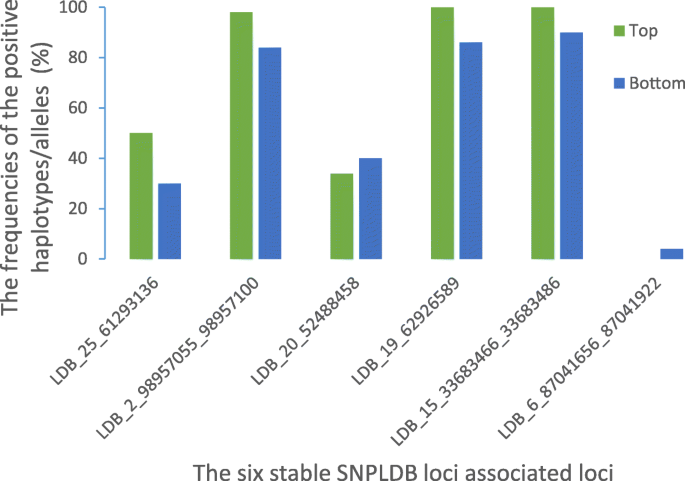
在多个环境中与LP相关的显著SNPLDB中,在两个单一种植环境中检测到5个稳定的SNPLDB位点,在单个种植环境中也观察到7个SNPLDB位点(图2)。gydF4y2Ba4gydF4y2Ba一个;表格gydF4y2Ba1gydF4y2Ba)。5个稳定的SNPLDB位点中,峰值位点LDB_2_98957055_98957100位于2号染色体(A02)上,−log值最高gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba)值(46.97)和表型变异解释(3.98%),在两种单一种植环境(E3和E4)中同时鉴定;第二高的位点LDB_20_52488458分布在第20染色体(D07),具有−对数gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba)值为40.68,表型变异最大,为5.09%。在与LP相关的68个位点中,23个为多snp位点,其余为单snp位点。124个阳性等位基因的等位基因效应值范围为0.0034 ~ 3.73%,平均为0.67%;131个阴性等位基因的等位基因效应值范围为- 0.012 ~ - 6.65%,平均为- 0.64%。gydF4y2Ba4gydF4y2Bab).如SNPLDB峰值位点LDB_2_98957055_98957100存在Hap1、Hap2和Hap3三个单倍型,效应值分别为0.47、−1.86和−1.39%(图2)。gydF4y2Ba4gydF4y2Bab);与携带Hap2(CTAG)和Hap3(TTGG)的材料相比,携带Hap1(CCAA)的材料LP显著增加(图2)。gydF4y2Ba4gydF4y2Bac).在另一个关联位点LDB_20_52488458中,存在AA、AG和GG 3个等位基因,效应分别为- 0.26、- 1.40和1.66%(图。gydF4y2Ba4gydF4y2Bab);而含有AA和GG等位基因的株系之间没有观察到显著的表型差异(图2)。gydF4y2Ba4gydF4y2Bad).另一个显著信号LDB_6_87041656_87041922具有Hap1(CCAATT)、Hap2 (CCGGCC)、Hap3(CTAATT)、Hap4(CTAGCT)和Hap5(TTAATT) 5个单倍型,且含有Hap2和Hap5的LP值显著高于含有Hap1的LP值(图1)。gydF4y2Ba4gydF4y2Bag)。对于另外两个稳定的SNPLDB位点(LDB_19_62926589和LDB_15_33683466_33683486),具有有利单倍型/等位基因的LP值显著高于具有不利单倍型/等位基因的LP值(图2)。gydF4y2Ba4gydF4y2Bae, f).此外,包含156个单倍型/等位基因的68个LP SNPLDB显著位点的效应可建立为一个68 × 315(位点×接入)的LP性状遗传变异信息矩阵(gydF4y2Ba图S1gydF4y2BaC)。gydF4y2Ba
LP的RTM-GWAS结果及单倍型分析。gydF4y2Ba一个gydF4y2Ba曼哈顿区(左)和LP的分位数-分位数图(右)。gydF4y2BabgydF4y2BaLP显著SNPLDB位点的等位基因效应分布。数字2-6分别表示5个有效SNPLDB位点LDB_2_98957055_98957100、LDB_20_52488458、LDB_19_62926589、LDB_15_33683466_33683486和LDB_6_87041656_87041922。gydF4y2BacgydF4y2Ba-gydF4y2BaggydF4y2Ba5个稳定SNPLDB位点在不同单倍型/等位基因间的LP差异。gydF4y2BacgydF4y2BaLDB_2_98,957,055_98957100, Hap1(CCAA), Hap2(CTAG), Hap3(TTGG);gydF4y2BadgydF4y2BaLDB_20_52488458、Allele1-3分别代表AA、AG和GG;gydF4y2BaegydF4y2BaLDB_19_62926589、Allele1-3分别代表CC、CT和TT。gydF4y2BafgydF4y2BaLDB_15_33683466_33683486, Hap1(CCCC), Hap2 (CTCT), Hap3(TTTT);gydF4y2BaggydF4y2BaLDB_6_87041656_87041922, Hap1(CCAATT), Hap2 (CCGGCC), Hap3(CTAATT), Hap4(CTAGCT)和Hap5(TTAATT)gydF4y2Ba
从3个目标性状的qtl -等位基因矩阵中,我们发现优势种质阳性单倍型/等位基因频率高于劣势种质阳性单倍型/等位基因频率的规律性。在产量最高和最低的品种之间,可以直观地观察到这种现象。以LP性状为例,50个最高株系(1943个,平均38.86个/系)阳性单倍型/等位基因的累积数量大于50个最低株系(1818个,平均36.36个/系)。相应的,50个高LP的累计负单倍型/等位基因数(1796个,平均35.92个/份)低于50个低LP的累计负单倍型/等位基因数(1998年,平均39.96个/份)。尽管315份材料之间存在表型差异,但任何一份材料都同时含有阳性和阴性等位基因。以优质棉花品种“鲁绵研28”为例,其BN、BW和LP的阳性等位基因数分别为34、44和44;阴性等位基因数分别为32、42和42。此外,我们还关注了SNPLDB的重要位点。对于稳定的BW SNPLDB位点LDB_25_61293136, BW 50株顶部的阳性等位基因频率明显高于底部50株。在LP的5个稳定SNPLDB位点中,有3个(ldb_98957055_98957100、LDB_19_62926589、LDB_15_33683466_33683486)符合LP前50个株系阳性单倍型/等位基因频率明显高于后50个株系的规律(图2)。gydF4y2Ba5gydF4y2Ba)。gydF4y2Ba
通过多环境RTM-GWAS程序分别鉴定出53、70和68个与BN、BW和LP相关的显著SNPLDB位点,并将这些显著SNPLDB位点的效应组织到三个目标性状的qtl -等位基因矩阵中;(2)分别检测到1个和5个与BW和LP稳定相关的SNPLDB位点;(3)效应极高或极低的QTL等位基因不一定来自-log最大的SNPLDB峰值位点gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba)值和表型变异解释;(4)对于显著相关的位点,优势位点的阳性等位基因频率普遍高于劣势位点,但并非所有位点都完全符合相关规律。研究结果为研究陆地棉产量相关性状的遗传基础提供了重要依据。gydF4y2Ba
潜在候选基因的鉴定和预测gydF4y2Ba
基于上述结果,通过RTM-GWAS程序鉴定了6个与BW和LP相关的稳定SNPLDB位点,它们应该是控制棉花产量相关性状的主要效应QTL。因此,我们主要研究了6个稳定的SNPLDB位点。参考以往大部分棉花研究中关联组snpldb的LD衰减距离和SNP-linked片段(200 kb) [gydF4y2Ba4gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba],推荐6个稳定SNPLDB位点周围±200 kb的基因组片段作为候选基因的靶点范围。根据陆地棉TM-1参考基因组v2.1 [gydF4y2Ba23gydF4y2Ba],在6个目标基因组片段中共注释了115个基因(gydF4y2Ba表S5gydF4y2Ba)。通过比对基因的基因组位置,我们发现稳定的SNPLDB位点(LDB_15_33683466_33683486)位于该基因的编码序列(CDS)内(gydF4y2BaGH_D02G1202gydF4y2Ba)。同时,我们发现SNPLDB位点LDB_19_62926589位于候选基因的启动子区gydF4y2BaGH_D06G2161gydF4y2Ba.gydF4y2Ba
根据南京农业大学(NAU)基因的归一化片段每千碱基的转录每百万的映射reads (FPKM)值,RNA-seq [gydF4y2Ba27gydF4y2Ba], 115个基因中有93个在20个陆地棉组织中至少一个组织中高表达,并被分为5个不同的组(组1-5)。5组中,第1组9个基因主要表达于花后5和10 d的纤维中,第2组22个基因主要表达于茎和花器官中,第3组3个基因主要表达于花后20和25 d的纤维中,第4组21个基因主要表达于胚珠发育的5个阶段,第5组38个基因主要表达于花器官中(图5)。gydF4y2Ba6gydF4y2Baa).另外,根据中国农业科学院棉花研究所(CRI) RNA-seq基因的其他FPKM值,115个基因中有100个在16个陆地棉组织中至少一个组织中高表达,并可分为5个不同的类群(I-V类群)。在这五组中,有10个基因被分配到I组,它们在纤维组织中高度表达;II组53个基因在茎、叶和花器官中高表达,III组5个基因在20或25 DPA的胚珠中高表达,IV组14个基因在根中高表达,V组18个基因主要在胚珠发育的5个阶段表达(图2)。gydF4y2Ba6gydF4y2Bab).通过NAU和CRI RNA-seq分别鉴定了主要表达于纤维和胚珠的两组基因(各33组)(gydF4y2Ba表S6gydF4y2Ba);其中16个基因在两个RNA-seq数据中是相同的,应该是棉花产量相关性状的潜在候选基因(图2)。gydF4y2Ba6gydF4y2Bac;表格gydF4y2Ba2gydF4y2Ba)。另外,将16个基因分别分配到- 1、3、4组和- I、III、V组(图2)。gydF4y2Ba6gydF4y2Bac).因此,我们推测分配给- 1、3、4族和- I、III、V族的基因可能与目标性状密切相关。gydF4y2Ba
鉴定与BW或LP相关的潜在候选基因。gydF4y2Ba一个gydF4y2Ba利用南京农业大学(NAU)的RNA-seq数据对20个陆地棉(TM-1)组织中93个候选基因的表达谱进行热图分析。红色表示高表达,绿色表示低表达。gydF4y2BabgydF4y2Ba利用中国农业科学院棉花研究所(CRI) RNA-Seq数据绘制了16个陆地棉(TM-1)组织中100个推测候选基因的表达谱热图,图中红色部分为16个基因gydF4y2Ba一个gydF4y2Ba而且gydF4y2BabgydF4y2Ba.gydF4y2BacgydF4y2Ba两个RNA-seq数据(NAU和CRI)之间的共同基因的文尼图。gydF4y2BadgydF4y2Ba基因结构gydF4y2BaGH_D06G2161gydF4y2BaSNPLDB位点LDB_19_62926589在基因启动子区的位置。E.转录组水平gydF4y2BaGH_D06G2161gydF4y2Ba在20个不同的组织中,基于每千碱基的转录本每百万映射reads (FPKM)的片段值gydF4y2Ba
16个潜在候选基因中,有14个具有生物学功能注释。7个基因编码与膜、锌指、抗病等相关的结合蛋白;6个基因编码酶,如氧化酶、硫酯酶、二酰基甘油胆碱磷酸酶、E3泛素-蛋白连接酶、天冬酰胺- trna连接酶等;剩下的基因之一编码MYB转录因子(表gydF4y2Ba2gydF4y2Ba)。利用基因本体论(GO)和京都基因与基因组百科(KEGG)路径项对16个基因进行功能预测。GO结果显示,四种基因(gydF4y2BaGH_D12G2952gydF4y2Ba,gydF4y2BaGH_D12G2950gydF4y2Ba,gydF4y2BaGH_A02G1555gydF4y2Ba而且gydF4y2BaGH_D12G2926gydF4y2Ba)与基因产物与DNA选择性、非共价相互作用的分子功能有关(GO:0003677);和两个基因(gydF4y2BaGH_D06G2164gydF4y2Ba而且gydF4y2BaGH_D06G2161gydF4y2Ba)参与与任何蛋白质或蛋白质复合物选择性地、非共价相互作用的过程(GO:0005515) (gydF4y2Ba表S7gydF4y2Ba)。通过KEGG通路分析,在2个基因(gydF4y2BaGH_D06G2180gydF4y2Ba而且gydF4y2BaGH_D12G2926gydF4y2Ba)和它们共同的代谢通路(ko01100) (gydF4y2Ba表S8gydF4y2Ba)。更重要的是基因gydF4y2BaGH_D06G2161gydF4y2Ba属于ii类氨基酰基- trna合成酶家族,这是一种普遍存在且进化保守的酶,主要在胚珠发育早期表达,它催化氨基酸的高度特异性酰化以同源trna,在其启动子区检测到SNPLDB位点LDB_19_62926589。gydF4y2Ba6gydF4y2Bad, e).基因同源性分析表明gydF4y2BaGH_D06G2161gydF4y2Ba的同源词gydF4y2BaasngydF4y2Ba参与天冬酰胺- trna连接酶活性的基因gydF4y2Ba支原体arthritidisgydF4y2Ba[gydF4y2Ba28gydF4y2Ba].的gydF4y2BaasngydF4y2Ba基因(gydF4y2BaAT5G32440gydF4y2Ba)是泛素系统成分提示蛋白mRNA中的一种gydF4y2Ba拟南芥gydF4y2Ba[gydF4y2Ba29gydF4y2Ba].然而,这些基因的功能还不清楚。gydF4y2Ba
讨论gydF4y2Ba
表型性状差异gydF4y2Ba
目前,世界前五大棉花生产国是印度、美国、中国、巴基斯坦和巴西;2019年,这些国家的棉花产量占全球的70%以上。中国在棉花收获面积上排名第四,而它的棉花产量在世界上排名第二。即便如此,中国仍是棉纤维进口和国内消费量最大的国家(USDA-FAS 2019) [gydF4y2Ba30.gydF4y2Ba].此外,中国棉花收获面积至少已经下降了5年,因此提高纤维产量仍然是中国棉花育种实践的主要目标。在4种种植环境下,对棉花产量相关性状(BN、BW和LP)进行了评价。我们发现,这三个与产量相关的性状的表型测量值表现出高度的多样性。本研究的表型研究为今后棉花产量育种提供优良种质资源具有重要意义。此外,3个与棉花产量相关的性状,特别是BN性状受种植环境的影响显著,E3-4环境的表型测量值极显著低于E1-2环境。这是因为除了光、温度和水等气候因素外,植物密度可能是影响每株植物氮含量的最重要因素之一。在田间试验中,E1-2环境的植株密度为每0.18 m 1株gydF4y2Ba2gydF4y2Ba)明显低于E3-4环境(每0.043 m 1株)gydF4y2Ba2gydF4y2Ba)。较高的植株密度通常会增加株内对光、水和养分等资源的竞争,并降低单株棉纤维产量[gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba].因此,对于单个棉花植株而言,其纤维产量可能会受到植株密度的很大影响。在本研究中,氮化硼调查值受两个试验点植物密度差异的影响。gydF4y2Ba
GWAS结果的比较gydF4y2Ba
棉花纤维产量是棉花产量的重要性状之一,受数量性状基因的控制。数量性状往往受环境的影响很大。因此,大多数QTL只能在单个环境中检测到,而极少数稳定QTL可以在多个种植环境中同时检测到。以往的研究认为在多个环境中同时检测到的QTL为稳定QTL [gydF4y2Ba33gydF4y2Ba].在本研究中,通过RTM-GWAS程序可以同时检测到多个环境和两个或两个以上单一环境的显著snpldb被认为是客观性状的稳定QTL。通过多环境和单环境综合分析,通过RTM-GWAS程序检测到6个与BW或LP相关的稳定SNPLDB位点。gydF4y2Ba
此前,通过GWAS方法在陆地棉中检测到一些与产量相关性状相关的SNP位点[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba], GWAS结果列于gydF4y2Ba表S9gydF4y2Ba.根据最近的研究,一些稳定BN-QTL (d02:55.00 - 58.20 Mb, a02:79.10 - 79.45 Mb,和d08:2.80 - 3.10 Mb), BW-QTL (d09:47.70 - 47.75 Mb和a02:74.94 - 75.56 Mb),和LP-QTL (a02:74.71 - 75.56 Mb, a02:79.10 - 79.45 Mb, a06:102.50 - 103.60 Mb, a08:52.09 - 52.10 Mb, d02:1.30 - 1.60 Mb, D02: 2.20 - -2.36 Mb, d03:30.00 - 40.00 Mb, d08:2.80 - 3.10 Mb,和d12:55.00 - 58.20 Mb)被发现在过去的5年(gydF4y2Ba1gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba].在以往的研究中,利用基于单位点模型的MLM-GWAS研究了3个产量相关性状的QTL,其中大部分主要定位在染色体A02、D02和D08上。本研究通过RTM-GWAS程序鉴定了1个稳定的BW-SNPLDB位点LDB_25_61293136 (D12)和5个稳定的LP-SNPLDB位点ldb_98957055_98957100 (A02)、LDB_20_52488458 (D07)、LDB_19_62926589 (D06)、LDB_15_33683466_33683486 (D02)和LDB_6_87041656_87041922 (A06)。为了验证与目标性状相关的重要SNPLDB位点的真实性和新颖性,将本研究中确定的稳定QTL与之前的GWAS结果进行了比较。结果表明,除ldb_98,957,055 _98957100位点外,其余5个稳定的SNPLDB位点可能是BW或LP性状的新QTL。此外,利用两组RNA-seq数据,在稳定snpldb的邻近基因组片段中预测了16个在胚珠或纤维中特异高表达的潜在候选基因。我们推测这些基因可能有助于调控棉花胚珠或纤维的生长发育,并与皮棉产量密切相关。gydF4y2Ba
RTM-GWAS手术的优越性gydF4y2Ba
在植物分子育种过程中,利用GWAS检测育种资源中的全基因组QTL非常重要,但以往的单位点GWAS程序主要集中在鉴定一个主要QTL等位基因点[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].本研究的目的不仅是在棉花种质群体中检测几个主要QTL,而且通过改进的GWAS程序鉴定完整的QTL-等位基因系统。因此,为了揭示棉花3个产量相关性状的全基因组QTL,本研究应采用检测效率高、功率大的GWAS方法。近年来的研究证明,RTM-GWAS程序在大豆中具有比MLM等其他GWAS模型更高的QTL检测效率和强度[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba34gydF4y2Ba].由于RTM-GWAS程序在整个QTL等位基因中具有明显的优势,本研究将其用于产量相关QTL等位基因的检测。与以往的单位点GWAS方法相比,RTM-GWAS方法检测到了更多显著的棉花产量相关性状QTL。在本研究中,通过多环境RTM-GWAS程序共鉴定出68个与LP相关的显著SNPLDB位点,而在我们之前的报道中,通过MLM-GWAS方法仅检测到12个与LP相关的显著SNP位点[gydF4y2Ba6gydF4y2Ba].同样,通过RTM-GWAS程序检测到的稳定LP-QTL(5)比通过MLM-GWAS检测到的LP-QTL(2)要多。对比结果表明,RTM-GWAS方法在解剖更多客观性状的QTL方面比MLM-GWAS更有优势。gydF4y2Ba
此外,RTM-GWAS程序还可以为qtl -等位基因矩阵提供比较全面的遗传信息,包括SNPLDB显著位点及其等位基因效应、群体等位基因组成以及每个位点的频率分布。本研究利用SNPLDB显著位点的正、负等位基因建立了3个产量相关性状(BN、BW和LP)的qtl等位基因矩阵。在qtl等位基因矩阵中,优势种质的阳性单倍型/等位基因比劣势种质的阳性单倍型/等位基因多。这一现象可以直接在表型值上端和下端之间观察到。但是,传统的GAWS模型不能直接组织qtl -等位基因矩阵。gydF4y2Ba
结论gydF4y2Ba
通过多环境RTM-GWAS程序,在315份陆地棉材料中分别检测到53个、70个和68个与BN、BW和LP相关的SNPLDB显著位点。计算显著SNPLDB位点的单倍型/等位基因效应,并组织qtl -等位基因矩阵,以提供自然群体的简短遗传组成。在多个环境的显著SNPLDB位点中,有6个位点同时在两个或多个单一种植环境中被鉴定出来,被认为是稳定的SNPLDB位点。因此,我们对6个稳定的SNPLDB位点进行了重点定位,在其附近区域共标注了115个基因,并通过2个RNA-seq数据预测了16个控制其目标性状的潜在候选基因。16种基因之一(gydF4y2BaGH_D06G2161gydF4y2Ba)主要在胚珠发育早期表达,并在其启动子区定位了SNPLDB基因座LDB_19_62926589。本研究鉴定了陆地棉产量相关性状的QTL等位基因和候选基因,为研究陆地棉产量相关性状的遗传基础提供了重要依据。gydF4y2Ba
方法gydF4y2Ba
植物材料和生长条件gydF4y2Ba
协会小组包括从中国收集的290个品系,从美国引进的21个品系和从前苏联引进的4个品系。中国棉花品种的种子来自我们的种质资源库,其余种质系的种子来自中国农业科学院棉花研究所(gydF4y2Ba表S10gydF4y2Ba)。所有陆地棉材料分别在E1和E2[2014年和2015年分别在河南省安阳(36.13°N, 114.80°E)]和E3和E4[2014年和2015年分别在新疆生产建设集团石河子(44.52°N, 86.02°E)] 4个自然环境中种植。所有的供试材料都是按照随机的完全块设计实验生长的,在每个环境中重复3次。这些品种在单行地块(长400 cm,宽80 cm,株距20 cm)中种植,在E1和E2中种植约20株,而所有材料在双行地块(长200 cm,宽76 cm,株距10 cm)中种植,在E3和E4中种植约40株。田间管理遵循常规耕作方法。gydF4y2Ba
表型性状测量gydF4y2Ba
在各环境下测定了3个与产量相关的性状(BN、BW和LP)。在每个试验年的9月初,对每一行中间的10株连续植株进行BN性状调查。在每个试验年的10月,从每个品种的植株中心部分人工采收20个常开的铃,并称重计算BW性状。然后用轧棉机仔细彻底剥去纤维样品,用电子天平计算纤维质量(FW, g)。然后按LP (%) = FW (g)/BW (g) × 100%计算各株系的LP值。对于3个与产量相关的性状,采用广义线性模型(GLM)计算其广义遗传力(BSH),并用IBM SPSS 22.0软件对表型数据进行方差分析。gydF4y2Ba
SNP调用和SNPLDB组装gydF4y2Ba
采用校正的十六烷基三甲基溴化铵(CTAB)方法从幼叶中提取所有系的基因组DNA [gydF4y2Ba35gydF4y2Ba],测序reads在Illumina HiSeq 2500系统(Illumina, Inc, San Diego, CA, USA)中通过SLAF-seq方法获得[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba].然后,我们使用BWA mem软件对一个新的陆地棉TM-1参考基因组v2.1的315个接入的高质量reads进行比对[gydF4y2Ba23gydF4y2Ba],所有变体调用管道都基于GATK最佳实践,并进行了一些修改。首先,我们使用BWA mem软件使用三个默认参数(−M -a -t8)来对齐读取到引用基因组,第二picard用于标记重复,第三我们使用GATK haplotyer - gvcftype管道调用变体,并通过broad机构提供的硬参数进行过滤(QD < 2.0 || FS > 60.0 || MQ < 40.0 || MQRankSum <−12.5|| ReadPosRankSum <−8.0 || SQR > 3.0) [gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba].筛选标准为最大缺失> 0.10,最小等位基因频率(MAF) < 0.05。gydF4y2Ba
使用Haploview软件计算单倍型块和多标记类型的后验概率,以确定最终的SNPLDB数据集[gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba].Haploview中使用置信区间法定义默认设置的区块,基因组距离超过200 kb的SNP区块除外[gydF4y2Ba42gydF4y2Ba].与单SNP等位基因一样,同一LD块内的多个SNP制造者被组织成具有多个单倍型的SNPLDB。gydF4y2Ba
RTM-GWAS程序gydF4y2Ba
基于全基因组SNPLDB标记估算的GSC矩阵,利用Mega7.0软件构建了315个陆地棉品种的邻接树。然后,利用全基因组snpldb估计的GSC矩阵的前10个特征值对群体结构偏差进行校正。在研究中,我们使用了基于弹性遗传相似系数(GSC)矩阵(弹性GSC矩阵和GSC特征向量示于)的RTM-GWAS过程与群体结构复现相结合gydF4y2Ba表S11gydF4y2Ba),以及单基因座模型下snpldb的初步筛选和多基因座模型下的逐步回归分析两阶段关联分析。RTM-GWAS程序由报告的参考文献进行[gydF4y2Ba20.gydF4y2Ba].以9244个多等位基因snpldb为基础,利用4种种植环境的表型值,分别对3个与产量相关的性状(BN、BW和LP)进行了多环境和4个单一环境的RTM-GWAS处理。为了降低Bonferroni校正的严格选择标准,并检测全基因组QTL等位基因,植物的snpldbgydF4y2BaPgydF4y2Ba值< 0.05(正常显著性水平)被认为是RTM-GWAS程序两阶段关联分析中的显著位点,参考以往大豆的研究[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].gydF4y2Ba
对于与目标性状相关的SNPLDB显著位点,通过RTM-GWAS程序的第二阶段关联分析计算每个位点的等位基因效应值[gydF4y2Ba20.gydF4y2Ba].然后根据等位基因效应值,对所有SNPLDB显著位点进行阳性和阴性等位基因的测定。最后,通过RTM-GWAS程序的交叉软件(如前所述),利用所有重要snpldb的等位基因效应值的计算数据,通过R Version 3.6.0创建了客观性状的qtl等位基因矩阵[gydF4y2Ba20.gydF4y2Ba].gydF4y2Ba
潜在候选基因表达分析gydF4y2Ba
少数能在两种或两种以上种植环境中同时检测到的SNPLDB位点被认为是与客观性状相关的稳定位点。对于稳定和顶部SNPLDB位点,每个单倍型/等位基因的平均表型值由每个SNPLDB类型的棉花品系的表型值计算。利用IBM SPSS 22.0软件对各单倍型/等位基因的表型值进行双尾t检验。利用稳定性状相关SNPLDB位点的染色体位置,在陆地棉TM-1参考基因组v2.1中寻找潜在的候选基因[gydF4y2Ba23gydF4y2Ba].根据以往棉花研究中检测到的snp连锁候选基因区域[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba],筛选稳定SNPLDB位点的候选基因的基因组片段在本研究标记周围±200 kb。gydF4y2Ba
NAU从20个棉(TM-1)组织(包括根、茎、叶、环面、花瓣、雄蕊、雌蕊、花萼、Ovule_0 DPA、Ovule_1 DPA、Ovule_3 DPA、Ovule_5 DPA、Ovule_10 DPA、Ovule_20 DPA、Ovule_30 DPA、fiber-5 DPA、fiber-10 DPA、fiber-20 DPA和fiber-25 DPA)中获得了一组RNA-seq数据,并可在NCBI SRA数据库(SRA: PRJNA248163)上查询[gydF4y2Ba27gydF4y2Ba],并利用FPKM值反映基因表达水平。另一组RNA-seq数据的FPKM值来自16个棉花(TM-1)组织(包括根、茎、叶、花瓣、环部、萼片、苞片、花药、Ovule_10 DPA、Ovule_15 DPA、Ovule_20 DPA、Ovule_25 DPA、fiber_5 DPA、fiber_10 DPA、fiber_20 DPA和fiber_25 DPA),通过一个网站(gydF4y2Bahttp://grand.cricaas.com.cn/page/tools/expressionVisualizationgydF4y2Ba)。假定候选基因表达方式的热图和表型值的箱形图由软件中心绘制(gydF4y2Bahttps://www.omicshare.com/tools/home/index/index.htmlgydF4y2Ba)。对所有潜在候选基因进行GO富集分析和KEGG分析,利用棉花生物信息网站(gydF4y2Bahttps://cottonfgd.org/gydF4y2Ba),高级参数为显著性水平0.01,每个分析项的最小基因数为3。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
从SLAF-seq分析中获得的陆地棉序列序列数据可在序列读取档案(gydF4y2Bahttp://www.ncbi.nlm.nih.gov/bioproject/PRJNA314284/gydF4y2Ba)。gydF4y2Ba
缩写gydF4y2Ba
- 方差分析:gydF4y2Ba
-
方差分析gydF4y2Ba
- BN:gydF4y2Ba
-
棉子数量gydF4y2Ba
- BSH:gydF4y2Ba
-
广义遗传gydF4y2Ba
- BW:gydF4y2Ba
-
铃重gydF4y2Ba
- cd:gydF4y2Ba
-
编码序列gydF4y2Ba
- 分区:gydF4y2Ba
-
花后天数gydF4y2Ba
- FPKM:gydF4y2Ba
-
每千碱基的片段每百万片段映射读取gydF4y2Ba
- 弗兰克-威廉姆斯:gydF4y2Ba
-
纤维的重量gydF4y2Ba
- 全球语言监测机构:gydF4y2Ba
-
广义线性模型gydF4y2Ba
- 走:gydF4y2Ba
-
基因本体论gydF4y2Ba
- GSC:gydF4y2Ba
-
遗传相似系数gydF4y2Ba
- GWAS:gydF4y2Ba
-
全基因组关联研究gydF4y2Ba
- KEGG:gydF4y2Ba
-
京都基因和基因组百科全书gydF4y2Ba
- LD:gydF4y2Ba
-
连锁不平衡gydF4y2Ba
- LP:gydF4y2Ba
-
线头百分比gydF4y2Ba
- LW:gydF4y2Ba
-
皮棉重gydF4y2Ba
- 加:gydF4y2Ba
-
小等位基因频率gydF4y2Ba
- ML-GWAS:gydF4y2Ba
-
多位点GWASgydF4y2Ba
- 传销:gydF4y2Ba
-
混合线性模型gydF4y2Ba
- 山:gydF4y2Ba
-
关联分析gydF4y2Ba
- QTL:gydF4y2Ba
-
数量性状位点gydF4y2Ba
- SLAF-seq:gydF4y2Ba
-
特异性位点扩增片段测序gydF4y2Ba
- SNP:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
- SNPLDBs:gydF4y2Ba
-
SNP连锁不平衡阻滞gydF4y2Ba
- SL-GWAS:gydF4y2Ba
-
Single-locus GWASgydF4y2Ba
- 主成分分析:gydF4y2Ba
-
主成分分析gydF4y2Ba
- 电脑:gydF4y2Ba
-
主成分gydF4y2Ba
- RTM-GWAS:gydF4y2Ba
-
限制性两阶段多位点多等位基因GWASgydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
方林,王强,胡勇,贾勇,陈杰,刘波,张震,关霞,陈松,周波,等。棉花基因组分析鉴定了与纤维品质和产量性状相关的选择特征和位点。Nat Genet, 2017;49:1089。gydF4y2Ba
- 2.gydF4y2Ba
Naoumkina M, Thyssen G, Fang D, Jenkins J, Mccarty FC。棉花纤维长度性状的遗传和转录组分析(gydF4y2Ba陆地棉gydF4y2Bal)神奇人口。BMC基因组学,2019;20:112。gydF4y2Ba
- 3.gydF4y2Ba
陈铮,Scheffler B, Dennis E, Triplett B,张涛,郭伟。gydF4y2BaGossypiumgydF4y2Ba)的基因组。植物物理学报,2007;gydF4y2Ba
- 4.gydF4y2Ba
孙忠,王旭,刘忠,顾强,张勇,李忠,柯辉,杨洁,吴娟,吴林,等。一项全基因组关联研究发现了陆地棉产量相关性状的新基因组区域和候选基因。理论应用,2018;131:2413-25。gydF4y2Ba
- 5.gydF4y2Ba
赛娟,宋敏,王辉,林志,张旭,方东,张娟。种内QTL的比较元分析gydF4y2Ba陆地棉gydF4y2Ba和种间gydF4y2Bag .分子gydF4y2Ba×gydF4y2Bag .取得gydF4y2Ba人群。Mol Genet Genomics 2015; 290:1003-25。gydF4y2Ba
- 6.gydF4y2Ba
苏娟,范松,李林,魏华,王超,王辉,宋明,张超,顾玲,赵松,等。GWAS法检测中国陆地棉衣分有利QTL等位基因和候选基因。前沿植物科学,2016;7:1576。gydF4y2Ba
- 7.gydF4y2Ba
马震,何松,王旭,孙杰,张勇,张刚,吴林,李震,刘震,孙刚,等。对陆地棉核心集落进行重测序,确定了影响纤维质量和产量的基因组变异和位点。Nat Genet, 2018; 50:803-13。gydF4y2Ba
- 8.gydF4y2Ba
马旭,王喆,李伟,张勇,周旭,刘勇,任忠,裴旭,周凯,张伟,等。对一个家系的核心材料进行重测序,可以确定棉花改良过程中基因组片段的来源和关键农艺性状位点。中国生物工程学报,2019;17(4):366 - 366。gydF4y2Ba
- 9.gydF4y2Ba
沈超,王楠,黄超,王敏,张旭,林哲。群体基因组学揭示了棉花遗传改良的精细规模重组格局。植物J. 2019; 99:494-505。gydF4y2Ba
- 10.gydF4y2Ba
兰德尔E, Schork N.复杂性状的遗传解剖。科学。1994;265:2037-48。gydF4y2Ba
- 11.gydF4y2Ba
Campbell C, Ogburn E, Lunetta K, Lyon H, Freedman M, Groop L, Altshuler D, Ardlie K, Hirschhorn j。Nat Genet, 2005; 37:868-72。gydF4y2Ba
- 12.gydF4y2Ba
李志强,李志强,李志强。结构化群体关联映射的研究进展。胡姆·热内。2000;67:170-81。gydF4y2Ba
- 13.gydF4y2Ba
Price A, Patterson N, Plenge R, Weinblatt M, Shadick N, Reich D.主成分分析校正全基因组关联研究中的分层。Nat Genet, 2006; 38:904-9。gydF4y2Ba
- 14.gydF4y2Ba
张震,Ersoz E, Lai C, Todhunter R, Tiwari H, Gore M, Bradbury P, Yu J, Arnett D, as J,等。线性模型方法适用于全基因组关联研究。Nat Genet, 2010; 42:355-60。gydF4y2Ba
- 15.gydF4y2Ba
Atwell S, Huang Y, Vilhja’l B, Willems G, Horton M, Li Y,孟D, Platt A, Tarone A, Hu T,等。107种表型的全基因组关联研究gydF4y2Ba拟南芥gydF4y2Ba自交系。Nat。2010;465:627-31。gydF4y2Ba
- 16.gydF4y2Ba
赵魏黄X, X,唱T, Q,冯Q,赵Y,李C,朱C,陆T,张Z, et al。水稻地方品种14个农艺性状的全基因组关联研究。Nat Genet, 2010; 42:961-7。gydF4y2Ba
- 17.gydF4y2Ba
李辉,彭震,杨旭,王伟,付杰,王杰,韩勇,柴勇,郭涛,杨楠,等。玉米籽粒油脂生物合成基因结构的全基因组关联研究。Nat Genet, 2013; 45:43-50。gydF4y2Ba
- 18.gydF4y2Ba
毛瑞平,李志刚,张志刚,张志刚。高粱农业气候性状的群体基因组和全基因组关联研究。中国科学院学报(自然科学版),2013;gydF4y2Ba
- 19.gydF4y2Ba
孟S,何娟,赵涛,邢刚,李勇,杨松,陆娟,王勇,盖杰。中国大豆地方群体种子异黄酮含量qtl等位基因系统的检测及优化杂交设计和基因系统探索。理论应用,2016;129:1557-76。gydF4y2Ba
- 20.gydF4y2Ba
何俊,孟S,赵涛,邢刚,杨松,李勇,管锐,卢杰,王勇,夏强,等。全基因组关联分析是一种创新的方法,适用于种质群体和植物育种研究。理论应用,2017;130:2327-43。gydF4y2Ba
- 21.gydF4y2Ba
倪艳,张艳,张艳。迭代确定独立筛选EM-Bayesian LASSO算法在多位点全基因组关联研究中的应用。科学通报,2017;13:e1005357。gydF4y2Ba
- 22.gydF4y2Ba
张艳,何娟,王艳,邢刚,赵娟,李艳,杨松,Palmer R,赵涛,盖杰。大豆种质群体100粒重数量性状位点-等位基因矩阵的建立及其在大豆育种中的优化重组设计。中国机械工程学报,2015;gydF4y2Ba
- 23.gydF4y2Ba
胡勇,陈杰,方林,张震,马伟,牛勇,鞠林,邓杰,赵涛,连杰,等。gydF4y2Ba海岛棉gydF4y2Ba而且gydF4y2Ba陆地棉gydF4y2Ba基因组为异体四倍体棉花的起源和进化提供了见解。Nat Genet, 2019; 51:739-48。gydF4y2Ba
- 24.gydF4y2Ba
孙忠,王旭,刘忠,顾强,张勇,李忠,柯辉,杨洁,吴娟,吴林,等。通过全基因组关联研究,发现了水稻纤维品质性状的遗传变异和候选基因gydF4y2Ba陆地棉gydF4y2Ba中国生物技术学报,2017;29(8):982-96。gydF4y2Ba
- 25.gydF4y2Ba
李涛,马霞,李宁,周玲,刘震,韩辉,桂颖,鲍艳,陈娟,戴霞。陆地棉抗黄萎病候选基因的全基因组关联研究(gydF4y2Ba陆地棉gydF4y2Bal .)。中国生物工程学报,2017;3(2):344 - 344。gydF4y2Ba
- 26.gydF4y2Ba
苏娟,李玲,张超,王超,顾玲,王慧,魏华,刘强,黄玲,于生。中国陆地棉植株结构组成性状遗传变异及候选基因的全基因组关联研究理论应用,2018;131:1299-314gydF4y2Ba
- 27.gydF4y2Ba
张涛,胡勇,姜伟,方林,关霞,陈杰,张杰,Saski C, Scheffler B, Stelly D,等。同种异体四倍体棉花(Gossypium hirsutum L. acc。TM-1)为光纤改进提供了资源。生物工程学报。2015;33:531-7。gydF4y2Ba
- 28.gydF4y2Ba
Kevin D, Cao Z, Ping L, David S, French C, Anh-Hue T, Ann egydF4y2Ba支原体arthritidisgydF4y2Ba.《感染Immun》2008;76:4000-8。gydF4y2Ba
- 29.gydF4y2Ba
Swarbreck D, Wilks C, Lamesch P, Berardini TZ, Garcia-Hernandez M, Foerster H, Li D, Meyer T, Muller R, Ploetz L,等。的gydF4y2Ba拟南芥gydF4y2Ba信息资源(TAIR):基因结构和功能注释。核酸决议2008;36:D1009-14。gydF4y2Ba
- 30.gydF4y2Ba
USDA-FAS(2019)美国农业部,外国农业服务局。gydF4y2Bahttps://apps.fas.usda.gov/psdonline/app/index.html#/app/advQuerygydF4y2Ba.访问2020年3月10日。gydF4y2Ba
- 31.gydF4y2Ba
董宏,孔霞,李伟,唐伟,张东。植株密度和施氮钾对不同肥力田棉花产量和主要养分吸收的影响。农田作物杂志2010;119:106-13。gydF4y2Ba
- 32.gydF4y2Ba
毛玲,张磊,Evers J, Werf W,刘松,张松,王波,李忠。间作棉花产量及品质对氯胺磷和密度的响应。农田作物学报2015;179:63-71。gydF4y2Ba
- 33.gydF4y2Ba
孙芳,张娟,王胜,龚伟,史勇,刘安,李娟,龚杰,尚华,袁勇。陆地棉多代多环境纤维品质性状QTL定位。Mol Breed. 2012; 30:569-82。gydF4y2Ba
- 34.gydF4y2Ba
李松,曹颖,何娟,赵涛,盖杰。大豆嵌套关联定位群体中qtl -等位基因系统的检测。理论与应用,2017;gydF4y2Ba
- 35.gydF4y2Ba
Paterson AH, Brubaker CL, Wendel JF。一种快速提取棉花的方法(gydF4y2BaGossypiumgydF4y2Baspp.)适合RFLP或PCR分析的基因组DNA。植物分子生物学杂志1993;11:122-7。gydF4y2Ba
- 36.gydF4y2Ba
孙旭,刘东,张旭,李伟,刘辉,洪伟,蒋超,管宁,马超,曾华,等。SLAF-seq:一种利用高通量测序大规模从头SNP发现和基因分型的有效方法。科学通报。2013;8:e58700。gydF4y2Ba
- 37.gydF4y2Ba
苏娟,庞超,魏华,李玲,梁波,王超,宋敏,王辉,赵松,贾欣,等。通过GWAS鉴定陆地棉早熟相关性状的有利SNP等位基因和候选基因。BMC Genomics. 2016;17:687。gydF4y2Ba
- 38.gydF4y2Ba
李华,杜斌。基于钻穴-轮变换的快速精确短读对准。生物信息学,2009,25(14):1754 - 60。gydF4y2Ba
- 39.gydF4y2Ba
Van der Auwera GA, Carneiro MO, Hartl C, Poplin R, Del Angel G, levi - moonshine A, Jordan T, Shakir K, Roazen D, Thibault J,等。从FastQ数据到高置信变量调用:基因组分析工具包最佳实践管道。生物信息学,2013;11:11-33。gydF4y2Ba
- 40.gydF4y2Ba
巴雷特JC,弗莱B,马勒J,达利MJ。单倍图:LD和单倍型图的分析和可视化。生物信息学。2005;21:263-5。gydF4y2Ba
- 41.gydF4y2Ba
Wall JD, Pritchard JK。人类基因组中的单倍型阻滞和连锁失衡。Nat Rev Genet, 2003; 4:587-97。gydF4y2Ba
- 42.gydF4y2Ba
Gabriel SB, Schaffner SF, Nguyen H, Moore JM, Roy J, Blumenstiel B, Higgins J, DeFelice M, Lochner A, Faggart M,等。人类基因组中单倍型块的结构。科学。2002;296:2225-9。gydF4y2Ba
确认gydF4y2Ba
特别感谢余树勋教授和他的团队(中国农业科学院棉花研究所),他们为我们的实验提供了很好的指导。感谢林海、余宇、宁新竹、李吉莲和董成光(新疆农垦科学院棉花研究所)在2014 - 2015年为我们提供了试验田,并对石河子的表型性状进行了研究。gydF4y2Ba
资金gydF4y2Ba
基金资助:甘肃农业大学科技创新基金(GAU-KYQD-2018-01)、国家自然科学基金(31660409、31971986)、龙源青年创新创业人才计划(2020RCXM182)。资助机构在设计、分析、解释数据和撰写手稿方面没有任何作用。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
CXW、JJS和XFM设计了研究方案;JJS、AZ、JJL和SCH对数据进行分析并进行GWAS;JJS、XLZ和QM进行田间试验,鉴定性状;DLY、JJS和CXW撰写了手稿。所有的作者都阅读并批准了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
这篇文章不包含任何作者对人类参与者或动物进行的研究。gydF4y2Ba
发表同意书gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称不存在利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:图S1。gydF4y2Ba
分别与BN (A)、BW(B)和LP(C)相关的显著snpldb的qtl等位基因矩阵。gydF4y2Ba
附加文件2:表S1。gydF4y2Ba
315个陆地棉材料3个产量相关性状的表型分布范围。gydF4y2Ba
附加文件3:表S2。gydF4y2Ba
3个产量相关性状的相关性分析。gydF4y2Ba
附加文件4:表S3。gydF4y2Ba
4种种植环境下3个产量相关性状的方差分析。gydF4y2Ba
附加文件5:表S4。gydF4y2Ba
SNPLDB的显著位点分别与BN、BW和LP相关。gydF4y2Ba
附加文件6:表S5。gydF4y2Ba
在稳定的SNPLDB位点的6个相邻基因组片段中共注释了115个基因。gydF4y2Ba
附加文件7:表S6。gydF4y2Ba
两份RNA-seq数据显示,基因名主要在陆地棉纤维或胚珠中表达。gydF4y2Ba
附加文件8:表S7。gydF4y2Ba
16个潜在候选基因控制目标性状的GO结果。gydF4y2Ba
附加文件9:表S8。gydF4y2Ba
16个控制目标性状的潜在候选基因的KEGG结果。gydF4y2Ba
附加文件10:表S9。gydF4y2Ba
利用GWAS法在陆地棉中检测到与产量相关性状相关的SNP位点。gydF4y2Ba
附加文件11:表S10。gydF4y2Ba
315个陆地棉品种资料。gydF4y2Ba
附加文件12:表S11。gydF4y2Ba
弹性GSC矩阵和GSC特征向量。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.创作共用公共领域奉献弃权书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条所提供的资料,除非在资料的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
苏,J。,王、C。妈,Q。gydF4y2Baet al。gydF4y2Ba利用RTM-GWAS方法,研究了陆地棉3个产量相关性状的QTL等位基因和候选基因。gydF4y2BaBMC植物生物学gydF4y2Ba20.gydF4y2Ba416(2020)。https://doi.org/10.1186/s12870-020-02613-ygydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-020-02613-ygydF4y2Ba
关键字gydF4y2Ba
- 陆地棉gydF4y2Ba
- 产量性状gydF4y2Ba
- RTM-GWASgydF4y2Ba
- QTL等位基因gydF4y2Ba
- 候选基因gydF4y2Ba