- 研究文章
- 开放访问
- 发表:
转录组从头组装和全基因组鉴定揭示了GA3.参与黄麻纤维形成的胁迫响应WRKY转录因子(Corchorus capsularis.)
BMC植物生物学体积20.文章编号:403(2020)
摘要
背景
WRKY是一组在植物生长发育和抗逆性中发挥重要作用的转录因子。到目前为止,没有发现黄麻WRKY (CcWRKY)基因,即使黄麻(Corchorus capsularis.)是世界上最重要的天然纤维作物之一。关于黄麻中WRKY基因的研究还远远不足以理解韧皮纤维生物合成的分子机制。
结果
Illumina配对端测序共获得244,489,479条clean reads。重新组装得到90,982个ungenes,平均长度为714 bp。通过对已知蛋白的序列相似性搜索,共注释了48,896条(53.74%)ungenes。为了挖掘CcWRKY的tf并识别它们的潜在功能,需要搜索CcWRKYs针对黄麻的转录组数据进行,共计43次CcWRKYs在这项研究中确定了。通过GSDS2.0,MEGA7.0,DNAMAN5.0,Weblogo 3和Swiss-Model的生物信息工具分析了蛋白质的基因结构,系统发育,保守结构域和三维结构。系统发育分析表明43CcWRKYs根据WRKY保守域特征和用?进行进化分析,将其分为3组:I、II和III,分别包含9个、28个和6个成员拟南芥.基因结构分析表明这些外显子数量CcWRKYs从3到11。在43CcWRKYs、10、2、2和14个基因分别在营养生长旺盛的叶、茎杆、茎皮和根中表达量较高。其中21的表达式为43CcWRKYs利用FPKM和RT-qPCR对次级细胞壁生物合成基因进行显著调控,GA3.强调典型的GA3.敏感侏儒种质相比于在黄麻一个优良的栽培品种。的独联体-元素分析表明,这21个启动子CcWRKYs有1至4名涉及胃肠杆菌素响应性的CIS-元素,这表明他们可能会对GA的回应调节Bast纤维的发展3.压力。
结论
共43岁CcWRKYs首次在黄麻中发现。系统发育关系和基因结构分析表明CcWRKYs可能有功能上的差异。表达分析显示21个TFs为GA3.应激反应基因。这些的识别CcWRKYs对其表达模式的研究将为进一步阐明其在黄麻韧皮纤维发育中的作用提供依据。
背景
WRKY基因家族是仅存在于植物中的转录因子。主要参与植物的转录调控和信号转导过程[1].在前者中,WRKY转录因子结合特定的DNA序列,抑制或激活大量重要基因的转录[2,3.].保守的WRKY结构域由60个aa残基组成,WRKY结构域内的一个保守的WRKYGQK六肽序列通常伴有c2hc型或C2H2锌指结构。根据WRKY结构域的数量和锌指结构的类型,WRKY家族可分为3组:组I、组II和组III [4].第二组可进一步分为五个亚组:II-a, II-b, II-c, II-d和II-e。组I包含两个WRKY域和C域2H2锌指结构,组II包含一个WRKY结构域和一个C2H2锌指结构,ⅲ组包含一个WRKY结构域和一个C2-H-C锌指状结构[5].
自从第一个WRKY基因(SPF1)是从甘薯中克隆出来的[6, WRKY基因已经在许多植物中被发现,例如拟南芥[7)、大米(8],大麦[9],油菜籽[10.,大豆11.),玉米(12.],马铃薯[13.),烟草(14.],杨树[15.],棉花[16.],黄瓜[5]和葡萄[17.].目前,已发现腕骨转录因子在植物防御反应对环境压力和生长发育中发挥着重要作用。WRKY70 TF(转录因子)是JA(JasmOxic)和SA(水杨酸)介导的信号通路中的必需组分[18.].水杨酸激活WRKY70,茉莉酸抑制其表达[18.].在拟南芥,一些WRKY转录因子是ABA(脱落酸)介导的气孔关闭和干旱响应的正调控因子[19.].AtWRKY75in.拟南芥和OsWRKY31两者都与水稻侧根的生长有关[20.,21.].在糊粉细胞中,aba诱导的WRKY基因协同作用控制gamyb介导的脱落酸和赤霉素信号,从而建立了脱落酸和赤霉素信号交叉对话的新过程[22.].
黄麻(Corchorus capsularis.L)为二倍体(2n = 14) natural fiber plant, which belongs to the Malvaceae family [23.].它主要在孟加拉国,印度和中国种植。黄麻的韧皮纤维主要用于制造织物,绳索和螺纹,这是人类生命中不可或缺的一部分。由于植物纤维是可再生的,环保和100%可生物降解,越来越多的人对植物纤维纺织品和生产技术感兴趣[24.].近几十年来,黄麻受到了相当大的关注。
众所周知,WRKY基因在植物生长发育、逆境耐受性,特别是在纤维发育中发挥着重要作用[25.].和生物活性赤霉素(气)是也是一种重要的植物生长调节剂,这在干发展中发挥的关键作用。然而,没有任何的WRKY(CcWRKY)的基因已被鉴定黄麻至今。因此,当务之急是在基因组草图和转录[发布之后在黄麻识别和分析WRKY转录因子23.].本研究的目的是确定CcWRKYs并分析它们在不同组织中的表达模式,尤其是BAST纤维响应GA3.压力。
方法
植物材料及处理
两种基因型,典型的GA3.敏感的矮人种质(“AIDIANYehuangma”)和精英品种(“黄马179”),被用作本研究的植物材料。2017年5月1日在福建农业和林业大学农场生长了这两种基因型。播种后10天(DAS),只用“黄玛179”的三个植物中的下胚轴。当60 das,黄麻植物生长期的三个“黄马179”的三个植物中,叶子,根,茎吠钩和茎棒被独立收集。和茎吠戒独立于“AIDIANYehuangma”的三种植物中取样。当120 DAS时,茎吠须独立收集来自“黄玛179”的三植物。
探讨GA的作用3.以“黄麻179”为对照品种,“爱甸野黄麻”为试验品种。按MS培养基方法[26.],标准培养基(CK)和0.1 mg·L- 1遗传算法3.(GA3)外源性激素培养基。优良品种“黄麻179”和典型的GA3.本研究采用敏感矮秆种质“爱甸叶黄麻”。每瓶30粒种子在MS培养基下种植,在第一片叶片生长时测定下胚轴长度。
为了识别的表达CcWRKYs在Bast纤维中回应Ga3.在胁迫条件下,用100 mg·L处理6株2个基因型的植株- 1外源遗传算法3.在60 DAS。经过4 h和72 h的GA3.胁迫处理后,分别从“黄麻179”和“爱电野黄麻”中获得各植株的茎皮。并分别取3株2个基因型的植株作为对照。每个组织的3个样本立即在液氮中冷冻,并分别作为3个生物复制保存在−80°C,用于后续的RNA分离(附加文件)1:表S1)。
转录组测序组装
使用Plant Total RNA Isolation kit (Tiandz Inc.;中国北京),分别。将每个样本组织的rna均匀混合在一起进行文库构建和Illumina测序,Illumina测序由北京诺沃金生物信息技术有限公司,中国北京(https://www.novogene.cn/).总之,我们收集了10 μg的优质RNA,用于构建转录组文库。12个测序库的mrna(附加文件1表S1)用Poly-T oligo-attached magnetic beads从每个样本组织的总RNA中纯化。然后,在Illumina专利的碎片缓冲液中,在高温下用二价阳离子将这些mrna片段化。利用上标II和随机寡核苷酸合成第一cDNA链,用RNase H和Polymerase I合成第二cDNA链。通过凝胶纯化和Illumina聚合酶链反应(PCR)扩增筛选片段,获得cDNA文库。分别采用安捷伦生物分析仪2100和AMPure XP系统对PCR产物进行定量和纯化。参照制造商的建议,使用Illumina的cBot聚类生成系统(TruSeq PE Cluster Kit v3-cBot-HS)对样本进行指数编码聚类。聚类生成后,利用Hiseq 2000平台Illumina进行转录组测序,配对序列为90 bp。
原始数据(序列)读取过滤的质量通过修整读取Perl脚本房子保持适配器(聚-N和低质量序列读数)。提供高品质的组装的序列数据是从头组装到使用三一包含所有的考虑所有默认设置,并映射到转录物(短读 - 组件)程序的转录。即具有小于10×覆盖转录物去除和最后质量好的原始数据存放在NCBI序列读数存档(SRA)数据库(BioProject ID:PRJNA555734)与到SAMN12327440 SAMN12327429的加入代码。
通过同源性搜索,BLASTx对基因功能进行注释,E值< 10- 5)针对NCBI的Pfam、注释蛋白家族(http://pfam.janelia.org/);Swiss-Prot (http://www.expasy.ch/sprot);Nr,非冗余蛋白序列(http://www.ncbi.nlm.nih.gov);去,基因本体(http://www.geneontology.org).使用Blast2GO程序进行GO和nr标注。利用WEGO程序进行基因本体功能分类。Unigene序列也与同源蛋白组(COG/KOG,http://www.ncbi.nlm.nih.gov/COG)来预测和分类可能的功能。路径分配按照KEGG、京都基因和基因组百科全书路径数据库(http://www.genome.jp/kegg).
鉴定、序列比对和基因结构分析CcWRKYs
根据Islam等人的方法进行CcWRKY基因的组装和ORF分析[23.Zhang等[27.].中所有WRKY蛋白的全长氨基酸序列拟南芥(http://www.arabidopsis.org/)用作查询序列。使用一种E-value阈值为10- 6.只有在包含Wrky域名的情况下才接受从爆炸搜查中识别的所有潜在的CCWrky基因。并且使用多个序列对齐确认预测壁的保守域。此外,进行了使用PFAM的保守域预测,以确保所有候选基因含有衣服保守域[28.].通过使用SWISS-MODELL (http://swissmodel.expasy.org/)[29.].将全长蛋白序列及其相关编码序列加载到gds (Gene Structure Display Server) 2.0版本中,获得ccwrky的内含子/外显子信息。的同源蛋白拟南芥作为查询序列,黄麻次生细胞壁生物合成基因也被本地BLASTP鉴定。
系统发育分析CcWRKYs
由于涉嫌基因的完全蛋白质序列的可变长度,从预测的蛋白质中提取的腕部域用于绘制系统发育树。由MEGA7软件构建邻居加入进化树[30.].用p-距离法计算进化距离,用来估计每个位点的氨基酸取代数。进行1000个自举抽样步骤[12.]来建立每棵树的可靠性。系统发育树在此基础上分为不同的类群拟南芥注释和分类。WRKY家族序列来自拟南芥的资料,从TAIR下载(http://www.arabidopsis.org/).
RNA提取和基因表达分析
使用Plant Total RNA Isolation kit (Tiandz Inc.;中国北京),分别。参照制造商的说明书,使用GoScript™反转录系统(Promega, Madison, USA)从rna中转录出cdna。的Cc肌动蛋白基因作为内参基因,使CcWRKY表达正常化[31.].三个独立的反应使用快速启动通用SYBR®绿色硕士(ROX)用7500实时PCR仪(ABI)指的是制造商的公认CcWRKY基因进行。的reaction systems were: 0.4 μl of right primer (10 μM), 0.4 μl of left primer (10 μM), 10 μl of GoTaq® qPCR Master Mix, 7.2 μl of Nuclease-Free Water, and 2 μl of cDNA. Amplification conditions were programmed as follows: 30 s of denaturation at 95 °C, followed by 15 s of 40 cycles at 95 °C, and 30 s at 60 °C, with a melting curve temperature ranged from 55 to 99 °C. Each reaction was carried out in three biological replicates and the relative gene expression was determined with 2- - - - - -△△CT方法[32.].为了获得可靠的结果,每个样品进行了3次生物学重复和2次技术重复。数据为三个独立实验的平均值±标准差。误差条表示平均值的标准偏差。
根据基因长度和定位到该基因的reads数计算基因表达量为FPKM: FPKM =总外显子片段/[定位reads(百万)×外显子长度(kb)]。的表达分析CcWRKYs根据下胚轴-10d、叶-60d、根-60d、茎皮-60d、茎棒-60d、茎皮-120d的不同基因的FRKM值进行测定(附加文件1:表S1)。在遗传算法3.压力,精英品种“黄马179”用作参考品种来估计表达CcWRKYs及相关基因,而一个典型的GA3.以敏感矮秆种质“爱甸叶黄麻”为试验种质。热图是用R语言根据转换后的日志数据生成的2(FPKM + 1)值。以FDR < 0.05和|log2(fold change) | > 1为标准,估算处理样品间差异表达基因。所有数据分析使用SPSS统计软件20。的独联体的上游启动子5kb区域内的-元件CcWRKYs预测并由Plantcare进行分析。
结果
转录组从头组装
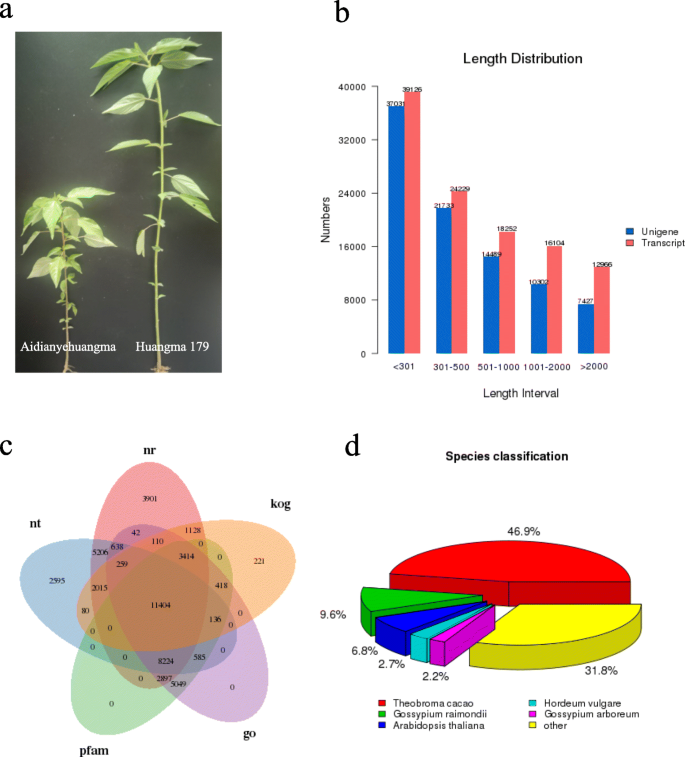
为了获得和表征GA的表达模式3.在黄麻的胁迫响应WRKY转录因子中,有12个测序库,分别来自2个基因型的下胚轴、叶片、根、茎皮和茎棒的不同生长阶段,是典型的GA3.对敏感矮秆种质“爱甸野黄麻”和优良品种“黄麻179”进行了比较研究。1a). Illumina配对端测序发现244,489,479条clean reads, GC比率为44.13%,Q30碱基为93.81% (Additional file .2:表S2)。此外,通过trinity程序从头组装了110,677个转录本,其中N50长度为1710 bp,平均长度为862 bp。1b,额外的文件3.:表S3,附加文件4:表S4)。其中73.73%的转录本长度在200 ~ 1000 bp之间。此外,90,982个unigenes的N50长度为1357 bp,平均长度为714 bp。1b,额外的文件3.:表S3,附加文件4:表S4)。unigenes的长度在201 ~ 15570 bp之间。58,764个长度在201 ~ 500 bp的unigenes, 14,489个长度在501 ~ 1000 bp的unigenes,大于1000 bp的unigenes为17,729个(19.49%)3.:表S3)。
为评价所鉴定的unigenes的质量,以trinity新组装的转录组序列作为参考序列。所有获得的干净序列通过RSEM软件映射到组装的unigenes上。对组装的unigenes功能注释,利用BLASTx对NCBI不同数据库Nr(非冗余蛋白序列)进行序列同源性搜索,e值为10- 5(图。1C,附加文件5:表S5)。在90,982个unigenes中,与SwissProt数据库和非冗余蛋白数据库中常见基因的同源性分别为39,238(43.12%)和33,410(36.72%)。总共有48,896(53.74%)的unigenes在不少于一个公共数据库(如GO, Nr, KOG, KO, PFAM或Swiss Prot)中进行了注释。最后,根据注释特征确定与黄麻相匹配的不同物种的序列数量(附加文件)6:表S6)。如图所示。1D,最丰富的三个物种是Theobroma可可(11457 46.9%),大麦芽(11071 9.6%)和Gossypium raimondii.(8274个,6.8%),约占总注释种的63.3%。
识别的长篇CcWRKYs和系统发育分析
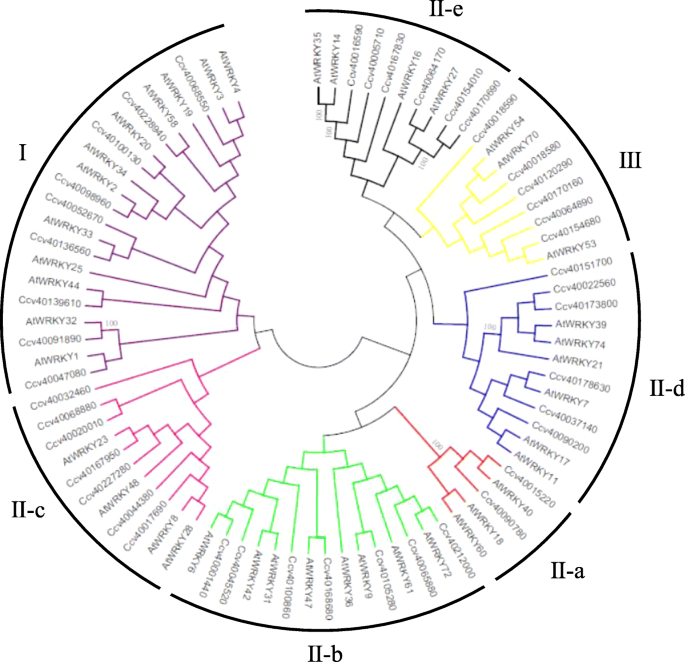
在黄麻中获得完整的鸦片成员(ccwrky.),BLASTP搜索,使用进行拟南芥TAIR的全长衣物蛋白序列(http://www.arabidopsis.org)查询。并利用Pfam检测其保守域。共鉴定出43个含有WRKY结构域的候选基因(命名为CcWRKY),如表所示1.
为了进一步评估CcWRKYs,采用DNAMAN5.0软件进行保守域识别,采用Weblogo进行保守结构,如图所示。2和额外的文件7.的保守域CcWRKYs可以分为三组:I、II和III,分别包括9、28和6名成员。组I可进一步分为I- c和I- n亚群,包含两个WRKY结构域和锌指结构,锌指结构为CX4C月22日至23日HXH。第II组又分为第II-a、第II-b、第II-c、第II-d和第II-e四个亚组,成员分别为2、7、7、6和6个。在II-a、II-b、II-d和II-e亚群中,WRKY c端七肽结构域和锌指结构为WRKYGQK和CX5C23.而II-c亚群中WRKY c端七肽结构域和锌指结构为WRKYGQK和CX4C23.HXH。ⅲ组为七肽结构域,WRKY c端锌指结构为WRKYGQK和CX7C23.HXC。
此外,目前的研究结果表明,尽管WRKY转录因子具有保守的WRKY结构域,但其蛋白序列仍然存在突变。在43CcWRKYs在黄麻中鉴定,一个基因(Wrkygqk)的保守结构域和四个基因的锌指结构都是突变的(附加文件7:表S7)。这些变异表明,尽管WRKY基因家族在结构上高度保守,但其WRKY结构域仍然存在一些变异,这也说明了植物WRKY基因家族在进化过程中具有功能多样性。
通过比较WRKY的tfA. Thaliana.与CcWRKYs,系统发育关系分析表明CcWRKYs可以分为三组:I,II和III。和II组可分为II-A,II-B,II-C,II-D和II-E子组(图。3.).系统发育树分析证实了保守域分析的结果CcWRKYs(图。2,表格1).
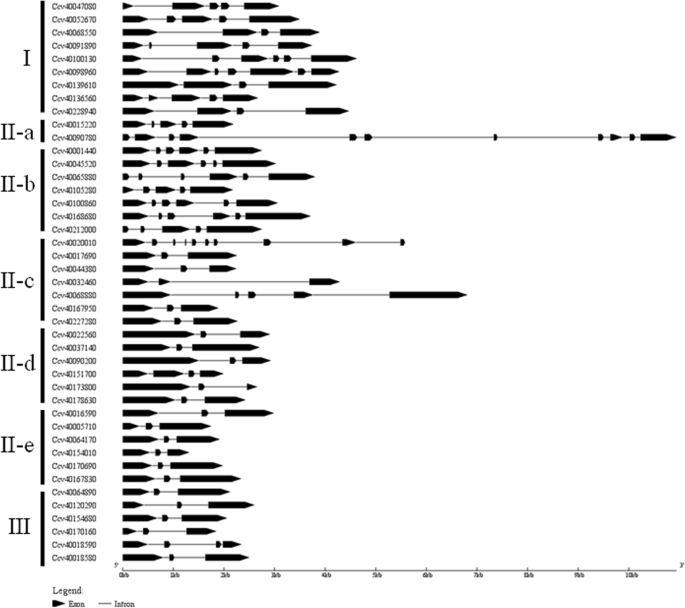
基因结构分析CcWRKYs
的外显子和内含子的数量CcWRKYs分析,如图所示。4.外显子的数量从3个到11个不等。21个wrky(48.84%)包含3个外显子,5个wrky(11.63%)包含4个外显子,8个wrky(18.60%)包含5个外显子,6个wrky(13.95%)包含6个外显子。不同组中,II组c + d + e和III组的外显子数相对保守,而I组和II组a + b + c的外显子数差异显著,差异较大。大部分的CcWRKYsin.group II c + d + e and group III contain 3 exons except Ccv40151700 (4 exons) and Ccv40018590 (4 exons).
基于二级结构,Wrkys的三级结构进一步卷曲和折叠。第三次结构CcWRKYs由SWISS-MODEL进行。大部分43的CcWRKYs有相似的三维结构。一个代表性的同源建模来自CcWRKYs显示在附加文件8(图S1),由几个beta折叠组成。这种三级结构与…相似拟南芥[33.,这证实了CcWRKYs在三级结构中高度保守。
表达式的分析CcWRKYs在不同的茎生长阶段
基因的组织特异性表达通常被认为是该组织中特定基因功能的标记。由于WRKY基因与植物韧皮纤维发育有关[34.,35.,表示CcWRKYs对茎生长的不同阶段进行了分析。用R语言绘制出表达模式的热图CcWRKYs在不同的茎生长阶段(附加文件9基于所述RNA-SEQ数据图S2):基因表达的差异通常通过不同的颜色来表示,即意味着红色代表高表达,蓝色之一代表低表达。全部CcWRKYs在茎生长的不同阶段都有表达,而CcWRKYs不同茎秆生长期不同。结果表明43例无假基因CcWRKYs.从热图的表达方式来看,43CcWRKYs分为两类(附加文件9:图S2)。表达43中的13CcWRKYs在黄麻各组织中含量较低,而在其他组织中含量较高。完全,10CcWRKYs在Leaf-60d、3CcWRKYs高度表达在缺杆萼-10d,2CcWRKYs在Stem stick-60d、2CcWRKYs在茎皮-60d、14CcWRKYs在Root-60d高表达,12CcWRKYs在茎皮-120d中高表达。可以看出,这些CcWRKYs主要表现在黄麻的茎皮中。随着黄麻茎生长期的延长,黄麻的韧皮纤维逐渐积累在茎皮中。因此,WRKY基因可能参与了黄麻韧皮纤维的发育。如Ccv40032460在下胚轴-10d高表达,在茎皮-60d低表达,在茎皮-120d无表达。提示该基因可能在黄麻纤维积累中起负调控作用。
遗传基因表达模式3.压力响应ccwrky.营养生长时期的基因
经0.1 mg·L的MS培养基处理后- 1外源遗传算法3.其中,“爱甸野黄麻”的下胚轴长度与未施外源激素的“爱甸野黄麻”相比差异显著,达到了优良品种“黄麻179”的下胚轴长度(附加文件)10.:图S3)。《爱dianyehuangma》是典型的GA3.敏感的矮化种质,如我们以前的研究所示[36.].鉴于生物活性赤霉素(GAs)在茎秆发育中起关键作用,利用“爱电叶黄麻”和“黄麻179”两个基因型对其表达模式进行了表征CcWRKYs在韧皮纤维中对GA的反应3.营养生长期(60das)的胁迫。
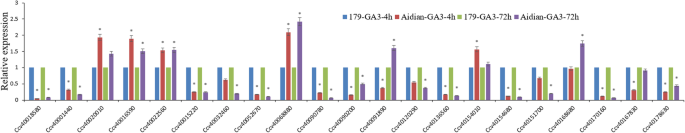
用GA处理茎皮3.在60 DAS上的“Huangma 179”和“Aidianyehuangma”的压力。然后,在喷涂GA之后,在两种不同的时间点中取样茎吠粒3.,分别。茎树皮没有GA3.治疗作为对照。对这些样本的RNA-seq数据进行分析,然后使用FPKM绘制相应的直方图(附加文件)11.:图。S4,附加文件12.:图S5)。以上x轴列显示基因表达上调,以下x轴列显示基因表达下调。大多数的表达CcWRKYs在GA3.压力,尤其是对下调基因而言。通过比较CcWRKYs在不同时间点(4和72 h后)喷涂GA3., 33的表达43CcWRKYs在“黄马179”和“AIDIANYEHUANGMA”中,茎吠戒是在同一趋势中调节。其中,它发现21CcWRKYs在GA下得到了显著的监管3.“爱甸野黄麻”与“黄麻179”的茎皮应力比较5和额外的文件13.:表S8),除了Ccv40020010、Ccv40015220、Ccv40068880、Ccv40154010和Ccv40090780。其中21CcWRKYs, 14的转录水平CcWRKYs被下调了7CcWRKYs在GA下,茎皮被上调了吗3.压力。为了验证基因表达的准确性,9CcWRKYs随机抽取进行RT-qPCR分析(附加文件14.:图S6)。RT-qPCR检测9CcWRKYs与FPKM的结果类似。
评价遗传算法3.参与细胞壁形成胁迫应答基因CcWRKY
评价遗传算法3.参与细胞壁形成胁迫应答CcWRKY基因,一些次生细胞壁生物合成基因,包括CesA1(中国极限运动协会,纤维素合酶),CesA4,CesA7,CesA8,CCoAOMT(咖啡酰辅酶甲基转移酶),和4 cl(4-香豆酸:辅酶A Ligase)作为标记基因。并筛选了一些赤霉素生物合成基因,如ent - copyll二磷酸合成酶、Ent-kaurene氧化酶、Ent-kaurene合成酶、Ent-kaurene酸氧化酶、ga20氧化酶、赤霉素2,3-羟化酶和赤霉素C13氧化酶。
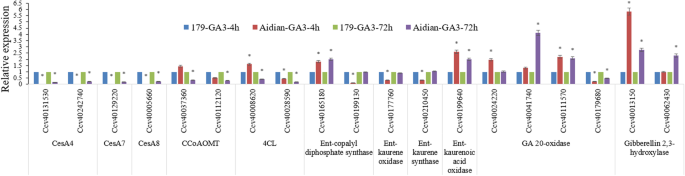
在“AIDIANyehuangma”中,大多数这些嗜酸性嗜酸性蛋白生物合成基因的表达的变化(测试:典型的GA3.敏感的矮化种质)比“黄马179”(对照:精英品种)更重要(图。6和额外的文件15.:表S9),表明遗传算法3.通过这些赤霉素生物合成基因的表达调控,在茎发育中发挥一定作用。值得注意的是,与黄麻179相比,“爱电液黄麻”中大部分次生细胞壁生物合成基因的表达明显下调(图2)。6).木质素合酶基因(CCoAOMT和4 cl)和纤维素合酶基因(CesA4,CesA7和CesA8)与黄麻179相比,爱甸野黄麻表达下调。结合21 GA的表达模式3.压力响应CcWRKYs(图。5),这就暗示了7个上调了CcWRKYs可能在应对GA中发挥积极作用3.而应力14则下调CcWRKYs可能在GA下的“AIDIANYEHUANGMA”的茎吠声的发展中发挥负面作用3.压力。
为了证实这一点暗示,独联体这21个促销员 - 元素分析CcWRKYs采用PlantCARE进行分析(表2).这些21的推动者CcWRKYs1到4独联体参与赤霉素应答性-elements,如GARE基序,P-箱和TATC盒。这21个CcWRKYs给予答复GA的压力3.并能提高“爱甸野黄麻”的株高等纤维相关性状,提示它们可能参与了黄麻韧皮纤维发育对赤霉素的响应3.压力。
讨论
CcWRKY转录因子在黄麻
WRKY转录因子是植物中最大的转录调控因子家族之一。它们在植物发育、生物和非生物胁迫中发挥重要作用[1,37.,38.,39.].本研究在黄麻的全基因组中鉴定了43种腕表。黄麻的腕表数量与糖甜菜(40)中的一致(40)一致[40]、油菜籽(43)[41.]和蓖麻(47)[42.].然而,在其他植物的基因组中,它是不一致的拟南芥(74),棉花(116)[16.]、大豆(188)[43.],杨树(104)[15.],西红柿(81)[44.卷心菜(145)[45.],其成员不仅仅是黄麻的成员。通过与不同的物种进行比较,不同物种中的腕部数量与其基因组大小不成比例。最近,研究人员提出,基因重复,细分重复和全基因组重复在基因家族的大规模生产中起重要作用[43.].不幸的是,由于黄麻研究的数据不足,特别是基因组数据。唯一公布的黄麻基因组草案不足以为许多问题提供令人满意的解决方案。我们怀疑黄麻基因组WRKY基因比其他物种少,可能是因为它们不像其他物种那样经历整个基因组复制。但是,随着基因组测序和组装的改进,我们认为WRKY的新成员也存在于黄麻基因组中。
通常,基因组中内含子和外显子的位置可以为其进化关系提供重要证据。在这项研究中,它全面分析了外显子和内含子的分布和长度CcWRKYs.通过分析基因结构CcWRKYs发现其成员由3 ~ 11个外显子组成,其中近一半为3个外显子。此外,黄麻中WRKY基因家族成员具有相似的三维结构,由多个β折叠构成。它类似于区域的三维结构拟南芥数据库中的WRKY蛋白[33.].这些结果为WRKY基因家族在不同物种中的进化提供了有价值的信息。
遗传算法3.参与细胞壁形成胁迫应答基因CcWRKY
第III组基因是WRKY基因家族中的一个,在植物的进化和适应过程中起着至关重要的作用,因此被认为是进化中最先进的[46.].但是,根据表达分析CcWRKYs在不同的组织中,可以发现大多数CcWRKYsⅲ组除Ccv40018590和Ccv40064890两个基因外,其余基因(Ccv40018580、Ccv40120290、Ccv40170160和Ccv40154680)均在纤维发育过程中高表达。这些年代结果在黄麻是一致的,以在棉花[47.].有人建议CcWRKYs在黄麻中的III族中可能对纤维作物具有一定的功能分化,这可能对作物的纤维发育可能一定的影响。近年来,王等人的研究。[48.]表明拟南芥WRKY转录因子参与了次生细胞壁的形成,可以显著增加植物的生物量,如AtWRKY12.在本研究中,大多数CcWRKY基因在茎皮和下胚轴中表达不同。这一有趣的现象使我们相信WRKY转录因子与韧皮纤维发育之间存在一定的联系。
植物激素在植物纤维素生物合成中起着重要的作用。赤霉素是植物在整个生命周期中生长发育的关键激素。赤霉素对棉纤维的起始、分化和发育有很大的影响,可以诱导更多的纤维初始细胞,刺激纤维伸长。因此,43CcWRKYs在遗传算法3.系统地分析了本研究中的应力。因为GA的影响很大3.在植物高度上,是典型的GA3.选择敏感矮秆种质“爱甸叶黄麻”作为赤霉素的靶标3.压力治疗。表达模式显示43例中有21例表达CcWRKYs在“AIDIANYEHUANGMA”中受到严重监管。它表示这些CcWRKYs在GA下是否可能参与韧皮纤维的发育3.压力。目前,已经表明,吉布林素介导的信号级联调节纤维素合成[49.].因此,Gibberellin基因的突变可以改变CesAs(纤维素合酶)基因的转录。GA对陆生植物次生壁纤维素的合成具有调控作用。已有研究报道WRKY转录因子参与表皮毛发发育、葡萄糖和赤霉素信号转导[50.].因此,进一步研究CcWRKY基因与GA之间的关系将是非常有意义的3.压力和纤维发育。
此外,由于黄麻的经济重要性,研究wrky基因的转录调控对黄麻的改良具有重要意义。这些CcWRKY基因对纤维发育和对GA的响应有明显的影响3.压力。目前,国内外对转录因子的研究较多拟南芥只是其中的一部分,它无法完全揭示每个转录因子的功能。由于不同的进化方向,相同的转录因子在不同物种中具有不同的功能。总体而言,本研究为随后研究CCWrky基因及其对纤维发育的影响提供了一种理论依据,并通过CCWrke基因的鉴定和生物信息分析分析。
结论
对12个测序库的转录组进行从头组装,获得GA的表达模式并进行表征3.黄麻的逆境响应WRKY转录因子。完全,43岁CcWRKYs在黄麻中被发现。这些CcWRKY基因的外显子数量差异很大,表明它们可能具有功能多样性。它们的表达概况CcWRKYs在茎发育的不同阶段表现出不同的表达模式。21的43CcWRKYs次级细胞壁生物合成基因对GA3.压力。这表明CcWRKYs可能与黄麻韧皮纤维的生长发育有关。
数据和材料的可用性
未加工的Illumina序列数据存放在国家生物技术信息中心(NCBI)中,并在序列读取归档(SRA)数据库中访问(https://www.ncbi.nlm.nih.gov/sra).登录号为PRJNA555734 (https://www.ncbi.nlm.nih.gov/bioproject/PRJNA555734).本研究中产生或分析的所有数据均包含在本文及其补充信息文件中。
缩写
- 遗传算法3.:
-
赤霉素
- CcWRKY:
-
黄麻(Corchorus capsularis.) WRKY
- AtWRKY:
-
拟南芥WRKY;
- 中国极限运动协会:
-
纤维素合成酶
- CCoAOMT:
-
咖啡酰辅酶A甲基转移酶
- 4 cl:
-
4-香豆酸:辅酶连接酶
- TF:
-
转录因子
- FPKM:
-
片段每千碱基每百万短片段
参考文献
- 1。
罗斯顿P,Somssich I,Ringler P,Shen JQ。腕骨转录因子。趋势植物SCI。2010; 15(5):247-58。
- 2。
关键词:烟草,WRKY, DNA结合蛋白,W-box元件植物科学。2001;161(4):655 - 64。
- 3.
转录因子复合物。中国生物医学工程学报。2002;12(2):225-30。
- 4.
wang j, wang j, wang j, et al . WRKY转录因子的研究进展。植物科学进展,2000;5(5):199-206。
- 5。
玲j,姜w,张y,yu h,mao z,顾x,黄s,谢b。衣服基因家族的基因组分析Cucumis巨大成功.BMC基因组学。2011;12(1):471。
- 6。
Ishiguro S, Nakamura K.编码一种新的dna结合蛋白SPF1的cDNA特征,该蛋白识别编码甘薯孢子素和β -淀粉酶基因的5 '上游区域的SP8序列。Mol Gen Genet MGG。1994, 244(6): 563 - 71。
- 7。
Riechmann JL, Heard J, Martin G, Reuber L, Jiang ZC, Keddie J, Adam L, Pineda O, Ratcliffe OJ,等。拟南芥转录因子:真核生物全基因组比较分析。科学。2000;290(5499):2105 - 10。
- 8.
Ramamoorthy R,Jiang S-Y,Kumar N,Venkatesh Pn,Ramachandran S.在各种非生物和植物精神治疗下填充水稻腕骨基因家族的综合转录分析。植物细胞生理。2008; 49(6):865-79。
- 9.
Mangelsen E,Rilian J,Berendzen K,Kolukisaogluü,Harter K,Jansson C,WANKE D.大麦的系统发育和对比基因表达分析(大麦芽) WRKY转录因子家族揭示了单子叶和双子叶之间潜在的保留功能。BMC基因组学。2008;9:194 - 200。
- 10。
杨波,蒋永强,Rahman M, Deyholos M, Kav NNV。油菜WRKY转录因子的鉴定与表达分析(芸苔属植物显著对真菌病原体和激素处理的响应。BMC植物BIOL。2009; 9(1):68-70。
- 11.
宋庆林,陈建军,程建军,等。古多倍体大豆的基因组序列。自然。2010;465(7278):178 - 83。
- 12.
魏k-f,陈俊,陈yf,wu l-j,xie d-x。玉米完全腕骨转录因子家族的分子系统发育及表达分析。DNA Res。2012; 19(2):153。
- 13。
Dellagi A能力HJ Avrova AO,能力AA, Montesano M,能力MM, Palva ET,能力PE、斯图尔特,他能力SH,托斯本土知识,能力TI,库克德,德能力C,里昂GD,能力LG、桦树公关、桦木公关。马铃薯基因编码一个WRKY-like转录因子诱导与Erwinia carotovora无性系种群的相互作用。atroseptica和5种与I类几丁质酶的表达协同调控。植物生态学报,2000;13(10):1092-101。
- 14。
王志强,杨鹏,范斌,陈志强。植物防御反应中序列特异性dna结合活性的寡聚选择方法。植物j . 1998; 16(4): 515 - 22所示。
- 15。
何华,董强,邵永强,姜辉,朱胜,程斌,向阳。中国水稻WRKY基因家族的全基因组调查与鉴定杨树trichocarpa.植物学报2012;31(7):1199-217。
- 16。
窦磊,张旭,庞超,宋敏,魏华,樊胜,于胜。棉花WRKY基因家族的全基因组分析。中国生物医学工程学报。2014;33(6):591 - 598。
- 17。
Wang L, Zhu W, Fang L, Sun X, Su L, Liang Z, Wang N, Londo JP, Li S, Xin H. WRKY家族基因的全基因组鉴定及其对低温胁迫的响应vitis Vinifera.植物学报。2014;14(1):103。
- 18.
wang J, wang J, wang J, ET al . WRKY70转录因子在植物防御中的表达。植物细胞。2004;16:319-31。
- 19.
Rushton DL,Tripathi P,Rabara Rc,Lin J,Ringler P,Boken Ak,Langum TJ,Smidt L,Boomsma DD,EMME NJ,等。WRKY转录因子:脱钙信号中的关键组分。植物Biotechnol J. 2012; 10(1):2-11。
- 20.
张军,彭勇,孙志贤。在转基因水稻植株中,病原菌诱导OsWRKY31的组成型表达增强了抗病能力,并影响了根系生长和生长素反应。细胞研究》2008;18(4):508 - 21所示。
- 21.
Devaiah BN, Karthikeyan AS, Raghothama KG。WRKY75转录因子是磷酸获取和根发育的调节因子拟南芥.植物理性。2007; 143(4):1789-801。
- 22.
谢智,张志林,邹旭,杨刚,小松松,沈启军。两种脱落酸诱导的WRKY基因抑制糊粉细胞中赤霉素信号的相互作用。植物j . 2006; 46(2): 231 - 42。
- 23.
Islam MS, Saito JA, Emdad EM, Ahmed B, Islam MM, Halim A, Hossen QMM, Hossain MZ, Rasel Ahmed MSH, Kabir SMT,等。两种黄麻的比较基因组学和纤维生物发生的洞察力。Nat植物。2017;3(1):16223。
- 24.
利用激光扫描系统测定亚麻纤维细度的方法。作物学报。2006;23(3):273-87。
- 25.
Al-Ghazi Y, Bourot S, Arioli T, Dennis SE, Llewellyn DJ。纤维发育过程中的转录谱鉴定了可能影响棉纤维质量的次生代谢和细胞壁结构的途径。植物生理学报。2009;50(7):1364。
- 26.
Green CE, Phillips RL。玉米组织培养植株再生研究。作物科学。1975;15:417-21。
- 27.
关键词:黄麻,纤维素生物合成,转录组,基因序列Corchorus capsularis.l .)。BMC基因组学。2015;16(1):164 - 79。
- 28.
田义,东Q,吉泽,志法,康吉,周Z.苹果疯子箱基因家族的基因组鉴定与分析。基因。2015; 555(2):277。
- 29.
阿诺德K,Bordoli L,柯普Ĵ,Schwede T的SWISS-MODEL工作区:蛋白质结构同源性建模的基于web的环境。生物信息学。2006; 22(2):195-201。
- 30.
MEGA7:用于更大数据集的分子进化遗传学分析7.0版本。中国生物医学工程学报。2016;33(7):1872。
- 31。
钮X,齐Ĵ,张G,徐军,陶A,方P,可靠的参考基因在黄麻定量实时PCR的基因表达分析苏J.选择(Corchorus capsularis.)在压力处理下。植物科学学报2015;6:848。
- 32。
利用实时荧光定量PCR技术分析相关基因表达。方法(圣地亚哥,加利福尼亚州)。2002; 25(4): 402 - 8。
- 33。
段Mrnj,Lu L,Li Lf,Wei Ch,Lai Lh,Li Y,Su XD。高分辨率晶体结构揭示了DNA装订机构拟南芥WRKY1蛋白质。核酸学报2007;35(4):1145-54。
- 34。
王浩,中岛君,韩明国,陈芳,Dixon RA。在双子叶植物中,WRKY转录因子的突变启动髓次生壁的形成,增加茎的生物量。中国科学(d辑:地球科学)2010;
- 35。
BF guo - y, LIU Wei, BI Cai-li。木质素生物合成中转录因子调控的研究进展。中国农业科学,2015;48(7):1277-87。
- 36。
张立伟,陶爱芬,林立辉,徐建堂,方平平,戚建民。黄麻矮秆种质植物株高外源激素敏感性研究(Corchorus capsularis.l .)。科学科学罪。2015; 48(10):1892-9。
- 37。
Ulker B, somsich I, Ülker B, somsich IE。WRKY转录因子:从DNA结合到生物学功能。植物学报。2004;7(5):491-8。
- 38。
谢震,张志龙,邹旭,黄杰,Ruas P, Thompson D,沈庆军。水稻WRKY基因超家族的注释和功能分析揭示了糊粉细胞中脱落酸信号的正调控和负调控。植物杂志。2005;137(1):176 - 89。
- 39。
王志强,王志强。WRKY转录因子在防卫信号转导中的作用。植物学报。2007;10(4):366-71。
- 40.
孔维,龙YK,NaiZhen DAN,ShaoZong杨,满珠包,向荣黄,小鹏FU。WRKY转录因子在非生物胁迫下的全基因组鉴定和表达分析甜菜属.科学科学罪。2017; 50(17):3259-73。
- 41.
杨B,姜Y,拉赫曼MH,Deyholos MK,NNV K.鉴定及在低芥酸菜子WRKY转录因子基因表达分析(芸苔属植物显著对真菌病原体和激素处理的响应。植物学报。2009;9(1):68。
- 42.
李海龙,张立波,郭东,李长志,彭世强。WRKY转录因子家族的鉴定和表达谱萝藦.基因。2012;503(2):248。
- 43.
于宇,王宁,胡荣华,向峰。大豆WRKY转录因子对盐胁迫响应的全基因组鉴定。SpringerPlus。2016; 5(1): 920。
- 44.
黄胜,高艳,刘建平,彭晓霞,牛晓霞,菲志,曹胜,刘玉兰番茄.基因组学杂志。2012;287(6):495-513。
- 45.
唐军,王芳,侯学林,王智,黄志宁。大白菜WRKY转录因子的全基因组分离与鉴定芸苔属植物拉伯ssp。揭示了在非生物和生物胁迫下的共线性及其表达模式。植物学报。2014;32(4):781-95。
- 46.
谢涛,陈超,李超,刘静,朝阳,何玉梅。菠萝WRKY基因家族的全基因组研究:发育和胁迫过程中的进化和表达谱。BMC基因组学。2018;19:490。
- 47.
玲玲d,亚宁G,埃文斯O,超你P,恒玲W,梅珍S,疏利女,树迅Y.鉴定及棉组III WRKY转录因子的表达分析.ĴINTEGR AGRIC。2016; 15(11):2469-80。
- 48.
王华,Avci U,中岛君,Hahn MG,陈芳,Dixon RA。在双子叶植物中,WRKY转录因子的突变启动髓次生壁的形成,增加茎的生物量。中国科学(d辑:地球科学)2010;
- 49.
黄D,王某,张b,尚冠k,shi y,张d,刘x,wu k,xu z,fu x等。吉布林蛋白介导的Della-NaC信号传导级联调节水稻中的纤维素合成。植物细胞。2015; 27(6):1681-96。
- 50.
拟南芥的毛状体和种皮发育基因GLABRA2编码WRKY转录因子。植物细胞。2002;14(6):1359 - 75。
确认
不适用。
涉及植物的研究
这项工作中对植物的实验研究和实地研究符合国际自然保护联盟关于濒危物种研究的政策声明和《濒危野生动植物种贸易公约》。
资金
本研究由国家自然科学基金项目(no . 31771369)和国家韧皮叶纤维作物农工技术研究体系项目(no . nycytx-19-E06)资助。资助数字为研究项目提供了资金支持,但未涉及工作设计、数据收集、分析和稿件准备。
作者信息
从属关系
贡献
LZ设计并指导研究;XW和YX对数据进行了分析;LZ在所有作者的输入下撰写了手稿;SN、JQ和LZ贡献了新的种质和技术。所有的作者编辑并同意最终的手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
RNA样品为黄麻(c . capsularis)。
附加文件2:表S2。
黄麻转录组序列的统计摘要。
附加文件3:表S3。
转录本和Unigenes的长度间隔及其分布。
附加文件4:表S4。
拼接长度分布。
附加文件5:表S5。
公共数据库中的Unigenes注释。
附加文件6:表S6。
Nr数据库中注释的unigenes。
附加文件7:表S7。
的七肽WRKYGQK和锌指结构的变化WRKY结构域CcWRKYs.
附加文件8:图S1。
黄麻WRKY家族蛋白三维结构预测
附加文件9:图S2。
表达谱的CcWRKYs在不同的组织和发育阶段。的FPKM值ccwrky.基因通过转化的log 2和热图中由R语言构造。
附加文件10:图S3。
赤霉素处理“黄麻179”和“爱甸野黄麻”幼苗平均下胚轴长度的比较3..用竖线(*)表示标准差P< 0.05,学生的t以及)。
附加文件11:图S4。
成绩单丰度CcWRKYsga3.压力。的相对表达水平ccwrky.将各品种各处理时间点的基因与正常条件下各处理时间点的基因进行比较。用竖线表示标准差。以FDR < 0.05和|log2(fold change) | > 1为标准,估计处理样品间的差异表达基因。179:优良品种“黄麻179”,爱电:GA3.敏感矮秆种质“爱甸叶黄麻”,ga3 - 4h3.应激处理,GA3-72 h: GA 72 h后3.压力治疗。
附加文件12:图S5。
GA后次生壁生物合成基因的转录本丰度3.压力。CESA:纤维素合成酶;CCOAOMT:Caffeioyl辅酶甲基转移酶;4CL:4-香豆素:辅酶是一个连接酶。将每个种类的每个处理时间点中的二次壁生物合成基因的相对表达水平与正常条件下的每次点的相对表达水平进行比较。用竖线表示标准差。以FDR < 0.05和|log2(fold change) | > 1为标准,估计处理样品间的差异表达基因。179:优良品种“黄麻179”,爱电:GA3.敏感矮秆种质“爱甸叶黄麻”,ga3 - 4h3.应激处理,GA3-72 h: GA 72 h后3.压力治疗。
其他文件13:表S8。
表达水平CcWRKYs选育优良品种“黄麻179”和GA3.敏感矮秆种质“爱甸叶黄麻”对赤霉素的响应3.压力。
附加文件14:图S6。
9表达模式的RT-qPCR测定CcWRKYs对GA的回应3.治疗方法。数据是三个生物重复计算的平均值±SD。179:优良品种“黄麻179”,爱电:GA3.敏感矮秆种质“爱甸叶黄麻”,ga3 - 4h3.应激处理,GA3-72 h: GA 72 h后3.压力治疗。
附加文件15:表S9。
对优良品种“黄麻179”和赤霉素几个次生细胞壁生物合成基因的表达量进行了研究3.敏感矮秆种质“爱甸叶黄麻”对赤霉素的响应3.压力。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
张磊,万旭东,徐勇。et al。转录组从头组装和全基因组鉴定揭示了GA3.参与黄麻纤维形成的胁迫响应WRKY转录因子(Corchorus capsularis.).BMC植物BIOL.20.403(2020)。https://doi.org/10.1186/s12870-020-02617-8
收到:
接受:
发表:
关键字
- 黄麻
- WRKY转录因子
- 遗传算法3.压力
- 表达式模式
- 系统发育分析