抽象的
背景
C2H2锌手指蛋白(C2H2 ZFPS)在塑造植物生长和适应环境的许多方面起着重要作用。植物基因组港口数百C2H2 ZFPS,其构成了高等植物中最重要和最大的转录因子家族之一。虽然在几种植物物种中报道了C2H2 ZFP基因家族,但尚未在模型豆科物种中描述Medicago Truncatula..
结果
在这项研究中,我们确定了218个C2H2型ZFP,其中337个单独的C2H2主题M. Truncatula..我们表明,局部基因重复的高率显着促进了C2H2基因家族的扩张M. Truncatula..所识别的ZFPS表现出高变型的基序布置和表达模式,表明短的C2H2锌指丝膜被用作具有不同功能的多种转录因子来识别顺式元素的转录因子。通过分析公共表达数据集和定量RT-PCR(QRT-PCR),我们鉴定了几种组织中特异性表达的几种C2H2 ZFP,例如结节,种子和花。
结论
我们的基因组 - 在一个重要的豆科植物中揭示了扩展的C2H2 ZFP基因家族M. Truncatula.,并为高等植物中C2H2 ZFP的多样化和扩展提供了新的见解。
背景
锌指蛋白(ZFPs)在植物界中已经进化并多样化成为一个庞大的转录因子家族[1那2].ZFP含有一至少量锌指,其长度约为23至30个氨基酸,并且具有几种半胱氨酸和组氨酸残基。基序中的半胱氨酸和组氨酸残基通过氢键坐标,形成一个或多个锌离子,形成稳定的指状结构,可以与核酸序列相互作用以调节靶基因表达。基于半胱氨酸和组氨酸残基的组合,ZFPS可以分为不同类型,例如C 2 H 2,C 2 C 2,C 2 HC和C3H [3.那4.那5.那6.].
C2H2型锌手指是ZFPS中最常见的基序之一,它可以由序列X2-Cys-X2,4-Cys-X12-His-X3,5-His-X3,4,5-(X表示任何氨基酸残留物和数字代表残留量)[7.那8.].这个大约23 - 30个氨基酸基序包含两个β薄片链和一个α- 分别在N-末端和C-末端,其围绕中心锌离子折叠以建立与靶DNA序列的主凹槽结合的稳定的简单结构[8.].ZFPs中C2H2锌指的数量是可变的,从一个到几十个,从而提供了灵活的亲和力与核酸结合[7.那9.].通过与DNA段相互作用的能力,C2H2 ZFP主要在控制靶基因的转录方面发挥作用,用作典型转录因子。有趣的是,乙烯响应元件结合因子相关的两亲性抑制基序(耳),其与转录核心压制器裸照(TPL)结合并抑制下游基因的表达,通常在C2H2 ZFP中发现,从而能够实现C2H2的转录抑制函数zfps。此外,一些C2H2 ZFP可以结合RNA或其他蛋白质参与RNA代谢,基因表达或蛋白质活性调节[9.那10那11那12那13].
C2H2 ZFP首次在非洲爪青蛙报道(非洲爪蟾蜍光滑的)转录因子IIIa(TFIIIA)蛋白,其由九个重复串联C2H2基序组成[14].在较高的植物种类中,在测序的基因组中发现了数百个C2H2 ZFP [1那12那15那16],包括一些功能表征的。C2H2 ZFPS在调节植物生长和发育,植物激素信号传导和非生物和生物应激反应的许多方面,表现出广泛的作用。[13那17那18那19那20.那21那22].
已经提出,C2H2基序通常可以分为两种类型:Q型和C型。Q型C2H2具有核心序列'Qalggh',其中'H'是C2H2基序的第一个组氨酸。几次Q型C2H2 ZFPS已经很好地研究,包括超人(sup)在拟南芥中,1 .耐干旱、耐盐(DST1)和像棕榈酸酯的pentafoliata1(Palm1./锈病抑制剂分化1(IRG1) 在M. Truncatula.[17那18那21那23那24].据报道,这种类型的C 2 H 2锌手指是特异于植物的,而在所有生物体中呈现的C型蛋白质并没有含有保守的核心序列[1那12那15那16那25那26].除了Q型和C型C2H2 ZFP外,一些最近的基因组研究还揭示了植物物种中新型C2H2的存在,例如改进的Q型(QM型)和Z型C2H2图案[15那26那27].
Medicago Truncatula.(也称为Barrel Medic)是一种模型豆类物种,靠近饲料物种苜蓿(Medicago Sativa.).在M. Truncatula.,只有少数C2H2 ZFPs被表征,如Palm1./IRG1和Symbiosome分化的监管机构(标准偏差)[23那24那28那29],两者在N-末端和C-末端的耳动术中具有单个C2H2基序。Palm1./IRG1在确定这一点方面发挥着重要作用M. Truncatula.复合叶型和表皮蜡质代谢[23那24那29那30.], 然而标准偏差控制结节开发期间的Symbiosome分化[28].
大多数C2H2 ZFPsM. Truncatula.尚未表征,缺乏基因组 - 范围的序列和C2H2 ZFP的表达分析。在这项研究中,共鉴定了总共218c 2 H 2类型的ZFPM. Truncatula.它们中的大多数只包含一个C2H2基序。系统分类了C2H2基序和ZFPs基序,并分析了所鉴定基因的基序排列。我们的结果表明,除了守恒的图案,乙炔ZFPs显示高序列变异在家庭成员中,展示功能多样化和乙炔主题支持的概念一直采用植物基因组提供多功能脚手架蛋白识别目标DNA序列。利用序列分析和同源搜索的方法,我们鉴定了许多C2H2 ZFPs与其他植物中特征良好的ZFPs (indetermined -domain, IDD)基因具有很高的相似性。与序列变异一致,微阵列表达分析显示C2H2 ZFPs的表达模式高度可变,进一步支持该家族基因的功能多样化。定量RT-PCR (qRT-PCR)的表达分析也证实了少数C2H2 ZFPs在某些组织中特异性表达,为进一步研究C2H2基因功能提供了有价值的候选基因。我们的工作鉴定和分类了一个C2H2型ZFPs大家族M. Truncatula.并且对鉴定的ZFP进行了基因结构和表达分析。本工作中提出的基因家族的图片对C2H2 ZFP的扩展和多样化进行了见解M. Truncatula.,并有助于了解C2H2 ZFP在此模型豆类中的角色。
结果
C2H2 ZFPS概述了M. Truncatula.基因组
Medicago转录因子数据库中列出了112个C2H2型ZFP(http://planttfdb.cbi.pku.edu.cn/index.php?sp=Mtr).鉴于在其他物种中报道了大量的C2H2 ZFPs [15那16那27,例如,拟南芥和大豆基因组中分别有176和321个C2H2 ZFPs [16那27],我们认为应该有更多的C2H2 ZFPM. Truncatula.基因组。到目前为止,C2H2 ZFPs的报道很少M. Truncatula..因此,文献调查没有返回关于C2H2 ZFP系列的许多有用信息M. Truncatula..为了广泛识别C2H2 ZFP,我们使用了一个组合的策略,并搜索了基因组数据库Mtruna17r5.0(https://medicago.toulouse.inra.fr/MtrunA17r5.0-ANR)和mt4.0v2(http://www.medicagogenome.org.)[31那32那33].基于报告的C2H2主题序列模式,我们在编程语言Python中使用正则表达式模块开发了自定义脚本。结果,发现了含有381c2H2基序的272个候选C2H2 ZFPS。为了消除潜在的假阳性,候选者进一步扫描了Prosite蛋白质数据库和PFAM平台以确认。通过C2H2主题扫描的候选者在俯视率和PFAM平台中或显示出显着的相似之处拟南芥或水稻C2H2 ZFPsM. Truncatula.乙炔ZFPs类型。该步骤排除了55个候选项,得到217个C2H2 ZFPs,其中包含328个C2H2 motifM. Truncatula.(无花果。1,附加文件1,附加文件2和附加文件3.).
TFIIIA是植物中含有C2H2基序最多的ZFP [14那16那34].对确定的217 ZFP的初步检查发现,TFIIIA不存在M. Truncatula.最新基因组(Jemalong A17)。我们怀疑它可能是由排序或注释误差引起的。使用其他品种R108的基因组序列(http://www.medicagohapmap.org/downloads/r108)和Affymetrix微阵列探测数据,我们识别并手动测序TFIIIA同源物M. Truncatula.(如下面所描述的)。
总之,我们鉴定了含有337C2H2基序的218C2H2 ZFPS(图。1,附加文件1,附加文件2和附加文件3.)远远超过植物转录因子数据库中列出的基因。所识别的C2H2 ZFPM. Truncatula.显示长度和分子量的变化,范围为小于100至1700多氨基酸,分别为7.3至197千杆(KDA)。然而,大部分家庭(总共218 ZFPS的182个)非常短(少于500个氨基酸)。所鉴定的C2H2 ZFP还显示出4.14至9.92的宽范围的等电点(PI),表明蛋白质的多样化物理化学性质。许多C2H2 ZFP具有剪接变体,进一步添加了基因家族的蛋白质多样性(附加文件1).
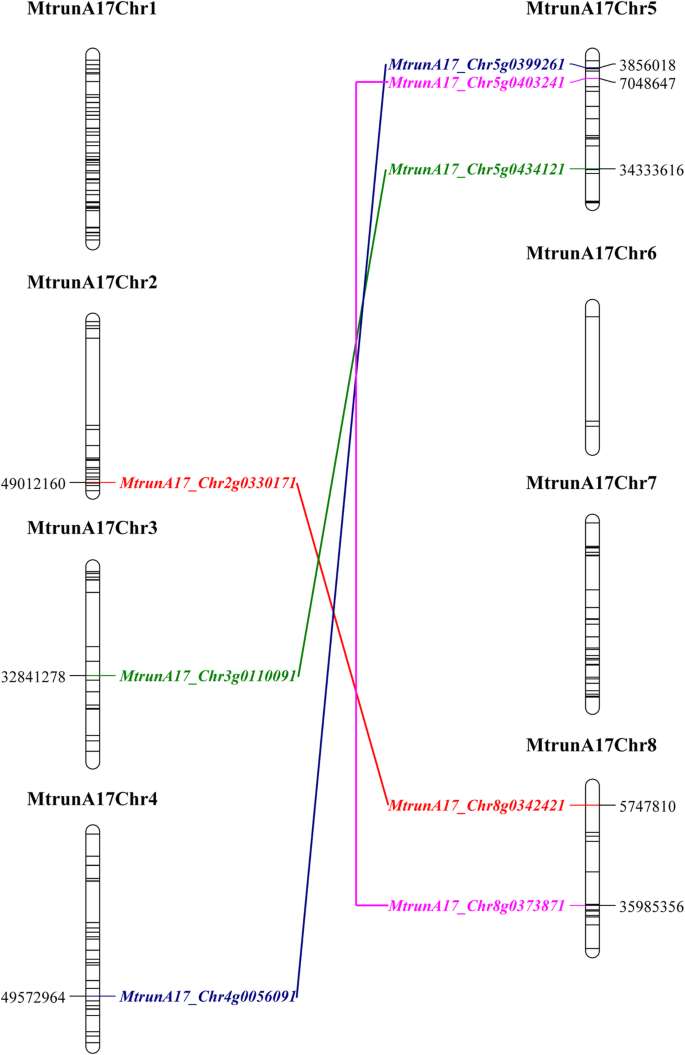
根据MTRUNA17R5.0和MT4.0v2基因组数据库,所有C2H2 ZFP都在染色体上找到。染色体1患有最多(59 C2H2 ZFP),代表全家族的约27%,其次是染色体7(36C2H2 ZFP)和染色体4(35C 2 H 2 ZFP)。染色体6和染色体8分别含有3和14 C2H2 ZFPS(图。1,附加文件4.).
在M. Truncatula.C2H2 ZFP基因家族,发现46个含有两到六C2H2 ZFP的局部重复组成(附加文件5.).例如,邻居基因MTRUNA17_CHR3G0086321 / MEDTR3G023750和MTRUNA17_CHR3G0086331 / MEDTR3G023760均编码C2H2型ZFPs;1号染色体14.30 M ~ 14.31 M的6.5 kb片段包含3个C2H2型ZFPs:mtruna17_chr1g0164501那mtruna17_chr1g0164511和MtrunA17_Chr1g0164521(附加文件5.).C2H2 ZFPs的局部重复显著增加了C2H2 ZFPs的总数M. Truncatula.,并提供了C2H2 ZFP的子官能化和多样化的潜力。
作为唇膏物种,M. Truncatula.经历了全基因组重复(WGD)5800万年前(MYR)[35那36那37].WGD产生基因对,其位于同时性基因组区域,并通过基因组水平分析检测[35].通过基因位置分析,MCScanX分析,并与以前的数据集进行比较[38[我们发现,来自WGD的4对C 2 H 2 ZFP(图。1,附加文件6.).同意复制,C2H2主题类型和布置在来自基因对的两个副病剂之间相同(附加文件6.).
获得C2H2 ZFPs在M. Truncatula.,我们试图执行序列对齐并重建C2H2 ZFP的系统发育树。然而,序列相似性分析表明,除了具有共享显着相似性的MTRuna17_CHR8R0156030 / METRUNA17_CHR3G0081131 / MEDTR3G011990之外,除了诸如MTRUNA17_CHR8R015603030 / METRUNA17_CHR3G0081131 / MEDTR3G011990,C2H2 ZFP系列的成员的顺序和结构是不小于15%的相似性检测到超过97%的基因,表明C2H2 ZFP的功能多样化(附加文件7.).
鉴定TFIIIA同源物M. Truncatula.基因组
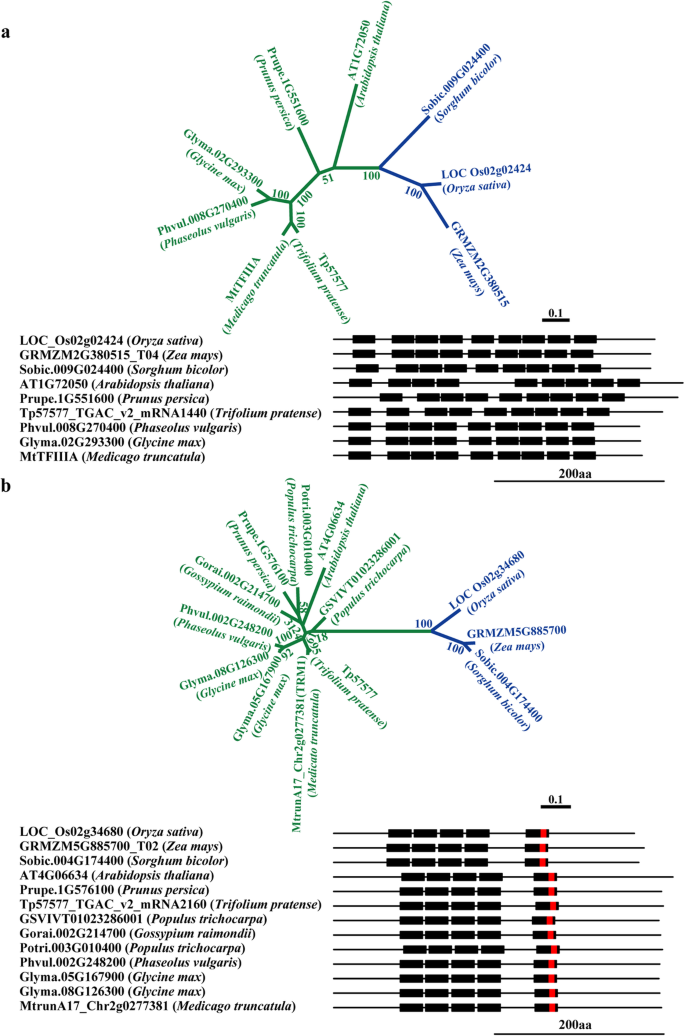
这拟南芥同族体的TFIIIA已被报道调节5S RNA基因的转录[16那34].这M. Truncatula.通过BLAST搜索找不到TFIIIA同源物拟南芥Tfiiia作为jemalong a17 mt4.0v2和mtruna17r5.0基因组序列的查询蛋白,可能是因为那个m . truncatula TFIIIA基因模型尚未在基因组序列中进行预测。BLAST搜索M. Truncatula.Affymetrix微阵列探针发现MTR.14440.1.S1_AT.,这对应于位于脚手架MWMB01000024上的基因组区域M. Truncatula.R108基因组(http://www.medicagohapmap.org/downloads/r108),非常相似attfiia.,建议TFIIIA同源物也存在M. Truncatula.R108基因组。在其他植物物种中进行BLAST搜索也发现了其他植物基因组中的TFIIIA同源物。为了证实Medicago R108基因组中TFIIIA的存在,我们对其编码区进行了扩增和测序M. Truncatula.TFIIIA同族体。所确定的M. Truncatula.TFIIIA同性恋与HMMER的TFIIIA同源物(HMMER)构建的隐马尔可夫模型(HMM)概况一致。http://hmmer.org/),确认鉴定TFIIIA同源物M. Truncatula..基因序列分析表明,类似于Arabidopsis TFIIIA,M. Truncatula.和其他植物TFIIIA同源物由九个C 2 H 2锌手指组成(图。2一个)。系统发育和序列相似性分析表明,所有植物TFIIIAs共有高序列同一性(图。2a),表明在调节5S RNA基因转录方面具有保守功能。在拟南芥TFIIIA中,2-4和5-9指串联排列,而C2H2锌指在n端相对分离。在其他植物TFIIIA同源物中也观察到这种模式,但第一和第二指链之间的连接序列稍短,尤其是在单子叶植物中(图1)。2一个)。然而,这种类似的主题布置模式表明植物TFIIIAs的保守进化祖先。
TRM1同源物的分析M. Truncatula.基因组
TRM1.首先是C4植物玉米的克隆和特征(Zea Mays.),其中TRM1结合到RBCS-M3基因,并发挥转录抑制作用[39].这TRM1.同源物也在拟南芥中鉴定[16].与TFIIIA一样,TRM1最初不是由BLAST搜索在M. Truncatula.Mt4.0v2基因组数据库以拟南芥或玉米TRM1蛋白序列作为查询对象。不过,一个BLAST搜索对M. Truncatula.MTRUNA17R5.0基因组数据库发现MTRUNA17_CHR2G0277381显示了拟南芥TRM1的高相似性。我们发现一个yefymetrix probesetMTR.25069.1.S1_S_AT.表现出高度相似于TRM1蛋白,表明TRM1也存在M. Truncatula.基因组。确认存在TRM1.在M. Truncatula.,我们扩增并测序M. truncatula trm1.同源物。像玉米trm1,M. Truncatula.TRM1同源物含有五个C2H2基序(图。2b).根据连接子长度,TRM1的C2H2基序可以分为两类(图5)。2b).前4个基序由29或30个连接残基组成串联重复序列。第5个基序与第4个基序相距65个残基。2b). HMM分析表明M. Truncatula.TRM1与HMMER用已知TRM1同源物构建的HMM轮廓一致。在其他植物物种中进行的序列搜索也发现了公认的TRM1同源物,这些同源物具有较高的序列同源性和保守的系统发育关系,表明TRM1在植物物种中具有保守的作用(图1)。2b)。有趣的是,来自单子叶种类的TRM1的N-末端比单萝卜物种的对应物略短。在水稻,玉米和高粱中,从第一个残留物到第一个C 2 H 2基序开始的N-末端的长度为61或63个氨基酸,而它在Dicot植物中的76至80个氨基酸范围(图。2b)。
令人惊讶的是,在第5个C2H2基序中发现了疑似EAR基序的LKLHLK序列M. Truncatula.在其他植物物种中的TRM1和TRM1同源物种,包括拟南芥,大豆,水稻,玉米和高粱(图。2b)。
C2H2主题M. Truncatula.
218 C2H2型ZFP识别M. Truncatula.基因组包含337个C2H2基序。我们一致M. Truncatula.C2H2主题并将其分为五个主要组,基于C2H2域内的保守核心序列(附加文件8.).
在71个基因的93个基序中发现q型C2H2s。q型C2H2的核心序列为QALGGH,其中H为C2H2基序的第一个组氨酸。这种类型的C2H2锌指已在以前的报道中被认为是植物特有的[1那12那15那16那25].除了核心序列之外,两个组织之间的三个残基还显示了某些Q型图案中的高保留。Regarding the residues between the two histidines, we further subclassified Q-type C2H2s into six subgroups, including 28 motifs with the consensus ‘QNA’ between the two histidines (Q1 subgroup), 22 motifs with ‘(K/R)AS’ (Q2 subgroup), 12 motifs with ‘MR(R/K)’ (Q3 subgroup), 10 motifs with ‘MN(I/V)’ (Q4 subgroup), 10 motifs with ‘KR(C/S)’ (Q5 subgroup), and 11 motifs without the consensus sequence between the two histidines (Q6 subgroup) (Additional file8.).
在一些C2H2基序中,Q型基序的核心序列略微或中度修改,例如“RAGGH”或“QGLGGH”。这些C2H2也已在其他植物物种中报道,本文被指定为Q改性(QM)类型。在里面M. Truncatula.基因组,57型QM型C2H2基序从45个ZFP鉴定出来。基于核心序列的模式和前一份报告中使用的方法[15],我们将qm类型的C2H2主题划分为9个子组(附加文件8.).
在19个ZFPs中,发现了一种新型保守的C2H2锌基序。从苯丙氨酸(F)开始,这种类型基序甚至比q型更加保守。在23个氨基酸中,16个在所有基因中是相同的(图。3.,附加文件8.).在拟南芥中,IDD10/JKD基因(AT5G03150)、BALDIBIS基因(AT3G45260)、IDD5基因(AT2G02070)和IDD7基因(AT1G55110)中发现了这一类型的c2h2。第一个IDD基因ID1是在玉米中克隆的,它控制着向生殖生长的过渡[40].植物中IDD家族由多个成员组成,在调节植物生长或代谢方面起着不同的作用。序列比较表明M. Truncatula.含有这种类型C2H2S的ZFPS与拟南芥IDDS相似。例如,含有这种类型的C2H2锌手指的MTRuna17_CHR8G0342421 / MEDTR8G017210显示了拟南芥IDD11(AT3G13810)的高相似性。由于在IDD基因中的普遍存在,这种类型的锌手指被命名为IDD型C2H2。虽然IDD基因尚未报告M. Truncatula.,IDD-型C2H2的鉴定证实了这一重要基因家族的存在和推定的保守功能M. Truncatula..
Z型图案代表显示高度保守的核心序列的C2H2,不属于Q,QM和IDD型C2H2。我们确定了25个zfps的41个C2H2主题,分为Z型(附加文件)8.).基于C2H2基序内的核心序列的模式,Z型C2H2S进一步分为八个亚组,每个子组中有2到15个单独的图案(附加文件8.).以前的报告将不包含任何保守核心序列的C2H2s归为c型[1那15那26].在这项工作中,我们确定了127个C型C2H2S。
C2H2 ZFPs的分类M. Truncatula.
所识别的C2H2 ZFPM. Truncatula.根据ZFPs中C2H2锌指数自然分为组。在218个C2H2 ZFPs基因中,151个基因具有单一的C2H2基序,占该家族的69%。单C2H2基序基因的优势丰度M. Truncatula.基因组与其他植物物种中的ZFP系列一致[16],强调了C2H2 ZFP家族在植物和动物基因组中的差异,其中C2H2基序基因很少被发现[7.].在具有多个C2H2S的67个ZFP中,38个基因有两个锌指状物,16种锌手指,9种锌手指,一个(TRM1)有五个锌手指,两只有六个,一(TFIIIA)有九个锌手指C2H2主题(附加文件9.).
含有一个C2H2基序的ZFPsM. Truncatula.
大多数C2H2 ZFPS发现M. Truncatula.基因组仅包含一个C2H2主题。共有218例ZFPS发现,151只有一个C2H2锌手指基序。耳动基序连接转录抑制器TPL / TPL样蛋白,因此能够实现编码基因的转录抑制函数。耳序通常在ZFP基因中发现。基于耳廓的存在,我们将151个单个C2H2 ZFP分类为三个亚组,即没有耳朵,ZFP,ZFP,一个耳朵和ZFP,具有多个耳朵。在151个单一C2H2 ZFP中,74含有耳朵,60个含有一只耳朵,17个包含多个耳朵图案(附加文件10).
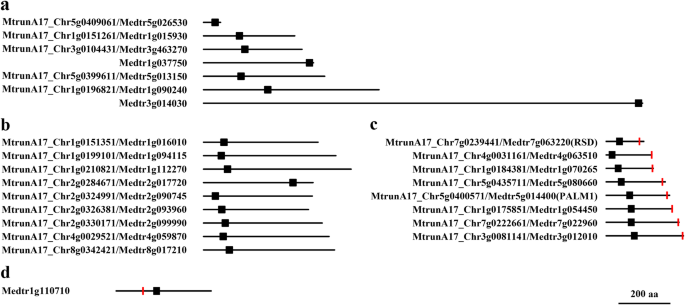
没有耳图案的74个单C2H2 ZFP长度变化,范围为69至1890个氨基酸(图。4.一个)。C2H2基序的分布也是可变的,从非常n-末端到C-末端的末端(图。4.一个)。这些功能突出了该子组的序列分流,并意味着成员之间的功能多样化。不幸的是,任何成员都没有表征到目前为止。为了更好地理解该亚组中基因的推定功能,我们在其他物种中进行了序列分析和同性恋搜索。所得基因注释表明,该亚组的功能显着多样化。一些成员是推定的锌指转录因子或C2H2样锌指蛋白,如MTRUNA17_CHR1G0151261 / MEDTR1G015930和MTRUNA17_CHR3G0104431 / MEDTR3G463270;一些成员作为含氦酶结构域的蛋白质被注释,例如MEDTR3G014030和MEDTR1G037750;一些人被预测为Polycomb蛋白的VEFS盒,例如MTRuna17_CHR1G0196821 / MEDTR1G090240和MTRUNA17_CHR5G0399611 / MEDTR5G013150(图。4.一个)。在该亚组中发现了十五型IDD型C2H2基序(图。4.b),其与拟南芥IDD基因高度相似,表明含有IDD型C2H2主题的成员M. Truncatula.可能对拟南芥同源物具有类似的功能。
MTRUNA17_CHR8G0355141 / MEDTR8G043980,带推导的746持续时间,高度相似于拟南芥锯齿状叶子(SE,AT2G27100)[41],建议它可能是M. Truncatula.SE的相同器官。拟南芥PCF11P-SIMILAR PROTEIN 4 (PCFS4, AT4G04885)属于C2H2 ZFPs基因家族,参与调控开花时间[42].这M. Truncatula.PCFS4同源物尚未鉴定。我们发现,该亚群中的一个成员MtrunA17_Chr6g0452881/Medtr6g011450与拟南芥PCFS4具有高度相似的C2H2基序排列,位于c端。这一结果提示MtrunA17_Chr6g0452881/Medtr6g011450可能是PCFS4的同源物。
60个含有单一EAR基序的C2H2 ZFPs中,有59个C2H2基序位于上游,而EAR基序位于下游。C2H2锌指靠近n端,而EAR位于c端远端,如报道的PALM1和RSD所示。这个C2H2 ZFPs亚群通常很短,有50个(83%)成员的长度小于350个氨基酸(图1)。4.C)。
除Palm1(MTRUNA17_CHR5G0400571 / MEDTR5G014400)和RSD(MTRUNA17_CHR7G0239441 / MEDTR7G063220)外,尚未表征该亚组中的所有基因。通过相似性分析,我们确定了几种非常相似的基因拟南芥表征C2H2 ZFPS。MTRUNA17_CHR1G0175851 / MEDTR1G054450在C-Terminus的N-末端和耳中携带一个C2H2,似乎是Arabidopsis ZFP8(AT2G41940),大豆ZFP基因Glyma.09G107400和水稻C2H2 ZFP LOC_OS08G36110的密切同源物。MTRUNA17_CHR5G0435711 / MEDTR5G080660和MTRUNA17_CHR1G0184381 / MEDTR1G070265非常类似于Arabidopsis兔耳(REB,AT5G06070)[43]和超人(Sup,AT3G23130)[19], 分别。始终如一地,所有这些同源物具有类似的基序结构,即位于可比位置的一个C2H2和一只耳朵(图。4.c,额外的文件10).
MEDTR1G110710是唯一具有位于C2H2锌手指上游的耳动图案的单个C2H2 ZFP(图。4.D,附加文件10).Medtr1g110710与拟南芥JAGGED (JAG, AT1G68480)高度相似,似乎是其同源物,而JAGGED在塑造侧面器官和促进叶片组织发育方面发挥着重要作用[44那45].序列分析还显示MEDTR1G110710与大豆Glyma.10G273800高度相似,水稻无限1(SL1,LOC_OS01G03840)和其他植物物种中的一些其他密切相关的基因,其与MEDTR1G110710共享类似的基因结构。SL1已被据报道是Jag的同源物[46].SL1不是调控叶片发育,而是控制花的发育,显示了同源基因的功能分化[46].连合在一起,Jag,Medtr1G110710和其他同源物可以代表C2H2型ZFP的特殊簇,具有C2H2锌手指上游的耳动图。
17个单基因含多个ear的C2H2 ZFPs,其中16个基因含两个ear, 1个基因含三个ear10).16个ZFPs包含两个ear,其中9个ZFPs与C2H2 motif位于两个ear之间,另外7个ZFPs与C2H2 motif位于ear的下游或上游(附加文件11).
具有多个C2H2主题的ZFPSM. Truncatula.
在里面M. Truncatula.基因组,67个ZFPS Harbored多个C2H2基序,包括含有两个C2H2的38个ZFPS,含有含有四个C 2 H 2 S,MTTRM1(五C 2 S),2个ZFPS的3个C 2 H 2 S,9 ZFPS,含有6个C 2 H 2 S和MTTFIIIA(九C 2 S)的3个ZFPS。具有多个C2H2S的ZFP已被广泛发现在其他植物物种中[1那15那16那27].基于图案之间的间隔长度,C2H2的组织可以分类为分散的分布式图案或串联阵列[16那27].利用该方法,我们将67个含有多个C2H2s的ZFPs分为2个亚组,其中15个含有串联C2H2s, 52个不含串联C2H2s。
15个C2H2基元串联阵列的ZFPs包括3个含有2个C2H2基元的ZFPs、2个含有3个C2H2基元的ZFPs、6个含有4个C2H2基元的ZFPs和2个含有6个C2H2基元的ZFPs,以及MtTRM1和MtTFIIIA。5.).数组中C2H2基序之间的间隔从0到11个残基不等。在MtrunA17_Chr8g0392331/Medtr8g106220中,两个C2H2之间没有连接残基,导致n端有两个连续的C2H2基序(图1)。5.).MTRUNA17_CHR7G0225252525251 / MEDTR7G029095和MTRUNA17_CHR4G0075341 / MEDTR4G132670分别组成的两个C2H2S之间的链接分别由5和10个残基组成(图。5.).MEDTR7G029090和MTRUNA17_CHR4G0075321 / MEDTR4G132610具有三C2H2S,显示了不同的阵列模式。在MTRUNA17_CHR4G0075321 / MEDTR4G132610中,连杆由10个残基和三个C2H2组成,形成连续串联阵列。相比之下,MEDTR7G029090中的第三C2H2与由第一和第二C2H2形成的阵列分离(图。5.).
在具有四个C2H2S的九个ZFP中,其中三个(MTRUNA17_CHR5G0403241 / MEDTR5G018850,MEDTR8G07849和MTRUNA17_CHR8G0373841 / MEDTR8G078700)具有相同的阵列模式。前三个C2H2S串联连接10或9个残基,并从阵列中分离第四C 2 H 2。值得注意的是,所有三种蛋白质都表现出与Arabidopsis Su(VAR)3-9相关蛋白5(SUVR5,AT2G23740)的相似之处。其中两个,MTRuna17_CHR1G0199511 / MEDTR1G094740和MTRUNA17_CHR3G0116441 / MEDTR3G075210分别与拟南芥早期开花6(ELF6,AT5G04240)和早期开花6(REF6,AT3G48430)的相似性高。尽管功能性分歧,但ELF6和REF6依次相似,它们均在C-Terminus的含有四个串联C2H2基序的jumonji级转录因子进行高度相似的jumonji级转录因子[47].类似于ELF6和REF6,MTRUNA17_CHR1G0199511 / MEDTR1G094740和MTRUNA17_CHR3G0116441 / MEDTR3G0116441 / MEDTR3G075210还有一系列位于C-末端的四个C2H2基序,包括降级的C2H2,其在ELF6和REF6中也已发现[16].MtrunA17_Chr1g0199511/Medtr1g094740和MtrunA17_Chr3g0116441/Medtr3g075210与最近报道的大豆Glyma.20G181000和Glyma.04G191900高度相似[27].
对于没有串联C2H2阵列的52个ZFP,C2H2锌指状物均分别定位。迄今为止,他们都没有表征。同源物搜索显示其中三个(MTRuna17_CHR1G0207451 / MEDTR1G106730,MTRUNA17_CHR1G0152661 / METTR1G018420和MTRUNA17_CHR3G0134711 / MEDTR3G102980)与拟南芥盐耐锌手指进行序列相似性(STZ / AT1G27730)[48),表明3种ZFPs可能介导的盐耐受性M. Truncatula..此外,来自本地串联重复的两个成员MtrunA17_Chr4g0049671/Medtr4g093260和MtrunA17_Chr4g0049701/Medtr4g093270与拟南芥HVA22家族成员表现出相似性,表明它们可能参与了HVA22家族的应激反应M. Truncatula..
表达式的分析M. Truncatula.乙炔ZFPs
的空间表达模式M. Truncatula.我们分析了C2H2 ZFPs的组织特异性表达数据集M. Truncatula..通过对C2H2 ZFPs和M. Truncatula.Affymetrix微阵列探针,我们发现,总共218个ZFPS的119具有相应的微阵列探针,使我们能够进行进一步的表达分析M. Truncatula.基因表达阿特拉斯(https://mtgea.noble.org/v3/) (图。6.A、b、c) [49那50].
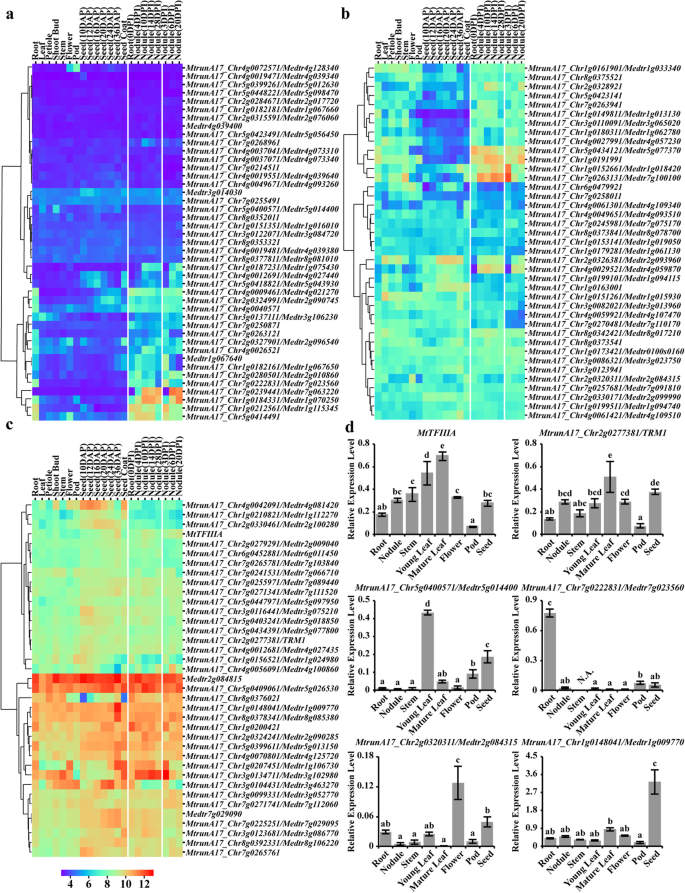
C2H2 ZFP一般表现出高度可变的空间表达模式,表明它们可能在调节组织开发方面发挥不同的作用M. Truncatula..11个基因,包括MtrunA17_Chr4g0019471 / Medtr4g039340那MtrunA17_Chr5g0399261 / Medtr5g012630 MtrunA17_Chr5g0448221 / Medtr5g098470 MtrunA17_Chr1g0182181 / Medtr1g067660, 和MTRUNA17_CHR5G0423491 / MEDTR5G056450,在所有组织中没有显着检测到,暗示它们可能在调节组织发育方面发挥着次要作用(图。6.一个)。相比之下,MEDTR2G084815.那MTRUNA17_CHR5G0409061 / MEDTR5G026530那MTRUNA17_CHR1G0148041 / MEDTR1G009770.和MTRUNA17_CHR8G0378341 / MEDTR8G085380在检查的所有组织中显示出高表达水平(图。6.C)。我们还检测到一些在某些组织中特异性表达的几种基因。例如,MtrunA17_Chr7g0222831 / Medtr7g023560除了根和结节之外的所有组织中表达了极低的表达,其中显着表达了(图。6.一个)。MtrunA17_Chr5g0418821 / Medtr5g043930仅在成熟的种子、种皮和结瘤中表达,提示它可能参与了种子的成熟和结瘤(图1)。6.a).与之前的报告一致,Palm1.(MTRUNA17_CHR5G04005571 / MEDTR5G014400)仅在叶,芽芽和种子中显示表达(图。6.一个)。
为了验证这些C2H2 ZFP的表达模式,我们使用QRT-PCR检查了25个基因的表达(图。6.D,图。7.和附加文件12).结果表明TFIIIA和TRM1.在所有组织中显示出高水平的表达,而其他基因如MTRUNA17_CHR5G0400571/Medtr5g014400 MtrunA17_Chr7g0222831 / Medtr7g023560 MtrunA17_Chr2g0320311 / Medtr2g084315而且,MTRUNA17_CHR1G0148041 / MEDTR1G009770.在某些组织中表达量较高(图。6.D,图。7.和附加文件12).尽管表达模式变化了选择基因,但它们显示了一致性M. Truncatula.基因表达图谱结果证实了已鉴定的C2H2 ZFPs的变异空间表达模式。
鉴定在Rhizobium symbioSomes中特异性表达的C 2 H 2 ZFPS
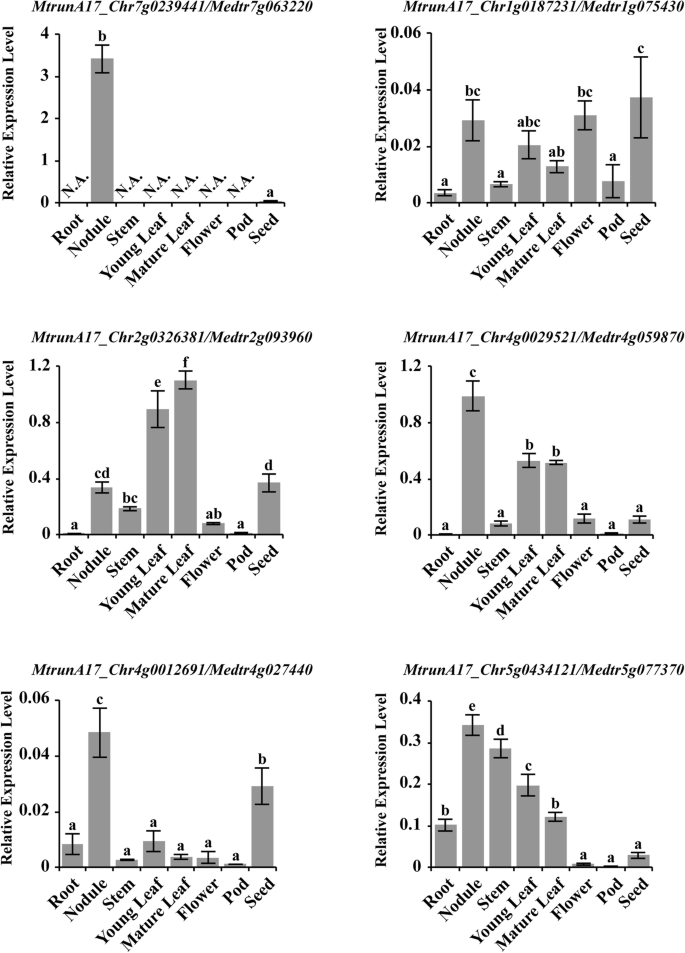
豆类共生结节固定约2亿吨氮,从而在可持续农业中发挥了至关重要的作用[51那52那53].识别专门表达的ZFPM. Truncatula.我们分析了共生结节的时间历程M. Truncatula.根瘤为C2H2 ZFPs。119个对应探针的ZFPs中,14个在结节中有明显的诱导,其中9个具有单一C2H2基序,2个具有2个C2H2基序,2个具有3个C2H2基序,1个具有4个C2H2基序。强烈支持前一份报告[28],标准偏差(MTRUNA17_CHR7G0239441 / MEDTR7G063220)ZFP是否显示结节中最强的诱导(图。6.这样的)。标准偏差那MTRUNA17_CHR1G0187231 / MEDTR1G075430在C-末端的N-末端和耳动基序中含有单个C2H2基序,在结节中也显示出显着的诱导(图。6.a). MtrunA17_Chr2g0326381/Medtr2g093960和MtrunA17_Chr4g0029521/Medtr4g059870均含有单一的C2H2基元,与拟南芥BIRD家族成员BALDIBIS (BIB)高度相似,且在结核中表达强烈(图。6.b)。Bib通过转录调节促进拟南芥根系的组织规范SHR.[54].由于结节发育涉及组织规范,MTRUNA17_CHR2G0326381 / MEDTR2G093960和MTRUNA17_CHR4G0029521 / MEDTR4G059870可能在结节发育中起重要作用。MtrunA17_Chr5g0434121 / Medtr5g077370在结节中表达,以及叶片,叶柄和芽芽如植物组织(图。6.b).有趣的是,在结节中诱导了4种ZFPs,分别是MtrunA17_Chr2g0326381/Medtr2g093960、MtrunA17_Chr4g0029521/Medtr4g059870,MtrunA17_Chr4g0012691 / Medtr4g027440, 和MtrunA17_Chr5g0418821 / Medtr5g043930,还显示出种子或种子涂层中的强烈表达,暗示它们可能调节一些结节和种子中的一些常见途径(图。6.a,b)。
为了验证C2H2基因的结节特异性表达模式,我们对6个基因进行了qRT-PCR检测(图。7.).标准偏差(MTRUNA17_CHR7G0239441 / MEDTR7G063220)在结节中显示出极定的表达,而在其他组织中没有检测到任何信号。其余的五个基因,MTRUNA17_CHR1G0187231 / MEDTR1G075430那MTRUNA17_CHR2G0326381 / MEDTR2G093960那MTRUNA17_CHR4G0029521 / MEDTR4G059870那MtrunA17_Chr4g0012691 / Medtr4g027440和MtrunA17_Chr5g0434121 / Medtr5g077370在结节中显示出高表达,尽管它们在其他组织中也显示出或多或少表达。然而,QRT-PCR检测的表达模式显示出与基因的表达结果良好的一致性。
讨论
在拟南芥、水稻和杨树等植物中,已经总结出C2H2锌基因家族。之前的研究表明,拟南芥基因组中有176个C2H2 ZFPs,水稻和杨树基因组中有189个C2H2 ZFPs [1那15那16那27].在重要的豆科植物大豆中,已鉴定出321种C2H2 ZFPs [27].本研究在典型豆科植物中鉴定了218个C2H2型锌指蛋白,含337个C2H2基元M. Truncatula..它们都属于凤蝶类,M. Truncatula.和大豆密切相关,共享WGD约为5800万年(MYR)以前[35那37那55].大豆接受了另一种WGD 13 Myr以前,导致古代多倍性基因组,其中近75%的基因具有重复的副本,解释为什么在大豆基因组中鉴定了更多C2H2 ZFP。在M. Truncatula.,在许多基因家族中据报道了基因的局部重复,例如热休克转录因子家族和F型箱基因家族[56那57那58].基因组测序数据已经证明了这一点M. Truncatula.具有很高的局部基因重复率,这有显着促进基因家族的扩展[35].这可能是我们检测到大量C2H2 ZFP的原因。作为证据,我们发现局部基因复制事件导致局部重复的C2H2 ZFPS。
我们将337个C2H2基元分为q型、IDD型、qm型、z型和c型5大类。q型C2H2s在植物中已被广泛发现,但这两种组氨酸之间的保守残基尚未被描述。在这项工作中,我们确定了某些保守的残基模式,例如q型C2H2锌指的两个组氨酸之间的共识“QNA”。尽管组氨酸之间的残基的功能尚不清楚,但它们很可能有助于q型C2H2基序的功能多样化。因此,q型C2H2基序根据两组氨酸残基之间的保守性进一步划分为亚组是合理的。Q-type的核心序列可以被改变,产生一个修正的Q-type (QM-type),其中包含57个成员M. Truncatula..我们发现IDD基因中的C2H2基元表现出独特的保护模式,与家族相区别,因此我们将其命名为IDD型C2H2指。z型C2H2指的是除胞嘧啶和组氨酸外的残基具有一定的保守模式的基序。由于对z型c2h2的功能研究不足,我们目前无法对z型c2h2内的亚群进行功能解释。
耳朵是转录镇压主题,使抑制作用能够对转录因子。我们还在单一C2H2基序ZFP中发现了耳动仪的宽,通常在ZFP的C末端,如Palm1 / IRG1和RSD所示。我们还发现了一个位于TRM1的第五C2H2主题中的推定耳图案M. Truncatula.和其他植物种类。TRM1被称为转录阻遏物。在TRM1同源物中识别推定的耳动图案非常有趣,尽管需要进一步的实验验证来解决这个推定的主题的作用。
首先确定了非洲爪蟾蜍光滑的, TFIIIA已在植物中被鉴定为植物中含C2H2锌指最多的ZFP [16那34那59那60].鉴于5S rRNA转录调控的重要作用,很可能M. Truncatula.哈勃tfiiia的同源物。最初,我们没有找到TFIIIA同源物M. Truncatula.基因组数据库。通过包括分析Affymetrix探针在内的综合方法,我们成功地鉴定出TFIIIA同源物M. Truncatula..此外,我们克隆并测序了编码序列M. Truncatula.TFIIIA同族体。C2H2基序的基因结构和排列在高等植物TFIIIA同源物中表现出高度的保守性,表明其在植物中的保守功能和进化历史。我们克隆并分析了TRM1M. Truncatula.,并从其他高等植物物种中鉴定出TRM1。TRM1最初克隆于玉米中,作为yy1样转录抑制因子,具有特定的抑制作用RBCS-M3在叶片细胞中。TRM1同源物的保守序列和图案布置M. Truncatula.和其他植物表明TRM1介导的抑制RBCS-M3可能在高等植物中高度保守。
虽然C2H2基序证明了高序列相似性,但C2H2 ZFP的整个蛋白质序列在整个家庭中显示出非常低的相似性。由于其结合DNA的能力,许多转录因子已经采用了C2H2基序,具有非常明显的功能来识别顺式元素。C2H2基序外的各种序列可以反映家庭中ZFP的功能多样化。为了支持这种观念,基因阿特拉斯分析和QRT-PCR显示的表达模式表明,ZFP表现出组织特异性表达模式的显着变化。
结论
总之,我们在模型豆类中识别了218个C2H2 ZFP,其中337 C2H2主题M. Truncatula..基于C2H2主题和耳奇图案的数量和排列,我们将这些ZFP分为不同的群体。使用基于相似性的分析,我们在鉴定的C2H2 ZFP基因家庭中发现了许多重要基因M. Truncatula..我们还分析了C2H2基序的序列,并确定了一种新型的C2H2基序,IDD型C 2 H 2。此外,我们识别并克隆了TFIIIA和TRM1同源物M. Truncatula.基因组。表达分析表明,经鉴定的C2H2 ZFPs可根据其在不同组织中的转录丰度分为三类。我们还发现并证实了一些C2H2 ZFP基因在某些组织中特异性表达,如花、种子和结节。我们的全基因组研究为C2H2 ZFPs在高等植物中的多样性和扩增提供了新的视角。
方法
植物材料
本研究所用种子采自华南农业大学草地科学系仓储中心。的种子M. Truncatula.(Jemalong A17和R108)在纸影上被摩擦,并在潮湿的滤纸上发芽。萌发后,植物在温室中生长以进行进一步的实验。
RNA提取和qRT-PCR
使用Trizol试剂(Invitrogen,15,596,026)和快速RNA分离试剂盒,从根,茎,未开封的幼叶,成熟的叶子,未开封的幼叶,成熟的叶子,花,未成熟的幼小叶,成熟叶,花,未成熟的豆荚,未成熟的种子和16天接种(DPI)结节(DPI)结节(Huayue yang Biotech, 0416–50) according to the manufacturers’ instructions. The reverse transcription was performed with TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix (Transgen, AT311–03) using 1.6 μg total RNA. The qRT-PCR was performed with ChamQ™ Universal SYBR qPCR Master Mix (Vazyme, Q711–02) on Bio-rad CFX Connect™ Real-Time System. Three biological replicates and three technical replicates of each biological replicate were used for qRT-PCR. Relative expression level of tested genes was normalized using the geometric mean ofMtActin(MtrunA17_Chr3g0129641 / Medtr3g095530)MtGAPDH(MTRUNA17CHR3G0122971/Medtr3g085850),mthelicase.(MTRUNA17CHR4G0077061./MEDTR4G134790.)三个基因[61那62那63].样本间统计学差异采用单因素方差分析(One-way ANOVA)和Tukey’s检验P.在SPSS 13.0软件中= 0.05 (https://www.ibm.com/analytics/spss-statistics-software.).QRT-PCR引物列于补充材料中(附加文件13).
C2H2 ZFPs的鉴定
只有112个M. Truncatula.C2H2 ZFP在植物转录因子数据库中(http://planttfdb.cbi.pku.edu.cn/index.php?sp=Mtr)和77 c2h2型zfpsM. Truncatula.基因表达阿特拉斯(https://mtgea.noble.org.).鉴于其他植物物种中的基因组大小和C2H2 ZFP基因数,我们认为应该有更多的C2H2 ZFPM. Truncatula.基因组。广泛识别C2H2 ZFPM. Truncatula.基因组,我们下载并分析了两个版本的M. Truncatula.基因组序列,Mtruna17r5.0(https://medicago.toulouse.inra.fr/MtrunA17r5.0-ANR)和mt4.0v2(http://www.medicagogenome.org.)[31那32那33].开发了一个python脚本(python 3.6)来搜索注释的蛋白质。在脚本中,正则表达式(https://docs.python.org/3/library/re.html)[64]“。{2} C. {2} C. {12} H. {3,5} H”或“。{2} C. {4} C. {12} H. {3,5} H”用于匹配C2H2主题。从该步骤中进一步扫描来自该步骤的候选C2H2 ZFP(https://prosite.expasy.org)和pfam(https://pfam.xfam.org.)的确认平台。通过确认的候选体被认为是C2H2型ZFPs。开发了一个自定义Python脚本来绘制基因中C2H2基序的基因结构和排列。
C2H2 ZFPS的剪接变体和物理化学性质分析
根据注释标记C2H2 ZFP的剪接变体M. Truncatula.mt4.0v2基因组数据库[32那33].利用ExPASy计算pI/Mw工具(http://web.expasy.org/compute_pi.).
鉴定TFIIA和TRM1同源物M. Truncatula.
识别M. Truncatula.TFIIIA和TRM1同源物,拟南芥TFIIIA和玉米TRM1蛋白序列被用作搜索的疑问M. Truncatula.在1E-15的截止E值下通过BLASK(MT4.0V2和MTRUNA17R5.0)蛋白质数据库(MT4.0V2和MTRUNA17R5.0)[65].不幸的是,爆炸计划没有返回TFIIIa的积极成果,但是MtrunA17_Chr2g0277381被发现是MTRUNA17R5.0蛋白质数据库而不是MT4.0v2的ATTRM1的同源物。接下来,我们使用了与TFIIIA的相同查询序列来搜索M. Truncatula.R108基因组序列数据库和M. Truncatula.Affymetrix probesets。TBLASTN结果表明,位于支架MWMB01000024上的一个区域与拟南芥TFIIIA具有很高的相似性。两个M. Truncatula.概念,MTR.14440.1.S1_AT.和MTR.25069.1.S1_S_AT.,分别与拟南芥TFIIIA和玉米TRM1具有很高的相似性。接下来,我们进一步交叉搜索M. Truncatula.R108基因组序列和探针。结果表明探测器MTR.14440.1.S1_AT.对应于支架MWMB01000024上与TFIIIA序列相似的片段,探针MTR.25069.1.S1_S_AT.对应于2号染色体上的位点(MtrunA17_Chr2g0277381)显示与玉米TRM1的序列相似性。根据上面的结果,我们注释了m . truncatula TFIIIA正轨。要从其他物种中识别TFIIIA和TRM1同源物,我们使用MTTFIIIA和MTTRM1蛋白序列作为查询和搜索植物血统数据库(https:////phytozome.jgi.doe.gov/pz/portal.html#).下载了与MTTFIIIA和MTTRM1高相似的基因进行序列和基序分析。我们还使用其他物种的TFIIIA和TRM1同源物,以构建嗯剖面并搜索蛋白质数据库M. Truncatula.由hmmer(http://hmmer.org/).结果证实鉴定出的MtTFIIIA和mttrm1是TFIIIA和TRM1的同源物。
cDNA克隆和测序
的互补M. Truncatula.R108或A17叶子用于克隆MtTFIIIA和MtTRM1, 分别。编码序列MtTFIIIA和MtTRM1通过标准PCR方案使用Q5®高保真DNA聚合酶(NEB#M0491)放大如下:98℃,10s,60℃,30℃,72℃,45℃,35秒。编码序列的PCR产物是Sanger测序。PCR引物列于补充材料中(附加文件13).
系统发育分析
TFIIIA,TRM1同源物或其他C2H2 ZFP的蛋白质序列由CLUSTALX程序排列(www.clustal.org.)[66].对于多种蛋白质序列比对,使用GONNET系列作为蛋白质重量Matirx,将间隙开口设定为10.00,将间隙延伸设定为0.20,延迟发散序列设定为30%。基于Dayhoff矩阵的最大似然方法用于重建Mega 7.0软件中的系统发育树[67].Bootstrap Repicate设置为1000。
主题分类和分析
为了对C2H2主题进行分类,使用CLUSTALX程序对齐主题序列(www.clustal.org.)[66].通过手动观察和与先前方法的比较[12那14],C2H2主题基于核心序列进行分类(附加文件8.).C2H2阵列被定义为C2H2,序列链的少于11个残基[15].
C2H2 ZFPs的分类和染色体定位M. Truncatula.
根据C2H2基序在蛋白质中的数量,自然将C2H2 ZFPs分为若干组。鉴定出的C2H2 ZFPs的染色体配位M. Truncatula.基因组注释文件。利用MapChart 2.32程序生成C2H2 ZFPs染色体定位图M. Truncatula.[68].
鉴定WGD的局部基因复制和基因对
我们使用先前报告的方法来识别局部基因重复M. Truncatula.[35].简而言之,位于100邻相邻基因模型内的C2H2基因被视为局部基因重复。McScanx程序用于识别WGD引起的C2H2基因对[38].简而言之,所有蛋白质M. Truncatula.被搜查了M. Truncatula.蛋白质数据库。利用MCScanX ' duplicate_gene_classifier '编码对结果进行进一步分析,鉴定WGD中的基因对。通过搜索之前发表的基因对数据集,进一步确认了已识别的基因对[35].
表达分析
首先搜索所有C2H2 ZFPSM. Truncatula.Affymetrix微阵列probesets。对应探针的ZFPs的组织水平表达数据从M. Truncatula.基因表达阿特拉斯(MTGEA,https://mtgea.noble.org/v3.)[48那49].开发了一种自定义Python脚本以构建ZFPS的LOG2变换表达式级别的热图。
可用性数据和材料
本研究中使用的数据集包含在附加表中。基因组DNA,cDNA和蛋白质序列MtTFIIIA和MtTRM1可以在登录号MT799184,MK830055,QJS95126,MT799185,MK830056和QJS95127中找到在NCBI Genbank。
缩写
- 乙炔ZFPs:
-
Cys(2)他(2)锌手指蛋白
- ZFPs:
-
锌指蛋白
- 耳朵:
-
乙烯反应元件结合因子相关的两亲性抑制基序
- QM-type:
-
修改Q型
- QRT-PCR:
-
定量RT-PCR
- WGD:
-
全基因组复制
- MYR:
-
百万年前
- 国际直拨电话:
-
Indeterminate-domain
参考
- 1.
杨树C2H2锌指家族转录因子的全基因组分析及其对非生物胁迫的响应(Populus Trichocarpa.).Plos一个。2015; 10(8):E0134753。
- 2.
锌指蛋白:经典的锌指蛋白出现在当代植物科学中。植物生理学杂志。1999;39(6):1053 - 1058。
- 3.
Bohm S, Frishman D, Mewes HW。酵母基因组中C2H2锌指基序的变化及锌指蛋白的分类。微生物学与微生物学杂志1997;25(12):2464-9。
- 4.
Chung Hr,Schafer U,Mackle H,Bohm S.基因组膨胀和含有含Zad C2H2锌 - 手指基因的基因组膨胀和聚类。Embo Rep。2002; 3(12):1158-62。
- 5.
Fedotova AA, Bonchuk AN, Mogila VA, Georgiev PG. C2H2锌指蛋白:最大但尚未开发的高级真核转录因子家族。Acta Nat。2017;9(2):47-58。
- 6.
李W,王继,孙Q,李波,余y,赵m等。玉米双B箱锌指蛋白的基因表达分析。Funct综合基因组学。2017; 17(6):653-66。
- 7.
Iuchi S.三类C2H2锌手指蛋白。细胞mol寿命。2001; 58(4):625-35。
- 8.
Pabo Co,Peisach E,Grant Ra。新型Cys2HIS2锌手指蛋白的设计与选择。annu rev biochem。2001; 70:313-40。
- 9.
Tsai Ry,Reed Rr。鉴定多Zn-Finger蛋白RoAZ的DNA识别序列和蛋白质相互作用域。Mol细胞Biol。1998年; 18(11):6447-56。
- 10。
Gamsjaeger R,Liew CK,Loughlin Fe,Crossley M,Mackay JP。粘性手指:锌手指作为蛋白质识别主题。趋势Biochem SCI。2007; 32(2):63-70。
- 11.
Lee BM, Xu J, Clarkson BK, Martinez-Yamout MA, Dyson HJ, Case DA等。转录因子IIIA锌指诱导5S RNA的匹配和“锁和钥匙”识别。中国生物医学工程学报,2006;
- 12.
CIFTCI-YILMAZ S,Mittlle R.植物的锌手指网络。细胞mol寿命。2008; 65(7):1150-60。
- 13.
Czarnocka W,Van der Kelen K,Willems P,Szechynska-Hebda M,Shahnejat-Bushris,Balazadeh S等人。病变模拟疾病1作为病症依赖性支架蛋白和转录调节剂的双重作用。植物细胞环境。2017; 40(11):2644-62。
- 14.
Miller J,McLachlan Ad,Klug A.来自Xenopus卵母细胞的蛋白质转录因子IIIa中的重复锌结合结构域。Embo J. 1985; 4(6):1609-14。
- 15.
Agarwal P,Arora R,Ray S,Singh Ak,Singh vp,Takatsuji H,等。水稻中C2H2锌 - 手指基因家族的基因组及其系统发育及表达分析。植物mol biol。2007; 65(4):467-85。
- 16.
锌指蛋白(C2H2)的保护、多样化和扩展拟南芥蒂利亚纳基因组。BMC基因组学。2004; 5(1):39。
- 17.
作者简介:Bowman JL, Sakai H, Jack T, Weigel D, Mayer U, Meyerowitz EM. SUPERMAN,拟南芥花同种异型基因的调节因子。发展。1992;114(3):599 - 615。
- 18.
Gaiser JC, Robinson-Beers K, gasasser CS。拟南芥超人基因介导胚珠外形的不对称生长。植物细胞。1995年; 7(3):333-45。
- 19.
Sakai H, Medrano LJ, Meyerowitz EM超人在维持拟南芥花螺纹边界。自然。1995年; 378(6553):199-203。
- 20。
崔LG,山爵士,石米,高JP,林HX。DCA1充当DST的转录共激活剂,有助于水稻中的干旱和耐盐性。Plos Genet。2015; 11(10):E1005617。
- 21。
黄晓燕,超达元,高建平,朱敏贞,石敏,林慧霞。一种未知的锌指蛋白,DST,通过气孔控制调控水稻的干旱和耐盐性。基因Dev。2009;23(15):1805 - 17所示。
- 22。
李士,赵湾,元D,段M,Qian Q,Tang L等。水稻锌指蛋白DST通过控制增强谷物生产Gn1a./OSCKX2.表达式。国家自然科学基金资助项目:国家自然科学基金资助项目。
- 23。
陈俊,玉吉,葛,王H,Berbel A,Liu Y等。通过Cys(2)他(2)模型豆类的锌指转录因子对解剖叶片形态的控制Medicago Truncatula..中国科学:地球科学;2010;
- 24。
摘要/ abstractMedicago Truncatula..植物信号表现。2010; 5(9):1134-7。
- 25。
Kiełbowicz-Matuk A.植物c2h2型锌指转录因子参与胁迫反应。植物科学。2012;185 - 186:78 - 85。
- 26。
法拉吉S,Rasouli Sh,Kazemitabar Sk。基因组探索杜兰姆小麦C2H2锌手指家庭(Triticum Turgidum SSP..杜伦姆):洞察生物过程中的作用,尤其是压力反应。生物素线。2018; 31(6):1019-42。
- 27。
袁,李X,李河,王l,张c,chen l等。大豆C2H2锌指蛋白的基因组鉴定与分类及豆科植物分解中的表达分析。前微生物。2018; 9:126。
- 28。
Sinharoy S,Torres-Jerez I,Bandyopadhyay K,Kereszt A,Pislariu CI,Nakashima J,等。Symmiosom分化的C2H2转录因子调节因子抑制分泌途径基因的转录Vamp721a并促进Symmiosome发展Medicago Truncatula..植物细胞。2013;25(9):3584 - 601。
- 29。
Uppalapati SR,Ishiga Y,Doraiswamy V,Bedair M,Mittal S,Chen J,等。损失缺陷叶片侵占蜡Medicago Truncatula Irg1./Palm1.突变体导致炭疽病孢子瘤和非黑锈病病原菌的孢子分化降低。植物细胞。2012; 24(1):353-70。
- 30.
Peng J,Berbel A,Madueno F,Chen R. Auxin响应因子3通过直接压制调节复合叶图案像棕榈酸酯的pentafoliata1表达式Medicago Truncatula..植物科学与技术,2017;
- 31。
Pecrix Y,Staton Se,Sallet E,Lelandais-Briere C,Moreau S,Carrere S等人。全基因组景观Medicago Truncatula.共生基因。Nat植物。2018; 4(12):1017-25。
- 32。
Tang HB,Krishnakumar V,Bidwell S,Rosen B,Chan An,Zhou Sg等人。用于模型豆类的改进的基因组释放(版本MT4.0)Medicago Truncatula..BMC基因组学。2014;15:1。
- 33。
Krishnakumar V,Kim M,Rosen BD,Karamycheva S,Bidwell SL,Tang HB等。MTGD:Medicago Truncatula.基因组数据库。植物生理学杂志。2015;56:1。
- 34。
Mathieu O,Yukawa Y,Prieto JL,Vaillant I,Sugiura M,Tourmente S.转录因子IIIA和核糖体蛋白L5的鉴定和表征拟南芥蒂利亚纳.核酸RES。2003; 31(9):2424-33。
- 35。
Young ND, Debelle F, Oldroyd GE, Geurts R, Cannon SB, Udvardi MK等。苜蓿基因组提供了深入了解根瘤菌共生的进化。大自然。2011;480(7378):520 - 4。
- 36.
Pfeil,Schlueter Ja,Shoemaker Rc,Doyle JJ。将古聚倍性与分类差异有关:使用39个基因家族的豆类植物发育分析。系统中的生物学。2005; 54(3):441-54。
- 37.
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W,等。古多倍体大豆基因组序列。自然。2010;463(7278):178 - 83。
- 38.
王y,唐,debarry jd,tan x,li j,wang x等。McScanx:一种用于检测和进化分析的工具包,对基因同步和共线性分析。核酸RES。2012; 40(7):E49。
- 39.
Xu T,Purcell M,Zucchi P,Helentjaris T,Bogorad L. TRM1,A YY1样抑制器RBCS-M3在玉米培养基细胞中的表达。Proc Natl Acad SCI U S A. 2001; 98(5):2295-300。
- 40.
Colasanti J ., Yuan Z ., Sundaresan V.这一不确定基因编码一个锌指蛋白,调控玉米向开花过渡所需的叶片信号。细胞。1998;93(4):593 - 603。
- 41.
Clarke JH, Tack D, Findlay K, Van Montagu M, Van Lijsebettens M. SERRATE位点控制拟南芥早期幼叶的形成和相长。植物j . 1999; 20(4): 493 - 501。
- 42.
邢D,赵H,徐克,李QQ。拟南芥PCFS4,酵母多腺苷酸化因子PCF11p的同源物,调节FCA替代处理并促进开花时间。工厂J. 2008; 54(5):899-910。
- 43.
克里泽克BA,刘易斯MW,弗莱彻JC。兔耳朵是一个第二轮压缩机钦am这在拟南芥花中保持空间边界。工厂J. 2006; 45(3):369-83。
- 44。
Ohno CK,Reddy GV,Heisler Mg,Meyerowitz Em。拟南芥锯齿状的基因编码促进叶片组织发育的锌指蛋白质。发展。2004; 131(5):1111-22。
- 45。
Dinneny JR,Yadegari R,Fischer RL,Yanofsky MF,Weigel D.的作用锯齿状的在整形横向器官。发展。2004; 131(5):1101-10。
- 46。
肖辉,唐杰,李勇,王伟,李旭,金玲,等。STAMENLESS 1编码单一C2H2锌手指蛋白,调节水稻中的花器官身份。工厂J. 2009; 59(5):789-801。
- 47。
Noh B,Lee Sh,Kim Hj,Yi G,Shin Ea,Lee M等。一对同源Jumonji /锌 - 手指级转录因子蛋白在拟南芥开花时间调节中的发散作用。植物细胞。2004; 16(10):2601-13。
- 48。
谢y,毛,赖D,张w,沉w.h(2)通过操纵Zat10 / 12介导的抗氧化防御和控制钠排除来增强拟南芥耐盐性。Plos一个。2012; 7(11):E49800。
- 49。
bendito VA, Torres-Jerez I, Murray JD, Andriankaja A, Allen S, Kakar K等。模型豆科植物基因表达图谱Medicago Truncatula..工厂J. 2008; 55(3):504-13。
- 50。
何静,王敏,赵px,唐勇,等。这Medicago Truncatula.基因表达式ATLAS Web服务器。BMC生物素。2009; 10:441。
- 51。
格雷厄姆pH,Vance Cp。豆类:重要性和约束更大使用。植物理性。2003; 131(3):872-7。
- 52。
主要研究方向为生物信息学、生物信息学、生物信息学和生物信息学。固氮作物豆科对农业系统生产力的贡献。共生关系。2009年,48(1 - 3):1。
- 53。
Biswas B,Gresshoff PM。共生氮固定在生物燃料可持续生产中的作用。int j mol sci。2014; 15(5):7380-97。
- 54。
Long Y, Smet W, Cruz-Ramirez A, Castelijns B, de Jonge W, Mahonen AP等。拟南芥BIRD锌指蛋白通过限制细胞命运调控因子SHORT-ROOT和促进命运规范共同稳定组织边界。植物细胞。2015;27(4):1185 - 99。
- 55。
Lavin M,Herendeen PS,Wojciechowski MF。Leguminosae的进化率分析暗示了在第三大学期间的快速多样化。系统中的生物学。2005; 54(4):575-94。
- 56。
关键词:热激转录因子,热激转录因子,全基因组分析Populus Trichocarpa.和Medicago Truncatula..MOL BIOL REP。2012; 39(2):1877-86。
- 57。
Trujillo di,Silverstein Ka,Young Nd。LEED的基因组特征.. PEEDS,一种基因家族Medicago谱系.G3(贝塞斯达)。2014; 4(10): 2003 - 12所示。
- 58。
Bellieny-Rabelo D,Oliveira Ae,Venancio TM。全基因组和串联重复对豆类F箱家族的扩展和功能多样化的影响(Fabaceae)。Plos一个。2013; 8(2):E55127。
- 59。
Wyszko E,Radlowski M,Bartkowiak S,Barciszewska MZ。玉米TF IIIA - 来自单子圈子的第一个转录因子IIIa。纯化和性质。Acta Biochim Pol。1997年; 44(3):579-89。
- 60。
关键词:高等植物,转录因子,IIIA,分离纯化中国生物医学工程学报。1997;249(1):107-12。
- 61。
acta ecologica sinica, 2011, 38(5): 689 - 693 .葛玲,彭杰,陈锐Phantastica.在Medicago Truncatula..植物理性。2014; 164(1):216-28。
- 62.
acta photonica sinica, 2011, 38(5): 732 - 736 .葛霖,于军,王浩,Luth D,白刚,王凯,等。通过操纵提高种子的大小和质量大SEEDS1在豆类物种中。Proc Natl Acad Sci U S A. 2016; 113(44):12414-9。
- 63.
Satge C,Moreau S,Sallet E,Lefort G,Aurac Mc,Rembliere C,等。DNA甲基化的重新编程对于结节发育至关重要Medicago Truncatula..Nat植物。2016; 2(11):16166。
- 64.
掌握正则表达式。O ' reilly media, Inc .);2006.
- 65.
Altschul SF,Madden TL,Schaffer AA,张J,张Z,米勒W等人。Papped Blast和Psi-Blast:新一代蛋白质数据库搜索程序。核酸RES。1997年; 25(17):3389-402。
- 66.
Larkin Ma,Blackshield G,Brown NP,Chenna R,McGettigan Pa,McWilliam H,等。Clustal W和Clustal X 2.0版。生物信息学。2007; 23(21):2947-8。
- 67.
Kumar S, Stecher G, Tamura K. MEGA7:针对更大数据集的分子进化遗传学分析7.0版。中国生物医学工程学报。2016;33(7):1870-4。
- 68。
vooorres重新。MapChart:用于链接地图和QTL的图形演示软件。j hered。2002; 93(1):77-8。
致谢
不适用。
资金
这项工作由中国国家科学基金(NSFC,项目3171345)和南中国农业大学提供给GE的实验室的启动基金。融资机构没有参与研究的设计;收集,分析,数据诠释和写作稿件。
作者信息
隶属关系
贡献
ZJ和LG构思,设计,并进行了研究。LW,HD,YW,WW,JL,JH和WH在研究中贡献并修订了稿件。ZJ和LG分析数据。LG写了稿件。所有作者均阅读并批准了手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者们宣称他们没有相互竞争的利益。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1。
乙炔ZFPs在Medicago Truncatula..每个C2H2 ZFPs的基因ID、位置、探针、理化性质、剪接变异、同源位点、基序和蛋白序列。
附加文件2。
C2H2 ZFPS的蛋白质序列。
附加文件3。
C2H2 ZFPS的C2H2基序序列。
附加文件4。
C2H2 ZFPs在染色体上的分布M.Truncatula..
附加文件5。
C2H2 ZFPs基因的局部复制M.Truncatula..
附加文件6。
C2H2 ZFP基因对源于全基因组复制M.Truncatula..
附加文件7。
C2H2 ZFPS配对序列相似性分析。
附加文件8。
C2H2主题分类M.Truncatula..
附加文件9。
C2H2 ZFPs的分类M.Truncatula..
额外的文件10。
在C2H2 ZFP中用一个锌手指耳廓。'N.A.'表示,该C2H2 ZFP不含耳动。
额外的文件11。
具有代表性的C2H2 ZFPs具有一个C2H2 motif和两个EAR motifM. Truncatula..黑线代表蛋白质序列。黑色和红色矩形分别表示C2H2和耳奇图案。
额外的文件12。
QRT-PCR中十五C2H2 ZFP的组织特异性表达分析M. Truncatula..13个选择的C2H2基因的QRT-PCR结果。显示的是三种生物重复的平均值±标准偏差,以及每个生物复制的三种技术复制。通过三个内源性对照基因的几何平均值标准化测试基因的相对表达水平。'N.A.'表示未定例的表达。栏上方的小写字母表示显着差异(P.< 0.05),样本间采用方差分析(ANOVA)和Tukey’s检验.
额外的文件13。
引物列表。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
焦忠,王磊,杜洪。等等。锌指C2H2基因家族的全基因组研究Medicago Truncatula..BMC植物杂志20,401(2020)。https://doi.org/10.1186/s12870-020-02619-6
已收到:
公认:
发表:
关键词
- 乙炔
- 锌指
- 局部基因重复
- 基因家族
- 耳朵的主题
- 表达