摘要
背景
矿质营养素在生物系统的生化和生理功能中起着至关重要的作用。通过遗传改良提高种子矿物质含量被认为是满足饲料需求的最有希望和最具成本效益的方法。本研究的总体目标是对137个大豆品系的种子进行矿物质含量(Ca, K, P和S)的GWAS,这些品系代表了加拿大种植的早熟大豆(成熟度组000-II)的多样性。
结果
137个大豆品系在5种环境中生长,并使用便携式x射线荧光光谱仪测量种子矿物质含量。关联分析使用三种统计模型和从测序基因分型和全基因组测序组合数据集中获得的一组220万个snp进行。在使用的三种统计模型中(在两种环境中),至少有两种模型鉴定出8个与Ca、K、P和S含量显著相关的qtl,每个qtl对表型变异的贡献率从17%到31%不等。这8个qtl中有7个在其他3个环境中具有很强的再现性。总共鉴定出三个候选基因参与这些矿物元素的运输和同化。
结论
很少有GWAS研究鉴定与大豆种子矿质元素含量相关的qtl。此外,本研究鉴定的qtl和候选基因将为大豆营养品质的遗传改良提供有益的标记辅助选择。此外,本研究还提供了在加拿大大豆种质中遇到的表型变异范围的详细信息。
背景
大豆被广泛用于食品、饲料和工业用途,使其成为最通用的粮食作物之一。事实上,大豆是蛋白质、油脂和碳水化合物的重要来源,也是其他有益营养物质的重要来源,例如矿质元素,这些元素会影响油脂和蛋白质部分的最终用途特性,以及用于种植后续作物的种子的品质特性[1]。植物获得矿物质营养是一个非常动态和复杂的过程,受生物和非生物因素及其相互作用的影响[2]。在农业方面,据报道,缺乏必需元素可导致产量损失、疾病易感性增加、代谢受损、正常发育中断和种子质量差[3.]。为此,了解矿物元素在各种环境条件下的吸收、调节、运输和储存对于解读植物与其环境之间的复杂关系至关重要。因此,种子基因组谱是将植物的遗传特征与其对环境扰动的反应相匹配的有力工具[4]。
通过基因改良提高种子矿质营养素含量被认为是确保满足消费者饮食需求的最有希望和最具成本效益的方法。由于任何性状的育种都依赖于表型变异的存在,这就需要鉴定出具有有用的谷物矿物遗传变异的品种,并了解这些种子性状的遗传结构[5]。
自从植物营养的概念被建立以来,人们一直在努力开发定量测量生物体元素组成的方法和工具。6]。与火焰原子吸收光谱(FAAS)等湿化学方法相比,能量色散x射线荧光(ED-XRF)等光谱方法操作更容易,而且是一种非破坏性的工具,因此越来越受到人们的青睐[7]。近年来,ED-XRF已成功地用于测定大豆中Ca、K、P和S的浓度[j]。8,9],在可可[7]和豌豆种子[10]。在这些研究中,通过光谱学获得的测量结果与以前使用更常见但昂贵的分析方法进行的研究一致。此外,这些研究表明,植物中矿物质元素的含量可以跨越很大的范围,而这种含量是由遗传和环境因素共同决定的[2,4,11,12,13]。
数量有限的研究旨在确定控制种子中矿物元素积累的遗传结构,并得出结论认为这些性状很可能由许多基因控制[2,14]。Zhang等。[15[j]利用148个SSR标记和178个F / s标记,报道了4个与大豆种子钙含量相关的qtl(分别位于第7、8和20染色体上)2:3和157 F2:4行。King等。[13]利用916个SSR标记和92个F标记,报道了3个与磷含量相关的qtl(分别位于第7、12和17染色体上)2:4行。最近,Ramamurthy等人。[2]利用1536个单核苷酸多态性(SNP)标记和288个大豆重组自交系(RILs),鉴定出7个与Ca、K和S含量相关的qtl(位于4、6、15、16和18号染色体上)。使用GWAS方法,Ziegler等。[4]利用美国农业部大豆种质资源收集的1653份大豆材料和36489个snp,鉴定出大豆种子中与Ca、K、P、S含量相关的9个qtl(位于染色体1、2、5、9、10和13上)。最后,Dhanapal等。[16]报道了与大豆茎部Ca、K、P和S含量相关的20条染色体上的65个qtl,使用了31748个snp和104个大豆基因型。总的来说,在这些研究中发现的qtl几乎没有重叠。这可能是由于大豆种子中矿质元素含量的遗传决定因素在不同的种质间不同,或者一些研究受到基因组覆盖不足的影响,未能检测到共享的qtl。
在控制大豆种子矿质元素积累的qtl鉴定不完整且经常不一致的背景下[4],特别是在早熟大豆(MG000-II)中,我们试图表征一组137个加拿大短季大豆品种的表型多样性,并鉴定该组种质中控制Ca, K, P和S含量的qtl。利用大量SNP标记和三种分析方法(CMLM、MLMM和FarmCPU),我们共鉴定出32个控制这四个重要元素积累的qtl,其中8个qtl通过至少两种方法共同鉴定。相信本研究结果将为今后大豆种子品质和营养成分的遗传改良研究提供新的思路。
结果
湿化学与能量色散x射线荧光法的相关性
为了验证我们选择的分析方法(能量色散x射线荧光,ED-XRF),用ED-XRF和火焰原子吸收光谱(FAAS)对30个样品进行了Ca和K的分析,并用分光光度法对P含量进行了分析。如图3所示。1,两种方法的相关系数(r)均为正且极显著(P< 0.001),范围为0.91 (Ca) ~ 0.94 (P)。结果表明,ED-XDF方法适用于大豆种子中Ca、P、K含量的定量测定。
表型变异及性状间的相关性
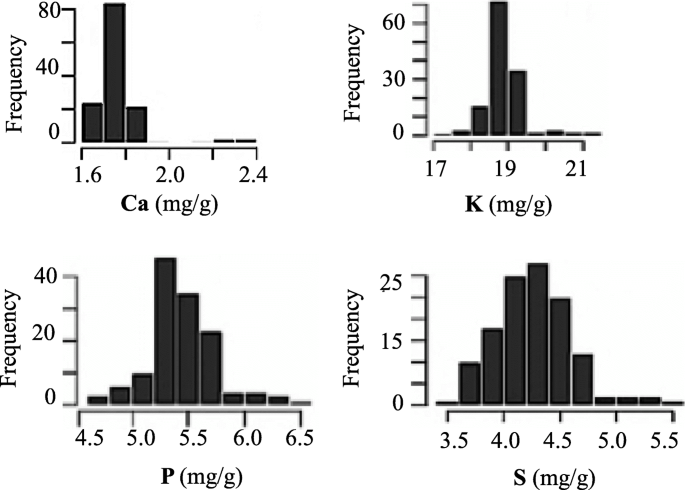
采用ED-XRF装置测定了2013年2个试验点(2个重复/试验点)137个大豆品系上Ca、K、P和S的浓度。频率分布表现为近似正态分布,并且在数量上似乎是遗传的(图2)。2)。如表所示1在干重基础上,4种元素的种子矿物质含量变化范围为:Ca 1.6 ~ 2.4 mg/g, K 17 ~ 21 mg/g, P 4.5 ~ 6.5 mg/g, S 3.5 ~ 5.5 mg/g。在所有137个品系中,Ca、K、P和S的平均值分别为1.8、18.7、5.3和4.3 mg/g。2个基因型平均值之间的最不显著差异(LSD)为Ca含量0.03 mg/g, K含量0.44 mg/g, P含量0.28 mg/g, S含量0.09 mg/g。广义遗传率在81% (K) ~ 99% (S)之间,具有较大的表型变异和较高的遗传率,表明这些性状和关联面板可以很好地揭示这些性状的遗传结构。
如表所示2,方差分析显示,基因型和环境都有极显著的影响(P≤0.001)对除Ca外的所有性状的表型变异均有显著影响,其中基因型效应是变异的唯一显著来源。在任何性状中均未观察到显著的x基因型环境相互作用。观察到的表型值显著(p< 0.001)在两个试验点之间相关,相关性范围在0.75到0.98之间。不同矿物质中的种子含量也被证明是相关的(表S1,粗体)。这些两两比较均有统计学意义(p< 0.05), K与S的相关性最高(r2= 0.67,p< 0.001)以及P和K之间(r2= 0.65,p< 0.001)。
基因分型和SNP呼叫
关联小组的细胞系最初通过GBS方法进行基因分型,共产生56 K高质量snp。在第二步中,使用包含4.3万个snp的参考面板对原始gbs衍生snp集进行缺失位点插入。去除InDels后,MAF < 0.05,杂合度> 0.1的标记,共保留2.18 M个SNP,平均标记密度为每435个碱基1个SNP。这些2.18 M snp在大豆20条染色体上的物理分布如图所示。S1。由此获得的基因型数据随后用于表征该面板内的群体结构并寻找标记-性状关联。
人口结构
这137个加拿大大豆核心系的群体结构最初是通过fastSTRUCTURE和亚群数(k)为7(图。3)。此外,从图中可以看出。3b和c系统发育树和基于pca的种群结构分析结果与fastSTRUCTURE分析结果一致。总之,这些结果表明k= 7可以很好地评价种群结构,采用相应的Q矩阵进行GWAS。
大豆种子矿质元素含量的全基因组关联扫描
为了发现导致表型变异的染色体区域,我们使用了三种分析工具来测量标记-性状关联:FarmCPU、CMLM和MLMM。如分位数-分位数(QQ)图所示(图2)。S2),三个模型都成功地限制了观察到的混淆效应p -值只偏离对角线(预期)p-值)在最极端的值(超过3E-03的几乎所有性状)。
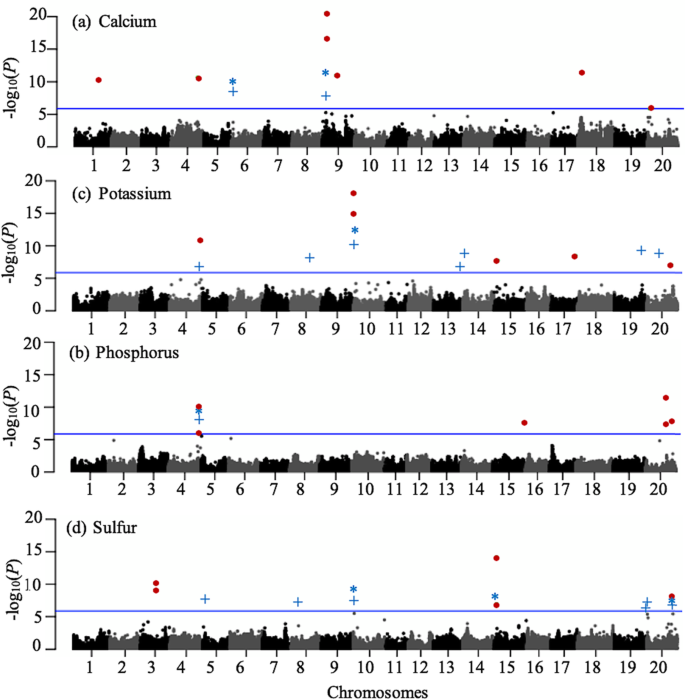
这些关联分析的结果显示为图中FarmCPU、CMLM和MLMM的曼哈顿图。4。根据错误发现率阈值(蓝色水平线,FDR≤0.05),我们检测到32个qtl,其中7个与Ca含量相关,10个与K含量相关,5个与P含量相关,10个与S含量相关(表S2)。有趣的是,有一个共同的QTL对K (K_#1)和P (P_#1)都有贡献。的不正确p -Ca含量为1.35E-06 ~ 2.84E-21, K含量为1.89E-05 ~ 8.05E-19, P含量为1.17E-06 ~ 3.61E-12, S含量为1.75E-05 ~ 6.63E-15。
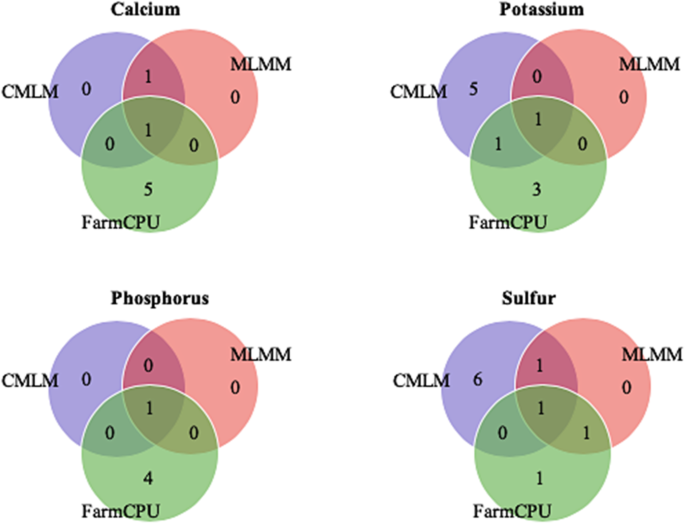
在这32个qtl中,共有8个qtl被至少两个模型共同识别(图2)。5),这8个稳健qtl的特征总结于表中3.。表型变异部分解释(R2), Ca为20% ~ 21%,K为17% ~ 31%,P为22%,S为18% ~ 23%。Ca、K、P和S的等位基因效应大小分别为0.06 ~ 0.07 mg/g、0.30 ~ 0.57 mg/g、0.30 mg/g和0.15 ~ 0.46 mg/g。遗传变异(加性)解释为狭义遗传力(h2Ca为41%,K为82%,P为78%,S为93%。
八个共鉴定的QTL在三种环境中的验证
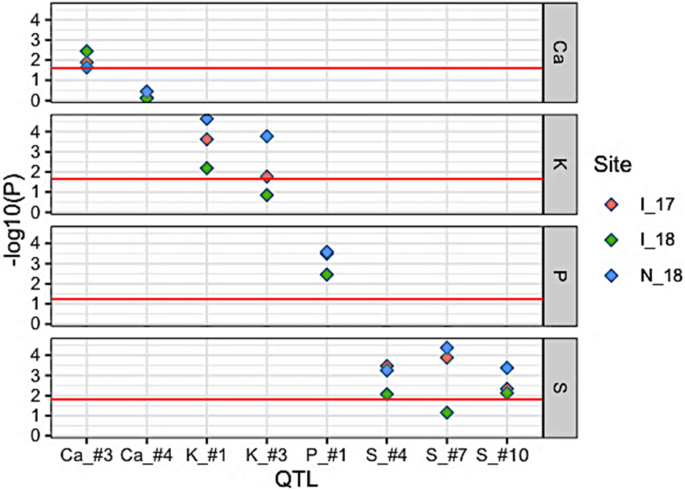
为了验证至少两个模型检测到的8个qtl中的每个qtl的稳定性,从另外三个试验中获得数据。总的来说,在三个新环境中,七个qtl在至少两个环境中得到了验证(图2)。S3和表S3)。只有Ca的QTL#4无法在三种新环境中的任何一种中验证。I_18环境的验证率最低,在该环境中成功检测到5个qtl(图2)。6)。在24个可能的qtl -环境组合(8个qtl × 3个环境组合)中,有18个在对照SNP峰值的株系平均表型之间存在显著差异。这些结果表明,所鉴定的qtl在广泛的环境中具有鲁棒性。
改进共鉴定QTL的GWA扫描
为了更深入地探索这些健壮qtl的变异,我们从2.2万个snp的大目录中提取了七个最健壮qtl周围的单倍型区域内的所有snp。将这些数据与修剪后的数据集(243 K)合并,再次使用三个模型进行GWAS。在这7个例子中的6个,观察到更强的关联信号,前一个和新的峰值SNP之间的物理距离在1到311 kb之间(表S4),但总是位于同一单倍型块内。
稳健QTL区域内候选基因的预测
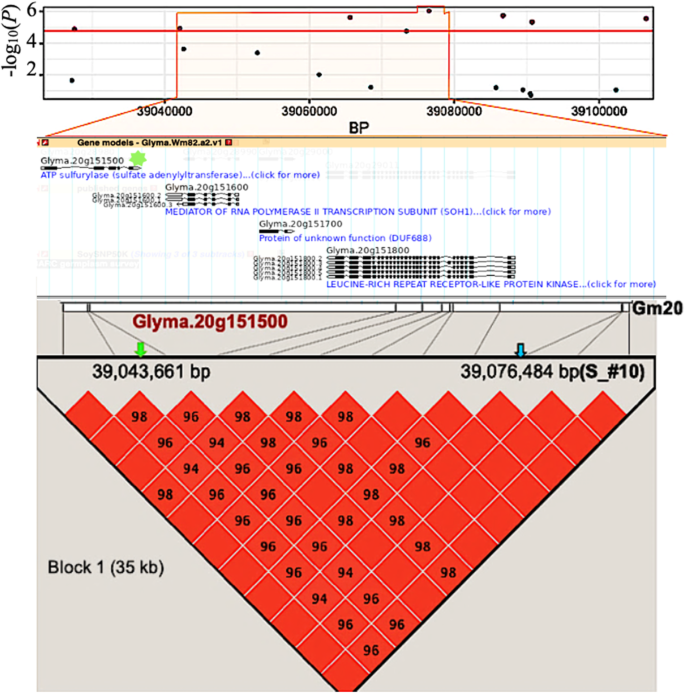
基于GWAS结果,我们研究了大豆基因组中标注的基因,以便从与每个性状显著相关的位点中鉴定出可能的候选基因。为了建立候选基因列表,我们只关注那些位于由最左和最右侧标记所划分的区域内的基因,这些标记处于完美LD (D ' = 1),具有上述七个qtl的峰值SNP。这些基因组区域(大小在32到360 kb之间)是从Wm82.a2中提取的。我们检测了v1和这些区域内基因的GO注释(表1)4)。这种方法的一个例子如图2所示。7。每个区域中(全部或部分)存在的基因数量在4到43个之间,这些基因的完整列表及其注释见表S5。
20号染色体单倍型区QTL S_#10候选基因的鉴定。上图:Gm20在约80kb区间(39,027-39,106 Kb)内的标记-性状关联。中间图:在35kb区域中存在的四个基因模型的位置和方向,该区域由最左(Gm20: 39,042,071)和最右(Gm20: 39,076,880)标记定义,这些标记与峰值SNP (Gm20:39,076,484)处于完美LD中。最有可能的候选基因(Glyma.20G151500,硫酸盐同化)用绿色星号突出显示。下图:在所定义的感兴趣的基因组区域内的标记之间的成对LD。LD表示为D ' × 100,空方块表示完整LD (D ' = 1)。图中为SNP峰(蓝色箭头)和候选基因(绿色箭头)的位置
为了确定候选基因,我们寻找符合以下两个标准之一的基因:1)被注释为参与给定矿物元素的运输并在根、芽或叶中表达的基因;2)被注释为参与感兴趣元素的摄取、易位和/或平衡并主要在种子中表达的基因。总共确定了三个有希望的候选基因,这些基因参与了这些矿物元素的运输或同化。我们首先发现Glyma.06G046000(位于Ca_#3 SNP峰上游132 kb), Glyma.10G020000(位于K_#3 SNP峰下游222 kb)。这两个基因都被注释为参与运输,并在根尖和根毛中表达。此外,Glyma.06G046000在嫩叶、花、主根、豆荚和种子中均有表达(图2)。S4B和d)。最后,Glyma.20G151500 (S_#10下游32 kb)被注释为参与硫酸盐同化,并在花、根、根瘤和种子中表达(图2)。S4f). qtl K_#1、P_#1、S_#4和S_#7未找到符合标准的候选基因。
候选基因的结构和核苷酸变异及其预测的功能影响
为了确定候选基因内或重叠的遗传(结构或核苷酸)变异是否可能构成因果变异,我们检查了56个品系的全基因组测序数据中建立的此类变异目录。没有发现与这三个候选基因完全或部分重叠的结构变异(> 51 bp)。在核苷酸变异方面,在两个基因的编码区共发现18个snp(1个在Glyma.06G046000中,17个在Glyma.20G151500中)。所有这些变异都被预测为对蛋白质功能具有“修饰”或“低”影响。因此,观察到的表型变异不太可能是由于这些候选基因的功能丧失。
为了更深入地了解这些候选基因在观察到的表型变异中的作用,进行了单倍型分析。如图1所示。S5显著的表型差异(P各候选基因的单倍型间差异均< 0.05)。例如,在Glyma.06G046000中鉴定的4个单倍型(A、B、C和D)中,携带B单倍型的7个品系与携带A、C或D单倍型的130个品系相比,Ca含量显著不同。同样,Glyma.10G020000中携带C单倍型的5个品系,其K含量高于携带其他4个单倍型中的一个的品系。在Glyma.20G151500的例子中,它是更常见的单倍型A (n= 99), S含量显著高于其他4个单倍型。这些结果支持每个候选基因都很有希望,因为它们都对表型变异有贡献。
讨论
表型变异及性状间的相关性
在最初发现标记-性状关联的两种环境中,所有元素(Ca、K、P和S)的种子含量均呈正态分布,表明它们是定量遗传的。在干重基础上,本研究的表型变异范围为Ca含量1.6 ~ 2.4 mg/g, K含量17 ~ 21 mg/g, P含量4.5 ~ 6.5 mg/g, S含量3.5 ~ 5.5 mg/g。在以前的研究中,不同的范围被报道过。Otaka等。[8]和Homura等人。[17Ca(分别为1.5 ~ 3.5 mg/g和1.5 ~ 3.2 mg/g)和K(分别为19.8 ~ 22.1 mg/g和16.7 ~ 21.2 mg/g)的种子含量相似。同样,King等人。[13]报告的P含量值范围相当(3.9至5.6 mg/g),而Dhanapal等人[16]报告的数值范围较低(1.3至4.9 mg/g)。然而,本研究中S含量的范围略高于以往报道。我们的S含量值普遍高于Fageria [18]和Bellaloui等人。[19(分别为2.5至4.0毫克/克和1.6至3.1毫克/克)。总体而言,本文报告的值与以往研究报告的值相当一致,范围的细微差异可能只是反映了在不同环境中生长的特定植物组以及测量方法的选择所造成的差异。尽管如此,在关联研究的背景下,表型值的准确性不如充分捕捉值在整个小组中的变化那么重要。
在我们的研究中,两种环境的广义遗传力估计都很高(H2> 0.80)。这种相对较高的广义遗传力表明,表型在很大程度上是由基因型效应决定的[20.]。在以前的研究中已经报道了这些性状的类似遗传力(H2= 0.48 ~ 0.93) [14,16]。此外,K和P浓度之间存在显著的正相关关系(0.65,表S1)。这与先前报道的0.80至0.94的结果一致[2,19]。
大豆种子矿质元素含量的全基因组关联扫描
共鉴定出32个与Ca、K、P和S含量相关的qtl(表S2),其中8个qtl被至少两个模型检测到。更重要的是,这8个qtl中有7个被证明是非常健壮的,因为它们可以在另外3个试验中成功地确认为影响矿物元素含量。在之前的GWAS研究中,Ziegler等。[4]和Ning等。[14]分别报道了22个和9个qtl。使用链接映射方法,Ramamurthy等。[2]报道7和Bellaloui等人。[19]报道了11个与大豆种子钙、钾、磷、硫含量相关的qtl。在这项工作中检测到的大量初始qtl(32个)可能归因于更详尽的基因组覆盖(2.2 M snp)以及使用多种模型来检测标记-性状关联。这不太可能是由于我们的关联小组中表型值的范围特别大,因为这个范围与以前研究报告的范围相当。
本研究中描述的qtl通常解释了相当大一部分表型变异(18-31%)。先前报道的qtl解释的表型变异从2%到18%不等。有趣的是,我们发现其中一个qtl对钾和磷含量都有影响。考虑到钾和磷元素之间的高度正相关,这种钾和磷含量的共享QTL并不奇怪。同样,Dhanapal等人。[16]也报道了一个与这两个元素的内容相关的QTL。这种相关性可能是由于共同的生理机制和代谢途径[4,21]。换句话说,它可能是通过控制这些矿物质浓度的同一基因的多效性发生的,如共转运蛋白[20.或者仅仅是因为在同一区域存在独立的基因。
为了将我们的结果与之前确定的QTL区域进行比较,我们在SoyBase基因组浏览器上查询了之前的QTL,并定义了它们的物理位置。本研究中鉴定的7个强效QTL均与先前报道的基于家族的定位或GWAS中鉴定的QTL间隔不一致。因此,当前的qtl可以被认为是新颖的。通过这项工作确定的qtl与之前报道的qtl之间没有重叠,这可能反映了这些性状是由不同作图实验中使用的实验材料中的不同基因决定的。
矿物元素积累的候选基因及其功能
如上所述,我们关注的是在根、芽或叶中也表达的运输相关基因,以及主要在种子中表达的参与营养吸收、转运和/或平衡的基因。对于钾含量,在Gm 10上的QTL K_#3上发现了一个与转运相关的基因Glyma.10G020000。该基因被标注为K+钾转运蛋白及其同源物答:芥(AT4G13420.1)编码高亲和力的K+转运体5 (HAK5)。在水稻方面,Yang等。[22研究表明,HAK5在控制K的流入和K的迁移中都起着重要作用+进入根部并将其输送到植物的空中部分。我们的候选基因的两个类似基因(Glyma.02 g154100和Glyma.07 g042500)被功能表征为参与根对K的吸收+在大豆中[23,24]。有趣的是,在Dhanapal等人的研究中。[16], K含量的qtl确实与这些HAK5相似物的基因组位置重叠。在我们的研究中,我们没有发现这些平行体与钾含量之间的任何关联,这表明是10号染色体(Glyma.10G020000)上HAK5平行体的变异导致了加拿大早熟大豆品系钾积累的差异。
对于S含量,我们发现了一个编码ATP硫化酶1的基因(Glyma.20G151500),这是已知的第一个参与硫酸盐同化途径的酶拟南芥(AT3G22890.1;ASA1) [25]。一个一个芥互补脱氧核糖核酸编码ASA1成功地补充了酿酒酵母ATP硫化酶突变体(met3),从而恢复蛋氨酸异养和硫酸盐运输[26]。有趣的是,我们的候选基因在Gm10上被[16(Glyma.10 g242600)。这再次表明,相同的酶活性有助于S积累,但不同的基因拷贝控制不同的种质资源S含量。
最后,对于Ca含量,候选基因Glyma.06G046000被注释为参与钙运输的钙转运atp酶。这个基因与an是同源的答:芥轨迹(AT1G27770.1;ACA1)编码叶绿体包膜Ca2+- atp酶,已知与钙调蛋白结合,导致Ca2激活+泵(27]。研究表明Ca2+- atp酶是真核细胞中主动运输Ca2+的酶[28]并参与植物生命周期的所有阶段,包括生长和发育[29]。
结论
与以往的研究相比,本研究中使用的高密度标记有助于重复检测与大豆种子中矿质元素含量相关的几个新位点。除了提供有关加拿大大豆种质中种子矿质元素含量的表型变异范围的详细信息外,本研究还提供了有关其积累的遗传结构的更多信息。本研究鉴定的标记和基因将为大豆通过标记辅助选择进行遗传改良提供参考。
方法
植物材料和实验设计
根据Sonah等人的群体结构分析,从304份更大的群体中选择了137份早熟大豆品系(成熟度组000-II, MG000-II) [30.来代表加拿大短季大豆的遗传多样性。大豆品系来源于博士。Louise O 'Donoughue (CÉROM, St-Mathieu-de-Beloeil, QC), Elroy Cober(加拿大农业和农业食品部,渥太华,安大略省),Istvan Rajcan(圭尔夫大学,圭尔夫,安大略省)和Éric Gagnon先生(Semences program Inc., st - c辽阔,QC)。在第一阶段(发现),品系在两种环境中表型化,即2013年在加拿大的Woodstock (ON)和St-Mathieu-de-Beloeil (QC)。试验设计为广义格型,所有品系种植在单行地块上,每个位置有两个重复。在第二(验证)阶段,2017年(17)和2018年(18)在渥太华(ON)的中央实验农场的三种环境中种植了相同的品系。这些品系以改良的增强型设计种植,作为四行地块,有一个重复。在每一年内,采用两种不同的处理:不灌溉(N)或滴灌(I)。由于N_17试验没有完整的品系/种子,因此仅使用I_17、I_18和N_18试验的数据对发现的qtl进行鲁棒性检验。
校正及验证
能量色散x射线荧光(ED-XRF)光谱仪的校准采用经验校准方法[31],其中使用了与感兴趣的样品具有相似成分和形态的标准物。在这里,使用火焰原子吸收光谱法(FAAS)测量了一组样品中Ca和K的元素浓度。32]和分光光度法测定磷含量,按[33]。对于S的含量,我们连续加入Na2所以4参考物质(WEPAL, IPE 885 (Maize))由美国国家标准与技术研究所(NIST)提供,其S含量已知。由此获得的值可作为校准ED-XRF装置(Niton XL3t955 GOLDD)标准曲线的基线。为了确定我们的ED-XRF测量的准确性和可靠性,用ED-XRF和湿化学方法分析了30个种子样品中Ca、K和p的浓度,而不需要湿化学验证S的含量。
表型和统计分析
对于每个样品,使用研磨机(Foss a /S: Cyclotec™1093 sample Mill)研磨10g整粒种子。从每条生产线上取0.3 g的均匀细粉样品,在液压制粒机(Carver 4350.L)中使用不锈钢制粒模具压制,以生产紧凑的13毫米(~ 0.2毫米厚)的颗粒。待用ED-XRF测定钙、钾、磷、硫含量时,将微球保存。
利用R软件包«lmer»进行描述性统计、基因型变异、环境和环境效应基因型以及这些矿物质含量之间的相关性分析。为了结合来自不同环境的信息,使用META-R中的限制最大似然来计算最佳线性无偏预测(blup) [34]。广义的遗传性H2跨环境计算如下:
在哪里\ ({\ upsigma} _ {\ mathrm {g}} ^ 2, {\ upsigma} _ {\ mathrm {g} \ mathrm {e}} ^ 2 \)和\({\upsigma}_{\ mathm {e}}^2 \)分别为基因型、基因型与环境交互作用和误差方差分量。nEnv是环境的数量,nrep是复制的数量。
基因分型和snp归算
来自测序192 plex GBS文库的总计约2.03亿个100 bp Illumina HiSeq2000单端reads可用于137个系(如先前详细介绍]30.])。简而言之,限制性内切酶猿K1用于生成单个192 plex GBS文库(包含额外的不相关样本),该文库在Illumina HiSeq2000测序仪的单线上测序。137个种群共获得2.03亿个100-bp单端reads。Fast-GBS管道[35]和Wm82.a2。V1参考基因组[36]用于最小读取深度为2次的SNP调用,并删除丢失数据超过80%的位点。使用BEAGLE v5对这组gbs衍生的snp进行缺失基因型的第一步插入[37]。其次,使用从102个部分重叠(56个共享)系的全基因组重测序中获得的4.3万个snp的参考面板,对gbs衍生的snp集进行缺失位点插入[38]。以前曾评估过这种未分型基因座的输入准确性[38的比例为96.4%。
人口结构与亲属关系分析
对于种群结构分析,我们使用了修剪后的(r)2> 0.5)使用Plink v1.9获得的14 K snp目录[39]。程序fastSTRUCTURE中实现的贝叶斯模型[40]用于人口分层分析。亚种群的数量(k)设为1 ~ 12,独立迭代3次。亚种群的数量(k)是使用fastSTRUCTURE中实现的python脚本(" choosek ")确定的。除了fastSTRUCTURE之外,还使用了两种不同的方法来推断种群结构:(i)基于2000次迭代的Tamura-Nei模型计算的共识系统发育树,在MEGA7中实现[41]及(ii)在GAPIT计划中实施的主成分分析(PCA) [42]。
全基因组关联分析
在GAPIT中,利用243个K snp的修剪目录评估了标记与表型之间的全基因组关联2> 0.9)和各性状的BLUP值。单位点GWAS采用CMLM,多位点GWAS采用MLMM和FarmCPU。通过亲属矩阵(K)和通过fastSTRUCTURE估计的群体结构矩阵(Q)传递的系间遗传亲缘关系用于控制假阳性关联。三种模型(CMLM、MLMM和FarmCPU)的标记-性状关联显著性阈值进行了调整p-值,错误发现率(FDR)设为≤0.05,根据Benjamini和Hochberg程序[43]。我们假设所有显著的标记-性状关联标记在同一单倍型区(由最左和最右的标记所划分的区域,这些标记处于完美LD (D ' = 1), SNP峰值)。当不同模型检测到的峰值不同时,选择两种模型检测到的峰值。此外,当至少两个模型检测到相同的峰值SNP时,最低的未校正p-value报告。我们选择仅报告和研究共检测的qtl,即每个性状至少有两个模型检测到的qtl。为了鉴定候选基因,将SNP峰值周围的感兴趣基因组区域定义为在与SNP峰值处于完美LD (D ' = 1)的最左和最右侧标记之间延伸。
三种环境下共检测qtl的等位基因效应验证
为了评估在发现阶段鉴定的qtl的可重复性,我们使用同一组137个品系,验证了qtl在三种不同环境(I_17、I_18和N_18)中的等位基因效应。标记-性状相关性采用t检验。根据SNP峰值的等位基因类别将群体分为两组。我们对两组的平均表型值进行t检验。标记-性状关联的显著性阈值根据多个检验进行调整(α = 0.05/n,其中n为一个性状的qtl总数)。显著性检验采用R 3.5.1版本中实现的t检验函数,根据[44]: Y = μ + f(标记)+误差,其中Y等于性状值,μ等于总体均值,f(标记)是显著标记的函数。
候选基因及其功能分析
通过使用数据挖掘算法[45],从SoyBase浏览器中提取了所有存在于感兴趣的单倍型块中的基因,并检查了它们的GO注释。在确定候选基因后,使用电子荧光象形文字(eFP)浏览器(www.bar.utoronto.ca)。
候选基因列表中由潜在功能缺失(LOF)等位基因引起的转录本改变通过检查[46]。LOF分析仅使用全基因组测序数据集(56个品系)。对于基因区域内核苷酸变异的研究,SnpEff [47]与全套核苷酸变异(修剪前,2.18 M snp)一起使用。
为了进一步支持候选基因参与观察到的表型变异,采用基因中心单倍型(GCH)方法,使用HaplotypeMiner识别候选基因内定义单倍型的多态性标记[48]和2.18个snp的完整目录。然后对每个单倍型的平均表型值进行t检验。
数据和材料的可用性
本研究中使用和分析的数据集可应通讯作者的合理要求向其提供。
缩写
- BLUP:
-
最好的线性无偏预测器
- CMLM:
-
压缩混合线性模型
- FarmCPU:
-
固定与随机模型循环概率统一
- GWAS:
-
全基因组关联研究
- H2:
-
广义遗传
- h2:
-
狭义遗传
- LD:
-
连锁不平衡
- LOF:
-
功能丧失
- 加:
-
次要等位基因频率
- MLMM:
-
多轨迹混合线性模型
- QTL:
-
数量性状位点
- R2:
-
表型变异解释
- SNP:
-
单核苷酸多态性
参考文献
- 1.
杨建军,杨建军。高温条件下大豆籽粒矿物含量的研究进展。植物学报,2001;31(1):595 - 596。
- 2.
李建军,李建军,李建军,李建军。大豆种子矿物质、半胱氨酸和蛋氨酸浓度新qtl的鉴定[j]稳定)。动物学报,2014;34(4):431 - 445。
- 3.
与离子吸收和利用基因型差异有关的生理机制。HORTSCIENCE。1989; 24:559 - 64。
- 4.
Ziegler G, Nelson R, Granada S, Krishnan HB, Gillman JD, Baxter I.大豆种质资源不同群体的全基因组关联研究。植物导报,2018;2:e00033。
- 5.
李建军,李建军,李建军,等。硬粒小麦×野生二粒小麦RIL群体籽粒矿质养分含量的数量性状位点。理论与应用,2009;119:353-69。
- 6.
盐DE, Baxter I, Lahner B.离子经济学和植物离子素的研究。植物学报,2008;39(5):779 - 779。
- 7.
陈建军,陈建军,陈建军,等。便携式ED-XRF直接测定可可粉样品中矿物质特征。食品科学与技术,2019;38(8):373 - 9。
- 8.
大taka A, Hokura A A, Nakai I.大豆中微量元素的x射线荧光分析及其在产地鉴定中的应用。食品科学与技术,2014;47(1):1 - 6。
- 9.
Tadeu Costa Junior G, Nunes LC, Feresin Gomes MH, Almeida E, Pereira de Carvalho HW便携式x射线荧光光谱法在体内条件下直接测定大豆叶片中的矿质营养素。x射线光谱。2019;:xrs.3111。
- 10.
Bamrah RK, Vijayan P, Karunakaran C, Muir D, Hallin E, Stobbs J,等。x射线荧光光谱技术在豌豆种子营养成分分析中的应用评价。作物科学,2019;59:2689。
- 11.
王晓明,王晓明,王晓明,等。提高巴西酸性土壤养分利用效率对作物可持续生产的影响。土壤学报,2001;32。
- 12.
vreugdenhill D, Aarts MGM, Koornneef M, Nelissen H, Ernst WHO。拟南芥种子阳离子矿物质含量的自然变异及QTL分析。植物学报。2004;27:828-39。
- 13.
柯金,劳特N,林顺生,史考特MP,舒梅克RC。大豆种子磷含量评价及QTL定位。Euphytica。2013;189:261-9。
- 14.
宁玲,孙鹏,王强,马东,胡忠,张东,等。大豆生物强化性状的遗传结构(大豆L. Merr.)通过关联分析和连锁映射揭示。Euphytica。2015;204:353 - 69。
- 15.
张波,陈平,石安,侯安,石桥涛,王东。大豆种子钙含量相关数量性状的推测位点。[J]中华医学杂志,2009;10(1):1 - 6。
- 16.
Dhanapal AP, Ray JD, Smith JR, Purcell LC, Fritschi FB大豆茎部组织宏量和微量营养素浓度相关新基因位点的鉴定。植物基因组学2018;11:0。
- 17.
贺建平,李建平,李建平,等。用无机元素组成测定黑豆“丹巴黑”籽粒的地理来源。食品科学与技术学报。2006;53:619-26。
- 18.
Fageria NK。作物对养分的利用。博卡拉顿:CRC出版社;2009.
- 19.
Bellaloui N, Khandaker L, Akond M, Kantartzi SK, Meksem K, Mengistu A,等。大豆“Spencer”重组系“md96 -5722”种子大量营养物质积累相关QTL的遗传定位AJB。2015; 3:224-35。
- 20.
刘建军,刘建军,刘建军,等。全基因组关联研究揭示了合成六倍体小麦中与10种谷物矿物质相关的新基因组区域。IJMS。2018; 19:3237。
- 21.
马勇,阚刚,张旭,王勇,张伟,杜华,等。大豆甘氨酸和β-甘氨酸含量的QTL定位(大豆l .稳定)。中国农业科学学报,2016;44(4):444 - 444。
- 22.
杨涛,张生,胡勇,吴峰,胡强,陈刚,等。钾转运体OsHAK5在低钾供应条件下水稻钾获取和根向茎转运中的作用植物生理学报,2014;16(6):945 - 959。
- 23.
陈红,陈霞,顾红,吴波,张红,袁霞,等。GmHKT1;4,一个调节大豆根系Na+/K+比例的新基因,增强转基因植物的耐盐性。植物学报,2014;33(3):391 - 391。
- 24.
Rehman HM, Nawaz MA, Shah ZH, Daur I, Khatoon S, Yang SH,等。对大豆5个K+转运基因家族的基因组和转录组学分析证实了它们在结瘤过程中的差异表达。植物科学,2017;8:804。
- 25.
王晓明,王晓明,王晓明,等。拟南芥ATP硫酰化酶基因的克隆及其在酿酒酵母中的功能表达。植物生理学报。1994;105:897-902。
- 26.
Logan HM, Cathala N, Grignon C, david J-C。克隆一种由一个成员编码的cDNA拟南芥ATP硫酰化酶多基因家族在酵母中的表达及其与植物硫营养的关系。生物化学学报,1996;27(1):1227 - 1233。
- 27.
Singh A, Kanwar P, Yadav AK, Mishra M, Jha SK, Baranwal V等。水稻非生物胁迫和发育过程中钙转运元件的全基因组表达和功能分析。[j] . 2014;28:894 - 915。
- 28.
KMdK H, MstSA B, Tuteja R, Tuteja N.全球钙传感器p型Ca2+−atp酶调控胁迫信号为农业开辟新途径。[J] .中国生物医学工程学报,2013;33(4):444 - 444。
- 29.
王勇,Itaya A,钟旭,吴勇,张杰,van der Knaap E,等。调控Ca的MicroRNA的功能和进化2 +−atp酶,在番茄生殖生长过程中触发阶段性小干扰rna的形成。植物学报,2011;23(3):385 - 203。
- 30.
Sonah H, O 'Donoughue L, Cober E, Rajcan I, Belzile F.大豆8个农艺性状的GBS-GWAS定位及QTL定位验证。植物学报,2014;13(3):521 - 521。
- 31.
Paltridge NG, Palmer LJ, Milham PJ, Guild GE, Stangoulis JCR。水稻和珍珠粟籽粒中锌和铁浓度的能量色散x射线荧光分析。植物学报。2012;31(1):361 - 361。
- 32.
优秀的。原子吸收光谱分析方法。珀金-埃尔默公司,美国1982: 1 - 300。
- 33.
墨菲J,赖利JP。测定天然水中磷酸盐的改进方法。化学学报,1962;27:31-6。
- 34.
Alvarado G, Marco L, Mateo V, Angela P, Francisco R, Juan B,等。META-R (Multi - Environment Trail Analysis with R for Windows) 6.01版。2015.
- 35.
张建军,张建军,张建军,等。基于基因分型的基因多态性分析方法的研究进展。中国生物医学工程学报,2017;18。https://doi.org/10.1186/s12859-016-1431-9。
- 36.
宋强,詹金斯,贾刚,Hyten DL, Pantalone V, Jackson SA,等。构建高分辨率遗传连锁图谱改进大豆基因组序列组装Glyma1.01。17. BMC Genomics. 2016;https://doi.org/10.1186/s12864-015-2344-0。
- 37.
陈建平,陈建平。基于局部单倍型聚类的全基因组关联研究的快速准确单倍型分期和缺失数据推断。[J] .中国生物医学工程学报。2007;31(1):1 - 4。
- 38.
李建军,张建军,张建军,等。植物基因组分型策略及其应用。科学通报,2018;31(1):491 - 491。
- 39.
李建军,李建军,李建军,等。PLINK:全基因组关联和基于群体的连锁分析的工具集。[J] .中国生物医学工程学报。2007;31(1):559 - 559。
- 40.
Raj A, Stephens M, Pritchard JK。fastSTRUCTURE:大型SNP数据集中种群结构的变分推断。遗传学。2014;197:573 - 89。
- 41.
MEGA7:用于更大数据集的分子进化遗传学分析版本7.0。中华生物医学杂志,2016;33:1870-4。
- 42.
Lipka AE,田芳,王强,Peiffer J, Li M, Bradbury PJ,等。GAPIT:基因组关联与预测集成工具。生物信息学。2012;28:2397-9。
- 43.
刘建军,刘建军。控制错误发现率:一种实用且强大的多重测试方法。[J] .中国生物医学工程学报。2003;22(1):389 - 391。
- 44.
Francis DM, Merk HL, Namuth-covert D.单标记分析(SMA)概述。2011: 1 - 3。
- 45.
Morales AMAP, O 'Rourke JA, van de Mortel M, Scheider KT, Bancroft TJ, borsamm A,等。转录组分析和病毒诱导的基因沉默鉴定了rpp4介导的亚洲大豆抗锈病途径中的基因。植物学报,2013;39(4):444 - 444。
- 46.
李建军,李建军,李建军,等。短季大豆全基因组核苷酸和结构变异的综合描述。植物学报,2018;16(6):749 - 759。
- 47.
王立林,王立民,王立民,王磊。一个用于注释和预测单核苷酸多态性影响的程序,SnpEff:基因组中的SNPs黑腹果蝇应变w1118;iso-2;iso-3。飞。2012;6:80 - 92。
- 48.
李建军,李建军,李建军,李建军,李建军。一个系统的基因中心的方法来定义单倍型和识别等位基因的基础上密集的单核苷酸多态性数据集。植物基因组。2019;12:180061。
致谢
作者要感谢来自加拿大魁省拉瓦尔大学Richard Belanger实验室和franois Belzile实验室的Jean Martin和Martin Lacroix。
资金
本研究由马里粮食安全农业培训(FASAM in French)通过合作研究与发展赠款资助。资助者在研究设计、分析或手稿准备中没有任何作用。
作者信息
从属关系
贡献
S.M.和F.B.构思了这个项目,解释了数据并撰写了手稿。S.M.进行了大部分表型分析和所有数据分析。M.M.进行了用于验证qtl的试验(三个地点)。所有作者已阅读并同意稿件。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者声明不存在利益冲突。
额外的信息
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
Malle, S, Morrison, M.和Belzile, F.大豆种子中矿质元素浓度控制位点的鉴定。BMC Plant Biol20.419(2020)。https://doi.org/10.1186/s12870-020-02631-w
收到了:
接受:
发表:
关键字年代
- 大豆
- 矿物质
- 光谱仪
- GWAS
- QTL