摘要
背景
GRAS蛋白是关键转录因子,其是植物特异性的,并参与各种植物生物过程。由于整个基因组测序技术的快速进展,肝广泛探索和研究了不同植物中的基因家庭。但是,对大豆的全面研究(甘氨酸最大)肝基因家庭相对滞后。
结果
在这项研究中,117甘氨酸最大肝基因(GmGRAS)被确定。进一步的系统发育分析表明GmGRAS基因可分为9个亚家族:DELLA、HAM、LAS、lisl、PAT1、SCL3、SCL4/7、SCR和SHR。基因结构分析表明GmGRAS基因缺乏内含子,相对保守。结构域和基序模式GmGRAS同一亚家族或思工的成员表现出相似之处。值得注意的是,扩张的GmGRAS基因家族因基因串联和节段性重复事件而被驱动。虽然,细分重复在生成新的作用方面取得了重要作用GmGRAS基因。此外,大豆和不同物种(两种单焦点和四个Dicots)中GRAS蛋白的同步和进化约束分析提供了更详细的证据GmGRAS基因进化。CIS.-元素分析表明GmGRAS基因可能响应于不同的环境压力并调节不同的生物过程。此外,表达模式GmGRAS在不同的组织中,在盐碱和脱水胁迫以及种子萌发过程中,基因存在差异。
结论
我们对其进行了系统的调查肝大豆基因,这可能对未来铺平道路有价值GmGRAS基因研究和大豆育种。
背景
GRAS转录因子(TFs)是植物特异性调控蛋白,在过去的十年中得到了广泛的研究[1那2那3.那4.].GRAS蛋白的名称来源于该基因家族中最早发现的三个成员:赤霉素不敏感(GAI)、GA1-3突变体抑制因子(RGA)和稻草人(SCR) [5.].通常,GRAS蛋白序列由400-770个氨基酸残基组成,其表现出高度保守的C末端区域和可变N末端[6.那7.].通常,GRAS结构域由保守的羧基末端区域确定,并且可以分为五个图案:富含亮氨酸的区域I(LHRI),VHIID,富含少数区域II(LHRII),PFYRE和SAW。值得注意的是,这五个主题在GRAS与其他蛋白质之间的相互作用中发挥了重要作用[8.].根据早期的研究,LHRI和LHRII对于GRAS蛋白的同源二聚化是至关重要的。VHIID基序是GRAS蛋白的核心成分,它包含一个非常保守的P-N-H-D-Q-L单元,以L-R-I-T-G结尾。3对保守的蛋白序列特征P、FY和RE可以被识别并组装到PFYRE基序中,可能与磷酸化有关。SAW基序由3个保守氨基酸残基组成:R-E、W-G和W-W [4.那6.那9.].与此相反,GRAS蛋白质的善变N-末端可以折叠和修饰成特定的分子的结合结构。基于这些,该GRAS蛋白质广泛地参与了诸如信号传导,根径向伸长,副梢分生组织的形成和应激反应中的植物[许多关键过程10.那11.那12.那13.].在此之前,肝基因家族在拟南芥被分为8个亚科,包括DELLA、SCL3、LAS、SCR、HAM、SHR、lisl和PAT1 [14.].Della Subfamily包含了时至今日那RGA和RGL,被报道为赤霉素信号转导的主要抑制因子[15.].重要的是,SCL3蛋白被验证为介导根伸长的开关[16.].此外,SCL3蛋白可与DELLA蛋白协同,通过IDD蛋白调节赤霉素反馈[17.].此外,SHR和SCR蛋白倾向于形成SCR/SHR复合物,该复合物被确定与根径向模式相关[18.那19.].据报道,LAS蛋白与拟南芥营养生长阶段的侧枝形成紧密相连[11.].此外,过表达VaPAT1(一种肝基因的血管血管血管)提高了转基因拟南芥对非生物胁迫的耐受性[20.].另一项研究结果ATSCL13(Pat1亚家族的成员拟南芥)参与光敏色素A (phyA)信号转导,并在下胚轴伸长中起主要作用[21.].在Medicago Truncatula., HAM亚家族基因MTNSP2.和我们一起MTNSP.1(SHR亚家族基因),形成了DNA结合复合物,以诱导染色信号传导期间的基因表达[22.].在矮牵牛,PhHAM基因以非细胞自主方式作用于邻近组织,并维持顶端分生组织的活动[23.].随着测序技术的快速发展,DLT、SCL4/7、Os19、Os4和PT20等几个新的亚家族逐渐丰富了前者肝不同植物中的基因亚毛利语[24.].到目前为止,单子叶和双子叶植物有30多种,如水稻、玉米、拟南芥、棉花、Malus Domestica.蓖麻已经在全基因组范围内进行了研究肝基因家族鉴定及分析[3.那8.那19.那25.那26.].
大豆(甘氨酸最大L.)是高品质蛋白质和油丰富的主要作物之一,其还含有各种营养素,如卵磷脂和异黄酮[27.].许多大豆转录因子家族,如WKRY [28.], MYB [29.),南京(30.], HD-Zip [31], ARF [32疯了[33]已经被调查和研究过。但是,综合研究大豆肝基因(GmGRAS)家庭相对滞后。由于重要性肝植物发育和生理过程中的相关基因,必须进行相关的探索和分析,以弥补这一空白。在这项研究中,我们系统地识别了117GmGRAS大豆基因组中的基因成员。首先,我们研究了已鉴定的GmGRAS成员的系统发育关系、基因结构、基序组成、染色体位置和基因复制事件。接下来,我们对大豆和四种双子叶植物(拟南芥那甘氨酸Soja.那Vigna Unguiculata.和茄属植物lycopersicum),以及两个单子叶植物(奥雅萨苜蓿和高粱二色的).此外,我们分析了CIS.-元素的启动子区域GmGRAS基因。此外,我们探索了表达模式GmGRAS在不同的组织,在盐和脱水胁迫和种子萌发过程中的基因。特别是由于种子萌发在大豆生产中的重要性,18种大豆具有代表性肝进一步选择基因并进行定量RT-PCR分析。集体,目前的研究提供了对未来功能研究的见解GmGRAS基因对大豆育种可能是有价值的。
结果
的识别GmGRAS基因大豆
完全117GmGRAS从大豆中鉴定基因Wm82.a2.v1植物基因组(https://phytozome.jgi.doe.gov/pz/portal.html#).其中116个基因被定位在20条不同的大豆染色体上,1个基因(Glyma.U013800.1.Wm82.a2.v1)位于大豆基因组中未归属的脚手架d_21上,更名为Gmgras117..根据染色体名称和染色体位置,其余116GmGRAS基因从GmGRAS1来gmgras116,分别(附加文件1:表S1)。
GmGRAS家族成员的基本特征列于表S1(附加文件)1),包括开放阅读框(ORF)长度、蛋白质大小、蛋白质分子量(MW)、等电点(pI)、预测的亚细胞定位、推定的保守结构域、其他物种的同源性。如表S1所示(附加文件1), GmGRAS55是最小的蛋白质,有169个氨基酸(aa),而GmGRAS111是最大的蛋白质(843个aa)。分子量范围为18975.84 ~ 91543.91 Da, pI范围为4.76 ~ 9.21 (GmGRAS55)。预测的亚细胞定位结果显示,74个GmGRAS蛋白位于核区,29个位于细胞质区,10个位于质膜区,2个位于胞外区,1个位于叶绿体区,1个位于线粒体区。以及所鉴定的编码序列和蛋白序列GmGRAS基因成员列于表S2中(附加文件2).
系统发育分析和分类GmGRAS基因成员
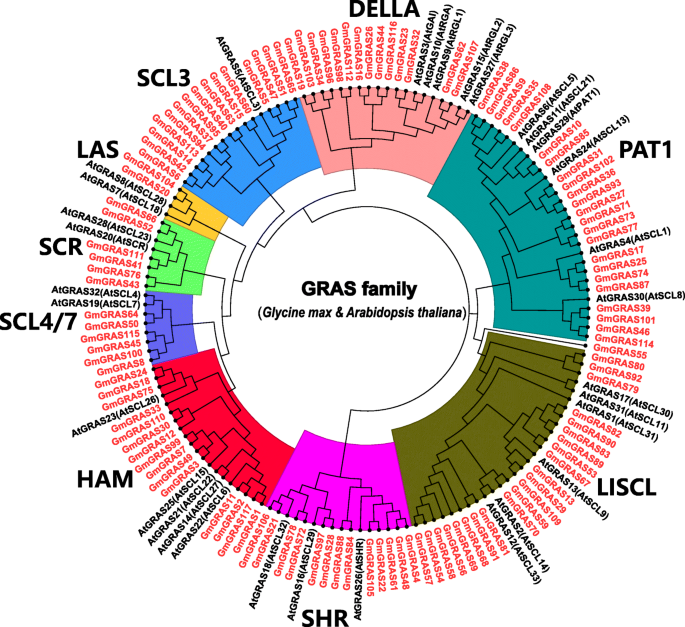
为了分类大豆甘蛋白的系统发育关系,我们基于本研究中鉴定的117个Gmgras构建了一种系统发育树,32名据报道了来自Tair的拟南芥蛋白质(https://www.arabidopesis.org/index.jsp.jsp.)(额外的文件3.:表S3)。系统发育分析表明116GmGRAS基因成员分为9个亚家族:DELLA、HAM、LAS、lisl、PAT1、SCL3、SCL4/7、SCR、SHR。GmGRAS55相对独立,不属于任何一个肝基因亚科(图。1).如图1所示。1和表S1(附加文件1),Pat1亚家族在此含有23个成员并且是最大的成员GmGRAS基因亚家族。LISCL亚家族比PAT1亚家族少一个基因成员。巧合的是,HAM和SCL3亚家族都有15个成员。此外,还有14、13、6、6和2GmGRASDella,ShR,SCR,SCL4 / 7和Las Subfamilies的基因成员分别。
基因结构和基序模式GmGRAS基因成员
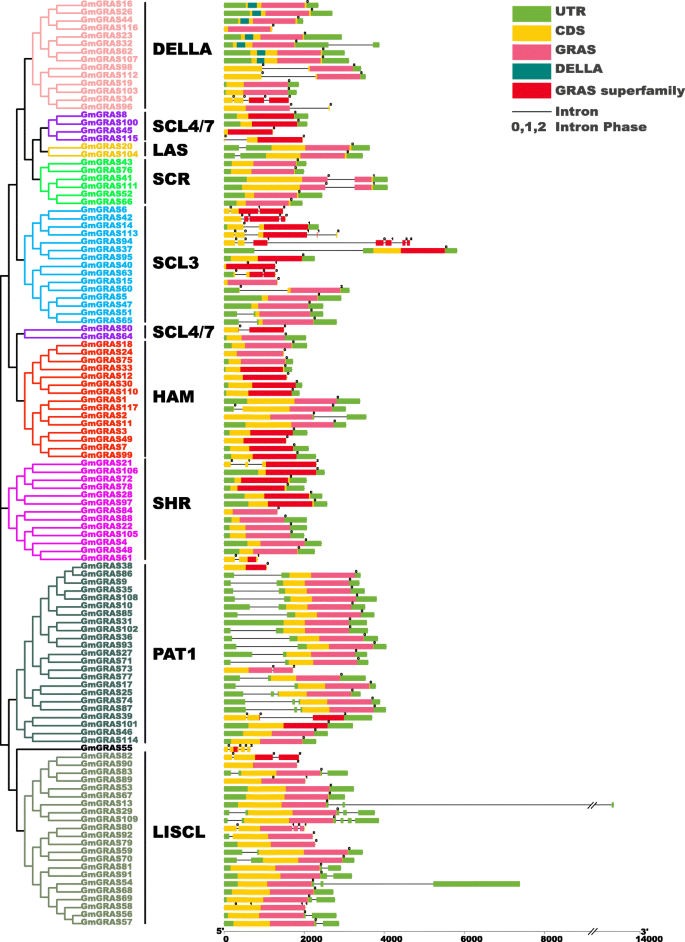
通过筛选相应的基因组DNA序列和注释文件,所识别的外显子内部模式GmGRAS获得基因。如图1所示。2,GmGRAS基因显示一到七个外显子(91个带有一个外显子,有两个外显子,有三个外显子,五个有四个外显子,一个有五个外显子,一个有五个外显子,一个有七个外显子,缺乏内含子。对于蛋白质保守的域,所有117个Gmgras成员至少具有一只克拉斯或GRAS超家族域。同一亚家族或思工中的成员具有类似的基因结构和蛋白质保守结构域。例如,Gmgras16,Gmgras23,Gmgras26,Gmgras32,Gmgras44和Gmgras62属于Della subfamily,并且它们中的每一个都包含一个外显子和没有内含子的Della蛋白质结构域(图。2).
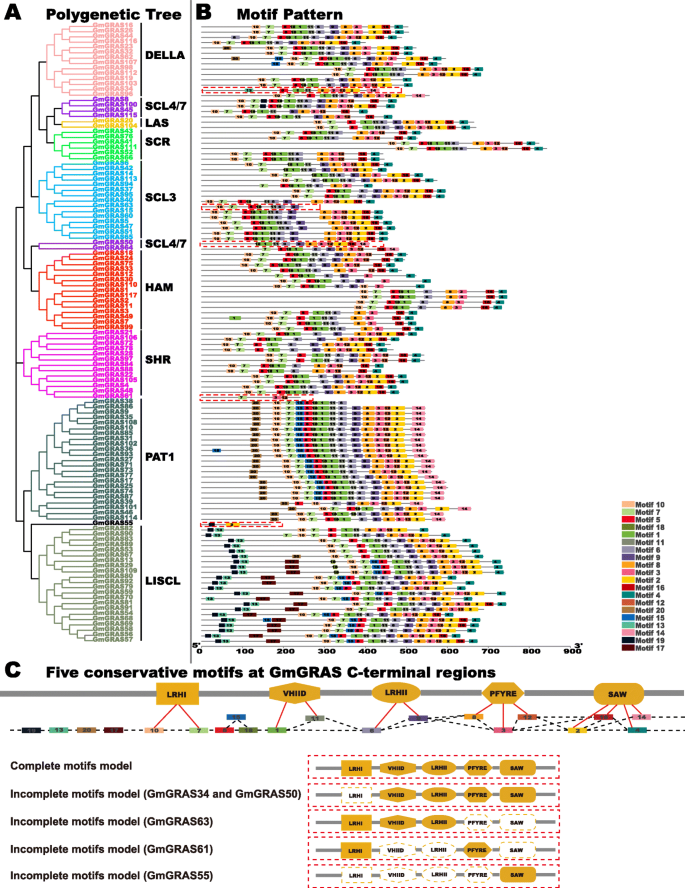
为了进一步证明Gmgras蛋白的结构,基于MEME-MOTIF扫描结果构建了示意图。如图1所示。3.b,展示了20个不同的模因-主题(命名为Motif 1-20)。此外,这些主题的详细信息见表S4(附加文件)4.)和20个模因主题的Seq标志展示在图S1(附加文件5.).参考Quan等人的分类。在胡桃regia,对模因-基序进行进一步评估,并将其分为5个GRAS特定的c端基序:LRHI、VHIID、LRHII、PFYRE和SAW [34].因此,motif 7和10被归为LRHI motif;motif 1和11属于VHIID motif;motif 6和9与LRHII motif相关;PFYRE基序包含基序3,8和12;Motifs 2,4,14和16在SAW motif中(图2)。3.b和无花果。3.c).此外,motif 5、15和18位于LRHI和VHIID motif之间。值得注意的是,GRAS特定c端基序中的模因-基序并不是固定的。有时,c端保守基序中仅存在一个模因基序。而c端保守基序则对应着两个或三个模因基序。有趣的是,一些大豆GRAS亚科包含独特的模因-motif。例如,Motifs 13、17和19只在lisl亚家族中发现。如图1所示。3.b和无花果。3.C,大多数Gmgras蛋白质包含C终端的五个保守基序的完整组成部分,但是,Gmgras34,Gmgras50,Gmgras55,Gmgras63和Gmgras61缺少五个保守的主题的一到四个亚基(用红色点缀表示盒子)。总的来说,特定的MEME-MOTIFGmGRAS基因亚科或分支表现出相似的组成和顺序。
染色体分布,同步和进化分析GmGRAS基因成员
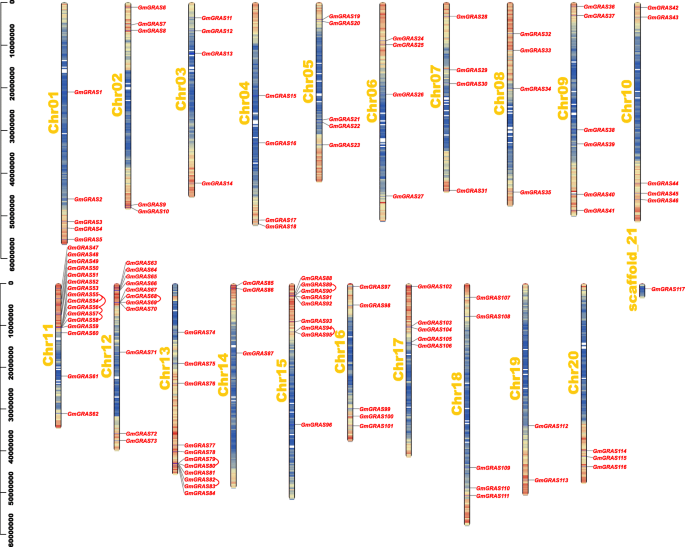
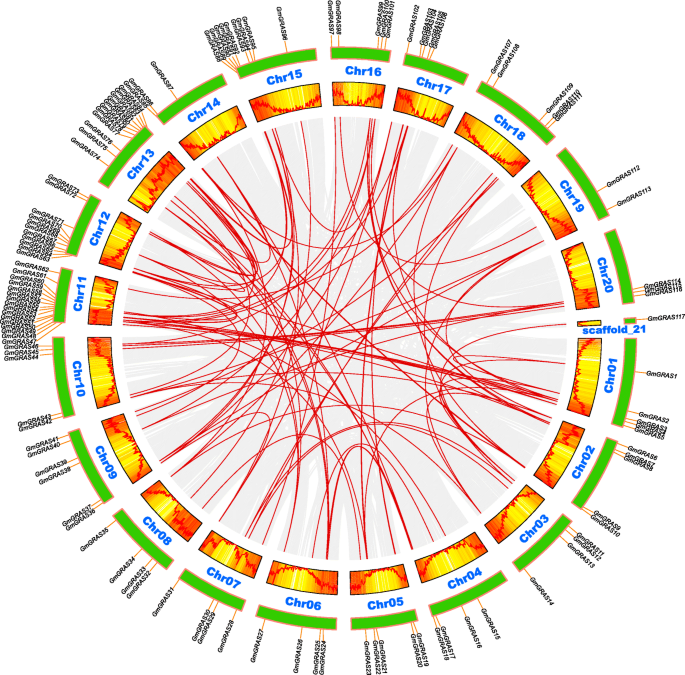
这GmGRAS根据大豆基因组的基因物理位置信息绘制基因染色体位置(图。4.).重要的是,每个染色体或支架的基因密度也通过在表S5(附加文件)中设置300 kb的遗传间隔来评估6.)通过从蓝色(低基因密度)到红色(高基因密度)的梯度颜色进一步说明。4..染色体或支架上的空白区域表明遗传区域缺乏基因分布信息。如图1所示。4., 117年GmGRAS基因在20条大豆染色体(Chr01 - Chr20)和scaffold d_21上分布不均匀。也是最确定的GmGRAS基因倾向于在高基因密度区域中定位。值得注意的是,CHR11包含最多GmGRAS基因和16个基因位于该染色体上。一些染色体(例如CHR12,CHR13和CHR15)具有相当大的GmGRAS而有些(如Chr19)成员很少,只有一个GmGRASscaffold_21基因。类似于之前的研究肝在其他物种中,在染色体长度和数量之间发现了无明显相关性GmGRAS基因(4.那7.].
早期研究表明,基因复制对于新基因功能的出现和基因家族的扩展至关重要[35].因此,我们进一步探索了117例的重复事件GmGRAS基因(图。4.和无花果。5.).在之前的一项研究中,Holub将串联复制事件定义为一个包含多个(两个或更多)基因家族成员的200 kb (kilobase)基因间区域[36].类似地,节段复制经常发生在植物中,因为大多数植物是二倍体多倍体,并在其基因组中保留大量重复的染色体块[37那38].通过多倍体和染色体重排的节段复制多个基因[39].重要的是,两种分段和串联重复被认为是植物中基因家族扩张的两个代表性主要原因[37那38].在这项研究中,是15GmGRAS在9个串联复制事件中发现了基因(gmgras54/Gmgras55.那Gmgras55./gmgras56.那gmgras56./gmgras57.那gmgras57./gmgras58那gmgras68/gmgras69那GmGRAS79/Gmgras80.那GmGRAS82/gmgras83那Gmgras89./Gmgras90.和gmgras94/gmgras95)分布在4条大豆染色体(Chr11、Chr12、Chr13和Chr15)上,表明这些区域可能是研究的热点GmGRAS基因分布(附加文件7.:表S6)。值得注意的是,除了用于的Liscl亚家族中,大多数串联复制事件发生在gmgras94/gmgras95,发生在SCL3亚家族。此外,104个节段重复事件与107个相关GmGRAS还检测到基因(图。5.和额外的文件7.:表S6)。总之,最GmGRAS基因可能来源于基因复制,而片段复制事件可能在产生新基因中起关键作用GmGRAS基因。
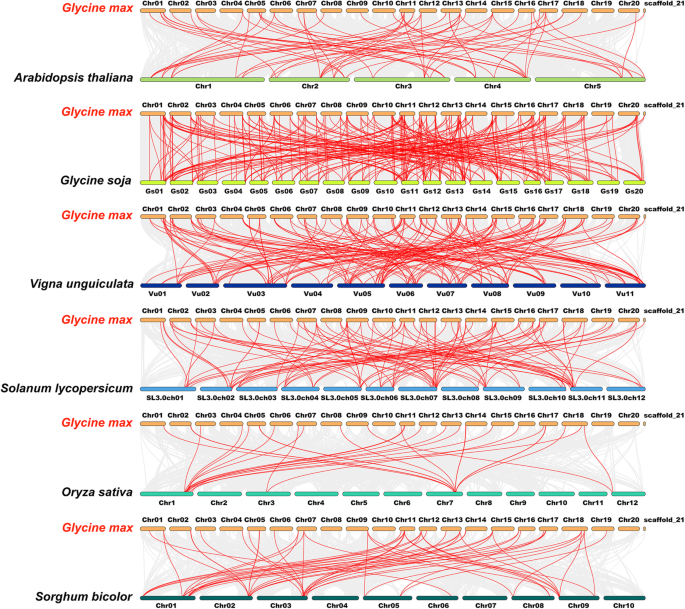
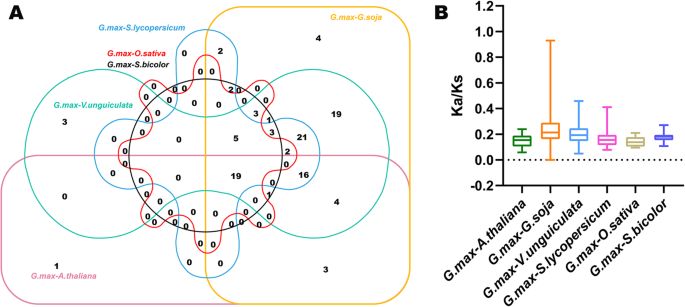
来探索大豆的进化线索肝基因家族,我们构建了6个比较同线图来显示基因家族的同线肝大豆和6个代表种类(图之间的基因成员。6.).六种代表性物种含有四个Dicots(拟南芥那甘氨酸Soja.那Vigna Unguiculata.和茄属植物lycopersicum)及两个单子叶植物(奥雅萨苜蓿和高粱二色的).完全109.GmGRAS基因成员与中、外基因成员呈共线关系甘氨酸Soja.(101),Vigna Unguiculata.(49),茄属植物lycopersicum(31)拟南芥(22),高粱二色的(21)和奥雅萨苜蓿(9)(附加文件8.:表S7)。和数量GmGRAS直向同源基因甘氨酸Soja.那Vigna Unguiculata.那茄属植物lycopersicum那拟南芥那高粱二色的和奥雅萨苜蓿分别为289、162、104、64、57和29。总的来说,GmGRAS与单子叶植物相比,双子叶植物的基因由更多的同同基因对组成。此外,作为。的祖先甘氨酸最大(大豆),甘氨酸Soja.与大豆表现出较强的同源性。重要的是,如交互式维恩图所示肝不同物种的基因(图。7.), 19GmGRAS基因syntenic肝所有六个物种的基因对。和19GmGRAS在表S8中以粗体突出显示基因(附加文件9.).大豆与其他物种间的同同基因对可能对阐明大豆的进化有价值肝基因。
在本研究中,KA / KS(非同义替代/同义替代)比率GmGRAS计算六种物种中的正交基因对以评估作用于大豆的进化约束肝基因家族(附加文件8.:表S7)。如图1所示。7.球GmGRAS直言基因对显示Ka / ks <1。因此,我们推测大豆肝基因家族可能在进化期间经历强烈的净化选择性压力[35].
CIS.-元素分析GmGRAS基因
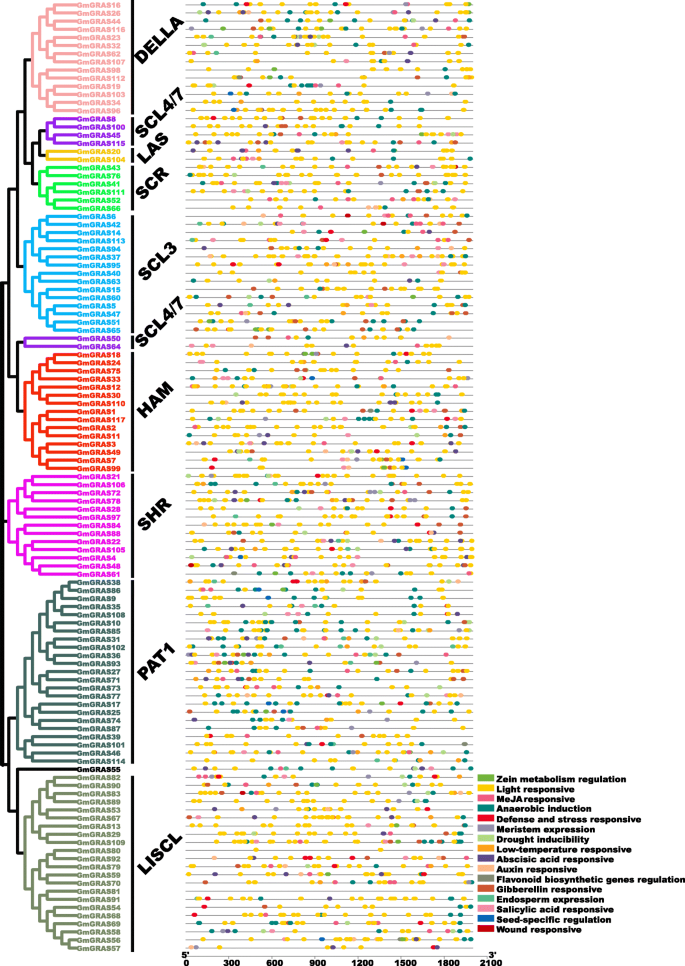
这CIS.-元素在基因表达的转录调控中起重要作用[40].在本研究中,鉴定了2000 bp的上游序列GmGRAS从大豆基因组中提取基因,然后CIS.-Element分析通过使用PlantCare进行(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)(额外的文件10.:表S9)。如图所示。8.和表S9(附加文件10.),16种类型的CIS.-元素在2000 bp的启动子区域获得GmGRAS基因。值得注意的是,CIS.-Elements等光响应性,毒素响应,胃肠杆菌素响应,脱落酸响应,MejA响应,防御和应力响应,干旱诱导性和厌氧诱导广泛分布,这表明了GmGRAS基因对不同的非生物胁迫有不同的反应,调控不同的生物过程。
表达谱的GmGRAS各组织基因与基因表达的相关性分析
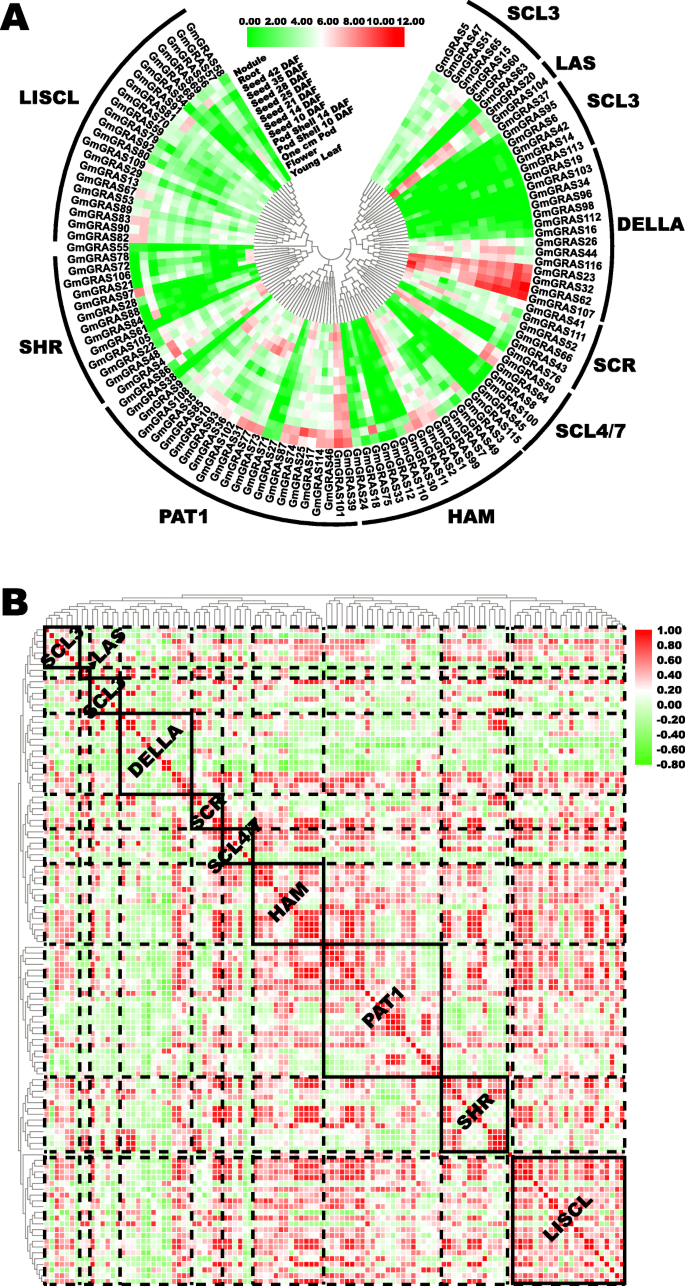
目的:研究GmGRAS本研究提取并分析了大豆不同发育阶段不同组织中基因成员的转录水平GmGRAS种子10 DAF,种子14 DAF,种子21 DAF,种子25 DAF,种子28 DAF,种子35 DAF,种子42 DAF,根和根瘤[41].结果是114GmGRAS基因(除了GmGRAS40那gmgras94和Gmgras117.)被招募了(附加档案11.:表S10)。114的表达数据GmGRAS基因的日志2归一化以描绘的热图GmGRAS不同组织的基因表达谱(图。9.a).根据图。9.,不同的GmGRAS基因亚属植物显示了不同的表达模式。例如,大多数GmGRASLISCL、SHR和SCL3亚家族的基因在所有组织中均表现出低表达,但部分基因(gmgras67那GmGRAS82和gmgras83在lisl亚家族中;GmGRAS97在SHR亚家族中;GmGRAS15和Gmgras65SCL3亚家族)在根和结节中表现出较高的表达水平。值得注意的是,9个基因(GmGRAS39和GmGRAS101PAT1亚家族;GmGRAS11HAM亚家族;GmGRAS50和gmgras64SCL4/7亚家族;GmGRAS23那gmgras32那gmgras62和GmGRAS107在Della subfamily中,在所有组织中呈现高表达,这可能表明在大豆植物发育过程中的重要功能。有趣的是,有两个基因成员(GmGRAS20和GmGRAS104)在Las亚家族中,它们两者在种子的叶子,花,豆荚,根和早期发育阶段的组织中显示出高表达。此外,最重要的是GmGRAS基因与其他组织相比,在根表现出相对高的基因表达。
另外,我们分析并计算了114之间的表达相关系数GmGRAS基因在表S11(附加文件12.).基于相关系数描绘了热图,并且通过不同的Gmgras亚壳进一步聚集了热线图(图。9.b)。如图1所示。9.b,根据GmGRAS亚族,用虚线将相关热图划分为不同的块。因此,显示了不同亚科之间的相关性。为了强调每个GmGRAS亚科成员之间的相互关系,我们用实心框将这些亚科围起来,并用粗体标注它们的名称。大多数GmGRAS基因显示与内部或外部亚属的成员的正相关。虽然,Della Subfamily中的相当大的Gmgras成员倾向于与其他Gmgras成员相对独立或呈现负相关性。总的来说,这些结果表明了GmGRAS基因可以广泛地相关,并且在大豆组织变化。
表达谱的GmGRAS盐水和脱水应力和基因表达相关分析中的基因
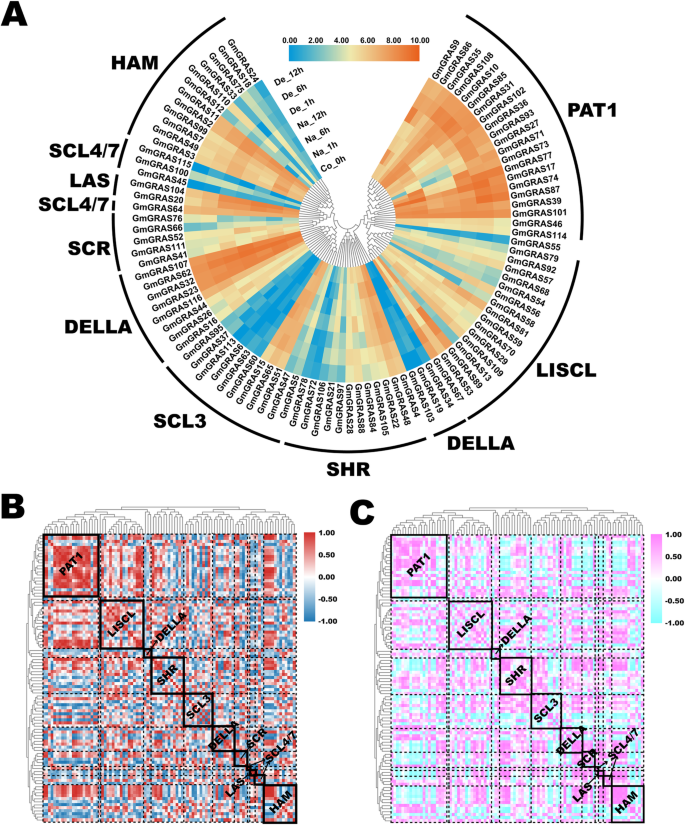
先前的研究报道肝基因广泛地对各种非生物压力作出反应[4.那19.那42].在本研究中,我们探讨了GmGRAS盐和脱水胁迫下大豆根的基因表达谱[j]。43].结果确定了95名成员GmGRAS基因提取,相关表达信息列于表S12(附加文件)13.).基于日志2标准化表达式数据,热图被描述(图。10.一种)。如图所示,在非生物应激期间存在四次点(0h,1 h,6 h和12h)。和亚属含有高表达和低表达GmGRAS基因。特别是,大多数GmGRASPAT1亚家族的基因表达水平较高。一般来说,属于同一遗传支系的基因在生理盐水和脱水胁迫下表现出相似的高或低转录水平。
此外,我们在表S13中进行了生理盐水和脱水胁迫下的基因表达相关性分析(附加文件)14.)和表S14(附加文件15.).根据获取的相关系数矩阵,构建两张相关热图(图2)。10.b和无花果。10.c).总体而言,无论是在生理盐水胁迫还是在脱水胁迫下,GmGRAS内部或外部亚家族成员之间普遍存在正相关和负相关,且呈交错关系。然而,在生理盐水和脱水胁迫下,基因表达的相关模式明显不同。例如,在生理盐水和脱水胁迫下,PAT1和SCL3亚家族的GmGRAS成员表现出不同的相关模式。总之,是不同的GmGRAS基因成员在生理盐水和脱水胁迫下广泛相关和不同表达。
表达谱的GmGRAS种子萌发过程中豆豆胚胎轴的基因和基因表达相关分析
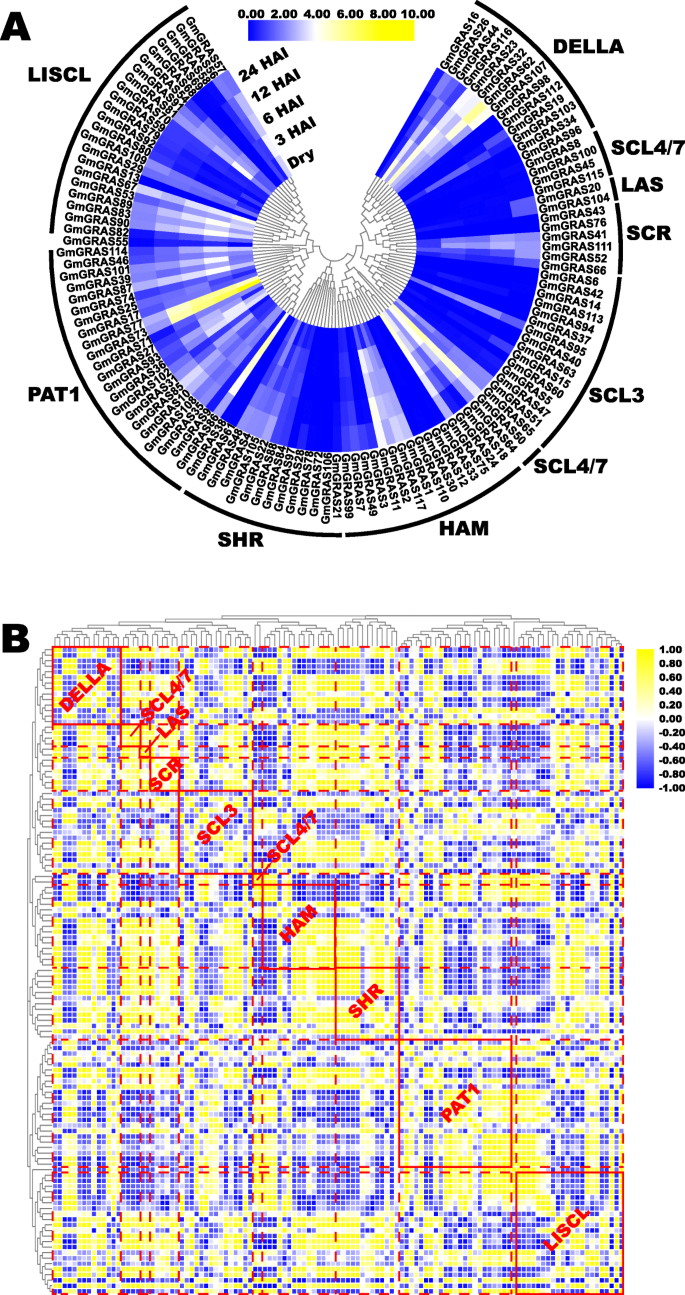
种子萌发是影响作物产量和质量的至关重要。高活力大豆胚胎轴对于确保萌发率是必不可少的[44].以往的研究对大豆胚轴的研究非常关注,从不同角度论证了其在大豆种子萌发中的作用[45那46].此外,肝家族基因也深刻地影响了种子的萌发[15.].因此,我们分析了相关的表达谱GmGRAS大豆萌发过程中的基因(附加文件16.:表S15) [47].提取的表达数据为Log2归一化,构建基因表达热图。11.a).如图所示。11.a,对5个时间点(干燥、3-HAI(吸胀后小时)、6-HAI、12-HAI和24-HAI)的大豆萌发过程进行了研究。值得注意的是,一些大豆肝基因具有相对高的基因表达和显示时间偏好。例如,gmgras73和GmGRAS77在Pat1中,亚家草在24-hai的前四次点和低表达中显示出高转录水平。相比之下,表达gmgras62在Della亚家族中连续上调并在24海中表达高表达。此外,进一步利用所提取的表达数据来构造热图,该热图是用零一对方法划分的,以显示每个的表达式模式GmGRAS基因在种子萌发过程中的作用(附加文件17.:图S2)。一般来说,不同GmGRAS基因成员表现出各种表达模式。
同时,我们进行了表S16(附加文件)中基因表达相关性分析18.),并根据基因表达的相关系数构建热图(图。11.b)。如图1所示。11.b,将热图进行系统聚类,用红色虚线将热图划分为不同的亚基,以显示不同GmGRAS亚科成员之间的相关性。此外,特定亚科的成员在红色实心框中突出显示,并以各自的亚科名称标注。总的来说,GmGRAS在不同的GmGRAS亚家族中,基因内外均呈正相关或负相关。综上所述GmGRAS在大豆种子萌发期间,基因广泛相关,不同地表达。
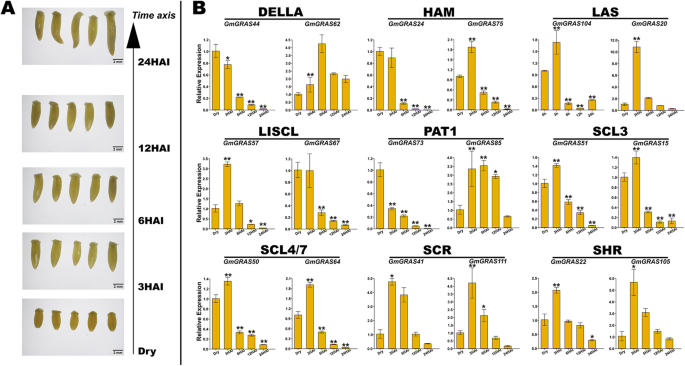
大豆种子萌发测定和代表的定量RT-PCR分析GmGRAS基因
在大豆种子萌发试验中,从每个时间点出发,用图中的时间轴排列五个具有代表性的大豆胚轴。12.一种。采样的大豆胚胎轴进行RNA提取和定量RT-PCR实验。进一步调查潜在的调节作用GmGRAS大豆种子萌发过程中的基因,18个代表GmGRAS基因,其表达水平在不同时间点相对较高(附加文件16.表S15),均是从已鉴定的大豆中精心挑选的肝基因。所选基因包含两个表达模式不同的基因成员(附加文件)17.:从每个图S2)。GmGRAS基因亚家族覆盖不同表达的GmGRAS作为代表的基因尽可能。代表性的特定引物GmGRAS用于定量RT-PCR测定的基因是表S17中的列表(附加文件19.).根据定量RT-PCR实验的结果,所选基因在不同的基因GmGRAS基因亚家族倾向于表现出不同的表达模式(图。12.b)然而,在某些亚家族中选择的基因表现出相似的表达模式。例如,的表达水平GmGRAS41和gmgras111在SCR亚家族以及GmGRAS22和GmGRAS105在SHR亚家族中,从0-HAI(干)上调至3-HAI,从6-HAI逐渐下调至24-HAI。此外,同一亚家族也存在不同的基因表达模式。例如,的表达式级别GmGRAS44在DELLA亚家族中,从0-HAI连续下调至24-HAI。相比之下,表达水平gmgras62从0-HAI上调至6-HAI,从12-HAI上调至24-HAI。因此,代表性基因的表达模式可以概括其多样性GmGRAS基因表达倾向,其中突出的调控GmGRAS种子萌发过程中大豆胚轴的基因。
讨论
GRAS蛋白是植物特异性的,并在不同的植物发育和生理过程中起重要作用。最近,通过全基因组测序技术的快速发展,逐渐实现了不同物种中GRAS成员的基因组鉴定。然而,关于大豆的相关研究肝基因家族仍然缺乏。在本研究中,我们对大豆GRAS转录因子进行了系统发育关系、基因结构、保守结构域、基序模式、基因染色体分布、基因复制、同源性分析和基因进化分析等方面的研究。此外,表达模式GmGRAS并对不同组织、盐碱和脱水胁迫下以及种子萌发过程中的基因进行了研究。
在这次调查中,117GmGRAS基因被确定。大豆中GRAS成员数量超过拟南芥(32个)[8.],蓖麻籽(48个部件)[25.]、番茄(53个成员)[48]及食米局(60名委员)[8.].这一结果也与物种的基因组大小相平行,这可能表明特定基因组大小与GRAS成员数量之间存在正相关关系。在AtGRAS分类的基础上,系统发育分析将GmGRAS蛋白分为9个亚家族(图。1).分类在同一基本家族或思工中的GRAS成员可能表明它们在不同物种中的类似功能。
以前的研究表明,植物GRAS系列来自原核基因组并水平转移以及基因重复[49].大豆是一种古老的多倍体,具有高度复制的基因组,大约75%的基因存在于多个副本中[50].根据表S6(附加文件7.),除了串联复制外,GmGRAS家族中相当多的成员来源于节段复制事件。我们的研究结果证实了片段复制和串联复制都是扩增的重要因素肝基因家族。此外,内含子被推测在植物进化中至关重要,它们倾向于在基因扩展的早期阶段产生,然后随着时间的推移逐渐消失[51那52].然而,也有例外,内含子在进化过程中可能更大,被认为是获得新基因功能的必要途径[19.那53].如图1所示。2发现,大多数GmGRAS基因成员缺乏内含子,其平行于关于保守特征的早期报告肝基因(4.那7.].值得注意的是,大多数PAT1亚家族成员(16个成员)在5 ' uts(非翻译区域)中都有内含子,这可能表明PAT1亚家族是大豆基因组中一个新进化的GRAS分支。GRAS蛋白的特定结构域或基序确保了蛋白质相互作用和DNA结合修饰的特征[54].在本研究中,我们将模因基序综合分为GRAS蛋白c端5个保守基序(图2)。3.b和无花果。3.c). 5个保守的c端基序中的模因-基序在不同的亚家族中波动,这可能反映了不同GmGRAS亚家族成员的不同生物学功能。总之,GmGRAS成员之间结构的一致性和差异性可以直接或间接地说明其功能的相似性和差异性。
在这次调查中,确认了GmGRAS基因不均匀地分布在20条染色体和一个大豆基因组支架上(图。4.).值得注意的是,CHR11包含最多GmGRAS16个基因,主要来源于DELLA、HAM、lisl、SCR、SHR、SCL3和SCL4/7亚家族(Additional file)1:表S1)。在早期的研究中,Sun等人。精心总结植物GRAS结构域的功能多态性:Della结构域与转录共激活,光信号,胶酸或茉莉酸信号传导,非生物应激的积分器,植物素和乙烯信号相关。火腿结构域与染色信号传导相关,转录共激活响应植物蛋白,拍摄公司维护;Liscl结构域响应于不同信号与转录激活或共激活相关;SCR域参与了根或射线径向图案;SHR结构域与转录调节,根或射线图案化和染色信号传导相关联。SCL3结构域可以与Della和ShR / SCR作用,以介导根内胚层中的细胞伸长率;SCL4 / 7结构域是响应环境压力的转录调节因子[55].此外,在16个基因中,GmGRAS48从shr subfamily,GmGRAS50来自SCL4/7亚家族,GmGRAS51来自SCL3亚家族,Gmgras52.来自SCR亚家族gmgras53和gmgras56.来自liiscl子族和gmgras62来自DELLA亚家族的基因在多个组织或器官的不同发育阶段均表现出较高的基因表达(附加文件)11.:表S10)。综上所述,我们推测一些关键GmGRASCHR11上的基因可能会对调节大豆植物生长和发育的潜在作用。
为了探讨不同物种间GRAS成员的进化关系,我们招募了4个双子叶植物和2个单子叶植物,并进行了同源性分析。结果,大豆和甘氨酸Soja.表现出最佳的正相关。相比之下,在大豆和奥雅萨苜蓿在本研究中进行了最弱的正相关。总的来说,双子叶植物的GRAS成员与鉴定出的GmGRAS成员表现出比单子叶植物更好的同源性(图2)。6.).因此,我们推测GRAS构件之间的同步相关性可以与物种的进化分歧联系起来。值得注意的是,发现了19个Gmgras成员与遍布多种物种的GRAS构件同步(图。7.a和附加文件9.表S8),表明这些同源对是保守的,可能在祖先分化之前就存在[35].因此,不同物种间同源GRAS成员的交叉可能对开展GRAS进化的相关研究具有重要价值。
据报道,GRAS成员广泛参与调节植物发育和应激反应[4.那19.].在本研究中,我们分析了CIS.-元素的启动子区域GmGRAS基因。如图1所示。8.那CIS.- 与植物生长和发展相关联的 - 诸如光响应元件,蟾蜍敏敏元素,分素表达元素的促进。我们还检测到非生物应力相关CIS.-防御和胁迫反应元件、干旱诱导元件、低温反应元件和创伤反应元件。因此,CIS.-elements分析提供的基因功能研究的线索,尤其是对相关的基因,以不同的应力响应和调节植物的发展。
寻求的表达模式GmGRAS不同组织中的基因,我们提取并分析鉴定的转录水平GmGRAS不同组织中的基因[41].作为一个整体,大多数GmGRAS基因处于相对较低的转录水平,同一基因亚家族中相当多的基因表现出相似的表达模式(图2)。9.一种)。值得注意的是一些GmGRAS基因,像GmGRAS39和GmGRAS101在整个PAT1组织和发育阶段亚科显示高的基因表达水平。这些GmGRAS基因可能是至关重要的,并广泛参与大豆植株发育过程。此外,一些GmGRAS基因,像GmGRAS20和GmGRAS104在LAS亚家族中表现出组织或发育阶段偏好,这突出了时空规定GmGRAS大豆生长发育过程中的基因。此外,根据图中的基因表达相关性分析。9.B,大多数GmGRAS基因间正相关,说明GmGRAS基因在相关调控中广泛合作。此前,人们对不同植物中的DELLA亚家族成员进行了广泛的研究,它们是GA (gibberellic acid)信号转导途径的关键负调控因子,抑制植物的生长发育[56那57].在这项研究中,大多数GmGRASDella Subfamily的基因显示与其他人的负相关GmGRAS基因或显示的相对独立性。相关联的与图1中的DELLA亚家族基因的基因表达概况。9.a,我们推测DELLA亚家族中的几个关键基因可能持续表达并调控大豆的生长发育。总的来说,确定GmGRAS基因在大豆植株发育中具有潜在的调控功能。
GRAS成员与调节其生化活动以应对非生物压力有关[12.].在本研究中,研究了一些GmGRAS成员在生理盐水和脱水应激中的作用[43].重要的是,相当大的GmGRAS基因在不同GmGRAS基因亚脲受到盐水和脱水应力的影响。作为一个整体,所识别的应力反应(高或低基因表达水平)GmGRAS基因在盐水和脱水应力期间呈现相似之处(图。10.A),这可能突出关键的作用GmGRAS响应非生物胁迫的基因。例如,大多数GmGRAS在生理盐水和脱水胁迫下,PAT、LAS和DELLA亚家族的基因呈现高表达(图2)。10.a).值得注意的是,DELLA蛋白被报道在调控植物的抗逆性中起重要作用[57],我们的调查进一步验证了以前的研究结果。然而,盐水和脱水应力期间的基因表达相关图案明显不同(图。10.b和无花果。10.c).因此,我们推测功能性GmGRAS基因可以在不同机制下对盐水和脱水应力进行广泛配合。
特别地,我们研究了GmGRAS种子萌发过程中豆豆胚胎轴的基因[47].有趣的是,大多数GmGRAS基因表达相对较低。然而,也有一些高度表达GmGRAS不同GmGRAS亚家族的基因(图。11.)。和GmGRAS基因在种子萌发过程中显示不同的表达模式(附加文件17.:图S2)。重要的是,如图所示。11.B,截然不同GmGRAS基因普遍相关,说明其功能GmGRAS基因可以广泛地相互作用和协调一致影响大豆种子发芽。此外,我们选择了18代表GmGRAS并进行RT-PCR定量分析。与报道的表达模式进行比较GmGRAS图S2中的基因(附加文件17.),GmGRAS24那GmGRAS41那GmGRAS44那GmGRAS50那GmGRAS51那gmgras57.那gmgras64那gmgras67那gmgras73和GmGRAS75经定量RT-PCR验证,结果基本一致(图2)。12.b)。然而,GmGRAS15那GmGRAS20那GmGRAS22那gmgras62那gmgras85那GmGRAS104那GmGRAS105和GmGRAS104从报告的转录组数据的那些显示不同的表达式模式[47].例如,在转录组数据中,转录水平gmgras62从0-hai到24-hai逐渐上调(附加档案17.:图S2)。然而,在定量RT-PCR检测结果中,表达水平gmgras62首先从0-hai到6-hai上调,然后从6-hai到24-hai下调(图。12.b)。对于目前研究中的大豆种子Gemination测定,与早期研究相比,我们添加了种子预制作过程[47],加速大豆种子吸收。这可能导致前面的相关表达GmGRAS基因。另一个可能的原因可能是本研究中大豆品种间的差异。“威廉姆斯82”)和之前的研究(cv。BRS 284) (47].总之,GmGRAS基因在大豆种子萌发过程中具有潜在的调控作用。
本研究提供了系统调查肝大豆的基因,可能有利于进入其生物学功能的见解。然而,目前的研究仅提供了初步表征GmGRAS应该进行基因和进一步的功能验证,以了解不同的角色GmGRAS各种生物过程中的基因。
结论
在这项研究中,我们确定了117GmGRAS基因大豆。这些GmGRAS在大豆基因组中位于20染色体上的基因和一个支架。所识别的Gmgras成员进一步分为九个Gmgras亚属。基因结构分析最多GmGRAS基因缺乏内含子,说明GmGRAS基因高度保守。保守结构域和基序模式分析表明,GRAS成员在同一亚家族或分支中表现出广泛的相似性,这可能表明它们的基因功能是平行的。而且,新事物的出现GmGRAS基因主要由基因复制驱动,其中节段复制事件起主导作用GmGRAS基因家族扩张。除此之外,CIS.-元件和基因表达分析揭示了所鉴定的潜在调控GmGRAS在不同的组织,在盐和脱水胁迫和大豆种子萌发过程中的基因。综上所述,我们全面研究了其特点肝大豆基因,结果为了解基因生物学功能和未来研究提供了有价值的线索GmGRAS基因。
方法
大豆中GRAS家族成员的开采
用于大豆(soybean)中GRAS基因的鉴定Wm82.a2.v1基因组版本),我们获得的所有AtGRAS蛋白序列从TAIR数据库查询序列(https://www.arabidopsis.org/)[4.那58].大豆基因组和基因组注释文件从植物血统v12下载。1.6数据库(https://phytozome.jgi.doe.gov/pz/portal.html).基于这些数据,我们利用TBtools软件提取出最具代表性的GmGRAS成员序列[59], GmGRAS蛋白序列在NCBI蛋白数据库中通过BLASTp (https://blast.ncbi.nlm.nih.gov/Blast.cgi?程序= blastp&page_type = blastsearch&link_loc = blasthome)。对于域组成分析,我们使用了NCBI保守的域数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi).手动删除缺乏GRAS相关域的蛋白质。此外,还除去了它们基因长度或小于100AA(氨基酸)的明显误差的蛋白质序列。扩展网站(http://expasy.org/tools/),用于鉴定GmGRAS蛋白的分子量(MW)、等电点(pI)和氨基酸数。为GmGRAS基因亚细胞本地化预测,我们使用大提琴(http://cello.life.nctu.edu.tw/)在线工具。
GmGRAS蛋白的系统发育分析与分类
拟南芥和大豆的GRAS蛋白序列通过使用Mega 7.0的肌肉方法(https://www.megasoftware.net/),使用默认参数。序列对齐后,采用neighbor joining (NJ)方法构建系统发生树,参数如下:Poisson模型、两两缺失和1000次bootstrap重复[35].根据TAIR数据库中AtGRAS亚家族成员的记录,将鉴定的GmGRAS蛋白进一步分类为不同的亚家族(https://www.arabidopsis.org/).改进的系统发育树用FigTree v1.4.3 (http://tree.bio.ed.ac.uk/software/figtree/)和Adobe Illustrator CC 2019(https://www.adobe.com/products/illustrator/free-trial-download.html).
基因结构和保守基序分析
基因结构由TBTOOLS描绘[59]随着大豆基因组的GFF3文件。通过MEMEV5.1.1进行了GMGRAS蛋白的保守基序扫描(http://meme-suite.org/tools/meme.),结果显示20个模因。模因图形的可视化和Seq标志是由TBtools创建的。参照Quan等对GRAS c端保守基序的分类,将鉴定出的模因基序进一步分为特异性LHR I、VHIID、LHR II、PFYRE和SAW基序[34].输出图形经Adobe Illustrator CC 2019修改。
GmGRAS基因的染色体位置,重复和同线性分析
的染色体的位置和复制GmGRAS根据植物血红型的可用大豆基因组信息映射基因,并由TBTOOLS软件显示[59].通过TBTOOLS计算每种染色体或支架的基因密度信息。探索正交结果的同时关系肝此外,我们还下载了大豆和其他物种的基因组数据和基因注释文件拟南芥(TAIR注释版本10),甘氨酸Soja.(v1.1),Vigna Unguiculata.(v1.1),茄属植物lycopersicum(ITAG3.2),奥雅萨苜蓿(MSU注释版本7.0)和高粱二色的来自植物血红制的)(v3.2)。通过在TBTOOLS中使用双向同步绘图仪功能来构建同步分析图。同步的Venn图肝TBtools描述了不同物种的基因。非同义替换(Ka)和同义替换(Ks)的复制肝TBtools进行基因计算。输出图形经Adobe Illustrator CC 2019修改。
CIS.-element分析GmGRAS基因
所鉴定的序列为上游2000 bpGmGRAS基因被TBtools提取[59].然后提取提取的序列提交给Plantcare网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测CIS.-元素在启动子区域。的图CIS.-要点GmGRAS基因由TBtools显示,并由Adobe Illustrator CC 2019修改。
表达谱分析GmGRAS基因
鉴定的转录水平GmGRAS从大豆的转录组数据中检索不同组织中的基因(https://soybase.org/soyseq/)[41],这是由RPKM(每千碱基百万读数)值评估的。的表达式概况GmGRAS通过已发表的研究数据中的RPKM值获得生理盐水和脱水胁迫下的基因[43].转录水平GmGRAS在发芽过程中大豆胚轴基因从Bellieny-雷贝洛等人,这是由该FPKM得分的早期研究提取的值(每千碱基百万片段)[47].的热图GmGRAS基于日志来说明基因表达式2使用TBTOOLS归一化RPKM或FPKM值[59].探讨GmGRAS基因表达相关性,提交并计算了提取的RPKM和FPKM值,并在OMICSHARE在线工具上计算(https://www.omicshare.com/tools/home/soft/getsoft.).此外,相关热图GmGRAS基因表达通过TBtools进行系统聚类分析。输出图形经Adobe Illustrator CC 2019修改。
植物材料和大豆种子萌发测定
本研究以大豆基因组参考品种Williams 82为材料。本研究随机选取3株独立的大豆植株,采收种子,进行3次独立的生物学重复。所有试验大豆种子均于2019年秋季在南京农业大学国家大豆改良中心当土试验站收获。参考前人的研究,我们重点研究了大豆种子萌发的5个时间点:干燥、3-HAI(吸胀后小时)、6-HAI、12-HAI和24-HAI [47].Importantly, we adopted the germination pouches (medium size, 18 cm height and 12.5 cm width, PhenoTrait Technology Co., Ltd.) to conduct the soybean seed germination experiment. For each time point, 40 healthy seeds were selected from the independent soybean plant harvesting seeds. And the seeds were placed in a 40 °C drying oven for 3 days to ensure the uniformity of seed moisture. The selected seeds were then disinfected by 0.05% potassium permanganate solution for 5 min and washed with deionized water. According to the instruction book of the germination pouch, the disinfected soybean seeds were soaked in deionized water for 2 hours to accelerate seed imbibition and ensure seed germination. Next, we placed eight seeds in each germination pouch and added 15 mL deionized water. Then germination pouches were put on the germination pouch shelf and placed in the dark in a temperature-controlled incubator at 25 °C. Embryonic axes were carefully separated from cotyledons for RNA extraction at each time point. Five representative seed embryonic axes at each time point were photographed by using a SPOT-RT digital camera (Diagnostic Instruments, Sterling Heights, MI) and an OLYMPUS SZ61 stereomicroscope (Olympus, Melville, NY, USA).
RNA分离和定量RT-PCR分析
为了验证大豆种子萌发过程中胚轴的表达模式,我们选择了18个有代表性的种子GmGRAS进行定量RT-PCR分析。采用3个独立的生物学重复,包含3个独立的植物进行定量RT-PCR。所选特异性定量RT-PCR引物GmGRAS基因由底漆总理5设计并列于表S17中(附加文件19.).参考Bellieny-Rabelo等人的前一项研究,从每个独立植物的40个胚胎轴上随机选择20个健康的种子胚轴,每次RNA分离的时间点[47].通过使用来自冷冻胚胎轴的RNAPREP纯植物套件(天根,北京)提取总RNA。通过电泳分析所有RNA,然后用Nanodrop Nd-1000分光光度计(Nanodrop,Wilmington,De,USA)量化。采用标记二二链CDNA合成试剂盒(Vazyme Biotech,南京,中国)去除基因组DNA,并将总RNA转化为cDNA [60].采用SYBR QPCR主混音(Vazyme Biotech,南京,中国)对Biorad CFX96实时系统进行定量RT-PCR测定[60].家政服务GmActin基因被确定为内部控制。对每个cDNA样品进行三次定量分析,并用2-△△CT.方法 [61].
统计分析
学生的T.-试验采用Graphpad Prism 8 (https://www.graphpad.com/scientific-software/prism/).测试的差异被确定为有显著P.-值截断0.05。所有误差棒均为独立生物重复的标准差。
数据和材料的可用性
本研究期间产生或分析的所有数据均包含在本文及其附加文件中。
缩写
- GM.:
-
甘氨酸最大
- TFs:
-
转录因素
- 盖:
-
赤霉酸不敏感
- RGA:
-
GA1-3突变体的阻遏因子
- SCR:
-
稻草人
- LHRI:
-
富亮氨酸地区我
- lhrii:
-
富亮氨酸地区二世
- 弗吉尼亚州:
-
血管血管血管
- 在:
-
拟南芥
- 巴:
-
Phytochrome A.
- 太:
-
Medicago Truncatula.
- AA:
-
氨基酸
- MW:
-
分子量
- pI:
-
等电点
- NJ:
-
Neighbor-joining
- 卡:
-
非同义替代
- Ks:
-
同义替换
- RPKM:
-
读每公斤万
- FPKM:
-
碎片每千碱基百万
- 海:
-
小时后自吸
- SD:
-
标准偏差
- 子:
-
开放阅读框
- cd:
-
编码序列
- 装备:
-
染色体
- kb:
-
千轴
- GS.:
-
甘氨酸Soja.
- Vu:
-
豇豆属unguiculate
- Sl:
-
茄属植物lycopersicum
- 某人:
-
高粱二色的
- OS.:
-
奥雅萨苜蓿
- 厘米:
-
厘米
- DAF):
-
几天后开花
- UTRS:
-
未翻译区
参考文献
- 1。
王YX,刘ZW,吴ZJ,李H,王WL,Cui X,壮族基因组鉴定及茶树族族转录因子的表达分析(茶树).SCI批准。2018; 8(1):3949。
- 2。
Ho-PlágaroT,Molinero-Rosales N,FariñaFloresD,VillenaDíazM,García-Garrido JM。鉴定和表达分析肝番茄中丛枝菌根发育中的转录因子基因。前植物SCI。2019; 10:268。
- 3.
风扇,张D,高C,赵M,吴总部,李YM,沈YW,汉族我。的识别、分类和表达分析肝基因家族在Malus Domestica..前面的physiol。2017; 8:253。
- 4.
刘明元,黄玲,马志涛,孙文军,吴强,唐泽忠,布托林,李春林,陈辉肝苦荞基因家族(Fagopyrum tataricum).BMC植物BIOL。2019; 19(1):342。
- 5.
Pysh Ld,Wysocka-Diller JW,Camilleri C,Bouchez D,Benfey Pn。拟南芥的GRAS基因家族:序列表征和基本表达分析稻草人样子基因。植物j . 1999; 18(1): 111 - 9。
- 6.
植物信号转导和发育中GRAS蛋白的作用。足底。2004;218(5):683 - 92。
- 7.
陈勇,朱鹏鹏,吴淑英,卢勇,孙军,曹庆华,李志勇,徐涛肝甘薯野生亲戚的转录因子Ipomoea Trifida..BMC基因组学。2019; 20(1):911。
- 8.
天cg,万p,sun sh,李杰,陈女士。基因组范围分析肝基因家庭在水稻和拟南芥中。植物mol biol。2004; 54(4):519-32。
- 9.
李MH,金B,宋sk,许祖,刘乃良,李史,金M,金DG,孙素,林策,等。大规模分析肝基因家族在拟南芥.植物学报。2008;67(6):659-70。
- 10.
蔡志强,蔡志强。PAT1是GRAS家族的新成员,参与光敏色素的信号转导。基因Dev。2000;14(10):1269 - 78。
- 11.
Greb T,Clarenz O,Schafer E,Muller D,Herrero R,Schmitz G,Theres K.分子分析侧抑制器基因揭示了腋生分生组织形成的保守控制机制。基因Dev。2003;17(9):1175 - 87。
- 12.
牛玉玲,赵婷婷,徐小燕,李建峰。全基因组鉴定和特征肝番茄转录因子(茄属植物lycopersicum).peerj。2017; 5:e3955。
- 13。
李P,张B,苏TB,李PR,鑫XY,王WH,赵XY,俞YJ,张DS,俞SC等人。BrLAS,一个鹅肝从转录因子Brassica Rapa.,与转基因拟南芥的抗旱性有关。植物学报2018;9:1792。
- 14.
Hirsch S,Oldroyd Ged。调节植物开发的GRAS结构域转录因子。植物信号表现。2009; 4(8):698-700。
- 15.
泰勒L,汤姆斯SG,胡俊华,迪尔A,阿隆索JM,小埃克,孙TP。DELLA蛋白和赤霉素调控拟南芥种子萌发和花发育。植物杂志。2004;135(2):1008 - 19所示。
- 16.
HEO JO,Chang Ks,Kim Ia,Lee Mh,Lee Sa,Song Sk,Lee Mm,Lim J.Gibberellin信号传导的Gibberellin Simbering Simprow样3拟南芥根。美国国立科学院。2011, 108(5): 2166 - 71。
- 17.
Yoshida H,Ueguchi-Tanaka M. Della和SCL3通过利用不确定的域蛋白作为转录支架,通过不确定的域蛋白来平衡胃肠杆菌蛋白的反馈调节。植物信号表现。2014; 9(9):E29726。
- 18.
崔春生,Levesque MP, Vernoux T, Jung JW, Paquette AJ, Gallagher KL, Wang JY, Blilou I, Scheres B, Benfey PN。一种进化上保守的机制界定了SHR运动在植物中的单层内皮层。科学。2007;316(5823):421 - 5。
- 19.
张波,刘杰,杨泽,陈安英,张昌杰,张学勇,李方成。猪GRAS转录因子基因家族的全基因组分析陆地棉L. BMC基因组学。2018; 19(1):348。
- 20.
袁益,方立立,卡鲁戈斯克,张立,高益,李世,鑫惠普。过度表达VaPAT1,一个鹅肝从转录因子血管血管血管,赋予拟南芥抗非生物胁迫能力。植物学报2016;35(3):655-66。
- 21.
GRAS蛋白SCL13是光敏色素依赖性红光信号的正调节蛋白,但也可以调节光敏色素的反应。基因组学杂志。2006;276(1):13-30。
- 22。
Hirsch S, Kim J, Muñoz A, Heckmann AB, Downie JA, olddroyd GED。在结瘤信号转导过程中,GRAS蛋白形成DNA结合复合物诱导基因表达Medicago Truncatula..植物细胞。2009; 21(2):545-57。
- 23。
关键词:茎分生组织肝-来自分化细胞的基因介导的信号。基因Dev。2002;16(17):2213 - 8。
- 24。
刘学勇,王伟民。中国猪GRAS基因家族的全基因组比较分析杨树那拟南芥和米饭。植物mol biol报告。2014; 32(6):1129-45。
- 25。
徐伟,陈志新,Ahmed N,韩波,崔庆华,刘雅娜。水稻全基因组鉴定、进化分析及胁迫响应肝蓖麻的基因家族。中华医学杂志。2016;17(7):1004。
- 26.
郭YY,吴HY,李X,李青,赵XY,段XQ,一个YR,吕W,一个HL。鉴定及玉米GRAS家族基因表达(Zea Mays.l .)。《公共科学图书馆•综合》。2017;12 (9):e0185418。
- 27.
王L,Cheng Yb,Ma QB,Mu Yh,Huang Zf,夏QJ,张Gy,Nian H. QTL精细映射大豆(甘氨酸最大两个ril居群的叶型相关性状。BMC基因组学。2019;20(1):260。
- 28.
Bencke-Malato M, Cabreira C, Wiebke-Strohm B, Bücker-Neto L, Mancini E, Osorio MB, Homrich MS, Turchetto-Zolet AC, De Carvalho MCCG, Stolf R, et al.;大豆WRKY家族的全基因组注释和响应相关基因的功能特征Phakopsora pachyrhizi.感染。中国生物医学工程学报。2014;14(1):236。
- 29.
杜H,杨SS,梁Z,冯谢,刘L,黄烨,唐yx。大豆中超法的基因组分析。BMC植物BIOL。2012; 12:106。
- 30.
Le DT, Nishiyama R, Watanabe Y, Mochida K, Yamaguchi-Shinozaki K, Shinozaki K, Tran LSP。大豆发育和脱水胁迫过程中植物特异性NAC转录因子家族的全基因组调查和表达分析DNA研究》2011;18(4):263 - 76。
- 31.
陈X,陈泽,赵立,赵y,程富,湘玉。盐度和干旱处理下的大豆HD-ZIP基因家族的基因组自分析。Plos一个。2014; 9(2):E8715。
- 32.
Ha CV, Le DT, Nishiyama R, Watanabe Y, Sulieman S, Tran UT, Mochida K, Dong NV, Yamaguchi-Shinozaki K, Shinozaki K,等。大豆生长素反应因子转录因子家族:发育和水分胁迫过程中的全基因组鉴定和表达分析。DNA研究》2013;20(5):511 - 24。
- 33.
范春梅,王旭,王永文,胡荣斌,张学明,陈建新,付云飞。大豆MADS基因的全基因组表达分析显示其在种子发育中的潜在功能。《公共科学图书馆•综合》。2013;8 (4):e62288。
- 34.
Quan SW,NIU JX,周L,XU H,MA L,Qin Y.基因组鉴定,分类,表达和重复分析肝家庭基因胡桃regia中国科学(d辑:地球科学);
- 35。
谢涛,陈昌华,李超,刘建荣,刘春英,何玉华。菠萝WRKY基因家族的全基因组研究:发育和胁迫过程中的进化和表达谱。BMC基因组学。2018;19(1):490。
- 36。
Holub EB。在野生植物拟南芥(Arabidopsis)中,军备竞赛早已成为历史。[j] .地理科学进展,2001;2(7):516-27 .]
- 37。
引用本文:王志刚,王志刚,王志刚。基因复制在大基因家族进化中的作用[j]拟南芥.植物学报。2004;4(1):10。
- 38.
朱y,吴妮,歌曲wl,尹gj,秦yj,yan ym,hu yk。大豆(甘氨酸最大) expansin基因超家族起源:在亚家族中发生的节段性和串联复制事件导致了分化选择。植物学报。2014;14(1):93。
- 39.
于军,王军,林伟,李绍成,李华,周军,倪px,董伟,胡森,曾春强,等。的基因组奥雅萨苜蓿:复制的历史。公共科学图书馆杂志。2005;3 (2):e38。
- 40。
王W,江W,刘Jg,李义,葛jy,李玉。大豆醛脱氢酶基因的基因组鉴定及其在干旱胁迫反应中的潜在作用。BMC基因组学。2017; 18(1):518。
- 41.
Severin AJ, Woody JL, Bolon YT, Joseph B, Diers BW, Farmer AD, Muehlbauer GJ, Nelson RT, Grant D, Specht JE,等。RNA-Seq阿特拉斯的甘氨酸最大:大豆转录组指南。BMC Plant Biol. 2010;10:160。
- 42.
单志宇,罗新林,吴美美,魏利明,范正平,朱玉梅。全基因组鉴定和表达肝木薯的基因家族成员。植物学报。2020;20(1):46。
- 43.
大豆HD-zip转录因子家族的综合表征和RNA-Seq谱分析(甘氨酸最大)在脱水和盐胁迫下。BMC基因组学,2014;15(1):950。
- 44.
在吸胀和以下发芽大豆胚轴生育酚含量 - Simontacchi男,卡洛A,弗拉加CG,Puntarulo S.氧化应激影响α。植物理性。1993; 103(3):949-53。
- 45.
Gazara RK, de Oliveira EAG, Rodrigues BC, Nunes da Fonseca R, AEA O, Venancio TM。大豆的转录景观(甘氨酸最大)在Paclobutrazol的萌芽期间胚胎轴,胃蛋白酶生物合成抑制剂。SCI批准。2019; 9(1):9601。
- 46。
Sangi S, Santos MLC, Alexandrino CR, Da Cunha M, Coelho FS, Ribeiro GP, Lenz D, Ballesteros H, hememerly AS, Venâncio TM,等。大豆胚轴萌发过程中的细胞壁动态及基因表达。足底。2019;250(4):1325 - 37。
- 47。
Bellieny Rabelo D,De Oliveira Eag,Ribeiro Es,CostaEP,Oliveira Aea,Venancio TM。转录组分析在发芽过程中揭示了大豆胚胎轴的关键调节和代谢方面。SCI REP。2016; 6:36009。
- 48。
黄伟,冼志强,康旭,唐宁,李志刚。全基因组鉴定、系统发育和表达分析肝番茄的基因家族。BMC Plant Biol. 2015;15:209。
- 49。
细菌GRAS结构域蛋白揭示赤霉素的反应机制。生物信息学,2012,28(19):2407 - 11所示。
- 50。
Schmutz J, Cannon SB, Schlueter J, Ma JX, Mitros T, Nelson W, Hyten DL, Song QJ, Thelen JJ, Cheng JL, et al.;古多倍体大豆的基因组序列。自然。2010;463(7278):178 - 83。
- 51。
剪接体内含子的进化:模式、谜题和进展。中国农业科学。2006;7(3):211-21。
- 52。
Roy SW,Penny D.在内含内的硅藻中的一个非常高的内含子位置这种pseudonana显示广泛的内含子增益。中国生物医学工程学报。2007;24(7):1447-57。
- 53。
Iwamoto M, Maekawa M, Saito A, Higo H, Higo K.从外显子-内含子结构推断植物过氧化氢酶基因的进化关系:单子叶和双子叶分离后的同工酶差异。应用计算机学报1998;97(1):9-19。
- 54。
刘少麟,白MJ,麦克雷TH。高等植物的转录因子及其基因。acta Biochem. 1999;262(2): 247-57。
- 55.
Sun XL, Xue B, Jones WT, Rikkerink E, Dunker AK, Uversky VN。在植物发育过程中,GRAS蛋白的n端结构域在分子识别中起着重要作用。acta botanica sinica(云南植物学报). 2011;47(3):457 - 461。
- 56.
王艳娟,邓东东。GA-GID的分子基础及进化模式1德拉管理模块。中国生物医学工程学报。2014;29(1):1 - 9。
- 57.
植物生长的刹车:气体如何关闭DELLA蛋白。中国生物医学工程学报。2009;60(4):1085-92。
- 58.
刘志军,郑涛,方建国。葡萄多半乳糖醛酸酶(PG)和果胶甲基酯酶(PME)基因的全基因组鉴定和表达谱分析(葡萄l .)。中华医学杂志。2019;20(13):3180。
- 59。
TBtools -一种用于大生物数据交互分析的集成工具。摩尔。2020;13(8):1194 - 202。
- 60。
李超,刘学勇,阮华,张建勇,谢凤峰,盖建勇,杨树平。gmwrky45增强转基因拟南芥对磷酸盐饥饿和盐胁迫的耐受性,并改变其育性。Front Plant Sci. 2020;10:1714。
- 61。
Livak KJ, Schmittgen TD。利用实时荧光定量PCR和2−PCR方法分析相关基因表达数据ΔΔCT方法。方法。2001;25 (4):402 - 8。
致谢
不适用。
资金
国家重点研发计划项目(no . 2016YFD0101500, no . 2016YFD0101504);中央高校基本科研业务费专项资金资助(no . KYT201801);长江学者与创新团队资助项目(no . PCSIRT_17R55)。
作者信息
从属关系
贡献
Spy和LW构思和设计了这项研究。LW进行了数据分析,实验,并完成了原稿。LW,XLD和YQG进行了定量RT-PCR分析。LW和Spy写道并修改了手稿。所有作者都阅读并批准了最终手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:表S1
.117确定GmGRAS本研究中的基因。
附加文件2:表S2
.鉴定的大豆的编码序列和蛋白质序列肝基因成员。
附加文件3:表S3
.相应的名称为32AtGRAS在拟南芥的基因。
附加文件4:表S4
.从MEME网站分析了大豆GRAS蛋白的主题。
附加文件5:图S1
.用于鉴定的Gmgras蛋白的20 meme-icif的SEQ标志。
附加文件6:表S5
.大豆基因组每条染色体或支架的基因密度。
附加文件7:表S6
.串联和分段复制GmGRAS基因对。
附加文件8:表S7
.一对一的正非关系肝大豆和其他六种物种的基因成员。
附加文件9:表S8
.非冗余GmGRAS与大豆和其他六种物种之间的同步关系相关的基因ID。
附加文件10:表S9
.CIS.-元素分析GmGRAS基因启动子区域。
附加文件11:表S10
.表达谱的GmGRAS在不同发育阶段的多个组织中的基因。
附加文件12:表S11
.两两相关系数不同表示GmGRAS不同组织中的基因。
附加文件13:表S12
.表达谱的GmGRAS脱水和盐胁迫期间大豆根系中的基因。
附加文件14:表S13
.两两相关系数不同表示GmGRAS盐胁迫下的基因。
附加文件15:表S14
.两两相关系数不同表示GmGRAS基因在脱水应激。
附加文件16:表S15
.表达谱的GmGRAS种子萌发过程中大豆胚轴的基因。
附加文件17:图S2
.表达的系统聚类表达模式GmGRAS基于已报道的转录组数据的大豆胚轴萌发过程中的基因。用0 - 1方法对FPKM值进行行缩放,以显示每个表达式的模式GmGRAS基因在种子萌发过程中的作用。
附加文件18:表S16
.两两相关系数不同表示GmGRAS种子萌发过程中大豆胚轴的基因。
附加文件19:表S17
.本研究中使用的引物的序列。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
王,L.,丁,X.,高,Y.等等。全基因组鉴定和特征肝大豆基因(甘氨酸最大).BMC植物杂志20.415(2020)。https://doi.org/10.1186/s12870-020-02636-5
收到了:
公认:
发表:
关键词
- 大豆
- 肝
- 全基因组
- 进化分析
- 表达模式
- 盐和脱水应力
- 种子萌发