抽象的
背景
高加索三叶草(三叶草ambiguum是一种根茎性强,低冠的多年生豆科和地被草。本种可作为观赏植物使用,由于发育良好的地下根茎系统和强大的克隆繁殖能力,可抵抗寒冷、干旱温度和放牧。然而,对白花三叶草根茎系统发育的转录后机制尚未进行全面的研究。此外,该植物的参考基因组尚未发表,这限制了对该植物许多重要生物学过程的进一步探索。
结果
我们采用PACBIO测序和illumina测序,以鉴定五种组织中的差异表达基因(DEG),包括TAPROOT(T1),水平根茎(T2),根茎肿胀(T3),根茎芽(T4)和根茎芽尖(T5)组织,在高加索三叶草根茎中。总共获得了19.82 GB清洁数据,并分析了80,654个非冗余成绩单。另外,我们鉴定了78,209个开放阅读帧(ORF),65,227个编码序列(CDSS),58,276个简单序列重复(SSR),6821替代剪接(AS)事件,2429长的非分量RNA(LNCRNA)和4501个推定转录因子(TFS)从64个不同的家庭。与其他组织相比,T5表现出更多的次数,T5中的共同上升基因主要被引入苯丙醇丙烷生物合成。我们还确定了甜菜碱醛脱氢酶(BADH)作为一个高表达基因特异性的T5。结合转录因子和生理指标的加权基因共表达网络分析(WGCNA),发现11个hub基因(MEgreen-GA3)在T3中表达上调,其中3个hub基因属于HB-KNOX家族。我们分析了276个参与激素信号转导的deg,其中与生长素(IAA)信号通路相关的基因最多,在T2和T5中显著上调。
结论
该研究有助于我们对五种不同组织的基因表达的理解,并为高加索三叶草的根茎生长和发育提供初步洞察。
背景
高加索三叶草(三叶草ambiguum,也被称为库拉三叶草,是一种低冠,强根状多年生豆科植物[1];原产于高加索地区的俄罗斯、土耳其东部和伊朗北部[2].白种三叶草作为一种开花植物和观赏植物,可以保护草坪[3.].与白三叶草相比,白三叶草具有较低的纤维浓度、较高的蛋白质浓度和饲料消化率[4]优于高铝土壤[5]在白三叶草增长贫困时,在年内提供高质量的饲料[6].高加索三叶草有深的,半木质的,通常是分枝的主根,许多分枝的根生长出新的植株,在茎的末端或节上[7,8,9].物种可以耐受牛的连续放牧(牛)[7,冬季极端气温[8],季节性水分缺陷和许多影响其他类型三叶草的严重疾病[9,10.].这些特征归因于其突出的主要根部,低展冠和发达的根茎系统[11.].
在没有参考基因组的植物中,高通量RNA测序(RNA-SEQ)技术已成为掌握研究目的的植物分子机制的快速有效方法[12.,13.,14.].该技术已应用于各种根状茎植物的研究,如高粱(高粱霍尔德和高粱propinquum)[15.,16.], 竹子 (早)[17.],oryza longistaminata.[18.,19.,20.],木贼属hyemale[21.],Panax Ginseng.[22.],芦苇南极光[23.],热带莲花(Nelumbo Nucifera)[24.), CangZhu (白术lacea)[25.],当归chuanxiong[26.], 生姜 (Zingiber Officinale.)[27.),而miscanthus lutarioriparius[12.].研究表明,植物根茎富含生长调节剂,其与根茎发育的能量,新陈代谢和激素途径有关。例如,属于BHLH,YABBY,NAM,TCP,故事和AP2系列的48个重要的转录因子(TFS)特异性地或在射尖和伸长区域的丰富oryza longistaminata.[18.,19.,20.].
近年来,越来越多的全长转录组被太平洋生物科学(PacBio)测序产生。PacBio测序是一种单分子测序技术,读长比二代测序长,平均读长可达15 KB,又称单分子实时(single-molecule, real-time, SMRT)。该技术不需要装配,可以完全保留RNA的3 '到5 '末端的整个序列,但错误率较高;此外,第二代逐步方法可以纠正错误[28.,29.,30.,31.].PacBio已被用于检测超过42280种不同的剪接亚型,并发现大量的替代剪接(AS)事件与根茎系统相关,并协助果园草基因组注释[32.,33.].结果表明,后术中规例在根茎系统中起着重要作用。此外,Illumina和Pacbio测序的组合适用于各种根组织,特别是Periderm,提供了丹参的更完整的视图(丹参)转录组[34.].PACBIO测序和RNA-SEQ分析也被用来沿着从拍摄顶点到第五个专区的发育梯度鉴定差异表达的转录物杨树P .摘要×P。euramericana简历.'nanlin895',显示15,838个差异表达的转录物,其中1216个是TFS [35.].
与传统牧草豆类相比,如白三叶草和红三叶草,根茎是白种人三叶草最独特的特征之一[9,36.].根茎系统在储能,运输和营养繁殖中具有重要功能[37.,38.,39.,40].我们结合Illumina测序和PacBio测序,通过分析高加索三叶草5个不同组织中的基因表达,并鉴定了高加索三叶草根茎发育的相关基因,构建了一个完整的高加索三叶草转录组。这些结果为进一步研究其在根茎分化和生长发育中的作用提供了良好的参考。
结果
PACBIO测序数据集分析
完成了高加索三叶草根茎的转录组测序,使用一个细胞获得了19.82 GB的干净数据。我们共鉴定了658,323 reads of inserts (ROIs),平均长度为2286 bp,质量为0.94,从720,832个聚合酶reads中筛选出12个通过,其中全通过的reads > = 0,预测的一致性精度>为0.81).结果表明,从5 '到3 '末端和polyA尾的整个转录组序列中,roi分别为62.87%(449,460)和29.4%(193,513)个全长(FL) reads。此外,全长非嵌合(FLNC) reads的数量为441,885个,平均FLNC reads长度为1969 bp(见表)1).cDNA和ROIs的主要数量分布在附加文件中1:图S1a和S1b。
随着PACBIO测序结果具有很高的错误率,FLNC读取使用迭代聚类进行纠错(ICE)算法并校正Illumina Hiseq2500平台来纠正错误。我们生成了227,516个共识同种型,平均共识同种型长度为2086年的BP,其中包括148,836种高质量的同种型(表1).我们使用CD-HIT成功获得80,654份非冗余转录本[41.]对于高加索三叶草根茎分析。
ORFS,SSR和LNCRNA的预测和识别事件
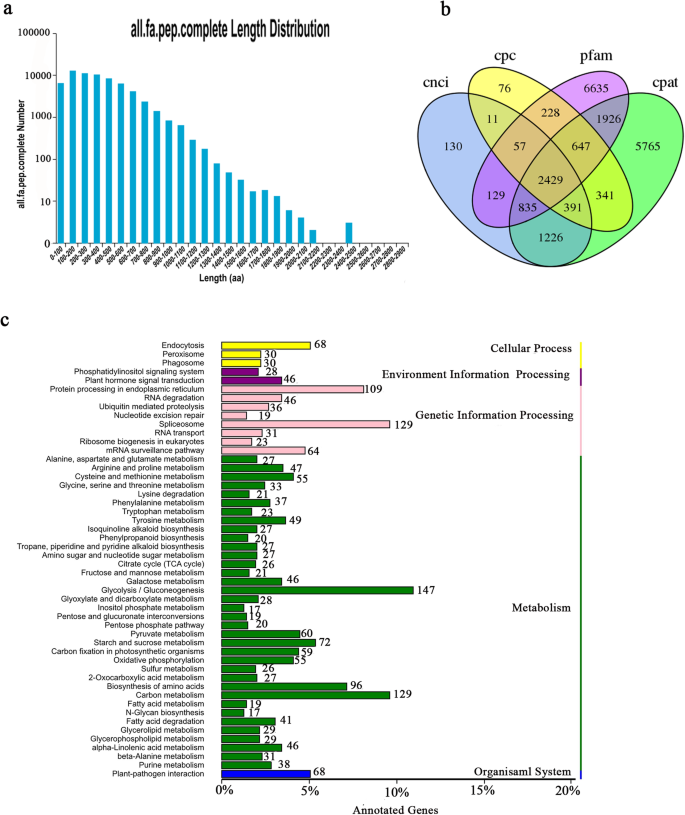
为了识别推定的蛋白质编码序列,我们使用TransDecoder预测了78209个开放阅读框(ORFs)。共鉴定出65 227个CDSs的起始密码子和终止密码子,完整CDSs的数量和长度分布如图所示。1a.其中,分布在100 - 200 bp范围内的转录本有12630份。
检查总共79,424个序列(167,351,883bp),包括58,276个简单的序列重复(SSR)和含36,110SSR的序列(附加文件2:表S1)。含有1个以上SSR的序列数为13856条,以化合物形式存在的SSR数为10041条。此外,大多数序列为单核苷酸(33,533)、双核苷酸(8610)和三核苷酸(14026)。
本研究通过编码潜能计算器(CPC)、编码非编码指数(CNCI)、pfam蛋白结构域分析和编码潜能评估工具(CPAT)预测2429个长链非编码rna (lncrna)(图)。1b),揭示未来研究的候选lncrna。
检测到总共6821个。因为没有参考基因组可用于高加索三叶草,所以我们无法识别出的类型。尽管如此,作为调节基因表达和产生蛋白质组多样性的重要机制,我们将这些结果显示为KEGG富集中的事件(图。1c),发现以下基因高度富集:“糖酵解/糖异生”(147)、“剪接体”(129)、“碳代谢”(129)、“内质网蛋白加工”(109)和“氨基酸生物合成”(96)。
文本注释
在七个数据库中的至少一种,包括NCBI非冗余蛋白(NR),Swiss-prot(手动注释和审查的蛋白质序列数据库),基因本体(GO),外科群组的至少一个(COG),真核直核群体(KOG),蛋白质(PFAM)和基因和基因组的京都百科全书(KEGG)。五个数据库的详细注释数(Go,Cog,NR,Kegg和Swiss-Prot)在Venn图中显示(附加文件3.:图S2)。
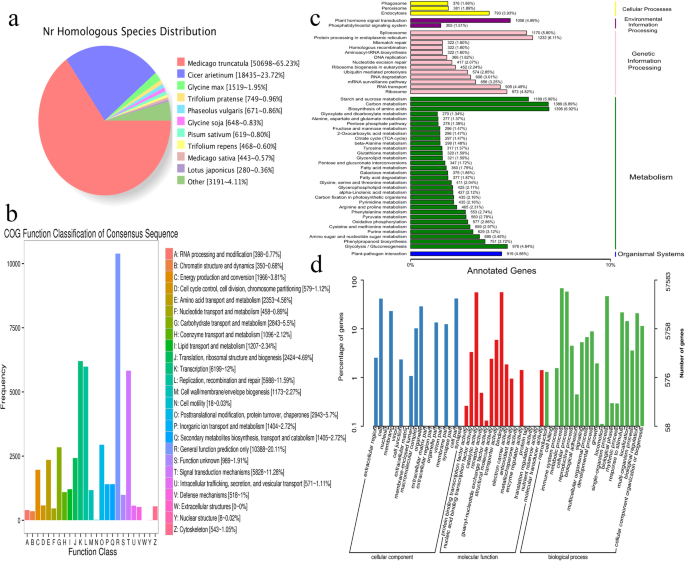
通过同源物种分析比较NR数据库中转录组序列,共注释了77721个转录本。约65.23%(50,689)的序列被对齐Medicago truncatula序列,然后是中投arietinum(23.72%,18,435)(图。2一种)。
对所有组装的转录物进行所有组装的转录物针对COG数据库进行搜索以评估转录组注释的有效性和完整性,结果分为26个主要类别(图。2b)。簇“一般函数仅预测”(10,388),“转录”(6199)和“复制,重组和修复”(5988),代表了三个最大的组,其次是“信号转导机制”(5828)和“后期改性,蛋白质转换和伴侣“(2943)。
KEGG数据库共匹配了33383个(42.84%)转录本,并进一步划分为128个KEGG通路(图)。2C);“氨基酸的生物合成”(1396),“碳代谢”(1389),“内质网的蛋白质加工”(1232),“淀粉和蔗糖代谢”(1189)和“抗磷酸体”(1170)是最代表性的途径.
根据GO分析,在三个本体中富集了57,583个转录本(图)。2D)“生物过程”、“分子功能”和“细胞成分”。参与生物过程的转录本主要有“代谢过程”(39,010)、“细胞过程”(33383)和“单生物过程”(26,933)。在分子功能方面,转录本主要富集“结合”(32,350)、“催化活性”(32,206)和“转运活性”(3404)。在“细胞成分”类别中,转录本的主要类别与“细胞部分”(24,076)、“细胞”(23,984)和“细胞器”(16,648)有关。
专门表达DEGS的基因和统计数据
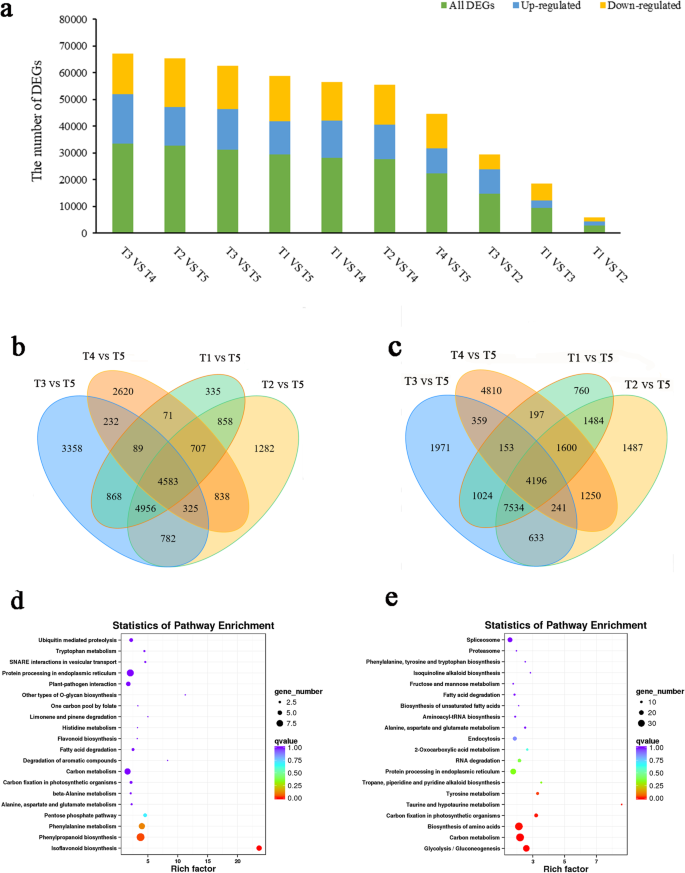
我们研究了五个组织中的转录表达水平,包括眩光(T1),水平根茎(T2),根茎肿胀(T3),根茎芽(T4)和根茎芽尖(T5)组织,以及T1的数量最多表达基因(76,124),其次是T4(75.978),T2(75,885),T3(74,396)和T2(74,327)(附加文件4:图S3A和附加文件4:图S3B。)。在每个组织中共同表达的基因数量为68,241。将通过illumina测序获得的清洁读数与非冗余转录物进行比较,以获得关于转录物和定量表达水平的位置信息(附加文件5:表S2)。每百万碱基对测序(FPKM)值的数千碱基序列的片段用于表示基因的表达水平。为了确定在组织中的特异性表达基因并向专业发育过程提供洞察,选择具有前5个平均表达水平的这些基因(至少两次重复和FPKM> 0.1)进行调查(表2).其中,T2和T5的FPKM值较高:F01_cb16574_c994/f1p0/1592(丝裂原活化蛋白激酶,MAPK3)和F01_CB71 58_C 94 / F1P0 / 1000(甜菜碱醛脱氢酶,BADH).
为了鉴定白三叶草根茎发育过程中的基因表达差异,我们重点对Illumina测序获得的差异表达基因(DEGs)进行了鉴定。DEGs在不同根茎组织中的分布示意图(图。3.a)、T3和T4的DEGs数量最多(33,612),其中上调基因18,372个,下调基因15,240个。T5及其他组织(T1、T2、T3、T4)中DEGs含量较高;与不同组织比较,T5中共发现4585个共上调基因和4196个共下调基因(图)。3.B和c)。关于T5中的上调基因,在KEGG分析中仅富集65例:“苯丙醇生物合成”(9),“内质网的蛋白质加工”(8),“苯丙氨酸代谢”(7),“碳代谢“(7)和”异黄酮生物合成“(6)(图。3.d).在T5中下调的基因中,注释了231个基因,主要涉及“碳代谢”(35)、“氨基酸生物合成”(34)、“糖酵解/糖异生”(29)、“内质网蛋白加工”(25)和“剪接体”(21)(图。3.e)。
TFs预测和WGCNA
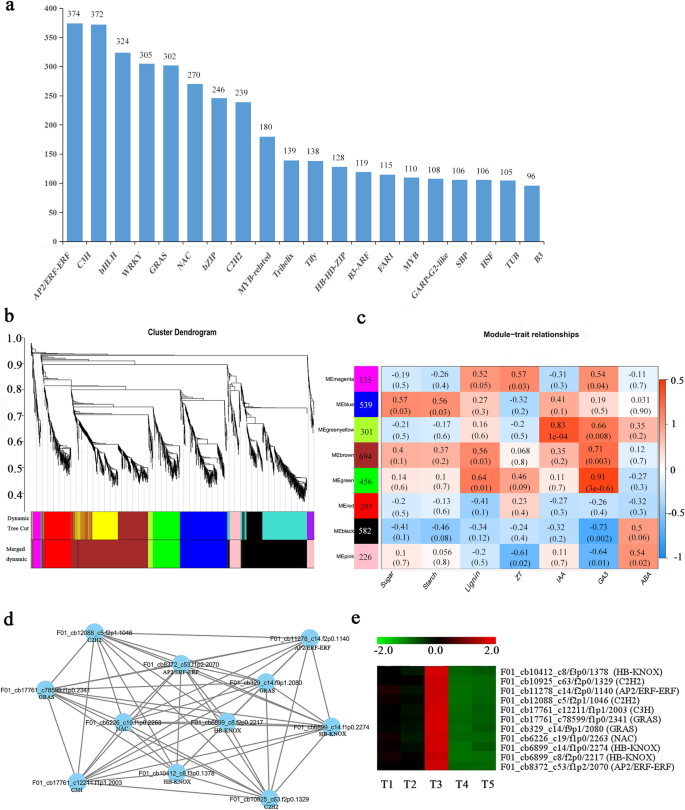
转录因子在植物生长发育中起着重要作用。我们检查了来自64个不同家族的4501个假定的tf(附加文件)6:表S3),前20个TF系列如图2所示。4a.大多数转录本属于AP2/ERF-ERF(374)、C3H(372)、bHLH(324)、WRKY(305)、GRAS(302)、NAC(270)、BZIP(246)、C2H2(239)和myb相关(180)家族。这些转录因子广泛参与植物的生长和胁迫反应,并与根茎发育有关。
我们使用加权基因共表达网络分析(WGCNA)进一步探索TFs之间的关系(过滤FPKM值< 1和K-ME< 0.7的TFs)6:表S3)和白种人三叶草根茎的生理特性(附加档案7:表S4)。高度相关的tf集群被定义为模块,同一集群中的tf高度相关。WGCNA识别出八个不同的模块(图中用不同颜色标记)。4b)。10个不同模块和每个不同样品(特征)的每个模块的特征基因之间的相关系数如图4所示。4C。值得注意的是,蟾蜍素(IAA)-trait和GA3-Trait与Megreenylow模块和Megreen模块显着相关(r2> 0.8,p < 10- 4.).除ABA外,绿色模块的大部分基因在6个性状中均上调,其中大部分转录因子主要在T3中上调(附加文件)8:图S5)。我们基于基于Eigengene的连接(KME)值≥0.99的标准,确定了11个轮毂基因,基于调节网络的绿色模块中的边缘重量值≥0.5(图。4d)。这些轮毂基因主要属于HB-CNOK,AP2 / ERF-ERF,GRAs,C2H2,C3H和NAC系列(MeGreen-Ga3);此外,这些TF系列在T1,T2和T3中上调,特别是在T3中(图。4E),可能与根茎中结节的形成有关。
根茎发育中激素信号转导相关基因的鉴定
植物激素在发育的各个方面起着重要的作用。据此,我们将DEGs定位于白花三叶草的激素信号转导途径,并分析了它们在不同组织中的表达。共有276个DEGs与8种激素的合成和代谢有关,包括IAA、ABA、乙烯(ETH)、细胞分裂素(CTK)、赤霉素(GA)、油菜素内酯(BR)、茉莉酸(JA)和水杨酸(SA)(图)。5).发现了IAA合成和代谢的最大基因数(62),然后在28,eth在28,Br为24,eth在24,eth时为52,eC,18,Ga为10,在10中。所有这些重要基因与激素合成和代谢相关的相关表现出不同组织中的不同表达模式。此外,比其他组织在T2和T5中清晰地调节更多基因。关于SA信号,几乎所有基因属于TGA家族,仅在T4中具有上调。与BR信令相关的大多数基因在T4和T5中显示出更高的水平。对于CTK转导,与CTK信号通路相关的所有关键基因被鉴定为DEG。只有三种基因在T3中显示出上调趋势,这可能是细胞分裂大量的组织。只有十台与GA信号有关;五个基因被归类为GID1.在T2中显着上调。与ABA信令和转导相关的基因显示出明显变化。对于JA信号传导,52个基因被注释为Jaz,在T1,T2和T3中具有上调。相反,大多数Eth信号传导基因在T3中表现出更高的表达,并且在T2中均在下调。
qRT-PCR验证基因表达
为了确认通过RNA-SEQ,12种基因获得的基因的准确性,包括六种植物激素信号转导基因,三种TFS和属于其他类别的三种基因,用于定量实时RT-PCR(QRT-PCR)分析(每个基因的三次重复)。QRT-PCR和RNA-SEQ结果之间的良好再现性由Pearson的相关性分析表示,从而验证RNA-SEQ数据的准确性和可靠性(附加文件9:表S5)。
讨论
在豆科植物中,高加索三叶草的根茎是独一无二的,赋予了该植物独特的克隆繁殖特性和抗逆性。本研究通过PacBio和Illumina测序,获得了高加索三叶草根茎的高质量转录序列,这一结果将有助于我们了解根茎的生长发育,并为进一步研究提供分子基础。
AS是调节基因表达和产生蛋白质多样性的重要机制[42.].发现作为第一次在高加索三叶草根茎中识别的事件的数量低于Medicago Sativa.(7568)[43.]但高于那个三叶草pratense(5492)[13.].我们对白种人三叶草根茎的特殊特征的研究受到参考基因组缺乏阻碍的,并且作为事件的类型是不可能确定的。
许多氨基酸(苯丙氨酸,酪氨酸和色氨酸)不仅是蛋白质的重要组成部分,而且是许多次级代谢物的前体。这些次生代谢物对植物生长至关重要[44.].类似于其他植物的根茎(oryza longistaminata.和miscanthus lutarioriparius)[12.,19.一些基础代谢在高加索三叶草的根茎中起重要作用,例如,碳代谢(1389)和淀粉和蔗糖代谢(1189)(图。2c)。
通过分析每个组织中特定表达的基因,我们发现MAPK3(F01_cb16574_c994/f1p0 /1592)主要具体表达式为T2(表2)2).MAPK家族已经在烟草中进行了研究,可能参与生长,发展和对植物激素和环境信号的反应[45.].T5与根组织(T1、T2和T3)有很大的不同(图3)。3.a),具有较多的deg,并且是形成新的地上部分的场所。在T5中观察到一个FPKM最高的特异基因BADH(F01_cb7158_c94 / f1p0/1000)(表2).BADH基因参与甜菜碱的合成,并作为植物渗透调节剂,在非生物胁迫中发挥重要作用[46.].实验表明BADH c增加甘薯和胡萝卜的耐受性,避免盐胁迫,氧化应激和低温应激等非生物应激,从而保持细胞膜完整性[47.,48.].BADH在T5的高水平上特异性表达,可能是因为T5比其他组织更脆弱,和BADH可以保护T5,促进新植物的生长。因此,我们推测,防御和胁迫反应在高加索三叶草的发育中起着至关重要的作用,这可能是它能够在极端冬季温度下生长的原因,或者这种反应可能是一个大根茎系统的必要条件。
TFS可以对控制发展方案的基因调节网络提供深入了解,并被认为是对响应内部生长调节剂以及环境信号而更好地理解根系组织分化和根部发展的主要贡献者[49.,50.].据报道,在莲花根茎形成的形成中,据报道,参与激素代谢,纤维素合成能量,代谢物质合成和运输应力以及膨胀相关蛋白基因和TFS,如BHLH,TCP,WRKY,BZIP,MYB和NAC[24.].此外,AP2 / ERF家族占白种人三叶草根茎中最大的数量(图。4A)、乙烯反应因子,如BBM / PLT4和PLT1-3,已被描述为根业企业启动和维护的主监管机构拟南芥[51.,52.].在Raphanus Sativus和Medicago Sativa,AP2/ERF调控的非生物应激反应机制已被认真研究[53.,54.].这拟南芥NAC家族成员NAC1.转导IAA信号的下游Tir1.促进横向根系发展[55.].在WRKY家族中,WRKY75据报道,涉及调节营养饥饿反应和根茎生长[56.].值得注意的是,我们对TFS进行了WGCNA聚类,发现11个枢纽基因中的3个属于MeGreen-Ga3中的HB-Knox家族(图。4d). GA可以抑制KNOX蛋白表达升高的影响,修饰KNOX可能通过GA水平的局部变化改变植物结构[57.].在拟南芥,GA20ox1过表达KNOX蛋白STM或BREVIPEDICELLUS的叶片mRNA水平降低[56.].此外,在模型植物,如拟南芥,玉米和烟草,诺克斯基因表达局限于茎分生组织和茎[58.].然而,在高加索三叶草的地下根茎中,诺克斯这些基因被鉴定为枢纽基因,特别是在膨大主根(T3)的主要组织中。这些发现是否与诺克斯的监管拟南芥公司,茎和芽是值得进一步的调查[59.].西程发现,在梨植物中共同表达诺克斯和pbknox1,这些因素涉及细胞壁增稠和木质素生物合成,抑制来自木质素合成的关键结构基因[60.].
越来越多的证据表明,激素影响分蘖的生长和贮藏器官的形成[12.,61.].易坤的研究表明,600 mg·L- 1GA3能促进白草根茎生长发育,增加内源IAA、ZT和GA含量3.[62.].T1和T2显示出高GID1.表达和GA可能参与光周期诱导和调控贮藏器官的形成和根茎伸长[63.];因此,Ti和T2可以是用于营养储存的关键器官。此外,jaz蛋白在根中累积(t1,t2和t3)。当病原体入侵和非生物应激发生时,据信Jaz-Myc旨在形成免疫网络,其次是Jaz蛋白质降解和Myc释放[64.].T3与ETH和ctk介导的通路密切相关,可能与根肿胀有关。IAA已被证明能激活根的形成,而CTKs则介导根的认同、早期原始解体和早期芽发育起始缺失[65.].CTKs有利于根茎膨大,但不利于根茎诱导。
与IAA合成代谢途径相关的基因在T3和T4中下调。IAA可能不会参与根本扩大或诱导芽的生产,但可能与侧向根发育密切相关。T1,T2和T3可能主要用作储存器官,为植物生长提供能量。此外,T4可能相对脆弱,需要SA途径来介导免疫以防止病原体感染并生长新植物。BR信号主要涉及植物生长和植物形态学发育,相关基因在T5中上调[66.].T4和T5主要与抗应激和次级代谢途径有关。当然,激素并不是调控顶端分生组织和侧生器官发育的唯一因素;它们经常与tf合作,平衡分生组织和器官发生的维持。
结论
总之,我们提供了基于PACBIO测序和Illumina测序的高加夫人三叶草根茎的全长转录组,揭示了不同组织的基因表达模式和注释。我们强调了激素和TFS在白种人三叶草的根茎中的作用,研究了白种人三叶草的不同组织中激素途径相关基因的表达,并通过WGCNA确定了11种中的11个枢纽基因。在这项研究中,确定了与根茎发育有关的一组基因,奠定了进一步的功能基因组学研究根茎发展的基础。
方法
植物材料及RNA制备
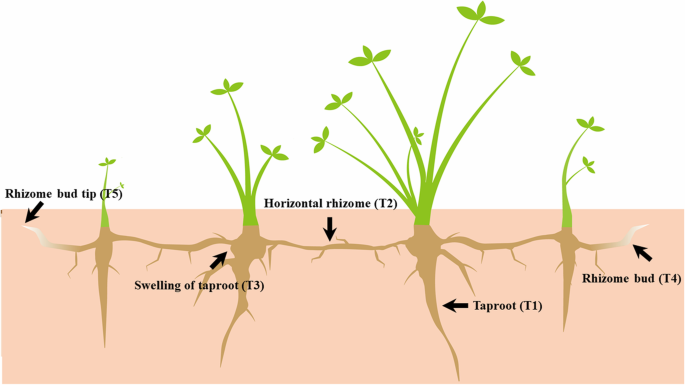
3岁的白种人三叶草T1,T2,T3,T4和T5组织(图。6)从东北农业大学的测试领域收集(E 126°14'; 2018年8月45°05')。我们将每个样品放入1.5ml离心管中,用3个单独的植物复制为每种组织,以确保RNA量。每个样本显示出良好的相关性(R2 > 0.8; Additional file10.:图S5)。植物材料的原始来源是从内蒙古农业大学内蒙古草品种工程技术研究中心引入的。内蒙古内蒙古草地农业大学的内蒙古草品种工程技术研究中心进行了正式的样品鉴定,提供了沉积的标本的细节并允许收集。白种人三叶草的IPNI生命科学标识符(LSID)是URN:LSID:IPNI.ORG:名称:522843-1。从土壤床中除去植物,用自来水轻轻洗涤根,在液氮中冷冻并立即储存在-80℃。使用Trizol提取总RNA。通过1.2%琼脂糖凝胶电泳监测RNA降解和污染。使用纳米玻璃和Agilent 2100生物分析仪(Santa Clara,CA)测定提取的总RNA的数量和完整性[13.].对于每个RNA样品,并行合并1μg单分子长读测序(PACBIO equel,MENLO PARK,USA)和Illumina测序(Illumina Novaseq6000,California,U.S.A)进行测序。
PacBio cDNA文库的制备与测序
用SMARTerTM PCR cDNA Synthesis Kit合成全长cDNA,然后进行全长cDNA PCR扩增和cDNA末端修复。使用Qubit 2.0荧光仪和Agilent 2100生物分析仪测定cDNA文库的浓度和质量[67.].通过PACBIO续集测序1-6 kB文库。
Illumina cDNA文库的构建与测序
首先,将15份真核细胞mRNA标本用含寡核苷酸(oligo, dT)的磁珠富集,并在碎片缓冲液中随机破碎成小片段。以小片段的mRNA为模板,用六碱基随机六聚体合成了cDNA第一链。经AMPure XP beads合成第二链(加入buffer、dNTPs、RNase H和DNA聚合酶I)后,用AMPure XP beads纯化cDNA,然后对双链cDNA进行末端修复,加入一条尾和测序连接子,用AMPure XP beads确定片段大小[41.].通过PCR评估最终cDNA文库,使用Agilent 2100生物分析仪测定cDNA文库的质量。使用Illumina Novaseq的5'和3'末端测序文库。
PACBIO测序数据分析
ROI序列提取使用Iso-seq管道,minFullPass = 0和minPredictedAccuracy = 0.80。在roi中,含有poly(A) tail信号和5 ' -和3 ' - cdna引物的reads被认为是全长非嵌合(FLNC)转录本[68.].采用ICE算法获得了一致的测序亚型。最后,使用Quiver对高质量的FL转录本进行分类,准确率大于99%。使用CD-HIT (identity > 0.99)去除高质量FL转录本中的冗余序列,最终获得非冗余转录本序列[69.].
Illumina测序数据分析
所有下游分析都使用高质量的清洁数据(从原始数据中卸下含有适配器,Poly-N和低质量读数)进行所有下游分析[70].这些干净的reads随后被映射到PacBio参考基因组序列,并通过将片段计数转换为FPKM值进行标准化。使用DESeq (v 1.10.1)进行差异表达分析;基于DESeq的fold change≥4 and FDR < 0.01被认为是差异表达的指标[71.].
SSR、ORFs、AS、lncRNA检测
使用MISA鉴定转录组中的SSRS(http://pgrc.ipk-gatersleben.de/misa//),仅检测到≥500 bp的转录本。
TransDecoder软件(https://github.com/transdecoder/transdecoder/releases.使用PFAM数据库中的ORF长度,对数似然评分和氨基酸序列比较来鉴定来自转录序列的可靠潜在CDS。
我们使用Iso-SeqTM数据直接运行具有高身份设置的all-vs-all BLAST [72.].符合所有标准的BLAST排列被认为是候选AS事件的产品[73.],两个HSP(高分割对)在对准中> 1000bp。两个HSP具有相同的前向/反向方向,并且一个序列在相同的对准中应邻接,或者具有小于5bp的小重叠。其他序列应该与显示“作为间隙”不同,并且连续序列应与不同的序列完全对齐。AS间隙应大于100bp,3'/ 5'端的至少100bp。
中国共产党(74.],CNCI,CPAT [75.]和pfam [76.用于将非蛋白编码RNA与推定的蛋白质编码RNA区分开来。选择长度大于200nt和两个以上外显子的转录物作为LNCRNA候选基因。
功能注释
有关所得NOREDRUMED转录序列的注释信息基于以下数据库中的BLAST:NR,PFAM [76.], KOG [77.],COG,瑞士 -78.],kegg [79.] 去 [80].
实时RT-PCR
存在是在10 -μl进行卷包含0.5μl稀释cDNA、0.2μl的底漆,0.2μl的反向引物,和1×SYBR预混料交货Taq II(豆类)与下列条件:95°C的180年代,紧随其后的是40 95°C的周期15秒,59°C 15和72°C 15 s。2-△△Ct法计算相对表达水平[81.].用三个重复进行所有反应。使用的所有引物都显示在附加文件中11.:表S6。
可用性数据和材料
在本研究中产生的一个组合的PACBIO库和一个Illumina RNA山库的原始读数可从NCBI的Bioproject获得(https://www.ncbi.nlm.nih.gov/bioproject/),分别为PRJNA586585和PRJNA588309。
缩写
- 雌花:
-
开放阅读框
- CDSS:
-
编码序列
- SSRS:
-
简单序列重复
- 作为:
-
可变剪接
- LncRNA:
-
长期非编码RNA
- TF:
-
转录因子
- KB:
-
千碱基
- 投资回报:
-
读取插件
- 冰:冰:
-
迭代聚类用于纠错;
- FL:
-
总长度
- FLNC:
-
全身non-chimeric
- CNCI:
-
Coding-Non-Coding指数
- CPC:
-
编码潜力
- CPAT:
-
编码潜在评估工具计算器
- FLNC:
-
全长非嵌合体
- COG:
-
同源组的簇
- NR:
-
NCBI非冗余蛋白质
- 去:
-
基因本体论
- Kog:
-
真核外科群体
- PFAM:
-
蛋白家族
- KEGG:
-
Kyoto基因和基因组的百科全书
- 度:
-
差异表达基因
- FPKM:
-
每百万碱基对的转录序列每千碱基测序
- WGCNA:
-
加权基因共表达网络分析
- IAA:
-
养阴
- 阿坝:
-
脱落酸
- 乙:
-
乙烯
- CTK:
-
细胞分裂素
- 遗传算法:
-
赤霉酸
- BR:
-
Brassinosteroid
- JA:
-
茉莉酸
- 山:
-
水杨酸
- BADHs:
-
甜菜碱醛脱氢酶
- MAPK:
-
促丝糖型活化蛋白激酶
- QRT-PCR:
-
实时定量rt - pcr
参考文献
- 1。
黑色AD,卢卡斯RJ。在干旱条件下混合草种中,白草苜蓿的产量高于白草苜蓿。NZGA。2000; 62:183-8。
- 2。
戴维斯博士。土耳其和东爱琴群岛的植物群。q Rev Biol。1965年; 35(1):103。
- 3。
I Lane, Watkins E, Spivak M.草坪草的种类影响库拉三叶草(Kura clover)的建立和开花三叶草ambiguum)在草坪。Hortscience。2019; 54(5):824。
- 4.
Andrzejewska J,Contreras-Govea Fe,Pastuszka A,Albrechtc Ka。库拉苜蓿与中欧传统栽培的多年生牧草豆类的性能比较。Acta Agr Scand。2016; 66(6):516-22。
- 5。
Caradus JR,Crush Jr,Ouyang L,Fraser W.耐铝合金白三叶草评估(Trifolium Repens.)选择在东奥塔哥高地土壤。NZGA。2001; 44:141-50。
- 6。
Watson RN, Neville FJ, Bell NL, Harris SL.在沿海的Plenty湾,白种三叶草作为旱地乳制品的牧草豆类。NZCA。1996; 58:83-8。
- 7。
Brummer EC,Moore KJ。牛肉牛持续放牧下常年凉爽季节草和豆类品种的持久性。Agron J. 2000; 92(3):466-71。
- 8。
谢弗CC,马腾GC。库拉苜蓿饲草产量、品质及林分动态。acta botanica yunica(云南植物研究)1991;71(4):1169-72。
- 9。
Taylar Nl,Smith Rr。库拉三叶草(三叶草ambiguum(M. B.)育种、培养和利用。阿阿格龙。1997;63(08年):153 - 78。
- 10。
孔特拉斯-戈维亚,帕斯图斯卡,阿尔布雷希特库拉苜蓿与中欧传统栽培的多年生牧草豆类的性能比较。Acta Agr Scand B-S P. 2016;66(6): 516-22。
- 11.
sper GS, Allinson DW。库拉三叶草(三叶草ambiguum):用作饲料和土壤保持的豆类。经济学机器人。1985;(2):165 - 76。
- 12.
胡rb,yu cj,王xy,jia cl,pei sq,他k,他g,kong yz,周gk。de novo转录组分析miscanthus lutarioriparius鉴定根茎发育中的候选基因。前植物SCI。2017; 8:482。
- 13。
超艳华,袁建平,李顺生,贾树青,韩立波,徐立新。红三叶草转录本和剪接亚型的分析(三叶草pratenseL.)通过单分子长读取测序。BMC植物BIOL。2018; 18:300-12。
- 14。
黄LK,闫高清,赵XX,张XQ,王j,frazier t,yin g,huang x,yan df,zang wj等。在热应激下鉴定差异表达基因,在果园紫杉中发育分子标记物(Dactylis glomerata通过转录组分析。生态学报。2015;151(5):497-509。
- 15.
jang cs,kamps tl,唐h,鲍德·je,lemke c,paterson啊。非根根瘤菌中根茎特异性基因的进化命运高粱基因型。遗传。2009; 102(3):266-73。
- 16。
张涛,赵晓强,王文生,黄丽丽,刘学勇,宗毅,朱丽华,杨大才,付斌,李志坤。根茎和空中芽的深层转录组测序高粱propinquum.植物mol biol。2014; 84(3):315-27。
- 17。
王凯,彭赫兹,林EP,金QY,华X,盛Y,卞HW,宁H,潘杰,王jh。鉴定竹状根茎芽发展的基因。J Exp Bot。2010; 61(2):551-61。
- 18。
胡凤英,陶大勇,Sacks E,傅斌,徐鹏,李俊,杨勇,Mcnally K, Khush GS, Paterson AH。水稻多年生性的趋同进化高粱.美国国立科学院。2003年,100(7):4050 - 4。
- 19。
通过基因组微差异表达分析鉴定根茎特异性基因的方法oryza longistaminata..植物学报。2011;11(1):18-32。
- 20。
He RF, Salvato F, Park JJ, Kim MJ, Nelson W, Balbuena TS, Willer M, Crow JA, May GD, Soderlund CA,等。红米全系统比较(oryza longistaminata.)的组织识别根茎特定的基因和蛋白质,这些基因和蛋白质是水稻改良的目标。植物学报。2014;14(1):46-67。
- 21.
Balbuena Ts,他rf,Salvato F,Gang Dr,Thelen JJ。古代血管植物发育根茎的大规模蛋白质组比较分析木贼属hyemale.前植物SCI。2012; 3:131。
- 22.
杨宝伟,咸日东。利用de novo RNA-seq转录组分析比较不同环境下栽培的人参根。植物生长学报。2018;84(1):149-57。
- 23.
瑞丰H,Min-Jeong K,William N,Balbuena Ts,Ryan K,Robin K,Crow Ja,May Gd,Thelen Jj,Soderlund Ca.基于下一代测序的常见簧片的转录组和蛋白质组学分析,芦苇南极光(禾本科),揭示了涉及入侵和根茎特异性的基因。acta botanica sinica(云南植物学报),2012;
- 24.
杨明,朱丽,潘超,徐丽,刘艳,柯伟,杨平。温带和热带荷花根茎形成调控的转录组分析(Nelumbo Nucifera).SCI REP。2015; 5:13059。
- 25.
Huang QQ, Huang X, Deng J, Liu HG, Liu ywen, Yu K, Huang BS。叶与根茎基因表达差异白术lancea:比较转录组分析。植物学报2016;7:348。
- 26.
宋T,刘泽,李杰,朱QK,谭河,陈杰,周JY,廖H.根瘤菌和叶片的比较转录组当归Chuanxiong.植物生态学报。2015;301(8):2073-85。
- 27.
Koo Hj,McDowell等,MA XQ,Greer Ka,Kapteyn J,谢ZZ,Descour A,Kim H,Yu Y,Kudrna D等,et al。姜和姜黄表达的序列标签鉴定根茎身份和发育的签名基因以及姜黄素,姜素和萜类化合物的生物合成。BMC植物BIOL。2013; 13(1):27-44。
- 28.
POP M,Salzberg SL。新测序技术的生物信息学挑战。遗传趋势。2008; 24(3):142-9。
- 29.
梅森队。更快的序列仪,更大的数据集,新挑战。基因组Biol。2012; 13(3):314。
- 30.
Rhoads A, Au KF。PacBio测序及其应用。生物信息学。2015;13(5):278-89。
- 31.
Li PH, Ponnala L, Gandotra N, Wang L, Si YQ, Tausta SL, Kebrom TH, Provart N, Patel R, Myers CR, et al.;玉米叶片转录组的发育动态。Nat麝猫。2010;42(12):1060 - 7。
- 32.
Wang T, Wang H, Cai D, Gao Y, Zhang H, Wang Y, Lin C, Ma L, Gu L. analysis of alternative splicing and alternative polyadenylation in moso bamboo(毛竹)植被类型).植物j . 2017; 91(4): 684 - 99。
- 33。
黄力坤,冯国英,严宏达,张志刚,Bushman BS,王金平,Bombarely A,李明志,杨志富,聂刚,等。基因组组装提供了深入了解的基因组进化和开花调控果园草。中国生物技术学报(英文版);2020;18(2):373-83。
- 34。
徐ZC,彼得·雷,韦瑟J,罗哈姆,廖博,张X,朱yj,吉阿,张b,胡sn,等。通过施加到不同根组织的测序平台的组合获得的全长转录组序列和接头变体萨尔维亚米尔蒂希萨和tanshinone生物合成。工厂j . 2015, 82(6): 951 - 61。
- 35。
赵琦,高紫育,张达,赵博,侗FQ,富思,刘立志,王立。发展动态杨树阻止转录组。植物生态学报;2019;17(1):206-19。
- 36。
林夫。WHR,斯蒂芬S. 5种的生理反应三叶草干旱压力。Chin J Appl Environ Biol。2011; 17(4):580-4。
- 37。
Schmid B, Bazzaz FA。多年生植物的克隆整合和种群结构:切断根茎连接的影响。生态。1987;68(6):2016 - 22所示。
- 38。
Schmid B, Bazzaz FA。根状茎植物对施肥和干扰的生长响应。Oikos。1992;65(1):24里面。
- 39。
Humphrey Ld,Pyke da。多年生小麦草中的克隆觅食:一种利用斑块土壤营养的策略。j eco。1997年; 85(5):601-10。
- 40。
张志强,张志强,张志强,等。根系对根状茎草本植物克隆可塑性的影响。生态。1998;79(7):2267 - 80。
- 41。
陈H,冯WW,Chen K,邱XC,徐H,毛古墓,赵T,丁YY,XY WU。转录组分析揭示了斑马鱼(Danio Rerio)卵巢中邻苯二甲酸二丁酯和邻苯二甲酸二异丁酯的组合暴露的潜在毒性机制。Aquat毒蛇。2019; 216:105290。
- 42。
Chaudhary S,Khokhar W,Jabre I,Reddy Asn,Byrne LJ,Wilson Cm,Syed NH。替代剪接和蛋白质多样性:植物与动物。前植物SCI。2019; 10(10):14。
- 43。
赵毅,元jb,guo t,xu lx,mu zy,han lb.转录物分析和接头同种型Medicago Sativa.L.通过单分子长读测序。acta botanica sinica(云南植物学报),2019;49(3):457 - 461。
- 44。
Tzin V,Galili G.新的洞察植物中的Shikimate和芳香氨基酸生物合成途径。莫尔植物。2010; 3(6):956-72。
- 45.
张淑娟。MAPK在植物防御信号中的级联作用。植物科学进展。2001;6:520-7。
- 46.
Fitzgerald TL,Waters Dle,Henry RJ。甜菜碱醛脱氢酶在植物中。植物BIOL。2009; 11(2):119-30。
- 47.
Kumar S,Daniell H.Prastid-Expressedededededede在胡萝卜培养细胞,根和叶中的醛醛脱氢酶基因赋予增强的耐盐性。植物理性。2004; 136(1):2843-54。
- 48.
范文军,张敏,张红霞,张鹏(Ipomoea Batatas.表达菠菜甜菜碱醛脱氢酶。《公共科学图书馆•综合》。2012;7(5):14。
- 49.
金金平,张浩,孔林,高刚,罗建军。植物转录因子功能和进化研究的门户。42(D1): D1182-7。
- 50。
Montiel G,Gantet P,Jay-allemand C,Breton C.转录因子网络。根本发展知识的途径。植物理性。2004; 136(3):3478-85。
- 51.
乙烯响应因子(AP2/ERF)转录因子:胁迫反应和发育程序的中介。新植醇。2013;199(3):639 - 49。
- 52.
AP2/ERF家族转录因子在植物非生物胁迫反应中的作用。acta optica sinica, 2012; 32(2): 369 - 372。
- 53.
Karanja BK,Xu L,Wang Y,Tang M,M'mbone Muleke E,Dong J,Liu L.萝卜的AP2 / ERF基因家族的基因组鉴定(Raphanus Sativus揭示进化和响应非生物胁迫的模式。基因。2019;718:144048。
- 54.
金晓燕,尹晓峰,Ndayambaza B,张志生,闵晓燕,林欣欣,王艳芳,刘文霞。ERF基因家族的全基因组鉴定和表达谱分析Medicago Sativa.L.在各种非生物胁迫下。DNA细胞BIOL。2019; 13(10):1056-68。
- 55.
谢Q,Frugis G,Colgan D,Chua NH。拟南芥NAC1在TIR1下游转导生长素信号,促进侧根发育。基因Dev。2000;14(23):3024 - 36。
- 56.
Devaiah BN, Karthikeyan AS, Raghothama KG。WRKY75转录因子是磷酸获取和根发育的调节因子拟南芥.植物杂志。2007;143(4):1789 - 801。
- 57。
Singh DP, Filardo FF, Storey R, Jermakow AM, Yamaguchi S, Swain SM。过表达赤霉素灭活基因改变种子发育,KNOX基因表达和植物发育拟南芥.physiol植物。2010; 138(1):74-90。
- 58。
诺克斯基因:植物发育和多样性的多功能调控因子。发展。2010;137(19):3153 - 65。
- 59。
徐L,沉Wh。polycomb on knox基因的沉默限制射击茎细胞利基拟南芥.咕咕叫医学杂志。2008;18(24):1966 - 71。
- 60.
程旭,李明莉,Abdullah M,李光华,张建勇,马曼佐,王辉,金强,蒋天生,蔡艳萍,等。在对梨(Pyrus Bretschneideri.)Knox家族和Pbknox1的功能表征,拟南芥Brevipedicellus原基因,参与细胞壁和木质素生物合成。前群体。2019; 10(17):632。
- 61.
Dello Ioio R,Linhares FS,Scacchi E,Casamitjana-Martinez E,Heidstra R,Costantino P,Sabatini S. cytokinins确定拟南芥通过控制细胞分化来脱位尺寸。Curr Biol。2007; 17(8):678-82。
- 62。
易k,赵呀,胡y,刘jx,他tt,李x,歌p,cui gw,yin xj。GA的效果3.6 -BA对白草根茎段生长和内源激素含量的影响。草业学报。2020;29(2):22-30。
- 63。
Sun T.Gibberellin-Gid1-Della:植物生长和发展的枢轴调节模块。植物理性。2010; 154(2):567-70。
- 64。
Garrido-Bigotes A,Valenzuela-Riffo F,Figueroa Cr。植物中的jaz蛋白的进化分析:一种寻找祖先序列的方法。int j mol sci。2019; 20(20):5060。
- 65。
Pernisova M, Grochova M, Konecny T, Plackova L, Harustiakova D, Kakimoto T, Heisler MG, Novak O, Hejatko J.细胞分裂素信号通路通过AHK4受体调控器官身份拟南芥.发展。2018;145(14):11。
- 66。
NIE SM,Huang Sh,Wang SF,毛YJ,刘JW,MA RL,王XF。通过SLBRI1过表达增强的芸苔类固醇信号强度使用通过在番茄中的外源BR应用的方式对抗抗旱性。植物理性生物化学。2019; 138:36-47。
- 67。
acta optica sinica, 2010, 31 (4): 558 - 564 . acta optica sinica, 2010, 31 (4): 558 - 564 . acta optica sinica, 2010, 31 (4): 558 - 564 . acta optica sinica, 2010, 31 (4): 558 - 564 . acta optica sinica, 2010, 31(4): 558 - 564。《公共科学图书馆•综合》。2015;10 (7):e0132628。
- 68。
杨永强,蒲英,尹旭,杜建军,周志林,杨丹,孙旭东,孙勇,杨艳萍。剪接的变体BrrWSD1在萝卜(芸苔属rapa var..及其在干旱胁迫下蜡酯合成中的可能作用。中国农业科学。2019;67(40):11077-88。
- 69.
Li W,Godzik A. CD-his:用于聚类和比较大套蛋白质或核苷酸序列的快速计划。生物信息学.2006; 22(13): 1658 - 9。
- 70。
张宁,张海军,赵波,孙qq,曹云英,李锐,吴xx, Weesda S,李丽,任淑霞,等。用RNA-seq方法鉴别黄瓜侧根形成中褪黑激素响应的基因表达谱。J Pineal Res. 2014; 56:39-50。
- 71。
Anders S,Huber W.序列计数数据的差异表达分析。基因组Biol。2010; 11(10):106-18。
- 72。
altschul sf,madden tl,schaffer aa,张俊,张z,米勒w,lipman dj。Papped Blast和Psi-Blast:新一代蛋白质数据库搜索程序。核酸RES。1997年; 25(17):3389-402。
- 73。
刘xx,梅文彬,Soltis PS, Soltis DE, Barbazuk WB。从没有参考基因组的单分子长读序列中检测选择性剪接的转录异构体。生态学报。2017;17(6):1243-56。
- 74。
孔L,张Y,叶ZQ,刘XQ,赵SQ,Wei L,GAO G. CPC:使用序列特征和支持向量机评估转录物的蛋白质编码潜力。核酸RES。2007; 35:345-9。
- 75.
王磊,王树清,李伟。CPAT:一种基于无对齐逻辑回归模型的编码潜力评估工具。核酸学报2013;41(6):7。
- 76.
Lamb J, Jarmolinska AI, Michel M, Menendez-Hurtado D, Sulkowska JI, Elofsson A. pconfam:一个交互式的家庭结构预测数据库。中华医学杂志。2019;431(13):2442-8。
- 77.
Koonin EV, Fedorova ND, Jackson JD, Jacobs AR, Krylov DM, Makarova KS, Mazumder R, Mekhedov SL, Nikolskaya AN, Rao BS等。在完整的真核生物基因组中编码的蛋白质的一个全面的进化分类。基因组医学杂志。2004;5(2):28。
- 78.
Renaux A, UniProt C. UniProt:通用蛋白质知识库。核酸学报2018;46(5):2699。
- 79.
Kanehisa M,Goto S,Kawashima S,Okuno Y,Hattori M.用于解密基因组的Kegg资源。核酸RES。2004; 32:D277-80。
- 80.
Ashburner M,球,Blake Ja,Botstein D,Butler H,Cherry JM,Davis AP,Dolinski K,Dwight SS,EPPIG JT等人。基因本体:生物学统一的工具。NAT Genet。2000; 25(1):25-9。
- 81.
Livak KJ, Schmittgen TD。利用实时荧光定量PCR技术分析相关基因表达数据-△△Ct方法。方法。2001;25:402-8。
确认
我们感谢国家自然科学基金(31802120)支持这项工作和生物标志物公司(北京,中国),用于PACBIO和Illumina测序和原始数据分析。
资金
本研究由国家自然科学基金项目(no . 31802120);大型人工草地植物与循环模式组合研究与示范项目(no . 2017YFD0502106);东北农业大学学术骨干基金项目资助。关键词:人工草地,植物,循环模式,生物多样性,生物多样性资助方没有设计实验,也没有起草和修改手稿。
作者信息
隶属关系
贡献
XY设计了实验并对手稿进行了修改。KY进行了数据处理并起草了手稿。YZ, YH和XC准备了材料并进行了实验。TH和JL协助稿件准备。GC构思了这项研究。所有作者阅读并批准了手稿的最终版本。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
ROI的输出。一个ROI读长度分布为1-6 KB大小的bin。bROI分类为1-6 kB。
附加文件2:表S1。
SSR的预测。
附加文件3:图S2。
NR,瑞士 - Prot,Cog,Go和Kegg的Venn图。
附加文件4:图S3。
抄本的数量。一个五种组织中转录物的数量统计。b不同组织中表达转录物的Venn图。
附加文件5:表S2。
illumina测序数据与非冗余转录物之间的比较结果统计
附加文件6:表S3。
白草根茎转录因子(TF)家族的预测。
附加文件7:表S4。
不同生理指标的结果。糖,淀粉,木质素,zt,ga的条形图3.,IAA和ABA内容。
附加文件8:图S4。
绿色模块为tf的热图。
附加文件9:表S5。
QRT-PCR的结果。
附加文件10:图S5。
每种样品的Pearson相关系数的热线图。
附加文件11:表S6。
用于QRT-PCR的引物。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
尹旭东,易坤,赵永强。et al。揭示高加索三叶草根茎发展的全长转录组。BMC植物BIOL.20,429(2020)。https://doi.org/10.1186/s12870-020-02637-4
收到了:
接受:
发表:
关键字
- 高加索三叶草(三叶草ambiguumM. Bieb。)
- 全长转录组
- RNA-SEQ.
- 粉末
- 植物激素
- TFs