摘要
背景
淡紫色(紫丁香oblata是一种重要的木本植物,具有很高的观赏价值。然而,目前可利用的遗传标记资源非常有限,对重要观赏性状的遗传结构知之甚少美国oblata这阻碍了它的基因研究。因此,开发有效的分子标记和了解复杂花性状的遗传结构对植物的遗传研究具有重要意义美国oblata.
结果
在本研究中,从9864条单基因序列中共获得10,988个SSR,平均每8.13 kb获得1个SSR,其中双核苷酸重复序列为优势类型(32.86%,3611)。共验证2042对引物,其中932对(45.7%)扩增成功,8对引物中248对(12.1%)多态美国oblata个人。此外,利用30个多态EST-SSR标记对192个栽培品种的遗传多样性和群体结构进行了评价美国oblata个人。共检测到234个等位基因,PIC值为0.23 ~ 0.88,平均值为0.51,表明该栽培群体具有较高的遗传多样性。群体结构分析表明,伴生群体有两个主要亚群。最后,利用混合线性模型鉴定出20个显著关联,涉及17个标记和9个花性状。此外,标记SO104、SO695和SO790与多个性状具有显著的亲缘关系。
结论
结果表明,新开发的标记为丁香的遗传育种提供了宝贵的资源和有力的工具。本研究为进一步促进丁香花性状的遗传改良提供了有效的基础。
背景
丁香属紫丁香(榆科),是一种重要的木本观赏植物。大约有27种野生的紫丁香被描述过的,其中大部分原产于中国[1].中国是世界丁香的集散地和产地中心,有1000年的栽培历史。经过近100年的培育,丁香品种约有2000个,形态和颜色各不相同,在世界各地普遍种植[2].s . oblata一种多年生落叶灌木,因其花期早,色泽优雅,香气独特,抗旱性强,在中国北方被广泛栽培为园林植物[3.].作为北京地区土生土长的早开花树种,美国oblata被确定为庭院绿化的重要树种[1].
与其他木本花卉类似,许多丁香新品种都是用传统育种方法培育出来的[4].虽然传统的杂交育种在改善丁香观赏性状方面发挥了重要作用,但由于育种周期长,培育新品种需要大量资源,已不能适应现代丁香产业的快速发展[5].与此同时,美国oblata作为一种异交植物,许多重要的观赏性状是定量的,很容易受到环境的影响。此外,对菊科植物重要观赏性状的遗传结构还知之甚少S.oblata,如花期、花瓣颜色、花序等。因此,这意味着迫切需要了解重要性状的遗传结构,利用现代工具缩短丁香育种周期,提高育种过程。
标记辅助选择(MAS)育种通过直接选择目标基因型,缩短所需时间,从而加快过程,提高育种效率[6].MAS的一个重要基础是确定与目标性状相关的分子标记。然而,由于缺乏与目标性状相关的标记,在一定程度上阻碍了丁香的遗传选择和育种。关联作图是了解复杂数量性状及其遗传变异的有效途径[7].近年来,它已被应用于观赏植物中重要数量性状位点的挖掘研究向日葵[8),李属却已[9),而Lagerstroemia籼[10].这些研究表明,关联作图可以有效地找到与控制目标性状的基因密切相关的分子标记,为MAS奠定了基础。关联映射依赖于已有的丰富的自然变异,映射精度高[11].
随着分子生物学的发展,许多类型的分子标记被开发出来并广泛应用于观赏植物的MAS育种[12,13,14].分子标记如等位酶和扩增片段长度多态性(AFLP)已被用于分析植物的遗传多样性紫丁香及其亲缘种[15,16,奠定了理论基础紫丁香繁殖。然而,与这些DNA标记相比,简单序列重复序列(SSRs)标记因其共显性、多等位基因、稳定性、广泛的基因组覆盖和易于检测等优点而被认为是MAS中理想的分子标记[17].SSR标记又称微卫星标记,是1 ~ 6个核苷酸的短序列重复序列,广泛分布于真核生物基因组的编码区和非编码区[18].近年来,通过数据挖掘,在不同的观赏植物中成功地发现了SSRs木兰ashei[19),Lagerstroemia籼[20.),牡丹[21),而罗莎矮牵牛[22].此外,SSRs还广泛应用于植物遗传育种中进行遗传多样性分析[23],品种鉴定[24], DNA指纹[25,链接映射[26,以及关联映射[27].到目前为止,仅开发了14对多态SSR标记美国寻常的[28),美国josikaea[29],然而,由于普遍缺乏基因组和转录组信息,它在很大程度上受到限制。目前还没有关于SSR标记的报道美国oblata。因此,在丁香育种中开发MAS的SSR标记仍然是必要的。
近年来,基于丰富的转录本序列,下一代测序(NGS)技术使大量SSR得以发展[30.,31,32].我们实验室之前研究了美国oblata利用RNA-seq技术对不同发育阶段的花进行分析,共鉴定出104,691个unigenes(接入号:SRP063913) [33].同时,一组192个不相关的个体培养美国oblata在花性状中表现出丰富的表型变异,采样进行关联映射。本研究的目的是(I)开发表达序列标记- ssr (EST-SSR)标记美国oblata转录组序列;(二)评价植物的遗传多样性和种群结构美国oblata耕地数量;(3)鉴定与花性状相关的SSRs标记;(4)探讨等位基因对花性状自然变异的影响。我们的研究结果为我国的群体遗传学研究提供了宝贵的资源美国oblata,从而加快基因型筛选的速度,为MAS育种提供平台。
结果
EST-SSRs的频率和分布美国oblata
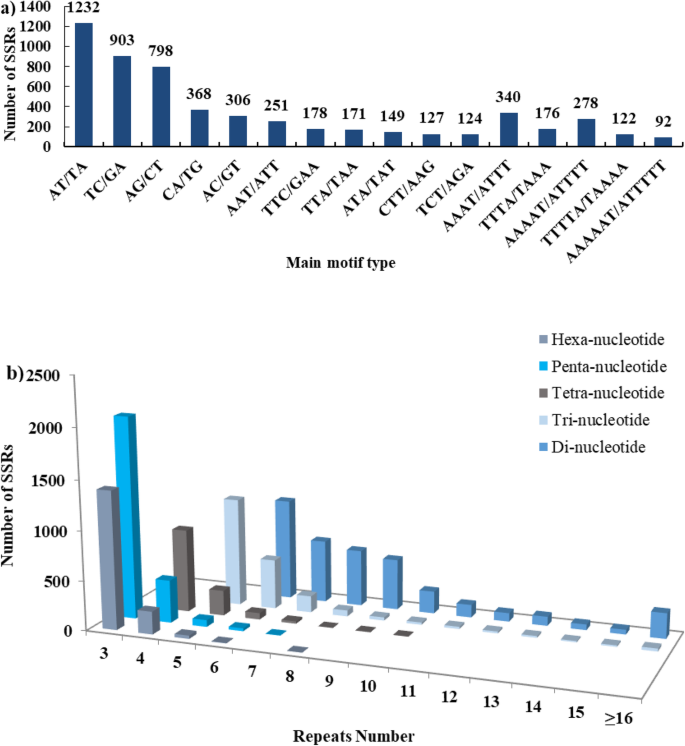
在本研究中,使用简单序列重复识别工具(SSRIT)扫描了104,691个unigenes,总长度为89.3 Mb [34从9864个(9.4%)unigenes中发现了10,988个潜在EST-SSRs,平均每8.13 kb有1个SSR。其中977个unigenes包含一个以上的EST-SSR位点。在这些潜在SSRs中,双核苷酸重复序列最丰富,频率为32.86%(3611),其次是五核苷酸重复序列(23.25%,2555)、三核苷酸重复序列(18.08%,1986)、六核苷酸重复序列(15.06%,1655)和四核苷酸重复序列(10.75%,1181)(表4)1).
无花果。1重复基序中以A为主。以AT/TA基序(11.21%,1232)最多,其次为TC/GA(8.2%, 903)、AG/CT(7.3%, 798)、CA/TG(3.3%, 368)、AAAT/ATTT(3.1%, 340)、AC/GT(2.8%, 306)、AAAAT/ATTTT(2.5%, 278)和AAT/ATT(2.9%, 251)(图4)。1a). 96.6%的SSRs长度为12 ~ 30 bp,其中最常见的是18 bp, 3.4%的SSRs长度为31 ~ 75 bp。数字1B表示3个串联重复序列(31.1%,3418)最多,其次是6个串联重复序列(14.7%,1610)、4个串联重复序列(1490,13.6%)和5个串联重复序列(13.1%,1440)。然而,超过15个串联重复序列的基序却非常罕见(2.16%)(图1)。1b)。
EST-SSR标记开发及多态微卫星位点筛选
除去侧翼序列短或不符合引物设计条件的est后,共筛选出2042个EST-SSRs用于引物合成。其中932对(45.7%)引物条带清晰可重复,其中二核苷酸324对,三核苷酸223对,四核苷酸88对,五核苷酸161对,六核苷酸136对。关于932个EST-SSR引物的信息见表S1.另外,245对引物产生的片段比预期的要大。其余865对引物在不同的扩增条件下没有产生条带或产生多个条带,这可能是由于序列或引物的装配错误。因此,不考虑进一步的分析。所有932对引物在8个引物中进行多态性筛选美国oblata248个(12.1%)基因型通过PAGE产生可重复的多态产物,包括110个二核苷酸、49个三核苷酸、18个四核苷酸、47个五核苷酸和24个六核苷酸。多态性比分别为34.0、22.0、20.5、29.2和17.6%。
遗传多样性与种群结构
根据Hardy-Weinberg平衡法,利用30个多态EST-SSR标记对192个品种的遗传多样性和群体结构进行了评价美国oblata个人。结果表明,共检测到234个等位基因,平均等位基因数(N一个)为7.8,介乎3至162).此外,观察到的杂合度(HO)和期望杂合度(HE)分别为0.21至0.87(平均0.52)和0.25至0.89(平均0.56)。其中,何低于HE,说明近交主要影响栽培种群美国oblata.这些位点的Shannon信息指数在0.53 ~ 2.33之间,平均值为1.15。SO711 ~ SO525的多态指数含量(PIC)为0.23 ~ 0.88,平均值为0.51。近93%(28个)EST-SSR标记具有较高或中等水平的遗传信息(PIC > 0.25),只有两个标记(SO310和SO711)多态性水平较低(PIC < 0.25)。这表明这些基因座包含了丰富的遗传信息,可用于遗传多样性研究紫丁香种质。
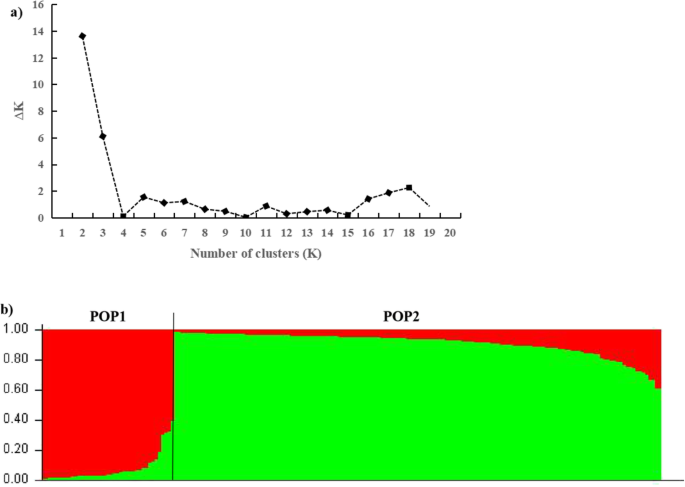
群体结构的存在会导致连锁不平衡(LD)水平的增加,这可能导致目标性状与不相关位点之间的相关性。因此,对种群结构进行分析和调整是进行关联分析的前提。利用structure 2.3.4对192个个体的30个多态标记进行种群结构分析,在该值处得到一个清晰的峰值K= 2。2a)使用Evanno等人的统计模型。[35].因此,这192个个体可分为两个亚群体,即红色指定的POP1(41个)和绿色指定的POP2(151个)。如图所示。2B,每个个体用一条细竖线表示,并根据其估计的成员概率(问),用于基于结构的关联映射。主成分分析(PCA)作为STRUCTURE算法的一种替代策略,被广泛用于种群亚种群的识别。PCA将关联人群分为两个亚人群(图S1),其聚类结果与使用STRUCTURE得到的聚类结果相似。
表型性状分析与单标记关联
目标性状在群体中的变异程度是关联映射的重要参数。数量性状变异系数为19.72 ~ 30.22%,分布统计值见表S2.不同性状间的相关分析显示有14个显著相关(P< 0.05),其中12个呈高度显著相关(P< 0.01S3).其中,花冠长度、花冠宽度、花冠裂片长度、花冠裂片宽度和花冠管长度均呈极显著正相关。另外,花冠叶(状态)与花冠叶(外围)之间呈极显著正相关。表中列出了关联群体中9个性状间表型相关性的更多细节S3.同时,对192个个体的9个表型性状进行Q聚类分析,发现群体分为两个亚群,这与基于EST-SSR标记的结构分析结果相似(图1)S2).
对于关联映射,理解LD的模式是一个重要的前提。利用119个小等位基因频率(MAF) > 1%的多态标记分析了192个栽培品种的LD水平美国oblata个人。结果表明r2所有位点对的范围从0.0001到0.5154。LD水平较低,多数标记处于连锁平衡状态(r2< 0.1;P< 0.001)。899个位点对存在连锁不平衡(P< 0.001), 830例r2> 0.005(83.1%)(图5)3.).当然,也发现在一些SSR位点中存在较强的LD水平,如标记SO015-SO428 (r2> 0.3;P< 0.001)。
基于混合线性模型(MLM)对119个ssr与9个性状进行关联分析。共进行了1071项单标记相关性试验,其中20项相关性显著(P的显著性水平(< 0.01)问< 0.01,涉及9个性状,17个ssr。对于每个特征,显著关联的数量从0到4个不等。这些位点解释的表型方差范围为0.36 ~ 20.76%,平均率为5.69%(表2)3.).其中4个SSR标记分别与花冠叶(状态)和花冠叶宽度显著相关。花冠叶(外围)有三个显著的关联;花序长度、花冠叶长度和花瓣颜色各有显著的相关关系;在关联群体中,花冠宽度、花冠筒和花期均有显著相关性(问< 0.01;表格3.).在本研究中,3个ssr(标记SO104、SO695和SO790)表现出与多个性状的显著相关性,表明某些性状的基因组区域具有多效性或连续性。在20个关联中,有8个关联的基因作用模式与过显性或过显性一致;其余12个标记在基因作用模式中分离,分别为加性(7)或部分至完全显性(6)3.).
讨论
深度转录组测序以指数级的方式产生大量的数据,为开发新的SSR标记和识别新基因提供了全面的信息和良好的资源[38,39,40].目前,大量基于转录组序列的SSR标记已被成功开发,并用于许多生物的功能变异检测和基因相关遗传分析,加速了MAS育种的进展[41,42,43].在本研究中,我们首次报道了美国oblata基于NGS技术生成的转录组序列。
从9864个转录组序列中共鉴定出10988个EST-SSR位点,约占该基因转录组的9.4%美国oblata,这与以往双子叶植物基因座频率为2.65 ~ 16.82%的报道一致[44].分布密度为1个SSR / 8.13 kb,低于文献报道大sibiricus(1/6.2 kb) [45),黎豆属pruriens(1/5.3 kb) [46,但高于紫花苜蓿(1/12.06 kb) [47),莲属椰子(1/13.04 kb) [48].SSR位点的出现频率在不同物种间差异很大,这可能与数据集的规模、SSR搜索标准以及SSR搜索使用的挖掘工具有关。先前的研究表明双核苷酸重复序列是双子叶植物中最常见的SSR重复类型[44,49,50,51],以及本研究,可能是由于过度表达的非翻译区域(utr) [49].在观察到的双核苷酸中,AT/TA(34.1%)最多,与芸苔属植物[52],这可能是由于Ile (AUA)和Tyr (UAU)氨基酸的高频率,因为它们的密码子携带这些基序[53].此外,与双子叶植物GC重复序列的低覆盖率一致,GC/CG的频率最低(0.2%)出现在美国oblata.这可能是由于胞嘧啶的甲基化,它抑制了许多植物的转录[54].通过比较发现,不同的物种有不同的重复类型和母题可能是由于不同的选择标准。
设计的2042条EST-SSR引物中,932条(45.7%)EST-SSR标记被成功扩增。扩增率高于芸苔属植物定(16.68%) (55,但低于虹膜(76%) (56),野菊花(88.6%) (57].本研究扩增率略低,可能与内含子含量和引物筛选标准严格有关。据我们所知,几乎没有任何关于开发广泛的EST-SSR标记的研究紫丁香.此外,有12.1%(248)个EST-SSR标记具有多态性s . oblata这低于Catharanthus roseus也叫[5852.7%的人Melilotus白色[59),分别。在本研究中,EST-SSRs的多态性比例处于相对较低的水平,可能受所用材料数量和地理来源狭窄的影响。
为了建立关联作图和MAS育种计划的基础,我们根据HWE平衡选择了30个多态标记来评估遗传多样性和群体结构美国oblata.192个个体共获得234个等位基因,平均每个位点等位基因数为7.8个,高于N一个(3.44)在75个基因型美国寻常的它们是由9个多态SSR标记确定的[28].平均等位基因数(NA)为7.8可能与较大的样本量和使用的多态SSR标记有关。然而,与美国josikaea(14.2),每个位点低7.8个等位基因[29].这一HE低于HO9个SSR位点的杂合子数量显著超标。但HO低于HE在其他20个SSR位点上,表明在群体中存在近亲繁殖。PIC值也是衡量遗传多样性的重要指标,SSR标记多态性介于0.23 ~ 0.88之间,均值为0.51,与前人研究结果一致[60].对30个多态SSR标记在转录组序列中的位置分析表明,18个标记(60%)存在于UTR中。本研究所使用的SSR位点的多态性程度相当可观,这可能是由于大多数多态SSRs都发生在UTR中。同时,30条PIC大于0.5的引物中,有15条引物作为标记具有良好的信息性。这表明,新开发的EST-SSR多态性标记为进一步的遗传分析提供了信息和有效的方法。
估计种群结构可以避免假阳性关联,提高关联映射的效率[61,62].因此,亚群体的选择对关联结果至关重要。例如,有报道称,水稻亚群体结构的选择决定了该标记是否与性状强烈相关[63].结构模型通过引入种群结构并试图寻找处于平衡状态的子群来解释HWE或LD的存在[64].因此,在结构分析中的群体亚群符合HWE,在192个个体中得到两个不同的亚群体。被分配到子组的个体模式与它的来源一致。在未来的研究中,将利用更多不同的野生基因型,在遗传多样性的基础上设计LD制图方案,以提高现有遗传资源的利用率美国oblata.此外,详细了解关联群体中物种的LD水平是提高标记-性状关联分辨率的重要前提。在本研究中,LD的低水平可能是由于美国oblata是一种异交植物,其重组率高。以前的研究表明木本植物的LD很低[65,66].例如,火炬松的LD在几个碱基的范围内迅速下降[67],在其他针叶树物种中候选基因中也存在类似的LD有限的结果[68,69,70,71].
美国oblata因其花期早、色泽淡雅、香味独特而广受欢迎。丰富的种质资源是现代育种的物质基础,种质资源的多样性直接关系到种质资源的有效利用。表型性状分析是接触种质资源最基本的方法[72].5个数量性状的变异系数(CV)在19.72 ~ 30.22%之间,与前人的研究结果一致美国oblata[73,74].改善紫丁香花性状,培育紫丁香新品种是育种工作者长期面临的课题。关联映射被广泛用于确定潜在目标位点和重要性状之间的关联[75,76,77].与单个显著标记产生的基于单倍型的关联相比,单标记关联在这种低LD树种中可能更强大[66].本研究以群体结构和亲缘关系为协方差,在单标记关联的基础上,采用MLM模型确定花性状的关联创客。同时,为了进一步提高关联结果的准确性,采用FDR方法对关联结果进行校正P价值观对所有人的交往有很多场合,这就大大降低了通货膨胀的程度P值。最终鉴定出17个SSR标记,这些SSR标记与杂交种的花性状存在显著相关。我们确定了多个显著标记可能是由于标记是发展自转录组序列美国oblata花(33].对于许多植物,特别是在关联分析中含有复杂数量性状的植物,一个巨大的挑战是大量的基因座几乎没有影响[78].发现了许多显著的关联,部分解释了一小部分表型变异,这表明许多基因控制位点与相对较小的个体效应相关。木本植物数量性状的遗传研究也报道了类似的结果,反映了一个多基因定量模型[79,80,81,82].
有趣的是,我们发现标记SO104、SO695和SO790与多个性状显著相关,这与这些性状之间显著的表型相关是一致的。这些多效性关联可能有助于识别重要的基因组区域[7].了解这些植物区系性状的遗传控制,可以进一步推断与性状相关的SSR的重要性[83].花冠筒长与加性模型的SO649相关,解释了20.76%的表型方差,使该标记在MAS育种中具有吸引力。花期和花瓣颜色是其重要的观赏性状,在很大程度上决定了其观赏价值。标记SO790与花期显著相关,显示出与超显性一致的基因作用模式。花瓣颜色与标记SO104和SO805高度显著相关,表现为基因作用模式,表型累积方差为6.2%。利用累积了多个SSR遗传效应的相加模型来解释显著的性状遗传力,使MAS在植物育种中具有吸引力[11].在20个关联中,有8个基因的作用模式符合过/劣势优势。与相应的纯合子个体相比,杂合子个体在超显性等位基因效应中对花的观赏价值贡献更大。这样的个体在群体遗传学的背景下有利于保存遗传变异,并可能由于包含两种等位基因而拥有自然优势(超显性选择)[84].同时,为了探索这17个含有ssr的多态序列的潜在功能,在GenBank中使用BLASTX对17个转录组序列进行了搜索S4).十个序列与齐墩果欧洲公司var。结果表明蛋白质。SO649标记的转录组序列被注释为E3泛素蛋白连接酶。SO649标记的转录组序列是一个含有B3结构域的蛋白,在胁迫反应和植物生长发育中起着极其重要的作用[85].SO415的转录组序列被注释为转录抑制子OFP15与花的发育有关拟南芥[86].综上所述,鉴定出与花性状相关的EST-SSR标记美国oblata利用关联映射法,为进一步分析以改良花性状为目标的MAS育种方案奠定基础。
结论
综上所述,共开发了932个SSR标记美国oblata其中多态性引物248条。这些新开发的EST-SSR标记为植物的遗传多样性、比较基因组学、基于基因的关联研究和标记辅助选择提供了重要的资源紫丁香。这是第一个关于关联映射的报告美国oblata共鉴定出17个与花性状相关的SSR标记。这些结果将在未来的MAS育种计划中发挥重要作用。
方法
植物材料和DNA提取
收藏1900年栽培美国oblata采自华北河北、辽宁两地,在北京农学院科研单位北京安吉明劳务有限公司(40°15′n, 116°60′e)田间,采用标准农艺栽培措施栽培。所有植株均为9龄左右,生长良好,开花正常,花性状组合稳定。八个美国oblata随机选取个体进行SSR标记开发和多态性标记筛选。一套192个美国oblata个体从收集中选择作为关联种群,展示各种花朵的颜色和形状。使用DNA安全植物试剂盒(天根生物技术,北京,中国)从幼叶中提取基因组DNA。使用2%琼脂糖凝胶和NanoDrop ND-1000紫外/可见分光光度计(Wilmington, DE)分别检测DNA的质量和数量。DNA工作浓度稀释至25 ng/μL。
表型的测量
在192个群体个体中对9个性状进行了评分,其中包括5个数量性状和4个质量性状,每个基因型至少有5个重复。花期性状包括花长、花宽、花冠叶长、花冠叶宽、花冠管长、花冠叶(状态)、花冠叶(外围)、花期和花瓣颜色。5个定量性状均采用数字卡尺(YB5001B, kraf十二维实业有限公司,中国)测定。使用色度计(CR-400, Konica Minolta Holdings, Inc., Japan)在每个基因型的5个花序(包括25个小花)测量花瓣颜色值,并将其分为6组(表1)S5)根据L *,一个*,b *测量生成。花冠叶(状态)、花冠叶(外围)和花期的测量依据是《清晰度、均匀性和稳定性测试指导方针-》紫丁香。所有测量结果见表S5.采用SPSS统计软件20对各性状进行方差分析、表型相关和Q聚类分析。
est - ssr的鉴定和验证
共得到104,691个unigenes美国oblata花发育三个不同阶段的转录组序列。RNA-seq数据已经提交到NCBI序列读取档案(SRP063913,https://trace.ncbi.nlm.nih.gov/Traces/sra/?study=SRP063913) [33].简单序列重复识别工具程序(SSRIT) (http://www.gramene.org/db/markers/ssrtool)用于在转录组序列中搜索SSRs [34].SSR检索标准基于完美二、三、四、五、六核苷酸基序的最小重复数分别为6、5、4、3、3。Primer Premier 5.0软件(Premier Biosoft International, Palo Alto, CA, USA)用于在ssr的侧翼区域设计引物。引物由北京瑞比奥生物技术有限公司合成。为了预测SSR是否存在于编码序列(CDS)或非翻译区(UTR), ORF Finder软件(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)用于识别EST序列中的起始和终止密码子。
聚合酶链式反应(PCR)扩增在10 μL反应体系中进行,其中包括1 μL 25 ng/μL DNA, 0.5 μL 10 μM每个引物,5 μL 2 × Power Taq PCR Master Mix (Aidlab,北京,中国)和3 μL无菌蒸馏水。PCR在以下条件下进行:95℃5min,然后在95℃30s循环30次,适当的退火温度(表S1) 30秒,72°C 1分钟,最后在72°C延长10分钟。PCR产物在2%琼脂糖凝胶上进行检测。成功的扩增产物用6%变性聚丙烯酰胺凝胶电泳(PAGE)分离,然后用银染色观察。为了确定结果的重复性,每对引物扩增3次。最后,鉴定了一个多态SSR引物子集,并将其指定为“验证标记”。
随机选取119条多态SSR引物加入荧光染料进行群体基因分型。所有荧光引物均由北京瑞博生物技术有限公司(北京,中国)合成。使用ABI3730xl DNA分析仪(Applied Biosystems, Carlsbad, CA, USA)进行毛细管电泳分离产物。多态位点分析使用GeneMapper 2.2.0版本的LIZ 600尺寸标准(Applied Biosystems)软件进行。
遗传多样性与群体结构分析
遗传多样性参数,包括等位基因数量(N一个),有效等位基因数目(NE),观察到的和预期的杂合度(何而且HE,分别),以及各标记的Shannon信息指数,采用POPGENE 1.31版本计算[87].利用PowerMarker 3.25版本计算各标记的多态性信息含量(PIC)、Hardy-Weinberg平衡(HWE)和小等位基因频率(MAF) [88].
192人的遗传结构美国oblata采用基于马尔可夫链蒙特卡罗(MCMC)算法和贝叶斯框架的STRUCTURE v2.3.4对个体进行评估。假设一个外加剂模型样本,为的每个值执行20个独立的运行K从1到20,每个都有10万的磨合周期,然后进行50万次迭代。通过这种方式,种群成员估计(问矩阵),为关联映射提供隶属度百分比。使用多变量统计包(MVSP) 3.1版本(科瓦奇计算服务公司,安格尔西,威尔士,英国)评估PCA。
Marker-trait协会
采用TASSEL 2.1版软件进行连锁不平衡分析5排列来计算r2对SSR标记之间的值(次要等位基因频率> 1%)[89].对位点被认为有一个显著的LD时P< 0.001。混合线性模型(MLM)考虑了问利用TASSEL 2.0.1版本的亲缘矩阵和亲缘矩阵来揭示花性状与SSR标记的相关性。的问矩阵由STRUCTURE得到。亲属矩阵(K)的估计,采用的是SPAGeDi软件版本1.2 [90].选择等位基因与性状间关联的显著阈值为P< 0.01。最后,使用错误发现率(FDR)对多个比较进行校正问R的值[91].显性基因(d)与加性基因(a)的比值用来衡量显著位点的基因效应。|d/a|≤0.5定义为加性效应,0.50 < |d/a| < 1.25为部分或完全优势,|d/a| > 1.25为不足或过度优势[92,93].计算基因效应的具体算法和公式如前所述。为了确定含有多态微卫星位点的单基因序列的推定功能,对这些序列进行了分析www.ncbi.nlm.nih.gov)非冗余蛋白数据库使用BLASTX搜索。
数据和材料的可用性
本文设计的引物包含在本文及其附加文件中。支持这项研究发现的RNA-seq数据已存入NCBI序列读取档案(SRP063913,https://trace.ncbi.nlm.nih.gov/Traces/sra/?study=SRP063913).支持这些研究结果的材料均不含野生资源,均为植物的栽培种质资源美国oblata.北京安吉明劳务有限公司完全遵守机构、国家或国际的指导方针,并获得相应的许可和营业执照。
缩写
- MAS:
-
分子标记辅助选择
- 苏维埃社会主义共和国:
-
简单序列重复
- RAPD:
-
随机扩增多态DNA
- 妊娠:
-
扩增片段长度多态性
- 美国东部时间:
-
表达序列标记
- 门店:
-
新一代测序
- N一个:
-
观察值
- NE:
-
有效的数量
- HO:
-
观察到的杂合性
- HE:
-
预期的杂合性
- 我:
-
香农信息指数
- 图片:
-
多态信息内容
- 传销:
-
混合线性模型
- 罗斯福:
-
错误发现率
- 密度:
-
马尔可夫链-蒙特卡洛
- LD:
-
连锁不平衡
- 加:
-
次要等位基因频率
- HWE:
-
哈迪温伯格平衡
参考文献
- 1.
崔海霞,蒋刚,臧思云。的分布、起源和演变紫丁香.公牛学报2004;24:2。
- 2.
张少云,崔海霞。《丁香》第一版。上海:上海科学技术出版社;2000.
- 3.
魏泽平,李秋峰。丁香种质资源的保存、育种与推广研究。农业科学2007;24(2):77-80。
- 4.
十、采收及景观应用紫丁香北京市种质资源分析。《北京花园》第二卷;2011.
- 5.
薛超,焦华,张伟,孙煜,曲松,庄铮,何敏紫丁香繁殖。天津农业科学2015;21(10):121-5。
- 6.
戴荣强,张亮,胡德强,赵文伟,潘刚,刘磊。植物分子育种研究进展。安徽农业科学,2009;32:073。
- 7.
杜启智,徐波波,潘伟,龚春荣,王启新,田建新,李伯林,张德强。纤维素合酶基因的等位基因变异(PtoCesA4)与生长和木材特性有关Populus tomentosa.G3基因学报,2013;3(11):2069-84。
- 8.
Fifippi CV, Aguirre N, Rivas JG, Zubrzycki J, Puebla, Cordes D, Moreno MV, Fusari CM, Alvarez D, Heinz RA, Hopp HE, Paniego NB, Lia VV。向日葵群体结构及遗传多样性特征的SSR和SNP标记定位群体。BMC Plant Biol. 2015;15:52。
- 9.
马凯峰,孙立德,程建华,潘海涛,王杰,张庆祥。在分布广泛的观赏树居群中,表观遗传变异与遗传结构具有协同作用李属却已植物科学2018;9:41。
- 10.
郑涛,秦波,李淑贞,蔡明,潘海涛,王杰,程建华,张庆祥。与紫薇匍匐性状相关的SSR分子标记筛选。森林。2019;10:429。
- 11.
吴军,程飞,蔡芳芳,钟勇,杰新。栽培植物花性状的关联定位芍药属rockii基于SSR标记。中国生物医学工程学报,2016;29(1):414 - 414。
- 12.
苏俊杰,张芳,李培瑞,关志智,方伟明,陈fd。菊花耐涝性的遗传变异与关联定位。足底。2016;244:1241-52。
- 13.
杨燕,宣林,余春春,王振英,徐建华,范卫伟,郭建军,尹永良。水稻生长性状高密度遗传图谱构建及数量性状位点鉴定Taxodium distichumvar。distichum×t . mucronatum)×t . mucronatum.BMC Plant Biol. 2018;18:263。
- 14.
张芳,康建民,龙景荣,于丽霞,王铮,赵志祥,张廷杰,杨qc。同源四倍体紫花苜蓿产量和产量组成成分的高密度连锁图谱构建及RAD-seq QTL定位。BMC Plant Biol 2019;19:165。
- 15.
明杰,顾wc。自然居群的遗传多样性紫丁香oblataAFLP标记检测。园艺学报2006;33:61269-74。
- 16.
廖红,顾伟,明杰。天然居群遗传多样性的确定紫丁香oblata使用等位酶标记。北京林业大学学报2009;31(5):84-9。
- 17.
Jose AG, Concepción M, Francisco M.植物分子标记研究的趋势。足底。2017;247:543-57。
- 18.
Gupta PK, Balyan HS, Sharma PC, Ramesh B.植物中的微卫星:一类新的分子标记。科学通报1996;45:145-54。
- 19.
Kohn CV, Conrad K, Kramer M, Pooler M.木兰ashei以SSR标记为特征。2018; 19:4923-36。
- 20.
刘勇,何东,蔡明,唐伟,李晓霞,潘海涛,张庆祥。微卫星标记的研制Lagerstroemia籼(荔枝科)及其相关种。应用植物学报2013;1:2。
- 21.
郭丽丽,郭德龙,赵伟,侯小刚。新开发的SSR标记揭示了植物的遗传多样性和地理聚类特征芍药属suffruticosYea根据花朵的颜色。中国生物医学工程学报。2018;93:4。
- 22.
齐卫军,陈旭,方福平,石淑春,李建军,刘晓婷,高晓强,赵楠,郝海辉,李玉军,韩玉军,张卓罗莎矮牵牛为花卉育种、性状改良和分类学研究提供微卫星标记。BMC Plant Biol. 2018;18:119。
- 23.
加迪萨,Tesfaye K, Dagne K, Geleta M.植物遗传多样性与种群结构分析属可食的(Vatke) Agnew使用新开发的EST-SSRs标记系统从埃塞俄比亚的不同农业生态中收集。BMC Genet. 2018;19:92。
- 24.
Pinto MV, Poornima HS, Sivaprasad V, Naik VG。一套应用于桑树品种鉴定和DUS检测的新SSR标记。J Genet。2018;97。
- 25.
甜樱桃近缘品种DNA指纹图谱研究。中华科学杂志2018;143:4282-8。
- 26.
Fenton ME, Owens BF, Lipka AE, Ortiz D, Tiede T, mateo - hernandez M, Ferruzzi MG, Rocheford T.玉米籽粒维生素E含量的高密度连锁定位。2018;31。
- 27.
李培瑞,苏建军,关志英,方伟明,陈丰德,张峰。切花菊耐旱性的关联分析(菊花)在苗期。生物技术。2018;8:226。
- 28.
Juntheikki-Palovaara I, Antonius K, Linde 'n L, Korpelainen H.普通紫丁香的微卫星标记(紫丁香寻常的l .)。植物遗传资源,2013;11:3279-82。
- 29.
Lendvay B, Pedryc A, Höhn M.狭窄地方病核微卫星标记的表征紫丁香josikaeaJacq。费尔。Rchb交货。植物学报,2013;41:1301-5。
- 30.
Park JH, Ahn SG, Choi YM, Oh HJ, Ahn DC, Kim JG, Kang JS, Choi YW,郑BR。玫瑰(罗莎矮牵牛l .)est衍生微卫星标记及其在草莓(草莓属spp)。科学通报,2010;125:733-739。
- 31.
叶玉军,冯丽,梁晓红,刘婷婷,蔡明,程建华,王杰,张庆祥,潘海涛。EST-SSR标记的鉴定、验证和跨种转移性及其在紫薇遗传评价中的应用(Lagerstroemiaspp)。Mol Breed. 2019;39:26。
- 32.
引用本文王晓亮,陈卫武,罗杰,姚志祥,于强,王永良,张淑珍,刘志刚,张锐,沈永明。EST-SSR标记的开发及其在濒危植物遗传多样性分析中的应用木兰sinostellata.Mol Gen Genomics, 2018; 294:135-47。
- 33.
郑军,胡振华,关晓玲,窦德强,白刚,王赟,郭东涛,李伟,冷培生紫丁香oblata采用。花序识别与色素生物合成和气味代谢相关的基因。科学通报,2015;10(11):e0142542。
- 34.
Temnykh S, DeClerck G, Lukashova A, Lipovich L, Cartinhour S, McCouch S.水稻微卫星的计算和实验分析(栽培稻L.):频率、长度变异、转座子关联和遗传标记电位。基因组学报2001;11(8):1441-52。
- 35.
王晓燕,王晓燕,王晓燕,等。利用结构化软件检测个体簇数的仿真研究。分子生物学杂志2005;14(8):2611-20。
- 36.
埃克特AJ,鲍尔AD,韦格琴JL,潘德B,杰姆斯塔德KD,克鲁托夫斯基KV, StClair JB,尼尔DB。海岸花旗松(黄豆属门齐西属变种松科)。一、抗寒相关性状。遗传学。2009;182:1289 - 302。
- 37.
Wegrzyn JL, Eckert AJ, Choi M, Lee JM, Stanton BJ, Sykes R, Davis MF, Tsai CJ, Neale DB。2010.黑杨木木质素和纤维素生物合成控制性状的关联遗传(杨树trichocarpa,水杨科)次生木质部。新植物学报2010;188:515-32。
- 38.
陈宏,王磊,王松,刘超,Blair MW,程欣。绿豆转录组测序(豇豆属辐射L.)基因和EST-SSR标记的鉴定。2015;10。
- 39.
安敏,邓敏,郑ss,宋永刚。橡树SSR标记的转录组从头组装与开发Quercus austrocochinchinensis而且问:kerrii(壳斗科).树Genet基因组。2016;12:103。
- 40.
邢伟,廖建勇,蔡爱梅,夏秋峰,刘勇,曾伟杜鹃latoucheae利用Illumina测序和开发新的EST-SSR标记进行杜鹃属植物遗传多样性分析。树Genet基因组。2017;15:53。
- 41.
马建强,姚明志,马春林,王晓春,金建强,王晓明,陈磊。基于ssr的茶树儿茶素含量遗传图谱构建及qtl鉴定(茶树).PLoS One, 2014;9:3。
- 42.
蔡凯,朱丽芳,张奎奎,李亮,赵志英,曾伟,林小春。从植物RNA-Seq数据中开发EST-SSR标记并进行表征植被类型violascens.植物科学进展2019;10:50。
- 43.
阿里N,李德龙,Eltahawy MS, Abdulmajid D, Bux L,刘伯伯,党新军,洪德龙。挖掘有利于种子储备利用效率的等位基因栽培稻通过关联映射的方法。BMC Genet. 2020;21:4。
- 44.
双子叶植物表达序列标签中简单序列重复的挖掘与研究。基因组。2005;48:6985 - 98。
- 45.
张振英,谢文伟,赵永强,张景春,王宁,Ntakirutimana F,闫建军,王yr .基于rna测序的EST-SSR标记开发E sibiricus及其在17种植物系统发育关系分析中的应用大物种。BMC Plant Biol 2019;19:235。
- 46.
Sathyanarayana N, Pittala RK, Tripathi PK, Chopra R, Singh HR, Belamkar V, Bhardwa PK, Doyle JJ, Egan AN。药用豆科植物的转录组资源黎豆属pruriens: EST-SSR标记的从头转录组组装、注释、鉴定和验证。BMC基因组学,2017;18:409。
- 47.
王铮,闫辉,付旭,李旭,高辉。苜蓿简单序列重复标记的开发及多样性分析(紫花苜蓿l .)。Mol Biol rep 2013; 40:4291-3298。
- 48.
潘磊,夏强,全智,刘宏,柯伟,丁勇。圣莲新型EST-SSRs的研究进展(英文)莲属椰子Gaertn)及其在植物遗传多样性分析中的应用n .椰子.中华医学杂志2010;101(1):171-82。
- 49.
吴娟,蔡凤峰,程飞,崔海林,周华。牡丹EST-SSR标记的转录组序列分析与开发。动物育种杂志,2014;34(4):1853-66。
- 50.
张爱梅,范丽,刘启忠。一套新的梨EST衍生SSR标记及其蔷薇科植物跨种转移性。植物生物学杂志2014;32:290-302。
- 51.
夏尔玛,王晓燕,王晓燕,王晓燕。茶叶中单基因微卫星标记的鉴定、鉴定及应用(英文)茶树l .)。植物生物学2009;9:53。
- 52.
石晶,黄思敏,詹建平,余建勇,王晓峰,华伟,刘少思,刘光华,王喆。全基因组微卫星的表征和测序标记的开发芸苔属植物作物物种。DNA Res. 2014; 21:53-8。
- 53.
Varshney RK, Thiel T, Stein N, Langridge P, Graner A.几种谷物est中微卫星频率和分布的硅分析。细胞生物学杂志2002;7:537-46。
- 54.
Katti MV, Ranjekar PK, Gupta .真核基因组序列中简单序列重复序列的差异分布。中华分子生物学杂志2001;18:1161-7。
- 55.
陈建峰,李荣荣,夏玉云,白光华,郭培刚,王志林,张华,Siddique KHM。大白菜EST-SSR标记的开发(芸苔属植物定l . ssp。chinensis var. utilis Tsen et Lee)的转录组组合。科学通报,2017;12(9):e0184736。
- 56.
Tang SX, Okashah RA, Cordonnier-Pratt MM, Pratt LH, Johnson VE, Taylor CA, Arnold ML, Knapp SJ。EST和EST- ssr标记资源虹膜.植物生物学,2009;9:72。
- 57.
韩喆,马晓霞,魏明,赵涛,詹仁涛,陈伟伟。SSR标记的开发与种内遗传差异的探讨野菊花基于转录组分析。BMC基因组学。2018;19:291。
- 58.
Kumar S, Shah N, Garg V, Bhatia S.从从头组装的转录组中大规模的硅内识别和表征简单序列重复(SSRs)Catharanthus roseus也叫(l)g .堂。植物细胞学报2014;33(6):905-18。
- 59.
阎卓,吴峰,罗凯,张艳芳,闫强,张艳芳,王雅尔,张建勇。番木瓜EST-SSR标记的跨种转移性Melilotus及其在群体遗传学研究中的应用。科学通报2017;7(1):17959。
- 60.
于海平,程飞,钟勇,蔡春芳,吴娟,崔海林。SSR (simple sequence repeat)标记的开发芍药属ostii研究芍药科牡丹的亲缘关系。科学通报,2013;164:58-64。
- 61.
King RA, Harris SL, Karp A, Barker JH。用于异源四倍体白柳-脆弱柳复合体种群研究的核微卫星位点的表征和遗传。植物学报,2010;6(2):247-58。
- 62.
丁杰,Ali F,陈刚,李宏,Mahuku G,杨宁,闫杰。全基因组关联图谱揭示了玉米对北方玉米叶枯病抗性的新来源。植物生物学杂志,2015;15(1):1。
- 63.
王旭,贾明明,盖平,李fn,贾勇。水稻稻瘟病抗性与产量相关成分的全基因组关联。生物学报2015;28:1383-92。
- 64.
王晓燕,王晓燕,王晓燕,等。基于多位点基因型数据的群体结构推断。遗传学。2000;155:945-59。
- 65.
杜启志,潘伟,徐波波,李伯林,张德强。纤维素合成酶中多态的简单序列重复位点(SSR) (PtoCesA)的基因与生长和木材特性有关Populus tomentosa.新植物学报,2013;197:763-76。
- 66.
杜启智,潘伟,田建新,李伯林,张德强。葡萄糖醛酸脱羧酶基因家族杨树:结构、表达和关联遗传学。公共科学图书馆。2013;8:4。
- 67.
布朗GR,吉尔GP,昆兹RJ,兰利CH,尼尔DB。火炬松核苷酸多样性与连锁失衡。中国科学(d辑:自然科学版)2004;
- 68.
Dvornyk V, Sirvio A, Mikkonene M, Savolainen o抗旱性.生物学报2002;19:179-99。
- 69.
针叶树复杂性状的关联遗传。植物学报2004;9:325-30。
- 70.
克鲁托夫斯基KV,尼尔DB。道格拉斯冷杉耐寒性和木材品质相关候选基因的核苷酸多样性和连锁失衡。遗传学。2005;171:2029-41。
- 71.
冈萨雷斯-马丁内斯SC,惠勒NC,厄索兹E,尼尔森CD,尼尔DB。关联遗传学松果体taeda木支柱性状遗传学。2007;175:399-409。
- 72.
Maryam F, Hossein SA, Ali K, Morteza A.桑属阿尔巴var。黑质多变量分析揭示的基因型。科学通报2019;248:41-9。
- 73.
明杰,顾wc。的表型变异紫丁香oblata采用。Res. 2006; 02:199-204。
- 74.
张旭,王媛媛,车德东。植物形态特征及变异分析紫丁香oblata在不同的生物群落中。中国观赏园艺研究进展。北京:中国林业出版社;2010.
- 75.
Zarbafi SS, Rabiei B, Ebadi AA, Ham JH。水稻叶瘟病相关性状的关联定位(栽培稻l .)。澳大利亚植物病理学。2020;49:31-43。
- 76.
引用本文聂刚,唐磊,张玉军,黄力克,马旭,曹旭,潘磊,张旭,张晓强。基于转录组测序的多年生草SSR标记及其与耐旱性的关联分析芒草来自中国。植物科学进展2017;8:801。
- 77.
易强,刘艳红,侯小斌,张小刚,李红,张建军,刘敏敏,胡艳芳,于格夫,李艳萍,王亚斌,黄亚斌。玉米产量相关性状的遗传解剖及其中亲本杂种优势(玉米l .)。BMC Plant Biol 2019;19:392。
- 78.
孙鑫,杜铮,任杰,Amombo E,胡涛,付杰。高羊茅不同品种热胁迫功能性状SSR标记的关联。植物生物学杂志,2015;15(1):116。
- 79.
González-Martínez SC,惠勒NC,厄索兹E,尼尔森CD,尼尔DB。关联遗传学松果体taeda李木的特性。遗传学。2007;175(1):399 - 409。
- 80.
Beaulieu J, Doerksen T, Boyle B, Clément S, Deslauriers M, Beauseigle S, Rigault P.松白云杉木材物理性状的关联遗传及其与基因表达的关系。遗传学。2011;188(1):197 - 214。
- 81.
Dillon SK, Brawner JT, Meder R, Lee DJ, Southerton SG。关联遗传学Corymbia citriodora变异亚株鉴定了影响木材生长和纤维素浆产量的单核苷酸多态性。植物学报,2012;29(3):596-608。
- 82.
Porth I, klapete J, Skyba O, Hannemann J, McKown AD, Guy RD, Friedmann MC.杨树木材特征的全基因组关联映射确定了一系列候选单核苷酸多态性。植物学报(自然科学版)2013;29(3):369 - 369。
- 83.
杜启智,王磊,周德龙,杨海军,龚春荣,潘伟,张德强。s -腺苷-低同型半胱氨酸水解酶基因家族等位基因变异与中国白杨木材性质相关(Populus tomentosa).BMC Genet 2014;15(增刊1):S4。
- 84.
变异大小种群中的超优势等位基因。遗传学。1999;152(2):775 - 81。
- 85.
罗光,叶立夫,陈小波。研究进展拟南芥B3转录因子基因超家族。化学生命。2013;33(3):287-93。
- 86.
黄建平,李海林,常燕。植物卵形家族蛋白的全基因组分析拟南芥.东北农业大学(英文版)。2012; 19(3): 49-59。
- 87.
叶长峰,杨景荣,波义涛,叶志华,毛建新。POPGENE,版本132:用于群体遗传分析的用户友好软件。分子生物学与生物技术中心。加拿大:埃德蒙顿阿尔伯塔大学;1999.
- 88.
刘凯,缪斯公司SV。PowerMarker:遗传标记分析的集成分析环境。生物信息学。2005;21(9):2128 - 9。
- 89.
Yu J, Pressoir G, Briggs WH, Bi IV, Yamasaki M, Doebley JF, Kresovich S.一种统一的混合模型关联映射方法。Nat Genet, 2006; 38:203-8。
- 90.
SPAGeDi:一种在个体或群体水平上分析空间遗传结构的通用计算机程序。分子生物学杂志,2002;2(4):618-20。
- 91.
Storey JD, Tibshirani R.全基因组研究的统计学意义。中国科学(d辑:自然科学版)2003;30(4):557 - 557。
- 92.
埃克特AJ,鲍尔AD,韦格琴JL,潘德B,杰姆斯塔德KD,克鲁托夫斯基KV,尼尔DB。海岸道格拉斯杉木(黄豆属门齐西属变种I.抗寒性相关性状。遗传学。2009;182:1289 - 302。
- 93.
Wegrzyn JL, Eckert AJ, Choi M, Lee JM, Stanton BJ, Sykes R, Neale DB。黑杨木木质素和纤维素生物合成控制性状的关联遗传(杨树trichocarpa,水杨科)次生木质部。植物生态学报,2010;29(2):359 - 359。
确认
基金资助:北京市教委科研项目(KM201810020)、国家自然科学基金项目(31800602)、北京市高校创新团队建设与教师职业发展项目(IDHT20180509)、北京市教委项目(cff - pxm2018_014207_000024)。我们也感谢北京市教委对生态修复工程跨学科创新项目的资助。
资金
基金资助:北京市教委科研项目(KM201810020)、国家自然科学基金项目(31800602)、北京市高校创新团队建设与教师职业发展项目(IDHT20180509)、北京市教委项目(cff - pxm2018_014207_000024)。我们也感谢北京市教委对生态修复工程跨学科创新项目的资助。资助者在研究的设计、收集、分析和解释数据以及撰写手稿中没有任何作用。
作者信息
从属关系
贡献
JW, PL和YY构思和设计了该研究。YY和RH进行了研究,JZ和ZH分析了数据,YY和JW撰写了这份手稿。所有作者阅读并批准了该手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1:图S1。
基于30个EST-SSR标记的192个个体的主成分分析。
附加文件2:图S2。
Q聚类分析f192美国oblata基于9个表型性状。
附加文件3:表S1。
932对SSR标记信息发育于美国oblata.
附加文件4:表S2。
表型性状的描述性统计扁桃树.
附加文件5:表S3。
关联群体中9个性状的表型相关性估计。
补充文件6:表S4。
17个多态EST-SSRs的推定功能及其与蛋白质编码基因的同源性。
附加文件7:表S5。
本研究关联群体的9个调查性状及测量标准。
权利与权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
杨勇,何锐,郑俊杰,杨勇。et al。EST-SSR标记的开发及其与花性状的关联定位紫丁香oblata.植物生物学20.436(2020)。https://doi.org/10.1186/s12870-020-02652-5
收到了:
接受:
发表:
关键字
- 紫丁香oblata
- EST-SSR
- 遗传多样性
- 人口结构
- 关联映射