摘要
背景
植物已经适应了在不利条件下生存,或利用有利条件来应对环境。在植物适应的过程中,植物激素被进化为有效利用有限的资源。植物激素包括生长素、茉莉酸、水杨酸和乙烯,通过对激素受体的突变和处理/不处理等实验设计进行表型观察,揭示了它们在植物适应环境中的作用。
随着下一代测序(Next Generation Sequencing, NGS)技术的发展,对样本植物的总基因表达进行评分,并估计植物激素对基因表达的影响程度成为可能。这使我们能够通过植物激素推断信号通路,这极大地刺激了使用突变体进行功能基因组学的研究。由于NGS技术和分析技术的不断发展,许多植物激素相关研究产生并积累了基于NGS的数据,特别是RNAseq数据已存储在以NCBI、EBI和DDBJ为代表的序列读取存档中。
描述
本研究采用mock、SA和MeJA处理,收集野生型基因型拟南芥(Col0)的激素处理RNAseq数据。通过机器学习方法确定了受激素影响的基因。对受影响基因的表达程度进行量化,使用d3 (data-driven-document)将其可视化为箱线图,并使用Django构建数据库。
结论
使用这个数据库,我们创建了一个web应用程序(http://pgl.gnu.ac.kr/hormoneDB/),列出激素相关或激素影响基因,并将所选基因的基因表达的箱线图可视化。这个网络应用程序最终帮助功能基因组学研究人员谁想要收集激素的基因反应的情况。
背景
植物是无固着生物,它们能适应大量的外界刺激,以使自己进入有利的环境,或在周围环境的不利条件下生存。为此,植物在叶、茎或根的细胞中产生少量的内源性调节因子,如植物激素,并将其转运到其他部位,利用它们来控制植物的代谢[1].因此,即使是少量的植物激素对植物代谢也非常重要,许多科学家通过遗传方法在模型植物中研究了植物激素的功能、合成、运输和信号通路,答:芥,使用各种测量方法。因此,植物激素的某些作用已得到很好的研究。例如,生长素、赤霉素和细胞分裂素主要参与植物生长,乙烯参与果实成熟,脱落酸参与种子休眠[2],茉莉酸(JA)诱导害虫抗性[3.],水杨酸(SA)诱导病原菌抗性和植物系统抗性[4]和油菜素类固醇在维管束分化中的作用[5]而独脚金内酯则主导土壤微生物的反应[6].
因此,遗传方法答:芥利用突变体可以通过基因丢失/获得后发生的表型变化来揭示基因的功能,估计植物激素及其信号通路的作用。除了针对有限数量基因的遗传方法外,微阵列还用于分析大基因集的转录表达,以了解更复杂的基因表达途径。此外,随着新一代测序(NGS)技术和分析方法的不断发展,RNA-seq真正革命性地检测了全基因和小rna等其他调控元件的转录表达[7].基于这些技术进步,对细胞分裂素各生长阶段及激素浓度的基因表达分析[8],以及生长素和脱落酸[9]对种子萌发、叶片形成、莲座生长、开花等表型进行了研究。由于组织在不同条件下的转录谱是动态的,许多研究小组不断生成RNAseq数据,以确定植物激素如何影响植物以及哪些基因与它们相关。
与此同时,研究人员为了其他分析目的或使用新开发的方法再生已发表的结果,不断地重新访问或请求已发表的原始数据。研究人员将他们的研究材料存放起来变得非常普遍;NGS原始数据、分析数据、实验信息元数据上报公共数据库;NCBI、EBI和DDBJ [10,11].目前,大量的NGS数据是在未经特别许可的情况下通过公共数据库公开的。随着上传的NGS数据激增,研究人员试图利用原有的生物学知识和沉积的NGS数据推导出新的含义;但是,含有样本详细信息的元数据往往不一致或缺失,因此,在数据生成方法上,对元数据进行修正和列出可比数据变得十分费力。此外,如果没有训练有素的生物信息学家,很难处理大量沉积的NGS数据。
因此,我们从NCBI序列读取档案(sequence read archive, SRA)中收集了植物激素治疗研究的RNAseq数据,并手工检查了元数据信息。经过充分检查的数据被处理成规范化单元中的表达式值,例如m值规范化的修剪平均值(TMM),在分组箱线图和web应用程序的描述面板中可视化。我们将SA、JA和它们的模拟处理作为数据库建设的目标,因为与其他激素相比,它们目前保存良好,可以形成大型数据集。基因表达水平使用d3可视化平台以分组箱线图显示[12].此外,我们应用机器学习算法随机森林,将样本分为JA, SA和mock处理。由此产生的模型清楚地指出了与治疗密切相关的分类的重要特征(基因),这一结果也列在了我们的web应用程序中。
我们的应用将是一个有用的应用,谁感兴趣的基因激素反应答:芥基因组,特别是SA和JA。此外,所选择的上调和下调明显的基因集将是参与SA和JA信号通路的强候选基因。此外,在我们的数据库中,其他激素治疗的RNAseq数据将每年更新。
结构与内容
数据库构建工作流
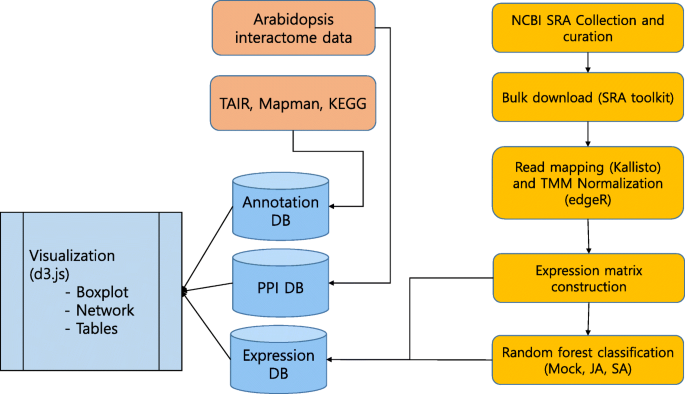
为了构建SA/ ja诱导的基因表达数据库,我们构建了一个简单的数据准备、数据库构建和web应用程序设计方案(图2)。1).数据准备步骤包括检查ncbi保存的RNAseq研究,以确定要托管的目标数据集。对于DB构建步骤,我们实现了Mapman、KEGG、Protein-protein interaction的良好准备数据和公共数据的sqlite3导入答:芥.最后,我们设计了web应用程序的用户界面,以方便知识挖掘。
从NCBI SRA采集样品
我们尝试收集NCBI SRA中RNAseq数据的元数据信息。元数据应包含对样本的描述,如组织、处理、生态型、诱导突变、测序平台等。然而,并不是每一项研究都包含足够的信息,可以在样本之间进行比较。因此,我们手动检查元数据并过滤掉不具有基本信息的SRA条目。我们定义了用于填充以下列的基本信息;研究ID,运行ID,检测类型,基因型,治疗,发育阶段,组织和布局(补充表1).共收集了1000份与激素相关的RNAseq研究(补充图1A)以SA和JA处理为主。我们将“基因型”限定为Col0,“处理”限定为对照组(245)、SA_1.0 mM(224)、SA_2mM(1)、MeJA_0.1 mM(224)、JA_1mM(1),处理后采样时间为15 min至72 h(补充图)1B).我们排除了多处理实验。作为对照,我们收集了经过水或DMSO处理的实验。我们还将研究范围缩小到叶片组织。发育阶段仅限于标记为“5 w-old plant”,“3w-old”,“营养生长”的样品。经过这次审查,我们可以列出695个具有SRR ID的实验,其中包括3个SRA研究;SRP031882、SRP112501和SRP125543 [13,14].
表达式矩阵的构造,分析,存储
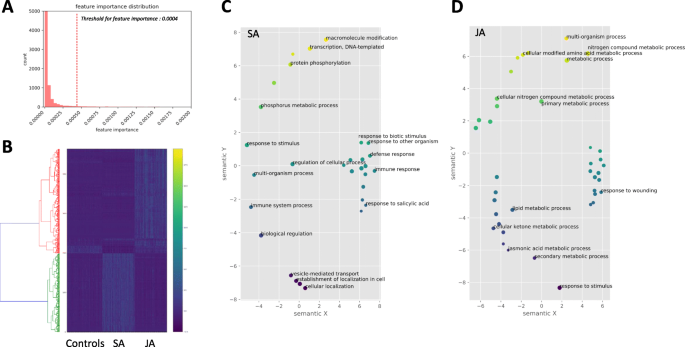
收集的SRA研究资料使用SRA-toolkit下载[15根据收集到的Run ID。在将SRA文件转换为基于文本的NGS读取格式Fastq之后,我们将读取映射到的参考编码序列答:芥(Araport 11) [16],利用EdgeR和Kallisto软件计算转录本的表达值(m值归一化的修剪平均值,TMM) [17,18].在处理的基础上,采用随机森林(RF)分类方案对样本进行分类;Control, JA, SA使用scikit-learn软件包[19].射频训练采用100次迭代,检索每个转录本的特征重要性分布。从RF训练中,每个基因将被分配100个特征重要性,以解释基因对对照、SA和JA组分类的重要性。我们平均了100个特征的重要性,并绘制了它们的直方图(图2)。2a).我们选取特征重要性值的阈值为0.0004。阈值被确定为具有来自分布的前1%的重要基因。从RF模型训练中共提取了463个候选基因。根据不同的处理,过滤后的转录本表现出不同的表达模式(图。2b).使用REVIGO的GO注释[20.]表示SA和JA特定条款;“对水杨酸的反应”和“茉莉酸的代谢过程”。SA特异性基因集还包含“免疫系统反应”和“叶片衰老”。JA特异性基因集包括“损伤反应”和“激素代谢过程”(图2)。2c和d). RF训练得到的基因总表达值和候选基因列表存储在sqlite3数据库中。
web应用程序构建和可视化
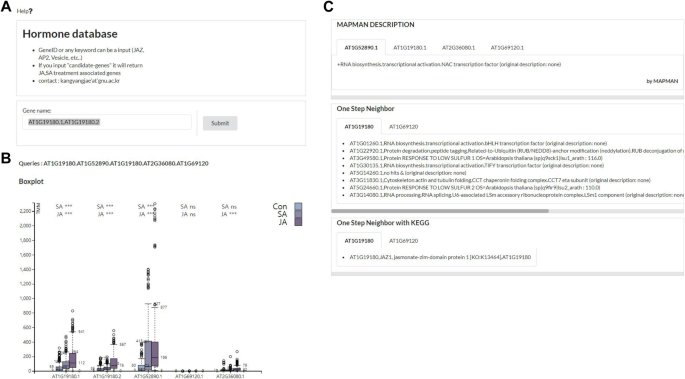
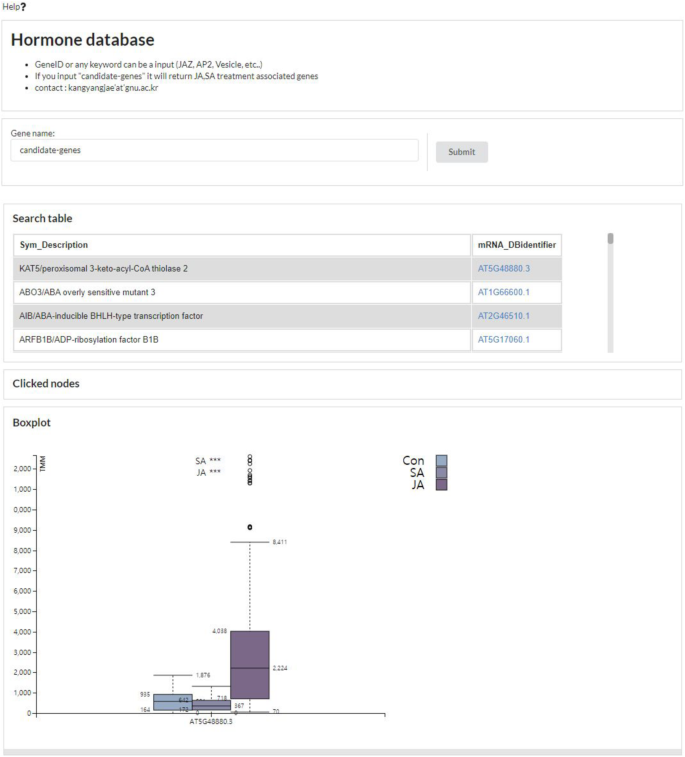
基于Django web框架(https://www.djangoproject.com)和d3.js (https://d3js.org/) [12],我们可以在网络应用程序上可视化基因表达和候选基因的结果。此外,网页的设计是使用语义UI (https://semantic-ui.com/).对于查询表单,用户可以输入成绩单ID(例如。AT1G19180.1),并输入基因ID(例如;AT1G19180)此外,用户可以输入任何搜索关键词,如“JAZ”、“Vesicle”、“Transcription”等。3.a).我们尝试显示查询相关信息;如分组箱线图中的转录本表达(图;3.B)、基因描述[16], KEGG通路[21], Mapman本体[22],拟南芥互作组[23(图。3.c).此外,在箱线图中,各组箱线图的顶部显示对照与处理之间的每个t检验显著性。我们使用Mapman本体直观地提供转录本的基因分类信息。此外,我们添加了PPIN和KEGG通路的邻居信息,以呈现查询基因的丰富信息。为了列出RF训练中候选基因提取的结果,我们允许搜索关键词“candidate-genes”。如果用户输入“candidate-genes”,它会在搜索表面板中列出明确表达的基因,点击可以直接看到表达模式和许多注释(图2)。4).
实用和讨论
自NGS技术发展以来,许多基于该技术的基因组研究在模型植物中进行,答:芥.在NCBI SRA中,总共累积了74tb的NGS数据答:芥.我们检查和收集了已研究的植物激素的NGS数据,并通过基于RF训练的特征选择方案探索了处理依赖的候选基因。为了使它们成为web应用程序,我们使用了一个web框架Django,它允许我们管理数据库,并托管我们的网页,提供有用的功能;它们的基因表达和网络的可视化,基因的分类和描述。由于NCBI SRA中用于其他激素治疗的RNAseq存款也在增加,我们正计划每年更新和更新我们的web应用程序,以扩大可用性。我们希望许多研究人员在没有生物信息学专业知识的情况下,能够找到他们的目标基因在JA和SA处理方面的表达模式,并从我们的候选基因列表中检索到新的有趣的基因。
结论
在这项工作中,我们开发了一个可搜索的数据库,重点是RNAseq数据答:芥受到SA和JA路径的影响,导致通过大型数据集的机器学习改进web应用程序。该数据库将每年更新一次,以反映NCBI中RNAseq的累积数据。总的来说,这个新的网络平台将帮助植物研究人员更容易地找到转录谱中的激素反应基因。
数据和材料的可用性
应用程序的网址是http://pgl.gnu.ac.kr/hormoneDB/.本研究分析的数据集列在补充表中1.
缩写
- 走:
-
基因本体论
- 是:
-
茉莉酸
- KEGG:
-
京都基因和基因组百科全书
- NCBI:
-
国家生物技术信息中心
- 门店:
-
下一代测序
- 射频:
-
随机森林
- 山:
-
水杨酸
- SRA:
-
序列读取存档
- TPM:
-
每百万抄本数
参考文献
- 1.
Santner A, Calderon-Villalobos LIA, Estelle M.植物激素是植物生长的多功能化学调节剂。生物化学学报。2009;5:31 1 - 7。
- 2.
Davies PJ,编辑。植物激素:生理学、生物化学和分子生物学。多德雷赫特:施普林格;1995.ISBN 9780792329855。
- 3.
浏览J. Jasmonate:一种在植物中具有多种作用的氧脂素信号。维塔姆。2005;72:431-56。
- 4.
Loake G, Grant M.植物防御中的水杨酸——参与者和主角。植物学报。2007;10:466-72。
- 5.
李志刚,李志刚,李志刚,李志刚。植物甾体激素信号转导的分子机制。细胞发育生物学,2005;21:177-201。
- 6.
Shinohara N, Taylor C, Leyser O.直脚金内酯通过触发生长素外排蛋白PIN1从质膜快速消耗来促进或抑制芽分枝。《公共科学图书馆·生物学》2013;11:e1001474。
- 7.
Kukurba KR, Montgomery SB. RNA测序与分析。《寒泉哈布议定书》2015;2015:951 - 69。
- 8.
Bhargava, Clabaugh I, To JP, Maxwell BB, Chiang Y-H, Schaller GE, Loraine A, Kieber JJ。利用微阵列元分析和RNA-Seq鉴定拟南芥细胞分裂素反应基因。中国生物医学工程学报,2013;
- 9.
陈娟,毛琳,陆伟,应涛,罗忠。草莓采后果实对外源生长素和脱落酸响应的转录组分析。足底。2016;243:183 - 97。
- 10.
埃德加R,多姆拉切夫M,鞭策AE。基因表达综合:NCBI基因表达和杂交阵列数据存储库。核酸Res. 2002; 30:207-10。
- 11.
Petryszak R, Keays M, Tang YA, Fonseca NA, Barrera E, Burdett T, Füllgrabe A, Fuentes AM-P, Jupp S, Koskinen S,等。基因表达图谱更新——人类、动物和植物基因和蛋白质表达的综合数据库。核酸研究,2016;44:D746-52。
- 12.
李国强,李国强,李国强3.:数据驱动文档。电子工程学报。2011;17:2301-9。
- 13.
Hickman R, Van Verk MC, Van Dijken AJH, Mendes MP, Vroegop-Vos IA, cararls L, Steenbergen M, Van der Nagel I, Wesselink GJ, Jironkin A,等。茉莉酸基因调控网络的结构与动态研究。《植物科学》2017;29:2086-105。
- 14.
曹明杰,张彦林,刘旭,黄辉,周雪霞,王文林,曾安,赵成忠,司涛,杜军,等。结合化学和遗传方法来提高植物的抗旱性。Nat Commun. 2017;8:1183。
- 15.
列诺宁R, Sugawara H, Shumway M.国际核苷酸序列数据库合作序列读取档案。核酸研究,2011;39:D19-21。
- 16.
程春阳,陈ap, Thibaud-Nissen F, Schobel S, Town CD.拟南芥参考基因组的完整重标注。植物J. 2017; 89:789-804。
- 17.
Bray NL, Pimentel H, Melsted P, Pachter L.近最优概率rna序列定量。生物工程学报。2016;34:525-7。
- 18.
Robinson MD, McCarthy DJ, Smyth GK。edgeR:用于数字基因表达数据差异表达分析的生物导体包。生物信息学。2010;26:139-40。
- 19.
Pedregosa F, Varoquaux G, Gramfort A, Michel V, Thirion B, Grisel O, Blondel M, pretenhofer P, Weiss R, Dubourg V,等。Scikit-learn: python中的机器学习。J Mach Learn Res. 2011; 12:2825-30。
- 20.
Supek F, bosnjak M, Škunca N, Šmuc T. REVIGO总结并可视化了基因本体术语的长列表。科学通报。2011;6:e21800。
- 21.
KEGG:京都基因和基因组百科全书。核酸Res. 2000; 28:27-30。
- 22.
Thimm O, Bläsing O, Gibon Y, Nagel A, Meyer S, Krüger P, Selbig J, Müller LA, Rhee SY, Stitt M. MAPMAN:一个用户驱动的工具,将基因组学数据集显示在代谢途径和其他生物过程的图表上。植物学报,2004;37:914-39。
- 23.
拟南芥互作组制图联盟。拟南芥相互作用组图中网络进化的证据。科学。2011;333:601-7。
确认
不适用
资金
整个研究工作包括研究设计、数据收集和分析都是在“下一代生物绿色21计划”(项目编号:;pj013333901)“韩国农村发展局。
作者信息
从属关系
贡献
DUW和YJK确定了实验设计,进行了分析,构建了web应用程序并撰写了手稿。HP、YL、HHJ和JHP收集了可用的SRA转录组数据,并对所选SRA数据的标签进行了策划。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
我们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
吴D.U,全H.H,朴H。et al。数据库:对水杨酸(SA)和茉莉酸甲酯(MeJA)处理的RNAseq累积数据的可视化web应用程序拟南芥.BMC植物生物学20.453(2020)。https://doi.org/10.1186/s12870-020-02659-y
收到了:
接受:
发表:
关键字
- 水杨酸
- 茉莉酸
- 的一点
- 拟南芥
- RNAseq
- 数据库