抽象的
背景
生态环境形状植物建筑和改变生长季节,为小麦遗传改进提供了基础。因此,了解特定生态环境中籽粒产量和产量相关性状的遗传基础是重要的。
结果
在中国河南省河南省高产区的96个精英小麦品种结构化面板使用Illumina Iselect 90 K SNP测定进行了基因分型。从山前和平原区域的生态环境中获得的选择压力为人口分歧提供了最初的推动力。这确定了两个亚群中的显性性状(尖峰数和尖峰百分比在贫民窟2:1;千核重量,谷物填充率(GFR),到期日(MD)和生育期(FP)在亚贫民中占优势2:2),这也是与捐助者父母的遗传相一致。基因组宽协会研究确定了12个屈服相关的性状的107个重要的SNP,10个区域是多种性状的磷酸盐。特别是,GY与MD / FP,GFR和HD共同定位qtl-ple5a.那QTL-ple7A.1和QTL-PLE7B.1.地区。进一步的选择性扫描分析揭示了选择的区域是这些特征的QTL。特别是,谷物产量(GY)与MD / FP呈正相关,它们共同位于VRN-1A轨迹。此外,检测到选择性扫描信号VRN-1B位于对MD / FP的意义的基因座。
结论
结果表明,生态选择驱动的等位基因频率广泛差异,在产量改进期间具有塑造植物建筑和生长季节。本研究中检测到的产量和产量组分的QTL可能选择性地应用于分子育种中。
背景
小麦,玉米和大米是世界上最重要的粮食作物。随着全球人口的持续增加,气候变化和降低耕地的可用性,每年的收益率约为2%,累计增加50%,以满足预测的全球需求。
河南省西北至中东部地区位于黄淮河流域以南,是中国最大的小麦产区和高产地区。该区小麦产量占全国总产量的四分之一,高产是该区小麦生产的核心目标。小麦主产区位于北亚热带,四季分明,是中国第二梯田和第三梯田的过渡带。因此,这些复杂的生态环境使得不同生长季节(半冬和弱春)和植物构型类型的小麦品种能够在该地区生长。
21世纪初,河南省小麦产量增长迅速,粮食增产较前一时期取得显著进展(Zhou et al. 2007)。推荐一批高产稳产、适应性强的品种在河南栽培,并被认可为创始亲本。如玉麦2号、周8425B、偃市4号在不同区域被多次不同程度地用作供体亲本。玉麦2号是弱春弱冬品种,分蘖力强,抗寒性强,但粒重较低(Zheng et al. 2011;Gao et al. 2017)。烟石4号是由阜诺和马拉衍生而来的春小麦品种,穗大早熟,分蘖能力较弱。1B/1R易位系周8425B是一个高产、强抗病的小麦品种,具有大穗和矮秆习性(Gao et al. 2015;Zhao et al. 2008;Li et al. 2006;Wang et al. 2017)。 Numerous progeny bred from these cultivars inherited desirable characters and were approved for commercial release. Yumai 25, Yumai 41, and Yumai 49 were selected from the cross between 394A and Yumai 2, and inherited the early maturity of Yumai 2. Zhoumai 9 (Yumai 21), which was bred by double-crossing Yumai 2 and Yanshi 4, exhibits high grain weight and semi-dwarfism but shows later maturity, and thus is suitable for planting in central Henan with early sowing. Further pyramiding of Zhou 8425B resulted in a series of Zhoumai-family wheat cultivars (e.g., Zhoumai 13, Zhoumai 16, and Zhoumai 22). In addition, a number of cultivars imported from other regions (e.g., Shaanxi) have been used as parents to shorten the fertility period, introduce disease resistance, improve grain end-use quality, and have contributed to an increase in genetic diversity.
基于分子标记和表型分析的群体遗传学被广泛用于检测在物种进化中重要的染色体区域,识别与对人类健康有益的性状相关的遗传变异,动物的生长特征,以及对重要性状有贡献的基因组区域[1那2那3.那4.那5.那6.].一种方法是通过将基因型与表型联系,对定量性状基因座(QTL)发现进行的基因组多样性研究,以确定所需性状的基础遗传基础。特别是,它在解剖型QTL的解剖中实现了实质性进展,以了解复杂性状的潜在遗传基础[7.那8.那9.那10.].一种替代方法是选择性扫描分析,其涉及等位基因频率之间的差异。选择性扫描是核苷酸序列之间变异变异显着降低的结果,这些核苷酸序列在驯化期间适用于适应性或适应期间的健身[11.那12.].该方法已广泛应用于植物群体遗传学,以识别与果实质量改善相关的信号[12.,不同生态型的开花时间差异[13.[越冬习惯[14.].
了解河南不同地区小麦品种间表型变异的遗传基础和环境适应的遗传足迹,并将这些信息整合到未来的小麦品种开发中,对小麦产量的持续提高具有重要意义。为了实现这一目标,我们将河南省小麦主产区现代小麦品种中遗传多样性最高的优良育种品种组成一个群体,进行表型评价。通过群体遗传分析评估群体结构,确定影响植物结构或生长季节以及环境适应的基因组区域。
结果
人口结构和种群之间的表型
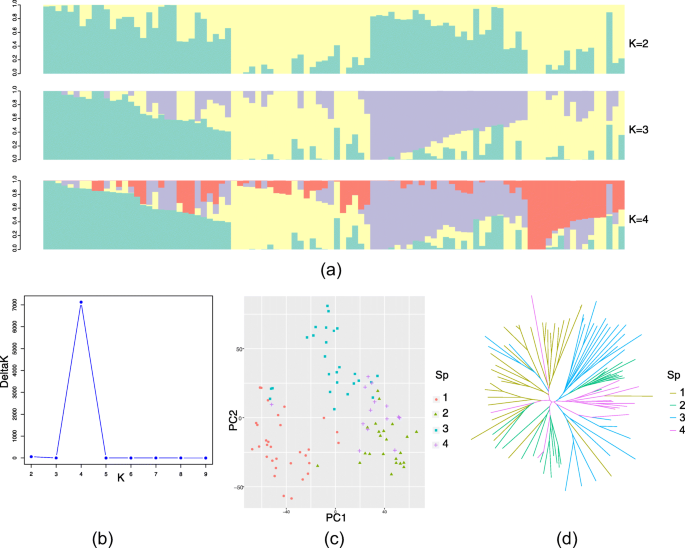
共有81,088个SNP标记用于群体结构评估。对所有品种进行了鉴定K.= 2K.= 4(无花果。1表S1)。在K. = 2, wheat cultivars in subpopulation 2:1 were derived from the donor parents Yumai 2, Yanshi 4, and Shaanxi, and were mostly selected in the northwest to central region of Henan, whereas subpopulation 2:2 exclusively comprised cultivars with the pedigree of Yumai 2, Zhou 8425B, and Yanshi 4 harboring the rye 1RS chromosome arm and were selected in the central region of the southeastern plains (Fig.2).在K.= 3时,亚群2:1的品种被分解为2个亚群体:亚群体3:1 (Sp1)和亚群体3:3 (Sp3)。3:1亚群的品种主要来自豫麦2号、偃师4号和内乡82C6,由正常小麦1BS染色体臂组成;3:3亚群的品种由豫麦2号、偃师4号和内乡82C6组成。和其他地区(如陕西)的品种,存在1B/1R染色体易位。在K. = 4, cultivars in subgroup 2 (K. = 2) were divided into an additional two subpopulations: subpopulation 4:2 (Sp2) and subpopulation 4:4 (Sp4). The majority of cultivars in Sp4 were second-generation derivatives (Zhoumai 13 and Zhoumai 16) of Yanshi 4 bred by pyramiding the donor parents with Zhou 8425B, whereas cultivars in Sp2 were derived from donor parents in Henan other than Zhou 8425B and Yanshi 4. In addition, cultivars grouped in Sp1 and Sp3 were suitable for growth in northwest-central Henan, whereas cultivars in Sp2 and Sp4 were suitable for cultivation in central-east Henan. The plot of the mean likelihood L(K.)和每次方差K.值表示K. = 4 was the most likely number of subgroups among the 96 cultivars (Fig.1b)。
对四个亚种群的分配叠加在主成分分析的结果上,与STRUCTURE的结果相似。在主成分分析中,PC1从其他品种中分离出Sp1 (1BS染色体臂的品种),没有对其他三个亚群体进行区分(图1)。1C)。PC2分离的SP2和SP3和SP4由PC3区分(图S1一种)。
为了研究品种中的系统发育关系,基于96种品种的基因分型数据构建系统发育树。在SP1和SP3中分组的品种显示了与结构和PCA的结果的一致关系,少数例外情况下(图。1d). Sp2分类的品种分为两类:一类与其他三个亚居群分离,另一类与Sp3相连,并伴有Sp4聚类。
可变部位数,核苷酸多样性(π)和平均核苷酸差异在SP3(35,722,0.13862和11,230.98265)中最高,其次是SP1(31,897,0.12774和10,348.97462)和SP2(27,206,0.11351,和9196.18937),在SP4(27,684,0.10994和8906.97984)中最低(表S3)。
平均LD估计范围从R.2 = 0.82 (0–0.5 Mb) toR.2 = 0.12 (436–436.5 Mb) (Fig. S1b). LD评分从0迅速衰减到10 Mb,并呈现近似的拐点R.2 > 0.6. The LD decay showed a moderate decrease within 10–60 Mb withR.2范围从0.6到0.4。拟合的回归在大约30 MB的阈值与平均LD衰减相交R.2 = 0.5.
表型性状评价与相关
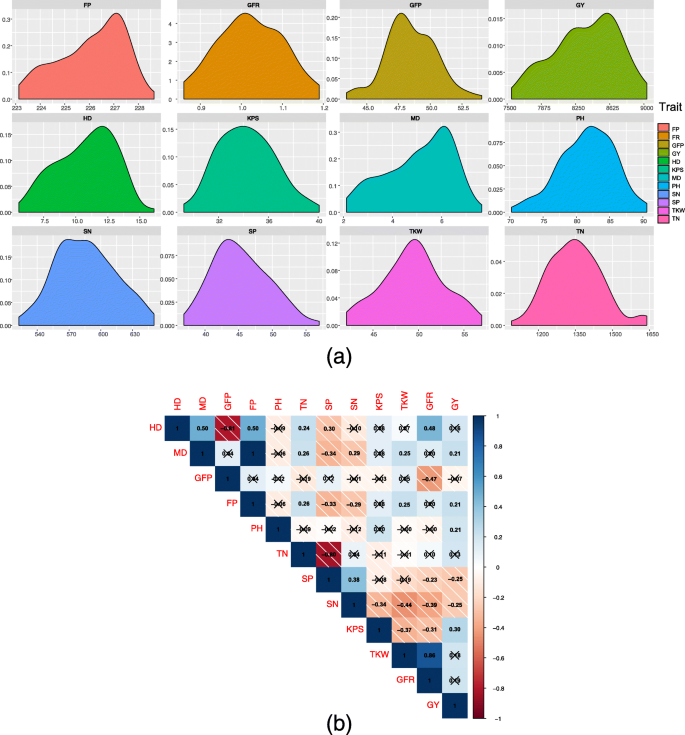
两种环境中的表型是显着相关的(P. < 0.01) and the kernel density distribution of phenotype BLUP values showed that all traits exhibited a continuous distribution (Fig.3.一种)。在测试的12个特征上的广义遗传性范围为0.53(TN)至0.98(MD和FP)。对于七个特征(HD,MD,GFP,FP,SN,TKW和GFR),H2大于0.9,而H2对于GY,KPS,TN,pH和SP分别为0.684,0.656,0.531,0.531和0.473(表1).
进行Pearson相关分析以检查12个特征之间的成对相关性(图。3.b)。其中,GY与MD,FP,pH和KPS正相关(R.= 0.21、0.21、0.21、0.30),与SP、SN呈负相关(R.=−0.25和−0.25)。GY与TKW和GFR呈弱正相关(0.19,但不显著)。TKW与KPS之间存在较强的负相关(R.=−0.37)。SN与TKW呈负相关(R. = − 0.44) and KPS (R. = − 0.34).
对于与周期相关的特征,MD和FP与TKW呈正相关(R. = 0.25), negatively correlated with SN (R.=−0.29),且与KPS无显著相关。此外,两个性状均与HD呈正相关(R. = 0.50) but no significant correlation with GFP. On the other hand, HD showed a strong negative correlation with GFP (R. = − 0.81).
群体中的表型特质优势
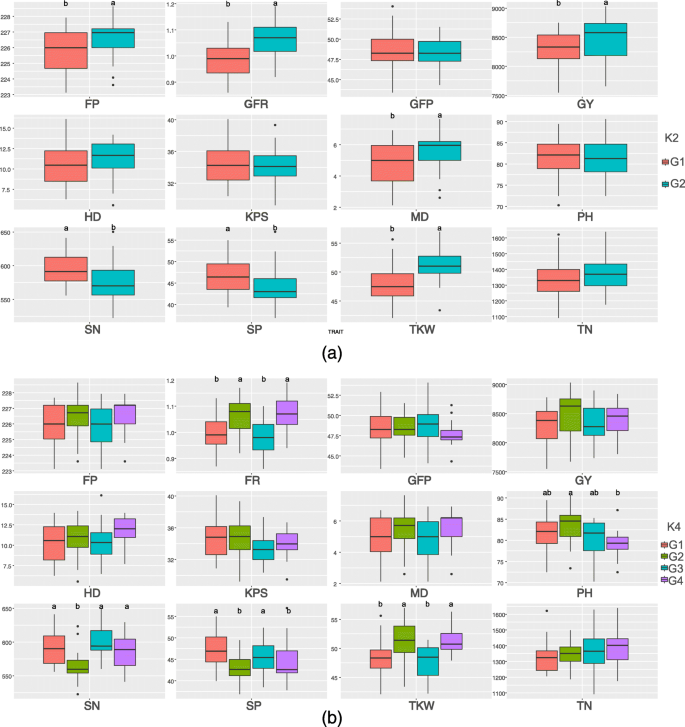
亚种群间的表型优势在K. = 2 andK. = 4 was assessed, and was more strongly observed atK.= 2(图。4.a)除下K.= 4(无花果。4.b)。
GFP、HD、KPS和TN的表型优势均不显著K. = 2 orK.= 4。2∶2亚群体TKW和GFR的表型优势显著高于2:1以下亚群体K. = 2, with the reverse trend observed for SP and SN. However, these phenotypic differences were not detected together underK.= 4。2种群中GY、MD和FP的表型值显著高于2亚种群,但在2亚种群中未检测到显著性K.= 4。此外,the predominant phenotype for PH was observed in subpopulation 2:2 underK. = 4, for which the plants grouped in Sp4 were shorter than those in Sp2.
基因组 - 12个农艺性状的协会研究
共107个显著snp (P. < 0.001) distributed across all 21 chromosomes except 3D, 4A, 4D, and 6D were detected for GY and related traits. With regard to the individual traits, 13, 11, 15, 10, 7, 3, 13, 6, 21, 7, 12, and 6 significant SNPs were detected for FP, GFR, GFP, GY, HD, KPS, MD, PH, SN, SP, TKW, and SN, respectively. These significant loci showed logarithm of the odds scores ranging from 3.00 to 5.74 and contributed 10.45–24.19% of the effects on the corresponding traits (Table S4.,图.. s2).
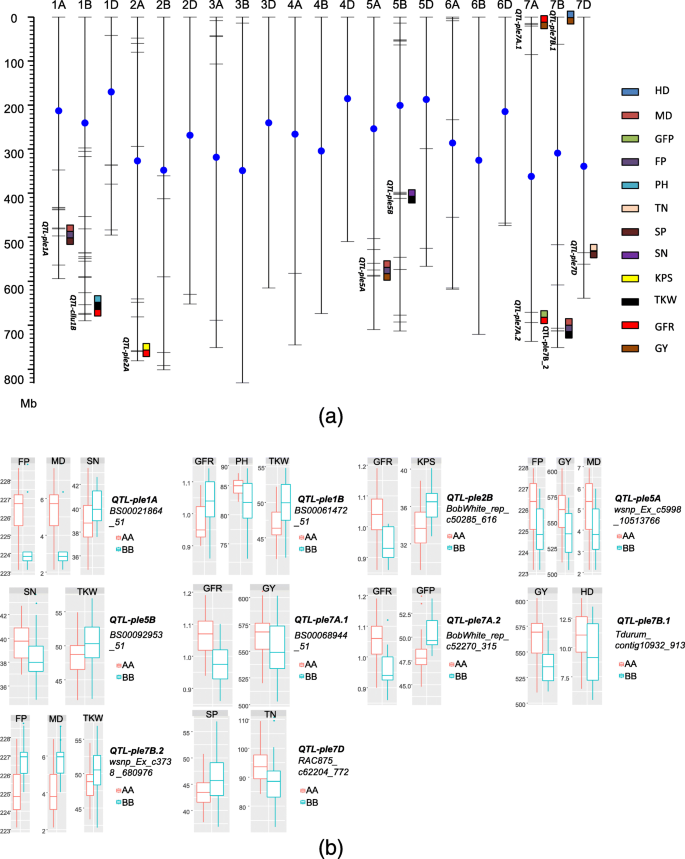
十QTL区域中显示出与考虑的平均LD衰减距离的各种性状的多效性的贡献。的QTL被分发在染色体1A,1B,2A,5A,5B,7A(2),7B(2),和图7D(图5.).
三个QTL区域对GY显示出一种或两个其他特征。QTL.QTL-ple7A.1,由标记表示BS00068944_51,控制GY和GFR,效果方向一致。HD和GY也观察到相同方向的效果,以及FP,MD和GY解释的QTL-PLE7B.1.和qtl-ple5a.,分别用SNP标记表示tdurum_contig10932_913和wsnp_Ex_c5998_10513766, 分别。
MD/FP与其他性状在另外两个QTL区域共定位:SN在QTL-ple1A(代表BS00021864_51),与TKW位于同一位置QTL-PLE7B.2(代表wsnp_ex_c3738_6809767)具有一致的效果方向。
GFR在具有不同效果方向的QTL区域内共同定位四个其他特征。分别控制特征KPS和GFPQTL-ple2B(bobwhite_rep_c50285_616) 和QTL-ple7A.2(bobwhite_rep_c52270_315)与对比效果方向的GFR。QTL.QTL-ple1B(BS00061472_51)与TKW共同定位,具有相同的效果方向,但在相反的效果方向上具有pH值。
等位基因的TKW值QTL-ple5B_BB高于那个QTL-ple5B_AA,而SP观察到相反的结果。此外,观察到互补效果方向QTL-PLE7D.控制SP和TN。
选择性扫描信号的全基因组扫描
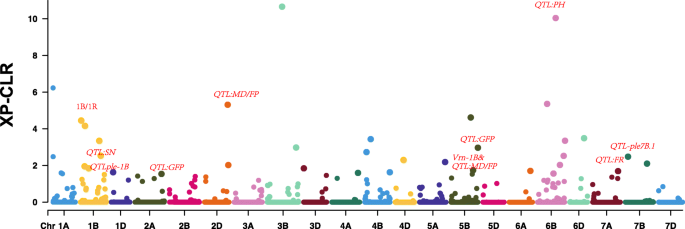
为了研究候选选择性扫描对整个基因组的性状发散的影响,我们通过比较了两个群体的选择签名K. = 2. The footprints of selection were detected in a total of 62 genomic regions across all 21 chromosomes except 5D and 7D, with a span of 0.83% of the wheat genome. The mean strength of selection was 0.0305 (Table S5., 无花果。6.).
十个选择性扫掠地区是Agranomic特征的QTL。两个选择信号位于623251480 BP和637,251,480 BP侧翼qtl-ple1b。染色体2a和5b上的两个信号从两个QTL用于GFP的QTL位于约〜10mb。类似地,在535251480bp的染色体1b上的选择性扫描信号从QTL的SN为〜18kb。染色体5B的信号在562087488 BP和580,087,488bp flanded qtls的MD和FP,其覆盖VRN-B1先前据报道的基因区域参与生长习惯的调节[15.].此外,在1BS上检测到五种选择信号,表明1B / 1R易位是人口劣质的重要签名。
讨论
群体之间的植物结构由生态塑造
以往对小麦的研究表明,亚种群结构取决于地理来源或驯化状态[16.那17.那18.].在本研究中,河南商业品种小组的人口结构主要是通过来自供体父母的特征的遗传来决定,这显着影响了与育种目标和环境适应有关的表型变化。1B / 1R易位和外部遗传资源是对人口结构影响的额外因素。
河南的温度通常从南部到南部南部减少,但不同地区的生态因子提供不同的选择压力,具有成形的植物建筑和生长季节。河南西北地区经历了更长的冬季,但温度较高,因为太极山脉从西伯利亚堵塞了冷空气的渗透,这是在该区域中选择的亚洛杉矶中群中的品种继承了yanshi 4的早期成熟的原因来自榆叶的强烈分蘖能力2.相比之下,河南中心东部地区是一个平坦的平原,易受中等纬度的冬季季风气候的影响。在亚贫民中分组的品种适用于东南平原的培养,被选为来自周8425B的理想谷物填充性状[19.].亚种群的进一步细分K.= 2K. = 4 only slightly influenced phenotypic differentiation, which indicated that introduction of cultivars from other regions probably contributed to other traits, such as end-use quality and cold resistance.
产量的遗传基础及其改良
提高产量是现代小麦品种繁殖的重要目标。产量包括三个主要特征:核心重量,每个尖峰核数和尖峰号。然而,这三种组分中的每一个都有助于产生对不同程度的改善[20.那21.那22.].在本研究中,产量的三个组成部分是高度协调的。籽粒产量与KPS呈显著正相关,与SN呈显著负相关,与TKW呈弱正相关,表明产量的提高依赖于穗对营养生殖库容量的增加。
籽粒产率的测定是复杂的,并且生长周期的任何阶段的因素可能影响大陆小麦生产区域的最终产量[23.].在目前的研究中,GY与FP,MD和HD呈正相关,HD暗示了长期生殖增长期是源供应的重要因素[24.那25.].相比之下,缩短FP有利于提高复种指数(玉米,小麦种植系统在河南普遍实行),这表明产量的提高是通过加快生长速度是一个挑战。然而,MD / FP和KPS,提示增加KPS用于提高产量比TKW到MD / FP较不敏感之间没有观察到相关性。
以前的研究表明,通过极大地将pH从100厘米到〜80cm的pH值下降,GY增强与降低的植物高度呈正相关。26.那27.那28.那29.那30.那31].目前研究中的pH和GY之间的正相关可能是因为低植物高度会导致生物质的丧失和疾病风险的增加,而植物高度不是小麦住宿的主要原因[32那33那34那35那36那37].
关联作图群体中多效qtl的鉴定是有效分析相关性状遗传基础的一种有前景的方法[7.那38那39那40那41那42].控制穗性状间权衡的qtl的鉴定已被广泛报道[10.那43].QTL.QTL-ple2B对本研究中的GFR和KPS显示了肺炎效应。最近克隆的基因,GNI.,在染色体2A上显示对KPS和TKW的抗脂肪效应[44].然而,由于其同源疗法的位置,QTL-ple2B不是相同的基因[44那45].鉴于GFR和TKW之间的高相关,QTL-ple2B是解释权衡TKW(GFR)和SN之间的遗传基础的新的轨迹。此外,QTL-PLE5B.互补调节千粒重和SN,这是两个负相关的特征。这个QTL区间似乎是内核相关性状的新等位基因。然而,5A染色体上最近报道的稳定的QTL千粒重6.9%的增幅相关[46].此QTL间隔可能位于共线区域中QTL-PLE5B.但需要进一步探索。额外的QTL,qtlple-7d.,对比方向控制TN和SP,这表明早期发育期间的耕地形成不能完全转化为有效的尖峰。此外,qtlple-7d.是一个小说QTL,因为没有QTL已经记录在一起来控制分蘖发展。
等位基因的qtl-ple5a.那QTL-PLE7B.2, 和QTL-ple1A这有助于更长的FP与较高的GY,更高的TKW和下部Sn相关,其为成熟时期对籽粒产量和产量组分的影响提供遗传基础。特别是,qtl-ple5a.和QTL-PLE7B.2位于类似的基因组位置VRN-1A和VRN-3A根据中国春天参考组装基因组,这与关于结合绘制群体不同面板的几个报告一致,表明这一点VRN1.和VRN3.与GY有关[38那47那48那49那50].此外,基因底层QTL-ple1A尚未被鉴定,这表明该位点代表一个新基因。
选择法
对植物的广泛研究已经在显著相关的位点周围发现了群体遗传特征,并对不同的亚群体进行了划分,如驯化与野生资源[12.那51]而且该工厂建筑是塑料响应生态变化[13.或亚种分化[52].
在本研究中鉴定了几种已知的功能基因侧翼的选择性扫描信号,包括先前报道的适应基因VRN-1B,它表现出优势效应VRN-1A对籽粒产量[49那53].在本研究中,通过选择性等位基因延长MD / FPVRN-1B对GY有非显着影响。相比之下,qtlple-5a.(VRN-1A这些结果表明,采用金字塔法可能是一种探索早熟与高产平衡的育种策略VRN-1B只要。
Several selective sweep regions that flanked QTLs for agronomic traits (e.g., TN, GFR, MD, and FP) were identified in the present study, which reflected that these alleles were contributed by utilization of characters of the donor parents, possibly in response to ecological environments. In particular, no selective sweep signals were detected around QTLs for KPS, which suggested that alleles contributing to increase in KPS may be further applicable in breeding.
结论
全基因组关联分析,鉴定与籽粒产量和产量构成要素相关联的若干个QTL,并提供了深入了解的产量相关性状的遗传基础。一些所标识的QTL的是在选择下,这意味着在等位基因频率广泛变化通过生态压力到形状植物结构和生长季节驱动。我们探讨人口结构,生态适应性,为的QTL农艺性状和选择区域之间的关系。该鉴定的QTLqtlple-7a.1.和qtlple-5a.分别通过加速灌浆和适度延迟成熟期来提高小麦产量。未受选择压力影响的qtl可用于性状改良育种中的标记辅助选择。
方法
植物材料和田间试验
一个96个小麦品种面板(表S1)为研究对象。[54那55].这些品种在西北地区培养到河南省的中心东部地区,并挽救了河南农业科学院小麦研究所。使用随机嵌段实验设计,在两个位置(新乡和安阳)的种子生长,种子机械播种。对于所有实验,每种品种建立每个绘制150,000个种子的两种重复图,每种绘图为150,000种。
基因分型和物理映射
使用十六烷基三甲基溴化铵法从幼叶组织中萃取每种品种的基因组DNA,并使用IlluminaIselect®90KSNP测定的基因分型,该分析在加利福尼亚州戴维斯基因组中心(戴维斯,CA,USA)。.源自小麦中国春季参考基因组序列(IWGSC V1.0;https://wheat- urgi.versailles.inra.fr/)使用BLAST + 2.2.25封装(国家生物技术信息中心,Bethesda,MD,USA)构建,用于搜索单核苷酸多态性(SNP)标记的所有序列的顶部命中以确定其物理位置(表S2).
每个SNP的基因型簇是使用Genome Studio version 2011.1软件(Illumina, https: //)确定的www.illum.ina.com).根据81,587个探针对所有样本的基因型进行了分类,根据双等位snp的基因型,分为两个纯合子(AA&BB)和一个杂合子(AB)。去除缺失率> 5%的SNPs,保留80540个SNPs。
人口分析
使用在不同数量的群集下的结构版本2.3软件中实现的基于模型的方法估计了人口结构(K.)范围从K. = 1 toK.= 10。为了精确估计参数,我们使用了一个包含10,000次老化迭代和10,000次马尔可夫链蒙特卡罗迭代的混合模型,每个模型有五个独立的重复K.值(56].结构收割机在线程序(http://taylor0.biology.ucla.edu/structureHarvester/)用于整理STRUCTURE输出文件,以使用evans方法检测最可能的种群细分水平[57].采用GAPIT采用默认参数进行主成分分析(PCA),计算前三个主成分(PCs) [58].使用Mega V7.0.14使用Kimura 2参数模型构建邻接树[59].使用1000个复制进行引导测试,以评估树拓扑的统计支持。使用“GGTree”包来可视化最终树[60]使用R脚本。
使用相关系数的平方进行连接不平衡(LD)分析(R.2)每对染色体上的每对标记,使用Tassel版本5.2.55 [61那62].计算在当前站点周围使用50个标记的滑动窗口和截止值P. < 0.001. The meanR.2在一个0.5 Mb的窗口中绘制了物理距离,并对数据进行了指数曲线拟合。对应的物理距离R.2衰减到最大值的50%被确定为平均衰减距离。
使用DNASP版本6.12.03计算可变位点,核苷酸多样性(π),核苷酸多样性(π),平均核苷酸差异的平均核苷酸差异和单倍型数量的数量[63].
表型评估和分析
记录了抽穗期(HD)、成熟期(MD)、灌浆期(GFP)、肥育期(FP)、分蘖数(TN)、穗率(SP)、穗数(SN)、株高(PH)、每穗粒数(KPS)、千粒重(TKW)、灌浆速率(GFR)和籽粒产量(GY)等12个农艺性状。
播种日期在两个地方都相同(10月7日)。HD和MD日期分别确定为整个图的峰值标题和成熟度。GFP计算为MD减去高清;由于播种日期,FP等于MD。通过比较真实和最早的值,MD和HD被记录为相对值。对于TN,KPS,pH,Sn和TKW的评价,从幼苗阶段监测1米长度的样品排直至收获。TN被计算为联系前的分蘖总数;SN被测量为尖峰的总数。TN和SP的值被转换为每个图的总数。测量pH作为尖峰顶部与根部的距离; KPS and TKW were calculated as follows:
GFR被确定为TKW除以GFP;GY计算为每种绘图的总粒重。
在两个位置的每个性状最佳线性无偏预测(BLUP)中的溶液使用“lme4”包对于R推断和使用线性混合模型[嵌合64]:
在哪里y表型,G是基因分型效果,L.是位置效果,R.是复制的数量,*表示两个因素之间的交互,%表示位置因子中的复制嵌套E.是残余的。
在广义遗传力(H2)用下面的公式估算:
在哪里\({\ sigma} _g ^ 2 \)是基因型的变异部分,\({\西格玛} _ {绿\ AST L} ^ 2 \)是基因型和位置之间的相互作用的方差分量,\({\ sigma} _e ^ 2 \)是残余的方差,R.是每个位置的复制数量L.是每年的谐波平均值和每个测试品种的位置[65].
用这些特征的成对相关系数的计算进行了cor在5%显著性水平下,使用“皮尔逊”方法(P. < 0.05) and was visualized using the “corrplot” package in R [66].
为了单独处理四个亚组之间的表型优势,基于5%意义水平的Scheffe多比较方法使用单向分析差异(ANOVA)(P. < 0.05) using IBM SPSS Statistics version 22 (IBM Corporation, Armonk, NY, USA).
协会映射
使用具有Tassel版本5.0的混合线性模型进行关联映射[62].总共使用11,930个已知物理位置和缺失值缺失的值,并且所有杂合基因型被排除在标记物性关联之外。人口结构(问:矩阵),定义为基因型间的差异亲缘关系,亲缘矩阵(K.代表每种人口中所有成对比较的共同等位基因比例的矩阵被列入统计数据。使用MLM模型(混合线性模型)进行基因组宽关联映射,如Tassel中的描述[62].每个性状的结合值用于表型观察。阈值-log10(P.- 对于基因组 - 宽协会映射,每个标记的值≥3.0被认为是重要的。
考虑到表型测定的系统误差,筛选多效QTL的标准如下:1)将平均LD距离内的不同性状标记视为一个QTL;2)所有品种按每个双等位基因SNP分组,采用方差分析(ANOVA)检验表型显著性(P.< 0.05),目的是在接下来的单倍型分析中选择一个代表QTL的标记。或者,在平均LD中所有未通过方差分析的显著标记将被排除在多效QTL筛选之外。
对于随后的单倍型分析,分析了与代表性标记的每个纯合等位基因对应的表型参数的子集,以揭示对该性状的QTL效应(使用Scheffe的方法,P. < 0.05).
选择性扫描分析
在XP-CLR版本1.0中实现的跨人物复合似然比测试[67]用于扫描选择性扫描,在亚组下的比较中K. = 2 representing trait dominance. The SNPs with neither more than 80% deletion nor minor allele frequency less than 5% were excluded from the analysis. Genetic distances between adjacent SNPs were calculated on the basis of the proportionally increased physical distance of adjacent surrounding markers in an integrated genetic map. For each chromosome, the XP-CLR score was calculated with the following command: ‘-w1 0.5 200 $blockSize 1 -p0 0.95’. Regions with region-wise XP-CLR scores in the top 1% were considered to be candidate selective sweeps.
数据和材料的可用性
目前研究的表型数据可根据电子邮件发送到相应作者。基因分型数据可用在附加文件中4..
缩写
- QTL:
-
数量性状位点
- GWAS:
-
基因组协会研究
- LD:
-
连锁不平衡
- SNP:
-
单核苷酸多态性
- BLUP:
-
最好的线性无偏见预测
- PCA:
-
主成分分析
- 高清:
-
标题日期
- MD:
-
成人礼
- GFP:
-
粮食灌装期
- 外交政策:
-
生育时期
- TN:
-
分蘖号码
- SP:
-
上升的百分比
- SN:
-
钉号码
- pH:
-
株高
- KPS:
-
每穗粒数
- 千粒重:
-
千粒重量
- GFR:
-
颗粒填充率
- GY:
-
粮食产量
参考文献
- 1。
Karlsson Ek,Kwiatkowski DP,Sabeti PC。人口的自然选择和传染病。NAT Rev Genet。2014; 15:379。
- 2。
Atanur Santosh S,Diaz Ana G,Maratou K,Sarkis A,Sarkis A,Retival M,Game L,Tschannen Michael R,Kaisaki Pamela J,Otto Georg W,Man M,Chun J等人。基因组测序揭示了人工选择下的基因座,在实验室大鼠中涉及疾病表型。细胞。2013; 154(3):691-703。
- 3.
Liedvogel M,Åkessons,博赫斯。迁移迁移的遗传。趋势ECOL EVOL。2011; 26(11):561-9。
- 4.
Zhao K, Tung CW, Eizenga GC, Wright MH, Ali ML, Price AH, Norton GJ, Islam MR, Reynolds A, Mezey J, et al.;全基因组关联作图揭示了水稻复杂性状丰富的遗传结构。Nat Commun。2011;2:467。
- 5.
李魏赵黄X, Y, X, C,王,赵问,李W,郭Y,邓L,朱C, et al。水稻开花时间与籽粒产量性状的全基因组关联研究。Nat麝猫。2011;(1):44 32-9。
- 6.
郭志,陈丹,Alqudah AM, Röder MS, Ganal MW, Schnurbusch T.小麦小花育性的多位点关联分析。新植醇。2017;214(1):257 - 70。
- 7.
李飞,文伟,刘军,张勇,曹胜,何震,金辉,张超,闫军,等。基于全基因组关联研究的面包小麦籽粒产量遗传结构。植物学报。2019;19(1):168。
- 8。
Quintero的A,莫勒罗G,雷诺MP,DF卡尔代。权衡千粒重和粒数之间在小麦取决于GXE相互作用:一个精英CIMMYT面板(CIMCOG)为例。Jagron欧元。2018; 92:17-29。
- 9。
萨德拉斯vo。种子大小与作物中数量之间的折衷方程。现场作物RES。2007; 100(2):125-38。
- 10。
Sukumaran S,Lopes M,Dreisigacker S,Reynolds M.多环境春小麦试验的遗传分析鉴定了谷物重量和晶粒数的特定轨迹特异性折衷的基因组区域。Al Appl Genet。2018; 131(4):985-98。
- 11.
鲁克,魏莉,李X,王义,吴j,刘米,张c,陈z,小z,jian h等。全基因组重构揭示了芸薹属植物来源和遗传基因座的改善。NAT Communce。2019; 10(1):1154。
- 12.
郭某,赵某,孙h,王x,吴s,林t,任y,gao l,邓y,张j等。414种栽培和野生西瓜附加的Resequence确定了果实质量特征的选择。NAT Genet。2019; 51(11):1616-23。
- 13.
吴d,梁Z,颜T,徐Y,轩L,唐Ĵ,周G,Lohwasser U,华S,汪H,等人。全基因组重测序油菜种质的全球集合的揭示生态型差异的遗传基础。莫尔植物。2019; 12(1):30-43。
- 14.
Cavanagh Cr,Chao S,Wang S,Huang是,斯蒂芬S,Kiani S,Forrest K,Saintenac C,Brown-Guedira GL,Akhunova A等。基因组的比较多样性揭示了多种选择六倍体小麦体力样品和品种的改进目标。PROC NATL ACAD SCI。2013; 110(20):8057-62。
- 15.
在大麦和小麦中,VRN-1第一个内含子的大缺失与春季生长习性相关。中国生物医学工程学报。2005;27(1):54-65。
- 16.
周y,陈z,程m,陈俊,朱t,王r,刘y,qi p,chen g,jiang q等。揭开了中国小麦的分散历史,自适应演化和选择。植物Biotechnol J. 2018; 16(1):280-91。
- 17.
基于高密度SNP标记的埃塞俄比亚硬粒小麦遗传多样性和群体结构分析Triticum Turgidum SSP。杜伦姆).BMC Genet。2020; 21(1):18。
- 18。
Balfourier F,Bouchet S,Robert S,De Oliveira R,Rimbert H,Kitt J,Choulet F,Paux E,Consortium IWGS,Consortium B:全球遗传地理和小麦遗传多样性历史。SCI ADV 2019,5(5):EAAV0536。
- 19。
焦健,康伟。河南省年降水量的区域特征分析[J]。华北水利水电科学研究院。2007:4。
- 20。
Sidwell RJ,Smith El,McNew RW。硬红色冬小麦交叉1中籽粒产量和所选产量相关性状的遗传与相互关系。农作物科学。1976; 16(5):Cropsci1976.0011183x001600050013x。
- 21。
穆罕默迪男,谢里菲P,Karimizadeh R,Shefazadeh MK。在面包小麦籽粒产量和产量组件之间的关系在不同水可用性(旱地和补充灌溉条件下)。Notulae牡丹土壤杆菌荷兰国际花卉园艺克卢日纳波卡。2012; 40(1):195-200。
- 22。
Ferrante A, Cartelle J, Savin R, Slafer GA。在广泛的环境中,传统小麦和当代小麦的产量决定、主要成分之间的相互作用和产量稳定性。农田作物Res. 2017; 203:114-27。
- 23。
斯拉夫·帕。小麦品种阶段开发率的差异与光周期和春化的反应无关。内在早期假设的观点。j农业sci。1996年; 126(4):403-19。
- 24。
关键词:热胁迫,春小麦,产量,生理形态,性状,灌溉条件.acta botanica botanica(云南植物研究). 1994;21(6):717-30。
- 25.
Shearman VJ,Sylvester-Bradley R,Scott Rk,Foulkes MJ。与小麦产量进展相关的生理过程。农作物科学。2005; 45(1):175-85。
- 26.
Peng J, Richards DE, Hartley NM, Murphy GP, Devos KM, Flintham JE, Beales J, Fish LJ, Worland AJ, Pelica F. ' Green revolution '基因编码突变型赤霉素响应调节剂。自然。1999;400(6741):256 - 61。
- 27.
贝鲁奇A,托普AM,布鲁恩S,马吉德Ĵ,安德森SB,Rasmussen的SK。关联映射在斯堪的纳维亚冬小麦产量,株高和性状重要的是第二代生物乙醇生产。前植物SCI。2015; 6(1046)。
- 28.
Beche E,Benin G,Da Silva Cl,Munaro LB,Marchese Ja。20世纪巴西小麦生理性状的产量和变化的遗传增益。Jagron欧元。2014; 61:49-59。
- 29.
Rebetzke G,Ellis M,Bonnett D,Condon A,Falk D,Richards R. RHT13矮化基因可减少花梗长度和植物高度,以提高小麦的晶粒数和产量。现场裁剪。2011; 124(3):323-31。
- 30.
Gale Md,Youssefian S,Russell G.小麦的Dwarfing基因。Prog植物品种。1985; 1:1-35。
- 31。
FLINTHAM J,BörnerA,伍德兰A,Gale M.优化小麦籽粒产量:rHT(葛霉素不敏感)矮化基因的影响。JAS。1997年; 128(1):11-25。
- 32。
Miralles D,Slafer G.在推荐和晚期播种日期下的矮种,半矮种和春小麦的高肌菌和高度上源线的产量,生物质和产量组分。植物品种。1995; 114(5):392-6。
- 33。
GENT MP。冬小麦降低高度Isolines的冠层轻型拦截,气体交换和生物量。农作物科学。1995年; 35(6):1636-42。
- 34。
张X,杨氏,周y,He Z,XIA X. rhT-B1b,rHT-d1b和rhT8在分子标记检测到秋播中国小麦中的高度基因的分布。Euphytica。2006; 152(1):109-16。
- 35。
Lopes MS,Reynolds MP,Manes Y,Singh Rp,Crossa J,Braun HJ。“历史性”集合繁殖的“历史”集中CIMMYT弹簧面包小麦相关性状的遗传产量增益及变化。农作物科学。2012; 52(3):1123-31。
- 36。
MA D-Q,王X-W,XU L-J,ZHU Y-P,K-H Z,王D-M。黄淮小麦地区一些小麦种质衍生的矮化基因的分布。J河南大学大学。2009; 2。
- 37.
小麦赤霉病抗性的类型和组成。植物品种。1995;114(5):377 - 86。
- 38.
Kato K,Miura H,Sawada S.测绘QTL控制谷物产量及其在小麦染色体5A上的组分。Al Appl Genet。2000; 101(7):1114-21。
- 39.
高楼文W,刘俊,拉希德A,尹G,夏X,吴X,QTL对产量构成,株高和产量相关生理性状在中国小麦跨周8425B他Z.全基因组连锁分析/中国春。前植物SCI。2015; 6:1099。
- 40。
Kumar N,Kulwal PL,Balyan HS,Gupta PK。QTL映射,用于产量和产量有助于面包小麦的绘制群体中的贡献性状。mol繁殖。2006; 19(2):163-77。
- 41。
李胜,贾军,魏旭,张旭,李丽,范勇,孙辉,雷涛,等。小麦产量性状的品种间遗传图谱及QTL分析。摩尔繁殖。2007;20(2):167 - 78。
- 42。
科特A,法洛A.与GWAS的优势和特点分析的局限性:回顾。植物的方法。2013; 9(1):29。
- 43。
翟H,Feng Z,Du X,Song Y,Liu X,Qi Z,Song L,Li J,Li L,Peng H,等。Tagw2-A1的新等位基因位于精细映射的QTL中,该QTL增加了粒重,但在小麦(Triticum aestivum L)中减少晶粒数。Al Appl Genet。2018; 131(3):539-53。
- 44。
Sakuma S,Golan G,Guo Z,Ogawa T,Tagiri A,Sugimoto K,Bernhardt N,Brassac J,Mascher M,Hensel G等人。通过Homeobox基因的突变释放小麦的小麦的小麦生育。Proc Natl Acad Sci U S A. 2019; 116(11):5182-7。
- 45。
戈兰G,阿亚隆I,佩里A,就是心兰G,ADE-阿贾伊T,Mosquna A,Distelfeld A,法勒Z. GNI-A1介导在四倍体小麦权衡之间穗粒数和千粒重。Al Appl Genet。2019; 132(8):2353-65。
- 46。
Brinton J,Simmonds J,Minter F,Leverington-Waite M,Snape J,Uauy C.增加了肝细胞长度为六倍体小麦中粒重的主要定量性状基因座。新植物。2017; 215(3):1026-38。
- 47。
Stelmakh af。VRN基因对面包小麦标题和农艺性状的遗传效应。Euphytica。1992年; 65(1):53-60。
- 48。
张丽玲,刘大川,郭小龙,杨伟林,孙建忠,王德伟,张安。普通小麦产量及产量相关性状数量性状位点的基因组分布。acta botanica yunnanica(云南植物研究). 2010;52(11):996-1007。
- 49.
Ogbonnaya FC, Rasheed A, Okechukwu EC, Jighly A, Makdis F, Wuletaw T, Hagras A, Uguru MI, Agbo CU。春小麦农艺和生理性状的全基因组关联研究。中国科学:地球科学。2017;130(9):1819-35。
- 50。
Bennett D,Reynolds M,Mullan D,Izanloo A,Kuchel H,Langridge P,Schnurmusch T.在热量,干旱和高产潜在环境下检测面包小麦(Triticum Aestivum L.)中的两个主要谷物产量QTL。Al Appl Genet。2012; 125(7):1473-85。
- 51。
Hufford MB,Xu X,Van Heerwaarden J,Pyhajarvi T,Chia JM,Cartwright Ra,Elshire RJ,Glaubitz JC,Guill Ke,Kaeppler Sm,等。玉米驯化与改进的比较群体基因组学。NAT Genet。2012; 44(7):808-11。
- 52。
杜旭,黄光国,何胜,杨志,孙光国,马旭,李宁,张旭,孙军,刘敏,等。通过对243份棉花二倍体材料基因组的重新测序,确定了关键农艺性状的遗传基础。Nat麝猫。2018;(6):796 - 802。
- 53。
Sehgal D,Autrique E,Singh R,Ellis M,Singh S,Dreisigacker S.鉴定谷物产量和产量稳定性及其认证相互作用的基因组区域。SCI批准。2017; 7(1):41578。
- 54。
杨杰,周y,吴q,陈y,张p,张y,胡w,王x,zhao,dong l等。小麦(Triticum Aestivum L.)的新型塔尔3-5A等位基因的分子表征及其与粒度的关系。Al Appl Genet。2019; 132(6):1799-814。
- 55。
杨杰,周Y,张烨HW,吴q,陈y,王x,guo g,liu z,ca t等。标签3的克隆,表征与小麦内核特征相关的等位基因变异的鉴定(Triticum Aestivum.L.)。BMC Genet。2019; 20(1)。
- 56。
Evanno G,Regnaut S,Goudet J.使用软件结构检测个人的簇数:模拟研究。Mol Ecol。2005; 14(8):2611-20。
- 57。
Earl DA, vonHoldt BM。结构收割机:一个用于可视化结构输出和实现埃万诺方法的网站和程序。植物保护学报。2012;4(2):359-61。
- 58。
Lipka Ae,Tian F,Wang Q,Peiffer J,Li M,Bradbury PJ,Gore Ma,Buckler Es,张Z. Gapit:基因组关联和预测综合工具。生物信息学。2012; 28(18):2397-9。
- 59。
Kimura M:通过核苷酸序列(1980)PROC的比较研究估算基取代的进化率的简单方法。在:美国国家科学院.1181-1188。
- 60.
于国平,朱洪波,关勇,陈立平。系统发育树及其协变量和相关数据可视化和注释的R包。生态学报。2017;8(1):28-36。
- 61。
WEIR BS,OTT J.遗传数据分析II。趋势类型。1997年; 13(9):379。
- 62。
Bradbury PJ,张Z,Kroon de,Casstevens TM,Ramdoss Y,Buckler Es。TASSEL:多样化样本中复杂性状的关联映射软件。生物信息学。2007; 23(19):2633-5。
- 63。
罗萨斯Ĵ,费雷尔-马塔A,桑切斯 - DelBarrio JC,Guirao-波多黎各S,迎战Librado P,拉莫斯 - Onsins SE,桑切斯 - Gracia的A. DnaSP 6:大数据集的DNA序列多态性分析。mol Biol Evol。2017; 34(12):3299-302。
- 64。
Bates D, Mächler M, Bolker B, Walker S:线性拟合使用lme4混合效应模型.arXiv预印本arXiv: 140658232014年。
- 65。
Piepho H-P,MöhringJ.计算遗传性和植物育种试验的遗传学和选择响应。遗传学。2007年; 177(3):1881。
- 66。
Wei T,Simko V,Levy M,Xie Y,Jin Y,Zemla J.包'Corrplot'。统计员。2017; 56:316-24。
- 67。
Chen H,Patterson N,Reich D.人口分化为选择性扫描的测试。Genome Res。2010; 20(3):393-402。
确认
作者感谢浙江大学农业部Dezhi Wu教授,用于对这款手稿的关键审查。
资金
这项研究受到了河南农业研究系统的专项基金(S2010-01-G03),中国国家重点研发项目(2016 yfd0100402),河南科技项目(192102110138),河南省科学技术的特殊项目的基础和条件。国家重点研发计划项目(2016YFD0100402)和河南省科技基金及条件专项项目资助。河南省农业科研体系专项(S2010-01-G03)资助栽培、表型及手稿编制。GWAS分析和选择性扫描分析由河南省科技计划项目(192102110138)资助。
作者信息
隶属关系
贡献
ZL和TC构思了这项研究。YZ,YZ,WH,YC,XW和Hz收集现场数据。JY,YZ和TC进行了人口结构分析和GWA。YZ进行了选择性扫描分析。JY,YZ和ZL写了稿件。所有作者均阅读并批准了手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者声明他们没有竞争利益。
附加信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1。
(a)PCA分析(PC1和PC3,PC2和PC3)的曲线图示出了分配在结构结果中的四个群体。(b)基因组LD(R.2)抗遗传距离的分布。虚线标记每个0.2R.2.
附加文件2:图S2。
全基因组关联分析和12个农艺性状位数 - 分位数图。性状名称被标记在上部或底部各图。曼哈顿地块,负-log10P.来自基因组扫描的值绘制在21个染色体中的每一个上的物理位置。红线表示基因组显着性阈值。
附加文件3:表S1。
96个河南小麦品种的信息,包括章程及其结构中确定的亚贫瘠作业。
附加文件4:表S2
96个小麦品种的snp和基因型信息。染色体和位置信息参照中国春季参考基因组(CS Ref v1.0, IWGSC)的物理位置。
附加文件5:表S3。
四种亚流域群体遗传参数的比较。
附加文件6:表S4。
标志性关联,磷酸QTL和12个农艺性状的候选基因的信息。
附加文件7:表S5。
全基因组扫描选择性扫描信号,用于k = 2的亚步骤。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
杨,J.,Zhou,Y.,Hu,W。et al。解锁人口结构,植物建筑,生长季节,河南小麦品种环境适应的关系。BMC植物杂志20,469(2020)。https://doi.org/10.1186/s12870-020-02674-z.
已收到:
公认:
发表:
关键字
- 精英小麦品种
- 人口结构
- 收益率
- 格干
- 选择扫描