摘要
背景
籽粒行数是玉米驯化改良的一个重要性状。探索KRN的遗传基础具有重要的研究意义,可为分子辅助选择提供有价值的信息。
结果
本研究采用一种单基因座法(MLM)和六种多基因座法(mrMLM、FASTmrMLM、FASTmrEMMA、pLARmEB、利用全基因组关联研究(GWASs)的pKWmEB和ISIS EM-BLASSO)方法,在639个玉米自交系的关联小组中鉴定了KRN的显著数量性状核苷酸(QTNs),这些自交系由MaizeSNP50 BeadChip分型。在3种表型环境下,7种GWAS方法以最佳线性无偏预测(BLUP)值显示不同数量的krn相关QTNs,范围在11 ~ 177。在此基础上,通过至少三种方法和至少两种环境鉴定出了位于染色体1、2、3、5、9和10上的7个重要的KRN区域。此外,来自7个区域的49个基因在不同的玉米组织中表达。在49个基因中,ARF29(Zm00001d026540,编码生长素反应因子29)CKO4.(Zm00001d043293,编码细胞分裂素氧化酶蛋白)与KRN显著相关。我们还对KRN进行了全基因组预测(WGP),发现KRN相关的标记snp具有较高的预测精度。最好的策略是整合所有GWAS模型识别的所有krn相关的标记snp。
结论
这些结果有助于我们理解玉米KRN的遗传结构,并为玉米KRN的基因组选择提供有用的信息。
背景
玉米(玉米大刍草(teosinte)起源于大约9000年前墨西哥南部的野生祖先大刍草(teosinte)的一次驯化事件,现在是世界上最重要的谷类作物之一。1].在驯化过程中,其形态特征,特别是花序结构发生了深刻的变化[2那3.].现代玉米从大刍草的小穗向大穗的转变伴随着粒行数(KRN)的急剧增加[4.].因此,人们一直在努力探索玉米花序结构和KRN差异显著的遗传基础。
KRN是一种重要的穗型性状,在雌性花序发育过程中由多个分生组织类型形成,包括花序分生组织(IMs)、小穗对分生组织(SPMs)、小穗分生组织(SMs)和花分生组织(FMs) [5.].到目前为止,通过研究突变体,已经克隆了一些基因,并发现它们参与了负责分生组织发育和KRN修饰的复杂调控网络[6.那7.那8.那9.那10].然而,这些古典突变体显示出与植物建筑有关的其他性状的阴性胸膜复制,并且难以直接在玉米育种中使用[11].因此,连锁定位和关联定位已经在不同的自然群体中进行,目的是鉴定更多的控制KRN的优秀自然等位基因。
通过连锁定位,在双亲本分离群体中发现了许多与KRN相关的数量性状位点(qtl),但由于其遗传效应较小,尚未成功克隆KRN4[12),KRN1[13].KRN的全基因组关联研究(GWASs)也已进行,并发现许多数量性状核苷酸(QTNs) [14那15那16].同时,GWAS结果很容易受到种群结构和自然种群中罕见变异的影响[17].因此,在使用GWAS方法时,已经开发了许多统计模型来提高识别基因型-表型关联的能力,如单位点混合线性模型(MLM)方法[18那19]和多位点方法mrMLM [20.ISIS em-blasso [21], pLARmEB [22], FASTmrEMMA [23], pKWmEB [24]和fastmrmlm [25].MLM方法是一种单位点固定单核苷酸多态性(SNP)效应方法,用于多基因背景的情况下控制群体结构[18那19].为了降低假阳性率(FPR),严格的Bonferroni校正用于传销方法中的多次检测校正[26].多位点方法是一种基于随机snp效应模型的备选GWAS程序,不需要多次测试校正[26].在这个模型中有两个步骤。首先,通过不同的算法选择较少的SNPs,然后将SNPs用于多位点模型中检测真信号[20.那21那22那23那24那25那26].近年来,少数研究采用上述GWAS方法检测水稻不同性状的重要基因座[27)、玉米(28],亚麻[29,面包小麦[30.]和陆地棉花[31那32].
以往的研究表明,KRN是定量遗传的,单个遗传位点的效应一般较小,这对玉米育种的遗传改良提出了挑战。因此,最好的方法是通过对分布在整个基因组的更多标记的综合分析来提高预测KRN的能力。基因组选择(GS)或全基因组预测(WGP)能够利用全基因组数据提高育种效率[33].在以往的研究中,对F进行KRN的WGPs1重组自交系之间的杂交种[34,相互联系的双亲本玉米种群[35]和339个玉米自交系[36,表明KRN是一个适合全基因组预测的性状。Liu等[15[展示大约300个顶部Krn相关的TybsnPS足以使用RIDGE回归最佳线性无偏析预测(RR-BLUP)预测近交系和混合动力车的KRN。基于这些分析,我们面临着确定如何选择更少的标记以准确预测KRN。几项研究报告说,从GWAS的结果中选择关联标记,并将其作为WGP模型中的固定效果产生更好的性能,而不是单个WGP模型实现的性能[37那38那39].这可能提供了一种同时对遗传结构的不同方面建模的方法,对育种者来说尤其容易[39].
在这项研究中,我们通过使用一种单个基因座方法,MLM方法和六种多层方法,MRMM,FastMRMLM,FastMMA,PlarmeB,PKWMEB和ISIS,执行了基于MaizeNP50 Beadchip的一个关联面板的GWA,包括基于MaizeNP50 BeAdchip的麦克解,包括639麦小读线。em-blasso。分析了通过不同方法和跨越不同环境的常见显着的QTN,并进一步预测与KRN相关的候选基因。使用各种KRN相关的TACSNPS还进行WGP来描述KRN的遗传结构。
结果
关联面板中KRN的自然变异
2011年XX(河南省新乡,35.19°N, 113.53°E)、BJ(北京,39.48°N, 116.28°E)和GZL(吉林省公竹岭,43.50°N, 124.82°E)共639个玉米自交系测定了KRN(表S)1).结果表明,KRN在各环境中呈正态分布,各环境之间的KRN呈高度正相关,XX与BJ之间的相关系数为0.73,XX与GZL之间的相关系数为0.79(图1)。1a). KRN表现出较高的广义遗传力(H2= 0.90,表1),这与以前的研究结果相似[14那16].通过对不同环境的KRN进行比较,我们发现XX的KRN平均值最小(13.69),最小值(8.60)和最大值(20.60),所有植物均种植在夏季(6月)。随着纬度的增加,春季(5月)的平均KRN增加(北京14.65,GZL 14.59)。KRN的最大-最小范围出现在日长最长的GZL(12.60)中1).根据以往的研究结果[40],我们的协会面板可分为五个亚组:Reid,Tangsipingtou(TSPT),Lvdahonggu(LRC),Lancaster和P。各个子组的KRN统计分析结果显示在表S中2.5个亚组间KRN无显著差异(图。1b).这些结果表明,KRN是一个数量性状,在关联面板中检测的自交系之间的表型变异有助于剖析KRN的遗传结构。
不同方法鉴定的KRN的qtn
KRN (MLM)单基因座分析
基于MaizeSNP50 BeadChip,我们获得了分布在10条玉米染色体上的42,667个高品质SNPs。下P.< 0.0001和P.< 0.001阈值,在XX中发现3/56、3/46、1/24和3/51 krn相关QTNs(图。2a),在bj中(图。2b),在GZL中(图。2c)和BLUP(图。2分别d)。为了解释这个模型中的过度修正,P.选择< 0.001阈值来识别krn相关qtn。共发现177个QTNs与KRN相关,这些QTNs解释表型变异的比例为1.84 ~ 4.01%(表S3.).
KRN的多位点分析
使用不同的多基因座模型,我们在XX,BJ和GZL中识别出KRN的不同数量的QTN,以及所有位置的Blup。这些QTN在10个染色体上不均匀地分布,最多QTNS上的CHR。1和最少的CHR。8(图。2e).具体来说,XX的15个(FASTmrEMMA)-177个(mrMLM) QTNs, BJ的11个(FASTmrEMMA)-30个(ISIS EM-BLASSO) QTNs, GZL的12个(FASTmrEMMA)-55个(mrMLM) QTNs, BLUP的11个(FASTmrEMMA)-106个(mrMLM) QTNs(表S)4.).对比分析不同统计方法的GWAS结果,FASTmrEMMA在所有环境中检测到的qtn最少,而mrMLM在所有环境中检测到的qtn最多,除了BJ(表S)4.).7种方法之间的QTN重叠分析表明,至少两种方法共同检测的共同QTNs占不同环境QTNs的40%以上(图S)1A和表格5.XX为42%,BJ为62%,GZL为58%,BLUP为47%)。例如,65普通本考察团代表30位点被两种方法在XX, codetected和39常见本考察团代表13个位点,28常见本考察团代表7个位点,25常见本考察团代表5位点和6常见本考察团代表1轨迹codetected三、四、五和六个方法,分别(图1A和表格5.).7种方法均未在不同部位鉴定出qtn。总体而言,ISIS EM-BLASSO检测到的qtn数量第三多,识别出的共检测qtn最多,其次是FASTmrMLM(图S)1A和表格5.).对不同环境中的GWA结果的比较分析表明,在不同位置反复检测到MLM方法和ISIS EM-Blasso鉴定的大部分QTNS(图S1b,表6.).
综上所述,将我们的GWAS结果与前人的研究结果进行比较,我们发现一些控制玉米花序结构的重要基因位于显著qtn的200 kb范围内,包括CT2(Zm00001d027886),FEA3(Zm00001d040130),BAD1(Zm00001d005737),RA1(Zm00001d020430),VT2(Zm00001d008700)(表13).
KRN候选基因的注释和表达
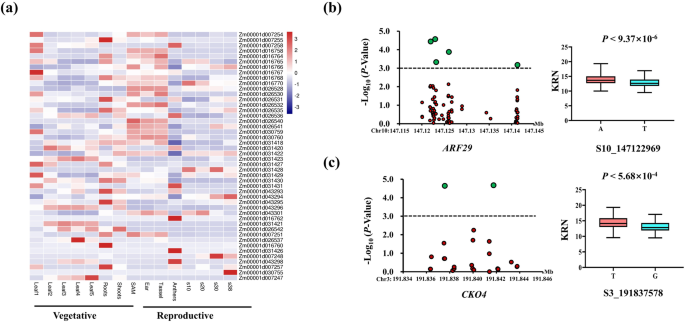
为了获得可靠的显著QTNs并预测KRN的候选基因,我们只使用至少三种方法(单位点或多位点)和至少两种环境下同时鉴定的QTNs进行下一步分析。最后得到7个控制KRN的qtn(表)2).7个QTNs分别位于第1、2、3、5、9和10号染色体上,其PVE范围为1.06 ~ 5.21%。基于关联面板中的连杆不平衡(LD)(图S)2),获得QTNS(上游和下游)周围的49个基因,它们的表达在不同的玉米组织中广泛变化(图。3.A和表格7.).例如,Zm00001d016760编码脱落酸压力成熟6蛋白,根中高度表达,和Zm00001d031426编码丝氨酸/ threonine-protein激酶,Zm00001d043298,编码一个P-loop包含三磷酸核苷水解酶超家族蛋白,流苏和花药中高度表达。49个基因中,22个在不同穗发育突变体中有差异表达(表S8.);即ra1那红警2和ra3中突变体具有异常的高度分枝的流苏和耳朵,耳朵显示非常大的KRN [41];的kn1突变体有更小的穗和更少的小穗[42].这一结果表明,这22个基因可能参与玉米中的耳发。
有趣的是,我们发现Zm00001d026540(编码生长素响应因子29,ARF29)位于Chr. 10上pse -110106563下游200 kb内,可通过MLM法和所有6种多位点GWAS方法检测(表1)2),在sam和耳中表达高于其他组织(表S7.).同时进行候选基因关联作图。的单核苷酸多态性ARF2910-kb启动子和10-kb区域的下游ARF29从玉米HapMap3 [43].测量了282个自交系在6个环境下的KRN(见方法),计算BLUP值。MLM定位结果显示,5个SNPs(2个SNPs在基因内,3个SNPs在基因上游区域)左右ARF29与KRN显著相关(图。3.b和表3.).ARF29可以绑定Bif1(与SAM发育和最终KRN相关)启动子通过识别TTTCGG motif [44那45].位于基因体内的S10_147,122,969 SNP位点与KRN显著相关。该SNP (A/T)有两个等位基因,其中A等位基因具有更高的KRN。细胞分裂素也在未成熟尖刺的发育和最终KRN的形成中发挥重要作用[46].例如,UB3通过细胞分裂素途径和CLAVATA-WUSCHEL途径调控KRN [46].在这项研究中,CKO4.(Zm00001d043293,编码细胞分裂素氧化酶蛋白)通过四种GWAS方法(MLM、mrMLM、pLARmEB和pKWmEB)检测到位于Chr. 3上的put -163a-110、967、306-138上游200 kb内2)和候选基因关联作图CKO4.也进行了。SNP和KRN也从HAPMAP3和282近交系中获得。MLM结果表明,两个SNP位于上游CKO4.与KRN显著相关(图。3.c和表3.).S3_191,837,578 SNP有两个等位基因(T/G),其中T等位基因与较高的KRN相关,但频率较低。因此,该等位基因在玉米育种中可能没有广泛的应用。
KRN的全基因组预测
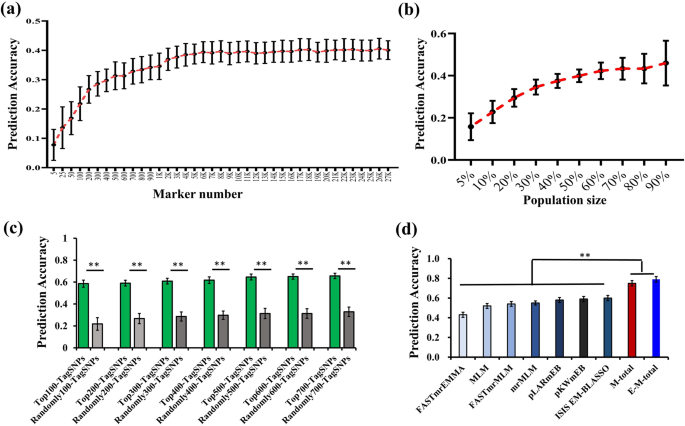
我们首先使用阈值对所有标记的LD块进行分析r2> 0.2,在我们的关联面板中获得27,688个tagSNPs。然后,我们在全基因组中随机选取5 ~ 27000个不同数量的tagSNPs,计算自交系KRN的预测精度,即预测值与模拟值的相关性。结果表明,随着tagSNPs数量的增加,预测准确率增加(图2)。4.A和表格9.).具体来说,当tagSNPs数量从5个增加到500个时,预测准确率急剧上升,当tagSNPs数量从400个增加到2000个时,预测准确率缓慢上升。一旦数字超过2000,预测的准确性就一直保持在较高水平。虽然使用了大量的tagSNPs对KRN进行预测,但预测精度仍小于0.5。训练群体大小对预测准确性的影响也基于14000个标记(约占总标记snp的50%)进行评估。在关联面板中,预测精度随着训练人群规模的增加而提高。当训练人口规模从50增加到90%时,预测精度有轻微的提高。4.b和表s10).
为了更好地了解KRN的遗传结构,提高对KRN的预测能力,我们根据MLM方法获得的与KRN相关的显著性对27688个tagSNPs进行排序,得到最高的tagSNPs。我们发现,这些顶级的tagSNPs的预测精度(范围从0.58的前100个tagSNPs到0.66的前700个tagSNPs)高于随机选择的tagSNPs(范围从0.22的100个随机tagSNPs到0.33的700个随机tagSNPs)。4.c及表S11).
我们收集了基于BLUP的不同模型检测到的代表显著QTNs的tagsnp,并在我们的关联面板中计算KRN的预测精度。结果表明,不同方法识别的这些tagSNPs的预测精度从0.43 (FASTmrEMMA)到0.60 (ISIS EM-BLASSO)不等(图2)。4.d和表S12).我们还发现,同一方法识别的与KRN相关的tagSNPs在不同环境下的预测精度不同(图S)3.和表年代12).为了探究在不同GWAS方法中使用共检测的QTNs是否能够提高KRN的预测精度,我们选取了至少2、3、4、5或6种方法鉴定的常见QTNs进行预测。结果表明,只有两种以上方法鉴定的共同QTNs(共同≥2)才能保持较高的可预测性;其他常见QTN在预测KRN方面没有优势,这可能是由于QTN数量较小(图S)3.和表年代12).
另外,为了提高预测能力,我们将7种方法检测到的KRN相关的tagSNPs(204个在XX, 87个在BJ, 118个在GZL, 167个在BLUP)放在一个单一环境中,即M-total tagSNPs,进行KRN预测。结果,我们发现预测精度有了很大的提高,XX的预测精度达到了0.74,BJ的预测精度达到了0.66,GZL的预测精度达到了0.75,BLUP的预测精度达到了0.75(图3)。4.d和表S12).在每个环境中,这些可预测性都比单一方法高得多(表S12).然后,我们收集了所有方法和所有环境中与KRN相关的tagsnp,即E-M-total tagsnp,共得到439个tagsnp。然而,当我们使用更多的E-M-total tagSNPs而不是更少的M-total tagSNPs时,预测的准确性只有轻微的提高(从BJ的0.68到BLUP的0.79,对于439个tagSNPs)。4.d和表S12).
讨论
迄今为止,GWAS方法已经被广泛用于研究许多物种重要性状的遗传基础,通过计算基因型和相应表型变异之间的关联[47].为了识别真关联信号,人们建立了许多基于不同算法的统计方法。在本研究中,我们选择了一种单基因座方法(MLM)和六种多基因座方法(mrMLM、FASTmrMLM、FASTmrEMMA、pLARmEB、pKWmEB和ISIS EM-BLASSO),在我们的关联panel中对KRN进行全面的GWAS mapping。在这7种方法中,mrMLM识别出的QTNs数量最多,FASTmrEMMA识别出的QTNs最少,ISIS EM-BLASSO识别出的共检测的QTNs最多,这与Cui等报道的结果一致[27水稻耐盐基因座。因此,多位点模型是研究玉米KRN遗传变异的有价值的替代方法。此外,在Peng等人的研究中也观察到少数通过不同方法共同检测的常见QTNs [30.研究面包小麦中游离氨基酸的含量。
将我们的GWAS结果与前人的研究结果进行比较,我们发现一些控制玉米花序结构的重要基因位于200 kb的显著QTNs范围内(表S13),包括CT2(Zm00001d027886),FEA3(Zm00001d040130),BAD1(Zm00001d005737),RA1(Zm00001d020430),VT2(Zm00001d008700)。在这些基因中,CT2[7.),FEA3[10]在CLAVATA-WUSCHEL反馈信号中起作用,它们的突变导致耳原基增大、筋膜化和KRN增加。BAD1[48),RA1[41],它们都编码转录因子,通过ROMASO途径参与玉米花分支系统的遗传调控。VT2[49对植物营养和生殖发育有显著的影响,且突变体穗表现出明显的缺陷。此外,在之前的花序发育GWAS作图中,LD区域内约60%的重要QTNs是共同检测到的,其中一些位点是多效性的[14那15].
WGP也是动物育种和植物改良的一种有效方法[50].因为KRN主要由附加基因座控制,所以我们选择了RRBLUP添加剂模型来进行WGP [51].正如预期的那样,随着随机选择的tagsnp数量的增加,预测的准确性也会增加,这与Liu等人的发现是一致的[15],并由标记密度对WGP的影响确定[50].然而,随机选取的tagsnp预测能力较低,因此,我们决定将GWAS结果与WGP结合,探索最佳的KRN预测标记数据集。因此,在使用显著标记snp时,很容易达到较高的预测水平,且中高值与Liu等报道的值一致[15, Guo等[34, Riedelsheimer等[35Xu等人[36].这一结果表明,将GWASs的显著信号作为固定效应整合到WGP模型中可以有效提高KRN的预测。Liu等人也得出了类似的结论[15]为KRN,由边和荷兰[52玉米对南方叶枯病(SLB)、灰斑病(GLS)和株高(PHT)的抗性[39用于热带水稻改良。尽管在玉米和高粱中对含有峰值GWAS信号的WGP模型进行了不同的评估[53,我们的研究表明,使用上述GWAS方法中通过某一阈值的QTNs作为rrBLUP模型中的固定效应,是预测KRN的有力工具,这在本研究中是给定群体中特定性状的考虑。
基于本研究结果,我们认为KRN是由许多加性位点控制的,rrBLUP模型可用于玉米自交系的KRN预测。不同GWAS方法的联合利用有助于玉米育种中候选基因和KRN的预测。
结论
本研究采用多种GWAS方法鉴定玉米KRN的显著QTNs。7种GWAS方法发现不同数量的krn相关qtn,范围在11 ~ 177。在此基础上,通过至少三种方法和至少两种环境鉴定出了位于染色体1、2、3、5、9和10上的7个重要的KRN区域。此外,来自7个区域的49个基因在不同的玉米组织中表达。在49个基因中,ARF29(Zm00001d026540,编码生长素反应因子29)CKO4.(Zm00001d043293,编码细胞分裂素氧化酶蛋白)与KRN显著相关。我们还对KRN进行了WGP,我们发现KRN相关的标签snp具有较高的预测准确率。最好的策略是整合所有GWAS模型识别的所有krn相关的标记snp。这些结果将有助于我们了解KRN的遗传基础,并为进一步研究这一重要性状提供重要的候选基因。
方法
植物材料与表型
639个玉米自交系组成的联合小组,代表了中国温带自交系的广泛遗传多样性[54],被收集用于GWASs。我们声明,本研究中的所有植物材料均符合《濒危野生动植物物种贸易公约》。本研究使用的植物材料在我们实验室保存。
2011年在吉林省拱竹岭(43.50°N, 124.82°E)、河南省新乡市(35.19°N, 113.53°E)和北京市(39.48°N, 116.28°E) 3个环境下进行3个重复的随机区组设计。为了便于描述,这三个环境分别被指定为GZL、XX和BJ。在每个地点,田间试验包括单行3米长,相邻行0.6米,每行12株。中国农业科学院作物科学研究所在上述地点均建立了田间试验基地。作物科学研究所批准了大田试验,大田管理遵循当地的玉米管理做法。本研究未涉及濒危或保护物种。
每个品系采穗5个,在穗中部评价KRN [54].BLUP值采用SAS PROC MIXED模型计算,随机效应为基因型、环境和复制[14那55].广义遗传力(H2),根据Wu等人计算[40].变异系数计算为简历(%) = SD/均值,其中SD和均值分别为KRN在各环境中的标准差和均值[55].
DNA提取和基因分型
采集各玉米株系5株幼嫩叶片进行基因组DNA提取。我们采用十六烷基三甲基溴化铵(CTAB)法提取基因组DNA [56].所有样品都是用麦格纳普50珠芯片检查和基因分型的质量,这是从B73参考序列开发的56,110玉米SNP的Illumina Beadchip阵列[57].然后,将缺失率超过20%、次要等位基因频率(MAF) < 0.05的成功称为SNPs的基因分型数据集排除[58].在此之后,使用42,667个高质量的SNPs进行进一步分析。
GWAS映射
本研究采用1种单基因座法、MLM和6种多基因座法,分别为mrMLM、FASTmrMLM、FASTmrEMMA、pLARmEB、pKWmEB和ISIS EM-BLASSO。对每一个频率较小的多态性位点的等位基因进行进一步分析。利用TASSEL 5.2程序计算亲缘关系矩阵并进行主成分分析(PCA) [59].在TASSEL 5.2中也产生了一个控制种群结构(Q)和亲属关系(K)的传销(MLM Q + K) [18那19].6种多位点GWAS作图方法与mrMLM软件包一起使用。在R环境中的GUI v3.2 (http://127.0.0.1:5846/)[26].所有参数均设置为默认值,MLM显著关联的临界阈值设置为-log10P.≥3,设置6种多位点方法的LOD评分≥3 [26].
候选基因分析
使用关联面板中的物理距离的LD衰减在Tassel 5.2中计算为200 kB(图S2).基于MaizeGDB的B73参考基因组V4 (https://www.maizegdb.org/).这些基因的表达数据是从以前的研究中收集的[42那60].从玉米HapMap3数据集中获得了包含选定基因内SNPs的基因组片段,包括10 kb启动子区、基因体和基因下游10 kb区域[43].在282个不同品系的全球玉米协会图谱上进行候选基因定位分析。该关联面板的表型已在我们之前的报告中提供[40],并于2009年和2010年在北京、河南新乡和新疆乌鲁木齐等6个环境中测定了KRN。采用TASSEL 5.2中的MLM方法进行关联分析,控制了种群结构(Q)和亲缘关系(K)。前三个主成分(PCs)在之前的研究中分析[40,作为共变异,控制282行关联图面板中现有的种群结构。显著的标记-性状关联在-log中声明10P.> 3。
KRN的基因组预测
为了预测自交系的KRN,我们估计了WGP的可预测性。我们在PLINK软件中将LD模块分组[61使用阈值r2> 0.2,根据LD块识别tagsnp。采用岭回归最佳线性无偏预测(rrBLUP)软件包进行R [62].我们随机选取协会面板中一半的自交系作为训练群体(320个自交系),其余319个自交系作为验证群体[15].我们使用不同方法标识的KRN相关的TAGSNPS在四个条件下对近交线进行KRN的基因组预测(XX,BJ,GZL和BLUP)。Simultaneously, 5 to 27,000 randomly selected tagSNPs, the total tagSNPs related to KRN identified by the seven methods in a single environment (M-total tagSNPs), the total tagSNPs for KRN from all methods and environments (E-M-total tagSNPs) and the common tagSNPs for KRN detected by at least two, three, four, five, or six methods were also used for the same procedure. The random sampling of tagSNP numbers, the training and validation populations and the predictions were all repeated 100 times.
数据和材料的可用性
在当前研究期间使用和/或分析的数据集可以在合理的请求时从相应的作者获得。
缩写
- BJ:
-
北京
- FMs:
-
植物分生组织
- GWAS:
-
全基因组关联研究
- GZL:
-
公主岭
- 即时通讯:
-
花序分生组织
- KRN:
-
内核行号
- LD:
-
连锁不平衡
- 传销:
-
混合线性模型
- QTL:
-
数量性状位点
- 本考察团:
-
定量特征核苷酸
- 短信:
-
小穗分生组织
- SNP:
-
单核苷酸多态性
- spm:
-
一对小穗分生组织
- XX:
-
新乡
参考文献
- 1.
关键词:玉米,多位点微卫星基因分型,单驯化美国国家科学研究院2002;99:6080-4。
- 2.
Iltis HH。从大刍草到玉米:灾难性的性转化。科学。1983;222:886 - 94。
- 3.
Iltis HH。同源性易位与玉米的起源(Zea Mays.那禾本科):用新眼光看待旧问题。经济学机器人。2000;54:7-42。
- 4.
玉米进化的遗传学。2004; 38:37-59。
- 5.
王志强,王志强。拟南芥的转录生物学研究进展[j]。植物杂志。2009;149:38-45。
- 6.
研究发现,玉米穗2基因编码一个富含亮氨酸的重复受体样蛋白,调控玉米茎分生组织增殖。基因Dev。2001;15:2755 - 66。
- 7.
玉米Galpha基因COMPACT PLANT2在CLAVATA信号通路中调控茎分生组织大小。大自然。2013;502:555-8。
- 8.
Bommert P, Nagasawa NS, Jackson D.玉米籽粒行数的数量变化受FASCIATED EAR2位点控制。Nat麝猫。2013;45:334-7。
- 9.
玉米SBP-box转录因子unbranched2和unbranched3通过调控侧原基起始率来影响产量性状。中国科学技术大学学报(自然科学版)2014;111:18775-80。
- 10.
李永强,李永强,李永强,等。玉米器官原基信号通过FASCIATED EAR3调控干细胞增殖和产量性状。Nat麝猫。2016;48:785 - 91。
- 11.
李敏,钟伟,杨飞,张志强。玉米花序结构数量性状基因座的遗传和分子机制。植物生理学杂志。2018;59:448-57。
- 12.
刘丽,杜勇,沈旭,李敏,孙伟,黄建军,等。KRN4控制玉米籽粒行数的数量变异。公共科学图书馆麝猫。2015;11:e1005670。
- 13.
王军,林铮,张旭东,刘洪,周磊,钟胜,等。KRN1是玉米籽粒行数的主要数量性状位点。新植醇。2019;223:1634-46。
- 14.
杨志强,杨志强,杨志强,等。玉米雄性和雌性花序性状的不同遗传结构。公共科学图书馆麝猫。2011;7:e1002383。
- 15.
刘丽,杜勇,霍东,王敏,沈欣,岳波,等。玉米粒行数的遗传结构及全基因组预测。Theor Appl Genet. 2015; 128:2243-54。
- 16.
肖勇,童华,杨旭,徐胜,潘强,乔飞,等。利用多群体对玉米穗遗传结构的全基因组分析。新植醇。2016;210:1095 - 106。
- 17.
余建平,王志强,王志强,等。玉米关联群体:数量性状位点分析的高分辨率平台。植物j . 2005; 44:1054 - 64。
- 18.
张艳梅,毛勇,谢超,Smith H,罗磊,徐松。玉米商品自交系自然发生遗传变异的数量性状位点定位玉米l .)。遗传学。2005;169:2267 - 75。
- 19.
于建军,王志强,王志强,等。一种用于关联映射的统一混合模型方法,该方法考虑了多个层次的相关性。Nat麝猫。2006;38:203-8。
- 20.
王少波,冯建勇,任文龙,黄波,周磊,温永杰,等。通过多位点混合线性模型方法提高全基因组关联研究的能力和准确性。Sci众议员2016;6:19444。
- 21.
谭巴CL .,倪永林,张永明。多位点全基因组关联研究的迭代确定独立筛选EM-Bayesian LASSO算法。公共科学图书馆。2017;13:e1005357。
- 22.
张娟,冯建勇,倪玉玲,温雅娟,牛勇,Tamba CL . pLARmEB:最小角度回归与经验贝叶斯在多位点全基因组关联研究中的集成。遗传(Edinb)。2017; 118:517-24。
- 23.
温宇军,张辉,倪玉玲,黄波,张健,冯建勇,等。混合线性模型在多位点全基因组关联研究中的方法学实施。短暂的Bioinform。2018;19:700-12。
- 24.
任文龙,温永杰,邓伟尔JM,张永明。pKWmEB:在多基因背景控制下将Kruskal-Wallis检验与经验贝叶斯相结合进行多位点全基因组关联研究。遗传。2018;120:208-18。
- 25.
谭巴CL,张永明。多位点全基因组关联研究的快速mrMLM算法。bioRxiv。2018.https://doi.org/10.1101/341784.
- 26.
张永明,贾泽,邓伟尔。多位点GWAS新方法在复杂性状遗传解剖中的应用。植物学报。2019;10:100。
- 27.
崔勇,张飞,周勇。多位点GWAS在水稻耐盐基因座检测中的应用。植物科学学报2018;
- 28.
徐勇,杨涛,周勇,尹胜,李鹏,刘军,等。基于单位点和多位点模型的玉米淀粉糊化特性全基因组关联定位。植物学报。2018;9:1311。
- 29.
他,肖j,拉希德ky,姚z,李p,jia g等。基因组 - 亚麻抗脂肪抗性的协会研究(亚麻属植物usitatissimuml .)。前面。植物科学。2019;9:1982。
- 30.
彭勇,刘华,陈建军,史涛,张超,孙东,等。面包小麦自由氨基酸水平的6个多位点模型的全基因组关联研究。植物科学学报,2018;
- 31.
陆地棉纤维品质性状遗传分析的单位点和多位点关联研究陆地棉l .)。植物科学学报2018;
- 32.
苏军,马强,李敏,郝芳,王超。中国早熟陆地棉纤维品质相关性状的多位点全基因组关联研究。植物科学学报2018;
- 33.
Heffner E, Sorrells M, Jannink J-L。作物改良的基因组选择。作物科学。2009;49:1-12。
- 34.
郭涛,李华,严军,唐军,李军,张震,等。2个优良玉米自交系重组自交系间F1杂种性能预测。Theor Appl Genet. 2013; 126:189-201。
- 35.
Riedelsheimer C, Endelman JB, Stange M, Sorrells ME, Jannink JL, Melchinger AE。相互联系的双亲本玉米群体的基因组可预测性。遗传学。2013;194:493 - 503。
- 36.
徐勇,徐超,徐胜。玉米农艺性状多组学预测及关联作图。遗传(Edinb)。2017; 119:174 - 84。
- 37.
张志刚,高宁,何军,等。利用全基因组关联研究的结果提高复杂性状全基因组预测的准确性。《公共科学图书馆•综合》。2014;9:e93017。
- 38.
Aruda MP,Lipka Ae,Brown PJ,Krill Am,Thurber C,Brown-Guedira G,等。比较基因组选择和标记辅助选择对小麦的镰刀镰刀菌钝性抗性(小麦l .)。摩尔品种。2016;36:84。
- 39.
作者相关论文:shindel JE, Begum H, Akdemir D, Collard B, Redona E, Jannink JL, et al.;包含全新GWAS的全基因组预测模型是热带水稻改良的一个强有力的新工具。遗传(Edinb)。2016; 116:395 - 408。
- 40.
吴旭,李勇,石勇,宋勇,张东,李超,等。联合连锁定位和GWAS揭示了大量调控玉米雄性花序大小的遗传位点。植物生物技术学报2016;14:1551-62。
- 41.
王志强,王志强,王志强,等。玉米及相关禾本科植物花枝系统结构的研究。大自然。2005;436:1119-26。
- 42.
Bolduc N, Yilmaz A, Mejia-Guerra MK, Morohashi K, O’connor D, Grotewold E,等。玉米分生组织KNOTTED1调控网络的解开。基因Dev。2012;26:1685 - 90。
- 43.
郭旭东,卢勇,邹超,何波,荣志,等。建设第三代玉米单倍型映射。傻瓜。2017; 7:1-12。
- 44.
玉米营养和花序发育过程中不育花序在器官发生中的作用。遗传学。2008;179:389 - 401。
- 45.
陈志强,陈志强,陈志强,陈志强,等。玉米生长素反应因子家族的DNA结合景观。Nat Commun。2018;9:4526。
- 46.
杜勇,刘丽,李敏,方胜,沈欣,褚军,等。UNBRANCHED3通过调节玉米和水稻细胞分裂素的生物合成和信号转导来调节分支。新植醇。2017;214:721-33。
- 47.
肖勇,刘洪,吴磊,严建军。玉米全基因组关联研究:赞美和凝视。摩尔。2017;10:359 - 74。
- 48.
杜兰蒂尼;凯洛格(Kellogg EA);玉米穗分枝角的正常形成需要TCP转录因子——分枝角缺陷1 (BRANCH ANGLE DEFECTIVE 1, BAD1)。中国科学院院刊2012;109:12225-30。
- 49.
等。消失的tassel2编码一种草特异性色氨酸转氨酶,这是玉米营养和生殖发育所必需的。植物细胞。2011;23:550 - 66。
- 50.
王志强,王志强,王志强,等。基因组选择:植物改良的全基因组预测。植物科学进展。2014;19:592-601。
- 51.
张志强,何建平,高宁,等。利用遗传结构增强的方差-协方差矩阵进行全基因组预测的准确性。G3(贝塞斯达)。2015; 5:615-27。
- 52.
卞颖,荷兰JB。多亲本玉米群体全基因组关联研究增强基因组预测。遗传(Edinb)。2017; 118:585 - 93。
- 53.
水稻B, Lipka AE。玉米和高粱中包含全基因组峰值关联研究信号的RR-BLUP基因组选择模型的评估。植物基因组。2019;12:180052。
- 54.
李旭,李勇,吴旭,白宁,宋勇,张东,等。我国重要玉米自交系籽粒相关性状的特征分析。植物遗传资源学报。2016;17:1-5。
- 55.
李应祥,李超,刘旭,陆峰,罗梅,等。利用极其庞大的多遗传背景群体鉴定与玉米开花时间相关的遗传变异。植物j . 2016; 86:391 - 402。
- 56.
穆雷MG,汤普森WF。快速分离高分子量植物DNA。核酸Res. 1980; 8:4321-5。
- 57.
等。大玉米(玉米l .)SNP基因分型:开发和种质基因分型,并与B73参考基因组进行遗传作图。《公共科学图书馆•综合》。2011;6:e28334。
- 58.
吴旭,李艳,石艳,宋艳,王涛,黄艳,等。利用高通量SNP基因分型研究玉米优质种质的优良遗传特性。Theor Appl Genet. 2014; 127:621-31。
- 59.
张志,kon DE, Casstevens TM, Ramdoss Y, Buckler ES。TASSEL:用于不同样本中复杂性状关联制图的软件。生物信息学。2007;23:2633-5。
- 60.
易锋,顾伟,陈静,宋宁,高旭,张旭,等。早期玉米种子发育的高时间分辨率转录组景观。植物细胞。2019;31:974 - 92。
- 61.
王志强,王志强,王志强,等。PLINK:全基因组关联和基于群体的连锁分析的工具集。Am J Hum Genet. 2007; 81:559-75。
- 62.
利用R包rrBLUP进行基因组选择的Endelman J. Ridge回归和其他核。植物基因组。2011;4:250-5。
致谢
不适用。
资金
在国家自然科学基金(91735306,31801373)的支持下,我们得以进行关联图谱的基因分型。得益于国家科技部(2016YFD0100303, 2016YFD0100103)和中国农业科学院创新计划,我们能够进行关联图谱面板的表型鉴定。这些资助机构与这份手稿的出版没有任何关系。
作者信息
从属关系
贡献
Y. A.和L. C.进行了GWAS和WGP,并起草了手稿;x。L.和C. L.构思了这项研究,并帮助讨论了结果。Y. S.和D. Z.领导本次研究的策划。T. W.和Y. L.设计了研究并编辑了手稿。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参加
不适用。
同意出版
不适用。
利益争夺
作者宣称,这项研究是在没有任何商业或财务关系的情况下进行的,这些关系可以被解释为潜在的利益冲突。
额外的信息
出版商的注意
Springer自然仍然是关于发表地图和机构附属机构的司法管辖权索赔中立。
补充信息
附加文件1:图S1。
常见qtn与不同的模型和不同的环境共同检测。a,不同方法共检测的常见QTNs。x轴表示不同的环境。y轴表示仅一种方法和至少两种、三种、四种、五种、六种或七种方法检测到的显著qtn的相应数量。b,在不同位置共同检测的共同QTNs。
附加文件2:图S2。
在我们的协会面板中LD随物理距离衰减。
附加文件3:图S3。
自交系KRN的全基因组预测。不同颜色的柱状图表示使用不同模型识别的tagsnp时KRN的预测精度。P.-值的估计基于双尾学生的t检验。***:P.值< 0.0001;NS:P.值> 0.05。
附加文件4:表S1
.在我们的协会面板的材料信息的列表。
附加文件5:表S2。
关联小组中各亚组KRN的描述性统计。
附加文件6:表S3。
用传销法确定的KRN的显著qtn。
附加文件7:表S4。
6种多位点方法鉴定的KRN的显著qtn。
附加文件8:表S5
.常用的QTNs采用不同的检测方法。
附加文件9:表S6
.共同的QTNs在不同的位置共同检测。
附加文件10:表S7
.候选基因在不同玉米组织中的表达。
附加文件11:表S8。
玉米穗突变相关基因。
附加文件12:表S9
.对随机选择的不同数量的标签snp(从5到27000)的KRN预测精度。
附加文件13:表S10
.KRN预测不同培训人口尺寸的准确性。
附加文件14:表S11
.top标记snp与随机标记snp预测准确率比较。
附加文件15:表S12
.利用代表不同方法鉴定的显著qtn的标记snp对自交系KRN的预测精度。
附加文件16:表S13
.比较我们的GWAS结果与以往研究中检测到的QTNs。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
安永,陈丽,李永祥。et al。基因组 - 宽协会研究与全基因组预测揭示了玉米Krn的遗传建筑。BMC植物杂志20.490(2020)。https://doi.org/10.1186/s12870-020-02676-x
收到了:
公认:
发表:
关键字
- 玉米
- 内核行号
- 全基因组关联研究
- 定量特征核苷酸
- 全基因组预测