摘要
背景
花生是全球最重要的油田种类之一。NAC转录因子(TF)基因通过激活或压制靶基因表达,在盐和干旱应激应答中起重要作用。然而,小关于花生中的NAC基因很少。
结果
我们对来自二倍体野生花生品种的NAC基因进行了全基因组鉴定落花生duranensis和落花生ipaensis,包括分析染色体位置,基因结构,保守的主题,表达模式和CIS.- 启动子区域内的元素。总共81和79种NAC基因被鉴定出来A. Duranensis.和答:ipaensis基因组。花生NACs及其系统发育分析拟南芥水稻对应物将这些蛋白质分类为18个不同的亚组。鉴定了五十一体原因对,并且发现46种异质过程在两者的染色体上都是高度同义的A. Duranensis.和答:ipaensis.基于RNA测序(RNA-seq)的分析表明,盐胁迫和干旱胁迫下,43个NAC基因表达上调或下调。其中17个基因在栽培花生中的表达(arachis hypogaea.)在两次压力下都在或下调。此外,基于定量逆转录PCR(RT-QPCR)的分析表明,大多数随机选择的NAC基因的表达往往与对比RNA-SEQ结果一致。
结论
我们的研究结果有助于花生NAC基因的功能鉴定,本研究发现的参与盐胁迫和干旱胁迫反应的基因可能是花生改良的潜在基因。
背景
种植花生(arachis hypogaea.)是世界范围内一种重要的经济油料作物,为人类营养提供植物油和蛋白质[1].在花生植株的生长过程中,产量受到多种环境因素的不利影响,如盐胁迫和干旱胁迫,使植株无法充分发挥其遗传潜能[2]. 筛选抗逆品种是实现作物产量目标的重要保证[3.].抗性基因的鉴定和利用是新品种生产的基础。转录因子(Transcription factors, TFs)通过与特定的基因结合来激活或抑制基因的表达CIS.- 靶官能基因的启动子内的元素,调节许多生物过程[4.那5.].NAC[无顶端分生组织(no apical meristem, NAM)]是最大的植物特异性TF家族之一,拟南芥已显示转录激活因子(ATAF1 / 2)和杯形子叶(CUC2)]蛋白质调节几种生物方法,包括对盐和干旱胁迫的反应[6.那7.那8.].值得注意的是,NAC TFs被认为对植物适应陆地非常重要[9.].NAC蛋白通常在n端有一个保守的NAM结构域,在c端有一个高度可变的结构域,后者与特定的生物学功能有关。NAC家族基因已经在包括裸子植物和胚胎植物在内的多种植物中得到了广泛的研究[10.那11.那12.那13.那14.那15.那16.那17.那18.那19.].然而,直到最近,对花生NAC家族基因的综合分析及其对盐和干旱胁迫的反应模式受到限制。
提高证据表明NAC蛋白参与植物生物和非生物反应。例如,杨树NAC13基因在盐应激反应中起着至关重要的作用[20.].麦子的过度表达n(Tanacl-D1)增强对镰刀菌头的抗性枯萎病[21.],TaNAC30负调节小麦对条纹锈的抵抗力[22.], 和TaNAC29可以通过增强抗氧化系统提供盐胁迫耐受性[23.].表达的TsNAC1从盐土植物Thellungiella halophila被证明可以提高非生物胁迫抗性,特别是耐盐性[24.].SLNAC35从…起茄属植物lycopersicum可以促进盐和干旱胁迫下的根生长和发育[25.),和米饭Onac033.由干旱诱导,可以对转基因植物中的盐和干旱胁迫提供强烈的抗性[26.].在花生中,已知NAC TFS参与反应对非生物胁迫的反应。例如,Ahnac2.和Ahnac3.可以改善转基因中的盐和耐旱性拟南芥和烟草[27.那28.], 和Ahnac4.提高转基因烟草的耐旱性[29.].此外,过度表达的MuNAC4从骑马图中显示转基因对转基因花生的增强的抗旱耐受性[30.].
异源四倍体的基因组答:hypogaea(AABB)及其两个野生二倍体祖先落花生duranensis(aa)和落花生ipaensis(BB)最近测序[1那31那32那33那34那35].两个二倍体物种的A和B基因组类似于培养花生的A和B次基因组,可用于鉴定候选性抗性基因[32那35].基因组信息的可用性为全基因组分析NAC基因和探索涉及花生生物和非生物反应的潜在基因提供了机会。随着RNA测序(RNA-seq)成本的降低,转录组测序已成为一种强大的高通量敏感的差异表达基因分析技术。一些花生RNA-seq数据集包含了不同组织或对不同治疗的反应的信息已经发表[36那37那38那39].例如,从22种不同的组织和二倍体花生物种的发育阶段产生的RNA-SEQ数据A. Duranensis.和答:ipaensis使花生NAC同源表达谱分析变得方便[36].还分析了鉴于盐和干旱胁迫的差异基因表达,这有助于鉴定盐和干旱反应中涉及的NAC基因[37那39].
在本文中,我们介绍了来自野生花生基因组的全基因组鉴定和表征NAC基因的结果,以及栽培花生盐和干旱胁迫的盐和干旱胁迫。我们分析了它们的系统发育关系,结构特征,染色体地点和基因局部基因对。我们还确定了不同组织中的表达特征,并在RNA-SEQ数据的基础上响应盐和干旱胁迫[36那37那39].通过RT-qPCR (quantitative reverse transcription PCR, RT-qPCR)对17个参与盐胁迫和干旱胁迫的基因进行了鉴定。本研究旨在为进一步分析花生中NAC蛋白的功能提供理论依据,并探索NAC基因在花生对盐和/或干旱胁迫响应中的同源性。
结果
NAC蛋白的鉴定A. Duranensis.和答:ipaensis
NAC基因共81个和79个1,其他文件1和2)从二倍体中鉴定出来A. Duranensis.(〜1.25 GB)和答:ipaensis(~ 分别为1.56 Gb),低于拟南芥(105)[40]及米(141)[41].然而,164个NAC蛋白(附加文件3.4)在培养的同种异体情况下鉴定出来答:hypogaea(~ 2.54 Gb)。这个数字接近于来自A. Duranensis.和答:ipaensis.NAC基因的密度A. Duranensis.(0.07 / mb)大于(0.05 / mb)答:ipaensis.NAC基因的密度答:hypogaea是0.06/Mb,这大约是A. Duranensis.和答:ipaensis.
由于缺乏NAC基因的指定标准注释arachis.,我们将这些基因命名为这些基因AdNAC1-AdNAC81和Ainac.1-AiNAC79.确定的NAC基因A.duranensis和A.IPAensis.编码的蛋白质长度为95-681个氨基酸残基,平均345个氨基酸,分子量为11 kDa至77.4 kDa公司。预测蛋白的等电点在4.57~10.25之间。NAC基因的详细信息A.duranensis和答:ipaensis在表中提供1,包括基因位置和推定拟南芥直系学家。
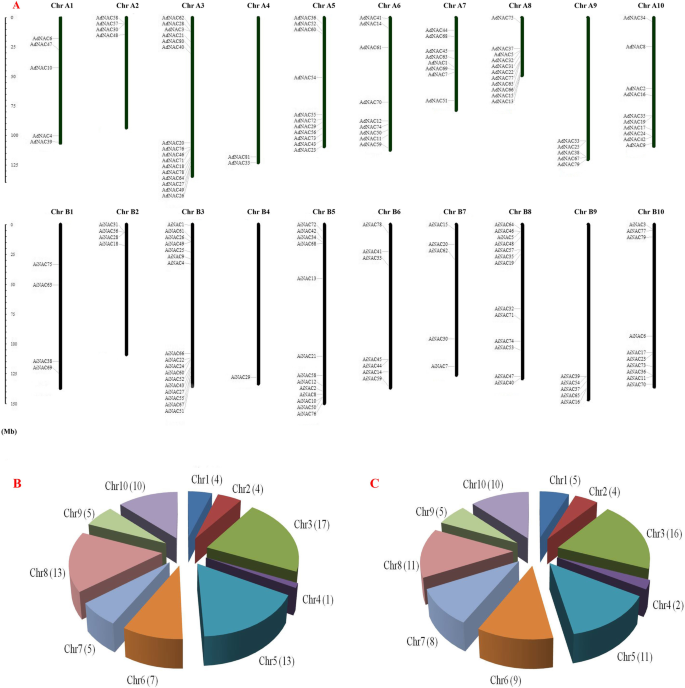
如图1所示。1,AdNAC和Ainac.基因在10个染色体上被非随机分布A. Duranensis.(基因组)和答:ipaensis基因组(B)。在这些物种中,A3染色体包含的NAC基因最多(16个),A4染色体包含的NAC基因最少(2)。1b)。在答:ipaensis,将17个基因分布在B3染色体上,而在染色体B4上只发现一个NAC基因(图。1C)。
NAC同源体位于同位基因座A. Duranensis.和答:ipaensis基因组
根据系统发育关系,检测到51个同源基因对AdNAC和Ainac.基因(图。2、表2)并通过染色体定位和基因结构进一步证实。在这些同源基因对中,有46对位于同线基因座上A. Duranensis.和答:ipaensis染色体(图。1a) 是的。但是9号的位置AdNAC基因与其同源基因的位置不对应答:ipaensis.例如,adnac7.位于A7染色体上,而其直译a . ipaensis AiNAC53,位于B8染色体上。这一发现表明二倍体花生基因组中的大型染色体重排已经发生。此外,具有低身份的基因对可能来自不同的剪接图案或源自释放的不完全基因组草稿的过早止血密码子[1].
系统发育分析,基因结构和保守的主题arachis.NAC基因
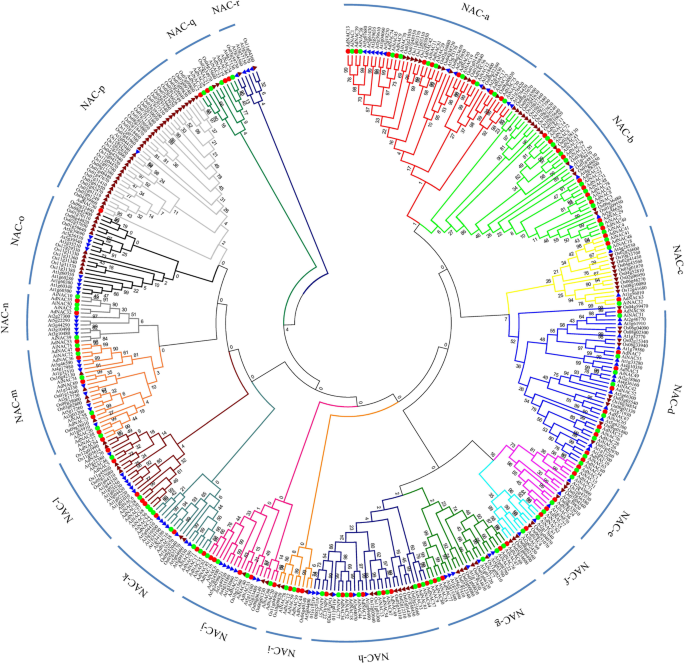
探讨两个野生动物的nac之间的关系arachis.并预测其潜在的功能,全长NAC蛋白A. Duranensis.(附加文件5.),答:ipaensis(附加文件5.),拟南芥(双子叶植物)(附加文件6.)和水稻(单子叶)(附加文件7.)进行多序列比对。系统进化树将野生花生nac分为18个亚群(NAC-a ~ NAC-r)拟南芥和水稻同源物(图。2).一般来说,拟南芥,水稻和花生NAC蛋白质在所有亚组中均匀分布。但是,仅包含NAC-O和NAC-R子组拟南芥和米饭NAC和没有花生NACS。值得注意的是,NAC-P亚家族包括36种水稻NAC,但只有1个AdNAC和1拟南芥NAC,而NAC-n亚组中未发现水稻NAC。另一种基于保守的NAM域的系统发育树显示在附加文件中8..
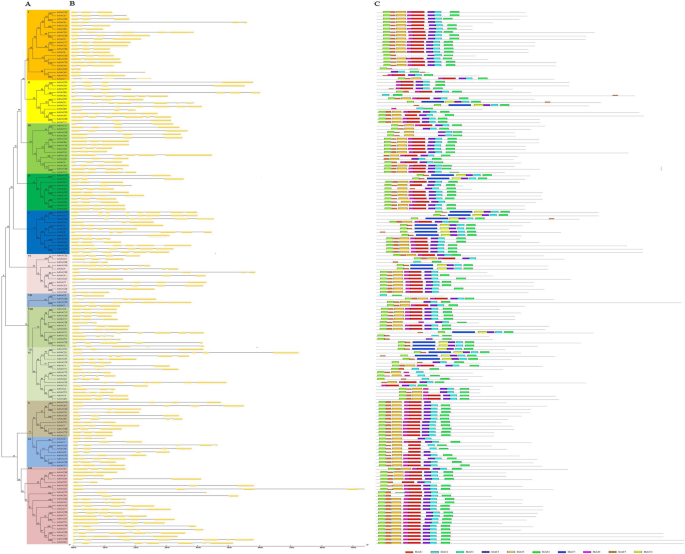
为了研究花生NAC基因的结构多样性,我们分析了花生NAC基因的外显子/内含子结构及其系统发育相似性(图1)。3.).所有的NAC基因来自A.duranensis和答:ipaensis分为十二个亚属(图)3.a).通常,同源基因来自A.duranensis和答:ipaensis例如,共用类似的外显子/内含子结构,包括内含子数和外显子长度,AdNAC80和AiNAC9在亚家族里,adnac59.和AiNAC59在亚家族III,而AdNAC81和Ainac29.在亚家族IV(附加文件9.).基因结构分析表明,花生NAC基因内的内含子分布是多样的,从1到9变化(图。3.b).一般来说,大多数ns含有2-3内含子;例如,77个基因含有2个内含子,43个基因包含3个内含子。
NAC基因的基因结构和基因组成A. Duranensis.和答:ipaensis.一种来自两个野生花生的NAC蛋白通过MEGA 6.0使用NJ方法分为12个系统发育子组,其中NJ方法具有1000个引导复制,指定为I到不同颜色背景中的XII。B.使用在线GSDS工具分析了花生NAC基因的基因结构。外显子和内含子分别由黄色盒子和黑线表示。底部的尺度代表外显子和内含子的尺寸。CMEME探讨了花生NAC蛋白内保守基序的分布。每个图案都是通过彩色框中的数字来区分。黑线显示了非保守的序列。每个图案的详细信息列于其他文件中10.
为了进一步确定NAC基因的多样化,预测推定的主题,以及内部的十个保守的主题arachis.NAC蛋白分析(附加文件10.). 正如预期的那样,密切相关的成员之间的基序组成是共同的。例如,亚家族XII中的大多数NAC蛋白含有8个基序。值得注意的是,大多数预测的基序位于NAC结构域的N-末端区域,这表明N-末端区域对NAC基因的功能至关重要(图。3.C)。
CIS.-基因启动子区的作用元件arachis.NAC基因
NAC基因在对众多应力的反应中起重要作用。推定CIS.- 在起始密码子(ATG)上游的2.5 kB序列内响应生物或非生物应力的反应的元素(附加文件11.)分析了。如其他文件所示12.那14.已知的与压力相关的CIS.确定这些NAC基因的启动子内的元素。数字的数量CIS.- 传递因子范围从0到10,有10种不同类型的CIS.的启动子区域内的-作用元件AdNAC34那AdNAC30,AiNAC30.只有4个基因的启动子(adnac7.那AdNAC15那adnac44,Ainac15.)含有富含TC的主题,涉及防御和压力响应[42].在160名启动子的中,133篇的ARES拷贝,这对于厌氧诱导至关重要[43].CGTCA基序参与茉莉酸甲酯(MeJA)激素介导的应激反应[44],存在于93个基因范围内。在这些2.5kb启动子区域中也发现了与非生物和生物应激反应相关的其他与非生物和生物应激反应相关的其他元素,例如TGA,W1,HSE和LTR元素。这些结果表明NAC基因响应于生物和非生物胁迫而转录调节。
NAC基因在不同组织中的表达谱A. Duranensis.和答:ipaensis
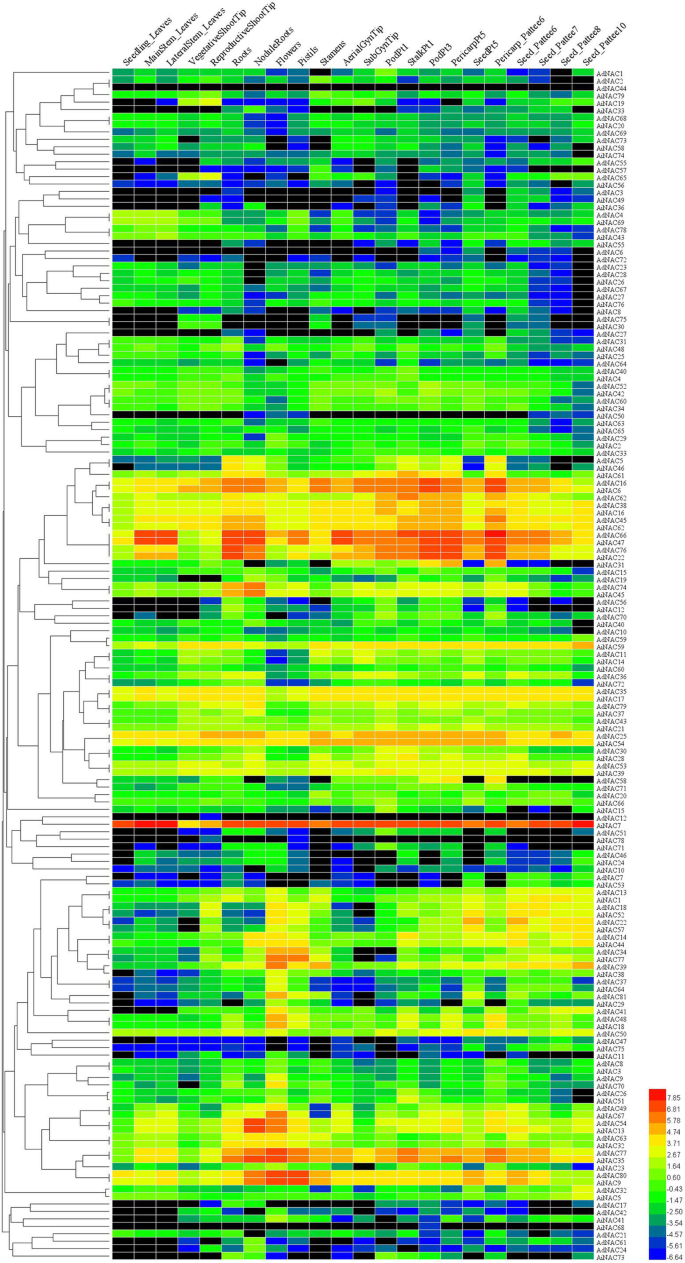
为了研究NAC基因的组织特异性表达谱,我们利用了Clevenger等人的转录组数据[36].检查的22个组织包括几乎所有组织和发育阶段。如图1所示。4.,没有发现adnac44在22种组织中表达。在22个组织中有23个NAC基因高水平表达。在这23个基因中,AiNAC7在所有22种组织中表现出相对高的表达水平,而其同源物AdNAC12仅在生殖茎尖组织中表达。例如,具有相同表达模式的基因,AdNAC16和AiNAC6,分为同一组(组V,图。3.).此外,一些NAC基因表现出组织特异性或优先表达模式。例如,adnac58没有在种子,雌蕊或雄蕊中表达。该组织特异性表达数据分析最终可能有助于确定NAC基因的调节功能的位置。
22种不同组织中NAC基因组织特异性表达模式及两种野生花生的发育。来自Clevenger等人的Illumina RNA-SEQ数据。[36[重新分析,平均FPKM值是LOG2转化的,使用HEMI获得热图。表达强度以不同的颜色显示(红色,高表达;绿色,低表达;黑色,没有表达)。顶部的棒代表22种不同的组织和发育阶段;NAC基因来自A. Duranensis.和答:ipaensis显示在右侧
采矿NAC基因参与盐和干旱胁迫的反应
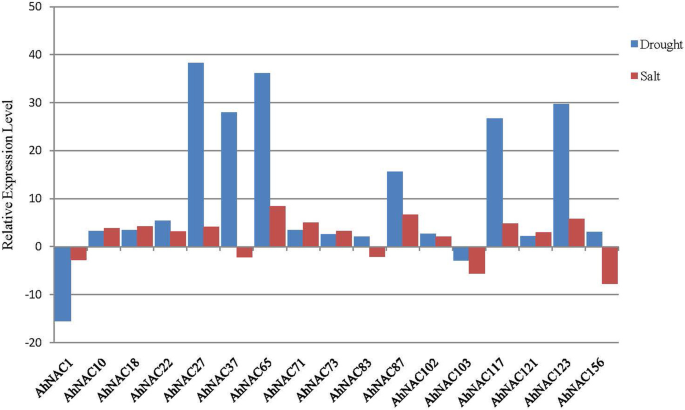
许多NAC基因被认为是非生物胁迫应答基因。为了探索盐和/或干旱胁迫应答中涉及的NAC基因,我们分析了已发表的盐胁迫下栽培花生转录组测序结果[39]和干旱[37)治疗。盐处理下,28个基因表达量上调2倍,15个基因表达量下调2倍以上。8个基因表达量显著上调超过5倍,最高表达量达到17倍,6个基因表达量下调超过5倍(图)。5.,附加文件13.).在干旱处理中,30个基因的表达上调超过2倍,将9个基因的表达上调大于10倍,最大的表达达到38倍。13基因的表达下调超过2倍,最大的表达达到15倍(图。5.,附加文件14.).17个基因的表达被发现对盐和干旱胁迫都有响应。四个基因(Ahnac1.那AhNAC37型那Ahnac83和AhNAC156)对盐胁迫和干旱胁迫的反应相反(图5)。5.).培养的这些NAC基因的信息答:hypogaea在附加文件中列出3.. 这些观察结果表明,一些NAC蛋白可能在多种应激反应中发挥作用。
NAC基因的RT-QPCR在盐和干旱胁迫下栽培花生
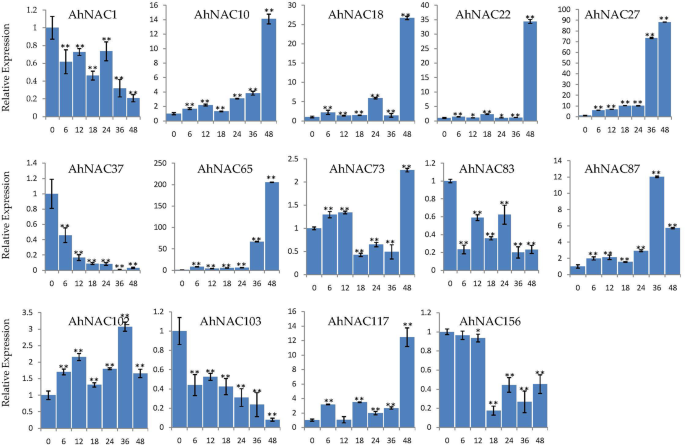
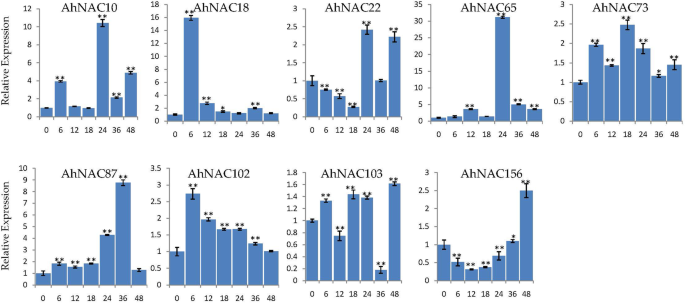
为了确定哪些基因对胁迫有响应,从而进一步提高花生的抗逆性,我们对花生根系进行了RT-qPCR表达分析。从17个涉及盐胁迫和干旱胁迫的NAC基因中随机选取了几个基因。盐胁迫(51.33 mM)处理下,根系中检测到的大部分nac的表达趋势(除nac的表达趋势AhNAC73型)与RNA-SEQ结果相同。例如,表达Ahnac1.那AhNAC37型那ahnac103,AhNAC156在所有检测到的时间点都在盐胁迫下下调,而表达水平AhNAC10型那AhNAC18型那AhNAC22型那AhNAC27型那AhNAC65型那ahnac87那ahnac102,Ahnac117被调节。值得注意的是AhNAC10型那AhNAC18型那AhNAC22型那AhNAC27型那AhNAC65型,Ahnac117在48岁时达到顶峰 盐胁迫处理后h,和对照组表达增加AhNAC65型达到了200多倍(图。6.). 在20%PEG6000处理下,表达水平AhNAC10型那AhNAC18型那AhNAC65型那AhNAC73型那ahnac87,ahnac102治疗后各时间点均增加,且表达水平AhNAC65型在处理24小时后增加近30倍(图。7.).这些结果与RNA-seq结果一致(图。5.). 总的来说,这些结果表明这些基因对盐和干旱处理的反应有可能改善花生。
讨论
表征arachis.NAC基因
NAC基因是最大的植物TF家族之一的成员,在许多应激反应中起着关键作用[4.那5.].NAC基因家族已经从一些植物基因组中得到了特征[10.那11.那12.那13.那14.那15.那16.那17.那18.那19.那40那41].然而,对NAC基因知之甚少arachis.物种。栽培花生答:hypogaea由两个二倍体野生花生杂交而成。野生花生A和B基因组A. Duranensis.(aa)和答:ipaensis(BB)与栽培花生(AABB)的A和B次基因组高相同[32].野生花生二倍体只包含一组基因组(AA或BB),比异源四倍体栽培花生(含A和B亚基因组)更容易克隆基因。利用现有的22种不同组织类型的野生花生的RNA-seq数据A.duranensis和A.IPAensis.便于基因表达谱分析[36].因此,在这项研究中,我们对来自野生花生的NAC TFS进行了基因组分析,并探讨了培养花生盐和干旱胁迫的原始基因潜在功能。可以从本研究中从野生花生的正交基因推导出来自栽培花生的NAC基因的信息(例如,染色体位置,基因结构,组织表达谱)。
总共,81,79和164个NAC TFS被野生花生物种鉴定出来A.duranensis那答:ipaensis和栽培花生答:hypogaea,分别。每个同源基因中都发现了两个或两个以上的花生NAC基因拟南芥.有关arachis.NAC基因家族,包括模型名称,位置,核苷酸长度,分子量和理论PI,以及拟南芥表中列出了直系同源物1和附加文件3..已有研究表明,四倍体花生中生物抗逆性基因核苷酸结合位点(NBS)结构域的数量小于它们的总和A. Duranensis.和答:ipaensis并导致栽培花生部分抗性能力丧失[32].然而,在我们的研究中,nac的数量(164)A.HypoGaea.几乎是野生动物的总和吗A. Duranensis.(81)和答:ipaensis(79)。这种扩展可能来自多种基因复制事件,包括全基因组重复arachis.谱系后跟多个分段和串联复制事件[27.那32].这些结果与来自栽培棉的NAC相同Gossypium Barbadense.和两个二倍体棉种类,雷氏棉和木本棉[45].以往的研究表明,NAC基因的参与在转录调控中起主要作用[45].因此,我们推测nac可能是通过调节抗胁迫相关基因或蛋白来发挥功能,而不是像“开关”一样发挥功能。栽培花生中NAC基因的数量(164个)高于其他植物(105个)拟南芥[40,大米141元[41]和101在大豆[46),大约是后者的1.56倍拟南芥,并发现了类似的结果杨树[10.]. 大豆NAC基因密度的研究A.duranensis那答:ipaensis和A.HypoGaea.(0.07 / MB,0.05 / MB,0.06 / MB)低于拟南芥(0.87/Mb)及米(0.37/Mb) [11.].这可能归因于arachis.大型基因组大小,这表明NAC家族的基因组大小和数量并不总是相关的。这些NAC基因在每个NAC基因上不均匀分布arachis.染色体(图。1).各染色体上NAC基因数为1 ~ 17,染色体长度与NAC基因数无正相关关系。一些NAC基因,如adnac58那adnac57.和AdNAC30,倾向于位于染色体上的簇中,因此这些基因可能会合作地运作[47].
组织特异性表达谱是有用的,因为它确定了与定义单个组织的确切性质有关的基因[48]. 在这项研究中,我们利用已发表的22种组织类型的RNA-seq数据来检测基因的特异表达模式arachis.NAC基因(36].普遍地表达了二十三种NAC基因,其可以用作调节大量基因的平台,随后被特定调节剂微调的细小。值得注意的是,我们发现了adnac58在种子、雌蕊和雄蕊中均未表达,表明其启动子可用于非种子基因工程。
系统发育分析和表达谱分析arachis.盐和干旱胁迫下的NAC基因
进行系统发育分析arachis.NAC与单子叶(米)和Dicot(拟南芥)模型植物物种,以研究进化关系和预测干旱或盐反应基因。在本研究中,这些nac被分为18个亚组,这与之前的分析结果基本一致[10.那40那41].nap亚家族中有36个水稻nS,但只有1AdNAC和1拟南芥南汽(无花果。2),这表明他们可能已经在米饭中获得或迷失拟南芥和arachis.当他们与共同的祖先分离时。与此相反,NAC-n亚家族中没有水稻NAC基因。2),暗示在单子叶 - Dicot发散后发生这种亚组的多样化和扩张。这种现象也在萝卜中发现,杨树和其他物种[10.那11.].
如果AdNAC和Ainac.在系统发育树中基因成对聚类,这些基因对被认为是同源基因[49那50].本研究根据花生的系统发育关系,从两种野生花生中鉴定出51个同源基因AdNAC和Ainac.基因(图。2、表2),其占整个家庭的57%以上,序列标识范围从61到99%(表2),四十六个基因位于同步基因座,并在上面表现出高的共线性A. Duranensis.和答:ipaensis染色体(表格2, 图。1).一些假定的同源基因对表现出低编码DNA序列(CDS)或低蛋白质身份,这可能是由于基因组测序错误(例如,adnac55.和它的直系同源Ainac10).两种野生花生的一些NAC基因均未位于相应的染色体区域,提示二倍体基因组发生了较大的染色体重排。同源基因通常表现出相似的特征和表达模式[49那51].由两个野生种衍生而来的栽培种的同源NAC基因功能可能是冗余的。例如,adnac54和Ainac13.从亚家族VIII有3个外显子和共享相同的保守基序。两者在根瘤和花中均有高表达,但在其他器官中表达量相对较低,与相应的结果相似拟南芥直接同源NAC2在根和鲜花中表达,针对调节盐应激反应和侧根发育[52].此外,anac2.也可以由脱落酸(ABA),1-氨基环丙烷-1-羧酸(ACC)和1-萘乙酸(NAA)诱导[52].它们在培养的花生中的相应外置基因可以一起起作用。来自不同植物物种的正交基因显示出落入一个亚组和共同的类似功能的趋势。许多NAC基因已经在功能性上表征拟南芥和他们的直译的基因arachis.在本研究中确定了(表1).结合系统发育结果,可以根据花生NAC基因的功能来预测其功能拟南芥水稻同源物,也有可能用于进一步的功能研究。例如,adnac77.那AiNAC9,AiNAC35和他们一起拟南芥直向同源基因,ANAC19(At1g52890)基因聚集在同一个NAC-g亚家族(图。2).的表达ANAC19是由干旱、高盐和脱落酸(ABA)诱导的。在同一亚族中,表示Arabidopsis Anac55(At3g15500)和anac72.(At4g27410)也受到干旱和高盐度的诱导[8.].因此,我们推测了这一点adnac77.那AiNAC9,AiNAC35是干旱和高盐反应基因,调节花生在不利生长条件下的生存。毫不奇怪,ahnac87的同源基因adnac77.和AiNAC35通过RNA-seq分析,在盐和干旱处理下均诱导了花生(图1)。5.)和基于RT-QPCR的结果证实,在栽培花生中,表达ahnac87在盐和干旱胁迫处理下上调(图。6.和7.).此外,Arabidopsis Anac2.(At1g01720,也称为ATAF1),它是同源的AdNAC22,是由干旱胁迫引起的[53].他们的正端表达AhNAC37型,根据比较RNA-seq分析,在干旱胁迫下上调约27.5倍(图。5.).这些调查结果强烈支持职能arachis.从这些同源基因中可以推断出NAC基因拟南芥和米饭。
以前的报道提供了基于基于基于基因族的应力相关功能的系统发育分析的有力证据。脱水诱导的基因Ahnac3.(EU755022Ahnac117在我们的研究中,提供了超抗脱水和干旱胁迫的抗性[27.].在我们的研究中,表达Ahnac117根据比较RNA-seq数据,在盐处理下诱导(图。5.),并通过RT-QPCR确认(图。6.和7.).类似的结果也出现在Ahnac4.的同源物HM776131ahnac87在我们的研究中,与彻底主义adnac77.和AiNAC35) 和Ahnac2.(EU755023)[28.那29.].这两个基因相似于97.78%,受干旱和盐胁迫高度诱导,并赋予转基因植物的干旱和耐盐性。
方法
序列数据库搜索
从Peanutbase数据库中检索本研究中所有NAC基因的序列(www.peanutbase.org)使用NAM域(PF02365)作为搜索查询。我们使用NCBI数据库手动验证了推测的候选蛋白(https://www.ncbi.nlm.nih.gov/)确认NAM域的存在。通过简单的模块化结构研究工具(SMART;http://smart.embl-heidelberg.de/)域分析程序和Pfam (Protein family:http://pfam.xfam.org/)数据库确认搜索结果的可靠性。只保留含有这些域的序列。通过扩展蛋白质组学服务器上的蛋白质组学和序列分析工具预测每个蛋白质的MWS和PIS(http://web.expasy.org/compute_pi/).推定拟南芥通过BLASTP搜索鉴定了花生NAC的正交。
序列对准和系统发育分析
研究从花生和单点的NAC蛋白之间的系统发育关系拟南芥单子叶水稻拟南芥NAC蛋白序列从拟南芥信息资源(TAIR;https://www.arabidopsis.org/)和水稻NAC蛋白序列从水稻基因组注释项目(RGAP;http://rice.plantbiology.msu.edu/).全长氨基酸序列通过CLUSTALW程序进行多次对准。使用Mega 6.0软件的邻近加入(NJ)方法构建了无根植物的系统发育树,并使用1000次迭代进行引导测试。
染色体位置、基因结构及保守基序分析
从Peanutbase网站中检索NAC基因的染色体位置信息(www.peanutbase.org).这些基因通过MapInspect程序映射到染色体上(http://mapinspect.software.informer.com).花生NAC基因的mRNA和gDNA信息均来自于花生数据库(www.peanutbase.org).我们使用了GSDS(http://gsds.cbi.pku.edu.cn.)在线计划探索NAC基因的外显子/内含子组织。meme(http://meme-suit.org)程序对NAC蛋白序列进行基序分析。所有蛋白序列的结构域均通过Pfam 31.0 (http://pfam.xfam.org/),基于隐马尔可夫模型。
预测CIS.-启动器内的作用元素
启动子序列(长度为2.5 kB)从PeanutBase网站下载(www.peanutbase.org) 为了CIS.表演元素分析。通过New PLACE (https://sogo.dna.affrc.go.jp/cgi-bin/sogo.cgi?lang=en&pj=640&action=page&page=newplace)[54].
基于RNA-SEQ在花生中NAC基因的表达分析
Clevenger等人的研究获得了22种不同组织类型和发育阶段的平均每千碱基每百万reads片段图谱(FPKM)值[36].每个NAC基因的FPKM值通过HemI进行log2转化,并以热图的形式显示[55].
为了研究NAC基因在盐和干旱胁迫下的表达模式,盐胁迫下各基因的平均FPKM值[37]和干旱[39]的处理是从我们以前的工作中获得的。这些表达变化2倍以上的NAC基因的平均FPKM值通过Excel软件进行比较,HemI转化log2并以热图的形式显示[55].
植物材料、生长条件和胁迫处理
本课研组培育的花生“华育9303”,除另有说明外,在恒温20℃,光照16小时,黑暗8小时的条件下生长。大约1个月后,用51.33 mM NaCl(盐处理)或20%聚乙二醇(PEG) 6000(干旱处理)处理植株。收集处理0、6、12、18、24、36和48 h后的根系,立即用液氮冷冻,保存在−80℃。
RNA提取和RT-QPCR基于分析
总RNA采用MiniBEST植物RNA提取试剂盒(Takara,中国大连)提取。利用PrimeScript RT-PCR试剂盒(Takara)合成第一链cdna,采用qPCR检测盐和干旱处理下AhNAC基因的表达水平。反应混合物为2 μL cDNA (10.3 ng/μL),正、反引物(400 nM)各2 μL, TB Green Premix Ex Taq II (Takara) 10 μL,加入无菌水,总体积为20 μL。在ABI 7500快速实时系统(Applied Biosystems, CA, USA)上进行扩增,方法如下:50℃,2 min;95°C保温2分钟;95°C 15 s和60°C 34 s循环40次。通过熔融曲线分析验证了反应的特异性。RT-qPCR中检测到的每个NAC基因的基因特异性引物均根据其他基因的差异设计,并列于附加文件中15..每种基因进行三种生物重复进行。使用基因转录水平使用ΔΔCT方法[56].执行学生的T检验以使用SPSS软件计算P值。当P <0.05时,我们认为NAC基因是差异表达基因的。归一化所选NAC基因的表达水平,施基因被用作内部对照[47].
结论
本文从系统发育、染色体定位、基因结构、保守基序、CIS.启动子区内的元素,两倍二倍体中Nac基因家族成员的表达分析arachis.进行了物种鉴定。这些结果为今后的研究提供了有益的基础。arachis.NAC基因。在基于比较的RNA-SEQ和RT-QPCR的分析的基础上,我们还确定了参与干旱和/或盐应激反应的NAC基因,这可能是适用于花生改善。
可用性数据和材料
本研究期间生成或分析的所有数据都包含在本发布的文章及其补充信息文件中。
缩写
- 南京:
-
NAM,ATAF1 / 2和CUC2
- 南:
-
没有顶端商品
- ATAF1/2:
-
拟南芥转录激活因子
- 铜2:
-
的杯状容器子叶
- TF:
-
转录因子
- RNA-SEQ:
-
RNA序列
- RT-QPCR:
-
实时定量聚合酶链反应
- Gb:
-
千兆碱基对
- m:
-
百万碱基对
- AA:
-
氨基酸
- kDa:
-
千达尔顿
- 惩罚:
-
茉莉酸甲酯
- 阿坝:
-
脱落酸
- ACC:
-
1-aminocyclopropane-1-carboxylic酸
- naa:
-
1-萘乙酸
- cd:
-
编码DNA序列
- 兆瓦:
-
分子量
- PI:
-
等电点
- RGAP公司:
-
水稻基因组注释项目
- TAIR:
-
拟南芥信息资源
- NJ:
-
Neighbor-joining
- FPKM:
-
每百万百万映射的成绩单每千碱基映射的碎片
- 挂钩:
-
聚乙二醇
参考
- 1。
Bertioli DJ,Cannon SB,Froenicke L,Huang G,Farmer Ad,Cannon Ek等。基因组序列落花生duranensis和落花生ipaensis,栽培花生的二倍体祖先。NAT Genet。2016; 48(4):438-46。
- 2。
VAHDATI K,LOTFI N.核桃干旱和盐度应力的植物中的非生物胁迫耐受性。在:Vahdati K,Leslie C,编辑。非生物胁迫植物应对与农业应用。里耶卡:Intech;2013. p。307-65。
- 3.
阿拉伯MM,Marrano A,Abdollahi-Arpanahi R,Leslie Ca,Cheng H,Neals DB,Vahdati K.结合表型,基因型和环境在波斯核桃中揭示水性利用效率的灭绝成分。J Exp Bot。2020; 71(3):1107-27。
- 4.
朱茹非生物应激信号和植物反应。细胞。2016; 167(2):313-24。
- 5.
曲丽娟,朱应祥。转录因子家族拟南芥:主要研究进展及有待进一步研究的问题。植物生物学杂志。2006;9(5):544-9。
- 6.
Nakashima K,Tran LS,Van Nd,Fujita M,Maruyama K,Todaka D,Ito Y,Hayashi N,Shinozaki K,Yamaguchi-Shinozaki K. NAC型转录因子的功能分析OsNAC6参与水稻非生物和生物胁迫应答基因的表达。51植物j . 2007;(4): 617 - 30。
- 7.
侯赛因rm,阿里米,冯x,李x。的本质n抗旱大豆栽培的基因家族(甘氨酸臂l .稳定)品种。中国生物医学工程学报。2017;17(1):55。
- 8.
关键词:抗氧化剂,抗氧化剂,抗氧化剂,分离,功能分析拟南芥胁迫诱导NAC转录因子与干旱应答CIS.-元素在脱水胁迫的早期响应1启动子。植物细胞。2004;16(9):2481 - 98。
- 9
徐B,Ohtani M,Yamaguchi M,Toyooka K,Wakazaki M,Sato M,Kubo M. NAC转录因子对植物适应土地的贡献。科学。2014; 343(6178):1505-8。
- 10。
8 .胡锐,齐刚,孔勇,孔丹,高强,周刚。大豆中NAC结构域转录因子基因家族的综合分析Populus Trichocarpa..中国生物医学工程学报。2010;10:15 15。
- 11.
Karanja Bk,Xu L,Wang Y,Muleke Em,Jabir Bm,谢Y,Zhu X,Cheng W,Liu L.在萝卜中非生物胁迫下的NAC转录因子基因的基因组宽的表征和表达分析(萝卜l .)。同行j . 2017; 5: e4172。
- 12.
关键词:木本植物,NAC,转录因子,全基因组分析,表达模式,低温胁迫prunusmume.基因。2018;9(10):494。
- 13。
Guérinc,roche j,allard v,ravel c,mouzeyar s,bouzidi mf。在面包小麦下的干旱和热应力下NAC家族的基因组分析,扩张和表达(T. Aestivum.L.)。公共科学图书馆一号。2019;14(3):e0213390。
- 14.
孙H,胡米,李杰,陈L,李米,张某,张X,杨奇。NAC转录因子的综合分析在棉花纤维开发和应力反应期间揭示其作用。BMC植物BIOL。2018; 18(1):150。
- 15.
刁W,斯奈德JC,王某,刘j,潘b,gu g,ge w,dawood mh。辣椒中NAC转录因子基因家族的基因组分析(甜椒L.):染色体定位、系统发育、结构、表达模式、CIS.- 启动子和互动网络中的元素。int j mol sci。2018; 19:1028。
- 16.
沉S,张Q,石y,Sun z,张q,侯S,吴r,姜l,zhao x,guo y。Nac结构域转录因子基因系列的基因组分析Theobroma可可.基因。2020;火灾。
- 17.
Pascual M,Belen C,Francisco M,Concepcion A. NAC转录因子家庭在海洋松树(松果体松树):涉及胁迫反应的两个基因的分子调控。中国生物医学工程学报,2015;
- 18.
查克拉波蒂R,罗伊S。不同胚状植物类群4个参考种NAC转录因子成员多样性及其系统发育意义的评价。生理分子生物学植物。2019;25:347–59.
- 19.
Pascual MB, Llebres MT, Craven-Bartle B, Canas RA, Canovas FM, Avila C。PpNAC1,苯丙氨酸生物合成和海洋松树利用的主要调节剂。植物Biotechnol J. 2017; 16(5):1094-104。
- 20.
张X,程Z,赵K,姚W,孙X,蒋T,周B。杨树的功能特性NAC13耐盐基因。植物科学。2019;281:1-8。
- 21.
Perochon A、Kahla A、VranićM、Jia J、Malla KB、Craze M、Wallington E、Doohan FM。小麦NAC与一种孤儿蛋白相互作用,增强了小麦对赤霉病的抗性。植物生物技术杂志。2019;17(10):1892–904.
- 22.
王乙、魏杰、宋恩、王恩、赵杰、康Z。一种新的小麦NAC转录因子,TaNAC30对小麦条锈病抗性有负调控作用。综合植物生物学杂志。2018;60(5):432–43.
- 23.
关键词:小麦NAC转录因子TaNAC29与对盐胁迫的反应有关。植物生理学杂志。2015;96:356-63。
- 24.
刘超,王斌,李忠,彭忠,张杰。TSNAC.是抗非生物胁迫和生长的关键转录因子。植物杂志。2018;176(1):742 - 56。
- 25.
王克,张某,马X,王Y,孔福,萌Q.一种应激相关的NAC转录因子(SLNAC35)在生物和非生物胁迫中起着积极的作用。杂志。2016;158(1):45 - 64。
- 26.
胡H,戴米,姚J,小B,Li X,张Q,熊L.过表达NAM,ATAF和CUC(NAC)转录因子提高水稻的抗旱和耐盐性。PROC NATL ACAD SCI。2006; 103(35):12987-92。
- 27.
刘X,Hong L,Li Xy,Yao Y,Hu B,Li L.改进转基因的干旱和耐盐性拟南芥过表达一个NAC转录因子arachis hypogaea..生物化学与生物技术。2011;75(3):443-50。
- 28.
刘旭,刘松,吴军,张斌,李旭,闫燕,李磊花生NAC3在烟草中,通过增加超氧化物清除来增强脱水和抗旱能力。植物生理学杂志。2013;70:354-9。
- 29。
关键词:花生,NAC基因,过表达Ahnac4.,增强烟草的耐旱性。植物生理学杂志。2017;64(4):525-35。
- 30.
Pandurangaiah M,Lokanadha Rg,Sudhakarbabu O,Nareshkumar A,Kiranmai K,Lokesh U,Thapa G,Sudhakar C.过表达骑马ramp(Macrotyloma uniflorumLam.Verdc)。NAC转录因子(MuNAC4)能增强花生的耐旱性。生物科技摩尔》。2014;56(8):758 - 69。
- 31.
陈X,Lu Q,刘H,张j,hong y,lan h等。栽培花生的测序,arachis hypogaea.,产生对基因组进化和石油改进的见解。莫尔植物。2019:1-15。
- 32.
庄W,陈H,杨米,王j,pandey mk,张c等。栽培花生的基因组提供了熟悉豆科核型,多倍体演化和作物驯化的洞察力。NAT Genet。2019; 51(5):865-76。
- 33。
陈晓,李浩,潘德明,杨青,王旭,等。花生a -基因组前体细胞基因组草案(落花生duranensis)提供了洞察地质果皮、石油生物合成和过敏原。自然科学学报。2016;113(24):6785-90。
- 34。
尹d,ji c,ma x,李h,张w,李思,刘f,赵k,李f,李克,宁l,他j,王y,zhao f,xie,zheng h,zhang x,张y,张j.同种异体一体化野生花生的基因组arachis monticola.:De Novo集会。傻瓜。2018; 7(6):GIY066。
- 35。
Bertioli DJ, Jenkins J, Clevenger J, Dudchenko O, Gao D, Seijo G等。分段异源四倍体花生基因组序列arachis hypogaea..51 Nat麝猫。2019;(5):877 - 84。
- 36。
Clevenger J,Chu Y,Scheffler B,Oziasakins Pa。AllotetroTaploid的发育转录组MAParachis hypogaea..前植物SCI。2016; 7:1446。
- 37。
赵霞,李超,万盛,张涛,闫超,单顺。转录组学分析及基因的发现arachis hypogaea.干旱压力。mol biol rep。2018; 45(2):119-31。
- 38.
关键词:花生,黄曲霉毒素,抗感,收获后种子,转录组分析黄曲霉. BMC植物生物学。2016;16:54.
- 39.
张H,赵XB,孙QX,燕CX,王继,元CL,李CJ,山SH,刘FZ。比较转录组分析显示分子防御机制arachis hypogaea.以应对盐胁迫。13.国际基因组学杂志;
- 40。
大田H,佐藤K, Doi K, Nagata T, Otomo Y, Murakami K, Matsubara K, Osato N, Kawai J, Carninci P, Hayashizaki Y, Suzuki K, Kojima K, Takahara Y,山本K, Kikuchi S.综合分析n家族的基因栽培稻和拟南芥.DNA研究》2003;10(6):239 - 47。
- 41.
方y,y j,谢k,谢w,xiong l。Nac转录因子家庭中Nac转录因子家族中组织特异性或应激反应基因的系统序列分析及鉴定。Mol Gen Genomics。2008; 280(6):547-63。
- 42.
Yan J,Wang B,Jiang Y,Cheng L,Wu T.gg Gmfnsii控制的大豆黄酮新陈代谢对非生物胁迫进行反应,并调节植物耐受性。植物细胞生理。2014; 55(1):74-86。
- 43.
Germain H, Lachance D, Pelletier G, Fossdal CG, Solheim H, Seguin a青云杉防御素1启动子保持在拟南芥,表示缓解植物植物和裸子植物之间的信令途径。J Exp Bot。2012; 63(2):785-95。
- 44.
Mark R,Olive JC,Walker Ks,Elizabeth SD,James P.玉米Anaerobic响应元件的功能性质ADH1基因。植物分子生物学。1990;15(4):593–604.
- 45.
关键词:NAC,转录因子,系统分析,转录因子Gossypium Barbadense.揭示他们的角色verticillium.枯萎。同行j . 2019; 7: e7995。
- 46.
Pinheiro GL, Marques CS, Costa总经理,Reis PA, Alves MS, Carvalho CM, Fietto LG, Fontes EP。大豆NAC转录因子的完整清单:序列保存和表达分析揭示了它们在胁迫反应中的独特作用。基因。2009;444(1 - 2):10-23。
- 47.
袁春林,孙庆祥,孔永忠。全基因组挖掘花生种子特异性候选基因用于启动子克隆。《公共科学图书馆•综合》。2019;14 (3):e0214025。
- 48.
关键词:大豆,生长素,转录因子,水分胁迫,转录因子DNA研究》2013;20(5):511 - 24。
- 49.
宋辉,王鹏飞,林军,赵传忠,毕彦平,王学军。全基因组鉴定和鉴定WRKY花生基因家族。植物科学与技术,2016;
- 50。
Altenhoff AM, Dessimoz C.推断orthology和谬误。中国生物医学工程学报,2012;
- 51.
宋,王pf,侯l,赵sz,赵cz,xia h,li pc,张y,卞xt,王xj。全局分析WRKY大豆脱水和盐胁迫的基因及其响应。植物科学,2016;7:9。
- 52.
何晓,穆荣,曹伟,张震,张杰,陈松。AtNAC2是乙烯和生长素信号通路下游的转录因子,参与盐胁迫反应和侧根发育。植物J。2005;44:903–16.
- 53.
刘勇、孙杰、吴勇。Arabidopsis Ataf1.提高了转基因水稻对盐胁迫和ABA的耐受性。植物学报。2016;129:955-62。
- 54.
Higo K, Ugawa Y, Iwamoto M, Korenaga t PlantCIS.-作用调节DNA元素(PLACE)数据库:1999。核酸研究1999;27(1):297–300.
- 55.
邓伟,王勇,刘卓,程辉,薛永和。一种绘制热图的工具。《公共科学图书馆•综合》。2014;9 (11):e111988。
- 56.
Rao X,Huang X,周Z,林X.改进了2-ΔΔct定量实时聚合酶链反应数据分析方法。Biostat Bioinforma生物科技公司。2013;3(3):71–85.
确认
我们感谢在公共数据库中获得的花生基因组和转录组数据的贡献者以及匿名审查员担任稿件的评论。
资金
本研究由中国天然科学基金(32001585,31601336),山东省农业工业技术研究体系(SDAIT-04-02)和农业创新项目(SDAIT-04-02)和农业创新项目(TS201712080)资助山东农业科学与技术(CXGC2016A01)。
作者信息
从属关系
贡献
QXS和SHS构思和设计了研究。CLY和CJL进行了生物信息分析。CLY进行了实验并起草了手稿。XDL和XBZ参与了分子分析。CJL,CXY,JW和QXS促成了稿件的修订。所有作者都读过了最后的手稿。
相应的作者
伦理宣言
伦理批准并同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
作者们宣称他们没有相互竞争的利益。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
额外的文件1。
两种野生花生NAC基因mRNA序列分析。
额外的文件2。
两种野生花生NAC基因的gDNA序列
额外的文件3。
n栽培花生TF基因家族成员的研究。
额外的文件4。
花生NAC基因的mRNA序列。
额外的文件5。
两个野生花生的NAC蛋白。
附加文件6。
拟南芥南汽的蛋白质。
附加文件7。
大米NAC蛋白质。
附加文件8。
NAC蛋白的系统发育树分析arachis.那拟南芥基于保守的NAM域的稻米。
附加文件9。
外显子内部结构之间的比较adnac59.那AdNAC80那AdNAC81以及它们的直系同源物AiNAC59那AiNAC9那Ainac29..
附加文件10。
NAC蛋白内保守基序的序列标志。
附加文件11。
来自两个野生花生的NAC基因2500磅培训促进剂区。
额外的文件12。
NAC基因启动子内存在的不同顺式作用元件的数量。
附加文件13。
基于比较RNA-seq数据参与盐反应的基因。y轴表示与未处理植株相比的褶皱变化。x轴显示盐处理下花生中表达上调和下调2倍以上的基因。
额外的文件14。
基于比较RNA-seq数据的干旱反应相关基因。Y轴代表与未处理植物的水平相比的褶皱变化。X轴显示干旱处理下栽培花生中表达上调或下调超过2倍的基因。
额外的文件15。
本研究中使用的引物。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
袁超,李超,陆晓明。等等。NAC转录因子家族的综合基因组特征及其对花生盐和干旱胁迫的反应。BMC植物杂志20.454(2020)。https://doi.org/10.1186/s12870-020-02678-9
收到了:
公认:
发表:
关键词
- 花生
- NAC基因家族
- 基因组的特征
- RNA-seq
- RT-QPCR.
- 盐胁迫
- 干旱胁迫