摘要
背景
大豆食品的生产要求大豆种子具有特定的物理和化学特性。通过标记辅助选择(MAS)技术鉴定与种子重量、种子蛋白质和蔗糖浓度等增值性状相关的数量性状位点(QTL),可以加速高蛋白大豆品种在食品级市场上的竞争。本研究的目的是在两个高蛋白重组自交系(RIL)群体中鉴定和验证与这些增值性状相关的QTL。
结果
RIL群体来源于高蛋白品种‘AC X790P’(蛋白质含量49%,干重基础)和两个高产商业品种‘S18-R6’(蛋白质含量41%)和‘S23-T5’(蛋白质含量42%)。14个大效应QTL (R2> 10%)与种子蛋白浓度相关。其中7个QTL在两个群体中均被检测到,其中8个与种子蔗糖浓度或种子重量相关的QTL共定位。在两个居群中均未发现蛋白质相关QTL与种子产量相关。在7个蛋白质相关区域中鉴定出16个在蛋白质代谢中可能起作用的候选基因:qPro_Gm02-3, qPro_Gm04-4, qPro_Gm06-1, qPro_Gm06-3, qPro_Gm06-6, qPro_Gm13-4和qPro-Gm15-3。
结论
来自高蛋白亲本的RIL群体的使用为鉴定四个可能被来自不同亲本品种的群体的大效应QTL分离所掩盖的新QTL创造了机会。总的来说,我们已经确定了9个蛋白质QTL,这些蛋白质QTL在当前的研究中或在其他研究中都被检测到。这些QTL对大豆新品种的选育具有一定的参考价值。
背景
大豆(大豆(l)梅里尔]是植物性膳食蛋白质的主要来源。西方国家对豆腐和豆浆等全豆食品需求的增加,引起了研究人员、大豆种植者和大豆食品加工商的注意。大豆制品需要大豆种子具有特定的物理和化学特性,包括最佳的种子蛋白质浓度、种子蔗糖浓度和种子重量[1,2,3.,4,5,6,7],这些对商品大豆育种计划并不重要。由于食品加工者需要一致的种子成分来维持生产程序,开发环境稳定、具有最佳增值性状的高产大豆品种已成为一个重要的育种目标。
种子组成和产量组成性状受许多基因和环境因素的影响[8,9,10,11,12,13].种子蛋白质浓度与种子产量呈负相关,这阻碍了具有竞争力的高蛋白大豆品种的发展[9,14,15,16,17,18,19,20.,21,22,23].额外的增值特性,如高种子蔗糖浓度和高种子重量,也是大豆食品加工商感兴趣的。众所周知,蔗糖浓度会影响许多大豆食品的适口性和质地[24].然而,种子蛋白与蔗糖浓度呈显著的反比关系[25].这种关系对于豆腐等需要高浓度蛋白质和蔗糖才能达到最佳产量的大豆食品是有害的。5].鉴定和使用与提高种子蛋白质浓度和附加附加值性状相关的数量性状位点(QTL),可以通过将理想的等位基因积累到共同的遗传背景中,加速开发具有竞争力的高蛋白大豆品种,以适应北美食品级市场。
许多研究试图确定大豆种子蛋白质积累的遗传基础。SoyBase已经索引了248个与种子蛋白质浓度相关的双亲本QTL,这些QTL涵盖了超过35个独立研究的结果[37].这些QTL位于每条大豆染色体上,尽管染色体6、15、18和20特别受青睐[38].Qi等进行的QTL-meta分析[39]在多种遗传背景和生长环境中鉴定出51个共识QTL,这些QTL位于除16号染色体外的所有连锁群上。许多因素,如大置信区间,小加性效应,与其他理想性状的负相关,环境稳定性差,QTL-by-genetic背景相互作用效应,限制了这些QTL在标记辅助选择程序中的有效性[40,41,42,43,44].许多QTL也被确定为其他感兴趣的性状,包括在50多项独立研究中确定的318个种子重量相关QTL,以及在32项独立研究中确定的188个种子产量相关QTL [37].蔗糖浓度受到的关注相对较少,在4项独立研究中鉴定出37个蔗糖相关QTL [37].
RNA-seq数据的全球分析显示,Kunitz胰蛋白酶抑制剂1、凝集素家族蛋白、种子存储2S白蛋白超家族蛋白、bZIP同源物和myb样转录因子与种子蛋白质积累有关[39].在之前的研究中,这些转录本也与种子蛋白质积累有关[45,46,47].特定基因,如ABI3, ABI4而且LEC1也与种子蛋白质积累有关[48,49].
一种可能用于改善多基因性状的QTL检测方法是利用来自精英亲本[46]的分离群体。以往旨在检测蛋白质相关QTL的研究大多使用来自外来种质或期望性状表型差异较大的亲本品种的定位群体[50].利用来自精英系的群体可以增加在不同群体中发现被常见的大效应QTL所掩盖的新QTL的机会。这些QTL有利于大豆高蛋白新品种的开发。
本研究利用3个蛋白质含量高至中高的高产大豆品种杂交获得的两个重组自交系(RIL)群体,鉴定了与食品级大豆重要性状相关的QTL。研究了与种子蛋白质浓度相关的显著基因组区域与种子蔗糖浓度、种子重量和产量的关系。确定多种增值性状的基因组区域,有利于同时改良食品级大豆新品种的优良性状。为了更好地理解调节大豆种子储存蛋白质积累的潜在机制,还对这些区域进行了候选基因的筛选。
结果
蛋白质和其他增值食品级性状的表型分析
在2015年和2016年田间季节的多环境试验中,对RIL群体的种子重量、产量、蛋白质和蔗糖浓度进行了评估。1;补充表S1- s4).采用高通量近红外反射率(NIR)法测定种子蛋白质和蔗糖浓度,该方法是目前测定大豆种子成分性状的常用方法[51,52].虽然高效液相色谱法(HPLC)是一种更准确的测量种子蔗糖含量的方法,但以往的研究表明,近红外法也可以对大豆种子蔗糖浓度进行可靠和无偏倚的估计,这适用于区分不同蔗糖水平的基因型,也适用于QTL研究[52].在本研究中,两个群体亲本品种之间的种子蛋白质浓度有显著差异。在POPn_1中,' AC X790P '在五个测试环境中的平均蛋白质浓度为48.08%(±0.19%,标准误差),而' S18-R6 '的平均蛋白质浓度为40.93%(±0.19%)。在POPn_2。在五个测试环境中,‘AC X790P’的平均蛋白质浓度为48.24%(±0.21%),而‘S23-T5’的平均蛋白质浓度为42.60%(±0.21%)。
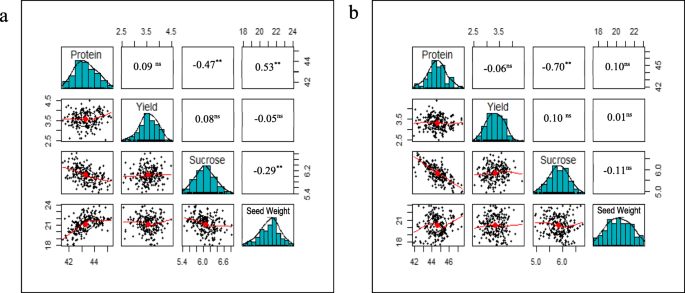
每个群体中RIL系之间蛋白质浓度的差异在个体和联合多环境中都是显著的(图2)。1;补充表S1).POPn_1种子蛋白质浓度变化范围为41.53 ~ 45.27%,平均蛋白质浓度为43.31%(±0.03%)。在POPn_2中,种子蛋白浓度变化范围为41.93 ~ 47.46%,平均蛋白浓度为44.60%(±0.03%)。1;补充表S1).在一些单独的环境中观察到了海侵分离,但在考虑多环境数据时没有观察到海侵分离(补充表S1).正态分布(图;2LSMEAN估计表明蛋白质浓度是由多个基因控制的。
亲本品种在种子产量、种子重量和种子蔗糖浓度方面也存在差异,并且在两个群体的多环境数据组合中也注意到相当大的差异。1).在POPn_1中,估计入口种子重量(每100粒种子克)从18.08 g到23.88 g不等,平均种子重量为21.18 g(±0.055 g)。种子产量也从2.55吨公顷不等−1增至4.49 t ha−1,平均种子产量为3.57 t ha−1(±0.025 t ha−1),种子蔗糖浓度变化范围为5.44 ~ 6.82%,平均蔗糖浓度为6.06%(±0.016%;补充表S2- s4).在POPn_2中也有类似的变化(图2)。1).种子重17.67 ~ 22.95 g,平均20.34 g(±0.057 g)。种子产量从2.52吨公顷不等−1至4.40 t ha−1,平均种子产量3.34 t ha−1(±0.024 t ha−1),种子蔗糖浓度变化范围为4.95 ~ 6.75%,平均蔗糖浓度为5.84%(±0.014%)。两个居群的种子产量和种子蔗糖浓度均表现为海侵分离。虽然一些RILs在单个环境中表现出海侵分离的种子重量,但当考虑组合多环境数据时,没有观察到这一点(补充表S2- s4).
我们之前的研究显示了显著的差异(p基因型、环境和基因型x环境处理对这些群体蛋白质浓度和产量的影响均< 0.01)[53],表明遗传因素对这些目标性状的表现起着重要作用。蛋白质浓度和百粒重(H2= 0.93-0.95, 0.87-0.89;补充表S5).蔗糖浓度(H2= 0.70 - -0.81;补充表S5种子产量遗传力较低(H2= 0.22-0.36)(补充表S .5).
性状之间的关系
采用Pearson相关系数法测定了单个环境和复合多环境下种子蛋白质浓度与蔗糖浓度、种子重量与产量之间的关系。综合多环境数据,两居群种子蛋白与蔗糖浓度呈极显著(α = 0.05)负相关(POPn_1: r =−0.47;POPn_2: r =−0.70;无花果。2).在POPn_1中,种子蛋白质浓度与种子重量呈正相关(POPn_1: r = 0.53),种子重量与蔗糖浓度呈负相关(POPn_1: r =−0.29)。有趣的是,两个居群的种子蛋白质浓度与种子产量之间均无显著相关性(POPn_1: r = 0.09;POPn_2: r =−0.06)(图;1而且2).各环境目标农艺性状与种子品质性状之间的线性关系见补充表S6.
大豆基因组的SNP定位
从每个群体的多态SNP标记构建连锁图。在POPn_1中,利用807个多态性SNP标记建立连锁图谱,并将其划分为39个连锁群。在POPn_2上建立了包含40个连锁群上1406个SNP标记的连锁图谱。大豆基因组全部20条染色体均有表达,大多数染色体由两个或多个连锁基团组成。POPn_1和POPn_2的连锁图谱长度分别为2385和2690 cM。连锁群数量的增加归因于分布在大染色体区域的亲本基因型之间缺乏多态性标记,因为加拿大大豆优良品种可能具有相似的谱系。
与种子蛋白浓度相关的QTL
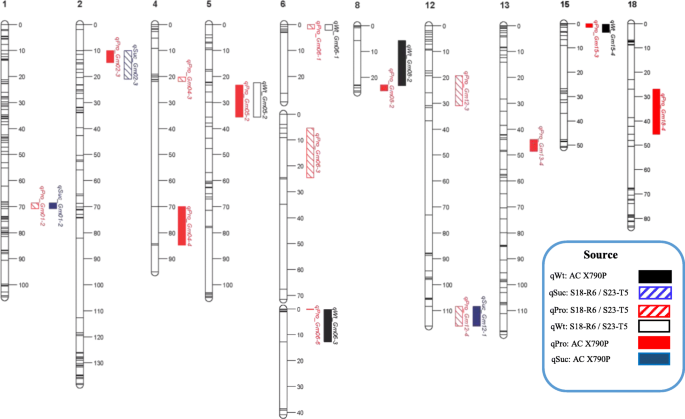
结合多环境数据,在染色体1、2、4、5、6、8、12、13、15和18上鉴定出14个与种子蛋白浓度相关的大效应QTL。所有QTL至少在4个独立环境中与蛋白质相关。这14个QTL解释了10.4 - 21.9%的种子蛋白质浓度表型变异1).其中6个QTL -qPro_Gm01-2,qPro_Gm04-3,qPro_Gm06-1,qPro_Gm06-3,qPro_Gm12-3,qPro-Gm12-4-携带了来自“S18-R6”或“S23-T5”的有益等位基因,其余8个QTL -qPro_Gm02-3,qPro_Gm04-4,qPro-Gm05-2,qPro_Gm06-6,qPro-Gm08-2,qPro-Gm13-4,qPro_Gm15-3,而且qPro_Gm18-3-携带来自“AC X790P”的有利等位基因。蛋白质相关QTL等位基因在不同遗传背景下均呈阳性,提示有可能通过叠加有利等位基因来获得高蛋白优良子代。
在本研究确定的14个QTL中,9个QTL -qPro_Gm01-2(r2 = 10.4%),qPro-Gm04-4(r2 = 13.7%),qPro-Gm05-2(r2 = 14.2%),qPro_Gm06-1(r2 = 21.9%),qPro_Gm06-3(r2 = 12.6%),qPro_Gm08-2(r2 = 12.3%),qPro-Gm12-3(r2 = 11.6%),qPro-Gm12-4(R2 = 12%),和qPro_Gm13-4(R2 = 11.6%) -为以前未报道的QTL,因此被认为是新的QTL1;26)。这些新QTL中有4个在两个映射群体中都被检测到(表2)1).表中列出了与先前在SoyBase上报道的蛋白质相关QTL共定位的其余QTL1;补充表S7.
附加增值性状相关QTL
利用多重QTL作图(MQM)算法的复合区间作图分析,对含有假定的与种子蛋白质浓度相关的大效应QTL的基因组区域与种子产量、蔗糖浓度和种子重量的关系进行了评估。(表2;补充表S8).在14个蛋白质相关QTL中,有8个与其他性状相关QTL共定位。3个蛋白质相关QTL -qPro_Gm01-2,qPro_Gm02-3,qPro_Gm12-4-与种子蔗糖浓度相关的QTL共定位(表2).这些有利的等位基因分别来自不同的亲本来源,这支持了本研究中观察到的种子蛋白质和蔗糖浓度之间的显著负相关关系。(表2;无花果。3.).其余5个与蛋白质相关的QTL与种子重量相关,其中3个区域与种子重量呈正相关2;无花果。3.).所有感兴趣性状的有利等位基因均由亲本品种提供。蛋白质相关QTL没有与种子产量的显著区域共定位,这与两个居群中蛋白质浓度与种子产量的不显著关系一致。在种子重量(9个)、种子油浓度(5个)和种子产量(2个)方面,SoyBase将我们的7个蛋白质相关QTL与先前确定的QTL进行了关联(补充表S)7[37].
在蛋白质QTL区域内挖掘候选基因
为了进一步验证与种子蛋白浓度相关的QTL,使用Williams 82在SoyBase上的Glyma 2.0 Assembly (Wm82.a2.v1),根据其功能知识,编制了候选基因列表[37].每个QTL侧翼区域的基因数量从4个到74个不等。在侧翼区域对应qPro_Gm13-4(跨度26 kb),鉴定出5个基因。这些基因包括Glyma.13G167800和Glyma.13G167900,分别位于SNP峰(28246299)下游6和9 kb处,分别被标记为核糖体蛋白和核糖体生物发生调节蛋白(表2)3.).这些基因在蛋白质合成中起间接作用。Severin等人提供的基因表达数据[54]指出,Glyma.13G167800在开花后10 - 21天在种子中表达(DAF)。Glyma.13G167900也在种子中表达,尽管表达水平低于Glyma.13G167800。两个候选基因,Glyma.06G004500和Glyma.06G001800qPro_Gm06-1被确定。这些基因分别位于QTL峰值上游74 kb和下游148 kb,编码跨膜氨基酸转运蛋白和核糖体家族蛋白(表2)3.).先前的转录组分析发现,在14至17岁的种子中,Glyma.06G004500的表达增加,21 DAF [54].
Glyma.04G212500和Glyma.04G214500在qPro_Gm04-4时间间隔.这些基因分别与cupin超家族和核糖体蛋白家族相关(表2)3.).cupin超家族与种子储存蛋白有关[55],而核糖体蛋白家族基因与mRNA翻译相关。候选基因Glyma.04212500恰好位于SNP峰位置,支持cupin与种子蛋白浓度相关的作用。Glyma.06G113700, Glyma.06G116400, Glyma.06G119700位于qPro_Gm06-3地区(表3.).Glyma.06G113700编码40S核糖体蛋白的一个潜在结构成分。Glyma.06G116400和Glyma.06G119700分别与一个跨膜氨基酸转运蛋白和一个细胞内转运蛋白相关3.).
从中鉴定出3个候选基因Glyma.15G129800、Glyma.15G130000和Glyma.15G134800qPro_Gm15-3它们参与核糖体的结构成分(表3.).下鉴定了翻译起始因子蛋白Glyma.06G225600和Glyma.06G225700qPro_Gm06-6时间间隔(表3.).其中Glyma.02G220000和Glyma.02G221500有助于核糖体的结构完整性并在翻译中发挥作用qPro_Gm02-3地区(表3.).根据之前的转录组分析,Glyma.02G220000在种子14 ~ 17、21、25、28和35 DAF中表达[54].
候选基因也被假定为与蛋白质相关区域共定位的蔗糖和种子重量相关QTL。鉴定出4个候选基因:Glyma.06G004400和Glyma.06G007900qPro_Gm06-1而且qWt_Gm06-1地区和Glyma.15G133600和Glyma.15G133800qPro_Gm15-3而且qWt_Gm15-4地区.这四种基因都参与碳水化合物代谢(GO:0005975)(表4).
讨论
以大豆为基础的食品制造商需要大豆种子特定的物理和化学特性来维持他们的生产实践。例如,最佳的豆腐生产需要大豆种子中含有高浓度的蛋白质和蔗糖。而蛋白质与蔗糖浓度呈负相关[38,52,56,57,58].种子蛋白浓度与其他增值性状之间的这些显著负相关,已成为通过传统育种方法培育具有竞争力的食品级大豆品种的主要障碍[14,15,16,17,18,19,20.,21,22,23,59].对蔗糖没有影响或对其他增值性状有积极影响的蛋白质相关QTL的鉴定将是主要的好处。在本研究中,种子蛋白质浓度、种子重量和产量之间的关系表明,目前这两个群体都适合选择具有竞争力的产量和大种子大小的最佳蛋白质浓度。另一方面,种子蛋白质与蔗糖浓度呈负相关,说明蛋白质浓度的选择可能以牺牲种子蔗糖浓度为代价(反之亦然)。这些关系可以归因于分别控制这些性状的紧密连接位点,或者归因于特定位点的多效性效应[19].
本研究的广义遗传力估计证实,所观察到的种子蛋白质浓度、种子蔗糖浓度和种子重量的表型变异有很大一部分归因于基因型。因此,表型选择可能是增加这些性状遗传增益的成功工具。这与先前的研究一致,在这些研究中,已经报道了种子蛋白质浓度(H2= 0.81 - -0.92;[16,60],种子蔗糖浓度(H2= 0.46 - -0.86;[60,61]和种子重量(H2= 0.73 - -0.89;[60]跨越不同的基因背景和环境。
使用MAS可以为多个感兴趣的性状“堆叠”理想的QTL,这使育种家能够筛选早期世代材料以获得最佳的性状组合。这种方法已被用于育种项目,特别是育种抗病品种[62,63,64].马鲁夫等人。[65]讨论了利用MAS构建大豆花叶病毒抗性基因的价值,其中涉及到优化多重抗性的特定遗传组合。这种方法增加了育种计划选择具有多重抗性的纯合植物的能力,因为抗病基因之间的上位相互作用使得疾病反应的表型筛选不可靠[65].Jiang等人也采用了这种策略。[66],不同亲本来源的阳性等位基因的聚合显示出提高大豆种子蛋白质填充率和整体种子质量。
在本研究中,确定了14个与种子蛋白质浓度相关的大效应QTL,阳性等位基因来自每个亲本来源。这可能是由于本研究中使用了独特的映射种群。以前的QTL研究使用的定位群体来自具有较大表型差异的外来种质或亲本品种,以获得所需的感兴趣性状[50].然而,许多现代优良大豆品种已经具有较高的蛋白质浓度(约40%,干基),并可能固定为在不同群体中鉴定的大效应QTL。在目前的研究中,利用中等和高蛋白的优良亲本品种,可以识别可能在其他群体中被掩盖的新QTL [60,67,68],也会导致大多数染色体上出现两个或多个连锁基团,并且缺少与种子蛋白浓度相关的主要QTL区域,例如15号和20号染色体上的QTL区域。这些区域的消除也可能限制了这些群体中QTL相互作用的全部范围,并夸大了已确定的QTL对感兴趣性状的影响[67,69,70].此外,许多QTL作图程序在小效应和中效应QTL的识别上存在困难。这些小的和中间的QTL主要与数量性状有关,如种子蛋白浓度[71,72].Beavis效应表明,在较小的映射群体(< 1000个子代;61),这可能进一步夸大了本研究中确定的QTL的影响。
最近,黑格雷等人。73]利用直接分子辅助选择技术改善大豆种子碳水化合物的组成。棉子糖合成酶3基因的自然变异(rs3 snp5)与超低棉子糖家族低聚糖(UL RFO)碳水化合物特征相关,从而提高了豆粕的蔗糖浓度和有效代谢能[74,75].棉子糖和水苏糖的减少归因于一种特定的遗传组合平日W331+6 . Rs3 snp5/ Rs3 SNP单倍型C -由RS3基因缺陷引起。开发了分子标记试验来检测这些变异,使其向优良大豆品种的渐渗更加流畅[73].
为了进一步了解大豆种子中蛋白质富集的潜在机制,我们从蛋白质相关QTL的侧翼区域鉴定了候选基因,并对其在蛋白质积累中的功能进行了筛选。在这项研究中,通过对491个基因的生物学过程和功能注释,在SoyBase (www.soybase.org;[76])。许多假定的候选基因被确定(表4通过GO注释:与蛋白质翻译过程相关的基因有16个(GO:0006412, GO:0015171, GO:0006413, GO:0042254, GO:0006886, AT6G61750, PF01490),与碳水化合物代谢相关的基因有8个(GO:0005975),与脂类代谢相关的基因有3个(GO:0006629),其余参与信号转导、转运、生物合成过程、核酸代谢、光合作用等多种功能。蛋白质、油脂和蔗糖之间的显著关系[38,52,55,57]支持脂质和碳水化合物代谢相关基因的作用,这些基因也在这些蛋白质相关QTL的侧翼区域被鉴定出来。
Severin等人提供的转录组分析数据,[54]表明,Glyma.13G167800(核糖体生物发生)、Glyma.13G167900(核糖体生物发生)、Glyma.06G004500(跨膜氨基酸转运蛋白)和Glyma.02G220000 (60S核糖体蛋白)在大豆种子中表达,这支持了它们在大豆种子蛋白质积累中的作用。Glyma.04G212500与cupin超家族相关,其中包括11S(甘氨酸)和7S (ß-conglycinin)种子存储蛋白。11S和7S种子贮藏蛋白占大豆种子贮藏蛋白的70% [54,77].因此,Glyma.04G212500可能与大豆种子蛋白积累有很强的相关性。张等。[78]在第15和20号染色体上鉴定了13个可能参与蛋白质生物合成的候选基因,并对核糖体结构成分、60S核糖体蛋白、氨基酸跨膜转运和翻译起始因子3进行了功能注释。在我们的研究中,这些注释也与七个候选基因相关,这有力地支持了它们在我们人群中蛋白质积累中的作用。张等。[78]还对核糖体、翻译起始因子3和氨基酸跨膜转运基因进行了基因表达分析,结果表明高蛋白亲本在荚果生殖生长阶段表达显著上调。这与它们在大豆种子蛋白质积累中的作用是一致的[78].李等人。[79]也在9号染色体上一个蛋白质QTL的侧翼区发现了一个候选基因,该基因被注释为氨基酸转运体基因。在另一项研究中,一种氨基酸转运基因的过度表达野豌豆属narbonensis豌豆导致种子蛋白质浓度显著增加[80].对这些候选基因及其可能的变异的进一步探索,将进一步加深我们对大豆种子蛋白质积累途径的认识,并可能为改进标记或分子辅助育种技术以改善大豆种子成分性状提供依据。
结论
综上所述,本研究中鉴定的9个蛋白质相关QTL被验证,可能适用于标记辅助选择程序。每一个都为同时改善多个性状提供了重要信息。它们的价值将由个体育种计划的目标决定。例如,qPro_Gm06-1、qPro_Gm06-6、qPro_Gm08-2和qPro_Gm15-3与种子重QTL呈正相关。这些QTL可能不适合纳豆育种计划,这将有利于较小的种子大小。在这种情况下,qPro_Gm05-2一个与蛋白质相关的QTL与种子重量呈负相关。一个多性状QTL的策划小组可以让育种家筛选早代种质,以获得大豆食品加工所需的特定物理和化学特征。
未来的研究可能会考虑蛋白质的生物合成、储存和代谢对大豆种子蛋白质浓度的影响,如本研究中假设的候选基因功能所示,以促进对大豆种子蛋白质积累途径的更好理解。育种家也可能希望更深入地研究这些候选基因的潜在变异,以及它们在植物代谢中的作用。本研究提供的QTL可作为利用标记辅助选择的食品级大豆育种项目的工具,并可作为发现蛋白质生物合成途径变异的起点。
方法
映射的数量
F的两个种群4利用重组自交系(RILs)进行种子组成、性状和产量的数量性状推测位点(QTL)鉴定。第一个群体(POPn_1)由190个来自“AC X790P”和“S18-R6”杂交的RILs组成。“AC X790P”是一个2.2相对成熟度组(MG)品种,由加拿大农业和农业食品部门在安大略省哈罗开发,具有高而稳定的种子蛋白质浓度(48.6%,干重基础;[49])。' S18-R6 '是一个1.8 MG的商业品种,具有中等的种子蛋白浓度(40.4%),由安大略省阿尔瓦的先正达加拿大公司开发[81].
第二个群体(POPn_2)由‘S23-T5’和‘AC X790P’杂交的193个RILs组成。“S23-T5”是由明尼苏达州奥瓦托纳市的先正达种子公司开发的高产2.3 MG种子蛋白中等(41.3%)的优良品种[82].与安大略省西南部的历史产量相比,亲本品种被认为产量高[83].这两种RIL人群都是在圭尔夫大学里奇敦校区开发的。
实验设计
2015年和2016年,RIL种群在安大略省西南部的五个环境中生长:查塔姆2015年(CHA15)、梅林2015年(MER15)、查塔姆2016年(CHA16)、梅林2016年(MER16)和巴尔米拉2016年(PAL16)。田间试验采用随机完全区组设计,并进行两次重复,其中通过最近邻分析(NNA),利用五个环境中每个环境中最近邻小区的信息,对小区性能进行空间变异性调整[53].样地由5个4米行组成,行距43厘米,出苗后修剪至3.8米长。样地按69粒/m播种2或者说每亩500粒种子。试验继续采用标准的耕作和栽培方法,每个地块中间的三行进行收获,用于种子产量估计和收获后评估。
表型数据收集
使用Perten DA 7250 SD近红外反射(NIR)分析仪(Perten Instruments Canada, Winnipeg, MB)测定每个收获地块的种子蛋白质和蔗糖浓度,校准方法由Perten Instruments提供[84,85,86,87].不同种子成分性状(包括种子蛋白质和蔗糖浓度)的校准统计数据见补充表S9.每次近红外测量是三次技术复制的平均值。种子产量(吨公顷−1在水分13%时)和种子重量(每100粒种子克)也记录在每个收获地块。
统计分析
使用SAS 9.4 (SAS Institute Inc., Cary, NC)进行统计分析。进行方差分析(ANOVA),使用PROC MIXED为每个环境生成lsmean,“基因型”为固定效应,“块”为随机效应。PROC MIXED还使用以下模型对种子重量、蛋白质和蔗糖浓度进行组合方差分析:
在哪里Yij表示感兴趣的性状(种子蛋白质积累量、种子蔗糖积累量、种子重量或种子产量),α我代表“基因型”效应,βj表示“环境”效应,αβij表示“环境基因型”效应和εij表示剩余效应。“基因型”、“环境”和“由环境决定的基因型”被认为是固定效应,“块(环境)”被认为是随机效应。采用PROC CORR法检验各输入性状估计之间的关系。
基因型数据收集
从Palmyra 2016地点的每个群体的第一个复制块中收集年轻的三叶叶组织。每个RIL的叶片组织从每个地块的多个植物中取样,并保存在2 mL螺旋盖管中。使用Savant ModulyoD Thermoquest (Savant Instruments, Holbrook, NY)将样品冷冻干燥72小时,然后在−80°C保存以备将来使用。基因组DNA从冷冻干燥的组织样本中提取,使用Sigma GenElute™DNA提取试剂盒(Sigma®,圣路易斯,密苏里州)方法的改进程序。DNA质量使用1%琼脂糖凝胶电泳验证,而数量使用Qubit®2.0荧光计(Invitrogen, Carlsbad, CA)验证.
DNA样品(30 μl / 10 ng μl−1DNA)转移到plate - formme D 'analyses Génomiques, Université Laval (Laval, Quebec, Canada)进行基因测序分型(GBS),使用Fast-GBS管道Gmax_275_v2参考基因组[88].Fast-GBS管道鉴定出24,738个高质量单核苷酸多态性(SNPs)。杂合snp被认为是缺失数据。> 20%缺失数据或最小次要等位基因频率小于0.3的snp在用Beagle归一之前被丢弃[89].
连锁图谱构建和QTL定位
采用JoinMap 5.0软件构建各群体遗传连锁图谱[90].分离畸变水平显著且与卡方检验(α = 0.01)预期的1:1比例不同的SNP标记被从进一步分析中移除。在种群内分离相同的标记被减少为一个标记,用于连接图的构建。利用最小似然概率(LOD)≥3将标记分组为每个染色体内的连锁组,并使用Kosambi映射函数计算遗传距离。此后,这些标记的遗传位置被锚定在物理位置上。
利用MapQTL®6中的多QTL映射(MQM)算法对感兴趣的性状进行复合区间映射(CIM) [91].经验LOD阈值通过1000次置换试验计算,I型错误率为0.05。使用自动辅助因子选择函数来识别MQM的重要辅助因子。使用MapChart 2.32创建了重要QTL的图形表示[92].
同时还筛选了与种子蛋白质浓度相关的QTL区域,发现了与种子重量、种子产量和种子蔗糖浓度相关的显著QTL。利用SoyBase将假定的QTL与已发表的与种子蛋白浓度相关的基因组区域进行比较[38].在SAS 9.4 (SAS Institute Inc., Cary, NC)中,使用单标记分析(SMA)在交替群体中也确认了假定的QTL。以LSMEAN估计值为因变量,以SNP标记物为自变量,采用PROC GLM方法鉴定显著单标记物效应(α < 0.0001)。通过基因分型测序得到的SNP位置用于表示MQM和SMA中的标记名称。
候选基因搜索
每个QTL的侧翼标记是根据每个峰值标记周围的LOD值选择的。为了确保实际QTL位于所选范围内,选择QTL峰值两侧LOD阈值以下的第一个标记作为侧翼标记。对于每个与蛋白质相关的QTL,利用侧翼标记之间的区域根据其功能来识别候选基因。利用SoyBase大豆遗传图谱从侧翼区域共提取了491个基因。每个基因的功能注释通过TAIR (www.arabidopsis.org/),去(http://geneontology.org/), pfam (http://pfam.xfam.org/)和PANTHER (http://www.pantherdb.org/)透过SoyBase (https://soybase.org/).这种功能知识用于减少基因数量和确定假定的候选基因。
大豆电子荧光象形文字浏览器(www.bar.utoronto.ca)用于生成关于候选基因的附加信息,例如组织和发育阶段依赖的表达(基于Severine等人的转录组数据。[54])。Pfam,蛋白质结构域和家族的综合集合,以及NCBI用于获得候选基因的额外信息。
数据和材料的可用性
支持本研究结果的表型和基因型数据可在figshare网站上公开获取https://doi.org/10.6084/m9.figshare.13008467.
缩写
- QTL:
-
数量性状位点
- MAS:
-
分子标记辅助选择
- 瑞来斯:
-
重组自交系
- DAF):
-
开花后翌日
- POPn_1:
-
第一个人口
- POPn_2:
-
第二个人口
- MG:
-
成熟的组织
- CHA15:
-
查塔姆2015
- MER15:
-
梅林2015
- CHA16:
-
查塔姆2016
- MER16:
-
梅林2016
- PAL16:
-
巴尔米拉2016
- NNA:
-
最近邻分析
- 近红外光谱:
-
近红外反射
- 方差分析:
-
方差分析
- 单核苷酸多态性:
-
单核苷酸多态性
- CIM:
-
复合区间映射
- 统一民族运动党:
-
多QTL映射
- LOD:
-
概率
- SMA:
-
单标记分析
- eFP:
-
电子荧光象形文字
参考文献
- 1.
王海林,赫塞尔廷。豆腐加工中的凝血条件。生物化学,1983;17:7-12。
- 2.
沈cf, L DeMan, RI Buzzell, DeMan JM。大豆和豆浆特性对豆腐产量和品质的影响:葡萄糖酸-δ-内酯混凝剂食品科学,1991;56:109-12。
- 3.
谢斐MJ,爱。大豆成分与豆腐质地的关系。食品质量,1992;15:53-66。
- 4.
蔡涛,常kc .加工对大豆贮藏蛋白的影响及其与豆腐品质的关系。食品化学学报。1999;47:720-7。
- 5.
大豆种子成分的稳定性及其对豆浆和豆腐产量和品质的影响。食品科学,2002;35:337-45。
- 6.
大豆品种影响豆浆和豆腐的品质和功能。中国食品农业科学,2005;85:2514-8。
- 7.
Stanojevic SP, Barac MB, Pesic MB, Vucelic-Radovic BV。大豆基因型及加工方法对豆腐脑品质的影响。农学通报。2011;49:7368 - 76。
- 8.
卡弗BF,伯顿JW,卡特TE,威尔逊RF。改变不饱和脂肪酸组成的大豆系对环境变化的响应。作物科学1986;26:1176-81。
- 9.
王晓明,王晓明,王晓明,等。中欧地区大豆种子蛋白质含量的遗传变异及其影响因素。科学通报2000;1306:1300-6。
- 10.
苏达林,李志强,王志强,等。东南欧洲大豆育种中环境相互作用对基因型的影响。植物育种。2006;125:191-4。
- 11.
刘志强,刘志强,刘志强,刘志强,等。大豆蛋白质、油脂及5种主要脂肪酸含量的QTL鉴定植物科学学报,2014;5:568 - 568。
- 12.
王志强,王志强,王志强,等。拓展大豆种子成分性状改良的组学资源。前沿植物学报2015;6:1021。
- 13.
马勇,阚刚,张旭,王勇,张伟,杜辉,等。大豆(Glycine max L. Merr)中甘氨酸和β -同源甘氨酸含量的QTL定位农业化学学报。2016;64:3473-83。
- 14.
Shannon G, Wilcox JR, Probst AH。6个大豆群体F4代蛋白质选择和产量的估计收益。作物科学,1972;12:24 - 6。
- 15.
伯顿JW。提高蛋白质数量和质量的大豆育种。在:Shibles R,编辑。第3届大豆研究会议,1984年8月12-17日,美国艾姆斯。博尔德:西景出版社;1985.361 - 7页。
- 16.
伯顿JW。定量遗传学:与大豆育种有关的结果。进:Wilcox JR,编辑。大豆:改良、生产和使用。麦迪逊:美国农学学会;1987.211 - 47页。
- 17.
Wilcox JR, Cavins JF。高种子蛋白回交大豆品种。作物科学,1995;35:1036-41。
- 18.
赫尔姆斯TC,奥尔夫JH。选择增加蛋白质的大豆系的蛋白质、油和产量。作物科学,1998;38:457 - 457。
- 19.
郑杰,李文杰,李文杰,等。大豆种子蛋白质、油脂与产量QTL的研究。作物科学,2003;43:1053-67。
- 20.
崔z, James AT, Mizazaki S, Wilson RF, Carter TE。培育传统和新型大豆食品专用大豆。在:刘科,编辑。大豆作为功能性食品和配料。香槟:AOCS出版社;2004.p . 264 - 322。
- 21.
尹晓霞,吴志强。大豆种子中异黄酮、油脂和蛋白质与产量的关系。艾力龙。2005;97:1314-21。
- 22.
王志强,王志强,王志强,等。美国农业部大豆种质资源的群体结构和全基因组关联分析。植物基因组。2015;8:1-13。
- 23.
Kim M, Schultz S, Nelson RL, Diers BW。大豆种子蛋白15号染色体PI 407788A QTL的鉴定及精细定位。作物科学,2016;56:219。
- 24.
曾杰,陈鹏,石安,王东,张波,Orazaly M,等。大豆种子蔗糖含量数量性状位点的鉴定。作物科学2015;54:554-64。
- 25.
海莫维茨T Collins FI Panczner J Walker WM。大豆种子中油、蛋白质和糖含量的关系。Agron J. 1972; 64:613-6。
- 26.
Brummer EC, Graef GL, Orf J, Wilcox JR, Shoemaker RC。8个大豆居群种子蛋白质和油脂含量的QTL定位。作物科学,1997;37(2):370-8。
- 27.
Fasoula VA, Harris DK, Boerma HR。来自两个大豆群体的种子蛋白质、种子油和种子重量数量性状位点的验证和指定。作物科学2004;44(4):1218-25。
- 28.
李旭,马贝利,马勉,卡特,ER施佩尔,达希利,等。与大豆种子蛋白质和油脂含量相关的RFLP基因座在人群和地点的分布应用理论。1996;93(5-6):649-57。
- 29.
沃林顿CV, Abdel-Haleem H, Hyten DL, Cregan PB, Orf JH, Killam AS,等。贝宁×丹白空大豆群体种子蛋白质和氨基酸的QTL分析应用物理学报,2015;28(5):839-50。
- 30.
毛涛,姜志,韩艳,滕伟,赵霞,李伟。大豆多遗传背景和环境下蛋白质和油脂含量数量性状位点的鉴定。植物营养学报,2013;29(6):344 - 344。
- 31.
齐震,侯敏,韩旭,刘超,姜华,辛东,等。大豆种子蛋白质浓度QTL的鉴定及其在多种环境下的加性效应和上位性效应分析植物营养学报,2014;29(4):344 - 344。
- 32.
斯托博SK, Orf JH, Jung HG, Chase K, Lark KG, Somers DA。大豆种子细胞壁多糖相关数量性状位点的研究。作物科学2004;44(6):2101-6。
- 33.
王旭,姜桂兰,Green M, Scott RA,宋q, Hyten DL, Cregan PB。大豆两个重组自交系群体籽粒产量、油脂和蛋白质含量数量性状位点的鉴定与验证。中国生物医学工程学报。2014;29(5):339 - 339。
- 34.
刘丽娟,刘丽娟,董忠,刘丽娟。大豆在加拿大×中国杂交两个独立居群中适应北美和亚洲巨环境的遗传基础。《理论与实践》,2013;26(7):379 - 379。
- 35.
Palomeque L, Li- jun L, Li W, Hedges B, Cober ER, Rajcan I.超大环境QTL: 2。在高产适应性大豆与高产外来大豆系杂交群体中发现农艺性状QTL与种子产量QTL共定位。应用理论,2009;119:429-36。
- 36.
梁兆辉,于玉良,王顺丰,云莲,王廷峰,魏玉良,等。大豆异黄酮、油脂和蛋白质含量的QTL定位。中国农业科学,2010;9(8):1108-16。
- 37.
SoyBase。美国农业部大豆基因和基因组数据库。2019.https://soybase.org/.2019年4月5日访问。
- 38.
陈鹏,陈晓明,王晓明,陈晓明,等。大豆种子蛋白分子图谱和基因组学研究进展及展望。《理论与实践》,2017;
- 39.
齐震,张震,王震,于洁,秦宏,毛旭,等。元分析和转录组分析揭示了大豆种子发育过程中储存组成的枢纽基因。植物细胞环境学报,2018;41:2109-27。
- 40.
Panthee DR, Pantalone V, West DR, Saxton AM, Sams CE。大豆种子蛋白质、油脂含量及种子大小的数量性状位点。作物科学(英文版);2005;
- 41.
植物数量性状的育种。第二版。伍德伯里:斯坦玛出版社;2010.
- 42.
齐震,吴强,韩旭,孙勇,杜旭,刘超,等。大豆油含量QTL定位与配体分析方法的整合。Euphytica。2011;179:499 - 514。
- 43.
张志强,张志强,张志强,等。下一代大豆囊线虫抗性育种的基因组辅助系统发育分析和标记开发。植物科学进展(英文版);
- 44.
王旭,姜刚,宋强,Cregan P, Scott R,张杰,等。大豆两个重组自交系群体种子含硫氨基酸的数量性状位点分析。Euphytica。2015;201:293 - 305。
- 45.
乔福KD,戈德堡RB。Kunitz胰蛋白酶抑制剂基因在大豆生命周期和转化烟草植株中有差异表达。植物学报。1989;1:1079-93。
- 46.
Walling L, Drews GN, Goldberg RB。大豆种子蛋白mRNA水平的转录和转录后调控。中国科学院学报(自然科学版)1986;83:2123-7。
- 47.
叶文伟,陈建春,林密,陈玉梅,林春春。甘薯孢子粉的功能活性:一种具有胰蛋白酶抑制活性的块茎贮藏蛋白。植物化学学报,1997;33:565 - 566。
- 48.
田罗堂,马大藤,马西西,罗瑞,邝瑞文,等。拟南芥叶状子叶1在营养细胞中足以诱导胚胎发育。细胞。1998;1:1195 - 205。
- 49.
Soderman EM, Brocard IM, Lynch TJ, Finkelstein RR。拟南芥ABA-insensitive4基因在种子和脱落酸反应信号网络中的调控和功能。植物学报。2000;124:1752-65。
- 50.
Hyten DL, Pantalone VR, Sams CE, Saxton AM, Landau-Ellis D, Stefaniak TR,等。大豆优良群体的种子质量QTL。应用理论,2004;109:552-61。
- 51.
Baianu I, You T, Costescu D, Lozano P, Prisecaru V, Nelson R.高分辨核磁共振(NMRS)和近红外(NIRS)测定大豆种子中大豆油、蛋白质和氨基酸残留量。导刊。2012;7:1-1。
- 52.
王晓燕,王晓燕,张晓燕,等。利用高密度连锁作图技术分析大豆种间作图群体中隐含种子蛋白质、油脂和蔗糖含量的基因组热点。生物科技植物》。j . 2018; 16:1939-53。
- 53.
Whaley R, Eskandari M.基因型主效应和基因型-环境互作效应对食品级大豆种子蛋白质浓度和产量的影响(大豆(l)美林)。Euphytica。2019;215:33。
- 54.
Severin AJ, Woody JL, Bolon YT, Joseph B, Diers BW, Farmer AD,等。Glycine max的rna序列图谱:大豆转录组指南。中国生物医学工程学报。2010;
- 55.
Dunwell JM。Cupins:一个功能多样的蛋白质新超家族,包括发芽蛋白和植物贮藏蛋白。生物技术,1998;15:1-32。
- 56.
Nichols D, Glover K, Carlson S, Specht J, Diers B.大豆种子蛋白QTL的精细定位及其对农艺性状的影响。作物科学2006;46:834。
- 57.
陈鹏,陈志强,陈志强。大豆食品级性状的遗传力与相关性。植物育种,2011;130:647-52。
- 58.
Sonah H, O 'Donoughue L, Cober E, Rajcan I, Belzile F.利用GBS鉴定8个农艺性状的基因座?WAS方法在大豆的QTL中有效。2015; 12:211-21。
- 59.
Poysa V, Buzzell RI。AC X790P大豆。植物科学进展(英文版);2001;
- 60.
Eskandari M, Cober ER, Rajcan I.大豆籽油的遗传控制:一、中等高油亲本杂交大豆籽油居群的QTL及其与种土浓度相关的基因。《应用理论》2013;126:483-95。
- 61.
王晓明,王晓明,王晓明,等。大豆蔗糖含量控制基因座的鉴定。Mol Breed, 2000; 6:105-11。
- 62.
Kelly JD, Afanador L, Haley SD。大豆普通花叶病毒抗性的金字塔基因。Euphytica。1995;82:207-12。
- 63.
Miklas PN, Delorme R, Stone V, Daly MJ, Stavely JR, Steadman JR,等。细菌、真菌和病毒抗病基因座在重组自交系普通豆群体('Dorado ' /XAN 176)中定位。自然科学进展,2000;
- 64.
李志刚,黄宁,李志刚,张志刚,等。利用标记辅助选择技术将3个抗白叶枯病基因(xa5、xa13和Xa21)聚合到籼稻品种PR106中。《应用理论》2001;13:1011-5。
- 65.
Maroof S, Jeong SC, Gunduz I, Tucker DM, Buss GR, Tolin SA。大豆花叶病毒抗性基因的金字塔标记辅助选择。作物科学,2008;48:517-26。
- 66.
蒋震,韩勇,滕伟,张震,孙东,李勇,等。大豆种子不同发育阶段蛋白质填充率QTL的鉴定。Euphytica。2010;175:227-36。
- 67.
正如乔丹。植物育种中数量性状位点分析的现状与未来。植物育种,2002;121:281-91。
- 68.
Winter S, Shelp BJ, Anderson TR, Welacky TW, Rajcan I.大豆PI464925B抗大豆囊线虫水平抗性的QTL分析。应用理论,2007;14(4):461 - 72。
- 69.
Hyne V, Kearsey MJ。QTL分析:标记回归的进一步应用。应用理论,1995;91:471-6。
- 70.
王晓明,王晓明,王晓明,等。植物QTL分析研究进展?遗传。1998;80:137-42。
- 71.
导演WD。QTL分析:功率、精密度和准确度。进:帕特森,编辑。复杂性状的分子解剖。佛罗里达州博卡拉顿:CRC出版社;1998.p . 145 - 62.5。
- 72.
徐松。比维斯效应的理论基础。遗传学。2003;165:2226-59。
- 73.
刘志强,王志强,王志强。大豆种子碳水化合物分子辅助育种的研究进展。理论应用,2020;133:1189-200。
- 74.
哈雷利KB,帕姆奎斯特D,比利尤KD。大豆种子碳水化合物特征的分类。农学通报。2013;61:1105-11。
- 75.
施林格,约翰·A,艾米丽·c·迪尔金,克里斯汀·d·比利尤。发芽率高、棉子糖和水苏糖含量超低的大豆。美国专利8,471,107,2013年6月25日发布。
- 76.
莫拉莱斯AM, O 'Rourke JA, Scheider K, Bancroft T, Borem A, Nelson R,等。转录组分析和病毒诱导的基因沉默鉴定了rrpp4介导的亚洲大豆抗锈病途径中的基因。植物功能生物学,2013;4:1029-47。
- 77.
Yaklich RW, Helm RM, Cockrell G, Herman EM.大豆种子主要过敏原在Glycine max种质核心收集中的分布分析。作物科学1999;39:1444-7。
- 78.
张涛,吴涛,王磊,姜波,甄超,袁松,等。结合连锁和GWAS分析确定了与大豆种子蛋白质和油含量相关的qtl。中国生物化学杂志,2019;
- 79.
李霞,邵志,田锐,张辉,杜辉,孔勇,等。多环境和背景下大豆种子蛋白质和油脂含量qtl和候选基因的挖掘。Mol Breed. 2019;39:139。
- 80.
罗瑞奇,李志强,李志强,等。氨基酸转运体(VfAAP1)在豇豆和豌豆种子中的异位表达增加了贮藏蛋白质。植物物理学报,2005;1:1236-49。
- 81.
加拿大食品检验局(CFIA)。作物报告:S18-R6。2011.http://www.inspection.gc.ca/english/plaveg/pbrpov/cropreport/soy/app00006462e.shtml.2016年10月13日访问。
- 82.
加拿大食品检验局(CFIA)。作物报告:S23-T5。2011.http://www.inspection.gc.ca/english/plaveg/pbrpov/cropreport/soy/app00007153e.shtml.2016年10月13日访问。
- 83.
安大略省农业、食品和农村事务部。省级农田作物产量及价格。2016.http://www.omafra.gov.on.ca.2017年12月21日访问。
- 84.
Kovalenko IV, Rippke GR, Hurburgh CR.近红外光谱法测定大豆脂肪酸:线性和非线性定标方法。石油化学学报,2006;83): 421 - 7。https://doi.org/10.1007/s11746-006-1221-z.
- 85.
用近红外光谱法测定全大豆中的脂肪酸。油脂技术,2007;19:88-90。https://doi.org/10.1002/lite.200600029.
- 86.
黄晓明,杨晓明,杨晓明。大豆种子组成对播种速度和行距影响的研究。作物科学(英文版);https://doi.org/10.2135/crop-sci2013.07.0463.
- 87.
杨晓明,李志强,李志强,等。植物密度对亚麻酸还原大豆品种的影响。Agron J. 2010;102:348。https://doi.org/10.2134/agronj2009.0167.
- 88.
Torkamaneh D, Laroche J, Bastien M, Abed A, Belzile F. Fast-GBS:一种从基因分型测序数据中高效、高精度调用snp的新管道。BMC生物信息学。2017;18:5。
- 89.
Browning BL, Browning SR.数百万个参考样本的基因型植入。胡姆。热内。2016;98:116-26。
- 90.
van Ooijen JW。JoinMap 4.0:实验群体遗传连锁图谱计算软件;2006.
- 91.
van Ooijen JW。MapQTL®6,二倍体物种实验群体数量性状位点定位软件;2009.
- 92.
MapChart:用于链接映射和qtl的图形化表示的软件。赫瑞德,2002:77-8。
确认
作者要感谢Bryan Stirling, Chris Grainger, John Kobler,以及Guelph大学Ridgetown校区的整个大豆团队,感谢他们的技术支持。
资金
该项目部分由SeCan和安大略省农业食品创新联盟(之前的OMAFRA- u of G Partnership)资助,该联盟是安大略省农业、食品和农村事务部(OMAFRA)和圭尔夫大学(G of G)之间的合作。资助机构在研究设计、数据收集、分析和解释以及撰写手稿方面没有发挥任何作用。
作者信息
从属关系
贡献
RMW和ME构想并设计了实验;我监督了这项研究;RMW进行实验,收集表型数据,并进行统计分析;ST进行候选基因鉴定;RMW和ST起草了手稿;LL和ME监督统计分析并修改手稿。所有作者均已阅读并批准最终稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
补充文件1:补充表S1。
在查塔姆2015年、查塔姆2016年、梅林2015年、梅林2016年和帕尔米拉2016年这五个环境中,' AC X790P ' x ' S18-R6 '和' AC X790P ' x ' S23-T5 '这两个RIL群体的大豆种子蛋白质浓度(%,以干重为基础)的平均值、标准误差(α = 0.05)、范围和亲本平均值。补充表S2。大豆种子产量的平均值、标准误差(α = 0.05)、范围和亲本平均值(吨公顷)−1’AC X790P’x’S18-R6’和‘AC X790P’x’S23-T5’这两个RIL群体在Chatham 2015、Chatham 2016、Merlin 2015、Merlin 2016和Palmyra 2016这五个环境中均存在。补充表S3。在查塔姆2015年、查塔姆2016年、梅林2015年、梅林2016年和帕尔米拉2016年这五种环境中,' AC X790P ' x ' S18-R6 '和' AC X790P ' x ' S23-T5 '两个RIL群体的大豆种子重量(100粒种子重量,单位为克)的平均值、标准误差(α = 0.05)、范围和亲本平均值。补充表S4。在Chatham 2015、Chatham 2016、Merlin 2015、Merlin 2016和Palmyra 2016这五种环境中,' AC X790P ' x ' S18-R6 '和' AC X790P ' x ' S23-T5 '两个RIL群体中大豆种子蔗糖浓度的平均值、标准误差(α = 0.05)、范围和亲本平均值(%,干基)。补充表S5。在5种环境(CHA15、CHA16、MER15、MER16和PAL16)中,评价了2个RIL群体的蛋白质浓度、蔗糖浓度、种子重量和种子产量的广义遗传力。补充表S6。在5种环境(Chatham 2015、Chatham 2016、Merlin 2015、Merlin 2016和Palmyra 2016)以及重组自交系群体的组合环境中,分析了种子蛋白质和蔗糖浓度、100粒重和种子产量的Pearson相关系数。补充表S7。主要假定QTL (R2> 10.0%)与大豆种子蛋白浓度、蔗糖浓度和种子重量相关,在先前的研究中有可能用于标记辅助选择、候选基因和共定位QTL。补充表S8。主要假定QTL (R2> 10.0%)与大豆种子蔗糖浓度、产量和种子重量相关,具有潜在的标记辅助选择应用价值,通过多QTL作图(MQM)在2015年和2016年在安大略省联合环境下对RIL群体进行了检测。补充表S9。全籽(干基)油、蛋白质、水分和脂肪酸成分的校准值,由Perten Instruments提供。交叉验证的决定系数(R2CV)解释了在参考化学和预测值之间可以预测的方差的比例。最小值和最大值是预测范围的极限。SECV是交叉验证的标准误差,其中从验证集中移除样本并进行预测,并计算数据集的总误差。因子表示一个给定性状的校准方程中包含的因子的数量。样品显示校准中使用的样品总数。列出了每个特性的校准文件日期。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
怀廷,r.m.,托拉比,S,卢肯斯,L。et al。与食品级大豆重要种子品质性状相关的基因组区域。BMC植物生物学20.485(2020)。https://doi.org/10.1186/s12870-020-02681-0
收到了:
接受:
发表:
关键字
- 食品级大豆
- 蛋白质
- 蔗糖
- 种子重量
- 连锁分析
- 候选基因