摘要
背景
芍药属decomposita是中国特有的植物,具有重要的观赏、药用和经济价值,被认为是濒危植物。遗传多样性和种群结构很少被描述。目前还没有保护管理计划。
结果
本研究利用16对SSR引物对其遗传多样性和群体结构进行了分析。共获得122个等位基因,平均每个位点有7.625个等位基因。期望杂合度(He)的变化范围为0.043 ~ 0.901(平均0.492)。中度遗传多样性(He= 0.405),以丹巴为遗传多样性中心。曼特尔测试显示,种群间的地理距离和遗传距离呈正相关(r= 0.592,P= 0.0001),表明与距离隔离模型的一致性。分子方差分析(AMOVA)表明,主要的分子方差存在于群体内(73.48%)而不是群体间(26.52%)。贝叶斯结构分析和主坐标分析(PCoA)支持种群划分为3个聚类。
结论
这是第一次研究的遗传多样性和群体结构p . decomposita利用SSR。提出了三个管理单元作为保护措施。研究结果将有利于该物种的保护和开发利用,为其进化和系统地理学的进一步研究提供理论依据。
背景
属芍药属L.(芍药科)包括32种木本和草本植物,主要分布于北半球。芍药属分为三个部分:Onaepia,牡丹,芍药属[1,2].的牡丹部分包括八个中国本地及特有种[2],在汉语中通常被称为牡丹或牡丹。在中国,牡丹花被认为是“花中之王”,这种植物因其药用价值和观赏价值而受到珍视。1,3.].牡丹种子中含有脂肪酸,可提取籽油,因此牡丹已成为重要的木本油料作物。4].
芍药属decompositaHandel-Mazzetti是来自牡丹部分。主要分布在四川西北部的偏远山区,是中国本土特有的,分布零星,分布狭窄,种群规模小。它生长在稀疏的Cupressus chengiana海拔2000-3100 m有森林、幼次落叶阔叶林和灌丛n= 10条染色体。它是由昆虫异花授粉的[5]并通过种子繁殖[6].在过去,p . decomposita由两个亚种组成:p . decomposita无性系种群。rotundiloba和p . decomposita无性系种群。decomposita[7,8,9].基于形态学特征和分子数据,它们现在被认为是独立的物种[2,10,11].
p . decomposita是一种著名的观赏花,因为它的花大,艳丽,色彩鲜艳,芳香四溢。因此,当地人收集植物用于观赏园艺。它也是一种传统的药用植物,因为它的根皮(“丹皮”)被用作传统的中药,具有多种治疗特性,例如清热、凉血、活血、化瘀[12].它最近被认为是一种重要的木本油料植物。籽粒平均含油量为32.23±1.96%,主要由7种脂肪酸组成。大部分油(91.94-93.70%)由不饱和脂肪酸组成,其中亚麻酸占40.45-47.68% [13].从种子中提取的油可以用作油脂化学品、化妆品和药物。14].因此,p . decomposita它不仅是一种观赏植物,而且是一种重要的药用植物,是一种有价值的木本油料作物。
由于栖息地破坏、种子过度采收、中药误用根皮以及再生能力自然较差等多重威胁,p . decomposita自然生境日益破碎化,自然种群规模和植物个体数量急剧减少,导致遗传资源大量流失。目前,大多数种群规模小,分散,分散,增加了近亲繁殖的可能性和遗传漂变的可能性。此外,种子产量低、幼苗更新困难以及缺乏特定的长距离种子传播机制导致种群更新能力差,因为许多群落缺乏幼苗和树苗。根据其分布、生物学特征和生存状况,p . decomposita已列为濒危植物(英文)[15,16].因此,保护这一物种至关重要。遗传资源保护和植物育种计划需要对濒危物种的遗传多样性和遗传结构进行评估[17,18].然而,由于缺乏遗传背景知识,很难制定保护策略。
分子标记的使用可以精确地估计遗传多样性。过去,研究人员利用扩增片段长度多态性(AFLP)、序列相关扩增多态性(SRAP)、简单序列重复序列(ISSR)和随机扩增多态性dna (RAPD)等多种分子标记来研究Section中物种间的遗传关系牡丹[19,20.,21,22,23].与AFLP、SRAP、ISSR和RAPD相比,SSR标记具有共显性、分布广、高可转移性、高多态性、高再现性和高可靠性等显著优势,且成本相对较低[j]。24,25],因此它们通常被认为是理想的分子标记。它们已被广泛用于研究不同植物物种的遗传多样性、种群结构和遗传关系[26,27,28,29,30.],包括牡丹[31,32,33].
到目前为止,对p . decomposita一直局限于物种间的遗传关系和issr的遗传多样性[34],没有研究探索SSRs的遗传多样性,也没有研究探索这种重要的木本油籽物种的遗传关系或群体结构。没有制定育种计划,从中选择最佳的种质资源或资源保护策略,阻碍了植物的保护p . decomposita。从而,准确地认识种群结构和遗传多样性p . decomposita是迫切需要的。
因此,鉴于其在医疗、工业和观赏方面的应用价值,对该植物进行了遗传研究。本研究首先选取了16对多态性SSR标记,通过群体间和群体内的分子变异分析,确定了遗传多样性和群体结构,为制定合理的种质资源保护管理策略、资源配置和未来的指向性育种策略提供了重要信息。
结果
SSR标记多态性
在本研究中,16个多态微卫星位点共122个等位基因[35,36,37,38,39,40,41,42,43)(表1)在11个自然种群的258株植物中扩增。每个基因座观察到的等位基因数目(N一个基因座间差异较大,从2个等位基因(PSMP2)到20个等位基因(PAG1)(平均= 7.625)。每个基因座的有效等位基因数目(Ne)从1.045 (PSMP2位点)到9.929 (PAG1位点)不等(平均= 3.208)。观察到的每个位点杂合度(Ho)范围从0.027 (WD09位点)到0.992 (73A位点)(平均= 0.385),而期望杂合度(He)范围为0.043 (PSMP2位点)~ 0.901(PAG1位点),平均0.492。多态信息内容(图片值)的变化范围为0.042 (PSMP2位点)~ 0.891 (PAG1位点),平均值为0.456。共有21个私人等位基因(NP除M2和M4外,其余9个群体均通过16个标记鉴定出(表5)1).
在位点水平上,遗传分化系数(F圣)和基因流动(N米)由F-各基因座的统计量差异显著。群体间遗传分化的配对比较表明F圣和N米分别为0.430 (50F, R位点)和2.735 (PSESP5位点)。遗传分化系数(F圣),估计16个位点的近交系数为0.193(范围从PSESP5位点的0.084到50F, R的0.430)。F是)为0.038(表1)2).
群体遗传多样性
在种群水平上,遗传多样性指数(以PPL,N一个,Ne,我,Ho,He,F)在不同的种群中有所不同p . decomposita,如表所示3.。平均而言,多态基因座(PPL)在11个种群中均有较高的阳性率(80.68%),其中DB1在68.75% ~ 93.75%之间,多数种群(10/11)≥75%。观察到的等位基因数目(N一个),从2.563 (M4)到4.813 (DB2)不等,平均值为3.637。有效等位基因数目(Ne)的值为2.322,从1.811 (M1)到2.813 (DB2)不等。平均杂合度(He)和观察到的杂合性(Ho)的平均值为0.329 (M1) ~ 0.538 (DB1)和0.314 (M2) ~ 0.464 (DB2),平均值分别为0.405和0.394。香农信息指数的平均值(我)在0.580 (M1)至1.017 (DB1)的区间内为0.777。固定指数(F)平均为0.032,在种群水平上的范围为- 0.160 (JC5)至0.154 (DB1)。大多数位点符合Hardy-Weinberg平衡(HWE),但个别种群不完全满足HWE,特别是种群DB2和M3中有许多位点偏离HWE(分别为7个和8个),表明种群结构为泛型。基因座56A和73A在所有人群中均偏离HWE。
群体间的遗传分化和基因流动
基因分化的差异(F圣)是极显著的(P< 0.001),从0.041(在JC1和JC2之间)到0.234(在DB2和M2之间)不等,平均值为0.098 (P< 0.001;表格4),根据16种标记在11个种群中进行测量。相反,基因流动的值(N米),从0.820 (DB2和M2之间)到5.890 (JC1和JC2之间)不等,平均值为2.781(表2)4).
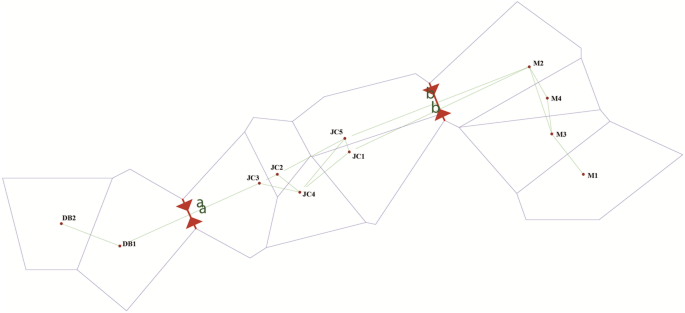
Nei’s遗传距离通过两两比较计算得到,基于SSR标记的Nei’s遗传距离从0.058 (M2与M4之间)到0.462 (DB2与M2之间)不等,平均值为0.178,大部分两两遗传距离发生在0.1 ~ 0.3范围内(表1)5).进行的曼特尔试验p . decomposita表明种群之间的地理距离和遗传距离呈正相关(r= 0.592,P< 0.001)。1),符合IBD(距离隔离)模型。AMOVA结果表明,81.70%的总分子变异是由区域内差异引起的,其余的(18.30%)发生在区域间(P< 0.001)。在群体水平上,73.48%的总分子变异主要来自群体内的个体分化,其余的(仅26.52%)来自群体间的分子变异P< 0.001)。当将总分子方差分成三个层次时,AMOVA分析显示,最大分子方差的比例(70.61%)仍然来自群体内的遗传分化(P< 0.001),而13.39% (P< 0.001), 16% (P< 0.001),分别为区域间遗传分化和区域内群体遗传分化导致的总分子方差(表2)6).根据的矩阵确定区域间推断的基因流屏障的位置F圣值(无花果。2).
基因流动的障碍。灰线对应种群之间的假设边界,并用相应的代码标记。带箭头的红色实线表示基因流动的障碍,种群缩写如表所示7
群体结构和遗传关系
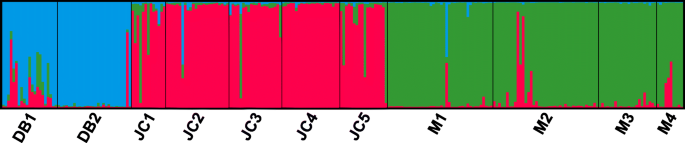
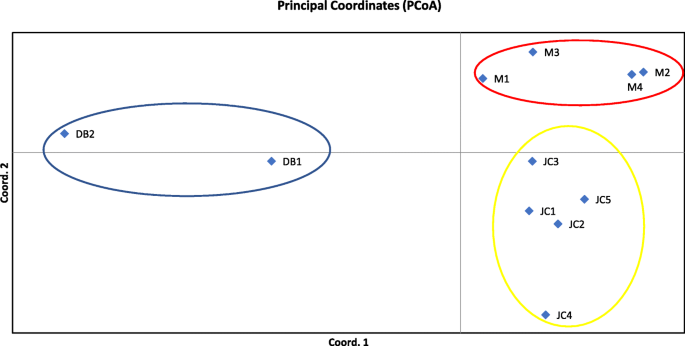
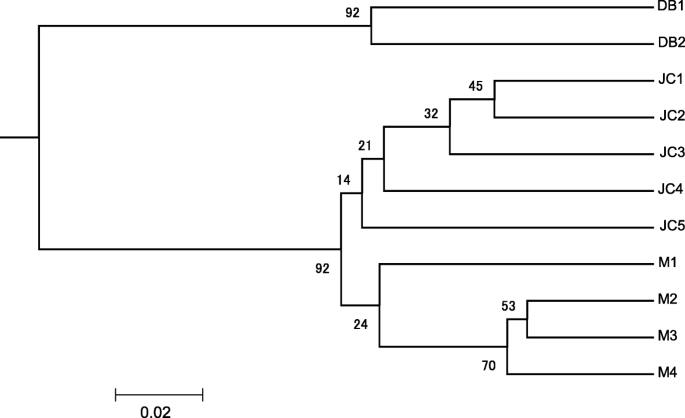
当Δ时,最佳遗传簇数为3K最大值是多少K= 3(图S1).因此,所研究的所有11个种群被分成三个不同的遗传簇(图2)。3.).聚类1采集丹巴县2个种群的49株单株,聚类2采集金川县5个种群的97株单株,其余112株采集梅尔康县4个种群的单株分配到聚类3。很明显,这三个遗传簇与PcoA中鉴定的簇相同,代表了自然分布p . decomposita。根据群体间的遗传距离进行主坐标分析(PCoA),揭示了如图所示的遗传结构。4。3个主要坐标轴的方差百分比为76.66%(轴1-50.71%,轴2-16.95%,轴3-9.00%)。此外,PCoA的结果与结构分析的结果一致,支持UPGMA聚类树,如下所述。
UPGMA树形图是根据Nei的遗传距离值构建的,它准确地反映了种群间和种群内的遗传关系。UPGMA树显示,11个种群可分为两个主要集群:1和2(图2)。5).聚类1包括DB1和DB2 2个种群,聚类2由其余9个种群组成,聚类2又分为2个短分支:金川县的5个种群(JC1、JC2、JC3、JC4和JC5)为1个短分支,马尔康县的4个种群(M1、M2、M3和M4)为1个短分支。
讨论
重要的是保持自然种群的遗传多样性,以确保一个物种的持续生存、适应和进化潜力[44].传统上,植物形态和生理性状的差异被用来评价多样性。然而,由于这些性状在不同的环境条件下并不稳定,因此这些方法只能获得有限的信息。最近,一系列DNA分子标记技术已被用于分析牡丹,包括使用RFLP [21], rapd [45], issr [46]和AFLP标记[47].然而,这些研究主要集中在研究种间或野生种间的系统发育关系,普遍认为需要更多的分子标记来进行遗传研究芍药属物种。SSR是群体遗传学研究中最实用的分子标记,因为它可以测量共显性等位基因并显示出高水平的多态性。本研究首次研究了植物的遗传多样性和种群结构p . decomposita通过微卫星标记,对保护、管理和更好地了解其遗传关系具有重要意义。
遗传多样性
遗传多样性的差异可能是少数因素造成的,例如,一个物种的生活史或地理特征[48].一般来说,与广泛分布的物种相比,不广泛分布的特有物种的遗传多样性较少[49],通常是因为它们的种群数量有限,而且由于它们与其他种群隔绝,所以它们适应了它们特定的栖息地[50].
本研究表明p . decomposita是温和的(Ho= 0.394,He= 0.405)芍药属物种,即使它是稀有和濒危物种。与以往的野生树牡丹研究相比,本研究中观察到的遗传多样性参数略低于野生树牡丹p . jishanensis(Ho= 0.446) [31),p . rockii(Ho= 0.459,He= 0.492) [33,但高于p . ostii(Ho= 0.343,He= 0.321) [51],p . jishanensis(He= 0.340) [31],p .成为(Ho= 0.334,He= 0.369) [52),p . ludlowii(Ho= 0.014,He= 0.013) [52].ISSR标记遗传多样性分析表明p . decomposita这并不高[34],也低于本研究的结果。
的遗传多样性水平p . decomposita(He= 0.405)均低于两种“特有种”(He= 0.420)及“广泛分布”的物种(He= 0.620) [48].造成这一结果的可能原因是,其分布范围零星狭窄,种群规模小,种群间空间距离大,限制了种群间传粉,导致自交和近交,遗传多样性可能较低。
目前的分析方法已大大提高了对遗传多样性的认识p . decomposita,多态性水平在不同种群之间存在差异。在本研究中,遗传多样性(我,Ho,He,图片)在种群水平上较为均匀,且种群DB1和DB2相对高于其他种群,这与丹巴人为干扰程度低、种群规模大有关。因此,丹巴是该物种主要的遗传多样性中心。固定指数(F)结果显示,3个居群(JC1、JC4、JC5为负值)的杂合子数量过多,表明有近交现象,其余8个居群(正值)的纯合子数量过多,表明有近交现象。平均正近交系数(F是)值(0.051)表明纯合子过多p . decomposita(表3.).
研究结果加强了一个假设,即在一个狭窄的分布范围内的濒危植物通常是可再生的。遗传变异的减少可能意味着对不断变化的环境的适应能力下降,从而导致灭绝的危险增加和近亲繁殖的增加[44,53].
基因流动与遗传分化
基因流和遗传分化系数这两个重要参数被用来评估群体的遗传结构[54].基因流与遗传分化系数呈负相关[55].
基因流动是一种基本的微观进化现象,它阻止种群间的遗传分化,影响遗传多样性的维持[56,57].许多濒临灭绝的植物是孤立的,狭窄地分布在几个小种群中,可能是以前广泛分布的物种的残余,这些物种有大量连续的种群[56,58].在目前的研究中,基因流(平均)N米值)之间p . decomposita居群> 1,理论上可以防止遗传漂变导致的遗传分化[59].遗传漂变尚未成为影响遗传结构的主要因素p . decomposita。然而,p . decomposita种群现在受到碎片化和破坏的影响,在大多数种群中发生遗传交换。由于这些原因,再加上自然种群在空间上相距遥远(被山脉和河流屏障隔离),遗传漂变可能逐渐发生。
尽管多样性似乎主要发生在种群内部,但种群间的遗传分化除了DB2与金川和马尔康种群间的遗传分化程度较高外,大部分发生在中低水平(表1)4),根据Wright提出的尺度[60].的意思是F圣表明该物种种群间存在适度的遗传分化。利用Barrier 2.2对传播障碍进行识别,发现不同地理区域间复杂地形对基因交换的抑制作用。
AMOVA结果(P< 0.001)也支持种群分化。AMOVA分析表明,种群间和种群内存在分子变异,种群内而非种群间存在较大的分子变异,这与中国其他异花授粉物种的情况相同芍药属[33,51,52]和其他使用ISSR标记的研究[34].一般来说,在异交和长寿植物中,它们的大部分遗传变异存在于群体内,而自交植物的大部分遗传变异保持在群体内[48].
群体结构和遗传关系
用于检测遗传多样性和种群结构的方法多种多样[61,62,63,64,65].建议将三种有效的技术结合起来,因此我认为PCoA, Structure和UPGMA分析的结合能够产生可靠的结果。UPGMA能够阐述直观的关系,尽管它不能完全分类人口。相反,Structure软件可以客观地对种群进行分类,并制定育种计划。因此,该方法被认为是最适合的种群分类方法。
本研究通过UPGMA聚类分析,将来自3个不同地区的11个居群划分为2个聚类,表明这些地区存在两个不同的遗传群。结构的结果清楚地表明,采样地点表现为三个簇,其中有一些混合个体的例子。这些混合的迹象表明,基因流动可能在某些地方仍然存在(这被更大的N米对某些种群的估计)。这表明Structure软件的分析是可靠的。此外,PCoA结果与Structure结果一致,支持UPGMA聚类树。
此外,种群间的遗传关系反映了种群的自然地理位置,这得到了使用Mantel检验构建的IBD(距离隔离)模型的支持。这个IBD模型p . decomposita表示正相关(r= 0.592,P群体间地理距离和遗传距离之间的差异< 0.001)。遗传分化的差异主要是由于地理屏障隔离了不同的基因库。花粉流动效率低、种子传播不密切和发芽率低是导致三种不同类型植物生长的潜在原因p . decomposita基因池。
原位种群和非原位种群的保护
了解种群的遗传多样性、结构和基因流动对制定适当的管理和保护策略至关重要。用于再引进的种群资源,包括繁殖材料和种质收集,必须在遗传变异方面是最佳的。
遗传资源的收集和保存管理必须保证大多数现有变异得到保存。保护种群间的多样性必须集中于保持遗传上最独特的种群,而保护种群内的多样性必须保护不因遗传漂变而丧失多样性的大型核心种群[66].在…的情况下p . decomposita,保护不仅要考虑种群之间的地理距离,还要考虑不同集群的存在及其不同的生长生境。在每个集群中,必须根据遗传多样性水平、种群再生状态和威胁程度来选择种群保护的优先级。在每个群体中建立几个种群的大型储备,可以保证基因库的样本,可以包含所有种群中存在的独特性和多样性。
遗传多样性对于一个物种保存潜在的进化能力以应对不断变化的环境尤为重要[67].维持遗传多样性和进化潜力是濒危物种保护管理计划的首要目标[68,69].因此,濒危和珍稀植物种群内部和种群之间的遗传变异信息在制定保护和管理策略的过程中起着重要作用[70].因此,我建议三个自然分布区对应三个保护管理单元。鉴于目前的情况是种群数量迅速减少,它们的自然生境受到极端威胁,必须采取就地和移地养护行动。所有种群,特别是遗传多样性高或遗传差异大的种群,都应受到保护。就地保护被认为是保护濒危植物最有效的方法,通过就地保护可以在自然栖息地保护整个基因库。由于栖息地的破坏和环境的波动,小种群更有可能灭绝。为了尽可能地保存遗传变异,必须就地保存所有植物单株和种群。传统的保护方法主要集中在原地保护,如改善再生、控制过度放牧和保护自然栖息地,可能足以维持种群的规模。因此,防止种群的遗传同质性是至关重要的。必须立即实行就地保护,在核心分布区划定和引进保护区,并严格禁止野生采伐p . decomposita。种群DB1和DB2具有较高的遗传多样性,必须优先进行原位保护。先前的许多研究表明,杂合性是确保种群适应性和适应潜力的最佳方法[71].然而,在包括DB1、JC3和M2在内的一些群体中发现了明显的杂合子缺陷,这可能是群体片段近交的结果。
的数量p . decomposita由于放牧(M2, JC5, DB1, DB2)、过度采收(M2, M3)、滥采种子(JC2-5)、靠近村庄、农田和果园生长(JC2-5)或被城市扩张破坏的地区(M2, M4)而面临栖息地破坏、丧失或破碎化的问题。鉴于这一挑战,除了就地保护外,非常建议在野外和实验室为每一个需要保护的濒危植物种群建立非就地基因库[72].保育策略p . decomposita在采取保护措施时,应以保护检测到的3个遗传簇为目标,并考虑到具有私有等位基因的群体(M2和M4除外)。种群DB1和DB2具有相对较高的遗传多样性,必须成为迁地保护的具体目标。由于群体间的遗传分化程度较低,每个群体可能代表一个物种遗传变异的很大组成部分。因此,可以设计种子收集策略,在不同遗传簇的自然地理分布范围内尽可能多地收集各种群的种子种质资源库,并利用植物组织培养技术对种质资源进行保存。在迁地保护过程中,必须对遗传差异较大的群体进行人工杂交,以迅速提高杂合性。从田间采集的种子进行移地栽培后,应将树苗引入源地。综上所述,保护珍贵的遗传资源应结合原位保护和非原位保护。
结论
这项详细的遗传信息研究提供了遗传多样性和种群结构的第一手资料p . decomposita,这有利于制定保护和管理濒危植物的措施。自然居群保持中低水平的遗传多样性、高水平的基因流动和低水平的群体间遗传分化。11个自然种群被划分为3组/群,可作为3个保护管理单位。这些种群是未来育种计划和保护策略的宝贵遗传资源。这是第一次发现遗传多样性p . decomposita研究结果为种质改良和亲本选择制定育种策略提供参考。
本研究中所使用的标记可用于研究植物的种群结构、遗传多样性、种质收集和保护策略p . decomposita。这些标记提供了有关该物种遗传结构的重要信息,对该物种未来的改良和育种计划具有重要意义。通过SSR分析作物的遗传多样性、群体结构和群体间的亲缘关系,将有助于作物育种、种质资源管理和保护。这些结果为今后研究遗传多样性、保护、管理和研究计划提供了重要的参考资料。
方法
植物材料
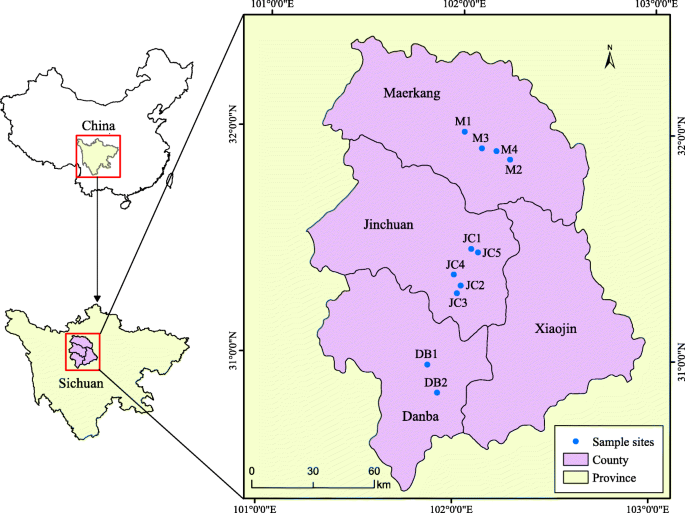
本研究使用的植物材料均采自野外,并获得了采集样本的许可。植物材料的收集也符合机构、国家或国际准则。共采集了11个自然居群的258株单株p . decomposita在开花季节之前的2017年,几乎覆盖了中国的整个区域分布。从每个种群中取样10 - 40株相距至少10m的单株。抽样的详情列于表中7和无花果。6。在野外分别取样新鲜、嫩嫩和健康的叶子,然后立即放入塑料中。将袋子密封,用显色硅胶干燥,保存在- 20°C,直到分离出DNA。本研究所用样品的正式鉴定由王世权完成。代金券标本存放于海南师范大学植物标本室。
DNA提取及PCR扩增
使用植物基因组DNA试剂盒(Tiangen Biotech,北京,中国)按照制造商的说明,从每个样品的叶片中提取总基因组DNA。在1%琼脂糖溶液中分别用分光光度法和凝胶电泳法测定DNA浓度和质量。将提取的DNA稀释至工作浓度为50 ng/μl,然后在- 20℃保存至需要时使用。选用已有文献记载和开发的牡丹引物进行筛选。这些引物在12个有代表性的样品上进行筛选,初步筛选后得到16对多态性微卫星引物[35,36,37,38,39,40,41,42,43[源自genomic, Table1),选择产生高度多态性和高水平扩增的基因进行后续分析。
用5 μl 2 × Taq PCR MasterMix (0.1 U/μl Taq DNA Polymerase)、dNTPs各0.5 mM、Tris-HCl 20 mM、KCl 100 mM、MgCl 3 mM的总容积为10 μl进行SSR-PCR扩增反应2),每次加入基因组DNA模板1 μl (50 ng/μl), ddH 3 μl2O,每个引物1 μl[用5 '荧光标记(TGTAAAACGACGGCCAGT, FAM, HEX, TAMRA)标记]。PCR扩增使用Bio-Rad热循环仪(Applied Biosystems)进行,采用两种不同的循环方案,如下所示:95°C预变性5分钟,然后95°C变性30秒,62-52°C退火30秒(每个循环下降1°C), 72°C延伸30秒。2.在95℃下变性30秒,在52℃下退火30秒,然后在72℃下延伸30秒,最后在72℃下延伸20分钟,共25次循环。所有PCR产物在ABI 3730XL DNA分析仪上用毛细管电泳进行基因分型。利用GeneMarker Version 4.0 (Softgenetics, USA)软件对所有基因座的等位基因相对于内部产品尺寸标准LIZ 500进行评分。
数据分析
多态信息内容(图片)用Cervus 3.0软件计算[73],具有等位基因丰富度(一个r)和种群内近交系数(F是),使用FSTAT 2.9.3.2软件计算[74].多态位点的百分比(PPL)、私人等位基因数目(NP)、观察到的等位基因数目(N一个)、有效等位基因数目(Ne)、香农资讯索引(我),观察到的杂合度(Ho),期望杂合度(He)、固定指数(F)、遗传分化系数(F圣)、基因流动(N米)、Nei’s遗传距离(NGD)、遗传同一性(NGI)、F-统计、Hardy-Weinberg平衡(HWE)、Mantel检验、分子方差分析(AMOVA)和主坐标分析(PCoA)用GenAlEx 6.5软件进行计算[75].在structure 2.3.4软件中采用贝叶斯聚类分析方法对群体遗传结构进行分析[76].总共十次独立运行(K= 2-10),运行长度为1 × 105马尔可夫链蒙特卡罗(MCMC)在1 × 10的熟透期后重复5具有相关等位基因频率的混合模型的迭代。Δ的K方法(77来选择最合适的K值和最优遗传簇数在Structure Harvester V6.0软件上的应用[j]78].利用philips V3.67软件对1000个bootstrap重复进行聚类分析,得到了一种带算术平均值的非加权对群方法(UPGMA)树形图[79的遗传距离,然后用来评估种群之间的遗传关系。
BARRIER 2.2版本使用Monmonier最大差分算法[80]探索种群间遗传不连续性最大的地理位置。使用该程序根据地理坐标绘制采样位置,并通过在种群对遗传距离矩阵中评估最大值在地图上指示障碍。
数据和材料的可用性
16条多态微卫星引物的测序数据在文中列出,其他DNA序列未应用于本研究。
缩写
- 妊娠:
-
扩增片段长度多态性
- AMOVA:
-
分子方差分析
- ISSR:
-
简单序列重复
- PcoA:
-
主坐标分析
- RAPD:
-
随机扩增的多态DNA
- RFLP:
-
限制性片段长度多态性
- SRAP:
-
序列相关扩增多态性
- 苏维埃社会主义共和国:
-
简单序列重复
参考文献
斯特恩FC。属的研究芍药属。伦敦:皇家园艺学会;1946.
洪德耀。世界牡丹:分类与植物地理学。伦敦,圣路易斯:皇家植物园邱园出版社,邱园和密苏里植物园出版社;2010.
程财政年度。牡丹品种群选育及品种体系研究进展。植物学报,2007;1(1):89 - 104。
张玉玲,韩晓霞,牛丽霞,张健,何丽霞。9种野生牡丹种子油中脂肪酸含量分析。中国粮油协会,2015;30:72-9。
杨勇,罗金涛,张丰峰,宋红霞,刘国良,曾晓玲。花性状及育种体系研究芍药属decomposita。植物资源与环境学报,2015;24(1):97 - 104。
程飞,李建军,陈大志。中国野生牡丹树种的自然繁殖特性。植物学报。1997;24:180-4。
洪达元:关于芍药属decompositahand.-Mazz。动物学报。1997;52:957-63。
洪德耀,潘凯。属植物的分类历史及修订芍药属教派。牡丹(芍药科)。植物学报,1999;37:351-68。
洪德耀,潘凯,特兰约。中国植物志。北京:科学出版社、密苏里植物园出版社;2001.
香港DY。芍药属rotundiloba(洪德耀)洪德耀:芍药(芍药科)研究的新进展。[J] .生态学报,2011;49(4):464 - 467。
周立林,邹晓华,周志强,刘健,徐超,余健,等。多种野生牡丹产生了“花之王”,牡丹安德鲁斯。中国科学院学报(自然科学版);2014;31(1):391 - 391。
李康,周宁,李海燕。牡丹花和牡丹种子的成分及功能研究。粮食资源开发,2012;33:228-30。
杨艳,刘建军,曾晓玲,吴艳,宋红霞,刘国良。几种野生油菜种子油脂肪酸组成的比较研究芍药属decomposita。植物学报,2015;42(2):1807 - 14。
韩建刚,李晓青,刘志,胡永华。牡丹作为油料植物的潜在应用。中国粮油学报,2014;27:21-5。
洪达元,周立林,何晓军,袁建辉,张玉玲,程飞,等。野生芍药品种现状及保护专题。生物工程学报,2017;25(5):781 - 793。
秦汉,杨勇,董世义,何强,贾勇,赵琳,等。中国高等植物濒危物种名录。生物工程学报,2017;25:696-744。
Cohen JI, Williams JT, Plucknett DL, Shands H。非原位植物遗传资源保护:全球发展与环境问题。科学。1991;253:866 - 72。
Ouborg新泽西。整合基因组时代的群体遗传学和保护生物学。生物通报,2010;6:3-6。
引用本文:林启波,周志强,赵霞,潘凯,洪达元芍药属教派。牡丹直流。基于Adh基因家族的DNA序列。植物学报。2004;31:627-32。
孟磊,郑国光。中国野生种与栽培品种的系统发育关系分析芍药属教派。牡丹使用RAPD标记。林业科学,2004;40:110-5。
赵鑫,周志强,林启波,潘凯,洪达元。植物种间关系的分子证据芍药属教派。牡丹:甘油-3-磷酸酰基转移酶(GPAT)基因PCR-RFLP和序列分析。植物学报,2004;42(2):236 - 44。
赵鑫,周志强,林启波,潘凯,李美明。的系统发育分析芍药组。山(芍药酶)基于多个DNA片段和形态学数据。[J] .生态学报。2008;46(6):563 - 572。
周志强,潘凯,洪达元。野生牡丹品种关系与栽培牡丹起源研究进展。植物学报。2003;30:751-7。
Agarwal M, Shrivastava N, Padh .分子标记技术及其在植物科学中的应用进展。植物学报,2008;27:617-31。
Varshney RK, Graner A, Sorrells ME。植物微卫星基因标记:特征与应用生物技术进展。2005;23:48-55。
Kumar GS, Singh R, Choudhury DR, Bharadwaj J, Gupta V, singded A.鸡腿遗传多样性和种群结构研究(辣木属鉴定),利用形态和SSR标记。作物学报,2014;60:316-25。
Aboukhalid K, Machon N, lambourdire J, Abdelkrim J, Bakha M, Douaik A,等。濒危物种遗传多样性及种群结构分析牛至属植物紧统来自摩洛哥,利用SSR标记:对保护的启示。生物保护学报,2017;22(2):172 - 82。
李建军,李建军,李建军,等水松baccata:支持波兰保护和恢复计划的案例研究。生态学报,2018;39(9):1249 - 1249。
倪建林,朱国光,王晓峰,徐勇,孙志明,陈建辉,等。苎麻的遗传多样性与群体结构(苎麻l .)。作物学报,2018;33(5):344 - 344。
吴峰,张元元,马建新,罗凯,狄海燕,刘振平,等。本属植物遗传多样性及群体结构分析Melilotus。作物学报,2016;35(5):591 - 591。
徐XX,程飞,冼海龙,彭丽萍。濒危特有种遗传多样性与种群结构芍药属jishanensis及其保护意义。生物化学学报,2016;66:319-25。
于海鹏,程飞,钟毅,蔡春春,吴军,崔海林。植物SSR标记的开发芍药属ostii研究芍药科三株芍药的亲缘关系。植物科学学报,2013;44(4):558 - 564。
袁建辉,程飞,周世林。牡丹的遗传结构(芍药属rockii)和秦岭山脉是导致大量人口破碎化的地理屏障。科学通报,2012;7(7):334 - 355。
童峰,谢峰,曾晓明,何晓军。遗传多样性芍药属decomposita和芍药属decomposita无性系种群rotundilobaISSR标记检测。西北学报,2016;36(6):1968 - 76。
张建军,舒庆勇,刘泽,任红霞,王立林,凯泽德。牡丹品种鉴定的两种est标记体系。植物科学进展,2012;31(3):391 - 391。
高志明,吴杰,刘泽,王立林,任红霞,舒庆。牡丹微卫星的快速发展及其启示。生物医学工程学报。2013;14(4):886 - 96。
Homolka A, Berenyi M, Burg K, Kopecky D, Fluch S.牡丹微卫星标记牡丹(芍药科)。[J] .中国生物医学工程学报。2010;31(2):444 - 444。
王建新,夏涛,张建民,周世林。牡丹14颗微卫星的分离与特性分析(牡丹).环境保护学报,2009;10:1029-31。
侯晓刚,郭丽玲,王静。油菜EST-SSR标记的开发与鉴定牡丹(芍药科)。[J] .中国生物医学工程学报,2011;22(3):391 - 391。
侯晓光,郭磊,程培平,张建勇。30个新型多态微卫星引物的开发aeonia suffruticosa。杆菌的杂志。2011; 55:708-10。
王大东,马华,张玉玲,段爱华,李文杰,李振华。芍药属(芍药科)表达序列标签衍生的微卫星标记转移到芍药属成为。中国生物医学工程学报,2013;12(2):481 - 481。
Gilmore B, Bassil N, Nyberg A, Knaus B, Smith D, Barney DL, Hummer K.牡丹微卫星标记的下一代测序研究。中国生物医学工程学报,2013;38(4):564 - 564。
蔡春芳。牡丹表型性状的高密度遗传连锁图谱构建及qtl分析。博士学位论文北京:北京林业大学;2015.
Frankham R, Ballou JD, Briscoe DA。保护遗传学概论。剑桥:剑桥大学出版社;2002.
苏鑫,张宏,董琳,张建强,朱小涛,孙凯。RAPD分类与鉴定芍药属rockii栽培于甘肃省的品种。西北学报,2006;26(6):696 - 701。
索志林,张海军,张志明,陈富夫,陈富华。种间杂交的DNA分子证据芍药属rockii和p . suffruticosa基于ISSR标记。云南学报,2005;27:42-8。
刘鹏,王志超,尚峰。植物遗传多样性的AFLP分析牡丹栽培于河南省。植物学报。2006;33:1369-72。
Nybom H.估算植物种内遗传多样性的不同核DNA标记的比较。中国生物医学工程学报,2004;13(3):1145 - 1145。
嗯M,嗯HW。无性系草本植物遗传多样性与群体结构格局蕨麻fragarioidesvar。sprengeliana(蔷薇科)在韩国。生物学报。1999;42:64-70。
张建军。植物种群规模小的遗传与进化结果:对保护的启示。In: Falk DA, Holsinger KE,编辑。珍稀植物的遗传与保护。纽约:牛津大学出版社;1991.3-30页。
彭丽萍,蔡春春,钟艳,徐晓霞,冼海龙,程飞,毛建峰。遗传分析揭示了新兴油料作物的独立驯化起源芍药属ostii是一种栽培历史悠久的牡丹。科学通报,2017;7:534 - 552。
张建民,López-Pujol J,龚霞,王洪峰,Vilatersana R,周世林。喜马拉雅-横断地区牡丹种群遗传动态芍药属subsect Delavayanae。生物化学学报。2018;25(5):564 - 564。
陈建平,陈建平。中国野生动物RAPD基因多态性研究进展Leuca-dendron elimense(变形科):对其保护的启示。生物保护。2000;95:39-48。
Hamrick JL, Godt MJW。植物物种中同工酶的多样性。见:AHD B, Clegg MT, Kahler AL, Weir BS,编辑。植物群体遗传学、育种与遗传资源。Sinauer Associates, Sunderland, MA;1990.43 - 63页。
格兰特V.进化过程:对进化论的批判性研究。科学研究。1986;17:65-98。
M.基因流动与种群结构。真实的洛杉矶,编辑。生态遗传学。普林斯顿:普林斯顿大学出版社;1994.3 - 17页。
姚晓华,叶庆国,康敏,黄宏伟。微卫星分析揭示了濒危树种的种群间差异和基因流动Changiostyrax dolichocarpa(Styracaceae)在中国中部有破碎分布。新植物学报。2007;17(6):472 - 80。
田村a,田村a,田村a,田村a,田村a,田村aTricyrtis ishiiana(缬草科)在日本的遗存种群。植物保护学报,2011;12:491-501。
引用本文:Hamrick JL, Godt MW, Sherman-Broyles SL.木本植物物种遗传多样性的影响因素。新。1992;6:95-124。
《种群的进化和遗传学》第4卷:自然种群内部和种群之间的变异。芝加哥:芝加哥大学出版社;1978.
张鹏,周正成,金国强,范海红,胡海鹏。种源带分配与遗传多样性分析木荷在中国使用RAPD标记。林业科学,2006;42:38-42。
宗晓霞,关金平,顾军,王华,马勇。中国陆生种与国际遗传资源组成的豌豆核心种质群体结构与遗传多样性的分化。[J] .植物资源学报,2009;10:347 - 353。
谭晓军,吴志康,程卫东。关联分析及其在植物遗传研究中的应用。植物学报,2011;46:108-18。
张建军,杨云云,吴超,胡志勇,王荣光,郭玉英,任晓玲。烤烟种质资源遗传多样性、群体结构及亚群体特异性研究。烟草学报,2012;18:21-49。
赖国光,赖志祥,刘文辉,叶伟,林玉林,刘世成,等。基于NTSYS和STRUCTURE的闽中野生香蕉3个自然居群ISSR分析中国作物学报,2014;35:223-31。
Namkoong G.种质资源采集取样。植物科学。1988;23:79-81。
李建军,李建军,李建军,李建军,李建军,李建军。遗传多样性对种群生存的影响。植物学报。2000;2:379-95。
马格勒斯CR,普雷西RL。系统的保育规划。大自然。2000;405:243-53。
罗利奎,李建平,李建平,等。濒危物种遗传多样性及结构分析各种cervina种群及其对保护的影响。生物化学学报,2013;50(5):551 - 561。
李建军,李建军,李建军。保护遗传学:超越维持标记多样性。中华医学杂志。1994;3:423 - 431。
李建平,李建平。遗传多样性与适应性的关系。生态学报,2003;17(2):393 - 398。
海伍德VH,伊里昂多JM。植物保护:老问题,新视角。生物保护学报。2003;13(3):391 - 391。
加里诺夫斯基ST,锥ML,马歇尔TC。修改计算机程序CERVUS如何适应基因分型错误增加了亲子鉴定的成功。中国生物医学工程学报,2007;16(1):1099 - 1096。
Goudet J. FSTAT,估计和测试基因多样性和固定指数的程序(版本2.9.3)。洛桑:洛桑大学,Disponívelem:http://www.unil.ch/izea/softwares/fstat.html(2001年10月22日查阅);2001.
Peakall R, Smouse PE。GenAlEx 6.5:遗传分析excel。用于教学和研究的种群遗传软件-更新。生物信息学。2012;28:2537-9。
Pritchard JK, Stephens M, Donnelly P.基于多位点基因型数据的种群结构推断。遗传学。2000;155:945-59。
李建军,李建军,李建军,等。基于结构的个体聚类数量检测方法研究。中国生物医学工程学报,2005;14(2):444 - 444。
Earl DA, von Holdt BM。结构收割机:一个可视化结构输出和实现埃文诺方法的网站和程序。自然资源学报,2012;4:359-61。
Felsenstein J. phillip (Phylogeny Inference Package)版本3.6.7。西雅图,华盛顿州:华盛顿大学基因组科学系;2007.
Manni F, guard E, Heyer E.(遗传、形态、语言)变异的地理模式:如何使用Monmonier算法检测障碍。生物学报。2004;36(6):773 - 790。
致谢
我非常感谢四川当地相关机构和工作人员在森林内提供研究许可,并帮助收集实验材料。感谢钱朝菊博士的屏障分析。我特别感谢编辑和审稿人的宝贵意见和建议。
资金
本研究得到国家自然科学基金(资助号:31670345、31860085)和海南省自然科学基金(资助号:318MS047、2019RC172)的资助。资助者在研究的设计、数据的收集、分析、解释和撰写手稿方面没有任何作用。
作者信息
从属关系
贡献
S-QW构思研究,设计实验,进行实验,分析数据并撰写论文。作者阅读并批准了最后的手稿。
相应的作者
道德声明
伦理批准并同意参与
由于这项研究是在一种濒危物种上进行的,我确认我遵守了所有相关的机构、国家和国际准则。本研究由国家自然科学基金和海南省自然科学基金资助,主要包括处理这些植物和采集样本。
发表同意书
不适用。
相互竞争的利益
不适用。
额外的信息
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1:图S1。
ΔK在K = 1-10上的分布。
附加文件2:表S1。
各人群私人等位基因综述。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
王,平方。濒危物种的遗传多样性和种群结构芍药属decomposita中国特有物种及其保护意义BMC Plant Biol20.510(2020)。https://doi.org/10.1186/s12870-020-02682-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-020-02682-z
关键字
- 保护策略
- 遗传多样性
- 基因的关系
- 芍药属decomposita
- 人口结构
- 简单序列重复(SSR)