摘要
背景
适当的种质遗传多样性和群体结构信息可以提高植物育种的效率。尼泊尔面包小麦的低生产力(小麦尼泊尔是全球第四大最易受气候变化影响的国家。尼泊尔春小麦的遗传多样性和群体结构尚未见报道。这项研究旨在改进对更多样化和未充分利用的遗传资源的开发,为当前和未来的全球粮食安全育种工作做出贡献。
结果
我们使用基因分型-测序(GBS)对来自尼泊尔的318个春小麦品系进行了表征,其中包括166个地方品种、115个CIMMYT先进品系和34个尼泊尔发布品种。我们鉴定了95k个高质量snp。本地品种的遗传多样性最大,其次是CIMMYT品系和释放品种。虽然我们预计只有3个类群对应这3个种子起源,但种群结构显示了两个大的、不同的亚种群,以及两个较小的、分散的亚种群之间,有显著的混合。主成分分析(PCA)和UPGMA距离聚类验证了这一结果。不同亚居群之间的LD衰减模式不同,范围在60 ~ 150kb之间。我们讨论了20世纪70 - 90年代的种质探索可能错误地收集了外来种质,而不是当地的地方品种和/或收集了自20世纪50年代开始引进外来种质以来已经交叉杂交的材料。
结论
我们认为,在这项研究中,尼泊尔只有一个小麦“地方品种”的子集是真实的。瞄准这些地道的本地品种可能会加速当地的育种计划,以改善这个气候脆弱国家的粮食安全。总的来说,这项研究提供了对尼泊尔小麦遗传多样性的新认识,这可能有助于全球小麦育种计划。
关键信息
本研究对尼泊尔春小麦种质资源的群体结构和遗传多样性进行了研究。这一发现出乎意料,提供了对种质的新认识。
背景
现代植物育种家面临的一个重大挑战是,在应对气候变化对作物生产力造成的影响的同时,需要在人口不断增加的情况下提高作物产量[1].目前世界人口在2019年约为80亿,预计到2050年将超过96亿,这意味着粮食产量必须至少增加33%,才能满足不断增长的需求[2].然而,要实现联合国可持续发展目标所确定的到2030年消除全球饥饿的目标,目前的研发状况似乎无法应对这一重要挑战[3.].虽然增加粮食生产面积的空间有限,但通过遗传改良提高产量是克服未来粮食危机相关问题的关键手段。目前有关气候变化的研究概述了越来越多的热、冷和干旱胁迫对农作物有害[4].这种情况对提高面包小麦(小麦l .)。面包小麦是世界第三大主要谷物作物,2017年全球产量为7.57亿公吨[5].在全球范围内,小麦提供了谷类总热量摄入的41%,在发展中国家占谷类热量摄入的35%,在发达国家占74% [6].总体而言,在膳食摄入量方面,小麦在全球排名第二,大部分作物(68%)被用作食物,约19%用于饲料和生物燃料[7].
与全球情况类似,小麦也是尼泊尔的主要谷物之一。自1960年以来,尼泊尔的小麦种植面积增加了5倍[8如今占全国谷物种植面积的1/5 [9].尼泊尔目前的小麦产量为每公顷2.2吨[9],而美国为3.1吨/公顷[10印度等邻近国家的产量为3.2吨/公顷[10].到2030年,尼泊尔对小麦的需求预计将增长约89万吨[11].然而,尼泊尔在全球最易受气候变化影响的国家中排名第四,尤其容易受到干旱和其他气候相关灾害的影响[12].这种情况表明,尼泊尔可以从小麦育种的进一步进步中受益。为了加快这方面的工作,彻底了解现有小麦种质的遗传多样性和群体结构可能有助于更有效地部署现有遗传资源[13].种质分子特性可以指出群体中独特的等位基因来源,并防止基因冗余的种质在育种项目中被用作亲本[14].此外,这种分析揭示了种质资源过去的地理流动、它们的遗传隔离和/或混合程度[15,16].此外,这种分析可以发现种质护照信息中的潜在错误。在这种情况下,分析尼泊尔现有小麦种群的遗传多样性是有价值的。尼泊尔春小麦的种群结构此前没有报道过。
现代面包小麦通过遗传变异狭窄的供体种杂交进化,造成了遗传瓶颈,导致遗传多样性狭窄[17].狭窄的遗传变异也是驯养过程中瓶颈的结果,加上过去几十年的密集育种努力[18,19].因此,小麦育种者总是对多样化和扩大作物遗传多样性的机会感兴趣。传统农民在低投入条件下种植的地方品种被认为是种质多样性的主要来源之一[20.].现代育种计划的结果是当地适应的精英种质,可用于定向引入特定的等位基因[18,19,20.].在尼泊尔,国家农业遗传资源中心(NAGRC)有数百个地方品种,该中心是尼泊尔的国家基因库,隶属于尼泊尔农业研究委员会(NARC)。自20世纪50年代以来,尼泊尔一直从国际玉米和小麦改良中心(CIMMYT, Mexico)引进优质种质,这些种质构成了尼泊尔国家小麦研究计划(NWRP)发布的品种的基础。
分子水平上的遗传多样性表征得益于基因组序列。面包小麦的基因组很大(约17000 Mb) [21其中约80%由重复序列组成[22,23].六倍体面包小麦参考基因组序列注释于2018年发布[24],覆盖所有21条染色体,包括107,891个高置信度基因。高质量参考基因组的存在为利用现代工具和方法开发小麦遗传资源开辟了道路。基因组序列使高密度全基因组标记的开发成为可能[25].基因测序分型(GBS)就是这样一种基于下一代测序(NGS)的技术,能够产生高密度的全基因组标记[26,27].在各种降低复杂度的方法中,考虑到成本、易于操作和较少的净化步骤,GBS方法更有效[28,29].它大大降低了基因组的复杂性,并为测序创建了更同质的文库。由于异源多倍体和大基因组尺寸是阻碍小麦分子标记发展的两个关键因素,GBS标记的使用为实现当前和未来的基因组探索迈出了一步。GBS标记包括SNP标记,这些标记广泛应用于需要大量标记的遗传研究中,如种群结构的确定、QTL定位、标记-性状关联、基因组选择和基于地图的克隆[30.,31].
本研究旨在通过评估尼泊尔春小麦的遗传多样性和种群结构,为尼泊尔乃至全球的植物育种工作做出贡献。本研究利用GBS衍生的snp对318个春小麦品系(包括地方品种、释放品种和先进育种系)进行了评价。研究的主要目的是:(i)确定尼泊尔小麦遗传材料的遗传多样性和连锁不平衡(LD)面板特征,(ii)确定尼泊尔小麦遗传材料群体结构的基础。
结果
面包小麦基因组间多态GBS标记的密度不同
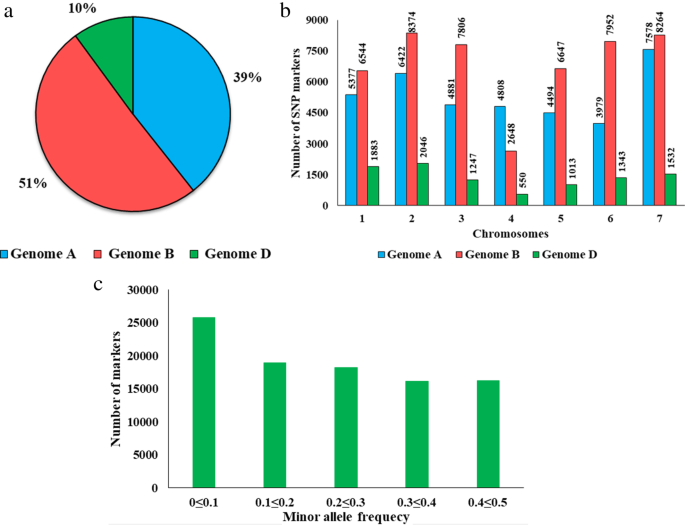
我们对318份资料进行了基因分型-测序(GBS)1:表S1),获得约8亿次阅读,平均每次访问250万次。使用Fast-GBS管道处理原始读取并调用snp。在对缺失数据进行补入后,我们获得了横跨a、B和D基因组的95,388个多态标记的最终数据集2:表S2,图1a, b). SNP标记的最高比例(51%)来自b基因组,其次是a基因组(39%),其余10%来自D基因组。每条染色体的SNP标记数从550 (4D)到8374 (2B)不等2:表S2,图1b).在每个基因组中,每个基因组识别的最低和最高标记数量分别为3979 (6A) - 7578 (7A), 2648 (4B) - 8374 (2B)和550 (4D) - 2046 (2D)2:表S2,图1b).该目录中约26%的snp(25,820)具有可称为罕见的小等位基因(MAF < 0.1)(图1)。1c)。
不同来源的种群在遗传多样性上存在差异
采用两个不同的指标:核苷酸多样性和Tajima’s d来评估NWDP内的遗传多样性。用NWDP的三个组成部分(三个加拿大基因型包括在“释放品种”组中)中的每个组成部分测量该小组的核苷酸多样性。正如预期的那样,本地品种(166个)的遗传多样性最高,其次是CIMMYT品系(115个)和释放品种(37个)(见表)1).
亚群分离与种子来源无关
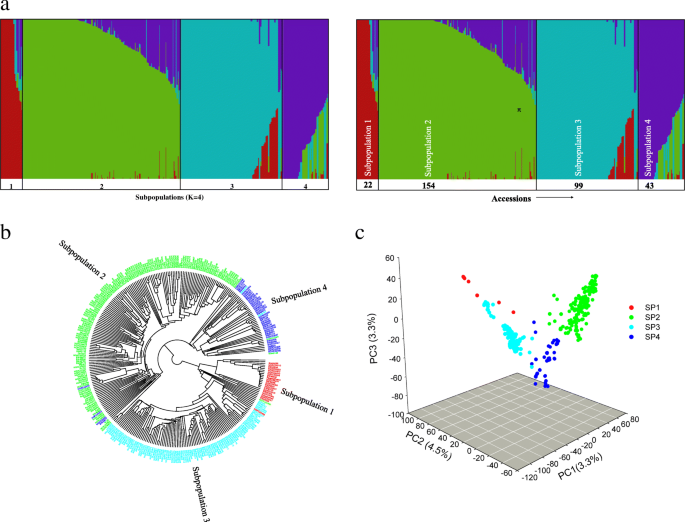
fastSTRUCTURE分析确定4个亚群体(K = 4)是NWDP中使用95 K高质量SNP标记的318份材料的最佳聚类数量(图1)。2a).在这4个亚居群中,亚居群2和亚居群3差异明显,数量较多,而另外两个亚居群(1和4)数量较少。结果表明,亚群体1、2、3和4分别包含22、154、99和43个群体。然而,无论是亚种群的数量还是隶属度的分配都不能很好地反映NWDP所包含的三种隶属度的存在和组成(图1)。2一个额外的文件1:表S1)。
然后使用UPGMA进行聚类分析,并基于亲缘关系或亲属关系的估计。得到的树状图(图5)。2B)是使用估计的遗传距离得到的,该距离提供了每个加入在特定集群和顺序中的位置。聚类与上述种群结构高度一致(图。2a),尽管在某些情况下,分配给一个子群的一个加入者没有与同一子群的其他个体分组(用不同于图中各自亚群的颜色标签表示)。2无花果。2b;额外的文件3.:表S3)。
为验证上述结果而进行的主成分分析证实了两个较大且不同的亚种群以及两个较小且重叠较多的亚种群的存在。2c).本分析使用了三台pc机,其中三台pc机约占变异的14%。结果表明,PC1、PC2和PC3的双标图将基因型分为两个不同的组(亚群2和亚群3),而另外两个组(亚群1和亚群4)则分散在这两个亚群之间。亚群3的亲缘关系更明显,而亚群2和亚群4的亲缘关系略接近,尽管亚群2有亲缘关系。在亚群体2和3中表现为紧密聚集,而亚群体1和4较小且较分散。最小的聚类,即亚居群1,与其他亚居群相比似乎高度分散(图1)。2c)。
连锁不平衡在整个种群中衰减较快
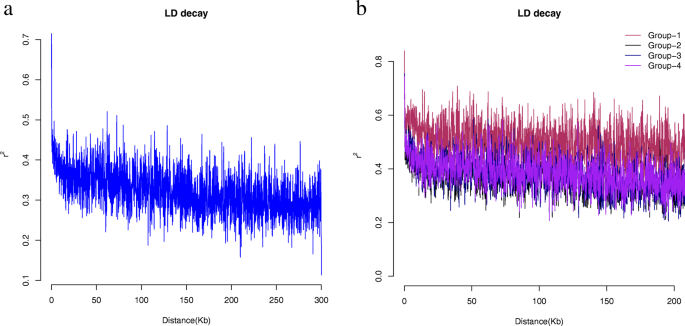
对整个群体和从群体结构分析得到的每个亚群体分别进行LD衰减分析。估计的等位基因频率相关性(r2)与基因座对的物理距离作图,以评估LD衰变的模式。平均而言,劳工处(r2= 0.72)已衰减一半(r2= 0.36),整个种群的物理距离为~ 60kb(图5)。3.一个)。
结果还显示了四个不同亚群体的LD模式的变化(图。3.b).第1亚群体的LD最高(r2= 0.85)其次是亚群3 (r2= 0.75),亚群体4 (r2= 0.75),然后亚群体2 (r2= 0.6)。亚群体1在物理距离大于150kb时,LD下降到一半。同样,在亚群体2中,LD降至其值的一半(r2= 0.3)在约75 Kb的物理距离。在亚群体3中,LD在物理距离约90kb处衰减到一半。亚居群4的LD衰减模式与亚居群3相似,均为~ 90 Kb。因此,结果明显表现出差异r2整个种群和已确定的亚种群之间的价值。平均而言,第1和第4亚群的死亡率更高r2而全居群和亚居群2的值较低r2值。与其他亚居群相比,全居群的LD衰减率(~ 60kb)较低。
讨论
种群结构可能是由大量的种质引进尼泊尔所决定的
本研究使用的小麦材料是尼泊尔春小麦多样性的代表。群体结构分析将尼泊尔小麦多样性研究小组(NWDP)的318个小麦品种划分为4个亚群体(两个大的亚群体和两个分散在中间的小亚群体)。所有使用的三种方法(总体结构、UPGMA聚类和PCA)一致地导致了这个分组。在早期的研究中也观察到使用这些方法分组的一致性[32,33,34].通过fastSTRUCTURE将种群分化为不同的亚种群是基于假设的每个亚种群的基因型的相关频率[14,35,36].类似地,UPGMA聚类根据遗传距离将种群划分为不同的亚种群[37而PCA则说明了基于遗传距离的亚群体分化[38].
根据种子来源,我们推测有3组,第1组包括来自尼泊尔的本地品种,其他的包括CIMMYT系列和商业发行的尼泊尔品种。这种预期是基于这样一种信念:在尼泊尔这个多山的民族国家的不同地区生长的地方品种,在基因上比新引进的现代品种更加多样化[20.,39].然而,与我们的预期相反,所获得的结果并没有产生基于材料来源的如此明显的聚类,而是基于混合物[40]在所有四个亚群体中均可见。第一个主成分只解释了总基因型变异的6.3%,表明种群中存在大量的混合。我们观察到亚群体2拥有最高的CIMMYT品系比例(60%)和释放品种比例(65%),而亚群体3拥有最高的地方品种比例(42%)(附加文件)4:表S4)。类似地,从q矩阵(fastSTRUCTURE中贝叶斯聚类的输出)中观察到的结果显示,在四个子组中,每个子组中都有许多与其他组中的其他个体不相关(相关度频率为> 99%)的条目(附加文件)3.:表S3)。具体而言,在划分为第3亚群的99份资料中,69份资料与其他类群的个体无亲缘关系,其中49份资料为本地人种。结果表明,这49个品种可能是真正的地方品种(补充文件:表S1;额外的文件3.:表S3)。而在亚群体2中,154个品系中有56个与其他亚群体的品系无亲缘关系,其中28个是CIMMYT品系,其次是地方品种和释放的尼泊尔品种。有趣的是,结果显示,具有相同或至少一个或多个共同祖先的一些亲缘聚集在同一组中。例如,CIMMYT系BW49342、BW49351、BW49392、BW49394和BW49954有一个共同的祖先“KACHU”,它们被分组在子种群2中(附加文件:表S1)。同样,被释放的品种Tilottama和拥有共同祖先“VIVITSI”的CIMMYT品系BW48137、BW49108和BW49456被分组在亚群体2中。至于加拿大的属地,这三个属地都没有与西北发展地区的其他属地有任何共同的祖先。加拿大的“诺威尔”被分组在亚群体2中,它的祖先可能与本研究未包括的其他CIMMYT种质有某种联系(附加文件:表S1)。
意外的群体结构(4个亚群体而不是3个;)的结果可能是由于影响种质群体结构的许多因素,如品种的年龄、植物育种者的活动[41,42,地理来源[36,38,43,44],市场等级[37,45和倍性水平[39].从古代起,尼泊尔就开始种植小麦(主要生长在西部山区)[46].但正式的由公共部门进行的小麦育种始于1951年的尼泊尔;Lerma 52是尼泊尔于1960年释放用于商业种植的第一个小麦品种[46].1960年以前,小麦在尼泊尔是一种次要作物;在约10万公顷的小麦种植面积中,主要是本地品种和一些印度品种占主导地位[46,47].但在接下来的几十年里,情况发生了巨大的变化,从20世纪60年代中期开始,大量的半矮秆现代品种被引入尼泊尔,同时政府促进了改良品种的种植[48].尼泊尔的小麦种质资源主要来自国际小麦改良中心、墨西哥和印度[45,46,48]和美国国际开发署[45].从1960年到1990年,小麦种植面积增加了500%以上[8,也被称为“绿色革命”[48].文章还指出,一些在20世纪60年代中期以前广泛种植的小麦品种也是印度和墨西哥小麦育种项目开发的现代小麦品种[8].因此,从20世纪50年代的一次小丰收开始[48],小麦现已成为尼泊尔的主要谷物之一,占全国谷物覆盖率的22% [9].这种情况很可能导致尼泊尔本地品种的种植严重减少。
研究了自1960年以来在该国发布的43个品种,尼泊尔在品种开发方面高度依赖外国种质[46].平均而言,目前尼泊尔采用改良品种的比例约为97% [46,49].2001年之前在尼泊尔公布的35个小麦品种使用了来自22个国家的89个祖先,而这些品种不包括任何尼泊尔种质[45].截至2016年,在尼泊尔释放的小麦品种中,约80%的小麦品种并非尼泊尔起源(附加文件:表S1),而这些释放品种的祖先明显来自美国(13%)、印度(13%)、法国(12%)、阿根廷(6%)和意大利(6%)[50].这一证据清楚地强调了外国材料在尼泊尔小麦种质池中的主导地位。
该研究中心成立于2010年,一直致力于通过对有价值性状的表征、评估和鉴定来维护农业遗传资源。全国小麦研究中心收集和保存的小麦品种约有1700个[51].共开展了18个种质资源收集项目,收集包括小麦在内的不同作物品种,而在尼泊尔西部仅开展了2次小麦遗传资源收集[45这可能是尼泊尔不同地方民族的主要来源。这些遗传资源是从海拔720至3353米的不同海拔收集的。由于尼泊尔崎岖的地形和偏远的农业生态条件造成的困难,这些收集的大部分不是直立的作物,而是来自农民的谷仓[51].因此,在收集的过程中,偶然可能会导致遗传资源的重复,此外,在种质探索过程中收集到的一些护照信息可能具有误导性,即有些收集到的可能不是“真实的”地方品种。这里的论点是,在20世纪70年代、80年代和90年代进行的种质探索很有可能错误地收集了外来种质,并将其标记为当地的地方品种,因为农民已经种植了几十年(在20世纪50年代和60年代初引入外来材料之后)。还有第二个潜在的原因:尽管尼泊尔不是小麦的起源中心之一,但种质的传播和进化与人类迁移是同步的。52].在这种情况下,尼泊尔外来小麦遗传多样性的一个来源可能与农民从尼泊尔遥远的西部和中西部来回于印度西北部的季节性迁移有关,这种迁移对许多家庭来说至少持续了2-3代[53].印度和尼泊尔之间历史上由农民主导的种子共享,加上CIMMYT材料的正式引入,可能加剧了尼泊尔小麦种群结构的扭曲,因为国际小麦育种计划(如CIMMYT)中对印度小麦遗传资源的广泛合作和使用,这是合理假设的结果,认为适应于印度北部的性状也会对尼泊尔有利。后一种说法可以从观察结果中得到证实,在2001年之前尼泊尔释放的35个小麦品种中,有16个品种来自印度,14个来自墨西哥,4个来自尼泊尔,其余1个来自肯尼亚(补充文件:表S1) [45].尼泊尔与中国接壤,一个有趣的问题是中国小麦遗传资源在多大程度上影响了尼泊尔小麦。中国小麦对尼泊尔小麦种质资源的影响尚未得到广泛报道。然而,根据现有资料,至少有一个尼泊尔的放生品种(Kanti, 1997年放生)有一个祖先(FUFAN17)源自中国[46].尽管与印度的南部边境是自由开放的,但喜马拉雅山脉阻断了中国和尼泊尔之间的人员和种子流动,而且两国之间有限的过境点受到更严格的控制。就“本土”和外来种质之间的广泛混合而言,由于种质收集始于20世纪70 - 90年代,而CIMMYT种质引进始于20世纪50年代,最简单的解释是,在收集之前就发生了交叉杂交。所有这些论点都需要验证。
地方人种遗传多样性较高
我们根据种子来源比较了不同组间的核苷酸多样性,发现地方品种的遗传多样性比其他两组(精英系和高级系)更丰富。这些地方民族更有可能拥有一些与生物和非生物抗压能力相关的有价值的等位基因。与现代优良品种相比,地方品种通常表现出更高的遗传多样性[39].地方种族的遗传多样性基本上是由农民的活动、环境因素以及包括随机突变、种群间基因流动和遗传漂变在内的进化力量所塑造的[54].与此相反,现代品种在改良过程中的选择压力、重组率和一些分离畸变导致某些基因的丢失,导致遗传多样性低[20.,39,55].此外,与现代精英种质相比,地方品种受到的选择不那么严格,因此更有机会保持较高的遗传多样性[56].由于长年生长在极低投入的环境中,使得与适应性状相关的等位基因得以自然选择,因此地方品种拥有一些与适应不同压力相关的宝贵基因[20.].例如,由于环境压力和个体选择努力,在一个中国小麦地方种群中观察到较高的遗传差异[57].地方品种促进从远亲处引入新的遗传变异,从而使作物从长远来看得到改善[55包括耐压性相关特征,如干旱[58].例如,克里奥尔小麦本地品种(从欧洲传入墨西哥的本地品种)已被用作不同非生物胁迫(包括干旱)的等位基因来源[59].虽然目前的结果没有显示出高水平的稀有等位基因(Tajima’s D)的存在,但当地品种可能已经适应和选择了一些与尼泊尔小麦生长环境相关的特定性状。
D基因组是预期中多态性最少的基因组
高密度SNP标记的应用目前广泛用于评估遗传多样性、种群结构和各种进化问题[60].在这项研究中,我们在三个小麦基因组中鉴定了95,388个SNP标记。在B基因组中发现了最多的多态性标记(约51%),这与最近对230个小麦资源群体的研究相似[61].此外,在不同的合成六倍体小麦(SHWs)群体中观察到的B基因组中snp数量最高[14,43,62].snp数目最多的是2B染色体,其次是7B和6B染色体。这也与其他对不同小麦群体进行的研究一致[14,63].观察到的D基因组SNPs比例(约10%)较低,但与早期研究一致[64].D基因组内的低水平多态性可能是由于在供体D基因组杂交(即杂交)时出现的遗传瓶颈造成的。Ae。tauschii)进入六倍体基因组,与四倍体小麦祖先(AABB)的基因组进行比较[65].换句话说,六倍体小麦的多样性在野生小麦中所占的比例较低Ae。tauschii与野生的A和B基因组相比另一种解释是,与A和B基因组相比,D基因组中罕见等位基因的比例更高,可能在现代品种发育过程中更容易受到遗传漂变的影响,在D基因组中造成了最近的遗传瓶颈[66].使用gbs衍生SNP标记获得的不同基因组对SNP多样性的相对贡献也与过去使用DArT标记在六倍体小麦上进行的研究结果一致[14,67,68和SSR标记[34,38,41,69].
连锁不平衡(LD)模式的变化可能表明选择压力的水平
LD衰减距离可以指示重组率,决定了关联和QTL定位的精度[35,44,69].多种因素影响种群中等位基因的随机组合,导致LD模式的变化,包括选择、非随机交配、突变、混合、种群大小和遗传漂变[70,71].同样,标记的类型和数量也会影响LD的测量[35],即有限的标记数量导致LD在基因组中的分布分辨率有限[42].最近,大量SNP标记被用于生成高分辨率地图,从而实现对LD的精确估计,以促进遗传资源的开发[44].在本研究中,估计的整个种群和亚种群的LD距离表明,与最近的一项研究相比,LD衰减块更短[40]证明了在322个软红冬小麦群体中LD在大约1 Mb的距离上衰减,这比我们在本研究中观察到的要高得多,尽管群体大小大致相似。这一结果可能是由于尼泊尔小麦种质中存在较高的遗传多样性。此外,我们观察到,与亚人群相比,整个研究人群的LD更低,LD衰减更快,这当然是由于前者的规模更大[72];较小的人口通常有较高的LD [14,44].与这一观察结果一致的是,亚群体1(最小的只有22份)的LD最大,尽管该群体中约50%的资源是地方种族。根据[73],地方品种的等位基因多样性较高,LD衰减距离较精粹系短。与亚居群2相比,亚居群3具有较高的LD和较慢的LD衰减,尽管该群体中本地种族的比例更高(约71%)。在这里,LD值和LD衰减程度的变化也可能是由于遗传漂变和/或遗传材料上的选择压力[36,71,74特别是CIMMYT的品种和尼泊尔释放的品种,它们构成了约50%的种群。
结论和未来展望
本研究为了解尼泊尔春小麦的遗传多样性提供了新的思路。特别是,许多被视为本地品种的品种与CIMMYT先进品种的关联性是一个令人惊讶的结果。这一发现表明,从20世纪50年代到最近几年,通过交叉杂交和/或收集错误,遗传资源的引入显著地改变了目前被标记为尼泊尔本土春小麦的种群结构。这项研究中产生的信息,包括遗传多样性、种群结构和LD,可以指导尼泊尔未来的育种计划。特别是,由于尼泊尔42%以上的农业用地是雨养土地[9仅干旱就造成了20-30%的产量损失[75],预计会因气候变化而恶化[76,77],尼泊尔正在优先发展新的耐旱小麦品种,这将依赖于开发种质多样性。在这种情况下,这项研究表明,尼泊尔的小麦“地方品种”中只有一小部分是真实的。利用这些真正的地方品种可能是利用有用的适应性特征来改善这个气候脆弱国家未来的粮食安全的一条途径。此外,利用这些遗传资源可能有助于全球小麦育种工作,旨在减少由包括气候变化在内的各种因素造成的粮食不安全。
方法
植物材料
研究人员从不同来源收集了318个春小麦品种,并将其命名为尼泊尔小麦多样性研究小组(NWDP)1:表S1)。该小组包括166个尼泊尔地方种族,由尼泊尔国家农业遗传资源中心(NAGRC)提供。国际玉米和小麦改良中心(墨西哥CIMMYT)提供了115个先进育种系:这些系是根据在尼泊尔进行3年(2011-12季节至2013-14季节)田间试验后的表现(包括疾病和粮食产量)选择的。由于cimmyt培育的种质资源对尼泊尔改良品种的池有显著贡献,我们将这些先进育种系纳入了NWDP。截至2014年,尼泊尔国家小麦研究计划(NARC)还提供了34个在尼泊尔发布的用于商业种植的品种。目的是使多样性小组成为尼泊尔春小麦遗传资源的代表性。出于兴趣,该研究小组还包括三种加拿大春小麦基因型,这些基因型可从加拿大圭尔夫大学的小麦育种实验室获得。
DNA提取和基因测序分型(GBS)
根据制造商的协议,使用DNeasy Plant Mini kit (Qiagen, Hilden, Germany)从田间收集的叶片组织中提取每一种小麦的基因组DNA。采用GBS方法进行SNP基因分型太平洋标准时间我/MspI库是按照Poland等人(2012)编写的。在pleforme d’analyses的离子质子测序仪上对多重GBS文库进行单端测序(每个96 plex GBS文库有两个PI芯片)Génomiques[生物研究所Intégrative et des Systèmes (IBIS), Université Laval(魁北克,QC,加拿大)]。
GBS数据分析
离子激流序列读取(50-160 bp)使用Fast-GBS管道处理[27].简单地说,FASTQ文件是基于条形码序列进行多路复用的。对多路复用reads进行修剪,然后根据小麦参考基因组进行定位[78].然后从映射的reads中鉴定出核苷酸变异。然后,如果它们满足以下任何一个标准,就删除变异:(i)它们有两个以上的等位基因;(ii)阅读质量(QUAL)总分< 32;(iii)映射质量(MQ)评分< 30;(iv)读取深度< 2;(v)杂合度为> 50%;丢失的数据为> 80%。缺失数据补入使用BEAGLE v4.1执行[79]由[80].
SNP分布与遗传多样性分析
使用TASSEL v.5.2.48可视化分析基因组中snp的分布[81].利用VCFtools 0.1.2 b估算核苷酸多样性(Pi)和分化度(Tajima’s D)来表征遗传多样性[82].在这些分析中,我们使用了小等位基因频率(MAF)≥0.01和窗口大小为1000 bp的snp。计算所有窗口的平均Pi和Tajima’s D,以获得每个源群体的全基因组平均值。
人口结构分析
基于模型的贝叶斯聚类软件fastSTRUCTURE v 1.0 [83]被用来推断研究小组中的亚群体数量。为了测试子种群的数量(K), fastSTRUCTURE使用默认设置运行100次交叉验证,在318次访问中将K的值从K = 1到10变化。使用fastSTRUCTURE包中包含的Python脚本选择最大化边际可能性的子种群数量。结构图的排序基于fastSTRUCTURE生成的q值。
此外,TASSEL v.5.2.48 [81]使用协方差法从相同的SNP数据中生成主成分(pc)。每个PC解释的变化比例由程序中估计的特征值决定。然后只使用前三台PC创建PC图。材料的颜色编码是基于使用fastSTRUCTURE计算的估计种群结构。
流苏v.5.2.48 [81]还用于估计所有配对组合的基于标记的遗传距离矩阵,使用来自95,388个标记的数据。在R [84]通过使用hclust函数(85].采用无加权算术平均对组法(UPGMA)进行聚类。树状图是用as.dendrogram函数,树状图的自定义使用dendextend[86),circlizeR[的包87].最后,根据使用fastSTRUCTURE计算出的估计人口结构,对每个添加项进行颜色编码。
连杆不平衡衰减分析
为了研究研究群体中的连锁不平衡(LD)衰减,等位基因频率平方相关(r2)通过使用TASSEL v.5.2.48中具有比较意义的1000个排列得到[81].然后,将LD衰减绘制为两者之间的关系r2值和基因组中SNP标记的物理距离。通过种群结构分析,测定了整个种群和四个亚种群的LD衰减。
数据和材料的可用性
本研究中产生的基因型数据(95 K GBS-SNPs)和种群结构数据集可在figshare公开获取:https://figshare.com/projects/Population_Structure_of_Nepali_Spring_Wheat_Germplasm/90923
缩写
- 国际玉米和小麦改良中心:
-
国际玉米和小麦改良中心
- GBS:
-
Genotyping-by-sequencing
- LD:
-
连锁不平衡
- 刑警:
-
尼泊尔农业研究委员会
- NAGRC:
-
国家农业遗传资源中心
- NWDP:
-
尼泊尔小麦多样性小组
- NWRP:
-
国家小麦研究计划
- SNP:
-
单核苷酸多态性
- UPGMA:
-
算术平均的非加权对组法
参考文献
Raza A, Razzaq A, Mehmood S,邹X,张X,吕燕,等。气候变化对作物适应的影响及其应对策略:一项综述。植物。2019;8:34。
联合国。2019年世界人口展望:要点(ST/ESA/SER.A/423):经济和社会事务部。纽约:联合国;2019.
粮农组织。作物前景和粮食状况。季度全球报告。罗马:联合国粮食及农业组织;2018.
联合国政府间气候变化专门委员会。摘要为决策者。见:2014年气候变化:影响、适应和脆弱性。a部分:全球和部门方面。In: Field CB, Barros VR, Dokken DJ, Mach KJ, Strandrea MDM, Bilir TE, Chatterjee M, Ebi KL, Estrada YO, Genova RC, Girma B, Kissel ES, Levy AN, macracken S, Mastrandrea PR, White LL,编辑。第二工作组对政府间气候变化专门委员会第五次评估报告的贡献。剑桥和纽约:剑桥大学出版社;2014.
粮农组织。世界粮食和农业:统计袖珍。罗马:联合国粮食及农业组织;2018.
B . Shiferaw, Smale M, Braun HJ, Duveiller E, Reynolds M, Muricho G.粮食养活世界小麦在全球粮食安全中所起作用的过去的成功和未来的挑战。食品安全内核。2013;5:291 - 317。
粮农组织。粮食展望:全球粮食市场半年度报告。罗马:联合国粮食及农业组织;2018.
尼泊尔稻麦系统中小麦生产的经济学。In: Teng PS, Kropff MJ, ten Berge HFM, Dent JB, Lansigan FP, van Laar HH,编辑。在农场和区域两级应用系统方法。Dordrecht: Kluwer学术出版社;1997.45-52页。
MoAD。尼泊尔农业统计资料。加德满都:尼泊尔政府、农业发展部、农业企业促进和统计司、Singha Durbar农业统计处;2017.
FAOSTAT:联合国粮农组织的统计数据。[http://www.fao.org/faostat/en/#search/Wheat].2019年12月访问。
普瑞德。尼泊尔粮食供应与需求分析,国际粮食政策研究,2011。新德里:环境和生产司;2011.
联合国:尼泊尔的脆弱性概况。发展政策委员会第二十次全体会议,2018年3月12日至16日。纽约:联合国;2018.
Mwadzingeni L, Figlan S, Shimelis H, Tsilo TJ。提高小麦耐旱性的遗传资源和育种方法。作物改良学报2017;31:48 - 72。
王晓燕,王晓燕,王晓燕,王晓燕。欧洲六倍体面包小麦的遗传多样性及群体结构分析(小麦l .)品种。《公共科学图书馆•综合》。2014;9:e94000。
霍辛格·柯,威尔·布尔。地理结构人群的遗传学:定义、估计和解释FST。Nat Rev Genet。2009;10:639-50。
李伟,刘亮,王勇,张强,范刚,张松,等。杏的遗传多样性、群体结构与亲缘关系(李属)基于限制性内切位点相关DNA测序。Hortic杂志2020;7:69。
李军,万胜,杨宇。合成六倍体小麦在育种过程中增强了面包小麦的变异和适应性进化。中国科学d辑(英文版)。
张宏,Mittal N, Leamy LJ, Barazani O.回归野外——利用未开发的野生亲缘遗传多样性进行作物改良。另一个星球达成。2017;10:5-24。
傅YB。了解现代植物育种下的作物遗传多样性。《应用科学学报》2015;128:2131-42。
Lopes MS, El-Basyoni I, Baenziger PS, Singh S, Royo C, Ozbek K,等。利用小麦本地品种的遗传多样性以适应气候变化。J Exp Bot. 2015; 66:3477-86。
小麦基因组学成熟。植物生物学杂志2017;36:142-8。
史鑫,凌华。普通小麦及其祖先种基因组测序研究进展。作物j . 2017; 6:15-21。
Brenchley R, Spannagl M, Pfeifer M, Barker GLA, Amore RD, Allen AM,等。面包小麦全基因组霰弹枪测序分析。大自然。2012;491:705-10。
IWGSC。利用完全注释的参考基因组改变小麦研究和育种的限制。科学(80 -)。2018; 361: eaar7191。
Torkamaneh D, Boyle B, Belzile F.作物全基因组基因分型策略与数据整合。应用科学学报2018;131:499-511。
Elshire RJ, Glaubitz JC, Sun Q, Poland JA, Kawamoto K, Buckler ES,等。一种针对高多样性物种的稳健、简单的基因测序分型(GBS)方法。《公共科学图书馆•综合》。2011;6:e19379。
Torkamaneh D, Laroche J, Bastien M, Abed A, Belzile F. Fast-GBS:从测序基因分型数据中高效高精度调用snp的新途径。BMC生物信息学。2017;十八5。
Poland J, Endelman J, Dawson J, Rutkoski J, Wu SY, Manes Y,等。基因测序分型在小麦育种中的应用。植物基因组。2012;5:103-13。
Davey JW, Hohenlohe PA, Etter PD, Boone JQ, Catchen JM, Blaxter ML.利用下一代测序发现全基因组遗传标记和基因分型。Nat Rev Genet。2011;12:49 - 510。
Huq A, Akter S, Sup I, Hoy N, Kim T, Jin Y,等。基因中功能性snp的鉴定及其对植物表型的影响。植物生物技术学报,2016;43:1-11。
Kumar S, Banks TW, Cloutier S.通过下一代测序发现SNP及其应用。植物基因组学杂志2012;2012:831460。
陈旭,闵东,亚西尔塔,胡颖。利用SSR标记研究中国优秀冬小麦的遗传多样性、群体结构和连锁失衡。《公共科学图书馆•综合》。2012;7:e44510。
Tascioglu T, Metin OK, Aydin Y, Sakiroglu M, Akan K, Uncuoglu AA。面包小麦遗传多样性、群体结构与连锁失衡(小麦l .)。生物化学麝猫。2016;54:421-37。
Ya N, Raveendar S, Bayarsukh N, Ya M, Lee J-R, Lee K-J,等。基于SSR标记的蒙古小麦遗传多样性和群体结构:保护和管理的意义。植物育种。2017;5:213-20。
Chao S, Dubcovsky J, Dvorak J, Luo M, Baenziger SP, Matnyazov R,等。春小麦和冬小麦连锁失衡和SNP变异的群体和基因组特异性模式(小麦l .)。BMC基因组学。2010;11:727。
赵胜,张伟,杜bcovsky J, Sorrells M.美国小麦遗传多样性及全基因组连锁不平衡评价(小麦L.)代表不同市场类别的种质资源。作物科学。2007;47:1018-30。
刘旭,王辉,王辉,郭铮,徐旭,刘杰,等。玉米基因组选择的影响因素。作物j . 2018; 6:341-52。
Khan MK, Pandey A, Thomas G, Akkaya MS, Kayis SA, Ozsensoy Y,等。印度和土耳其小麦的遗传多样性与群体结构。AoB植物。2015;7:plv083。
吕文根,李文华,李文华,李文华,等。小麦地域性基因组多样性。遗传学。2017;205:1657 - 76。
Ward BP, Brown-Guedira G, Kolb FL, Van Sanford DA, Tyagi P, Sneller CH,等。弗吉尼亚生长的软红冬小麦产量相关性状的全基因组关联研究。《公共科学图书馆•综合》。2019;14:e0208217。
brbakliic L, Trkulja D, kondiic -Špika A, mikiic S, tomiiich M, Kobiljski B.关联制图中小麦核心群体结构的确定。谷物公社。2015;43:22-8。
Benson J, Brown-guedira G, Murphy JP, Sneller C.抗枯萎病软质冬小麦群体结构、连锁失衡与遗传多样性。植物基因组。2012;5:71 - 80。
Bhatta M, Morgounov A, Belamkar V, Poland J, Baenziger PS.揭示合成六倍体小麦的新遗传多样性和群体结构。BMC基因组学。2018;19:591。
张丹,白刚,朱超,余杰,卡佛BF。美国优秀冬小麦的遗传多样性、群体结构与连锁失衡。植物遗传学报(英文版);
Joshi BK, Mudwari A, Bhatta M.尼泊尔小麦遗传资源尼泊尔农业研究。2006;7:1-9。
Vijayaraghavan K, Majumder R, Naithani M, Kapur R, Kaur P.小麦遗传增益:小麦种子在印度、孟加拉国、尼泊尔和不丹的现状和机会。纽约:博洛格全球防锈倡议;2018.
Morris ML, Dubin HJ, Dubin HJ, Pokhrel T.回到尼泊尔的小麦研究。卷92 - 04。墨西哥:国际玉米和小麦改良中心;1992.
Morris ML, Dubin HJ, Thaneshwar P.回到尼泊尔的小麦育种研究。阿格利司经济。1994;10:269 - 82。
Timsina KP, Gairhe S, Magar DBT, Ghimire YN, Gauchan D, Padhyoti Y.农业研究是技术验证、传播和采用的可行手段:尼泊尔小麦研究的一个例子。Agron J Nepal. 2016; 4:9-24。
尼泊尔的植物育种:过去、现在和未来。高等学校农业学报2017;1:1-33。
paulo MN, Joshi BK, Ghimire KH。尼泊尔农业植物遗传资源管理现状。Agron J Nepal. 2016; 4:75-91。
巴傅里叶F, Bouchet S, Robert S, De OR, Rimbert H, Kitt J,等。世界小麦遗传多样性的系统地理学和历史。Sci放置2019;5:eaav0536。
Bruslé T.选择目的地和工作:尼泊尔工人在印度北部北阿坎德邦的移民策略。Mt Res Dev. 2008; 28:240-7。
Mercer KL, Perales HR。作物多样性中心地方种族对气候变化的进化响应另一个星球达成。2010;3:480 - 93。
Cavanagh CR, Chao S, Wang S, Huang BE, Stephen S, Kiani S,等。全基因组比较多样性揭示了小麦六倍体地方品种改良的多重选择靶点。《美国国家科学院学报》上。2013; 110:8057 - 62。
Monteagudo A, Casas AM, Cantalapiedra CP, Contreras-Moreira B, Gracia MP, Igartua E.利用来自地方品种的新多样性来改善优质大麦品种。植物科学进展2019;10:434。
周勇,陈铮,程明,陈杰,朱涛,王锐,等。揭示了小麦在中国的分散历史、适应性进化和选择。植物生物技术学报。2017;16(4):385 - 391。
Mwadzingeni L, Shimelis H, Rees DJG, Tsilo TJ。干旱胁迫与非胁迫条件下小麦农艺性状的全基因组关联分析。《公共科学图书馆•综合》。2017;12:e0171692。
潘伟杰,李海燕,李海燕,等。解锁克里奥尔小麦的遗传多样性。Sci众议员2016;6:23092。
Oliveira HR, Hagenblad J, Leino MW, Leigh FJ, Lister DL, Penã-Chocarro L,等。用优质面包小麦单核苷酸多态性标记对地中海重访的四倍体小麦地方品种进行评价。BMC麝猫。2014;15:54。
Eltaher S, salam A, Belamkar V, Emara HA, Nower AA, Salem KFM,等。内布拉斯加州冬小麦F3:6基因型的遗传多样性和群体结构麝猫。2018;9:76。
Gordon E, Kaviani M, Kagale S, Payne T, Navabi A.合成六倍体衍生小麦的遗传多样性与群体结构(小麦l .)登记入册。作物学报2018;66:335-48。
于宏,邓卓,向春,田杰。小麦b基因组农艺性状多样性分析及连锁不平衡定位。J基因组。2014;2:20-30。
林柏特H, Darrier B, Navarro J, Kitt J, Choulet F, Leveugle M,等。六倍体小麦高通量SNP的发现与基因分型。《公共科学图书馆•综合》。2018;13:e0186329。
基因组可塑性是多倍体小麦驯化成功的关键因素。科学(80 -)。2007; 316:1862-6。
晁硕,张伟,Akhunov E, Sherman J,马勇,罗MC,等。美国小麦基因来源SNP标记多态性分析(小麦l .)品种。摩尔品种。2009;23:23-33。
El-Esawi MA, Witczak J, Abomohra AEF, Ali HM, Elshikh MS, Ahmad M.基于DArT标记的奥地利和比利时小麦种质资源遗传多样性和群体结构分析。基因。2018;愚人节。
张丽丽,刘德东,郭晓丽,杨文力,孙建忠,王德伟,等。利用DArT标记研究中国北方常见小麦品种的遗传多样性和群体结构BMC麝猫。2011;42。
Abbasabad EZ, Mohammadi SA, Moghaddam M, Kamli MRJ。利用SSR标记分析伊朗小麦地方品种的遗传多样性、群体结构和连锁失衡。植物种质资源特征直到2017;15:327-34。
Vos PG, Paulo MJ, Voorrips RE, Visser RGF, van Eck HJ, van Eeuwijk FA。四倍体马铃薯模拟和snp -阵列数据中LD衰减和各种LD衰减估计量的评价。《应用科学》2017;130:123-35。
Flint-Garcia SA, Thornsberry JM, Buckler ES。植物连锁不平衡结构。植物生物学,2003;54:357-74。
王硕,黄D, Forrest K, Allen A, Chao S,黄BE,等。利用高密度90000单核苷酸多态性阵列分析小麦多倍体基因组多样性。植物工程学报(英文版)2014;
郝超,王磊,葛辉,董艳,张旭。中国面包小麦遗传多样性与连锁失衡(小麦L.)。《公共科学图书馆•综合》。2011;6:e17279。
Hassan NM, El-bastawisy ZM, El-sayed AK, Ebeed HT,真主安拉MMN。脱氢酶基因在小麦抗旱性中的作用。J Adv Res. 2015; 6:179-88。
Li X, Waddington SR, Dixon J, Joshi AK, de Vicente MC.南亚农业系统中干旱和其他与水相关的约束对主要粮食作物的相对重要性。食品安全内核。2011;3:19-33。
Khatiwada KR, Pandey副总裁。尼泊尔喜马拉雅地区水文气象干旱特征:以卡纳利河流域为例。极端气候气候。2019;26:100239。
Dahal P, Shrestha NS, Shrestha ML, Krakauer NY, Panthi J, Pradhanang SM,等。尼泊尔中部干旱风险评估:时空分析。Nat危险。2016;80:1913-32。
Torkamaneh D, Laroche J, Belzile F.从测序基因分型(GBS)数据调用全基因组SNP: 7个管道和两种测序技术的比较。《公共科学图书馆•综合》。2016;11:e0161333。
Browning BL, Browning SR.用数百万参考样本进行基因型归责。Am J Hum Genet。2016;98:116-26。
Torkamaneh D, Belzile F.扫描和填充:结合测序基因分型、SNP阵列和全基因组重测序数据的超密集SNP基因分型。《公共科学图书馆•综合》。2015;10:e0131533。
Bradbury PJ, Zhang Z, Kroon DE, Casstevens TM, Ramdoss Y, Buckler ES。TASSEL:用于不同样本复杂性状关联映射的软件。生物信息应用笔记2007;23:2633-5。
Danecek P, Auton A, Abecasis G, Albers CA, Banks E, Depristo MA,等。变量调用格式和VCFtools。生物信息应用笔记2011;27:2156-8。
j: fastSTRUCTURE:大SNP数据集中种群结构的变分推断。遗传学。2014;197:573 - 89。
核心团队R. R:统计计算的语言和环境。维也纳:R统计计算基金会。https://www.r-project.org/.2018年7月访问。
Müllner D. Fastcluster:用于R和python的快速分层、聚合的集群例程。J Stat soft . 2013; 53:1-18。
数据和文本挖掘denextend:一个用于可视化、调整和比较层次聚类树的R包。生物信息学。2015;31:3718-20。
顾志,顾磊,顾磊。基于生物信息学的环形可视化方法。2014;30:2811-2。
致谢
我们把这篇论文献给我们的导师、同事和朋友,已故的Alireza Navabi教授,他在准备这篇手稿时突然去世了。作者要感谢Nick Wilker和Katarina Bosnic (Guelph大学)在种子处理和田间种植方面的帮助。我们感谢A.K. Joshi博士、Madan Bhatta先生、Krishna Hari Ghimire先生和Deepak Pandey先生协助从尼泊尔获得种子,以及墨西哥CIMMYT的Thomas Payne博士。国家遗传资源中心(NGRC,尼泊尔)和农业研究理事会(NARC,尼泊尔)慷慨提供了尼泊尔地方品种种子。释放品种的种子由国家小麦研究项目(NWRP隶属于尼泊尔国家小麦研究中心)提供。CIMMYT育种系由墨西哥CIMMYT提供。我们也感谢P. Stephen Baenziger博士(内布拉斯加大学)和Andrew J. Burt博士(加拿大农业和农业食品部)提供的宝贵意见。
资金
这项研究得到了加拿大国际粮食安全研究基金(CIFSRF)对MNR的慷慨支持,该基金由国际发展研究中心(IDRC,渥太华)和加拿大全球事务部联合资助SeCan安大略农业适应委员会和粮食农民。资助机构在研究的设计、收集、分析和解释数据以及撰写手稿方面没有发挥作用。
作者信息
从属关系
贡献
KK和AN对论文进行了概念化。KK写了手稿。DT对数据分析和技术编辑做出了贡献。MK支持植物取样和DNA分离。FB提供了技术投入。MNR负责技术和语言编辑。作者已阅读并批准了手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
相互竞争的利益
所有的作者都宣称不存在竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
卡德卡,K.,托尔卡马内,D.,卡维亚尼,M.。et al。尼泊尔春小麦种群结构(小麦l .)种质。BMC植物杂志20.530(2020)。https://doi.org/10.1186/s12870-020-02722-8
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-020-02722-8
关键字
- 尼泊尔
- 长白猪
- Genotype-by-sequencing (GBS)
- 遗传多样性
- 人口结构
- 连锁不平衡