摘要
背景
Gnetum是一种经济上重要的热带和亚热带裸子植物,具有各种膳食,工业和药用用途。许多碳水化合物,蛋白质和纤维在成熟过程中积聚Gnetum种子。然而,与此过程相关的分子机制尚不清楚。
结果
因此,我们组装了一个全长转录组从未成熟和成熟g . luofuense使用PACBIO测序读取的种子。我们鉴定了总共5726个新型基因,9061个替代剪接事件,3551LNCRNA,2160种转录因子,并发现8512个基因具有至少一种聚(A)位点。另外,由illumina测序产生的六种转录om的基因表达比较显示,14,323个基因与未经7891个基因的未成熟阶段差异地表达到成熟阶段,上调和6432个基因下调。QRT-PCR验证了14个差异表达的转录因子,AUX / IAA和BHLH系列的表达,表明它们可能在种子成熟中具有重要作用g . luofuense.
结论
这些发现为了解裸子植物种子发育提供了有价值的分子资源。
背景
Gnetum是热带和亚热带裸子植物乔木和灌木的一个属,分布在南美洲、非洲东部和亚洲[1].Gnetum在饮食和工业方面具有巨大的经济潜力:它的叶子可以用作蔬菜,它的茎和树皮可以制成线、网和纸,它的种子可以用来做油和饮料。一个Gnetum种子来自雌性生殖器的雌性生殖器,在雌性斯特罗氏菌的衣领上产生[1那2].一个Gnetum种子由三层膜组成,最外层是种皮-假种皮[3.那4.].Gnetum种子富含多种化学物质,如碳水化合物、蛋白质和纤维[5.那6.].初级代谢(如碳水化合物代谢)可能与种子成熟过程(假种皮颜色由绿色变为红色)有关。1) 在Gnetum但是未仔细调查该过程的分子机制。
以往对转录因子(TFs)的研究为深入了解生殖器官发育的分子机制提供了宝贵的见解Gnetum.包含I型和II型MADS-BOX基因的MADS箱体基因,编码调节种子植物生殖器官发育的基本转录因子[7.].以前的工作已经表明II型MADS-boxAG例如,TM8.- 样基因高度表达g . luofuense种子(8.].此外,AUX / IAA.基因通过响应激素毒型/吲哚-3-乙酸而参与种子植物中的各种器官的发展[9.那10].最近的一项研究表明AUX / IAA.雌球果的发育与基因有关g . luofuense[11].另一项研究表明bHLH基因促进…的发展g . luofuense叶子(11].此外,bHLH和MYB转录因子能够形成一种复合物,调节雄蕊发育和种子生产[12].因此,编码MADS盒,AUX / IAA和BHLH TFS的基因可能会发挥重要作用Gnetum种子成熟,这些可能性需要进一步研究。
除关键基因/TFs外,调控雄球果发育的其他分子机制g . luofuense也值得注意。例如,以前的研究表明g . luofuense利用选择性剪接(AS)和选择性聚腺苷酸化(APA)来增加叶片和雌球果发育过程中转录组的复杂性[11].AS被认为是真核生物发育的重要调节因子[13那14].此外,APA通过产生不同编码序列或3 ' utr的不同亚型,促进了靶rna的稳定性、翻译和定位[15].裸子植物中的AS和APA还有很少的调查,但是在Agiospers中的研究更丰富(例如[16那17那18那19那20.那21])。此外,长链非编码rna (long noncoding RNAs, lncRNAs)至少具有200个核苷酸,也可能在调控中发挥作用Gnetum种子发展。lncrna参与几乎所有真核生物的转录和转录后基因调控[22那23那24].LNCRNA的存在仅在叶子中报道银杏l . (25那26],在叶和雌球花g . luofuense[11].迄今为止,在裸子植物中的LNCRNA已经支付了很少的注意[11那27].
为了调查APA和LNCRNA,PACBIO测序提供比Illumina测序更好的性能,因为单分子转录组测序在CDNA分子的5'和3'末端提供更大的序列完整性,识别替代方案的更高精度同种型,以及增加RNA单倍型的力量[11那16那28].因此,在本研究中,我们从两个发育阶段(不成熟和成熟)生成了一个全长转录组g . luofuense种子使用的参考基因组g . luofuense(=g . montanum) [29].采用单分子数据对AS、APA、lncrna及相关tf进行研究。此外,我们利用Illumina RNA测序技术分别生成了两个种子发育阶段的转录组,以揭示调控种子成熟过程的关键基因Gnetum.
结果
PacBio测序和纠错
成熟和不成熟的全长转录组g . luofuense种子共有12869,707个子reads (19.81 Gb),平均长度为1540 bp(表S)1,无花果。1A).以ROIs > 0.8为精度值进行自校正后,得到384,042个平均长度为1919 bp的环状共识序列(circular consensus sequences, CCSs),其中全长非嵌合(FLNC) reads占81%(312,444,图S)1B).使用ICE算法对FLNC读进行聚类,并对非FLNC读进行抛光。FLNC reads和经过处理的非FLNC reads被合并,得到165,883个经过处理的一致亚型,长度从167到13,816 bp不等(图S1C).使用Illumina测序数据和LoRDEC软件对165,883个经过优化的共识reads进行进一步校正。经过校正后,平均长度和N50、N95值略有变化(表S)2).
基因组测绘和新型基因检测
经过修正的经过润色的共识解读被映射到g . luofuense使用GMAP参考基因组。162,887个(98.19%)reads被映射到参考文献(图S1D);其中,参考基因组正链上特异mapping reads 63,049条(占总mapping reads的38.01%),负链上特异mapping reads 60292条(占36.35%),多重mapping reads 39,546条(占23.84%),未特异mapping reads 2996条(占1.81%)。每个支架上的映射密度g . luofuense基因组见图S1E. 98%以上的图谱reads与参考基因组具有相似性,图谱reads的覆盖率均在80%以上(图S1F).删除未映射和冗余的reads后,剩余41151个reads,其中7899个是已知基因的新亚型,5726个reads来自新基因。
新基因的注释和分类
5726个新基因在ncbi NR、KEGG、GO、SwissProt、KOG和Pfam等6个数据库中进行了检索。共注释了4099个新基因,其中NR数据库注释了2588个(表S3.).五种 -云杉sitchensis(649个基因),Amborella trichopoda.(116),葡萄(88),Elaeis Guineensis.(80)和莲属椰子(61) - 提出了最大的击中数g . luofuense新基因(图S2一种)。用Kegg途径注释两千四百八十七种新基因(表S.3.),富集程度最高的途径为“信号转导”(169个基因)、“碳水化合物代谢”(83个基因)和“翻译”(69个基因),图S2B). GO分析将2069个基因分为三类:“生物过程”、“细胞成分”和“分子功能”(图S)2C).被分类为生物过程范畴的新基因主要以“代谢过程”(1052)、“细胞过程”(1037)和“单生物过程”(581)进行注释。细胞成分类的新基因主要标注为“细胞”(519)、“细胞部分”(519)和“膜”(367)。在分子功能分类中,新基因主要被标注为“结合”(1192)、“催化活性”(942)和“转运体活性”(132)。瑞士Prot、KOG和Pfam数据库分别注释了一千九百30个基因、1315个基因和2069个基因(表S3.).
AS和APA分析
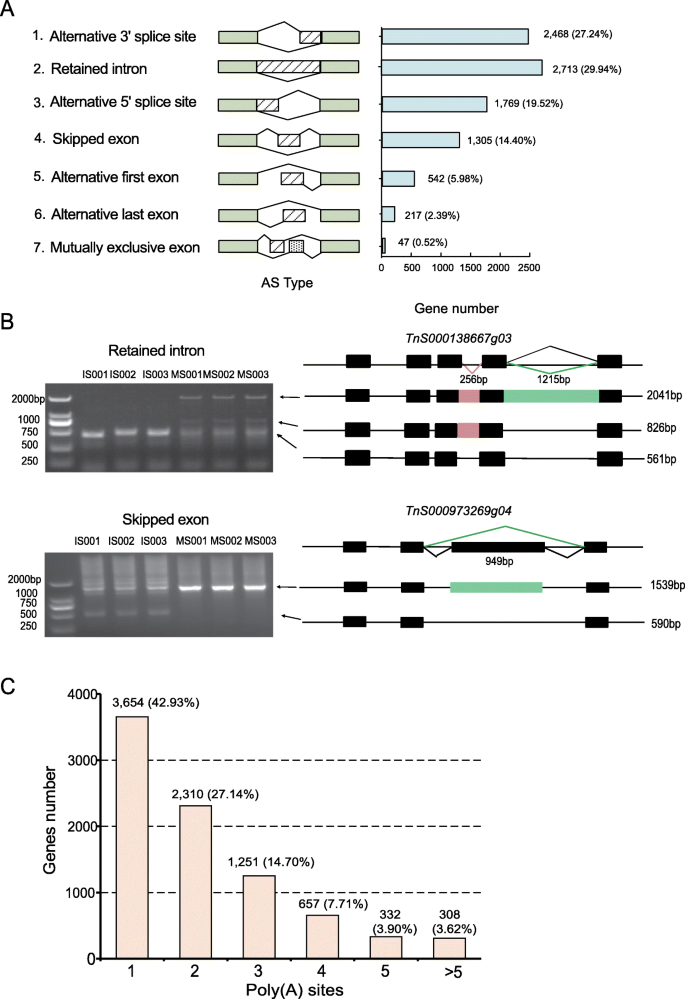
映射到参考基因组后g . luofuense,共检测到9061个AS事件。这些可以分为七种类型(图。2A):保留内含子(2713,29.94%),3’可变剪接位点(2468,27.24%),5’可变剪接位点(1769,19.52%),跳过外显子(1305,14.40%),第一外显子(542,5.98%),最后外显子(217,2.39%),互斥外显子(47,0.52%)。
为了验证AS事件的识别,两个基因的表达,即。TNS00138667G03.和TNS000973269G04通过qRT-PCR进行验证(图。2b,附加文件1).此外,共有8512个基因来自g . luofuense种子至少有一个支持聚(A)位点。其中3654个位点(42.93%)为单一poly(a)位点,640个位点(7.52%)为至少5个poly(a)位点。2C)。在基因中发现了最多的聚(a)位点-21-11-21TnS000670009g01.
识别TFS和LNCRNA
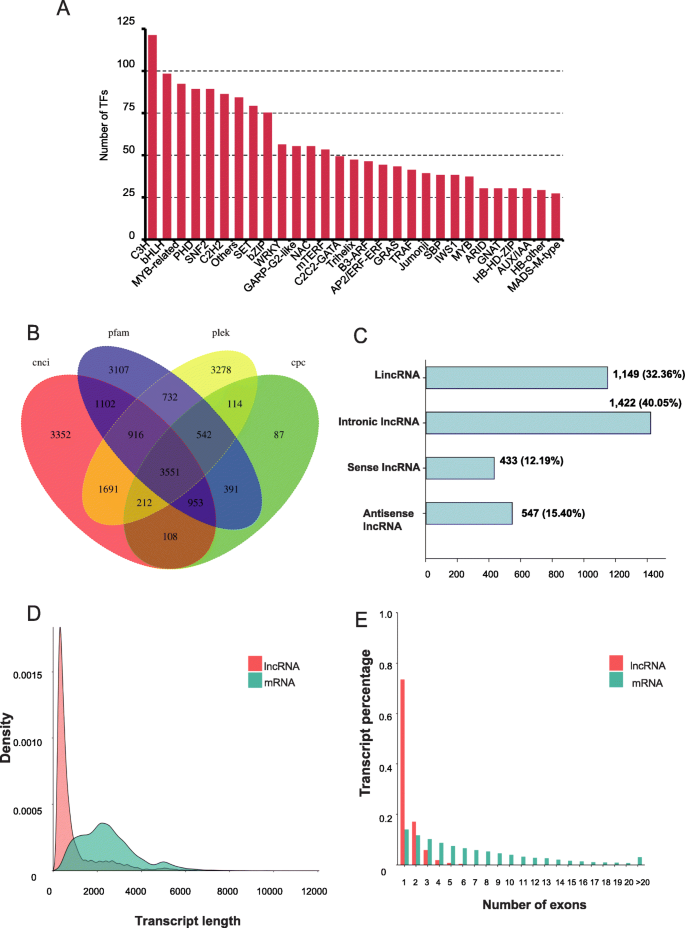
使用ITAK检测来自86个基因家族的2160个转录因子(TFS)。所识别的TFS的最大部分来自C3H(5.6%),BHLH(4.53%)和MyB相关(4.26%)系列(图。3.a).此外,通过CNCI、CPC、PFAM和PLEK方法分别鉴定出11,885、5958、11,294和11,037个lncrna。四种方法共鉴定出3551个lncrna(图)。3.B.),长度范围从200到7840 bp。LNCRNA进一步分为四种类型(图。3.c):1422(40.05%)感应内肠道LNCRNA,1149(32.36%)长的非基础非编码RNA,547(15.40%)反义LNCRNA和433次重叠的LNCRNA(12.19%)。所识别的LNCRNA的长度分布比从中预测的MRNA的长度分布相当窄幅g . luofuense基因组(图。3.d)。此外,大多数鉴定的LNCRNA具有五个或更少的外显子,而从参考基因组预测的MRNA倾向于具有较大数量的外显子(图。3.e)。
一种在全基因组转录组中鉴定的部分转录因子(前28个基因家族)g . luofuense种子。B.维恩图显示了使用四种不同方法识别的lncrna的数量:CPC (Coding Potential Calculator)、CNCI (Coding- non -Coding Index)、CPAT (Coding Potential Assessment Tool)和Pfam (Protein Family)。C四种lncRNA类型的功能分类和编号。D.鉴定的lncrna在参比基因组上的长度密度分布g . luofuense与全长转录组中鉴定的LNCRNA相比。E.参考基因组预测的mrna外显子数量分布及全长转录组中已识别的lncrna
Illumina对两个发育阶段种子样品的测序
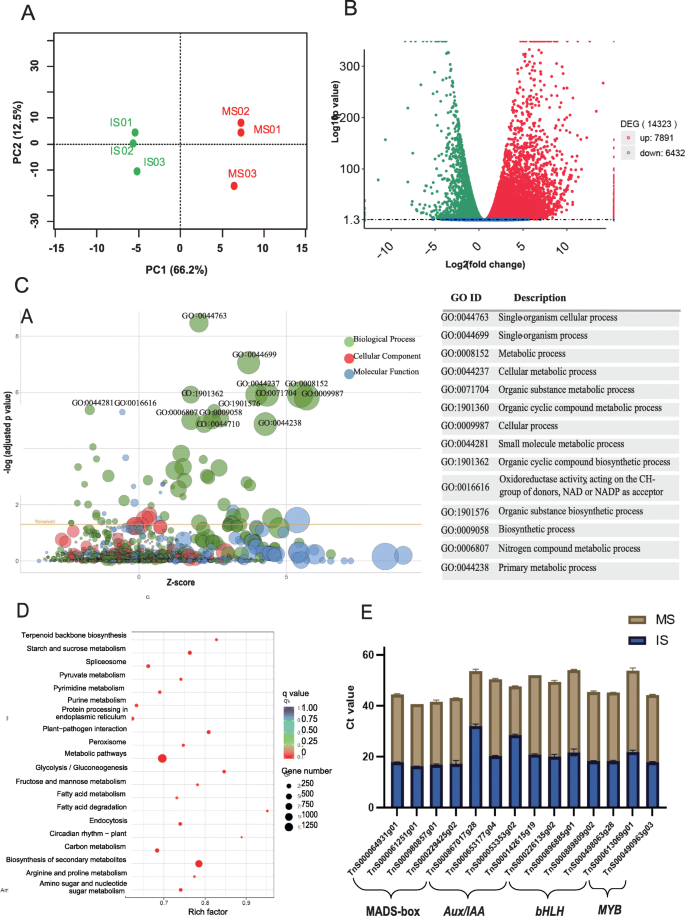
探讨种子发育过程中的基因表达模式g . luofuense,3个未成熟种子样本(IS)和3个成熟种子样本(MS)生成了306,900,384个干净Illumina读取(46.04 Gb的原始数据),Q30值从93.54到94.07%(表S4.).缺失适配器和低质量reads后,6个样品的平均GC含量为47.08%。PCA分析显示,未成熟种子和成熟种子重复样本的基因表达高度相关(相关效率值= 0.95,PC1和PC2解释的累积变异比例= 78.7%)(图4)。4.a).映射到g . luofuenseIS基因组的作图率(平均89.44%,表S5.)明显大于MS(平均84.46%)T.-测试P.值= 0.003)。两个发育阶段的RNA-seq分析共获得23,977个基因(IS中19,010个,MS中20,737个),其中2970个为新基因。
deg检测及qRT-PCR验证。一种三种未成材种子样品(IS01-03)和三种成熟种子样品(MS01-03)中基因表达的PCA分析。B.未成熟和成熟种子样品基因表达差异的火山图g . luofuense,在红色和下调基因中具有上调基因,以未成熟的种子到成熟的种子。C富集的GO条款的泡沫图;这X设在代表了Z.-core,这y-axis表示调整后的负对数p -值,圆尺寸与富集的基因数量成比例,圆形颜色表示三个GO术语类别。右侧的表格描述了类别生物过程中的前14个术语,这些过程已经在左侧的泡沫图中标记。D.富含Kegg术语的泡沫图;这X-axis代表丰富的因素,圆尺寸与富集的基因数成比例,圆形颜色对应于调整后的负对数P.-值为每个KEGG路径。E.14个TF基因的表达(即Mads-Box,AUX / IAA.那bHLH和MYB基因)从不成熟和成熟的种子g . luofuense进行qRT-PCR验证,表达值与△△CT-方法
富含QRT-PCR验证的富集分析
在IS(对照组)和MS之间共鉴定了14323个差异表达基因(DEGs):我们发现了7891个上调基因和6432个下调基因(图)。4.b)从IS到ms。DEGs也用三类氧化石墨烯术语进行了注释,其中“生物过程”类中的多个氧化石墨烯术语相对于显著富集Z.分数和调整P.值(无花果。4.C)。前五个富集的GO术语是“单生体蜂窝流程”(GO:0044763),“单有机物过程”(GO:0044699),“代谢过程”(GO:0008152),“蜂窝代谢过程”(GO:0044237)和“有机物质代谢过程”(GO:0071704)。参考的egs也富含多克GEGG途径拟南芥.富集程度最高的5个KEGG途径分别是“代谢途径”(KEGG ID: ath01100, 1229个基因)、“次生代谢产物生物合成”(ath01110, 844个基因)、“碳代谢”(ath01200, 179个基因)、“核糖体”(ath03010, 164个基因)和“淀粉和蔗糖代谢”(ath00500, 154个基因)。4.d).利用qRT-PCR验证14个感兴趣基因的相对表达:4个MADS-box基因,4个MADS-box基因AUX / IAA.基因,四bHLH基因,和两个MYB基因。图中显示了14个基因在两个种子发育阶段的相对表达。4.e。
讨论
全长转录组的结构分析
全长转录组的结构分析
事件分析
据报道,在被子植物中,AS模式的百分比在不同器官之间存在显著差异,玉米的营养组织(如根和穗)比生殖组织(如花粉和胚乳)表现出更高的内含子保留百分比(见图)。3.17)。此外,在草莓中的水果发育过程中,保留的内含子百分比显着下降[30.].在裸子植物中,银杏基因(如。gb_12621和GB_20198)显示营养器官和生殖器官之间以及叶和种子的未成熟和成熟阶段之间AS的差异[26].在本研究中,保留内含子占所有AS事件的29.94%(图2)。2a),该图显着低于叶片中报告的那些[41.5%,12]和雌性斯特罗毕[46%,11]g . luofuense.这一结果表明,不同AS模式的频率可能不仅是物种特有的,而且可能在不同器官之间存在差异。
APA分析
在被子植物中,选择性聚腺苷酸化参与开花时间的调节[31那32].在裸子植物中,研究APA可以改善基因组注释,促进对类黄酮生物合成的理解g . biloba[26].在本研究中,将总共8512个基因鉴定为具有至少一种聚(a)位点,随着聚(a)位点从一个增加的数量增加,具有各种数量的聚(a)位点的基因的数量急剧下降到五(图。2c).不同数量poly(A)位点的基因数目模式与中观察到的一致g . luofuense叶和雌球果[11].我们的结果表明,选择性聚腺苷酸化丰富了蛋白质组学的复杂性,并影响种子的成熟过程g . luofuense.
lncRNA分析
在被子植物中,lncrna参与草莓果实发育和颜色变化[33],以及红茶中的香气形成[34].在裸子植物中,LNCRNA在叶片发展的调节中具有重要作用[27]叶子颜色变化g . biloba[25].四种lncRNA类型的百分比已经被证明在g . biloba:LincrNA(50.6%),感测LNCRNA(21.6%),内肠道LNCRNA(20.9%),反义LNCRNA(6.9%)[26].最近的一项研究表明,在全长转录组中,lincRNA最高(40.8%),反义lncRNA最低(1.67%)g . luofuense雌性斯特罗伯[11].而在本研究中,我们发现内含子lncRNA的数量(40.05%)最高,意义lncRNA的数量(2.1%)最低(Fig. 2)。3.因此,似乎g . luofuense使用不同的lncrna来调节不同的生殖器官。该场景与报告的结果类似P. Abies.[35].此外,LNCRNA往往比蛋白质编码基因更短,并且具有更少的外显子[27]这个发现与先前的裸子植物研究一致,如g . biloba[27],挪威云杉[35], 和g . luofuense[11].
马铃薯种子成熟关键转录因子/基因g . luofuense
MADS-box基因
MADS-box转录因子根据保守的MADS结构域序列分为I型和II型两类[36那37].与II型基因相比,I型基因在先前的研究中受到更少的关注,尽管它们在雌性配子体的发育中的作用,在Anuiaperms中突出了胚胎和种子[36那38].I型基因在裸子植物中的功能尚不清楚,I型基因在针叶树的枝、针和球果中的广泛表达被认为是“转录噪声”[39].I型基因进一步细分为Mα、Mβ和Mδ亚群;Mα基因一般在针叶树的不同枝组织中表达,而Mβ/Mδ基因则在胚、芽和雄球果中表达[39].在g . luofuense,共11个I型基因,(7个Mα基因,3个Mβ基因,1个Mδ基因)已被报道[8.].IDS-Box TFS的二十七种类型g . luofuense种子(图。4.e)和基因TnS000803113g11在未成熟和成熟的种子之间差异表达,表明在种子成熟中的重要作用g . luofuense.
在II型MADS-box基因中TM8.基因最早是在番茄花中发现的[40)和TM8.-like Gene Eraf17显示在女性花中表达,但不是黄瓜的雄花[41].在裸子植物中,TM8.例如基因(如。GBMADS11和GbMADS6在g . biloba和TbTM8在Taxus Baccata.)影响雄球果假种皮发育和种子假种皮发育[42].在g . luofuense那TM8.类基因几乎占了具有不同表达模式的基因的一半[8.].例如,TNS013912549G01在女性和男性斯特罗比中表达,而TNS001008199T01仅在雄球藻中表达[8.].先前的研究表明GpMADS1, 一种TM8.Hou et al. (2019b)定义的类基因参与了雌球果的发育g . parvifolium[43].到目前为止,已经发现38个II型基因g . luofuense, 其中TM8.类基因构成了几乎一半已确认的基因数目[8.].在本研究中,有两个TM8.例如基因TNS000061251G01和TNS000980857G01差异表达。此外,其他II型MADS-box基因,如AG- 麦基基因TNS000064931G01和AGL6.- 麦基基因TnS000229425g02在两个发育阶段有差异表达g . luofuense种子。我们的结果与之前的研究一致AG那AGL6.和TM8.类基因调控种子的发育g . biloba和t . baccata[42].
辅助/ IAA基因
AUX / IAA.TFS在种子植物的植物态反应中发挥着重要作用[9.那10那44].例如,在被子植物中,FaAux / IAA1和辅助/ IAA2参与草莓果实的开发[45], 和EgrIAA4被认为是调控次生细胞壁和纤维发育的关键桉树[46].另一项研究表明IAA9番茄中的水果和叶形态发生[47在裸子植物中AUX / IAA.基因LaIAA2似乎对根系发育和生长素信号转导具有重要作用[48].此外,六AUX / IAA.基因(格鲁亚1-6.)已被确认g . luofuense,所有这些都与雌球果的发育有关[11].在本研究中,四个AUX / IAA.的基因,TnS000653177g04(格鲁娅2.),TnS000867017g28(格鲁娅3.),TnS000053353g02(格鲁娅4.), 和TnS000142615g19(格鲁娅5.),通过qRT-PCR在两个发育阶段的差异表达和验证g . luofuense种子(图。4.这些结果表明AUX / IAA.基因可能也很重要g . luofuense种子成熟。
bHLH基因
在被子植物中,bhlh编码基因抹刀已被报道控制花和果实的发展拟南芥[49那50, bHLH TF已被证明可以确定种皮颜色Brassica Rapa.[51].此外,bHLHTFs与MYB和WDR TFs一起参与类黄酮生物合成的调控[52那53那54].二的表达式MYB有关的基因,即。Osmyb1和Osmyb4,在开花后14天达到饱和水平,表明它们在水稻种子成熟中起重要作用[55].据报道,在裸子植物中,三种bHLH转录因子对紫杉醇生物合成途径中的基因表达有负调控作用水松cuspidata[56].此外,据报道,BHLH和MYB TFS在根中参与黄酮类生物合成而不是种子银杏叶[26].在g . luofuense, 67年bHLH通过全长转录本鉴定叶片中的基因;30例进行系统发育分析,分为4个亚群[57].此外,110个BHLH TFS是在雌性斯特罗伯的开发期间是最丰富的TFSg . luofuense[11].在本研究中,鉴定了98个BHLH,其中四个BHLH基因,即TNS000226135G02那TnS000896885g01那TnS000889809g02,TnS000498063g28差异表达,并通过qRT-PCR验证其表达(图。4.这些结果表明bHLH TFs在种子成熟过程中也可能起重要作用g . luofuense.
与碳水化合物新陈代谢相关的基因
Gnetum种子富含碳水化合物g . africanum(87.62%) (5.),g . gnemon(64.1%)[6.].碳水化合物的积累Gnetum种子使它们可口和营养丰富,从而吸引了各种食草动物来促进种子分散[58那59].在本研究中,未成熟和成熟种子之间的DEGs富集于多个KEGG途径,如碳代谢、淀粉和蔗糖代谢、糖酵解/糖异生、果糖和甘露糖代谢(图2)。4.d).在氧化石墨烯条件下,如初级代谢过程、代谢过程和细胞代谢过程中也富集了deg(图)。4.c).这些结果表明,参与碳水化合物代谢的基因在种子成熟过程中也是必不可少的g . luofuense.
结论
我们生成了一个全长的转录组g . luofuense利用Pacbio测序技术对两个发育阶段的种子进行测序。我们共鉴定了5726个新基因,9061个可变剪接事件,3551个lncrna, 8512个基因至少拥有一个poly(a)位点。转录因子MADS-box、Aux/IAA和bHLH在紫花苜蓿种子成熟过程中发挥重要作用g . luofuense.这些发现提供了裸装器官裸体开发的有价值的分子资源。
方法
植物材料和RNA提取
Gnetum luofuense2018年9月2日和28日,在中山大学竹园栽培的雌性个体(凭单号“CH003”,SYS)中分别采集了未成熟(IS)和成熟(MS)发育阶段的种子。1a)孙中山大学的权限。为了获得两种发育阶段的全长转录组,将相同的量(10g)的成熟和未成熟的种子合并,在液氮中温育,并在-20℃下冷冻以进行PACBIO SMRT测序。另外,六个样本g . luofuense收集种子(“IS001-003”和“MS001-003”)用于illumina测序,从未成熟阶段(对照组)和来自成熟阶段的三个。使用RNA试剂盒(QIAGEN,VALENCIA,CA,USA)提取每个样品的RNA,遵循制造商的说明书。使用RNA酶DNA酶(QIAGEN)去除遗物DNA,并通过1%琼脂糖凝胶电泳评估样品的RNA浓度。用于纳米玻璃分光光度计(Thermofisher Scientific,Wilmington,De,USA)和Agilent 2100 BioAnalyzer(Agilent Technologies,Palo Alto,USA)用于评估提取的RNA的纯度和完整性。g . luofuense本研究所使用的样本来自仅为教学和研究而栽培的植物。因此,该植物的种子采集和试验研究均符合国家指导方针。
植物材料和RNA提取
Gnetum luofuense2018年9月2日和28日,在中山大学竹园栽培的雌性个体(凭单号“CH003”,SYS)中分别采集了未成熟(IS)和成熟(MS)发育阶段的种子。1a).为了获得两个发育阶段的全长转录组,将相同数量(15 g)的带有假种皮的成熟和未成熟种子混合在一起,在液氮中培养,并在−20℃冷冻,用于PacBio SMRT测序。此外,六个样本g . luofuense为illumina测序收集种子,来自未成熟阶段(对照组)和来自成熟阶段的三个。使用RNA试剂盒(QIAGEN,VALENCIA,CA,USA)提取每个样品的RNA,遵循制造商的说明书。使用RNA酶DNA酶(QIAGEN)去除遗物DNA,并通过1%琼脂糖凝胶电泳评估样品的RNA浓度。用于纳米玻璃分光光度计(Thermofisher Scientific,Wilmington,De,USA)和Agilent 2100 BioAnalyzer(Agilent Technologies,Palo Alto,USA)用于评估提取的RNA的纯度和完整性。
Library construction and PacBio sequel sequencing
当提取的RNA完整性满足最低要求(> 7.0)时,使用SMARTer PCR cDNA Synthesis kit (Clontech, Takara Bio Inc.,志贺,日本)合成全长cDNA。用KAPA HIFI PCR试剂盒(KAPA Biosystems, Boston, MA, USA)对合成的cDNA进行PCR扩增。PCR扩增后,采用QIAquick PCR纯化试剂盒(Qiagen, Hilden, Germany)对cDNA进行质量控制和纯化。RNA样本进行末端修复和SMRT哑铃型适配器的连接。在PacBio测序之前,我们建立了2个bins (1 - 4kb, 4 - 6kb)优先对较小的cdna进行测序。
文库建设和Illumina测序
在Illumina测序之前,所有6个含有poly(A)的RNA样本都用oligo (dT)磁珠富集。用碎片缓冲液将富集的RNA随机减少为小片段。用六聚体和逆转录酶(上标III, Invitrogen)生成第一链cDNA。用AMPure XP beads纯化后,用DNA聚合酶I、RNase H和dNTPs (Sigma-Aldrich)合成第二链cDNA。对双链cDNA进行末端修复和poly(A)拖尾,然后进行Illumina适配器连接。经过第二轮纯化和PCR扩增,最终获得cDNA文库。在Illumina HiSeq 4000平台测序之前,使用Qubit 2.0荧光仪评估6个cDNA文库的质量。
PacBio数据处理和纠错
使用PACBIO SMRTLINK V.5.1软件分析了PACBIO测序数据。首先,我们使用以下参数从平台生成的BAM文件获得了读取的插入(ROIS):最大跌落分数-0.8,最小长度-20,不抛光,最小Z.-CORE-9999,最小通过-1,最小预测精度-0.8,最大长度为18,000。基于存在和不存在5'和3'cDNA引物和3'多(a)尾,参见[中,将ROI分为全长读取(FLS)和非全长读取(NFL)和3'多(a)尾部,参见[11].使用异构级聚类(ICE)算法,聚集FLS和NFL以实现共有同种型。为了获得全长非嵌合(FLNC)同种型,使用Quiviver软件校正来自氟的高质量同种型,其后校正精度高于99%。来自NFL的低质量共识同种型是用rolyec进行纠正的[60使用了两份经过illumina测序的样本(一份来自成熟种子,另一份来自未成熟种子)。
基因组测绘和新型基因检测
所有FLNCs和校正后的nFLs均被定位到参比基因组g . luofuense(=g . montanum) [29]使用GMAP [61].GMAP输出文件用于后续分析。使用以下参数去除冗余flnc:最小识别- 0.9,最小修剪覆盖- 0.85,允许接近indel-0。5 '端不同长度的映射FLNCs不认为是冗余的。将FLNCs映射到注释基因g . luofuense基因组被认为是已知的基因;否则,它们被归类为新的基因和新型同种型的已知基因。
功能标注与分类
所有新发现的基因均通过BLASTX v.2.2.26搜索(E-value < 1 × 10)注释−5)的基因本体论(GO,http://www.geneontology.org.)、京都基因和基因组百科全书(KEGG,http://www.genome.jp/kegg/),蛋白质(PFAM),Kog / Cog(蛋白质簇的簇,http://www.ncbi.nlm.nih.gov/cog/)、NCBI非冗余蛋白序列(NR, http//www.ncbi.nlm.nih.gov/)和瑞士语(http //http://www.expasy.org/sprot/)数据库和Hmmer v.1b2搜索(e-value <1×10−10)pfam(蛋白质家庭,http://pfam.xfam.org/)数据库(62那63].此外,使用在R中实现的GoseQ包进行富集分析[64], KEGG富集分析采用KOBAS 2.0版本[65].
AS和APA分析
使用Tapis管道进行基因结构分析[16].首先,我们确定了7种类型的可变剪接(AS)事件:可变3 '剪接位点、保留内含子、可变5 '剪接位点、跳过外显子、可变第一外显子、可变最后外显子和互斥外显子。此外,进行选择性聚腺苷酸化(alternative polyadenylation, APA)分析,并根据其聚(A)数对基因进行分类。
识别TFS和LNCRNA
通过使用TransDecoder对PFAM数据库进行搜索来识别具有开放阅读帧(ORF)的编码序列(CDS)[66].根据鉴定的CDS,通过植物转录因子数据库v.4.0 (http://planttfdb.cbi.pku.edu.cn)使用iTAK版本15.03 [67].使用四种方法识别LNCRNA:PC(编码潜在计算器),CNCI(编码 - 非编码指数),CPAT(编码潜在评估工具)和PFAM。LNCRNA,其长于200nt并具有至少两个外显子,不编码蛋白质,并分为四组:LincrNA,内肠道LNCRNA,感测LNCRNA和反义LNCRNA。
DEG识别和QRT-PCR验证
将Illumina测序的带有poly(N)和low scores的raw reads删除,并对其余reads的两端适配器进行修剪。被清理的读取被映射到g . luofuense基因组使用HISAT2 v.2.1.0 [68].使用R包edgeR通过一个缩放归一化因子计数和调整映射读数[69].映射读的数量被转换为每千个转录片段的片段数(FPKM)。为了识别差异表达基因(DEGs),我们将成熟和不成熟种子的三个复制样本的RNA数据分别合并,然后使用R package EBSeq v. 1.20.0 [70].检测结果符合以下要求:已校正P.-Value(由Benjamini&Hochberg方法调整)-0.005和日志2(褶皱变化)值1。
QRT-PCR验证
为了验证AS事件的发生,在全长转录组中,两个基因的表达,即。TNS000138667G03和TNS000973269G04通过qRT-PCR进行验证。其中两微克的RNA提取自成熟和未成熟的种子G. lufuens.e并根据制造商的协议进行cDNA合成。在执行中存在下列条件:10分钟在95°C(1周期),10 s在95°C, 30年代在55°C和15秒72°C(40周期),温度下降从95°C到60°C(0.5°C / 10 s)和终止在30年代25°C。进行基因电泳,检测不同长度的qRT-PCR产物。选择14个deg进行qRT-PCR验证。这g . luofuense使用actin基因作为内源性对照来估计ΔΔct方法的靶基因的相对表达[71].对每个样品进行3次重复,计算qRT-PCR基因表达值的平均值和标准差。
可用性数据和材料
来自合并的种子样本和来自六个样品的RNA-SEQ数据的PACBIO测序数据全部沉积在BIOPROMET登录号PRJNA622631下的NCBI序列读取归档(SRA)中。支持结果的数据包含在文章和其附加文件中。其他相关材料可在合理的请求上获得相应的作者。
缩写
- 度:
-
差异表达基因
- cd:
-
编码序列
- ORFS:
-
开放阅读框
- TFs:
-
转录因素
- 个人电脑:
-
编码潜在的计算器
- CNCI:
-
Coding-non-coding指数
- CPAT:
-
编码潜能评估工具
- APA:
-
可变聚腺苷酸化
- 读者:
-
全长读取
- nfls:
-
非全长读取
- 辅助/ IAA:
-
蟾蜍/吲哚-3-乙酸
- bHLH:
-
基本helix-loop-helix
参考
- 1.
侯c,汉弗莱。m,杜伯恩。欧。赖丁。C。对进化历史的新见解Gnetum(买麻藤目)。分类。2015; 64(2):239-53。
- 2.
HOU C,Wikströmn,Strijk J,Rydin C.使用高通量NGS,Sanger测序和形态地解决密切相关的裸子植物中的系统发育关系和物种分歧。植物系统中的Evol。2016; 302(9):1345-65。
- 3.
Thoday毫克。的雌花序和胚珠Gnetum africanum与笔记Gnetum飞.安机器人。1911;25(4):1101 - 35。
- 4.
麻粒类与贝内特类种子的一些相似之处。新植醇。1911;10(4):140 - 4。
- 5.
化学成分的测定Gnetum africanum(AFANG)种子。植物营养学报。2007;6(1):40-3。
- 6.
Bhat R, Binti Yahya N.评估belinjau (gnetum gnemon.种子面粉质量是开发新型食品和食品配方的基础。食品化学。2014;156:42-9。
- 7.
王永强,王永强。裸子植物生殖和被子植物花起源的基本知识。细胞生物学杂志。2010;21(1):118-28。
- 8.
侯c,李l,刘z,苏y,wan t.疯子箱基因的多样性和表达模式Gnetum luofuense- 用于功能多样性和演化的可执行。Trop Plant Biol。2020; 13:36-49。
- 9.
罗军,周军,张军。AUX / IAA.植物基因家族:分子结构、调控和功能。中国生物医学工程学报。2018;19(1):259。
- 10.
吴文婷,刘应祥,王玉强,李慧敏,刘建新,谭建新,何东东,白建文,马海林。演化分析AUX / IAA.植物基因家族具有双重起源和可变的核定位信号。中华医学杂志。2017;18(10):107。
- 11.
侯超,邓宁,苏玉芳。PacBio长读测序揭示了转录组的复杂性AUX / IAA.基因进化Gnetum(买麻藤目)。森林。2019;10(11):1043。
- 12.
齐涛,黄红梅,宋松,谢东东。一种bHLH-MYB复合物对茉莉酸介导的雄蕊发育和种子生产的调控拟南芥.植物细胞。2015;27(6):1620 - 33所示。
- 13.
杨志强,杨志强,杨超,王志强,陈东,谢勇,吴勇。番茄开花期长链非编码rna选择性剪接的鉴定和遗传分析。基因组学,2020;112(1):897 - 907。
- 14.
王志强,王志强,王志强。植物选择性剪接的全基因组比较分析。中国科学院院刊2006;103(18):7175-80。
- 15.
Elkon R,Ugalde AP,Agami R.替代裂解和多腺苷酸:范围,调节和功能。NAT Rev Genet。2013; 14(7):496。
- 16.
Abdel-Ghany SE, Hamilton M, Jacobi JL, Ngam P, Devitt N, Schilkey F, Ben-Hur A, Reddy AS。用单分子长读法对高粱转录组的调查。Nat Commun。2016;7:11706。
- 17.
王T,王H,Cai D,GAO Y,张H,王Y,林C,MA L,GU L.摩尔竹中的根茎相关替代剪接和替代多腺苷酸的综合性分析(植被类型).植物j . 2017; 91(4): 684 - 99。
- 18.
刘xx,梅文彬,Soltis PS, Soltis DE, Barbazuk WB。从没有参考基因组的单分子长读序列中检测选择性剪接的转录异构体。生态学报。2017;17(6):1243-56。
- 19.
赵强,高志飞,张丹,赵宝邦,董凤琴,付彩霞,刘丽娟,王宝兵。发展的动力杨树茎转录组。J Pl Biotech。2019; 17(1):206-19。
- 20。
赵颖,袁娟,李树华,贾树华,韩磊,徐磊(三叶草pratense通过单分子长读测序。植物学报。2018;18(1):300。
- 21.
胡,杨y,郑泽,牛z,杨y,万德,刘j,ma t.分析替代剪接和替代多腺苷酸杨树阿尔巴var锥体通过单分子长读取测序。前群体。2020; 11:48。
- 22.
默瑟TR,丁格ME,马特克JS。长链非编码rna:功能研究。中国农业科学。2009;10(3):155-9。
- 23.
lncrna在植物中的遗传和表观遗传效应。生物技术生物tec Eq. 2019;33(1): 429-39。
- 24.
刘杰,王鹤,楚纳。长度非编码RNA转录组植物。J Pl Biotech。2015; 13(3):319-28。
- 25.
吴y,郭j,王t,曹f,王g.叶片突变关联的长度非编码RNA的转录分析银杏叶L.BMC植物BIOL。2019; 19(1):527。
- 26.
叶杰,程胜,周旭,陈志金,谭军,郑军,徐飞,张伟,廖玉玲银杏叶揭示涉及类黄酮生物合成的转录变体。Ind Crop Prod. 2019;139:111547。
- 27.
王L,夏X,江H,卢Z,崔j,曹F,金B.新型LNCRNA的基因组鉴定与表征银杏叶.树木。2018;32(5):1429 - 42。
- 28.
MINOCHE AE,DOHM JC,Schneider J,Holtgrawe D,Viehover P,Montfort M,Sorensen TR,Weisshaar B,Himmelbauer H.利用用于真核基因预测的单分子转录物测序。基因组Biol。2015; 16(1):184。
- 29.
万涛,刘志明,李立峰,Leitch AR, Leitch IJ, Lohaus R,刘志军,辛海平,龚玉波,刘勇,等。麻藤属植物和种子植物早期进化的基因组。Nat植物。2018;4(2):82 - 9。
- 30.
李艳艳,戴超,胡超,刘志强,康晨。基于SMRT-和Illumina-based RNA-seq的草莓选择性剪接的全局鉴定。植物j . 2017; 90(1): 164 - 76。
- 31.
反义转录因子触发的靶向3 '处理拟南芥方法染色质沉默。科学。2010;327(5961):94 - 7。
- 32.
Simpson GG, Dijkwel PP, Quesada V, Henderson I, Dean C. FY是一种RNA 3 '末端加工因子,与FCA相互作用以控制拟南芥植物的过渡。细胞。2003;113(6):777 - 87。
- 33.
关键词:长链非编码rna,果实发育,颜色变化,转录组分析草莓属pentaphylla.Hortic杂志2019;6(1):42。
- 34.
Varshney D, Rawal HC, Dubey H, Bandyopadhyay T, Bera B, Kumar PM, Singh NK, Mondal TK。组织特异性长链非编码rna参与红茶香气的形成。2019; 133:79-89。
- 35.
Nystedt B, Street NR, Wetterbom A, Zuccolo A, Lin Y-C, Scofield DG, Vezzi F, Delhomme N, Giacomello S, Alexeyenko A.挪威云杉基因组序列和针叶树基因组进化。自然。2013;497(7451):579 - 84。
- 36.
Gramzow L,Theissen G.一个Hitchhiker的植物疯狂世界的指南。基因组Biol。2010; 11(6):214。
- 37.
花器官身份的发展:来自MADS房子的故事。植物学报。2001;4(1):75-85。
- 38.
Masiero S,Colombo L,Grini Pe,Schnitter A,拼写架。I型疯子箱转录因子的新兴重要性植物繁殖。植物细胞。2011; 23(3):865-72。
- 39.
MADS研究针叶树的基因组:用于确定种子植物MADS-box基因的祖先集。安机器人。2014;114(7):1407 - 29。
- 40。
Pnuuli L,Abu-Abeid M,Zamir D,Nacken W,Schwarz-Sommer Z,Lifschitz E.番茄中的Mads Box基因家族:与归属基因的花卉发育,保存的二级结构和同源性期间的时间表达Antirrhinum.和拟南芥.植物J. 1991; 1(2):255-66。
- 41。
Ando S, Sato Y, Kamachi S, Sakai S. MADS-box基因的分离ERAF17)及其表达与乙烯诱导黄瓜植株雌花形成的相关性(Cucumis sativusl .)。足底。2001;213(6):943 - 52。
- 42。
Lovisetto A,Guzzo F,Tadiello A,Toffali K,Favretto A,Casadoro G. Mad-Box基因的分子分析追溯到裸子植物的肉质果实发明。分子Biol Evol。2012; 29(1):409-19。
- 43。
新道,伊藤,上田,加藤,Hasebe M.裸子植物中MADS基因的特性gnetum parvifolium.及其对种子植物生殖器官进化的启示。另一个星球Dev。1999;1(3):180 - 90。
- 44。
桂福特TJ。AUX / IAA.蛋白质与生长素信号转导。植物科学进展,1998;3(6):205-7。
- 45.
刘德,陈继,鲁W.早期疾病响应的表达和调节AUX / IAA.草莓果实发育过程中的基因。中国人民大学学报(自然科学版)2011;38(2):1187-93。
- 46.
Yu H, Soler M, San Clemente H, Mila I, Paiva JA, Myburg AA, Bouzayen M, Grima-Pettenati J, Cassan-Wang H.全基因组分析AUX / IAA.基因家族在桉树:作用的证据EgrIAA4在木材的形成。植物生理学报。2015;56(4):700-14。
- 47.
Wang H, Jones B, Li Z, Frasse P, Delalande C, Regad F, Chaabouni S, Latche A, Pech J, Bouzayen M.番茄AUX / IAA.转录因子IAA9参与果实发育和叶片形态发生。植物细胞。2005;17(10):2676 - 92。
- 48.
冯军,齐磊,孙旭,林勇,董军,张胜AUX / IAA.基因(LaIAA2从落叶松属)。生物技术学报。2012;11(47):1064 - 9。
- 49.
Heisler MG, Atkinson A, Bylstra YH, Walsh R, Smyth DR。抹刀,一种控制心皮边缘组织发育的基因拟南芥,编码A.bHLH蛋白质。发展。2001;128(7):1089 - 98。
- 50。
Groszmann M,Paicu T,Smyth Dr。刮刀的功能域,涉及Carpel和水果开发的BHLH转录因子拟南芥.植物j . 2008;(1): 55 40-52。
- 51.
李旭,陈磊,洪敏,张勇,祖飞,文杰,易斌,马超,沈杰,屠建军bHLH转录因子BRTT8导致黄色种子外套Brassica Rapa..《公共科学图书馆•综合》。2012;7 (9):e44145。
- 52.
徐W,Dubos C,Lepiniec L. myB-BHLH-WDR复合物对类黄酮生物合成的转录控制。趋势植物SCI。2015; 20(3):176-85。
- 53.
张志强,张志强,张志强,等。植物MYB、bHLH和WDR蛋白竞争蛋白复合物形成的进化比较。acta photonica sinica, 2019; 40(12): 3197-209。
- 54.
Nemesio-Gorriz M,Blair PB,Dalman K,Hammerbacher A,Arnerup J,Stenlid J,Mukhtar SM,Elfstrand M.识别挪威MyB-BHLH-WDR转录因子复杂构件与类黄酮途径的调节相关联。前植物SCI。2017; 8:305。
- 55.
Suzuki A, Suzuki T, Tanabe F, Toki S, Washida H, Wu C-Y, Takaiwa F. myb相关基因的克隆和表达。基因。1997;198(1 - 2):393 - 8。
- 56.
Lenka Sk,Nims Ne,Vongpaseuth K,Boshar Ra,Roberts SC,Walker El。紫杉醇生物合成基因的茉莉酮响应表达水松cuspidata培养的细胞由BHLH转录因子Tcjamyc1,Tcjamyc2和Tcjamyc4负调节。前植物SCI。2015; 6:115。
- 57.
邓n,侯c,刘c,李米,巴特我,田y,陈文,杜c,江泽民,诗歌,什叶思光合特性在亚洲演变中的意义Gnetum(买麻藤目)。植物学报。2019;10:39。
- 58.
香港脊椎动物分散果实的特征。热带生态学报。1996;12(6):819-33。
- 59.
忘记P-M,Hammond DS,Milleron T,托马斯R.培训森林中的啮齿动物囤积的季节性和食物囤积:种子分散和幼苗招募的后果。在:忘记P-M,Hammond DS,Milleron T,Thomas R,编辑。种子分散和节俭:生态演化与保护。瓦莱德福:CAB INTERINATIONS;2002. p。241-56。
- 60。
Salmela L,竞争对手E.Rordec:准确,高效的长读误差校正。生物信息学。2014; 30(24):3506-14。
- 61。
吴丹丹,渡边CK。GMAP: mRNA和EST序列的基因组定位和比对程序。生物信息学。2005;21(9):1859 - 75。
- 62。
Finn Rd,Clement J,Eddy SR。HMMER Web服务器:交互式序列相似性搜索。Nucl酸res。2011; 39:29-37。
- 63。
Albert Va,Barbazuk WB,Der JP,Leebens-Mack J,Ma H,Palmer JD,Rounsley S,Sankoff D,Schuster Sc,Soltis de。这Amborella基因组和开花植物的演变。科学。2013; 342(6165):1241089。
- 64。
r核心团队。R:统计计算版本3.2.0的语言和环境。维也纳:统计计算基金会;2018.检索从https://www.r-project.org..
- 65。
谢C,毛X,黄家,丁Y,吴家,董某,孔L,高G,Li C-Y,Wei L. Kobas 2.0:用于富集途径和疾病的批量识别和鉴定的Web服务器。Nucl酸res。2011; 39:316-22。
- 66。
Haas BJ,Papanicolaou A,Yassour M,Grabherr M,血液PD,Bowden J,Couger MB,Eccles D,Li B,Lieber M.新创利用trinity平台从RNA-seq中重建转录本序列,进行参考文献的生成和分析。Nat Protoc。2013;8(8):1494。
- 67。
Zheng Y, Jiao C, Sun HH, Rosli HG, Pombo MA, Zhang PF, Banf M, Dai XB, Martin GB, Giovannoni JJ, et al. iTAK:一种植物转录因子、转录调节因子和蛋白激酶的全基因组预测和分类程序。摩尔。2016;9(12):1667 - 70。
- 68。
HISAT:低内存要求的快速拼接对准器。自然冰毒。2015;12(4):357。
- 69.
罗宾逊MD,麦卡锡DJ,SMYTH GK。编辑:用于数字基因表达数据的差异表达分析的生物导体包。生物信息学。2010; 26(1):139-40。
- 70.
Leng N,Dawson Ja,Thomson Ja,Ruotti V,Rissman Ai,Smits BM,Haag JD,Gould Mn,Stewart RM,Kendziorski C. Ebseq:RNA-SEQ实验中推断的经验贝叶斯分层模型。生物信息学。2013; 29(8):1035-43。
- 71。
Livak KJ, Schmittgen TD。利用实时荧光定量PCR技术分析相关基因表达数据−ΔΔCT方法。方法。2001;25(4):402 - 8。
确认
我们感谢TopEdit有限责任公司在本手稿的准备过程中对语言的编辑和校对。我们感谢曾西进博士Gnetum种子的照片。我们还感谢三位审稿人对文本提出的宝贵意见。
资金
This work was supported by the Special Fund for Hunan Forestry Science and Technology (XLK201812 to N.D.), Forestry Science and Technology Innovation Project of Hunan Province (HNGYL-2019-01 to N.D.), and the Natural Science Foundation of Guangdong Province, China (No. 2018A0303130163 to C.H.). The funding body had no influence over the design of the study and collection, analysis, and interpretation of data and in writing the manuscript.
作者信息
隶属关系
贡献
ND、CH、CL和YT构思设计了实验。ND和CH写了手稿。CH、BH、SS和SQ进行了实验。BH, FM和QS分析数据。CL和YT管理该项目。FM和SS对软件的使用提供了帮助。BH, MF, SS采集植物样品。ND, CH, CL和YT对手稿进行了修改。所有作者均已阅读并最终同意发表。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明没有竞争的经济利益。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
额外的文件1:
图。S1。完整成绩单的统计和质量控制。一种底部的长度分布。B.flnc的长度分布。C使用同构型级聚类算法的一致性同构型长度分布。D.统计和分类完整的文本对照g . luofuense参考基因组。E.映射上的读密度g . luofuense支架;这X-轴表示脚手架位置(Mb)y-AXIS表示中值读取密度(LOG2),绿色和红线分别代表支架的正股和负股。F所有映射全长转录物的比例和标识范围。红色和蓝色条分别表示全长读数的覆盖范围和身份。
额外的文件2:
图。S2。新型基因的注释概述g . luofuense种子。一种不同种子植物种类中NR注释的分布,X-axis表示注释读数的数量。B.Kegg浓缩注释的新型基因,X-axis表示注释读数的数量。C全长转录本的基因本体(GO)注释和分类。
额外的文件3:
表S1。详细信息处理PacBio测序数据。
额外的文件4:
表S2。PacBio测序数据中的详细信息经Illumina测序数据校正。
额外的文件5:
表S3。6个数据库注释的新基因数量摘要。
附加文件6:
表S4。详细信息由Illumina排序数据六g . luofuense种子样本。
附加文件7:
表S5。详细的基因组图谱信息来自六个转录组g . luofuense种子样本。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
邓娜,侯c,何博。等等。全长转录组和基因表达分析揭示了在种子发育过程中表达的基因和分子元件Gnetum luofuense.BMC植物杂志20,531(2020)。https://doi.org/10.1186/s12870-020-02729-1
收到了:
公认:
发表:
关键词
- 买麻藤目
- 全长转录组
- 功能基因
- 种子
- lncRNA