抽象的
背景
土壤盐分是影响作物生长和产量的主要非生物胁迫因子之一,严重制约着农业的可持续发展。藜麦营养价值高,对极端天气和土壤条件的适应性强,被认为是未来最有前途的作物之一。然而,藜麦对盐胁迫的适应性反应的分子机制仍然知之甚少。为鉴定耐盐候选基因,采用内参引导组装法,比较了在300 mM NaCl处理0、0.5、2和24 h时,两种不同耐盐性藜麦基因型在根中的表达情况。
结果
耐盐剂(ST)基因型显示出较高的种子萌发率和植物存活率,以及盐胁迫下的盐敏感(SS)基因型的育苗潜力更强。平均产生了38,510,203个高质量的清洁读数。鉴定了基因和基因组(Kegg)途径的重要基因本体(GO)术语和京都百科全书以更深地理解差异反应。转录组分析表明藜麦盐响应基因主要与次生代谢物,α-亚麻酸代谢,植物激素信号转导和代谢途径有关。此外,在ST基因型中的差异表达基因(DEGS)中显着富集了几种途径,例如苯基丙醇生物合成,植物 - 病原体相互作用,异喹啉生物碱生物合成和酪氨酸代谢。两种基因型的各个阶段都常见,鉴定为核心盐响应基因的各个阶段,包括一些转录因子成员,如MYB,怀疑和南汽,以及一些植物激素信号转导相关基因,如塔,PP2C和tify10a.,这在适应本物种的盐条件方面发挥着重要作用。通过定量实时PCR(QRT-PCR)检测21℃的表达模式,并确认了RNA-SEQ结果的可靠性。
结论
参与藜耐盐我们确定候选基因,以及一些在DEGS ST基因型完全表达。的DEGS共同对盐胁迫下两种基因型可能是关键基因藜适应盐度环境。这些候选基因主要通过参与活性氧调节的耐盐性(ROS)清除系统,蛋白激酶的生物合成,植物激素信号转导和其它重要的生物学过程。这些发现为深入理解监管机制基本藜的耐盐性网络提供了理论依据,也为提高未来育种计划的耐受盐度奠定了基础。
背景
土壤盐分是一个主要的非生物胁迫,以现代农业在干旱和半干旱地区,严重影响种子萌发,植物生长和发育,以及作物生产力[1,2].据估计,约有8亿公顷,占世界土地总面积的6.5%,受到盐胁迫的影响[3.].更糟糕的是,全球变暖和非理性开采和土地利用一直在加速土壤盐渍化,这将导致2050年耕地的一半损失[4,5].土壤盐渍化的扩大和世界人口的增长给农业发展带来了巨大的压力。6].因此,我们对筛选耐盐品种或通过基因工程改善植物的耐盐性来解决盐度问题是非常迫切的。
高盐度破坏体内平衡水势和离子分布,引起高渗应激,离子不平衡和氧化损伤,并最终危及植物生命[7,8].降低的叶片尺寸,气孔闭合,光合抑制和生物质减少是应对盐胁迫的反应策略[9].为了在盐胁迫下生存,植物进化出了一系列的反应机制,包括复杂的生理特性、代谢和信号通路以及分子网络,以增强其耐盐性[10.,11.].不同品种和品种的耐盐性存在明显差异。一般来说,耐盐植物保持高钾+/ Na+细胞质中的比例,对细胞功能非常重要,这些离子调节植物的生理和发育过程[12.].植物应对和适应盐胁迫主要由多个生物合成和信号通路通过基因表达调控[13.].在分子水平下,已经确定了与耐盐性有关的许多关键基因,并且详尽分析了它们的调节机制。AtHKT1(高亲和钾转运体1)驱动适应拟南芥通过限制根到茎的钠含量来降低盐度+交通运输(14.].过度表达四SOS1盐敏感基因提高了转基因菊花的耐盐性[15.].ZFP179锌指蛋白179通过调节氧化应激反应增强水稻的耐盐性[16.].SLCBL10(Calcineurin B样蛋白10)通过调节Na介导番茄中的耐盐耐药性+和Ca2+液泡中的通量[17.].过度表达GsPRX9(过氧化物酶基因)增强了大豆盐胁迫和抗氧化反应的耐受性[18.].异位表达PtCYP714A3(一种细胞色素p450单氧基酶基因)来自杨树trichocarpa抑制转基因水稻茎部生长和抗盐胁迫[19.].等离子体膜结合SbNHXLP(Na+/H+从高粱中提取的反转运蛋白(antiporter-like protein)与钠有关+排除,维持离子稳态,并减轻转基因番茄中的NaCl胁迫,更值得注意地增加了番茄的果实产量[20.].此外,已显示各种转录因子(TFS)参与盐应激反应,例如NAC [21.],基本亮氨酸拉链(BZIP)[22.]、APETALA2/乙烯响应因子(AP2/ERF) [23.], WRKY [24., basic helix-loop-helix (bHLH) [25.]和myb [26.].它们调节多个下游基因的表达水平,这可能最终影响植物的耐盐性。植物激素是复杂的发育过程和应力响应信号网络的重要调节因子[27.],和脱落酸(ABA)的作用[28.,乙烯(ET) [29.)、生长素(30.]、茉莉酸酯(JA) [31.芸苔类化合物(Br)[32.[植物对盐压力的关键部件进行了很好的研究。这些研究还证明,通过盐诱导的基因操纵的不同的生物途径和代谢过程是相互关联的并且在一起以抵抗盐胁迫。
藜麦 (Chenopodium藜麦Willd。)是一年一度的阔叶草本植物,属于苋科科家族,起源于南美安第斯泊州安第斯泊州地区[33.].藜麦是一种具有高营养价值,富含必需氨基酸,矿物质,膳食纤维,维生素和抗氧化成分的营养价值的假细胞作物[34.].对干旱、霜冻和盐胁迫的高度耐受性使藜麦能够适应各种极端气候和土壤条件[35.,36.,37.],对发展节水农业,提高旱地利用效率,改良滨海盐碱土,维护农业生态系统多样性具有重要的现实意义。由于其优异的谷物营养品质和对各种非生物胁迫的强耐受性,藜麦这一潜在作物引起了全世界的关注,联合国粮食及农业组织宣布2013年为国际藜麦年[38.].自7000年前自7000年前以来作为Andes的原住民居民的主要食物,奎奴亚藜多样性可以分为五个主要的生态文:索拉斯,安安兰省,高地,余山脉和沿海低地39.],而沙拉雷斯地方品种被认为具有最高的耐盐性[40].到目前为止,在30个国家的59个基因库中,超过16,400名奎奴亚藜及其野生亲属的加入[41.].然而,不同藜麦品种的耐盐性差异很大,有的甚至可以在与海水相似的盐浓度下生长,而有的则相对较弱[42.].藜麦耐盐性是近年来藜麦研究的热点,但主要是对其农艺性状和生理反应的研究。鉴定耐盐基因是通过基因工程进行耐盐作物育种的重要组成部分[43.].在许多植物中已经发现了许多与高盐度反应相关的基因和信号通路,但在藜麦中只有少数被记录下来。高质量藜麦参考基因组的发表和相关转录组研究的完成促进了盐反应基因的鉴定,并为加速藜麦基因改良提供了见解[44.,45.,46.].通过整合生理数据,RNA-SEQ和单核苷酸多态性(SNP)分析,在藜麦中鉴定了预测编码跨膜蛋白的一些候选耐盐基因[45.].此外,研究人员还对表皮膀胱细胞(一种与毛状体同源的细胞结构)进行了转录组测序,以揭示藜麦耐盐的机制[46.].然而,迄今为止,很少有人努力阐明藜麦适应盐胁迫的分子机制。
在本研究中,使用RNA-SEQ进行根部的比较转录组分析,以在分子水平下揭示盐度胁迫下的缩小响应,在两个染色藜基因型中:耐盐性QQ056和盐敏感37TE。我们在每次盐处理时间点(0.5,2和24小时)处的两个基因型中分析了差异表达的基因(DEGS)。鉴定了在盐胁迫下三个时间点的两种基因型共同的核心DEG,并通过定量实时PCR(QRT-PCR)验证它们的表达模式。还分析了这些盐响应基因所涉及的可能的生物方法和代谢途径,提供了有价值的见解,揭示了对盐胁迫适应的分子机制,以及在未来的育种计划中发育耐盐奎奴亚藜。
结果
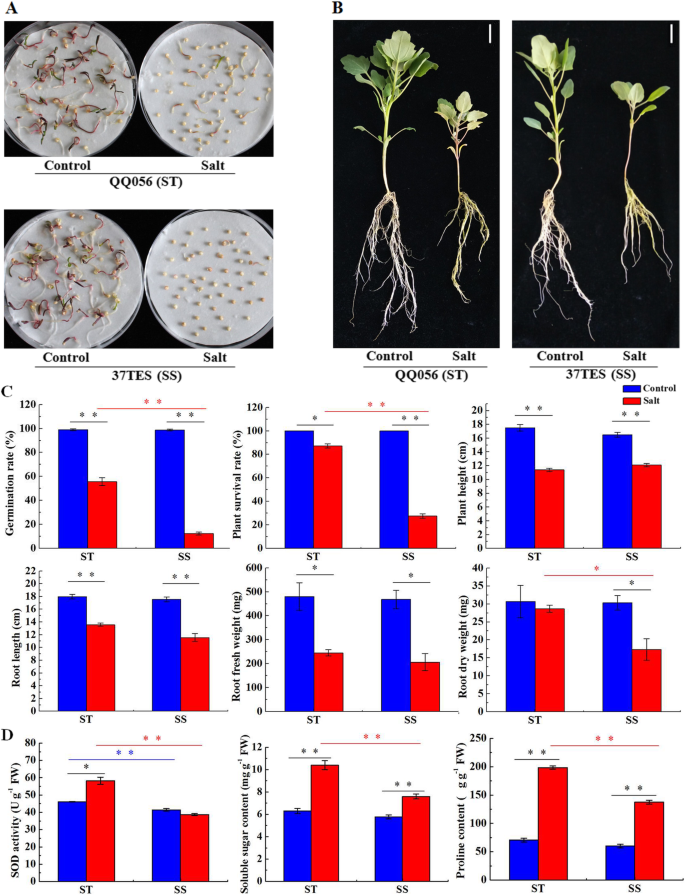
盐度胁迫下耐盐性和盐敏基因型之间的表型差异
与对照相比,300 mM NaCl处理下QQ056(耐盐性,ST)和37TES(盐敏感,SS)的种子萌发率均显著降低,但SS的下降明显高于ST(图2)。1a和c)。在盐胁迫下,ST的萌发率显著高于SS。同样,SS幼苗对盐胁迫的抵抗能力也弱于ST。1b).在对照条件下,虽然两种基因型植株成活率、株高、根长、根鲜重和根干重无明显差异,ST基因型植株成活率和根系干重的降低低于SS,这两项指标在盐处理下均显著高于SS(图2)。1C)。总之,ST基因型拥有比SS基因型更好的耐盐性。与对照,可溶性糖含量和两个基因型的脯氨酸含量相比均被显著增加,超氧化物歧化酶(SOD)活性在ST被显著惜售但在SS(图略有下降。1d)。除SOD活性外,在控制条件下,可溶性糖含量和两种基因型的脯氨酸含量没有显着差异。但是所有ST的所有三种生理指标都高于SS在盐处理下的SS。
RNA测序和转录组件
利用对照和盐处理(300 mM NaCl, 0.5, 2, 24 h)条件下28日龄ST和SS幼苗根系的RNA样本进行Illumina Genome Analyzer深度测序。平均生成41,470,921个原始读,每个样本经过清洗和质量检查后获得38,510 203个干净读(见表)S1).平均清除率可高达92.86%,证明了测序结果的高质量。约85.30%的reads被映射到藜麦参考基因组,33182448个reads被映射到唯一区域,2192951个reads被映射到多个区域(见表)S1).
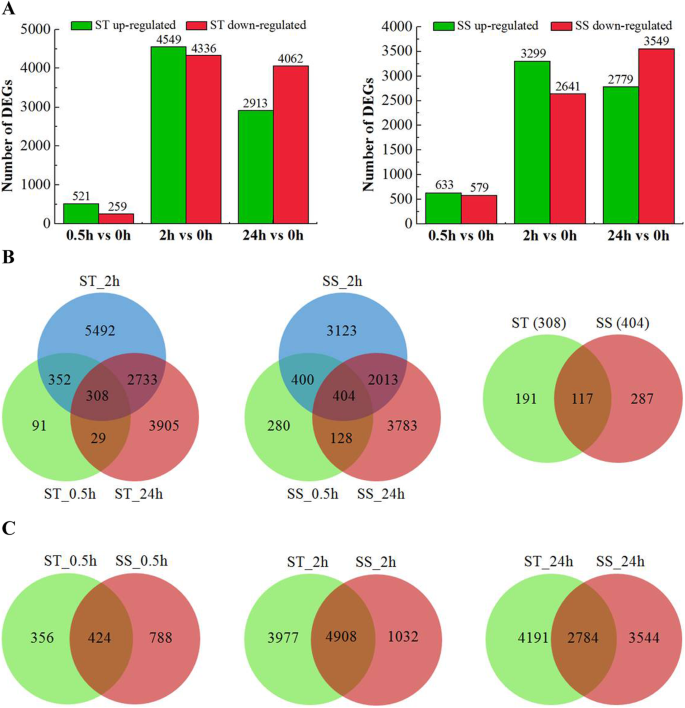
盐处理下二歧杆菌的鉴定与分析
与对照相比,在0.5、2和24 h ST/SS中分别有780/1212、8885/5940和6975/6328基因的差异表达(图2)。2a).盐胁迫下,ST组的DEGs总数大于SS组。然而,不同盐胁迫时间点ST和SS中上调和下调的DEGs数量也存在差异。其中,521/633、4549/3299和2913/2779在0.5、2和24 h ST/SS分别上调,而259/579、4336/2641和4062/3549则相应下调(图2)。2a).维恩图分析表明,盐反应基因具有基因型特异性和时间特异性,这可能是ST和SS基因型耐盐性差异的原因(图2)。2B和c)。
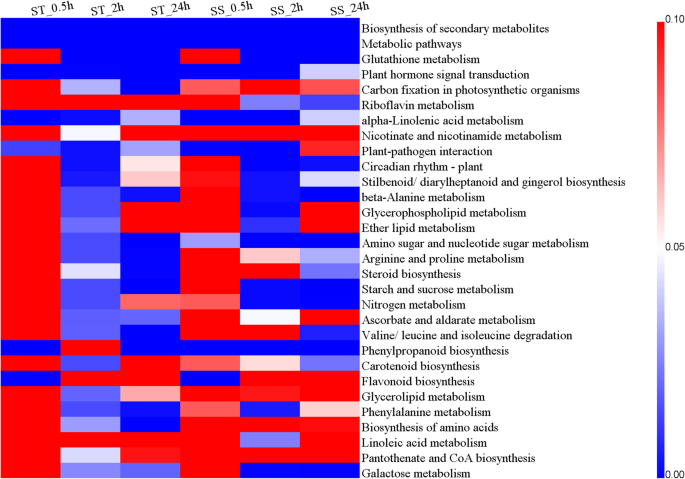
通过KEGG途径富集分析,揭示了几种重要的盐相关途径。这些途径主要涉及次生代谢产物的生物合成、氨基酸代谢、维生素代谢、脂质代谢、植物激素信号转导、类胡萝卜素的生物合成和碳水化合物代谢(见表1)S2).其中,在每个盐应激时间点的两种基因型中显着富集了次级代谢物,代谢途径,植物激素信号转导和α-亚麻酸代谢的生物合成(图。3.).植物激素信号转导的显着富集揭示了植物激素在盐胁迫中的重要性。基于271个背景基因在奎奴亚藜,25 / 30,133 / 106中的植物激素信号转导,分别在0.5,2和24小时的本途径中富集在该途径中(表S2).此外,一些途径最好丰富在圣或者党卫军盐胁迫时间点,如植物-病原互作在圣、氨基糖和核苷酸糖代谢和phenylpropanoid生物合成在党卫军,表明盐胁迫的响应机制的差异两个基因型(无花果。3.).
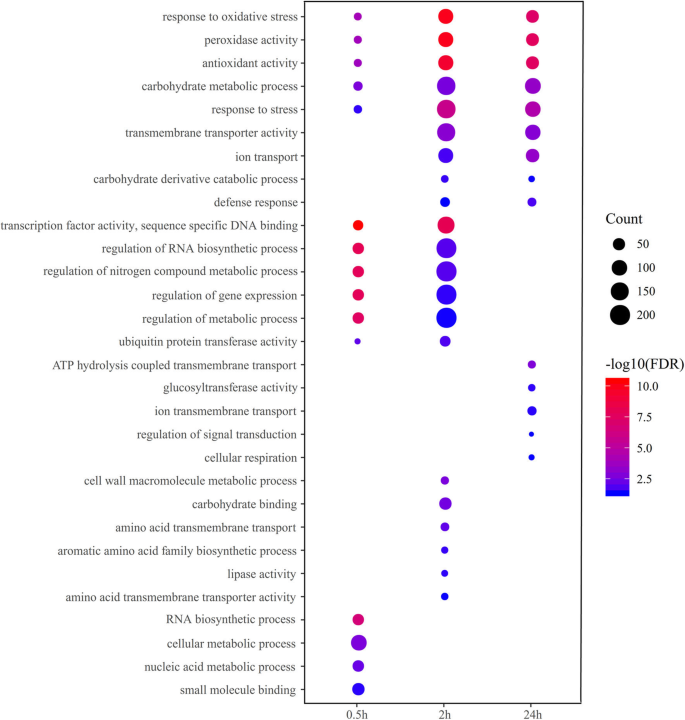
采用氧化石墨烯富集法对其生物学功能进行表征。结果表明,在所有盐胁迫时间点,两种基因型的氧化石墨烯中,包括对氧化应激的响应、氧化还原过程、氧化还原酶活性和抗氧化活性等多个氧化石墨烯项均显著富集,这已被证明与植物的耐盐性有关(见表)S3和S4).0.5 h(300毫米氯化钠压力,转录因子的活动,特定的DNA序列绑定,RNA生物合成过程的监管,和调节基因表达明显丰富在这两种基因型,而去氧化应激反应,过氧化物酶avtivity,抗氧化活性在盐胁迫2 h和24 h显著增强(图2)。4).这些表明,对盐胁迫的初始阶段主要涉及转录激活或表达一些重要基因的分子官能,随着盐处理时间的延伸,诱导了一些生物过程。同时,还揭示了转录因子对盐胁迫反应的重要作用。
盐胁迫响应于ST基因型的egs分析
在ST和SS的所有采样时间点分别通常鉴定了总共308和404℃,而191和287分别在NaCl治疗的ST和SS中专门发现(图。2b)。进一步分析显示,87个基因上调圣在191度以上,包括calcium-dependent蛋白激酶,ethylene-responsive转录因子,热休克因子蛋白,UDP-glycosyltransferase,转录因子WRKY MYB,可能对提高耐盐候选基因在圣(表S5).在ST的191个deg中,最具代表性的GO类别是与氧化应激反应相关的生物过程、对刺激的反应和对应激的反应、与过氧化物酶活性、抗氧化活性和序列特异性DNA结合相关的分子功能,与细胞外区和细胞壁相关的细胞成分(见表1)S6).显着富集的Kegg途径包括苯丙醇化生物合成,二次代谢物的生物合成和代谢途径(表S7).在苯丙醇化生物合成中富集11个基因,其中大部分与过氧化物酶有关。
盐胁迫下ST和SS的共同差异
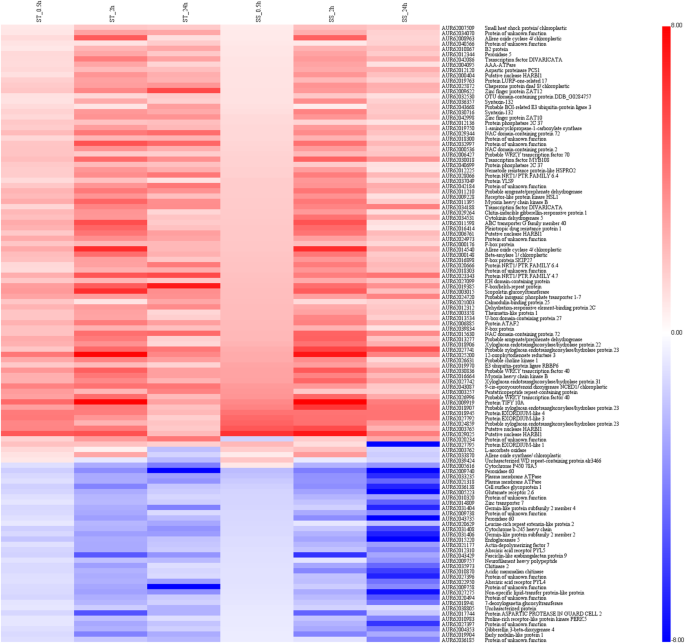
尽管在每个基因型的特异表达差异基因的,117度的视角是共同的两种基因型的不同阶段(图2b)。这些基因(83升至和34个下调)组成型活性,并且仍称为核心耐盐水平,包括许多过氧化物酶(PODS),蛋白质磷酸酶2Cs(PP2CS),锌指蛋白(Zats),F盒蛋白质和血浆膜ATP酶(表S8).特别是,还包括几种转录因子,例如Wrky,NaC和MyB,表明其在转录调节中的重要作用是有助于藜氨基藜耐盐性。
此外,组成型活性的基因的表达模式在盐胁迫条件下,两种基因型进行了分析。如图1所示。5,以热图显示117个核心DEGs的时空表达模式。虽然两种基因型的耐盐性存在较大差异,但这些基因表达模式相似,表明这些基因通常是在该物种响应盐胁迫时被诱导的。
盐胁迫响应ST和SS共常数的富集分析
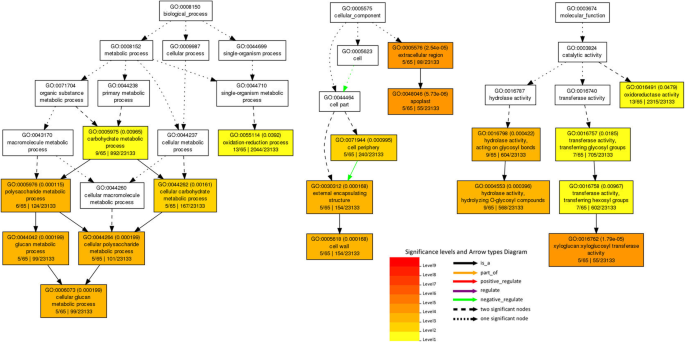
基于氧化石墨烯富集分析,将117个核心基因分为3个氧化石墨烯本体和32个术语(图2)。6).在多糖代谢过程、细胞葡聚糖代谢过程、细胞多糖代谢过程、葡聚糖代谢过程、细胞碳水化合物代谢过程、碳水化合物代谢过程、氧化还原过程等生物过程中,大量基因显著富集(图)。6和表S9).此外,一些氧化石墨烯类别在deg中的分子功能显著富集,包括木葡聚糖:木葡糖苷转移酶活性,水解酶活性,水解o -糖基化合物,水解酶活性,作用于糖基键,转移酶活性,转移己糖基,转移酶活性,转移糖基和氧化还原酶活性(图。6和表S9).因此,藜麦的耐盐过程非常复杂。
盐胁迫响应ST和SS共常数的Kegg途径浓缩分析
核心基因与KEGG数据库对齐,并分配到16 kegg途径(表S10).显著富集的途径有α -亚麻酸代谢、次生代谢产物生物合成、苯丙素生物合成、植物激素信号转导、代谢途径和泡囊运输中的SNARE相互作用(见表)S10).代谢途径表现出最大数量的DEG,表明激活许多生化反应,以增强对盐环境的适应。富含苯丙烷化生物合成的四个含量,其中三个被鉴定为豆荚。
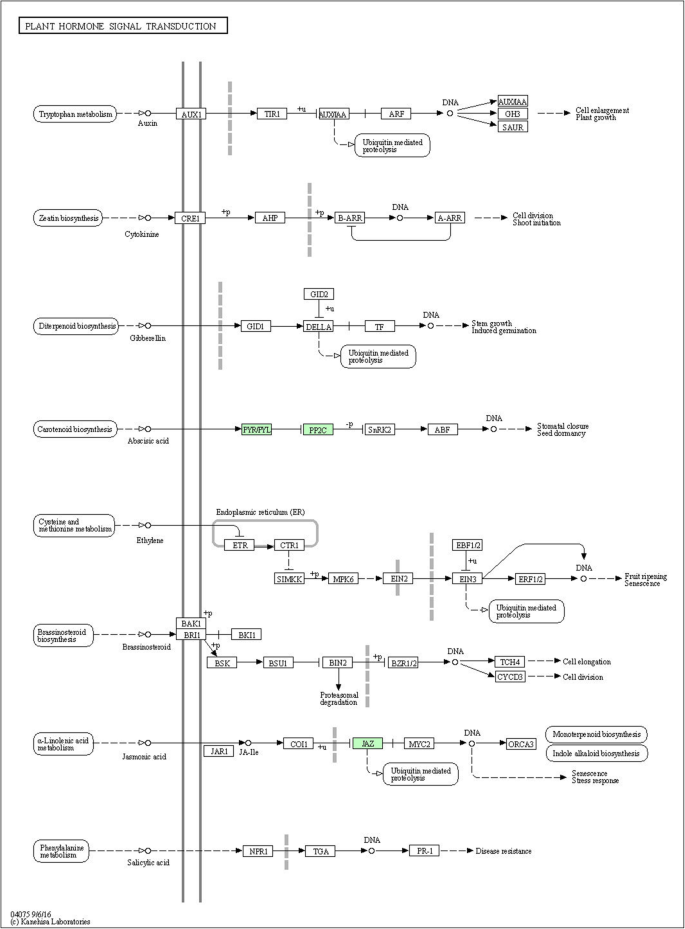
预计五种差异表达的转录物参与植物激素信号转导,包括两个PP2Cs(Aur62012136,Aur62040699),一个Tify10a(Aur62009919)和两个ABA受体Pyls(Aur62012310,Aur62022950)(表S8和S10).PP2C和PYL是ABA信号通路的重要参与者,它们的参与最终导致气孔关闭以响应盐胁迫(图2)。7).JA响应蛋白Tify10A可以立即诱导泛素介导的蛋白水解或间接促进单萜类化合物生物合成和吲哚生物碱生物合成以应对刺激(图。7).这PP2CS.和tify10a.在300 mM NaCl处理下,两种基因型均受到正调控,而所有供试负调控,表明它们在高盐度的适应中发挥着不同的作用。同时也揭示了植物激素在藜麦抗盐胁迫中的重要作用。
qRT-PCR验证RNA-Seq数据
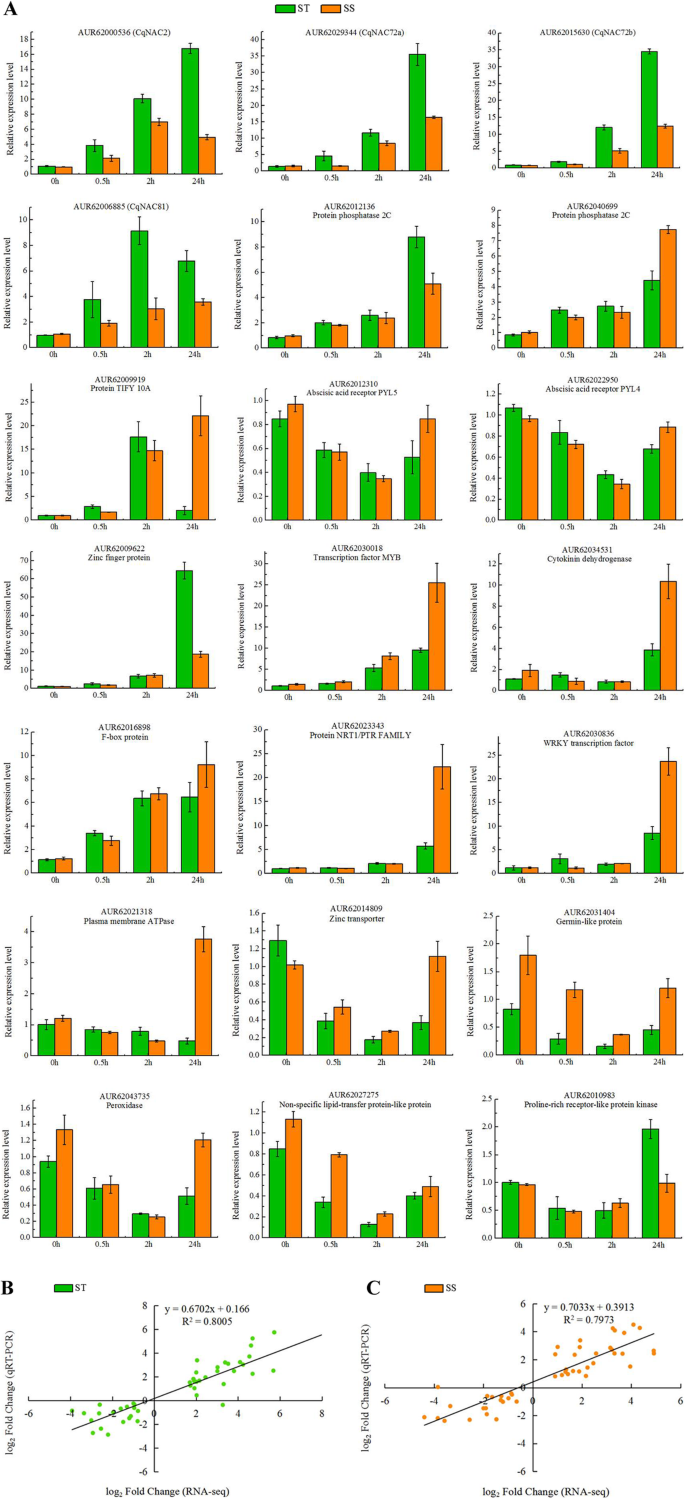
为了进一步验证RNA-SEQ的表达数据,在最初用于下一代测序的相同RNA样品上进行QRT-PCR。选择在盐胁迫下的两种基因型中常见的二十一段用于QRT-PCR分析,包括六种转录因子(四个NACS., 一MYB和一个怀疑)、5个植物激素相关基因(2个PP2CS., 一tify10a.和两个所有供试)和其他10个关键基因(图。8一种)。在这些中,13个上调和8表达下调(表S8).特定引物列于表中S11.要比较RNA-SEQ和QRT-PCR之间的表达数据,将相对表达水平变换为日志2折叠变化。QRT-PCR结果与ST中的RNA-SEQ数据强烈相关(R2 = 0.8005) (Fig.8b)及物料供应员(R2= 0.7973)(图8c),证明了本研究中RNA-Seq表达谱的可靠性。
讨论
土壤盐分是一种主要的环境压力,已成为全球作物生产和粮食安全的主要问题。虽然藜麦比其他作物更耐盐,但高于一定浓度阈值的高盐度显著影响其产量和质量。因此,有必要研究藜麦在高盐环境下的重要盐响应机制和耐盐机制,以开发高产稳产藜麦新品种。在本研究中,我们在300 mM NaCl胁迫0、0.5、2和24 h对两种不同基因型藜麦幼苗进行转录组分析,并鉴定出两种基因型中特别常见的、仅在ST中存在的DEGs,并进行进一步分析。这可能会为藜麦耐盐的候选基因和代谢途径提供深入的见解。其中,与ROS清除系统、蛋白激酶、植物激素信号转导和转录因子相关的基因占主导地位。
盐胁迫下活性氧清除系统
盐胁迫可以显着增加ROS的产生[47.,对细胞结构造成氧化损伤,最终导致程序性细胞死亡[48.].ROS生成和猝灭之间的平衡是细胞存活的先决条件,而细胞存活是由复杂的抗氧化系统维持的[49.].在适应外部环境应力中,植物已经开发出各种非酶促和酶的ROS解毒机制。抗氧化酶如荚,超氧化物歧化酶(SOD),抗坏血酸过氧化物酶(APX)和过氧化氢酶(猫)在清除ROS中起重要作用,并且可以在盐度应力下保持低水平的ROS [50.].本研究在两种基因型中均发现了大量与氧化还原酶活性和氧化还原过程相关的deg,提示氧化应激可能是由短期盐胁迫引起的。POD活性、抗氧化活性以及上述两项氧化石墨烯活性均在ST中显著富集,表明它们在提高耐盐性方面的重要性。POD是活性氧清除系统中重要的抗氧化酶,可由某些基因编码。我们发现POD5.(AUR62012344)在盐胁迫下,两种基因型中POD的编码基因均明显上调,表明清除ROS系统在藜麦耐盐性不高的情况下,在保护藜麦免受盐胁迫方面具有重要作用。
参与盐胁迫的蛋白激酶
蛋白激酶在植物开发和环境压力反应中起重要作用。除拷贝蛋白激酶(RLK)是膜锚定蛋白,通常携带细胞外亮氨酸的重复(LRR),其充当传感器和受体介导信号转导的[51.].过度的ATRPK1转基因拟南芥中的耐盐性增强[52.].在这里,一个rpk.(AUR62009228)和五个LRR-RPKS.(AUR62039001,AUR62021617,AUR62018804,AUR62007198和AUR62040226.)分别在两种基因型和ST基因中被鉴定为上调的DEGs,表明它们在藜麦抗盐胁迫中发挥重要作用。类受体激酶的另一个子类是富含脯氨酸的类扩张类受体激酶(PERKs),这是一组在细胞壁起传感器/受体作用的蛋白质。特别是在环境胁迫下,它们会引起植物细胞壁的变化,并抑制根的生长[53.].我们发现一个差异表达Perk.基因(AUR62010983)ST和SS均在盐处理下强烈调节,暗示其在适应盐水环境中的可能负调节功能。钙依赖性蛋白激酶(CDPK)对于调节非生物胁迫耐受性,包括干旱,冷,热和盐度是必不可少的[54.].在这项研究中,三个cdpks.(AUR62029467,AUR62024429.和AUR62026022)在盐胁迫下被发现在ST中有显著上调,这表明它们在提高藜麦耐盐性方面有巨大的潜力。
盐胁迫下的植物激素信号转导
植物激素在增强对环境胁迫的抵抗力方面起着关键作用[55.].在本研究中,在盐处理的不同时间点,ST和SS植物激素信号转导通路中都显著富集了大量的DEGs。ABA是植物适应非生物胁迫的重要信使[56.].ABA信号转导有三个重要组成部分:ABA受体PYR/PYL/RCARs、负调控因子PP2Cs和正调控因子SnRKs。之前的研究表明TaPP2C1通过激活抗氧化系统和不依赖aba的基因转录过程,使转基因烟草抵抗盐胁迫[28.].同样的,几PP2CS.在奎奴亚藜基因型中被发现高度上调(两个PP2CS.),并专门在ST (4PP2CS.)在盐胁迫下,表明其在ABA信号通路中的负调节作用,但抗盐性阳性作用。Tify蛋白,作为JA反应的阻遏物,在植物发育中发挥重要作用,对非生物应激的反应[57.].在目前的研究中,一个差异表达基因tify10a.在ST和SS共同响应于盐胁迫,这可能通过调节JA信号通路保护藜对盐胁迫被强烈诱导。此外,我们还发现了一个赤霉素(GA)有关的基因GA 3-β-双加氧酶(Ga3ox4.)(AUR62004353.)被认为参与了赤霉素生物合成途径,但在盐胁迫下,藜麦中赤霉素信号被强烈抑制,提示赤霉素信号可能也参与了藜麦的盐胁迫信号途径。然而,这些植物激素相关基因的分子机制仍需进一步研究。
藜麦盐胁迫相关转录因子的研究
NAC,BZIP,AP2 / ERF,WRKY,MYB和BHLH等转录因子调节盐响应基因的表达,并最终确定植物的耐盐水平[21.,22.,23.,24.,25.,26.,58.].转录组分析发现,在NaCl处理下,两种基因型有12个差异表达的转录因子,其中3个mybs.(AUR62042086,AUR62034188和AUR62030018),四个NACS.(AUR62000536,AUR62029344,AUR62015630和AUR62006885.), 三WRKYs(AUR62030836.,AUR62026996和AUR62006427.)和两个ZFPs(AUR62042998和AUR62009622).已经证明了两个ZFP.成员Zat10.和Zat12.在拟南芥中可能被盐胁迫诱导[59.,60.],而藜麦中的两个同源基因在本研究中也被高度上调,表明它们可能在盐胁迫中发挥作用。除了四个wrky和四个myb,还有一个小块土地(AUR62028234)和一个Bzip.(AUR62010368)在ST基因型的DEGs中也发现,所有这些DEGs都在盐胁迫反应中被强烈诱导,表明它们可能在提高耐盐性方面发挥作用。一些小块土地通过调节ABA和ET水平,提高对高盐度的耐受性[61.]但是,在盐胁迫的初始阶段,有些是通过扩增ROS活化的丝裂剂活化蛋白激酶(MAPK)级联信号[62.].这些结果表明,耐盐是一个极其复杂的过程,涉及多种生物过程和代谢途径,调控基因的转录网络和相互作用值得进一步研究。
结论
在本研究中,通过转录组分析鉴定了奎奴亚藜中耐盐性耐盐性的许多候选基因,这可能对未来奎奴亚藜的耐盐性育种具有很大的价值。其中大多数这些基因包括一些TFS,参与ROS清除,植物激素信号转导,次级代谢物的生物合成,以及代谢途径,表明藜麦的耐盐性是一种复杂的机制。这些发现提供了理解奎奴亚藜的分子调节网络的有价值的信息,并为奎奴亚藜的基因功能和耐盐性改善提供遗传资源和理论依据。
方法
植物材料和NaCl处理
从美国农业部(USDA)耐盐QQ056(ST)和盐敏感37TES(SS)的两种藜份材料用于这项研究。QQ056来源于智利与USDA号PI 584524,和源自37TES在美国新墨西哥州与USDA数埃姆斯13723.将种子表面消毒,用3%的过氧化水合物1分钟并漂洗使用无菌水数次,和then germinated at 28 °C under long-day conditions with a photoperiod of 16 h/8 h (day/ night) in a phytotron. Seven days later, the uniform seedling were transferred to half-strength Hoagland nutrient solution. 28-day-old seedlings were treated with 300 mM NaCl. After exposure for 0 (control), 0.5, 2, and 24 h, the root samples were frozen in liquid nitrogen and stored at − 80 °C for transcriptome sequencing and qRT-PCR validation. Phenotypic changes were observed after 14 d of NaCl treatment. Each treatment was set three biological replicates.
表型特征
盐胁迫对两种基因型的种子萌发的影响如下设计:将灭菌的种子与蒸馏水(对照)和300mm NaCl在28℃下的照明培养箱中的蒸馏水(对照)和300mM NaCl放置7d,分别,然后记录种子萌发率。在幼苗阶段,测量植物高度,植物高,根长,根部的植物高度,根长,根部新鲜重量和根系干重测量,以证明表型变化。同时,还通过杨等人的方法来确定SOD活性,可溶性糖含量和脯氨酸含量。[63.].对28日龄幼苗进行盐胁迫28 d,记录其成活率。采用SPSS Statistics 17软件进行统计分析。
RNA分离,图书馆施工和转录组测序
使用RNAprep Pure Plant Kit分离总RNA,并按照制造商的说明用DNase I(天根,北京,中国)处理。RNA浓度和质量由Thermo 2000生物分析仪和RNA NanoDrop (Thermo Scientific,美国)测定。使用TruSeq链mRNA LT样本准备试剂盒(Illumina, Cat)进行RNA-Seq文库制备。RS-122-2101,美国)根据制造商协议。最终文库采用QuantiFluor dsDNA系统(美国Promega)进行定量,并在Illumina HiSeq 2500平台(Illumina Inc.)上进行测序。贝瑞基因组公司,北京,中国。
读取映射、序列汇编和差分表达式
筛选适配器序列和低质量碱基后,将clean reads映射到藜麦基因组v1.0 (https://www.ncbi.nlm.nih.gov/genome/?term=quinoa)使用Tophat软件[64.].库读取对齐文件用作Cufflinks软件的输入[65.],它将读入晶状体组成。基因表达水平由每百万映射的读数(FPKM)每千碱基的碎片确定。显着的虚假发现率调整P值FDR <0.05,日志2以Fold Change≥1作为鉴别DEGs的标准。
富集分析
使用奇异富集分析(SER)分析工具与Agrigo V2.0的FDR <0.05进行富集分析DEG的富集分析[66.,并以藜麦基因组为背景。DEGs的KEGG通路分析采用KOBAS 2.0进行[67.].使用Benjamini-Hochberg校正法确定了显著富集的途径P- value <0.05。并且通过KAAS服务器获得植物激素信号转导途径的概述[68.].
Heatmap与聚类分析
利用HemI软件对DEGs进行层次聚类分析[69.].KEGG通路热图使用FDR值绘制。和LOG2在基因表达热图中使用折叠变化值。
QRT-PCR验证
对于RNA-seq的图书馆建设同样的RNA样品进行起诉定量RT-PCR分析。第一链cDNA,根据制造商的说明书,使用PrimeScript TM RT-PCR试剂盒(Takara,日本)合成。定量RT-PCR与根据制造商的方案,使用的FastStart通用的SYBR Green硕士(罗氏,德国)的实时PCR系统的StepOne版本2.1(Applied Biosystems公司,USA)进行。CqEF1a(AUR62020767.)作为内对照,使基因表达水平正常化[70].相对表达量以2——∆∆CT方法。QRT-PCR的引物呈现在表中S11.
数据和材料的可用性
本次研究的数据集以登录号(SRR11921127-SRR11921149)的形式保存在公开访问的NCBI序列读取归档(SRA)数据库中。
缩写
- 圣:
-
耐盐
- SS:
-
食盐过敏
- 可见:
-
差异表达基因
- 走:
-
基因本体论
- Kegg:
-
基因和基因组的京都百科全书
- ROS:
-
反应性氧气
- 荚:
-
过氧化物酶
- rlks:
-
Recptor-like蛋白激酶
- CDPK基因:
-
钙依赖性蛋白激酶
- PP2C:
-
可能是蛋白磷酸酶2C
- 阿坝:
-
脱盐酸
- JA:
-
贾斯莫酸盐
- TF:
-
转录因子
- 存在:
-
定量实时PCR
参考
- 1。
黄志强,王志强,王志强,等。植物耐盐机制的新认识及其在植物育种中的应用。植物学报2016;7:1787。
- 2。
Julkowska MM,Koevteet,Mol S,Hoefsloot H,Feron R,Tester Ma,等。根系结构响应盐胁迫的根系结构遗传成分。植物细胞。2017; 29(12):3198-213。
- 3.
Rozema J, Flowers T.作物盐碱化的世界。科学。2008;322:1478 - 80。
- 4.
Albaladejo I,Egea I,Morales B,Flores FB,Capel C,Lozano R等人。鉴定表型改变的关键基因res(通过盐度恢复细胞结构)番茄突变体及其在盐胁迫下的恢复。生物谷bioon . 2018;18(1):213。
- 5。
朱镕基JK。植物的非生物胁迫信号与响应。细胞。2016;167:313-24。
- 6。
盐囊:它们重要吗?植物科学进展。2014;19:687-91。
- 7。
朱镕基JK。植物耐盐。植物科学进展,2001;6(2):66-71。
- 8。
lv xy,陈sx,王yg。理解甜菜对盐胁迫的生理分子应答的研究进展。前植物SCI。2019; 10:1431。
- 9。
张JL,石H.生理和植物耐盐的分子机制。Photosyn Res。2013; 115:1-22。
- 10。
Gupta B,Huang B.植物盐度耐受机制:生理学,生物化学和分子表征。int j基因组学。2014; 2014:701596。
- 11.
朱镕基JK。植物耐盐性的遗传分析。植物杂志。2000;124:941-8。
- 12.
Kim MJ,Shin R,Schachtman DP。核因子调节拟南芥中的脱落酸反应。植物理性。2009; 151:1433-45。
- 13。
盐胁迫下植物的基因表达谱分析。植物科学学报2011;30:435-58。
- 14。
D,Chen Jg,Gao YQ,Li X,Chao ZF,Chen Zr等。AtHKT1驱动器的改编拟南芥通过减少花卉钠含量来盐度。Plos Genet。2017; 13(10):E1007086。
- 15.
高J,Sun J,Cao P,Ren L,Liu C,Chen S等人。组织na的变异+内容和活动SOS1两种物种和两个相关的菊花中的基因。BMC植物BIOL。2016; 16:98。
- 16。
孙淑娟,郭树清,杨旭,鲍玉梅,唐慧娟,孙辉,等。水稻耐盐新基因Cys2/ his2型锌指蛋白的功能分析中国生物医学工程学报。2010;61(10):2807-18。
- 17。
Egea I, Pineda B, Ortíz-Atienza A, Plasencia FA, Drevensek S, García-Sogo B,等。SlCBL10钙调磷酸酶b样蛋白通过调节钠来保证植物在盐胁迫下的生长+和Ca2+体内平衡。植物杂志。2018;176(2):1676 - 93。
- 18。
Jin T,Sun Y,Zhao R,Shan Z,Gai J,Li Y.过氧化酶基因的过度表达GsPRX9在大豆中赋予耐盐性。int j mol sci。2019; 20(15):3745。
- 19。
王c,杨y,王h,ran x,li b,张j等。细胞色素p450单氧化酶基因的异位表达PtCYP714A3从杨树trichocarpa减少射击生长,提高转基因水稻中盐胁迫的耐受性。植物Biotechnol J. 2016; 14(9):1838-51。
- 20。
Kumari PH, Kumar SA, Sivan P, Katam R, Suravajhala P, Rao KS,等。质膜中Na的过度表达+/H+脱紫甲样蛋白质(SbNHXLP)通过维持离子平衡来提高番茄的耐盐性和产量。植物学报2017;7:2027。
- 21。
黄Q,王Y,Li B,Chang J,Chen M,Li K,等。小麦的NAC转录因子Tanac29增强了转基因中的盐和耐旱性拟南芥.BMC植物BIOL。2015; 15:268。
- 22.
梁超,刘志强,王志强,等。bZIP转录因子GhABF2赋予棉花耐旱和耐盐性(gossypium hirsutumL.)。SCI批准。2016; 6:35040。
- 23.
张克,陈米,李兰,徐Z,陈X,郭继,等。大豆的过度表达GMERF3.基因,AP2 / ERF型转录因子用于增加对转基因烟草中的盐,干旱和疾病的耐受性。J Exp Bot。2009; 60(13):3781-96。
- 24.
梁启元,吴永华,王凯,白志云,刘清良,潘玉珍,等。菊花WRKY基因DgWRKY5增强转基因菊花中盐胁迫的耐受性。SCI批准。2017; 7(1):4799。
- 25.
OEEM M,Rong X,Li Z.解剖基本螺旋环 - 螺旋转录因子的作用,SLBHLH22在盐胁迫和干旱胁迫下,转基因植株的生长状况茄属植物lycopersicumL.前植物SCI。2019; 10:734。
- 26.
郭h,王y,王l,胡p,王y,jia y等。MYB转录因子基因的表达BPLMYB46.影响非生物胁迫耐受性和次级电池壁沉积Betula Platyphylla..生物技术学报2017;15(1):107-21。
- 27。
Bari R,Jones JDG。植物激素在植物防御反应中的作用。植物mol biol。2009; 69:473-88。
- 28。
胡伟,严燕,侯旭,何颖,魏勇,杨光,等。TaPP2C1,F2蛋白磷酸酶2C基因,赋予转基因烟草中的盐胁迫抗性。Plos一个。2015; 10(6):E0129589。
- 29。
曹文辉,刘军,何晓军,穆瑞林,周海林,陈少英,等。乙烯反应的调节影响植物的盐胁迫反应。植物杂志。2007;143(2):707 - 19所示。
- 30.
jung jh,公园厘米。拟南芥种子萌发中盐应激信号的植物素调节。植物信号表现。2011; 6(8):1198-200。
- 31.
伊斯梅尔A,黎曼男,葡萄藤尼克P的茉莉酮酸酯途径介导的耐盐性。J Exp Bot。2012; 63(5):2127-39。
- 32.
崔飞,刘丽,赵强,张志强,李强,林波,等。拟南芥泛素缀合物UBC32是一种Erad组分,其在芸苔类固醇介导的盐胁迫耐受性。植物细胞。2012; 24(1):233-44。
- 33。
Yasui Y, Hirakawa H, Oikawa T, Toyoshima M, Matsuzaki C, Ueno M, et al.;拟制的自交系的基因组序列Chenopodium藜麦,具有巨大环境适应性和突出营养特性的同种异体一种倍性作物。DNA Res。2016; 23(6):535-46。
- 34。
Koziol MJ。藜麦化学成分和营养评价(Chenopodium藜麦Willd)。食品学报,1992;5(1):35-68。
- 35。
Jacobsen S-E,Monteros C,Christiansen JL,Bravo La,Corcuera LJ,Mujica A.奎奴亚藜的植物反应(Chenopodium藜麦Willd。)以各种鉴生阶段冻结。Jagron欧元。2005; 22:131-9。
- 36。
Adolf VI,Jacobsen S-E,Shabala S.藜麦毒性耐盐机制(Chenopodium藜麦Willd)。Environ Exp Bot。2013; 92:43-54。
- 37。
Razzaghi F,Ahmadi Sh,Adolf Vi,Jensen Cr,Jacobsen S-E,安德森MN。奎奴亚藜水关系与蒸腾(Chenopodium藜麦野生的)盐碱化和土壤干燥。作物学报,2011;197(5):348-60。
- 38。
Zurita-Silva A, Fuentes F, Zamora P, Jacobsen S-E, Schwember AR.育种藜麦(Chenopodium藜麦):潜力和前景。摩尔品种。2014;34:13-30。
- 39。
Risi JC, Galwey NW。安第斯山脉的藜属谷物:印加现代农业作物。应用生物学1984;10:145-216。
- 40.
蔡志强,高强。5个高原藜麦品种耐盐生理生化机制的比较。植物学报。2020;20(1):70。
- 41。
奎奴亚藜的全球扩张:趋势与限制。植物学报2016;7:622。
- 42。
Hariadi Y,Marandon K,Tian Y,Jacobsen S-E,Shabala S. Ionic和Quinco的渗透关系(Chenopodium藜麦野生的)植物生长在不同的盐度。acta photonica sinica . 2011;62(1): 185-93。
- 43。
彭志鹏,何胜,龚伟,孙军,潘志,徐飞,等。盐胁迫诱导的两种棉花基因型差异表达基因及其转录调控的综合分析BMC基因组学,2014;15(1):760。
- 44。
贾维斯DE,何YS,Lightfoot的DJ,SchmöckelSM,李B,BORM TJ等。基因组Chenopodium藜麦.大自然。2017;542:307-12。
- 45。
Schmöckelsm,Lightfoot dj,Razali R,Tester M,Jarvis de。鉴定施用型跨膜蛋白的盐度耐受性Chenopodium藜麦通过集成生理数据,RNASEQ和SNP分析。前植物SCI。2017; 8:1023。
- 46.
邹超,陈安,肖磊,等。高质量的藜麦基因组组装提供了深入了解盐囊耐盐的分子基础和特殊的营养价值。细胞研究》2017;27(11):1327 - 40。
- 47.
Miller G,Suzuki N,Ciftci-yilmaz S,Mittller R.反应性氧气物种稳态和干旱和盐度应力的信号传导。植物细胞环境。2010; 33(4):453-67。
- 48.
张浩,韩斌,王涛,陈胜,李辉,张勇,等。植物盐反应机制:来自蛋白质组学的见解。蛋白质组学杂志2011;11:49-67。
- 49.
Chaturvedi Ak,Patel Mk,Mishra A,Tiwari V,JHA B.的SBMT-2来自嗜睡症的基因赋予非生物胁迫耐受性,并调节转基因烟草中的ROS清除。Plos一个。2014; 9(10):E111379。
- 50.
Habib Sh,Kausar H,Saud HM。植物生长促进relizobacteria通过ROS-Scavenging酶提高秋葵中的盐度胁迫耐受性。BioMed Res Int。2016; 2016:6284547。
- 51.
袁N,袁S,李ž,周男,吴P,胡Q,等人。应力诱导因子2,富含亮氨酸重复激酶对基础植物病原体的防御。植物理性。2018; 176(4):3062-80。
- 52.
田中平,Maruyama K, Osakabe K, Todaka D,等。膜结合类受体蛋白激酶1 (RPK1)的过度生产增强了拟南芥的非生物胁迫耐受性。[J] .中国生物医学工程学报。2010;28(12):9190-201。
- 53.
Humphrey TV,Haasen Ke,Aldea-Brydges Mg,Sun H,Zayed Y,Indriolo E等人。perk-kipk-kcbp信号负极调节根系生长拟南芥.J Exp Bot。2015; 66(1):71-83。
- 54.
Atif RM, Shahid L, Waqas M, Ali B, Rashid MAR, Azeem F,等。植物钙依赖蛋白激酶(CPKs)信号通路对非生物胁迫耐受性的影响中华医学杂志。2019;20(21):5298。
- 55.
杨X,赵T,饶P,高K,杨X,陈Z,等人。的转录谱Populus tomentosa冷应激。2019; 135:283-93。
- 56.
邓胜,马军,张磊,陈峰,桑志,贾志,等。新转录组测序和基因表达谱分析Magnolia Wufengensis.响应冷应激。BMC植物BIOL。2019; 19(1):321。
- 57。
2 .中国小麦(小麦)TIFY家庭和角色Triticum Durum TDTify11a.在盐胁迫耐受性。Plos一个。2018; 13(7):E0200566。
- 58。
Deinlein U,斯蒂芬AB,堀江T,罗W,许G,施罗德JI。植物的耐盐性机制。趋势植物SCI。2014; 19:371-9。
- 59。
锌指蛋白Zat12在拟南芥胞质抗坏血酸过氧化物酶1的表达中起重要作用。中国生物化学杂志。2004;27(12):11736-43。
- 60.
Sakamoto H, Maruyama K, Sakuma Y, Meshi T, Iwabuchi M, Shinozaki K,等。拟南芥Cys2/ his2型锌指蛋白在干旱、寒冷和高盐胁迫条件下具有转录抑制因子的功能。植物杂志。2004;136(1):2734 - 46所示。
- 61.
刘德,陈X,刘j,ye j,guo z.大米ERF转录因子OSERF922.负面调节抵抗力magnaporthe oryzae.和耐盐性。J Exp Bot。2012; 63(10):3899-911。
- 62.
王志强,王志强,王志强,等。盐响应ERF1调控水稻对盐胁迫的初始反应中活性氧依赖的信号通路。植物细胞。2013;25(6):2115 - 31所示。
- 63.
杨a,戴x,张wh。一个R.2R3.MYB型基因OsMYB2参与水稻耐盐、耐冷和耐脱水。中国生物医学工程学报。2012;63(7):2541-56。
- 64.
Kim D,Pertea G,Trapnell C,Pimentel H,Kelley R,Salzberg SL。TOPHAT2:在存在插入,缺失和基因融合的情况下,转录om的精确对准。基因组Biol。2013; 14(4):R36。
- 65。
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR,等。TopHat和袖扣RNA-seq实验的差异基因和转录本表达分析。Nat Protoc。2012;7(3):562 - 78。
- 66。
天t,刘y,yan h,你q,yi x,du z等。Agrigo v2.0:2017年农业社区的GO分析工具包。核酸RES。2017; 45(W1):W122-9。
- 67。
谢超,毛欣,黄建平,丁勇,吴军,董胜,等。KOBAS 2.0:用于标注和识别富集途径和疾病的网络服务器。核酸学报2011;39(Web服务器版):W316-22。
- 68。
KAAS:一种自动基因组注释和路径重建服务器。核酸Res. 2007;35(Web服务器版):W182-5。
- 69。
邓炜,王Y,刘Z,程H,薛Y.Hemi:用于说明热插拔的工具包。Plos一个。2014; 9(11):E111988。
- 70.
刘建军,王瑞,刘伟,张华,郭颖,温瑞。热休克蛋白70的全基因组特性Chenopodium藜麦和表达分析Cqhsp70s对干旱胁迫的反应。基因。2018;9(2):35。
致谢
我们衷心感谢韩钊先生对我们实验室的帮助和有益的讨论。
资金
这项工作得到了江苏农业科技创新基金(CX(19)3116)的支持。融资组织在这项研究中没有作用。
作者信息
隶属关系
贡献
SP和GM构思和设计了实验;SP进行了实验,分析了测序数据,并写了纸张。这位作者都读到并批准了最终手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
RNA-Seq结果总结。
附加文件2:表S2。
在盐胁迫下显著富集ST和SS中DEGs的KEGG通路(校正)P值< 0.05)。
附加文件3:表S3。
不同时间点氧化石墨烯在ST/SS中的富集分析。
附加文件4:表S4。
在不同时间点的ST和SS中常见的富集术语分析。
附加文件5:表S5。
在所有盐胁迫时间点,ST基因型只上调的基因列表。
附加文件6:表S6。
GO术语在ST基因型中仅在DEG中显着富集。
附加文件7:表S7。
仅对ST基因型的DEGs进行KEGG途径分析。
附加文件8:表S8。
参与藜麦对盐胁迫反应的核心基因集。
附加文件9:表S9。
GO术语在奎奴亚藜的核心面积中显着富集。
附加文件10:表S10。
奎尼诺藜的核心Degs的丰富的Kegg路径。
附加文件11:表S11。
QRT-PCR的引物序列。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
史鹏,顾国华,王志强,等。不同基因型藜麦在盐胁迫下的转录组分析和基因表达谱分析。BMC植物BIOL.20,568(2020)。https://doi.org/10.1186/s12870-020-027531.
收到了:
接受:
发表:
关键字
- RNA-SEQ.
- 转录组分析
- 藜麦
- 盐胁迫
- 候选基因
- 微分表达式