摘要
背景
生长素是植物生长发育和胁迫反应的关键。小型植物(Saur.)是高等植物中最大的早期生长素应答基因家族。然而,功能却很少Saur.基因是已知的由于许多家庭成员之间的功能冗余。
结果
在这项研究中,我们进行了一个系统发育分析,利用795个SAURs的蛋白质序列来自中国Anthoceros Angustus.,Marchantia polymorpha,Physcomitrella金属盘,江南卷柏,银杏叶,Gnetum Montanum.,Amborella trichopoda.,拟南芥蒂利亚纳,栽培稻,玉米,大豆,截形苜蓿和Setaria Italica..系统发育树表明,Saur蛋白可以分为10分钟和三种亚壳,并且三种苔藓物种的Saur蛋白仅位于亚家族III中,这表明他们可能是祖先。从苔藓植物到植物病,Saur.家庭出现了很大的扩张。的数量Saur.FABACEAE物种中的基因显着高于其他植物中的基因,其可以与FABACEAE谱系中的独立全基因组重复事件相关。系统发育树也表明了Saur.在被子植物中,单子叶和双子叶的基因独立扩展。保守基序和蛋白结构预测表明,高等植物的SAUR蛋白具有高度保守性,几乎所有的SAUR蛋白中都存在两个亮氨酸基序I残基,这说明这些残基对SAUR蛋白的稳定性和功能起着至关重要的作用。表达式的分析Saur.使用来自水稻和大豆的公共可用RNA-SEQ数据的基因表明了同一思工中的成员的功能相似性,其也通过QRT-PCR进一步证实。SAUR职能的概述还表明SAUR函数通常在亚克士内一致。
结论
本研究提供了进入的进化和功能的见解Saur.从苔藓植物到花生植物的基因家族,特别是在豆科植物中。未来的调查了解Saur.家庭成员应该使用一个分支作为学习单元。
背景
作为固着生物,植物必须通过综合发育信号和环境信号来适应环境变化。生长素是最早被发现的植物激素,在植物生长发育和胁迫反应中发挥着重要作用[1.].3个基因家族被认为是生长素反应的早期和主要基因:生长素/吲哚乙酸(AUX / IAA.),格雷琴Hagen 3(GH3),小型植物(Saur.)[2.,3.].Saur.最初可以在2-5分钟内最初诱导基因[4.].有趣的是,大多数Saur.基因缺乏内含子[3.,5.],并且可以在许多水平下监管;例如,Saur.由于位于3′非翻译区的一个高度保守的下游元件(DST),基因可以在转录后受到调控,这导致mRNA以不依赖生长素的方式不稳定[1.,6.,7.,8.].
第一个Saur.基因从绵延处理的大豆伸长的胚源鉴定出来(大豆)[9].在过去的30年里,会员们Saur.在许多其他的心血管中已经鉴定了基因家族,因此包括植物中的大型基因家族,例如拟南芥蒂利亚纳(81.Saur.基因,包括两个假基因)[2.],栽培稻(58包括两个伪原)[10],玉米(79) (3.],Gossypium raimondii就(145) (11],柑橘(70) [12],植被类型(44)13],Boehmeria Nivea.(71) (14]以及Citrullus lanatus(65)[15].许多成员Saur.基因家族是高频率串联和片段复制的结果,这导致了类似基因之间的功能冗余[6.].
直到现在,几Saur.基因已被证明在植物生长、发育和胁迫反应的不同过程中发挥作用。例如,AtSAUR63促进细胞伸长,从而产生更长的下胚轴、雄蕊丝、花瓣和花序梗[16].过度的AtSAUR36和AtSAUR49促进叶片衰老[17,18,19].的阿斯图尔62.和阿尔斯库75.突变体在离体和体内显示正常花粉活力,但花粉管生长存在缺陷[20.]. 类SAUR50蛋白参与向日葵的向日葵运动(向日葵[21].许多Saur.同源物在睡莲花昼夜节律的开放和关闭中起关键作用[22,23].的索尔41该亚家族由脱落酸诱导调控细胞的增殖和耐盐性拟南芥蒂利亚纳苗(24].的thermo-responsiveAtSAUR26亚家族表现出高频率的基因变化,与适应局部温度气候相关[25].
提高我们对生物进化和功能的认识Saur.在本研究中,我们首先利用植物中的795条SAUR蛋白序列进行系统发育分析Anthoceros Angustus.,Marchantia polymorpha,Physcomitrella金属盘,江南卷柏,银杏叶,Gnetum Montanum.,Amborella trichopoda.,拟南芥蒂利亚纳,栽培稻,玉米,Setaria Italica.,大豆和截形苜蓿. 基因的保守基序和蛋白质结构预测及基因表达谱分析Saur.以家庭为例,探讨其可能的功能Saur.基因。此外,我们总结了所有识别的功能Saur.基因。
结果
Saur蛋白质从藻类鉴定到更高的植物种类
我们从植物血统12数据库的爆炸搜查中识别出来自以下植物物种的Saur蛋白家族成员:Physcomitrella金属盘(18)成员,拟南芥蒂利亚纳(81个,包括两个假基因),栽培稻(58,包括两个假基因)和玉米(79)。这与以前的研究结果一致[2.,3.,10,26].同时,首次鉴定了3、5、15条SAUR蛋白序列Anthoceros Angustus.,Marchantia polymorpha和江南卷柏,分别。在裸子植物,Saur.家庭成员银杏叶(42)和Gnetum Montanum.(37)为首次鉴定。在花植物中,首次鉴定出26个和58个SAUR蛋白序列Amborella trichopoda.和Setaria Italica.,分别。有趣的是,共鉴定出141和236条SAUR蛋白序列截形苜蓿和大豆。这些结果表明Saur.在进化的过程中,家庭成员大大增加了。此外,没有一个Saur.在7种绿藻的基因组中发现了同源基因(Chlamydomonas Reinhardtii.,盐藻,Volvox Carteri.,Coccomyxa子面素软糖c - 169,微单胞菌CCMP1545,微单胞菌sp。rcc299和Ostreococcus lucimarinus.)及四种新近获批注的链藻(spirogloea muscicola,中间人Endlicherianum.,Mesostigma Viride和Chlorokybus atmophyticus)使用Saur蛋白序列拟南芥蒂利亚纳作为查询。来自不同物种的所有已鉴定的SAUR蛋白序列用于下游分析(补充表1.).
编码的Saur蛋白是长度为55-423氨基酸的多肽,预测分子量范围为6.38-46.81kDa和理论等电点(PI)的范围为4.58至12.在进一步的分析中,我们使用了植物-MPLOC服务器 (http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)[27]预测所识别的Saurs的可能蛋白质定位。超过60.8%的Saur蛋白含有核靶向信号,预计其他蛋白质将局部定位于细胞膜,细胞质,叶绿体或线粒体等(补充表2.).
Saur蛋白的系统发育分析
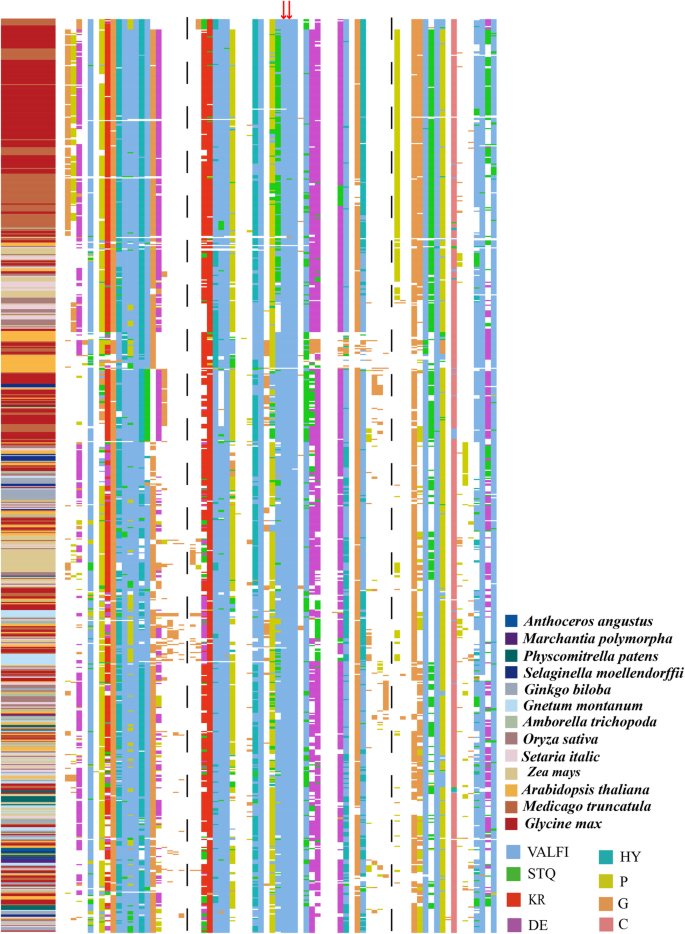
深入了解Saur.我们使用SAUR蛋白序列,排除假基因,从植物Anthoceros Angustus.(3),Marchantia polymorpha(5),Physcomitrella金属盘(18),江南卷柏(15),银杏叶(42),Gnetum Montanum.(37),Amborella trichopoda.(26),拟南芥蒂利亚纳(79),栽培稻(56)玉米(79),Setaria Italica.(58),大豆(236)和截形苜蓿(141)分别通过FastTree v2.1和IQ-TREE v2.0.6构建SAUR蛋白家族的系统发育树。在这两棵树的基础上,我们将SAUR蛋白划分为3个亚家族和10个分支,并将其划分为1 ~ 10组。1.补充图。1.,补充数据表1.,2.).3种苔藓植物的SAUR蛋白均分布在ii亚科的2个分支中,表明这2个分支可能是它们的祖先。单子叶植物和真子叶植物的大部分序列都聚在一个进化支系中Saur.基因家族在单子叶植物和真子叶植物中独立扩展。SAUR的大多数成员银杏叶,Gnetum Montanum.已经与红细胞中的一个平行形成独立的分支,这与他们的进化状态一致。Fasttree和IQ树树中的大多数Clade 1成员都是大豆的蛋白质截形苜蓿(无花果。1.补充图。1.).这可能是豆科植物分支中基因复制或独立基因组复制的结果。此外,FastTree和IQ-TREE均显示Fabaceae物种形成了一个独特的进化谱系(枝1),而在其他共享分支中,真子叶植物SAUR蛋白成员的数量大致相同(图1)。1.补充图。1.).我们进一步重建系统发生树Saur.基因家族成员来自含羞草和cercis canadensis.,这是Fabaceae家族以外的姐妹分类群,在一起拟南芥蒂利亚纳,Glycine Max,Medicago Truncatula.系统发育树可分为10个分支Saur.基因家族从截形苜蓿和大豆只有在分支1有非常大的扩张(补充图。2.).Synteny分析是一种有用的工具,用于建立基因之间的正非关系和功能联系。然后我们进行了同步分析Saur.基因拟南芥蒂利亚纳,大豆和截形苜蓿(补充图。3.).结果表明Saur.染色体上的基因簇拟南芥蒂利亚纳与多重有明显的共时关系吗Saur.的多个染色体上的基因簇截形苜蓿和大豆.这些结果表明,发生了许多Saur.Fabaceae的成员是由于独立的基因组复制而产生的。
生长素小rna的最大似然系统发育树(畜群)在十三个植物物种中。从所有Saur蛋白的多个序列对齐构建树Anthoceros Angustus.,Marchantia polymorpha,Physcomitrella金属盘,江南卷柏,银杏叶,Gnetum Montanum.,Amborella trichopoda.,拟南芥蒂利亚纳,栽培稻,玉米,Setaria Italica.,大豆和截形苜蓿利用FastTree v2.1中的极大似然法,结合JTT + CAT模型。然后在FigTree v1.4.4中澄清SAUR成员。SAUR科划分为3个亚科和10个支。在相应的分支标记Bootstrap支持率。文章中所报道的SAUR亚支系和提到的成员也被标注在上面。根据左上角简化的陆生植物系统发育,不同门的基因分支被着色。用不同的彩色圆点代表不同物种的SAUR成员
SAUR蛋白特有的保守核心区域和结构
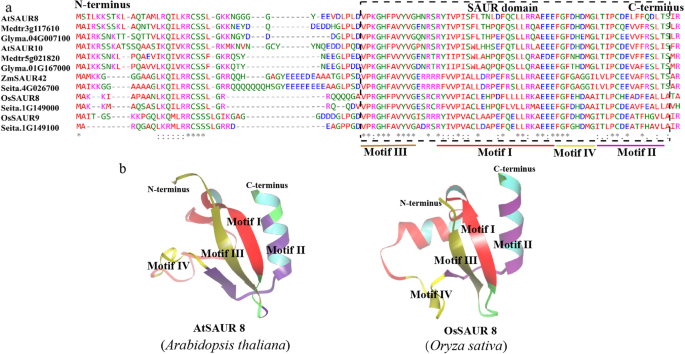
所有Saur蛋白的多序列对齐Anthoceros Angustus.,Marchantia polymorpha,Physcomitrella金属盘,江南卷柏,银杏叶,Gnetum Montanum.,Amborella trichopoda.,拟南芥蒂利亚纳,栽培稻,玉米,大豆,截形苜蓿和Setaria Italica.结果表明,该序列共有一个约60个氨基酸残基的保守核心区,称为SAUR结构域(fig .)。2.,3.),与以往的报告相符[1.,6.].此外,我们还观察到,与一般单子叶植物相比,真子叶植物中大多数SAUR成员含有更多的保守氨基酸位点。几乎所有SAUR蛋白序列中都发现了两个形成疏水核心的亮氨酸残基,这说明这两个残基在SAUR蛋白的折叠和基本功能中起着关键作用(图1)。2.).
我们在SAUR蛋白的保守区域内鉴定了五个基序Physcomitrella金属盘,拟南芥蒂利亚纳,栽培稻,玉米,Setaria Italica.,大豆和截形苜蓿(补充图。4.). 大多数SAUR成员都包含了母题I到IV和 ~ 20%的SAUR序列含有motif V。值得注意的是,SAUR蛋白Physcomitrella金属盘仅包含基序I ~ III,表明高等植物中的SAUR蛋白进化出了新的基序。然而,这些母题的功能目前尚不清楚,有待进一步研究。
先前的研究表明,这两棵树的枝2中的AtSAUR10亚枝可以诱导细胞伸长(图1)。1.补充图。1.)[28].枝2中与AtSAUR10具有密切同源关系的SAUR成员高度保守,尽管N-和c -端区域保守程度低于核心区域(图1)。3.a).大部分SAUR蛋白如图所示。3.a被预测定位于叶绿体(补充表2.),其可以与N-或C末端区域的相似性相关联。Atsaur8和Ossaur8的保守序列显示了类似的三级结构和类似的基序(图。3.b) ,正如使用SWISS-MODEL服务器预测的那样[29]并用GROMACS2019.1进行分子动力学模拟(http://manual.gromacs.org/2019.1/index.html.)[30.motif I为β-sheet, motif II为典型的α-螺旋,motif III为β-sheet, motif IV为motif I和motif II之间的连接体(图1)。3.b) 说明AtSAUR8和OsSAUR8的功能可能相似。然而,每个基序在维持蛋白质稳定性或活性方面的确切功能还需要进一步研究。
表达式的分析Saur.基因
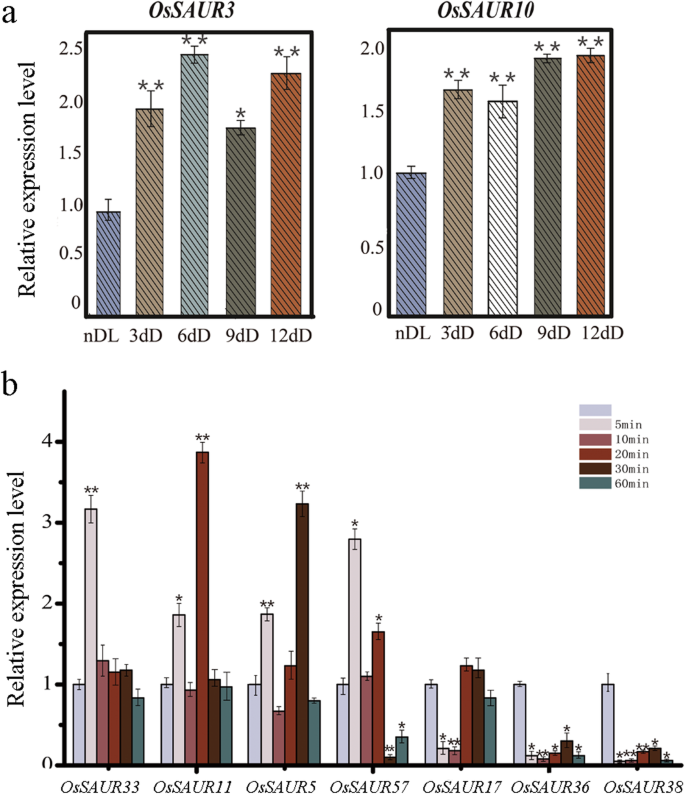
表达式分析是理解基因功能的有用工具。使用RNA-SEQ数据进行综合表达分析,以探索表达式轮廓Saur.水稻和大豆的基因(图。4.补充图。5.).对所有的表达水平进行了估计OsSAUR从11个组织和器官中检测到基因,除了8个基因(OsSAUR39,OsSAUR37,OsSAUR4,OsSAUR48,OsSAUR47,OsSAUR46,OsSAUR33和OSSAR21.)(图。4.).大多数的OsSAUR枝支5的基因在营养期表达量高,而在生殖期表达量低,这与它们的叶绿体定位有很好的一致性。1.,4.补充图。1.,补充表2.).奥梅尔2012.和OSSAR27.在授粉后25天在胚胎(DAP)后25天在胚胎中表达两棵树的植物8(图。1.,4.补充图。1.),这表明该分支的成员参与了水稻的胚胎发育。几乎所有的格姆索尔FastTree树枝3 (IQ-TREE树枝6)的基因表达模式相同,即开花后25 ~ 42天(DAF)的种子中表达量高,而14天(DAF)的荚壳和花中表达量低。1.,补充无花果。1.,5.).四个格姆索尔基因(格莱玛。3. g259700型,7 g051700 Glyma。,16 g020700 Glyma。和glyma。1 G137500.)在两个树的枝4中主要表达于种子(图。1.,补充无花果。1.,3.),这意味着该思想家的成员参与了种子发展。和....相比OsSAUR基因,数量更多格姆索尔基因表现出相似的表达模式。此外,基于数字基因表达库,我们观察到表达了许多OsSAUR基因是由非生物和生物胁迫诱导的,特别是盐胁迫、干旱胁迫和稻瘟病(补充图。6.),这表明Saur.基因介导应激反应,尽管迄今为止很少有研究调查这种功能。然后我们研究了表达模式OsSAUR暗处理后的fastree树(IQ-tree树的第7分支)的第6分支中的基因。结果表明OsSAUR3和奥梅尔10.在硫酸盐幼苗中表达高度表达(图。5.a). Jain et al.(2006)观察到OsSAUR7在同一枝系中,黄化苗的含量也显著高于正常苗[10这表明它们在光线和激素反应中发挥着类似的作用。这些结果表明,同一支系成员的表达模式往往相似。
澄清回应Saur.生长素基因的表达模式OsSAURs以10 μM IAA处理的水稻叶片为对象,随机选择不同枝系的基因。结果显示了极其不同的生长素响应表达模式(图。5.b) 是的。三个基因(OsSAUR33,奥梅尔11.和OsSAUR5)在养肝治疗后5分钟迅速诱导,而三个基因(奥美拉目州,OsSAUR36和OsSAUR38)在5 min内受到外源生长素的强烈抑制。奇怪的是,OsSAUR5710 μM IAA处理后,在30 min时受到强烈抑制。这些结果表明,表达Saur.基因与外源性养肝施用时间密切相关并表明Saur.基因在生长素介导的信号通路中起着极其复杂的作用。
确定的功能Saur.基因
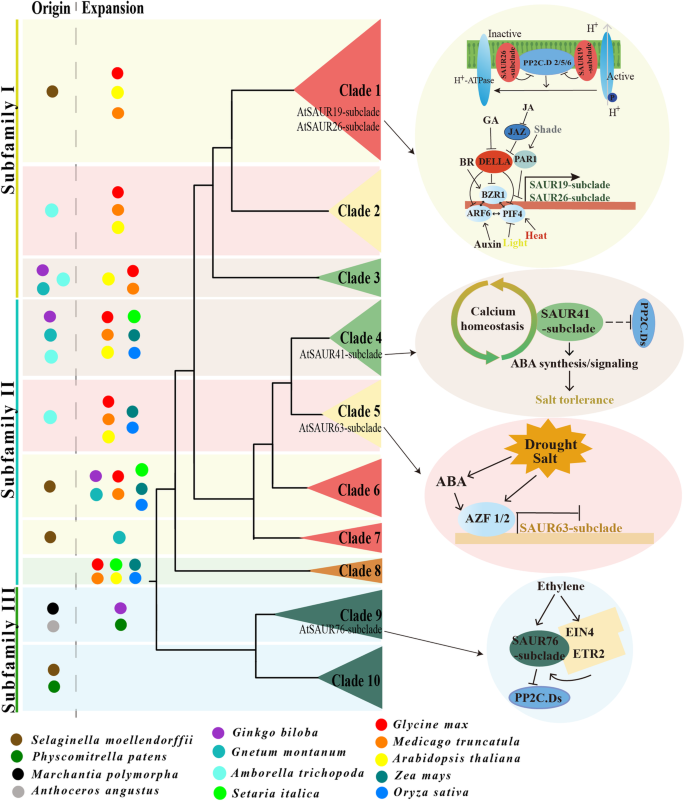
为了探索SAUR的功能,我们总结了所有目前确定的功能Saur.基因(图。6.,补充表。3.). 虽然SAUR蛋白影响植物生长发育的各个方面,但其分子机制可归纳为相对较少的过程。这两个树的分支1中的AtSAUR19亚基和AtSAUR26亚基抑制PP2C-D磷酸酶,然后激活质膜H+-ATP酶活性促进细胞扩张(图。1.,6.补充图。1.)[25,31].atsaur76亚分支通过与乙烯不敏感4 (EIN4)和乙烯受体2 (ETR2)的相互作用介导乙烯信号转导,促进植物的生长。1.,6.补充图。1.)[32].AtSAUR49通过抑制衰老抑制蛋白磷酸酶(SSPP)激活衰老相关受体样激酶(SARK)介导的叶片衰老信号[19]. AtSAUR70在体内以钙依赖的方式与钙调素结合[42],表明生长素信号转导与第二信使Ca之间存在联系2+/钙调蛋白。OsSAUR39过度表达对抗毒素生物合成和运输[43].此外,AbSAUR1过度表达显著提高了临床上被视为抗胆碱能药物的托烷生物碱的总产量,其产量是对照的3.55倍[44].研究人员还观察到,在同一进化谱系的基因显示出相似的功能,因此研究Saur.atsaur19 -亚支和atsaur26 -亚支,atsaur10 -亚支2,atsaur63 -亚支5,atsaur41 -亚支4,atsaur76 -亚支9。1.,6.补充图。1.).
简化的模式和函数Saur.植物进化中的基因。在植物进化的过程中,Saur.从苔藓植物到花生植物的基因家族扩张较大,各分支均经历了不同程度的扩张(左图)。右面板的图。6.显示功能Saur.响应激素和环境信号的基因:基于质膜H介导的酸生长+- atpase,生长素应答的SAUR蛋白(枝1中的AtSAUR 19-和AtSAUR 26亚支)激活质膜H+- atp酶通过抑制PP2C-D磷酸酶促进细胞扩张[31].进化枝9中的atsaur76亚分支通过与乙烯不敏感4 (EIN4)和乙烯受体2 (ETR2)相互作用介导乙烯信号转导促进植物生长[32]. SAUR41亚基被脱落酸诱导,调节细胞增殖和耐盐性,维持钙稳态[24].植物生长素反应因子6 (ARF6)、油菜素唑抗性因子1 (BZR1)和光敏色素相互作用因子4 (PIF4)可直接调控转录AtSAUR19-子代码和AtSAUR26-Subclade独立或复杂[33]感应激素和环境信号包括生长素,油菜素内酯(BR),赤霉素(GA),茉莉酸(JA),光,热和阴影[25,28,33,34,35,36,37,38,39,40].拟南芥aba反应通路中的锌指蛋白1 (zinc-finger protein 1, AZF1)和AZF2可以抑制AtSAUR63亚分支的表达[41]
讨论
在适应不同陆地环境的过程中,植物已经进化出一套激素来响应发育和环境信号[45].与细胞分裂素和乙烯相比,生长素信号在进化中出现的较晚,最初发现于链植物藻类[46,47].但没有S毛同源物在链光藻和其他藻类中发现,猜测Saur蛋白似乎相对出现。Saur.苔藓植物的基因首次被鉴定Physcomitrella金属盘[26]. 在本研究中,我们首先确定Saur.基因的参考基因组序列Anthoceros Angustus.(3),Marchantia polymorpha(5),江南卷柏(15),银杏叶(42),Gnetum Montanum.(37),Setaria Italica.(58),含羞草(62),cercis canadensis.(48)截形苜蓿(141),和大豆(236)。如此大规模的扩张Saur.从苔藓植物到花生植物的科应该有助于极大地平衡发育和环境信号,这对陆生植物的开花至关重要(图。6.).与数量相比Saur.在其他双子叶植物中,大量的基因存在于大豆和大豆的基因组中截形苜蓿(无花果。1.,6.补充图。1.).结合Saur.豆科植物与水稻基因的同源性分析拟南芥蒂利亚纳(补充图。3.),这表明Saur.家庭扩张可能与Fabaceae植物的全基因组重复相关[48].我们观察到单子叶植物的扩张少于一般的真子叶植物,这也被以前的研究证实Saur.棉花等植物的基因数(145)[11],柑橘(70)[12,竹(44)[13],苎麻(71)[14和西瓜(65)[15](补充图。7.).
了解蛋白质结构对于确定蛋白质的功能及其与其他蛋白质的相互作用是至关重要的。蛋白质结构预测对生物学研究具有深刻的理论和实践影响[49].在本研究中,我们鉴定了SAUR蛋白中5个保守的基序,发现大多数SAUR蛋白包含基序I到IV,约20%的SAUR家族成员包含基序V(补充图)。4.). 这一结果与棉花的预测结果相似[11]. AtSAUR8和OsSAUR8的三级结构表明基序I到IV产生了类似的结构(图。3 b),但各基序在维持蛋白质稳定性和活性方面的精细功能有待进一步研究。此外,我们发现motif I的两个亮氨酸残基位于疏水核心位置,几乎存在于所有SAUR蛋白中(图1)。2.),并可能参与蛋白质相互作用,如与PP2C的相互作用。D (31].然而,它们在SAUR蛋白生化功能中的关键作用有待于未来通过点突变遗传实验来证实。
SAUR蛋白的功能仍然是一个谜,尽管之前的重大突破(图。6.,补充表3.). 我们检测了7个随机选择的基因的表达水平OsSAUR对IAA治疗反应的基因。IAA处理5min后,4个基因表达量上调,3个基因表达量下调(图。5.b).数字基因表达分析也表明许多OsSAUR这些基因对盐、干旱和稻瘟病有响应。4.). 功能Saur.抗稻瘟病基因值得进一步研究。最近的一项研究报告说SLSAur69.通过抑制极性养蛋白运输来利用番茄果对乙烯的敏感性,以影响不处理的致成熟的过渡,这揭示了SLSAur69在养肝和乙烯之间介导串扰[50]. AtSAUR26亚家族被鉴定为生长热反应性的QTL,表现出与温度相关的自然多态性,影响植物结构的热反应性以适应当地温度[25].这些结果表明Saur.来自不同的植物的基因参与植物生长和应激反应的多种过程。
本表达分析Saur.基因表明表达模式在同一思工的成员之间相似。例如,OsSAUR3,奥梅尔10.和OsSAUR7FastTree树枝6 (IQ-TREE树枝7)的黄化幼苗中表达量较高(图1)。5.)(10].大多数的OsSAUR在植物相中高度表达植物相的基因,并在叶绿体中局部化(图。4.,补充表2.).四个格姆索尔基因(Glyma.3 g259700,格莱玛7 g051700型,Glyma.16 G020700和格莱玛1 137500元)在胰液体中,主要在种子中表达(图。1.补充图。1.补充图。5.). 这些结果表明,同一进化谱系中的基因具有功能相似性,这一点得到了功能实验的证实。例如,分支1中的AtSAUR19亚基和AtSAUR26亚基通过调用质膜H参与所谓的“酸生长”+atp酶激活(51]第4亚类中的AtSAUR41亚类维持钙稳态并调节干旱和耐盐性(图。1.,6.补充图。1.).
方法
鉴定和序列分析Saur.家庭成员
SAUR蛋白序列拟南芥蒂利亚纳用作植物血统12数据库的爆炸搜索(得分> 50,电子值<0.01)中的查询(https:////phytozome.jgi.doe.gov.).的SAUR成员的序列拟南芥蒂利亚纳,大豆,截形苜蓿,Marchantia polymorpha,Physcomitrella金属盘,江南卷柏和Amborella trichopoda.已下载.获得的SAUR成员序列栽培稻,玉米和Setaria Italica.具有相同的方法,但使用Saur蛋白序列栽培稻作为疑问。我们使用的是Saur蛋白序列拟南芥蒂利亚纳作为搜索SAUR成员标识的查询Anthoceros Angustus.,Gnetum Montanum.,银杏叶,含羞草和cercis canadensis.通过本地BLASTP(得分> 50,电子值<0.01)的NCBI-BLAST-2.10.1 +(ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/latest/).这五种物种的蛋白质序列从Datead网站下载(https://datadryad.org/stash/dataset/doi:10.5061/dryad.msbcc2ftv,https://datadryad.org/stash/dataset/doi:10.5061/dryad.0vm37)以及GigaScience GigaDB存储库(https://db.cngb.org/search/literature/27871309.,http://gigadb.org/dataset/101044,http://gigadb.org/dataset/101049分别)。Saur.会员鉴定叶绿素物种(Chlamydomonas Reinhardtii.,盐藻,Volvox Carteri.,Coccomyxa子面素软糖c - 169,微单胞菌CCMP1545,微单胞菌sp。rcc299和Ostreococcus lucimarinus.)及链藻(spirogloea muscicola,中间人Endlicherianum.,Mesostigma Viride和Chlorokybus atmophyticus的SAUR蛋白序列进行BLAST搜索拟南芥蒂利亚纳在Phytozome 12数据库(https:////phytozome.jgi.doe.gov.)及NCBI数据库(https://www.ncbi.nlm.nih.gov.)分别。795个SAUR蛋白序列Anthoceros Angustus.,Marchantia polymorpha,Physcomitrella金属盘,江南卷柏,银杏叶,Gnetum Montanum.,Amborella trichopoda.,拟南芥蒂利亚纳,栽培稻,玉米,大豆,截形苜蓿和Setaria Italica.与clustalx 2.1对齐[52]并用Jalview手动检查[53]. 保守区域Saur.基因家族(〜60个氨基酸)大大对齐,在非保守区域中存在局部同源性。手动移除一些极具间隙的位置,并且保持了几乎所有与局部同源性的非保守区域。最后获得序列对准用于构建FastTree和IQ树树。使用protparam分析Saur蛋白的物理化学参数(http://web.expasy.org/protparam)[54].Plant-mPLoc在线工具(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)[27用来预测Saur蛋白的亚细胞定位。
Saur家族的系统发育分析
对SAUR蛋白的多重序列比对进行了系统发育分析。利用fastreev2.1构建系统发育树[55]与JTT + CAT模型和IQ-TREE v2.0.6 [56]分别使用JTT + R9模型。通过执行1000次重复的引导分析,评估了对每个节点的支持。
SAUR蛋白的多序列对准与基序预测
SAUR家族成员的蛋白质序列被用于产生多重序列比对和使用Clustal Omega的可视化分析(https://www.ebi.ac.uk/Tools/msa/clustalo)[57]和Jalview [53], 分别。保留每个序列的保守部分,然后使用MEME施加用于基序预测的基序预测(http://meme-suite.org).
植物材料和生长条件
亚洲栽培米(oryza sativa subsp。japonica.简历。本研究使用的是日本裸(Nipponbare),该公司于2004年完成了基因组测序。该水稻品种的收集符合中国的制度和国家指南,种子存放在我们的实验室。水稻种子按前面所述的方法处理和生长[58].对于养肝治疗,将2周龄幼苗孵育在吉达营养溶液中[59,分别在5、10、15、30和60 min取样。暗处理时,将幼苗置于无光培养箱中,分别于3、6、9和12天取样。对照(CK)植株均在正常培养箱中生长。
实时荧光定量PCR分析
通过在正常条件和不同治疗的水稻幼苗的叶片叶片中用Trizol试剂萃取总RNA。对于植物来说,生长的零件(如根和叶)通常可以产生大量的养肝剂以维持植物生长的需要。基因OsSAUR3和奥梅尔10.被选为对光有反应的受试者。因此,从叶片中提取RNA进行qRT-PCR。cDNA是用PrimeScript RT试剂盒和gDNA擦除器(日本京都Takara)合成的。在CFX96上使用TransStart Tip Green qPCR SuperMix(TransGen Biotech,中国北京)进行PCR扩增™ 实时PCR检测系统(Bio-Rad,Hercules,CA,USA)。实验中使用的基因特异性引物见补充表4..对每种反应进行三种生物重复。使用2的QRT-PCR数据计算相对基因表达水平−△△CT方法 [60].
基于RNA-seq和数字基因表达数据的表达分析
表达数据来源于水稻基因组注释项目(http://rice.plantbiology.msu.edu/expression.shtml.)及大豆数据库(https://www.soybase.org/soyseq/)[61].这些数据在不同的植物组织内进行基因标准化,并根据皮尔逊相关系数采用加权配对法与Genesis (v1.7.6)软件进行层次聚类[62].使用白色和黑色在成因中可视化数字基因表达数据,以表示“未抑制”和“表达”的基因。
蛋白质三级结构预测与优化
利用瑞士模型对SAUR蛋白的三级结构进行了预测(https://swissmodel.expasy.org/)[29].AtSAUR8和OsSAUR8与模板序列的同源性分别为23.40和21.15%。采用gromacs2019.1软件包(http://manual.gromacs.org/2019.1/index.html.用魅力27强制场和提示3P水模型在足够大的盒子中包围整个蛋白质,然后通过50,000步的最陡血液进行能量最小化。最小化的结构与NVT和NPT模拟平衡。最后,我们进行了1 ns长的NVT MD模拟和结构参数,如RMSD在800 PS后融合。辅助示出了对结构优化的RMSD和Ramachandran图谱的分析。8..用NOC 3.01进行蛋白质结构的可视化(http://noch.sourceforge.net/).
FABACEAE植物与植物植物的基因同步分析拟南芥蒂利亚纳
基因组组装序列及基因注释拟南芥和截形苜蓿已从plant数据库(http://plants.ensembl.org/index.html).利用MCScanX构建基因共通性分析[63] TBTOOLS的程序[64].同时关系Saur.基因用蓝线标出。
可用性数据和材料
支持本文结论的数据集包含在文章及其补充信息文件中。用于FastTree和IQ-TREE树的多序列比对数据可在TreeBASE Web (http://purl.org/phy/treebase/phylows/study/tb2:26122).
缩写
- Saur.:
-
小型植物
- GH3:
-
格雷琴黑根3
- AUX / IAA.:
-
生长素/吲哚乙酸
- QRT-PCR:
-
定量实时聚合酶链反应
- DGE:
-
数字基因表达
- 毫升:
-
最大似然
- PP2C:
-
2C型蛋白磷酸酶
- EIN:
-
乙烯不敏感
- ETR:
-
乙烯受体
- 萨克:
-
Senescence-associated受体激酶
- SSPP:
-
衰老抑制蛋白磷酸酶
- 衣冠楚楚的:
-
几天后授粉
- DAF):
-
几天后开花
- CK:
-
细胞分裂素
- QTL:
-
数量性状位点
- 下午H+atp酶:
-
血浆膜H.+atp酶
- 阿坝:
-
脱落酸
- BR:
-
油菜素内酯
- GA公司:
-
赤霉素
- 是:
-
茉莉酸
- AZF:
-
拟南芥锌指蛋白
- ARF:
-
生长素响应因子
- BZR:
-
铜唑唑抗性
- 个人所得税:
-
Phytochrome互动因子
- 史:
-
拍摄
- fs:
-
幸运阶段幼苗
- TL:
-
二十年叶
- PoI:
-
出苗后花序
- 脉波重复间隔:
-
出苗前花序
- 答:
-
花药
- 病人:
-
雌蕊
- FDS:
-
五DAP的种子
- TDEM:
-
25个衣冠楚楚的胚胎
- TDS公司:
-
十DAP的种子
- TDEN:
-
25 DAP胚乳
- WGD:
-
全基因组复制
参考文献
- 1.
任洪林,刘国强。SAUR蛋白在植物生长中作为激素和环境信号的效应因子。摩尔。2015;8(8):1153 - 64。
- 2.
生长素响应基因的表达:基因、启动子和调控因子。植物生物学杂志。2002;49(3-4):353 - 356。
- 3.
小生长素上调RNA (Saur.)玉米的基因家族:鉴定,进化及其系统发育比较拟南芥、水稻和高粱。中国生物医学工程学报(英文版),2014;
- 4.
Franco AR, Gee MA, Guilfoyle TJ。用生长素和蛋白质合成抑制剂诱导和超诱导生长素响应mrna。中国生物医学工程杂志。1990;265(26):15845-9。
- 5.
关键词:水稻,全基因组,全基因组分析Saur.Solanaceae物种的基因家族。基因。2012; 509(1):38-50。
- 6.
Stortenbeker N, Bemer M. TheSaur.基因家族:植物适应生长发育的工具箱。实验机器人。2019;70(1):17-27。
- 7.
沙利文ml,绿色pj。烟草细胞和转基因植物中DST元素的突变分析:鉴定mRNA不稳定性的残留物。RNA。1996; 2(4):308-15。
- 8.
Newman TC、Ohme Takagi M、Taylor CB、Green PJ。DST序列在植物中高度保守Saur.基因,针对烟草中快速腐烂的目标报告转录物。植物细胞。1993年; 5(6):701-14。
- 9.
一种生长素诱导的大豆聚腺苷化rna的鉴定。植物生物学杂志。1987;9(6):611-23。
- 10.
Jain M, Tyagi AK, Khurana JP。全基因组分析,进化扩展,和早期生长素反应的表达Saur.水稻基因家族(栽培稻). 基因组学。2006;88(3):360–71.
- 11.
李X,刘克,耿y,吴m,pei w,zhai h,zang x,li x,张俊,yu s等。对小型植物瘤RNA的基因组分析(Saur.)棉花的基因家庭。BMC基因组学。2017; 18(1):815。
- 12.
谢尔、董丙、马Y、邓L、何S、易S、吕Q、郑Y。综合分析Saur.柑橘基因家族及其与脱落区的水果滴的转录相关性。Funct综合基因组学。2015; 15(6):729-40。
- 13.
白Q,Hou D,Li L,Cheng Z,Ge W,Liu J,Li X,Mu S,GaO J.基因组 - 宽分析和小型胃辘RNA的表达特征(Saur.)莫斯竹子的基因(植被类型).基因组。2017;60(4):325 - 36。
- 14.
黄X、鲍妍、王B、刘立杰、陈杰、戴立杰、苏宝乐、彭德祥。小生长素uprna的鉴定(Saur.招标植物中的基因:桑树(Morus notabilis.),大麻(大麻)及苎麻(Boehmeria Nivea.).J麝猫。2016;95(1):119 - 29。
- 15.
张n,黄x,宝y,王b,zeng h,cheng w,tang m,li y,ren j,sun y。基因组覆盖Saur.西瓜中的基因(Citrullus lanatus). 生理分子生物学植物。2017;23(3):619–28.
- 16.
Chae K, Isaacs CG, Reeves PH, Maloney GS, Muday GK, Nagpal P, Reed JW。拟南芥SMALL AUXIN UP RNA63促进下胚轴和雄蕊丝的伸长。植物j . 2012; 71(4): 684 - 97。
- 17.
STAMP P,Kumar PP。蟾蜍素和胃肠杆菌素响应拟南芥小型植物素UP rna36调节光线伸长率。植物细胞批准。2013; 32(6):759-69。
- 18.
侯凯,吴伟,甘思亮。索尔36,一个生长素向上的小RNA基因,参与促进叶片衰老拟南芥.植物理性。2013; 161(2):1002-9。
- 19.
文忠,梅燕,周杰,崔燕,王丹,王乃宁。SAUR49通过抑制SSPP正向调控叶片衰老拟南芥.植物细胞生理。2020; 61(3):644-58。
- 20.
他SL,谢洪林,Jauh GY。SMALL AUXIN UP RNA62/75是花粉管生长必需转录本翻译所必需的。植物杂志。2018;178(2):626 - 40。
- 21.
阿米亚人HS,Creux NM,Brown Ea,Garner Ag,Blackman BK,Harmer SL。昼夜调节向日葵升降机,花卉取向和粉碎机访问。科学。2016; 353(6299):587-90。
- 22.
植物生长素对睡莲花昼夜节律的调控。植物生物学学报。2018;18(1):143。
- 23.
陈张L F,张X, Z,赵Y, Lohaus R, Chang X,董W Ho SYW,刘X, et al。睡莲基因组和开花植物的早期进化。自然。2020;577(7788):79 - 84。
- 24.
邱t,齐米,丁x,郑y,周t,陈y,汉n,zhu m,bian h,王j。的索尔41小生长素UP RNA基因亚家族是脱落酸诱导的调控细胞增殖和耐盐性的基因拟南芥蒂利亚纳幼苗。Ann Bot。2020; 125(5):805-19。https://doi.org/10.1093/aob/mcz160.
- 25.
王Z,杨立,刘Z,鲁米,王米,孙q,兰y,shi t,wu d,hua J. Saur26 / 27/28蛋白确定的生长热反应性的自然变化拟南芥蒂利亚纳.新植醇。2019;224(1):291 - 305。
- 26
rensing sa,lang d,zimmer广告,terry a,萨拉玛娃,夏皮罗h,nishiyama t,perroud p-f,lindquist ea,kamisugi y等。Physcomitrella Genome揭示了植物征服土地的进化见解。科学。2008; 319(5859):64-9。
- 27
周凯奇,沈海斌。plant - mploc:一种自上而下的策略,用于增强预测植物蛋白亚细胞定位的能力。《公共科学图书馆•综合》。2010;5 (6):e11335。
- 28
van Mourik H, van Dijk ADJ, Stortenbeker N, Angenent GC, Bemer m拟南芥阿富汗二月基因:关注saur10分支。中国生物医学工程学报。2017;17(1):245。
- 29
Waterhouse A, Bertoni M, Bienert S, Studer G, Tauriello G, Gumienny R, Heer FT, de Beer TAP, remfer C, Bordoli L,等。瑞士模型:蛋白质结构和复合物的同源建模。核酸图谱(W1): W296-303。
- 30。
Abraham MJ, van der Spoel D, Lindahl E, Hess B,以及GROMACS开发团队。GROMACS用户手册版本2019.1。
- 31。
Spartz AK, Ren H, Park MY, Grandt KN, Lee SH, Murphy AS, Sussman MR, Overvoorde PJ, Gray WM。对PP2C-D磷酸酶的SAUR抑制激活质膜H+- atp酶促进细胞扩张拟南芥. 植物细胞。2014;26(5):2129–42.
- 32。
李子,陈鹤,李QT,陶吉,卞Xh,马b,张wk,陈鸣,张js。三个Saur蛋白Saur76,Saur77和Saur78促进植物生长拟南芥.Sci众议员2015;5:12477。
- 33.
哦,朱伊,白米,阿伦哈特,孙伊,王志。细胞的伸长是通过细胞内相互作用的转录因子的中央电路来调节的拟南芥下杆菌。Elife。2014; 3:47。
- 34.
Wiesel L, Davis JL, Milne L, Redondo Fernandez V, Herold MB, Middlefell Williams J, Morris J, Hedley PE, Harrower B, Newton AC等。马铃薯防御激素反应的转录参考图。Sci众议员2015;5:15229。
- 35.
不同的植物激素通过很大程度上不重叠的转录反应调节相似的过程。细胞。2006;126(3):467 - 75。
- 36.
欧阳福、毛建福、王杰、张S、李Y。转录组分析显示红光和蓝光调节挪威云杉的生长和激素代谢[挪威云杉(l)岩溶]。《公共科学图书馆•综合》。2015;10 (8):e0127896。
- 37.
Sun n,Wang J,Gao Z,Dong J,He H,Terzaghi W,Wei N,Deng XW,陈H.拟南芥Saurs对各种器官发展的差分光线调节至关重要。Proc Natl Acad Sci U S A. 2016; 113(21):6071-6。
- 38.
哦,朱耶,王泽。BZR1和PIF4之间的相互作用集成了芸苔类固醇和环境反应。NAT细胞BIOL。2012; 14(8):802-9。
- 39.
郝伊,欧娥,崔庚,梁兹,王志。HLH和bHLH因子之间的相互作用调节光调节植物的发育。摩尔植物。2012;5(3):688–97.
- 40。
Franklin KA, Lee SH, Patel D, Kumar SV, Spartz AK, Gu C, Ye S, Yu P, Breen G, Cohen JD,等。光敏色素相互作用因子4 (PIF4)在高温下调节生长素的合成。国家自然科学基金资助项目:国家自然科学基金资助项目。
- 41。
拟南芥Cys2/His2锌指蛋白AZF1和AZF2在非生物胁迫条件下对脱落酸抑制和生长素诱导基因具有负调控作用。植物杂志。2011;157(2):742 - 56。
- 42。
研究表明,钙调素相关蛋白与靶蛋白的差异结合通过高密度显示拟南芥蛋白质微阵列。Proc Natl Acad Sci U S A. 2007; 104(11):4730-5。
- 43.
康德S,Bi YM,Zhu T,Rothstein SJ。索尔39是一种生长素上升的小RNA基因,在水稻中起着生长素合成和运输的负调控作用。植物杂志。2009;151(2):691 - 701。
- 44.
白福,李思,杨c,赵t,张t,兰x,陈m,廖z。过度表达AbSAUR1基因提高小麦生物产量和生物碱产量颠茄.Ind作物项目2019;140。
- 45.
一千个植物转录组I.一千个植物转录组和绿色植物的系统基因组学。自然。2019;574(7780):679 - 85。
- 46.
王帅,李玲,李辉,萨胡SK,王辉,徐勇,先伟,宋斌,梁辉,程森,等。早期分化的链藻基因组揭示了植物的陆化。Nat植物。2020;6(2):95 - 106。
- 47.
程晟,先伟,付勇,Marin B, Keller J,吴涛,孙伟,李旭,徐勇,张勇,等。陆生接合线虫的基因组为研究陆生植物的进化提供了新思路。细胞。2019;179 (5):1057 - 67 e1014。
- 48.
王杰,孙平,李勇,刘勇,俞杰,马×,孙S,杨恩,夏R,雷T,等.豆科植物10个基因组的层次比对建立了一个家系级基因组学平台。植物生理学。2017;174(1):284–300.
- 49.
潘Y:基于机器学习方法的蛋白质结构预测及其理解.IEEE第7届生物信息学和生物工程国际研讨会2007:7-7。
- 50。
Shin JH, Mila I, Liu M, Rodrigues MA, Vernoux T, Pirrello J, Bouzayen M.索尔69通过增强对乙烯的敏感性,参与番茄果实的未成熟相转变。新植物。2019; 222(2):820-36。
- 51。
杜米,斯派克ep,灰色wm。快速的植物蛋白介导的细胞膨胀。Annu Rev植物Biol。2020; 71(1):379-402。
- 52。
Larkin M, Gordon B, Nigel C, Ramu M, Paul M, Hamish V, Franck W, Iain W, Andreas L, Rodrigo T, Julie G, Toby H, Desmond。Clustal W和Clustal X版本2.0。生物信息学。2007;23:2947-48。
- 53。
Waterhouse AM、Procter JB、Martin DM、Clamp M、Barton GJ。Jalview版本2——一个多序列比对编辑器和分析工作台。生物信息学。2009;25(9):1189–91.
- 54。
Wilkins Mr,Gasteiger E,Bairoch A,Sanchez JC,Williams KL,Appel Rd,Hochstrasser DF。扩展服务器中的蛋白质识别和分析工具。方法Mol Biol。1999; 112:531-52。
- 55。
价格Mn,Dehal PS,Arkin AP。FastTree:使用配置文件而不是距离矩阵计算大的最小进化树。mol Biol Evol。2009; 26(7):1641-50。
- 56。
阮L-T,施米特·哈塞勒,冯·哈塞勒,明。IQ-TREE:一种快速有效的估计最大似然系统发育的随机算法。中国生物医学工程学报。2014;32(1):268-74。
- 57.
Madhusoodanan N, Basutkar P, Tivey ARN, Potter SC, Finn RD等。2019年的EMBL-EBI搜索和序列分析工具api。核酸图谱2019;47(W1): W636-41。
- 58.
Jin Y,Pan W,Zheng X,Cheng X,Liu M,Ma H,Ge X.OSERF101.ERF家族转录因子,调节生殖组织的干旱胁迫反应。中国生物医学工程学报,2018;
- 59.
Yoshida S FD,Cock JH,Gomez KA:水稻生理研究实验室手册。国际水稻研究所,马尼拉,菲律宾1976:61–66。
- 60。
Livak KJ, Schmittgen TD。使用实时定量PCR和2(−Delta Delta C(T))方法。方法。2001;25(4):402 - 8。
- 61。
Severin AJ, Woody JL, Bolon YT, Joseph B, Diers BW, Farmer AD, Muehlbauer GJ, Nelson RT, Grant D, Specht JE等。RNA-Seq阿特拉斯的大豆:大豆转录组的指南。BMC植物BIOL。2010; 10:160。
- 62。
turn A, Quackenbush J, Trajanoski Z.起源:微阵列数据聚类分析。生物信息学(牛津大学)。2002; 18(1): 207 - 8。
- 63。
王艳萍,唐海斌,DeBarry JD,谭鑫,李建平,王兴耀,李天喜,金洪志,马勒B,郭浩,等。MCScanX:一个用于基因共线性和共线性检测和进化分析的工具包。中国生物医学工程学报(英文版);2012;
- 64。
关键词:生物信息学,大数据,交互分析,TBtools摩尔。2020;13(8):1194 - 202。
致谢
作者感谢王亚琼(复旦大学儿童医院)对本稿件的建议。我们还要感谢来自中国益智集团李文卞吉的罗伯特·麦肯齐博士(www.liwenbianji.cn/ac),以编辑该手稿草稿的英文文本。
资金
本研究得到国家自然科学基金青年基金(No. 31700208 ~ YJ)的资助。资助机构没有参与这项研究的设计、数据的收集、分析和解释以及手稿的撰写。
作者信息
从属关系
贡献
YJ和HZ构思并设计了实验。HZ进行系统发育分析,种植植株并进行qRT-PCR。ZY和XY总结Saur.功能。JC, XC和HZ在培养室采集叶片样品。HZ和YJ写了手稿。YL和FM对手稿的讨论做出了贡献。YJ修改了原稿。所有作者阅读并批准最终稿件。
通讯作者
道德声明
伦理批准并同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
作者声明他们在本文中没有竞争利益。
附加信息
出版商的注意事项
斯普林格自然保持中立,就管辖权的要求,在出版的地图和机构的联系。
补充信息
附加文件1:补充数据表S1
.FastTree V2.1的小型胃辘RNAS构建的最大似然性系统发育树(畜群)含有十三个植物物种的详细名称。
附加文件2:补充数据表S2
.利用IQ-TREE v2.0.6构建生长素小rna的最大似然进化树(畜群)含有十三个植物物种的详细名称。
补充文件3:补充表1
.鉴定的Saur蛋白序列来自Anthoceros Angustus.,Marchantia polymorpha,Physcomitrella金属盘,江南卷柏,银杏叶,Gnetum Montanum.,Amborella trichopoda.,拟南芥蒂利亚纳,栽培稻,玉米,Setaria Italica,Mimosa Pudica,cercis canadensis,甘氨酸最大, 和截形苜蓿.
附加文件4:补充表2
. 研究了13种植物小生长素RNA(SAUR)的长度、等电点和亚细胞定位。
附加文件5:补充表3
.所有功能标识Saur.基因。
补充文件6:补充表4
.本研究使用的基因特异性引物。
附加文件7:补充图1
.由IQ树构建的最大似然性系统发育树Saur.来自13种植物的基因家族。
附加文件8:补充图2
.最大似然系统发生树的FastTreeSaur.基因家族在拟南芥蒂利亚纳,含羞草,加拿大紫菀,断枝苜蓿和大豆.
附加文件9:补充图3
.FABACEAE植物与植物植物的基因同步分析拟南芥蒂利亚纳.
附加文件10:补充图4
.来自含7种植物种类的Saur蛋白的五个保守的主题拟南芥蒂利亚纳,栽培稻,玉米,大豆,截形苜蓿,Setaria Italica.,Physcomitrella金属盘.
附加文件11:补充图5
.生长素小RNA的表达谱(Saur.)大豆的基因(大豆).
附加文件12:补充图6
.数字基因表达OsSAURs不同于压力条件下水稻品种的不同发展阶段。
附加文件13:补充图7
.识别的数量Saur.单子叶植物和双子叶植物基因组的基因注释。
附加文件14:补充图8
. 结构参数RMSD和Ramachandran图的结构优化分析。
权利和权限
开放获取本文是根据知识共享署名4.0国际许可证授权的,该许可证允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供到知识共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可证中,除非在材料的信用额度中另有说明。如果材料未包含在文章的知识共享许可证中,并且您的预期用途不受法律法规的允许或超出允许的用途,您将需要直接获得版权持有人的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
张宏,于卓,姚晓明。等等。全基因组鉴定和表征小型胃引入RNA(Saur.)植物基因家族:正常生长和胁迫响应过程中的进化和表达谱。BMC植物杂志21日,4 (2021). https://doi.org/10.1186/s12870-020-02781-x
已收到:
认可的:
发表:
关键词
- Saur.
- 养阴
- 系统发育树
- 表达模式