抽象的
背景
苔属植物莎草属是莎草科植物中最大的属之一,是生态系统中重要的维管植物。然而,遗传背景苔属植物很复杂,分类尚不清楚。为了调查基因函数注释苔属植物,进行RNA测序分析。基于Illumina数据生成简单的序列重复(SSR),然后利用来研究79的遗传特征苔属植物种质。
结果
在本研究中,有36403个单基因,全长41724615 基于GO、KOG、KEGG、NR数据库获取bp并进行注释。研究结果为基因功能研究提供了理论依据。在8776个ssr中,随机选择了96对引物。利用42对多态性较高的引物,共扩增出180条多态性带,多态率为100%。平均条带数为4.3条,平均距离为0.548,多态信息含量为0.133~0.494。观察等位基因数(Na)、有效等位基因数(Ne)、Nei's(1973)基因多样性(H)和Shannon信息指数(I)分别为2.000、1.376、0.243和0.391。NJ聚类分为3个类群,新西兰材料表现出相似的遗传属性,聚类为一个类群。UPGMA和PCoA分析也显示了相同的结果。分子方差分析(AMOVA)表明,在地理起源聚类和NJ聚类的基础上,材料内部的遗传多样性优于材料之间的遗传多样性。更重要的是,79人的指纹苔属植物在本研究中建立了物种。引物对的不同组合可用于识别多个苔属植物同时,克服了传统识别方法的困难。
结论
转录组学分析揭示了注释基因的功能类别,为未来的基因功能研究提供了新的思路。遗传特征分析表明,79个品种间存在广泛的基因流动苔属植物物种。这些标记可以用来研究人类的进化史苔属植物以及相关物种,以及作为未来育种项目的指导。
背景
属苔属植物L属于莎草科,是一个巨大的属。它是环境中维管植物最重要的属之一[1],超过2000种物种普遍存在世界上[2[中国近500种3.].苔属植物物种广泛用作家庭草坪的地面封面以及由于冷水和耐水性的世界许多地区的坡度稳定[4.]践踏抗性和高装饰价值[5.那6.].
以前的研究侧重于地理分布,邮局和起源区苔属植物. 贝尼特斯贝尼特斯等人[7.]发现两者之间的明显遗传分化苔属植物西部地中海的姐妹种类,并指出地理障碍在限制基因流动中发挥了显着作用。míguez等。[8.]揭示了后期的迁徙 - 全茂 - 全茂的地中海形状的局面的局部摄影苔属植物教派。Rhynchocystis.Martín-Bravo等。[9.]证明了这一点苔属植物起源于东亚的后期eocene,其具有富有成效的多样性苔属植物. 以前的研究阐明了系统发育的结构苔属植物进入至少四个主要的林,包括siderostictae.分支,核心苔属植物那藜, 和加西激枝晶[10.].但是,组合ETS的超级涂覆分析,其和垫K DNA区域表明,对形态特征的过度依赖不适合自然群体的划界[11.].
分子标记是植物遗传多样性分析的有力工具,是加快植物育种进程的基础。目前常用的分子标记主要有ISSR、RAPD、RFLP、AFLP、SSR、SRAP等[12.]. 简单重复序列(simple repeat sequence,SSRs)具有丰富、多等位、高度多态性和共显性等优点,是揭示植物遗传基因作用机制的一个很好的选择[13.那14.]. RNA测序是获得非测序基因组和非模式生物可转移率较高的ssr的有效工具[15.那16.].已经证明,从一个物种获得的SSR可用于检测相关物种中的多样性,甚至在同一家族的其他属中[17.那18.].SSR已广泛应用于遗传作图、亲缘关系研究[19.]、品种鉴定[20.那21.]植物遗传多样性分析[22.].迄今为止,SSR标记已广泛应用于植物研究,如Zea Mays.[23.],西瓜[24],Triticum aestivum.和Cerasus属物种(25].
在先前的研究中,有许多分子标记研究苔属植物,其中还包括SSR法。M 'Baya等人[26]利用了14个分离的SSRsCarex Kobomugi.测试遗传结构Carex Hebes.和短叶苔草而且遗传变异程度很高。同时,其他有用的分子标记也被用于研究之间的差异苔属植物物种。Liu等人[3.]报告30个SSR标记C. Moorcroftii.,它提供了可用的工具来探索遗传结构和系统发育演化。Starr等人。[27]我发现了垫K条形码可以区分47%的苔属植物并明确区分了NJ进化树的系统发育多样性关系。Ning等人[28事实证明,ISSR分子标记是研究遗传多样性的强大工具苔属植物. 他们都发现了一个结果苔属植物物种是复杂的。Nagasawa等人[29]发现苔属植物群体可能导致低水平的多态性在群体中。Man等人[30]比较了15个品种的遗传变异和群体结构短尾线虫并表明基因流动是广泛的。虽然在研究方面已经取得了一定的进展苔属植物基于Illumina RNA测序的SSR分子标记遗传学研究,遗传学遗传学研究。在以前的研究中使用的材料数量较少。而且,与作物和模型植物的研究相比,分子研究苔属植物仍然缺乏。研究两者之间的关系和差异具有重要的经济价值苔属植物目前的物种。
在我们之前的研究中,我们使用单分子长阅读测序方法来研究基因的转录调控网络短叶苔草响应遮阳宽度[31].在本研究中,我们进一步根据先前的illumina测序数据集探索了SSRS。本研究的目的是:(1)丰富苔属植物转录组信息并更好地了解来自注释基因的功能类别,(2)以制定SSR标记并验证它们的多态性水平,(3)来调查之间的遗传背景苔属植物种质。
结果
Illumina测序和转录组的De Novo组装
转录组短叶苔草使用Hiseq进行测序TM值2000平台。总共获得43.67Gb的clean数据。每个样品的清洁数据达到6.32Gb, Q30基率在94.03%以上。共组装了36403个unigenes,其中12657个unigenes长度大于1kb。该unigene的N50为2016年,表明其具有较高的组装完整性。
基于不同数据库的基因注释
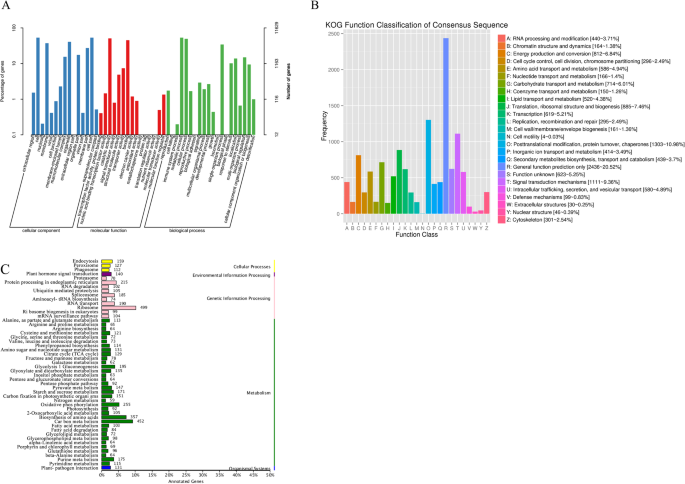
基于对序列的同源性分析,将11,629个未进剂(31.95%)分为三个主要的GO类别和50个子类别。GO分类包括“蜂窝过程”,“代谢过程”和“单体生物过程”。(图。1一种)。“细胞”是细胞组分组最大的亚组。下一个最大的组是“细胞部分”,后跟“细胞器”,“核心”和“大分子复合体”。十种不同分子函数类别中的类别“催化活动”和“绑定”苔属植物unigenes也很丰富。根据Kog数据库,11,871个unigenes分为25个官能团,20.52%的unigenes被注释为“一般职能”集群。“翻译后修改”,“蛋白质营业额”和“伴侣”(1303个未成年人,10.98%)是下一个最大的群体,其次是“信号转导机制”(1111人未进,9.36%)。或者,“核结构”(46人未进,0.39%),'细胞外结构'(30个未成熟,0.25%)。“细胞运动性”(4个未进剂,0.03%)组中的unigenes显着小于上述三组(图。1b)。基于Kegg数据库,发现总共8440个未成熟,包括属于五个大群体的40个生物途径(细胞过程,遗传信息处理,环境信息处理,新陈代谢和有机体系统)。包括核糖体(499,5.91%),碳水化合物代谢(452,5.36%)和氨基酸的生物合成(357,4.23%)在这50个途径中(图495,4.23%)(图。1C)。
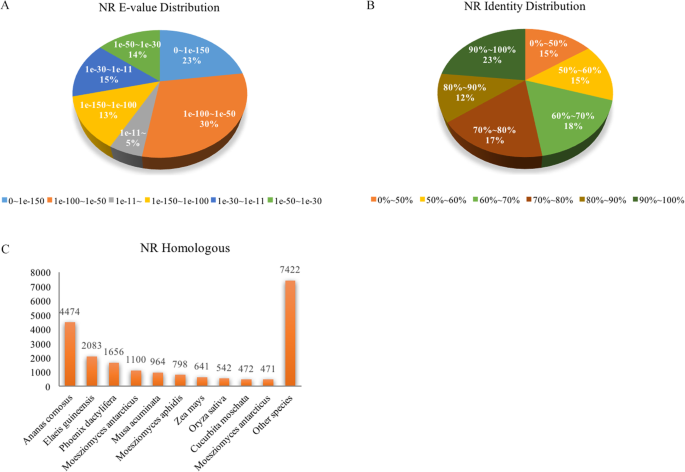
基于NCBI非冗余核苷酸数据库,E值分布显示23.00%的单基因产生显著的命中率(图。2a) 约35.00%的单基因表现出大于80%的同一性(图。2b) 是的。NR蛋白序列比对结果显示,21.69%的蛋白能与dna序列进行比对奥纳斯昏迷,10.10%可以与之一致Elaeis Guineensis.,8.03%可以与之对齐凤尾鱼(图。2C)。
转录组中SSRS的频率和分布
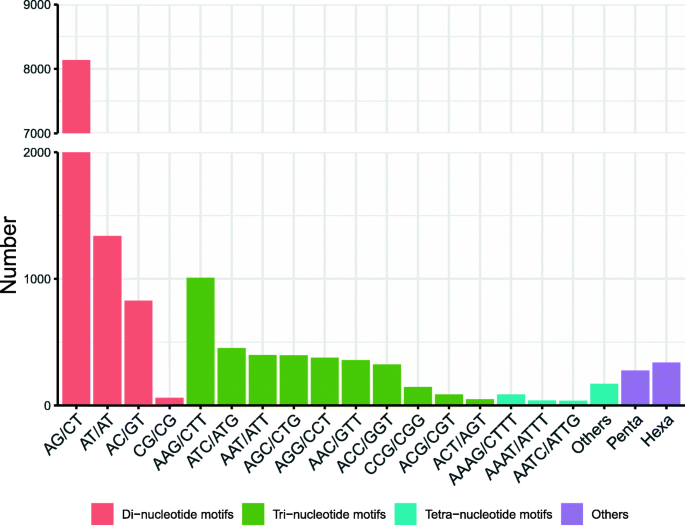
使用MISA软件扫描共扫描36,403个unigenes,并检测到8776个SSR基因座(表1).转录组中的SSR基因座具有六种类型,并且每个重复类型的数量变化很大。单一重复基序占64.93%的排名最丰富的类型,而六核苷酸占1.11%的态度是最小的类型。最丰富的二核苷酸重复是AC / GT(8138; 18.02%),然后在(1339; 3.10%),AC / GT(827; 1.83%)。最多的三核苷酸重复是AAG / CTT(1008; 2.23%),然后是ATC / ATG(454; 1.01%)。同时,最富裕的四重复基序类型是AAAG / CTTT(87; 0.21%)。六曲和五核苷酸基序的数量分别为399(0.88%)和377(0.83%)(图。3.).
新型SSRS的开发和转移性评估
我们设计和合成了96对以扩增11个表型差异苔属植物材料(表格S1).其中,42(43.75%)对可以扩增几个条带并具有高多态性。引物在不同之间表明良好的可转移性苔属植物物种。未扩增条带数占15.6%,其余条带呈低多态性或无多态性。
遗传多样性统计数据
在42个ssr位点中,我们在79份材料中识别了180个标记等位基因。42个SSRs中,PIC值为0.133 ~ 0.494,平均为0.259。ssr在材料间表现出广泛的遗传变异。两者的遗传多样性苔属植物对材料进行聚类分析、主成分分析。多态性率为100%,平均每引物扩增4.3条引物。观察等位基因数(Na)、有效等位基因数(Ne)、Nei’s(1973)基因多样性指数(H)和Shannon信息指数(I)分别为2.000、1.376、0.243和0.391苔属植物加入很高。我们还计算了所附的遗传距离(表S2),范围为0.222 ~ 1.000。遗传距离平均值为0.548。
集群分析苔属植物基于SSR标记
为了揭示分类信息苔属植物物种,我们根据我们的原始数据获得等位基因频率。我们使用NJ,PCOA和Coaronononomy等先验分类,而不是使用诸如出处或分类学,而是将结果组合起来探讨所有遗传信息和分类。
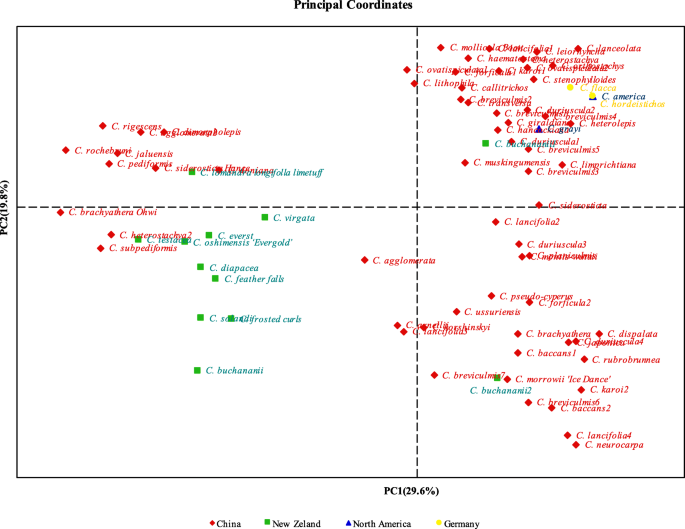
主坐标(PCOA)结果表明,轴2分离并产生两种遗传分化的组苔属植物加入(图。4.).第一主成分占29.6%,第二主成分占19.8%。
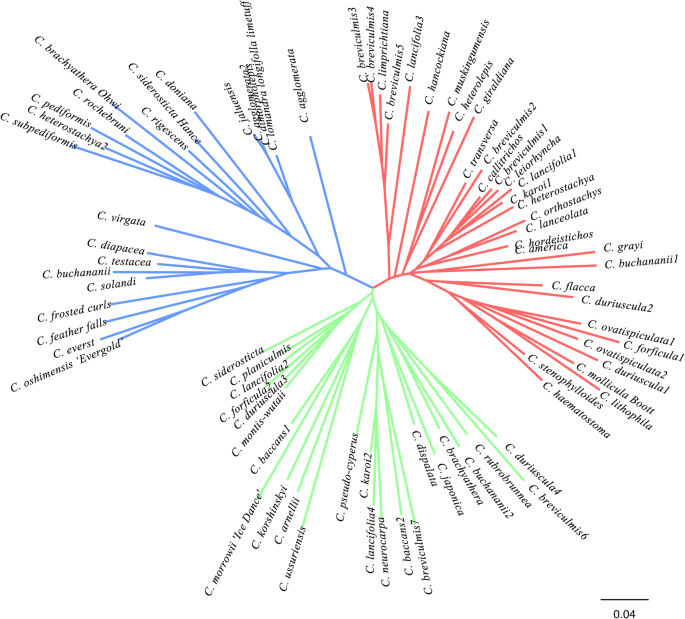
基于Shered等位基因的距离计算方法,Neibor连接系统发育分析将79个基因分为两组苔属植物材料分成三组(图。5.).一组共有22种材料和C. Jaluensis.那C. Dimorpholepis.那C. Agglomerata2.分为非常相似的类别。此外,来自新西兰的商业植物材料被归为第一类,包括C. Virgata.那C.磨砂卷发那C刺鲅那C. Oshimensis'Evergold'那C.羽毛瀑布那C. Buchananii.那C.雷维斯特那C. deapacea那c . testacea那C. Lomandra Longifolla跛行.第二组包括24种材料,C. empediformis.和C. Jaluensis.是最相似的品种。第三组包括33种材料,其中大部分来自中国各地,还有4种来自德国和北美。值得一提的是C短尾蛇从中国各省收集的都归为这一类。
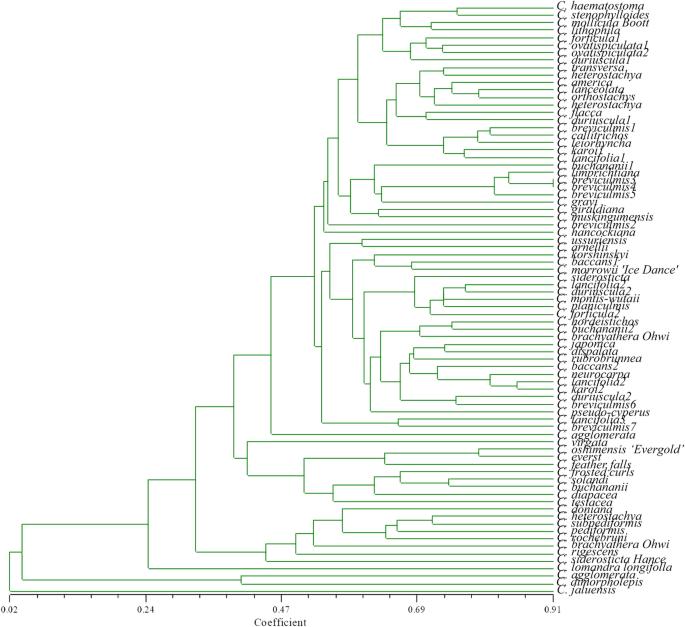
基于SSR原始数据,使用骰子系数方法来计算相似性。使用UPGMA方法聚类总共79种材料(图。6.). 基因型间的相似性为0.070 ~ 0.786. 通过UPGMA聚类,将79份种质资源分为两大类群。C. Jaluensis.被分配到一个单独的组。剩下的78个域名苔属植物材料被分为两个亚组c . agglomerata那C. Dimorpholepis.和C. Lomandra Longifolla跛行被分类为一组。当遗传距离为0.42时,新西兰的九个加入集聚到一个组中,但C. Lomandra Longifolla跛行不属于这一组。虽然与来自新西兰的材料没有聚类,但遗传相似性较高。关于收集来源苔属植物,有一定程度的基因交换苔属植物来自中国和新西兰,德国和北美的种质。但是,资源不能完全按地区划分。Sorensen骰子相关系数是R.= 0.941,表明聚类结果可靠。
AMOVA分析苔属植物基于不同方法的种质资源
为了评估这些种系之间的遗传差异,我们计算了f英石研究的两个级别对(起源和遗传分类)的所有载体之间的值。通过对来源的不同分析,Amova表示,群体内有79种遗传变异的88%,群体中有12%。Amova分析的结果表明,一些亚种在国家内有大的变化,这与来自不同国家的豁免的结果一致。虽然基于NJ集群分析的Amova分析结果显示,人群中的89%(表2). F英石值和概率P (rand≥data)分别为0.117和0.052。
指纹
结果表明,42对SSR标记能有效区分79份材料,并建立了一个一次能区分更多品种的指纹图谱(表1)S3).
讨论
我们之前的工作报道了基因的转录调控网络C短尾蛇使用单分子长读测序来应对阴影胁迫[31].在本研究中,我们利用Illumina RNA-SEQ数据来产生SSR标记,从而探讨其他79的遗传多样性苔属植物登记入册。我们释放了大量表达基因序列(36,403个Unigene序列)。为了更好地了解功能类别苔属植物基因,我们搜索了Go,Kog,Kegg Pathway映射数据库。总共获得了20,948个unigenes注释结果。结果表明,57.54%的基因被成功注释,反映了高转录组多样性苔属植物.基因本体是基因功能的全局分类系统,将基因的特征分类为“生物过程”,“细胞成分”和“分子函数”[32].Kog分析致力于对真核完整基因组进行分类的基本群。Kegg分析是分析基因代谢产品及其推定功能的最常用方法之一[33].与Li等人的结果一样。[34]GO数据库将注释信息分为三类:生物过程、细胞成分和分子功能。最大的子类别是“结合”,其次是“催化活性”。我们还发现,KEGG途径的基因群最大,属于代谢类,而遗传信息处理类是第二大类。基于NR比对结果,有21.69%的序列能与原始序列进行比对奥纳斯昏迷.它被认为是C短尾蛇和Bromeliaceae家族成员与蛋白质对准具有最接近的关系。奥纳斯昏迷凤梨科C短尾蛇属于Cyperaceae。考虑到植物的分类,A. Comsous.和C短尾蛇似乎是基因上的遥远物种。意外蛋白质对齐的原因可能是当前NR数据库中的缺乏有关的水芹酵母相关物种数据。结果表明,更新该属的遗传数据库是必要和迫切的。
在单基因文库的基础上进行了基因结构分析,共获得8776个SSR标记。在检测到的位点中,5699个单核苷酸重复序列占SSR位点总数的64.93%。其次是三核苷酸和二核苷酸(分别为1873和582,分别占21.37%和6.63%),这与萝卜中3个核苷酸重复序列最丰富的结果不同,频率为52%[35]. 说明植物中SSR位点的特异性不同。在303对功能性引物的重复序列中,AC/GT(18.02%)是最丰富的二核苷酸重复序列短叶苔草.
虽然有很多研究苔属植物,更多的研究是某种类型或受限制的属性。然而,遗传背景和遗传关系尚不清楚,特别是之间苔属植物物种。它限制了苔属植物资源、杂交亲本的合理选择和基因工程育种。特别是在基因水平上的研究苔属植物还没有发现诸如遗传关系之类的进化问题。
在本研究中,我们发现42对SSR引物在79对引物中扩增出178个等位基因苔属植物每个微卫星平均有4.3个等位基因。与以往研究相比,多态率100%高于研究Csempervirens公司用过的RAPD标记[36].它起诉通过转录组测序开发的SSR标记的发展效率和多态性比RAPD标记更有效。我们还发现本研究中的PIC值低于Ning等人报告的0.83的值。[28]类似于Nagasawa等人获得的值。[29].PIC值用于测量其他植物中的群体多态性水平,它们取决于所测试的换档。根据其信息内容,轨迹多态性可分为高水平(PIC> 0.5),中级(0.5张照片> 0.25)和低水平(PIC <0.25)[37].在数量上苔属植物在宁的研究中使用的分子标记很小,并且所有苔属植物品种均来自山东省同一地区。其次,Ning等人使用的ISSR [28]可能比使用特定特定的SSR标记更有效。PIC值类似于Nagasawa等人。[29]研究结果表明,20个EST-SSR标记在家系中表现出低多态性C. angustisquama.人口和King等人[38]结果鉴定出11个微卫星位点大头苔草. 由于物种的种群动态,在已开发的标记上,获得了较低的遗传多态性,而不是空等位基因。苔属植物细菌被认为具有较低的PIC值,该研究似乎反映了这种低多样性和低遗传变异苔属植物这与物种历史有关,而不是所用标记的特征有关。
在聚类分析中,我们采用了NJ、UPGMA和PCoA相结合的分析方法。首先,由于生长环境相似,9份材料均为新西兰引种驯化的商品材料,因此新西兰种质资源在分类上是一致的。与Ning等人的研究相同[28[来自同一区域的物种的结果可以分为一个类别,与地理分布和环境条件相关。其次,由于类似的遗传背景和形态特征或其共同的地理来源,C. Grayi.和C. HORDEISTICHONJⅲ组具有明显的特征。也C短尾蛇和C兰蔻从中国被分为近缘种Ⅲ组,这与传统的分类方法是一致的。这两个的形态苔属植物物种非常相似,甚至很难区分,但可以通过分子标记的聚类方法来实现。
在培养过程中,在培养过程中与中国测试材料进行了基因交换C. Buchananii1.和C. Buchananii2.与中国材料有不同程度的混合。由于样本量的原因,德国和北美的材料不能被归为一个单一的类别,但我们发现材料的收集地点会对材料的分类产生一定的影响carex。此外,通过主成分分析,可以看出苔属植物植物分类不清楚。也许同一属植物之间仍存在较大的遗传差异,且没有明确的结果。因此,为了更好地解释这一现象,还需要更多的外国种质进行进一步的研究。
此外,基于这些基因型数据的PCOA清楚地表明存在明显的遗传分化苔属植物加入。PCoA和UPGMA的聚类结果部分一致,在所分析的材料间表现出显著差异。指纹图谱的结果对指纹识别有一定的参考价值苔属植物物种具有极其类似的外观,不容易区分。以前的研究表明,SSR标记可用于植物物种多样性分析和指纹发展[39]. 以12对新开发的SSR引物为例,建立了36个枣品种的指纹图谱[40],并进一步推荐8个SSR位点作为茶树指纹图谱的核心标记集[41]. 在这项研究中,我们得出结论,不同的引物组合可以用来区分不同的物种苔属植物这对选育杂交种、培育新品种具有重要意义。
结论
转录组学分析揭示了注释基因的功能类别,为未来的基因功能研究提供了新的思路。遗传背景分析表明,基因流量在79中的广泛广泛苔属植物物种。这些标记可以用来评估遗传多样性和研究人类的进化史苔属植物以及相关物种,以及作为未来育种项目的指导。
方法
植物材料和DNA提取
总共79个苔属植物中国64份,新西兰11份,北美2份,德国2份。64份中国种质来源于北京植物园、泰山、西安植物园、河北、上海和北京市农林科学院基地3.).我们已经获得许可,可以在当地组织的指导下购买或收集所有的植物材料。样品的正式鉴定是由我们组的两位植物分类学家Chen和Dr. Xun参照《Flora Reipublicae Popularis Sinicae》,在商业引种的基础上进行的。用CTAB法分别混合各培养物的10株单株,提取健康幼芽的基因组DNA [42].使用1%琼脂糖凝胶电泳来检查基因组DNA的质量和完整性。通过Nanodrop 2000分光光度计(Nanodrop,Thermo Fisher Scientific,Wilmington,De,USA)测定DNA浓度。用DDH稀释提取的DNA2O至20 ng/mL,作为PCR扩增的模板和库。提取的DNA储存在-20°C[43].
转录组测序,DE Novo组装和功能注释
转录组测序短叶苔草采用Biomarker Technologies进行RNA提取,测序采用HiSeq™2000平台[31].在本研究中,使用TRINITY Version 2.5.1检测来自同一转录本的contigs,确定contigs之间的距离,并将它们连接在一起,获得端部不可扩展的contigs [44].TGICL软件用于接触这些单一基因并去除它们的冗余以获得非冗余单个基因。使用爆炸(http://blast.ncbi.nlm.nih.gov/blast.cgi.)已组装单基因的搜索(e值<1E-5)[45那46].将该序列与以下数据库进行比较:基因本体(GO),真核外科蛋白(Kog),基因和基因组(Kegg)的京都百科全书,以及非冗余蛋白(NR)的国家生物技术信息中心。
使用MISA鉴定转录组的SSR [47].使用primer3(http://primer3.sourceforge.net/releases.php)对于每个SSR底漆设计。优先选择SSR基因座含有两至六个核苷酸的术语。另一种选择原理是二核苷酸的六个最小重复单元,五三核苷酸和所有高阶动脉中的四个,包括四核苷酸,五核苷酸和六核苷酸和六核苷酸。
识别SSR和底漆设计
使用11的DNA进行SSR标记的扩增苔属植物具有大表型差异的物种。九十六个SSR引物,其被识别短叶苔草采用RNA-seq随机选取。所有引物均由瑞博生物技术公司合成。在所合成的96对引物中,有42对多态性水平较高,用于进一步的实验研究(表1)4.).使用Bio-Rad T100热循环仪进行所有反应。PCR反应体系的总体积为10μL,包括5μL2×Taq主混合物,0.2μl引物,2μl基因组DNA,2.6μlDDH2o .用于放大的热剖面由最初的10分钟变性在94°C,紧随其后的是34周期30年代在94°C, 30年代在优化退火温度,60年代在72°C扩展,最后5分钟在72°C的延伸。使用8.0%非变性聚丙烯酰胺凝胶与100 bp梯子标记(TRANSGEN BIOTECH,北京,中国)分离成功的PCR产物并通过银染色观察[48].DNA为79.苔属植物使用SSR引物扩增个体以分析遗传多样性。观察到光灯下的凝胶图像上的清晰条带,有或没有带(1)或(0)。
遗传多样性分析
利用POPGENE Version 1.3.2构建二元定性数据矩阵并进行分析。通过计算多态条带百分比(PPB)、有效等位基因数(Ne)、观察等位基因数(Na)、Nei’s基因多样性(H)和Shannon’s信息指数(I)来确定不同材料的遗传多样性。条带的多态信息(PIC)计算公式如下:\(\ mathrm {pic} = 1- \ sum \ limits_j ^ i {p_ {ij}} ^ 2 \)(P.一世和P.j是频率一世th和j一个基因座的等位基因)。我们使用了NTSYS版本2.1的软件[49]根据Nei的遗传距离构建所选组Q模式的集群分析描述[50]. 然后,采用算术平均的非加权两两法对参数进行分析。
群集分析和79种附加的Amova分析
相似之处苔属植物使用NTSYS 2.1进行评估。根据SSR数据集的相似性矩阵,使用UPGMA群集方法来构建树木图[51]. 利用PowerMarker版本3.25和MEGA-5,基于79个个体间的Dice差异矩阵构建了未加权系统发育树[52那53]. 基于FreeTree程序的Bootstrap函数,通过1000次重复Bootstrap分析,评价了系统发育树的稳健性[54]. 利用GenAlEx 6.5程序,基于两两距离矩阵,根据二元基因型之间的吻合差异进行主坐标分析(PCoA)[55]. 距离和协方差都是标准化的。
使用分子方差分析(Amova)分析地理基团之间和内部的涉及遗传变异[56].F统计学用于分析种群之间的遗传分化。使用Genalex 6.5软件进行两种分析[55].
数据和材料的可用性
本研究中生成的Illumina NGS读取已提交给国家生物技术信息中心的Bioproject数据库(Prjna488506)。
缩写
- AFLP公司:
-
扩增的片段长度多态性
- Amova:
-
分子方差分析
- 走:
-
基因本体论
- H:
-
基因多样性指数
- 我:
-
香农信息索引
- ISSR:
-
简单序列重复
- Kegg:
-
京都基因和基因组百科全书
- KOG:
-
真核矫形组
- na:
-
观察到的等位基因的平均数
- 不:
-
有效等位基因
- NJ:
-
邻居加入
- 编号:
-
NCBI非冗余蛋白质数据库
- PCoA:
-
主坐标分析
- 聚合酶链反应:
-
聚合酶链反应
- 照片:
-
多态信息内容
- RAPD:
-
随机扩增多态性
- RFLP:
-
限制片段长度多态性
- SRAP:
-
序列相关的扩增多态性
- 苏维埃社会主义共和国:
-
简单的序列重复
参考
- 1。
Pedersen Mat,Nowak MD,Brysting Ak,Elven R,BjoråCS。混合起源灰株苔草北方变种C. Stenolepis.,在Carex段Vesicariae(Cypetaceae)中的两个有问题的分类群。Plos一个。2016; 11(10):1。
- 2。
Gillespie EL、Pauley AG、Haffner ML、Hay NM、Estep MC、Murrell ZE。一个广泛分布的石灰岩地方病的14个多态性微卫星标记,Carex eburnea(莎草科:苔属植物教派。阿尔法).苹果厂SCI。2017; 5(8):1700031。
- 3.
关键词:微卫星,引物,引物引用本文Carex Moorcroftii.(莎草科),青藏高原草原的优势种。我是J Bot。2011;98(12):e382–4。
- 4.
叶烨,王伟林,郑春生,付军,刘华伟。4种野生植物的抗寒性评价苔属植物酷寒。应用生态学报。2017;28(1):89-95。
- 5.
王y,杨h,李x,王w,白c,刘g。苔属植物jianfengensis(苔属植物教派。菱形目(莎草科)海南一新种。《公共科学图书馆•综合》。2015;10 (9):e0136373。
- 6.
Léveillé-Bourret É, Starr JR, Ford BA。为什么这里有这么多莎草?苏门答腊石竹科,巨型石竹属进化之谜中缺失的一块苔属植物(Cypetaceae)。mol phylocyet evol。2018; 119:93-104。
- 7.
贝尼特斯·贝尼特斯C、埃斯库德罗M、罗德里格斯·桑切斯F、马丁布拉沃S、吉姆·梅耶斯P。上新世-更新世生态位演化形成了地中海植物群的系统地理学。摩尔经济。2018;27(7):1696–713.
- 8.
MíguezM,Gehrke B,Maguilla E,Jiménez-MejíasP,Martín-Bravo S.苔属植物教派。Rhynchocystis(Cypetaceae):西部亚热带依赖于西部的Palaearctic,显示了分散兰特群模式。J生物地理。2017年。
- 9。
Martín-Bravo S、Jiménez Mejías P、Villaverde T、Escudero M、Hahn M、Spalink D、Roalson EH、Hipp AL、Benítez Benítez C、Bruederle LP等,《世界性成功的故事:成功的幕后》苔属植物(莎草科)生物地理学和多样化。系统进化杂志。2019;57(6).
- 10。
GC。使苔草单系(莎草科,羊齿草科):一个新的更广泛的分界。acta botanica yunnanica(云南植物研究),2015;
- 11.
Group TGC,Jiménez-MejíasP,Hahn M,Lueders K,Starr Jr,Brown BH,Chouinard BN,Chung Ks,Escudero M,Ford Ba。越野发育标本级别的方法苔属植物(Cypetaceae)使用其,ETS和MATK序列的系统发育:对分类的影响。SYST BOT。2016年。
- 12.
Jones CJ,Edwards KJ,Castaglione S,Winfield Mo,Sala F,Wiel CVD,Bredemeijer G,Vosmejer G,Matthes M,Daly A.欧洲实验室网络RAPD,AFLP和SSR标记的再现性测试。mol品种。1997; 3(5):381-90。
- 13
Hokanson SC、Szewc McFadden AK、Lamboy WF、McFerson JR.微卫星(SSR)标记揭示了一个物种的遗传特性、遗传多样性和亲缘关系马鲁斯X家庭borkh。核心子集集合。Al Appl Genet。1998; 97(5-6):671-83。
- 14
Escudero M、Vargas P、Arens P、Ouborg NJ、Luceno M。东、西、北地中海及欧洲沿海植物的殖民史苔属植物extensa(莎草科)。摩尔经济。2010;19(2):352–70.
- 15
基于rna测序的EST-SSR标记开发[J]E西伯利亚及其对十七篇文化的系统发育关系分析Elymus.物种。BMC植物BIOL。2019; 19(1)。
- 16
关键词:EST-SSR标记,rna测序,草甘膦,草甘膦Lolium Multiflorum与相关物种交叉可转移性。mol品种。2018; 38(6)。
- 17
Alisoltani A,Ebrahimi S,亚洲人S,Hematyar M,Rafiei F.在非生物胁迫下对SSR和差异表达基因的并行考虑杏仁和相关的功能标志物的靶向发育李属物种。SCI Hortic-Amsterdam。2016; 198:462-72。
- 18
卡里亚RK、赖MK、卡里亚S、辛格R、达旺AK。微卫星标记:植物研究进展综述。大提琴。2011;177(3):309–34.
- 19。
Senthilvel S,Shaik M,Anjani K,Shaw Rk,Kumari P,Sarada C,Kiran Bu。蓖麻核心种质衍生的近交系集合中的遗传变异性和群体结构。j植物生物学习生物。2017; 26(1):27-34。
- 20。
Chapman Ma,Hvala J,Strever J,Matvienko M,Kozik A,Michelmore RW,Tang S,Knapp SJ,Burke Jm。来自红花的EST-SSR标记的开发,多态性和交叉分类效用(Carthamus tinctoriusL.)。Al Appl Genet。2009; 120(1):85-91。
- 21。
周S,王C,弗雷泽TP,严H,陈P,陈Z,黄L,张X,彭Y,马X。首次基于Illumina的甘草从头转录组分析及分子标记开发(Pennisetum purpureum.).mol品种。2018; 38(7):95。
- 22。
NIE G,唐L,张Y,Huang L,Ma X,Cao X,Pan L,Zhang X,Zhang X.基于转录组测序和常年草液耐旱性分析的SSR标记的开发芒草来自中国。前植物SCI。2017; 8。
- 23。
Choi JK、Sa KJ、Park DH、Lim SE、Ryu SH、Park JY、Park KJ、Rhee HI、Lee M、Lee JK。玉米DH群体遗传连锁图谱的构建及农艺性状相关qtl的鉴定(Zea Mays.L.)使用SSR标记。基因组织。2019; 41(6):667-78。
- 24。
Che Kp,Xu Y,Liang Cy,Gong Gy,Weng ML,Zhang Hy,Jin DM,Wang B. AFLP指纹和西瓜核心系列的瘢痕标记。Acta Bot Sin。2003; 45(6):731-5。
- 25。
日本樱花的起源(李属亚因子cerasus)使用核SSR标记显示的品种。树遗传学基因组织。2014; 10(3):477-87。
- 26。
Mbaya JBMJ,Hoffmann AA。遗传结构苔属植物来自澳大利亚高山地区的物种沿着海拔梯度:繁殖和基因流动模式。int j植物sci。2013; 174(2):189-99。
- 27。
小斯塔尔,纳齐RF,乔纳德BN。莎草属(苔草科,莎草科)植物DNA条形码与物种解析。分子经济资源。2010;9(s1):151–63。
- 28。
宁H,王W,郑C,李Z,朱C,张Q.遗传多样性分析胶凝(苔属植物SPP。)在山东,中国基于简单的序列重复。Biochem Systematics Ecol。2014; 56:158-64。
- 29
Nagasawa K,Setoguchi H,Maki M,Goto H,Fukushima K,Isagi Y,Sakaguchi S. EST-SSR标记的开发和特征苔属植物angustisquama(Cypetaceae),Solfatara领域的exprigophyte。苹果厂SCI。2018; 6(10):e01185-n / a。
- 30.
文赫,李赫,米什拉,洪赫。玉米的遗传变异与群体结构短叶苔草(Cypetaceae)在韩国。J植物BIOL。2000; 43(3):136-42。
- 31。
滕K,滕WJ,文HF,岳YS,郭伟,吴JY,范XF。PacBio单分子长阅读测序揭示了基因的复杂性短叶苔草转录组。BMC基因。2019;20(1):789.
- 32。
周F,王Z,YU G,Shi B,王X,强H,高H.基于RNA测序的简单序列重复(SSR)标记的开发与表征Medicago Sativa.在硅绘图到M. Truncatula.基因组。《公共科学图书馆•综合》。2014;9 (3):e92029。
- 33。
刘志杰、托瑞S、塔蒂尼M、布鲁内蒂C、芬斯基S、菲尼A、费里尼F、塞巴斯蒂亚尼F。毛白栎叶片RNA序列分析:从头转录组组装、注释和功能标记开发。公共科学图书馆一号。2014;9(11):e112487。
- 34。
李马,龙瑞聪,冯子瑞,刘福清,孙勇,张凯,康江明,王子忠,曹实。盐敏感基因的转录组分析及盐敏感基因的SSR标记探索Carex rigelens.使用RNA序列。综合农业杂志。2018;17(1):184–96.
- 35。
王S,王X,何Q,刘X,徐W,李L,高J,王F。利用Illumina双端测序技术对萝卜苗期和苗期根系进行转录组分析及EST-SSR标记的建立。植物细胞代表2012;31(8):1437–47.
- 36。
Yu F-H,Kruesi BO,Schneller JJ,Schuetz M,Tang M,Wildi O。植被异质性与植物遗传分化正相关苔草.植物群。2009; 204(9):651-7。
- 37
严D,赵X,程Y,马X,黄L,张X。植物的系统发育和多样性分析dactylis glomerata.利用SSR和IT-ISJ标记分析亚种。分子。2016;21(12):1459。
- 38
国王MG,罗尔森EH。中国11个微卫星位点的分离与鉴定大头苔草(莎草科)。保存基因。2009;10(3):531–3.
- 39
Bharti R,Kumar S,Parekh MJ。基因组简单序列重复(GSSR)标志物在孜然及其在多样性分析中的应用和交叉转移性。ind crop prod。2018; 111:158-64。
- 40.
马丽,孔德科,刘赫伯,王平,李伊,庞XM。36中国枣栽培的SSR指纹施工。Acta Horticulturae Sinica。2012; 39(04):647-54。
- 41.
谭立强,彭敏,徐丽丽,王丽丽,陈淑霞,邹勇,齐坚宁,程宏。128个中国无性茶树品种的SSR指纹分析及其家系关系。中国生物医学工程学报,2015;
- 42.
金钟君,崔鸿基。分子识别Schoenoplectiella使用微卫星标记物种(Cypetaceae)。植物系统中的Evol。2012; 298(4):811-7。
- 43.
Baruah J、Gogoi B、Das K、Ahmed NM、Sarmah DK、Lal M、Bhau BS。人群遗传多样性研究Cymbopogon.来自Ne-India的物种使用RAPD和ISSR标记。ind crop prod。2017; 95:235-43。
- 44。
Haynsen MS、Vatanparast M、Mahadwar G、Zhu D、Moger Reischer RZ、Doyle JJ、Crandall KA、Egan AN。从头转录组组装Pueraria Montana var。Lobata.和Neuustanthus phopsoloides用于开发ESSR和SNP标记:缩小侵入性Kudzu的美国起源。BMC基因组学。2018; 19(1):439。
- 45。
Altschul SF、Madden TL、Schäffer AA、Zhang J、Zhang Z、Miller W、Lipman DJ。Gapped-BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸研究,1997年;25:3389–402.
- 46。
Evangelistella C,Valentina A,Ludovisi R,Firrincieli A,Fabbrini F,Scalabrin S,Cattonaro F,Morgante M,Mugnozza Gs,Keurentjes JJB等。de novo集会,功能诠释和巨型芦苇的分析(Arundo Donax.L.)叶片转录元提供了用于开发生物燃料原料的工具。Biotechnol生物燃料。2017; 10:138。
- 47。
拜尔·S、蒂尔·T、门奇·T、舒尔茨·U、马斯彻·M。misaweb:用于微卫星预测的web服务器。生物信息学。2017;33(16):2583.
- 48。
ndjiondjop mn,semagn k,gouda ac,kpeki sb,tia dd,sow m,goungoulou a,sie m,perrier x,ghesquiere a等。遗传变异与人口结构选用格拉伯里马使用Dartseq开发迷你核心系列。前植物SCI。2017; 8。
- 49。
Tohme J,Gonzalez Do,Beebe S,Duque MC。野生豆核心集合的基因池AFLP分析。农作物科学。1996; 36(5):1375-84。
- 50.
Nie M,Bolland HR。来自东非的四种雷地血小鱼物种(Acari:Ixodidae)的精子发生,染色体和性别测定。genetica。1978; 48(3):233-8。
- 51。
Dawyndt P,Meyer HD,Baets BD.《重新审视UPGMA聚类:传递逼近的权重驱动方法》;2006
- 52。
Kumar S,Tamura K,Nei M. Mega:微型计算机的分子进化遗传分析软件。计算苹果生物。1994年; 10(2):189-91。
- 53。
刘凯,Muse SV。PowerMarker:用于遗传标记分析的集成分析环境。生物信息学。2005;9:2128-9。
- 54。
HAMPL V,PavlícekA,Flegr J.基于DNA指纹识别的系统发育树的施工和自卷草引导分析与Freeware Program FreeTreee:应用于Trichomonad寄生虫。INT J SYST EVOL Microbiol。2001; 51(PT 3):731。
- 55。
Speall R,Smouse Pe。Genalex 6.5:Excel中的遗传分析。教学和研究人口遗传软件 - 更新。生物信息学。2012; 28(28):2537-9。
- 56。
贾H,杨H,Sun P,Li J,Zhang J,Guo Y,Han X,Zhang G,Lu M,Hu J. de Novo转录组合,EST-SSR标记的开发和群体生物量柳树的遗传分析那柳树psammophila.SCI批准。2016; 6(1):39591。
致谢
我们感谢生物标记公司(中国北京)为Illumina平台在文库建设和测序方面提供的设施和专业知识。我们也感谢Editage公司(www.editage.com)用于语言编辑。
资金
该研究得到了北京自然科学基金(6204039),中国国家自然科学基金(31901397),北京农业科学研究基金和林业科学基金到KT。每个融资机构都根据研究提案授予基金。它们对实验设计,数据分析或解释或写作稿件没有影响。
作者信息
隶属关系
贡献
ZC和XF构思了该研究并设计了实验。LL进行实验。KT,PT和LL通过JW,Hz,CH,CC,LX和WG的建议分析了数据。LL和KT写了稿件。所有作者都阅读并批准了稿件的最终版本。
通讯作者
道德宣言
伦理批准并同意参与
本研究所用的材料是我们自己收集的。我们遵守了所有相关的机构、国家和国际准则,并规定了获得的适当许可。我们还获得了收集所有植物材料的许可。
同意出版
不适用。
相互竞争的利益
作者声明他们没有相互竞争的利益。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非信用额度中另有规定。
关于这篇文章
引用这篇文章
刘磊,范晓,谭平。等等。基于RNA测序的SSR标记的开发及其在内部的验证苔属植物L.物种。BMC植物生物学21,17(2021)。https://doi.org/10.1186/s12870-020-02792-8
已收到:
公认:
发表:
关键词
- 苔属植物l
- Illumina RNA排序
- 基因函数注释
- SSR标记
- 标记多态性
- 遗传群集