抽象的
背景
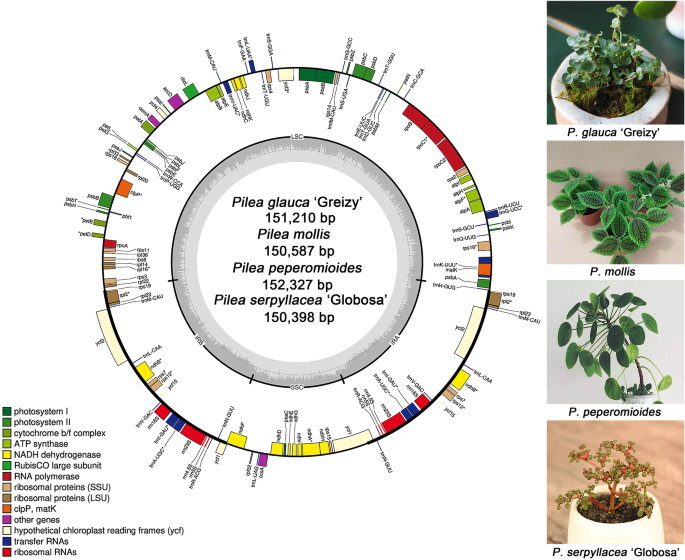
Pilea荨麻是荨麻科多年生草本植物的一个属,有些品种用作庭院观赏或药用。目前,尚无关于该菌质体基因组的信息Pilea,这限制了我们对这个属的理解。在这里,我们报道了4个质体基因组Pilea分类赛(Pilea mollis那Pilea glauca.“Greizy”,Pilea peperomioides和Pilea serpylacea.’Globosa’),并进行综合比较分析。
结果
四个质体基因组都具有典型的四分位数结构。质体基因组长度在150398 ~ 15237bp之间,每个基因组包含113个独特基因,其中79个蛋白质编码基因,4个rRNA基因,30个tRNA基因。比较分析表明,这4个基因组存在较高水平的序列差异。此外,识别出8个高可变区(宠物不-公安局M公安局Z -trn公司G-GCC公司,trn公司T-UGU-trn公司L-UAA公司,ACC.D-公安局一世,ndh公司F-rpl32,rpl32位trn公司L-UAG,ndh公司A-内含子和ycf1),建议作为DNA条形码区域使用。对荨麻科14属23种植物的质体基因组进行了系统发育分析Pilea在系统发生树的中部和下部,荨麻科的bootstrap支持为100%。
结论
我们的结果丰富了有关塑性基因组的资源。对比塑性分析提供了对体积基因组的三分特异性的洞察Pilea. 所鉴定的高变区可用于开发适用于各个研究领域的分子标记。
背景
Pilea种为荨麻科多年生草本植物,主要分布在热带和亚热带地区,部分种分布在暖温带地区。Pilea荨麻属是荨麻科中最大的一个,也是被子植物中比较大的一个属[1].许多种的叶子在Pilea有色斑,可用于园林栽培和观赏目的。它们往往是园林景观中荫凉潮湿环境中的主要植物类群。另一方面,在中国传统药典中,有几种植物被记录为药用植物,从中可以提取多种药理活性物质[2那3.那4.].例如,p . peperomioides《傣医》记载有消炎解毒作用,也用于丹毒和定骨。然而,这一群体却很少受到关注,关于它的报道也很少Pilea. 鉴于许多药用植物形态相似,特别是这些物种丰富的属,基于分子标记的准确物种鉴定对这些药用植物的合理利用尤为重要。
属Pilea在传统分类学中也是一个有争议的群体,以前的研究已经表明Sarcopilea.也属于这个属[5.]. 此外,近年来还报道了一些新种[6.那7.]. 对这一物种丰富的属进行修订,专家学者关注较少,难度较大。此外,对该属的研究报道相对较少,特别是在分子生物学和基因组学领域。虽然一些研究者已经用分子方法来探索属内的系统发育关系Pilea[1它在荨麻菌的系统发育位置[5.],所选择的DNA片段是单侧的、部分完整的,bootstrap支持值较低,具有一定的局限性。因此,我们有必要进一步研究它们的系统发育关系Pilea荨麻疹的种类。
叶绿体是光合作用的一种细胞器[8.植物和藻类的能量转化[9.那10]. 叶绿体基因组(在本文中称为质体基因组或质体体组)编码许多在光合作用和其他代谢特性中起重要作用的关键蛋白质[11]. 在以往的研究中,质体的一些独特特性已被广泛报道,如单系遗传等[12保守的编码区序列[13和基因组结构[14那15].这些可靠的资源为进化、DNA条形码、分类学和系统发育的研究提供了丰富的信息[16那17那18].虽然质体基因组高度保守,但在一些类群中观察到一些有趣的结构变化,如罕见的IR区域的扩张Strobilanthes[15的质体中线粒体DNA的插入Anacardium[19],一些豆科植物的IR区域完全或部分丧失[16那20那21].在最近的一项研究中,Wang等报道了荨麻科58个属中13个属的质体基因组,为该类群植物的研究提供了丰富的质体基因组资源[22].遗憾的是,目前还没有关于该菌质体基因组的报道Pilea植物。
在此,我们对4个质体基因组进行了测序、组装和分析Pilea种,包括一种罕见的本属肉质植物(p . serpyllacea).作为观赏或药用植物,这些植物在形态上(尤其是叶子)有很大的差异,是属的代表。我们的主要工作如下:(1)对4种植物的质体进行了测序和组装Pilea植物物种;(2)分析了中药材中质体的结构特征和序列分化Pilea; (3) 为进一步研究群体遗传结构,我们鉴定了简单序列重复(SSRs)位点和重复序列(4) 我们推断了Pilea荨麻科植物的全质体序列分析;(5)鉴定出高变区,可作为该属成员鉴定的DNA条形码。
结果
质体基因组的一般特征
使用Illumina HiSeq测序平台,每次5.38-5.89 GB清洁数据Pilea净读数为17,935118 ~ 19,627,967(补充文件)1:表S1)。质体是根据这些数据组装的。4个质体Pilea其特征是一个典型的环状DNA分子,长度在150398 ~ 15237bp之间。它们都具有保守的四分位结构,由一个大的单拷贝(LSC)区域(82063 ~ 83,292 bp)、一个小的单拷贝(SSC)区域(17487 ~ 18,363 bp)和一对反向重复(IR)区域(25,180 ~ 25,356 bp)组成(表1)1).质体的长度在这个属中是保留的。GC含量分析表明,4个质粒的GC含量在36.35 ~ 36.69%之间。值得注意的是,IR区GC含量(42.56 ~ 42.73%)显著高于LSC区(33.87 ~ 34.36%)和SSC区(30.01 ~ 30.81%)。原始测序数据和4条基因组序列已存入NCBI数据库(登录号:PRJNA675740和MT726015至MT726018)。
基因组注释
四个的塑性基因组Pilea所有物种均包含113个独特基因,包括79个蛋白质编码基因、4个rRNA基因和30个tRNA基因(附加文件)1:表S2)。这四个物种的基因顺序和基因数目非常相似,显示出保守的基因组结构。数字1的质体示意图Pilea.内含子在选择性基因剪接中起着重要作用[23]. 在注释的79个蛋白质编码基因中,有9个是独特的基因(石头剪刀16,rpoC1级,三磷酸腺苷F,宠物B,宠物D,rpl16,rpl2.ndh公司B,ndh公司a)含有一个内含子和两个独特的基因(ycf3.中电P) 包含两个内含子。此外,6个独特的tRNA基因(trn公司K-UUU,trn公司G-UCC公司,trn公司L-UAA,环境V-UAC,环境I-GAU,trn公司A-UGC)包含一个内含子。有7个蛋白质编码基因,4个rRNA基因和7个tRNA基因在IR区域完全复制,所以它们作为两个拷贝存在。的石头剪刀12基因是反式剪接基因,5'末端位于LSC区域中。然而,在IRA和IRB区域中发现了3'末端。这些结果与荨麻疹中的其他物种类似[22].
重复分析
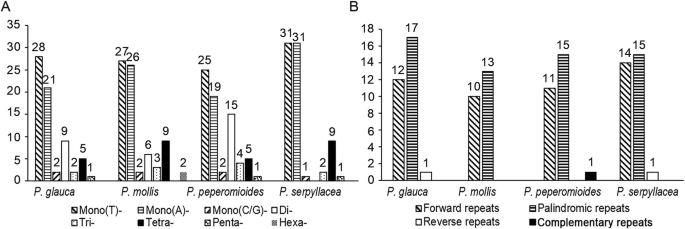
SSRs,又称微卫星序列,提供了大量的遗传信息[24那25那26].由于其高遗传多态性,SSRS通常用于分子标记的发展,并在物种鉴定中发挥重要作用[27那28].在本研究中,我们在4个被分析的物种中检测到68、75、71和80个SSRs(图。2一个,附加文件1:表S3)。SSRs多为单核苷酸均聚物,特别是A/T,占总数的70.75%。六核苷酸重复序列仅在p . mollis.这些SSR显示出高多态性,暗示识别潜力Pilea物种。
在塑性基因组中Pilea物种,我们检测到四种类型的分散重复。它们大多数是向前重复和回文重复(图。2b).相反,只有两个反向重复和一个互补重复。唯一的互补重复出现在p . peperomioides. 详细的序列显示在附加文件中1:表S4。这些短重复的长度主要分布在30 ~ 34 bp之间。我们注意到一个长度为102 bp的正向重复序列(在p . serpyllacea).
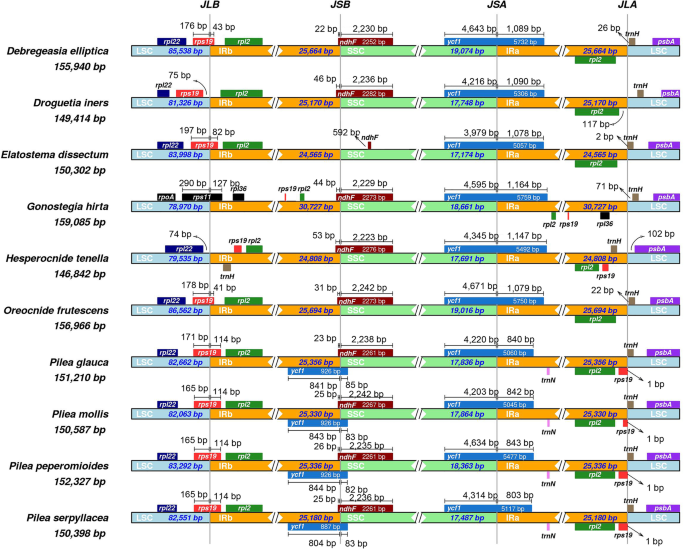
红外区域的收缩和膨胀分析
IR区的收缩和扩展被认为是质体基因组长度多样性的重要原因[29]. 此外,随着IR区域的扩张/收缩,靠近边缘的基因有机会进入IR或SC区域[30].我们检索了已发表的荨麻科6种植物的质粒,并与4种植物的质粒进行了比较Pilea物种。我们发现一些基因跨越或靠近IR和SC区域的边界。主要包括石头剪刀19,rpl22日,rpl2.ycf1,ndh公司F和trn公司H(图。3.).值得注意的是,观察到IR区域的异常扩展Gonostegia hirta. 红外区超过30000个 bp输入G. Hirta.,更多的基因可以进入IR区域(例如,rpl36和石头剪刀19)。然而,其他九种物种中的IR区域的长度约为25,000英镑,而且石头剪刀19个基因跨越LSC/IRb边界,除了在龙舌草和Hesperocnide tenella; 这个石头剪刀前者的19个基因在LSC区域,而后者在IR区域中完全。除此之外trn公司H基因完全进入细胞的IR区h . tenella,获得两份副本。可见,荨麻科部分物种的基因组结构、基因顺序和数量发生了明显的变化。
此外,ycf1基因穿过SSC / IRA边界,其中大部分位于SSC区域。的长度ycf四个基因Pilea物种差异很大,表明可能存在序列差异。令人惊讶的是,我们标注了两份ycf1在四个中Pilea植物;它们越过IRB / SSC边界,并不在其他物种中注释。序列对齐发现两个副本ycf1存在于其他分类群中,表明前一个注释是不完美的,尽管两份副本中的一个是一个片段ycf一般认为是假基因。有趣的是ndh公司F基因(小于100bp)跨越IRb/SSC区域,即第一个拷贝ycf1与ndh公司f in.Pilea物种。重叠区长度为108 bp。
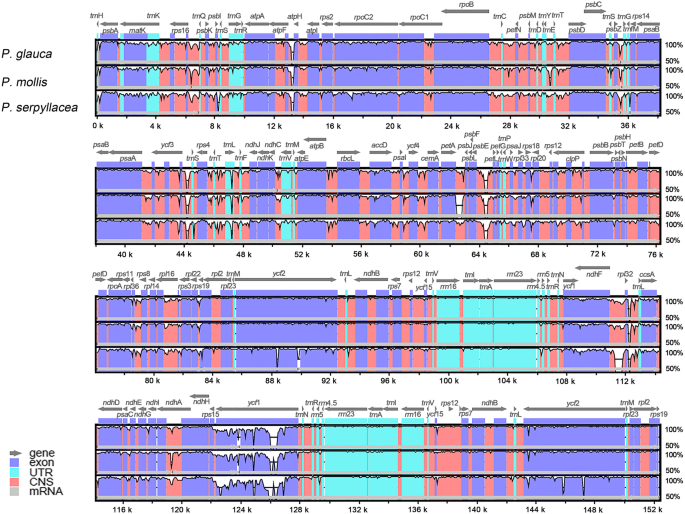
基因组差异
评估基于MVISTA的基因组发散,序列同一性分析[31]在4中进行Pilea物种,以质体为参照p . peperomioides.我们观察到不同程度的序列分歧,特别是在LSC和SSC区域中。相比之下,IR区域更加保守。在保守的非编码序列(CNS)中观察到大多数这些高度可变区域(图。4.).然而,序列差异最大的区域在蛋白质编码区,在基因ycf1存在。的编码区域ycf1在四个中Pilea物种间差异显著,部分片段相似性小于50%。总的来说,分析的基因组序列显示出相当高的水平的序列分歧整个属Pilea.
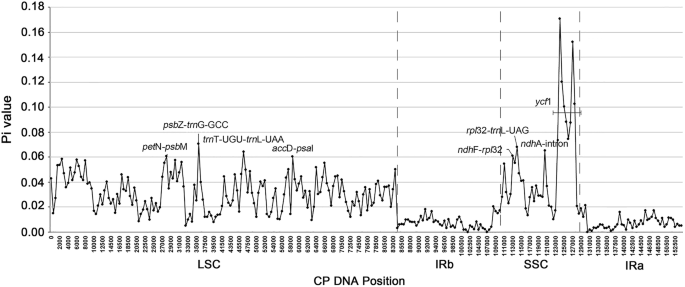
为了量化DNA多态性水平,用DnaSP v6.0对4个基因组进行比对分析[32].我们检测到8个高变区,其中Pi值超过0.06。5.),宠物不-公安局M (Pi = 0.06067);公安局Z -trn公司G-GCC (Pi = 0.07067);trn公司T-UGU-trn公司L-UAA (Pi = 0.06433);ACC.D-公安局我(pi = 0.06003);ndh公司F-rpl32(圆周率) = 0.06100);rpl32位trn公司L-UAG(圆周率) = 0.06800);ndh公司a内含子(Pi = 0.06533),和基因的大部分区域ycf1 (Pi值范围为0.07367 ~ 0.17067)。圆周率的值列在括号中。值得注意的是,大部分质体序列的Pi值都大于0.02 (IR区域除外),表明质体基因组具有丰富的多态性Pilea.
蛋白质编码基因的核苷酸变异
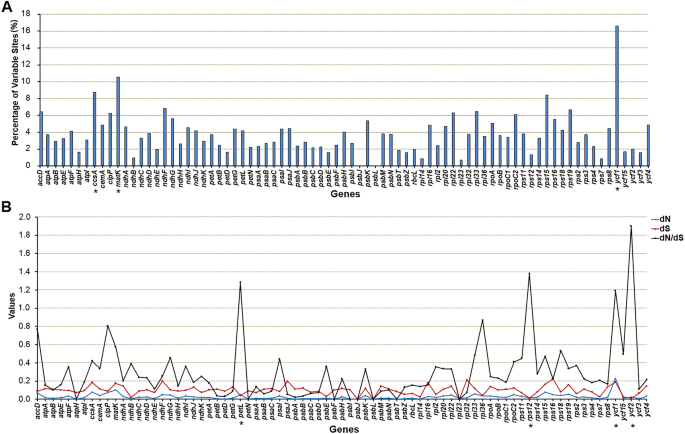
蛋白质编码区在质体基因组中高度保守[33].我们分析了4个鉴定出的79个独特的同源基因的蛋白编码序列Pilea分类群。令人惊讶的是,这些蛋白质编码基因也显示出高水平的变异(图。6.一个,附加文件1:表S5)。在79个共享基因中,63个的突变率超过2%,30个的突变率超过4%。突变率最高的基因是ycf1(16.62%),其次垫K (10.54%),CCS.a(8.74%)和石头剪刀15(8.42%)。只有两个基因(公安局J和公安局五十) 表现出极端的保守性,没有任何可变位点。此外,我们共观察到11个基因(ycf1,ndh公司F,石头剪刀19,ACC.D,rpo指挥中心,石头剪刀16,rpo一种,rpl20,ndh公司D,rpoC1和ycf2)在核苷酸序列中使用dnaps v6.0 [32].在这其中,ycf1人有35个索引,其次是ycf2 (9),ACC.D(4)和rpoC2(3)。考虑到蛋白质编码区的高度保守性,同一属中核苷酸突变率较高的蛋白质编码序列通常不多见,这些结果显示了植物质体基因组的种间多样性Pilea.
在这项研究中,使用pamlv4.9对79个共享基因的同义(dS)和非同义(dN)替换率以及dN/dS进行了平行估计[34].在79个基因中,ycf1,垫KCCS.A和石头剪刀15个有较高的dN值石头剪刀16,rpl32,ndh公司F和psaJ具有较高的dS值(图5)。6.b,附加文件1:表S6)。大部分基因表现出相当低的dN/dS值(小于0.6),表明大部分蛋白编码基因在进化过程中处于纯化选择状态。然而,三个基因的dN/dS比值(rpl36岁,中电P和ACC.d)在0.6和1.0之间。此外,DN / DS比率大于1.0宠物L石头剪刀12,ycf1和ycf2,表明它们在进化过程中处于正选择状态。这些结果清楚地表明,不同种类的植物中的质体基因Pilea可能受到不同的选择压力。
系统发育分析
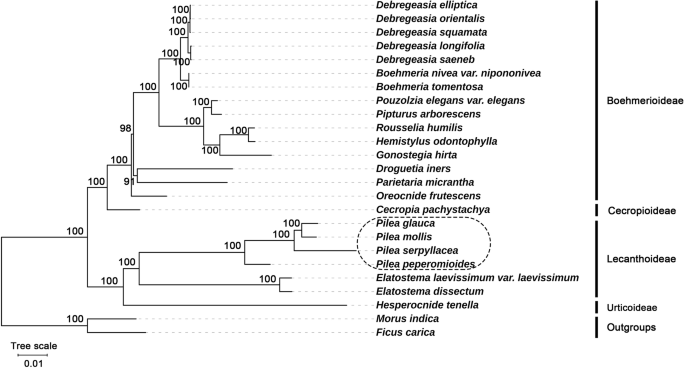
在本研究中,我们利用完整的质体序列作为数据集构建了最大似然树(ML)(详细资料见Additional File)1:表S7)。系统发生树在所有节点上都有很高的自举支持,显示了恢复的系统发生的可靠性(图5)。7.).
我们的系统发育树清楚地显示了两个分支,然后在100%bootstrap支持(ML)下进一步分化为四个子类。这四个亚纲对应于四个亚科:苎麻亚科、天蚕亚科、夜蛾亚科和荨麻亚科。这与传统的分类是一致的[5.].所有4Pilea聚集在一起的物种(所有节点都有B = 100为ML法),形成一个单系群,是Elatostema.它们都属于鸭嘴兽亚科。
讨论
保守的基因组结构和基因内容
在我们的研究中,我们报道了四个质体基因组Pilea首次分类。我们的组装结果显示,4个质体基因组的长度范围为150398 bp至152327 它们具有典型的四分体结构。在结构方面,这些结果与大多数荨麻科植物相似[35那36].本研究中,荨麻科的最长和最短质体基因组分别为159,085 bp (Gonostegia hirta)和146,842 bp(Hesperocnide tenella)分别是。这表明荨麻科的质体基因组可能经历了不同的进化过程。在我们四个人当中Pilea类群中最长的基因组序列是p . peperomioides(152,327 bp)和最短的是p . serpyllacea(150398 bp),我们没有检测到基因的获得或丢失,这表明质体仍然相对保守Pilea.
此外,我们检测了4个质体基因组的SSRs和重复序列。在294个SSRs中,有215个是单核苷酸重复序列,占所有SSRs的绝大部分(73.13%)。这些单核苷酸重复序列主要是A/T重复序列,它们对基因组的总体G/C含量有显著影响[37那38].这些SSR序列通常由简单的重复单元组成,例如聚腺嘌呤(多A)或多苯胺(Poly-T)重复。在不同物种中具有长度多态性,它们通常用作分子标记。塑性中的这些丰富的SSR基因座已应用于物种鉴定[16那39]. 散布的重复被认为是促进质体重排必不可少的[40那41].我们注意到,有一个重复重复,长度为102 bpp . serpyllacea,这对于增加质体的长度是有效的。这些重复是否导致了Pilea物种是一个有趣的问题。
IR区的变异是Agiospers中的常见现象。与整体缺失相比,没有一个IR区域[42那43那44],红外区扩张/收缩在被子植物中更为常见[45那46].通过对比分析,我们发现G. Hirta.显著扩大了IR区域,这也导致了质体总长度的增加。在我们测试的四个Pilea种,红外区长度为25180 bp至25356 血压,无显著差异。就IR/SC的边界区域而言,四个基因在边界附近的位置不同Pilea种与大多数被子植物相似。这表明Pilea物种在红外区没有明显的扩张/收缩。但是,我们观察到的重叠ycf1和SSC区(4634 bp)p . peperomioides与IRa的重叠部分相似(803 bp- 843 bp)。这表明有一个显著的差异ycf1基因序列。另外,第一个副本ycf1与ndh公司f in.Pilea分类群,这一结果也在拟南芥; 重叠部分约为30 英国石油公司[47].这些重叠是否影响这些蛋白质的转录或翻译也是一个有趣的课题。
序列差异揭示了植物质体基因组的种间多样性Pilea
在质体比较基因组学分析中,我们首先基于mVISTA对整个质体基因组进行比较。具体来说,我们还计算了可变位点的百分比,估算了79个正体蛋白编码基因中dN/dS的比例。像大多数被子植物一样,质体的非编码区Pilea显示出比编码区更高的多态性。令人惊讶的是,我们也发现了相当高水平的序列差异的编码区域的基因Pilea分类单元。在鉴定的79个鉴定的79个突变率的情况下,突变率超过2%,30个突变率超过4%。这在其他属中很少见,因为通常只有ycf1基因突变率高[48].突变率ycf四个基因Pilea物种是令人吃惊的16.62%。此外,共检测到35个indel,包括一个大片段插入p . peperomioides(177 bp,数据未显示)。这些因德尔造成了长度的增加ycf1个基因p . peperomioides.此外,还观察到异常高的核苷酸突变率垫KCCS.A和其他基因。
通常,DN改变受到各种突变率和选择性约束的双向效应。DN / DS大于1的比率被认为是基因经历了选择压力的标志。在我们的研究中,DN / DS比率表明四种基因(宠物L石头剪刀12,ycf1和ycf2) 可能经历了积极的选择Pilea.蛋白质编码基因的快速进化与物种的适应性进化密切相关[49那50),这表明Pilea物种可能经历过快速进化过程,导致富含物种的属。
八个高变区域可用作潜在的DNA条形码
此外,我们使用DNASP V6.0通过进行滑动窗口分析来定量DNA序列多态性(窗口长度,500bp;步长,500bp)。类似于MVISTA的结果,除IR区之外的大多数区域具有高PI值,这意味着几个区域具有分子标记的发育的可能性。我们推荐八个高变的地区,宠物不-公安局M (Pi = 0.06067);公安局Z -trn公司G-GCC (Pi = 0.07067);trn公司T-UGU-trn公司L-UAA (Pi = 0.06433);ACC.D-公安局我(pi = 0.06003);ndh公司F-rpl32(圆周率) = 0.06100);rpl32位trn公司L-UAG(圆周率) = 0.06800);ndh公司A-Intron(PI = 0.06533),几乎整个ycfPi值范围为0.07367 ~ 0.17067)Pilea分类单元。特别是基因ycf1,具有大量的InDels,可作为特异性的分子标记,这对我们正确鉴定和合理利用该属的药用类群具有重要意义。
系统发育分析Pilea基于质体基因组
与核基因组和线粒体基因组相比,质体基因组高度保守,已广泛应用于系统发育和进化研究[51那52那53].随着高通量测序技术的发展,叶绿体基因组序列在物种识别中起重要作用作为超级条形码[54那55].
的系统发育关系Pilea对荨麻科植物进行了完整的质体序列分析。在基于质体基因组的片面分析中,Pilea和Elatostema两个类群互为姐妹类群,均属于鸭嘴兽亚科。这与传统分类研究的结果一致[5.]. 然而,由于质体基因组的母体遗传[56,这些结果是有限的。准确的系统发育关系仍然需要对核基因和细胞器基因进行全面分析[57].此外,迄今为止仅测序了58个属植酸菌中的14个。需要更多基因组测序以确定关系中的关系Pilea和来自家庭荨麻科的其他物种。
结论
在这项研究中,四个质体基因组Pilea首次对该属植物进行了测序和组装。这4个质体具有与大多数被子植物相似的结构特征和典型的四分位数结构。不同寻常的是,这4个质体的序列,包括相对保守的蛋白质编码区,具有相当高水平的可变性,这为深入了解中国的质体基因组的种间多样性提供了基础Pilea.此外,鉴定出8个高变区,可作为鉴定该属植物的分子标记。本研究结果丰富了荨麻科植物质体基因组的资料,为荨麻科植物系统发育重建提供了依据Pilea.
方法
植物材料,DNA提取和测序
新鲜的四叶Pilea采自广州、昆明、宿迁等地的花卉市场。他们是由于杰教授指认的。这些物种是作为观赏植物栽培的,采集这些样品不需要许可。我们的实验研究,包括收集植物材料,符合机构,国家或国际准则。所有标本均保存于中国重庆西南大学标本室(凭证编号:UP60602至UP60605)。植物样品的详细信息见附加文件1:表S8。通过使用CTAB方法提取总基因组DNA [58].使用NEBNext®文库构建试剂盒构建插入体大小为350bp的DNA文库,并使用HiSeq Xten PE150测序平台进行测序。测序结果为每个物种提供了5.4-5.9 Gb的原始数据。去除质量较低的序列,包括质量值Q < 19且占总碱基比例超过50%的序列和5%以上的碱基为“N”的序列,得到干净数据。
基因组组装与注释
从清洁数据中使用Novo Genome组件采用Novoplasty V2.7.2完成[59], k-mer长度为39 bp,序列片段为RBC.以玉米L基因为种子序列。用Bowtie2(v2。0.1) [60]在默认设置下手动编辑和映射到组装的基因组序列的所有原始读取。通过使用CpGavas2最初通过CpGAVAS2进行补体基因组[61]与参考基因组(Elatostema解剖基因库:NC_047192.1)。然后使用GeSeq来确认注释结果[62].此外,使用apollo手动编辑问题的注释[63[ogdraw绘制的基因组图[64]. 原始测序数据和四个基因组序列已保存在GenBank(登录号:PRJNA675740;MT726015、MT726016、MT726017和MT726018)。
重复和SSR分析
通过使用压花提供的CUSP程序(V6.3.1)来确定GC含量[65].使用在线网站MISA识别简单的序列重复(SSR)(https://webblast.ipk-gatersleben.de/misa/),包括单,二 - ,三元,四核苷酸,最小数10,5,4,3,3和3的己核苷酸分别[66]. 此外,REPuter(https://bibiserv.cebitec.uni-bielefeld.de/reputer/)用于计算回文重复、正向重复、反向重复和互补重复,设置如下:汉明距离为3,最小重复大小为30 英国石油公司[67].
基因组比较
4的质体Pilea采用mVISTA中的shuffle-LAGAN模式进行物种比较[68那69]鉴定种间变异(http://genome.lbl.gov/vista/mvista/submit.shtml.).通过使用诸古化素鉴定并提取4种物种中的79个原基因[70].相应的核苷酸序列通过使用MaFFT(V 7.450)对齐[71在系统套件中实现。我们使用MEGA v6.0 [72]来计算蛋白质编码基因中可变位点的百分比。我们还使用DnaSP v6.0进行滑动窗口分析(窗口长度:500 bp,步长:500 bp) [32]来计算4个物种之间的核苷酸多态性(Pi)。最后,IRscope (https://irscope.shinyapps.io/irapp/)用来显示这些基因组的红外边界[73].
核苷酸取代率分析
前一步中的蛋白质编码序列是并行处理的。我们在pamlv.4.9中使用了CODEML模块[34]估计包括DN,DS的核苷酸取代的速率,以及DN至DS的比率。详细参数如下:CodonFreq = 2(F3×4型号);型号= 0(允许单个DN / DS值在分支之间变化);cleandata = 1(用模糊数据删除网站);和Codeml控制文件中的其他参数设置为默认设置。通过使用raxml v8.2.4中实施的最大可能性(ml)方法产生每个基因的系统发育树[74].
系统发育分析
从genbi (NCBI,https://www.ncbi.nlm.nih.gov/).这些种隶属于4个亚科1:表S7)。桑科二种(桑属籼和榕辣海参)被用作外组。用MAFFT对完整的质体序列进行比对(https://mafft.cbrc.jp/alignment/server/)在线版本7.471[71]. 利用raxmlv8.2.4中实现的最大似然(ML)方法,利用这些序列构建系统发育树[74].参数为“raxmlHPC-PTHREADS-SSE3 -f a -N 1000 -m GTRGAMMA -x 551314260 -p 551314260”。bootstrap分析采用1000个重复。
数据和材料的可用性
本研究产生的原始测序数据和4个质体基因组序列保存在NCBI (https://www.ncbi.nlm.nih.gov/),注册号:PRJNA675740、MT726015、MT726016、MT726017和MT726018。所有样品都保存在中国重庆西南大学植物标本馆。在合理的要求下,本手稿中产生的所有其他数据和材料均可从通讯作者处获得。
缩写
- 苏维埃社会主义共和国:
-
简单的序列重复
- 中枢神经系统:
-
守恒的非编码序列
- 国税局:
-
反向重复
- LSC公司:
-
大单副本
- SSC:
-
小单副本
- ML:
-
最大似然
- BS:
-
分支机构的支持
- 玻利亚:律
-
多腺苷
- PolyT:
-
多胸腺嘧啶
- dS编号:
-
同义替代率
- DN:
-
非同义词的替代利率
- 达克斯:
-
DNA序列多态性
- CTAB:
-
十六烷基三甲基溴化铵
- NCBI:
-
国家生物技术信息中心
- 圆周率:
-
核苷酸多样性/多态性
参考文献
- 1.
孟氏正义与发展党。植物丰富属的修订:基于cpDNA、nrDNA和形态学的皮莱亚(荨麻科)策略修订系统发育框架中国生物医学工程学报,2006;https://doi.org/10.3732/AJB.93.3.426.
- 2.
周Y,丽丽,任HC,秦路,李问,你PF,窦GF,张QY,梁h .整个植物的化学成分Pilea cavaleriei消减无性系种群。cavaleriei。Fitoterapia。2017;119:100-7。https://doi.org/10.1016/j.fitote.2017.04.010.
- 3.
Prabhakar Kr,Veerapur VP,Bansal P,Parihar VK,Reddy Kandadi M,Bhagath Kumar P,Priyadarsini Ki,Unnikrishnan MK。抗氧化剂和抗射线保护作用对LILEA微蛋白乙醇提取物的活性分数。Chem Biol相互作用。2007; 165(1):22-32。https://doi.org/10.1016/j.cbi.2006.10.007.
- 4.
Modarresi Chahardehi A,Ibrahim D,Fariza Sulaiman S.抗氧化剂,抗菌活性和毒性测试小叶菌毛。国际微生物学杂志2010;2010:826 - 830。DOI:https://doi.org/10.1155/2010/826830.
- 5.
吴征镒,王浩,易ts,刘杰,李德志。从三个基因组的多个位点和广泛的属样取样推断荨麻科(荨麻科)的分子系统发育。分子系统进化。2013;69(3):814-27。https://doi.org/10.1016/j.ympev.2013.06.022.
- 6.
Dorr LJ,斯特吉奥斯B。安第斯山荨麻属(荨麻科)四新种,附委内瑞拉属记述。植物键。2014;42:57–76.https://doi.org/10.3897/phytokeys.42.8455.
- 7.
Monro AK、Wei YG、Chen CJ。中国石灰岩岩溶中荨麻科三新种。植物键。2012;19:51–66.https://doi.org/10.3897/phytokeys.19.3968.
- 8.
Szabò I, Spetea C.叶绿体离子转运体对光合作用优化的影响。实验机器人。2017;68(12):3115-28。https://doi.org/10.1093/JXB/ERX063.
- 9。
Mullineaux PM,Exposito-Rodriguez M,Laissue PP,Smirnoff N.在植物和藻类中依赖于ROS依赖的信号通路,暴露于高光:与其他真核生物的比较。自由Radic Biol Med。2018; 122:52-64。https://doi.org/10.1016/j.freeradbiomed.2018.01.033.
- 10.
Pollari M, Ruotsalainen V, Rantamaki S, Tyystjarvi E, Tyystjarvi T.同时失活sigma因子B和D干扰了蓝细菌Synechocystis sp. PCC 6803的光适应。J Bacteriol。2009;191(12):3992 - 4001。https://doi.org/10.1128/JB.00132-09.
- 11.
王Z,朱旭光,陈Y,李Y,侯J,李Y,刘L。通过比较分析叶绿体和光合细菌的代谢网络来探索光合作用的进化。BMC基因组学。2006;7:100.https://doi.org/10.1186/1471-2164-7-100.
- 12.
Brandrud MK、Baar J、Lorenzo MT、Athanasiadis A、Bateman RM、Chase MW、Hedren M、Paun O。指根属(兰科)二倍体的系统基因组关系及异源四倍体的起源。系统生物学。2020;69(1):91–109.https://doi.org/10.1093/sysbio/syz035.
- 13
Shin DH, Lee JH, Kang SH, Ahn BO, Kim CK。野兔耳根,镰柴胡的完整叶绿体基因组:其分子特征。基因(巴塞尔)。2016; 7(5): 20。https://doi.org/10.3390/genes7050020.
- 14
Ebert D,Peakall R.叶绿体简单序列重复(CPSSR):将CPSSR发现和应用于各种植物物种的技术资源和建议。Mol Ecol Resour。2009; 9(3):673-90。https://doi.org/10.1111/j.1755-0998.2008.02319.x.
- 15
陈浩,邵杰,张浩,姜明,黄磊,张震,杨东,何敏,Ronaghi M,罗欣,等。对红花(Strobilanthes cusia (Nees) Kuntze)叶绿体基因组进行测序和分析,发现被子植物叶绿体的反向重复区同时收缩和扩张。植物科学与技术,2018;https://doi.org/10.3389/fpls.2018.00324.
- 16
[10]蒋敏,陈慧,何胜,王磊,陈爱安,刘超。玫瑰锦鸡儿种质质体的测序、鉴定和比较分析。植物分类学报,2011,30(3):457 - 464。国际分子科学杂志。2018;19(5):1419。https://doi.org/10.3390/ijms19051419.
- 17
刘晓,周斌,杨辉,李勇,杨强,陆勇,高勇菊花carinatumSchousb和Kalimeris indica..植物叶绿体全基因组显示两个逆转录和rbcL作为条形码。分子(瑞士巴塞尔)。2018; 23(6): 1358。https://doi.org/10.3390/molecules23061358.
- 18
[15]庞翔,刘慧,吴胜,袁勇,李辉,董建,刘忠,安聪,苏忠,李波。栎属植物的基因到基因组鉴定。植物分类学报,1999,15(3):467 - 474。国际分子科学杂志。2019;20(23):5940。https://doi.org/10.3390/ijms20235940.
- 19。
Rabah So,Lee C,Hajrah NH,Makki RM,Alharby HF,Alhebshi Am,Sabir Jsm,Jansen Rk,Ruhlman Ta。十种非典范庄稼物种的塑料测序揭示了腰果中的大量线粒体DNA。植物基因组。2017; 10(3):Plantgenome2017.03.0020。https://doi.org/10.3835/plantgenome2017.03.0020.
- 20
丢失和找到:在豆科系里由它的缺失定义的反向重复的返回。基因组生物进化。2019;11(4):1321-33。https://doi.org/10.1093/gbe/evz076.
- 21.
acta botanica yunnanica(云南植物研究中心),云南植物研究中心,云南植物研究中心,云南植物研究中心。植物保护学报(英文版);2014;https://doi.org/10.1111/pbi.12179.
- 22.
王子,Milne ri,du xy,刘j,吴泽。德布雷德西亚(荨麻藻藻)塑性的特征和突变热点。前群体。2020; 11:729。https://doi.org/10.3389/fgene.2020.00729.
- 23.
Plangger R、Juen MA、Hoernes TP、Nußbaumer F、Kremser J、Strebitzer E、Klingler D、Erharter K、Tollinger M、Erlacher MD等。结构域6中的分支位点凸起构象决定了II组内含子剪接中的功能性糖折叠。核酸研究,2019年;47(21):11430–40.https://doi.org/10.1093/nar/gkz965.
- 24.
MA SJ,SA KJ,HONG TK,LEE JK。基于简单序列重复(SSRS),中国北部和南部地区的紫苏作物及其杂草类型的遗传多样性及人口结构分析。基因基因组学。2019; 41(3):267-81。https://doi.org/10.1007/S13258-018-0756-3.
- 25.
seyoum m,du xm,他sp,jia yh,pan z,sun jl。用简单序列重复分析旱地棉(令人讨厌的L.)种质的遗传多样性和人口结构。J Genet。2018; 97(2):513-22。
- 26.
关键词:玉米,ssr, SNPs,亲缘关系遗传。2011;139(8):1045 - 54。https://doi.org/10.1007/s10709-011-9606-9.
- 27.
广奇,夏家,林际,余吉,万QH,方爵。IDSSR:一种用于从单个基因组序列识别多态微卫星的有效管道。int j mol sci。2019; 20(14):3497。https://doi.org/10.3390/ijms20143497.
- 28.
郭强,李旭,杨硕,杨智,孙勇,张娟,曹胜,董玲,Uddin S,李勇。刺槐遗传多样性及分化评价(刺槐L.)基于基因组和表达序列标签 - 简单序列重复。int j mol sci。2018; 19(9):2492。https://doi.org/10.3390/ijms19092492.
- 29.
Goulding SE, Olmstead RG, modern CW, Wolfe KH。叶绿体的潮起潮落反向重复。中国生物医学工程学报,1996;https://doi.org/10.1007/bf02173220.
- 30.
关键词:草本植物,叶绿体,红外光谱,突变热点,全基因组比较分子(瑞士巴塞尔)。2018; 23(7): 1537。https://doi.org/10.3390/molecules23071537.
- 31.
Frazer KA, Pachter L, Poliakov A, Rubin EM, Dubchak I. VISTA:计算工具比较基因组学。核酸研究2004;32(增刊2):W273-9。
- 32.
Rozas J,Ferrer-Mata A,Sánchez-Delbarrio JC,Guiroao-Rico S,Librado P,Ramos-Onsins Se,Sánchez-Gracia A.DNASP 6:大型数据集的DNA序列多态性分析。mol Biol Evol。2017; 34(12):3299-302。https://doi.org/10.1093/molbev/msx248.
- 33.
acta botanica yunyunica(云南植物研究). Hong Z, Wu Z, Zhao K, Yang Z, Zhang N, Guo J, Tembrock LR, Xu D. Comparative analysis of Five Complete叶绿体genome from Pterocarpus (Fabacaeae)。中国生物医学工程学报,2014;https://doi.org/10.3390/ijms21113758.
- 34.
杨卓。PAML 4:最大似然的系统发育分析。中国生物医学工程学报。2007;24(8):1586-91。https://doi.org/10.1093/molbev/msm088.
- 35.
傅立夫,辛子斌,文芳,李S,魏义庚。荨麻科(Elatostema dissectum)叶绿体基因组全序列研究。线粒体DNA第B部分。2019;4:838–9.https://doi.org/10.1080/23802359.2019.1567292.
- 36.
王瑞恩、刘杰、李志海、吴志勇。东方荨麻叶绿体基因组全序列研究。线粒体DNA第B部分。2019;4(1):1830–1.https://doi.org/10.1080/23802359.2019.1604186.
- 37
朱奇拉,阿沃加S,李Z,胡G,王Q,陈J。千里光科(菊科)11个完整叶绿体基因组的比较基因组学:DNA条形码和系统发育。底部螺柱。2019;60(1):17.https://doi.org/10.1186/s40529-019-0265-y.
- 38
李W,张建,郭X,刘Q,王克。山茶花粳稻结构的完整叶绿体基因组,对比和系统发育分析。Plos一个。2019; 14(5):E0216645。https://doi.org/10.1371/journal.pone.0216645.
- 39
苏艳,刘艳,李智,方志,杨玲,庄敏,张勇甘蓝抗裂头性的QTL分析(英文)Brassica oleracea.L利用SSR和InDel标记进行全基因组重测序。公共科学图书馆一号。2015;10(9):e0138073。https://doi.org/10.1371/journal.pone.0138073.
- 40
Lee HO, Joh HJ, Kim K, Lee SC, Kim NH, Park JY, Park HS, Park MS, Kim S, Kwak M,等。三种蜂科植物“邦邦”的叶绿体基因组动态重排及DNA条形码研究。国际分子物理学报,2019;https://doi.org/10.3390/ijms20092196.
- 41
关键词:美国雏菊(Melampodium sect. Melampodium, Asteraceae),异源多倍体,重复DNA进化系统医学杂志。2018;67(6):1010 - 24。https://doi.org/10.1093/sysbio/syy024.
- 42
Rousseau-gueutin M,Bellot S,Martin Ge,Boutte J,Chelaifa H,Lima O,Michon-Coudouel S,Naquin D,Salmon A,AInouche K,等人。六倍体孢子渣(Poaceae,Holidoideae)的叶绿体基因组:比较分析和分子约会。mol phylocyet evol。2015; 93:5-16。https://doi.org/10.1016/j.ympev.2015.06.013.
- 43
郑W,陈J,郝Z,史J。水稻叶绿体基因组信息的比较分析杉木(羊)。钩与柳杉属、台湾柳杉属和青杉属的同胞种。2016;17(7):1084。https://doi.org/10.3390/ijms17071084.
- 44
郝智,程涛,郑锐,徐辉,周勇,李敏,陆芳,董勇,刘鑫,陈杰,等。残存针叶树Glyptostrobus pensilis的全叶绿体基因组序列:柏科和松科叶绿体基因组重排的比较分析和动态洞见。《公共科学图书馆•综合》。2016;11 (8):e0161809。https://doi.org/10.1371/journal.pone.0161809.
- 45.
朱艾,郭伟,古普塔,风扇W,割草机jp。塑体倒置的进化动力学重复:膨胀,收缩和替代率损失的影响。新植物。2016; 209(4):1747-56。https://doi.org/10.1111/3/13743.
- 46.
他J,Yao M,Lyu Rd,Lin Ll,Liu Hj,Pei Ly,Yan SX,谢L,程J.完全叶绿体基因组和血管素(毛茛科)的全叶绿素基因组和塑性系统的结构变化。SCI批准。2019; 9(1):15285。https://doi.org/10.1038/s41598-019-51601-2.
- 47.
Asaf S, Khan AL, Khan MA, Waqas M, Kang SM, Yun BW, Lee IJ。拟南芥叶绿体基因组。gemmifera和拟南芥lyrata ssp。佩特拉-:结构和比较分析。SCI批准。2017; 7(1):7556。https://doi.org/10.1038/s41598-017-07891-5.
- 48.
东路,徐C,李成,孙俊,佐,施,程T,郭继,周S. YCF1,土地植物最有前途的塑植DNA条形码。SCI REP。2015; 5:8348。https://doi.org/10.1038/srep08348.
- 49.
董文伟,王仁荣,张妮妮,范文斌,方文敏,李振华。兰花种叶绿体基因组的分子进化:系统发育关系和适应性进化。国际分子科学杂志。2018;19(3):716。https://doi.org/10.3390/ijms19030716.
- 50.
黄毅,王杰,杨毅,范丙,陈杰。盐柳科叶绿体基因组的系统基因组分析和动态进化。前植物科学。2017;8:1050.https://doi.org/10.3389/fpls.2017.01050.
- 51.
杜yp,毕y,杨fp,张mf,陈xq,xue j,zhang xh。完全氯化石叶绿体基因组序列:进化动力学和系统发育分析的见解。SCI批准。2017; 7(1):5751。https://doi.org/10.1038/s41598-017-06210-2.
- 52.
关键词:凤仙花,白芍,叶绿体全基因组序列,系统发育分子(瑞士巴塞尔)。2018; 23(2): 246。https://doi.org/10.3390/molecules23020246.
- 53.
沈鑫,郭胜,尹勇,张杰,尹旭,梁超,王智,黄斌,刘勇,肖胜,等。叶绿体全基因组序列及系统发育分析艾斯特塔塔利卡斯.分子(瑞士巴塞尔)。2018; 23(10):2426。https://doi.org/10.3390/molecules23102426.
- 54.
FU CN,WU CS,YE LJ,MO ZQ,Liu J,Chang YW,Li Dz,Chaw Sm,Gao LM。全球紫杉(Taxus)塑性超级条形码的异构塑性和有效性的患病率。SCI批准。2019; 9(1):2773。https://doi.org/10.1038/s41598-019-39161-x.
- 55.
关键词:毛草,叶绿体,超条形码,物种识别引用本文:Sci众议员2018;8(1):1924。https://doi.org/10.1038/s41598-018-20399 -2198-018-20399-W..
- 56.
小克里斯蒂,比克曼M。单亲遗传促进细胞质基因组的适应性进化。分子生物学。2016;34(3):677–91.https://doi.org/10.1093/molbev/msw266.
- 57.
Górniak M, pan O, Chase MW。基于低拷贝核编码基因Xdh的兰科系统发育关系:与细胞器和核糖体DNA结果一致。中国生物医学工程学报,2010;https://doi.org/10.1016/j.ympev.2010.03.003.
- 58.
Arseneau JR,Steeves R,Laflamme M.从富含污染组织的高质量DNA改性低盐CTAB提取。Mol Ecol Resour。2017; 17(4):686-93。https://doi.org/10.1111/1755-0998.12616.
- 59.
Dierckxsens N,Mardulyn P,Smits G. Novoplasty:来自整个基因组数据的细胞器基因组的De Novo组装。核酸RES。2017; 45(4):E18-8。https://doi.org/10.1093/nar/gkw955.
- 60。
Langmead B,Trapnell C,Pop M,Salzberg SL.短DNA序列与人类基因组的超快和记忆有效比对。基因组生物学。2009;10(3):R25。https://doi.org/10.1186/gb-2009-10-3-R25.
- 61。
Shi L,Chen H,Jiang M,Wang L,Wu X,Huang L,Liu C. CpGavas2,综合塑料序列注释器和分析仪。核酸RES。2019; 47(W1):W65-W73。https://doi.org/10.1093/nar/gkz345.
- 62。
tilich M, Lehwark P, Pellizzer T, Ulbricht-Jones ES, Fischer A, Bock R, Greiner S. GeSeq -通用和准确的细胞器基因组注释。核酸研究。2017;45(W1): W6-w11。https://doi.org/10.1093/nar/gkx391.
- 63。
Misra S,Harris N.使用Apollo浏览和编辑基因组注释。Curr Protoc生物信息学。2005; 12(1):9.5.1-9.5.28。https://doi.org/10.1002/0471250953.bi0905s12.
- 64
GREINER S,LEHWARK P,BOCK R. UniterellarGenomedraw(OGDraw)版本1.3.1:扩展工具包用于细胞细胞基因组的图形可视化。核酸RES。2019; 47(W1):W59-W64。https://doi.org/10.1093/nar/gkz238.
- 65
赖斯P,朗登I,布莱斯比A。浮雕:欧洲分子生物学开放软件套件。趋势Genet。2000;16(6):276–7.https://doi.org/10.1016/s0168 - 9525 (00) 02024 - 2.
- 66
贝尔S、蒂尔T、芒克T、舒尔茨U、马斯彻M。misaweb:用于微卫星预测的web服务器。生物信息学。2017;33(16):2583–5.https://doi.org/10.1093/bioinformatics/btx198.
- 67
Kurtz S, Choudhuri JV, Ohlebusch E, Schleiermacher C, Stoye J, Giegerich R. REPuter:重复分析在基因组尺度上的多重应用。核酸杂志2001;29(22):4633-42。https://doi.org/10.1093/nar/29.22.4633.
- 68
Frazer KA, Pachter L, Poliakov A, Rubin EM, Dubchak I. VISTA:计算工具比较基因组学。核酸RES。2004;32(Web服务器问题):W273-W279。DOI:https://doi.org/10.1093/nar/gkh458.
- 69
蒂尔T,米哈勒克W,瓦尔什尼RK,格兰纳A。利用EST数据库开发大麦(Hordeum vulgare L.)基因源SSR标记及其特性。理论应用遗传学2003;106(3):411–422.内政部:https://doi.org/10.1007/s00122-002-1031-0.
- 70.
4 .张丹,高峰,李文新,王广德。基于分子序列数据管理的进化系统发育研究。系统科学与技术,2016,36(5):649 - 652。Mol Ecol Resour. 2020;20(1): 348-55。https://doi.org/10.1111/1755-0998.13096.
- 71.
关键词:MAFFT-DASH,整合蛋白序列,结构比对核酸研究2019;47(W1): W5-w10。https://doi.org/10.1093/nar/gkz342.
- 72.
Tamura K、Stecher G、Peterson D、Filipski A、Kumar S。分子进化遗传学分析6.0版。分子生物学。2013;30(12):2725–9.https://doi.org/10.1093/molbev/mst197.
- 73.
IRscope:一种可视化叶绿体基因组连接位点的在线程序。生物信息学。2018;34(17):3030 - 1。https://doi.org/10.1093/bioinformatics/bty220.
- 74.
斯塔马塔基斯公司。raxmlversion8:一个用于系统发育分析和大型系统发育后期分析的工具。生物信息学。2014;30(9):1312–3.https://doi.org/10.1093/bioinformatics/btu033.
确认
作者非常感谢Novogene(天津)提供的技术支持。
资金
国家自然科学基金项目(no . 31772260);重庆市留学创新工程项目(no . cx2019052)。关键词:岩石力学,岩石力学,岩石力学,数值模拟资助人没有参与研究设计、数据收集和分析、决定发表或手稿准备。
作者信息
从属关系
贡献
JieY构思了研究并设计了实验;JingY利用Illumina平台采集样品并提取DNA进行测序;FH对cp基因组进行组装和注释;JMT、SYZ和JLL进行叶绿体比较分析;JLL起草了手稿。所有作者已阅读并批准最终稿件。
相应的作者
道德宣言
道德认可和参与同意
这四个人Pilea种广泛分布在中国作为观赏植物。实验研究不包括遗传转化,保存所使用物种的遗传背景,以及任何其他需要伦理批准的过程。
同意出版
不适用。
相互竞争的利益
作者声明他们没有相互竞争的利益
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:表S1。
测序数据质量摘要。表S2。水稻质体基因组的基因组成Pilea.表S3。4个塑性基因组中简单序列重复(SSRS)的统计数据。表S4。重复(> = 30 (英国石油公司)在四个Pilea物种。表S5.4的可变部位和诱导的百分比Pilea物种。表S6.4个基因共有79个基因的dS、dN和dN/dS值Pilea物种。表S7.用于系统发育分析的质体基因组列表。表S8。植物样品的概要信息。
权利和权限
开放存取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非信用额度中另有规定。
关于这篇文章
引用本文
李,J.,唐,J.,曾,S。等等。四种植物质体基因组的比较Pilea(尿酸核酸)物种:深入了解三种体积基因组多样性Pilea.BMC植物生物学21,25(2021)。https://doi.org/10.1186/s12870-020-02793-7.
收到了:
公认:
发表:
关键词
- Pilea
- 质体基因组
- 种间多样性
- 高变区
- 系统发育关系