- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
基因组 - 盐胁迫条件下常见小麦产量及相关性状的研究gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba21gydF4y2Ba, 文章编号:gydF4y2Ba27gydF4y2Ba(gydF4y2Ba2021.gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
土壤盐渍化是对小麦生产的重大威胁。必须了解培育和选择具有增加小麦产量的新耐盐品种的盐耐盐性的遗传基础。gydF4y2Ba
结果gydF4y2Ba
在本研究中,对191份小麦材料进行了全基因组关联研究(GWAS),以鉴定与成年期性状相关的SNP标记。采用Wheat660K SNP序列对该群体进行基因分型,连续3年在低盐和高盐环境下对8个表型性状进行研究。389个SNPs代表11个qtl,与不同盐处理下的株高、穗数、穗长、粒数、千粒重、产量和生物质量呈极显著相关,表型解释率(gydF4y2BaRgydF4y2Ba2gydF4y2Ba),由9.14至50.45%不等。其中,4A、5A、5B和7A染色体上的重复和多效位点与低盐条件下千粒重、穗数、穗长、粒数等产量及产量相关性状显著相关。不同盐处理下,穗长相关位点主要位于1B、3B、5B和7A染色体上。4D和5A染色体上的两个位点分别与低盐和高盐环境下的株高有关。三个耐盐相关位点在两个双亲本群体中被证实是重要的。有利单倍型的分布表明,5组染色体上多效位点的优势单倍型被强选择,具有提高小麦耐盐性的潜力。为提高小麦耐盐育种的选择效率,在9个与产量及相关性状相关的位点上建立了14个KASP标记。gydF4y2Ba
结论gydF4y2Ba
利用Wheat660K单核苷酸多态性芯片,在天然小麦群体中检测了盐处理下产量及其相关性状的qtl。在RIL和DH人群中验证了重要的耐盐相关基因座。本研究为小麦耐盐育种提供了可靠的分子标记。gydF4y2Ba
背景gydF4y2Ba
全球受盐度影响的耕地面积超过8亿公顷,相当于全球陆地面积的约6% [gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba].此外,由于灌溉,受盐影响的土壤比例每年都在增加,这可能导致农业土壤的盐积累[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba4gydF4y2Ba].因此,盐渍化已成为粮食产量的主要限制因素[gydF4y2Ba2gydF4y2Ba].常见的小麦(gydF4y2Ba小麦gydF4y2BaL.)是一种重要的谷类作物,为世界上约20%的人口(gydF4y2Bahttp://www.fao.org/faostatgydF4y2Ba).为了满足不断增长的世界人口的粮食需求,小麦产量必须大幅度提高。gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba].常见的小麦是一种适度的耐盐作物,并且在盐渍土中生长时可以产生降低的产量[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].因此,了解普通小麦盐胁迫响应和适应的相关机制,利用耐盐品种在小麦遗传资源的选育和利用中具有重要意义。gydF4y2Ba
一些盐度适应相关机制,例如渗透调节,组织耐受过程和NAgydF4y2Ba+gydF4y2Ba在大米,玉米和几乎没有[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba].然而,由于其大(〜17 gb),复杂的和六倍(aabbdd)基因组,小麦的盐度耐受性的研究是有限的限制[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba].值得注意的是,随着高通量测序技术的发展,小麦全基因组信息的增加[gydF4y2Ba12gydF4y2Ba].同时,鉴定了许多单一核苷酸多态性(SNP)并置于SNP芯片上,该芯片已成为基因组关联研究(GWASS)和定量特征的基因分型平台(GWASS)和定量性状映射[gydF4y2Ba5gydF4y2Ba].例如,Wu等人揭示了控制颗粒形状和大小的qtl [gydF4y2Ba13gydF4y2Ba利用小麦Infinium iSelect 9k SNP基因分型技术对燕大1817/北农6重组自交系(RILs)进行基因分型研究。Ma et al. [gydF4y2Ba14gydF4y2Ba使用小麦90k阵列在衍生自小燕6的育种群体中进行了六个产量相关性状的Gwas。通过高密度小麦820k阵列表征了一系列二倍体,四倍体和六倍倍体涂层,并使用56,505个SNP标记构建小麦共有地图[gydF4y2Ba15gydF4y2Ba].最近,中国农业科学院设计了一个新的小麦SNP序列Wheat660K。Wheat660K序列包含约630,517个SNPs,这些SNPs来自192个普通小麦和相关物种的低覆盖测序(gydF4y2Bahttps://wheat.pw.usda.gov/gnpages/topics/wheat660_snp_array_developed_by_caas.pdf.gydF4y2Ba).Wheat660K基因具有基因组特异性和高密度的优势,在基因发现、单倍型定位、基因组选择和进化研究等方面具有广泛的应用前景。例如,利用Wheat660K SNP芯片在小麦品种Yaco“S”中检测到一个主要效应QTL,该QTL可能是一个新的成体植物抗条锈病基因[gydF4y2Ba16gydF4y2Ba].Cui等人。[gydF4y2Ba11gydF4y2Ba]在六倍体小麦高密度遗传图谱(Wheat660K array)上发现了一个主要稳定的穗粒数QTL。与矮化基因密切相关的标记gydF4y2Barht24.gydF4y2Ba被Tian等人确认[gydF4y2Ba17gydF4y2Ba利用Wheat660K阵列。gydF4y2Ba
Gwass基于连锁不平衡,该方法已成为基因发现和复杂性状的标记开发的流行和有效的工具[gydF4y2Ba18gydF4y2Ba].具有多种优点,例如使用各种种质作为材料和捕获繁殖实践所遗弃的优质等位基因的能力,Gwass已被广泛用于解剖调节小麦中生物和非生物胁迫耐受性的复杂遗传机制[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].例如,Riaz等人。[gydF4y2Ba21gydF4y2Ba[]利用10748个DArT-seq标记对295份面包小麦的叶锈病抗性进行了GWAS分析。Mwadzingei等人[gydF4y2Ba22gydF4y2Ba对93个不同的面包小麦基因型在干旱胁迫条件下的农艺性状进行了评价。然而,在盐胁迫条件下小麦主要稳定基因座的鉴定方面,迄今为止利用遗传算法进行的研究还很有限。Hussain等[gydF4y2Ba23gydF4y2Ba[关键词]小麦,微量元素,qtl定位gydF4y2Ba2gydF4y2Ba品系在苗期盐胁迫条件下生长。利用GWASs技术鉴定了冬小麦种子萌发期和苗期耐盐qtl [gydF4y2Ba20.gydF4y2Ba].此外,以往的研究大多局限于小麦苗期。有限的研究集中在小麦成虫期。gydF4y2Ba
在这里,在南浦生态农业实验站的三个连续赛期间评估了191个小麦种畜的表演,该季节在渤海周围的低平原中具有典型的盐渍化土壤。并且Wheat660K阵列用于鉴定与不同盐度条件下的小麦涂层的八种表型特征相关的SNP标记,目的是提供有关影响小麦产量和实际盐胁迫条件下相关性状的重要等位基因变异的有价值的信息,并找到可用的SNP小麦盐耐受育种计划的标记和品种。gydF4y2Ba
结果gydF4y2Ba
对盐胁迫的响应、表型性状的变异及其相关性gydF4y2Ba
对2014-2017年3个作物季节(低盐和高盐)的191份小麦材料进行表型分析。各性状在两种不同盐度处理下均存在显著差异。与LS条件相比,HS条件下8个表型性状的平均值均有不同程度的下降,说明HS条件下对小麦品种生长的抑制作用更为显著(表1)gydF4y2Ba1gydF4y2Ba).此外,对同一性状在同一处理下的3个田间试验进行了相关系数分析。所有成虫期性状均呈极显著正相关,田间试验重复性中等(见表)gydF4y2BaS2gydF4y2Ba).所有表型性状的遗传力估计均为中等至高遗传力水平。所有表型性状在LS条件下的遗传力均高于HS条件下的遗传力(表)gydF4y2Ba1gydF4y2Ba).PH在LS下的遗传力最高(gydF4y2BaHgydF4y2Ba2gydF4y2Ba = 88.58%) and HS (HgydF4y2Ba2gydF4y2Ba = 56.30%) conditions. SL and KPS have moderate and relatively stable heritability levels, withHgydF4y2Ba2gydF4y2BaLS条件下的56.83和39.5%的值分别为HS条件下的46.52和31.59%。与其他特征相比,YPP和GN在LS和HS条件下显示出类似但较低的遗传性水平(表gydF4y2Ba1gydF4y2Ba).Pearson在三个盐度条件下的3年间平均数据计算了特征之间的相关性的共同效率(表gydF4y2Ba2gydF4y2Ba).pH与Sn,Sl,KPS,BM,YPP和GN显着正相关,在盐胁迫条件下,pH和BM和GN之间的相关系数比较高于PH和其他两个盐度环境之间的其他性状.TKW与pH,Sn,KPS和GN显着显示出显着的负相关,但在盐度处理下与SL和YPP呈正相关。此外,BM和GN显示出LS和HS条件下的与YPP非常高,正相关。在盐度条件下,产率及其组分(Sn,KPS和TKW)之间的相关性显着呈阳性。重要的是,在两个条件下,YPP和Sn之间的相关系数最高,而YPP和TKW之间的相关系数与YPP和KPS之间的相关系数较低。有趣的是,与LS治疗相比,在HS处理下,YPP和Sn和KPS的相关系数略微增加。gydF4y2Ba
SNP基因分型gydF4y2Ba
根据PLINK软件提供的质量预处理结果(gydF4y2Bahttp://zzz.bwh.harvard.edu/plink/data.shtml.gydF4y2Ba除去8451和221,878个SNP,用于分别具有小于80%的呼叫率和小于0.05的次要等位基因频率。低基因分型未删除加入(MIND> 0.1)。最后,保留了191种含有387,657个SNP的含量以进行进一步分析。其中,322,590个SNP标记被映射到Wheat660K共识图(Prof.jizeng jia,Pers.comm。)。表中提供每麦染色体上呈现的SNP的数量gydF4y2BaS3gydF4y2Ba.191个品种的387657个SNPs共检测到740124个变异等位基因(表)gydF4y2BaS3gydF4y2Ba).21条小麦染色体的等位基因数平均范围为1.38 ~ 3.25。平均等位基因数最大的依次是6D、2D和4A染色体。3B染色体平均等位基因数最低(表)gydF4y2BaS3gydF4y2Ba).gydF4y2Ba
遗传结构和联系不平衡gydF4y2Ba
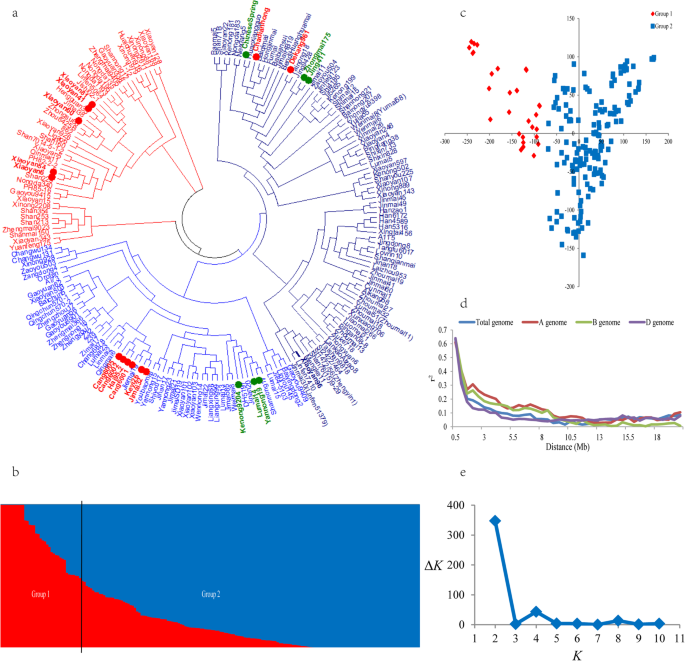
使用不同的方法来分析人口结构。基于Nei标准遗传距离的邻近方法[gydF4y2Ba24gydF4y2Ba用来分类191种附加,并表明它们分为两组(图。gydF4y2Ba1gydF4y2Ba一种)。第一组(45)主要由陕西的改善品种组成。第二组(146)主要由源自北京,河北,河南和山东的改善品种组成。通过PCA和人口结构分析进一步证实这两组。当群体的数量(gydF4y2BaKgydF4y2Bas)绘制δgydF4y2BaKgydF4y2Ba使用软件结构计算,最高ΔgydF4y2BaKgydF4y2Ba被观察到gydF4y2BaKgydF4y2Ba = 2 (Fig.1gydF4y2BaB和e),表明191份植物类群可分为2个亚群。PCA也显示了两个不同的聚类(图。gydF4y2Ba1gydF4y2Bac).利用邻接法构建的系统发育树,将耐盐小麦品种仓6001、仓6002、仓6005、德康961、济麦32和科艺26聚为第2类。gydF4y2Ba
利用A、B和D亚基因组的153,725、184,755和49,177个SNP标记计算连锁不平衡。基于批判的gydF4y2BargydF4y2Ba2gydF4y2Ba阈值0.1,整个基因组的LD衰减距离约为5.0 MB。在亚基因组中发现了8.5 MB的最高LD衰减距离,然后在B次基因组中进行8.0mb。D子基因组具有3.5mb的最低LD衰减距离(图。gydF4y2Ba1gydF4y2Bad)。gydF4y2Ba
特征和SNP之间的关联gydF4y2Ba
在成人阶段和SNP标记的表型性状之间进行关联分析。在六种环境中,在389个SNP中鉴定了389个SNP中的611个显着相关的信号,在六种环境中鉴定出八个表型特征gydF4y2BaP
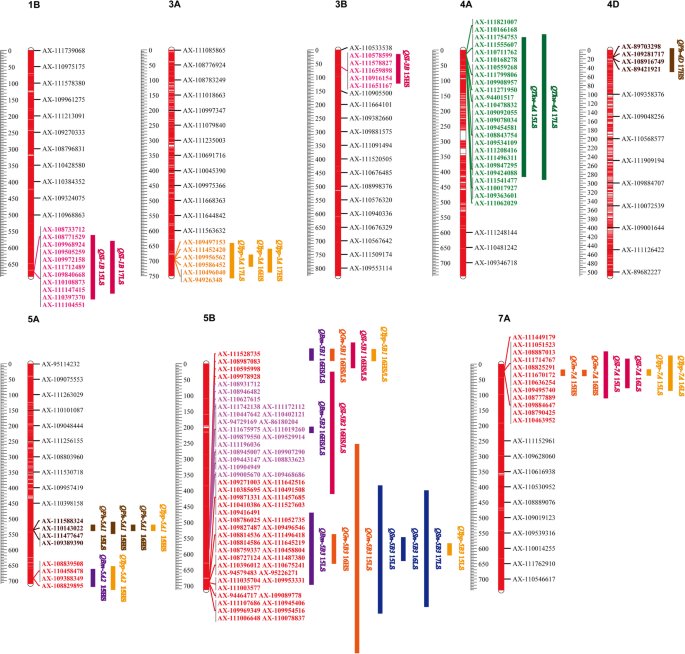
191份普通小麦品种表型性状主要qtl在染色体上的分布棕色、蓝色、玫瑰红、紫色、黄色、橙色和绿色条形分别代表株高(PH)、穗数(SN)、穗长(SL)、生物质量(BM)、小区产量(YPP)、粒数(GN)和千粒重(TKW)。条形图的长度表示检测到的QTL的snp数量。gydF4y2BaQPH.gydF4y2Ba为PH的QTL,gydF4y2BaQSN.gydF4y2Ba表示SN的QTL,gydF4y2BaQSL.gydF4y2Ba表示SL的QTL,gydF4y2BaQBmgydF4y2Ba表示BM的QTL,gydF4y2Baqypp.gydF4y2Ba代表ypp的qtl,gydF4y2BaQGngydF4y2Ba代表GN的QTL,gydF4y2Baqtkw.gydF4y2Ba代表tkw的qtl。LS表示低盐度处理;HS表示高盐度处理gydF4y2Ba
植物高度gydF4y2Ba
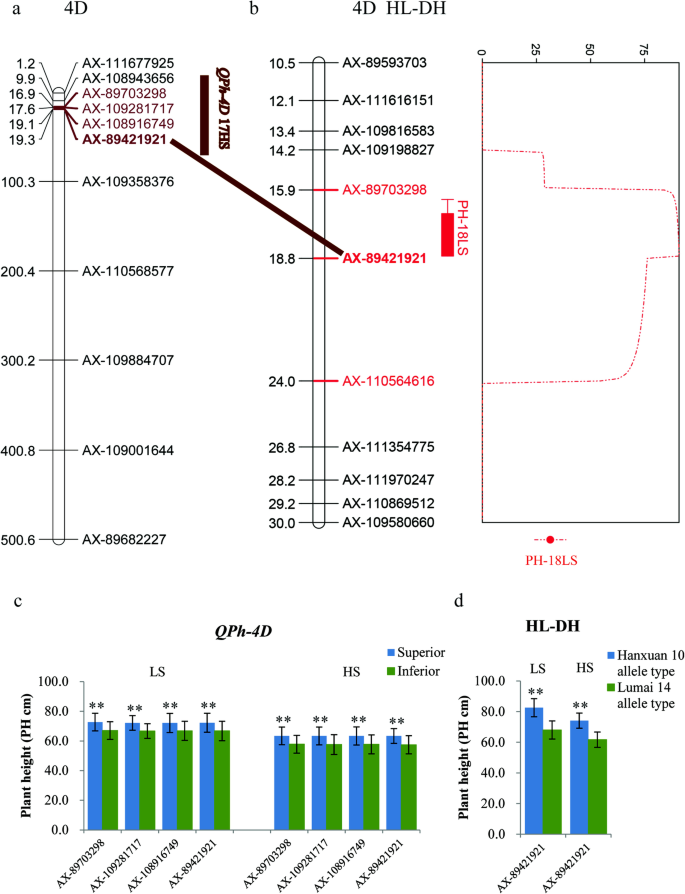
共检测到141个与PH相关的显著SNPs,主要分布在2B、2D、3D、4A、4D、5A、6B、7B和7D染色体上(表1)gydF4y2BaS4gydF4y2Ba).其中,在LS条件下的至少两个环境中检测到79个SNP,主要位于染色体5a上(表gydF4y2BaS5gydF4y2Ba).HS条件下,在同一环境中检测到8个与PH相关的SNPs,它们位于染色体4A(2)、4D(4)和5A(2)上(表)gydF4y2BaS5gydF4y2Ba).一个SNP位点被命名为gydF4y2BaQPH-5A1.gydF4y2Ba含有四个SNP并位于53.91-536.80 MB的染色体5A上,与15LS和15HS中的pH显着相关,解释表型变异的44.46-67.67%。更重要的是,一个SNPgydF4y2BaQPH-5A1.gydF4y2Ba与15HS中的YPP显着相关。在4D染色体上,位于间隔16.93-19.29 MB的四个SNP(图。gydF4y2Ba2gydF4y2Ba),定义为gydF4y2BaQPh-4DgydF4y2Ba,与17HS的PH相关,解释了52.81 ~ 53.62%的表型变异。gydF4y2Ba
穗长gydF4y2Ba
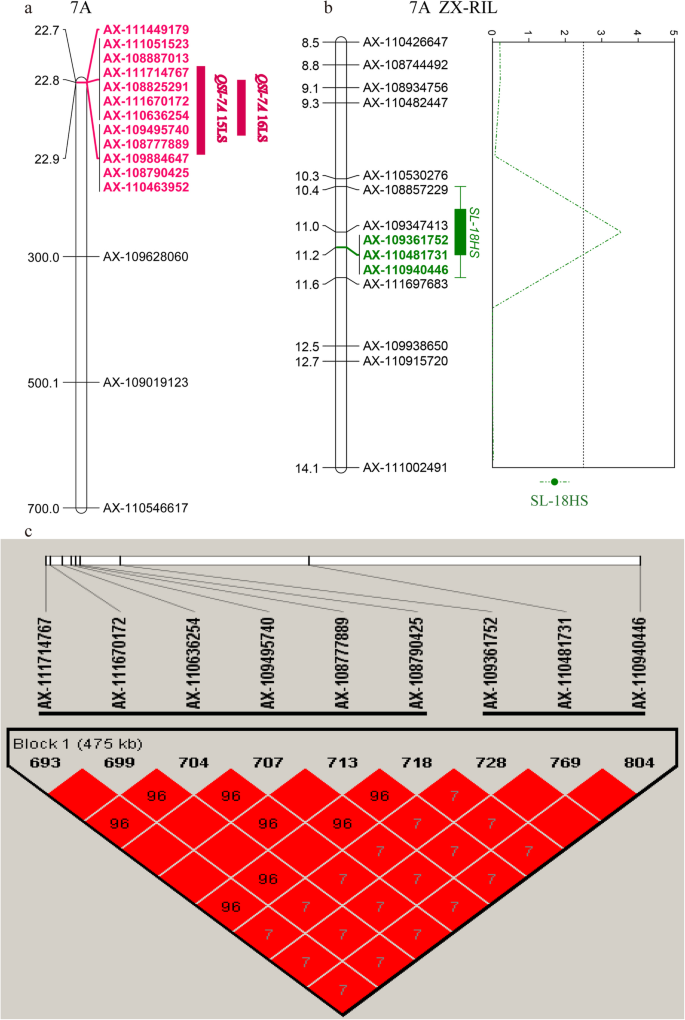
总共80个重要的SNP与SL相关,并分布在染色体1a,1b,2a,2d,3b,3d,5b,5d,6d和7a上(表gydF4y2BaS4gydF4y2Ba).其中,在LS条件下的两个环境中检测到染色体1B上的九个SNP和3个SNP(表gydF4y2BaS5gydF4y2Ba).同时,在HS条件下的两种环境中检测到染色体1a(1),1b(1),2a(1)和2d(1)上的四个SNP(表gydF4y2BaS5gydF4y2Ba).与SL相关的重要位点主要位于1B、3B、5B和7A染色体上。轨迹gydF4y2BaQSl-1BgydF4y2Ba,其中15LS和17LS有11个显著的SNPs,位于1B染色体的674.96-675.13 Mb(图2)。gydF4y2Ba2gydF4y2Ba)并解释了16.38-23.12%的表型变异。轨迹gydF4y2BaQSl-3BgydF4y2Ba由五个SNP组成,与15HS中的SL相关,位于3b上的62.13-62.14 MB(图。gydF4y2Ba2gydF4y2Ba),解释表型变异的12.18 ~ 12.82%。在5B染色体上,有4个SNPs位于574.06-578.35 Mb(图2)。gydF4y2Ba2gydF4y2Ba),命名为gydF4y2BaQ-5B1gydF4y2Ba,与SL(16HS / 16LS)的相对值相关联,并解释了表型变异的12.94-14.29%。重要的是,两个SNPgydF4y2BaQ-5B1gydF4y2Ba与BM、GN、YPP的相对值显著相关。轨迹gydF4y2BaQSl-5B2gydF4y2Ba,包含21个SNPs,位于5B染色体上583.48 ~ 584.89 Mb区间(图)。gydF4y2Ba2gydF4y2Ba).它与SL的相对值(16HS/16LS)显著相关,可解释13.03-15.28%的表型变异(表1)gydF4y2BaS5gydF4y2Ba).此外,一个SNPgydF4y2BaQSl-5B2gydF4y2Ba与BM的相对值(16HS/16LS)显著相关(表gydF4y2BaS5gydF4y2Ba).轨迹gydF4y2BaQ-7AgydF4y2Ba,共涉及12个SNPs,定位在7A染色体的22.75 ~ 22.88 Mb(图2)。gydF4y2Ba2gydF4y2Ba),其中15LS和16LS中有10个snp与SL显著相关,占穗长变异的20.20-28.47%。总共有7个snpgydF4y2BaQ-7AgydF4y2Ba与15LS和16LS的YPP显著相关,gydF4y2BaQ-7AgydF4y2Ba与15HS和16HS的GN显著相关(表gydF4y2BaS5gydF4y2Ba).gydF4y2Ba
粒数gydF4y2Ba
总共60个SNP与GN显着相关,主要位于染色体2A,3A,5B和7A上(表gydF4y2BaS4gydF4y2Ba).相关SNP的比较SN和GN的染色体5B累积的常见SNP呈大规模常见的SNP,其在公共轨迹处积累gydF4y2BaQ-5B3gydF4y2Ba(690.08 - -690.70 Mb)(图gydF4y2Ba2gydF4y2Ba).在该基因座,在LS环境中检测到34个SNP,在LS和HS条件下检测到五个SNP。值得注意的是,对成对性状的相关分析显示GN和Sn之间的相关性极高,分别为0.859和0.891以下的物质处理。gydF4y2Ba
生物质量gydF4y2Ba
高达18个重要的SNP与BM相关,它们主要分布在染色体4a,4b,5a和5b上(表gydF4y2BaS4gydF4y2Ba).其中15LS和16HS/16LS中分别检测到9个和3个5B染色体上的SNPs,分别能解释10.04-13.90%和12.61-12.80%的表型变异。gydF4y2Ba
单株产量gydF4y2Ba
共有48个显著的SNPs与YPP相关,主要分布在染色体2D、3A、5A、5B和7A上(见表)gydF4y2BaS4gydF4y2Ba).其中,六个snps,形成轨迹gydF4y2BaQYPP-3AgydF4y2Ba,位于染色体3A,694.01-694.96 MB。该基因座在16HS,17LS和17H中与YPP相关联,其表型变异为11.36-16.32%(图。gydF4y2Ba2gydF4y2Ba).另外,四个SNP,组成轨迹gydF4y2BaQ-5A2gydF4y2Ba,位于5A染色体692.16 ~ 692.39 Mb,在15HS中检测到,表型变异为10.69 ~ 12.70%(图2)。gydF4y2Ba2gydF4y2Ba).snpsgydF4y2BaQ-5A2gydF4y2Ba与15HS中的BM显着相关。与YPP和SL相关联的常见基因座gydF4y2BaQ-7AgydF4y2Ba(22.75 ~ 22.88 Mb),在7A染色体上观察到。gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
钉号码gydF4y2Ba
多达68个重要的SNP与Sn相关,并且大多位于染色体1a,1b,2a,2b,5a,5b,5d,6d和7b(表gydF4y2BaS4gydF4y2Ba).其中,LS条件下,5B染色体上有19个SNPs, HS条件下,5A染色体上有2个SNPs。轨迹gydF4y2BaQ-5B3gydF4y2Ba在5B染色体690.08 ~ 690.70 Mb处共发现36个SNPs,其中24个SNPs与15LS、16LS和17LS的SN相关(图1)。gydF4y2Ba2gydF4y2Ba)可以解释表型变异的17.29-32.82%。而且,SNPgydF4y2BaQ-5B3gydF4y2Ba在15LS(34)和16HS(5)中,不仅GN与15LS(9)中BM显著相关(表gydF4y2BaS5gydF4y2Ba).gydF4y2Ba
Thousand-kernel重量gydF4y2Ba
完全,与TKW相关的31个重要的SNP主要分布在染色体3B,3D,4A,4B和7B上(表gydF4y2BaS4gydF4y2Ba).其中,最多的SNP(最多25)来自染色体4A。这些SNP,形成轨迹gydF4y2BaQTkw-4AgydF4y2Ba在14.68-16.28 MB,在15LS和17LS中检测到,可以解释18.60-35.57%的表型变异。gydF4y2Ba
等位基因在自然群体中的表型效应gydF4y2Ba
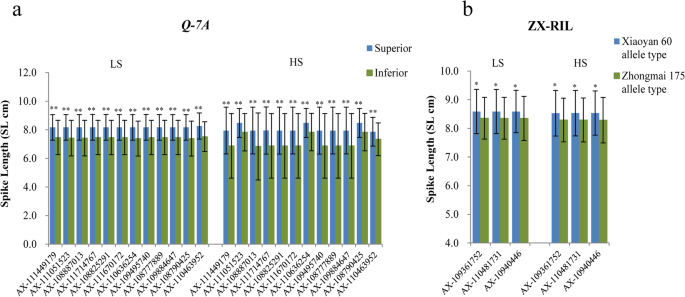
一个gydF4y2BatgydF4y2Ba进行测试以验证基于显着相关的SNP的天然群体中的有利等位基因,并且本研究中的一些基因座被确定为可能影响盐胁迫条件下小麦的农艺性状。例如,在基因座中具有有利等位基因的载体gydF4y2BaQSl-1BgydF4y2Ba(674.96-675.13 MB),gydF4y2BaQSl-3BgydF4y2Ba(62.13 - -62.14 Mb)gydF4y2BaQSl-5B2gydF4y2Ba(583.48-584.89 MB)显示较长的SLS(用LS增加0.54-0.97厘米,用HS×0.57-2.44厘米)。位点的有利等位基因gydF4y2BaQTkw-4AgydF4y2Ba(14.68-16.28 Mb)在LS下增加了4.06-5.32 g,在HS下增加了2.49-5.95 g。位点的有利等位基因gydF4y2BaQPh-4DgydF4y2Ba(16.93 ~ 19.29 Mb)在LS下增加了3.42 ~ 4.57 cm,在HS下增加了4.21 ~ 4.31 cm。具有有利等位基因的遗传gydF4y2BaQ-5B1gydF4y2Ba(574.06-578.35 Mb)的SLs长(1.06-2.46 cm), BMs大(4.72-5.45 g), GNs大(23.80-65.29)和YPPs大(2.58-8.69 g)。具有有利等位基因的遗传gydF4y2BaQ-5B3gydF4y2Ba(690.08 - -690.70 Mb)有更大的BMs(由1.49 - -4.54 g LS和HS下1.44 - -1.89 g), SNs (LS下0.46 - -1.50和0.35 - -0.64在HS), GNs (LS下14.49 - -60.75和16.16 - -25.68在HS)和YPPs(由2.71 - -5.73 g LS和HS下2.43 - -2.88 g)。位点的有利等位基因gydF4y2BaQ-7AgydF4y2Ba(22.75-22.88 MB)分别在LS下增加SL和YPP和2.16-3.39克,并在HS下增加了1.07-1.11厘米(表gydF4y2BaS6gydF4y2Ba).gydF4y2Ba
上述关键位点的等位基因频率范围为5.76 ~ 93.72%gydF4y2BaS6gydF4y2Ba).对有利等位基因分布的分析表明,引入的品种具有最高平均良好等位基因数,其次是改善的品种。Landrace中有利等位基因的平均数量是最低的(图。gydF4y2BaS1gydF4y2Ba一种)。除了甘肃和四川的加入,大多数是山地,河北,河南和陕西的加入较高的平均数量的有利等位基因,其次是来自山东,北京和山西的品种。然而,青海的栽培品种具有最低的有利等位基因数量(图。gydF4y2BaS1gydF4y2Ba因此,认为应利用引种品种和改良品种改良耐盐小麦的产量相关性状。此外,一些知名品种的相关性状的有利等位基因也存在差异。以科农181、良行99、早优504和锦河9123为例,虽然同源性相近,但科农181和良行99均含有位点上的有利等位基因gydF4y2BaQSl-1BgydF4y2Ba,gydF4y2BaQSl-5B2gydF4y2Ba和gydF4y2BaQ-7AgydF4y2Ba早油504和锦河9123具有较好的等位基因gydF4y2BaQSl-1BgydF4y2Ba,gydF4y2BaQSl-3B1gydF4y2Ba, 和gydF4y2BaQ-7AgydF4y2Ba.此外,Landrace Chadianhong和Baiqimai的YPP是相似的。查德洪含有洛基的有利等位基因gydF4y2BaQ-5A2gydF4y2Ba和gydF4y2BaQ-7AgydF4y2Ba而白旗麦在基因座中具有较好的等位基因gydF4y2BaQYPP-3AgydF4y2Ba和gydF4y2BaQ-5A2gydF4y2Ba.gydF4y2Ba
基于重要基因座的Kompetitive等位基因特异性PCR(KASP)标记gydF4y2Ba
总共设计了14个KASP标记,并用于检测ZX-RIL和HL-DH种群中的多态性(表gydF4y2BaS8gydF4y2Ba).八个kasp标记是多态的。其中,四(gydF4y2Bakasp - 5695gydF4y2Ba,gydF4y2Bakasp - 9337gydF4y2Ba,gydF4y2BaKASP-6491.gydF4y2Ba和gydF4y2Bakasp - 4516gydF4y2Ba)在ZX-RIL群体中显示多态性,2 (gydF4y2Bakasp - 9179gydF4y2Ba和gydF4y2Bakasp - 4767gydF4y2Ba)在HL-DH人群中具有多态性,gydF4y2BaKASP-8827gydF4y2Ba和gydF4y2BaKASP-9508.gydF4y2Ba)在ZX-RIL和HL-DH群体中均具有多态性。根据各KASP标记的基因分型结果,agydF4y2BatgydF4y2Ba对相关DH和RIL群体进行相应性状检测。gydF4y2Ba
KASP标记gydF4y2BaKASP-9508.gydF4y2Ba,来自轨迹gydF4y2BaQ-5A2gydF4y2Ba,在ZX-RIL以及HL-DH种群中是多态的。在HL-DH人口中,gydF4y2BaKASP-9508.gydF4y2Ba在两个环境(LS和HS)中与pH,BM和TKW显着相关,并且在HS条件下与YPP相关联(图。gydF4y2BaS2gydF4y2Ba广告)。对于轨迹gydF4y2BaQ-7AgydF4y2Ba,标记gydF4y2Bakasp - 9179gydF4y2Ba和gydF4y2Bakasp - 4767gydF4y2Ba在HL-DH人群中具有多态性。gydF4y2BaKASP-9179gydF4y2Ba在两个环境(LS和HS)中,在LS条件下的SL和HS条件下的BM和YPP有显着相关(图。gydF4y2BaS2gydF4y2Ba情况)。gydF4y2BaKASP-4767.gydF4y2Ba在HS条件下的pH,BM和YPP和LS条件下的SL显着相关(图。gydF4y2BaS2gydF4y2Ba我)。因此,基因座gydF4y2BaQ-5A2gydF4y2Ba和gydF4y2BaQ-7AgydF4y2Ba在盐胁迫条件下,对BM、PH、SL、TKW和YPP具有稳定性和多效性。gydF4y2Ba
讨论gydF4y2Ba
与以往研究比较及关键位点的基因预测gydF4y2Ba
利用Axiom Wheat660K基因芯片对191份小麦材料进行基因分型,利用GWAS技术对盐渍条件下与农艺性状相关的基因组区域和SNPs进行了鉴定。与株型、穗部性状、籽粒产量及其组成性状显著相关的SNPs主要存在于1B、3A、3B、4A、4D、5A、5B和7A染色体上。LS条件下显著相关SNPs的数量远多于HS条件下,这与干旱胁迫条件下的结果相似[gydF4y2Ba22gydF4y2Ba,gydF4y2Ba25gydF4y2Ba],表明环境相互作用的基因型可以高度影响小麦品种的盐度耐受性。总共,位于染色体1b(1),3a(1),3b(1),4a(1),4d(1),5a(2),5b(3)和7a(1)上的11个基因座在调制盐胁迫下调制小麦品种的农艺性状的重要作用(表gydF4y2BaS6gydF4y2Ba).gydF4y2Ba
已鉴定出相同或相关性状在相似位置的qtl [gydF4y2Ba22gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,证实了本研究中发现的位点的重要性(表gydF4y2BaS7gydF4y2Ba).例如,PH值是小麦形态发生和籽粒产量形成的关键农艺性状[gydF4y2Ba30.gydF4y2Ba].可能影响植物高度的因素包括孟德尔和定量基因,这些基因在所有21染色体上分布[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba].目前已有20多个影响PH的主要基因被鉴定为gydF4y2BaRhtgydF4y2Ba基因(gydF4y2Ba32gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba].著名的gydF4y2Barht4.gydF4y2Ba,gydF4y2Barht8.gydF4y2Ba,gydF4y2Barht5.gydF4y2Ba,gydF4y2Barht-b1gydF4y2Ba,gydF4y2Barhtd1.gydF4y2Ba,gydF4y2Barht9.gydF4y2Ba,gydF4y2Barht12.gydF4y2Ba和gydF4y2Barht13.gydF4y2Ba基因分别位于2BL、2DS、3BS、4BS、4DS、5AL、5AL和7BS染色体上[gydF4y2Ba14gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].在常见的育种实践中,短基因型是根据它们的抗倒伏性选择的。而在本研究中,在LS和HS条件下,PH与YPP呈一致的正相关关系,这证实了不同应激相关环境下的相似结果[gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba].在这里,基因座gydF4y2BaQPh-4DgydF4y2Ba在HS条件下,在16.93-19.29 Mb的位置在4D染色体上被鉴定,位于与gydF4y2Barhtd1.gydF4y2Ba基于与以往研究中检测到的qtl物理位置的比较[gydF4y2Ba27gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba].此外,HL-DH群体的QTL定位结果也验证了该位点的有效性gydF4y2BaQPh-4DgydF4y2Ba位于影响LS条件下PH的QTL区间(AX-89703298-AX-110564616)。gydF4y2Ba3.gydF4y2Baa,b)解释表型变异的25.90%(表gydF4y2Ba3.gydF4y2Ba).正加性效应来自Hanxuan10, AX-89421921中具有有利等位基因GG的品系在DH群体中也表现出较高的PHs,这与GWAS结果一致(表)gydF4y2Ba3.gydF4y2Ba,无花果。gydF4y2Ba3.gydF4y2Bac, d)另一个轨迹,gydF4y2BaQPH-5A1.gydF4y2Ba在LS和HS处理下,定位于5A染色体535.91 ~ 536.80 Mb。Díaz等[gydF4y2Ba44gydF4y2Ba]映射QTL(485.83 MB),影响间隔的pH和SLgydF4y2BaXmwg522gydF4y2Ba- - - - - -gydF4y2BaXCDO412C.gydF4y2Ba通过分析ITMI小麦绘制群体对盐度应力的反应来染色体5A。但这QTL很远gydF4y2BaQPH-5A1.gydF4y2Ba.基于中国春品种(IWGSC V1.1)的基因组序列,在535.91 ~ 536.80 Mb的位置区间发现了9个感兴趣的基因。基因预测和功能注释表明,这些基因与抗病基因和U-box E3泛素连接酶有关。近年来,在小麦中发现了多个具有抗病作用的qtl,研究表明,这些抗病基因不仅对病害的严重程度有影响,而且还影响小麦的生长和产量[gydF4y2Ba45gydF4y2Ba].例如,Zheng等人[gydF4y2Ba46gydF4y2Ba]映射了大效果QTL赋予gydF4y2Ba镰刀gydF4y2Ba冠倍细麦芽腐蚀性,发现QTL对植物高度和标题日期具有显着影响。虽然,报告表明,U-Box E3 Ligases在非生物应激反应中发挥了重要作用。例如,Min等人。[gydF4y2Ba47gydF4y2Ba)建议gydF4y2BaCaPUB1gydF4y2Ba,辣椒U-box E3泛素连接酶可增强转基因水稻的耐寒性。gydF4y2Ba
普通小麦群体和HL-DH群体株高(PH)的QTL定位在4D染色体上。gydF4y2Ba一个gydF4y2Ba染色体的位置gydF4y2BaQPh-4DgydF4y2Ba在191份普通小麦种质中。gydF4y2BabgydF4y2Ba“瀚轩10×Lumai 14”DH群体PH的QTL定位。gydF4y2BacgydF4y2Ba具有优势等位基因的亲本的表型值gydF4y2BaQPh-4DgydF4y2Ba在普通小麦种群中。gydF4y2BadgydF4y2Ba具有优势等位基因的亲本的表型值gydF4y2BaQPh-4DgydF4y2Ba在“汉轩10×Lumai 14”的人口中。**表示在0.01级的显着显着。LS:低盐度处理;HS,高盐度处理gydF4y2Ba
在本研究中,无论是LS还是HS条件下,TKW值都相对稳定。轨迹gydF4y2BaQTkw-4AgydF4y2Ba,在14.68 ~ 16.28 Mb的4A染色体上鉴定到与TKW显著相关的菌株。一个常见的QTL (gydF4y2BaXGWM160gydF4y2Ba)(719.26 MB)在营养 - ,干旱和盐胁迫条件下的96次数的群体中检测到控制产量和TKW [gydF4y2Ba39gydF4y2Ba].古典体重相关基因gydF4y2BaTaCWIgydF4y2Ba已经映射在染色体4a(610.04 mb)和5d(557.95 mb)的小麦[gydF4y2Ba48gydF4y2Ba].这些QTL都与之不同gydF4y2BaQTkw-4AgydF4y2Ba,暗示gydF4y2BaQTkw-4AgydF4y2Ba可以成为TKW的新型QTL。基因预测和功能诠释gydF4y2BaQTkw-4AgydF4y2Ba15个感兴趣的基因位于14.68-16.28 Mb的位置区间。此外,一个候选基因编码甘油-3-磷酸酰基转移酶(gydF4y2BaGPAT.gydF4y2Ba;找到了2.3.1.15)。当gydF4y2BaGPAT.gydF4y2Ba在一些植物中克隆了基因,例如gydF4y2Ba碱蓬莎莎gydF4y2Ba,gydF4y2Ba奥雅萨苜蓿gydF4y2Ba, 番茄,gydF4y2Ba拟南芥gydF4y2Ba, 等等 [gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba].研究表明gydF4y2BaGPAT.gydF4y2Ba基因与胁迫耐受性和种子发育密切相关[gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba].Ariizumi等[gydF4y2Ba52gydF4y2Ba研究发现,当过表达时,不饱和脂肪酸的含量增加gydF4y2Ba阿托帕特gydF4y2Ba在水稻中,在低温条件下可以提高转基因水稻幼苗的光合速率和生长。gydF4y2Ba
SL,一种稳定的表型特征,在两个盐应力环境中显示出与YPP的显着正相关。在这项研究中,基因座gydF4y2BaQSl-1BgydF4y2Ba和gydF4y2BaQSl-3BgydF4y2Ba在1B和3B染色体上分别鉴定出674.96 ~ 675.13 Mb和62.13 ~ 62.14 Mb。利用90k小麦SNP阵列,Gao等[gydF4y2Ba27gydF4y2Ba]也绘制了1B染色体上的SNPs (gydF4y2BaBS00070878_51gydF4y2Ba- - - - - -gydF4y2Bakukri_c1529_462gydF4y2Ba, 22.70 Mb)及3B (gydF4y2Baku_c12191_1202-excalibur_c3556_520gydF4y2Ba利用Zhou 8425B/Chinese Spring RIL群体与SL关联。此外,Mwadzingeni等人[gydF4y2Ba22gydF4y2Ba]与在干旱胁迫条件下使用93个面包小麦基因型的染色体1B上的SL相关的标记。刘等。[gydF4y2Ba53gydF4y2Ba]在染色体2a,2b和6a上的集成SL-相关QTLS在物理图上使用192个常见的小麦样品基于90k SNP芯片构建的物理图。基因座gydF4y2BaQSl-1BgydF4y2Ba和gydF4y2BaQSl-3BgydF4y2Ba本研究检测到的SL不同于以往的研究,可能是新的qtl。基因预测和功能注释表明,6个候选基因和2个候选基因在位置区间gydF4y2BaQSl-1BgydF4y2Ba和gydF4y2BaQSl-3BgydF4y2Ba.这些基因分别与角蛋白相关蛋白质和蓖麻素B样凝集素基因有关。以前的研究表明QTL(gydF4y2BaQPE.gydF4y2Ba1胜9负gydF4y2Ba)对于胰蛋白编码与稻米角蛋白相关蛋白5-4家族同源的蛋白质[gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba].而凝集素基因参与了植物的非生物胁迫反应[gydF4y2Ba57gydF4y2Ba].例如,小麦凝集素基因的表达水平gydF4y2BaHfr-1gydF4y2Ba是在干旱条件下上调的,而其他凝集素基因gydF4y2BaWci-1gydF4y2Ba在各种压力下显著上调[gydF4y2Ba57gydF4y2Ba].gydF4y2Ba
盐胁迫下籽粒产量及相关性状的QTL定位gydF4y2Ba
谷物产量是一种复杂的定量性状,由多种基因控制。仅基于谷物产量的选择很简单,但对小麦育种无效[gydF4y2Ba58gydF4y2Ba].因此,小麦育种者需要鉴定有助于籽粒产量的特征[gydF4y2Ba41gydF4y2Ba].因此,在任何条件下,与籽粒产量相关的性状都会自动被视为小麦育种的选择标准[gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba].在本研究中,BM,SL,GN和Sn显示在LS和HS条件下与YPP相似且非常高的相关性(表gydF4y2Ba2gydF4y2Ba).由Mwadzingeni等人支持[gydF4y2Ba22gydF4y2Ba研究发现,在干旱胁迫条件下,YPP与BM和GN的相关性相似。与盐胁迫相关的籽粒产量和组成性状的QTL聚类和共定位QTL已被报道[gydF4y2Ba26gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba59gydF4y2Ba,gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61gydF4y2Ba].此外,多效qtl控制的农艺性状之间存在异常高的相关性[gydF4y2Ba22gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba,gydF4y2Ba63gydF4y2Ba,gydF4y2Ba64gydF4y2Ba].两次出现可能负责QTL与不同特征相关的QTL的共定位:首先,影响各种特征或相同性状的基因被强烈连接;第二,单一基因可能对相关性状产生一系列影响或影响两个或更多个独立特征[gydF4y2Ba62gydF4y2Ba].在盐胁迫条件下,在5A、5B和7A上检测到影响籽粒产量相关性状的5个基因座(见表)gydF4y2BaS6gydF4y2Ba).gydF4y2Ba
-5族染色体似乎含有与非生物胁迫抗性相关的基因,并且在盐胁迫条件下的籽粒产量和相关性状的几个QTL尤其位于小麦的第5族染色体上[gydF4y2Ba26gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba60gydF4y2Ba,gydF4y2Ba62gydF4y2Ba].例如,基因gydF4y2BaVrn-A1gydF4y2Ba(587.41 MB),gydF4y2BaVRN2-5A / ZCCT1-5AgydF4y2Ba(698.180559 Mb),gydF4y2BaVRN-B1gydF4y2Ba(573.82 Mb)gydF4y2BaVrn-D1gydF4y2Ba(467.18 MB)通过控制冬季出苗的春化和耳朵血液化和耳朵时序的影响,位于染色体5A,5B和5D的长臂上[gydF4y2Ba33gydF4y2Ba,gydF4y2Ba65gydF4y2Ba,gydF4y2Ba66gydF4y2Ba].在目前的研究中,基因座gydF4y2BaQ-5A2gydF4y2Ba在HS条件下,对YPP和BM有影响,位于染色体5A的692.16 ~ 692.39 Mb。5B染色体上的三个位点,gydF4y2BaQ-5B1gydF4y2Ba(574.06 - -578.35 Mb),gydF4y2BaQSl-5B2gydF4y2Ba(583.48 - -583.89 Mb)gydF4y2BaQ-5B3gydF4y2Ba(690.08-690.70 MB),含有与YPP,SL,SN,GN和BM相关的SNP簇,在LS条件下。IWGSC数据库的BLAST搜索显示轨迹gydF4y2BaQ-5A2gydF4y2Ba影响ypp和bm接近gydF4y2BaVRN2-5A / ZCCT1-5AgydF4y2Ba(698.180559 MB)[gydF4y2Ba66gydF4y2Ba],也是轨迹gydF4y2BaQ-5B1gydF4y2Ba与YPP、SL、GN和BM的联系似乎位于附近gydF4y2BaVRN-B1gydF4y2Ba[gydF4y2Ba67gydF4y2Ba].更重要的是,ZX-RIL种群的QTL映射结果验证了该基因座gydF4y2BaQ-5B1gydF4y2Ba在HS条件下,每穗小穗数的QTL区间为AX-109996563-AX-111538681(表gydF4y2Ba3.gydF4y2Ba,无花果。gydF4y2BaS3gydF4y2Ba).另外,轨迹gydF4y2BaQ-5B3gydF4y2Ba接近QTL (gydF4y2BaQKNS.caas-5BLgydF4y2Ba, 695.99 Mb)由Li等人确定的每穗内核数[gydF4y2Ba68gydF4y2Ba].结果表明,这里鉴定的共同定位的QTL应在胁迫条件下生长的小麦品种的产量及其相关性状中起重要作用。gydF4y2Ba
基因预测和功能诠释gydF4y2BaQ-5A2gydF4y2Ba,gydF4y2BaQ-5B1gydF4y2Ba,gydF4y2BaQSl-5B2gydF4y2Ba和gydF4y2BaQ-5B3gydF4y2Ba表明,分别存在于每个基因座的位置间隔中包含的五个,45,19和12个候选基因。这些基因与脂质转移蛋白,植物精切蛋白合酶,丝氨酸/苏氨酸蛋白激酶和Mada-Box基因有关。脂质转移蛋白(LTPS)是一类小而无处不在的蛋白质,被认为在植物反应中具有关键作用[gydF4y2Ba69gydF4y2Ba,gydF4y2Ba70gydF4y2Ba].Kirubakaran等人[gydF4y2Ba70gydF4y2Ba)建议gydF4y2BaLTP.gydF4y2Ba小麦3F1基因可增强烟草对真菌的抗性。植物螯合素合成酶(Phytochelatin synthases, PCs)是一类富含半胱氨酸的硫醇反应性和重金属结合肽,参与植物的重金属积累和解毒[gydF4y2Ba71gydF4y2Ba].研究表明小麦的异性表达gydF4y2BaTAPCS1gydF4y2Ba增强了大米射击中的CD积累[gydF4y2Ba71gydF4y2Ba].丝氨酸/苏氨酸蛋白激酶(Serine/苏氨酸蛋白激酶,STK)是一个大型蛋白家族,在植物的胁迫信号传导和响应中起重要作用[gydF4y2Ba72gydF4y2Ba].Ge等[gydF4y2Ba73gydF4y2Ba[证明了过多的表达gydF4y2Ba品尝gydF4y2Ba基因能够增加根系生长gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba盐胁迫下。此外,MADS-box家族成员编码的转录因子不仅参与花器官的规范,还参与植物的生长发育[gydF4y2Ba74gydF4y2Ba].Ma et al. [gydF4y2Ba75gydF4y2Ba]对小麦中180个MADS-box基因进行了分析,发现它们参与了非生物和生物胁迫反应。gydF4y2Ba
此外,影响YPP的位点位于7A染色体(gydF4y2BaQ-7AgydF4y2Ba(22.75 ~ 22.86 Mb),在盐胁迫条件下也包含一个与SL相关的SNP簇。在7A染色体上,Gao等[gydF4y2Ba27gydF4y2Ba]识别QTL(gydF4y2Bawsnp_Ex_c200_391493gydF4y2Ba- - - - - -gydF4y2BaEx_c52798_415gydF4y2BaTKW,SN,SL和pH的244.27 MB)。此外,QTL映射导致ZX-RIL群体验证了该基因座gydF4y2BaQ-7AgydF4y2Ba可以形成LD块(475 kB),其QTL间隔(AX-108843150-AX-109347413)在HS处理下影响SL(图。gydF4y2Ba4gydF4y2Baa - c)。正加性效应来自于小燕60号,且具有良好等位基因的品种在关联群体中也表现出较长的SLs,这与GWAS结果一致(见表)gydF4y2Ba3.gydF4y2Ba,无花果。gydF4y2Ba5gydF4y2Baa、b)。gydF4y2Ba
小麦耐盐育种的有利单倍型gydF4y2Ba
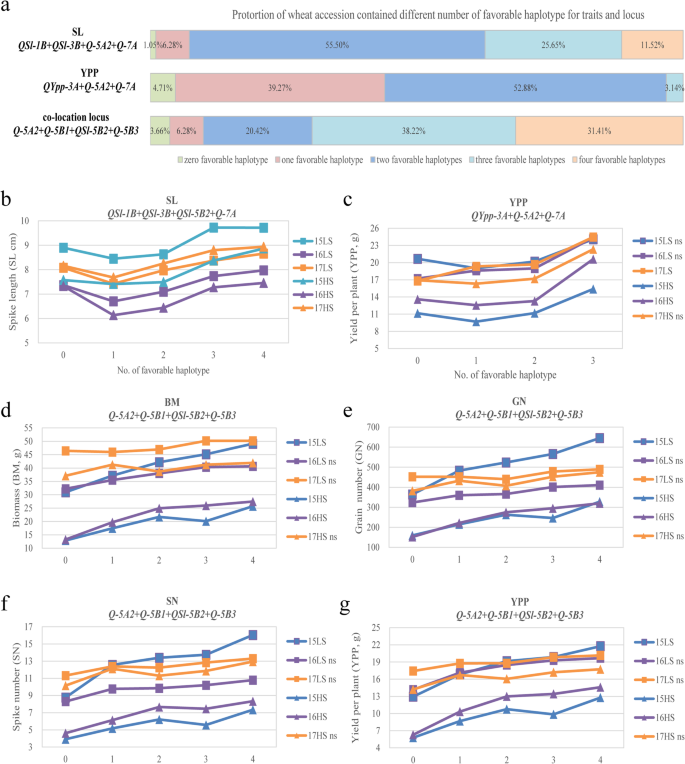
使用基本类型的SNP标记和与特征相关的等位基因分布,鉴定了一些单倍型,通过使用它们使用检测其表型值来确定有利的单倍型gydF4y2BatgydF4y2Ba- 最低。除去少于5%的单倍型后,每个基因座的主要单倍型用于进一步分析。当gydF4y2BatgydF4y2Ba- 最终表明,具有良好的单倍型的品种通常在盐胁迫条件下具有更理想的表型(表gydF4y2Ba4gydF4y2Ba,无花果。gydF4y2BaS5gydF4y2Ba).此外,每个位点的有利单倍型所占比例不同,说明这些重要位点在小麦育种过程中经历了不同程度的选择。例如,位点有利单倍型的比例gydF4y2BaQYPP-3AgydF4y2Ba和gydF4y2BaQTkw-4AgydF4y2Ba分别为5.24和8.90%,说明这些位点没有经历强选择。gydF4y2Ba
进一步的基因座效应比较分析表明,具有3个或3个以上有利基因座单倍型的品种gydF4y2BaQSl-1BgydF4y2Ba,gydF4y2BaQSl-3BgydF4y2Ba,gydF4y2BaQSl-5B2gydF4y2Ba和gydF4y2BaQ-7AgydF4y2Ba在LS和HS条件下,有明显的更长时间的SLs(图。gydF4y2Ba6gydF4y2Ba).在本研究中使用的群体中,如地方品种福麦和改良品种汉4589,约有37.17%的群体携带3个或3个以上的SL优良单倍型,这表明这些位点在小麦育种过程中已经聚集。gydF4y2Ba6gydF4y2Ba).在基因座中具有三个有利的单倍型品种gydF4y2BaQYPP-3AgydF4y2Ba,gydF4y2BaQ-5A2gydF4y2Ba, 和gydF4y2BaQ-7AgydF4y2Ba在盐胁迫条件下产生更大的产率(图。gydF4y2Ba6gydF4y2Ba).然而,只有3.14%的群体,如一些著名的耐盐品种仓6001和仓6005,在产量上包含3个单倍型,表明这些优良的单倍型在耐盐小麦育种中没有得到充分利用(图6)。gydF4y2Ba6gydF4y2Ba).为5组染色体的共定位位点,即gydF4y2BaQ-5A2gydF4y2Ba,gydF4y2BaQ-5B1gydF4y2Ba,gydF4y2BaQSl-5B2gydF4y2Ba和gydF4y2BaQ-5B3gydF4y2Ba在LS和HS条件下,具有4个有利单倍型的品种的SN、GN、BM和产量值最高(图2)。gydF4y2Ba6gydF4y2Ba).除仓6002、仓6005、德康961、科艺26、科农199等知名耐盐品种外,小燕6号、小燕926、小燕54、小燕101等小燕衍生品种均具有4个有利的单倍型。约31.41%的品种在5组染色体共定位位点中含有4个优势单倍型(图5)。gydF4y2Ba6gydF4y2Ba)表示,这些基因座在小麦育种期间已集成,并具有增加小麦耐油耐受性的潜力。一般而言,人口中有利单倍型分布的巨大差异表明,不同品种的各种耐盐性耐盐基础,以及具有优质单倍型的含量,如品种苍纲,沧6002,Cang 6005,Jimai 32和一些小燕衍生物,可以在小麦盐耐受育种中发挥关键作用。gydF4y2Ba
小麦品种的遗传比例和表型值包含不同数量的性状和位点的有利单倍型。gydF4y2Ba一个gydF4y2Ba小麦探剂的比例含有不同数量的有利的单倍型,用于穗长度(S1),每曲线(YPP)的产率和群体-5染色体上的共定位基因座。百分比代表小麦种的比例。gydF4y2BabgydF4y2Ba小麦种植的尖峰长度比较含有与SL相关的不同数量的基因座的合适单倍型。gydF4y2BacgydF4y2Ba小麦种的产量比较含有与ypp相关的不同数量的基因座的有利单倍型。gydF4y2BabgydF4y2Ba-gydF4y2BaggydF4y2Ba小麦5组染色体上共定位位点的有利单倍型数量不同。LS:低盐度处理;HS,高盐度处理。Ns表示无显著差异gydF4y2Ba
用于标记辅助选择的分子标记的开发gydF4y2Ba
一些研究强调,小麦的耐盐与排除NA的能力密切相关gydF4y2Ba+gydF4y2Ba从叶子[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba59gydF4y2Ba,gydF4y2Ba63gydF4y2Ba].Genc等[gydF4y2Ba59gydF4y2Ba]鉴定出5个与Na相关的qtlgydF4y2Ba+gydF4y2Ba在水培育苗试验中排除。这两个NagydF4y2Ba+gydF4y2Ba排除基因,gydF4y2Ba纳克隆gydF4y2Ba和gydF4y2BaNAX2.gydF4y2Ba,也已在杜伦姆小麦中发现[gydF4y2Ba76gydF4y2Ba,gydF4y2Ba77gydF4y2Ba].然而,本研究鉴定的主要qtl均未与定位基因或Na的qtl共定位gydF4y2Ba+gydF4y2Ba排除。报告表明na的qtlsgydF4y2Ba+gydF4y2Ba在水培中鉴定的排除对小麦产量进行了Dinky影响[gydF4y2Ba63gydF4y2Ba,gydF4y2Ba78gydF4y2Ba]与耐盐相关的QTL通常根据群体和环境而变化[gydF4y2Ba63gydF4y2Ba].实际上,与盐胁迫下产量及相关性状相关的qtl在小麦育种实践中发挥着更重要的作用。尽管,随着下一代测序的可用性,最近在小麦中发现了大量的SNPs [gydF4y2Ba79gydF4y2Ba].不同类型的高吞吐量SNP阵列已成功地用于研究小麦种群的标记特征联系[gydF4y2Ba79gydF4y2Ba].然而,由于对大量后代进行基因分型所需的时间和劳力,这些标记的验证和利用仍然是主要的挑战[gydF4y2Ba79gydF4y2Ba,gydF4y2Ba80gydF4y2Ba].最近,LGC有限公司开发了KASP基因分型技术[gydF4y2Ba81gydF4y2Ba进行SNP基因分型。然而,针对小麦重要农艺性状开发的KASP标记很少[gydF4y2Ba14gydF4y2Ba].在这项研究中,成功开发了14个KASP标记,用于九个基因座与产量和相关性状相关联。这些Kasp标记可以准确地对自然群体中的两种等位基因进行准确地分类(图。gydF4y2BaS6gydF4y2Ba).与BM、GN、SL和YPP相关的KASP标记来自两个多效位点gydF4y2BaQ-5A2gydF4y2Ba,影响HS条件下的BM和YPP,以及gydF4y2BaQ-7AgydF4y2Ba,影响LS条件下SL和YPP。这些KASP标记对筛选耐盐小麦种质资源和提高小麦耐盐育种的选择效率具有重要意义。gydF4y2Ba
结论gydF4y2Ba
本研究利用Wheat660K SNP序列对6个环境下的191份小麦材料进行了产量及相关性状评价。GWAS分析表明,在2种不同盐胁迫条件下,小麦8个性状共存在389个显著相关snp位点,其中11个位点在盐胁迫下对小麦株高、产量及相关性状具有关键作用。在5A染色体上的692.16 ~ 692.39 Mb和5B染色体上的574.06 ~ 578.35 Mb、580.03 ~ 584.89 Mb和690.08 ~ 690.70 Mb的位点上鉴定出4个有利等位基因频率较高的位点,分别为YPP、SN、SL、GN和BM。在9个与产量及相关性状密切相关的基因座中,成功开发了14个KASP标记,其中3个标记在韩选10和鲁麦14的双亲本群体中进行了验证。这些结果为今后利用标记辅助选择改良小麦耐盐性提供了有益的信息。gydF4y2Ba
方法gydF4y2Ba
植物材料gydF4y2Ba
植物材料收集了191份小麦材料,包括5个引种品种、10个地方品种和176个改良品种(见表)gydF4y2BaS1gydF4y2Ba).除了一个未知来源的一种改进的品种外,这些基因型起源于智利(1),意大利(2),罗马尼亚(1),苏联(1)和中国省,包括安徽(1),北京(17),甘肃(3),河北(40),河南(30),内蒙古(1),吉林(1),青海(6),陕西(39),山东(34),山西(8),四川(3),天津(1)和西藏(1)。每个加入的详细信息在表格中给出gydF4y2BaS1gydF4y2Ba.样品经中国科学院遗传与发育生物学研究所批准。植物材料均由中国科学院遗传与发育生物学研究所提供。gydF4y2Ba
GWAS检测到的显著的SNPs通过中脉175/小燕60和350 F的RIL群体进行验证gydF4y2Ba7gydF4y2Ba线(ZX-RIL)和双单倍体群汉轩10 / LUMAI 14(HL-DH),150条线。gydF4y2Ba
现场实验和表型gydF4y2Ba
在2014-2015,2015-2015,2015-2016和2015-2015和2015-2015和2015-2016的南浦生态农业实验站2016-2017种植季节。该站位于温暖的温带半湿润季风气候区,平均每年温度12.3°C和平均年降水量480毫米。南浦生态农业实验站的正常土壤在渤海地区周围干旱和盐压力典型,代表LS治疗。数十年来,根据检测值,Nanpi中正常土壤的盐度为0.18%(m / m)。HS治疗被设定为人工制作的盐池。通过将具有特定浓度的海盐溶液添加到正常土壤中,以0.3%(m / m)保证盐池的土壤盐度。土壤的总盐含量通过氯化物 - 硝酸银滴定法测定。因此,设定了两种处理,LS(0.18%,m / m)和Hs(0.3%,m / m)。完全,六个盐度环境被指定为15LS,15HS,16LS,16HS,17LS和17HS。 Each salt pool was a cement pond of 6.6 m × 2 m × 1.5 m, with waterproof cement thickness of 10 cm at the bottom. The bottom of the cement pond was paved with stones and river sand, and covered with non-woven fabrics. The soil evenly mixed with sea-salt solution was filled in the whole cement pond. The salt concentration of salt pool was checked from time to time, with the assurance that the salinity was maintained at 0.3% (m/m). The LS and HS treatments did not involve artificial irrigation during the cropping seasons. The field experiments were performed using a completely randomized design with five replications. Each block contained sixteen 200-cm-long rows with 10 cm between neighboring plants and 23 cm between neighboring rows. Hole sowing method was used and three sprouted seeds of any accession were sown in each plot. The three plants in one hole were harvest together and eight phenotypic traits at adult stage were investigated, including plant height (PH), spike number (SN), spike length (SL), kernels per spike (KPS), grain number (GN), thousand kernel weight (TKW), yield per plot (YPP) and biological mass (BM). Measurement methods for each trait were as follows: PH was the distance between the stem base and the top of the main spike excluding awns, SN was the number of spikes per plot, SL was the length of the main spike per plot, KPS was the kernel number of main spike per plot, GN was the number of grains per plot, TKW was the calculated by (YPP/GN) × 1000, YPP was the weight of grains per plot, BM was the aboveground weight per plot. The relative value of high salinity/low salinity (HS/LS) for each trait was also calculated and used for genome-wide association analysis.
ZX-RIL和HL-DH的表型用于验证GWA检测到的显着的SNP。在2016-2017(ZX-RIL)和2017-2018(ZX-RIL和HL-DH)下,在LS和HS条件下种植和收获这些两种群体。为低盐度处理设定了三个重复的行和十个复制的个体。种子播种用行空间20厘米,植物空间10厘米。如上所述研究了表型性状。gydF4y2Ba
基因分型gydF4y2Ba
利用CTAB法从191份供试材料的新鲜叶片中提取基因组DNA [gydF4y2Ba82gydF4y2Ba]。然后将DNA样本送往北京首都生物科技有限公司基因分型中心,使用Axiom Wheat660K基因分型芯片(gydF4y2Bahttps://wheat.pw.usda.gov/gnpages/topics/wheat660_snp_array_developed_by_caas.pdf.gydF4y2Ba),根据制造商的说明(Axiom 2.0分析手册工作流用户指南Rev3)。SNP呼叫率小于80%和次要等位基因频率小于0.05的标记被删除。使用PLINK软件完成基因分型数据的高质量预处理(gydF4y2Bahttp://pngu.mgh.harvard.edu/purcell/plink/gydF4y2Ba) [gydF4y2Ba83gydF4y2Ba].gydF4y2Ba
统计分析gydF4y2Ba
表型gydF4y2Ba
表型数据用SAS v.9.2软件(SAS Institute Inc., Cary, NC)进行分析。对所有性状进行方差分析、相关系数和重复性分析。遗传力估算采用以下公式计算:gydF4y2BaHgydF4y2Ba2gydF4y2Ba=σgydF4y2Ba2gydF4y2BaggydF4y2Ba/(σgydF4y2Ba2gydF4y2BaggydF4y2Ba + σ2gydF4y2BaGEgydF4y2Ba/gydF4y2BargydF4y2Ba + σ2gydF4y2Baε.gydF4y2Ba/gydF4y2Ba再保险gydF4y2Ba),其中σgydF4y2Ba2gydF4y2BaggydF4y2Ba,σ.gydF4y2Ba2gydF4y2BaGEgydF4y2Ba和σ.gydF4y2Ba2gydF4y2Baε.gydF4y2Ba分别是基因型,基因型×环境相互作用和残余误差差异的估计,E和R分别代表了环境的数量和重复。gydF4y2Ba
种群结构及亲属关系分析gydF4y2Ba
使用Tassel 5.0研究标记中标记中的链接不平衡(LD),其中标记是已知的标记[gydF4y2Ba84gydF4y2Ba].使用PLINK的所有载体共享的SNP建立距离矩阵[gydF4y2Ba83gydF4y2Ba].基于该距离矩阵,使用Phylip软件构建邻居连接树。建立亲属矩阵(K模型),以使用Tassel 5.0推断加入之间的关系[gydF4y2Ba84gydF4y2Ba].使用贝叶斯聚类程序structure V2.3.4计算种群结构(Q矩阵)[gydF4y2Ba85gydF4y2Ba].结构程序为每个亚潜水费(k)值的10倍,从1到15之间,烧伤时烧伤时间为100,000次迭代,并在[中的燃烧后的Markov Chain Monte Carlo(MCMC)的运行时间为500,000次运行gydF4y2Ba85gydF4y2Ba].亚种群数量用theΔK方法估计[gydF4y2Ba86gydF4y2Ba].同时,利用TASSEL 5.0进行主成分分析,推断种群分层[gydF4y2Ba84gydF4y2Ba].gydF4y2Ba
基因组关联分析gydF4y2Ba
采用TASSEL 5.0混合线性模型(MLM)检测snp与表型变异之间的关联[gydF4y2Ba84gydF4y2Ba].采用种群结构(Q矩阵)和亲属关系(K模型)两个因素作为混杂因素[gydF4y2Ba87gydF4y2Ba,gydF4y2Ba88gydF4y2Ba].单核苷酸多态性的前5个主成分被引入GWAS模型。资讯科技分类服务[gydF4y2Ba89gydF4y2Ba,以验证TASSEL结果的一致性和准确性。gydF4y2Ba
开发适用于关联SNPS的Kasp标记gydF4y2Ba
开发了竞争性等位基因特异性聚合酶链反应(KASP) (LGC Genomics LLC, Beverly, MA, USA)标记,用于检测和区分重要位点的关键snp的等位基因。除ph位点外,每个位点选取1 ~ 2个具有代表性的SNPs,共将9个位点的14个SNPs转化为KASP标记。利用SNP侧的Wheat660K序列,利用primer Premier 5软件设计KASP引物。由首都生物基因分型机构(中国北京)按照KASP手册进行KASP反应和荧光信号检测。gydF4y2Ba
可用性数据和材料gydF4y2Ba
支持本研究结果的数据集包括在文章及其附加文件中。本研究报道的实验数据已保存在生物信息和DDBJ中心的基因组表达档案(GEA)中,登录号为E-GEAD-406gydF4y2Bahttps://ddbj.nig.ac.jp/gea/reviewer.gydF4y2Ba,接入密钥为“bDhkCrXEb17jzYrxoMpt”。基因型数据已保存在中国科学院北京基因组研究所大数据中心基因组变异图(GVM)中,登录号为GVM000089 atgydF4y2Bahttp://bigd.big.ac.cn/gvm/getProjectDetail?project=GVM000089gydF4y2Ba.gydF4y2Ba
缩写gydF4y2Ba
- BM:gydF4y2Ba
-
生物质量gydF4y2Ba
- GN:gydF4y2Ba
-
粒数gydF4y2Ba
- GWAS:gydF4y2Ba
-
全基因组关联研究gydF4y2Ba
- KPS:gydF4y2Ba
-
内核穗gydF4y2Ba
- PH值:gydF4y2Ba
-
植物高度gydF4y2Ba
- QTL:gydF4y2Ba
-
数量性状位点gydF4y2Ba
- SN:gydF4y2Ba
-
钉号码gydF4y2Ba
- SNP:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
- SL:gydF4y2Ba
-
穗长gydF4y2Ba
- TKW:gydF4y2Ba
-
千粒重量gydF4y2Ba
- YPP:gydF4y2Ba
-
每个情节的产量gydF4y2Ba
参考gydF4y2Ba
- 1.gydF4y2Ba
Munns R,Tester M.盐度耐受机制。Annu Rev植物Biol。2008; 59:651-81。gydF4y2Ba
- 2.gydF4y2Ba
王M,夏G.小麦耐盐性分子机制的景观。作物J. 2018; 6(1):42-7。gydF4y2Ba
- 3.gydF4y2Ba
王德,张克,董立,东Z,李义,侯赛因A,翟海。中国小麦碾磨与终点利用的分子遗传和基因组分析:进展与观点。作物J. 2018; 6(1):68-81。gydF4y2Ba
- 4.gydF4y2Ba
开发耐盐作物:挑战与机遇。植物科学进展。2005;10(12):615-20。gydF4y2Ba
- 5.gydF4y2Ba
贾米,关吉,翟Z,耿思,张X,毛升,李A。小麦功能基因组学中的下一代测序时代:更新。作物J. 2018; 6(1):7-14。gydF4y2Ba
- 6.gydF4y2Ba
徐勇,何志强,何志强,何志强。作物基因分型技术研究进展与展望。摩尔。2017;10(8):1047 - 64。gydF4y2Ba
- 7.gydF4y2Ba
Abberton M, Batley J, Bentley A, Bryant J, Cai H, Cockram J, de Oliveira AC, Cseke LJ, Dempewolf H, de Pace C, et al.;气候变化期间的全球农业集约化:基因组学的作用。植物生态学报;2016;14(4):1095-8。gydF4y2Ba
- 8.gydF4y2Ba
王志强,王志强,王志强,等。提高小麦和其他谷物耐盐性的途径。昆虫学报。2006;57(5):1025-43。gydF4y2Ba
- 9.gydF4y2Ba
作物耐盐性的成本是多少?新植醇。2015;208(3):668 - 73。gydF4y2Ba
- 10。gydF4y2Ba
从基因组到基因:小麦研究的新时代?植物科学进展。2015;20(6):380-7。gydF4y2Ba
- 11.gydF4y2Ba
崔飞,张宁,范小龙,张伟,赵超,杨丽娟,潘荣强,陈敏,韩杰,赵新强,等。利用小麦660k SNP阵列衍生的高密度遗传图谱进行籽粒数主要QTL的高分辨率定位Sci众议员2017;7(1):3788。gydF4y2Ba
- 12.gydF4y2Ba
国际小麦基因组测序联盟(IWGSC)。使用完全注释的参考基因组转移小麦研究和育种的限制。科学。2018; 361:661。gydF4y2Ba
- 13.gydF4y2Ba
吴Qh,陈yx,周sh,fu l,陈俊,肖y,张d,欧阳sh,zhao xj,cui y等。高密度遗传联系地图结构和QTL纹理纹理和大小在小麦种群中的尺寸Yanda1817 x beinong6。Plos一个。2015; 10(2):E0118144。gydF4y2Ba
- 14.gydF4y2Ba
MA F,XU Y,MA Z,Li L,D. Geanome-ide-Gensi-Genfienci on Peaky相关性状的关键基因座的验证父母父母小燕6.莫尔品种。2018; 38(7)。gydF4y2Ba
- 15.gydF4y2Ba
Winfield Mo,Allen Am,Burridge Aj,Barker Gl,Benbow Hr,Wilkinson Pa,Coghill J,瀑布C,Davassi A,Scopes G,等。用于六倍体小麦的高密度SNP基因分型阵列及其二级和三级基因库。植物Biotechnol J. 2016; 14(5):1195-206。gydF4y2Ba
- 16.gydF4y2Ba
李H,王Q,徐L,Mu J,吴j,曾q,yu s,huang l,韩d,kang z.快速鉴定Qtl赋予成人植物抗性的主要效果QTIPE在小麦品种中的条纹锈病“。Euphytica。2017; 213(6)。gydF4y2Ba
- 17.gydF4y2Ba
田旭,文伟,谢磊,付磊,徐东,付超,王东,陈旭,夏旭,陈强,等。矮株高基因的分子定位gydF4y2Barht24.gydF4y2Ba在面包小麦。植物学报2017;8:1379。gydF4y2Ba
- 18.gydF4y2Ba
朱超,于军。植物关联图谱的研究现状与展望。植物基因组。2008;1(1):5 - 20。gydF4y2Ba
- 19.gydF4y2Ba
王R,陈继,安德森·贾,张继,赵W,惠勒J,Klassen N,See Dr,Dong Y. Genome-Wield抗性在太平洋西北和CIMMYT开发的春小麦抗性抗性。植物病理学。2017; 107(12):1486-95。gydF4y2Ba
- 20.gydF4y2Ba
OYIGA BC,Sharma RC,Baum M,Ogbonnaya Fc,LéonJ,Ballvora A.在小麦盐胁迫耐受性耐盐耐受性的定量特质位置检测到等位基因变化和差异表达。植物细胞环境。2018; 41(5):919-35。gydF4y2Ba
- 21.gydF4y2Ba
Riaz A, athiyanannan N, peryannan SK, Afanasenko O, Mitrofanova OP, Platz GJ, Aitken EAB, Snowdon RJ, Lagudah ES, Hickey LT, et al.;在瓦维洛夫小麦收集中解锁抗叶锈病的新等位基因。中国科学(d辑:地球科学)2018;gydF4y2Ba
- 22.gydF4y2Ba
Mwadzingeni L,Shimelis H,Rees DJ,Tsilo TJ。干旱胁迫下的小麦农艺性状的基因组关联分析。Plos一个。2017; 12(2):E0171692。gydF4y2Ba
- 23。gydF4y2Ba
关键词:小麦,苗期,耐盐性,微量元素,qtl定位Sci众议员2017;7(1):15662。gydF4y2Ba
- 24。gydF4y2Ba
Nei MCR。固定指数和基因多样性的估计。[j] .环境科学学报,1983;47(3):253-9 .]gydF4y2Ba
- 25。gydF4y2Ba
小麦生理、生化、表观遗传和分子生物学分析gydF4y2Ba小麦gydF4y2Ba)基因型与耐盐性差异较大。植物科学学报,2017;8:1151。gydF4y2Ba
- 26。gydF4y2Ba
Azadi A,Mardi M,Hervan Em,Mohammadi Sa,Moradi F,Tabatabaee Mt,Piresyedi Sm,Ebrahimi M,Fayaz F,Kazemi M等。在面包小麦的正常和盐胁迫条件下产量和产量组分的QTL映射(gydF4y2Ba小麦gydF4y2Bal .)。植物学报。2014;33(1):102-20。gydF4y2Ba
- 27。gydF4y2Ba
高峰,文文文,刘建军,刘建军,尹光国,夏旭,吴晓霞,何志强。小麦杂交周8425B/中国春产量构成、株高和产量相关生理性状QTL的全基因组连锁定位。植物学报。2015;6:1099。gydF4y2Ba
- 28。gydF4y2Ba
Sukumaran S, Dreisigacker S, Lopes M, Chavez P, Reynolds MP。温带灌溉条件下优质春小麦群体籽粒产量及相关性状的全基因组关联研究。中国科学:地球科学。2015;128(2):353-63。gydF4y2Ba
- 29。gydF4y2Ba
Chen H,Semagn K,IQBAL M,Moakhar NP,Haile T,N'Diaye A,Yang R-C,Hucl P,Pozniak C,扳手D.基因组区域与加拿大西春小麦表型特征相关的基因组区域。mol品种。2017; 37(11)。gydF4y2Ba
- 30.gydF4y2Ba
崔飞,李军,丁安,赵超,王丽,王昕,李松,包勇,李旭,冯东,等。小麦穗长和节间株高的QTL定位acta photonica sinica . 2011;122(8): 1517-36。gydF4y2Ba
- 31。gydF4y2Ba
刘勇,林勇,高胜,李志,马军,邓明,陈刚,魏勇,郑勇。中国小麦地方品种23个农艺性状的全基因组关联研究。工厂j . 2017, 91(5): 861 - 73。gydF4y2Ba
- 32。gydF4y2Ba
张n,风扇x,崔f,zhao,张w,zhao x,杨l,pan r,chen m,han j等。小麦的时间和空间表达的特征(gydF4y2Ba小麦gydF4y2BaQTL水平上的株高及其对产量相关性状的影响应用电子学报。2017;130(6):1235-52。gydF4y2Ba
- 33。gydF4y2Ba
5号染色体和5号染色体对小麦早熟时间的遗传控制。遗传。1975;36:49-58。gydF4y2Ba
- 34。gydF4y2Ba
陈胜,高锐,王浩,文敏,肖军,边宁,张锐,胡伟,程胜,别涛,等。一种新的减高基因的特性(gydF4y2Barht23.gydF4y2Ba)调控面包小麦穗部形态和植株构型。Euphytica。2014;203(3):583 - 94。gydF4y2Ba
- 35。gydF4y2Ba
鲁y,xing l,兴,胡p,cui c,张m,xiao j,王h,张r,王x等。表征推定的新半导体降低高度基因,gydF4y2BaRht_NM9gydF4y2Ba,以小麦(gydF4y2Ba小麦gydF4y2Bal .)。中国基因工程学报。2015;42(12):685-98。gydF4y2Ba
- 36.gydF4y2Ba
Peng Zs,Li X,Yang ZJ,Liao ML。四倍体半矮小小麦兰德·达凡凡省的新减少高度基因。genet mol res。2011; 10(4):2349-57。gydF4y2Ba
- 37.gydF4y2Ba
Ellis H,Spielmeyer W,Gale R,Rebetzke J,Richards A.“完美”标记gydF4y2BaRht-B1bgydF4y2Ba和gydF4y2Barht-d1b.gydF4y2Ba小麦的矮化基因。Al Appl Genet。2002; 105(6-7):1038-42。gydF4y2Ba
- 38.gydF4y2Ba
Li XPLS,Liu YP,Gale Md,Worland TJ。不同的影响gydF4y2BaRht-B1bgydF4y2Ba,gydF4y2Barht-d1b.gydF4y2Ba和gydF4y2BaRht-B1cgydF4y2Ba小麦农艺性状的矮化基因研究。谷物与社会。2006;34:919-24。gydF4y2Ba
- 39.gydF4y2Ba
Quarrie SA,斯蒂艾A,Calestani C,Semikhodskii A,Lebreton C,Chinoy C,Steele N,Pljevljakusic D,Waterman E,Weyen J等人。六倍体小麦的高密度遗传图(gydF4y2Ba小麦gydF4y2Ba从中国春x SQ1杂交获得的qtl及其在一系列环境下对粮食产量的qtl进行比较。应用计算机学报。2005;110(5):865-80。gydF4y2Ba
- 40.gydF4y2Ba
伊朗冷旱地条件下籽粒产量与一些农艺人物的关系。Pak J Biol Sci。2005; 8:959-62。gydF4y2Ba
- 41.gydF4y2Ba
Zarei Lckfe。下雨条件下杜伦小麦籽粒产量和一些农艺人物的评价。Aust J Croc SCI。2013; 7(5):609。gydF4y2Ba
- 42.gydF4y2Ba
王Z,吴X,任q,chang x,li r,jing r. qtl映射植物高度在小麦的发展行为(gydF4y2Ba小麦gydF4y2Bal .)。Euphytica。2010;174(3):447 - 58。gydF4y2Ba
- 43.gydF4y2Ba
吴X,王Z,常X,景族植物在不同水域下小麦植物高度发育行为的遗传解剖。J Exp Bot。2010; 61(11):2923-37。gydF4y2Ba
- 44.gydF4y2Ba
DíazdeLeónJL,Escoppinichi R,Geraldo N,Castellanos T,Mujeeb-Kazi A,RöderMs。与矿物成长面包小麦盐度耐受相关的定量特质基因座。Euphytica。2011; 181(3):371-83。gydF4y2Ba
- 45.gydF4y2Ba
郭金龙,翟世宁,李华生,刘爱芬,程德荣,韩锐,刘建军,孔仁仁,郑振华,宋建民。小麦抗白粉病基因和qtl的分子和物理定位研究进展农业科技。2017;18(6):965-70。gydF4y2Ba
- 46.gydF4y2Ba
郑志明,Stiller J,赵强,冯强,Choulet F, Feuillet C,郑亚利,魏玉梅,韩波,严国杰,Manners JM,刘chchj。六倍体小麦3B染色体长臂抗镰孢病QTL的精细定位BMC基因组学。2015;16:850。gydF4y2Ba
- 47。gydF4y2Ba
关键词:辣椒,U-box E3泛素连接酶,CaPUB1,耐寒性,干旱胁迫gydF4y2Ba奥雅萨苜蓿gydF4y2Bal .)。摩尔细胞。2016;(3):250 - 7。gydF4y2Ba
- 48。gydF4y2Ba
姜勇,姜强,郝超,侯军,王磊,张辉,张胜,陈旭,张晓霞。一种产量相关基因gydF4y2BaTaCWIgydF4y2Ba在小麦中:单倍型分析揭示了全球育种的功能,选择和演化。Al Appl Genet。2015; 128(1):131-43。gydF4y2Ba
- 49。gydF4y2Ba
Sui NTS,Wang W,Wang M,风扇H.来自Suaeda Salsa的甘油-3-磷酸酰酰转移酶的过度表达改善了耐盐性gydF4y2Ba拟南芥gydF4y2Ba.前植物SCI。2017; 8:1337。gydF4y2Ba
- 50。gydF4y2Ba
刘米,亨利·杰斯。高效平面杂交通过气相沉积钙钛矿太阳能电池。自然。2013; 501:395-8。gydF4y2Ba
- 51.gydF4y2Ba
Payá-Milans MA-MJ, Balbuena TS, Haslam RP, Gidda SK, Pérez-Hormaeche。向日葵gydF4y2Bahagpat9-1gydF4y2Ba在种子发育过程中是主要的GPAT。植物SCI。2016; 253:42-52。gydF4y2Ba
- 52.gydF4y2Ba
转基因水稻叶片磷脂酰甘油中脂肪酸不饱和的增加提高了光合速率和低温下的生长。植物生理学报。2003;43:751-8。gydF4y2Ba
- 53.gydF4y2Ba
刘建军,徐铮,范旭,周强,曹军,王峰,纪光,杨磊,冯波,王涛。中国小麦穗部相关性状的全基因组关联研究。植物学报。2018;9:1584。gydF4y2Ba
- 54。gydF4y2Ba
孙丕伟,王玉华,何强,舒芳,刘辉,王健,王建民,袁丽萍,邓海峰。gydF4y2BaOsGRF4gydF4y2Ba控制水稻中的晶粒形状,穗长和种子。J Intent Plant Biol。2016; 58(10):836-47。gydF4y2Ba
- 55。gydF4y2Ba
黄Xzqq,刘泽,孙海,河流,罗德,夏通用汽车,楚cc,李杰,傅xd。DEP1基因座的自然变化增强了水稻中的谷物产量。NAT Genet。2009; 41:494-7。gydF4y2Ba
- 56。gydF4y2Ba
周永忠,李志勇,易东成,刘杰,张海根,唐思智,顾明华,梁光华。数量性状基因的缺失gydF4y2BaQPE9-1gydF4y2Ba在水稻驯化过程中,与穗直立有关的性状可改善植株构型。遗传学。2009;183:315-24。gydF4y2Ba
- 57。gydF4y2Ba
Subramanyam S, Sardesai N, Puthoff DP, Meyer JM, Nemacheck JA, Gonzalo M, Williams CE。两个小麦防御反应基因的表达,gydF4y2BaHfr-1gydF4y2Ba和gydF4y2BaWci-1gydF4y2Ba,在生物和非生物胁迫下。植物SCI。2006; 170(1):90-103。gydF4y2Ba
- 58。gydF4y2Ba
辛格TPSK。绿豆分离居群中籽粒产量及其构成因素的关联。印度J Genet出版社1973;33:112-7。gydF4y2Ba
- 59。gydF4y2Ba
Genc Y,Oldach K,verbyla AP,Lott G,Hassan M,测试仪M,Wallwork H,McDonald GK。钠排除QTL与盐度胁迫下的面包小麦的改善幼苗生长相关。Al Appl Genet。2010; 121(5):877-94。gydF4y2Ba
- 60.gydF4y2Ba
马丽,周娥,霍宁,周锐,王刚,贾军。小麦重组自交系耐盐性遗传分析gydF4y2Ba小麦gydF4y2Bal .)。Euphytica。2006; 153(1-2):109-17。gydF4y2Ba
- 61.gydF4y2Ba
Masoudi B, Mardi M, Hervan EM, Bihamta MR, Naghavi MR, Nakhoda B, Amini A.面包小麦苗期耐盐性状的QTL定位。植物学报。2015;33(6):1790-803。gydF4y2Ba
- 62.gydF4y2Ba
Narjesi V,Mardi M,Hervan Em,Azadi A,Naghavi Mr,Ebrahimi M,Zali AA。粮食产量的定量特质基因座(QTL)分析,小麦农艺性状(gydF4y2Ba小麦gydF4y2Ba在正常和盐胁迫条件下。植物学报。2015;33(6):2030-40。gydF4y2Ba
- 63.gydF4y2Ba
Genc Y,Oldach K,Gogel B,Wallwork H,McDonald GK,Smith AB。在具有一系列盐度水平的环境中生长的面包小麦种群的农艺和生理性状的定量特质基因座。mol品种。2013; 32(1):39-59。gydF4y2Ba
- 64.gydF4y2Ba
Sukumaran S, Reynolds MP, Sansaloni C.全基因组关联分析确定了在产量潜力、干旱和热胁迫环境下生长的硬粒小麦产量和组成性状的QTL热点。植物学报。2018;9:81。gydF4y2Ba
- 65.gydF4y2Ba
春化的RFLP映射(gydF4y2BaVrnlgydF4y2Ba)和冰霜抗性(gydF4y2BaFrlgydF4y2Ba)小麦染色体5a的基因。Al Appl Genet。1995年; 90:1174-9。gydF4y2Ba
- 66.gydF4y2Ba
李晓东,李晓东,李晓东,等gydF4y2BaVRN-1gydF4y2Ba多倍体小麦的启动子区。Al Appl Genet。2004; 109(8):1677-86。gydF4y2Ba
- 67.gydF4y2Ba
La Yan L,Tranquilli G,Helguera M,Fahima T,Dubcovsky J。小麦春化基因的位置克隆gydF4y2BaVRN1.gydF4y2Ba.美国国家科学研究院2003;100:6263-8。gydF4y2Ba
- 68.gydF4y2Ba
李法吉WW,中湖H,金东L,惠J,双河C,洪威G,Jun Y,Pingzhi Z,yingXiu W,Xianchun X.基因组宽联系地区屈服相关性状的三个中文面包小麦种群的屈服相关性状密度SNP标记。Al Appl Genet。2018; 131(9):1903-24。gydF4y2Ba
- 69.gydF4y2Ba
徐y,郑x,宋y,zhu l,yu z,gan l,zhou s,liu h,wen f,zhu c.ntltp4,脂质转移蛋白增强盐和干旱胁迫耐受性gydF4y2Ba尼科尼亚塔哈瓦姆gydF4y2Ba.Sci众议员2018;8(1):8873。gydF4y2Ba
- 70。gydF4y2Ba
Kirubakaran Si,Begum Sm,Ulaganathan K,Sakthivel N.来自小麦的新型抗真菌脂转移蛋白的表征。植物理性生物化学。2008; 46(10):918-27。gydF4y2Ba
- 71。gydF4y2Ba
小麦植物螯合酶基因的异质表达gydF4y2BaTAPCS1gydF4y2Ba)增强了对镉的敏感性。生物物理学报。2012;44(10):886-93。gydF4y2Ba
- 72。gydF4y2Ba
al-mashhadani易卜拉欣嗨。用耐盐基因检测估计一些选定的小麦基因型中的耐盐程度(gydF4y2Ba品尝gydF4y2Ba)及其在盐度条件下的表达。int j applager gric sci。2015; 1(2):31。gydF4y2Ba
- 73。gydF4y2Ba
葛瑞聪,赵兵兵,沈永忠,黄志军。小麦丝氨酸/苏氨酸激酶基因的克隆及功能分析(gydF4y2Ba品尝gydF4y2Ba)与耐盐性有关。植物SCI。2012年; 173:55-60。gydF4y2Ba
- 74。gydF4y2Ba
水稻MADS-box基因家族在生殖发育和胁迫中的全基因组鉴定、组织和表达分析。BMC基因组学。2007;8:242。gydF4y2Ba
- 75.gydF4y2Ba
Ma J,Yang Y,Luo W,Yang C,Ding P,Liu Y,Qiao L,Chang Z,Geng H,Wang P等。面包小麦疯饮基因家族的基因组鉴定与分析(gydF4y2Ba小麦gydF4y2Bal .)。《公共科学图书馆•综合》。2017;12 (7):e0181443。gydF4y2Ba
- 76.gydF4y2Ba
lindsaymp, Lagudah ES, Hare RA, Munns R. A基因位点钠排斥(gydF4y2Ba纳克隆gydF4y2Ba),一个耐盐性状,在硬粒小麦上定位。植物生态学报。2004;31(11):1105。gydF4y2Ba
- 77.gydF4y2Ba
Byrt Cspj,Spielmeyer W,James Ra,Lagudah Es,Dennis Es,Tester M,Munns R. HKT1; 5种阳离子转运仪与小麦的Na +排除座,gydF4y2BaNAX2.gydF4y2Ba和gydF4y2BaKna1gydF4y2Ba.植物理性生物化学。2007; 143:1918-28。gydF4y2Ba
- 78。gydF4y2Ba
James Rabc,Zwart Ab,Hare Ra,Rathjen Aj,Munns R.祖先小麦钠排除基因的影响gydF4y2Ba纳克隆gydF4y2Ba和gydF4y2BaNAX2.gydF4y2Ba盐碱地硬粒小麦籽粒产量的研究。acta botanica botanica(云南植物研究). 2012;39:609-18。gydF4y2Ba
- 79。gydF4y2Ba
Chandra SSD,Pathak J,Kumari S,Kumar M,Poddar R,Balyan HS,Prabhu KV,Gupta PK,Mukhopadhyay K.来自下一代转录组测序数据的SNP发现,并在小麦中使用kasp测定的验证(gydF4y2Ba小麦gydF4y2Bal .)。摩尔2017:37品种。gydF4y2Ba
- 80.gydF4y2Ba
Rasheed Aww,Gao F,翟S,金H,Liu J,Guo Q,Zhang Y,Dreisigacker S,夏X,Z Z。基因KASP测定的基因kasp分析在面包小麦中关键经济特征的基因。Al Appl Genet。2016; 129:1843-60。gydF4y2Ba
- 81.gydF4y2Ba
基于竞争等位基因特异性PCR (KASP)的单核苷酸多态性基因分型技术及其在作物改良中的应用。摩尔品种。2013;33(1):1 - 14。gydF4y2Ba
- 82.gydF4y2Ba
大麦核糖体DNA间隔长度多态性:孟德尔遗传、染色体定位和群体动态。美国国家科学学院学报1984;81:8014-8。gydF4y2Ba
- 83.gydF4y2Ba
Purcell SNB,Todd-Brown K,Thomas L,Ferreira Mar,Bender D,Maller J. Plink:一种用于全基因组协会的工具和基于人口的连杆分析。我是j嗡嗡声的遗传。2007; 81:559-75。gydF4y2Ba
- 84.gydF4y2Ba
Bradbury Pjzz,Kroon de,Casstevens Tm,Ramdoss Y,Buckler Es。TASSEL:多样化样本中复杂性状的关联映射软件。生物信息学。2007; 23:2633-5。gydF4y2Ba
- 85.gydF4y2Ba
结构种群的关联映射。美国之音2000;67:170-81。gydF4y2Ba
- 86.gydF4y2Ba
Evanno Grsgj。使用软件结构检测个体的群集数:模拟研究。Mol Ecol。2005; 14:2611-20。gydF4y2Ba
- 87.gydF4y2Ba
yu jpg,Briggs wh,Bi IV,Yamasaki M,Doebuly JF,McMullen MD。一种统一混合模型方法,用于占多级相关性的映射。NAT Genet。2006; 28:203-8。gydF4y2Ba
- 88.gydF4y2Ba
张泽,赖春卿,Todhunter RJ, Tiwari HK, Gore MA, Bradbury PJ。适用于全基因组关联研究的混合线性模型方法。Nat麝猫。2010;42:355-60。gydF4y2Ba
- 89.gydF4y2Ba
Lipka Aetf,Wang Q,Peiffer J,Li M,Bradbury PJ,Gore Ma。Gapit:基因组关联和预测综合工具。生物信息学。2012; 28:2397-9。gydF4y2Ba
致谢gydF4y2Ba
感谢中国农业科学院作物科学研究所的景瑞莲教授和张学勇教授为我们提供HL-DH群体和部分小麦种质资源。gydF4y2Ba
资金gydF4y2Ba
基金资助:国家自然科学基金面上项目(No. 5130459);XDA24030302 &不。XDA08030105)。支持者为实验设计、数据收集、数据分析和文稿撰写提供了资金支持。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
ZSL和QZ构思了这项研究;QZ,QLL,pH和WT设计了实验;QLL开发了ZX-RIL人口;QZ,pH和QLL在两个环境下进行ZX-RIL群体的表型;QZ,WT和pH在六种环境下进行191个小麦涂抹的表型;pH执行GWAS分析并写了稿件;QZ还提出了许多建设性建议并修改了手稿。BL和HWL在材料制备和实验性能方面提供了很多帮助。所有作者阅读并认可的终稿。gydF4y2Ba
通讯作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用gydF4y2Ba
同意出版物gydF4y2Ba
不适用gydF4y2Ba
相互竞争的利益gydF4y2Ba
两位作者宣称他们没有相互竞争的利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:表S1。gydF4y2Ba
本研究中使用的191份常见小麦养殖的名称和起源。gydF4y2Ba
附加文件2:表S2。gydF4y2Ba
2015 - 2017年低矿化度和高矿化度处理现场试验对比分析PH值,株高;SN,穗数;SL,穗长;KPS:每穗粒数;GN,粒数;TKW:千粒重;YPP:每地块产量;BM、生物质量。**表示确定的显著差异gydF4y2BapgydF4y2Ba< 0.01。LS:低盐度处理;HS,高盐度处理。gydF4y2Ba
附加文件3:表S3。gydF4y2Ba
利用小麦660k单核苷酸多态性芯片的基因型数据,计算了低盐和高盐处理下191份小麦材料的遗传多样性。gydF4y2Ba
附加文件4:表S4。gydF4y2Ba
对191份普通小麦在低盐和高盐处理下8个农艺性状的GWAS结果进行了分析。PH值,株高;SN,穗数;SL,穗长;KPS:每穗粒数;GN,粒数;TKW:千粒重;YPP:每地块产量;BM、生物质量。LS:低盐度处理; HS, high salinity treatment; UN, unknown.
附加文件5:表S5。gydF4y2Ba
Gwas在191年透露的候选人SNP在191年在低盐度处理下的常见小麦涂抹。PH值,株高;SN,穗数;SL,穗长;KPS:每穗粒数;GN,粒数;TKW:千粒重;YPP:每地块产量;BM、生物质量。LS:低盐度处理; HS, high salinity treatment.
附加文件6:表S6。gydF4y2Ba
在低和高盐度处理下的11个基因座的支持等位基因和遗传效应。PH值,株高;SN,穗数;SL,穗长;KPS:每穗粒数;GN,粒数;TKW:千粒重;YPP:每地块产量;BM、生物质量。LS:低盐度处理; HS, high salinity treatment. “ns” represents no calculation for no significantly difference of traits between alleles.
附加文件7:表S7。gydF4y2Ba
snp位点与目前和以前的研究中发现的产量相关性状显著相关。PH值,株高;SN,穗数;SL,穗长;KPS:每穗粒数;GN,粒数;TKW:千粒重;YPP:每地块产量;BM、生物质量。gydF4y2Ba
附加文件8:表S8。gydF4y2Ba
14个KASP标记的信息为9个基因座提供了信息。PH值,株高;SN,穗数;SL,穗长;KPS:每穗粒数;GN,粒数;TKW:千粒重;YPP:每地块产量;BM、生物质量。gydF4y2Ba
附加文件9:图S1。gydF4y2Ba
普通小麦品种有利等位基因的平均数目。(a)不同类型普通小麦材料的有利等位基因的平均数量。(b)不同地区普通小麦种质有利等位基因的平均数量。gydF4y2Ba
附加文件10:图S2。gydF4y2Ba
DH群体“瀚轩10×Lumai 14”中HX等位基因与LM等位基因在不同性状上的平均差异,其中HX为“环轩10”等位基因,LM为“鲁麦14”等位基因(**为极显著水平)。gydF4y2Ba
附加文件11:图S3。gydF4y2Ba
QTL位于常见小麦种群和ZX-RIL群中刺激性5B的刺激物长度(SL)。(a)染色体位置gydF4y2BaQ-5B1gydF4y2Ba在191份普通小麦种质中。(b)“中麦175×Xiaoyan 60”RIL群体中SPS的QTL定位。(c) LD图gydF4y2BaQSl-5B1gydF4y2Ba在191年的常见小麦牧草和QTL中的QTL群中的“中迈175×小燕60”。gydF4y2Ba
附加文件12:图S4。gydF4y2Ba
每个基因座的不同单倍型的比例。该百分比代表了191份常见小麦涂抹中单倍型的比例。gydF4y2Ba
附加文件13:图S5。gydF4y2Ba
重要位点的单倍型分析。PH值,株高;SN,穗数;SL,穗长;GN,粒数;TKW:千粒重;YPP:每地块产量;BM、生物质量。LS:低盐度处理;HS,高盐度处理。gydF4y2Ba
附加文件14:图S6。gydF4y2Ba
14个KASP标记对9个关键位点的散点图分析。红点表示遗传材料具有fam型等位基因;蓝点表示遗传材料具有hex型等位基因。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.创作共用及公共领域专用豁免书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文提供的数据,除非在数据的信贷额度中另有说明。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
胡,P.,郑,Q.,罗,Q.gydF4y2Baet al。gydF4y2Ba盐胁迫条件下常见小麦产量和相关性状的基因组基因型研究。gydF4y2BaBMC植物杂志gydF4y2Ba21,gydF4y2Ba27(2021)。https://doi.org/10.1186/s12870-020-02799-1gydF4y2Ba
已收到gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-020-02799-1gydF4y2Ba
关键字gydF4y2Ba
- 常见的小麦gydF4y2Ba
- 有利的等位基因gydF4y2Ba
- GWASgydF4y2Ba
- 盐胁迫gydF4y2Ba
- Wheat660K SNP芯片gydF4y2Ba
- Yield-related特征gydF4y2Ba