摘要
背景
当归伞形花属(伞形花科)是一重要的经济属,约110种。当归物种在北半球的所有大陆上都有发现,东亚拥有最多的物种。形态特征,如果实解剖,叶片形态和地下结构当归物种表现出极端的多样性。因此,分类学的分类当归物种是一种复杂且有争议的物种,以往基于形态和分子的分类方法存在很大的不一致性。此外,还对各主要进化支的系统发育关系进行了研究当归集团,特别是在当归尚不清楚。叶绿体(cp)基因组序列已广泛应用于系统发育研究和遗传多样性评估。
结果
本研究对来自22个种、2个品种和2个品种的28个cp基因组进行了测序和组装当归.结合Genbank的36个可用的CP基因组,来自亚家族Apioideae的特征和进化模式当归对Cp基因组进行了研究,并对其系统发育关系进行了研究当归物种解决。的当归CP基因组具有典型的四脚石结构,包括由大单拷贝区域(LSC:76,657-103,161bp)和小单拷贝区域(SSC:17,433-21,794)分离的一对倒反转的重复(IRS:5836-34,706bp)。 bp). Extensive expansion and contraction of the IR region were observed among cp genomes of当归种,cp基因组的多样性格局与系统发育位置高度一致当归物种。种当归被分为两个主要的碎片,大多数物种被分组当归集团和答:omeiensis和答:中国分组的Sinodielsia与当归tenuissimum。
结论
我们的结果进一步证明了体积系统托儿科在增强复杂属的系统发育重建方面的能力,并在塑料进化中提供新的见解当归l
背景
多年生草本属当归L.(家庭Apiaceae)是一种分类论复杂和争议组,包含大约110种具有极端多态性的叶形形态,果实解剖和地下结构的极端多态性[1,2,3.].的成员当归分布在整个北半球,其中最多的物种(约55种)集中在东亚[3.,4,5].45当归种分布在中国,其中32种是特有种[3.,6];有些物种在野外极其罕见,只能从有限的标本中得知[7].
有些是地方性的当归这些品种具有巨大的经济价值,几百年来一直被用作中药[3.,8].一些种类的当归是官方的原药记录在中国2010年人民共和国的中国药典委员会,包括答:中国(中药名称:唐贵),答:biserrata(Duhuo),白芷(Baizhi)[7].另外15种当归也用作民间补救措施中草药材料(http://frps.eflora.cn.).
先前的研究的当归Systematics专注于KaryotaCicalical分析[2,9,10.、花粉形态[11.,12.,13.],叶柄和水果解剖学[14.]和植物化学[15.,16.].先前的分子系统发育分析当归专门基于DNA序列的系统发育分析,特别是在核核糖体(NR)DNA内部转录的间隔物(其)区域,以及相对较少的中国代表当归已纳入分析[6,17.,18.,19.,20.].Xue et al.(2007)使用了来自种的44条ITS序列当归和来自东亚的盟友,并提出当归是多源的。Feng et al.(2009)提出当归包括后,S.s.为单系Coelopleurum.,Czenaevia.,Ostericum koreanum在分析中但排除了先前认可的其他几种物种当归s.l. Liao等(2013)重建了植物的系统发育当归sll和不友好的关系当归有更广泛的样本当归(包括已知55种中的44种),并对nrDNA (its, ETS)、cpDNA (rps16基因内区,rps16-trnK代际垫片,rpl32-trnl.基因间的间隔,trnL-trnT基因间间隔)和形态数据。他们的分析表明许多种类的当归下降外当归S.S.那个四种物种当归发生在当归组。然而,赛利亚族内部的分支关系,特别是在当归但仍有争议,且大部分问题尚未解决。
叶绿体是植物光合作用和其他生化途径的关键细胞器[21.,22.].叶绿体基因组是植物中三个DNA基因组(包括核基因组和线粒体基因组)之一,具有115 - 165kb的相对保守的四部环结构[23.,24.].由于其相对稳定的基因组结构,完整的cp基因组序列已被广泛接受,为理解进化生物学提供了有价值的和信息丰富的数据来源,并已成为研究植物系统发育的有力工具[24.,25.,26.,27.,28.,29.,30.,31.,32.,33.,34.,35.].
在本研究中,我们报道了28个新测序的完全cp基因组当归(22种、2个变种和2个栽培品种),并对其基因组结构多样性进行了研究当归通过比较叶绿体基因组分析。此外,我们还测试了完全cp基因组在解决有争议的和不太好解决的系统发育方面的能力当归通过综合分析与另外36个已发表的CP基因组,从NCBIGegank从Apioideae的代表性植物(亚猿的亚家族)提供。
结果
的特点当归质体系
由Illumina Hiseq 4000系统获得的成对终端原始读数的数量范围为8,616,334至22,518,619〜28当归样本。映射每个对端读取后当归共提取52,277 ~ 1,673,010 reads,获得59 ~ 1445×叶绿体基因组覆盖率(表2)1).人工检测组装的叶绿体基因组的IR连接区,以避免可能的注释错误。从而获得了高质量的叶绿体基因组序列,便于下游分析。28当归叶绿体基因组序列存入GenBank(登录号:MT921958-MT921985)。
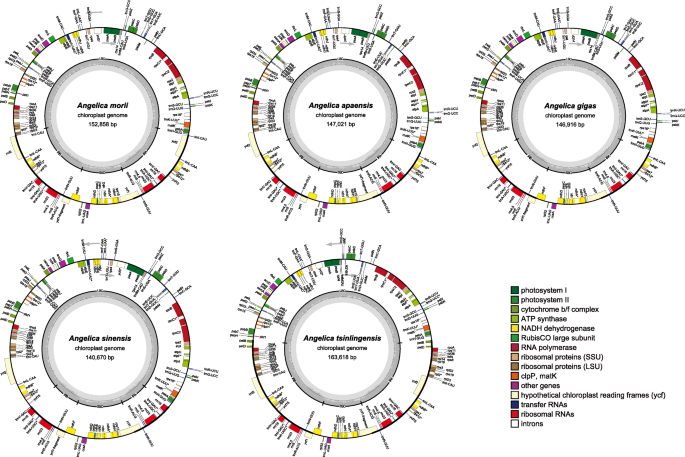
完全叶绿体基因组的长度范围从140,670 bp(答:中国)至163,618 bp(A. Tsinlingensis.)的33个cp基因组当归种(变种或品种)。所有cp基因组均具有典型的四部结构,包括一对被大单拷贝区(LSC: 76,657-103,161 bp)和小单拷贝区(SSC: 17433 - 21,794 bp)分隔的反向重复区(IRs: 5836-34,706 bp)(图1)。1;表格2).平均GC含量为37.5%,33完成之间几乎相同当归cp基因。基因总数为121 (答:中国)至144 (A. Tsinlingensis.)在这33中完成当归cp基因。在去除IR区域的重复基因后,33当归cp基因组包含113 ~ 114个不同的基因,包括80个蛋白质编码基因和4个rRNA基因1).大多数cp基因组含有29个tRNA基因,7个cp基因组含有1个tRNA基因(trnGucc或trnGgcc)(附加文件1:表S1)。在此基础上,研究了大豆中cp基因组的组织、基因顺序和GC含量当归与其他高等植物高度相同和相似(Fig。1).
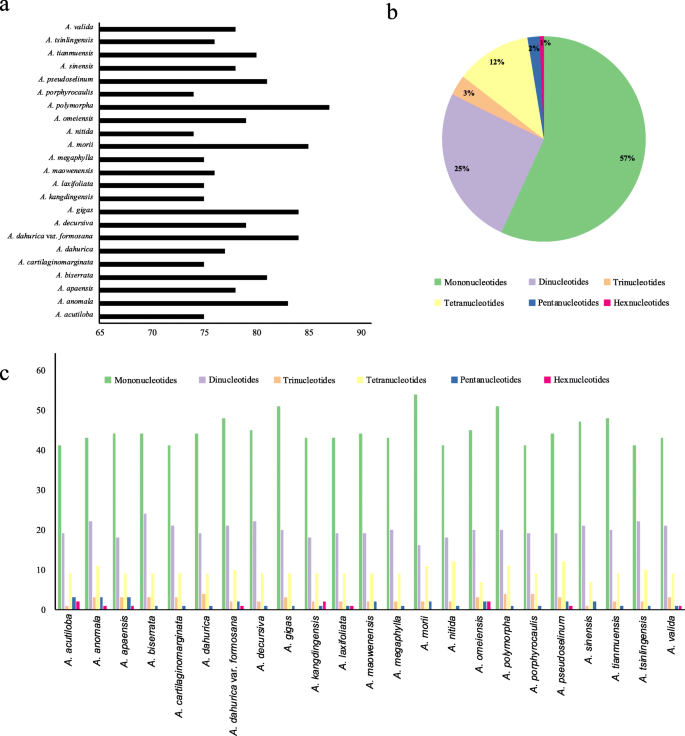
简单序列重复的数量(SSR)范围为68(答:nitida87(A. Polymorpha.)。当归cp基因(图。2一种)。大多数SSRS是单核苷酸重复(58%),而二核苷酸,三核苷酸,四核苷酸,四核苷酸和六核苷酸SSR组成的所有SSR的24,4,11,2和1%组成分别(图。2b).变异程度最高的单核苷酸重复数,范围为38 (答:nitida)至54(答:morii),而其他重复类型的数量在33人之间没有显著差异当归cp基因组(附加文件2:表S2,图2c)。
红外区域的扩张和收缩
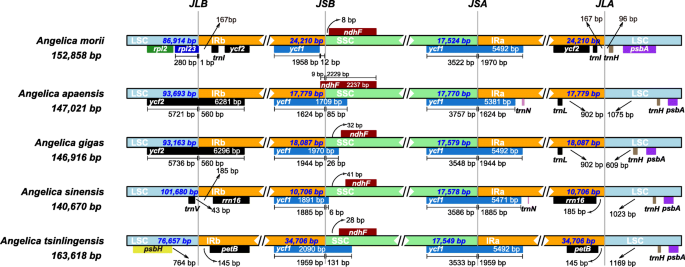
尽管CP基因组在基因组结构和尺寸方面高度保守,但由于IR / SC边界区域的膨胀和收缩引起的IR / SC结的大小的变化被认为是在CP基因组中产生长度变化的主要机制更高的植物[26.,36.,37.,38.].33例患者的IR区有广泛的扩张和收缩当归本研究检测的cp基因组根据IR/SC连接区和有无倒置的特征可分为五种不同类型。的红外区域答:morii扩展并包含一个副本的ycf2基因(I型);在大多数(25/33)当归在cp基因组中,IR/SSC的连接位点位于ycf1的基因,IR/LSC的连接位点位于trnL和trnH(II型)(图。3.).逆温约490 bptrny-trnd-trne的cp基因组中观察到基因答:牡蛎(类型III)和答:moii(图。4).红外区明显收缩答:中国(10,706 bp),并以RRN16.IR区基因(IV型);最大的膨胀区域观察到在A. Tsinlingensis.最后以petB基因在IR区域(类型V)(图。3.).
系统发育分析
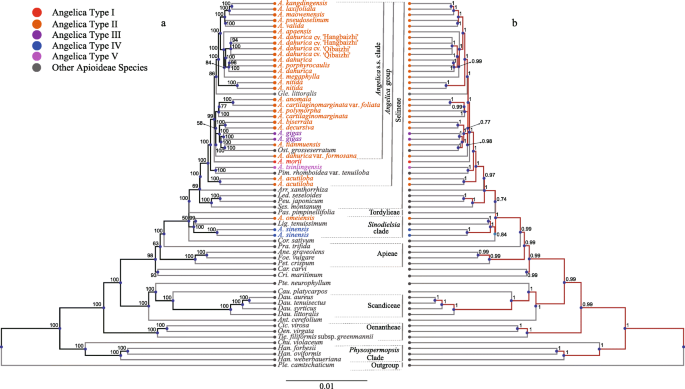
ML树和贝叶斯树产生了高度相似的拓扑。的成员当归主要分为两个主要的血统:(1)当归在Selineae部落(BS = 100, PP = 1)和(2)Sinodielsia(BS = 100, PP = 1)5).遵循以前研究确定的主要行员的名称[1,39.,40,41.].的当归小组成员占了大多数当归加入(30/33)和26当归加入形成了良好的支持当归s.s. clade (BS = 99, PP = 1)Glehnia littoralis和Ostericum grosseserratum(图。5).在这内当归sss分支,发现了四种主要的谱系(答:kangdingensis至答:有效霉素,答:apaensis至答:megaphylla,答:是至答:cartilaginomarginata,答:biserrata至新颖性。grosseserratum).支枝位置的支撑值答:是至白芷var。formosana较低(BS = 58, PP = 0.77)。的滨海当归物种答:morii,居住在东亚沿岸地区或岛屿,A. Tsinlingensis.,这显然不同于当归以其薄翼的背脊和犁沟上的三棱状分枝[1,被放在外面当归s.s.演化支(图1)5).答:acutiloba也被隔离了当归S.S.剥夺并占据早期分歧的分支当归集团(无花果。5).的Sinodielsia思工组成答:omeiensis,答:中国和当归tenuissimum。大多数进化枝在当归组接受了很高的BS/PP支持,除了包括的分支答:是至白芷var。formosana(BS = 58, PP = 0.77)5).大多数登记入册白芷(白芷,白芷简历。杭巴迪和白芷简历。兴安县城内)被放置在一个良好支持的支系中,还包括A. Porphyrocaulis.,除了白芷var。formosana,它被放置在一个相对遥远的分支,包括答:是至答:tianmuensis和Ostericum grosseserratum(图。5).的演化支,非当归物种一般与先前研究的推断一致[1,6,40,42.,43.].
讨论
中红外区域的扩张和收缩当归质体系
在这项研究中,我们测序了28个叶绿体基因组当归利用Illumina HiSeq-4000平台,将这些基因组与GenBank中其他5个已发表的相同属的叶绿体基因组进行了比较分析。的叶绿体基因组当归具有高等植物典型的四部分结构,基因顺序和含量均较为保守,由113 ~ 114个不同的基因组成。cp基因组当归GC含量相近,但LSC和SSC区域的GC含量显著低于IR区域,原因是IR区域包含了8个GC含量较高的rRNA基因。IR区域被认为是叶绿体基因组中最保守的区域[44.].
叶绿体基因组长度差异的主要原因被认为是IR、LSC和SSC区域的扩张和收缩,这在进化中是比较常见的[45.].
cp基因组的长度在140,670个碱基对(答:中国)至163,618个碱基对(A. Tsinlingensis.).已提出收缩,扩建或丧失IR地区的主要原因之一,解释CP基因组大小的变化[46.].据报道,伞形科植物的红外区出现了大规模的扩张和收缩;的确,在Apioideae cp基因组中记录的JLB变化的频率和规模在被子植物中是前所未有的[47.].在我们的研究中,红外区广泛的扩张和收缩检测到当归结果发现,叶绿体基因组的红外和红外与SSC或LSC的边界发生了五种类型的变化(图1)。3.).大多数当归种(25/33)具有II型cp基因组,IR/SSC的连接位点位于ycf1IR/LSC的连接位点位于基因之间trnL和trnH并显着聚集在当归组,除了答:omeiensis,它被归为Sinodielsia思.IR区域的扩展导致该区域包含额外的基因;例如,这一地区的沿海物种的扩张答:morii导致包含了一个复制的副本ycf2基因(I型)。在CP基因组中观察到IR区域的最大膨胀A. Tsinlingensis.(34,706 bp),以petB基因在IR区域。观察到红外区明显收缩答:中国(10704个基点)。观察到的变异模式与当归在系统发育分析中恢复的物种,反映了这种有涉及属的物种和CP基因组的高多样化(图。5).
属的系统发育当归
利用全cp基因组序列从33当归通过ML和Bayesian分析,得到了一个高度一致的拓扑结构(图1)。5).Apioideae的主要分支(如:酒仙亚科、Scandiceae、Apieae、tordyliae和Selineae)的分配与先前的研究结果一致[18.,40,42.,43.].种当归不属于一个单系支系,而是分布在四个支系中,大多数当归种聚集在一个良好支持的支系(当归(组),支持先前研究的系统发育拓扑[1,41.].该组还包括来自属的物种Glehnia(克林顿。littoralis)、Ostericum(新颖性。grosseserratum)和Pimpinella (Pim。rhomboideivar。tenuiloba)。
的当归Liao et al.(2013)中的s.s. clade主要包含东亚当归物种和来自Ostericum(新颖性。Koreanum,新颖性。huangdongensis)和Czemaevia(CZE。拉维塔塔var。larvigata)但是被排除在Glehnia (Gle。littoralisvar.littoralis,克林顿。var.Leiocarpa).基于整个cp基因组数据,克林顿。littoralis被归类于当归支持度较高(BS = 86, PP = 1)。答:是之前被归为Ostericum grosseserratum和物种Peucedanum在Acronema进化枝(6,43.],然后放入当归当同时使用nrITS、nrETS、cpDNA和形态学特征时[1].在这项研究中,答:是分组了A.边缘软骨,边缘软骨var。foliata,和A. Polymorpha.在一支枝内当归党卫军进化枝。的分配答:morii和答:tsinglingensis在当归小组,但在外面当归S.s. clade也与以前的研究一致,并得到了形态学特征研究的支持(如每条沟的背肋和三棱)[1].由于其独特的果实特征,其分类地位答:acutiloba多年来一直备受争议。答:acutiloba之前被置于其中当归基于nrDNA ITS序列的s.s.c leade [6,然后被放置在当归基于nrDNA、cpDNA和形态学特征的s.s.c leade [1].在我们的研究中,答:acutiloba也被孤立于当归S.S.剥夺并占据早期分歧的分支当归组。答:ameiensis之前被归为答:apaensis和答:nitida在一支枝内当归基于nrDNA ITS和cpDNA序列的s.s.c leade [1,6而是和他在一起答:中国和当归tenuissimum在Sinodielsia通过本研究的全cp基因组数据获得高支持度(BS = 100, PP = 1)。
本研究报告了33例的比较分析结果当归CP基因组在IR区中发现的广泛膨胀和收缩当归.CP基因组的变化可以分为五种类型,这与这些的一般系统发育放置一致当归物种。的关系当归这里检查的物种是清晰的,谱系在内当归与既往研究相比,分组的分辨率更高。我们认为我们的研究结果有助于我们理解人类的进化史当归物种;然而,更广泛的cp基因组取样(例如,蔷薇属植物,蔷薇属植物,多毛植物,和答:oncosepala)可能是必要的,以进一步表征之间的关系当归物种。这些发现也提供了信息丰富和有价值的遗传来源当归帮助物种鉴定和未来分类重建的种质资源当归.
结论
我们的分析不仅揭示了广泛的扩张和收缩的cp基因组之间的IR区域当归sp物种,但也显示了质体的力量解决关系,目前不解决和有争议的群体。红外区域的变化规律可分为五种不同类型,与类群基本一致当归系统发育分析中的物种。的关系当归这里调查的物种主要是清楚地分类和内部的谱系当归类群的分类比以往的研究有了更好的解析性,我们相信这将有助于理解类群的进化史当归物种,更广泛的CP基因组采样可能是必要的,以进一步说明物种的关系当归.
方法
分类抽样
我们采集了24个品种(包括2个品种和2个栽培品种)当归分布于14个省,约占85%种,覆盖大部分分布当归在中国(http://freps.eflora.cn/).本研究中收集的28个样本的采样信息的详细信息显示在支持信息附加文件中3.:表S3。基于形态特征,在中国医学院中国中医院国家资源中心的植物商品中,所有样品都是由Nian-Hu-Hotany,江苏省和中国科学院和中国科学院研究所)鉴定。科学。收取这些样本的许可是没有必要的,这些样本没有包含在国家重点保护植物名单中。将来自每种加入的新鲜叶子立即用硅胶干燥以进一步DNA提取。
植物材料和DNA提取
采用中国天根生物技术(北京)有限公司的植物基因组DNA试剂盒(DP305)提取每个样本的总基因组DNA。都是NanoDrop分光光度计(ND-1000;使用美国赛默飞世尔科学公司(Thermo Fisher Scientific, USA)和Qubit 2.0荧光计(Invitrogen, Life Technologies)评估DNA的质量和数量。
Illumina公司测序
用Covaris S2对总基因组DNA (30 - 150ng)进行片段分割,平均片段大小为550 bp。TruSeq DNA纳米文库制备试剂盒(Illumina)用于按照制造商的说明制备DNA文库。文库使用KAPA Illumina Library Quantification Kit (KAPA Biosystems)通过定量聚合酶链反应进行定量,并使用Illumina HiSeq 4000平台(Illumina, San Diego, CA)对汇集的文库进行测序(2 × 150 bp)。
叶绿体基因组的组装与注释
对原始测序reads进行定性评估并使用GetOrganelle version 1.6.4进行组装[48.使用默认设置。通过人工修订,确定叶绿体基因组序列中的模糊核苷酸或间隙以及IRs和SSC/LSC之间的四个连接区。使用GeSeq version 1.79对叶绿体基因组进行注释[49.].注释结果使用genes8.0.2版本(http://www.Geneious.com)避免潜在的注释错误。绘制叶绿体基因组的基因图用OGDDRAW版本1.3.1绘制[50.].
简单序列重复分析
使用Perl脚本MISA 2.0版本搜索CP基因组中的简单序列重复(SSR或MicroStaTellites)LOCI [51.].单核苷酸、二核苷酸、三核苷酸、四m五核苷酸和六核苷酸的ssr最小值分别为10、5、4、3、3和3。对重复进行了人工验证,删除了大量的结果。
cp基因组的比较分析
使用内部python脚本对基因组大小、GC含量、LSC/SSC/IR大小和基因数量进行统计。cp基因组结构和基因含量的比较分析采用Mauve version 2015-02-13 [52.]来定位可能的重排(如倒置)和基因顺序的变化Tiedemannia filiformis.无性系种群。格林曼尼伊(加入基因库:HM596071)。用IRscope可视化方法检查LSC-IRa/b和SSC-IRa/b的结合点[53.].
系统发育分析
对33个样本进行系统发育分析当归cp基因组连同来自Apoideae亚科主要世系的31个种(附加文件4:表S4)。使用PartitionFinder 2版本2.1.1选择了最合适的替代模型[54.]的最大似然(ML)和贝叶斯推理(BI)。ML分析采用RAxML-NG 0.9.0版本[55.]采用一般时间可逆(GTR) + G模型,并通过2000个bootstrap重复评估节点支持。BI分析采用MrBayes版本3.2.7a [56.].为马尔可夫链蒙特卡罗(MCMC)分析进行了两种5,000,000代的两种链,每1000代采样树木。前25%的采样树被抛弃,因为烧伤和剩余的树木用于建立50%的多数规则共识树。当分流频率的平均标准偏差保持在0.001以下时,考虑了实质性。
缩写
- BI:
-
贝叶斯推理
- GTR:
-
一般时间可逆
- 红外光谱:
-
反向重复
- 其:
-
核糖体DNA内部转录间隔区
- LSC:
-
大单副本
- ml:
-
最大似然
- 核糖体rna:
-
核糖体核糖核酸
- SSC:
-
小单副本
- tRNA:
-
转移RNA.
参考文献
- 1。
关键词:归当归,形态学,nrDNA, cpDNA,系统发育,归当归属系统机器人。2013;38(1):266 - 81。
- 2。
Vasil'eva MG, Pimenov MG。标题伞形科当归属的核型分类学分析。植物生态学报,1991;
- 3.
Meng-Lan S,Fa-Ting P,Zehui P,Watson MF,Cannon JFM,Holmes-Smith I等。在:吴泽,乌鸦博士,编辑。亚梨膜。PP。1-205在中国植物区,卷。14.圣路易斯:密苏里植物园新闻和北京:科学出版社;2005年。
- 4.
Wen J.开花植物中东亚和东部北美疏散分布的演变。Annu Rev Ecol Syst。1999; 30(1):421-55。
- 5。
廖春英,余勇,何晓娟。从nrDNA和cpDNA序列分析推断当归属(伞形科tribe Selineae)的历史生物地理学。系统演化。2012;50(3):206-17。
- 6。
冯涛,余勇,张旭,陈伟,何秀娟,等。基于ITS序列的中国横断山当归属及其近缘属(伞形科)的分子系统学研究:系统发育亲缘关系和生物地理意义。植物学报;2009;
- 7。
袁庆军,张斌,蒋丹,张文杰,林天泰,王乃华,等。药材种类及内药材鉴定当归L.(Umbelliferae)基于从DNA条形码推断的系统发生。Mol Ecol Resour。2015; 15(2):358-71。
- 8.
山猕猴。见:Shan RH, Sheh ML, editors。伞形科。《中华Popularis Sinicae》,第55卷,第13-62页。北京:中国科学院;1992.
- 9.
acta botanica yunbotanica(云南植物研究). P(泽慧),L - Xin-Tian, S menglan, S琅然。四川8种当归(伞形科)核型及其地理分布。植物分类学报。1991;29(5):431-8。
- 10.
张秋云,何兴进,张玉成,彭玲,吴宁。四川当归属6种植物核型研究。云南植物学报。2005;27(5):539-44。
- 11.
陈伟伟,何晓军,张学明,蒲建新。西南当归属植物花粉形态及其系统进化分析。西北植物学报.2007;27(3): 1364 - 72。
- 12.
兰思敏,苏平,潘志华。东亚与北美当归花粉形态的比较研究。植物资源与环境,1997;6(1):41-47。
- 13。
D.花粉形态Peucedanum来自四川及其系统意义。Acta Bot Boreal Endidation Sin。2004; 24(12):2341-45。
- 14。
张启云,邢锦鹤,张永昌,罗平,宁武。8种植物果实和叶柄的解剖学研究当归利用四川。武汉植物保护学报2005;23(6):549-54。
- 15.
陈旭,常启英。紫花千狐的属位及其与韩国当归的比较分类学研究。南京大学学报1987;23(1):23-31。
- 16。
Shneyer VS, Kutyavina NG, Pimenov MG。系统的内部和之间的关系Peucedanum和当归(伞形科-前藤科)从种子蛋白质的免疫学研究中推断出来。植物生态学报,2003;
- 17。
Katz-Downie DS,Valiejo-Roman Cm,Terentieva Ei,Troitsky Av,Pimenov Mg,Lee B等。朝向Apiaceae亚家族的分子发育亚己二酸:核核糖体DNA的附加信息其序列。植物系统中的Evol。1999年; 216(3-4):167-95。
- 18.
关键词:蜂科,蜂亚科,分子系统发育,核糖体DNA中国生物医学工程学报,1996;
- 19.
关键词:前藤属(Peucedanum sensu lato),近缘属,系统发育位置,Selineae(伞形科,伞形亚科)植物生态学报,2004;
- 20.
雪HJ。东亚当归的分类研究:核糖体DNA ITS序列的推断。植物分类学报。2007;45(6):783-95。
- 21.
关键词:叶绿体标记,植物系统发育,遗传多样性,DNA条形码《公共科学图书馆•综合》。2012;7 (4):e35071。
- 22.
植物叶绿体全基因组序列:基因组织、比较分析及与其他被子植物的系统发育关系。植物科学与技术,2016;
- 23.
王H-X,Liu H,Moore MJ,Landrein S,Liu B,Zhu Z-X等。塑体系统核糖中的洞察Caprifoliaceae S.L的演变。(Dipsacales)。mol phylocyet evol。2020; 142:106641。
- 24。
徐超,董伟,李伟,吕勇,谢旭,金鑫,等。六项的比较分析Lagerstroemia完整的叶绿体基因组。植物科学与技术,2017;
- 25。
1 .董伟,赵晓,陶超,林凯,周胜。被子植物质体基因组测序技术的研究:一套完整的通用引物和虎砂属植物系统发育的实例研究。基因组生物学进化。2013;5:5。
- 26。
东路,徐C,Li D,Jin X,Li R,Lu Q等。亲完全叶绿体基因组序列在乳糖中的比较分析Haloxylon物种(Amaranthaceae)。peerj。2016; 4:E2699。
- 27。
鲁尔曼(Ruhlman T, Lee S-B, Jansen RK, Hostetler JB, Tallon LJ, Town CD,等。完整的质体基因组序列胡萝卜胡萝卜:对被子植物生物技术和系统发育的启示。BMC基因组学。2006;7(1):222。
- 28。
黄迪,Hefer CA, Kolosova N, Douglas CJ, Cronk QCB。全质体测序揭示了密切相关的凤仙花的深层质体分化和核不一致,杨树balsamifera和p . trichocarpa(杨柳科)。新植醇。2014;204(3): 693 - 703。
- 29。
关键词:叶绿体基因组,生物地理结构,遗传变异,野生居群这将季L.在西北欧洲。GCB生物能源。2017; 9(1):46-56。
- 30.
胡萝卜属植物的全质体系统发育(Daucus.(蜂科):符合核数据和线粒体和核DNA插入质体。中国生物医学工程学报,2017;
- 31.
Liu H, Moore M, Landrein S, Liu B, Zhu Z,等。盘山蕨科(双翅目)质体系统基因组的进化洞见。Mol系统进化。2019;142:106641。
- 32.
姚刚,金军,李海涛,杨建斌,Mandala V, Croley M,等。石竹属质体系统基因组的进化洞见。分子系统进化。2019;134:74-86。
- 33.
刘丹,涂晓东,赵卓,曾明义,张松,马玲,等。质体系统基因组数据产生了新的和健全的见解,cleisostomus - gastrochilus支系(兰科,Aeridinae)。分子系统进化。2020;145:106729。
- 34.
张辉,魏荣,向清平。基于质体的系统基因组学解决了sanguinolenta类群在石松类(卷柏科)穗苔中的位置问题。系统进化。2020;106788。
- 35.
张锐,王永华,金娟,Moore M,张世东,陈世勇,等。质体系统基因组冲突的探索为豆科植物的深层关系提供了新的见解。系统医学杂志。2020;69(4):613 - 22所示。
- 36。
Kim KJ, Lee HL。陆生植物叶绿体基因组中普遍存在小逆转录。摩尔细胞。2005;19(1):104 - 13所示。
- 37。
Sajjad A, Khan AL, Khan AR, Muhammad W, Kang SM, Khan MA等。耳穴烟叶绿体全基因组及其与近缘种的比较。植物科学与技术,2016;
- 38。
关键词:栎属植物,叶绿体,全基因组,比较分析植物科学与技术,2016;
- 39。
唐尼斯SR,Katz-Downie DS,Watson MF。基于叶绿体DNA RPL16和RPOC1内含子序列的开花植物家族苹果藻的系统发育:甲壳化亚家族分类的超级分类。我是J机器人。2000; 87(2):273-92。
- 40.
1 .周健,龚欣,彭辉。中国蜂科蜂亚科蜂亚科的分子系统发育:来自ITS和cpDNA内含子rpl16和rps16序列的进一步证据。中国生物医学工程学报,2009;
- 41.
downnie SR, Spalik K, Katz-Downie DS, Reduron J-P。基于ITS序列的系统发育分析推断蜂科蜂亚科的主要分支。植物多样性研究进展。2010;
- 42.
1 .龚欣,刘志伟,彭慧,周杰。基于核糖体DNA内转录间隔序列的中国蜂科蜂亚科系统发育研究。植物分类学报,2015,34(5):751 - 754。分类单元。2008;57(2):402 - 416。
- 43.
唐妮斯,斯宾利克,凯茨 - 唐妮DS,雷隆JP。基于ITS序列的系统发育分析推断蜂科蜂亚科的主要分支。植物多样性evol。2010; 128(1):111-36。
- 44.
关键词:五分树科,叶绿体,全序列分析,系统发育《公共科学图书馆•综合》。2013;8 (10):e78568。
- 45.
薛胜,石涛,罗伟,倪鑫,Iqbal S,倪z,等。叶绿体全基因组的比较分析李属却已,p . armeniaca,p . salicina.园艺杂志2019;6(1):89。
- 46.
Bock R,Knoop V,编辑器。叶绿体和线粒体的基因组学,ADV。Photosyn Resp,Vol。35. DONDRECHT:斯普林克荷兰;dordrecht;2012. p。1-458。
- 47.
蜂科蜂亚科叶绿体反向重复序列的扩增和收缩。系统机器人2000;25(4):648-67 620。
- 48。
金建军,于文斌,杨建斌,宋勇,dePamphilis CW,易廷生,等。GetOrganelle:一个快速和通用的工具包,用于准确的重新组装的细胞器基因组。基因组生物学。2020;21(1):241。
- 49。
tilich M, Lehwark P, Pellizzer T, Ulbricht-Jones E, Fischer A, Bock R等。通用的、准确的细胞器基因组注释。核酸研究进展[j] .中国生物医学工程学报,2017;
- 50.
Greiner S, Lehwark P, Bock R. OrganellarGenomeDRAW (OGDRAW) version 1.3.1:用于细胞器基因组图形化可视化的扩展工具包。核酸图谱2019;47(W1): W59-64。
- 51.
利用EST数据库开发大麦(Hordeum vulgare L.)基因衍生ssr标记。理论应用,2003,(3):411-22。
- 52.
亲爱的ACE, Mau B, Blattner FR, Perna NT。淡紫色:保守基因组序列的多重排列与重排。基因组研究》2004;14(7):1394 - 403。
- 53.
Amiryousefi A,HyvönenJ,Poczai P. Irscope:一个在线计划,用于可视化叶绿体基因组的结位。生物信息学。2018; 34(17):3030-1。
- 54.
关键词:分子系统发育,进化模型,分割模型,分子系统发育中国生物医学工程学报。2016;34(3):772 - 73。
- 55.
Kozlov Am,Darriba D,Flowi T,莫雷利B,Stamatakis A. Raxml-NG:用于最大似然性的最大似然推理的快速,可扩展和用户友好的工具。生物信息学。2019; 35(21):4453-5。
- 56.
Ronquist F,Teslenko M,Van der Mark P,Ayres DL,Darling A,HöhnaS等人。MRBAYES 3.2:高效的贝叶斯系统发育推理和跨大型模型空间的模型选择。系统中的生物学。2012; 61(3):539-42。
致谢
不适用。
资金
本研究由国家重点研发计划(no . 2017YFC1703700;国家自然科学基金项目(NSFC: 81891010和81891014);中央政府重点项目:名优中药资源可持续利用能力建设(2060302);资助机构没有参与实验的设计、分析和数据的解释,也没有参与手稿的撰写。
作者信息
从属关系
贡献
LH,QY和WD构思和设计了这项研究。WD进行了De Novo组装,基因组注释,系统发育和其他分析。QY,XW,ML和YG收集了叶片材料。ML,JS,XW和YW执行了实验。QY,WD和JS起草了稿件。作者阅读并批准了最终手稿。
相应的作者
伦理宣言
伦理批准并同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者们宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充信息
附加文件1:表S1。
64个阿片亚科叶绿体基因组的基因含量。
附加文件2:表S2。
对64份Apoideae样品进行SSR位点检测。
附加文件3:表S3。
24种当归(包括2个变种和2个品种)的取样资料。
附加文件4:表S4。
31种本倍异物种叶绿体基因组特征的比较。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
王敏,王晓,孙洁。et al。反向重复序列的系统基因组学和进化动力学当归塑料。BMC植物BIOL.21,26日(2021年)。https://doi.org/10.1186/s12870-020-02801-w
收到了:
接受:
发表:
关键字
- 当归
- 质体基因组进化
- 系统核糖组织
- 倒置重复