摘要
背景
奎奴亚藜(藜藜麦野生小麦是一种古老的粮食作物,它能耐受非生物胁迫,具有良好的营养特性。霜霉病是藜麦的主要病害,是由生物营养性卵菌感染引起的霜霉属摘要Gaum。由于该疾病造成重大产量损失,在遗传资源中识别霜霉病抗性来源并了解其遗传基础是藜麦育种的重要目标。
结果
我们对132个南美基因型、3个丹麦品种和杂草亲缘种进行了侵染c .专辑用一个单独的p .摘要在温室条件下观察到疾病特征的巨大变化,如感染的严重程度,从5%到83%不等。线性混合模型显示基因型对疾病性状有显著影响,遗传力为0.72 ~ 0.81。产地海拔、种子皂苷含量等因子与耐霉性不相关,但气孔宽度与感染严重程度呈弱相关。尽管基因型对耐霉性有很强的影响,但88个基因型的全基因组关联映射未能发现显著的标记-性状关联,表明耐霉性的多基因结构。
结论
对耐霉性的强大遗传效应使我们能够识别遗传资源,这是未来藜麦育种中宝贵的抗性来源。
背景
奎奴亚藜(藜藜麦野生)是一种谷物作物,在南美洲被驯化,从智利到南哥伦比亚被种植了数千年。1].西班牙人到来后,许多地区的玉米被欧洲作物所取代。2].最近,藜麦作为替代粮食作物在世界范围内重新引起了人们的兴趣,成为玻利维亚等国的重要出口商品。2014年,玻利维亚出口了约4万吨藜麦[3.].人们对藜麦的兴趣源于它的营养特性和对高盐、干旱和霜冻等非生物胁迫的耐受性[2,4].对藜麦日益增长的需求和其在本土以外的成功种植,导致了旨在提高产量、抗病性和对新种植区域或气候变化的适应能力的多个育种项目[5,6,7].植物病害易感性是限制作物产量的一个重要生物因素。霜霉病是藜麦最主要的病害,是由生物营养性卵菌引起的霜霉属摘要Gaüm,以前称为霜霉属farinosaf.sp。chenopodii[8].它导致耐受性品种产量损失高达30 - 50%,在高湿和缺乏化学防治措施的条件下,易感品种几乎完全减产[9].该疾病在种植藜麦的大陆上广泛传播,可能是由受病原体污染的种子传播的[10,11,12,13].p .摘要也感染密切相关和广泛的杂草c .专辑(被称为鹅脚、肥母鸡或羊羔角)[14,15,它可以充当辅助主机。c .专辑发生在整个欧洲,经常被霜霉引起的病原体感染,似乎与p .摘要感染c .藜麦.因此,c .专辑可以替代主持p .摘要并构成了这种病原体的宿主[8,16].其他杂草藜属植物,如c . murale有售(荨麻叶藜属植物),c . ambrosioides(印度脚病)也容易感染[17,18,19],但交叉感染c .藜麦目前还没有报道。由于该病是种子传播的,对这种病原体的耐受性是开发新藜麦品种的一个关键性状[20.].
目前,人们对其生理机制所知甚少p .摘要-藜麦相互作用,或关于霜霉病耐受性的遗传基础和其他表型性状在疾病易感性中的作用。以往利用温室实验、幼苗、离体叶片和田间评分对藜麦耐受性的研究主要侧重于通过评分疾病症状来定量测量[9,13,21,22].对霉菌感染的反应利用对疾病严重程度的视觉评分,这是由病原体引起的叶片组织损伤的比例[9].另一个衡量标准是病原体产生孢子的程度。通过离体叶分析法和叶片表面孢子体的鉴定对其进行测定[23].可靠、高效的霜霉病抗性评价是藜麦良种开发的关键。
本研究的目的是研究来自南美洲(玻利维亚、秘鲁、厄瓜多尔和智利)的藜麦基因型变异对霜霉病病原体接种的反应。我们研究了在受控条件下表型评分的稳健性,并描述了疾病性状、感染严重程度、产孢量和发病率与其他表型性状的关系。这些性状包括气孔的大小和密度,因为p .摘要通过气孔进入叶片组织[8,24],以及种子皂苷含量,因为皂苷提取物具有抗真菌的特性[25,26].我们估计的遗传变异成分和遗传力的反应p .摘要并对一个子集的全基因组序列进行全基因组关联研究(GWAS),以确定可能具有耐受基因的基因组区域。
结果
耐霉性变异高
在3个独立的温室试验中,共培养了132个基因型(5个对照品种,21个栽培品种,106个品系),并进行了霉菌接种、表型鉴定和评分。感染严重程度由5.0% (藜产孢率为0.2%(品种Puno) ~ 83.6%(品种CV21),平均42.6%。不同基因型间感染发生率差异较小,为36.8%(加位G41) ~ 92.0%(加位G92),平均为71.6%。
基于线性混合模型的霉菌耐受性分析
感染的严重程度和产孢量以比例表示。因此,我们拟合了LMM在Eq. (1),包括原始数据和用logit和angular函数转换的数据,这些函数经常用于比例。我们的目标是评估数据转换和包含对照品种对估计方差分量、遗传力和基因型平均值的影响。此外,为了验证方差分量的估计不受对照品种的影响,我们通过使用虚拟变量从模型中删除重复检验,来评估每个区块复制对照品种和未复制基因型的随机块设计的效果。这些参数的组合得到6个符合性状严重性和产孢量的LMMs1).在所有模型中,基因型被拟合为固定效应,以估计基因型均值并测试基因型对疾病性状的影响。REML比率检验表明,实验的异质误差方差结构提供了更好的模型拟合(p< 0.05),但单个模型除外1).
在残差直方图和q -图(补充图)中,未转换的感染严重程度数据没有明显偏离正态性。S2A和B)。另一方面,残差与拟合图显示沿x-轴表示方差的异质性(补充图S2C).变化的一个来源是实验(补充图。S2D),这与表中REML比率检验结果一致1.基因型对感染严重程度的固定影响Wald检验对所有6个拟合模型均高度显著,而无对照品种的检验显著较低p值(表2).
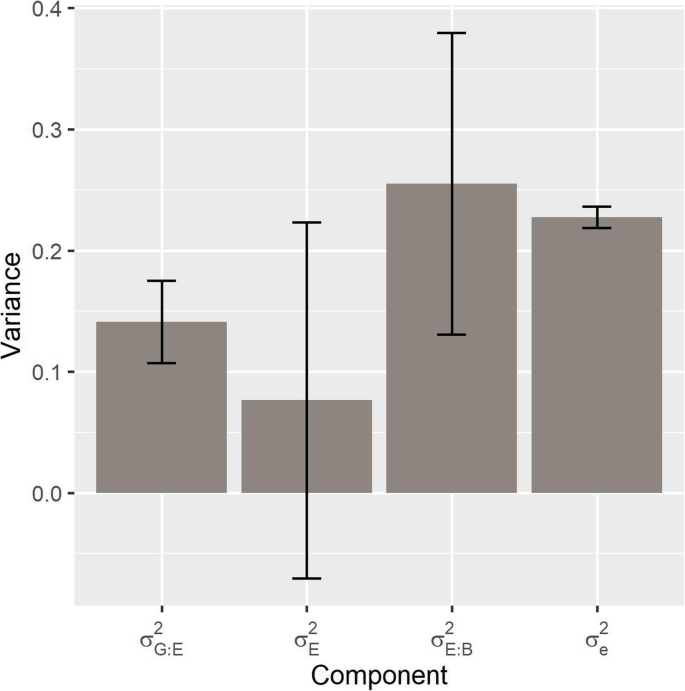
对方差分量的估计可以为变异来源建模,并解释实验设计的结构[27].对于感染的特征严重程度,各模型间的变异分量比例高度相似。我们得到了实验间方差的最高估计和置信区间(\({\σ}_E ^ 2 \))和基因型的实验互作方差(\({\σ}_ {G: E} ^ 2 \))组件。实验1 ~ 3方差分量的估计(\({\σ}_ {e1} ^ 2 \),\({\σ}_ {e2} ^ 2 \),\({\σ}_ {e3} ^ 2 \))和实验内嵌套块的方差(\({\σ}_ {E: B} ^ 2 \))则低得多(补充图。S3得了,补充表S2).在有和没有对照品种的模型比较中,估计的方差分量高度相似,因为95%置信区间重叠,但有对照品种的模型的方差比没有对照品种的模型的方差小(补充图。S3a - c)。
感染后性状产孢的结果与感染后的严重程度非常相似。固定效应Wald f检验在每一个以产孢为响应变量的拟合模型中均高度显著,这表明宿主的产孢基因型不同2).对照品种的去除明显减少p值。对于产孢,通过实验交互作用,每个模型中最大的方差成分是基因型,\({\σ}_ {E: B} ^ 2 \),但与感染严重程度相比,实验之间的方差估计(\({\σ}_E ^ 2 \))和实验内块的方差(\({\σ}_ {E: B} ^ 2 \))在所有模型的方差分量中最低(补充图。S3D-F)。综合这些分析为基因型对感染和产孢严重程度的影响提供了强有力的证据,这与数据转换和阻塞设计的影响有关。
发病率资料的广义线性混合模型分析
对于感染的特征发生率,我们使用GLMM,因为它允许拟合非正态分布的数据,如离散比例,并包括随机效应。我们使用一个分对数连接函数和假设实验之间的齐次方差,如条件皮尔逊残差所示,即没有迹象表明实验是需要解释的变化来源(补充图。S4).在两个因纳入和排除对照品种而不同的glmm中,使用发病率作为响应变量,两个模型的固定效应检验均显著(p< 0.001)。实验确定基因型\({\σ}_ {G: E} ^ 2 \)模型中控制品种的方差分量较大(图2)。1).反映实验设计的方差分量,\({\σ}_E ^ 2 \)而且\({\σ}_ {E: B} ^ 2 \),两个模型的标准误差最大。另外,在无对照品种的GLMM上进行了试验\({\σ}_E ^ 2 \)方差为零,表明两者之间具有可比性。无论使用哪种模型,后一个方差分量和残差方差分量都是最大的。综上所述,与其他两个疾病性状一样,感染发生率也显示出基因型对性状变异的强烈影响。
特征之间的相关性
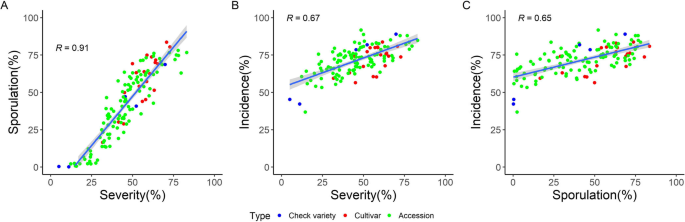
三种疾病性状相似的变异分量结构(图。1和补充图。S3)表明它们是相关的。感染严重程度和产孢量的性状调整平均值与对照品种高度相关(R= 0.91,p< 0.001;无花果。2)和不包括对照品种(R= 0.9,p< 0.001)。霉菌发病率与严重程度和产孢量的相关性明显较低(R= 0.67,R分别为= 0.65,p< 0.001)。
遗传力分析
我们还估计了遗传力,\(\眉题H ^ {2} \),并评估了数据转换对这些估计的影响。差异的平均标准误差(s.e.d),用来衡量在每个模型中两两比较的精度,和遗传方差,当基因型作为随机效应拟合时估计,都是\(\眉题H ^ {2} \)(Eq。3.))。这两个参数在有和没有对照品种的模型之间都有小幅增加,我们观察到所有数据转换的这种差异(表2).较高的平均s.e.d值表明,删除对照品种降低了两两比较的精度。结果是,\(\眉题H ^ {2} \)在没有对照品种或有反正弦根变换的模型中的估计结果分别略高于有对照品种或有其他变换的模型的估计(表2).综上所述,数据变换和排除重复对照品种对的估计影响不大\(\眉题H ^ {2} \),因为感染严重程度的遗传力估计值保持在0.72至0.78的狭窄范围内,孢产率的遗传力估计值在0.78至0.82的所有分析模型范围内。在有对照品种的GLMM模型中,估计遗传力为0.40,遗传方差为0.10,平均s.e.d为0.55,无对照品种的GLMM模型为0.08和0.48。
对霜霉病感染反应的基因型排序
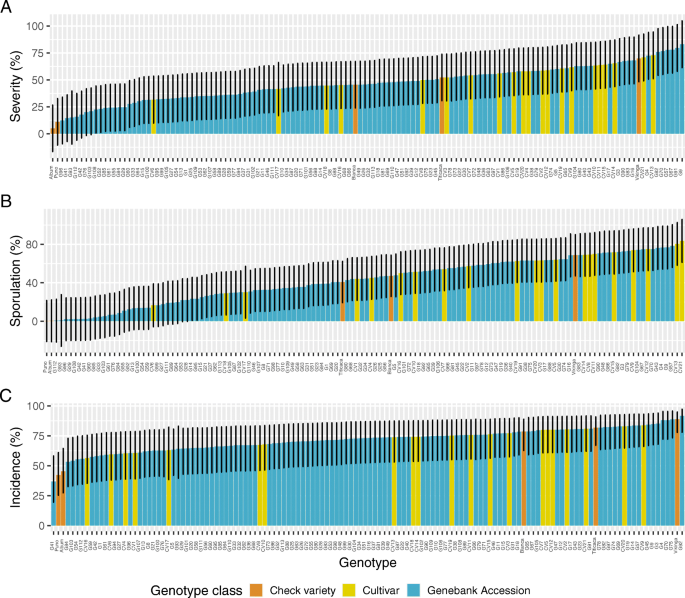
基因型对三种病原性状的强烈影响表明,在我们的样本中,基因库的来源和品种在耐霉性方面是高度可变的。因此,我们比较了所有三个性状的均值,并确定了基因型之间的实质性差异。在未转换数据和对照品种的LMM中,感染严重程度的调整平均值范围从5%到83%,在其他模型中显示出非常相似的范围。没有对照品种的模型导致该性状的范围较小,因为有两个对照品种c .专辑普诺对严重程度的估计最低。3.a).我们没有观察到对照品种和数据转换类型对感染严重程度的基因型排序有很强的影响。因此,该性状的基因型之间的差异是明显的,并允许识别耐受性和易感性基因型。控制品种c .专辑普诺和基因库品系G41、G42、G76、G93、G96和G112的耐受性最强,而对照品种Vikinga、CV13和CV21以及品系G4、G9、G57、G67、G82和G91的耐受性最强(图1)。3.一个)。
基因型与产孢有相似的模式。未转化产孢率的调整平均值(包括对照品种)在0.2% (Puno)到83.6% (CV21)之间。产孢数据的转换对估计平均值的分布有很小的影响,独立于是否包括或排除对照品种(图。3.b).产孢量最低的基因型为对照品种c .专辑产孢量最高的基因型为对照品种Vikinga、栽培品种CV12、CV13和CV21以及栽培品种G4、G9、G43、G67、G70和G104(图1)。3.b)。
该性状发病率的基因型变异低于其他两种疾病性状(图2)。3.c).加入或排除对照品种对调整平均值影响不大。发病率低的基因型包括对照品种普诺、c .专辑和加入G41;而对照品种Vikinga和基因型G75和G92的发病率最高(图2)。3.c)。
疾病性状与基因库护照数据和种子皂苷含量的关系
实验中所包含的基因型材料是利用护照数据中关于霉菌耐受性的信息进行筛选的,以获得一组该性状具有多态性的材料。在基因库访问的护照数据中记录的严重程度和发病率数据与我们的结果高度不一致。例如,106次访问中有35次被记录在护照数据的0%严重性类别下,但没有一次在我们的分析中被归类为此类;0.1 ~ 25%组16例,本研究组14例;26个词条占26 - 50%,而我们的数据集为56个词条。根据护照数据,26份护照属于最易受影响的类别(75-100%),而在本研究中只有6份护照。
疾病与气孔性状的唯一显著相关性是气孔的严重程度与宽度(r= 0.18,p= 0.041)。我们还测试了种子中皂素含量是否与疾病易感性相关,并对在两个地点收获的种子进行了泡沫试验,因为皂素含量在物候阶段和环境中不同。105个基因型的种子可从玻利维亚和丹麦两个地点获得,而26个基因型仅在丹麦收获。泡沫高度测量在种子来源之间没有相关性(皮尔逊的r= 0.16,p= 0.11)。在有皂苷和无皂苷的基因型的估计平均严重程度和产孢量的比较中,我们没有观察到任何基于种子来源的系统模式,也没有观察到如果分别比较品种和品种(t以及与p> 0.05)。
隔离p .摘要从c .专辑和相互传染c .藜麦
的隔离p .摘要本研究中使用的原分离物c .专辑然后进行植物性繁殖c .藜麦(简历。布兰卡和Vikinga)。孢子从这些植物中收获并接种土荆芥小孢子菌病情严重程度低(4%),产孢量最低(0.4%)c .专辑相比c .藜麦基因型(无花果。3.).这是第一次交叉感染c .专辑已经被报道。用于验证分离物的ITS DNA序列的BLAST比较显示与从c .藜麦简历。2001年收集的地图集(EU 113305)。
全基因组测序
鉴于对霉菌感染的反应具有高度显著的基因型效应,我们对代表表型变异范围的88个品种和品种的子集进行了测序,以进行GWAS。序列基因型包括2个对照品种(Puno和Titicaca), 18个栽培品种,68个基因库,其中39个来自玻利维亚,19个来自秘鲁,2个来自厄瓜多尔,1个来自智利。测序基因型的平均严重程度在11%到83%之间,平均为47%。
88个样本测序结果为7.9 × 1011Bp在2.6 × 109读取长度为150bp的对。将处理后的序列reads映射到藜麦参考基因组版本1后,每个基因型的序列覆盖率在0.38 X(对照品种Titicaca)到9.17 X (Accession G37)之间,平均为3.24 X。每个样品的测序reads比例在99.3%到99.9%之间,平均为99.8%。与核基因组相比,我们的样本中叶绿体和线粒体基因组的比例过高,分别为109 X和32.6 X。如此高的覆盖率是意料之中的,因为每个单倍体核基因组都有多个线粒体和质体基因组的副本。
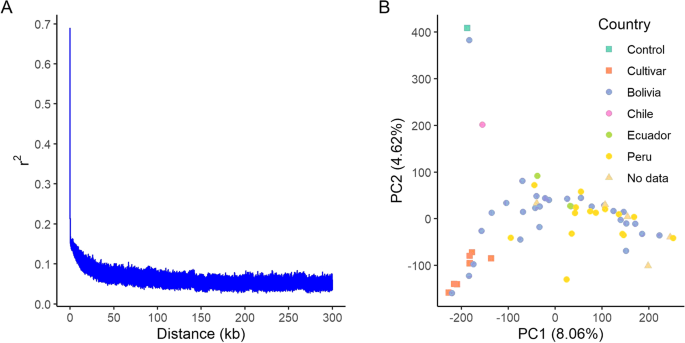
变异呼叫GATK在基因组中鉴定出18,017,831个双等位snp。在对小等位基因频率和样本缺失进行过滤,并对偏离Hardy-Weinberg平衡进行测试后,仍有4,131,562个变体。我们对覆盖<8 X,样本和位置缺失<0.7的基因型进行了归一化,从61个样本中获得606,791个SNPs,基于5000个隐藏标记估计准确率为96.9%。无花果。4A表示连杆不平衡,用r2,在测序的基因型中22 ~ 25kb范围内降至0.1。
主成分分析(PCA)表明,所有被分类为栽培品种的基因型聚集在一起(图。4B,左下角)。对照品种Puno和G71(原产于玻利维亚)从其他基因型中分离出来。入世G101也脱离了主要类群,起源于智利,表明它是沿海生态型。加入G42,似乎与主组分离(图。4b,中下),严重程度和产孢量都很低,与普诺品种相当。其余来自玻利维亚、秘鲁和厄瓜多尔的品种是混合的,不形成明显的群体。
霜霉病严重程度关联映射
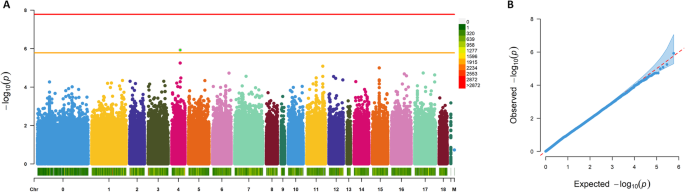
我们进行了两种不同的GWAS分析,以检测与霜霉病感染严重程度相关的基因组区域。由于严重程度与产孢量高度相关,我们只对两个性状中的第一个性状进行了GWAS。对61个FarmCPU基因型中603,871个SNPs的分析没有发现与平均严重程度有统计学意义的相关性,当模型与主成分(pc)拟合时(图)。5a和补充图。S5一个,分别)。4号染色体上的单个变异(S04_33782670)位于阈值(1.656 × 10)以上−06),未使用主成分对种群结构进行校正(图。5).QQ图显示没有通货膨胀或通货紧缩的迹象p-相对于理论期望的值(图。5b和补充图。S5B,分别),因此支持不存在显著关联。
我们还使用了k基于-mer的方法,因为它允许包含较低序列覆盖率的附加基因型(n= 88)并且不偏向于包含在参考序列中的基因组区域。这一分析是基于平均570,741,731k-每个样本长度为31的聚合物。对照品种“地的卡卡”(67,365,628)和基因库供试品种G37(814,316,239)的基因型数量最少和最多k分别即计数。总的来说,992946 .265k-mers通过过滤器,并被纳入亲属矩阵估计和后续的GWAS。GWAS第一阶段为880,137,481人k-mer进行了测试,10,001个通过了第一个过滤器,以适合GEMMA。最小的p值为任何k-mer为9.19 × 10−10为一个k- m。因此,该分析也没有检测到任何与感染严重性特征的显著关联,以排列为基础的5%p- 1.505 × 10的阈值−10为k- m分析。
讨论
用霜霉病病原菌分离物接种藜麦品种和基因库资料霜霉属摘要揭示了三个感染相关性状的显著差异,感染的严重程度,产孢量和霉菌发病率(图。3.).使用混合模型方法,我们验证了遗传效应、方差分量和遗传力的估计在数据转换和实验之间包含或排除对照品种方面是稳健的。霉菌感染易感性的差异有很强的遗传成分,正如高遗传方差成分估计和高遗传力所示,但我们无法在GWAS中确定与霉菌易感性强烈相关的单个基因组区域。在以往的研究中,生态类型、环境或生理参数,如产地海拔、种子皂苷含量或气孔的大小和密度,被假设与耐病性相关。除了气孔宽度外,我们没有发现三种疾病性状与任何这些参数的强相关性。
耐霉性遗传效应大
在选材和栽培品种的情况下,对每个区块的单一植物进行耐霉性评分,而对照品种有多个植物,因为一个人对所有植物的每个叶子进行评分,而且劳动密集型过程有评分窗口的时间限制(max。12 h)。为了评估参数估计的鲁棒性,我们使用线性混合模型和数据转换的各种组合以及包含或排除控制品种。尽管数据转换的使用仍在争论中[28,29,30.],它对残差分布或固定效应检验没有很大影响。这为我们估计的稳健性提供了强有力的证据,因为我们对每个基因型的植物表型数量大致相同,这减少了异方差的影响[31],以及从LMMs的鲁棒性到异质误差方差[32].三次实验中重复对照品种对模型拟合和参数估计的影响较小,因为它们的去除只造成了很小的增加\(\眉题H ^ {2} \)估计。这种鲁棒性源于平衡的实验设计,其中差异的变化很小[33].
拟合LMM和GLMM的一个限制是,对随机效应方差的可靠估计至少需要五个层次,即位置、实验、年份等。[27].在我们的实验中组数少解释了设计方差分量的大置信区间\({\σ}_E ^ 2 \)而且\({\σ}_ {E: B} ^ 2 \)(补充表S2而且S3),因此这并不是实验或区块之间变化的可靠估计。尽管如此,我们还是将这些影响建模为随机的,以使用块间信息并解释数据的非独立性,因为基因型嵌套在块中,在实验中嵌套[27,34].
之前关于藜麦产量性状的研究发现,基因型-环境变异成分的估值偏高[35].基因型-实验互作方差(\({\σ}_ {G: E} ^ 2 \))在我们的研究中,与跨性状和模型的其他方差成分相比也很大,这反映出即使是环境的细微差异也会导致基因型对疾病的不同反应。从植物育种的角度来看\({\σ}_ {G: E} ^ 2 \)变异成分很重要,因为它掩盖了表型变异的基因型成分。观察到的高\({\σ}_ {G: E} ^ 2 \)建议未来使用不同藜麦基因型的耐霉性研究应包括多个地点和年份[36],或使用复制对照品种进行不平衡设计,从而可以测试许多基因型[37].
疾病相关性状之间的高度相关性
为了识别宿主植物的不同反应,我们测量了三种不同的与疾病相关的性状。比较感染和产孢的严重程度是有意义的,因为病原体群体通常在毒力和繁殖力方面都有高水平的遗传变异[38,39].一些藜麦宿主基因型可能允许病原体快速结实,而另一些可能抑制其增殖[40].我们发现严重程度和产孢率之间有很强的正相关关系,前者的得分似乎是后者的一个很好的预测指标。由于这个原因,单独的严重程度可以用来评估一个基因型组,因为它也显示出比任何一种孢芽孢都更小的误差方差(图。S3D-F)或发生率(图。1).本研究疾病性状的相关系数(R= 0.9)大于黄瓜霜霉病评分方法评价中报道的系数[41].然而,应该在感染严重程度<5%的高耐药基因型中测量产孢,因为在这样的遗传背景下,正如在野生亲缘中观察到的那样,病原体的增殖可能被强烈削弱c .专辑,对照品种普诺和多个基因库资料。
遗传力分析
与遗传方差的估计一致,模型之间的遗传力估计中等高,非常相似,严重程度的范围从0.72到0.78,产孢率的范围从0.78到0.81。相比之下,估计葡萄霜霉病产孢的遗传力(葡萄)约0.40 [42],玉米对高粱霜霉病系统侵染的抗性在0.61 ~ 0.68之间[43].虽然估计\(\眉题H ^ {2} \)温室试验可能不同于田间试验[44],疾病性状的高遗传力表明有可能选择较高的耐霉性。在这方面,我们的结果与以前的估计一致\(\眉题H ^ {2} \)生理性状、形态性状和产量性状也较高(≈0.85)[45],表明通过植物育种可以显著改善藜麦的多个性状。
对霜霉病耐受性的鉴定
尽管我们的研究小组包括主要的藜麦生态类型[46],升高与感染或产孢的严重程度无相关性。由于感染需要较高的湿度(图。8),缺乏这样的相关性反映了高海拔地区湿度的小气候变化。例如,玻利维亚高原(Altiplano)北部比南部更潮湿。47].此外,来自安第斯山谷(2500-3500 masl)的生态类型,湿度也往往较高,是最耐受性的品种之一[47,48,49].这些观察结果与我们的结果一致,因为来自南部高原的5个品种感染的严重程度最高(来自玻利维亚的G4、G9和G82,以及来自秘鲁的G67和G99)。
我们观察到,根据其耐霉性对基因型进行排序,在疾病性状和分析方法方面是稳健的,这允许确定高耐受性的品系,以便进一步调查或在育种中利用。耐受性基因型包括野生亲缘c .专辑普诺对照品种是霜霉病严重程度、产孢量和发病率最低的基因型之一(图1)。3.a-c),以及一套玻利维亚品种和基因库资料。例如,栽培品种Mañiqueña (CV21)和Phisankalla (CV10)在玻利维亚南部Altiplano等干旱地区表现良好,但在潮湿环境中容易霉变[3.,7,48].这种表型在我们的研究中得到了证实,因为这两个品种在我们实验的潮湿条件下都是最敏感的(图。3.).有人提出,在干燥环境中表现良好而在潮湿环境中具有高疾病易感性的基因型,要么在驯化过程中没有被选择用于抗病,要么在干燥环境中具有优势,这可能是由于抗性成本[47].目前的证据是矛盾的,并没有建立这种关系,因为栽培品种“Rosa Blanca”(CV6)是在干旱地区培养的[3.],但对该病害的耐受性较高,平均严重程度为32%,而更敏感的品种' Jach ' a Grano ' (CV15;64%的严重程度)和“Aynoka”(CV20;严重程度58%)源自同一育种计划。如此高的变异可能是基因型与实验相互作用的结果。S3),并与相似的GxE相互作用解剖和产量相关性状一致c .藜麦[45,50].
我们研究的一个重要局限性是只使用了单一的分离物p .摘要对于接种,因为它不允许测试耐霉性是种族特异性的还是反映数量抗性。初步证据支持后一种假设,因为栽培品种Kurmi (CV16)和Mañiqueña Real (CV21)接种了玻利维亚分离的p .摘要根据霜霉病的进展情况,分别分为耐受性和易感性p .摘要[51].我们的结果证实了这两个品种与不同的分离株之间的差异,因为Kurmi(45.1%的感染严重程度)比Maniqueña(70.7%)更不敏感(图1)。3.).由于库尔米被开发用于高原(3600-3800 masl)栽培,并在抗霜霉病的田间试验中被选中[52],库尔米对两个不同分离株的高耐受性支持定量的疾病耐受性。我们的结果也支持先前的研究,发现重组自交系来自智利和秘鲁的分离,在一个F2:6人口(23].
护照数据与我们的结果的比较由于缺少评分方法、植物表型阶段、材料的遗传组成(如杂合程度)和玻利维亚田间试验的信息而受到限制。此外,我们关于感染严重程度的数据是基于一种致病分离物,而护照数据是基于当地种族的自然感染,其毒性可能与本研究中使用的丹麦分离物不同[14].以前的研究表明,当使用高度敏感的多态性鉴定方法时,安第斯分离株在遗传上是不同的[14].PCR测序结果表明,该分离物与丹麦Tåstrup采集的标本EU 113305在ITS区序列完全相同c .藜麦简历。2001年地图集[8].
病害性状与气孔性状和种子皂苷含量的关系
p .摘要通过气孔进入宿主组织,吸器从气孔孔中出来释放孢子[8,24],这可能解释了病原体对潮湿环境的偏好,因为在这种条件下气孔通常是打开的[53].因此,我们测试了病原体性状是否与气孔性状相关,发现只有感染的严重程度与气孔宽度呈弱相关。这一结果应该谨慎对待,因为我们的测量仅基于每个基因型的一片叶子,因为获取数据需要付出很高的努力。未来的表型分型应该利用自动图像分析方法来获得更大的气孔性状数据集。
霜霉病在种子的果皮中保持休眠状态[54种子的皂苷含量可影响感染早期基因型的反应。但是,我们没有发现种子皂苷含量与霉病严重程度之间的关系。对缺乏相关性的一种解释可能是泡沫试验揭示了GxE效应,因为从不同地区种植的植物中获得的相同基因型种子样品中皂角苷含量的得分不同。这是意料之中的,因为皂角苷的含量随时间的推移而变化,并取决于植物的水分状况[55,56].
因此,我们的研究结果不支持气孔特性或皂苷含量等其他性状对霉菌耐受性有显著影响的假设。
霜霉病严重程度的GWAS解释
我们根据霉菌感染的严重程度和全基因组重测序数据进行GWAS,以检验观察到的基因型之间的差异是否由少数基因组区域引起。两种GWAS方法都未能检测到显著的变异或关联k-感染严重的中东呼吸综合征。GWAS的能力取决于关联面板的样本量和感兴趣性状的遗传结构[57].我们的分析受限于61 (FarmCPU)和88 (k由于表型值的分布表明这是一种多基因数量性状(图1)。3.a)藜麦对p .摘要包括温室试验和田间试验在内的多项研究都支持感染[13,23,40,58,59,60,61].
我们的研究结果为藜麦更有效的抗性育种提供了思路。耐霉性的大变异和疾病性状的高遗传力使得通过跨越分布两端的基因型(例如,丹麦品种Puno和Titicaca;无花果。3.).先前的工作表明,QTL定位可能识别出主要的R基因,这些基因可能在藜麦育种中有用,因为霉抗性是由不完全基因效应调节的[62],这取决于病原体的侵袭性,正如在厄瓜多尔材料上观察到的那样[21].此外,研究结果还表明,各菌株霉病严重程度的分离比F2从苦和甜(即不含种子皂苷)基因型的杂交中提取的群体图谱显示,霉菌耐受性显示显性遗传[63].
通过更多的高通量表型分型方法,对耐霉性遗传变异的分析和利用将得到加强。我们的实验建立在单一分离物的靶向接种上,这有助于对耐受性进行稳健和可重复的估计,但这是一项密集的工作,并限制了用于遗传定位的基因型的数量。然而,对离体叶片进行评分或随机选择田间叶片等替代方法可能会产生误导,因为田间宿主基因型x病原体基因型x环境(GxGxE)的影响难以控制。此外,病原体感染的症状受叶片组织的位置和年龄的影响[64,这是由于诱导抗性不仅发生在最初的感染部位,而且也发生在远端未感染部位[65].因此,除了本研究中使用的受控温室实验外,利用深度学习等现代表现型技术对病原体感染进行评分的分离种群的多位置田间试验是抗性育种的一种补充方法[66].这两种方法与遗传分析相结合,将有助于在本地种植范围内外开发改良的藜麦品种。
结论
我们的研究揭示了奎奴亚藜品种和品种的高度变异霜霉属摘要感染。我们已经证明交叉感染来自c .专辑来c .藜麦反之亦然是可行的这种广泛分布的杂草很可能是病原体的蓄水池也是病原体的备用宿主p .摘要,这对藜麦种植有影响c .专辑.耐霉性在基因型之间的显著差异具有很强的遗传成分,因此在育种计划中是适合选择的。然而,基于单一实验-或单一位置田间试验-的推论应该谨慎,因为通过实验相互作用发现了一个大的基因型,所以未来的工作是关于抗c .藜麦来p .摘要必须在设计和规划阶段考虑到这一点。
方法
植物材料
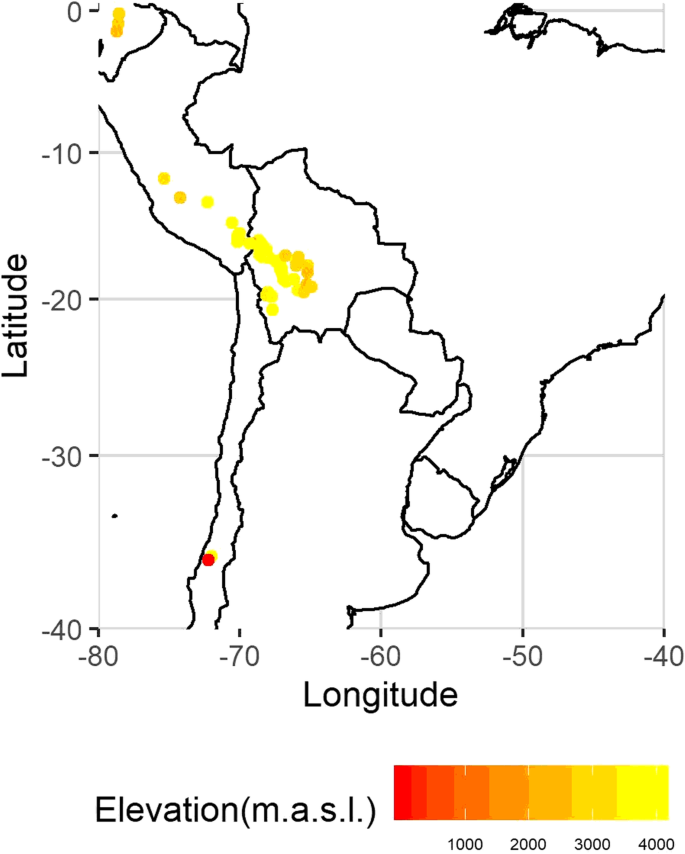
本研究分析的藜麦基因型由玻利维亚国家种质银行储存的106个品种组成。它们包括在玻利维亚(55个)、秘鲁(33个)、厄瓜多尔(7个)和智利(4个)收集的地方物种,海拔从2海里/平方英尺不等。4082 m.a.s.l。(无花果。6).有七件藏品没有来源信息。我们还包括21个玻利维亚品种,玻利维亚品种“布兰卡”(“Blanquita”)和三个丹麦品种“普诺”,“Vikinga”和“提提卡”。入选名单及其护照资料载于补充文件1(来源:http://germoplasma.iniaf.gob.bo).
根据护照数据,按海拔分布的南美种质库。地理坐标来源:玻利维亚国家种质库(http://germoplasma.iniaf.gob.bo).映射是用R包创建的mapdata该数据库使用CIA世界数据库II的数据坐标
选择的基因库既代表藜麦的地理多样性,又代表耐霉性的变异,这些变异是在拉巴斯(玻利维亚)的田间试验中根据自发感染进行评分的p .摘要.从护照数据中无法获得附加信息,包括遗传状态、评分方法、评分时的物候阶段和试验地点。
四种藜麦品种和野生相对常见藜麦(藜L。)作为温室试验的对照品种。这四个藜麦品种包括“Blanca”品种,它适应于玻利维亚的北部高地和安第斯间山谷,并部分抵抗霜霉病[3.,52].另外三个对照品种“提提卡”、“普诺”和“维金加”是在哥本哈根大学的藜麦育种项目中开发的。品种Puno (KVL 37)和Titicaca是从智利和秘鲁的地方品种中育种而来,并选择了早熟和适应欧洲条件的品种[67,68].他们在田间试验中表现出不同水平的霜霉病易感性(s.e。Jacobsen,个人沟通),这在本研究的一个试点实验中得到了证实(补充图。S1).藜与藜麦密切相关,是一种分布广泛的杂草。c .专辑本研究中使用的种子于2017年和2018年在哥本哈根大学理学院试验站的一个前藜麦育种场(Højibakkegaard, Tåstrup)收集。
对这些植物的实验研究,包括植物材料的收集,完全符合机构、国家和国际的指导方针。根据当地法例进行温室研究。玻利维亚国家种子研究所(iniafi -玻利维亚)获准将玻利维亚基因库的种子材料用于本研究的唯一目的,但有一项谅解,即所有种子将在项目完成后销毁。丹麦品种的种子是由QuinoaQuality Aps公司作为礼物提供的。
接种用变异庇诺孢菌分离株
先前的研究确认了交替宿主在病原类型进化和传播中的作用[69]p .摘要两者都是病原体吗c .藜麦而且c .专辑[8,14,15,18,20.].得到…的确定的分离p .摘要,离开C.album2018年9月下旬,在Højibakkegaard研究站的一个前藜麦养殖场收集了典型的霜霉孢子。利用Danielsen和Ames的协议,将分离株接种到两个藜麦品种(Blanca和Vikinga)中进行维持和繁殖[48].我们使用这两个品种,因为它们的潜伏期不同。40].Vikinga品种的潜伏期为5 d, Blanca品种的潜伏期为7-10 d。这些差异使得病原体在布兰卡上得以保持,并在Vikinga变种上快速繁殖。
我们使用内部转录间隔区(ITS)的DNA测序来确认分离物是p .摘要.孢子悬浮液(1 × 1011孢子/ml)从维持的接种体产生用尼龙过滤器过滤20μm孔径(Merck Millipore Ltd.)捕获p .摘要孢子被转移到1.5 ml含有玻璃微珠的微离心管中(425-600μM)并放在冰上。200μl加入裂解缓冲液(DNAeasy Plant Mini Kit, Qiagen),用无菌研棒将菌丝粉碎。另外200μL的裂解缓冲液与4μl的RNase。按照制造商的说明,用DNeasy Plant Mini Kit (Qiagen)提取DNA。引物设计用于扩增一个1150 bp的覆盖ITS-1和ITS-2区域的卵菌科成员,包括Peronospora, Pythium和Phytophthora的片段,通过聚合酶链反应(PCR)从基因组DNA中扩增,分别使用Oomyc fw1: 5 ' cggaaggatcattaccacac和Oomyc rv1: 5 ' cgcttattgatatgcttaagttca作为正向引物和反向引物。PCR扩增1个周期,95C, 3 min;35次94C 30秒,55C 30秒,72C 40秒,72C 1次3分钟。扩增产物用QIAquick PCR纯化柱(Qiagen)纯化,用NanoDrop Lite分光光度计测定DNA浓度。PCR扩增ITS的DNA测序在Eurofins Genomics进行。DNA序列提交到NCBI(登录MT895880),并使用BLASTN (https://blast.ncbi.nlm.nih.gov).
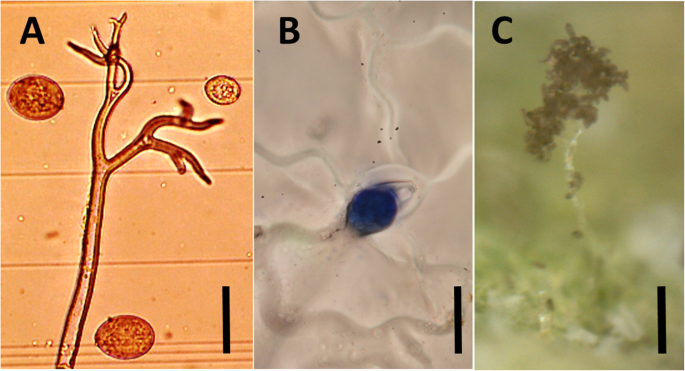
的识别p .摘要通过显微镜和组织病理学
通过显微镜和组织病理学切片的视觉分析也证实了病原体的身份。用样品进行了显微分析霜霉属溶液安装在玻片上。组织病理学切片取材于±5cm的布兰卡叶片2在感染后7天收集。叶片样品背面涂指甲油,晾干24 h。然后去除印迹,用乳酸酚苯胺蓝染色[70].显微镜和组织病理学准备均安装在徕卡MZ12.5显微镜上,用徕卡DFC420相机在40倍放大下拍照(图1)。7a, b)。此外,用数码显微镜(Dino-Lite,型号AM4113/AD4113 Dino-Lite,荷兰纳尔登)捕获活感染(图1)。7c).经过病原验证和校准后,分离物不断繁殖在足底维金加和布兰卡病原体分离和传播的详细规程见补充文件2.
表型表征的设计
表型数据于2019年2月至5月在哥本哈根大学的温室中收集。采用3个序列相同的试验评价了藜麦基因型对霜霉病接种的响应,每个试验包括132个基因型的完整集和4个对照品种,每个试验采用随机完全区组设计,每个区组4个。每个实验都占用一个专门用于实验的温室,以避免昆虫侵扰或交叉污染的风险,并提供生物防治剂。实验在前一个实验结束后2周开始。在区块内,品系和品种由一株植物代表,而对照品种由2 - 5株植物代表。
在实验之前,由于基因库的杂合度未知,玻利维亚的基因库材料进行了一次自花授粉以增加纯合度。2017年2月至8月在光周期为12 h、白天平均温度24℃、夜间平均温度18℃的温室内育苗,施肥水(NPK 14-3-23 + mg EC 1.9)灌溉。日照长度超过植物要求后,采用温室帘保持光周期。种子在自然条件下收获、清洗和储存,直到2018年1月。收获的种子被播种在含有泥炭的小罐中,以确保移植植株的供应,并生长7到10天。为了避免苍蝇的侵扰,堆肥用蚊蚋醇溶液(10% v/v)浇灌。然后将植株移栽到550厘米3.在同样的条件下在温室里种植3周。温室管理包括生物防治剂,以防治温室中常见的害虫。
移栽3周后,将植株移栽到不同的温室,用校准的溶液(1 × 105孢子/毫升)的p .摘要孢子和Tween 20(1%)。用高压喷枪将溶液全面喷到每一株植物上。每块用50ml溶液。接种后5天用塑料布覆盖接种过的植物块(图。8a)在完全黑暗和夜间温度为15摄氏度的条件下持续24小时,以创造刺激感染的条件。除去覆盖物后,植物在温室条件下生长。一旦观察到症状(图。8B),通常在接种后5 ~ 6天,再次覆盖植株24 h,以促进孢子生成。
植物对病原体感染的反应用三个变量来衡量:严重程度、产孢量和发病率。分别通过目视分析叶片正面被褪绿或其他颜色病变覆盖的叶面积和叶片背面可见孢子的病变组织面积,对严重程度和产孢情况进行评分。测量结果被记录为每片叶子的百分比,然后平均每株植物。发病率以有症状的叶子的比例计算。这些参数由同一个人测量,以避免操作偏差,所有的植物在评分时具有相同的年龄。
表现型的气孔
为了测量叶片表面气孔的宽度、长度和密度,我们制作了每个基因型叶片背面的树脂铸型[71].干燥后,在石膏上添加一层指甲油,晾干,取下,安装在玻璃载玻片上,用玻璃盖盖。载玻片安装在徕卡MZ12.5光学显微镜上,使用徕卡DFC420数码显微镜相机40倍放大拍摄三个视野。宽度和长度使用徕卡应用程序套件软件测量。气孔密度是通过每单位面积的气孔数量估算出来的,气孔计数是通过使用基于web的应用程序气孔计数器(stomata Counter)获得的,然后手动管理数据[72].
用线性混合模型(LMM)分析表型数据
使用ASREML-R软件包3.0版本,使用以下混合模型来估计面板中每种基因型的疾病的平均严重程度和产孢量[73]:
在哪里yijk是否有反应(严重程度或产孢)k-第th基因型j的第-块我th实验,μ是一般平均值,ρ我效果是我th实验,βij效果是j嵌套在我th实验,αk是基因型效应,ραij基因型-实验相互作用吗eijk是残差项。由于实验被认为是一个随机因素,因此所有涉及随机因素的影响都需要建模为随机,因此实验的影响、实验中的块以及基因型-实验相互作用被视为随机效应[74],而基因型的主要影响被视为固定的。为了避免异常值对遗传方差估计的影响,在拟合模型后检测异常值,并使用PLABSTAT包的默认方法从数据集中删除异常值[75],如在[76].
实验的残差最初建模为正态分布,独立于一个共同的方差分量,其中n是观察的总数。此外,我们拟合了误差向量(经实验排序)的方差-协方差矩阵为的模型\(R\sim N\左(0,{\sigma}_e^2{I}_n\右)\),在那里n是观察的总数。此外,我们为每个实验拟合了一个具有独立方差分量的模型[77]:
在哪里\ ({\ oplus} _ {j = 1} ^ {R} _j \)和的直接和是R1,R2而且R3.每个实验的方差-协方差结构都是这样的吗\({R}_j={\varSigma}_e^2(j)\ast {I}_{nj} \),在那里新泽西观察的次数是多少j−th实验。
广义线性混合模型分析
我们采用广义线性混合模型(GLMM)对藜麦霜霉病的发病率进行分析,并利用SAS软件的PROC GLIMMIX程序拟合如下模型:
其中logit是线性预测器和观测值之间的链接函数(pijk),ρ我效果是我th实验,βij效果是j嵌套在我th实验,αk基因型效应,和ρα本土知识是基因型-实验的相互作用。该模型通过SAS 9.0版本中PROC GLIMMIX的RANDOM语句中的residual关键字包含了一个scale参数来解释数据的过度分散[78].
遗传评估
特设广义遗传力估计为:
在哪里\({\σ}_g ^ 2 \)基因变异和\(\眉题{\ upsilon} \)是调整后均值之差的平均方差[33].估计\({\眉题{H}} ^ 2 \),模型与基因型进行随机拟合,使用ASREML-R包对严重程度和产孢情况进行拟合,使用PROC GLIMMIX对SAS的发病率进行拟合,以获得估计\({\σ}_g ^ 2 \).采用基因型为固定效应的模型进行估计\(\眉题{\ upsilon} \).
模型的比较
为了比较具有不同误差方差结构的模型,在asremlPlus R包中实现的受限似然比检验[79]用于检验异质误差方差是否改善了模型。通过在感染严重程度、产孢和发病率模型中添加一个虚拟变量,研究了重复对照品种对遗传方差估计的影响[80].这样一个模型被表述为
在哪里Y而且W为向量,0为参考品种,1为栽培品种和品系,αk是基因随机效应。其余的效果与方程式相同。(1)和(2).用平均标准差(s.e.d)和遗传力对这些模型进行比较。使用asremlPlus R包中的predictplus函数计算s.e.d值[79].
通过使用logit转换数据重复上述步骤来评估转换感染严重性和孢芽孢评分对遗传力估计的影响左(\ \ (\ mathit日志}{\ \离开[\压裂{p} {1 - p} \右]\)\)角变换,或者说反正弦根变换\ \离开({\罪}^{1}\离开[正确\ sqrt {p} \] \) \),在那里p是否观察感染的严重程度或产孢情况。因为logit函数在0或1处未定义,所以通过对原始值加减0.025来调整这些极限处的数据。基因型的固定效应通过ASREML R包中实现的Wald’s f检验和SAS的Proc GLIMMIX程序固定效应的II型检验进行检验。
对比方法
各基因型霜霉病感染的平均严重程度和产孢量,它们的置信区间和所有两两比较都用asremlPlus R包估计[79的严重性和产孢情况,以及SAS的PROC GLIMMIX程序的发病率。平均数之间的比较是基于t-具有显著性阈值的测试α= 0.05。
特征之间的相关性
为了确定表型性状和藜麦抗霉性相关性状之间的相关性,即严重程度、产孢量和发病率,我们使用了气孔(宽度、长度和密度)测量数据。对具有显著性阈值的每对变量估计Pearson相关系数α= 0.05,使用R包Hmisc [81].
皂苷含量与霜霉病严重程度的关系
用泡沫法测定种子皂苷含量[82,将0.5克种子和5毫升蒸馏水放在试管中,用力摇晃30秒。在摇晃后,泡沫高度记录到最近的0.1厘米。为了评估该皂苷分析的稳健性,对每个基因库的两个种子样本进行了评估,一个来自生长在玻利维亚的植物,一个来自Højbakkegaard繁殖的植物。所有读数为零(即摇晃后没有观察到泡沫)的原料都被标记为“不含皂角苷”,所有其他原料都被标记为“含皂角苷”。为了测试皂苷的存在或缺乏与霜霉病严重程度之间的关系,我们进行了一项研究t-测试使用从实验间具有异质方差的LMM中获得的调整平均值,使用未转换的数据,没有控制品种。之所以使用这组方法,是因为从先前的分析中没有迹象表明用转换后的数据拟合模型可以提高估计的准确性。
全基因组DNA测序
为了提取DNA,每个基因型在哥本哈根大学Taastrup校区的温室里种植了两株植物,并从一株两个月大的植物中收集了两片健康的叶子,用硅胶储存起来晾干。使用AX重力DNA提取试剂盒(A&A Biotechnology, Gdynia,波兰)按照制造商的说明进行DNA提取。用琼脂糖凝胶电泳控制DNA纯度和质量,用SYBR绿色染色量子位仪测定浓度。使用Baym等人的协议构建DNA测序文库。83].全基因组测序在Illumina NovaSeq机器(Novogene)上用短读Illumina测序完成。
基因组测序,变异调用和基因型归责
原始读取、映射和变量调用的处理都是通过定制的Snakemake管道完成的。84].原始读取用Trim_galore v 0.6.4修整[85(参数-q 30 -fastqc -paired)。然后用SAMTOOLS 1.10对读取进行排序和索引[86并用markduplduplments(参数remove_duplduplments = TRUE工具的PICARD v2.21.9 [87].得到的FASTQ文件与藜麦参考基因组1.0版本进行了比对[88和细胞器基因组[89]使用Burrows-Wheeler校准仪v0.7.17 [90使用默认参数。
使用GATK 3.8调用变体(snp和索引)[91]通过使用HaplotypeCaller工具,每个基础的最低质量分数为20,最低映射质量分数为30。每个样本的GVCF文件与GATK的带有默认参数的基因型GVCF工具合并。使用LinkImputeR 1.2.3对缺失数据进行估算和筛选,该方法允许用户定义一系列过滤器,并评估它们对估算标记的准确性和最终数量的影响[92].imputation的阈值为depth =8(包含位置的读取数)和missing =0.7(小于阈值深度的位置/样本的比例)。变异数据被过滤到较小的等位基因频率>= 0.05和偏离Hardy-Weinberg平衡p> 0.01采用似然比检验[93].用变异间的两两相关系数估计连锁不平衡,r2,使用最终的VCF文件作为PopLDdecay的输入[94使用默认参数。
全基因组关联研究(GWAS)
我们使用了两种关联映射方法,FarmCPU,用于序列变体(snp)和ak-mer-based方法。FarmCPU(固定和随机模型循环概率统一)在固定和随机效应模型的两步迭代过程中使用SNP,以提高计算时间,减少结构的混杂效应,并与其他方法相比提高识别显著标记-性状关联的能力[95,96].模型运行时,包含和不包含前三个主成分p-值阈值为0.01 (Bonferroni校正),包括在模型的第一次迭代期间的标记和全基因组显著性阈值。
的k基于-mer的方法97使用测序reads而不是分子变异来识别基因型-表型的关联,以解决参考基因组的缺乏或解释结构变异。我们使用以下参数在Snakemake管道中实现了该方法:k-mer长度=31个核苷酸,次要等位基因计数=3次要等位基因频率=0.05。该方法需要一个亲属矩阵,该矩阵由EMMA(高效混合模型关联)所使用的方法估计,并由一个按状态相同(IBS)的等位基因共享矩阵组成,假设每个变体对表型有小的随机影响[98].
数据和材料的可用性
在这项研究中产生或分析的所有表型数据都包含在这篇发表的文章中补充文件1(完整的霉菌感染数据集)。对测序数据进行鉴定霜霉属摘要可从NCBI基因库获取,ID为MT895880,https://www.ncbi.nlm.nih.gov/nuccore/MT895880.原始全基因组测序读取数据可从欧洲核苷酸档案馆(ENA)获得,项目编号为PRJEB39907,https://www.ebi.ac.uk/ena/browser/view/PRJEB39907.
缩写
- GLMM:
-
推广线性混合模型
- GWAS:
-
全基因组关联研究
- LD:
-
连锁不平衡
- LMM:
-
线性混合模型
- 主成分分析:
-
成分分析原则
- SNP:
-
单核苷酸多态性
参考文献
- 1.
穆希卡A,雅各布森s。e。La quinua (藜藜麦威尔德)我们的父母西尔维斯特。作者:Moraes RM, Øllgaard B, Kvist LP, Borchsenius F, Balslev H,编辑。Botánica Económica de los Andes Centrales。拉巴斯:圣市长大学Andrés;2007.449 - 57页。
- 2.
Gómez L, Aguilar E. Guía del culo de Quinua。第二。利马:拉莫利纳国立农业大学;2016.http://www.fao.org/documents/card/es/c/3a12f679-22a1-46a0-a91e-6853ca5bb5dd/.
- 3.
Gandarillas A, Rojas W, Bonifacio A, Ojeda N.玻利维亚藜麦:PROINPA基金会的视角在:巴齐勒D,贝尔泰罗D,涅托C,编辑。2013年世界藜麦最新进展报告。罗马:粮农组织拉丁美洲区域办事处;加勒比海地区。2015.p . 344 - 61。http://www.fao.org/3/a-i4042e.pdf.
- 4.
Zurita- A, Fuentes F, Zamora P, Jacobsen S-E, Schwember AR育种藜麦(藜藜麦):潜力和前景。摩尔品种。2014;34:13-30。
- 5.
Bazile D, Bertero D, Nieto C. 2013年世界藜麦现状报告:粮农组织;CIRAD;2015.http://www.fao.org/3/a-i4042e.pdf.
- 6.
Bazile D, Pulvento C, Verniau A, al - nusairi MS, Ba D, Breidy J,等。藜麦的全球评价:藜麦国际年后粮农组织在9个国家的项目的初步结果。植物科学进展2016;7:1-8。
- 7.
Murphy KM, Matanguihan JB, Fuentes FF, Gómez-Pando LR, Jellen EN, Maughan PJ,等。藜麦育种与基因组学。正确的做法:高盛一世,编辑。植物育种的评论。霍博肯:约翰·威利父子公司;2018.p . 257 - 320。
- 8.
崔永杰,丹尼尔森S, Lübeck M,洪S b, Delhey R,申h d。藜麦霜霉病病原菌的形态和分子特征(藜藜麦).Mycopathologia。2010;169:403-12。
- 9.
对藜麦疾病评估方法预测霜霉病造成的产量损失的能力的评价。作物保护。2004;23:219-28。
- 10.
丹尼尔森,雅各布森,霍肯霍尔,等。藜麦霜霉病的初步报道霜霉属farinosaf.sp。Chenopodii在丹麦。工厂说。2002;86:1175。
- 11.
Testen AL, McKemy JM, Backman PA。第一份报告由藜麦引起霜霉病霜霉属摘要在美国。工厂说。2012;96:146。
- 12.
崔玉杰,崔iy, Kim JS, Shin HD。第一份报告由藜麦引起霜霉病霜霉属摘要在韩国。工厂说。2014;98:1003。
- 13.
Khalifa W, Thabet M.霜霉病的变异(霜霉属摘要Gäum)一些藜麦的抗性(藜藜麦野生)埃及条件下的栽培品种。中东农业杂志2018;7:671-82。
- 14.
王志强,陈志强,陈志强,等。藜麦藜麦属藜麦长孢粉孢菌的pcr分析。中华微生物学杂志,2010;50:104-9。
- 15.
Kara M, Soylu EM, Uysal A, Kurt S, Choi Y-J, Soylu S.土耳其藜属变异Peronospora的霜霉病形态学和分子特征。《澳大利亚植物志》2020;15:10。https://doi.org/10.1007/s13314-020-0381-2.
- 16.
崔永杰,李志强。猪肠菌科的进化、多样性与分类,以猪肠菌属为重点。植物病理学®。2016; 106:6-18。https://doi.org/10.1094/PHYTO-05-15-0127-RVW.
- 17.
维尔玛S. C.和乔汉L. S.和马瑟R. S.。霜霉属farinosa(Fr) Fr。土荆芥murale有售印度的新纪录。咕咕叫科学。1964;33:720-1。
- 18.
Aragón L、Gutiérrez w、霜霉病上四henopodium物种。Fitopatologia。1992; 27:104-9。
- 19.
Baiswar P, Chandra S, Kumar R, Ngachan SV。霜霉属摘要在土荆芥murale有售在印度。植物科学,2010;5:45-7。
- 20.
Testen AL, Del M J-GM, Ochoa JB, Backman PA。分子检测霜霉属摘要南美和美国藜麦霜霉病病原体的系统发育。植物病理学。2014;104:379 - 86。
- 21.
Ochoa J, Frinking HD, Jacobs T.利用厄瓜多尔材料对藜麦/霜霉病病原系统的毒力群和抗性因子的假设。植物病理学研究。1999;48:425-30。
- 22.
索。土荆芥sp.:遗传资源、民族植物学和地理分布。2003; 19:1-7。https://doi.org/10.1081/FRI-120018863.
- 23.
Benlhabib O, Boujartani N, Maughan PJ, Jacobsen SE, Jellen EN。藜麦F2:6居群遗传多样性增强(藜藜麦)通过生态型间的杂交而形成。植物科学进展2016;7:1-9。
- 24.
霜霉病的评价(霜霉属farinosaf.sp。Chenopodii)藜麦基因型间的抗性及其抗性调查p . farinosa使用扫描电子显微镜生长。毕业论文:杨百翰大学;2008.https://scholarsarchive.byu.edu/etd/1512.
- 25.
雅各布森是。藜麦的全球潜力(藜藜麦Willd)。食品杂志2003;19:17 7 - 77。
- 26.
Tenorio R, Terrazas E, Alvarez MT, Vila JL, Mollinedo P. Concentrados de saponina de藜藜麦y deCaiphora andina:替代生物防治机构fitopatógenos。牧师Boliviana女性生殖器。2010; 27:33-40。
- 27.
Harrison XA, Donaldson L, Correa-Cano ME, Evans J, Fisher DN, Goodwin CED,等。简要介绍生态学中的混合效应建模和多模型推理。PeerJ。2018; 6:1-32。
- 28.
Milligan GW。反正弦变换在方差分析中的应用。梅亚斯;1987;47:563-73。
- 29.
O 'Hara RB, Kotze DJ。不要对计数数据进行对数转换。方法中华生物学报2010;
- 30.
迪华顿,许fkc。反正弦是逆正弦:生态学中对比例的分析。生态。2011;92:3-10。
- 31.
齐默尔曼DW。方差异质性对地理位置显著性检验的效度和威力的两个独立影响。统计Methodol。2006;3:351 - 74。
- 32.
Jacqmin-Gadda H, Sibillot S, Proust C, Molina J-M, Thiébaut R.线性混合模型对错种误差分布的鲁棒性。计算统计数据肛门2007;51:5142-54。
- 33.
Piepho hp, Möhring J.计算不平衡植物育种试验的遗传力和选择响应。遗传学。2007;177:1881-8。
- 34.
博尔克BM,布鲁克斯ME,克拉克CJ,乔治SW,小波尔森,史蒂文斯MHH等。广义线性混合模型:生态学和进化的实用指南。生态学报2009;24:27 - 35。
- 35.
Bertero HD, La Vega AJ, Correa G, Jacobsen SE, Mujica A.基因型和基因型-环境互作对藜麦产量和粒级的影响(藜藜麦国际多环境试验的模式分析结果表明。田间作物号2004;89:299-318。
- 36.
海沃德MD, Romagosa I, Bosemark NO, Cerezo M.入选:海沃德M, Bosemark NO, Romagosa I,编辑。植物育种:原理与展望:施普林格荷兰;1993.
- 37.
植物育种实验的不完全块设计。Agric Res J. 2017;54:607。
- 38.
Sacristán S, García-arenal F.植物致病菌种群的毒力和致病性演化。植物病理学杂志2008;9:369-84。
- 39.
张伟,张文华,张文华。植物宿主-病原相互作用的特异性和毒力连续性:原因和后果。新植醇。2009;183:513-29。
- 40.
Mhada M, Ezzahiri B, Benlhabib O.霜霉病抗性评价(霜霉属farinosa)在藜麦(藜藜麦Willd)种质。国际生物学杂志2015;6:4748-52。
- 41.
Pitrat M,编辑器。葫芦科2008:第ixeucarpia葫芦科遗传和育种会议记录,2008年5月21-24日,法国阿维尼农。阿维尼翁:INRA;2008.
- 42.
迪维洛夫K,芭芭P,卡德尔-戴维森L,赖施BI。种间葡萄抗霜霉病单表型和多表型QTL分析。应用科学学报。2018;131:1133-43。
- 43.
Lohithaswa HC, Jyothi K, Sunil Kumar KR, Puttaramanaik HS。高粱霜霉病抗性相关qtl的鉴定与导入(Peronosclerospora sorghi(Weston and Uppal) C. G. Shaw)在玉米中进行标记辅助选择。J麝猫。2015;94:741-8。
- 44.
加德纳KM,拉塔RG。生态类型内和生态类型间数量性状的遗传变异和遗传相关性燕麦属barbata.中华生物学报2008;21:737-48。
- 45.
王晓燕,王晓燕,王晓燕。藜麦形态性状和品质性状的遗传力及其与产量的关系(藜藜麦)在地中海环境中的基因型。谷物科学,2016;70:177-85。https://doi.org/10.1016/j.jcs.2016.06.003.
- 46.
塔皮亚M, Gandarillas H, Alandia S, Cardozo A, Mujica A. La quinua y La kañiwa: Cultivos Andinos。Bogotá:美洲农业科学研究所农业科学国际调查中心;1979.
- 47.
Danielsen S, Bonifacio A, Ames T.藜麦病害(藜藜麦).2003; 19:43-59。
- 48.
丹尼尔森,艾姆斯·埃尔米迪乌(霜霉属farinosade la quinua (藜藜麦) en la zona andina:手工实践para el estafmedad and el patogeno。利马:国际帕帕中心(CIP);2000.http://cipotato.org/wp-content/uploads/2014/10/AN60198.pdf.
- 49.
Gabriel J, Luna N, Vargas A, Magne J, Angulo A, La Torre J等。奎奴亚德瓦莱(藜藜麦wild .): Fuente valiosa de sistencia genética al mildiu (霜霉属farinosaWilld)。中华医学杂志2012;3:27-44。
- 50.
Al-Naggar A, El-Salam R, Badran A, El-Moghazi M.藜麦农艺、生理和产量性状的遗传力及其相互关系(藜藜麦在升高的水压力下。Arch Curr Res Int. 2017; 10:1-5。
- 51.
Rollano-Peñaloza OM, Palma-Encinas V, Widell S, Rasmusson AG, Mollinedo P.藜藜麦感染了霜霉属摘要这是藜麦霜霉病的致病剂。bioRxiv。2019.https://doi.org/10.1101/607465.
- 52.
藜麦育种与现代品种开发。在:巴齐勒D,贝尔泰罗D,涅托C,编辑。2013年世界藜麦最新进展报告。罗马:粮农组织拉丁美洲区域办事处;加勒比海地区。2015.p . 454 - 65。
- 53.
兰志明,李文华,李文华,等。植物气孔对湿度变化的响应。足底。1971;100:76 - 86。
- 54.
Danielsen S, Mercado VH, Ames T, Munk L.霜霉病种子传播(霜霉属farinosaf.sp。Chenopodii)和相对湿度对藜麦幼苗感染的影响。种子科学技术2004;32:91-8。
- 55.
Solíz-Guerrero JB, De Rodriguez DJ, Rodríguez-García R, Angulo-Sánchez JL, Méndez-Padilla G.藜麦皂苷的浓度和组成分析。在:Janick J, Whipkey A,其他人,编辑。新作物和新用途的趋势。亚历山大:灰机;2002.p . 110 - 114。
- 56.
Martínez EA, Veas E, Jorquera C, San Martín R, Jara P.将quínoa重新引入干旱的智利:在极低灌溉条件下栽培两个低地种族。农业科学学报2009;195:1-10。
- 57.
GWAS在性状分析中的优势与局限性:综述。工厂方法。2013;九29。
- 58.
Gandarillas A, Saravia R, Plata G, Quispe R, Ortíz-Romero R.主要藜麦病虫害。在:巴齐勒D,贝尔泰罗D,涅托C,编辑。2013年全球藜麦发展现状报告:罗马,粮农组织拉丁美洲区域办事处;加勒比海地区。2015.p . 192 - 215。http://www.fao.org/3/a-i4042e.pdf.
- 59.
Kumar A, Bhargava, Shukla S, Singh HB, Ohri d藜藜麦在印度的中东条件下对霜霉病的抗性。作物保护。2006;25:879 - 89。
- 60.
麦克尔希尼E, Mazón N,里维拉MM,佩拉尔塔IE。Líneas厄瓜多尔的早熟早熟早熟早熟。正确的做法:丹尼尔·DL,编辑。农业生物多样化组织producción在安地那的非正式部门través我的参与性。PREDUZA;2003: 40-47页。
- 61.
Curti RN, La Vega AJ, Andrade AJ, Bramardi SJ, Bertero HD。阿根廷西北部藜麦基因型产量及其生理决定因素的多环境评价。田间作物Res. 2014; 166:46-57。
- 62.
波兰JA, Balint-Kurti PJ, Wisser RJ, Pratt RC, Nelson RJ。灰色的阴影:定量抗病的世界。植物学报2009;14:21-9。
- 63.
Mastebroek HD, Loo R .育种奎奴亚藜的技术现状。在:Parente G, Frame J,编辑器。COST 814会议摘要/会议记录,欧洲阴湿地区作物发展。欧洲共同体官方出版物办事处;2000.491 - 6页。
- 64.
卡利斯特罗,Gómez-Pando L, Ibañez M. Evaluación de la电阻门de quinua al mildiú (霜霉属摘要)我们的国家,我们的国家,我们的国家,我们的国家,junín-perú。收录于:Resúmenes《第六届世界会议和第三届国际研讨会》,Perú 2017;2017.29页。
- 65.
防御启动的分子研究。植物学报2011;16:524-31。
- 66.
机器学习在植物-病原体相互作用中的应用:从田间规模到基因组规模的生物预测。新植醇。2019;228:35。
- 67.
Stikic R, Glamoclija D, Demin M, Vucelic-Radovic B, Jovanovic Z, Milojkovic-Opsenica D,等。藜麦种子的农艺学和营养评价(藜藜麦作为面包配方中的一种成分。谷物科学,2012;55:132-8。https://doi.org/10.1016/j.jcs.2011.10.010.
- 68.
孙毅,刘芳,Bendevis M, Shabala S, Jacobsen S- e。两种藜麦(藜藜麦野生)品种对渐进式干旱胁迫。中国农业科学(英文版)2014;https://doi.org/10.1111/jac.12042.
- 69.
Lewis CM, persons A, Bebber DP, Kigathi RN, Maintz J, Findlay K,等。英国小麦茎锈病重新出现的可能性。Commun杂志。2018;1。https://doi.org/10.1038/s42003-018-0013-y.
- 70.
Silva M, Rijo L, Rodrigues C Jr.Hemileia vastatrix关于山茱萸品种的研究Coffea阿拉比卡.见:第11届国际咖啡科学研讨会论文集。洛美:ASIC;1985.635 - 44页。
- 71.
植物气孔孔径的估算拟南芥使用硅橡胶印模。生物Protoc。2017;7:e2347。
- 72.
Fetter KC, Eberhardt S, Barclay RS, Wing S, Keller SR.气孔计数器:用于自动气孔识别和计数的神经网络。新植醇。2019;223:1671 - 81。
- 73.
DG Butler, Cullis BR, Gilmour AR, Gogel BJ。ASReml-R参考手册(版本3)。布里斯班,昆士兰:昆士兰州,第一工业和渔业部;2009.
- 74.
Piepho HP, Buchse A, Emrich K.随机实验混合模型的漫游指南。作物学报2003;189:310-22。
- 75.
伍兹高频。PLABSTAT:植物育种实验统计分析的计算机程序。斯图加特:霍恩海姆大学植物育种、种子科学和种群遗传学研究所;2001.
- 76.
Bernal-Vasquez A-M, Utz H-F, Piepho hp。广义格的离群点检测方法:从方差分析到REML转换的一个案例研究。2016; 129:787-804。
- 77.
第3章:ASReml中的方差建模。在:植物和动物育种的遗传数据分析。可汗:施普林格;2017.p . 87 - 106。
- 78.
Stroup WW。广义线性混合模型:现代概念、方法与应用。博卡拉顿:CRC出版社;2013.
- 79.
Brien C. asremlPlus::增加ASReml-R在拟合混合模型中的使用;2019.
- 80.
关于复杂处理结构的设计实验分析的注释。HortScience。2006; 41:446-52。
- 81.
Harrel FE, Dupont C. Hmisc: Harrel杂;2019.
- 82.
Koziol乔丹。藜麦苦度阈值皂素浓度的非洲色学估计(藜藜麦Willd)。食品与农业科学。1991;54:211-9。
- 83.
mbase - size基因组的廉价复用文库制备doi:https://doi.org/10.1371/journal.pone.0128036.
- 84.
Köster J, Rahmann S. snakemaker -一个可扩展的生物信息学工作流引擎。生物信息学。2012;28:2520-2。
- 85.
克鲁格F.修剪丰富!2015.https://github.com/FelixKrueger/TrimGalore.
- 86.
李浩,Handsaker B, Wysoker A, Fennell T,阮杰,Homer N,等。序列对齐/映射格式和SAMtools。生物信息学。2009;25:2078-9。
- 87.
广泛的研究所。皮卡德工具。访问日期:2018/02/21;2.17.8版本。http://broadinstitute.github.io/picard/.
- 88.
Jarvis DE, Ho YS, Lightfoot DJ, Schmöckel SM, Li B, Borm TJA,等。的基因组藜藜麦.大自然。2017;542:307。
- 89.
Maughan PJ, Chaney L, Lightfoot DJ, Cox BJ, Tester M, Jellen EN,等。线粒体和叶绿体基因组为藜麦的进化起源提供了见解(藜藜麦willd)。Sci众议员2019;9:1-1。
- 90.
李辉,杜斌。基于洞穴-轮变换的快速和精确的长读对准。生物信息学。2010;26:589 - 95。https://doi.org/10.1093/bioinformatics/btp698.
- 91.
麦肯纳A,汉娜M,班克斯E, Sivachenko A, Cibulskis K, Kernytsky A,等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组研究》2010;20:1297 - 303。
- 92.
钱曼,米基科夫斯基,加德纳,林坚:非模式生物的用户导向基因型调用与归咎。BMC基因组学。2017;18:523。
- 93.
从高通量测序数据中估计基因型频率。遗传学。2015;201:473 - 86。
- 94.
张超,董树生,徐建勇,何文明,杨天龙。PopLDdecay:一种基于不同调用格式文件的快速有效的链接不平衡衰减分析工具。生物信息学。2018;35:1786-8。
- 95.
刘鑫,黄敏,范斌,Buckler ES,张卓。固定和随机效应模型的迭代应用在强大和高效的全基因组关联研究中。公共科学图书馆麝猫。2016;12:1-24。
- 96.
Malik PL, Janss L, Nielsen LK, Borum F, Jørgensen H, Eriksen B,等。利用标记-性状关联进行生物质产量和品质性状的双用途小麦品种育种。《应用科学》2019;132:3375-98。
- 97.
识别没有完整基因组的植物表型变异的遗传变异。Nat麝猫。2020;52:534-40。
- 98.
Kang HM, Zaitlen NA, Wade CM, Kirby A, Heckerman D, Daly MJ,等。模式生物关联制图中种群结构的有效控制。遗传学。2008;178:1709-23。
致谢
我们感谢玻利维亚国家农林创新研究所(INIAF)和温室团队。作者要感谢哥本哈根大学的实验室技术员Lene Klem对病原体进行PCR分析。我们感谢Elisabeth Kokai-Kota建立了DNA测序库,感谢Mireia Vidal提供了生物信息学方面的建议。作者通过bwHPC感谢Baden-Württemberg国家的支持。我们对Sven-Eric Jacobsen提供的丹麦品种、支持和知识表示感谢。
资金
这项工作由多民族玻利维亚国通过“科学和技术主权奖学金”项目向CCL提供奖学金。这项工作的一部分是通过F.W. Schnell教授的Stifterverband向KS提供资金。资助机构在研究设计、数据收集和分析或数据解释方面没有任何作用。由Projekt DEAL支持和组织的开放获取资金。
作者信息
从属关系
贡献
CCL、MCA、HPP和KS设计了该研究。CCL进行了田间试验和表型分析工作。OSL设计了初步研究和病原体分离物的分子鉴定。CCL和MCA分析了表型数据。DBA、CA和SS为实验工作和数据分析提供建议。MCA分析测序数据并进行GWAS。CCL、MCA和KS撰写了手稿。所有作者都阅读、修改并同意了手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明他们没有竞争利益
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1:图S1至S5和表S1至S3
.补充图和表。
附加文件2:补充文件1。
基因库接入清单和护照数据;霉菌感染原始数据;皂苷和气孔的测定。
补充文件3:补充文件2。
霜霉病病原体分离和维持的详细规程霜霉属摘要。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
Colque-Little, C., Abondano, m.c., Lund, oset al。对霜霉病病原体耐受性的遗传变异霜霉属摘要藜麦遗传资源中(藜藜麦).BMC植物杂志21,41(2021)。https://doi.org/10.1186/s12870-020-02804-7
收到了:
接受:
发表:
关键字
- 藜藜麦
- 藜
- 霜霉属摘要
- 霜霉病
- 表现型
- 线性混合模型
- 定量的阻力
- 植物遗传资源