摘要
背景
石斛兰catenatum兰花是一种附生植物,是一种珍贵的中草药,生长在树干和悬崖上,经常面临各种环境压力。SET DOMAIN GROUP (SDG)蛋白作为组蛋白赖氨酸甲基转移酶,通过修饰染色质结构和调节基因转录参与多效性发育事件和应激反应,但其在植物发育中的作用d . catenatum是未知的。
结果
在本研究中,我们鉴定了44个SDG蛋白d . catenatum基因组。随后,我们从基因结构、蛋白结构域组织和系统发育关系等方面进行了综合分析d . catenatumSDG (DcSDG)蛋白,以及来自模型植物的经过充分研究的同源物拟南芥而且栽培稻以及新鉴定的42个来自近亲兰花植物的SDG蛋白蝴蝶兰属equestris.我们发现DcSDG蛋白可以分为8个不同的类(I~VII和M),这与之前的描述基本一致。基于已报道的SDG成员主要在拟南芥中的催化底物,预测I类(E(z)样)对H3K27me2/3的沉积,II类(ash样)对H3K36me的沉积,III类(Trx/ atx样)对H3K4me2/3的沉积,M类(ATXR3/7)对H3K4me的沉积,IV类(Su (var)样)对H3K27me1的沉积,V类(suv样)对H3K9me的沉积,以及VI类(S-ET)和VII类(RBCMT)对组蛋白和非组蛋白甲基化的沉积。RNA-seq衍生的表达分析显示DcSDG蛋白质通常在不同的组织和器官中有广泛而不同的表达。最后,通过环境应力测试,得到了DcASHR3,DcSUVR3,DcATXR4,DcATXR5b,DcSDG49与干旱恢复处理密切相关,表达DcSUVH5a,DcATXR5a而且DcSUVR14a都明显受低温影响,甚至达到61%DcSDG基因会对热休克做出反应。
结论
本研究对兰科植物SDG基因进行了系统的鉴定和分类d . catenatum揭示了它们在进化过程中的功能分化,发现了它们在发育过程和应激反应中的广泛作用。这些结果为兰科植物set蛋白的进一步功能研究和表观遗传机制解剖提供了建设性的线索。
背景
发育和环境刺激相关的一组基因的时空表达模式在很大程度上受到表观遗传修饰的调控,表观遗传修饰主要包括DNA甲基化、非编码rna、染色质重塑和组蛋白修饰[1].组蛋白修饰可能发生在翻译后组蛋白尾部和核心区域的各种残基上。这些修饰主要包括甲基化、酰基化、磷酸化、泛素化、瓜氨酸化、羟基化、O-GlcNAcylation和adp -核糖基化[2,3.],构成不同的组合,即“组蛋白密码”,在染色质模板化过程中起作用[4,5].在这些共价修饰中,组蛋白甲基化表现出复杂的特征,它不仅发生在不同的残基(赖氨酸和精氨酸)和位置上,而且涉及不同数目的甲基(1~3个)[6].组蛋白赖氨酸甲基转移酶(hkmase) / SET DOMAIN GROUP (SDG)蛋白催化组蛋白赖氨酸修饰,该蛋白通常具有一个进化保守的SET结构域作为催化模块[7],除了H3K79甲基转移酶DOT1缺乏SET结构域[8].sdg催化的特定赖氨酸残基组蛋白甲基化可对基因表达产生类似或相反的影响,如H3K4和H3K36与基因激活相关,而H3K9、H3K27、H4K20与基因抑制相关[9].
根据序列同源性和系统发育重建,植物SDG蛋白可分为7类:I类,E(z)同源(H3K27me writer);II类,ASH1同源物(H3K36me写入器);III类,Trx同源物及相关蛋白(H3K4me writer);IV类,具有SET结构域和PHD结构域的蛋白质;V类,Su (var)同系物和亲属(H3K9me作者);VI类,具有中断SET结构域(S-ET)的蛋白质;VII类核酮糖-1,5-二磷酸羧化酶/加氧酶(Rubisco)甲基转移酶(RBCMT)和其他SET相关蛋白用于靶向非组蛋白[10,11].西班牙基因已在细菌、病毒和真核生物中被发现[12,13].存在西班牙细菌中的基因最初被认为是真核宿主水平基因转移的结果[14,15].然而,对更多原核生物(不仅包括病原体和共生体,还包括自由生活的细菌和古细菌)释放的基因组的研究表明西班牙基因在原核生物中经历了独立的进化,这一事件与真核SDGs的进化无关,另一方面,细菌和古生菌之间发生了古老的水平基因转移[13,16].
SDG家族目前已在拟南芥基因组中被系统识别和分类(49个成员)[10,17],芸苔属植物拉伯(49)11],葡萄(33)18],杨树trichocarpa(59)19],玉米(43)20.],栽培稻(43)21],茄属植物lycopersicum(52)22],素类(47)23],Gossypium raimondii就(52)24], C4圆锥模型Setaria italica(53)25),而荔枝(48)26].然而,兰花物种的SDG家族,构成了一个极端进化的分支,仍然是难以捉摸的。
SDG蛋白及其相关的组蛋白甲基化标记广泛参与多种生长发育过程,如种子休眠、植物从营养到胚胎逆转的抑制、芽分枝、根系结构、叶绿体发育、开花时间、春化、花器官发育、胚球和花药发育、胚和胚乳发育、植物衰老、类胡萝卜素生物合成和thigmomorphogenesis [27,28,29,30.,31,32].它们也牵涉到对生物和非生物压力的反应。SDG8通过调控茉莉酸(JA)和/或乙烯信号通路中的一个基因子集,在植物防御坏死性真菌病原体中是必需的[27]以及拟南芥基础蛋白和R蛋白介导的对细菌病原体的抗性[33].功能丧失的突变sdg8结果增强了对真菌和细菌病原体的敏感性。拟南芥TRITHORAX-LIKE PROTEIN1 (ATX1)作为H3K4me3的作者,通过直接激活SA/JA信号介质的表达,在拮抗水杨酸(SA)/JA信号通路中调控防御反应基因的表达WRKY70通过在其核小体上建立H3K4me3标记[34].此外,ATX1参与干旱胁迫响应,其破坏导致植物对脱水胁迫的耐受性下降atx1植物(35].ATX1在ABA依赖和非依赖通路中调节脱水应激信号。在aba依赖通路中,脱水胁迫诱导ATX1结合NCED3该基因座编码ABA生物合成中的限速酶。随后H3K4me3标记沉积增加,RNA聚合酶II募集增加,导致增强NCED3ABA的表达和产生[36].脱水应激可引起全组蛋白H3K4me1/2/3模式的动态和特异性变化拟南芥,尤其是H3K4me3标记,在胁迫诱导基因的核小体上具有广泛的分布分布[37].同样,干旱胁迫引发水稻幼苗中多个位点(分别包括3927/910个沉积增加/减少的基因)上H3K4me3的富集发生巨大变化,并与其在干旱胁迫下的转录物变化呈正相关[38].然而,当拟南芥暴露在低温下时,两个冷反应基因的染色质中H3K27me3的沉积逐渐减少,COR15A而且ATGOLS3[39].
石斛兰catenatum(亦称石斛兰officinale)属兰科,是一种珍稀的中药材。的茎d . catenatum是传统方药保健中止胃、生津液、滋阴解热的主要药用部位[40].此外,现代药理学证实,植物茎中含有生物活性提取物,具有抗癌、保肝、降血脂、抗疲劳、抗氧化、抗便秘、降糖、保护胃溃疡、降压和增强免疫的作用[41].然而,鉴于其长期的广泛需求和过度开发,d . catenatum濒临灭绝,曾被列为中国濒危药用植物[42].在过去的20年里,d . catenatum已成功栽培,成为重要的保健经济作物。不幸的是,干旱、寒冷和高温等环境压力极大地限制了它的生长,导致严重的产量损失[43].因此,有必要筛选和鉴定抗不同环境胁迫的候选基因d . catenatum分子育种。
为了详细了解药用兰植物的SDG家族,我们在整个基因组中鉴定了SDG成员d . catenatum,随后对系统发育关系、基因结构、结构域组织、基因表达谱和对环境应激的响应进行了全面评估。我们的结果提供了关于进化和功能的见解西班牙药用兰花的基因。
结果
SDG蛋白的鉴定d . catenatum基因组
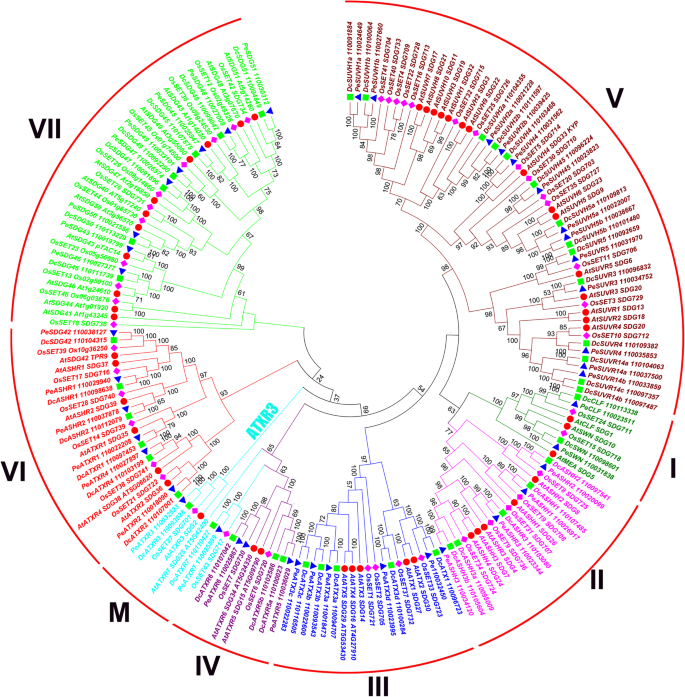
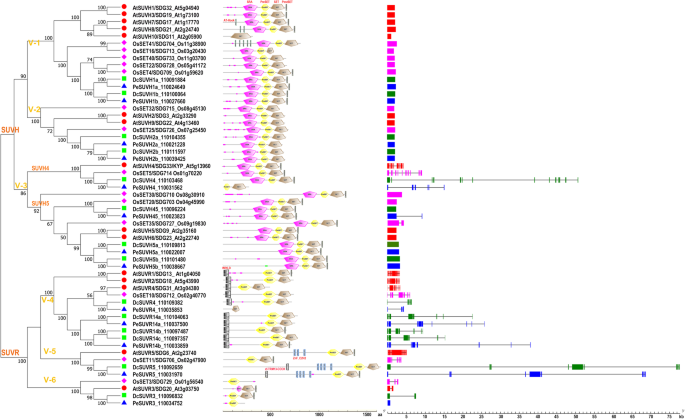
以获得SDG蛋白的所有成员d . catenatum,我们使用已知的拟南芥和水稻SDG蛋白进行BLASTP搜索d . catenatum基因组(INSDC: jsdn0000000002)。首先,我们在Superfamily 1.75数据库中检测了拟南芥和水稻的SDG基因(http://supfam.org/SUPERFAMILY/).我们发现了49个基因拟南芥,与文献报道相对应(附加文件1) [10,17].另一方面,46个基因被鉴定栽培稻(附加文件1), 3个(Os01g65730/OsSET44, Os01g74500/OsSET45, Os06g03676/OsSET46)超过43个报道基因[21].进行了对等BLAST以确认命中来自d . catenatum和它的近亲蝴蝶兰属equestris属于可持续发展目标大家庭。最终,我们获得了44个SDG基因d . catenatum(表1和无花果。1)和42个p . equestris(附加文件2),它们以拟南芥的同源物命名。
可持续发展目标的系统发育分析d . catenatum,p . equestris拟南芥和水稻。该树包括44个包含SET结构域的蛋白质d . catenatum, 42来自p . equestris49岁答:芥46个来自o .漂白亚麻纤维卷。结合进一步的系统发育分析,将181个含有SET结构域的蛋白质分为8类。4.SDGs氨基酸序列采用Clustalx比对,系统发育树采用MEGA7构建,设置如下:树推理作为邻居连接(NJ);包括位点成对删除选项的总序列分析;代入模型:p-distance;以及1000个重复的内部分支可靠性的Bootstrap测试。显示引导值> 50%。直流,d . catenatum;Pe、p . equestris;在,答:芥;操作系统,o .漂白亚麻纤维卷
的可持续发展目标家族的特征和分类d . catenatum,我们在双子叶模型植物中使用了SDG蛋白答:芥即单子叶模式植物o .漂白亚麻纤维卷,及其近亲p . equestris作为系统发育分析的参考。结果表明,上述4个物种的181个SDG蛋白可聚为8个类(I~VII和M),大多符合拟南芥的分类标准[10].相比之下,拟南芥TRITHORAX RELATED 3 (ATXR3)分支此前被归类为III类[10],但在本研究中它与其他分支分离,靠近IV类atxr7样蛋白(图。1).考虑到它们相似的底物特异性,我们将ATXR3分支和邻近的atxr7样蛋白结合在一起,并将它们归类为M类,进一步的III类、IV类和M类系统发育分析支持了这一点(图2)。4).鉴于VI类和VII类具有非组蛋白和组蛋白甲基化的潜在功能,本研究中我们主要关注I~V类和M类具有充分研究的组蛋白甲基化特异性的作用。
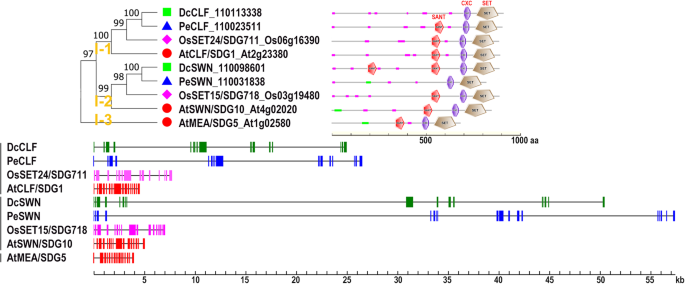
I类:E(z)类(H3K27me2/3)
I类在单子叶植物中含有两个E(z)同源体d . catenatum,p . equestris和水稻,以及三个特征明确的同源物,即卷曲叶(CLF), SWINGER (SWN)和MEAEA (MEA),它们代表了双褶叶植物拟南芥的三个不同分支(图。2).这类基因含有15~16个内含子,与拟南芥和水稻相比,这两种兰花的内含子长度都非常长。这一结果表明内含子总长度与相应的基因组大小呈正相关。在其他类别的成员中也观察到与内含子/外显子比例有关的类似现象,稍后将提到。这3个拟南芥E(z)蛋白是进化保守的Polycomb repression Complex 2 (PRC2)的催化亚基,参与了H3K27me3抑制标记在靶基因位点上的沉积[44].CLF(显性H3K27me3基因)和SWN在营养和生殖发育中起冗余作用,而MEA仅在抑制中央细胞增殖和胚乳发育中起作用[45,46,47].水稻E(z)同源物SDG711和SDG718参与调控开花时间的精确光周期[48].分枝I-1 (CLF-like)和分枝I-2 (SWN-like)在拟南芥中各含有一个同源物,而分枝I-3 (MEA-like)仅存在于拟南芥中。植物E(z)样蛋白一般在c端区域具有高度保守的结构域组织,包括一个SANT结构域、富半胱氨酸CXC结构域和签名SET结构域,但DcCLF和PeSWN缺乏SANT结构域,DcSWN在n端有一个额外的SANT结构域。
一类DcSDGs的结构域组织和基因结构。使用MEGA7生成NJ树,参数设置如图所示。1基于I类SDG蛋白的全长氨基酸序列d . catenatum,p . equestris拟南芥和水稻。沿着树枝的数字表示引导值。不同的保守蛋白结构域(SANT、CXC和SET)按图示着色。各物种SDGs的基因结构用不同的颜色表示。实框表示外显子,黑线表示内含子
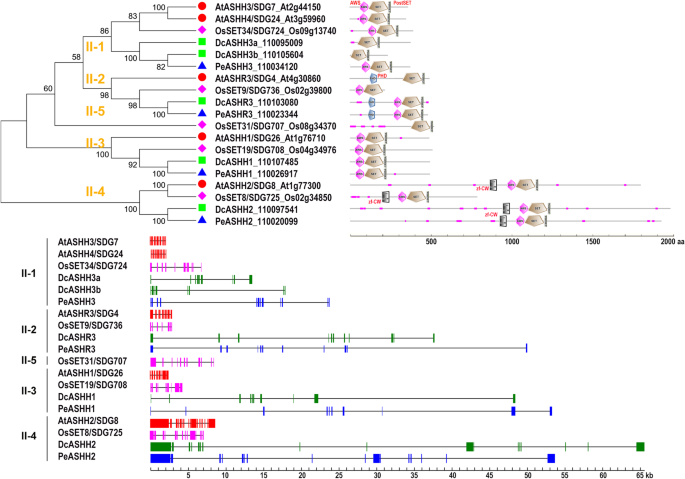
II类:类灰(H3K36me)
II类可进一步分为五个分支,每个分支由每个植物物种的一个成员组成,除了拟南芥中II-1分支的两个成员和d . catenatum(无花果。3.).分枝II-1至II-4存在于所有被研究的物种中,但分枝II-5只存在于水稻中,其中包含一个成员SDG707,功能未知。II类蛋白质通常共享三个保守结构域:Associated with SET (AWS)、SET和PostSET结构域[10,49].
第二类可持续发展目标的领域组织和基因结构。使用MEGA7生成NJ树,参数设置如图所示。1基于类SDG蛋白的全长氨基酸序列d . catenatum,p . equestris拟南芥和水稻。沿着树枝的数字表示引导值。不同的保守蛋白结构域(AWS, PHD, zf-CW, SET和PostSET)按指示着色。各物种SDGs的基因结构用不同的颜色表示。实框表示外显子,黑线表示内含子
分支II-1 (ASHH3-like)的成员比其他四个分支中的同源者相对较矮。拟南芥ASHH3/SDG7需要适当的时机来响应春化[50].SDG7缺乏可检测到的hkmase活性[51],但水稻直系物SDG724/LVP具有H3K36甲基化活性。损失SDG724导致花期晚[52].值得注意的是,DcASHH3a/3b在d . catenatum缺乏AWS域,不同于PeASHH3及其近亲p . equestris和ASHH3在拟南芥和水稻中的同源体,它们在物种形成过程中的功能差异是值得研究的。
除水稻SDG736外,分枝II-2 (ashr3样)成员的特征是在n端附近有一个额外的PHD结构域。ASHR3/SDG4参与调控花粉管生长和雄蕊发育,其过表达导致生长阻滞和雄性不育[53,54].ASHR3对H3K36me1和可能的H3K36me2具有催化活性,H3K36me2参与调节根分生组织中的细胞分裂能力[55].
分支II-3 (ASHH1-like)成员在n端具有均匀的蛋白长度和高度保守的AWS-SET-PostSET结构域组合。拟南芥ASHH1/SDG26敲除通过降低H3K4me3和H3K36me3水平导致晚开花表型SOC1轨迹(56,57].类似地,水稻的击倒直系化合物SDG708导致早期生长阶段H3K36me1/2/3水平的晚开花表型和全基因组下降[58].可以预见的是,d . catenatumDcASHH1具有类似的功能。
分支II-4 (ashh2样)蛋白比其他蛋白长得多,其特征是靠近n端三重结构域组合的额外CW结构域。拟南芥ASHH2/SDG8作为H3K36me2/3的主要作者[57,59],敲除该基因可导致营养期和生殖期的多效表型[60].一致地,水稻的击倒正交SDG725引起广泛的缺陷,包括矮化、叶片直立和种子小[32].在蛋白质结构方面,ASHH2同源于d . catenatum或p . equestris更像拟南芥SDG8而不是水稻SDG725。
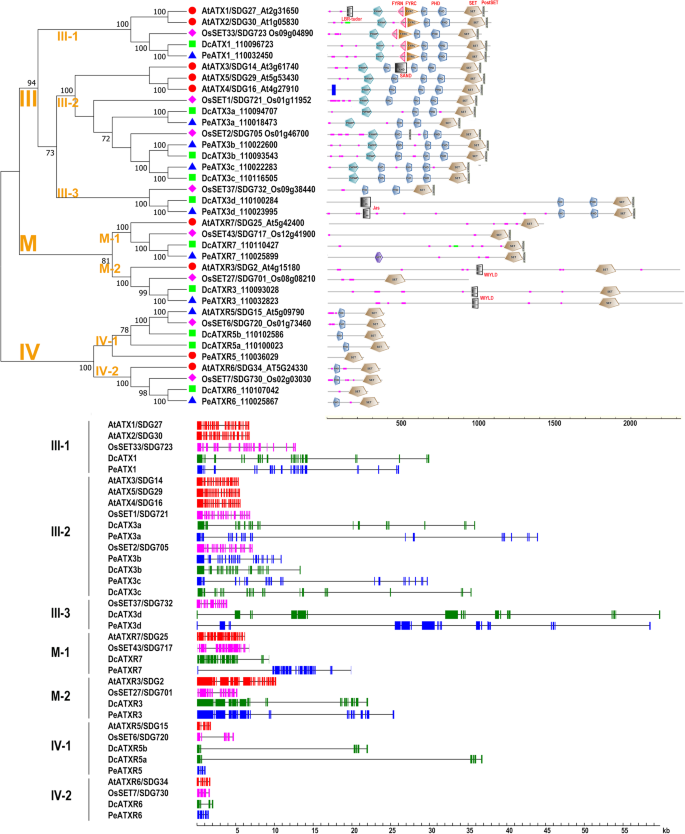
III类:Trx/ atx样(H3K4me2/3)
III类由5个成员组成,在每个被研究的植物物种中可以进一步分为3个分支(图2)。4).III类蛋白具有中间区域串联的PHD结构域和SET结构域的特征- - - - - -n端上的PostSET域组合。此外,一些演化支包含额外的不同结构域,如特定于演化支III-1/2的PWWP结构域和FYRN- - - - - -Clade III-1特有的FYRC域组合。
III类、M类和IV类DcSDGs的域组织和基因结构。使用MEGA7生成NJ树,参数设置如图所示。1基于SDGs III/M/IV类氨基酸全长序列d . catenatum,p . equestris拟南芥和水稻。沿着树枝的数字表示引导值。不同的保守蛋白结构域按图示着色。各物种SDGs的基因结构用不同的颜色表示。实框表示外显子,黑线表示内含子
Clade III-1 (ATX1-like)在拟南芥中包含两个成员,在其他三个物种中各有一个成员。在拟南芥中,ATX1和ATX2序列具有相似的结构域结构[61],但在大多数情况下具有不同的表达模式,并影响大部分不重叠的基因集的表达[62].对于共享靶标,ATX1和ATX2分别占H3K4me3和H3K4me2标记的沉积,[62].与拟南芥不同atx1早花表型[62],水稻同源突变体ostrx1/sdg723通过降低中央开花时间积分器的H3K4me3水平,显示花期晚Ehd1.OsTRX1可以拯救拟南芥atx1表型(63,64,65],表明atx1样蛋白在进化过程中表现出保守的生化和分子功能。然而,由于发育环境的差异,atx1样蛋白在不同的物种中产生特定的表型。因此,兰花中DcATX1和PeATX1可能在花期调控中发挥重要作用。
分支III-2 (ATX3-like)在每个测试种中包括三个成员。拟南芥ATX3/4/5聚在一起,从单子叶直系植物中分离出来。来自d . catenatum,p . equestris始终聚集在一起,与他们的亲密关系相一致。在拟南芥中,ATX3/4/5具有共同的进化起源,并在全基因组H3K4me2/3谱中发挥冗余作用。此外,Atx3 atx4 atx5三突变体显示侏儒症和生育力降低[66].在水稻中,atx3样蛋白SDG721和SDG705在调节H3K4甲基化水平中发挥冗余作用。这两种基因的缺失导致半侏儒症[67].考虑到拟南芥和水稻中atx3样突变体的矮秆表型,atx3样突变体在拟南芥和水稻中的同源性d . catenatum而且p . equestris可能与调节植物结构有关。
分支III-3对所检测的单子叶表现出特异性,每个物种含有一个副本。d . catenatumDcATX3d和p . equestris与水稻同源基因OsSET37/SDG732相比,PeATX3d在c端有一个额外的Jas结构域。对这支支系的进一步调查将为单子叶植物中SDG家族的进化提供深入的见解。
M类:ATXR3/7 (H3K4me)
M类包括两个分支,即M-1分支(类atxr7)和M-2分支(类atxr3)。每个分支包含一个拷贝每个植物物种(图。4).atxr7样蛋白通常缺乏额外的结构域,除了具有c端GYF结构域的PeATXR7。拟南芥ATXR7/SDG25作为H3K4单甲基化(H3K4me1)的作者,敲除其导致早花[59,68].atxr3样蛋白在每个物种中也只有一个拷贝,除了OsSET27/SDG701外,其特征是中间区域存在DUF4339结构域。拟南芥ATXR3/SDG2是H3K4me3的主要撰稿人,其缺失导致多向性发育缺陷[28,69,70].d . catenatumDcATXR3和p . equestrisPeATXR3与拟南芥ATXR3/SDG2相比水稻SDG701具有更相似的蛋白质结构。这一发现表明,兰花中的atxr3样蛋白可能保留了其祖先的作用,而水稻的同源蛋白可能由于特定结构域和部分序列的丢失而发生功能分化。
IV类:类Su (var) (H3K27me1)
IV类可以分为两个分支,分支IV- i(类atxr5)和IV- ii(类atxr6),除了PeATXR5外,它们除了定义的SET结构域外,还具有n端PHD结构域的特征。4).在拟南芥中,ATXR5和ATXR6的功能在很大程度上是重叠的,两者的缺失导致整体H3K27me1的减少和异染色质的解压缩[71,72].ATXR5/6参与维持DNA复制[73]和抑制转座因子的表达[74].ATXR5或ATXR6过表达均可导致男性不育[75].ATXR5和ATXR6可能扮演不同的角色,因为ATXR5在质体和细胞核中有双重定位,而ATXR6只在细胞核中[75].
V级:类suv (H3K9me)
类V在拟南芥中有15个成员,在水稻中有14个成员d . catenatum, 13英寸p . equestris;这些成员可以进一步分为SUVH和SUVR两个子类,分别包括V-1到V-3支和V-4到V-6支。5).V类蛋白质通常以预设值为特征- - - - - -集- - - - - -PostSET或PreSET- - - - - -设置域组合。SUVH蛋白通常包含另一个象征性的SET-和RING-ASSOCIATED (SRA)结构域,而进化枝V-4和V-5中的SUVH蛋白通常分别包含另一个wild结构域和串联ZnF_C2H2结构域。SUVH基因通常缺乏内含子,除了SUVH4分支和两个成员(PeSUVH45而且SDG727)的SUVH5分支,而SUVR基因包含不同数量的内含子。一般来说,V类成员负责组蛋白H3赖氨酸9 (H3K9me)的甲基化,其中H3K9二甲基化(H3K9me2)是基因沉默和DNA甲基化的关键标志,并参与异染色质的形成和基因表达的重编程[76].
V类DcSDGs的结构域组织和基因结构。使用MEGA7生成NJ树,参数设置如图所示。1基于中v类SDGs的全长氨基酸序列d . catenatum,p . equestris拟南芥和水稻。沿着树枝的数字表示引导值。不同的保守蛋白结构域按图示着色。各物种SDGs的基因结构用不同的颜色表示。实框表示外显子,黑线表示内含子
SUVH子类
在拟南芥V-1进化枝中,SUVH1/3/7/8/10 5个成员聚在一起,与水稻中的5个同源体和水稻中的2个同源体各有区别d . catenatum或p . equestris.这一结果表明,在双子叶和单子叶分化后,这些分支成员发生了重复。然而,这两个正交正交d . catenatum或p . equestris分别配对在一起,说明它们的基因复制发生在分裂之前石斛兰而且蝴蝶兰属.拟南芥SUVH1/SDG32具有明显的抗沉默功能,促进DNA甲基化靶向基因的表达。SUVH1敲除不会对H3K9me2水平产生影响,但会降低H3K4me3水平[77].此外,SUVH1结合rna定向DNA甲基化(RdDM)靶向的高度甲基化的基因组位点。然而,水稻suvh1样蛋白SDG728保留了其介导H3K9甲基化和参与反转录转座子抑制的经典功能[78].d . catenatum而且p . equestris包括两个suvh1样蛋白,远少于拟南芥和水稻中的5个成员。因此,SUVH1同源体在兰科植物中的功能及进化机制有待进一步研究。
分支V-2由每个被检测物种的两个成员组成,与分支V-1相比,这些成员缺乏PostSET结构域。在拟南芥中,SUVH2和SUVH9作为姐妹同源基因在RdDM和异色基因沉默中具有重叠功能[79,80].SUVH2过表达导致异位异色化,并伴有严重发育缺陷,如极度侏儒症[79,81].SUVH2和SUVH9可能具有不活跃的组蛋白甲基转移酶活性[82,83].然而,同时缺席的SUVH2而且SUVH9导致RdDM位点H3K9me2水平显著降低[80,84].SUVH2和SUVH9可与甲基化DNA结合,促进Pol V募集到RdDM位点[82,84].考虑到这些物种中suvh2样蛋白结构域高度相似,其功能应该是进化保守的。
分支V-3蛋白可进一步分为SUVH4和SUVH5两个分支。SUVH4分支在每个物种中都有一个成员,而SUVH5分支在拟南芥和水稻中有两个成员,在水稻中有三个成员d . catenatum或p . equestris.在拟南芥中,SUVH4/KRYPTONITE (KYP)、SUVH5和SUVH6作为H3K9甲基转移酶,是维持DNA甲基化所必需的[85,86,87,88,89,90].SUVH4作为主要的H3K9me1/2写入器[76].SUVH5和SUVH6作为SUVH5分支的姐妹序列,表现出具有位点特异性的hkmase活性[76,86,87].在水稻中,suvh4样蛋白SDG714介导H3K9甲基化,参与DNA甲基化、转座元件转座和基因组稳定性[91].值得注意的是,PeSUVH4蛋白p . equestris与其他三个物种的suvh4样蛋白相比,其结构域明显较短,缺乏SRA和PostSET结构域。SUVH4在兰花属间的分化石斛兰而且蝴蝶兰属是值得调查的。
SUVR子类
在进化枝V-4中,拟南芥中有3个成员p . equestris1个在大米里,4个在d . catenatum,分别。在拟南芥中,SUVR2以rddm依赖和-独立两种方式介导转录沉默[92].SUVR4通过引入H3K9me3标记来抑制潜在有害的转座子活性,从而参与表观遗传防御机制[93].SUVR4特异性地在常染色质中的转座子和伪基因上将H3K9me1转化为H3K9me3 [93,94],但SUVR1和SUVR2在体外未检测到组蛋白甲基转移酶活性[92,95].在这项研究中,d . catenatumDcSUVR4,p . equestrisPeSUVR4和水稻SDG712与拟南芥SUVR4被归为一类,而与SUVR1/2未被归为一类,这表明除了PeSUVR4序列明显较短且缺乏WIYLD和预设结构域外,它们都具有泛素结合和hkm酶活性。
除SDG706外,分支V-5在每个测试物种中都包含一个成员,其特征是额外的串联ZnF_C2H2结构域。在拟南芥中,SUVR5缺乏SRA结构域,但通过其锌指基序识别特定的DNA序列,并通过H3K9me2沉积以DNA甲基化独立的方式建立异色状态[96].敲除SUVR5导致开花延迟,在五元组中没有进一步增强的表型Suvr1 suvr2 suvr3 suvr4 suvr5突变体(96,97].这一发现表明SUVR5是SUVR亚类的主要发育调节因子。
演化支V-6成员在每个物种中都有一个副本,其编码蛋白明显短于该类其他演化支。拟南芥SUVR3包含一个额外的AWS域,与SET-PostSET域组合接近,DcSUVR3包含一个完整的PreSET-SET-PostSET域组合。然而,SUVR3在水稻和p . equestris只包含一个预设结构域,这表明进化支V-6中的基因可能受到更小的选择压力,在进化过程中变得越来越不同。到目前为止,该进化支的基因功能仍未确定。
组织和器官表达谱DcSDG基因
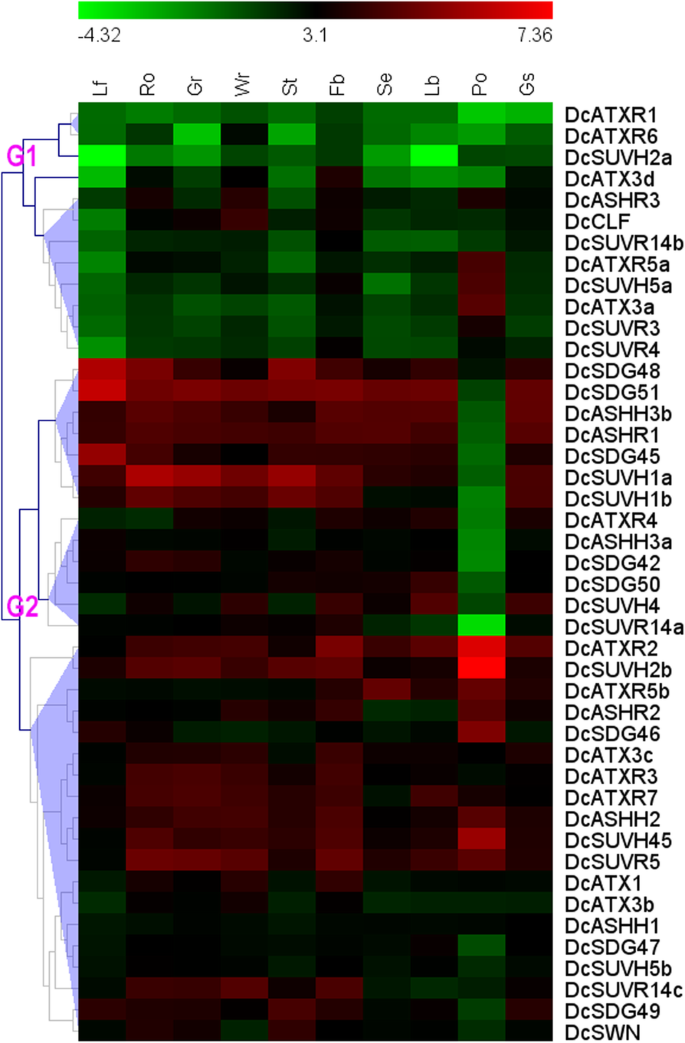
调查…的潜在作用DcSDGs在成长和发展的过程中d . catenatum,我们检测的表达谱DcSDGs通过重新分析不同植物组织和器官的RNA-seq数据,包括叶、根、绿色根尖、根的白色部分、茎、花蕾、萼片、唇瓣(唇)、花粉和合蕊(柱)[98].
基于层次聚类(图;6和附加文件3.)的表达模式DcSDGs可分为G1和G2两组。G1基因通常在大多数组织器官中低表达。然而,一些基因在特定的组织和器官中高度表达,如DcATX3d花蕾里,DcASHR3在根和花蕾,和DcATXR5a,DcSUVH5a,DcATX3a花粉块。G2基因在不同组织器官中表现出不同的表达谱。大多数G2组基因在大多数检测到的组织器官中都是高表达的,而有几个在大多数组织器官中都是中等表达的,如DcASHH3a,DcASHH1,DcSUVH5b,DcSDG47.此外,约86%(38/44)的基因在花芽中表达,而根和合石茎中表达率为77%(34),叶中表达率为57%(25),茎中表达率为45%(20),传粉中表达率为43%(19),萼片中表达率为55%(24),唇瓣中表达率为64%(28)。这些发现表明DcSDG家族在花蕾形成过程中起着至关重要的作用。值得注意的是,DcSDG51在叶,DcSUVH2b在花粉中,和DcSUVH1a在根和茎中显示最显著的表达,表明它们在特定组织中的突出功能。
六个重复的表达式配置文件DcSDG基因对进一步比较(附加文件4).总的来说,一个拷贝在所有组织中都表现出较高的表达水平,除了DcSUVH5a / 5 b这表明在植物的生长发育过程中,可能有一种旁缘细胞起主导作用。DcASHH3a / 3 b,DcATX3b / 3 c,DcATXR5a / 5 b而且DcSUVR14b / 14 c表现出相似的表达模式,而DcSUVH5a / 5 b在检测到的组织和器官中,对表现出不同的表达谱。这些结果表明,不同的重复基因对可能会经历不同的进化压力,并在不同的时间发散。
表达水平DcSDGs为了应对环境压力
d . catenatum是一种附生兰花,生长在树干和悬崖上,经常经历不同的环境压力,如干旱、寒冷和高温。检测…的反应DcSDG干旱胁迫相关基因的表达谱DcSDGs通过分析不同干旱处理下叶片的RNA-seq数据进行评估[99(图。7和附加文件5).总之,第1天灌溉,第2 - 7天不浇水,第8天恢复。分别在第2天(DR5和DR8)、第7天(DR6和DR10)、第9天(DR7和DR15)的06:30和18:30以及第8天(DR11)的18:30取样。结果表明,1周干旱胁迫显著抑制了DcCLF,DcASHR3,DcSUVR3,DcSUVR14c,但明显诱导的表达DcATXR5b,DcATXR4,DcSDG49在黎明和黄昏取样时。随后,复水恢复了DcASHR3,DcSUVR3,DcATXR5b,DcATXR4,DcSDG49.
为了探索SDG蛋白在低温胁迫响应中的可能作用,我们评估了SDG蛋白的表达水平DcSDGs通过分析叶子上的原始RNA-seq读数d . catenatum20°C(对照)和0°C处理20 h的幼苗[43(图。8和附加文件6).数据显示,32%DcSDG基因(14)在低温胁迫下表现出转录变化。例如,表达上调的基因包括DcASHH1(二),DcATX3b(3),DcSUVH4(V),DcSUVH5a(V),DcSUVH5b(V), DcSDG45 (VII), DcSDG51 (VII),而表达去调控基因也包括在内DcATXR5a(四),DcATXR5b(四),DcSUVR14a(V),DcASHR1(VI),DcASHR2(VI),DcATXR2(VI),DcSDG50(七).的表达水平DcSUVH5a,DcATXR5a,DcASHR2而且DcSUVR14a(折叠变化> 2或< 0.5)受寒冷影响显著。
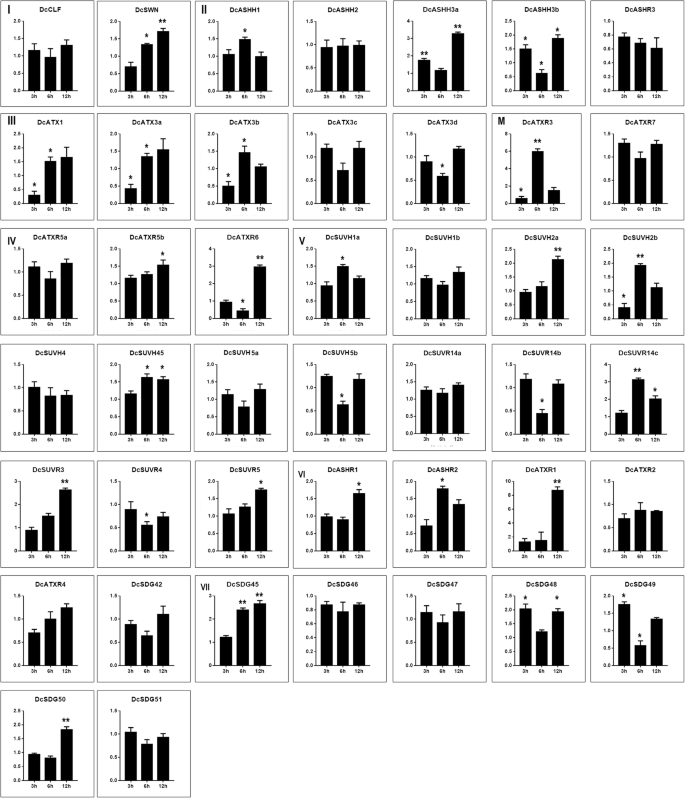
为了进一步了解DcSDG蛋白在高温(35°C)胁迫下的作用,研究了SDG基因在黄芪叶片中的表达谱d . catenatum对幼苗进行定量检测反转录-聚合酶链式反应(RT-qPCR)9).结果表明,不同的表达模式DcSDG热休克治疗过程中的基因。在处理后3 h (HAT),上调基因数量(10个)略高于下调基因数量(7个)。在处理后6 h,上调基因数量(10个)略高于下调基因数量(7个)DcSDG诱导基因(15个上调基因,10个下调基因)。在12 HAT时,上调基因的数量(27个)明显高于下调基因的数量(3个)。在热休克后检测的基因中,有3个II类基因(DcASHH3a / 3 b而且DcATX3a)、五个V类基因(DcSUVH2a b / 2,DcSUR14b / 14 c,DcSUVR3)、两个第VI类基因(DcATXR1和DcASHR1),两个VII类基因(DcSDG45/48)和一个M类基因DcATXR3至少在一个时间点(P< 0.05,图;9).
讨论
植物中SDG蛋白的鉴定和分类d . catenatum
d . catenatum在中国的食品服务业、制药、化妆品、保健品和观赏园艺中具有广泛的应用价值。最近成功的基因组测序d . catenatum其独特的发育和生活特征促进了其作为模型兰花的使用,具有相当大的理论相关性[One hundred.].在本研究中,有44种SDG蛋白d . catenatum经鉴定并分为8类:i类(H3K27me2/3)、ii类(H3K36)、iii类(H3K4me2/3)、m类(H3K4me)、iv类(H3K27me1)、v类(H3K9me)、vi类/S-ET(未定义)、vii类/ RBCMT(非组蛋白Rubisco)。
到目前为止,对第六类和第七类的理解是非常有限的。VI类包括六个分支(附加文件7),其成员具有被Zf-MYND结构域中断的长S-ET结构域,在植物中尚未有功能特征。在哺乳动物ASHR1同源物中,SET和MYND域包含3(SMYD3)可甲基化组蛋白H3K4和H4K5,而SMYD2可二甲基化H3K36并抑制基因转录[101,102,103].SMYD蛋白在细胞核和细胞质中也具有非组蛋白底物,如SMYD2用于p53和雌激素受体α, SMYD3用于VEGFR1和map3 K2 [104,105,106,107].第七类包括九个分支(附加文件8),其成员的特征是一个额外的Rubisco底物结合(Rubis-subs-bind)结构域,在植物和动物中保守。在开花植物中,叶绿体定位的Rubisco大亚基n -甲基转移酶(LSMT)在甲基化Rubisco小亚基和果糖-1,6-二磷酸醛缩酶(FBA)方面发挥着保守和古老的功能。此外,该酶还在豆科、葫芦科和蔷薇科中发挥了催化Rubisco大亚基K14三甲基化的新作用[108,109].基于His-Ala/Pro-Trp三联序的存在与Rubisco甲基化状态之间的严格相关性[108, LSMTs在d . catenatum而其他三个被研究的物种由于缺乏这种三联序,只保留了祖先的功能。在动物中,VII类同源物SETD3与额外的rubis -subs- binding结构域显示H3K36甲基转移酶活性[110].因此,植物中第VI类和第VII类成员可能同时参与组蛋白和非组蛋白的甲基化;这样的假设超出了以往的预期。
SDG蛋白的进化与功能分化
基因复制作为一种关键的驱动力,提供了额外的遗传物质副本,以耐受随机突变,在自然选择中生存下来,并在进化过程中创造新物种。基因复制,包括串联复制和多倍体(全基因组复制,WGD),在很大程度上有助于基因家族的形成[24],如SDG家族中的d . catenatum.d . catenatum已经经历了至少四轮WGD事件,包括被子植物从裸子植物分裂之前(ζ)和之后(ε)的两个古代WGD,大多数单子叶植物共有的τ WGD和兰科谱系特有的最近WGD [98,111].重复基因通常经历三种进化命运:具有沉默功能的非功能化、具有新功能的新功能化和具有部分功能的亚功能化[112].d . catenatum包含6对重复的DcSDG基因。在DcSUVH2a b / 2对,DcSUVH2a在所有检查的组织中均未表达,这意味着可能发生了新功能化。DcASHH3a / 3 b,DcATX3b / 3 c,DcATXR5a / 5 b而且DcSUVR14b / 14 c对显示相似的表达模式,表明他们可能经历了亚功能化。DcSUVH5a / 5 b配对在不同的组织和器官中表现出不同的表达谱,表明新的表达特异性和功能已经发展。
SDG蛋白在植物发育过程和环境胁迫反应中的参与
与相应组蛋白甲基化相关的SDG蛋白可以通过不同的机制输出精确的发育指令。(1)不同的SDG蛋白具有相似的催化特异性,但作用于不同的基因组。作为H3K4甲基化的作者,ATX3/4/5具有不同的靶点ATXR3/SDG2[66].(2)一些SDG成员涉及特定的保护综合体。I类E(z)同源物(CLF, SWN和MEA)与MSI1, FIE和Su(z)12同源物(FIS2, EMF2和VRN2)构成不同的PRC2复合体,在不同的发育阶段发挥作用[113].(3)特异性SDG蛋白可与其他类型的表观遗传调控因子相互作用。SUVH4/5/6与组蛋白去乙酰化酶HDA6相互作用,通过组蛋白H3K9甲基化和H3去乙酰化沉默一个转座子子集[114].进一步深入剖析SDG成员的分子结构d . catenatum具有特定的发育和生存模式,将丰富SDG基因家族的作用机制。
SDG蛋白建立的组蛋白甲基化广泛参与对环境应激和病原体挑战的响应[27,33,35,36,37,38].有研究报道干旱胁迫可引起植物组蛋白H3K4甲基化模式的全面改变拟南芥还有大米[37,38].拟南芥III类成员ATX1通过aba依赖和独立途径参与干旱胁迫响应[35,36].这里我们观察到的动态表达式的变化DcASHR3(二类),DcSUVR3(V),DcATXR5b(V),DcATXR4(VI),DcSDG49(VII)与干旱复水处理密切相关,表明H3K36和H3K9的甲基化也参与了干旱响应。H3K36me3和H3K27me3已被证实在拟南芥低温诱导的表观遗传开关中起拮抗作用开花位点c(方法)轨迹[115].在d . catenatum,我们确定了六个显著的冷反应DcSDG基因,包括DcASHH1(二类),DcATX3b / 3 d(3),DcATXR5a / 5 b(类),DcSUVH5a / 5 b而且DcSUVR14a / 3(V)(图8).结果表明,不同的组蛋白甲基化标记与特定的DcSDG蛋白在冷处理期间发挥一定的作用。通过高通量ChIP-seq方法结合针对各种组蛋白甲基化标记的优秀商业抗体,进一步研究其直接靶点将是有趣的。最近,Huang等人。[24彻底鉴定的棉花西班牙并注意到,这些基因中的大部分表达在高温条件下下降。在这项研究中,61%的DcSDG基因表现出对热休克的反应,但上调基因数量明显高于下调基因数量。这一发现可能与植物特有的附生生活方式和水芹酸代谢途径有关d . catenatum.
结论
在本研究中,我们鉴定了44个SDG蛋白d . catenatum它的近亲是42p . equestris,并将另外3个SDG成员(Os01g65730/OsSET44、Os01g74500/OsSET45和Os06g03676/OsSET46)补充到之前的水稻SDG基因家族(43个成员)中。根据系统发育关系和底物特异性,以拟南芥SDG成员为参考,将这些基因分为8类。此外,我们还分析了D.可持续发展目标不同组织和器官中的基因及其对不同环境压力的反应。我们的发现提供了全面的信息,分类和表达谱D.可持续发展目标基因,将为功能表征奠定基础西班牙兰花的基因家族。
方法
的识别西班牙基因家族d . catenatum而且p . equestris
所有SDG序列,包括基因组dna, CDS和蛋白质栽培稻而且拟南芥从植物基因组学资源Phytozome v12 (http://phytozome.jgi.doe.gov/pz/portal.html).其中,蛋白质序列被用作查询来搜索同源的d . catenatum而且p . equestris针对NCBI数据库使用BLASTp工具。所有的命中进一步证实了签名SET域的存在,使用在线生物软件PROSITE (http://prosite.expasy.org/)、SMART (http://smart.embl-heidelberg .de/)和PFam (http://pfam.xfam.org/search).
基因结构、结构域和系统发育关系分析
基因结构用基因结构显示服务器GSDS 2.0 (http://gsds.cbi.pku.edu.cn/) [116].利用SMART和Pfam数据库分析了域组织。系统发育分析使用MEGA7进行[117].利用SDG蛋白的全长氨基酸序列构建邻居连接树(neighbor-joining, NJ),设置如下:对缺失数据进行配对删除处理;代入模型的p-距离法;并进行了1000个重复的自举试验,以评价内部分支的可靠性。
在硅表达式剖析DcSDG基因
用于组织和器官表达谱分析DcSDG一个野生个体的叶片(SRR4431601)、根(SRX2938667)、绿色根尖(SRR4431599)、根白色部分(SRR4431598)、茎(SRR4431600)、花蕾(SRR4431603)、萼片(SRR4431597)、唇瓣(SRR4431602)、花粉(SRR5722145)和合蕊(SRR4431596)的原始RNA-seq数据d . catenatum均从Zhang等人提供的NCBI序列读取档案(SRA)中下载[98].用于8月龄小鼠干旱应激及应激消除实验d . catenatum植物(99],第1天灌水,第2 - 7天不灌水,第8天恢复灌水,每2天15时30分灌水一次。因此,从分别在第2天06:30和18:30 [DR5 (NCBI: SRR7223299)和DR8 (SRR7223300)]、第7天[DR6 (SRR7223298)和DR10 (SRR7223296)]、第9天[DR7 (SRR7223301)和DR15 (SRR7223297)]和第8天18:30 [DR11 (SRR7223295)]收获的叶片中获得RNA-seq原始reads。的表达式分析DcSDG基因对冷胁迫的响应,从Wu等提供的NCBI中获得叶片在20°C对照条件下(SRR3210630、SRR3210635和SRR3210636)和0°C冷处理20 h的原始RNA-seq reads (SRR3210613、SRR3210621和SRR3210626) [43].所有样本的读数都与NCBI一致石斛兰使用HISAT包的参考基因组[118每个样本的映射reads使用StringTie进行组装[119].然后,将所有样本的转录组合并,使用perl脚本构建一个综合转录组。生成最终转录组后,使用StringTie和edgeR估计所有转录本的表达水平。使用StringTie通过计算FPKM来计算mrna的表达水平。DcSDG选择基因,用log2 (fold change) > 1或<−1定义差异表达基因,差异有统计学意义(p值< 0.05)。热图使用TIGR MultiExperiment Viewer (MeV4.9)软件生成[120].
厂房材料及热休克处理
d . catenatum品种“京品NO.”1 "(品种号;哲R-SV-DO-015-2014)来自浙江省亚热带造林国家重点实验室。d . catenatum在温室中,20°C,光照12 h /暗12 h条件下生长。1年生幼苗在温控培养箱中,在35°C的热休克条件下处理指定时间(3 h、6 h和12 h),与20°C的模拟植株进行比较。然后收获叶片,在液氮中快速冷冻,并储存在−70°C下,用于进一步的表达分析。
实时定量RT-PCR (RT-qPCR)
使用TRIzol试剂(Invitrogen, USA)从叶片中提取总RNA,然后进行无rnase - DNase I处理。根据制造商的说明,通过PrimerScript RT Enzyme Mix I试剂盒(TaKaRa,日本)进行第一链cDNA合成。根据SYBR®Premix Ex Taq™II (Tli RNaseH Plus)试剂盒(TaKaRa, Japan)说明书制备RT-qPCR反应混合液(10 μl)。然后使用附加文件中列出的引物,在CFX96 Touch™实时PCR检测系统(BIO-RAD,美国)上对每个生物重复进行3个技术重复9.反应条件设定为:94℃,3 m, 40个循环,94℃,20s, 60℃,20s, 72℃。本构DcACTIN作为内参基因。将各被测基因的表达值用内参基因归一化,相对表达量用2计算——ΔΔCT方法(121].
数据和材料的可用性
在此研究过程中产生或分析的所有数据都包含在本文及其附加文件中。在当前研究中生成和分析的数据集可根据合理要求从通讯作者处获得。
缩写
- 阿坝:
-
脱落酸
- ATX1:
-
拟南芥TRITHORAX-LIKE PROTEIN1
- ATXR3:
-
拟南芥三胸相关3
- ChIP-Seq:
-
染色质免疫沉淀测序
- CLF:
-
卷曲的叶子
- E (Z):
-
Zeste
- 功能性:
-
的特性,6-bisphosphate醛缩酶
- 方法:
-
开花位点c
- 帽子:
-
治疗后1小时
- HKMTase:
-
组蛋白赖氨酸甲基转移酶
- 是:
-
茉莉酸
- KYP:
-
引火上身
- 意味着:
-
MEAEA
- NJ:
-
Neighbor-joining
- PRC2:
-
2 .多梳抑制复合体
- RBCMT:
-
核酮糖-1,5-二磷酸羧化酶/加氧酶甲基转移酶
- RdDM:
-
rna定向DNA甲基化
- RT-qPCR:
-
实时定量聚合酶链式反应
- Rubis-subs-bind:
-
二磷酸核酮糖羧化酶substrate-binding
- 山:
-
水杨酸
- 西班牙:
-
设置域组
- 设置:
-
Su (var) E(z)和三胸
- SMYD3:
-
SET和MYND域包含3
- SRA:
-
SET-和RING-ASSOCIATED
- 苏(var):
-
杂色抑制因子
- SWN:
-
赶时髦的人
- 硫氧还蛋白:
-
Trithorax
- WGD:
-
全基因组复制
参考文献
- 1.
Goldberg AD, Allis CD, Bernstein E.表观遗传学:景观形成。细胞。2007;128(4):635 - 8。
- 2.
黄华,Sabari BR, Garcia BA, Allis CD,赵颖。细胞。2014;159(2):458。
- 3.
李文杰,李志强,李志强,等。组蛋白修饰对基因表达的影响。遗传学趋势:TIG。32(1): 2016; 42-56。
- 4.
Jenuwein T, Allis CD.翻译组蛋白密码。科学。2001;293(5532):1074 - 80。
- 5.
Strahl BD, Allis CD。共价组蛋白修饰的语言。大自然。2000;403(6765):41-5。
- 6.
刘超,陆峰,崔霞,曹霞。高等植物组蛋白甲基化研究。植物学报,2010;
- 7.
Tschiersch B, Hofmann A, Krauss V, Dorn R, Korge G, Reuter G.果蝇位置效应杂色抑制基因Su (var)3-9编码的蛋白质结合了同源异型基因复合物的拮抗调控域。中国科学院学报。1994;13(16):3822-31。
- 8.
冯强,王宏,吴洪辉,erdjumem - bromage H, Tempst P, Struhl K,张艳。H3-lysine 79甲基化由一个新的无SET结构域hmts酶家族介导。目前生物学:CB。2002; 12(12): 1052 - 8。
- 9.
张艳,Reinberg D.组蛋白甲基化的转录调控:核心组蛋白尾部不同共价修饰之间的相互作用。基因工程学报2001;15(18):2343-60。
- 10.
Ng DW, Wang T, Chandrasekharan MB, Aramayo R, Kertbundit S, Hall TC。植物SET结构域蛋白的结构、功能与调控。生物化学学报,2007;1769(5-6):316-29。
- 11.
黄艳,刘超,沈文华,阮勇。油菜set结构域蛋白家族的系统发育分析与分类。BMC植物生物学2011;11:175。
- 12.
王晓明,周敏敏,王晓明,王晓明,等。病毒SET蛋白对细胞基因转录的抑制作用。细胞生物学杂志,2008;10(9):1114-22。
- 13.
Alvarez-Venegas R, Sadder M, Tikhonov A, Avramova Z.细菌SET结构域基因的起源:垂直还是水平?分子生物学杂志,2007;24(2):482-97。
- 14.
Aravind L, Iyer LM。set结构域组蛋白甲基转移酶的来源是通过一个简单结构单元的复制。细胞周期。2003;2(4):369-76。
- 15.
Stephens RS, Kalman S, Lammel C, Fan J, Marathe R, Aravind L, Mitchell W, Olinger L, Tatusov RL, Zhao Q,等。人类专性细胞内病原体:沙眼衣原体的基因组序列。科学。1998;282(5389):754 - 9。
- 16.
细菌SET结构域蛋白及其在真核染色质修饰中的作用。Front Genet. 2014;5:65。
- 17.
彭维安F,布莱文斯T,皮卡尔德CS。拟南芥组蛋白赖氨酸甲基转移酶。决议2010;53:1-22。
- 18.
aquaa F, Vega A, Timmermann T, Poupin MJ, Arce-Johnson P.葡萄SET DOMAIN GROUP家族的全基因组分析。植物细胞学报,2011;30(6):1087-97。
- 19.
雷磊,周世林,马红,张立生。白杨全基因组复制后SET结构域基因家族的扩展和多样化。BMC进化生物学。2012;12:51。
- 20.
钱艳,奚艳,程斌,朱山。玉米DNA甲基转移酶基因家族的全基因组鉴定与表达谱分析。植物细胞学报,2014;33(10):1661-72。
- 21.
陆震,黄霞,欧阳勇,姚娟。水稻OsSET基因家族的全基因组鉴定、系统发育及共表达分析。公共科学学报,2013;8(6):e65426。
- 22.
Aiese Cigliano R, Sanseverino W, Cremona G, Ercolano MR, Conicella C, Consiglio FM。番茄组蛋白修饰物的全基因组分析:深入了解其发育作用。BMC Genomics. 2013;14:57。
- 23.
徐娟,徐宏,刘勇,王霞,徐强,邓霞。甜橙组蛋白修饰基因家族的全基因组鉴定及其在果实发育和果蓝霉侵染过程中的表达分析。前沿植物科学,2015;6:607。
- 24.
黄艳,莫艳,陈萍,袁霞,孟F,朱松,刘震。雷蒙棉SET结构域蛋白的鉴定及其对高温胁迫的响应。科学代表2016;6:32729。
- 25.
亚达夫·CB, Muthamilarasan M, Dangi A, Shweta S, Prasad M.对谷子SET结构域基因家族的综合分析,确定了SiSET14在非生物胁迫耐受力中的可能作用。科学通报2016;6:32621。
- 26.
彭敏,应鹏,刘霞,李超,夏瑞,李娟,赵敏。荔枝果实脱落过程中组蛋白修饰基因的全基因组鉴定及其表达模式。植物科学,2017;8:639。
- 27.
张志刚,张志刚,张志刚,张志刚。拟南芥组蛋白甲基转移酶SET DOMAIN GROUP8介导植物对坏死性真菌的防御反应中茉莉酸酯/乙烯途径基因的诱导。植物科学进展。2010;29(3):344 - 344。
- 28.
张晓东,沈文华,张晓东,张晓东,张晓东。拟南芥SET DOMAIN GROUP2是H3K4三甲基化所必需的,对孢子体和配子体的发育都至关重要。植物学报。2010;22(10):3232-48。
- 29.
Cazzonelli CI, Cuttriss AJ, Cossetto SB, Pye W, Crisp P, Whelan J, Finnegan EJ, Turnbull C, Pogson BJ。染色质修饰组蛋白甲基转移酶SDG8对拟南芥类胡萝卜素组成和芽分枝的调控。植物学报。2009;21(1):39-53。
- 30.
Cazzonelli CI, Nisar N, Roberts AC, Murray KD, Borevitz JO, Pogson BJ。染色质修饰酶,SDG8,涉及形态,基因表达和表观遗传反应的机械刺激。植物科学进展(英文版);
- 31.
董刚,马丹萍,李娟。组蛋白甲基转移酶SDG8调控拟南芥幼苗分枝。生物化学学报。2008;39(4):359 - 64。
- 32.
隋萍,金健,叶松,穆超,高洁,冯辉,沈文华,于勇,董安。油菜素类固醇调控水稻H3K36甲基化的研究进展。植物学报,2012;30(2):344 - 344。
- 33.
Palma K, Thorgrimsen S, Malinovsky FG, Fiil BK, Nielsen HB, Brodersen P, Hofius D, Petersen M, Mundy J.拟南芥acd11的自身免疫是通过免疫受体的表观遗传调控介导的。公共科学学报,2010;6(10):e1001137。
- 34.
郭敏,王志刚,王志刚,王志刚。转录因子在两种拮抗途径横截面上的表观遗传控制。表观遗传学。2007;2(2):106 - 13所示。
- 35.
丁y, Lapko H, Ndamukong I, Xia Y, al - abdallat A, Lalithambika S, Sadder M, Saleh A, Fromm M, Riethoven JJ,等。拟南芥染色质修饰剂ATX1,肌小管蛋白样AtMTM和对干旱的反应。植物信号行为学。2009;4(11):1049-58。
- 36.
丁艳,张志军,张志军,等。拟南芥三胸样因子ATX1通过aba依赖和aba不依赖途径参与脱水胁迫响应。植物学报,2011;36(5):344 - 344。
- 37.
van Dijk K, Ding Y, Malkaram S, Riethoven JJ, Liu R, Yang J, Laczko P, Chen H, Xia Y, Ladunga I,等。拟南芥脱水胁迫下全基因组组蛋白H3赖氨酸4甲基化模式的动态变化植物学报。2010;10:238。
- 38.
宗伟,钟霞,游娟,熊玲。干旱胁迫下水稻组蛋白h3k4三甲基化及基因表达的全基因组分析。中国生物医学工程学报,2013;29(1):344 - 344。
- 39.
权cs,李迪,崔g,郑伟。拟南芥冷反应基因H3K27三甲基化的组蛋白占用依赖和独立去除植物学报,2009;30(1):344 - 344。
- 40.
闫玲,王旭,刘辉,田勇,连娟,杨蓉,郝松,王旭,杨松,李强,等。铁皮石斛(Dendrobium officinale)的基因组揭示了中国重要的传统兰科植物的生物学特性。植物学报。2015;8(6):922-34。
- 41.
唐华,赵涛,盛勇,郑涛,付林,张艳。铁皮石斛的民族药理学、植物化学、药理学及产业化研究进展。Evid Based Complement altermed . 2017;2017:7436259。
- 42.
吴涛,刘俊,黄俊华,叶欣,荣世生,童燕,张凯。一种珍贵的民间药物石斛的研究综述。中国生物医学工程学报,2012;29(5):379 - 379。
- 43.
吴志刚,蒋伟,陈世林,曼特里娜,陶志明,蒋彩霞。铁皮石斛冷转录组和代谢组的洞见:冷驯化过程中代谢和基因调控网络的全球重编程。前沿植物科学,2016;7:1653。
- 44.
郑波,陈霞。组蛋白H3赖氨酸27三甲基化在植物发育中的动态研究。植物学报。2011;14(2):123-9。
- 45.
郭丽娟,王晓明,王晓明,王晓明,王晓明,等。拟南芥中同源异型基因表达的研究进展。大自然。1997;386(6620):44-51。
- 46.
Grossniklaus U, Vielle-Calzada JP, Hoeppner MA, Gagliano WB。拟南芥多梳群基因MEDEA对胚胎发生的母系控制。科学。1998;280(5362):446 - 50。
- 47.
Kiyosue T, Ohad N, Yadegari R, Hannon M, Dinneny J, Wells D, Katz A, Margossian L, Harada JJ, Goldberg RB,等。拟南芥MEDEA多梳基因对非受精胚乳发育的控制。中国科学院学报(自然科学版),1999;26(3):366 - 366。
- 48.
刘X,周C,赵Y,周,王W,周DX。水稻zeste [E(z)]基因的增强子SDG711和SDG718分别参与长日和短日信号转导,介导对开花时间的精确光周期控制。植物科学进展(英文版);
- 49.
施普林格NM, Napoli CA, Selinger DA, Pandey R, Cone KC, Chandler VL, Kaeppler HF, Kaeppler SM。玉米和拟南芥SET结构域蛋白的比较分析表明,在单子叶和双子叶分化之前,SET结构域蛋白存在多个重复。植物科学进展。2003;32(2):339 - 344。
- 50.
李俊,尹建勇,赵伟,沈文华,Amasino RM。拟南芥春化反应适当时机所需的甲基转移酶。中国科学(d辑),2015,26(3):344 - 344。
- 51.
Sahr T, Adam T, Fizames C, Maurel C, Santoni V. o -羧基和n -甲基转移酶在植物水通道蛋白上的活性。植物生理学报。2010;51(12):2092-104。
- 52.
孙超,方杰,赵涛,徐斌,张峰,刘林,唐杰,张刚,邓霞,陈峰,等。组蛋白甲基转移酶SDG724介导H3K36me2/3在MADS50和RFT1的沉积,促进水稻开花。植物科学进展。2012;24(8):3235-47。
- 53.
Cartagena JA, Matsunaga S, Seki M, Kurihara D, Yokoyama M, Shinozaki K, Fujimoto S, Azumi Y, Uchiyama S, Fukui K。拟南芥SDG4通过成熟花粉中组蛋白H3 lysines 4和36的甲基化来调节花粉管的生长。生物工程学报。2008;32(2):355-68。
- 54.
Thorstensen T, Grini PE, Mercy IS, Alm V, Erdal S, Aasland R, Aalen RB。拟南芥set结构域蛋白ASHR3参与雄蕊发育,并与bHLH转录因子ABORTED micro孢子(AMS)相互作用。植物分子生物学杂志,2008;26(1):344 - 344。
- 55.
Kumpf R, Thorstensen T, Rahman MA, Heyman J, Nenseth HZ, Lammens T, Herrmann U, Swarup R, Veiseth SV, Emberland G,等。ASH1-RELATED3 SET-domain蛋白控制着拟南芥初生根分生组织的细胞分裂能力和静止中心。中国生物医学工程学报。2014;36(2):344 - 344。
- 56.
柏尔A,沙菲克S,皮农V,董A,沈文华。trxG家族组蛋白甲基转移酶SET DOMAIN GROUP 26通过独特的遗传途径促进开花。中国植物学报,2015;26(2):344 - 344。
- 57.
徐玲,赵钊,董a,苏碧古-塔康纳特L, Renou JP, Steinmetz A,沈文伟。组蛋白H3赖氨酸36的二甲基化和三甲基化而非单甲基化标志着拟南芥开花时间调控和其他过程相关基因的活跃转录。分子生物学杂志,2008;28(4):1348-60。
- 58.
刘波,魏刚,石娟,金娟,沈涛,倪婷,沈文华,于勇,董安。组蛋白H3赖氨酸36特异性甲基转移酶SET DOMAIN GROUP 708控制水稻开花时间。中国生物医学工程学报,2016;21(2):577 - 588。
- 59.
李松,傅飞,徐松,李雪雪,尹东杰,孟景涛。组蛋白赖氨酸甲基转移酶对植物免疫的调控作用。植物学报。2016;28(7):1640-61。
- 60.
赵智,于勇,梅耶D,吴晨,沈文华。通过表达开花位点C来预防早花需要组蛋白H3 K36的甲基化。中国生物医学工程学报,2005;7(12):1256-60。
- 61.
Pien S, Fleury D, Mylne JS, Crevillen P, Inze D, Avramova Z, Dean C, Grossniklaus U.拟南芥TRITHORAX1通过组蛋白3赖氨酸4三甲基化动态调控开花位点C激活。植物学报。2008;20(3):580-8。
- 62.
Saleh A, Alvarez-Venegas R, Yilmaz M, Le O, Hou G, Sadder M, al - abdallat A, Xia Y, Lu G, Ladunga I,等。三胸基因ATX1和ATX2高度相似的拟南芥同源基因编码具有不同生化功能的蛋白质。植物学报。2008;20(3):568-79。
- 63.
崔雪峰,李世峰,李世峰,李世峰,刘超,曹霞,安G.三胸族蛋白(Oryza sativa Trithorax1)通过与早抽穗期互作控制水稻开花时间。中国生物医学工程学报。2014;36(3):339 - 344。
- 64.
蒋萍,王松,姜红,程斌,吴凯,丁勇。compass样复合体促进水稻开花和穗部分枝。中国生物医学工程学报,2018;36(4):344 - 344。
- 65.
蒋鹏,王松,郑浩,李辉,张峰,苏勇,徐震,林华,钱强,丁勇。SIP1通过向Ehd1募集OsTrx1参与水稻开花时间的调控。中国生物医学工程学报,2018;19(1):422-35。
- 66.
陈丽强,罗建辉,崔志华,薛明,王玲,张晓霞,Pawlowski WP,何艳。ATX3、ATX4和ATX5编码H3K4甲基转移酶,对植物发育至关重要。中国生物医学工程学报,2017;29(3):344 - 344。
- 67.
蒋鹏,王松,Ikram AU,徐忠,姜华,程斌,丁勇。水稻生长需要SDG721和SDG705。中国生物工程学报,2018;29(7):528 - 528。
- 68.
柏尔A,徐琳,高洁,科涅特V,施泰因梅茨A,董安,沈文华。SET DOMAIN GROUP25编码组蛋白甲基转移酶,参与开花位点C的激活和抑制开花。植物科学进展。2009;29(3):344 - 344。
- 69.
江云,田田Y,叶康,Amasino RM。拟南芥三胸相关3/SET结构域GROUP2是拟南芥冬-年习性所必需的。中国生物医学工程学报,2012;29(5):344 - 344。
- 70.
皮农五,姚X,董安,沈文华。sdg2介导的H3K4me3在拟南芥雄性配子发生过程中染色质凝结和有丝分裂中起着至关重要的作用。中国生物医学工程学报,2017;29(2):344 - 344。
- 71.
Jacob Y, Feng S, LeBlanc CA, Bernatavichute YV, Stroud H, Cokus S, Johnson LM, Pellegrini M, Jacobsen SE, Michaels SD。ATXR5和ATXR6是染色质结构和基因沉默所需的H3K27单甲基转移酶。分子生物学。2009;16(7):763-8。
- 72.
Jacob Y, Stroud H, Leblanc C, Feng S,卓l, Caro E, Hassel C, Gutierrez C, Michaels SD, Jacobsen SE。组蛋白H3赖氨酸27甲基转移酶对异色DNA复制的调控。自然。2010;466(7309):987 - 91。
- 73.
Jacob Y, Bergamin E, Donoghue MT, Mongeon V, LeBlanc C, Voigt P, Underwood CJ, Brunzelle JS, Michaels SD, Reinberg D,等。组蛋白H3变体H3.1的选择性甲基化调控异染色质复制。科学。2014;343(6176):1249 - 53年。
- 74.
Ma Z, Castillo-Gonzalez C, Wang Z, Sun D, Hu X, Shen X, Potok ME, Zhang X。拟南芥锯齿菌协调组蛋白甲基转移酶ATXR5/6和RNA处理因子RDR6调控转座子表达。开发细胞,2018;45(6):769-84 e766。
- 75.
Raynaud C, Sozzani R, Glab N, Domenichini S, Perennes C, Cella R, Kondorosi E, Bergounioux C.拟南芥中两种细胞周期调控SET-domain蛋白与增殖细胞核抗原(PCNA)相互作用植物学报,2006;36(3):395-407。
- 76.
Jackson JP, Johnson L, Jasencakova Z, Zhang X, PerezBurgos L, Singh PB, Cheng X, Schubert I, Jenuwein T, Jacobsen SE。组蛋白H3赖氨酸9的二甲基化是拟南芥DNA甲基化和基因沉默的关键标志。Chromosoma。2004;112(6):308 - 15所示。
- 77.
李松,刘玲,李松,高玲,赵颖,金玉杰,陈霞。SUVH1是Su (var)3-9家族成员,促进DNA甲基化靶向基因的表达。中国生物医学工程学报,2016;44(2):608-20。
- 78.
秦福军,孙庆文,黄丽明,陈小生,周德胜。水稻SUVH组蛋白甲基转移酶基因在染色质修饰和反转录转座子抑制方面具有特异性功能。植物学报。2010;3(4):773-82。
- 79.
费晓东,王晓明,王晓明,等。拟南芥异染色质蛋白与异染色质基因沉默的调控。植物科学进展。2006;26(3):357 - 366。
- 80.
库尔曼M,梅特MF。拟南芥转录基因沉默需要发育非冗余的SET结构域蛋白SUVH2和SUVH9。植物分子生物学杂志,2012;39(6):623-33。
- 81.
Naumann K, Fischer A, Hofmann I, Krauss V, Phalke S, Irmler K, Hause G, Aurich AC, Dorn R, Jenuwein T,等。AtSUVH2在拟南芥异色组蛋白甲基化和基因沉默中的关键作用中国生物医学工程学报。2005;24(7):1418-29。
- 82.
Johnson LM, Du J, Hale CJ, Bischof S, Feng S, Chodavarapu RK, Zhong X, Marson G, Pellegrini M, Segal DJ,等。含有SRA-和set -结构域的蛋白质将RNA聚合酶V占用与DNA甲基化联系起来。大自然。2014;507(7490):124 - 8。
- 83.
Johnson LM, Law JA, Khattar A, Henderson IR, Jacobsen SE。drm2介导的DNA从头甲基化所需的sra结构域蛋白。公共科学学报,2008;4(11):e1000280。
- 84.
刘忠文,邵瑞荣,张长昌,周建新,张文文,李林,陈松,黄红伟,蔡涛,何新军。SET结构域蛋白SUVH2和SUVH9是pol V占据rna定向DNA甲基化位点所必需的。公共科学图书馆,2014;10(1):e1003948。
- 85.
Du J, Johnson LM, Groth M, Feng S, Hale CJ, Li S, Vashisht AA, Wohlschlegel JA, Patel DJ, Jacobsen SE。KRYPTONITE介导DNA甲基化的组蛋白甲基化机制。中国生物医学工程学报。2014;36(3):495-504。
- 86.
Ebbs ML, Bartee L, Bender J.通过SUVH6和SUVH4甲基转移酶的联合作用,H3赖氨酸9甲基化维持在转录的倒置重复序列上。分子生物学杂志,2005;25(23):10507-15。
- 87.
拟南芥SUVH5组蛋白甲基转移酶对DNA甲基化的特异性调控。植物学报。2006;18(5):1166-76。
- 88.
Jackson JP, Lindroth AM, Cao X, Jacobsen SE。KRYPTONITE组蛋白H3甲基转移酶对CpNpG DNA甲基化的控制。大自然。2002;416(6880):556 - 60。
- 89.
马娜娜F, Bartee L, Bender J.拟南芥SET结构域蛋白的DNA甲基化维持而不是建立。中国生物医学工程学报。2002;21(24):6842-52。
- 90.
Rajakumara E, Law JA, Simanshu DK, Voigt P, Johnson LM, Reinberg D, Patel DJ, Jacobsen SE。拟南芥SUVH5 SRA结构域识别5mC的双翻转机制及其对体内DNA甲基化和H3K9二甲基化的影响基因开发,2011;25(2):137-52。
- 91.
丁勇,王鑫,苏玲,翟娟,曹松,张东,刘超,毕勇,钱倩,程震,等。SDG714是一种组蛋白H3K9甲基转移酶,参与水稻Tos17 DNA甲基化和转位。植物细胞学报。2007;19(1):9-22。
- 92.
韩玉峰,窦坤,马志智,张文文,黄宏伟,李林,蔡涛,陈松,朱建科,何新军。SUVR2在拟南芥中通过与snf2相关的染色质重塑蛋白结合参与转录基因沉默。地球科学进展。2014;24(12):1445-65。
- 93.
Veiseth SV, Rahman MA, Yap KL, Fischer A, egg - jacobsen W, Reuter G, Zhou MM, Aalen RB, Thorstensen T.拟南芥SUVR4组蛋白赖氨酸甲基转移酶结合泛素并将H3K9me1转化为H3K9me3转座子染色质。公共科学图书馆,2011;7(3):e1001325。
- 94.
Rahman MA, Kristiansen PE, Veiseth SV, Andersen JT, Yap KL, Zhou MM, Sandlie I, Thorstensen T, Aalen RB。拟南芥组蛋白甲基转移酶SUVR4通过具有四螺旋束结构域与泛素结合。生物化学,2014;53(13):2091 - 100。
- 95.
Thorstensen T, Fischer A, Sandvik SV, Johnsen SS, Grini PE,路透社G, Aalen RB。拟南芥SUVR4蛋白是一种核仁组蛋白甲基转移酶,偏好单甲基化H3K9。核酸研究,2006;34(19):5461-70。
- 96.
Caro E, Stroud H, Greenberg MV, Bernatavichute YV, Feng S, Groth M, Vashisht AA, Wohlschlegel J, Jacobsen SE。set结构域蛋白SUVR5以DNA甲基化独立的方式介导H3K9me2在刺激反应基因上的沉积和沉默。公共科学学报,2012;8(10):e1002995。
- 97.
Krichevsky A, Gutgarts H, Kozlovsky SV, Tzfira T, Sutton A, Sternglanz R, Mandel G, Citovsky V. C2H2锌指set组蛋白甲基转移酶是一种植物特异性染色质修饰剂。中国生物医学工程学报。2007;36(1):339 - 339。
- 98.
张国强,刘广昌,李志,Lohaus R,肖友友,牛少生,王建勇,林玉春,徐强,陈丽娟,等。变位基因组和兰花的进化。自然。2017;549(7672):379 - 83。
- 99.
邹丽林,万霞,邓红,郑伯强,李宝军,王艳。石斛果苞果酸代谢途径的RNA-seq转录组分析。科学通报。2018;5:180252。
- One hundred.
张国强,徐强,边春,蔡卫强,叶春明,刘伟强,吉田康,张立林,常顺生,陈峰,等。catenatum Dendrobium。基因组序列为研究多糖合成酶、花的发育和适应性进化提供了新的视角。科学通报2016;6:19029。
- 101.
布朗MA,西姆斯RJ 3,戈特利布PD,塔克PW。Smyd2的鉴定和表征:一种分裂的SET/MYND结构域包含组蛋白H3赖氨酸36-特异性甲基转移酶,与Sin3组蛋白去乙酰化酶复合物相互作用。摩尔癌症,2006;5:26。
- 102.
Hamamoto R, Furukawa Y, Morita M, Iimura Y, Silva FP, Li M, Yagyu R, Nakamura Y. SMYD3编码与癌细胞增殖有关的组蛋白甲基转移酶。中国生物医学工程学报。2004;6(8):731-40。
- 103.
Van Aller GS, Reynoird N, Barbash O, Huddleston M, Liu S, Zmoos AF, McDevitt P, Sinnamon R, Le B, Mas G,等。Smyd3调节癌细胞表型,催化组蛋白H4赖氨酸5甲基化。表观遗传学。2012;7(4):340 - 3。
- 104.
黄娟,Perez-Burgos L, Placek BJ, Sengupta R, Richter M, Dorsey JA, Kubicek S, Opravil S, Jenuwein T, Berger SL. smyd2介导的甲基化抑制p53活性。大自然。2006;444(7119):629 - 32。
- 105.
Kunizaki M, Hamamoto R, Silva FP, Yamaguchi K, Nagayasu T, Shibuya M, Nakamura Y, Furukawa Y.血管内皮生长因子受体1的赖氨酸831是SMYD3甲基化的新靶点。癌症,2007;67(22):10759-65。
- 106.
Mazur PK, Reynoird N, Khatri P, Jansen PW, Wilkinson AW, Liu S, Barbash O, Van Aller GS, Huddleston M, Dhanak D,等。SMYD3将MAP 3K2的赖氨酸甲基化与ras驱动的癌症联系起来。大自然。2014;510(7504):283 - 7。
- 107.
张欣,田中坤,闫娟,李娟,彭东,姜艳,杨震,Barton MC,温宏,石霞。组蛋白甲基转移酶smyd2介导的雌激素受体α蛋白甲基化调控。中国科学:自然科学,2013;29(3):344 - 344。
- 108.
Ma S, Martin-Laffon J, Mininno M, Gigarel O, Brugiere S, Bastien O, Tardif M, Ravanel S, Alban C.植物叶绿体醛化酶/Rubisco赖氨酸甲基转移酶底物特异性的分子进化。植物学报,2016;9(4):569-81。
- 109.
Z颖,RM Mulligan, Janney N, Houtz RL。Rubisco小亚基和大亚基n -甲基转移酶。双功能和单功能甲基转移酶,甲基化Rubisco的小亚基和大亚基。中国生物医学工程学报,1999;29(5):366 - 366。
- 110.
金德伟,金凯波,金志勇,徐顺生。斑马鱼组蛋白H3K36甲基转移酶setd3的鉴定。中国生物工程学报。2011;29(2):329 - 329。
- 111.
克拉克JW,多诺霍PCJ。全基因组复制与植物宏观进化。植物科学进展,2018;23(10):933-45。
- 112.
林奇,康纳利,j。重复基因的进化命运和后果。科学。2000;290(5494):1151 - 5。
- 113.
莫兹戈瓦,柯勒,李海宁,等。多梳抑制复合体PRC2在发育过程中的作用。中国植物学报,2015;29(1):344 - 344。
- 114.
于长波,泰荣,王素生,杨萍,罗敏,杨松,程凯,王卫成,程玉生,吴凯。组蛋白脱乙酰lase6与组蛋白甲基转移酶SUVH4、SUVH5和SUVH6共同调控转座子沉默。植物细胞学报,2017;29(8):1970-83。
- 115.
杨海燕,杨海燕,陈晓明。H3K36me3和H3K27me3在拟南芥低温诱导表观遗传开关中的拮抗作用。中国生物医学杂志,2014;24(15):1793-7。
- 116.
胡斌,金俊,郭爱英,张宏,罗娟,高刚。GSDS 2.0:一种升级版的基因特征可视化服务器。生物信息学,2015,31(8):1296 - 7。
- 117.
Kumar S, Stecher G, Tamura K. MEGA7:更大数据集的分子进化遗传学分析版本7.0。分子生物学杂志,2016;33(7):1870-4。
- 118.
Kim D, Langmead B, Salzberg SL. HISAT:低内存需求的快速拼接对齐器。光子学报。2015;12(4):357-60。
- 119.
Pertea M, Pertea GM, Antonescu CM, Chang TC, Mendell JT, Salzberg SL. StringTie能够改善RNA-seq reads的转录组重建。中国生物工程学报。2015;33(3):290-5。
- 120.
Saeed AI, Sharov V, White J, Li J, Liang W, Bhagabati N, Braisted J, Klapa M, Currier T, Thiagarajan M,等。TM4:免费的、开源的微阵列数据管理和分析系统。生物学技术。2003年,34(2):374 - 8。
- 121.
Livak KJ, Schmittgen TD。使用实时定量PCR和2(−Delta Delta C(T))方法分析相关基因表达数据。方法。2001;25(4):402 - 8。
确认
感谢专业编辑公司EnPapers (http://www.enpapers.com/).
资金
本工作由国家重点研发计划项目(2017YFC1702201)资助,重点研究野生仿栽培及胁迫响应机制石斛兰,国家自然科学基金项目(31870310),重点研究植物的表观遗传机制石斛兰浙江农林大学人才创业项目科研基金(2017FR035),专注于组蛋白甲基化的功能解剖石斛兰亚热带造林国家重点实验室(ZY20180206),重点研究植物环境适宜性代谢机制石斛兰.资助机构没有参与研究的设计、数据的收集、分析和解释,也没有参与撰写手稿。
作者信息
从属关系
贡献
DHC和JPS计划并设计了本次研究。HLQ进行了实验。DHC, HLQ, YH和LZ对数据进行了分析。DHC和JPS撰写了这篇文章。所有作者都同意了这份手稿。
相应的作者
道德声明
伦理批准并同意参与
的d . catenatum本试验选用商品栽培品种“京品NO.”。哲r - sv - 015 - 2014。它不需要伦理上的批准。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1。
拟南芥和水稻SDG蛋白序列检索自Phytozome 12数据库。
附加文件2。
植物SDG基因的鉴定与分类p . equestris.序列及信息p . equestrisSDG基因和蛋白质来自NCBI数据库(https://www.ncbi.nlm.nih.gov/).
附加文件3。
表达式数据DcSDG基因来自不同的组织和器官d . catenatum.的FPKM值DcSDG用不同组织器官中的基因进行表达分析。6.Lf:叶,Ro:根,Gr:绿色根尖,Wr:根白色部分,St:茎,Fb:花蕾,Se:萼片,Lb:唇瓣,Po:传粉,Gs:合蕊。
附加文件4。
重复的表达模式西班牙基因对。复制的FPKM值DcSDG用不同组织器官中的基因进行比较。Lf:叶,Ro:根,Gr:绿色根尖,Wr:根白色部分,St:茎,Fb:花蕾,Se:萼片,Lb:唇瓣,Po:传粉,Gs:合蕊。
附加文件5。
表达式数据DcSDG不同干旱处理的基因。的FPKM值DcSDG利用不同干旱处理下无花果叶片中的基因进行表达分析。7.第1天浇水,第2 - 7天晾干,第8天复浇水。在不同的时间采集树叶;DR5/ DR8、DR6/DR10、DR7/DR15分别表示第2天、第7天、第9天的06:30、18:30采样,DR11表示第8天的18:30采样。
附加文件7。
vi类DcSDGs的结构域组织和基因结构。使用MEGA7生成NJ树,参数设置如图所示。1基于中vi类可持续发展目标的氨基酸全长序列d . catenatum,p . equestris拟南芥和水稻。沿着树枝的数字表示引导值。不同的保守蛋白结构域按图示着色。各物种SDGs的基因结构用不同的颜色表示。实框表示外显子,黑线表示内含子。
附加文件8。
第七类DcSDGs的结构域组织和基因结构。使用MEGA7生成NJ树,参数设置如图所示。1基于第七类可持续发展目标的氨基酸全长序列d . catenatum,p . equestris拟南芥和水稻。沿着树枝的数字表示引导值。不同的保守蛋白结构域按图示着色。各物种SDGs的基因结构用不同的颜色表示。实框表示外显子,黑线表示内含子。
附加文件9。
本研究用于热休克治疗表达分析的引物。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
陈,DH。,Qiu, HL., Huang, Y.et al。植物SET DOMAIN GROUP家族的全基因组鉴定及表达谱分析石斛兰catenatum.BMC植物生物学20.40(2020)。https://doi.org/10.1186/s12870-020-2244-6
收到了:
接受:
发表:
关键字
- 西班牙
- 设置域
- 组蛋白赖氨酸甲基化
- 表达分析
- 环境压力