抽象的
背景
蛋白质 - 蛋白质相互作用(PPI)在不同的生物过程中起非常重要的作用。实验验证或预测的PPI数据在不同的植物物种中越来越多地提供。为了进一步探索PPI的生物学功能,理解植物PPI的相互作用细节(例如,相互作用位点的3D结构上下文)是必要的。通过集成生物信息学算法,交互细节可以在不同的级别注释,然后编译成用户友好的数据库。在我们以前的研究中,我们开发了Atappisite,旨在为模型工厂提供PPI的交互网站信息拟南芥蒂利亚纳.考虑到arappisite的应用仅限于一个物种,它非常自然地应该进化到新的数据库中,该数据库可以在多个工厂中提供PPI的相互作用细节。
描述
plapisite(http://zzdlab.com/plappisite/index.php)是一个全面、高覆盖率和面向交互详细信息的13种植物相互作用的数据库。除收集121个实验验证的蛋白配合物结构外,还构建了13个植物中实验/预测PPIs的配合物结构,并对其相互作用位点进行了注释。对于三维结构无法建模的PPIs,我们推导了相关的域-域相互作用(ddi)和域-motif相互作用(dmi)。为了便于预测PPIs的可靠性评估,还提供了interolog模板的来源物种、GO注释、亚细胞定位和基因表达相似性。利用JavaScript软件包可视化蛋白质复合物、蛋白质相互作用位点和蛋白质相互作用网络的结构。我们还开发了一种用于蛋白质复合物同源性建模和蛋白质相互作用位点注释的在线工具。plappite中包含的所有数据也可以在下载页面免费获得。
结论
Plapisite为植物研究界提供了易于使用和全面的数据资源,用于搜索和分析13种重要植物物种中的蛋白质相互作用细节。
背景
蛋白质参与细胞中的大多数生物学过程,它们倾向于在稳定或短暂的复合物中而不是在分离中执行其生物学功能[1].因此,大规模识别蛋白质-蛋白质相互作用(PPIs)是全球了解整个蛋白质组格局的重要一步。到目前为止,大量的高通量实验已经被用于识别模式生物中的全基因组PPIs(也称为相互作用组),例如拟南芥,酿酒酵母,秀丽隐杆线虫,黑腹果蝇,智人和大肠杆菌K12[2,3.,4,5,6,7].关于已知的工厂PPI库存,36,099答:芥PPIs已储存于BioGRID (v3.4.155) [8].相比之下,来自其他植物的已知PPI的数量受到限制,因为实验方法仍然耗时和费力。
为了提高PPIs的覆盖范围,已经开发了多种计算方法来预测PPIs,包括interolog mapping [9,10.,基于基因/域融合的PPI推断[11.,12.],域名域/基序交互转移[13.,14.],基因共同表达[15.,机器学习方法[16.,17.]等。这些方法也被广泛应用于预测植物PPI [18.,19.],为植物科学家进一步研究植物蛋白的功能机制提供了有益的数据资源[20.,21,22,23,24].
为了进一步破译PPIs的分子机制,关键步骤是识别与PPIs相关的相互作用域、基序和位点。一些数据库集成了来自蛋白质数据库(PDB)的相互作用域和基母的信息[25],如三维交互域数据库(3did) [26].可以从蛋白质复合物的实验验证结构中鉴定蛋白质相互作用位点。然而,2018年释放PDB的植物中只有大约120种非冗余的异二聚体,这落后于实验验证的植物PPI的数量[8,27,28,29,30.].因此,生物信息学方法将在加速注释实验和预测PPIs的相互作用域、基序和位点方面发挥重要作用。
2016年,我们开发了Atappisite [31],提供有关模型厂7336种经实验确定的PPIs的详细信息答:芥.AraPPISite允许研究人员查询PPIs的三维结构、蛋白质相互作用位点、ddi和DMIs。此外,它还显示了丰富的相互作用位点的物理化学注释。但是,AraPPISite有一定的局限性。首先,AraPPISite只选取一种生物,答:芥考虑到。其次,Atappisite仅提供有限的实验验证PPI的蛋白质相互作用细节,同时忽略了预测的PPI,这缩小了arappisite的覆盖范围。此外,在出版arappisite后,实验性PPI的数量大大增加。第三,arappisite缺乏蛋白质复杂结构构造和交互站点分配的方便预测平台,当查询PPI不存在于Atappisite时是不方便的。在这种情况下,有必要将arappisite进入一个新版本,为多个工厂杂志组提供更高的覆盖范围的交互细节。
在这里,我们提供PlaPPISite (http://zzdlab.com/plappisite/index.php),一种自由和用户友好的植物蛋白质相互作用站点数据库。与其先例(即,Arappisite)相比,plapisite包含12个其他植物杂志。尽管PPI网络和相应的交互站点主要从计算方法推断出PLAPPISITE大大增加了PPI与交互站点注释的覆盖范围。此外,将方便的预测平台集成到plapisite中,用户可以仅提交一对蛋白质序列以获得蛋白质复杂结构和交互位点信息。
建筑和内容
数据库体系结构
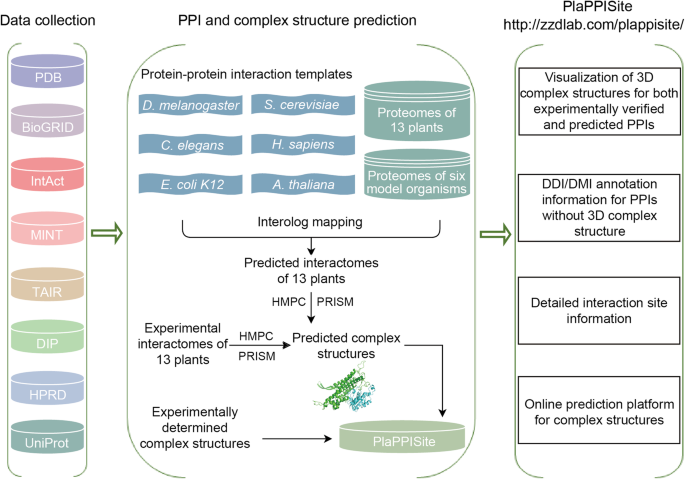
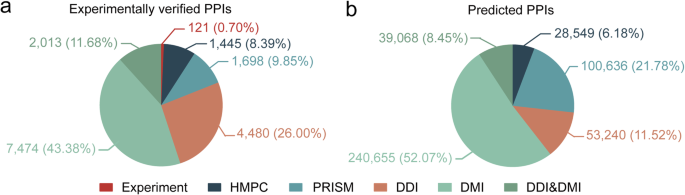
构建PlaPPISite的流程图如图所示。1.目前的plapisite包含17,231实验验证的PPI和462,148预测的PPI。PLAPPISITE中PPI的分布如图2所示。2和额外的文件1S1:表。在17231个实验验证的PPIs中,只有121个实验验证了蛋白复合物的结构,并保存在PDB数据库中。采用HMPC (Homology modeling of Protein Complex, HMPC)和PRISM (Protein Interactions By Structural Matching, PRISM)技术,分别获得了1445和1698个PPIs蛋白复合物的预测结构。其余的13967个实验验证的PPIs仅用DDIs/DMIs注释。在462,148个预测的PPIs中,HMPC和PRISM分别成功预测了28,549和100,636个蛋白质复合物结构。其余332 963 PPIs也用DDIs/DMIs注释。
采集和处理实验验证的PPI
我们收集了13种植物的实验验证的PPIs,包括答:芥,Chlamydomonas Reinhardtii.,萝藦,甘氨酸最大,栽培稻,卷柏moellendorffii,Solanum lycopersicum,茄属植物tuberosum,葡萄,玉米,Brachypodium distachyon,杨树trichocarpa和Medicago truncatula从五个公共数据库(BioGRID,https://thebiogrid.org/[8];完整,https://www.ebi.ac.uk/intact/[28];薄荷,https://mint.bio.uniroma2.it/[27];探底,https://dip.doe-mbi.ucla.edu/dip/Main.cgi[30.];TAIR,https://www.arabidopsis.org/[29])。删除了自我交互,冗余交互和非物理交互。为了统一这款13种植物的蛋白质ID,将不同类型的蛋白质ID转化为Uniprot ID。结果,获得了13个植物的49,007个非冗余PPI(附加文件1:表S2)。
植物PPIs全基因组预测
和....相比答:芥含有48,607实验验证的PPI,另一种12植物的实验验证PPI是罕见的。为了补充实验验证的PPI,通过Interogrom Mapping方法进行这13个植物的基因组PPI预测[9].简而言之,两种蛋白质(一个和B)在其中一种植物中,可以预测以在其各自的正非蛋白质之间存在实验验证的PPI(A'和B”)在其他物种中。蛋白质对(A',B”)也被认为是蛋白质对的孕系膜(一个,B).为了获得用于预测植物PPI的高质量的INTEROMIC模板,我们首先收集了六种模型生物的实验验证的PPI,包括答:芥,S. Cerevisiae.,秀丽隐杆线虫,D. Melanogaster.,H. Sapiens.和大肠杆菌K12,源自BioGRID, IntAct, MINT, DIP, TAIR和HPRD [32)(附加文件1:表S3)。然后,从UniProt数据库下载模式生物和13种植物的蛋白质序列[33].此外,InParanoid 8 [34用来鉴定13种植物和模型生物之间的矫形器。为了确保预测的PPI的质量,在[中使用的严格阈值(即,inparanoid得分= 1.0)中使用的严格阈值(即,inparanoid得分= 1.0)。35,以推断其同源关系。因此,通过间质作图,我们预测了这13种植物的蛋白质相互作用组,每个植物对应的PPIs数量如表所示1.
预测蛋白质互动网络的可靠性评估
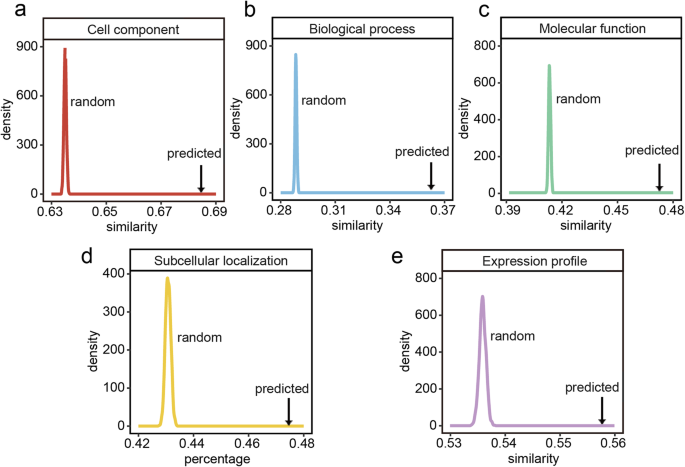
由于普遍缺乏足够的实验验证的植物PPI,间接证据包括基因本体学(GO)术语的相似性,亚细胞共同定位的比例和基因表达谱的相似性用于评估13预测蛋白的可靠性interactomes。作为重要的基因功能注释系统,GO注释包括三类,即分子功能,细胞组分和生物过程。据报道,两种共享类似GO注释的蛋白质可能互相互动。我们从Go数据库下载了这笔13个工厂的Go注释[36,37]并将它们映射到13个预测的杂志。从各种证据推断出Go数据库中的GO注释,包括实验和计算证据。实际上,一些GO术语通过立即的关系注释。对于每个预测的植物互联术,获得了高覆盖的GO注释(附加文件1:表S4)。此外,一个名为GOSemSim的R包[38]应用于计算任何两个相互作用蛋白之间的去相似性。为了评估预测的PPI网络的可靠性,通过使用称为iGraph的R包来基于相应的预测互联族来为每个工厂构建1000个随机网络。39].函数keeping_degseq被用来随机重新修整边缘,同时保留网络的原始程度分布。此外,为每个随机网络重新计算GO术语的相似之处。结果,预测网络中的平均转换相似度显着高于每个工厂构建的1000个随机网络中的相似性,这意味着13个预测网络具有可接受的可靠性。例如,预测的平均转到相似性答:芥网络比任何随机网络(经验的)都要高P值< 0.001;无花果。3.A-C)。在其他12个工厂中观察到相同的趋势(附加文件2:数字S1-S3)。
已经确定,相互作用蛋白倾向于具有相同的亚细胞定位(即,共定位)。考虑到大多数植物缺乏蛋白质组的亚细胞定位信息,我们通过众多称为multiloc2的流行预测工具预测了13个植物的蛋白质亚细胞局部化[40],为植物蛋白提供专门的预测模块。Multiloc2覆盖的预测10亚细胞局部化,包括核,细胞质,线粒体,叶绿体,细胞外基质,质膜,过氧化物体,内质网,高尔基装置和液泡。结果,大约50%的PPI在每种工厂中都是共同定位的(附加文件1:表S5),高于任意随机PPI网络中对应的比例(图5)。3.d,额外的文件2:图S4)。
此外,还应用转录组数据来执行可靠性评估[15.].在不同阶段或时间点呈现相似表达模式的蛋白质编码基因更可能是相互作用的[41].最常用的共表达措施是Pearson相关系数(PCC)。在我们的研究中,我们检索了九种植物的基因表达数据(G. Max.,o .漂白亚麻纤维卷,z梅斯,答:芥,S. lycopersicum.,诉酿酒用葡萄,B. Distachyon.,P. Trichocarpa.和m . truncatula)来自基因表达式omnibus(Geo)数据库[42,因为缺乏其他四种植物的可用表达数据。对每一株植物,人工筛选来自不同组织、器官或发育阶段属于同一基因型的20个表达谱样本。根据任意两个相互作用蛋白的基因表达谱计算PCC值。同样,预测网络的PCC均值显著高于为9个电厂构建的1000个随机网络的PCC均值,进一步说明预测的PPI网络具有合理的可靠性(图2)。3.E,附加文件2:图S5)。
关于互联症答:芥,实验PPIs的数量比较大,一些预测的相互作用体已经公开,这使得我们可以直接将我们的预测PPIs与一些现有的相互作用体进行比较。为此,我们收集了9065个预测结果,可信度很高答:芥PPI(S-PPI)来自[43,它是基于一个对接评分算法,使用实验确定和预测的蛋白质结构。自身的相互作用以及与蛋白质的相互作用都没有出现在我们的收集中答:芥除去蛋白质组,最终保留了8358个PPI。为了确保公平的比较,我们选择了预测的高质量答:芥其中包括38,610个可建立三维结构或可注释ddi /DMIs的interologi推断的PPIs。总体而言,我们预测的PPIs、S-PPIs和实验PPIs之间重叠的PPIs数量较低,但重叠程度显著(Additional file)2:图S6;超几何测验,都是成对的P-值< 2.2 × 10- 16).相比之下,与S-PPI相比,我们预测的PPI与实验PPI共享更高的重叠率。总的来说,上述直接比较进一步表明我们的预测具有可比性和合理的准确性。
实验/预测复杂结构和互动位点的注释
共实验验证了101个与复杂结构有关的答:芥,c . reinhardtii,o .漂白亚麻纤维卷和z梅斯从PDB数据库收集,涉及121个非冗余二进制PPI。考虑到一些PPI可以拥有来自不同结构确定源的多组复杂结构,保留了具有最佳分辨率的相应复杂结构。如果两个相互作用蛋白可以映射到候选复合结构的多个链,则进一步选择具有最大相互作用界面的两条链作为PPI的最终复杂结构。
大多数植物PPI不可用实验复杂结构。遵循我们以前的Atappisite策略,进一步应用了两种基于模板的方法(HMPC和棱镜)以预测实验验证和预测的PPI的复杂结构,主要包括三个步骤,即模板选择,单体建模和复杂建模。
为了模拟相互作用蛋白质对的复杂结构,我们首先通过BLAST搜索为每个蛋白质选择最佳的同源模板[44]针对PDB数据库。从爆炸推断的模板候选者应具有至少30%的序列同一性与查询蛋白质,并且对准应覆盖查询蛋白的至少40%的序列长度。通常,具有最高序列同一性的模板候选被认为是查询蛋白的最佳模板。在某些情况下,模板候选共享类似的序列标识,但不同的对准覆盖范围,最高对准覆盖范围优先考虑[45].其他模板选择标准与AraPPISite中使用的相同。下一步是根据选定的模板为相互作用的蛋白质对中的每个蛋白质构建一个单体模型。每个蛋白质由modeler生成5个模型(版本9.19)[46]选择具有最低涂料得分的模型。蛋白质的N-和C-末端的未对准残留物,即取向范围之外的残留物,被截断以确保预测蛋白质结构的质量。一旦获得了两个相互作用蛋白的预测结构,就进一步建模了相应的蛋白质复合物结构。我们首先使用HMPC来推断复杂结构,这需要相互作用蛋白对的两个模板来自PDB数据库中相同复杂结构的两个不同的接触链。否则,棱镜软件[47]来推断其复杂结构,这只需要两个单体结构与已知的复杂结构具有相似的结合界面即可。有关HMPC和PRISM实现的更多细节可在[31].
此外,可以从实验/预测的复杂结构中检索相互作用位点。简而言之,如果它们的最短原子距离小于或等于4.0,则将来自两个相互作用蛋白的残基分配为相互作用位点(残基)。两个相互作用蛋白之间的所有相互作用残基构成完整的相互作用界面。
网络实现
数据库结构基于MySQL 5.5.60和PHP 5.4.16。该服务在具有Linux操作系统CentOS 7.4的Apache 2.4.6服务器上运行。相似 [48,一个名为Cytoscape.js的JavaScript图形库[49,用于显示PPI网络。表格和交互图表是基于几个基于网络的JavaScript库生成的,比如datatable .js、charts.js和Highcharts.js。天然气凝析液(50[基于WebGL的3D查看器由MMTF提供支持,用于显示PPI的3D复杂结构。
效用和讨论
我们的目标是开发一个由多个功能模块组成的植物蛋白质交互站点的综合数据库。它允许用户从系统的角度和可视化蛋白质复合结构和相互作用位点来探索蛋白质之间的关联。与此同时,它还提供了一个在线预测平台。此外,它允许用户不仅直接从在线数据库访问数据,还允许下载完整的本地数据。
数据访问
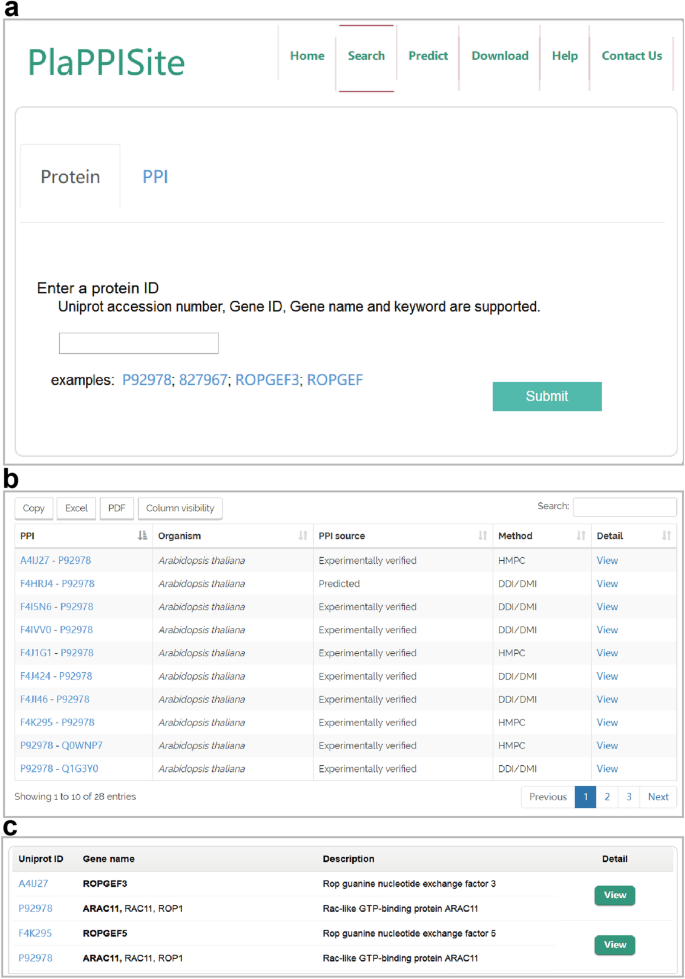
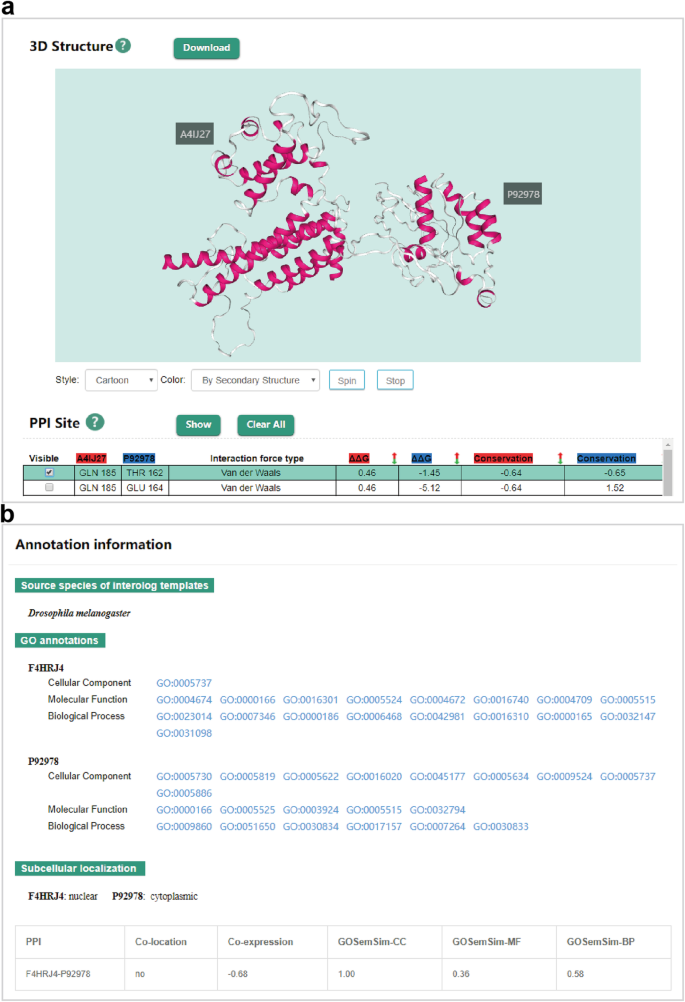
PlaPPISite提供了两种获取PPIs注释信息的方法(图。4a).用户可以输入单个蛋白ID或关键字。查询蛋白、来源生物、PPI测定方法和复杂结构预测方法相关的PPIs将在表格中列出(图)。4b)。同样,用户还可以通过搜索两个蛋白质ID或关键字直接访问感兴趣的PPI(图。4C)。
蛋白质复合结构和相互作用细节的可视化
与以前的版本相比,Plapisite将新的插件NGL应用于显示蛋白质复杂结构,这已广泛用于许多蛋白质结构数据库,例如PDB。新插件的利用增加了各种颜色方案和分子表示,例如骨干和太空填充物。复杂结构可以通过任何颜色方案和分子表示来呈现,并通过自动旋转从不同的角度看。符合先前的版本,可以在复杂结构上显示详细的交互站点,并且还列出了相应的物理化学特性,包括粘合类型,节约评分和Gibbs自由能(ΔΔG)的变化(图。5a).此外,我们为无法构造复杂结构的PPIs提供了DDI/DMI注释。值得注意的是,预测的PPIs列出了interolog模板、GO注释和亚细胞定位的源物种。用户可能希望使用GO注释、基因表达谱和亚细胞定位计算出的相似性度量来进一步判断PPIs的可靠性(图)。5b)。
PPI网络的可视化
考虑到Plapisite中每个植物互乱的大小很大,我们只为每个PPI提供子网可视化。简而言之,对于每个PPI,plapisite增加了一个子网,它由两个相互作用蛋白中涉及的第一层交互组成(附加文件2:图S7)。子网可以由各种布局呈现并导出以进一步分析。在子网中,可以根据需要调整边缘长度和节点间距。关于子网中的预测PPI,注意节点由亚细胞定位着色,边缘通过两个节点之间的基因表达相似性着色。
预测平台
为了帮助用户构建蛋白质复杂结构并为未存放在Plapisite中的自己的PPI分配交互站点,基于HMPC方法(附加文件)构建了在线预测平台(附加文件2:图S8a)。结果,两个查询蛋白的模板,模板和查询蛋白质,复杂结构和详细的交互站点之间的序列标识和覆盖率(附加文件2:图S8b)。
预测的PPI和预测蛋白质复杂结构的可靠性
为了增加这13种植物副间的覆盖率,虽然预测的PPI的可靠性始终是争议的,但是在Plapisite中收集了大量预测的PPI数据。即使三条间接证据和与预测的直接比较答:芥Interactome由[43],以证明PPI预测的可接受可靠性,PlaPPISite中预测的PPI可能不可避免地包含大量假阳性。为了有效地指导用户正确使用预测的PPI数据,我们做了两方面的努力。首先,只有高质量的PPIs被保留并沉积在PlaPPISite中。这里的高质量意味着可以构建那些PPIs的复杂结构,或者可以对DDIs/DMIs进行注释。其次,给出预测PPIs的GO注释、基因表达谱和亚细胞定位的相似性,指导用户正确使用预测PPIs。
尽管蛋白质复杂结构预测的生物信息学算法已被广泛开发,但是预测蛋白质复合结构的可靠性也难以定量评估。在我们以前的Atappisite出版中[31,我们主要使用了27个经实验确定的复杂结构答:芥PPIs作为评估HMPC和PRISM性能的测试集。虽然测试集的大小非常有限,但结果表明,HMPC和PRISM在构造复杂结构时都达到了合理的性能。相比之下,HMPC的精度优于PRISM。
此外,我们收集了4493个突变部位995答:芥来自两个来源的蛋白质[51,52]进一步判断预测互动站点的质量。从手动收集的突变中收集突变,通过表型效应,可以通过使用Google学者使用文献搜索来在Tair和其他突变中找到。我们预测的复杂结构中包含总共248个含有1110个突变残留物(279个中性突变和831个有害突变)的蛋白质(附加文件1:表S6)。因此,831个有害突变中有530个位于预测的相互作用界面,而279个中性突变中只有16个发生在相互作用界面。因此,与中性突变相比,有害突变在预测的相互作用界面显著富集(Fisher’s exact test,单侧检验)P-Value <2.2×10- 16;无花果。6).已经很好地确定,与中性突变相比,有害突变更可能位于蛋白质相互作用界面[53,54,55].例如,David和Sternberg(2015)报告了导致疾病的不同分布和性质,导致单一氨基酸变化(SEVS)和不同结构区域内的多态性[54].他们观察到,在相互作用界面处,1960年的人类疾病引起的4285个引起的,而在相互作用界面发生在没有已知的疾病关联的1699多态性中仅为1699多态性中的603。结果表明,与多态性相比,在相互作用界面上更容易发生疾病的群体(Fisher的确切测试,单尾P-Value <2.2×10- 16),这与我们的发现一致。因此,上述计算分析为预测的相互作用位点的可靠性提供了额外的证据。综上所述,我们目前和以前的计算分析支持了预测的复杂结构和相互作用位置的合理可靠性。
结论
PlaPPISite是一个免费的公共资源,提供了13种植物丰富的PPI细节。在结构层面,PlaPPISite不仅包括13种重要植物的实验/预测PPIs的三维结构和相互作用位点,还列出了相互作用位点的理化性质和残基保存。此外,对于那些无法成功构建三维结构的PPIs,也标注了DDI/DMI信息。需要强调的是,尽管我们已经进行了一系列计算实验,直观地提供了预测PPIs和蛋白复合物结构可靠性的证据,但PlaPPISite中沉积的PPI和相互作用位点信息可能不可避免地包含假阳性。考虑到潜在的假阳性,我们希望PlaPPISite能够成为一个重要的数据平台,加速我们对植物相互作用的全球理解。例如,它可以有效地指导诱变等实验工作,以查明植物PPIs的功能作用。
可用性数据和材料
数据库通过自由提供http://zzdlab.com/plappisite/index.php.
缩写
- ∆∆G:
-
吉布斯自由能的变化
- 3DID:
-
三维交互领域数据库
- ddi:
-
域域交互
- DMIS:
-
Domain-Motif交互
- 地理:
-
基因表达综合
- 走:
-
基因本体论
- HMPC:
-
蛋白质复合物的同源性建模
- PDB:
-
蛋白质数据库
- 质子泵抑制剂:
-
蛋白质相互作用
- 棱镜:
-
结构匹配的蛋白质相互作用
参考文献
- 1。
从分子水平到蛋白质组水平预测蛋白质相互作用。化学启116:4884 2016;909年。
- 2.
拟南芥互作图谱协会。网络演化的证据拟南芥interactome地图。科学。2011;333:601-7。
- 3.
Uetz P,Giot L,Cagney G,Mansfield Ta,Judson Rs,Knight JR等人。综合分析蛋白质 - 蛋白质相互作用酿酒酵母.自然。2000; 403:623-7。
- 4.
钟伟,斯特恩伯格PW。全基因组预测秀丽隐杆线虫遗传相互作用。科学。2006; 311:1481-4。
- 5.
爱情l,獾js,brouwer c,chaudhuri a,kuang b,li y等。蛋白质相互作用图黑腹果蝇.科学。2003;302:1727-36。
- 6.
贝兰G,Peregrin-Alvarez JM,Li J,Yang W,Yang X,Canadien V等人。含有保守和必需蛋白复合物的交互网络大肠杆菌.大自然。2005;433:531-7。
- 7.
李志刚,李志刚,李志刚,等。人类蛋白质-蛋白质相互作用网络:注释蛋白质组的资源。细胞。2005;122:957 - 68。
- 8.
Chatr-aryamontri a,outhtred r,boucher l,rust J,Chang C,Kolas NK等。BioGrid交互数据库:2017更新。核酸RES。2017; 45:D369-79。
- 9.
yu h,luscombe nm,lu hx,zhu x,xia y,han jd等。基因组之间的注释转移:蛋白质 - 蛋白质中的疑问和蛋白质-DNA调节。Genome Res。2004; 14:1107-18。
- 10。
何飞,张勇,陈辉,张震,彭玉玲。稻瘟病菌蛋白质相互作用网络的预测。BMC基因组学。2008;9:519。
- 11.
Enright j, Iliopoulos I, Kyrpides NC, Ouzounis CA.基于基因融合事件的全基因组蛋白相互作用图谱。大自然。1999;402:86 - 90。
- 12.
Morilla I, lee JG, Reid AJ, Orengo C, Ranea JA。在人类蛋白质相互作用网络预测中的蛋白质结构域融合评估:在人类着丝点模型中的应用。生物科技新》。2010;27:755 - 65。
- 13.
王志强,王志强。域共现网络的演化核心。BMC Evol Biol. 2005;5:24。
- 14.
王志刚,王志刚。结构域与基序相互作用的动态关系。公共科学图书馆。2012;8:e1002341
- 15.
GE H,Liu Z,Church Gm,Vidal M.转录组和互乱映射数据之间的相关性酿酒酵母.NAT Genet。2001; 29:482-6。
- 16.
Kotlyar M, Pastrello C, Pivetta F, Lo Sardo A, Cumbaa C, Li H, et al.。在硅预测物理蛋白质相互作用和鉴定相互作用组孤儿。Nat方法。2015;12:79 - 84。
- 17.
周勇,周永生,何飞,宋娟,张铮。简单密码子对的使用能否预测蛋白质相互作用?摩尔BioSyst。2012; 8:1396 - 404。
- 18.
丁yd,chang jw,guj,chen d,li s,xu q等。甜橙蛋白质 - 蛋白质相互作用网络的预测与功能分析。BMC植物BIOL。2014; 14:213。
- 19.
刘胜,刘勇,赵军,蔡胜,钱洪,左科,等。水稻复杂农艺性状相关基因优先排序的计算互作(栽培稻).植物j . 2017; 90:177 - 88。
- 20。
Brandão MM, Dantas LL, Silva-Filho MC. AtPIN:拟南芥蒂利亚纳蛋白质相互作用网络。BMC生物信息学。2009; 10:454。
- 21。
朱刚,吴安,徐晓军,肖鹏,陆璐,刘军,等。玉米蛋白质相互作用数据库。植物杂志。2016;170:618-26。
- 22。
林M,周X,沉X,毛C,陈X.预测拟南芥基于互乱的资源和网络拓扑系统生物学分析。植物细胞。2011; 23:911-22。
- 23。
Cui J,Li P,Li G,Xu F,Zhao C,Li Y等。ATPID:拟南芥蒂利亚纳蛋白质互乱数据库 - 植物系统生物学的一体化平台。核酸RES。2008; 36:D999-D1008。
- 24。
Lee K,Thorneycroft D,Achuthan P,Hermjakob H,IDEKER T.使用文献策划和预测的蛋白质 - 蛋白质相互作用数据集进行制图植物杂志。植物细胞。2010; 22:997-1005。
- 25.
玫瑰pw,prlića,altunkaya a,bi c,布拉德利ar,christie ch,等。RCSB蛋白质数据库:蛋白质,基因和3D结构信息的整合视图。核酸RES。2017; 45:D271-81。
- 26.
Stein A,Russell RB,Aloy P. 3Did:已知三维结构的相互作用蛋白质结构域。核酸RES。2005; 33:D413-7。
- 27.
李志强,李志强,李志强,等。MINT,分子相互作用数据库:2012更新。核酸学报2012;40:D857-61。
- 28.
Orchard S,Ammari M,Aranda B,Breuza L,Briganti L,Broackes-Carter F等人。Mintact项目 - 作为11个分子交互数据库的常见策策平台。核酸RES。2014; 42:D358-63。
- 29.
Lamesch P,Berardini Tz,Li D,Swarbreck D,Wilk C,Sasidharan R等人。拟南芥信息资源(TAIR):改进的基因注释和新工具。核酸RES。2012; 40:D1202-10。
- 30。
Salwinski L,Miller Cs,Smith Aj,Pettit Fk,Bowie Ju,Eisenberg D.互动蛋白质数据库:2004年更新。核酸RES。2004; 32:D449-51。
- 31。
李华,杨胜,王超,周勇,张志强。AraPPISite:蛋白质精细相互作用位点注释数据库拟南芥蒂利亚纳.植物学报。2016;92:105-16。
- 32。
Keshava Prasad Ts,Goel R,Kandasamy K,Keerthikumar S,Kumar S,Mathivanan S等人。人类蛋白质参考数据库-2009更新。核酸RES。2009; 37:D767-72。
- 33。
UniProt财团。UniProt:通用的蛋白质知识库。核酸Res. 2017;45: D158-69。
- 34。
Sonnhammer el,Östlundg. inparanoid 8:273蛋白蛋白质的矫形分析,大多是真核生物。核酸RES。2015; 43:D234-9。
- 35。
顾华,朱平,焦元,陈敏。PRIN:一种水稻相互作用网络的预测。BMC生物信息学。2011;12:161。
- 36。
基因本体联盟。基因本体(GO)数据库和信息学资源。核酸RES。2004; 32:D258-61。
- 37.
Ashburner M,Ball Ca,Blake Ja,Botstein D,Butler H,Cherry JM等。基因本体:生物学统一的工具。NAT Genet。2000; 25:25-9。
- 38.
于国刚,李飞,秦勇,波新,吴勇,王绍峰。基于语义相似度的GO词与基因产品语义相似度度量方法。生物信息学。2010;26:976-8。
- 39.
复杂网络研究的图形软件包。acta optica sinica . 2006; 29(3): 497 - 503。
- 40。
MultiLoc2:整合系统发育和基因本体论术语改进亚细胞蛋白定位预测。BMC生物信息学。2009;10:274。
- 41。
杨志强,杨志强,杨志强。全基因组表达数据与蛋白质相互作用的关系。基因组研究》2002;12:37-46。
- 42。
巴雷特等。NCBI GEO:功能基因组学数据集更新档案。核酸学报2013;41:D991-5。
- 43。
董胜,刘v,宋锐,吴勇,等。全蛋白质组,基于结构的蛋白质相互作用预测/新的分子相互作用观察器。植物杂志。2019;179:1893 - 907。
- 44。
altschul sf,gish w,miller w,myers ew,lipman dj。基本的局部比对搜索工具。J Mol Biol。1990; 215:403-10。
- 45。
Interactome3D:添加蛋白质网络的结构细节。Nat方法。2013;10:47-53。
- 46。
Sali A,Blundell TL。空间束缚满足的比较蛋白质模型。J Mol Biol。1993年; 234:779-815。
- 47。
丘脑N,Gursoy A,Nussinov R,Keskin O.通过使用棱镜的界面在界面的进化和结构相似性通过匹配进化和结构相似性来预测蛋白质 - 蛋白质相互作用。NAT PROTOC。2011; 6:1341-54。
- 48。
齐华,蒋志强,张凯,杨树华,何峰,张志强。PlaD:植物对病原菌的防御反应的转录组学数据库,为植物免疫系统的研究提供了新的认识。基因组学、蛋白质组学和生物信息学。
- 49.
Franz M, Lopes CT, Huck G, Dong Y, Sumer O, Bader GD。js:一个用于可视化和分析的图理论库。生物信息学。2016;32:309-11。
- 50。
玫瑰,希尔德布尔兹。NGL查看器:用于分子可视化的Web应用程序。核酸RES。2015; 43:W576-9。
- 51。
Kovalev MS, Igolkina AA, Samsonova MG, Nuzhdin SV。对农业植物有害编码突变进行分类的管道。植物学报2018;9:1734。
- 52。
科诺TJY, Lei L, Shih CH, Hoffman PJ, Morrell PL, Fay JC。比较基因组学方法可以准确预测植物中的有害变异。G3(贝塞斯达)。2018; 8:3321-9。
- 53。
Sahni N,Yi S,Taipale M,Fuxman Bass Ji,Coulombe-Huntington J,Yang F等人。人类遗传障碍的广泛大分子相互作用扰动。细胞。2015; 161:647-60。
- 54。
David A, Sternberg, MJ。蛋白-蛋白界面核心和边缘残基错义突变对人类疾病的贡献。中华医学杂志。2015;427:2886-98。
- 55。
王志强,王志强,王志强,等。蛋白质相互作用位点单核苷酸变异的分布及其与小等位基因频率的关系。蛋白质科学。2016;25:316-21。
确认
我们感谢那些通过使社区自由访问的数据/数据库/软件来构建Plapisite的科学家/开发人员。
资金
这项工作得到了北京自然科学基金会的批准(5172021)的支持。资金机构在研究设计,数据收集,分析和解释中没有作用,决定发布或准备稿件。
作者信息
隶属关系
贡献
XY和HL进行了分析,建立了数据库并起草了手稿。ZZ和HL构思并指导了这项研究。SY、HQ、TW协助数据库建设。ZZ修改了原稿。所有作者阅读并批准了提交手稿的最终版本。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:TableS1。
plapisite中13株植物的PPI编号分布。表S2。13植物的实验验证PPI的数量。表S3。六种模型生物的实验验证PPI的数量。表S4。用于13植物的GO注释团。表S5。13植物的亚细胞共定位比例。表S6。已知与预测交互站点相关的突变信息。
附加文件2:图S1。
1000随机网络和预测网络的平均蜂窝分量相似性的分布。图S2。1000个随机网络和预测网络的平均生物工艺相似性的分布。图S3。1000个随机网络与预测网络的平均分子函数相似度分布。图S4。1000个随机网络和预测网络的平均亚细胞共定位比例的分布。图S5。1000个随机网络与预测网络的平均表达式轮廓相似度分布。图S6。维恩图显示了两个预测PPI集和一个实验PPI集之间的重叠PPI的数量。图S7。PPI的主要子网。用户可以替代地导出子网进行进一步分析。图S8。复杂结构构建与交互站点分配的预测平台。(a)预测平台界面。用户可以提交查询PPI的两种蛋白质序列以检索复杂结构和相应的交互站点。(b)预测结果示例。可以在此页面上下载预测的复杂结构和相应的交互站点。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
杨,X.,杨,S.,Qi,H.等等。plapisite:植物蛋白质 - 蛋白质相互作用位点的综合资源。BMC植物杂志20,61(2020)。https://doi.org/10.1186/s12870-020-2254-4
已收到:
接受:
发表:
关键词
- 植物
- 数据库
- 蛋白质复合物的三维结构
- 蛋白质 - 蛋白质相互作用位点
- 域域交互
- Domain-motif交互
- Interolog