摘要
背景
Exserohilum turcicum是高粱和玉米的重要病原菌,引起高粱叶枯病和北方玉米叶枯病。由于同一病原体可以侵染两种最重要的粮食作物并造成重大损失,因此研究植物-病原体进化和研究两种植物的共同抗性机制是一种理想的病害系统.识别参与的高粱基因大肠turcicum对此,我们进行了全基因组关联研究(GWAS)。
结果
利用高粱转化面板在三个环境中进行评估,我们共鉴定了216个显著标记。基于与重要标记的物理连锁,我们共检测到113个独特的候选基因,其中一些基因在植物防御中具有已知的作用。此外,我们还比较了已知的在抵抗中起作用的玉米基因大肠turcicum随着关联映射结果,并发现在两种作物中赋予抵抗的基因的证据,提供了玉米和高粱之间的共同抵抗的证据.
结论
利用遗传学方法,我们确定了共同的遗传区域,赋予抗性大肠turcicum玉米和高粱都有。我们鉴定了几个有希望的高粱抗叶枯病候选基因,其中包括r基因介导的抗性相关基因。我们在理解寄主抗性方面取得了重大进展大肠turcicum,这对减少这种重要病原体造成的损失至关重要。
背景
将寄主植物的抗性从一种转化到另一种有利于抗病品种的发展。此外,病原体进化的知识可以为疾病管理提供信息,包括宿主抗性。微生物变成植物病原体的一个过程是病原体从一个宿主跳到一个新物种[1].当病原体转移到一个新的宿主时,来自原病原体系统的知识就可以转化到新的作物品种上。高粱是世界上第五大最重要的谷物作物,生物胁迫限制了高粱的产量。寄主抗性对生物胁迫的管理至关重要。高粱与玉米关系密切,但对高粱的生物抗逆性知之甚少。
叶面真菌病原体Exserohilum turcicum(通过)。伦纳德和萨格斯SetOphaeria Turcica(Luttr)。K. J. Leonard & Suggs),是玉米和高粱的病原菌,引起北方玉米叶枯病(NCLB)和高粱叶枯病(SLB)。玉米和高粱是两种最重要的谷类作物,都易受其影响大肠turcicum.在玉米中,NCLB被认为是美国最重要的疾病之一[2].据估计,NCLB导致2012年至2015年间损失了2790万吨玉米,这是由于疾病最广泛的损失。在高粱中,SLB被认为是一个重要的真菌疾病。如果感染发生在胰棒的出现前,它可能导致谷物产量损失高达50%[3.].这种疾病在高湿度和中等温度的地区最具破坏性[4].值得关注的是,一些地区已采用高度敏感品种进行生产[5].在玉米上,疾病首先出现在叶子上的小,棕褐色斑点,以及高粱,作为小红斑点。斑点扩大并与红线或棕色边界放入长期椭圆的病变。边框可以根据基因型在两个宿主中都有各种颜色。
大肠turcicum在墨西哥的玉米共同进化,随后跳跃导致高粱疾病[6].一个单一位点决定了玉米的寄主特异性第二个单一位点决定了高粱的寄主特异性[7].病原体能够在现场进行性繁殖,并使用混合生殖策略[8,9].宿主特异性的简单遗传架构和该领域的性繁殖发生率使主持人跳跃很可能。玉米和高粱之间观察到遗传分化大肠turcicum分离株,来自两个寄主的分离株之间的基因流动已经被观察到,表明玉米和高粱特异性分离株在自然界中交配[9].
这种病原体具有很高的进化潜力,其特征是在田间进行有性繁殖的能力和较大的种群规模,这强调了发展持久抗性的重要性,特别是对玉米和高粱都有效的抗性.虽然存在化学控制和文化方法以控制叶片枯萎,但种植抗性品种是最经济和环保的疾病控制方法[10].玉米的寄主抗性已经得到了很好的研究[11,12,13,14,15,16,但两种作物的抗性之间的关系尚不清楚。
对玉米的抗性进行了定性和定量的描述。已经确定了几个对NCLB有效的主要基因,包括Ht1, Ht2, Ht3, HtN和ht4[17].NCLB的数量抗性也得到了充分的研究,涉及的几个基因包括泛,ZMREM6.3.,以及咖啡酰辅酶a o -甲基转移酶[11,12,13].但是,抵抗力大肠turcicum对高粱的理解并不透彻。对高粱的寄主抗性的研究很少大肠turcicum[18,19].之前的研究假设玉米和高粱具有共同的抗性机制,即在高粱5号染色体上存在一个高度保守的CC-NB-LRR编码基因簇大肠turcicum[20.].然而,以前还没有研究发现玉米和高粱的抗性遗传变异。
高粱转换面板(SCP)是一系列线的集合,以便对精英线进行几代人进行异国情调的线路[21].该面板包括约800个已与Tx406回交的转换系,因此基因组大部分是具有早熟和矮化的渐渗的外来亲本[22].SCP由高粱所有5个亚群体的个体组成。SCP非常适合于抗病定位,因为这些株系是光周期不敏感和矮化的,创造了同质性来标准化抗病评估,但也有足够的等位基因多样性来识别新的抗病等位基因。
我们的中央假设是玉米和高粱占用机制。本研究的目的是i)鉴定高粱中与宿主抗梭菌相关的基因座;II)鉴别候选基因的SLB抗性;III)比较玉米和高粱的遗传建筑;iv)识别玉米和高粱之间的共同电阻机制.
结果
电阻的评估大肠turcicum
我们分别于2016年和2017年在田间和2018年在温室对SLB的SCP进行了评估。Line对所有现场环境都非常重要(P < 0.0001)温室潜伏期(P= 0.0464),但温室DLA测量值(P = 0.2187) (Table1).因此,我们在进一步的分析中没有包括温室DLA数据。在所有的野外环境中,我们都观察到正偏态,一些株系完全抵抗。对于2018年的IP数据,我们观察到一个双峰分布。尽管田间和温室数据的分布存在差异,但我们发现了显著的相关性(P< 0.05),且显著正相关(系数= 0.52;P < 0.01) between the two field-collected datasets (Table2).由于SLB开花后进展,因此根据开花时间将人口分成五组,以控制成熟度和疾病严重程度之间的关系。我们在现场环境中没有发现开花时间和疾病严重程度之间的显着相关性,但正相关性弱(系数= 0.07;P< 0.10)。
高粱由五个群组成,我们检查了每组的平均抵抗力。我们没有检测到亚步骤之间的显着差异(附加文件3.:图S1)。Kafir的平均抗性最高。所有群体都有高度敏感品系,说明没有一个品种具有一致的抗性。相反,所有的种族都含有有助于抗性和易感性的等位基因。
与抗病相关的显著snp大肠turcicum在高粱
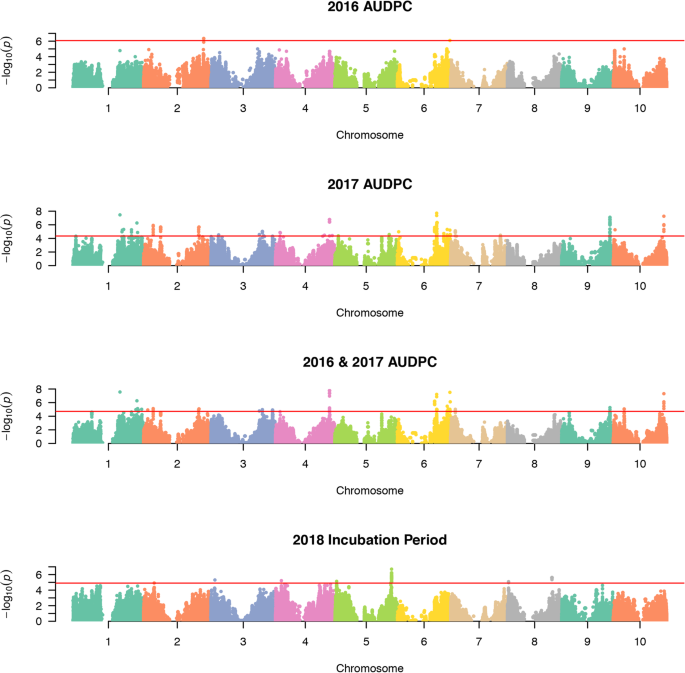
我们发现许多基因座参与了对SLB的抗性。我们分别使用2016年、2017年、2016年和2017年的AUDPC组合和2018年的IP数据集识别了3、152、66和43个有意义的标记(图)。1).我们在所有染色体上检测到显著的SNPs (FDR < 0.10)。在合并数据集中,4号染色体上62,185,882 - 62,289,470 bp的区域关联最显著(图)。2).在2017年的数据集中,6号染色体是最重要的。IP分析中最重要的SNPs位于第5染色体,从2017年数据集的重要关联中约有2mb。
高粱抗病基因大肠turcicum
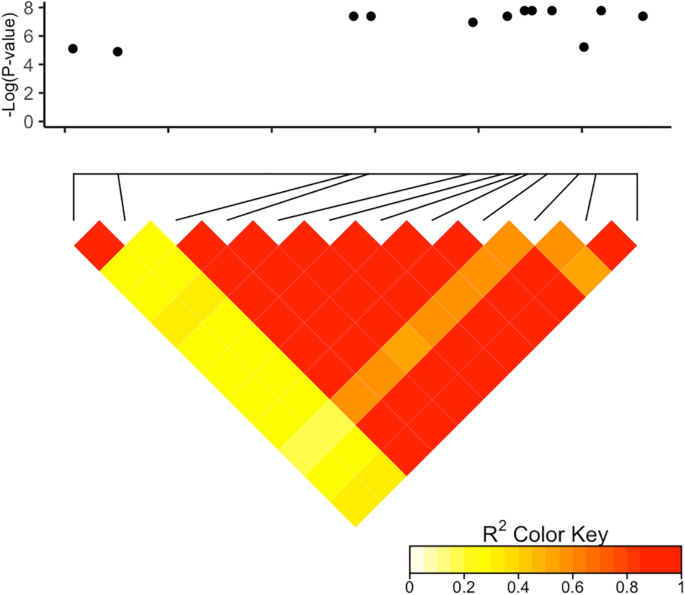
利用BTx623参考序列,根据显著snp的物理位置鉴定候选基因。我们总共鉴定了113个独特基因(附加文件)1:表S1)。根据罗斯福总统的调整,排名前十的基因P-values,如表所示3..在约62 MB的染色体4上鉴定了总共23个显着的SNP。牵伸区域为103kb并含有11个基因。它由2017和组合数据集涉及。我们在该区域内检查了LD,发现了两个LD块在该区域内具有重要关联(图。2).有几个基因可能参与位于103千克地区内的植物防御,包括氧化应激3(Sobic.004G279700.1)的高粱垂直,烟草病毒繁殖蛋白3,含有蛋白质的重金属相关结构域,以及蛋白质磷酸酶。氧化应激3邻牙和蛋白质磷酸酶存在显着的SNP。
其他区域的几个候选基因与植物防御有关,包括一个创伤反应家族蛋白,以及一个谷胱甘肽s -转移酶编码基因。生长素反应与生长素外排载体蛋白和生长素反应因子有关。信号传导还涉及到一个丝裂原活化蛋白激酶,在其他几个蛋白激酶。在涉及的蛋白激酶中,一个具有富亮氨酸结构域,另一个具有壁相关激酶编码结构域。此外,还包含了一个包含抗病蛋白的NB-ARC结构域的基因。氧化应激相关过程与过氧化物酶超家族蛋白编码基因有关。该基因在类胡萝卜素、叶绿素和赤霉素(GA)生物合成中起关键作用。
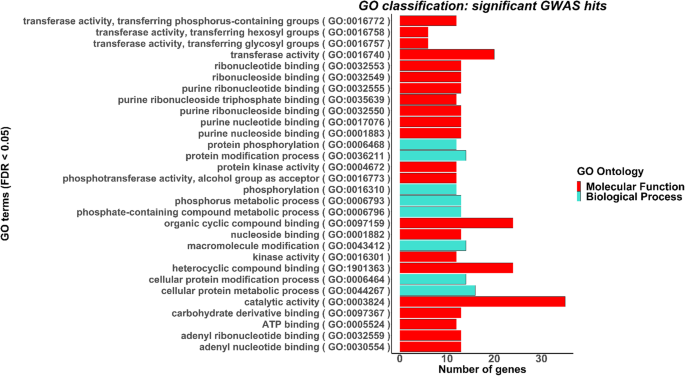
评估候选基因功能意义的SEA在GWAS中鉴定出30个显著富集的GO项,其中8个在生物过程中,22个在分子功能中。生物过程中丰富的氧化石墨烯术语主要包括蛋白质磷酸化过程、蛋白质修饰过程、蛋白质代谢过程和磷代谢过程(图1)。3.).分子功能丰富的氧化石墨烯活性包括激酶活性、磷酸转移酶活性、ATP结合、杂环化合物结合和催化活性(图)。3.).
玉米与高粱耐受的比较大肠turcicum
高粱抗病性的遗传结构与玉米相似,涉及的基因座较多。我们整理了一份36个玉米基因的名单,这些基因在玉米中发挥了最有力的作用大肠turcicum基于以前的地图研究的抵抗(附加文件2:表S2) [12,13,14,16,23,24,25,26].我们鉴定了这些玉米基因的高粱同源性,并将它们与我们的关联作图结果进行了比较。我们观察到SCP的长距离连锁不平衡高达1 Mb,因此考虑了玉米衍生候选基因1 Mb内的任何关联(附加文件)4:图S2)。在36个高粱候选基因中,有12个与玉米候选基因在1 Mb范围内存在显著相关(附加文件)2:表S2)。为了确定是否有明显多于预期的基因是偶然的,我们进行了排列测试。我们随机选择了36个基因,并找出了这些基因中有多少接近显著关联。基于排列检验,我们得出结论,我们在1 Mb范围内发现了12个显著相关基因,这是非常显著的(P < 0.01).
在比较分析中,最接近的候选地基因是锌指195 kB,来自最近的高粱协会[23].一种remorin (SORBI_3001G460300)与玉米抗NCLB有关[12]为394 Kb,是高粱中一个显著的关联。此外,在玉米中发现的基因类在高粱中也发现了。在高粱中发现了一个GST编码基因(Sobic.006G085100),在玉米中发现了一个GST基因,因为它参与了多种疾病的抗性[14].
讨论
我们开发了一种健壮的、高通量的方法来田间筛选高粱的SLB。利用这种方法,我们发现了基因型之间的显著差异和几个显著的关联,从而证明了它的实用性。我们发现kafir是,平均来说,最耐药的,这与之前的研究一致,kafir类型是最耐药的[5].
环境条件影响疾病的发展,特别是田间与温室。我们发现基于实地的数据集之间的显著关联是一致的。2016年,天气更有利于疾病的发展,观察到的疾病也更多。这可能解释了我们观察到的2016年和2017年结果之间的一些差异。孵化期是温室研究中更为可靠的表型测量指标。由于表型测量之间的内在差异以及两种环境之间缺乏相关性,我们没有进行联合温室田GWAS。田间和温室之间缺乏对应关系也在涉及的其他研究中观察到大肠turcicum,特别是在玉米中,在那里有NCLB QTL,在该领域有效但不在温室中有效[27].
已经为高粱的基因组关联映射开发了几种资源[22,28,29].关联映射已用于高粱以进行多种特征,已成功用于识别给予特征的基础知识的基因[30.,31].这里我们使用SCP,因为所有的株系都在伊利诺斯州中部开花,植株高度相对均匀。这些都是重要的因素,在评估面板上的疾病是叶面和加强后开花。值得注意的是,SCP的设计阻止了某些基因组区域的检测,因为用于生成SC系的杂交方案[22].因此,我们不会检测到在该群体中固定的那些基因座附近的关联。
初步证据表明,SCP中可能存在一个主要基因分离。IP数据的双峰分布表明,在这一人群中可能有基因延缓疾病症状的发作。NCLB主基因HtN1延长孵化时间和潜在的时期[32.].HtN1已经在玉米中克隆并编码一种壁相关激酶[24].高粱细胞壁相关激酶与细胞壁相关激酶最接近的显著关联为459 Kb [24].Martin等人发现玉米的主要基因存在于高粱中[20.,并且在高粱中观察到了品种结构大肠turcicum隔离(5].值得注意的是,并不是所有的主要基因都已在玉米中克隆出来,而未克隆的基因可能会在这个面板中产生抗性。对双亲群体中潜伏期不同的双亲群体进行进一步调查,以确定该群体中是否存在延迟疾病发作的主要基因,以及是否存在HtN1存在于高粱中。
我们在这项研究中鉴定了113个候选基因(附加文件)1:表S1)使用严格阈值并具有非常重要的FDR调整P-Values,表明它们可能是真正的积极成果。此外,在间隔内用几个重要关联聚类表示区域,其可以指示QTL下面的远程LD或多基因。对于对这种疾病的抗性抗性的多种基因是常见的[12,13].染色体4区中有趣的候选基因包括蛋白质磷酸酶和氧化应激的高粱。需要进一步的工作来跟进这些基因并辨别它们在SLB阻力中的作用。
通过GWAS鉴定出了几个有趣的候选基因,包括已知参与植物防御的蛋白质磷酸化在内的几个生物过程也与SEA有关。通过基因定位,筛选出一个编码PDS的基因(Sobic.006G177400)。PDS的破坏增强了病毒积聚[33.,对该基因的进一步研究是值得的。候选基因为含抗病蛋白(Sobic.002G053300)的NB-ARC结构域。抗性基因通常包含NB-ARC信号域[34.],在种群中观察到的完全抗性可能表明种群中存在主要基因。IP关联图中最重要的候选基因之一是F-box编码基因。F-box基因被认为可以调节R基因的表达[35.].在IP数据集中还有其他一些有趣的关联,包括氯喹抗性转运体样转运体2的高粱同源物。在Arabidposis,缺乏这种基因的突变体对疫霉感染(36.].
结果表明,高粱抗病性的遗传结构与玉米相似,且涉及多个位点。玉米完全抗大肠turcicum很少见[14,37.]但在这项研究中,我们发现了几条完全抗拒的线。这可能是由于本研究中使用的病原体菌株或环境不太有利于感染。在任何情况下,高粱可能会更具抵抗力大肠turcicum比玉米多,并具有抗性等位基因。
我们发现玉米和高粱对由高粱引起的病害有共同的抗性遗传区域大肠turcicum.这与其他系统的研究形成了对比。例如,黑麦草和谷物对真菌病原体的抗性QTL在全基因组水平上并不一致[38.].然而,值得注意的是,在这项研究中,我们检测了不同植物宿主对相同真菌的抗性。在基因层面,有数量疾病基因的例子,如POQR在多个主机中接受了会聚演化和赋予阻力[39.] 和LR34赋予小麦,玉米和高粱的几种疾病的抵抗[40,41.,42.,43.].还需要进一步的研究来确定是否相同的基因导致了抗药性大肠turcicum在玉米和高粱。
结论
综上所述,这是第一次利用全基因组关联作图技术来识别与SLB抗性相关的基因组区域。SCP包括高度抗SLB的行。这将有助于高粱抗病育种,因为已鉴定的标记可用于培育抗病品种。我们鉴定了113个候选基因,包括在植物防御中有已知作用的基因和几个与主要抗病基因有关的基因。我们比较了玉米和高粱的抗性,发现两种作物的抗性遗传结构相似。我们发现玉米和高粱之间有共同的抗性机制,12个候选高粱基因与已知的玉米抗性基因的同源性在1 Mb以内。
方法
表现型
植物材料
我们评估了SCP [222016年和2017年,在伊利诺伊州厄巴纳市作物科学研究和教育中心的田间,以及2018年在伊利诺伊州厄巴纳市植物护理设施的温室。转化系最初是由一个优秀的供体与外来的祖先系回交4次,在F选择2矮化和光周期不敏感植物的一代[21].SC系的种子最初从美国农业部作物系统研究实验室(Lubbock, TX, USA)获得。根据开花时间,每株SCP被分配到5个组,因为开花时间与抗性之间有很强的相关性大肠turcicum[14].集合独立于子群体。
实验设计
在现场试验中,使用R包创建了不完全块设计,每个组有两个重复。agricolae”[44.,45.]并且每个块在随机位置中用一个易感校验线(TX623)增强。每条线被种植在单行情节中,并遵循中央伊利诺伊州的标准农艺法。在种子上用围裙(Mefenoxam; syngenta,瑞士)和概念(fluxofenim; syngenta,瑞士)进行处理之前。地块是以50种种子/行的密度植物种植的,含有3.65米,含有0.91米的小巷。我们分别在2016年和2017年种植了705和679条线路。由于种子可用性,2017年评估了较少的线条。
对于温室实验,在每个块中的两个检查线,TX623(易感性)和SC0283(抗性)中的增强设计中评估了一个复制。温室评估是在Urbana,IL于2018年进行的,使用596系列,其中一株植物在一个加仑的锅中装满通用灌封混合物。将条件设定为12/12-H光暗循环和30/20°C的日夜温度。
疾病筛查
我们用了三个大肠turcicum从伊利诺斯州高粱叶片中分离15st003、15st008和16st001菌株,用固体接种物接种大田生长的植株。为了产生固体接种物,分离菌从−80℃储存的甘油转移到乳糖-酪蛋白水解琼脂(LCA),并在室温下进行12/12-h光照-黑暗循环2-3周的培养。固体高粱基质由2200 ml未经处理的高粱颗粒与1375 ml蒸馏水在高压釜袋中混合制成。谷物浸泡一夜,然后蒸压两次,每次20分钟。高压灭菌谷物接种一种大肠turcicum通过淹没每个LCA板的孢子悬浮液,其中约8毫升DDH2o,用玻璃棒脱落孢子,将约5ml未稀释的孢子悬浮液移液到每个袋中。每袋用单一分离物培养。用12/12-H光暗循环在室温下温育茵瓜2-3周。每天重新分配谷物以避免形成丛。用不同分离物的袋子紧接在接种中,以均衡袋中的孢子浓度。通过施加大约¼茶匙(1.5mL)的高粱谷物,在5-6叶阶段接种植物大肠turcicum在螺纹。
在温室试验中,4周龄植株接种0.5 ml大肠turcicum液体接种物放置在螺旋中[27].我们在LCA平板上培养相同的真菌菌株,如上所述,但将悬浮液的浓度调整为4 × 103.接种后,将植株置于雾室,保持高湿度过夜,每15 min头顶喷10 s。
疾病评估
患病叶面积(DLA)以目测方式评估,分级范围从0到100,增量为5% [46.].0分表示该样地所有植株均健康,未见病变,100分表示该样地所有植株完全坏死。病叶面积(DLA)在开花后,间隔7天,每样地进行3次评估。对于温室,我们评估了主要的DLA [47.]在接种后14、21和28 d。此外,每天检查植物的病变形成情况,并记录接种后第一次出现病变的天数。
基因分型
Fernandes等人通过基因分型测序获得了107,421个SNPs(以下称为目标集)的数据集[48.]和Thurber等人。[22].为了增加靶面板的标记密度,使用全基因组重新测序数据集(以下称为参考基因型集)用于抵御未键入的SNP [49.].参考集由239个个体和5,512,653个锚定在高粱双色参考基因组3.1(https://phytozome.jgi.doe.gov) [50.].我们过滤了杂合子(> 10%),SNP覆盖率(<4X)和缺失基因型(> 40%)的参考组。另外,在归属之前,还滤出了具有次要等位基因计数<3和深度<3的SNP。最终参考集包括239个个体和4,268,905个SNP。
在归样前,使用conformity -gt [51.].这一步排除了在参考基因型中不存在的目标snp,并调整了基因组位置和染色体链以匹配目标和参考集。因此,纳入用于imputation的34498个目标集SNPs的小等位基因频率为> 1%,位点与参考面板匹配。采用gt选项,window = 80000 bp, overlap = 10000 bp, ne = 150000 bp,对未分型snp进行排序。过滤后,使用Beagle 4.1版本来输入缺失的基因型(选项“gtgl”),然后是一个阶段(选项“gt”)步骤[52.].我们使用了一个1500 BP的窗口,以及两个步骤的重叠。通过除去r的变体,使用基于连杆不平衡来修剪基因型数据集2值大于0.9,使用窗口大小为20和步长5个SNP [53.].然后使用GAPIT对标记进行筛选,寻找频率为0.05的次要等位基因[54.].我们使用GAPIT 3.0版本对现场数据集进行关联分析[54.].共有338651个标记被纳入分析。
数据分析
疾病进展曲线下面积(AUDPC)从DLA数据使用绝对方法与R包'的“AUDPC”函数计算Agricolae.'[44.,45.].使用SAS版本9.4 (SAS Institute Inc., Cary, NC)中实现的PROC MIXED函数运行线性模型,所有因素均拟合为随机效应。每年分别进行分析,以及结合现场数据进行分析。由于田间和温室环境之间的内在差异,没有将田间和温室数据结合起来。最初,模型拟合,包括设计因素和线(表1).对于Field DataSets,Set在年内嵌套,复制在年内设置内嵌套,并且块嵌套在集合中的复制中。使用使用受限制的最大可能性(REML)方法(REML)方法实现的WALD的Z-Test Statistics来确定随机因子在模型中包含随机因子的意义[55.].此外,还进行了似然比检验,以确定是否将因素纳入模型。2016年的AUDPC模型包括行、集、块和复制。2017年的AUDPC机型包括线和集。结合2016年和2017年的模式包括线,年,集,代表,块,环境,以及由环境交互的年。我们计算了2016年、2017年、2018年IP以及2016年和2017年合并数据集的最佳线性无偏预测因子(BLUPs)。没有对2018年的AUDPC进行进一步分析,因为这条线在分析中不显著。基于blup将2018年IP数据集分为两类。效应小于0的株系视为抗性株系,效应大于0的株系视为敏感株系。表型数据可在附加文件5S1:文件。
采用“CMLM”方法进行GWAS,使用GAPIT [54.],共包含四个主成分。10%的错误发现率被用来确定关联是否显著[56.].因为IP数据集是分类的,所以我们使用Logistic回归来使用PLINK版本1.9进行关联映射[53.].通过Gapit计算的主要成分包括在PLINK分析中。
利用“Hmisc”包中的“rcorr”函数对开花时间、株高和亚群体进行Pearson相关[57.[R.从Thurber等人获得开花时间和植物高度的数据。[22].杜克大学的HSD测试使用的是Agricolae.' 包裹 [44.],以确定不同亚种群间联合野外数据是否存在显著差异。
候选基因的选择
与基因组特征显著关联的物理邻近性被用来识别候选基因。BEDTools工具包被用来根据重要的SNP位置识别候选基因[58.,59.].如果显著的SNP是基因的,则包含SNP的基因被报道为候选基因。如果SNP是基因间的,则报告最接近的基因特征作为候选基因。利用agriGO v2.0通过单一富集分析(SEA)确定候选基因的功能意义[60.].
玉米候选基因和同步高粱基因
基于之前的研究,我们整理了一份候选玉米抗性基因列表[12,13,16,23,24].高粱同线同源性的筛选玉米候选基因是使用[61.].为了确定高粱中接近关联的同系物的数量是否显著,进行了排列检验。我们随机选择了36个基因,并确定这些基因中有多少在1mb的显著关联范围内。我们对这个测试进行了1000次迭代。
数据和材料的可用性
支持本文结论的数据集包含在本文(及其附加文件)中。基因型数据集可通过Figshare获得(doi:https://doi.org/10.6084/m9.figshare.11288204).
缩写
- GWAS:
-
基因组协会研究
- LD:
-
连锁不平衡
- NCLB法案:
-
北玉米叶枯萎
- QTL:
-
数量性状位点
- QTN:
-
定量特征核苷酸
- SCP:
-
高粱转换面板
- SLB:
-
高粱叶枯病
- SNP:
-
单核苷酸多态性
参考文献
- 1.
Stukenbrock eh,麦当劳。农业生态系统植物病原体的起源。Annu Rev phytopathol。2008; 46:75-100。
- 2.
Mueller DS, Wise KA, Sisson AJ, Allen TW, Bergstrom GC, Bosley DB, Bradley CA, Broders KD, Byamukama E, Chilvers MI等。2012年至2015年,美国和加拿大安大略省因病害造成的玉米产量损失估计。植物健康进展。2016;17(3):211-22。
- 3.
Frederiksen RA, Odvody GN。高粱病害概要。第二版:圣保罗:APS出版社;2000.
- 4.
Hennessy GG,Demilliano Waj,Mclaren CG。原发性变量对南部非洲高粱叶枯病严重程度的影响。植物病理学。1990; 80(10):943-5。
- 5。
疾病严重程度、发病率及小种SetOphaeria Turcica在乌干达的高粱。acta botanica yuni sinica(云南植物学报). 2011;31(3):383-92。
- 6。
Borchardt DS, Welz HG, Geiger HH。基因的结构SetOphaeria Turcica热带和温带气候中的人口。植物病理学。1998年; 88(4):322-9。
- 7。
作者简介:哈密。AH, Aragaki M.SetOphaeria Turcica.植物病理学。1975;65(3):280 - 3。
- 8。
王志强,王志强,王志强,王志强SetOphaeria Turcica在自然玉米田在泰国。Kasetsart J. 2014; 48(2):175-182。
- 9。
Nieuwoudt A,Human MP,Craven M,Crampton BG。群体的遗传分化Exserohilum turcicum来自南非的玉米和高粱。植物病理学研究。2018;67(7):1483 - 91。
- 10。
利用复杂性培育抗病作物。中国科学(d辑:科学)2018;19(1):21-33。
- 11.
杨强,何勇,杨丽,等。一种编码玉米咖啡酰辅酶A o -甲基转移酶的基因对多种病原体具有定量抗性。Nat麝猫。2017;49(9):1364 - 72。
- 12.
Jamann TM, Luo X, Morales L, Kolkman JM, Chung CL, Nelson RJ。在玉米中,一个鮣鱼基因与数量抗病有关。中国科学(d辑:地球科学)2016;
- 13.
Jamann TM, Poland JA, Kolkman JM, Smith LG, Nelson RJ。玉米数量抗病位点的基因组复杂性研究。遗传学。2014;198(1):333 - 44。
- 14。
Wisser RJ,Kolkman JM,Patzoldt Me,Holland JB,Yu J,Krakowsky M,Nelson RJ,Balint-Kurti PJ。玉米疾病抗性的多变量分析表明了一种抗血液遗传基础,并意味着GST基因。PROC NATL ACAD SCI U S A. 2011; 108(18):7339-44。
- 15.
Wisser RJ,Balint-Kurti PJ,Nelson RJ。玉米抗病性遗传建筑:公布研究的合成。植物病理学。2006; 96(2):120-9。
- 16。
波兰贾,布拉德伯里PJ,克兰特州,纳尔逊rj。玉米北部叶片枯叶的全基因组嵌套术。Proc Natl Acad Sci U S A. 2011; 108(17):6893-8。
- 17。
Welz Hg,莱格尔哈哈。抗北方玉米叶枯萎病的基因在不同的玉米种群中。植物品种。2000; 119(1):1-14。
- 18。
高粱对炭疽病和烟草枯病的抗性。植物品种。2016;135(3):318 - 22所示。
- 19。
Sharma R, Upadhyaya HD, Manjunatha SV, Rao VP, Thakur RP。高粱种质资源对叶面病害的抗性。工厂说。2012;96(11):1629 - 33所示。
- 20。
Martin T,Biruma M,Fridborg I,Okori P,Dixelius C.一种高度保守的NB-LRR编码基因集群有效SetOphaeria Turcica在高粱。BMC植物BIOL。2011; 11:151。
- 21。
Stephens JC, Miller FR, Rosenow DT。外源高粱向早期组合基因型的转化。作物科学。1967;7(4):396。
- 22。
Thurber CS, Ma JM, Higgins RH, Brown PJ。高粱对温带粮食生产适应性的回顾性基因组分析。基因组医学杂志。2013;14 (6):R68。
- 23.
丁J,Ali F,Chen G,Li H,Mahuku G,Yang N,Narro L,Magorokosho C,Makumbi D,Yan J. Genome-Wide Association Mappping揭示了玉米北方玉米叶片抗性的新源。BMC植物BIOL。2015; 15:206。
- 24.
Hurni S, Scheuermann D, Krattinger SG, Kessel B, Wicker T, Herren G, Fitze MN, Breen J, Presterl T, Ouzunova M,等。玉米抗病基因Htn1反对北玉米叶枯萎编码壁相关的受体样激酶。Proc Natl Acad Sci U S A. 2015; 112(28):8780-5。
- 25.
李应祥,陈丽,李超,石玉英,宋勇,张德成,张志强,李勇,等。随着试验条件的增加和标记密度的增加,发现了更多与玉米抗南方和北方叶枯病相关的遗传位点。Sci众议员2018;8(1):6848。
- 26。
van Inghelandt D,Melchinger Ae,Martinant JP,Stich B.北玉米叶枯萎的基因组 - 宽协会映射(SetOphaeria Turcica)巨大的商业玉米种质组中的抗性。BMC植物BIOL。2012; 12:56。
- 27。
Chung CL, Longfellow JM, Walsh EK, Kerdieh Z, Van Esbroeck G, Balint-Kurti P, Nelson RJ。影响不同真菌发病阶段的抗性位点:利用渐渗系进行玉米QTL定位和表征SetOphaeria Turcicapathosystem。BMC Plant Biol. 2010;10:103。
- 28。
Bouchet S,Olatoye Mo,Marla SR,Perumal R,Tesso T,Yu J,Tuinstra M,Morris GP。利用嵌套关联映射群体增加了全球高粱多样性中的适应性特征的力量增加。遗传学。2017; 206(2):573-85。
- 29。
Brenton ZW, Cooper EA, Myers MT, Boyles RE, Shakoor N, Zielinski KJ, Rauh BL, Bridges WC, Morris GP, Kresovich S.高粱生物能源开发、改良和开发的基因组资源。遗传学。2016;204(1):21-33。
- 30.
Adeyanju A,Little C,Yu J,TESSO T. Genome-Incive endia型抗性抗茎秆腐蚀性的关联研究。G3(Bethesda)。2015; 5(6):1165-75。
- 31.
Cuevas He,Prom LK,Cooper EA,Knoll Je,Ni X. Anthracnose的基因组 - 范围协会映射(炭疽菌sublineolum)美国高粱协会面板的抗性。植物基因组。2018; 11(2):1-13。
- 32。
Raymundo广告,Hooker Al,Perkins JM。基因的影响Htn论北方玉米叶枯病流行的发展。工厂说。1981;65(4):327 - 30。
- 33。
DeBlasio SL, Rebelo AR, Parks K, Gray SM, Heck MC.通过下调叶绿素去饱和酶破坏叶绿体功能,增强了蚜虫传播的韧皮部限制病毒的系统性积累。植物与微生物相互作用学报,2018;31(10):1095-110。
- 34。
van der Biezen EA, Jones JDNB-ARC结构域:植物抗性基因产物和动物细胞死亡调控因子共享的一种新的信号基序。咕咕叫医学杂志。1998;8 (7):R226-7。
- 35.
GOU M,Shi Z,Zhu Y,Bao Z,Wang G,Hua J. F-Box蛋白CPR1 / CPR30负调节R蛋白SNC1积累。工厂J. 2012; 69(3):411-20。
- 36.
Maughan Sc,Pasternak M,Cairns N,Kiddle G,Brach T,Jarvis R,Haas F,Nieuwland J,Lim B,Muller C等人。植物同源物恶性疟原虫谷胱甘肽稳态和应激反应需要氯喹抗肺炎术。Proc Natl Acad Sci U S A. 2010; 107(5):2331-6。
- 37.
关键词:玉米,北方叶枯病,抗性,数量性状,基因座中国科学(d辑:地球科学)2011;23(2):307-26
- 38。
赵永强,Barker R, Pfender W, Warnke S, Sim SC, Jung GH。黑麦草与谷类作物多重抗性的比较分析。acta photonica sinica . 2008; 37(4): 531-43。
- 39。
植物抗病基因POQR prolyl oligo peptidase基因的平行进化。公共科学图书馆麝猫。2017;13 (12):e1007143。
- 40.
刘国强,刘国强,刘国强,刘国强,刘国强,刘国强,刘国强,刘国强,刘国强。小麦Lr34多病原菌抗性基因对高粱的炭疽病和锈病具有抗性。生物技术学报2017;15(11):1387-96。
- 41.
Sucher J,Boni R,Yang P,Rogowsky P,Buchner H,Kastner C,Kumlehn J,Krattinger SG,Keller B.耐用的小麦疾病抗性基因LR34使玉米具有普通锈病和北方玉米叶枯病的抗性。生物技术学报2017;15(4):489-96。
- 42.
spelmeyer W, Singh RP, McFadden H, Wellings CR, huertta - espino J, Kong X, Appels R, Lagudah ES。利用小麦抗病位点Lr34 /Yr18间质缺失突变体的精细遗传和物理定位。中国科学(d辑:地球科学)2008;116(4):481-90。
- 43.
kattinger SG, Lagudah ES, Spielmeyer W, Singh RP, Huerta-Espino J, McFadden H, Bossolini E, Selter LL, Keller B.一种假设的ABC转运体赋予小麦对多种真菌病原体的持久抗性。科学。2009;323(5919):1360 - 3。
- 44。
De Mendiburu F. Agricolae:农业研究的统计程序;2014.
- 45。
R核心团队。统计计算的语言和环境。出版日期:R基础统计计算;2018.
- 46。
波兰JA, Nelson RJ。评价者变异性和不同评价表对QTL作图的影响。植物病理学。2011;101(2):290 - 8。
- 47.
陈志强,陈志强,陈志强,等。玉米抗北方叶枯病基因的克隆及定位。中国科学(d辑:地球科学)2010;121(2):205-27。
- 48.
费尔南德斯SB,迪亚斯KOG,费雷拉DF,布朗PJ。多性状、间接和性状辅助基因组选择对高粱生物量改良的效率。中国科学(d辑:地球科学)2018;
- 49.
Valluru R,Gazave EE,Fernandes SB,Ferguson Jn,Lozano R,Hirannaih P,Zuo T,Brown PJ,Leakey Adb,Gore Ma,等。有害的突变负担及其与高粱复杂性状的关联(高粱双色).遗传学。2019;211(3):1075 - 87。
- 50.
McCormick RF, Truong SK, Sreedasyam A, Jenkins J, Shu S, Sims D, Kennedy M, Amirebrahimi M, Weers BD, McKinley B, et al.;的高粱双色参考基因组:改进的组装,基因注释,转录组图谱和基因组组织的签名。植物j . 2018; 93(2): 338 - 54。
- 51.
Browning SR, Browning BL.利用局部单倍型聚类对全基因组关联研究进行快速准确的单倍型定位和缺失数据推断。中国科学(d辑:地球科学)2007;81(5):1084-97。
- 52.
布朗宁BL,褐变SR。基因型归责用数百万参考样品。我是j嗡嗡声的遗传。2016; 98(1):116-26。
- 53.
Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D, Maller J, Sklar P, de Bakker PI, Daly MJ,等。PLINK:全基因组关联和基于群体的连锁分析的工具集。中国科学(d辑:地球科学)2007;
- 54.
唐y,刘x,王j,李m,王q,田f,su z,pan y,liu d,lipka ae等。Gapit版本2:一种增强的基因组关联和预测的集成工具。植物基因组。2016; 9(2):1-9。
- 55.
Littell RC。SAS系统用于混合模型。SAS Institute Inc.;1996.
- 56。
Benjamini Y,Hochberg Y.控制虚假发现率 - 一种实用而强大的多种测试方法。J Royal Stat Soc系列B-Stat方法。1995年; 57(1):289-300。
- 57。
小哈勒尔的包裹“Hmisc”。在:R包版本;2019.
- 58。
昆兰:瑞士陆军基因组特征分析工具。12 11-34。
- 59.
Paterson AH, Bowers JE, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov A, et al.;的高粱双色基因组与草类的多样化。大自然。2009;457(7229):551 - 6。
- 60.
田涛,刘勇,严海英,尤强,易旭,杜智,徐文英,苏哲。agriGO v2.0:一种面向农业社区的GO分析工具,2017年更新。核酸Res. 2017;45(W1): W122-9。
- 61.
张勇,Ngu DW, Carvalho D,梁志坤,邱玉梅,Roston RL, Schnable JC。高粱和玉米亚基因组的差异调控同源基因。植物细胞。2017;29(8):1938 - 51。
致谢
我们要感谢伊利诺伊大学作物科学研究和教育中心协助进行田间试验。
资金
本研究由美国能源部科学办公室、生物与环境研究办公室(BER)资助,批准号为。DE-SC0019189。
作者信息
隶属关系
贡献
TJ,PJ和SM构思了这项研究。XZ,CK和TJ收集了表型数据。SF和PJ生成基因型数据集。XZ和TJ进行了关联映射。XZ和PA进行了比较分析。TJ和XZ编写并修改了手稿。所有作者均批准最终提交。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
全基因组关联图谱显示的所有基因。(CSV 50 kb)
附加文件2:表S2。
候选玉米基因及其高粱同源基因的列表。(CSV 6 kb)
附加文件3:图S1。
5个亚种群2016年和2017年AUDPC合并的箱形图。拦截不添加到blup中。各亚群体间无显著差异。
附加文件4:图S2。
高粱转换面板的长程联动不平衡。
附加文件5:文件S1。
高粱转化面板的表型数据。每个环境和组合环境的数据以blup形式呈现。(CSV 28 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
张旭东,费南斯,s.b.,凯撒,C。et al。玉米和高粱的保守防御反应Exserohilum turcicum.BMC植物BIOL.20,67(2020)。https://doi.org/10.1186/s12870-020-2275-z
已收到:
接受:
发表:
关键字
- 高粱叶枯病
- 全基因组关联映射
- 定量抗病性
- 北玉米叶枯萎
- Exserohilum turcicum
- SetOphaeria Turcica
- 高粱