- 研究文章GydF4y2Ba
- 开放获取GydF4y2Ba
- 发表:GydF4y2Ba
组织特异性和冷应答型lncrna的鉴定GydF4y2BaMedicago Truncatula.GydF4y2Ba通过高通量RNA测序GydF4y2Ba
BMC植物生物学GydF4y2Ba体积GydF4y2Ba20.GydF4y2Ba文章编号:GydF4y2Ba99.GydF4y2Ba(GydF4y2Ba2020.GydF4y2Ba)GydF4y2Ba
抽象的GydF4y2Ba
背景GydF4y2Ba
长链非编码rna (lncrna)在植物对环境胁迫的响应中发挥着重要的作用,作为基因表达的重要调节器。然而,lncrna是否以及如何参与植物冷驯化依赖的抗冻性仍然是未知数。GydF4y2BaMedicago Truncatula.GydF4y2Ba是豆科植物基因组学研究的一个突出模式,其特点是冷驯化特性。为了确定lncrna在植物冷胁迫反应中的作用,我们在豆科植物模型植物中进行了全基因组高通量测序GydF4y2BaM. Truncatula.GydF4y2Ba.GydF4y2Ba
结果GydF4y2Ba
对非冷处理叶片和根、冷处理叶片和根4个处理的12个样本进行RNA-seq数据采集GydF4y2BaM. Truncatula.GydF4y2BaJemalong A17幼苗。生成了总数为1.04亿的原始读数。其中,在质量控制(QC)之后,11.5亿滤波读取进行下游分析。从十二个样品中鉴定了大量24,368个独特的LNCRNA。在这些LNCRNA中,983和1288分别对叶片和根部的冷处理敏感。我们进一步发现,内肾上腺-LNCRNA对冷处理最敏感。冷响应的LNCRNA不均匀地分布在八个染色体上GydF4y2BaM. Truncatula.GydF4y2Ba幼苗具有明显的位置偏好。进一步分析显示,叶子和根之间的冷响应LNCRNA不同。预计LNCRNA的推定靶基因主要涉及蛋白质翻译,运输,代谢和核酸转录的过程。此外,串联阵列的网络GydF4y2BaCBF / DREB1GydF4y2Ba根据基因表达和染色体定位,对6号染色体上已报道的主要耐冻QTL区域的基因及其相关lncrna进行解剖。GydF4y2Ba
结论GydF4y2Ba
我们确定了一套全面的LNCRNA,对冷治疗有响应于GydF4y2BaM. Truncatula.GydF4y2Ba并在叶片和根中发现了组织特异性的冷响应lncrna。我们进一步剖析了潜在的调控网络GydF4y2BaCBFGydF4y2Ba代际RNA(GydF4y2Bamtcir1.GydF4y2Ba),GydF4y2BaMTCBF.GydF4y2Ba在响应和适应方面发挥着关键角色GydF4y2BaM. Truncatula.GydF4y2Ba冷压力。GydF4y2Ba
背景GydF4y2Ba
非编码rna (ncrna)的发现,包括短的(22-33个核苷酸)和长的(> 200个核苷酸)ncrna,改变了基因的传统定义[GydF4y2Ba1GydF4y2Ba那GydF4y2Ba2GydF4y2Ba].通过缺乏任何明显的开放阅读框架(ORF)的长期非编码RNA(LNCRNA)主要由RNA POL II转录,剪接,5'封端,甚至在3'末端的聚酰胺化[GydF4y2Ba2GydF4y2Ba那GydF4y2Ba3.GydF4y2Ba].除了RNA Pol ii衍生的lncrna外,也有报道称,由两种植物特异性dna依赖的RNA聚合酶RNA Pol IV和RNA Pol V转录的其他种类的lncrna [GydF4y2Ba4.GydF4y2Ba].根据其基因组起源,LNCRNA可以分为以下类别。(1)反义,当另一种转录物的一个或多个外显子分别在相对的股线上重叠时。(2)内肾内,当它们完全来自第二个转录物的内含子内。(3)代否,当它们位于两个基因之间的基因组间隔内[GydF4y2Ba2GydF4y2Ba].lncrna参与众多生物现象的调控,包括印迹基因组位点、塑造染色体构象和变构调节酶活性[GydF4y2Ba3.GydF4y2Ba那GydF4y2Ba5.GydF4y2Ba].lncrna在生物过程中发挥着多种机制的关键调节作用,如作为支架、诱骗或通过基因组靶向信号等GydF4y2BaCIS.GydF4y2Ba或GydF4y2Ba反式GydF4y2Ba,并导致靶基因下调或过表达[GydF4y2Ba6.GydF4y2Ba那GydF4y2Ba7.GydF4y2Ba].对植物中lncrna的研究表明,它们在广泛的生物过程中发挥重要作用,特别是在生殖发育和对环境胁迫的响应中[GydF4y2Ba3.GydF4y2Ba那GydF4y2Ba8.GydF4y2Ba].GydF4y2Ba
在植物中,在玉米中进行了LNCRNA的基因组鉴定[GydF4y2Ba9.GydF4y2Ba),拟南芥GydF4y2Ba10GydF4y2Ba],杨树[GydF4y2Ba11GydF4y2Ba],GydF4y2BaMedicago Truncatula.GydF4y2Ba[GydF4y2Ba12GydF4y2Ba], 番茄 [GydF4y2Ba13GydF4y2Ba和其他人。早期功能特征的植物lncrna来源于的调控途径GydF4y2BaFLC.GydF4y2Ba,参与拟南芥开花的大师阻遏物基因。lncrnasGydF4y2Ba冷风GydF4y2Ba和GydF4y2Ba冷空气GydF4y2Ba消极的调节GydF4y2BaFLC.GydF4y2Ba通过不同的模型[GydF4y2Ba14GydF4y2Ba那GydF4y2Ba15GydF4y2Ba那GydF4y2Ba16GydF4y2Ba].近年来,大量参与植物对非生物胁迫的反应的LNCRNA已经表现了[GydF4y2Ba17GydF4y2Ba那GydF4y2Ba18GydF4y2Ba那GydF4y2Ba19GydF4y2Ba].GydF4y2Ba
生长在温带和寒冷地区的植物可以通过暴露在较低的、不结冰的温度下一段时间来增强对冰冻的耐受性,这被称为冷适应[GydF4y2Ba20.GydF4y2Ba那GydF4y2Ba21GydF4y2Ba].冷驯化过程中的许多分子变化是对冷驯化诱导的冷冻耐受性的增强负责[GydF4y2Ba22GydF4y2Ba].CBF / DREB1(C-重复结合因子/脱水响应元件结合因子1)激活含下游CRT / DRE的冷调节(COR)基因,这反过来有助于提高植物的耐受性以冻结[GydF4y2Ba23GydF4y2Ba那GydF4y2Ba24GydF4y2Ba那GydF4y2Ba25GydF4y2Ba].GydF4y2BaICE1GydF4y2Ba[GydF4y2Ba26GydF4y2Ba],GydF4y2BaICE2GydF4y2Ba[GydF4y2Ba27GydF4y2Ba]三个密切相关GydF4y2BaCAMTAsGydF4y2Ba[GydF4y2Ba28GydF4y2Ba]已被确定为正稳压因素GydF4y2BaCBFGydF4y2Bas。积极的调节GydF4y2BaRDM4.GydF4y2Ba上的表情GydF4y2BaAtCBF2GydF4y2Ba和GydF4y2BaAtCBF3GydF4y2Ba表明,GydF4y2BaRDM4.GydF4y2Ba对于Pol II转录很重要GydF4y2BaCBFGydF4y2BaS,它在拟南芥对冷压力的耐受性中起着关键作用[GydF4y2Ba29GydF4y2Ba].最近的研究表明,冷活血浆膜蛋白激酶CRPK1磷酸化14-3-3蛋白,触发其核转移以损害转录因子CBF的稳定,用于在拟南芥中冷应激期间过度冷防反应的反馈[GydF4y2Ba30.GydF4y2Ba].的lncRNAGydF4y2BaSVALKAGydF4y2Ba被确定为负调控因子GydF4y2BaCBFGydF4y2Ba拟南芥的表达和植物抗冻性[GydF4y2Ba31GydF4y2Ba,然而,如何GydF4y2BaCBFGydF4y2Ba在冷置的情况下,LNCRNA激活基因仍有待探索。GydF4y2Ba
Medicago Truncatula.GydF4y2Ba是一年一度的牧草作物[GydF4y2Ba32GydF4y2Ba],并已成为豆类基因组学的重要模型[GydF4y2Ba33GydF4y2Ba那GydF4y2Ba34GydF4y2Ba那GydF4y2Ba35GydF4y2Ba].考虑到GydF4y2BaM. Truncatula.GydF4y2Ba与普通豆科植物牧草苜蓿(GydF4y2Ba紫花苜蓿GydF4y2Ba),它是一种有价值的材料,用于研究豆类植物中的环境胁迫的分子生理[GydF4y2Ba36GydF4y2Ba那GydF4y2Ba37GydF4y2Ba那GydF4y2Ba38GydF4y2Ba].苜蓿是一种冰冷的耐受豆科物种,能够在冷适应时能够积累冷适应特异性(CAS)蛋白[GydF4y2Ba36GydF4y2Ba那GydF4y2Ba39GydF4y2Ba那GydF4y2Ba40GydF4y2Ba].的GydF4y2Ba卡斯商学院GydF4y2Ba是同源的GydF4y2Ba天哪GydF4y2Ba基因GydF4y2BaCIS.GydF4y2Ba- 通过CBF / DREB1因素进行了解[GydF4y2Ba36GydF4y2Ba那GydF4y2Ba38GydF4y2Ba].与生长在温带的其他植物类似,GydF4y2BaM. Truncatula.GydF4y2Ba植物有冷驯化性状[GydF4y2Ba36GydF4y2Ba那GydF4y2Ba41GydF4y2Ba].串联阵列GydF4y2BaCBF / DREB1GydF4y2Ba基因定位于6号染色体的主要耐冻QTL区GydF4y2BaM. Truncatula.GydF4y2Ba[GydF4y2Ba42GydF4y2Ba].以识别冷应答的lncrnaGydF4y2BaM. Truncatula.GydF4y2Ba对4个处理(非冷处理叶片、非冷处理根系、冷处理叶片、冷处理根系)进行全基因组高通量测序。我们确定了一套全面的LNCRNA,对冷治疗有响应于GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。此外,可能的监管网络GydF4y2BaCBFGydF4y2Ba代际RNA(GydF4y2Bamtcir1.GydF4y2Ba),GydF4y2BaMTCBF.GydF4y2Ba年代GydF4y2BaM. Truncatula.GydF4y2Ba被揭露了。GydF4y2Ba
结果GydF4y2Ba
识别冷响应LNCRNAGydF4y2BaM. Truncatula.GydF4y2Ba幼苗通过高通量测序GydF4y2Ba
识别冷应答型lncrnaGydF4y2Ba那GydF4y2Ba我们对12个cDNA文库进行了3个重复的RNA测序,分别处理了4个处理(非冷处理叶片、nt叶片;NT-roots Non-cold-treated根;Cold-treated叶子,CT-leaves;冷处理根,ct根)GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。以3周龄叶片和根部的mrna为载体,通过合成接头构建cDNA文库GydF4y2BaM. Truncatula.GydF4y2Ba分别在4°C冷处理和26°C非冷处理5 h。因为在动物和植物中lncrna的表达水平很低[GydF4y2Ba2GydF4y2Ba],进行深度RNA测序,从12个cDNA文库中获得超过1,204,140,634个原始reads(表GydF4y2Ba1GydF4y2Ba).为了评估RNA测序(RNA-SEQ)获取的数据质量,通过使用FASTQC通过PHRED样算法分配了读取中的每个基础的基础[GydF4y2Ba43GydF4y2Ba].结果表明,数据可信度高,Q20大于95% (Supplementary Table S1)。将测序结果映射到GydF4y2BaM. Truncatula.GydF4y2BaA17基因组(补充表S4),构建了一种综合管线以识别独特的LNCRNA。这导致识别超过30,000 mRNA和10,000个独特的LNCRNA,每增加12个cDNA文库(表GydF4y2Ba1GydF4y2Ba;附加文件GydF4y2Ba8.GydF4y2Ba:数据集S1;附加文件GydF4y2Ba9.GydF4y2Ba:数据集S2;附加文件GydF4y2Ba10GydF4y2Ba:数据集S3;附加文件GydF4y2Ba11GydF4y2Ba:数据集S4)。GydF4y2Ba
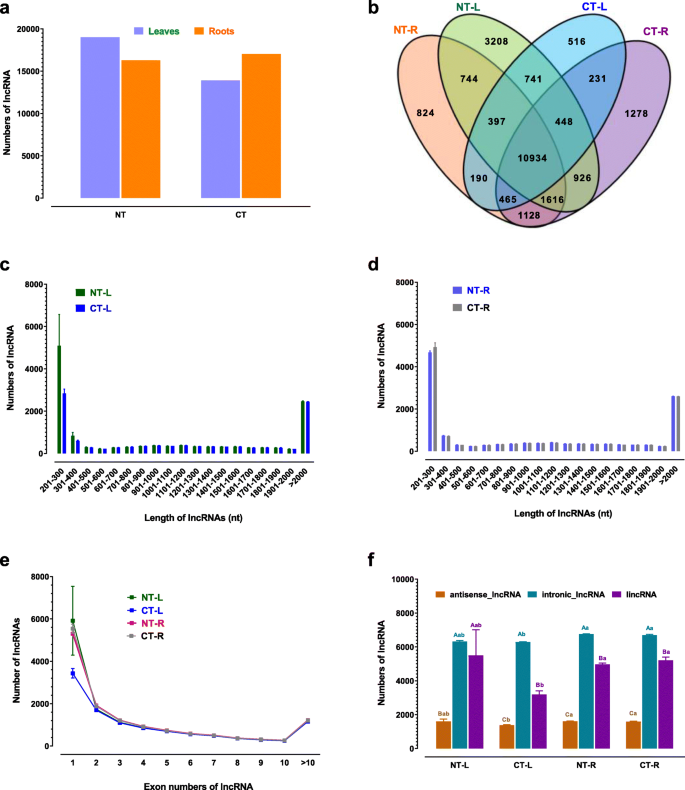
识别lncRNAs总数的四个处理样品,我们合并数据的三个生物学重复(相关系数是补充表S2和S3)所示为每个治疗不管重复性,到19014年,16298年,13922年和17026年独特lncRNAs确定了NT-leaves、NT-roots, CT-leaves CT-roots,分别(无花果。GydF4y2Ba1GydF4y2Ba一种)。叶中鉴定的LNCRNA的数量大于NT(19,014 / 16,298)场景中的根部,但冷处理后它们较少(13,922 Vs 17,026)(图。GydF4y2Ba1GydF4y2Ba一种)。CT叶中鉴定的LNCRNA的数量减少了5092,导致6494消失,与NT叶相比,1402外观。相比之下,CT根中鉴定的LNCRNA的数量增加了728,与NT根相比,2155次消失和2883个外观(图。GydF4y2Ba1GydF4y2Ba一种)。GydF4y2Ba
识别冷响应LNCRNAGydF4y2BaM. Truncatula.GydF4y2Ba幼苗。a: NT-L、CT-L、NT-R、CT-R中识别的常见/特异lncrna数量。b:叶(NT, CT)和根(NT, CT)中鉴定的lncrna总数。所示的值(GydF4y2Ba一种GydF4y2Ba) 和 (GydF4y2BaB.GydF4y2Ba)从汇集三个独立的RNAseq实验中获得。叶片中LNCRNA的长度分布(GydF4y2BaCGydF4y2Ba)和根(d)。E:在NT-L,CT-L,NT-R和CT-R中包含不同数量的外显子的LNCRNA数。F:NT-L,CT-L,NT-R和CT-R中鉴定的三种型LNCRNA的数量。大写字母表明了一个重要的不同GydF4y2BaP.GydF4y2Ba < 0.05 according toT.GydF4y2Ba- 在同一治疗样本中的三种型LNCRNA之间,小字母表示显着不同GydF4y2BaP.GydF4y2Ba < 0.05 according toT.GydF4y2Ba- 在NT-L,CT-L,NT-R和CT-R之间的最低,相同型LNCRNA。所示的值(GydF4y2BaCGydF4y2Ba), (GydF4y2BaD.GydF4y2Ba), (GydF4y2BaE.GydF4y2Ba) 和 (GydF4y2BaFGydF4y2Ba)是平均值±SE与三个独立的RNAseq实验。反义-lncRNA: lncRNA与相反链上的参考分子重叠;intronic-lncRNA:完全位于参考内含子内的lncRNA;incRNA:基因间lncRNA;NT: non-cold治疗;CT:冷治疗;L:叶子;接待员:根GydF4y2Ba
通过汇集来自4种处理和12个样本库的数据,我们识别出24,368个独特的lncrna(图)。GydF4y2Ba1GydF4y2Bab)。在这些LNCRNA中,我们同时鉴定了四种处理中的10,934(44.9%)独特的LNCRNA,4465(18.3%)和3230(13.3%)LNCRNA分别对叶片和根具有特异性(图。GydF4y2Ba1GydF4y2Bab).此外,在未冷处理和冷处理的叶片中特异性鉴定出3208(13.2%)和516 (2.1%)lncrna,在未冷处理和冷处理的根中分别鉴定出824(3.4%)和1278 (5.2%)lncrna(图2)。GydF4y2Ba1GydF4y2Bab)。GydF4y2Ba
在长度上,大部分的lncrna相对较短,短于2000 nt的lncrna在nt - leaf、nt -roots、ct - leaf和CT-roots中分别占81.1、77.4、80.5和80.8%(图2)。GydF4y2Ba1GydF4y2Bac和d)。具体来说,我们发现小于400 nt的lncrna是显性的。例如,nt -leaves、nt -roots、CT-leaves和CT-roots中小于400 nt的lncrna分别占总lncrna的42.6、31.6、40.8和41.8%(图2)。GydF4y2Ba1GydF4y2BaC和D)。GydF4y2Ba
我们还在LNCRNA中分析了外显子数量。如图1所示。GydF4y2Ba1GydF4y2Bae中,随着外显子数目的增加,lncrna的数量急剧减少,其中只有1个外显子的lncrna数量最多,nt - leaf、nt -root、ct - leaf和ct -root中分别约占42.4、39.8、31.5和41.0%(图3)。GydF4y2Ba1GydF4y2Bae)。GydF4y2Ba
在本研究中,我们根据其基因组来源,将lncRNA分为反义-lncRNA (lncRNA在相反链上与参考子重叠)、内含子-lncRNA (lncRNA完全位于一个参考内含子内)和lincRNA (lncRNA在基因间)三类(图)。GydF4y2Ba1GydF4y2BaF)。在三种类型的LNCRNA中,最多的叶片和根中的内肾上腺炎LNCRNA和反义-LNCRNA元(图。GydF4y2Ba1GydF4y2BaF)。GydF4y2Ba
染色体上LNCRNA分布的分析显示,鉴定的LNCRNA在八条染色体上不均匀分布GydF4y2BaM. Truncatula.GydF4y2Ba幼苗具有明显的位置偏好(附加文件GydF4y2Ba2GydF4y2Ba:图。S1;附加文件GydF4y2Ba3.GydF4y2Ba:图。S2)。GydF4y2Ba
冷响应LNCRNA的特征GydF4y2BaM. Truncatula.GydF4y2Ba幼苗GydF4y2Ba
我们根据以下五个方面的特征在于冷敏感LNCRNA:改变折叠(FC(日志)GydF4y2Ba2GydF4y2Ba)),外显子数,LNCRNA的长度,染色体的分布,以及叶子和根的LNCRNA的类型GydF4y2BaM. Truncatula.GydF4y2Ba.根据TPM(百万百万的转录物)和折叠倍数≥2,选择显示不同表达的LNCRNA。GydF4y2BaP.GydF4y2Ba-value< 0.05对于冷处理和非冷处理的叶子和根之间的3次生物重复(附加文件GydF4y2Ba8.GydF4y2Ba:数据集S1)。GydF4y2Ba
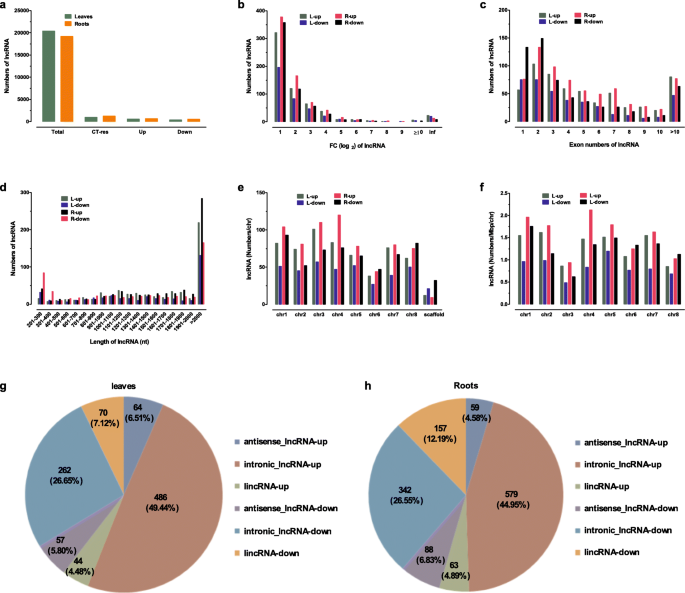
在鉴定的LNCRNA中,983和1288分别对叶片和根部的冷处理有响应(图。GydF4y2Ba2GydF4y2Baa).根中冷应答lncrna的数量大于叶中,冷处理后上调或下调的lncrna的数量也大于叶中(图2)。GydF4y2Ba2GydF4y2Baa).在叶和根中,上调的lncrna数量均大于下调的lncrna数量(图2)。GydF4y2Ba2GydF4y2Bab)。此外,只有少数LNCRNA被特异性发现通过冷处理叶片和根的冷治疗来上调(附加文件GydF4y2Ba8.GydF4y2Ba:数据集S1)。如图1所示。GydF4y2Ba2GydF4y2BaB,随着(FC(LOG)减少了具有上调或下调表达水平的LNCRNA的数量GydF4y2Ba2GydF4y2Ba))在叶子和根部。最高数量的上调或下调的LNCRNA是那些表达变化为FC的那些(日志GydF4y2Ba2GydF4y2Ba)<4.例如,它们在叶片上调和下调89.2%,分别在根中上调和下调93.6和95.2%(图。GydF4y2Ba2GydF4y2Bab)。GydF4y2Ba
冷敏感LNCRNA的特征GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2Ba一种GydF4y2Ba:叶和根中冷应答lncrna的数量GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2BaB.GydF4y2Ba:冷处理后不同FC(log2)的lncrna的数量GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。X轴数据显示(FC(log2))值从≥1到≥10不等。GydF4y2BaCGydF4y2Ba:紫花苜蓿叶片和根中含有不同外显子数的lncrna的数量GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。d:不同长度的lncrna的数量对冷处理有反应GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2BaE.GydF4y2Ba:冷处理后,叶和根染色体上分布的lncrna的数量GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2BaFGydF4y2Ba:8染色体上的LNCRNA的密度应对叶子和根的冷处理作出反应GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。CT-res:冷处理应答型lncrna;上调或下调:冷处理诱导或减少lncrna表达;L:叶子;接待员:根源。叶片中受冷处理调控的三种类型的lncrna(反义- lncrna、内含子- lncrna和lincRNA) (GydF4y2BaGGydF4y2Ba)和根源(GydF4y2BaHGydF4y2Ba) 的GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。反义-lncRNA: lncRNA与相反链上的参考分子重叠;intronic-lncRNA:完全位于参考内含子内的lncRNA;Lincrna:intercenic lncrNaGydF4y2Ba
除了在叶或根中含有两个外显子的lncrna外,显示或下调表达的lncrna数量也随着外显子数量的增加而减少(图)。GydF4y2Ba2GydF4y2Bac).不同于四种处理中鉴定出的只有一个外显子的lncrna数量最多的结果,冷应答的lncrna数量最多的是包含两个外显子的lncrna(图2)。GydF4y2Ba2GydF4y2Bac).虽然大多数(约40%)的lncrna在GydF4y2BaM. Truncatula.GydF4y2Ba幼苗短于400 nt(图。GydF4y2Ba2GydF4y2BaC和D),短于2000毫升的LNCRNA显示出对冷处理的相当响应,无论其长度如何(图。GydF4y2Ba2GydF4y2Bad)。GydF4y2Ba
在8条染色体的叶片中,响应冷处理,上调的lncrna数量多于下调的lncrna数量(图)。GydF4y2Ba2GydF4y2Bae和f)。相比之下,冷应答lncrna在根中表现出不同的模式。例如,根中chr 6和chr 8上上调的lncrna数量少于下调的lncrna,而剩余染色体上上调的lncrna数量多于下调的lncrna(图)。GydF4y2Ba2GydF4y2Bae和f)。进一步的分析表明,鉴定的LNCRNA的数量和密度增加的冷治疗诱导的增加在叶片和根中的8条染色体中不同(图。GydF4y2Ba2GydF4y2Bae和f)。在CHR3(17.0%)上发现最大数量,而最少的叶子中最少的叶子(图6.4%)(图。GydF4y2Ba2GydF4y2Bae)。通过冷处理上调的最多的LNCRNA是CHR4(20.2%),而最少数最少的ROORS(图)是最少的GydF4y2Ba2GydF4y2Bae).虽然叶片中lncrna上调和下调浓度最高的染色体与根中不同,但叶片和根中lncrna上调和下调浓度最低的染色体均在chr3上(图3)。GydF4y2Ba2GydF4y2Bae和f)。这些结果表明,每条染色体具有不同的模式,响应于LNCrans表达的冷治疗。GydF4y2Ba
最多的冷响应LNCRNA属于叶子和根中的内含性,但最少的叶片和根部中的叶片和反义-LNCRNA是叶子和根部(图。GydF4y2Ba2GydF4y2Bag和h)。此外,不同类型的lncRNAs冷处理的反应不同,这样的表达49.4和45.0%的intronic-lncRNAs叶子和根被冷处理向上调节,但antisense-lncRNAs只有4.9%和6.5,分别和linc-lncRNAs 4.9%和4.5(图。GydF4y2Ba2GydF4y2Bag和h)。类似于上调的LNCRNA,下调内肠道克朗的数量也是大多数,但最少的叶子和根部是反义-LNCRNA(图。GydF4y2Ba2GydF4y2Ba这些结果可能提示intronic- lncrna是对冷处理最敏感的lncrna。GydF4y2Ba
低温应答lncrna的功能特性GydF4y2BaM. Truncatula.GydF4y2Ba幼苗GydF4y2Ba
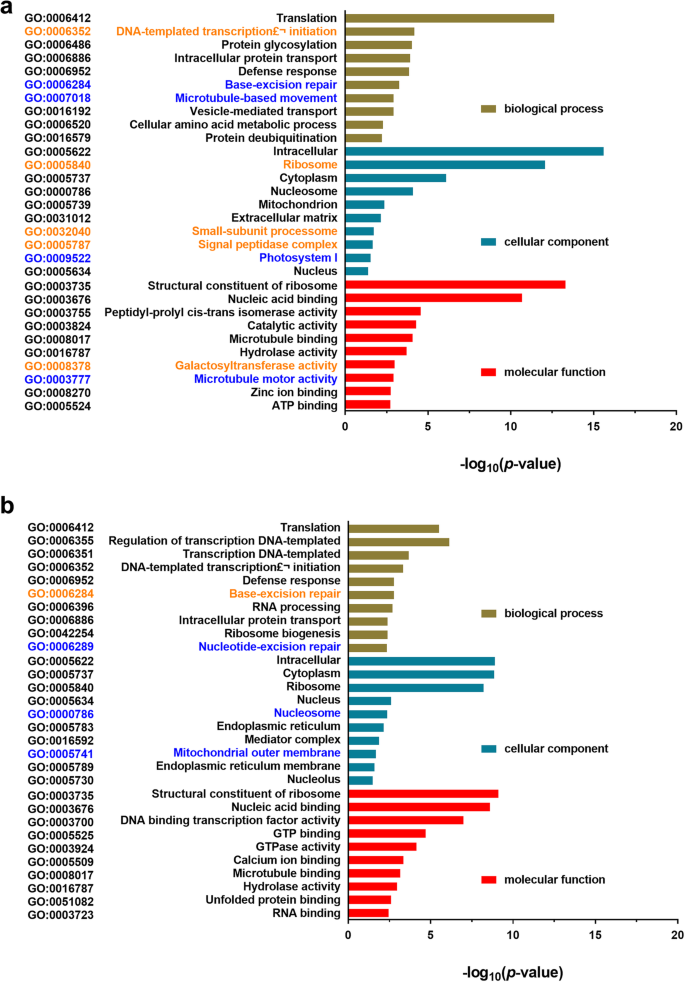
发现潜在的功能cold-responsive lncRNAs,我们分析了基因本体论(去)的基因中的和硝唑cold-responsive lncRNAs基于三个生物学重复的转录表达之间的寒冷和non-cold治疗根和叶在100 kb和基因组的位置。生物过程、细胞成分和分子功能的前10个术语根据显著富集(校正)进行了分析GydF4y2BaP.GydF4y2Ba < 0.05) in leaves and roots (Fig.3.GydF4y2Ba;附加文件GydF4y2Ba1GydF4y2Ba:表S5和S6)。GydF4y2Ba
基因本体(GO)分析。转移基因的基因术语,并基于叶片中的转录表达和基因组定位与冷响应性LNCRNA共局限(GydF4y2Ba一种GydF4y2Ba)和根源(GydF4y2BaB.GydF4y2Ba) 的GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。可靠性由-log10 (GydF4y2BaP.GydF4y2Ba< 0.05)。y轴上的黑色词表明,这些氧化石墨烯词的基因在低温处理下被上调或下调。y轴呈橙色的词表明这些GO词的基因在低温处理下上调。y轴上的蓝色词表示这些氧化石墨烯术语的基因在低温处理下下调。更多信息详见附加文件GydF4y2Ba1GydF4y2Ba:表S5和S6GydF4y2Ba
在前10个生物学过程中,叶片中与dna模板转录(起始)相关的基因(GO:0006352)和根中与碱基切除修复相关的基因(GO:0006284)均在低温处理下表达。冷处理降低了叶片中参与碱基切除修复(GO:0006284)和泡囊介导运输(GO:0007018)的基因和根中参与核苷酸切除修复(GO:0006289)的基因的表达量。大多数氧化石墨烯组的基因都在低温处理下上调或下调(图。GydF4y2Ba3.GydF4y2Ba).在叶片细胞组分前10项中,冷处理下叶片中核糖体、小亚基加工体和信号肽酶复合物(GO:0005840、GO:0032030、GO:0005787)三个组的所有基因均上调表达。而冷处理则导致根核小体(GO:0000786)和线粒体外膜(GO:0005741)中所有基因表达下调。其他GO细胞组分的基因在叶片或根部通过冷处理被上调或下调(图2)。GydF4y2Ba3.GydF4y2Ba).共表达并与叶子和根中冷响应LNCRNA共同局记的基因的分子函数非常复杂。诱导基因在半乳糖基转移酶活性组(GO:0008378)中的表达,并且通过冷处理降低了微管运动活性组(GO:0003777)中基因的表达(图。GydF4y2Ba3.GydF4y2Ba).这些MRNA编码的蛋白质在叶子和根部具有不同的功能,表明对冷治疗的器官特异性反应。这些结果突出了LNCRNA基因组定位的多样性和对其潜在靶基因作用的调控功能的复杂性。GydF4y2Ba
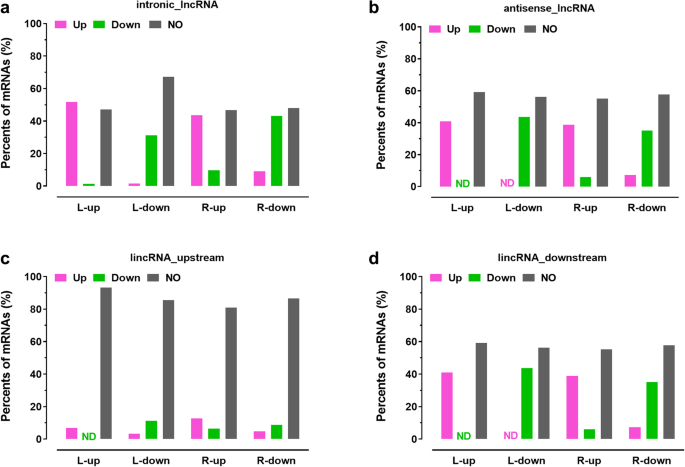
为了进一步表征LNCRNA与其潜在的靶蛋白编码基因之间的表达关系,分析了LNCRNA表达模式的相关性及其响应于冷处理的潜在靶基因的相关性(图。GydF4y2Ba4.GydF4y2Ba).如图1所示。GydF4y2Ba4.GydF4y2Ba,超过30%的潜在靶基因显示出具有内肾内-LNCRNA和反义-LNCRNA的类似表达模式(图。GydF4y2Ba4.GydF4y2Baa和b)。例如,在叶片中检测到的内含子- lncrnas冷诱导表达(L-up)的潜在靶基因约为51.7%,在根中检测到的内含子- lncrnas冷诱导表达(R-up)的潜在靶基因为43.6%。类似于诱使lncRNAs的表情,31.3%的潜在目标基因L-down(也就是说,表达式是减少冷治疗叶子)intronic-lncRNAs减少冷处理,虽然它是43.1%的潜在目标基因R-down(也就是说,表达式是减少冷治疗根)intronic-lncRNAs(无花果。GydF4y2Ba4.GydF4y2Baa).在反义lncrna的潜在靶基因中也发现了类似的情况(图2)。GydF4y2Ba4.GydF4y2Bab)。另外,与LincrNA相关的潜在靶基因的表达模式更复杂(图。GydF4y2Ba4.GydF4y2BaC和D)。上游潜在靶基因的表达模式与其相关的LincRNA略有相似之处(图。GydF4y2Ba4.GydF4y2BaC)。相反,下游潜在靶基因的表达与相邻LincrNA的表达呈正相关(图。GydF4y2Ba4.GydF4y2Bad)。例如,通过冷处理诱导约40.8%的下游潜在靶基因,诱导r-up Lincrnas的38.8%下游潜在靶基因诱导相同的冷处理。类似于LNCRNA的冷诱导的表达,通过冷处理降低了43.8%的下游LINCRNA的下游潜在靶基因(L-DOWN),而在根中下调LINCRNA的下游潜在靶基因的35.1%下游(R-下来)(图。GydF4y2Ba4.GydF4y2Bad)。GydF4y2Ba
lncrna与潜在靶基因的相关性分析GydF4y2BaM. Truncatula.GydF4y2Ba.内含子- lncrna表达模型(GydF4y2Ba一种GydF4y2Ba), antisense-lncRNAs (GydF4y2BaB.GydF4y2Ba)叶子和根部的潜在靶基因GydF4y2BaM. Truncatula.GydF4y2Ba幼苗对冷处理有反应。lincrna及其潜在上游靶基因的表达模型(GydF4y2BaCGydF4y2Ba),下游基因(GydF4y2BaD.GydF4y2Ba)的叶子和根GydF4y2BaM. Truncatula.GydF4y2Ba幼苗对冷处理有反应。RNA-seq (TPM)分析表达。反义-lncRNA: lncRNA与相反链上的参考分子重叠;intronic-lncRNA:完全位于参考内含子内的lncRNA;lincRNA:基因间lncRNA;L:叶子;R:根;Up:冷处理诱导表达;Down:冷处理降低表达;ND:没有检测到GydF4y2Ba
分析lncRNA -GydF4y2BaMTCBF.GydF4y2BaS网络GydF4y2Ba
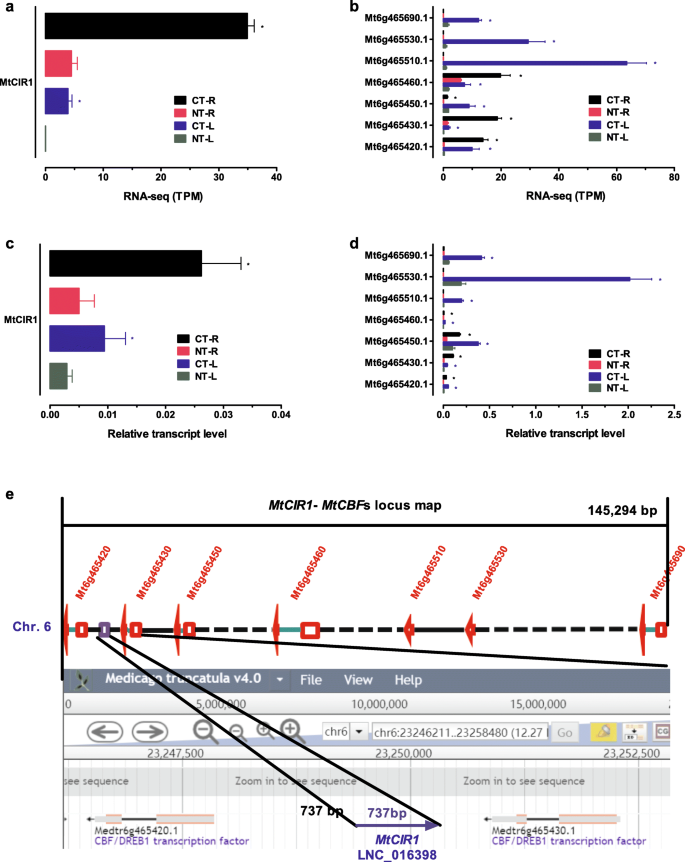
C-重复/ DRE结合蛋白(GydF4y2BaCBFGydF4y2BaS)已被识别为冷响应的枢转转录调节因素(GydF4y2Ba天哪GydF4y2Ba)基因在许多植物品种的低温处理[GydF4y2Ba22GydF4y2Ba].串联阵列GydF4y2BaCBF / DREB1GydF4y2Ba据报道,基因位于染色体6中的主要冰镇耐受性QTL区域中GydF4y2BaM. Truncatula.GydF4y2Ba[GydF4y2Ba42GydF4y2Ba].确定冷应答lncrna在调控中的作用GydF4y2BaMTCBF.GydF4y2Ba,我们检测了lncrna的表达模式GydF4y2BaMTCBF.GydF4y2Ba通过考虑它们在染色体上的位置。一个逆转录lncRNA (LNC_016398-GydF4y2Bamtcir1.GydF4y2Ba那GydF4y2BamGydF4y2Ba.GydF4y2BaT.GydF4y2Baruncatula.GydF4y2BaCGydF4y2BaBF.GydF4y2BaS.GydF4y2Ba一世GydF4y2BantergenicGydF4y2BaR.GydF4y2BaNA)(无花果。GydF4y2Ba5.GydF4y2BaA和c)和7GydF4y2BaCBFGydF4y2Ba基因(GydF4y2BaMT6G465420GydF4y2Ba那GydF4y2BaMT6G465430GydF4y2Ba那GydF4y2BaMT6G465450GydF4y2Ba那GydF4y2BaMT6G465460GydF4y2Ba那GydF4y2BaMt6g465510GydF4y2Ba那GydF4y2BaMt6g465530GydF4y2Ba和GydF4y2BaMT6G465690.GydF4y2Ba) (无花果。GydF4y2Ba5.GydF4y2Bab和d)由RNA-seq冷处理诱导(图。GydF4y2Ba5.GydF4y2Baa和b),并通过实时定量Q-PCR进行验证(图4)。GydF4y2Ba5.GydF4y2Bac和d).基因组定位分析GydF4y2Bamtcir1.GydF4y2Ba七GydF4y2BaCBFGydF4y2Ba基因表明它们在染色体上邻近分布(图。GydF4y2Ba5.GydF4y2Bae)。GydF4y2Bamtcir1.GydF4y2Ba(733 bp)位于基因间区GydF4y2BaMT6G465420GydF4y2Ba和GydF4y2BaMT6G465430GydF4y2Ba,它没有显示出这些基因的任何重叠(图。GydF4y2Ba5.GydF4y2Bae)。GydF4y2Ba
lncrna之间的关系GydF4y2Bamtcir1.GydF4y2Ba以及它们的潜在目标GydF4y2BaMTCBF.GydF4y2Ba基因GydF4y2BaM. Truncatula.GydF4y2Ba植物。转录回应GydF4y2Bamtcir1.GydF4y2Ba(GydF4y2Ba一种GydF4y2Ba),GydF4y2BaMTCBF.GydF4y2Ba基因(GydF4y2BaB.GydF4y2Ba) 在GydF4y2BaM. Truncatula.GydF4y2Ba通过RNA-SEQ(TPM)在4℃下冷处理植物5小时。转录回应GydF4y2Bamtcir1.GydF4y2Ba(GydF4y2BaCGydF4y2Ba),GydF4y2BaMTCBF.GydF4y2Ba基因(GydF4y2BaD.GydF4y2Ba) 在GydF4y2BaM. Truncatula.GydF4y2Ba通过实时定量Q-PCR验证,4°C低温处理5 h。NT: non-cold治疗;CT:冷治疗;L:叶子;接待员:根源。值为平均值±SE (GydF4y2BaNGydF4y2Ba = 3) and the asterisks indicate significant differences atP.GydF4y2Ba冷处理样品(CT)与非冷处理对照(NT)之间的差异< 0.05GydF4y2BaT.GydF4y2Ba-测试。E:基因组图GydF4y2BaMTCBF.GydF4y2Ba基因簇和GydF4y2Bamtcir1.GydF4y2Ba.GydF4y2Bamtcir1.GydF4y2Ba和GydF4y2BaMTCBF.GydF4y2Ba基因簇位于145.3 kB的染色体6GydF4y2BaM. Truncatula.GydF4y2Ba第A17 Jemalong。红色矩形表示外显子。红色箭头表示基因5′到3′的外显子和方向。绿线表示内含子。蓝色矩形表示lncRNA。黑线表示基因间区。黑色数字表示基因间区长度(bp)GydF4y2Ba
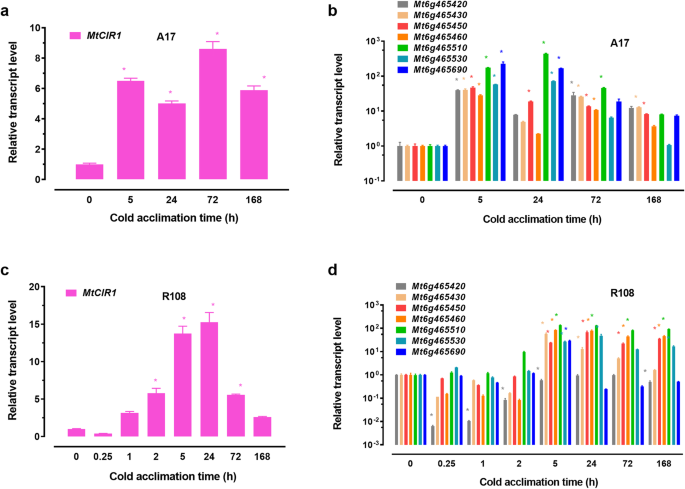
lncRNAGydF4y2Bamtcir1.GydF4y2Ba不仅是七个的邻近GydF4y2BaMTCBF.GydF4y2Ba染色体6(图。GydF4y2Ba5.GydF4y2BaE),其表达水平也与冷诱导相当GydF4y2BaMTCBF.GydF4y2Ba(图。GydF4y2Ba5.GydF4y2Ba模拟)GydF4y2Ba.GydF4y2Ba要确认结果,我们测试了成绩单响应GydF4y2Bamtcir1.GydF4y2Ba以及它们的潜在目标GydF4y2BaMTCBF.GydF4y2Ba通过定量实时PCR在叶片中进行冷处理的基因GydF4y2BaM. Truncatula.GydF4y2Ba(无花果。GydF4y2Ba6.GydF4y2Ba).表达GydF4y2Bamtcir1.GydF4y2Ba冷处理上调(图。GydF4y2Ba6.GydF4y2Baa).例如,冷处理5小时后,转录产物GydF4y2Bamtcir1.GydF4y2Ba增加了7倍(图。GydF4y2Ba6.GydF4y2Ba一种)。七的成绩单GydF4y2BaMTCBF.GydF4y2Ba它位于CHR6上并由邻居GydF4y2Bamtcir1.GydF4y2Ba在叶片中瞬时增加(图。GydF4y2Ba6.GydF4y2Bab).一个重要的问题是冷处理诱导的表达是否GydF4y2Bamtcir1.GydF4y2Ba在表达GydF4y2BaMTCBF.GydF4y2Ba为了回答这个问题,我们监测了GydF4y2Bamtcir1.GydF4y2Ba和GydF4y2BaMTCBF.GydF4y2Ba在低温处理初期(0 ~ 2 h),叶绿素含量显著增加。如图1所示。GydF4y2Ba6.GydF4y2BaA,转录水平GydF4y2Bamtcir1.GydF4y2Ba在冷治疗的2小时内明显诱导。冷诱导的增加GydF4y2Bamtcir1.GydF4y2Ba通过成绩单的积累进行5小时GydF4y2BaMTCBF.GydF4y2Ba(图。GydF4y2Ba6.GydF4y2Bab).与其他四种不同GydF4y2BaMTCBF.GydF4y2BaS,在5小时达到达到峰值,然后在冷治疗过程中落下,表达三个GydF4y2BaMTCBF.GydF4y2Ba年代,GydF4y2BaMt6g465510GydF4y2Ba那GydF4y2BaMt6g465530GydF4y2Ba和GydF4y2BaMT6G465690.GydF4y2Ba在5小时内也达到达到峰值,冷处理期间保持高水平,高达24小时(图。GydF4y2Ba6.GydF4y2Bab)。结果表达的结果GydF4y2Bamtcir1.GydF4y2Ba接着是GydF4y2BaMTCBF.GydF4y2Ba冷治疗过程中的转录物可能表明LNCRNA之间的串扰存在GydF4y2Bamtcir1.GydF4y2Ba和GydF4y2BaMTCBF.GydF4y2Ba基因。GydF4y2Ba
lncrna的反应GydF4y2Bamtcir1.GydF4y2Ba以及它们的潜在目标GydF4y2BaMTCBF.GydF4y2Ba冷治疗的基因。GydF4y2Ba一种GydF4y2Ba:的转录反应GydF4y2Bamtcir1.GydF4y2Ba在叶子中GydF4y2BaM. Truncatula.GydF4y2Ba幼苗经过冷治疗。GydF4y2BaB.GydF4y2Ba:的转录反应GydF4y2BaMTCBF.GydF4y2Ba叶片中的基因GydF4y2BaM. Truncatula.GydF4y2Ba幼苗经过冷治疗。从4℃处理4℃的3周龄幼苗中提取叶RNA以进行不同的时间段,然后使用实时定量RT-PCR检测相对表达。值是指±se(n = 3),星号表示显着差异GydF4y2BaP.GydF4y2Ba < 0.05 according toT.GydF4y2Ba-相对于室温控制的测试(0 h)GydF4y2Ba
讨论GydF4y2Ba
对植物lncrna的研究表明,lncrna在广泛的生物过程中发挥重要作用,特别是在调节植物对生物和非生物胁迫的反应,如玉米的干旱胁迫[GydF4y2Ba9.GydF4y2Ba),GydF4y2Ba杨树trichocarpaGydF4y2Ba[GydF4y2Ba11GydF4y2Ba,盐和干旱胁迫GydF4y2BaMedicago Truncatula.GydF4y2Ba[GydF4y2Ba12GydF4y2Ba],GydF4y2BaP. Infestans.GydF4y2Ba- 西红柿中的言论[GydF4y2Ba13GydF4y2Ba].然而,对于lncrna是否参与豆科模式植物冷驯化依赖的抗冻性研究尚不清楚GydF4y2BaMedicago Truncatula.GydF4y2Ba.在本研究中,我们确定了24,368个独特的LNCRNA,类似于所获得的数字GydF4y2BaM. Truncatula.GydF4y2Ba由盐和干旱胁迫引起LNCRNA [GydF4y2Ba12GydF4y2Ba].在24368个lncrna中,叶片和根部有983个和1288个lncrna对冷处理有响应。在我们的研究中发现的冷应答lncrna比在木薯中发现的要多,这可能是由于不同的测序方法和植物种类,例如不同的基因组大小和对低温胁迫的敏感性[GydF4y2Ba44GydF4y2Ba].GydF4y2Ba
我们的研究结果表明,叶片中鉴定出的唯一的lncrna和mrna的总数大于根,但根中冷应答lncrna和mrna的比例高于叶(fig . 2)。GydF4y2Ba1GydF4y2Ba和GydF4y2Ba2GydF4y2Ba;附加文件GydF4y2Ba4.GydF4y2Ba:图。S3;附加文件GydF4y2Ba5.GydF4y2Ba:图。S4)。这些结果可能表明,不同的器官对冷治疗的特定反应,使得RNCRNA和RNA在根中的MRNA可能对冷治疗更敏感。相对于叶子中的那些,根系中的冷响应性LNCRNA和MRNA的比例较高可能意味着响应于冷治疗的根部的重要性。然而,迄今为止,很少有研究侧重于根对冷应力的生理和分子反应[GydF4y2Ba45GydF4y2Ba那GydF4y2Ba46GydF4y2Ba].有人提出,在四个年度豆类中的冷置适应和冰霜抗性的能力与较高的根部以较高的芽比和较高浓度的溶质在根中[GydF4y2Ba47GydF4y2Ba].因此,根介导的生理过程可能在植物抗冻性的调控中发挥重要作用。GydF4y2Ba
我们的研究结果表明,大多数GO项参与了不同生物过程的调控,这与植物冷胁迫反应中存在复杂机制的报告相一致[GydF4y2Ba21GydF4y2Ba那GydF4y2Ba48GydF4y2Ba].氧化石墨烯参与防御反应的术语可能直接参与应激条件下细胞的保护。据报道,与转录调控相关的氧化石墨烯项0006355在响应冷冻胁迫中发挥作用GydF4y2BaM. Sativa.GydF4y2Ba[GydF4y2Ba49GydF4y2Ba].据报道,通过用作反应性氧(ROS)的清除剂(ROS)的清除剂,参与Cu / Zn超氧化物歧化酶的基因编码,其被植物中的mir398切割,参与冷应激反应的调节(ROS)[GydF4y2Ba50GydF4y2Ba那GydF4y2Ba51GydF4y2Ba].这些结果可能意味着在对冷应力的反应的调节中可能存在LNCRNA和MicroRNA之间的相关性。有趣的发现是基因在叶片切除修复(GO:0006284)中包括叶片,但通过冷处理在根部上调(图。GydF4y2Ba3.GydF4y2Ba).DNA破坏因子对基因组是一个巨大的威胁。为了保护DNA不受损伤,人们开发了许多DNA修复策略。基底-切除-修复途径在生物体中是一种高度保守的机制,并已被建议用于去除和修复诱变的氧化DNA损伤[GydF4y2Ba52GydF4y2Ba].虽然关于碱基切除修复系统在胁迫和植物激素信号转导中的作用已有广泛的研究[GydF4y2Ba53GydF4y2Ba那GydF4y2Ba54GydF4y2Ba,人们对它们对植物冷胁迫反应的影响知之甚少。本研究发现碱基切除修复的GO项表明碱基切除修复可能在冷处理DNA修复中发挥重要作用。GydF4y2Ba
作为关键的转录激活因子,脱水和冷反应基因家族(GydF4y2BaCBF / DREB1GydF4y2Ba)激活下游冷调节(GydF4y2Ba天哪GydF4y2Ba)基因,这反过来有助于增强对冰冻胁迫的耐受性[GydF4y2Ba22GydF4y2Ba那GydF4y2Ba24GydF4y2Ba那GydF4y2Ba55GydF4y2Ba].15个品种中,一个主要的耐冻QTL Mt-FTQTL6占表型变异的40%GydF4y2BaM. Truncatula.GydF4y2Ba入库已被映射到一个区域GydF4y2BaM. Truncatula.GydF4y2Ba6号染色体(GydF4y2Ba56GydF4y2Ba].串联阵列GydF4y2BaCBF / DREB1GydF4y2Ba基因位于染色体6中的主要冷冻耐受性QTL(MT-FTQTL6)区域中GydF4y2BaM. Truncatula.GydF4y2Ba[GydF4y2Ba42GydF4y2Ba].但是,作为低温刺激的信号传导级联的早期事件,如何表达GydF4y2BaCBFGydF4y2BaS由冷治疗诱导仍然很难尚不清楚。许多报告证明了LNCRNA通过与DNA和RNA分子的相互作用和转录因子来参与各种生物过程的调节,导致靶基因的改变[GydF4y2Ba5.GydF4y2Ba].此外,lncrna的表达模式通常与两者的mRNA的表达模式相关GydF4y2BaCIS.GydF4y2Ba和GydF4y2Ba反式GydF4y2Ba举止,表明某些LNCRNA可以在表达网络中共同调节[GydF4y2Ba57GydF4y2Ba].在本研究中,我们发现lncrna和GydF4y2BaMTCBF.GydF4y2Ba通过rna测序和基因表达分析,发现GO: 0005634在低温反应中的表达。我们证明了6的表达式GydF4y2BaMTCBF.GydF4y2Bas与一个lncrna的表达有关GydF4y2Bamtcir1.GydF4y2Ba.具体来说,我们发现GydF4y2Bamtcir1.GydF4y2Ba是位于近距离接近的基因间非分量RNAGydF4y2BaMTCBF.GydF4y2Ba基因的表达GydF4y2Bamtcir1.GydF4y2Ba和GydF4y2BaMTCBF.GydF4y2Ba通过冷处理诱导基因。该观察结果符合证明约一半的代苯基非编码RNA接近蛋白质编码基因[GydF4y2Ba57GydF4y2Ba].因此,这些数据可以提供关于在冷处理期间邻近基因对邻近基因的转录调节的重要线索。GydF4y2Ba
lncRNA转录水平的观察GydF4y2Bamtcir1.GydF4y2Ba低温2 h内增加,其次为累积GydF4y2BaMTCBF.GydF4y2BaS在5 h时可能提示在GydF4y2BaMTCBF.GydF4y2Ba年代和GydF4y2Bamtcir1.GydF4y2Ba.然而,这些结果不同于以往的研究,积累GydF4y2BaAtCBF1-3GydF4y2Ba低温胁迫下,15 min内检测到转录本GydF4y2Ba天哪GydF4y2Ba基因转录物在2小时[GydF4y2Ba58GydF4y2Ba].尽管这六种动物在序列上高度相似,在进化上也有着密切的关系GydF4y2BaMTCBF.GydF4y2Ba在本研究中确定的GydF4y2BaAtCBF1-3GydF4y2Ba,冷反应的差异可能意味着GydF4y2BaCBFGydF4y2BaS转录产物对低温处理是植物特有的。例如,拟南芥对冷胁迫比GydF4y2BaM. Truncatula.GydF4y2Ba植物(附加文件GydF4y2Ba6.GydF4y2Ba:图S5;附加文件GydF4y2Ba7.GydF4y2Ba:图S6)。最近的研究发现了一种lncRNAGydF4y2BaSVALKAGydF4y2Ba在拟南芥基因组的一个冷敏感区域。突变GydF4y2BaSVALKAGydF4y2Ba影响CBF1表达和抗冻性[GydF4y2Ba31GydF4y2Ba].是否lncRNAGydF4y2Bamtcir1.GydF4y2Ba在GydF4y2BaM. Truncatula.GydF4y2Ba在调节植物抗冻性方面与GydF4y2BaSVALKAGydF4y2Ba在拟南芥中,有必要进一步利用遗传学和生理学方法进行研究。GydF4y2Ba
许多研究发现了这一点GydF4y2BaICE1GydF4y2Ba那GydF4y2BaICE2GydF4y2Ba,三者密切相关GydF4y2BaCAMTAGydF4y2Ba年代,GydF4y2BaRDM4.GydF4y2Ba是积极的监管者GydF4y2BaCBFGydF4y2BaS,磷酸化的14-3-3蛋白溶解稳定CBF蛋白[GydF4y2Ba26GydF4y2Ba那GydF4y2Ba27GydF4y2Ba那GydF4y2Ba28GydF4y2Ba那GydF4y2Ba29GydF4y2Ba那GydF4y2Ba30.GydF4y2Ba那GydF4y2Ba59GydF4y2Ba].然而,是否GydF4y2Bamtcir1.GydF4y2Ba与这些蛋白的调控有关吗GydF4y2BaMTCBF.GydF4y2BaS和抗冻性有待阐明。舒等[GydF4y2Ba49GydF4y2Ba]确定了许多冻结和GydF4y2BaMedicagoGydF4y2Ba特异性miRNA参与调节凝固耐受性GydF4y2BaM. Sativa.GydF4y2Ba.lncrna转录后的一个重要机制是作为介质发挥作用,包括作为小rna的前体,与mirna共同作用调节mRNA的翻转[GydF4y2Ba60.GydF4y2Ba].是否对寒冷有反应GydF4y2BaMTCBF.GydF4y2Bas和lncran.GydF4y2Bamtcir1.GydF4y2Ba也与这些microRNA有关,有待于在未来的研究中发现。GydF4y2Ba
测序和QPCR的表达水平在我们的研究中不一致,特别是在LNCRNA的表达水平下(图。GydF4y2Ba5.GydF4y2Ba模拟)。主要有三个因素可以解释这些结果。首先,由于测序和qPCR是两种不同的检测方法,预计两种方法的结果会有一定程度的不一致,特别是对于低表达的rna。其次,利用qPCR验证测序结果,验证的是差异趋势,即上调或下调,而不是差异倍数。最后,测序过程中的复制可能部分干扰RNA转录本的定量结果[GydF4y2Ba61.GydF4y2Ba那GydF4y2Ba62.GydF4y2Ba].GydF4y2Ba
结论GydF4y2Ba
在本研究中,我们在豆科模式植物的叶和根中鉴定了大量的冷应答lncrnaGydF4y2BaM. Truncatula.GydF4y2Ba幼苗。我们进一步证明了冷响应性LNCRNA是组织特异性的,即叶片中鉴定的LNCRNA的数量大于非冷应激情景的根部,但在冷处理后它们较少。我们发现,三种类型的内肾上腺-LNCRNA在它们对冷应激的反应中不同,内含内克隆是对冷应激最敏感的。另一个有趣的发现是冷敏感的LNCRNA在基因组中的八条染色体上不均匀地分布GydF4y2BaM. Truncatula.GydF4y2Ba对地点有明显的偏好。此外,我们还解剖了lncRNA-的调控网络GydF4y2BaMTCBF.GydF4y2Ba这些基因可能通过整合lncRNA在植物对冷胁迫的响应和适应中发挥关键作用GydF4y2Bamtcir1.GydF4y2Ba他们的潜在目标GydF4y2BaMTCBF.GydF4y2Ba基因。GydF4y2Ba
方法GydF4y2Ba
植物材料和生长条件GydF4y2Ba
种子GydF4y2BaM. truncatula,GydF4y2Ba本研究中使用的Jemalong A17由美国农业部植物科学研究中心卡罗尔·万斯博士提供。Jemalong A17是豆科植物的模式植物,其基因组已经测序[GydF4y2Ba35GydF4y2Ba那GydF4y2Ba63.GydF4y2Ba].种子GydF4y2BaMedicago Truncatula.GydF4y2Ba用浓硫酸处理4分钟,然后用水彻底冲洗。种子在4°C冷却2 d后,播种在0.8%琼脂上,在25°C发芽,直到自由基长到2 cm左右。将秧苗种植在塑料桶中(一桶三棵秧苗),塑料桶中填满充氧营养液。全效营养液成分为:2.5 mM KNOGydF4y2Ba3.GydF4y2Ba,0.5毫米khGydF4y2Ba2GydF4y2Ba阿宝GydF4y2Ba4.GydF4y2Ba, 0.25 mM CaClGydF4y2Ba2GydF4y2Ba,1 mm mgsoGydF4y2Ba4.GydF4y2Ba, 100 μM Fe-Na-EDTA, 30 μM HGydF4y2Ba3.GydF4y2Ba博GydF4y2Ba3.GydF4y2Ba, 5 μM MnSOGydF4y2Ba4.GydF4y2Ba,1μmznsoGydF4y2Ba4.GydF4y2Ba,1μmcusoGydF4y2Ba4.GydF4y2Ba和0.7μmnaGydF4y2Ba2GydF4y2BaMoo.GydF4y2Ba4.GydF4y2BapH值为6.0。植物在26°C日/ 22°C夜晚的绿色房子中生长,14-H光周期,120μmolmGydF4y2Ba−2GydF4y2Ba年代GydF4y2Ba−1GydF4y2Ba状况。GydF4y2Ba
冷处理和样品收集GydF4y2Ba
在4°C,60μmolm的4°C,九个三周历史的Jemalong A17幼苗在三个塑料桶中生长。GydF4y2Ba−2GydF4y2Ba年代GydF4y2Ba−1GydF4y2Ba5小时(CT)。同时,九个三周大的jemalong a17幼苗在另外三个塑料桶中长到26°C,60μmolmGydF4y2Ba−2GydF4y2Ba年代GydF4y2Ba−1GydF4y2Ba5小时被用作对照(NT)。为一个生物学复制,收集九叶样品的九叶(随机化方法)和来自一个塑料桶中生长的三种植物的根样本。将三种生物学复制维持在相同条件下在单个生长室中重复三个单独的时间来进行样品收集。最后,收集12种用于四种非冷处理叶片和根(NT-L,NT-R),冷处理的叶片和根(CT-1,CT-R)的四种处理,用于RNA测序。同时,收集12个亚样品用于QRT-PCR试验。在收获后,将样品在液氮中冷冻,并在-80℃下储存以进行RNA提取。GydF4y2Ba
RNA提取和cDNA文库建设GydF4y2Ba
根据制造商的协议,使用Trizol(Invitrogen)从不同样品中提取总RNA。DNA消化用RNase的DNase I(Promega)。在1%琼脂糖凝胶上评价RNA降解和污染。Nanophotometer®分光光度计(Upplen,Ca,USA)检查RNA的纯度。RNA浓度通过Qubit®2.0矩阵(Life Technologies,CA,USA)中的Qubit®RNA测定试剂盒测定。RNA纳米6000测定试剂盒(BioAnalyzer 2100,Agilent Technologies,CA,USA)检查RNA完整性。GydF4y2Ba
对于每个样品,使用3μgRNA的量用于制备RNA样品。首先通过Epicenter Ribo-Zero™RRNA去除试剂盒(ePicenter,USA)除去核糖体RNA,并通过乙醇沉淀清洁RRNA游离残余物。此后,在制造商描述的协议之后,使用Nebnext®Ultra™定向RNA文库预备套件的Nebnext®Ultra™定向RNA文库制备试剂盒来产生测序文库,用于Illumina®(Neb,USA)。产品纯化(Ampure XP系统),用安捷伦生物分析仪2100系统测定文库质量。GydF4y2Ba
测序和数据分析GydF4y2Ba
在Illumina Hiseq 4000平台上对文库进行测序,生成150 bp的配对序列。12个样本的原始读数用于质量过滤。通过从原始数据中删除包含适配器、ploy-N和低质量读的读,可以获得干净读。同时测定干净数据的Q20、Q30和GC含量[GydF4y2Ba43GydF4y2Ba].低质量的阅读(Phred评分< 20;读取长度< 50个碱基)和带有适配器污染的读取被删除,以生成一组高质量的读取,随后称为干净数据。所有的下游分析都是基于高质量的干净数据。GydF4y2Ba
读取映射和转录组组装GydF4y2Ba
清洁读取映射到GydF4y2BaM. Truncatula.GydF4y2Ba基因组。指数的GydF4y2BaMedicagoGydF4y2Ba基因组序列V4.0(GydF4y2Bahttp://www.medicagohapmap.org/tools/blastformGydF4y2Ba)是使用bowtie2 v2.2.8构建的,对端干净读取与GydF4y2BaM. Truncatula.GydF4y2Ba基因组使用HISAT2 v2.0.4 [GydF4y2Ba64.GydF4y2Ba].为了构建转录组,使用Cufflinks重新组装映射的reads [GydF4y2Ba65.GydF4y2Ba].所有转录本的长度要求为> 200 bp。GydF4y2Ba
每个样本的映射读取由StringTie (v1.3.1)组装[GydF4y2Ba66.GydF4y2Ba]以基于参考的方法。Stringtie使用新颖的网络流量算法以及可选的DE Novo装配步骤来组装和定量表示每个基因座的多个剪接变体的全长转录物。GydF4y2Ba
编码潜在分析和识别LNCRNAGydF4y2Ba
使用以下工具进行编码电位的转录物的预测,包括CNCI(编码 - 非编码指数)(V2))[GydF4y2Ba67.GydF4y2Ba] CPC(编码潜在计算器)(0.9-R2)[GydF4y2Ba68.GydF4y2Ba], Pfam Scan (v1.3) [GydF4y2Ba69.GydF4y2Ba[Phylocsf(系统发育密码子替代频率)(V20121028)[GydF4y2Ba70GydF4y2Ba].其余未编码的未知转录本作为候选lncrna集合。GydF4y2Ba
靶基因预测GydF4y2Ba
我们首先鉴定了位于lncRNA上游和下游100 k的编码基因,然后分析了它们的功能。采用加权基因共表达网络分析(WGCNA)对来自不同样本的基因进行聚类[GydF4y2Ba71.GydF4y2Ba]为了搜索常见表达模块,通过功能丰富分析分析它们的功能。GydF4y2Ba
基因表达量的定量及差异表达分析GydF4y2Ba
Kallisto-Sleuth管道用于计算每个样本中LNCRNA和编码基因的TPM [GydF4y2Ba72.GydF4y2Ba].TPM是根据转录本的长度和映射到该转录本的读取计数计算的每千个外显子的每千个碱基的转录本。log2倍变化≥2(诱导)和/或≤- 2(减少)的差异表达基因(DEGs)GydF4y2BaP.GydF4y2Ba在每对比较中,任何一个样本的值小于0.05被认为是显著差异表达。GydF4y2Ba
实时定量PCR (qRT-PCR)GydF4y2Ba
使用Trizol (Invitrogen)按照制造商提供的协议提取总rna。用RNase-free DNase I (Promega)酶切DNA。将0.5 μg左右的RNA用PrimeScript®RT reagent Kit (TaKaRa)反转录成cDNA第一链。采用ABI Stepone Plus仪器进行实时定量PCR (qRT-PCR)。基因特异性引物和内控引物见Supplementary Table S7。我们从3个生物重复中进行了3个独立的qRT-PCR实验,每个cDNA在56℃退火条件下进行3次测量,共40个扩增周期。用比较CGydF4y2BaT.GydF4y2Ba方法。GydF4y2Ba
基因本体(GO)富集分析GydF4y2Ba
通过GOseq R包对差异表达基因或lncRNA靶基因进行基因本体(GO)富集分析,并校正基因长度偏差[GydF4y2Ba73.GydF4y2Ba].经修正的GO术语GydF4y2BaP.GydF4y2Ba值小于0.05为差异表达基因显著富集。我们利用Cytoscape软件基于共表达和基因组共定位构建了lncrna和蛋白编码rna之间的相互作用网络[GydF4y2Ba74.GydF4y2Ba].GydF4y2Ba
统计分析GydF4y2Ba
本研究中的所有实验只重复至少三次。结果给出了±SE。使用SPSS17.0软件(Chicago,IL,USA)进行统计分析。的GydF4y2BaT.GydF4y2Ba-检验用于确定治疗的效果是否在统计学上有差异GydF4y2BaP.GydF4y2Ba<0.05级。GydF4y2Ba
可用性数据和材料GydF4y2Ba
本研究期间生成或分析的所有数据都包含在本发布的文章及其补充信息文件中。GydF4y2Ba
缩写GydF4y2Ba
- Antisense-lncrna:GydF4y2Ba
-
LNCRNA在相反的股线上重叠GydF4y2Ba
- CAMTAGydF4y2Ba史:GydF4y2Ba
-
钙调素结合转录激活因子GydF4y2Ba
- 中科院:GydF4y2Ba
-
Cold-Acclimation-SpecificGydF4y2Ba
- CBF / DREB1GydF4y2Ba:GydF4y2Ba
-
C-重复结合因子/脱水响应元件结合因子1GydF4y2Ba
- CNCI:GydF4y2Ba
-
Coding-Non-Coding-IndexGydF4y2Ba
- cor:GydF4y2Ba
-
CRT / DRE-containing Cold-regulatedGydF4y2Ba
- 中国共产党:GydF4y2Ba
-
编码潜在计算器GydF4y2Ba
- CT:GydF4y2Ba
-
冷治疗GydF4y2Ba
- DEG:GydF4y2Ba
-
差异表达基因GydF4y2Ba
- FC(日志GydF4y2Ba2GydF4y2Ba):GydF4y2Ba
-
Log2-transformed褶皱变化GydF4y2Ba
- 走:GydF4y2Ba
-
基因本体论GydF4y2Ba
- IncRNA:GydF4y2Ba
-
代际lncra.GydF4y2Ba
- Intronic-lncraN:GydF4y2Ba
-
lncRNA完全属于一个参考内含子GydF4y2Ba
- lncRNAs:GydF4y2Ba
-
长不用的RNA.GydF4y2Ba
- mtcir1.GydF4y2Ba:GydF4y2Ba
-
m . truncatula CBFGydF4y2Ba代际RNA 1GydF4y2Ba
- NT:GydF4y2Ba
-
非冷治疗GydF4y2Ba
- ORFS:GydF4y2Ba
-
开放阅读框GydF4y2Ba
- PhyloCSF:GydF4y2Ba
-
系统发育密码子替换频率GydF4y2Ba
- QC:GydF4y2Ba
-
质量控制GydF4y2Ba
- RDM4.GydF4y2Ba:GydF4y2Ba
-
RNA定向DNA甲基化4GydF4y2Ba
- RNA-seq:GydF4y2Ba
-
RNA序列GydF4y2Ba
- TPM:GydF4y2Ba
-
每千碱基外显子每百万片段的转录本GydF4y2Ba
- WGCNA:GydF4y2Ba
-
加权基因共表达网络分析GydF4y2Ba
参考GydF4y2Ba
- 1.GydF4y2Ba
Mattick JS,Makunin IV。非编码RNA。哼唱mol tenet。2006; 15:R17-29。GydF4y2Ba
- 2.GydF4y2Ba
PONTING CP,OLIVER PL,REIK W.长的NOODING RNA的进化和功能。细胞。2009; 136:629-41。GydF4y2Ba
- 3.GydF4y2Ba
Chekanova JA。长期非编码RNA及其在植物中的功能。CurrOp植物BIOL。2015; 27:207-16。GydF4y2Ba
- 4.GydF4y2Ba
Wierzbicki at,haagjr,pikaard cs。RNA聚合酶POL IVB / POL V的非编码转录介导重叠和相邻基因的转录沉默。细胞。2008; 135:635-48。GydF4y2Ba
- 5.GydF4y2Ba
Chen Ll,Carmichael GG。解码核长期非编码RNA的功能。CurrOgin Cell Biol。2010; 22:357-64。GydF4y2Ba
- 6。GydF4y2Ba
Kornienko AE,Guenzl PM,Barlow DP,Pauler FM。长期非编码RNA转录作用的基因调节。BMC BIOL。2013; 11:59。GydF4y2Ba
- 7。GydF4y2Ba
长链非编码RNA生物发生和功能的独特特征。Nat Rev Genet, 2016; 17:47-62。GydF4y2Ba
- 8。GydF4y2Ba
Nejat N,Mantri N.长期非编码RNA在植物反应中的新兴作用对生物和非生物胁迫。CRIT Rev Bio Technol。2018; 38:93-105。GydF4y2Ba
- 9。GydF4y2Ba
张W,汉泽,郭QL,刘义,郑yx,吴佛尔等。响应干旱胁迫的玉米长期非编码RNA的鉴定。Plos一个。2014; 9:E98958。GydF4y2Ba
- 10.GydF4y2Ba
刘j,jung c,xu j,王h,邓sl,伯尔纳德L等。基因组 - 宽分析揭示了长期性非数性RNA的调节GydF4y2Ba拟南芥GydF4y2Ba.植物细胞。2012; 24:4333-45。GydF4y2Ba
- 11.GydF4y2Ba
帅P,梁D,唐S,张ZJ,叶迪,Su YY等。基因组识别和新型干旱响应LINCRNA的功能预测GydF4y2Ba杨树trichocarpaGydF4y2Ba.J Exp Bot. 2014; 65:4975-83。GydF4y2Ba
- 12.GydF4y2Ba
王TZ,刘M,赵毫克,陈RJ张WH。渗透胁迫和盐胁迫相关长链非编码rna的鉴定和特性GydF4y2BaMedicago Truncatula.GydF4y2Ba采用基因组高通量测序。BMC植物BIOL。2015; 15:131。GydF4y2Ba
- 13.GydF4y2Ba
崔吉,栾ys,姜n,包头,萌j。抗性和易感番茄之间的比较转录组分析允许识别抵抗抵抗力的LNCRNA16397GydF4y2BaPhytophthora Infestans.GydF4y2Ba共同表达glutaredoxin。植物j . 2017; 89:577 - 89。GydF4y2Ba
- 14.GydF4y2Ba
Liu F,Marquardt S,Lister C,Swiezewski S,Dean C.目标30加工反义转录物触发拟南芥FLC染色质沉默。科学。2009; 327:94-7。GydF4y2Ba
- 15.GydF4y2Ba
HEO JB,SUNG S. vernalization介导的介导的外膜遗传沉默,长内肾内含RNA。科学。2011; 331:76-9。GydF4y2Ba
- 16。GydF4y2Ba
低温诱导的反义rna的进化守恒GydF4y2Ba开花轨迹CGydF4y2Ba在GydF4y2Ba拟南芥GydF4y2Ba常年的亲戚。Nat Commun。2014;5:4457。GydF4y2Ba
- 17。GydF4y2Ba
Qin T,Zhao Hy,Cui P,Albesher N,Xiong LM。核心化长期非编码RNA增强干旱和盐胁迫耐受性。植物理性。2017; 175:1321-36。GydF4y2Ba
- 18。GydF4y2Ba
王tz,赵mg,张xx,刘米,杨cg,陈毅等。新型磷酸盐缺乏响应型长非编码RNA在豆科模型植物中GydF4y2BaMedicago Truncatula.GydF4y2Ba.J Exp Bot. 2017; 68:5937-48。GydF4y2Ba
- 19。GydF4y2Ba
王勇,罗晓军,孙峰,胡建辉,查晓军,苏伟,等。Overexpressing lncRNAGydF4y2Ba巢穴GydF4y2Ba提高稻米产量并调节邻近基因簇表达。Nat Commun。2018;9:3516。GydF4y2Ba
- 20.GydF4y2Ba
Graham D,帕特森BD。植物对低,非冻结温度的反应:蛋白质,新陈代谢和适应。Annu Rev植物理性。1982; 33:347-72。GydF4y2Ba
- 21.GydF4y2Ba
盖伊尔Cl。冷驯化和冷冻应力耐受性:蛋白质代谢的作用。Annu Rev植物植物植物Mol Biol。1990; 41:187-223。GydF4y2Ba
- 22.GydF4y2Ba
托马斯休曼氏金融。植物冷驯化:抗冻基因及其调控机制。植物生理学报1999;50:571-99。GydF4y2Ba
- 23.GydF4y2Ba
Jaglo-Ottosen KR, Gilmour SJ, Zarka DG, Schabenberger O, Thomashow MF。GydF4y2Ba拟南芥CBF1GydF4y2Ba超表达诱发GydF4y2Ba天哪GydF4y2Ba增强抗冻能力。科学。1998;280:104-6。GydF4y2Ba
- 24。GydF4y2Ba
Park SC, Lee CM, Doherty CJ, Gilmour SJ, Kim YS, Thomashow MF。监管的GydF4y2Ba拟南芥GydF4y2BaCBF调节由一个复杂的低温调节网络。植物j . 2015; 82:193 - 207。GydF4y2Ba
- 25。GydF4y2Ba
贾应旭,丁玉玲,史玉涛,张学勇,龚泽忠,杨树华GydF4y2BacbfGydF4y2Ba三重突变体揭示了基本功能GydF4y2BaCBFGydF4y2Ba在冷驯化中,允许CBF调节器的定义GydF4y2Ba拟南芥GydF4y2Ba.新植物。2016; 212:345-53。GydF4y2Ba
- 26。GydF4y2Ba
陈志强,黄志强,陈志强,等。ICE1:拟南芥冷诱导转录组和抗冻性的调节因子。基因Dev。2003;17:1043-54。GydF4y2Ba
- 27。GydF4y2Ba
弗尔索娃,Pogorelko, GV,塔拉索夫,弗吉尼亚州GydF4y2BaICE2GydF4y2Ba,参与冷驯化的基因,其决定了凝固耐受性GydF4y2Ba拟南芥GydF4y2Ba.基因。2009;429:98 - 103。GydF4y2Ba
- 28。GydF4y2Ba
Doherty CJ, Van Buskirk HA, Myers SJ, thomas MF。角色GydF4y2Ba拟南芥GydF4y2BaCAMTA转录因子在冷调节基因表达和抗冻性中的作用。植物细胞。2009;21:972 - 84。GydF4y2Ba
- 29.GydF4y2Ba
Chan ZL,王YP,Cao MJ,龚毅,亩Zx,王HQ等。GydF4y2BaRDM4.GydF4y2Ba调节冷应力阻力GydF4y2Ba拟南芥GydF4y2Ba部分通过CBF介导的途径。新植物。2016; 209:1527-39。GydF4y2Ba
- 30.GydF4y2Ba
冷信号从细胞膜向细胞核穿梭。摩尔细胞。2017;66:7-8。GydF4y2Ba
- 31.GydF4y2Ba
长链非编码RNA SVALKA的转录通读控制着植物的冷驯化。Nat Commun。2018;5:4561。GydF4y2Ba
- 32.GydF4y2Ba
Nichols PGH,Loi A,Nutt BJ,Evans PM,Craig Ad,Pengelly BC等。澳大利亚农业 - 15年革命的新年和短期多年生牧场牧场。现场作物RES。2007; 104:10-23。GydF4y2Ba
- 33.GydF4y2Ba
库克博士。GydF4y2BaMedicago Truncatula.GydF4y2Ba- 制作的模型!CurrOp植物BIOL。1999年; 2:301-4。GydF4y2Ba
- 34.GydF4y2Ba
Tadege M, Ratet P, Mysore KS。插入突变:功能基因组学的瑞士军刀GydF4y2BaMedicago Truncatula.GydF4y2Ba.植物科学进展。2005;10:229-35。GydF4y2Ba
- 35.GydF4y2Ba
年轻的Nd,Udvardi M.翻译GydF4y2BaMedicago Truncatula.GydF4y2Ba基因组学作豆类。CurrOp植物BIOL。2009; 12:193-201。GydF4y2Ba
- 36.GydF4y2Ba
Pennycooke Jc,Cheng HM,Stockinger EJ。比较基因组序列和表达分析GydF4y2BaMedicago Truncatula.GydF4y2Ba和苜蓿亚种GydF4y2Ba钩状的COLD-ACCLIMATION-SPECIFICGydF4y2Ba基因。植物理性。2008; 146:1242-54。GydF4y2Ba
- 37.GydF4y2Ba
张啊,赵田QY,张WH毫克。耐受性的比较研究GydF4y2BaMedicago Truncatula.GydF4y2Ba和GydF4y2BaMedicago Falcata.GydF4y2Ba冻结。足底。2011;234:445-57。GydF4y2Ba
- 38.GydF4y2Ba
张志强,胡新宁,张玉强,苗志勇,谢超,孟XZ,等。转录因子MYB61和MYB3的反向调控通过缓解C-repeat结合因子的抑制,提高了抗冻性。植物杂志。2016;172:1306-23。GydF4y2Ba
- 39.GydF4y2Ba
陈志强,王志强,王志强,等。紫花苜蓿低温驯化特异性基因的克隆及其与抗冻性的关系。植物杂志。1989;89:375 - 80。GydF4y2Ba
- 40。GydF4y2Ba
Wolfraim La,Langis R,Tyson H,Dhindsa Rs。CDNA序列,表达和特异性特异性基因的转录稳定性,GydF4y2BaCAS18GydF4y2Ba,由紫花苜蓿(GydF4y2BaMedicago Falcata.GydF4y2Ba)细胞。植物杂志。1993;101:1275 - 82。GydF4y2Ba
- 41。GydF4y2Ba
赵毫克,刘WJ、夏XZ王TZ,张WH。冷驯化诱导的抗冻性GydF4y2BaMedicago Truncatula.GydF4y2Ba幼苗被乙烯对抗调节。physiol platararum。2014; 152:115-29。GydF4y2Ba
- 42.GydF4y2Ba
等。CBF/DREB1基因串联序列位于水稻抗冻QTL区GydF4y2BaMedicago Truncatula.GydF4y2Ba6号染色体。BMC基因组学。2013;14:814。GydF4y2Ba
- 43.GydF4y2Ba
EWING B,Green P.使用PHRED的自动定序器痕迹的基础呼叫。II错误概率。Genome Res。1998年; 8:186-94。GydF4y2Ba
- 44.GydF4y2Ba
李某,yu x,雷n,程z,赵p,他。Cassava中冷和/或干旱反应LNCRNA的基因组鉴定和功能预测。SCI批准。2017; 7:45981。GydF4y2Ba
- 45.GydF4y2Ba
集装箱苗圃的根系冻害:对种植场地的影响——一项综述。新。2005;30:167 - 84。GydF4y2Ba
- 46.GydF4y2Ba
Suzuki K,Nagasuga K,okada M.高根温度在水稻幼苗叶子中诱导的冷冻损伤。植物细胞生理。2008; 49:433-42。GydF4y2Ba
- 47.GydF4y2Ba
Hekneby M, Antolín MC. Sánchez-DíazM。不同一年生豆科植物的抗寒性和生化变化。环境实验机器人。2006;55:305-14。GydF4y2Ba
- 48。GydF4y2Ba
低温感知导致高等植物的基因表达和耐冷性。新植醇。2012;195:737-51。GydF4y2Ba
- 49。GydF4y2Ba
舒yj,刘y,li w,歌曲ll,张j,guo ch。基于高通量测序的Medicago Sativa L的冷冻应激响应微小RNA及其靶的基因组调查。G3。2016; 6:755-65。GydF4y2Ba
- 50。GydF4y2Ba
陈勇,陈勇,德Beus M, Bowley SR, Bowler C, Inze D,等。超氧化物歧化酶提高转基因紫花苜蓿(GydF4y2Ba紫花苜蓿GydF4y2Bal .)。植物杂志。1993;103:1155 - 63。GydF4y2Ba
- 51。GydF4y2Ba
Sunkar R,Kapoor A,Zhu JK。两种Cu / Zn超氧化物歧化酶基因的术后诱导GydF4y2Ba拟南芥GydF4y2Ba由MiR398的下调和氧化应激耐受性重要。植物细胞。2006; 18:2051-65。GydF4y2Ba
- 52.GydF4y2Ba
等。碱基切除修复蛋白Ntg1中SUMO修饰位点的鉴定DNA修复。2016;48:51 - 62。GydF4y2Ba
- 53.GydF4y2Ba
陈慧聪,褚平,周玉玲,李勇,刘杰,丁勇,等。DNA糖基化酶/AP裂解酶AtOGG1的过表达提高了种子的寿命和抗非生物胁迫能力GydF4y2Ba拟南芥GydF4y2Ba.J Exp Bot。2012; 63:4107-21。GydF4y2Ba
- 54.GydF4y2Ba
江苏,朗奇桑德兰S.拓展机制及对编码DNA糖基酶的基因的进化史及其参与应激和激素信号传导。基因组Biol Evol。2016; 8:1165-84。GydF4y2Ba
- 55.GydF4y2Ba
Knight H,Mugford SG,Ulker B,Gao Dh,Thorlby G,Knight Mr。SFR6识别SFR6,在CBF函数上翻译后冷置的关键组分。工厂J. 2009; 58:97-108。GydF4y2Ba
- 56.GydF4y2Ba
等。水稻抗冻性及相关性状的遗传变异及QTL定位GydF4y2BaMedicago Truncatula.GydF4y2Ba.Al Appl Genet。2013; 126:2353-66。GydF4y2Ba
- 57.GydF4y2Ba
Guttman M, Rinn JL。大型非编码rna的模块化调控原理。大自然。2012;482:339-46。GydF4y2Ba
- 58.GydF4y2Ba
Gilmour SJ, Zarka DG, Stockinger EJ, Salazar MP, Houghton JM, Thomashow MF。低温调节GydF4y2Ba拟南芥GydF4y2BaAP2转录激活剂的CBF系列作为冷诱导的早期步骤GydF4y2Ba天哪GydF4y2Ba基因的表达。植物j . 1998; 16:433-42。GydF4y2Ba
- 59.GydF4y2Ba
刘征镒,贾应祥,丁玉玲,史玉涛,李铮,郭勇,等。冷反应中,质膜crpk1介导的14-3-3蛋白磷酸化诱导其核导入以微调CBF信号。摩尔细胞。2017;66:117-28。GydF4y2Ba
- 60。GydF4y2Ba
全美,陈建华,张迪强。探索长链非编码rna的秘密。中华医学杂志。2015;16:5467-96。GydF4y2Ba
- 61。GydF4y2Ba
Aird D,Ross Mg,Chen Ws,Danielsson M,Fennell T,Carsten R等。illumina测序文库中PCR扩增偏倚分析和最小化。基因组Biol。2011; 12:1-14。GydF4y2Ba
- 62。GydF4y2Ba
苏志强,李胜,李志强,等。由测序质量控制联盟对RNA-seq的准确性、重现性和信息内容进行综合评估。生物科技本质》。2014;32:903-17。GydF4y2Ba
- 63。GydF4y2Ba
年轻的ND,Cannon SB,Sato S,Kim D,Cour Dr,Town CD等。测序基因空间GydF4y2BaMedicago Truncatula.GydF4y2Ba和GydF4y2BaLotus粳稻GydF4y2Ba.植物理性。2005; 137:1174-81。GydF4y2Ba
- 64。GydF4y2Ba
Langmead B, Salzberg SL.快速间隙读对齐与领结2。Nat方法。2012;9:357-U54。GydF4y2Ba
- 65。GydF4y2Ba
Trapnell C,Williams Ba,Pertea G,Mortazavi A,Kwan G,Van Baren MJ,等。通过RNA-SEQ的转录程序组件和定量揭示了细胞分化期间未经发布的转录物和同种型切换。NAT BIOTECHNOL。2010; 28:511-5。GydF4y2Ba
- 66.GydF4y2Ba
Pertea M,Kim D,Pertea Gm,韭菜JT,Salzberg SL。RNA-SEQ实验与Hisat,Stringtie和Ballgown的转录级表达分析。NAT PROTOC。2016; 11:1650-67。GydF4y2Ba
- 67.GydF4y2Ba
Sun L,Luo Ht,Bu Dc,Zhao Gg,Yu Kt,Zhang Ch,等。利用序列固有组合物来分类蛋白质编码和长期非编码转录物。核酸RES。2013; 41:E166。GydF4y2Ba
- 68.GydF4y2Ba
kong l,zhang y,ye zq,刘xq,zhao sq,wei lp等。CPC:使用序列特征和支持向量机评估转录物的蛋白质编码电位。核酸RES。2007; 35:W345-9。GydF4y2Ba
- 69.GydF4y2Ba
Punta M,Coggill PC,Eberhardt Ry,Mistry J,Tate J,Boursnell C,等。PFAM蛋白质家族数据库。核酸RES。2012; 40:D290-301。GydF4y2Ba
- 70.GydF4y2Ba
林MF,Jungreis I,Kellis M.Hhylocsf:区分蛋白质编码和非编码区的比较基因组学方法。生物信息学。2011; 27:I275-I82。GydF4y2Ba
- 71。GydF4y2Ba
Langfelder P,Horvath S.WGCNA:用于加权关联网络分析的R包。BMC生物信息学。2008; 9:559。GydF4y2Ba
- 72。GydF4y2Ba
基于不确定度的RNA-seq差异分析。Nat方法。2017;14:687 - 90。GydF4y2Ba
- 73。GydF4y2Ba
Young MD, Wakefield MJ, Smyth GK, Oshlack A. RNA-seq的基因本体论分析:考虑选择偏差。基因组医学杂志。2010;11:R14。GydF4y2Ba
- 74。GydF4y2Ba
王金涛,王金涛,王金涛,等。Cytoscape:一个整合生物分子相互作用网络模型的软件环境。基因组研究》2003;13:2498 - 504。GydF4y2Ba
致谢GydF4y2Ba
我们感谢植被和环境变革的国家重点实验室测试中心的技术支持。GydF4y2Ba
资金GydF4y2Ba
该研究得到了中国国家自然科学基金(31671270)和中国科学院(KFJ-STS-ZTDTP-056)的支持。GydF4y2Ba
作者信息GydF4y2Ba
从属关系GydF4y2Ba
贡献GydF4y2Ba
作者提出了关于他们贡献的以下声明:构思了研究:Z. M.和Z. W-H。设计实验:Z.M.进行实验并分析了数据:Z.M。,W.T.,S.,Y.X.和T. R.撰写了本文:Z.M。和Z.W-H。所有作者都读过并批准了稿件。GydF4y2Ba
相应的作者GydF4y2Ba
道德声明GydF4y2Ba
伦理批准和同意参与GydF4y2Ba
不适用。GydF4y2Ba
同意出版GydF4y2Ba
不适用。GydF4y2Ba
相互竞争的利益GydF4y2Ba
作者简介:张文浩,副主编。GydF4y2Ba
附加信息GydF4y2Ba
出版商的注意GydF4y2Ba
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。GydF4y2Ba
补充信息GydF4y2Ba
附加文件1:表S1。GydF4y2Ba
实验样本RNA-Seq质量统计数据。GydF4y2Ba表S2。GydF4y2Ba实验样品LNCRNA的相关系数。GydF4y2Ba表S3。GydF4y2Ba实验样品MRNA的相关系数。GydF4y2Ba表S4。GydF4y2BaRNA-Seq结果的统计数据映射到GydF4y2BaM. Truncatula.GydF4y2BaA17基因组实验样本。GydF4y2Ba表S5。GydF4y2Ba叶片中冷响应LNCRNA的推定目标的增强GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2Ba表S6。GydF4y2BaGO增强了植物根中冷应答lncrna的假定靶点GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2Ba表S7。GydF4y2Ba引物序列用于实时定量RT-PCR。GydF4y2Ba
附加文件2:图S1。GydF4y2Ba
lncrna在8条染色体上的密度分布GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2Ba
附加文件3:图S2。GydF4y2Ba
LNCRNA对染色体的数量和密度分布GydF4y2BaM. Truncatula.GydF4y2Ba幼苗有无冷处理。GydF4y2Ba
附加文件4:图S3。GydF4y2Ba
通过高通量测序识别MRNAGydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2Ba
附加文件5:图S4。GydF4y2Ba
低温反应mrna的特征GydF4y2BaM. Truncatula.GydF4y2Ba幼苗。GydF4y2Ba
附加文件6:图S5。GydF4y2Ba
利用MAGE构建MtCBFs和AtCBFs的蛋白序列比对。GydF4y2Ba
附加文件7:图S6。GydF4y2Ba
采用MEGA软件构建MtCBFs和AtCBFs系统发育树。GydF4y2Ba
附加文件8:数据集S1。GydF4y2Ba
lncRNA TPM的结果。GydF4y2Ba
附加文件9:数据集S2。GydF4y2Ba
lncRNA序列。GydF4y2Ba
附加文件10:数据集S3。GydF4y2Ba
mRNA TPM结果。GydF4y2Ba
附加文件11:数据集S4。GydF4y2Ba
信使rna序列。GydF4y2Ba
权利和权限GydF4y2Ba
开放获取GydF4y2Ba本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问GydF4y2Bahttp://creativecommons.org/licenses/by/4.0/GydF4y2Ba.创作共用及公共领域专用豁免书(GydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/GydF4y2Ba)适用于本文提供的数据,除非在数据的信贷额度中另有说明。GydF4y2Ba
关于这篇文章GydF4y2Ba
引用这篇文章GydF4y2Ba
赵敏,王涛,孙涛。GydF4y2Ba等等。GydF4y2Ba组织特异性和冷应答型lncrna的鉴定GydF4y2BaMedicago Truncatula.GydF4y2Ba通过高通量RNA测序。GydF4y2BaBMC植物杂志GydF4y2Ba20.GydF4y2Ba99(2020)。https://doi.org/10.1186/s12870-020-2301-1GydF4y2Ba
收到了GydF4y2Ba:GydF4y2Ba
公认GydF4y2Ba:GydF4y2Ba
发表GydF4y2Ba:GydF4y2Ba
迪伊GydF4y2Ba:GydF4y2Bahttps://doi.org/10.1186/s12870-020-2301-1GydF4y2Ba
关键词GydF4y2Ba
- cbfGydF4y2Ba
- 冷应激GydF4y2Ba
- 长期非编码RNAGydF4y2Ba
- Medicago Truncatula.GydF4y2Ba
- mtcir1.GydF4y2Ba