摘要
背景
可变剪接(AS)在调节转录组和蛋白质组多样性中起着重要的调节作用。特别是,它增加了蛋白质的功能多样性。最近利用RNA-Seq对AS进行的全基因组分析表明,AS在植物中非常普遍。此外,已有研究表明,大多数AS事件受组织特异性调控。
描述
为了揭示AS和组织特异性剪接事件所诱导的功能特征,需要一个数据库来探索这些特征,特别是在植物中。为了实现这些目标,我们构建了一个由黄瓜选择性剪接生成的注释转录本数据库(CuAS:http://cmb.bnu.edu.cn/alt_iso/index.php.),集成了基因组注释、亚型功能、亚型特征和多个组织间的组织特异性AS事件。CuAS支持一个检索系统,可以识别唯一的ID(基因ID、亚型ID、UniProt ID和基因名)、染色体位置和基因家族,并支持一个浏览器来可视化每个基因。
结论
我们认为CUAS可以有助于揭示由黄瓜中的组织特异性诱导的新功能特征。CUAS自由地提供http://cmb.bnu.edu.cn/alt_iso/index.php..
背景
可变剪接(Alternative splicing, AS)是一个重要的转录后过程,由一个基因产生多个转录本。它在适应环境、发育和组织特异性方面发挥着关键作用[1那2那3.那4.].此外,它增加了蛋白质的功能多样性[2].
自第40年前的第一次发现以来[5.,越来越多的选择性剪接基因已被报道。随着测序技术的发展,人们发现AS在真核生物中普遍存在。最近,基于RNA-Seq数据,95%的人类基因[6.)和61%的拟南芥基因(7.据报道,据据报道是经历的。此外,已研究的功能。新兴的实验证据表明,可以调节蛋白质的以下性质:1)与其他蛋白质和核酸结合[8.[2)根据本地化信号的蛋白质定位[9., 3)酶的性质[104)与配体相互作用[11].总体而言,只要影响蛋白质功能的各个方面就可以影响[2].
多个AS数据库,如ASpedia [12], VastDB [13]和dbate [14]已经建立,但这些数据库是针对脊椎动物,尤其是人类,以及其中很少有植物的地址。在暴露于环境压力的植物中,通过替代剪接来调节许多生物学过程[3.].随着测序技术的发展,植物中AS的检测日趋成熟[15].因此,需要一个数据库,用于作为事件和检索系统查询和探索植物中的换档抄本的功能。
在这里,我们介绍了由黄瓜(CUA)生成的注释转录物数据库(Cucumis sativusl . var。缎点简历。9930年和Cucumis sativusvar。Hardwickii.PI 183967)。该数据库提供了五种类型的数据:(1)基因组注释,(2)作为从多种组织,(3)同种型特征,(4)同种型功能,(5)和组织之间的剪接事件。Web应用程序包括四个组件:注释数据库,检索系统,浏览器和工具。该用户友好的数据库将作为集线器,用于揭示由Cucumbers的事件和组织特异性诱导的功能特征。
结构和内容
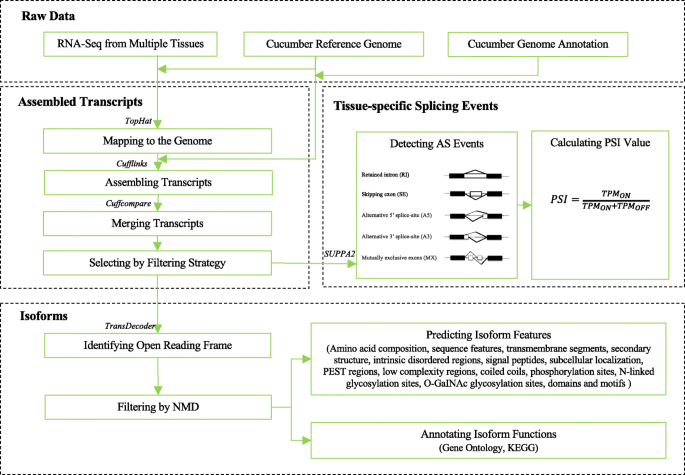
CuAS数据库集成了基因组注释、来自多个组织的AS事件、亚型功能、亚型特征和组织特异性剪接事件。积分步骤如图所示。1.
数据源
CuAS包括两个黄瓜品种的数据:Cucumis sativusl . var。缎点简历。9930年和Cucumis sativusvar。Hardwickii.PI 183967.从中收集了基因组序列和基因组注释http://cmb.bnu.edu.cn/Cucumis_sativus_v20/.来自十种组织的RNA-SEQ数据Cucumis sativusl . var。缎点简历。从SRA数据库下载9930(https://www.ncbi.nlm.nih.gov/sra/)(SRA:SRA046916),七种组织的RNA-SEQ数据Cucumis sativusvar。Hardwickii.PI 183967获取从网站获得http://cmb.bnu.edu.cn/Cucumis_sativus_v20/.这七个组织包括根、茎、叶、雄花、雌花、果实和卷须。
识别替代剪接事件和同种型
在之前的研究中基于RNA-Seq的十个组织来自Cucumis sativusl . var。缎点简历。9930,我们通过使用TOPHAT和袖扣组装成绩单[16], 分别。然后将这些成绩单与使用袖手组合的参考基因组注释文件进行比较。根据袖手保护的输出,转录物分为12类。然后,采用以下策略来获得高质量的转录物[17那18].首先,所有包含三个类代码(=,j, o) (http://cole-trapnell-lab.github.io/cufflinks/cuffcompare/)从Cuffcompare生成的输出中提取。“j”类和“o”类的成绩单被认为是新的成绩单。接下来,带有单个外显子的新转录本被移除,我们得到了一个组装好的黄瓜转录组。为了减少潜在的错误组装的转录本,每个新的剪接连接需要至少10个读取支持,每个已知的剪接连接需要至少一个读取支持。根据这些标准,获得了某些剪接连接reads支持的转录本。最后,使用Salmon(版本0.13.0)计算每百万读的转录本(TPM)值[19[至少一个样品中的具有大于或等于一个样品的TPM值的转录物用于分析[20.].随着一系列过滤器的实现,获得了高质量的推定转录组。基于所获得的转录物,通过使用Suppa2(2.3版)鉴定事件[21].AS事件分为5种类型:保留内含子(RI)、跳过外显子(SE)、可选3 '剪接位点(A3)、可选5 '剪接位点(A5)和互斥外显子(MX)。
为了更好地理解由单个基因编码的差异剪接亚型的影响,我们使用了TransDecoder (https://github.com/transdecoder/transdecoder.(版本3.0.1)以识别组装的转录本中的候选编码区域。TransDecoder对Pfam 30.0进行同源搜索[22]和Uniprot数据库(版本2016_11)[23为开放式阅读框架(ORFs)获取支持证据。我们使用参数“-single_best_orf”为每个转录本选择最佳的单一ORF。如果一个提前终止密码子位于距离最后一个剪接连接超过55个核苷酸的位置,则该转录本被认为是无意义介导的mRNA衰变(NMD)的结果[24那25那26].任何ORF长度大于或等于300 bp且未显示NMD的转录本都被保留以供进一步分析。采用相同的软件和参数Cucumis sativusvar。Hardwickii.PI 183967。
同种型水平的功能注释
首先,我们执行了一个Blast2Go [27为每个亚型分配基因本体论术语的分析。Blast2GO对UniProt (release 2017_06)数据库执行BLASTP搜索(E-value 1e-05)。然后,将鉴定出的同源异构体映射到京都基因和基因组百科全书(KEGG) (https://www.genome.jp/kegg/,版本90.1)[28].kaas(kegg自动注释服务器,https://www.genome.jp/tools/kaas/)用于分配KEGG通路。
在同种型水平上的特征预测
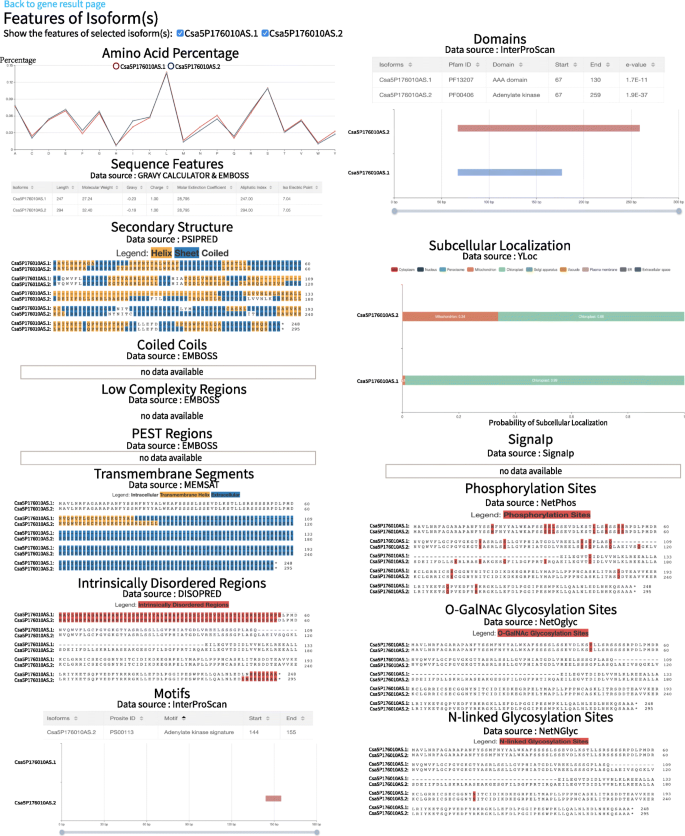
用于预测同种型功能的软件列于表中1.共预测了15种特征,包括氨基酸组成、序列特征、跨膜片段、二级结构、固有紊乱区、信号肽、亚细胞定位、PEST区、低复杂性区、卷曲区、磷酸化位点、n -连接糖基化位点、O-GaINAc糖基化位点、结构域和基序。
通过UniRef90数据集(release 2016_01)搜索固有紊乱的跨膜片段、二级结构和区域。使用InterProScan 5.24分配域和motif [29].
组织剪接事件
为了调查组织特异性剪接事件,作为事件量化,作为事件测量的代表性的剪辑索引(PSI)的百分比百分比(PSI)被量化。PSI测量从含有作为事件的特定形式的基因表达的mRNA的级分[30.].这些reads被用来量化Salmon的转录丰度[19],以及PSI值[31],用SUPPA2计算所有AS事件。
基因描述和基因家族的预测
基因的功能描述由AHRD工具提供(https://github.com/groupschoof/AHRD)基于BLASTP对ULIPROT和TAIR的搜索结果。关于基因家族,转录因子(TFS),转录调节剂(TRS)和蛋白激酶(PKS)被ITAK鉴定(版本1.7)[32].通过Orthofinder鉴定剪接相关基因(版本:2.3.1)[33与…的顺序相反拟南芥[34,包括小核核糖核蛋白、剪接因子、剪接调控相关蛋白、新型剪接体蛋白和可能的剪接相关蛋白。
网络实现
Web界面使用PHP编程,HTML和JavaScript实现。通过插件echarts生成所有图形[35].所有表都符合layui的风格(https://www.layui.com/).Poshy Tip(https://github.com/vadikom/poshytip)用于显示氨基酸的位置。
效用和讨论
CUAS系统包含四个组件:注释数据库,检索系统,浏览器和工具(BLAST和JBROWSE)。
数据库概述
总共有60,643份成绩单(36,274份来自Cucumis sativusl . var。缎点简历。9930和24,369来自Cucumis sativusvar。Hardwickii.PI 183967)。根据这些记录,10748个AS事件(6673个来自Cucumis sativusl . var。缎点简历。9930和4075从Cucumis sativusvar。Hardwickii.PI 183967)和49,018个亚型(28,588来自Cucumis sativusl . var。缎点简历。来自9930和20,430Cucumis sativusvar。Hardwickii.保留了PI 183967以分析特征和功能。同种型功能用基因本体进行注释[36]和kegg [28]。关于亚型特征,预测了15种类型的特征。此外,对所有AS事件的PSI值进行了量化(参见构建和内容)。
Web界面
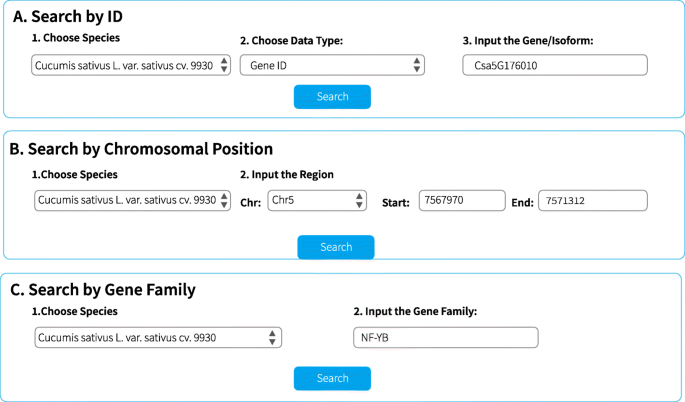
CUAS Web-界面提供对基因组注释的访问,同种型水平的功能注释,同种型水平的特征,以及组织特异性的事件。可以使用三种输入格式查询数据:ID(基因ID /同种型ID / UNIPROT ID /基因名称),染色体位置和基因家族(图。2,例如,CSA5G176010).这些输入数据可用于组织间AS事件及其相关标注的搜索。
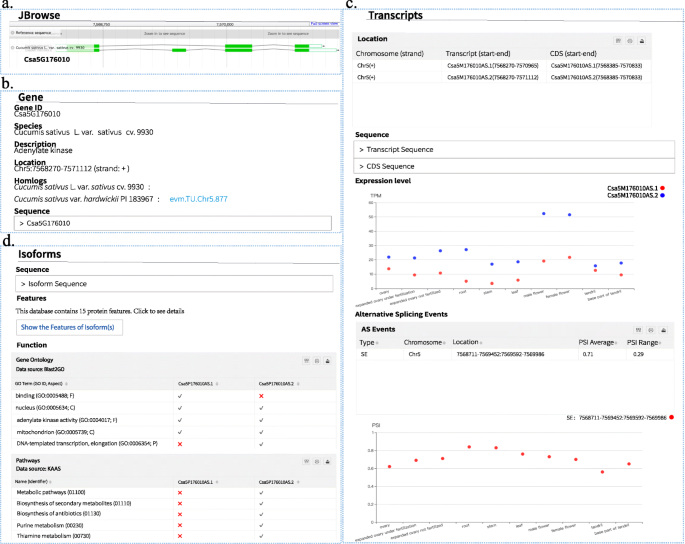
搜索结果在结果页面被分类和可视化,如图所示。3.通过使用的例子CSA5G176010.两个转录本的结构CSA5G176010由jbrowse显示(图。3.a).结果组织在三个水平,基因,转录本和亚型水平。在基因水平上,我们列出了该基因及其在两个黄瓜中的同源物的基本信息(图。3.b)。在组织中的每个查询基因报告,在组织中的每个查询基因报告了转录物水平的转录表达丰度,预测为这些事件的PSI值和PSI值。这也在图2中示出。3.c,其中检测到SE事件CSA5G176010.两种转录物在所有组织中表达。在同种型水平下,提供了同种型功能注释(GO注释和Kegg途径注释)和基因同种型的特征。如图1所示。3.的两种异构体CSA5G176010呈现一些不同的功能,例如“绑定”和“放大器救助”。
可以通过单击保留替代同种型的特征“显示同种型的特征”(图。3.d).特征列表显示在isoform特征页面上(图。4.),包括氨基酸组成、序列特征、跨膜片段、二级结构、固有紊乱区、信号肽、亚细胞定位、PEST区、低复杂区、卷曲区、磷酸化位点、n -连接糖基化位点、O-GaINAc糖基化位点、结构域、和主题(参见结构和内容)。如图1所示。4.使用CSA5G176010例如,CSA5P176010AS.1包括“腺苷激酶签名”图案,但CSA5P176010AS.2不包括基序。此外,两个转录物之间存在不同的功能特性。这些结果表明SE事件检测到CSA5G176010对异构体的功能有影响。
此外,还提供了两个工具:BLAST和JBrowse。利用BLAST技术寻找黄瓜的同源序列。用户可以在“查询序列”框中粘贴他们的DNA或蛋白质查询序列。用户可以设置搜索参数,如搜索数据库、搜索程序、最大命中数和e值。用户可选择“查询资料”以选择查询资料库。BLAST数据库包括两个黄瓜的基因、转录本、CDSs和亚型。通过选择“程序”,根据查询顺序和搜索数据库,可以选择搜索程序(BLASTN、TBLASTX、BLASTX、TBLASTN、BLASTP)。“高级选项”可用于设置最大命中次数和e值。使用JBrowse可视化黄瓜的基因组特征,包括黄瓜多个组织的转录本。
我们的数据库提供HTTP链接以在Fasta格式下载基因组序列,转录序列,推定CDS和蛋白质序列。基因结构注释可以以GFF3格式获得。可以获得映射到UNIPROT的ID列表。作为事件和PSI值也可以下载。数据文件列表(包括异形功能和同种型功能)也可以文本格式访问。CUAS网站上提供了详细的用户手册。
结论
RNA-SEQ的出现推动了转录组织的快速扩张。这增加了功能特征和转录物之间的差距,这是试图了解如何出现多样性的关键步骤。CUA提供用于探索功能特征与从黄瓜中多个组织预测的转录物的资源,并且可以从PSI值获得组织特异性。CUA将有助于揭示用作植物中的事件和组织特异性诱导的新功能特征。
CUA是一个正在进行的项目,我们计划在下一个版本中进一步发展它。特别是,我们将为作为网站添加变更注释,并探索变异与变化之间的关系。我们还计划包括与其他生物相关的数据,例如Cucumis Melo.l . (37] 和Citrullus lanatus.[38],这将有助于通过对葫芦科植物AS的比较分析,更好地了解AS。
数据和材料的可用性
CUAS自由地提供http://cmb.bnu.edu.cn/alt_iso/index.php..数据集可以从这里下载http://cmb.bnu.edu.cn/alt_iso/index.php/download..详细的用户手册可在http://cmb.bnu.edu.cn/alt_iso/index.php/help.该网站针对Internet Explorer,Mozilla Firefox,Google Chrome和Safari进行了优化。
缩写
- A3:
-
替代3'拼接 - 网站
- A5:
-
选择5 '杂交
- 为:
-
可变剪接
- CuAS:
-
通过黄瓜替代剪接产生的注释转录物数据库
- MX:
-
相互排斥的外影
- NMD:
-
nonsense-mediated mRNA衰变
- 子:
-
开放阅读框
- 战:
-
蛋白激酶
- PSI:
-
拼接百分比指数
- 国际扶轮:
-
保留内含子
- SE:
-
跳过外显子
- TFS:
-
转录因素
- TPM:
-
每百万读的成绩单
- TRs:
-
转录监管机构
参考文献
- 1.
洛佩兹AJ。pre-mRNA的选择性剪接:发育后果和调控机制。Annu Rev Genet 1998; 32:279-305。
- 2.
Kelemen O,Convertini P,Zhang Z,Wen Y,Shen M,Falaleeva M,STAMM S.替代拼接的功能。基因。2013; 514(1):1-30。
- 3.
Staiger D,棕色JW。生物定时,发展和应力响应交叉处的替代拼接。植物细胞。2013; 25(10):3640-56。
- 4.
Lee Y,RIO DC。替代前mRNA剪接的机制和调节。annu rev biochem。2015; 84:291-323。
- 5.
Berget Sm,Moore C,Sharp Pa。腺病毒2晚期mRNA的5'末端的拼接区段。Proc Natl Acad Sci U S A. 1977; 74(8):3171-5。
- 6。
潘Q,Shai O,Lee Lj,Frey Bj,Blencowe Bj。高通量测序对人体转录组替代剪接复杂性的深度测量。NAT Genet。2008; 40(12):1413-5。
- 7。
Marquez Y,Brown JW,Simpson C,Barta A,Kalyna M.转录Mome调查显示替代剪接景观的复杂性增加拟南芥.Genome Res。2012; 22(6):1184-95。
- 8。
Belfiore A,Frasca F,Pandini G,Sciacca L,Vigneri R.胰岛素受体同种型和胰岛素受体/胰岛素样生长因子受体杂种在生理学和疾病中。EndoCr Rev. 2009; 30(6):586-623。
- 9。
朱海英,王志强。下丘脑同源盒基因Bsx1a及其亚型Bsx1b的克隆与功能分析。中国生物医学工程学报。2007;27(10):3743-9。
- 10。
两个具有不同性质的肝素酶剪接变异在早期是必要的Xenopus.发展。J Biol Chem。2008; 283(23):16004-16。
- 11.
Ko J,Fuccillo MV,Malenka RC,Sudhof TC。LRRTM2用作促进兴奋性突触形成的新生素配体。神经元。2009; 64(6):791-8。
- 12.
亨达,金吉,赵世英,朴志明。人类选择性剪接的综合百科全书。核酸Res. 2018;46(数据库版):D58-63。
- 13.
Tapial J, Kch H, Sterne-Weiler T, Gohr A, Braunschweig U, Hermoso-Pulido A, Quesnel-Vallières M, Permanyer J, Sodaei R, Marquez Y.可选择剪剪谱和功能关联图谱揭示了同时表达多种主要异构体的新的调控程序和基因。基因组研究》2017;27(10):1759 - 68。
- 14.
Bianchi V, Colantoni A, Calderone A, Ausiello G, Ferrè F, Helmercitterich M. DBATE:替代转录本表达的数据库。数据库。2013;2013 (6):bat050。
- 15.
Syed NH, Kalyna M, Marquez Y, Barta A, Brown JW。植物的选择性剪接——成熟。植物学报,2012;17(10):616-23。
- 16。
Sun Y,Hou H,Song H,Lin K,Zhang Z,Hu J,Pang E.黄瓜多组织中的替代拼接比较。BMC植物BIOL。2018; 18(1):5。
- 17。
董超,何锋,Berkowitz O,刘建军,曹鹏,唐敏,史华,王伟,李强,沈志,等。选择性剪接在维持水稻矿质营养稳态中起着关键作用(栽培稻).植物细胞。2018;30(10):2267 - 85。
- 18。
王志刚,王志刚,李斌。干旱胁迫和玉米发育过程中选择性剪接的全基因组分析[J]。植物杂志。2016;170(1):586 - 99。
- 19。
Salmon提供了快速和偏知的转录本表达量化。Nat方法。2017;14(4):417 - 9。
- 20。
Wagner GP, Kin K, Lynch VJ。一个基于模型的基因表达标准调用RNA-seq数据。理论Biosci。2013;132(3):159 - 64。
- 21。
Trincado JL, Entizne JC, Hysenaj G, Singh B, Skalic M, Elliott DJ, Eyras E. SUPPA2:在多种条件下快速、准确和不确定性感知的差异拼接分析。基因组医学杂志。2018;19(1):40。
- 22。
Finn Rd,Coggill P,Eberhardt Ry,Eddy SR,Mistry J,Mitchell Al,Potter SC,Punta M,Qureshi M,Sangrador-Vegas A等。PFAM蛋白质家族数据库:迈向更可持续的未来。核酸RES。2016; 44(D1):D279-85。
- 23.
UniProt CT。UniProt:通用的蛋白质知识库。核酸学报2018;46(5):2699。
- 24.
纳吉E, Maquat LE。含内含子基因终止密码子位置的规则:当无意义影响RNA丰度时。生物化学进展。1998;23(6):198-9。
- 25.
基于nmd的基因调控——植物适应性增强的策略?植物生理学报。2019;60(9):1953-60。
- 26.
Kalyna M, Simpson CG, Syed NH, Lewandowska D, Marquez Y, Kusenda B, Marshall J, Fuller J, Cardle L, McNicol J,等。选择性剪接和无意义介导的衰退调节重要调控基因的表达拟南芥.核酸学报2012;40(6):2454-69。
- 27.
Blast2GO:植物基因组学功能分析的综合套件。植物基因组学杂志,2008;
- 28。
Kanehisa M,Goto S,Kawashima S,Okuno Y,Hattori M.用于解密基因组的Kegg资源。核酸RES。2004; 32(数据库问题):D277-80。
- 29。
InterProScan是InterPro签名识别方法的集成平台。生物信息学。2001;17(9):847 - 8。
- 30。
王等,桑德堡河,罗S,Khrebtukova I,张L,Mayr C,Kingsmore SF,Schroth GP,击败CB。人体组织转录om中的替代异构体调节。自然。2008; 456(7221):470-6。
- 31。
Alamancos GP,PagèsA,Trincado JL,Bellora N,Eyras E.利用转录程序量化,以便快速计算替代拼接轮廓。RNA。2015; 21:1521-31。
- 32。
Zheng Y Y, Jiao C, Sun H, Rosli HG, Pombo MA, Zhang P, Banf M, Dai X, Martin GB, Giovannoni JJ, et al. iTAK:一种植物转录因子、转录调节因子和蛋白激酶的全基因组预测和分类程序。摩尔。2016;9(12):1667 - 70。
- 33。
Emms DM, Kelly S. OrthoFinder:比较基因组学的系统发育直系学推断。基因组医学杂志。2019;20(1):238。
- 34。
Wang BB,Brendel V. ASRG数据库:识别和调查拟南芥蒂利亚纳参与前mrna剪接的基因。基因组医学杂志。2004;5 (12):R102。
- 35.
李丹,梅华,沈勇,苏胜,张伟,王军,祖敏,陈伟。基于图表的web可视化快速构建框架。视觉信息。2018;2(2):136 - 46所示。
- 36.
Ashburner M,球,Blake Ja,Botstein D,Butler H,Cherry JM,Davis AP,Dolinski K,Dwight SS,EPPIG JT等人。基因本体:生物学统一的工具。基因本体组织。NAT Genet。2000; 25(1):25-9。
- 37.
Garciamas J, Benjak A, Sanseverino W, Bourgeois M, Mir G, González VM, Hénaff E, Câmara F, Cozzuto L, Lowy ECucumis Melo.L.)。Proc Natl Acad Sci U S A. 2012; 109(29):11872。
- 38.
郭胜,张建军,孙浩,Salse J, Lucas WJ,张华,郑勇,毛磊,任勇,王铮,等。西瓜(Citrullus lanatus.)并重新排列20种不同的换人。NAT Genet。2013; 45(1):51-8。
- 39.
米P,Longden I,Bleasby A. Falmoss:欧洲分子生物学开放的软件套房。趋势类型。2000; 16(6):276-7。
- 40.
琼斯DT,泰勒WR,桑顿JM。一种用于预测全螺旋膜蛋白结构和拓扑结构的模型识别方法。生物化学。1994;33(10):3038 - 49。
- 41.
琼斯DT。基于位置特异性评分矩阵的蛋白质二级结构预测。中华医学会昆虫学分会。1999;292(2):195-202。
- 42。
Jones Dt,Cozzetto D. Dipred3:用注释的蛋白质结合活性精确无序区域预测。生物信息学。2015; 31(6):857-63。
- 43。
Bendtsen JD,Nielsen H,Von HG,Brunak S.改进了信号肽的预测:SignalP 3.0。J Mol Biol。2004; 340(4):783-95。
- 44。
Briesemeister S,RahnenführerJ,Kohlbacher O. Yloc - 一种可解释的Web服务器,用于预测亚细胞定位。核酸RES。2010; 38(Web服务器问题):497-502。
- 45。
Blom N,Sicheritz-PonténT,Gupta R,Gammettoft S,Brunak S.从氨基酸序列的翻译后糖基化和蛋白质磷酸化的预测。蛋白质组学。2004; 4(6):1633-49。
确认
我们感谢编辑和匿名审稿人进行令人欣赏稿件的洞察力反馈。
资金
这项工作得到了中国天然科学基金的支持(授予31571361)。资金机构在研究和收集,数据的设计方面没有作用,或者撰写稿件。
作者信息
隶属关系
贡献
ELP和YS对分析进行了构思和设计。YS、QBZ、BL参与了数据库并实现了用户界面。ELP, YS和KL起草了手稿。ZHZ为这项工作提供了必要的建议。所有作者阅读并批准了最终的手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
太阳,Y.,张,Q.,刘,B。et al。CUA:通过在黄瓜中替代剪接产生的注释转录物数据库。BMC植物杂志20,119(2020)。https://doi.org/10.1186/s12870-020-2312-Y.
已收到:
公认:
发表:
关键字
- 黄瓜
- 可变剪接
- Isoform-level函数
- Isoform-level特性
- 组织特异性可变剪接事件