摘要
背景
凤梨科被子植物有超过3500种,具有极高的形态和生态多样性,但遗传变异非常低。在许多属中,植物的营养非常相似,这使得很难确定不开花凤梨科植物。这对于活的植物来说尤其成问题,因为这些植物通常被种植了几十年而没有开花。因此,DNA条形码技术是一种非常有前途的方法,为物种测定提供可靠和方便的辅助。然而,在凤梨属中所观察到的标准条形码标记的低遗传变异导致了问题。
结果
在这项研究中,低拷贝核基因AGT1.被鉴定为适用于密切相关的溴尿液的分子鉴定的新型DNA条形码标志物。将与相邻的遗传高度可变的内含子组合相对慢慢地发展的外显子序列,正确匹配的基于Megablast基物种鉴定率为迄今为止使用其他条形码标记的溴脲报道的最高速率约为两倍。
结论
在目前的工作中,我们描述AGT1.作为一种新的植物DNA条形码标记,用于用于溴脲的条形尺寸,遗传变异低的植物组。此外,我们提供了一个全面的标记序列数据集,用于进一步用于Bromeliad研究界。
背景
迅速辐射的单子叶植物凤梨科目前被认为包括3597种和76属[1]主要分布主要限制在新生儿[2,3.].Bromeliadds是一个形态学上高度独特的植物组,对于大多数属于哪一个最新的专着。因此,确定需要良好的专业知识,这是一些有问题的群体可能只能被少数专家自信地识别的那一点。此外,测定通常依赖于生成人物,而开花很少发生在许多溴洲,特别是当在北半球的植物园中栽培时。因此,测定的辅助方法,例如DNA条形码,对Bromeliad研究界具有高兴趣。这特别适用于植物园背景,因为菠萝蛋白酶通常包括主要部分,因为他们作为观赏植物的普及以及他们的科学价值,例如他们的科学价值。作为一种型号组,用于快速辐射和凸轮光合作用的演变[3.,4,5].
在倡议的范围内,以改善德国植物园植物园生物植物植物和溴 - 群岛(Evoboga)的植物园植物植物收集和可用性[6],我们旨在开发一种易于处理和经济高效的DNA条形码协议,并为Bromeliad提供全面的条形码数据库。
DNA条形码方法广泛用于识别动物物种,并在寿命国际条码(Ibol)项目的范围内,可以使用遗传标记成功确定大量的动物物种[7].对于动物条形码,线粒体基因组的细胞色素C氧化酶I(COI)座位在植物中普遍使用,同样的轨迹不是信息性的,因此需要替代方案[8,9].塑性标记如matK和rbcl.以及植物条形码提出了核标记,如ITS1 / ITS2及其组合,但适用性并不普遍,因此不同的组需要不同的条形码标记[10.,11.,12.,13.].
近年来,已经进行了一些努力,以测试溴脲的一套规范植物条形码标志物。但是,证明了这一点rbcl.,trnh-psba.和matK是不足以决定物种的,因为遗传变异较低[14.].因此,开发凤梨特有的条形码程序需要评估新的潜在的DNA条形码标记,并最终考虑新的条形码方法。
在本研究中,我们研究了低拷贝核基因(LCNG)的潜力AGT1.作为Bromeliaceae DNA条形码的标志物。AGT1.编码参与光素途径的拟甲酰胺氨基转移酶拟南芥[15.,16.,17.]轨迹被建议作为低分类学水平的系统发育研究的标志物[18.].自那以后AGT1.已成功用于许多系统发育研究,涵盖了各种Angiosperm植物组[19.,20.,21.,22.,23.,24.,25.].在最近两项旨在修订“隐齿状体复合体”的研究中[23.]和属菠萝,包括其最亲近的亲属[25.],AGT1.被证明对溴脲基西/亚类水平的重建具有很大的价值,从而促进进一步调查其对条形码的潜在适用性。
基于这些目标,我们选择了来自主要溴脲亚属的良好和系统大学分类的代表并产生了AGT1.序列以:(1)检查遗传多样性,并评估存在“条形码 - 间隙”的内部变化;(2)阐明系统发育相关性和限制AGT1.序列信息;(3)评估基于Megablast的物种确定率并将其与matK;(4)评估以下物种水平的分辨率,用于人口研究和潜在援助AGT1.追踪溴皂甙症的杂交起源。
结果
AGT1.PCR扩增和测序成功率
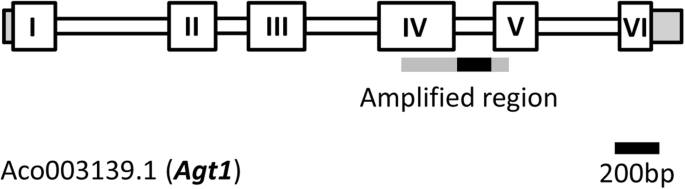
的AGT1.本研究使用的标记区域最初如既往研究所述进行放大[18.,23.].由于双条带出现频率较高,我们设计了凤梨属特异性引物,其退火温度较高,以获得更高的特异性,并阻止非特异性条带的出现。由此,我们可以得到300-700碱基对(bp)长AGT1.通过凝胶电泳和荧光定量验证了足够的DNA质量的415个案例(89.2%)的PCR片段。
此外,使用时,Sanger测序成功率相当低AGT1.特定的引物也用于测序。因此,我们在每个寡核苷酸上添加了通用测序引物位点(M13, SP6)。考虑到只有那些成功的情况下,测序工作的两个引物,从而导致一个全长一致序列比对,AGT1.排序成功率为370中的287人(77.5%)。我们一起获得,我们获得了579AGT1.Bromeliaceae Barcodes为本研究,覆盖236 Tillandsioideae,3 Hechtioideae,3 Navioideae,38 pitcairnioideae,10 puyoideae,1 brocchinioideae和288溴脲素分类,其中一些在早期的研究中已经发表[23.,25.].
在51例(约15%)中,我们无法成功序列AGT1.Intron IV部分作为电泳图显示双峰,可能是由于内含子内的等位基因变化。为了更详细地调查这一点,我们也克隆了AGT1.所选中的等位基因,并发现由于Intron IV中的插入/删除而发生的等位基因差异在所有检查的情况下(附加文件4:S4)。这些等位基因差异被限制在外显子IV序列部分内,我们无法检测到显着的序列变异。
AGT1.序列特性和序列聚类
部分AGT1.基因体扩增包括大约四分之三的外显子IV(264bp),整个内含子IV以及外显子v(14bp)的非常短的部分,如图2所示。1.与之相反AGT1.ortholog of.拟南芥蒂利亚纳凤梨属的基因体长约3025 bp,包含5个外显子而不是4个外显子(附加文件)7:S7)。溴脲AGT1.内含子IV区被证明是高度可变的AGT1.序列在我们的数据库,从大约100碱基对(bp)到大约400 bp。当试图比对所有的插入子IV时,我们发现插入/缺失的频率很高,不仅在远亲类群中,而且在同一亚科的物种中。因此,我们无法对整个凤梨科进行比对,也无法对凤梨科的亚科进行比对。
与之前的研究一样,表明序列分歧AGT1.Intron IV在Genera / Clade水平上相当低,因此保持良好[23.,25.,我们评估了AGT1.这两项研究中报道的进化支之间的序列相似性,发现值≥97%的序列一致性。鉴于此,我们的目标是在所有样本中找到序列一致性高于98%的簇AGT1.我们的数据库中存在的序列,使用DNA序列聚类软件CD-HIT-EST [28.].
如表所示1,我们发现52个序列一致性≥98%的簇AGT1.凤梨亚科、Tillandsioideae和Pitcairnioideae分别有34个和8个簇。
系统发育有效性AGT1.序列簇
支持系统发育相关性AGT1.凤梨亚科植物的序列聚类研究Portea./Gravisia复杂的(29.].对应的序列出现在AGT1.数据集集群,具有高于98%的标识值且没有非“Portea./Gravisia该集群中存在复杂的“成员”。另外六个集群代表最近被分配到“隐齿象复合体”的群体[23.],五是“nidularioid综合体”的成员[30.,31.[一个仅包括最近学习的属的分类群菠萝[25.].在剩下的溴脲中AGT1.序列我们发现了七个集群,这些集群仅包括以下任一属的成员:Wittmackia,Billbergia,Araeococcus,Hohenbergia.,Lymania要么Neoregelia..所有这些属性当前被认为是单噬细胞[29.,30.,31.,32.,33.,34.,35.,36.,37.,38.].
虽然我们得到了31种序列簇我们得到了系统发育载体,但检测到的溴脲簇中的9个具有不按照电流分类的物种组合物。十二个溴脲AGT1.集群由同一物种的成员组成。
如果是34AGT1.我们在Tillandsioideae成员中检测到的序列簇,我们参考了最近发表的两个研究,基于多基因座DNA序列和形态学数据[1,39.,40].如表所示1,我们发现有21个簇有文献支持,10个簇包含不同分支的分类群,与文献相矛盾(补充文件)2:表S2)。此外,在所有的类群中,我们发现了尚未被纳入任何系统发育研究的分类群。
的AGT1.我们从皮特证尼省成员获得的序列全部与同一属的成员聚集[1,41.].据此,我们发现5个簇只包含单系属中的任何一种Pitcairnia.,抚养名和德国ocohnia.(附加文件2:表S2)。
属级别遗传多样性和“条形码差距”评估
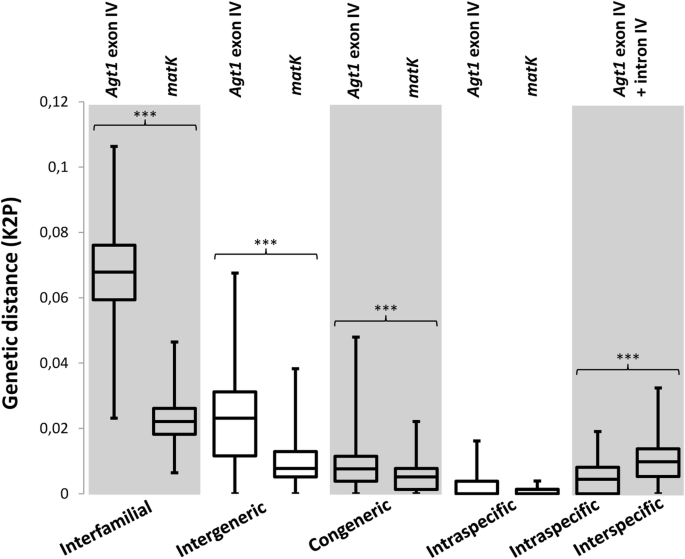
以评估其遗传多样性AGT1.EXON IV / Intron IV序列,我们产生了群集的对齐,序列相似度高于98%,并使用Kimura 2参数(K2P)模型计算遗传多样性(附加文件5:S5)。如图所示。2(最右边窗格),不同物种(种间)的K2P值在005至0015之间,离群值和中位数约为0.01。
遗传分化(K2P)AGT1.外显子IV(n= 234),matK序列(n = 233) among different taxonomic levels, as well asAGT1.外显子IV(n= 45,山苍子科)和matK(n= 58, Tillandsioideae)的不同来源的Tillandsioideae(种内)。最右边窗格:遗传分化之间AGT1.Tillandsioideae的一个子集的外显子IV +内含子IV检测“条形码缝隙”的存在(n = 97 Interspecific andn = 53 Intraspecific). Asterisks indicate statistical significance based on the Wilcoxon rank sum test (***p< 0.001)
为了评估K2P值在物种间和物种内部(种内)是否存在差异,我们还计算了同一物种的遗传多样性。由于实际原因,本分析仅限于Tillandsioideae亚科的11种,平均每种5种。如图所示。2(右上窗格),K2P值重叠,因此我们找不到“条形码间隙”。
家族水平遗传多样性及其比较matK
由于高的遗传变异AGT1.Intron IV部分和产生的对准产生的困难,我们分离出外显子IV / Intron IV序列并从外显子v移除了14bp部分以进一步分析。根据注释确定外显子/内含子边界ananas comosus agt1基因模型和肽序列[26.].
评估所获得的序列数据表明AGT1.外显子IV地区具有31%的可变地点的一部分,并且定义信息场所的速率约为23%(表2和额外的文件5:S5)。对于此评估,我们使用K80替代模型,因为它据报道是最准确的AGT1.Bromeliad的标记[23.].
作为matK是最有前途的凤梨科DNA条形码标记[14.,我们也包括在内matK在这个研究中。因此,我们下载了440matK覆盖来自Genbank的这项研究的溴脲属金属覆盖的序列并完全按照我们的作用评估遗传变异AGT1.(附加文件6: S6)。如表所示2,可变位点的比率(24%)和吝啬信息位点的比率(14.5%)matK是相当低的情况下AGT1..
我们接下来比较了遗传多样性AGT1.和matK基于K2P替代模型的序列。的AGT1.比较了不同亚科分类群(家族间)、亚科内种群(属间)和各亚科内种群(同属)间的外显子IV序列比对。如图所示。2相比,matK,AGT1.显示所有分类水平之间的遗传距离值相对高。平均间隙的距离AGT1.但是远高于内部距离,但值是重叠的,表明在许多情况下,序列分歧AGT1.种内标记区域不低于种间标记区域。
AGT1.使用megablast介导的物种识别
为了评估适用性AGT1.作为DNA条形码标记,并将其性能与其进行比较matK,我们生成了一个包含所有IV外显子/ IV内含子的序列数据库AGT1.序列。执行Megablast搜索此溴脲AGT1.数据库中,我们使用了来自多个不同种源的序列。数据库共包含567个不同物种,共检索到41/42个物种,包括Bromeliaceae亚科brochinioideae、Bromelioideae、Hechtioideae、Navioideae、Pitcairnioideae、Puyoideae和Tillandsioideae。如表所示3.和额外的文件2:表S2,正确的物种识别使用AGT1.Exon IV / Intron IV在41个测试病例中可能是可能的。
我们还测试了物种识别率matK使用42种作为对A的查询matK数据库共包含440个序列。如表所示3.和额外的文件2:表S2,在42个案例中有10个被识别成功,因此明显低于AGT1..
接下来,我们将正确的分配测试到物种组,因为它们被定义为Subfamilies Bromelioideae和Tillandsioideae [23.,39.].在42例中的32个中,AGT1.具有最高比特分数的序列被分配给相同物种或物种组中考虑的物种(表3.和额外的文件2:表S2)。
DNA条形码超出物种鉴定的应用
初步研究表明AGT1.序列多样性也适用于解决人口水平多样性,最终有助于调查杂交[23.,25.], 我们获得了AGT1.推定的杂种物种以及潜在父母的序列。
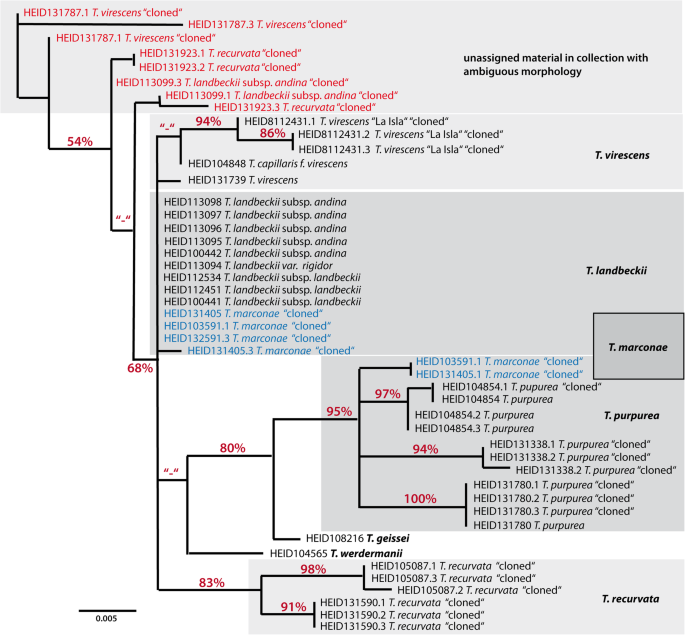
我们选择了蒂尔西亚subg。隔音瘤成员Tillandsia Marconae.为此目的,因为它已被描述为推定的杂种种类Tillandsia Paleacea和Tillandsia紫竹[42.].首先,我们克隆了AGT1.来自潜在父级物种的序列Tillandsia紫竹,T. Virescens.,t . recurvata和T. Landbeckii.这是在秘鲁南部沙漠的共现点收集的。随着AGT1.最大似然树如图所示。3.描述了,T. Virescens.,t . recurvata和T. Landbeckii.已被分配到蒂尔西亚subg。隔音瘤在以前的研究中[39.,40,43.,可以清楚地与Tillandsia紫竹复杂的成员T. purpurea.和T. Marconae..不同的T. Marconae Agt1.等位基因明确分配给T. Landbeckii.或者是T. purpurea.GenePool(相同的等位基因和/或高引导支持)。Tillandsia Marconae.由此,携带两种父母种类的Genopools的等位基因,其也与智利北部介于秘鲁的北部北部的局部流行病T. purpurea.而且很大程度上是智利T. Landbeckii..因此,我们解释T.Marconae.作为推定的杂交T. purpurea.和T. Landbeckii.从subg。隔音瘤作为早些时候提出的父母[42.,43.].
AGT1.外显子IV +内含子IV的各种加入的最大似然树蒂尔西亚亚属隔音瘤成员T. Virescens.,t . recurvata和T. Landbeckii.以及Tillandsia紫竹复杂的成员(包括T. Marconae.)."克隆"表示AGT1.克隆序列以测试AGT1.复制数字和模拟变奏。数字对应海德堡植物园的登录代码(登录详细信息见表)S1).异源多倍体杂交T. Marconae.用蓝色字体表示。红色字体的品种表明形态特征不明确,可能代表杂种来源的附加品种
讨论
遗传多样性和系统发育有效性AGT1.序列
我们获得了579.AGT1.外显子IV + Intron IV序列覆盖各种溴脲亚特征Bromelioideae,Tillandsioideae,Pitcairnioideae,Puyoideae,Brocchinioideae和Hechtioideae。由于较高的变化AGT1.内含子IV序列部分,由于仅对包含的物种系统发育信息的子集可用,我们无法生成全球和家族范围的比对。相反,我们使用CD-HIT-EST软件进行序列聚类分析,获得序列一致性值≥98%的组。我们选择98%的门槛是因为AGT1.以往研究中所报道的进化支之间的序列相似性约为98%以上[23.,25.].与发表的文学的群集进行比较时,我们发现在大多数情况下,群集代表目前接受的分类群(表1).这些群体要么由相同物种的个体组成,属于来自相同属的物种或物种,其中在文献中分配给子属或物种组。
因此,遗传多样性AGT1.序列对当前接受的多基因座来发育相当良好,并且似乎非常适合在整个溴脲中用作DNA条形码标记。然而,由于一些获得的簇包含不适合各组的物种,并且另外,如在非群集内,我们发现我们预期的所有物种 - 虽然存在于数据库中 - 但是使用的很明显AGT1.由于DNA条形码标记需要预防措施,因此不适用于所有物种的安全确定。
在内异性和间隙遗传变异(“条形码间隙”)之间具有清晰分离的大小被认为是良好DNA条形码标记的关键要求[44.,尽管这个问题引起了激烈的讨论[45.].我们评估了AGT1.外显子IVAGT1.外显子IV +内含子IV Kimura 2参数(K2P)值在物种之间和不同的登录之间。如图所示。2(最右侧窗格),我们发现在测试的情况下的内特异性K2P值之间的重叠,因此我们不能辨别不同的“条形码间隙”。然而,由于可以预期这种差距的发生是最真实的,因为它是现实的,或者是否可能只有采样不足的人工制品[46.,我们不认为缺乏“条码间隙”是反对使用的普遍理由AGT1.DNA条码技术。
主要是,我们认为由于基因流动,根据密切相关的物种的进化分歧,我们将不得不应对不足的问题AGT1.序列差异,在某些情况下,DNA条形码通常是具有挑战性的。例如,在基于MegaBLAST的物种鉴定试验中,我们发现41个测试案例中有4个存在歧义(见表)3.).在这些实例中,我们发现一个或多种显示与查询物种相同的比特分数。鉴于这些试验的物种数量相对有限,我们还必须假设将来会将更多的物种添加到数据库中,我们将越多,我们将不得不应对由于有限的有限而削弱此类歧义AGT1.序列分歧。
物种歧视使用AGT1.相对matK
对我们的Bromeliad的梅格瑙搜索所选分类群的绩效AGT1.序列数据库使我们能够正确识别41个被测物种中的22个(53%)。因此,使用AGT1.与先前的研究相比,我们可以增加溴脲对溴脲的DNA条形码的歧视性成功,其标记区域约为两倍,该研究报告了27.6%的识别成功matK,19%的人rbcl.和26%的人trnh-psba.[14.].当三个标记时,同一位作者最多报告了最多44.4%的识别成功rbcl.+matK+trnh-psba.的总和,仍然大大低于AGT1.[14.].
与旨在区分异质且不紧密相关的群体的研究相比,鉴定成功率约为53%,旨在歧视异质且不相关的群体,例如雨林补丁的医疗植物或物种,这成功鉴定了90%的物种[10.,47.].然而,最近发表的一些研究表明,在所有报道的研究中,对近缘物种的DNA条形码比对异质性更强的群体的效率要低得多。例如,使用ITS2可以正确识别76.4%的调查菊科物种[48.].在一项研究中,旨在将其用作属于属的条形码标志物Corydalis.(Papaveraceae)的鉴定成功率为65.2% [49.].另外两项研究调查了核标记ITS2的使用,用于密切相关的条形码姜黄物种(Zingiberaceae),发现46.7%分别确定了73%的物种[50.,51.].
因此,我们的正确物种鉴定率约为53%,在据报道的其他Agiosperm组的范围内,我们得出结论,由于上述原因,通常难以获得更高的分数,特别是在具有众所周知的低遗传变异的基团中如溴eliad [14.].
DNA条形码在Bromaliaceae使用中的适用性AGT1.
鉴于PCR成功率为89%,排序成功率为77%,同时考虑到我们能够恢复全长的事实AGT1.从凤梨科8个亚科中的7个亚科的序列中,我们认为条形码在整个科中普遍可用。在许多其他研究中,报道了标记物的放大效率,如matK和its2 /2在相同的范围内,甚至更低[52.,53.].
Intron IV部分的等位基因变异在大约15%的情况下涉及测序和所需克隆的事实是部分地与我们发现标记的先决条件,使我们能够开发廉价且易于处理的条形码程序。然而,它还有机会扩大其适用性,以扩展到植物收藏中的探讨以及有助于人口研究或检测潜在的F1或早期发电杂种。
为了检验高遗传变异的可能性AGT1.可能足以调查溴脲种类的网状物种的网状演化,我们进行了一个案例研究蒂尔西亚subg。隔音瘤成员Tillandsia Marconae.,因为它被描述为一种潜在的杂种物种Tillandsia Paleacea和Tillandsia紫竹[42.].从我们的结果中(图。3.)我们有良好的迹象表明Tillandsia Marconae.可能是一个混合Tillandsia紫竹和另一个子。隔音瘤成员,作为克隆物种的一种等位基因最接近各个家长的最大似然分析中的等位基因Genepool(图。3.).鉴于包括在内的分类群中的事实Tillandsia Landbeckii.共享栖息地Tillandsia Marconae.和Tillandsia紫竹[54.],我们认为很可能Tillandsia Marconae.通过杂交出现Tillandsia Landbeckii.和Tillandsia紫竹.这些调查结果支持早期的研究,使用其他低核标记来解决同一问题[43.].虽然这些假设需要通过其他分类相关数据和更全面的数据集进一步强调,但我们认为值得注意的是AGT1.序列分辨率足够高,也可以用于研究人口水平,以重建网状进化过程。
结论
一起携带,我们证明了这一点AGT1.允许评分约53%(41例中的22例中的22种)的溴脲基物种测定率,其对应于使用单个标记的先前报告的速率增加,其最高为27.6%和44.4%的精心制备组合三个标记[14.].通过在未来将数据集扩展到基因组规模,可以进一步使用其他规范标记来进一步增加正确的物种识别率。
我们打算开发一种易于处理的协议,可以使用低成本的努力和基本提供的实验室设施在植物园背景下应用,我们考虑使用AGT1.一个可能的解决方案,以协助凤梨属物种的测定,并可能也与其他低遗传变异的植物组。参考文献资料的相应知识数据库系统正在开发中。
方法
植物材料的采集和采样策略
植物材料收集自德国和奥地利的3个植物园(德国海德堡植物园;Alter Botanischer Garten Göttingen,德国;德国柏林植物博物馆;以及来自巴西(Refúgio dos Gravatás, Teresópolis,里约热内卢de Janeiro, Brazil)一位合著者(EMCL)的私人收藏。这些活标本是按照《生物多样性公约》(1993年)第9条所建议的迁地保护准则种植的。我们对取样材料进行充分分类的标准是由至少一位著名凤梨科专家进行可验证的评估和物种收集历史的完整文件(例如“Werner Rauh遗产项目”[55.])。植物材料的出处,确定植物材料的人的名称和关于凭证标本的可用性的信息列于附加文件1S1:表。
我们试图涵盖在最近修订的孕妇亚家族的最近修订中所代表的所有曲线[39.]以及在最近对溴脲的研究中恢复的所有曲线[23.,25.,32.,34.,35.,36.,56.].在亚科伯基替硝基菌,Hechtioideae,Navioideae,Pitcairnioideae和Puyoideae,我们添加了许多不同的物种,因为我们可以从上述任何收藏品中获得。这最终导致477种(3597)和目前接受的76属的51种[1].
我们另外从推定的杂交物种中获得植物材料Tillandsia Marconae.W. Till&Vitek以及许多潜在的家长物种(T. Paleacea.C. PRESL,T. purpurea.Ruiz&Pav。,T. Virescens.(Ruiz&Pav。)L. B. SM。,t . recurvata(L.)L.和T. Landbeckii.菲尔。)从秘鲁沙漠南部的遗址收集[42.].
DNA提取、PCR扩增、DNA测序、质粒克隆
根据制造商的说明,使用Qiagen (Venlo, NL) DNeasy Plant Mini Kit从新鲜或干燥的叶子材料中提取DNA。PCR扩增AGT1.marker region was performed using MyTaq DNA-polymerase (Bioline, London, UK) using 10–20 ng DNA template, 5x MyTaq reaction buffer (5 mM dNTPs and 15 mM MgCl), 10 pmol of each primer (AGT1_SP6_Fw: 5‘-ATTTAGGTGACACTATAGATTGATGTCGCATTAACCGGC-3‘ and AGT1_M13_Rev: 5’-AACAGCTATGACCATGGCAGTTCTTCAGTCCCCATG-3’). The cycling conditions were the following: 95 °C 3 min. [30 cycles: 95 °C 30 s.; 56 °C 20 s.; 72 °C 20 s.] 72 °C 5 min. Before designing bromeliad specific primers, we also used the canonicalAGT1.之前研究中使用的引物[18.,23.].PCR扩增和测序matK引物组合MatK 5F: 5 ' -ATACCCTGTTCTGACCATATTG-3 '和trnK2r 5 ' -AACTAGTCGGATGGAGTAG-3 '。内部排序matK使用底漆Tomatk 480f 5'-catctkgaaatttggtc-3'完成了[57.].
在测序之前,根据供应商的建议,用外切核酸酶I (New England Biolabs, Ipswich, MA, USA)和虾碱性磷酸酶(Thermo Fischer Scientific, Waltham, MA, USA)联合处理PCR反应。
使用标准测序引物(SP6_FW 5'-)使用标准测序引物(SP6_FW 5'-)使用ABI 3730平台(Senckenberg生物多样性和气候研究中心)的ABI 3730平台(Applied Biosystems,CA,USA)设备进行了Sanger测序(Senckenberg生物多样性和气候研究中心)进行atttaggtgacactatag-3'和m13_rev 5'-aacagctatgaccatg-3')。
AGT1.根据供应商的建议,使用CloneJET PCR克隆试剂盒(Thermo Fischer Scientific, Waltham, MA, USA)进行PCR片段克隆。然后在Eurofins Genomics (Eurofins Scientific, Luxemburg, LU)使用通用质粒测序引物M13正向和SP6反向对每个克隆试验进行4到5个质粒测序。
收购matK来自Genbank的序列
482.matK从Genbank下载序列。将所有序列对齐并修剪至782bp的长度。可以在附加文件中找到包含所有物种的GenBank登录号的列表1S1:表。
数据分析
对序列数据进行编辑和分析诅咒软件(BioMatters,奥克兰,NZ,版本11.0.5)[58.].序列比对采用诅咒实施的Mafft.序列对齐工具(版本7.388)[59.].序列比对进一步分析Mega7.软件包(60].利用Kimura 2参数距离(K2P)分析外显子IV序列比对Mega7.,采用Wilcoxon秩和检验。
AGT1.使用CD-HIT-EST软件进行序列数据库聚类分析[28.,61].最终序列标识截止设置为0.98,所有其他参数都设置为默认值(附加文件2:表S2)。
使用爆炸介导的物种鉴定使用诅咒实现自定义BLAST函数[59.].所有快餐序列AGT1.外显子IV被修剪至264 bp,内含子IV部分在外显子V边界被截断。使用MegaBLAST算法进行两两比较,使用以下设置进行数据库搜索:评分(Match Mismatch): 1-2;缺口成本(Open Extend):线性;最大价值:10;文字大小:28。输出是通过增加每一次命中的Bit-Score来排序的。我们认为,只有当最高位分对应于与查询相同的物种,而所有其他物种确实具有完全不同的较低位分时,识别才会成功(附加文件)2:表S2)。
使用Paup生成最大似然树* [62].对线总长度为492 bp,“间隙”和缺失数据未计算在内(图2)。3.和额外的文件3.:S3)。提供了从1000复制的引导支持值(> 50%)。
可用性数据和材料
本研究使用的数据集包括在手稿和补充材料中。本研究获得的所有DNA序列均提交至GenBank,登录号列于附加文件1S1:表。
改变历史记录
2020年12月30日
对本文的修订已发布,并可通过原始文章访问。
缩写
- BP:
-
底对
- Ibol:
-
国际条码生命
- K2P:
-
Kimura 2-参数
- LCNG:
-
low-copy核基因
- 聚合酶链反应:
-
聚合酶链反应
- subg:
-
亚属
参考文献
- 1。
Gouda EJ,屠夫D,Gouda CS。Bromeliadds的百科全书,版本4.乌得勒支:大学植物园;2019年。http://bromeliad.nl/encyclopedia/(2017年1月24日)。
- 2。
Givnish TJ,Barfuss MHJ,Van EE B,Riina R,Schulte K,豪华R等。Bromeliaceae的系统发育,自适应辐射和历史生物地理学:来自八个轨迹血浆系统的见解。我是J机器人。2011; 98:872-95。
- 3.
Givnish TJ, Barfuss MHJ, Van Ee B, Riina R, Schulte K, Horres R,等。凤梨科植物的适应辐射、相关和偶然进化及净物种多样化。分子系统学进展。2014;71:55-78。
- 4.
张军,刘军,明锐。菠萝CAM植物基因组分析。J Exp Bot. 2014; 65:3395-404。
- 5。
Ming R.菠萝基因组:单极管和凸轮光合作用的参考。趋势类型。2016; 32:690-6。
- 6.
Gardens4Science - Der Online-SammlungskatalogBotanischerGärten。http://gardens4science.biocase.org/.
- 7.
Ratnasingham S, Hebert PDN。基于dna的所有动物物种注册:条形码索引号(BIN)系统。PLoS ONE。8 (7): e66213。https://doi.org/10.1371/journal.pone.0066213.
- 8.
Fazekas AJ, Kesanakurti PR, Burgess KS, Percy DM, Graham SW, Barrett SCH等。使用DNA条形码标记,植物物种是否天生就比动物物种更难区分?摩尔生态资源。2009;9:130-9。
- 9。
通过DNA条形码进行生物鉴定。中国生物医学工程学报。2003;27(4):427 - 427。
- 10。
陈胜,姚洪,韩建军,刘超,宋建军,石磊,等。ITS2区域作为一种新的药用植物DNA条形码的验证。5 (1): PLoS ONE e8613。https://doi.org/10.1371/journal.pone.0008613.
- 11.
Hollingsworth Pm,Forrest Ll,Spouge JL,Hajibabaei M,Ratnasingham S,Van der Bank M等人。土地植物的DNA条形码。PROC NATL ACAD SCI。2009; 106:12794-7。
- 12.
克雷斯WJ。植物DNA条形码:今天和未来的应用。J Syst Evol. 2017;
- 13。
克拉克兰人kj,zimmer e e,Weigt La,Janzen DH。使用DNA条形码识别开花植物。PROC NATL ACAD SCI。2005; 102:8369-74。
- 14。
Maia VH,Da Mata CS,Franco Lo,Cardoso Ma,Cardoso Srs,血换为等等。DNA条形码Bromeliaceae:成就和陷阱。Plos一个7(1):E29877。https://doi.org/10.1371/journal.pone.0029877..
- 15.
Liepman啊。丙氨酸氨基转移酶同源物催化谷氨酸:拟南芥过氧化物酶体中的乙醛酸氨基转移酶反应。植物杂志。2003;131:215-27。
- 16.
Liepman啊,奥尔森LJ。过氧化合物喹啉丙氨酸:乙醛酸氨基氨基转移酶(AGT1)是拟南芥中具有多个底物的光致血管酶。工厂J. 2001; 25:487-98。
- 17.
张强,李志强,李志强,张志强。拟南芥丝氨酸氨基转移酶AGT1基因的克隆及表达分析。植物化学。2013;85:30-5。
- 18.
李敏,王文德,毕索利,等。COS基因作为普遍可扩增标记用于近缘植物物种系统发育重建的发展。支序分类学。2008;24:727-45。
- 19。
Babineau M,Gagnon E,Bruneau A.在紧密相关的属和含有CaeSalpinioid豆类中的19个低拷贝核基因的系统发育效用。南非J机器人。2013; 89:94-105。
- 20。
李志刚,李志刚,李志刚,等。缬草科(迭香科)植物间亲缘关系的研究:来自低拷贝核区的新见解和新假说。系统机器人。2015;40:327-35。
- 21。
Blanco-Pastor JL, Vargas P, Pfeil BE。结合模拟揭示了地中海Linaria的杂交和不完全谱系分选。PLoS ONE。7 (6): e39089。https://doi.org/10.1371/journal.pone.0039089.
- 22。
在第四纪时期,同源特征决定了地中海植物的两种进化策略:低分化和范围扩展与Linaria的地理形成。摩尔生态。2013;22:5651 - 68。
- 23。
凤梨科凤梨亚科和凤梨属的新分类。Phytotaxa。2017;318:1 - 88。
- 24。
Naumann J,Symmank L,Samain Ms,MüllerKF,Neinhuis C,Depamphilis CW,等。追逐野兔 - 在物种富集群辣椒(Piperaceae)内和低于物种水平的核唯一拷贝基因区域的系统发育效用。BMC EVOL BIOL。2011; 11:357。
- 25。
Matuszak-Renger S,Paule J,Heller S,LEME EMC,Steinbeiss GM,Barfuss MHJ等人。基于核,塑性和AFLP数据的厌氧和相关分类群(Bromelioideae,Bromeliaceae)的系统发育关系。植物系统中的Evol。2018; 304:841-51。https://doi.org/10.1007/S00606-018-1514-3.
- 26。
王志强,王志强,王志强,等。菠萝基因组与CAM光合作用的进化。Nat麝猫。2015;47:1435-42。https://doi.org/10.1038/ng.3435.
- 27。
Goodstein DM,Shu S,Howson R,Neupane R,Hayes Rd,Fazo J等人。植物血统:绿色植物基因组学的比较平台。核酸RES。2012; 40:D1178-86。
- 28。
李伟,高德泽。Cd-hit:一种用于聚类和比较大组蛋白质或核苷酸序列的快速程序。生物信息学。2006;22:1658-9。
- 29.
Heller S,Leme Emc,Schulte K,Benko-Iseppon Am,Zizka G.在Aechmea Alliance中阐明了系统发育关系:Portea和Gravisia综合体的AFLP分析(Bromeliaceae,Bromelioideae)。SYST BOT。2015; 40:716-25。
- 30.
Santos-Silva F,Venda Akl,Hallbritter HM,LEME EMC,Mantovani A,Forzza RC。嵌套在混乱:关于“尼凡综合体”关系的见解和Neoregelia(Bromelioideae-Bromeliaceae)的进化史。布里塔尼亚。2017; 69:133-47。
- 31。
凤梨科植物的分类、系统发育、进化和生物地理学。(论文)。歌德大学,法兰克福。德国法兰克福的歌德大学;2018.
- 32。
Aguirre-Santoro J,Stevenson D,Michelangeli F. Ronnbergia联盟(Bromeliaceae,Bromelioideae)的分子系统和关于其形态学的见解。mol phylocyet evol。2016; 100:1-20。
- 33。
Schulte K,Barfuss MHJ,Zizka G.从核和塑体DNA Loci推断出溴脲(Bromeliaceae)的系统发育揭示了亚家族内坦克习惯的演变。mol phylocyet evol。2009; 51:327-39。https://doi.org/10.1016/j.ympev.2009.02.003.
- 34。
埃文斯TM,Jabaily Rs,De Faria APG,De Sousa Ldof,Wendt T,棕色GK。基于叶绿体DNA序列数据的Bromeliaceae亚家族溴脲的系统发育关系。SYST BOT。2015; 40:116-28。
- 35。
德索萨LDOF, Wendt T, Brown GK, Tuthill de, Evans TM。基于形态和叶绿体DNA序列的凤梨科凤梨亚科单系和系统发育关系。系统机器人。2007;32:264 - 70。
- 36。
Aguirre-Santoro J. ronnbergia联盟的分类学(Bromeliaceae:Bromelioideae):Ronnbergia和Ronnbergia的新组合,概要和新的环节。植物系统中的Evol。2017; 303:615-40。
- 37。
Silvestro D,Zizka G,Schulte K.解开关键创新对溴脲(Bromeliaceae)多样化的影响。进化(n y)。2014; 68:163-75。
- 38。
sass c,specht cd。强调艾森菊(Bromeliaceae)强调核心染色体的系统发育估计。mol phylocyet evol。2010; 55:559-71。https://doi.org/10.1016/j.ympev.2010.01.005.
- 39。
Barfuss MHJ,直到W,LEME EMC,PinzónJP,Manzanares JM,Halbritter H,等。Bromeliaceae Subfam的分类系统修正。基于多基因座DNA序列系统发育和形态学的Tillandsioideae。Phytotaxa。2016; 279:1-97。
- 40。
Granados Mendoza C,Granados-Aguilar X,DonadíoS,Salazar Ga,Flores-Cruz M,Hágsatere等。两种高度多种谱系的Tirandsia(Bromeliaceae)的地理结构。植物学。2017; 95:641-51。
- 41。
Schütz N, Krapp F, Wagner N, Weising K. Pitcairnioideae s.s.(凤梨科)系统发育学:来自核和质体DNA序列数据的证据。Bot J Linn Soc. 2016; 181:323-42。
- 42。
Till W, Vitek E. Tillandisa marconae - eine neune Art aus der peruanischen Küstenwüste。acta botanica sinica(云南植物学报),1985;
- 43。
Barfuss MHJ。凤梨科的分子研究:质体和核DNA标记在系统发育、生物地理和特征进化中的意义,重点介绍凤梨科的一个新分类。维也纳大学博士论文;2012.
- 44。
Čandek K . Kuntner M. DNA条形码差距:基于形态学和地理尺度的可靠物种鉴定。摩尔生态资源。2015;15:268-77。
- 45。
董开,徐c,李c,sun j,zuo y,shi s等人。YCF1,土地植物最有前途的塑植DNA条码。SCI REP。2015; 5:8348。
- 46。
Wiemers M, Fiedler K. DNA条形码缺口存在吗?-以蓝蝴蝶为例(鳞翅目:灰蝶科)。黑旋风。2007;4:8:1-16。
- 47。
等。关键词:神经网络,神经网络,神经网络DNA条形码在识别非洲雨林树木方面有多有效?PLoS ONE。8 (4): e54921。https://doi.org/10.1371/journal.pone.0054921.
- 48。
高T,姚H,宋j,朱y,刘开,陈S,gao t。评估使用候选DNA条形码的可行性鉴别大型钙科家族的种类。BMC EVOL BIOL。2016; 10:324。
- 49。
任敏芬,王永伟,徐志超,李勇,辛天宇,周建国,等。罂粟科最复杂属紫堇属的DNA条形码研究。生态另一个星球。2019;9:1934-45。
- 50。
陈家,赵家,埃里克森dl,x j,kress wj。从缅甸和中国测试DNA条形码与缅甸和中国密切相关的姜黄素(Zingiberaceae)。Mol Ecol Resour。2015; 15:337-48。
- 51。
施立尔克,张俊,韩吉,宋杰,姚h,朱yj等。测试所提出的DNA条形码的潜力,用于Zingiberaceae的物种鉴定。J Syst Evol。2011; 49:261-6。
- 52。
Kress WJ, Erickson DL, Swenson NG, Thompson J, Uriarte M, Zimmerman JK。在波多黎各森林动态图中使用DNA条形码建立热带树木群落系统发育的进展。《公共科学图书馆•综合》。2010;5:1-8。
- 53。
Hollingsworth ML, Andra Clark A, Forrest LL, Richardson J, Pennington RT, Long DG,等。植物条形码基因座的选择:在3个不同的陆生植物类群中,用种水平抽样法评价7个候选基因座。生态学报2009;9:439-57。
- 54。
Zizka G,Schmidt M,Schulte K,Novoa P,Pinto R,Königk.智利溴脲:保护地位的多样性,分布和评估。生物方向保守。2009; 18:2449-71。
- 55。
Koch MA, Schröder CN, Kiefer M, Sack P. 20世纪植物生物多样性的宝库:海德堡植物园和植物标本馆的Werner Rauh遗产项目。acta botanica sinica(云南植物研究),2013;29(4):493 - 497。
- 56。
Almeida VR,Ferreira da Costa A,Mantovani A,Gonçalves-Esteves V,De Oliveira Rdc,Forzza RC。毒素(Bromeliaceae,Bromelioideae)的形态学系统。SYST BOT。2009; 34:660-72。
- 57。
杨志强,李志强,李志强,等。凤梨亚科分子系统发育及其对分子生物学和CAM进化的影响。Senckengergiana Biologica。2005;85(1):113 - 25所示。
- 58。
等。Geneious basic:一个集成的、可扩展的用于序列数据组织和分析的桌面软件平台。生物信息学。2012;28:1647-9。
- 59。
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30:772-80。
- 60。
分子进化遗传学分析第7版。0用于更大的数据集简短通信。Mol Biol Evol. 2016; 33:1870-4。
- 61。
黄颖,牛斌,高艳,付玲,李伟。CD-HIT套件:用于生物序列聚类和比较的web服务器。生物信息学,2010,26(5):680 - 82。https://doi.org/10.1093/bioinformatics/btq003.
- 62。
Swofford DL。使用规定(*和其他方法)的系统发育分析。J Mol Evol。2002年。
致谢
感谢Andreas Franzke和Simone Elfner(德国Heidelberg大学),Barbara Knickmann(Botanischer Garten derUniversitätWien,奥地利)以及Michael Schwerdtfeger和Leonard Georg(德国Göttingen大学植物园)用于植物抽样帮助;Heike Kappes(Grunelius-Möllgaard实验室,法兰克福,德国)和弗兰克拉佩(歌德大学,德国法兰克福,德国)为实验室提供帮助。
资金
这项研究是由德国联邦教育和研究部(BMBF)资助的,由授予Marcus a. Koch和Georg Zizka的一笔赠款(Evo-BoGa)。我们感谢德国科学、研究和艺术部(Baden-Württemberg)以及Ruprecht-Karls-Universität海德堡(Ruprecht-Karls-Universität Heidelberg)在开放获取出版资助项目中提供的资金支持。
作者信息
隶属关系
贡献
上海、广州、JP、MAK、FB是本次研究的构思和设计。FB, SH, NC, JP, AL, AN, MK和MAK对数据的获取、分析和解释做出了贡献。EMCL, WT, MHJB提供了资料并提供了分类方法。FB撰写论文,JP, GZ, MAK, MHJB和CL贡献。所有作者阅读,评论和批准的最终手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
本文已更新以添加正确版本的其他文件1。
补充信息
附加文件1。
supplaryary_table_s1_plant_material。
附加文件2。
Supplementary_Table_S2_CD_Hit_MegaBLAST。
附加文件3。
supplaryary_material_s3_agt1_alignment_tillandsia_ml_tree。
附加文件4。
supplareary_material_s4_agt1_cloning。
附加文件5。
Supplementary_Material_S5_Agt1_exonIV_alignment。
附加文件6。
Supplementary_Material_S6_matK_alignment。
附加文件7。
Supplementary_Material_S7_Agt1_genmodels_Acomosus_Athaliana。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Bratzel,F.,Heller,S.,Cyrannek,N。等等。低拷贝核基因AGT1.作为Bromeliaceae的新型DNA条形码标志物。BMC植物BIOL.20,111(2020)。https://doi.org/10.1186/s12870-020-2326-5
已收到:
接受:
发表:
关键词
- DNA条形码
- 低拷贝核基因
- 植物集合
- 凤梨