抽象的
背景
芒果,Mangifera indica.L.是一种重要的热带水果作物,因其果实甜而芳香。过去对该物种的改良主要依赖于来自印度次大陆1000多个品种的偶然幼苗,这些品种在果实大小、产量、生物和非生物胁迫抗性以及果实质量等性状上存在较大差异。从历史上看,芒果一直是一种分子信息非常有限的孤儿作物。直到最近,基于分子和基因组学的分析才使得建立连锁图谱、转录组和对大量集合进行多样性分析成为可能。此外,基因组和表型信息的联合分析有助于提高芒果育种效率。
结果
该研究测序,De Novo组装,分析,并向单枚乳头芒果品种“Tommy Atkins”的基因组。基因组序列草案在“Tommy Atkins”的高分子量DNA上使用NRGENE DE-Novo魔法产生,补充了10x基因组学长读取测序以改善初始组件。“Tommy Atkins”X'Nensington Pride'之间的混合群体用于生成分阶段的单倍型染色体和高度解决的相位的SNP地图。最终的“Tommy Atkins”基因组组装是一种共有序列,包括代表芒果20染色体的20个假调子,包括〜439 MB单倍体芒果基因组的〜86%。使用“Tommy Atkins”X'Nensington Pride'绘图人口识别略微测序〜3.3米SNP。重复掩蔽鉴定26,616个基因,中值3348 bp。整个基因组重复分析显示了与之共享的祖先的65 Mya多倍化事件青春痘occidentale..在LG4和LG7上的两个区域包含28个候选基因,这两个区域与定位群体中重要的果实大小特征相关。
结论
完整的“汤米·阿特金斯”芒果基因组的获得将有助于研究芒果遗传学的全球计划。
背景
芒果是在热带和亚热带区域的103个国家种植的重要果实作物。常见的芒果通常是一个大,热带和常绿树,直立,可以伸展致密的树冠,如果没有修剪,在一些气候中可以达到30米。据估计,芒果产量超过5000万公吨(MT),从5680万公顷的面积超过5000万吨(MT)[1,2].印度是迄今为止最大的芒果生产国,占世界生产的41.6%(18吨),随后中国有10%(4.5公吨)。生产的大部分产量并在本地产生,由于原产国的局部消费量高,仅出口大约9.5吨,并且果实的高度易腐烂性质[1,3.].
芒果(Mangifera indica.属于木犀科。根据形态特征可以认为是从45[4]至69 [5]物种Mangifera主要在热带亚洲起源的属,在麦利西亚西部发现最多的多样性地区[6]. 普通芒果,m .籼至少驯养了4000只 多年前,又从一个起源于阿萨姆河谷靠近缅甸西部边界的印支地区进一步发展起来,在第四纪时期遍布印度次大陆[6,7,8].另外26种也有可食用的水果,包括m . altissima,m . caesia,M. foetida.,M克芒,M. Laurina,m . odorata,M帕江和m . pentandra在不同的东南亚社区都是传统的消费方式[9,10,11].
虽然芒果品种的驯化和选择已经发生成数千年,但芒果的系统繁殖相对较近,与许多温带树果作物相比。由于长幼年,聚氧化物和非常低的果实保留,系统芒果繁殖是长期努力(长达25岁),从而降低育种效率并向繁殖生成周期增加时间[12].因此,对芒果遗传学和特质遗传性的一般性理解受到限制。最近,系统育种计划旨在开发各种生产,消费和可运输性的品种,更适合国家和国际市场。养殖芒果具有改进的特征,如降低的树脂活力,常规高产率,疾病耐受性,长的保质期,最佳的水果尺寸,形状,颜色和高饮食品质都是提高生产效率和消费者需求的主要兴趣[12,13].
“汤米阿特金斯”来自一个相对较新开发的品种群,起源于美国佛罗里达州,作为20世纪早期的偶然幼苗[14,15,16]. 它们的成功部分归功于它们相对较高的产量、较大的果实大小、强烈的腮红颜色、较低的活力以及在热带和亚热带地区的适应性。这个群体起源于1910年从印度进口到美国的高产单胚品种“Mulgoba”。从“Mulgoba”中选择的一个早期幼苗被命名为“Haden”,它本身产生了主导国际贸易的单胚品种“Keitt”、“Kent”和“Tommy Atkins”。另一个品种“肯辛顿骄傲”在过去的一个世纪里一直主导着澳大利亚的生产,直到现在才慢慢被新品种所取代,这些新品种通常在其系谱中有“肯辛顿骄傲”。“肯辛顿骄傲”在1885年至1889年间在丹尼森港(现在的鲍恩)出现之前的澳大利亚起源尚不清楚肯辛顿骄傲'有一个独特的风味和香气不常见的其他印度或佛罗里达品种。它的形状和红色腮红表明它起源于印度次大陆,而它的多胚性表明它起源于东南亚。有人认为,“肯辛顿骄傲”可能是印度和东南亚血统的杂交种[17].
早期驯化的芒果品种的果实大小通常较小,可以看到在较老的芒果品种m .籼在印度东北部、安达曼群岛和整个东南亚生长的变种和其他物种[8].果实大小一直是芒果育种的优先目标,随着时间的推移,选择已经使受欢迎的交易芒果品种的平均大小增加到400克[12,18,19,20,21].据估计,果实大小具有较高的遗传力[22].除了果实大小、硬度、颜色、香气产生和应激反应是这种跃变果实的品质特征,需要在基因组水平上进行研究以提高芒果果实的品质。保质期短、易受冷害和采后病害是影响芒果销售的主要挑战[23].质地软化是消费者接受的一个主要品质属性,它与细胞壁多糖及其降解酶有关[24].在与芒果软化(广泛的果胶降解)相关的细胞壁降解酶中,有外聚半乳糖醛酸酶,果胶甲基酯酶,(1-4)-葡聚糖酶和-半乳糖苷酶[25].
最近的研究改进了我们关于芒果的遗传和基因组信息,包括对种质资源的分子分析[26,27,28,29,30]、单核苷酸多态性基因分型分析[31,32],所有20个连锁组的遗传图谱[13,33,34,转录组[27,28,29,32,35,36,37,38,39,40].所有的遗传和基因组信息都支持芒果是一个拥有40条染色体的二倍体,这意味着单倍体的染色体数目为20条。这与20个连锁组的存在一致。单倍体基因组大小估计为~ 439 Mb [41].在缺乏纯合子品种的情况下,“汤米·阿特金斯”芒果品种被选中进行基因组测序,因为它是种植最广泛、国际贸易最广泛的品种之一。
一个高质量的基因组可以极大地帮助芒果育种家。重要园艺性状的候选基因同源物可以通过组合“组学”方法鉴定,包括与其他树木作物的比较基因组学。候选基因的单核苷酸多态性(SNP)标记可用于苗期筛选杂种,以确定标记基因型与园艺性状之间的关联。这将提高育种效率,并通过丢弃不携带有利等位基因的劣质幼苗来降低选择成本。本报告描述了初级商业芒果品种‘Tommy Atkins’染色体水平基因组的建立。通过对果实大小候选基因的鉴定,证明了基因组的实用性。该基因组将成为全球研究界推进芒果遗传育种研究的资源。
结果
基因组测序、组装和注释
的m .籼用NRGene de - novo Magic、10X Genomics长读测序和分阶段SNP图谱的组合,对“汤米·阿特金斯”芒果基因组进行了测序和从头组装。结果是在571个支架中得到了一个单倍体序列一致组装TA4,总序列长度为377 Mb(表1)1).87%的装配被锚定在20个平均长度为16.43 Mb的二倍体假分子上(补充表)1).TA4组件占芒果基因组估计大小的86% (~ 439 Mb) [41].GC含量为33.65%。
利用两种不同的方法评估了组装所捕获的基因空间的完整性。首先,BUSCO分析表明,97.4%的核心基因被完全捕获。BUSCO分析还发现12.6%的重复基因和0.4%的片段基因(补充表2).其次,对来自不同组织和条件下的3个公共RNA-Seq数据集(SRP066658、SRP179820、SRP192932)的映射率分别为92.57、87.33和78.95%。
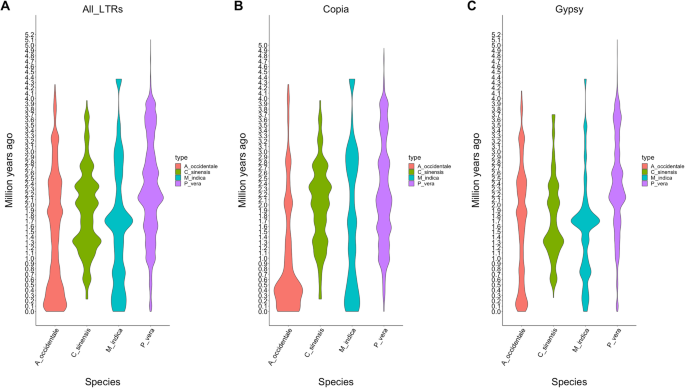
总基因组的181个(3748%)为重复序列 英国石油公司)。转座因子(TE)Ⅰ类是最丰富的成分,占基因组的34%(128898479) 英国石油公司)。由于在植物基因组中很常见,LTR/Copia和LTR/Gypsy是最丰富的TEⅠ类,分别占13%和14%。TE II类占总成的12%(45931312 bp)和Helitrons占总成的2%(6544060) bp)(补充表3.).LTR/Copia和LTR/Gypsy的插入年龄显示了两组不同的扩张年龄。对于LTR/Copia,在0.1和3.0 MYA左右有两个适度的扩张,而对于LTR/Copia,在1.7 MYA左右有一个强劲的扩张(补充图1).
预测了26,616个基因模型和30,344个转录本m .籼使用MAKER-P对TA4基因组进行组装(Table2).利用BUSCO胚胎植物10数据集对基因注释的完整性进行了评价。94.6%的BUSCO基因在m .籼基因模型结构注释。重复基因的比例略高于基因组序列,为17.7%,BUSCO片段的比例也略高于基因组序列,为1.7%(补充表)2).89.0%的转录本至少有一个功能注释,这些功能来自ARAPORT11、SwissProt和NCBI NR数据库的BLAST同源性搜索或InterproScan的蛋白结构域搜索。最高百分比的蛋白质结构域包括PPR重复,亮氨酸丰富重复和蛋白激酶结构域(补充表)4).
芒果基因组的进化分析
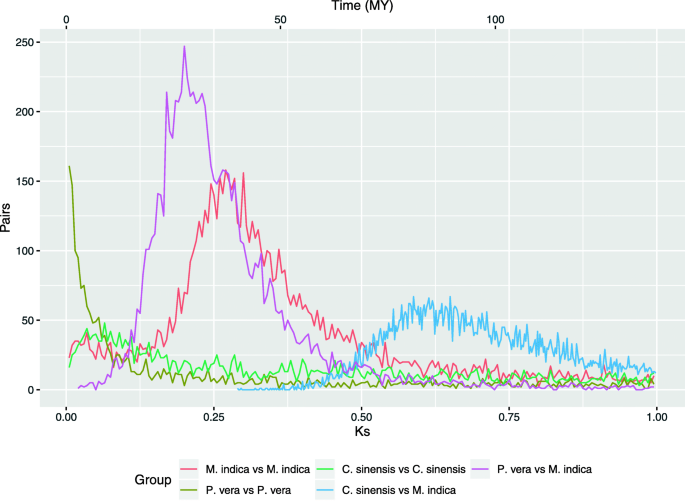
为了阐明芒果的部分进化历史,我们将芒果的基因组与自身和其他选定的基因组进行了比较。素类以无患子目(order Sapindales)为外类群种(不同科,相同目),以81 MYA [42]. 编码序列的Ks分布分析表明,芒果基因组有一个65mya(Ks)的全基因组复制(WGD) = 0.270). 不与共享开心果与其他系统发育研究结果一致(图1)。1) [42].有可能P维拉共享相同的WGD事件m .籼,但它被基因组组件期间的同源物基因崩溃所掩盖。替代方案是开心果祖先与共同的祖先分歧Mangifera在WGD事件前几百万年。这种替代方案是一致的一些核型在其他成员的金鱼草科。一些系统发育研究将其分为A1、A2和B三类[43].属像Mangifera,Anacardidium, 和Semecarpus在组A1中,具有20对或更多对染色体(2n = 2x = 40 [44],2牛 = 2倍 = 42 [45,和2n = 4x = 60 [46),分别)。属像开心果,石斛, 和Cotinus.A2组中有15对染色体(2n = 2x = 30) [47,48,49].在这种情况下,拥有15对染色体的A组祖先来自两个祖先物种A1和A2。A2保持相同数量的染色体,而A1有一个复制染色体到30对染色体的WGD事件。在诸如的属中Mangifera和Anacardidium,他们分别通过减少染色体的数量,直到二十二十二对产生。
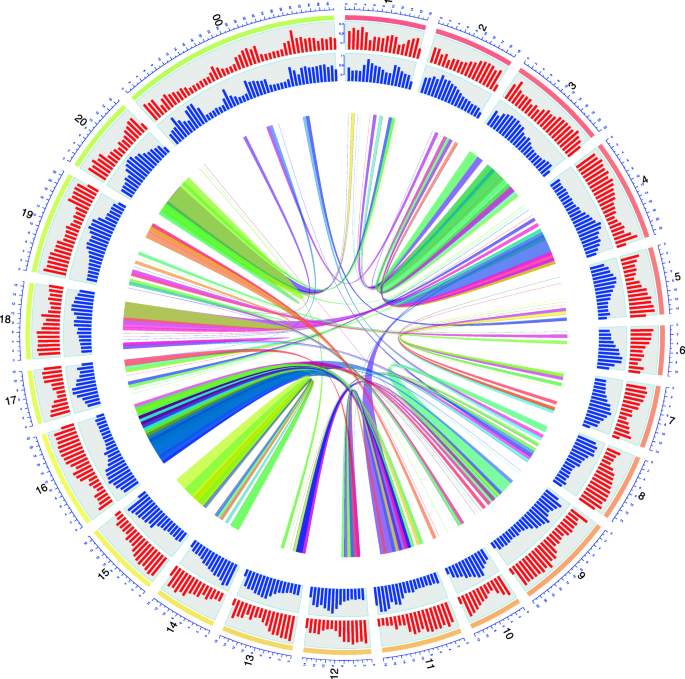
对来自WGD的同源基因对的分析表明,芒果染色体之间具有共线性(图。2).例如,14号染色体和15号染色体共享518对基因,11号染色体和16号染色体共享509对基因5).在为WGD分析的三种物种中也比较了重复景观。这三种物种显示出类似的重复元素水平范围C. Sinensis.(基因组组装的39%)到P维拉(基因组组装的66%)。LTR/Copia的含量比LTR/Gypsy的含量要小,但在芒果基因组中它们的含量比较接近(分别为13和14%)C. Sinensis.(10和14%)和P维拉(20和32%)。第II级TE含量显着高m .籼(12%)与其他三种物种相比(9%C. Sinensis.和P维拉)(补充表2).虽然这三个物种的LTR谱是相似的(补充图1),后5个MYA的插入时间不同。m .籼和C. Sinensis.LTR/Gypsy1.7mya和1.3mya最近分别出现了强劲的扩张(图。3.).
“汤米·阿特金斯”和“肯辛顿骄傲”的比较
来自芒果品种“Tommy Atkins”(单胚)和“Kensington Pride”(多胚)的序列被绘制到TA4二倍体一致组装上,利用snpEff识别变异和效应。对于“Tommy Atkins”,共鉴定了5,394,956个变异,包括3,946,854个SNPs, 1,051,633个MNPs(多核苷酸多态性),138,977个插入,146,654个缺失和110,838个混合变异。“汤米·阿特金斯”的平均变异率是每69 bp一个变异。类似地,“肯辛顿骄傲”的变异数是通过将“肯辛顿骄傲”芒果品种映射到TA4组装上来确定的。变异总数为9,030,142个,包括6,291,666个SNPs、568,959个MNPs、223,249个插入、245,632个缺失和700,636个混合变异。“肯辛顿骄傲”的变异数是“汤米·阿特金斯”的1.67倍,平均变异率为每41 bp一个变异。根据影响、函数类、类型和区域以及完整的snpEff摘要所产生的不同影响在补充数据中报告S1汤米·阿特金斯和补充数据S2“肯辛顿的骄傲”。
芒果基因家族分析
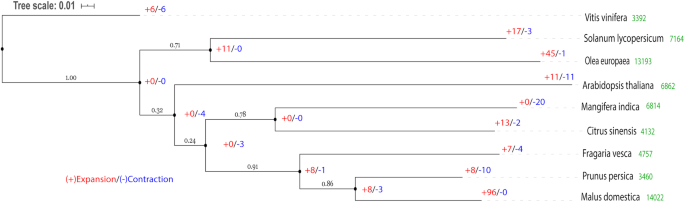
另外八种植物的蛋白质组用于与芒果的比较研究,包括拟南芥,四种跃期果实(Solanum lycopersicum,海棠,碧桃)和四种非跃期果实(葡萄,素类,Fragaria Vesca.,Olea Europaea). 使用Orthofinder共鉴定出17382个正交组,包含219431个基因(占总输入的75.3%)[50].用包含所有种的正群来推断种树,其中914个完全由单拷贝基因组成。基因复制事件以种树末端分支的数目来表示(图1)。4). 显示的是两个副本都保留在至少50%的后代物种的重复。芒果中没有显著扩展的基因家族,但与果实成熟有关的基因家族,如植物转化酶/果胶甲基酯酶抑制剂显著收缩(表1)3.).
利用芒果基因组研究单核苷酸多态性与果实重量的关系。
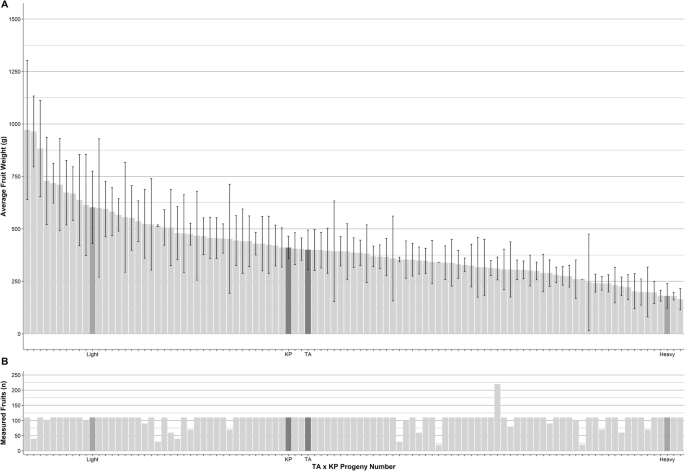
基因组的可用性使重要性状的遗传解剖成为可能。而“汤米·阿特金斯”和“肯辛顿骄傲”在许多相似的特征上是相似的4),其子代的果实质量变异较大。5和6).杂交群体“Tommy Atkins”和“Kensington Pride”亲本的平均果实重分别为400 g和410 g,杂交后代的平均果实重分别为165 g和971 g。据估计,单核苷酸多态性与果实重量的关联确定了两个QTL区域P- 值0.001。在LG4上从BP位置8,275,233至8,495,231中鉴定一个区域,其中一个ON LG7,来自BP位置3,831,615至3,914,160。
‘Tommy Atkins’x‘Kensington Pride’子代的果实重量表型数据。一个“汤米阿特金斯”,“肯辛顿骄傲”和99个混合人口后果的平均水果重量。深灰色酒吧代表了两个父母的汤米atkins'和'肯辛顿骄傲'。中灰色条表示图1中所示的光和果实。6.误差栏代表标准偏差。b用以计算各亲代和后代平均果重的总果数
果实质量QTL区基因
使用QTL区域的位置边界,在LG4区和LG7 QTL区域中的七个基因中发现总共28个基因(表5).报告了该区域中的所有基因,无需基于它们对水果重量的潜在影响而过滤。
单倍型与果实重量的关联
利用“Tommy Atkins”x“Kensington Pride”的阶段性遗传图谱,确定了每个贡献与果实重量相关的SNP次要等位基因的亲本的单倍型。“Tommy Atkins”x“Kensington Pride”群体中每个个体的所有snp的基因型是由阶段性遗传图谱和单倍型数据推断出来的。PLINK分析发现了QTL区域的snp及其相关的单倍型。结果表明,QTL区各个体可能存在TA1KP1、TA1KP2、TA2KP1、TA2KP2等4个双倍型(单倍型)。大多数个体在QTL区域只观察到2个单倍型(分别来自“Tommy Atkins”和“Kensington Pride”)。在该群体的LG4区和LG7区分别发现了3个可能的重组实例,但这些个体没有被包含在该区域各自的双倍型分析中。以双倍型为组的单果重数据方差分析显示LG4和LG7组间差异显著。事后Tukey多重比较均数检验显示,LG 4的TA2KP1和TA2KP2双倍型间差异显著,LG 7的配对组间差异不显著(补充数据S3).
芒果基因组学的Web资源
一个在线基因组资源被创建来支持芒果的研究(https://mangobase.org).Mangobase包含一个基因组浏览器,显示具有基因序列,注释,重复位置,SNP曲目和其他数据的芒果参考序列。包括BLAST的其他工具可用于与与基因组序列,CD和蛋白质数据集相关的数据集一起使用。还可以查看遗传地图芒果基地.org. 该系统基于SGN平台(https://solgenomics.net/) [51],它还包括许多其他功能,包括用于基因位点的社区管理系统,以及用于现场和基因分型实验的高级数据管理。所有数据都可以作为完整的数据集从FTP站点下载ftp://ftp.mangobase.org/.
讨论
生成一个注释的基因组组装树木作物,如芒果,提供了一个必要的遗传和基因组工具,通过植物育种改进。最终的“汤米·阿特金斯”二倍体一致组装,TA4,由20个假分子组成,与芒果的两个可用的遗传图谱一致。通过对87个“Tommy Atkins”x“Kensington Pride”杂交群体的个体进行分段测序,确定了基因组中已知核苷酸位置的~ 3 M个SNP变异。结果是为芒果社区和那些对比较基因组感兴趣的人提供了一个公开可用的资源。测序和数据分析的进步使科学发现成为可能,即使是对芒果等极有价值但历史上一直未被研究的物种。
最近,一个芒果品种“Alphonso”的基因组序列发表了[52].这两个基因组的测序、组装和注释方法有显著差异。然而,两个基因组装配使用相同的已发布的图谱来完成装配[33].然而,“汤米·阿特金斯”基因组使用了与发表的芒果地图相同的连锁基团编号来识别假分子,而“阿方索”发表的没有。相反,“Alphonso”基因组提供了每个伪分子对应的连接基团。利用这一信息,生成了一个表来显示两个装配的伪分子的对应关系(补充表S8).除了在“Alphonso”组装中有一个~ 7mb大的伪分子19外,每个伪分子的长度基本相似。两个组件的质量测量(例如BUSCO分析)也是相似的。这两个基因组的注释有显著差异,其中“Alphonso”的预测蛋白编码基因为41,251个,而“Tommy Atkins”的预测蛋白编码基因为26,616个。据报道,“阿方索”基因组的重复序列含量为40.5%,而“汤米·阿特金斯”基因组的重复序列含量为48%。
一般来说,“Alphonso”芒果基因组和我们的基因家族分析结果的主要差异是由于输入数据的数量和我们分析的重点不同。共有41,251个蛋白质编码基因注释了“Alphonso”,而“Tommy Atkins”基因组中有26,616个优质基因。这种差异导致有效的基因家族数量减少,用于Café分析“汤米·阿特金斯”基因组比较“阿方索”(分别为3281和3791)。此外,当排除可能的转座因子时,为“Tommy Atkins”基因家族分析调用的参数是非常严格的,而当包括代表超过100个基因的任何基因家族时则更加保守。例如,一些抗病基因家族(如:NB-LRR)可能被TE基因家族错误地描述,夸大实际数量,这可能影响基因家族结果。此外,本研究使用基于共享的pfam域的人工合并计数,而不是使用Orthofinder基于交互爆炸自动创建的基因家族。最后,本研究的重点是比较更年期和非更年期水果之间的差异,去除膨大基因家族使本研究的分析更准确地识别这些差异。这些差异可能导致扩缩基因家族分配结果的对比。
使用重新编号的伪模节,比较两种基因组的电流曲线,并且通常发现是一致的。一个很好的例子是Pseudomolure4和15之间的Synteny为“汤米阿特金斯”和相应的MIN11和MIN19为'alphonso'。用于产生代名代表的推断的系统发育树和频率分布的方法,以允许完全基因组重复的发生估计为“alphonso”和'tommyatkins'是不同的。考虑到常用基因组的数据,推断的系统发育树或估计芒果的全基因组重复事件没有显着差异。的一个nalysis of expansion or contraction of gene families after the whole genome duplication event for mango gave different results for ‘Alphonso’ where the majority of gene families showed expansion in comparison to other genomes and ‘Tommy Atkins’ where gene families exhibited contraction in comparison to other genomes as described below.
对芒果和果实物种(跃变型和非跃变型)的基因组分析表明,芒果没有明显扩展的基因家族,只有收缩型。果胶转化酶/果胶甲基酯酶抑制剂基因家族在芒果基因组中收缩最大。果胶转化酶/果胶甲基酯酶抑制剂蛋白已知参与果实成熟和软化。其他被承包的基因家族有糖基水解酶家族28 (GH28),包括聚半乳糖醛酸酶(内、外和鼠李糖醛酸酶),糖基水解酶家族1 (GH1),包括β -半乳糖苷酶和(1 - 4)- β -葡聚糖酶和其他蛋白质和未知的基因产物(表)3.).果胶甲基酯酶抑制剂(PMEI)是销售中的大型多岛家族,并调节PME活性和果实成熟期间的甲基酯化程度[53,54].拟南芥中鉴定出71个PMEI基因[55], 97英寸芸苔属植物拉伯[56]芒果中鉴定出18种PMEIs。这种家族收缩可能与芒果果实硬度的迅速丧失(果胶降解引起的早期软化)有关[37这是由于PME的抑制作用减弱。在PME作用下,聚半乳糖醛酸酶(PG)继续水解果胶;PG是植物中最大的水解酶家族之一A.拟南芥,芸苔属植物拉伯,马铃薯,Solanum lycopersicum,杨树trichocarpa,大豆,Citrullus lanatus和Cucumis巨大成功分别有68、99、49、54、76、98、62和53个基因[57].但是,在芒果基因组中鉴定了48页PGS [58];以前,在芒果CV的转录组中仅鉴定了17个PG基因。在成熟期间,肯特和9中的9个差异表达了[36,58].尽管与其他参考植物相比,PG家族在芒果基因组中收缩了,但其中有7个是外显PG,它们与芒果果实中的果胶修饰和软化有关[25].在“Tommy Atkins”芒果基因组中还鉴定了30pmes和二十五个β-半乳糖苷酶,并且还鉴定了对软化的细胞 - 壁改造酶重要性[25].
“汤米·阿特金斯”基因组的可用性将使以前的芒果基因组研究获得更大的见解。例如,之前的一项研究确定了涉及芒果果实成熟和果实品质相关代谢途径的蛋白质转录本[36].将这些reads映射到基因组将有助于进一步深入分析与多聚半乳糖醛酸酶、细胞壁蛋白和酶、代谢、乙烯生物合成和信号转导、蔗糖和类胡萝卜素代谢过程相关的转录本,并且多糖的分解代谢过程不仅为编码序列,而且为调控区域,以揭示调控基因表达的基因产物。
通过分析平均果实重量表型数据,我们证明了基因组组装和‘Tommy Atkins’x‘Kensington Pride’图谱的实用性。这对育种者和种植者来说都是非常有价值的特性。各亲本的果重基本相同(~ 400 g),而杂交种的果重在~ 165 g ~ ~ 965 g之间(表1)4,无花果。5和6),受到高度杂合的父母最有可能引起的近灾区的强烈示例。我们能够将果体重特征与两个区域上的果实重物与LG4上的两个区域相关联,其中一个关于LG7,具有重要关联(P值< = 0.001)。LG4区域的候选基因包括编码泛素蛋白连接酶的E3基因。该基因(Prupe.6G045900 E3泛素蛋白连接酶)在桃树6号染色体上的果实质量QTL中被发现。英仙李) [59]. 在桃中的表达研究表明,Prupe.6Go45900在果实发育期间的表达量增加了5倍,在果实重量QTL分析的19个候选基因中的表达量增加幅度位居第二。
从控制杂交中绘制群体图谱在芒果中并不常见,因为创建它们需要高水平的技术熟练度。大多数林木育种群体是由已知商业价值的开放授粉母本和通过遗传筛选确定父本并由两个已知亲本组成的杂交群体。在‘Tommy Atkins’x‘Kensington Pride’种群中,双亲都是有价值的商业品种,从其后代中选择有成为商业品种的潜力。两种亲本都是高度杂合子的,因为商业果树是从开放授粉母树的种子中选择的,以获得良好的园艺性状,然后进行营养繁殖。一般来说,与一年生作物相比,研究的F1种群数量较少,图谱的发展和性状的关联需要伪检验杂交方法(Grattapaglia和Sederoff, 1994)。我们的每个SNP的单倍型遗传重组图谱对我们的芒果定位群体是独一无二的。因此,我们对两个QTL区双倍型与果实质量的关联分析也是独一无二的。这使我们能够制定策略,以提高从任何一个亲本的开放授粉后代中确定最适合商业果实大小的子代的效率。
将果重与QTL区域中特定的双倍型联系起来,说明LG4区域是果重的一个重要指标。在LG4 QTL区域,双体型TA2KP1的平均单果重最低(329) g) TA2KP2平均单果重最高(499) g) 是的。由于TA2单倍型存在于最大和最小的果实中,对果实大小的主要影响是由于KP亲本KP1单倍型降低了果实重量,KP2单倍型增加了果实重量。在LG7的QTL中没有观察到明显的双倍型差异,但TA2KP1(450)的两个平均果重最高 g) 和TA2KP2(468 g) 而TA1KP1和TA1KP2两个双倍型几乎相同,分别为352和359 分别为g。因此,在LG7的QTL上,TA亲本的效应最大,TA1降低单果重,TA2增加单果重。每个QTL区域的双倍型可以用24个SNPs可靠地预测。因此,可以在苗期筛选“Tommy Atkins”自由授粉后代,以确定LG7 QTL区域是否存在TA2单倍型Kensington Pride的自由授粉后代可以在LG4 QTL区域筛选KP2的存在。其它园艺性状的QTL鉴定正在进行中,可以为‘Tommy Atkins’和‘Kensington Pride’的自由授粉后代在苗期进一步提高选择效率提供一套有利单倍型的标记。
结论
芒果基因组联盟成功开发了最具商业价值的品种“汤米·阿特金斯”的芒果基因组,这是向研究芒果遗传学的全球综合倡议迈出的一步。对“Tommy Atkins”基因组的测序被证明在鉴定与果实重量相关的qtl、基因和双倍型方面是有用的。我们预计,“汤米·阿特金斯”基因组和相关资源的可用性芒果基地.org将来会导致未来的额外发现。
方法
本研究的目的、设计及设定
本研究的目的是建立一个高质量的芒果基因组,并以果实重量为例进行性状分析。
水果重量测量
Fruit weight was measured in fully mature fruit at harvest on a sample of ten randomly picked fruit from the ‘Tommy Atkins’ x ‘Kensington Pride’ parents and each of the 99 individual progeny within the ‘Tommy Atkins’ x ‘Kensington Pride’ hybrid population. The measurements were repeated over 5 years between 2007 and 2012.
植物材料和DNA提取
“Tommy Atkins”芒果品种在美国佛罗里达州迈阿密的USDA亚热带园艺研究站策划和维护。A hybrid population from the cultivar ‘Tommy Atkins’ (TA, maternal parent) and cultivar ‘Kensington Pride’ (KP, paternal parent) consisting of 104 individuals was generated by hand pollination and maintained at the Department of Agriculture and Fisheries, Mareeba, Australia [60].通过裂解分离的细胞核,从“汤米·阿特金斯”叶物质中分离出高分子量DNA。从“汤米·阿特金斯”,“肯辛顿骄傲”和“汤米·阿特金斯”x“肯辛顿骄傲”杂交体的DNA从叶片材料中分离,使用mago - bind植物DNA DS 96试剂盒(Omega, M1130-01),根据制造商的协议略有修改,以尽量减少降解。40 3毫米叶拳(约40毫克)地面一旦CSPL提取缓冲和蛋白酶K Genogrinder 2000在1750转2分钟。30分钟后孵化在65°C,样本离心机在4000 x g 15分钟和500 ul的溶解产物被转移到一个新的96深井板。其余步骤均根据Omega制造商协议在Hamilton Microlab STARlet液体处理机器人上进行,所有混合液均采用涡流而不是吸气,最终洗脱液手动转移。
基因组测序、组装和注释
“汤米·阿特金斯”高分子量DNA在NRGene(以色列)进行了文库构建、测序和组装。脉冲场凝胶电泳验证了高分子量DNA质量。利用Chromium仪器(10X Genomics, Pleasanton, CA)分离长度大于50 Kb的DNA片段,构建Gemcode文库。该文库在HiSeqX平台上进行测序,获得2 × 150 bp的序列。构建了5个大小选择文库,大小选择范围为470bp ~ 10kb,其中2个猎枪文库大小选择范围为~ 470bp,未进行PCR扩增。这个片段大小被设计用来产生Hiseq2500 v2快速模式上的片段的2 × 265 bp的测序重叠,从而创造了一个机会产生长度约265 bp到520 bp的“拼接”reads。根据制造商的协议(Illumina, San Diego, CA),使用TruSeq DNA样品制备试剂盒版本2,不经PCR扩增,制备800 bp DNA片段大小的基因组文库。为了增加序列多样性和基因组覆盖率,使用Illumina Nextera配对样品制备试剂盒(Illumina, San Diego, CA)构建了3个独立的MP文库,跳跃量分别为2-5 Kb、5-7 Kb和7-10 Kb。使用Illumina HiSeq2500对800 bp的猎枪文库进行测序,结果为2 × 160 bp(使用v4 Illumina化学),MP文库在HiSeq4000上进行测序,结果为2X150 bp。对于10倍铬库,PE和MP库的构建和测序是在伊利诺斯大学厄巴纳-香槟分校Roy J. Carver生物技术中心进行的。 10X Chromium library construction and sequencing were conducted at HudsonAlpha Institute for Biotechnology, Huntsville, Alabama.
利用“Tommy Atkins”x“Kensington Pride”图谱绘制的高密度初步图谱,将新组装的支架排列成连锁组。使用来自10X Genomics测序(10X Genomics, Huntsville, AL)的数据进一步改进了组装。产生了一致组装(TA4),并减少到20个假分子(连接基团)加上未组装的支架。通过比较两个芒果SNP图中的SNP顺序,验证并修改了装配[13,33].
基因组组装使用DeNovoMAGIC™软件平台(NRGene, Nes Ziona, Israel)。这是一个基于DeBruijn图的汇编器,旨在有效地提取原始读取中的底层信息,以解决由于基因组多倍体、杂合性和重复性而导致的DeBruijn图的复杂性。这个任务是通过在图中使用基于精确读取的移动来完成的,该移动在局部重复上迭代连接连续的相控contigs来生成长相控脚手架[61,62,63,64,65]. 额外的原始铬10X数据被用于多倍体/杂合性相,支持支架验证和进一步延长相支架。利用denovogic进行杂合子基因组组装™ 产生两个装配版本:分阶段和非分阶段。
对于读预处理,PCR副本、illumina适配器和Nextera连接(用于MP库)被移除。将pe450 bp 2 × 265 bp的库重叠reads合并,最小需要的重叠为10 bp,以创建缝合reads。在预处理之后,合并PE读数据被扫描,以检测和过滤具有假定测序错误的读数据(包含在其他读数据中不会重复出现多次的子序列)。从所有PE和MP读取中构建一个De Bruijn图(kmer = 127 bp)。接下来,PE读取被用来在图中寻找contigs之间的可靠路径进行重复解析和扩展。10x条形码读取被映射到连续序列,确保只有在有证据表明这些连续序列来源于单个基因组序列的情况下,相邻的连续序列才被连接(来自同一两个或多个条形码的读取被映射到两个连续序列)。
对于分阶段/非阶段的装配过程,进行两个并行装配以完成分阶段和非阶段的装配结果。阶段组装过程利用完整的contigs集。在非阶段装配过程中,识别出同源连续体,过滤掉其中一个同源连续体,留下纯合子集和杂合子区中的一个同源连续体。在非阶段装配的装配过程中,两个同源contigs的连接信息被保存,通常使更长的非阶段支架成为可能。
对于脚手架组件,所有以下步骤都是平行的,用于分阶段和未分阶段的组件。Contigs与PE和MP信息连接到脚手架中,根据PE和MP链路的距离估计Contig之间的间隙。此外,使用10x数据来验证和支持在脚手架期间正确的相位。最终间隙填充步骤使用PE和MP链路和DE BRUIJN图信息,以检测连接间隙边缘的唯一路径。将10x条形码读数映射到组装的支架,并且用相同条形码映射到支架中的相同条形码的读数被鉴定为单个长分子的一部分。接下来,用20kb长度窗口扫描每个支架,以确保覆盖整个窗口的不同簇的数量(表示通过几个长分子的20kb连接的支撑)相对于簇的数量是统计学意义的跨越窗口的左侧和右边缘。在检测到潜在的脚手架组件误差的情况下,支架在可疑20kb窗口的两个边缘处破裂。最后,将被映射到支架边缘的条形码(第一和最后20 kB序列)进行比较,以产生具有连接两个支架的连杆与两个以上的常见条形码连接的支架曲线图。支架图中的线性支架路径被组成为组件的最终支架输出。
分阶段重组遗传图谱
TA4支架的组装(包括10X Genomics的数据)产生了一个部分阶段的组装,即每个支架都起源于单个单倍型。为了将这些分阶段的支架分类为单倍型组(例如找到支架之间的连接并将它们放在同一个LG中),我们制作了分阶段重组基因图谱。
Illumina测序(5倍覆盖范围)的DNA从“汤米阿特金斯”,“肯辛顿骄傲”,和每个“汤米阿特金斯”x“肯辛顿骄傲”杂交是由NRGene进行的。各亲本均鉴定出独特的杂合snp。所有的snp在一个亲本中均为纯合子,在另一个亲本中均为杂合子,这使得亲本单倍型在图谱的每个区域都能被识别出来,并且每个个体都有重组点。如(Grattapaglia和Sederoff, 1994)所述,NRGene使用伪检验杂交方法生成亲本图谱。将SNPs标记到TA4组装的部分阶段支架上,以确定它们的单倍型起源和位置。SNPs是根据其在TA4最终组装中的伪分子上的核苷酸位置命名的。建立了拥有20个连锁群的“Tommy Atkins”x“Kensington Pride”群体的阶段性SNP图谱,SNP数量约为3.3 M。根据Illumina序列数据和TA4阶段装配推断,确定了杂交个体在每个SNP位点上的基因型和单倍型。
重复屏蔽、标注和质量控制
使用Maker Wiki中的重复图书馆建设基础描述的策略创建了特定于芒果的重复库(http://weatherby.genetics.utah.edu/maker/wiki/index.php/repeat_library_construction -basic.). RepeatModeler 1.0.8版(http://www.repeatmasker.org/RepeatModeler.html)和先决条件RepeatMasker和库(http://www.repeatmasker.org.),侦察(http://www.reatmasker.org/RECON-1.08.tar.gz),重复侦察(http://repeatscout.bioprojects.org)基金会(http://tandem.bu.edu/trf/trf.html)及新界经济发展局(ftp://ftp.ncbi.nih.gov/pub/seg/nseg/)从适当的网站下载。上面引用的芒果特定的重复库和repeatmasker库与重复掩码器一起使用,以在TA组件上掩模重复。可能被错误掩蔽的潜在基因的手策序仅揭示了逆转录病毒基因,并在注释之前随后掩盖。
注释重复蒙版TA4组件和整体质量评估的注释是制造商-P [66遵循所描述的协议。
转录本证据来自用于SNP标记开发的RNA序列数据[29]以及NCBI Genbank(GAPC01,GBCV01,GBJO01,GBVW01,GBVX01)的五种转录om。推定的蛋白质编码基因使用来自抄本证据(mango_all_mains_protein.fasta.txt),uniprot_sprot_plants.fa,tair10_pep_20101214_updated和proteing.fa(素类). 基因发现者是受过芒果训练的奥古斯都和证据建模者。如前所述,在组装中分析了BUSCO基因[67].
特征与税率杂交种的单倍型和基因型的关系
“Tommy Atkins”x“Kensington Pride”群体的亲本和杂交后代的平均果实重量数据如前所述收集[13].根据分阶段重组遗传图谱,确定了各杂种个体各SNP位点的基因型和单倍型。性状与每个杂交个体在每个SNP上的基因型和单倍型的关联使用PLINK [68].PLINK分析使用qassoc或qassoc。费舍尔是相同的。使用qassoc验证初始输出。Perm, which permutated the genotype data for individuals and recalculated theP协会的价值。的改变P-值估计数经过过滤,产生一个与果实重量相关的SNP标记子集P-value <= 0.001,定义了果实重的QTL区域。
Orthofinder分析
八种物种的蛋白质组用于对比较的芒果研究。包括九种物种是拟南芥此外还有四种跃期水果和四种非跃期水果,即苹果(海棠), 桃 (碧桃)、番茄(Lycopersicum esculentum)、芒果(Mangifera indica.)、橙(素类),草莓佛手柑),橄榄(Olea Europaea)和葡萄(维达斯酿酒用葡萄).转座因子相关基因在基因家族扩增和收缩分析中产生噪声,因此利用转座因子(TransposonPSI) (http://transposonpsi.sourceforge.net)并过滤出来。基因在用PFAM结构域ID使用KINFIN在正交组中用纯注释[69]为了在分配Orthogroups时弥补矫正器的高严格性。使用类似功能的正交组手动合并计数,并根据PFAM结构域创建了基因家族列表。共有3281个基因家族用作CaféV3.1的输入[70],以估计计算基因出生率和死亡率所需的lambda值(补充表)S9和S10).
果实质量QTL区基因鉴定及单倍型鉴定
利用QTL区snp的核苷酸位置搜索芒果基因组组装注释,鉴定候选基因。利用分阶段遗传图谱确定供体的单倍型,并对整个QTL区进行单倍型累加,以确定供体的单倍型。最常见的结果是,在整个QTL区,每个亲本都有一个单倍型。来自母本或父本的一个以上单倍型的存在是该区域重组事件的证据。可以根据要求提供用于此分析的Perl脚本。通过方差分析和事后Tukey多重比较均值检验,将单倍体与果实重量进行关联。
全基因组复制分析
序列组件,CD和Gene注释GFF文件从NCBI下载(P维拉- Pisver_v2, GCA_008641045.1 [71]及植物线虫(C. Sinensis.- v1.1,参考JJOQ01000000)。使用WGD (commit a77f8f4 on Nov 26, 2019)估计Ks分布[72]. 时间树(http://timetree.org/)用来估计年龄之间的差异C. Sinensis.和anacardiaceae [42].
重复序列注释
四个基因组(芒果,腰果,开心和柑橘)中的转移元素(TES)通过结合基于同源的和基于Novo的方法来注释。对于De Novo方法,我们使用了RepeatModeler(Smit等,2015),LTR_Finder [73],ltrharvest [74,和ltr_retriver [75]来构建de novo TE库。对于基于同源性的方法,我们使用Repbase [76]每个物种的图书馆。组合来自这两种方法的TE图书馆。我们使用了重复克服[77]来识别所选基因组中的个体te。
数据和材料的可用性
在NCBI下沉积完整的芒果基因组序列WIOI00000000加入号码。用于构建基因组的原始序列读取可在序列读取存档(SRA)存储库prjna450143..
缩写
- LG公司:
-
连锁群
- gh:
-
糖基水解酶家族1
- m .籼:
-
Mangifera indica.
- 公吨:
-
吨
- 迈亚:
-
百万年前
- PME:
-
果胶methylesterase
- PMEI公司:
-
果胶甲基酯酶抑制剂
- 答:
-
聚半乳糖醛酸酶
- QTL:
-
数量性状基因座
- SNP:
-
单核苷酸多态性
- WGD:
-
全基因组重复
参考
- 1.
加兰SV。世界芒果生产和销售趋势。Acta Horticulturae。2017;(1183):351 - 64。https://doi.org/10.17660/ActaHortic.2017.1183.51.
- 2.
Faostat F.粮食和农业数据。2011年。
- 3.
GalánSaúcoV.世界芒果生产和营销的趋势。在:Xi International Mango研讨会1183;2015年。
- 4.
侯Mangifera。植物Malesiana。1978;8:423-40。
- 5.
Kostermans A, Bompard J-M。《芒果:植物学与命名》。伦敦:园艺与利用学术出版社;1993.
- 6.
分类学与系统学。简介:芒果:植物学、生产和用途。瓦林福德:出租车国际;2009.19-41。
- 7.
芒果的植物学、栽培、用途和未来的改进,特别是在印度观察到的。经济学机器人。1953;7(2):130 - 62。
- 8.
穆克吉,李茨,介绍:植物学和重要性。简介:芒果:植物学、生产和用途。第二版;2009.1–18.
- 9
BOPPARD,JM。分类和系统学。芒果:植物学,生产和用途,第二次。瓦莱特福德:Cabi;2009.19-41。
- 10.
Gruezo WS,芒果属。在:Verheij EWM,Coronel RE(eds)东南亚植物资源第2号:食用水果和坚果。瓦赫宁根:Pudoc DLO;1992年p。203–6.
- 11
导论:植物学和重要性。正确答案:Litz R, editor。芒果;植物学、生产和用途。瓦林福德:出租车国际;1997.p -。
- 12
巴利是,狄龙是NL。芒果(Mangifera indica.L.)繁殖。:植物育种策略进展:水果。Cham:Springer;2018. p。811-96。
- 13
Kuhn DN, Bally ISE, Dillon NL, Innes D, Groh AM, Rahaman J等。芒果遗传图谱:芒果育种的工具。植物科学与技术,2017;https://doi.org/10.3389/fpls.2017.00577PubMed PMID:WOS:0003995248000001。
- 14
Knight RJ, Schnell RJ。芒果在佛罗里达的引进和“哈登”品种对现代工业的意义。经济学机器人。1994;48(2):139 - 45。
- 15
热带和亚热带水果作物的起源和世界重要性;1980.
- 16
Schnell rj,骑士rj。用于估计遗传关系的随机扩增多态性DNA(RAPD)标记Mangifera indica.L霍特科学。1992;27(6):574摄氏度。
- 17
介绍芒果到澳大利亚。Proc R Soc Qld. 2000;109:83。
- 18
讨厌的伊势,编辑器。澳大利亚芒果育种活动。第四届热带和亚热带水果国际研讨会。茂物:国际园艺科学学会,园艺学报;2008.(印刷中)。3.-7 November 2008
- 19
Iyer C, Degani C.经典育种和遗传学。见:芒果植物学,生产和使用。Wallingford Oxon: CAB International;1997.49 - 68页。
- 20.
Iyer注册会计师,Schnell RJ。育种和遗传学。作者:Litz RE,编辑。芒果植物学,生产和使用第二版。沃林福德第二版,奥克斯:卡比国际;2009年。67–96.
- 21.
Kulkarni VJ, Bally ISE, Brettell RIS, Johnson PR, Hamilton D.澳大利亚国家芒果育种计划——寻找新千年的改良品种。Acta Hortic。2000;575:287 - 93。
- 22.
Hardner CM,Bally ISE,Wright CL.利用多元个体混合模型预测芒果平均果重的育种值。大提琴。2012;186(2):463–77.https://doi.org/10.1007/s10681-012-0639-7PubMed PMID:我们:000304698100017。
- 23.
Sharma RK、Singh PK、Joshi AK、Bhardwaj SC、Bains NS、Singh S。保护南亚小麦生产免受茎锈病(Ug99)流行的影响。植物病理学杂志。2013;161(5):299–307.
- 24.
Tharanathan rn,Yashoda HM,Prabha TN。芒果(Mangifera indica.“水果之王”——概述。《国际食品评论》,2006年;22(2):95–123.https://doi.org/10.1080/87559120600574493PubMed PMID:WOS:000236762800001。
- 25。
李志明,陈建平,李建平。热带水果成熟过程中壁降解酶、果胶改性及软化的比较研究。植物科学。2004;167(2):317 - 27所示。https://doi.org/10.1016/j.plantsci.2004.03.030PubMed PMID:我们:000222280600016。
- 26。
Warschefsky ej,von Wettberg EJB。芒果种群基因组分析(Mangifera indica.)建议驯化的复杂历史。新植物。2019; 222(4):2023-37。https://doi.org/10.1111/3ph.15731PubMed Pmid:WOS:000467301100030。
- 27。
Dillon NL、Bally ISE、Wright CL、Hucks L、Innes DJ、Dietzgen RG。澳大利亚国家芒果基因库的遗传多样性。园艺科学。2013;150:213–26.https://doi.org/10.1016/j.scienta.2012.11.003PubMed PMID:WOS:000315557600032。
- 28。
关键词:芒果,EST-SSR,表达序列标记,多样性分析多样性。2014;6(1):72 - 87。
- 29。
Kuhn D,Dillon N,Innes D,Wu L-S,Mockaitis K,编辑。芒果单核苷酸多态性(SNP)标记的建立(Mangifera indica.)转录组,进行遗传多样性的定位和估计。ISHS木瓜、菠萝和芒果2016年国际研讨会。
- 30.
(1)库恩(Kuhn DN, Dillon N, Bally I, Groh A, Rahaman J, Warschefsky M等。利用SNP标记和简化的目视分析方法估计芒果种质的遗传多样性和亲缘关系。Sci Horticulturae。2019;252:156 - 68。https://doi.org/10.1016/j.scienta.2019.03.037PubMed PMID:我们:000467513900021。
- 31。
Kuhn D, Livingstone D III, Richards J ., Manosalva P ., Van den Berg N ., Chambers A. A.基因组工具在鳄梨(Persea americana)育种中的应用:SNP的发现和基因分型。Sci Hortic。2019;246:1-11。
- 32。
谢尔曼,鲁宾士丹,贝尼塔,伊什-沙洛姆,沙拉比-施维格等。芒果(Mangifera indica.L.)基于从转录组的单核苷酸多态性的种质多样性。BMC植物生物学。2015; 15:277。https://doi.org/10.1186/s12870-015-0663-6PubMed Pmid:WOS:000364627700001。
- 33.
罗超,舒斌,姚启森,吴慧霞,徐文涛,王淑平。基于特异性位点扩增片段测序的芒果高密度遗传图谱构建。植物科学与技术,2016;https://doi.org/10.3389/fpls.2016.01310PubMed Pmid:WOS:000382123800001。
- 34.
党智,陈勇。基于SRAP、AFLP和ISSR标记构建芒果遗传连锁图谱。生物科技阿格利司》。2017;6(6):9到16。
- 35
Azim MK,汗IA,张Y.芒果的特征(Mangifera indica.转录组和叶绿体基因组。中国生物医学工程学报,2014;https://doi.org/10.1007/s11103-014-0179-8PubMed PMID:我们:000335756900014。
- 36
Dautt-Castro M, Ochoa-Leyva A, Contreras-Vergara CA, pachco - sanchez MA, Casas-Flores S S, Sanchez-Flores A等。芒果(Mangifera indica.L.)CV。Kent Fruit Mesocarp de Novo转录组大会识别对成熟的重要组织。前植物SCI。2015; 6:62。
- 37
Dautt-Castro M,Ochoa-Leyva A,Contreras-Vergara Ca,Muhlia-Almazan A,Rivera-Dominguez M,Casas-Flores S等人。Mesocarp RNA-SEQ芒果分析(Mangifera indica.l)鉴定检疫采后处理对基因表达的影响。Sci Horticulturae。2018;227:146-53。https://doi.org/10.3389/fpls.2015.00062PubMed PMID:我们:000414107700018。
- 38
Luria N,Sela N,Yaari M,Feygenberg O,Kobiler I,Lers A等。芒果果皮转录组的从头组装揭示了芒果对热水处理的反应机制。BMC基因组学。2014;15:957.https://doi.org/10.1186/1471-2164-15-957PubMed PMID:我们:000345250700001。
- 39
Pandit SS,Kulkarni Rs,Giri AP,Kollner TG,Degenhardt J,Gershenzon J,等。各种基因在果实开发期间的表达分析和芒果成熟。植物理性。生物学习。2010; 48(6):426-33。https://doi.org/10.1016/j.plaphy.2010.02.012PubMed PMID:我们:000278798900006。
- 40.
吴慧霞,贾慧明,马新文,王sb,姚qs,徐文涛,等。芒果的转录组和蛋白质组学分析(Mangifera indica.Linn)水果。j .蛋白质组学。2014;105:19-30。https://doi.org/10.1016/j.jprot.2014.03.030PubMed PMID:WOS:000338600000003。
- 41.
主要植物核DNA含量。植物生物学杂志。1991;9(3):208-18。
- 42.
[11]刘永强,杨晓东,杨晓东。时间树:一种用于时间轴、时间树和发散时间的资源。北京:科学出版社。摩尔。杂志。另一个星球。2017;34(7):1812 - 9。https://doi.org/10.1093/molbev/msx116.PubMed Pmid:WOS:000402754400023。
- 43.
皖南BS。椿科属间关系分析。中国医学杂志。2006;51(1):65 - 95 PubMed mid: WOS:000237710400009。
- 44.
Pierozzi NI, Rossetto CJ。两个品种的染色体特征Mangifera indica.L牧师胸罩。2011;33(1):546–51.https://doi.org/10.1590/S0100-29452011000500074PubMed PMID:我们:000297793700073。
- 45.
腰果的染色体研究(青春痘occidentale.l .)。生物技术学报。2007;6(2):131-6。
- 46.
中国山地木本类群的细胞学进化。细胞遗传学杂志1990;25:308-20。
- 47.
黄连木(黄连木科)的染色体数目。中国生物医学工程学报。2003;78(1):35-8。
- 48.
爱A. IOPB染色体数目报告XL。分类单元。1973;22(2):185 - 291。
- 49。
杨志明,张国华,张国华,等。线虫科一些成员的染色体研究。细胞遗传学。1990;25:36-42。
- 50.
OrthoFinder:解决全基因组比较中的基本偏差显著提高了正组推理的准确性。基因组医学杂志。2015;16:157。https://doi.org/10.1186/s13059-015-0721-2PubMed Pmid:WOS:000358984500001。
- 51.
Fernandez Pozo N,Menda N,Edwards JD,Saha S,Tecle IY,Strickler SR等。Sol基因组学网络(SGN)-从基因型到表型再到育种。核酸研究,2015年;43(D1):D1036–D41。https://doi.org/10.1093/nar/gku1195PubMed PMID:我们:000350210400152。
- 52.
王P,罗伊夫,黄杰夫,高世,朱GP,Dang Zg等。热带水果芒果的基因组演变和驯化。基因组Biol。2020; 21(1):60。https://doi.org/10.1186/s13059-020-01959-8PubMed Pmid:WOS:000519049000001。
- 53。
研究结果表明:①Balestrieri C, Raiola A, D’avino R, Tamburrini M, et al。果胶methylesterase抑制剂。Bba-Proteins Proteom。2004;1696(2):245 - 52。https://doi.org/10.1016/j.bapap.2003.08.011PubMed PMID:我们:000189080200012。
- 54。
等。关键词:cara IB, Lionetti V, Camardella L, D'Avino R, Giardina T, Cervone F,等。一种功能性果胶甲基酯酶抑制剂蛋白(SolyPMEI)在番茄果实成熟过程中表达,并与PME-1相互作用。acta botanica botanica(云南植物研究),2012;https://doi.org/10.1007/s11103-012-9921-2PubMed Pmid:WOS:000304913200008。
- 55。
王美娟,袁dj,高文华,李勇,谭建峰,张学利。PME和PMEI家族的比较基因组分析揭示了植物细胞壁中果胶代谢的演变。《公共科学图书馆•综合》。2013;8 (8):e72082。https://doi.org/10.1371/journal.pone.0072082PubMed PMID:我们:000323097300177。
- 56。
谭超,刘志勇,黄森,李春春,任军,唐晓燕,等。果胶甲基酯酶抑制剂(PMEI)家族可能与大白菜(菜心). 分子遗传学。基因组学。2018;293(2):343–57.https://doi.org/10.1007/S00438-017-1391-4PubMed PMID:我们:000427630400004。
- 57.
金杰、萧实、汤玛、李伟、彭定森。植物多聚半乳糖醛酸酶基因家族的扩展模式和表达差异。基因组生物学。2006;7(9):87.https://doi.org/10.1186/gb-2006-7-9r87PubMed PMID:我们:000242490400013。
- 58.
Dautt-Castro M, Lopez-Virgen AG, Ochoa-Leyva A, contrera - vergara CA, Sortillon-Sortillon AP, Martinez-Tellez MA等。芒果的全基因组鉴定(Mangifera indica.多聚半乳糖醛酸酶:在果实成熟过程中家族成员和总酶活性的表达分析。前植物科学。2019;10https://doi.org/10.3389/fpls.2019.00969PubMed Pmid:WOS:00047786860000001。
- 59
曹凯,李勇,邓春华,Gardiner SE,朱桂荣,方文伟,等。比较群体基因组学鉴定了桃果实驯化性状相关的基因组区域和候选基因。植物生理学学报(英文版);2019;https://doi.org/10.1111/pbi.13112PubMed Pmid:WOS:000486082300012。
- 60
吕平,庄臣公关,芒果育种。培育作物作物Trop物种。纽约:施普林格;2009.51–82.
- 61.
Apples R,Eversole K,Feuillet C,Keller B,Rogers J,Stein N等人。使用完全注释的参考基因组转移小麦研究和育种的限制。科学。2018; 361(6403):eaar7191。https://doi.org/10.1126/science.aar7191661 - +。PubMed PMID:我们:000442818200032。
- 62.
Luo MC, Gu YQ, Puiu D, Wang H, Twardziok SO, Deal KR,等。小麦D基因组前体的基因组序列山羊草属tauschii.自然。2017; 551(7681):498- +。https://doi.org/10.1038/nature24486PubMed Pmid:WOS:000416043700044。
- 63.
Hirsch CN、Hirsch CD、Brohammer AB、Bowman MJ、Soifer I、Barad O等。优秀自交系PH207的草案汇编提供了玉米基因组和转录组多样性的见解。植物细胞。2016;28(11):2700–14.https://doi.org/10.1105/tpc.16.00353PubMed Pmid:WOS:000390800000003。
- 64.
Lu F, Romay MC, Glaubitz JC, Bradbury PJ, Elshire RJ, Wang TY,等。玉米全基因组序列锚点的高分辨率遗传定位。Nat Commun。2015;6:6914。https://doi.org/10.1038/ncomms7914PubMed PMID:我们:000353704100022。
- 65.
杨宁,徐锡伟,王瑞荣,彭文丽,蔡丽玲,宋建民,等。的贡献玉米亚种墨西哥单倍型到现代玉米。纳特。公社。2017;8:1874.https://doi.org/10.1038/s41467-017-02063-5PubMed PMID:WOS:000416895400016。
- 66.
Campbell MS、Law MY、Holt C、Stein JC、Moghe GD、Hufnagel DE等。MAKER-P:植物基因组注释快速创建、管理和质量控制的工具包。植物生理学。2014;164(2):513–24.https://doi.org/10.1104/pp.113.230144PubMed PMID:我们:000331132300002。
- 67.
Simao Fa,Waterhouse Rm,Ioannidis P,Kriventseva EV,Zdobnov Em。BUSCO:评估基因组组装和注释完整性,单拷贝正端。生物信息学。2015; 31(19):3210-2。https://doi.org/10.1093/bioinformatics/btv351PubMed PMID:WOS:000362845400018。
- 68.
Chang CC, Chow CC, Tellier LCAM, Vattikuti S, Purcell SM, Lee JJ。第二代PLINK:迎接更大更丰富的数据集的挑战。Gigascience。2015;4。https://doi.org/10.1186/s13742-015-0047-8PubMed PMID:WOS:0003656620000001。
- 69.
Laetsch Dr,Blaxter ML。Kinfin:用于分类蛋白序列的分类群体感知分析软件。G3-基因基因类型。2017; 7(10):3349-57。https://doi.org/10.1534/g3.117.300233PubMed PMID:我们:000412549600012。
- 70.
De Bie T、Cristianini N、Demuth JP、Hahn MW。CAFE:研究基因家族进化的计算工具。生物信息学。2006;22(10):1269–71.https://doi.org/10.1093/bioinformatics/btl097PubMed Pmid:WOS:000237319300018。
- 71.
Zeng L,Tu XL,Dai H,Han FM,Lu Bs,Wang Ms,等。全基因组和转录瘤揭示了开心果的适应和驯化。基因组Biol。2019; 20:79。https://doi.org/10.1186/s13059-019-1686-3PubMed PMID:我们:000465145200002。
- 72.
Zwaenepoel A,Van de Peer Y. WGD-简单的命令线工具,用于分析古代全基因组重复。生物信息学。2019; 35(12):2153-5。https://doi.org/10.1093/bioinformatics/bty915PubMed PMID:WOS:000474844600023。
- 73.
LTR_FINDER:一种预测全长LTR逆转录转座子的有效工具。核酸杂志2007;35:W265-W8。https://doi.org/10.1093/nar/gkm286PubMed Pmid:WOS:000255311500049。
- 74.
Ellinghaus D, Kurtz S, Willhoeft U. LTRharvest,一种高效灵活的LTR反转录转座子从头检测软件。BMC生物信息学。2008;看去。https://doi.org/10.1186/1471-2105-9-18PubMed Pmid:WOS:000253685900001。
- 75.
关键词:长末端重复序列逆转录转座子;植物杂志。2018;176(2):1410 - 22所示。https://doi.org/10.1104/pp.17.01310PubMed PMID:我们:000424285500035。
- 76.
包维德,Kojima KK, Kohany O. Repbase更新,真核生物基因组重复元件数据库。移动DNA-Uk。2015; 6: UNSP 11。https://doi.org/10.1186/s13100-015-0041-9PubMed PMID:我们:000355907900001。
- 77。
应用RepeatMasker技术鉴定基因组序列中的重复元件。生物信息学。2009;25(1):4.10。1 - 4 . .4.
致谢
我们感谢P. Johnson, P. Lu, S. Blakie, R. Brettell, V. Kulkarni,感谢P. Johnson, P. Lu, S. Blakie, R. Brettell, V. Kulkarni,感谢P. Johnson, P. Lu, S. Blakie, R. Brettell, V. Kulkarni,感谢C. Maddox,感谢C. Maddox,感谢B. Freeman,感谢他们提供分子生物学技术支持。
基金
研究在一定程度上资助# 203 - 0859(授予和RO) 203 - 0110(授予YC)的首席科学家以色列农业部和由格兰特# - 5106 - 18 R巴德(美国-以色列两个民族的农业研究与发展基金)授予,DNK, YC和RO)。
AHC得到了美国农业部国家食品和农业研究所(USDA-NIFA 2018–51181-28375)的资助。
IB、ND和DI得到了Hort Innovation和农业部和渔业部的支持。
作者信息
从属关系
财团
贡献
AHC协调团队撰写稿件,完成稿件的撰写。DNK参与了论文的撰写,获得了基因组项目的资助,并进行了果实重量分析。IB开发了“肯辛顿骄傲”杂交种群的“汤米阿特金斯”,并参与了手稿的撰写。ND收集样本,提取DNA,开发并提供“肯辛顿骄傲”基因组序列,并参与撰写手稿。DI开发了“肯辛顿骄傲”基因组序列,并参与了手稿的撰写。AS, YC, RO,参与了基因组组装项目的战略规划,资助了基因组测序,并参与了手稿的撰写。MAIO, AR进行Orthofinder分析并参与撰写稿件。AB和HY进行了WGD和TE分析,并参与了论文的撰写。作者阅读并批准了最终稿件。
通讯作者
道德宣言
伦理批准并同意参与
不适用。
同意出版
这份手稿已经过所有作者的审查和批准。
相互竞争的利益
作者承认其中一个合著者(AB)是BMC植物生物学的编辑委员会成员。作者们宣称他们没有其他的竞争利益。
额外的信息
出版商的注意事项
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充资料
附加文件1:补充数据S1
.snpEFF报道“Tommy Atkins”
附加文件2:补充数据S2
.snpEFF报道“肯辛顿骄傲”
附加文件3:补充数据S3
.果重LG4 QTL报告。S4的补充数据.果重LG7 QTL报告。补充表1. 会议纪要m .籼基因组由染色体组装。补充表2.一致性二倍体基因组装配TA4的BUSCO分析。补充表3.芒果基因组中重复DNA序列的发生和分布。补充表4.蛋白质域的内容。补充表5.每条染色体Ks在0.1 ~ 0.6之间的同源对数。补充表6.重复元素比较分析。补充表7.用伪分子比较“Tommy Atkins”和“Kensington Pride”的SNP变异和变异率。补充表S8. “Tommy Atkins”和“Alphonso”之间链接组的并发性。补充图1. 重复作文籼稻和C. Sinensis.
附加文件4:补充表S9
,S10.orthofinder报告
权利和权限
开放存取本文是根据知识共享署名4.0国际许可证授权的,该许可证允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供到知识共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可证中,除非在材料的信用额度中另有说明。如果材料未包含在文章的知识共享许可证中,并且您的预期用途不受法律法规的允许或超出允许的用途,您将需要直接获得版权持有人的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/. 知识共享公共领域放弃(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
芒果基因组财团。,Bally, I.S.E., Bombarely, A.等。“汤米阿特金斯”芒果基因组揭示了水果品质的候选基因。BMC植物生物学21,108(2021)。https://doi.org/10.1186/s12870-021-02858-1.
已收到:
接受:
发表:
关键词
- Mangifera indica.
- 水果的重量
- QTL.
- De Novo组装