- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
重复寡核苷酸的物理定位有助于建立基于基因组图谱的核型,以鉴定花生染色体变异gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba21gydF4y2Ba,商品编号:gydF4y2Ba107gydF4y2Ba(gydF4y2Ba2021gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
染色体变异在作物育种和遗传研究中起重要作用。单链寡核苷酸(oligo)探针的发展简化了原位杂交(鱼)的荧光过程,并促进许多物种中的染色体鉴定。基因组测序为寡核糖探测器提供丰富的资源。然而,由于缺乏有效的染色体标记,花生在花生中取得了很小的进展。到目前为止,花生染色体变体的鉴定仍然是挑战。gydF4y2Ba
结果gydF4y2Ba
基于从花生品种Tifrunner(AABB,2)的参考序列中鉴定出的全基因组串联重复序列(TRs),共开发了114个新的寡核苷酸探针gydF4y2Ban =gydF4y2Ba4x = 40)和二倍体种gydF4y2Baarachis ipaensis.gydF4y2Ba(BB,2)gydF4y2Ban =gydF4y2Ba2x = 20)。基于其位置和染色体中的重叠信号,将这些寡聚探针分为28种。对于每种类型,用绿色荧光素6-羧基荧光素(FAM)或红色荧光素6-羧基甲基甲基溴胺(TMA)选择代表性寡核苷酸。通过汇集荧光团共轭探针开发了两种鸡尾酒,多路复用#3和多路复用#4。Multiplex#3包括FAM-Demified Oligo TIF-439,Oligo TIF-185-1,Oligo TIF-134-3和Oligo TIF-165。Multiplex#4包括TAMRA改装的寡核苷IPA-1162,Oligo IPA-1137,Oligo DP-1和Oligo DP-5。每个鸡尾酒都能够在顺序鱼/基因组的原位杂交(GISH)和Silico Mapping中建立基于基于地图的核型。此外,我们确定了由辐射暴露引起的花生的14个染色体变体。总共28个代表性探针进一步染色到新的核型上。在探针中,八个被映射在次要收缩,插入和终端区域中; four were B genome-specific; one was chromosome-specific; and the remaining 15 were extensively mapped in the pericentric regions of the chromosomes.
结论gydF4y2Ba
新的Oligo探针的开发提供了一种有效的工具,可用于区分花生的各种染色体。鱼类的物理映射揭示了花生染色体中重复寡核苷酸的基因组织。建立基于基于地图的核型并用于鉴定与其参考序列位置的比较之后花生的染色体变化。gydF4y2Ba
背景gydF4y2Ba
染色体变异,如易位,逆变,重复和缺失在作物育种和遗传研究中起重要作用。这种变化通常由物理诱导[gydF4y2Ba1.gydF4y2Ba,gydF4y2Ba2.gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba],化学[gydF4y2Ba4.gydF4y2Ba,gydF4y2Ba5.gydF4y2Ba]以及遗传因素[gydF4y2Ba6.gydF4y2Ba]. 大量的染色体变异在番茄中被创造和利用[gydF4y2Ba1.gydF4y2Ba)、小麦(gydF4y2Ba4.gydF4y2Ba]、玉米[gydF4y2Ba7.gydF4y2Ba)、棉花(gydF4y2Ba8.gydF4y2Ba]以及其他农作物。gydF4y2Ba
种植花生(gydF4y2Baarachis hypogaea.gydF4y2Ba左,2gydF4y2Ban =gydF4y2Ba4x = 40,基因组AABB)是一种主要的油籽和经济作物。2018年,全球花生总产量45950,900公吨,平均产量1900.20公斤/公顷[gydF4y2Ba9gydF4y2Ba].许多花生基因突变体已经创建并调查了[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba]. 控制荚果宽度的候选基因[gydF4y2Ba14gydF4y2Ba],种子涂色颜色[gydF4y2Ba15gydF4y2Ba和半侏儒状态[gydF4y2Ba16gydF4y2Ba已通过基因突变体的转录组测序鉴定。然而,由于缺乏检测方法和参考基因组,仅报告了少数染色体变化[gydF4y2Ba17gydF4y2Ba].gydF4y2Ba
传统的染色体变异细胞学分析方法包括染色体C显带[gydF4y2Ba17gydF4y2Ba],基因组原位杂交(GISH)[gydF4y2Ba18gydF4y2Ba]利用TRs质粒克隆进行荧光原位杂交(FISH)[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba或转座子[gydF4y2Ba22gydF4y2Ba].这些方法已被用于建立核型,并揭示染色体结构和演化。然而,基于这些方法的核型分辨率太低并且不足以鉴定花生中的染色体变体。此外,上述方法是耗时和昂贵的,从而限制了他们在实践中的广泛应用和效率。gydF4y2Ba
最近发展起来的单链寡核苷酸FISH(SSON-FISH)方法为包括小麦在内的许多物种的染色体鉴定提供了一种简单有效的方法[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba],黑麦[gydF4y2Ba25gydF4y2Ba],大麦[gydF4y2Ba26gydF4y2Ba],玉米[gydF4y2Ba27gydF4y2Ba)、大米(gydF4y2Ba28gydF4y2Ba),土豆gydF4y2Ba29gydF4y2Ba]还有黄瓜[gydF4y2Ba30.gydF4y2Ba]. 全基因组TR寡核苷酸的发展极大地提高了核型的分辨率,有助于准确鉴定染色体变异[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba]. 此外,TRs的物理作图不仅揭示了基因组的进化,而且可以指导高拷贝数重复序列的染色体区域的序列组装[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba33gydF4y2Ba].gydF4y2Ba
在花生中,杜等人[gydF4y2Ba34gydF4y2Ba]应用8个人工合成的寡核苷酸探针对不同花生品种进行核型分析,并鉴定染色体易位系。进一步发展了一种更有效的低聚染料染色方法,并应用于大量花生样品[gydF4y2Ba35gydF4y2Ba];然而,探针数量少的局限性阻碍了核型的分辨。因此,挖掘覆盖所有染色体区域的TRs基因组是提高核型分辨率的必要手段。gydF4y2Ba
最近,花生的基因组测序[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba]及其野生供体物种,gydF4y2BaA.硬脑膜gydF4y2Ba和gydF4y2Ba答:ipaensisgydF4y2Ba[gydF4y2Ba39gydF4y2Ba],已完成。全面的测序数据可以挖掘整个花生基因组中覆盖所有染色体的代表性TRs。根据花生参考基因组图中的遗传连锁图对同源染色体进行编号[gydF4y2Ba36gydF4y2Ba].然而,参考图中所分配的染色体数目与细胞遗传学核型的实际染色体数目并不一致[gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba34gydF4y2Ba].因此,遗传和细胞遗传学图之间的联系目前是不可用的。gydF4y2Ba
本研究旨在:1)基于花生品种Tifrunner(AABB)的参考序列,在全基因组水平上挖掘具有代表性的寡核苷酸[gydF4y2Ba36gydF4y2Ba] 和gydF4y2Ba答:ipaensisgydF4y2Ba(BB)[gydF4y2Ba39gydF4y2Ba];2)建立基于基于地图的Tifrunner核型,物理地图重复寡核苷酸,以揭示花生中的基因组组织;3)使用核型以鉴定花生辐射暴露引起的染色体变化。gydF4y2Ba
结果gydF4y2Ba
利用花生全基因组测序数据开发重复性寡核苷酸探针gydF4y2Ba
基因序列分析gydF4y2Ba答:ipaensisgydF4y2Ba使用串联重复发现(TRF)进行分析[gydF4y2Ba40gydF4y2Ba]分别产生4595个和894个重复序列。这些序列的长度在4 bp和723 而拷贝数在50到29162之间[gydF4y2Ba41.gydF4y2Ba],共有80和35 TRs属于Tifrunner和gydF4y2Ba答:ipaensisgydF4y2Ba,分别用于进一步开发寡核苷酸。gydF4y2Ba
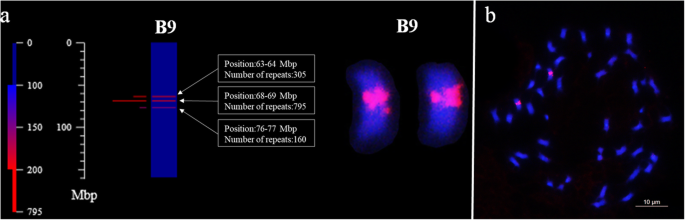
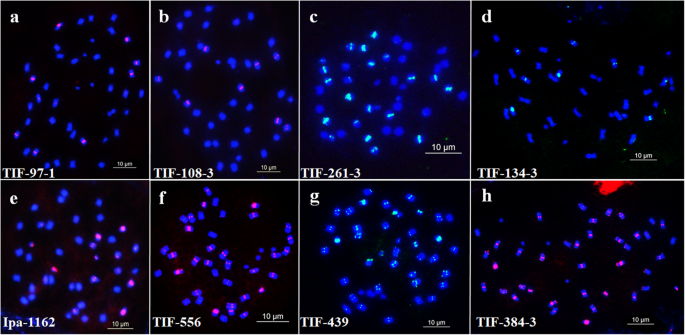
使用oligo7设计了249个oligos[gydF4y2Ba42.gydF4y2Ba],并利用B2DSC基于Tifrunner参考序列在硅上进行了映射[gydF4y2Ba31gydF4y2Ba].设计的寡核苷酸首先通过随机引物标记法进行标记[gydF4y2Ba27gydF4y2Ba]与花生染色体杂交。通过显微镜观察到设计的寡核苷酸114的114在TiFrunner的染色体的不同位置产生了明显的信号(图。gydF4y2Ba1.gydF4y2Ba,图。gydF4y2Ba2.gydF4y2Ba,表格gydF4y2BaS1gydF4y2Ba).基于鱼类观察的图案和位置分布,将114个寡聚探针分为28种类型(表gydF4y2BaS2gydF4y2Ba); 核型中具有相同位置和重叠信号的寡核苷酸被归类为同一类型。对于28种类型中的每一种,选择单个寡聚物并用6-羧基荧光素(FAM)或6-羧基四甲基罗丹明(TAMRA)进一步修饰。数字gydF4y2Ba1.gydF4y2Ba显示了oligo-Ipa-1463在电子地图和FISH之后的结果。在染色体图中,在B9染色体63–77 Mbp区域观察到1260个拷贝(图。gydF4y2Ba1.gydF4y2Baa) 是的。FISH分析证实,ligo-Ipa-1463的信号只存在于一对染色体的相似区域。因此,我们得出结论,该染色体与参考序列中的染色体B9相对应(图。gydF4y2Ba1.gydF4y2Bab) 是的。gydF4y2Ba
基于荧光原位杂交的Tifrunner品种各探针在染色体上的FISH (gydF4y2Ba一个gydF4y2Ba)TIF-97-1(红色,信号);gydF4y2BabgydF4y2Batif-108-3(红色,信号);gydF4y2BacgydF4y2BaTIF-261-3(绿色,信号灯);gydF4y2BadgydF4y2Batif—134 - 3(绿色信号);gydF4y2BaegydF4y2BaIpa-1162(红色,信号);gydF4y2BafgydF4y2Batif-556(红色,信号);gydF4y2BaggydF4y2BaTIF-439(绿色,信号灯);gydF4y2BahgydF4y2Batif—384 - 3(红、信号)。比例尺:10 μmgydF4y2Ba
发展基于基于地图的Tifrunner核型gydF4y2Ba
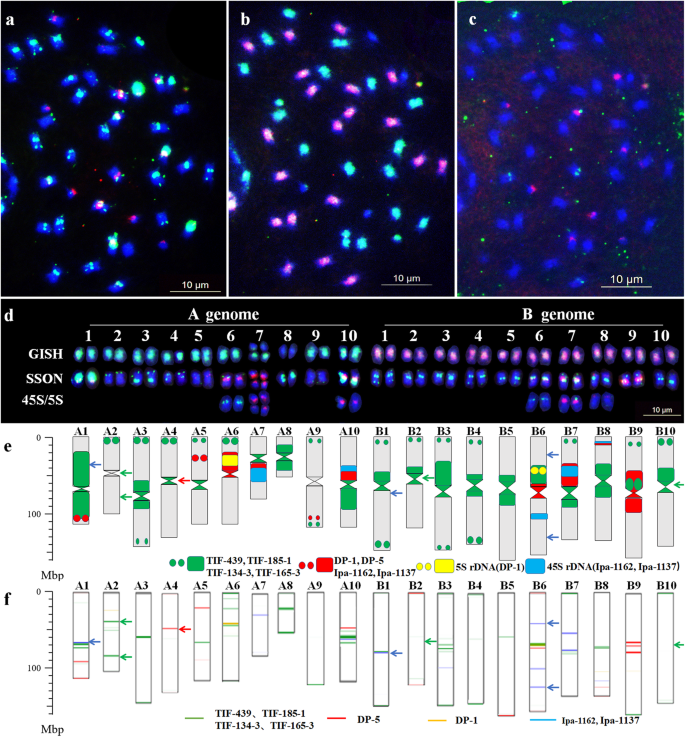
基于28个oligo探针的独特模式和序列组成,我们利用8个合成的oligo开发了两种新的oligo探针鸡尾酒,Multiplex #3和Multiplex #4。复用#3包括FAM-modified TIF-439, TIF-185-1, TIF-134-3和TIF-165-3。多路复用#4包括tamra改进的Ipa-1162, Ipa-1137, DP-1和DP-5。DP-1和DP-5均来源于Du等人之前的研究[gydF4y2Ba34gydF4y2Ba].在Multiplex #3和Multiplex #4的顺序FISH/GISH之后,总基因组dnagydF4y2BaA.硬脑膜gydF4y2Ba和gydF4y2Ba答:ipaensisgydF4y2Ba以45S和5S rdna为探针,建立了Tifrunner的强壮核型(图5)。gydF4y2Ba3.gydF4y2Baa–e)。gydF4y2Ba
序列荧光原位杂交/基因组原位杂交(FISH/GISH)品种的核型和染色体图。gydF4y2Ba一个gydF4y2BaFISH使用Multiplex #3(绿色)和Multiplex #4(红色);gydF4y2BabgydF4y2BaGISH基因的应用gydF4y2BaA.硬脑膜gydF4y2Ba(绿色)和gydF4y2Ba答:ipaensisgydF4y2Ba(红色);gydF4y2BacgydF4y2Ba鱼使用45s rdna(红色)和5s rdna(绿色)作为探针;gydF4y2BadgydF4y2Ba与基因组测序图中的染色体相对应的核型;SSON用多重#3(绿色)和多重#4(红色)表示鱼类的核型;GISH用鱼类的总基因组dna表示鱼类的核型gydF4y2BaA.硬脑膜gydF4y2Ba(绿色)和gydF4y2Ba答:ipaensisgydF4y2Ba(红色);45s / 5s表示使用45s rdna(红色)和5s rdna(绿色)的鱼核型;gydF4y2BaegydF4y2BaTifrunner的Idiogram Karyype;gydF4y2BafgydF4y2Ba染色体图;蓝色箭头(gydF4y2BaegydF4y2Ba)和(gydF4y2BafgydF4y2Ba)表明Ipa-1162和Ipa-1137寡核苷酸在核型和染色体图之间的非对应信号;中的绿色箭头(gydF4y2BaegydF4y2Ba)和(gydF4y2BafgydF4y2Ba)表明TIF-439、TIF-185-1、TIF-134-3和TIF-165-3寡核苷酸在核型图和染色体图之间的非对应信号;红色箭头(gydF4y2BaegydF4y2Ba)和(gydF4y2BafgydF4y2Ba)表示核型和染色体图之间的寡核苷酸DP-5的非对应信号。比例尺:10 μmgydF4y2Ba
比较8个寡核苷酸在核型和染色体图中的分布,我们发现实际染色体中的大多数信号位点和信号强度与其在参考序列中各自的位置和拷贝数很好地对应(图。gydF4y2BaS1gydF4y2Ba). 此外,还建立了一个基于基因组图谱的Tifrunner染色体组型。核型中的实际染色体被重新编号为A1 ~ A10和B1 ~ B10,根据它们在人类基因组图上的伪分子数[gydF4y2Ba36gydF4y2Ba]. 我们发现染色体组型中的一些染色体短臂,如A1和B7,在基因组图中没有定位在短臂(上臂)上。为了便于理解,本研究中的长臂(L)和短臂(S)是根据基因组图谱的上臂和下臂进行分配的。根据这一核型,在7条染色体上观察到9个显著的非对应信号(图。gydF4y2Ba3.gydF4y2Bae-f)。例如,寡核苷酸Ipa-1162和Ipa-1137在染色体图谱的A1和B1染色体上有明显的分布位点,但与实际染色体进行探针杂交时未观察到信号。相比之下,在染色体B2和B10的着丝粒区域,寡聚物tifi -439, tifi -185-1, tifi -134-3和tifi -165-3产生强烈的信号,但在染色体图谱中没有被定位(图1)。gydF4y2Ba3.gydF4y2Bad–f,图。gydF4y2BaS1gydF4y2Ba).gydF4y2Ba
为了验证基因组图核型,我们从基因组图中A1和A3染色体的短臂中选取了两个染色体特异的单拷贝序列寡核苷酸文库L1A-1和L3A-1进行了序列FISH/GISH分析。文库与Multiplex #3、Multiplex #4和总基因组dna结合gydF4y2BaA.硬脑膜gydF4y2Ba和gydF4y2Ba答:ipaensisgydF4y2Ba作为探针,用于后续的分析(图。gydF4y2BaS2gydF4y2Ba).在两个预期的染色体上清楚地观察到两种染色体特异性寡聚文库的具体信号,表明核型和基因组图之间的实际染色体之间的相当大。gydF4y2Ba
在该核型的A亚基因组染色体中,染色体A1和A8在着丝粒区均含有强烈的绿色信号,而A1在其长臂末端也有强烈的红色信号。染色体A6、A7和A10有45S或5srdna位点。染色体A2、A3和A4在短臂末端区域有绿色信号,而A3和A4在着丝粒区域有绿色信号。染色体A5在短臂内有红色信号,而A9在长臂的近端区有红色信号。在B亚基因组中,染色体B6、B7和B8显示了45srdna或5srdna的探针信号。染色体B9和B3在着丝粒区分别有红色和绿色信号。其他染色体在着丝粒或端粒区域有不同强度的绿色信号(图。gydF4y2Ba3.gydF4y2Bae).根据观察到的独特模式,所有染色体在核型中都可以明显分化。gydF4y2Ba
寡核苷酸探针的染色体分配gydF4y2Ba
为了在基于基于基于地图的Tifrunner核型中映射TR,通过鱼和硅映射分析28个代表性寡核苷酸(图。gydF4y2Ba4.gydF4y2Ba).28 oligos在B亚基组染色体上产生更多的信号,而不是亚基因组的信号(图。gydF4y2Ba4.gydF4y2Ba).在28个寡核苷酸中,六个(TIF-165-3,TIF-439,TIF-356,TIF-198-1,TIF-384-3和TIF-185-1)在染色体的间隙或末端区域中产生了信号.四个寡核苷酸(TIF-198-2,TIF-416-3,TIF-497和TIF-342-2)在B亚基上具有仅发信号,表明这些寡核苷酸是对B亚基组特异性的。两个寡核苷酸(IPA-1137和IPA-1162)仅在次级收缩时具有信号,其在连续鱼之后的45s RDNA的信号完全重叠。Oligo IPA-1463专门显示一对染色体上的信号,表明它是特异性染色体的。另外15个寡核苷酸具有位于渗透区域内的信号(图。gydF4y2BaS3gydF4y2Ba).gydF4y2Ba
在通过FISH物理映射的28个寡核苷酸中,22个寡核苷酸的分布与电子映射结果中的分布相同或相似(图。gydF4y2Ba4.gydF4y2Ba,图。gydF4y2BaS4gydF4y2Ba).而6个oligos (TIF-89-3、TIF-155-5、TIF-198-1、TIF-359-3、TIF-76-1和Ipa-1757)在两张图谱中存在显著差异。例如,tifi -89-3和tifi -155-5被硅定位到两对具有大量拷贝的染色体上。8对染色体显示明显的FISH信号。类似地,TIF-198-1在硅中只映射到一对染色体,但在16对染色体上产生了信号。相比之下,在13对染色体上发现了明显的tifi -76-1位点,但在5对染色体上产生了FISH信号。这可能表明TRs显然不是在Tifrunner基因组中组装的。gydF4y2Ba
辐射曝光诱导的Silihwong(SLH)染色体变化的鉴定gydF4y2Ba
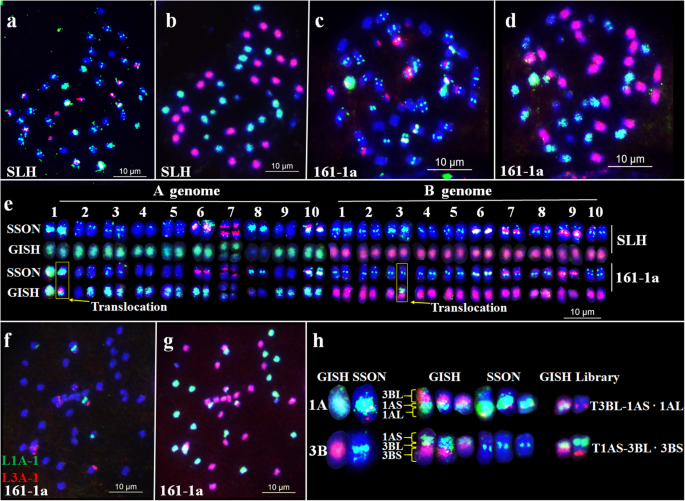
检查花生中的染色体变化,在中国品种SLH和70辐射诱导的M种中进行顺序鱼和GISHgydF4y2Ba1.gydF4y2BaSLH植物具有多重#3和多重#4,总基因组DNAgydF4y2BaA.硬脑膜gydF4y2Ba和gydF4y2Ba答:ipaensisgydF4y2Ba,45秒和5S RDNA作为探针(图。gydF4y2Ba5.gydF4y2Ba).14米gydF4y2Ba1.gydF4y2Ba本研究中使用的植物表现出染色体变异。例如,在植物161-1a中,一个相互易位是明显的,很可能发生在染色体1a和3B之间。来自A亚基因组的染色体易位片段在整个臂上有广泛的绿色信号,在染色体末端有红色信号。这种模式类似于1A的着丝粒和长臂上的信号。另一节段的信号分布模式为短臂末端到着丝粒的3B型,即t3bl-1AS·1AL和t1as·3BL-3BS。使用与1A和3A上臂完全杂交的单拷贝寡聚文库探针L1A-1和L3A-1进行额外的FISH。实验结果证实了两条易位染色体的同一性(图。gydF4y2Ba5.gydF4y2Baf–h)。gydF4y2Ba
在辐射诱导的M中检测到染色体变体gydF4y2Ba1.gydF4y2Ba花生品种四里红(SLH)161-1a植株经序列荧光原位杂交/基因组原位杂交(FISH/GISH)鉴定。gydF4y2Ba一个gydF4y2Ba–gydF4y2BadgydF4y2BaSLH中顺序鱼/ GISH的结果(gydF4y2Ba一个gydF4y2Ba–gydF4y2BabgydF4y2Ba)和MgydF4y2Ba1.gydF4y2Ba植物161-1A(gydF4y2BacgydF4y2Ba–gydF4y2BadgydF4y2Ba),使用复用#3(绿色)和复用#4(红色)(a和c);gydF4y2BaA.硬脑膜gydF4y2Ba基因组DNA (b和d;绿色);gydF4y2Ba答:ipaensisgydF4y2Ba基因组DNA (b和d;红色)。gydF4y2BaegydF4y2BaSLH和161-1a的核型。gydF4y2BafgydF4y2Ba–gydF4y2BaggydF4y2Ba使用探针库L1A-1(绿色)和L3A-1(红色)顺序鱼/ GISH。gydF4y2BahgydF4y2Ba161-1a易位染色体。SSON用Multiplex #3(绿色)和Multiplex #4(红色)表示FISH核型;GISH表示FISH染色体核型gydF4y2BaA.硬脑膜gydF4y2Ba(绿色)和gydF4y2Ba答:ipaensisgydF4y2Ba(红色)。比例尺:10 微米gydF4y2Ba
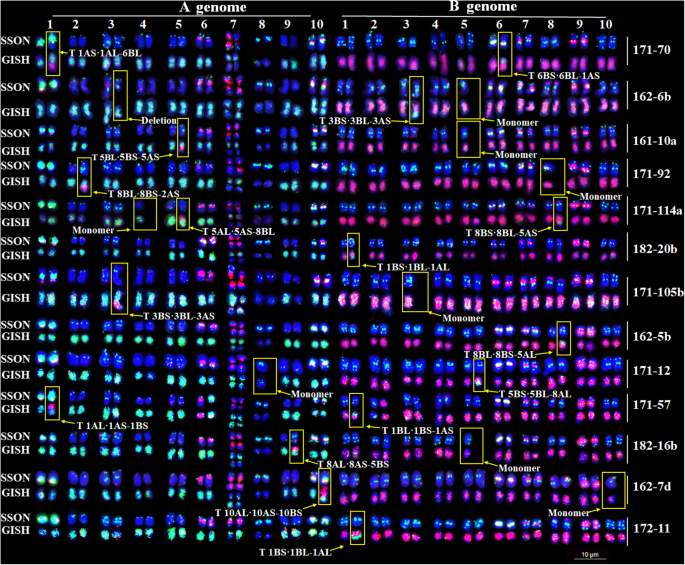
共有13种植物被鉴定出17个易位,1个缺失和8个单体染色体(图。gydF4y2BaS5gydF4y2Ba).在鉴定的染色体变化中,在非同源染色体之间观察到同源染色体之间观察到八个易位。染色体1,3和5显示了更多的易位数(图。gydF4y2Ba6.gydF4y2Ba).gydF4y2Ba
讨论gydF4y2Ba
新型寡聚探针的应用gydF4y2Ba
TRs在植物基因组中占很大比例。在过去,DNA重复被认为是基因组的“垃圾”[gydF4y2Ba43.gydF4y2Ba].然而,近年来各种研究表明,TRs可以用来更好地了解进化历史和基因组组成[gydF4y2Ba44.gydF4y2Ba].TRs也作为控制异染色质形成的表观遗传信号的占位符,并可能在修复双链DNA断裂中发挥作用[gydF4y2Ba44.gydF4y2Ba,gydF4y2Ba45.gydF4y2Ba].gydF4y2Ba
在不同物种之间,许多重复的DNA元素在染色体上产生特定的分布模式。这些元素是植物染色体鉴定和细胞遗传学研究中最常见的探针来源[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba46.gydF4y2Ba,gydF4y2Ba47.gydF4y2Ba]. Du等人[gydF4y2Ba34gydF4y2Ba]基于花生基因组测序技术,开发了8个花生寡核苷酸探针gydF4y2BaA.硬脑膜gydF4y2Ba,建立了高分辨率的核型,并揭示了八个之间的基因组关系gydF4y2Ba花生gydF4y2Ba物种。然而,他们研究中使用的寡核苷酸探针既不是基因组特异性的也不是染色体特异性。此外,他们没有覆盖花生染色体的间隙区域。gydF4y2Ba
在本研究中,114个新的寡核苷酸覆盖了花生染色体的不同区域。选择28个寡核苷酸,代表28种染色体分布模式,并将其定位到花生染色体上。8个寡核苷酸可能位于次级缩窄区、间质区或末端区;4例为B染色体特异性,1例为染色体特异性。这些结果表明,如果使用不同的寡聚探针组合,这些探针可能有助于以相当高的分辨率精确鉴定花生染色体易位。与以前报道的标记比较,如反转录转座子[gydF4y2Ba48.gydF4y2Ba]、45S和5srdna质粒克隆[gydF4y2Ba49.gydF4y2Ba[少数细菌人工染色体(BAC)克隆[gydF4y2Ba21gydF4y2Ba]新鉴定的28个寡核苷酸产生的信号覆盖了染色体的不同区域。此外,多重#3和多重#4分析的发展大大简化了FISH程序。这些新进展将有助于花生染色体工程的发展,并为今后的进一步应用提供支持。gydF4y2Ba
由于重复序列被认为是在不同的进化压力下进化而来的,所以通过FISH对重复序列进行比较制图可以促进系统发育分析[gydF4y2Ba33gydF4y2Ba]. 根据重复序列,推测了花生四倍体的基因组组成[gydF4y2Ba21gydF4y2Ba]51株A、B、K、F、E和H基因组之间的关系gydF4y2Ba花生gydF4y2Ba澄清了物种[gydF4y2Ba35gydF4y2Ba].此外,本研究中鉴定的28个寡核苷酸将为花生及其亲属的基因组进化研究提供有效的工具。gydF4y2Ba
基于基因组图谱的花生染色体组型揭示了花生重复序列分布的特殊性gydF4y2Ba
Tifrunner中的重复序列占组装基因组的74%[gydF4y2Ba36gydF4y2Ba].最近的花生基因组测序[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba]和花生染色体研究的进展[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba提供了有价值的信息。然而,参考基因组图和物理染色体核型之间的联系仍然缺乏。利用Tifrunner的参考序列,在硅定位寡核苷酸,并与产生的FISH核型进行比较。参考图中大部分重复序列位置和拷贝数与核型中FISH信号分布和强度相同或相似。据此,建立了Tifrunner的染色体核型,所有染色体按参考基因组图重新编号。gydF4y2Ba
此外,我们的发现表明,几个重复被不一致地定位与实际染色体和在硅序列。例如,tifi -89-3、tifi -155-5和tifi -198-1在更多的染色体中产生了FISH信号,而tifi -76-1在si中被定位到比我们的FISH实验所看到的更多的染色体位置。这些结果表明,Tifrunner参考序列的组装可能还需要进一步细化,特别是在TRs拷贝数较高的区域。在许多具有大基因组的物种中,高拷贝元件组装的模糊性是一个问题。目前的工作突出了进一步测序和组装花生基因组的需要。gydF4y2Ba
花生研究和育种染色体变体鉴定的重要性gydF4y2Ba
我们新开发的28个寡核苷酸的开发和物理映射大大改善了花生的核型分辨率,从而促进更有效地鉴定染色体变化。在本研究中,70个中的70个辐射诱导的mgydF4y2Ba1.gydF4y2Ba鉴定植物具有易迁移,缺失和单体染色体。这表明通过在花生染色体工程中辐射鉴定和诱导染色体突变的寡糖的潜力。染色体变异可用于易位或删除映射,如前所述,花生的辐射杂交映射和基因映射[gydF4y2Ba50.gydF4y2Ba,gydF4y2Ba51.gydF4y2Ba].gydF4y2Ba
此外,在常规育种中,部分同源染色体或异源染色体之间发生的易位促进了DNA在同源染色体之外的交换,从而产生新的有益性状。在未来的研究中,我们打算确定额外的易位和生产自交系易位创造纯合易位与有益的基因。本研究有助于花生的遗传改良。gydF4y2Ba
结论gydF4y2Ba
基于Tifrunner和gydF4y2Ba答:ipaensisgydF4y2Ba,开发114个新的重复序列寡核苷酸和染色体定位。它们分为28种类型,主要基于它们的位置和染色体的重叠信号。选择和与FAM和TAMRA共轭的总共28种代表28种类型的寡核苷酸。在Tifrunner参考序列中八个寡核苷酸的分布比较,构建了一种基于基于基于基于基于基于基于地图的鱼核型,其中将所有染色体重新编号为参考文献。目前的核型具有显着提高的分辨率,可以促进鉴定14染色体变化的SLH。然而,几个TRS产生了与Tifrunner的参考序列中的副本数字和位置不一致的信号。这表明Tifrunner的参考序列的组装仍然需要进一步改进。核型中的28个寡核苷酸的独特分布模式提供了可见的证据,以便校正此类轻微误差。因此,本新型探针和核型提供了对花生染色体工程和进化研究的有效工具。gydF4y2Ba
方法gydF4y2Ba
植物材料gydF4y2Ba
花生(gydF4y2BaA.海下gydF4y2Ba左,2gydF4y2Ban =gydF4y2Ba4x = 40)品种,Tifrunner和SLH,被河南作物分子育种,河南农业科学院,中国农业科学院。为了开发染色体变异,使用A的剂量为16 GyMa射线照射开花阶段的SLH植物gydF4y2Ba60.gydF4y2BaCo源(河南省科学院同位素研究所有限公司)。总共70米gydF4y2Ba1.gydF4y2Ba植物用于本研究。gydF4y2Ba
染色体准备gydF4y2Ba
染色体的制备,如杜等人所述[gydF4y2Ba52.gydF4y2Ba]稍作调整。当花生植株的根长到2 切下根尖,置于8-羟基喹啉溶液中3分钟 h在25 摄氏度。随后用一氧化二氮(NgydF4y2Ba2.gydF4y2BaO)在0.8-1.2MPa的压力下为1.5小时,并在25℃下以绝对乙醇 - 冰醋酸(3:1)溶液固定在12小时。然后将经处理的根尖端储存在冰箱中 - 20℃直至进一步使用。将顶点在45%冰醋酸中切除并崩解2-3分钟,压缩,然后在超低温度冰箱中冷冻在-80°C中24小时。将根材以绝对乙醇脱水12小时并对鱼干燥。gydF4y2Ba
寡聚探针的设计gydF4y2Ba
Tifrunner和gydF4y2Ba答:ipaensisgydF4y2Ba是从PeanutBase下载的(gydF4y2Bahttps://peanutbase.org/homegydF4y2Ba). 两个序列gydF4y2Ba答:ipaensisgydF4y2Ba使用tandem Repeats Finder (TRF, version 4.09)分析基因组以获得串联重复序列[gydF4y2Ba40gydF4y2Ba]基于Tang等人的方法[gydF4y2Ba31gydF4y2Ba]. 应用了以下参数:match = 2.不匹配 = 7.英代尔 = 7.匹配概率 = 80; indel概率 = 10; 最小得分 = 50; 最大周期 = 2000.周期大小> 4和DNA拷贝数 > 使用Python脚本获得了50个。串联重复序列> 用CD-HIT测定75%的同一性[gydF4y2Ba41.gydF4y2Ba]用于聚类并保持每个聚类的一致性。长度为40–45的寡头 然后根据串联重复序列和寡核苷酸7设计nt[gydF4y2Ba42.gydF4y2Ba].gydF4y2Ba
为了确定每个设计探针的有效性,首先将寡核苷酸物理地映射到染色体的参考序列上。每个寡核苷酸探针的序列在B2DSC2服务器上使用核苷酸基本局部比对搜索工具(blastn)与基因组比对。使用pident对blast结果进行过滤 = 90%和qcovhsp = 90%. 使用B2DSC中的Plot工具绘制物理位置信息。由通用生物系统公司(中国安徽)合成了具有丰富位点分布的寡糖。gydF4y2Ba
FISH和顺序FISH分析gydF4y2Ba
首先使用最终标记技术用Biotin-16-DUTP或Digoxigenin-11-DUTP标记寡核苷酸探针。包括10μl系统:2μl5×末端脱氧核苷酸转移酶(TDT)缓冲液;0.5μL数据;3.5μlddhgydF4y2Ba2.gydF4y2BaO;2μl CoClgydF4y2Ba2.gydF4y2Ba; 0.5 μl TDT酶;0.5 μl生物素-16-dUTP或地高辛-11-dUTP;和寡头(1 纳克/微升)1 微升;维持在37岁 °C持续15分钟 筛选后,获得28个寡核苷酸探针(表1)gydF4y2BaS2gydF4y2Ba). 通用生物系统公司(安徽,中国)用TAMRA或FAM对所有重复性寡核苷酸的5′端进行修饰,以进一步用于鱼类。为了建立染色体组型,我们研制了两种复合探针鸡尾酒,分别命名为复合3号和复合4号。复用#3包括FAM改性TIF-439、TIF-185-1、TIF-134-3和TIF-165-3;复用4包括TAMRA修饰的Ipa-1162、Ipa-1137、DP-1和DP-5。gydF4y2Ba
利用染色体A1和A3的基因组序列建立了两个单拷贝寡核苷酸文库(L1A-1和L3A-1)gydF4y2BaA.硬脑膜gydF4y2Ba根据Du等人描述的方法[gydF4y2Ba34gydF4y2Ba]. 每个单拷贝寡核苷酸文库来自每个染色体的远端区域。库由MYcroarray(美国密歇根州安阿伯)合成。根据MYcroarry\u MYtags标记方案,扩增得到的文库并用生物素-16-dUTP或地高辛-11-dUTP标记。gydF4y2Ba
鱼和顺序鱼类程序遵循Du等人描述的那些。[gydF4y2Ba34gydF4y2Ba]. 简言之,载玻片在70%甲酰胺中75%变性 70°C时 S杂交液,包括3 每支探针μl,变性13分钟 将载玻片浸入37℃的杂交溶液中 °C湿箱中至少12 h、 然后用2 × 生理盐水枸橼酸钠42 摄氏度。玻片用4′,6-二氨基-2-苯基吲哚(DAPI)染色,并用固定培养基固定。gydF4y2Ba
采用顺序FISH技术对寡核苷酸探针的信号进行定位,并将已测序的染色体与细胞学鉴定的染色体相关联。此后,使用重复或单拷贝寡聚探针进行FISH,然后在干燥前清洗以去除所有信号。GISH程序是用小鼠的总基因组dna进行的gydF4y2BaA.硬脑膜gydF4y2Ba和gydF4y2Ba答:ipaensisgydF4y2Ba以45S和5S rdna为探针进行FISH检测。gydF4y2Ba
捕获图像和分析gydF4y2Ba
FISH图像是用带冷却电荷耦合器件相机(徕卡)的徕卡DM6000荧光显微镜(徕卡)拍摄的。使用Adobe Photoshop对图像进行了对比度和亮度优化。每一种染色体增加约3-5个细胞,进行核型分析和染色体多样性分析。大多数核型是由单细胞发展而来,除非染色体有重叠。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
与本研究有关的所有数据均以表格和/或图表形式包含在手稿中,作者很高兴在合理要求下分享分析/原始数据和植物材料。支持本文结论的其他数据集包含在本文及其附加文件中。gydF4y2Ba
缩写gydF4y2Ba
- 益生元:gydF4y2Ba
-
寡核苷酸gydF4y2Ba
- 鱼:gydF4y2Ba
-
荧光原位杂交gydF4y2Ba
- TRs公司:gydF4y2Ba
-
串联重复gydF4y2Ba
- 家庭:gydF4y2Ba
-
6-carboxyfluoresceingydF4y2Ba
- 塔姆拉:gydF4y2Ba
-
6-羧丁苄甲基溴胺gydF4y2Ba
- 吉斯”:gydF4y2Ba
-
基因组原位杂交gydF4y2Ba
- SSON公司:gydF4y2Ba
-
单链寡核苷酸gydF4y2Ba
- 扶轮基金会:gydF4y2Ba
-
串联重复探测器gydF4y2Ba
- SLH:gydF4y2Ba
-
四里红gydF4y2Ba
- 美国银行:gydF4y2Ba
-
细菌人工染色体gydF4y2Ba
- NgydF4y2Ba2.gydF4y2Bao:gydF4y2Ba
-
一氧化二氮gydF4y2Ba
- TDT公司:gydF4y2Ba
-
末端脱氧核苷酸转移酶gydF4y2Ba
- 达皮:gydF4y2Ba
-
4',6-二氨基-2-苯基吲哚gydF4y2Ba
工具书类gydF4y2Ba
- 1.gydF4y2Ba
Bhala VP,Verma RC。伽玛射线在番茄中诱导染色体畸变(gydF4y2BaSolanum lycopersicum.gydF4y2Bal .)。染色体机器人。2018;12(4):86 - 90。gydF4y2Ba
- 2.gydF4y2Ba
浦j,王q,沉y,庄l,李c,tan m,bie t,chu c,qi z.染色体4j的物理映射gydF4y2Ba北偃麦草gydF4y2Ba利用伽马辐射引起的像差。中国生物医学工程学报,2015;gydF4y2Ba
- 3.gydF4y2Ba
张继,江y,郭y,李克,杨z,徐d,轩x鉴定鉴定新型染色体畸变gydF4y2Ba60.gydF4y2Ba有限公司-γ-射线在小麦gydF4y2Ba簇毛麦gydF4y2Ba行。国际化学学报,2015;16(12):29787-96。gydF4y2Ba
- 4.gydF4y2Ba
关键词:小黑麦,zebularine,染色体畸变,遗传变异基因组。2016;59(7):485 - 92。gydF4y2Ba
- 5。gydF4y2Ba
徐涛,边宁,温敏,肖军,袁超,曹安,张松,王旭,王华。一种普通小麦(gydF4y2Ba小麦gydF4y2Ba高分蘖矮化突变体。理论应用基因。2017;130(3):483–94.gydF4y2Ba
- 6。gydF4y2Ba
达尼洛娃电视,张克,刘开,弗雷贝B,吉尔BS。小麦条纹马赛克病毒和小麦马赛克病毒抗性基因WSM3的同优素重组转移和分子细胞遗传学映射gydF4y2Ba中间偃麦草gydF4y2Ba给小麦。理论应用基因。2017;130(3):549–56.gydF4y2Ba
- 7。gydF4y2Ba
Kynast RG、Okagaki RJ、Galatowitsch MW、Granath SR、Jacobs MS、Stec AO、Rines HW、Phillips RL。利用染色体加成和辐射杂交系对玉米基因组进行分析。美国国家科学院学报。2004;101(26):9921–6.gydF4y2Ba
- 8。gydF4y2Ba
高W,陈祖,约翰·ZY,Raska D,Kohel RJ,Womack Je,Betelly DM。宽交叉全基因组辐射混合型棉花(gydF4y2Bagossypium hirsutumgydF4y2Bal .)。遗传学。2004;167(3):1317 - 29。gydF4y2Ba
- 9。gydF4y2Ba
联合国粮食及农业组织。gydF4y2Bahttp://www.fao.org/faostat/en/#data/QCgydF4y2Ba. 访问20;2020年8月。gydF4y2Ba
- 10gydF4y2Ba
Mbaye G、Soumboundou M、Diouf L、Ndong B、Djiboune AR、Sy PM、Dieng SM、Diouf M、Diouf NN、Barry A。γ射线辐照花生籽粒对栽培效果的评价。打开J Biophys。2017;7(3):94–100.gydF4y2Ba
- 11gydF4y2Ba
Hake AA,Shirasawa K,Yadawad A,Nayak SN,Mondal S,Badigannavar AM,Nadaf HL,Gowda M,Bhat RS.从花生独立突变体关联小组鉴定与产量和品质性状相关的转座因子标记(gydF4y2Baarachis hypogaea.gydF4y2BaL.)。大提琴。2017;213(12):283.gydF4y2Ba
- 12.gydF4y2Ba
Joshi P,Jadhav MP,Shirasawa K,Yadawad A,Bhat RS.花生导入系叶片抗病性和生产性突变体(gydF4y2Baarachis hypogaea.gydF4y2Ba). 植物品种。2020;139(1):148–55.gydF4y2Ba
- 13.gydF4y2Ba
Nadaf HL、Biradar K、Murthy G、Krishnaj PU、Bhat RS、Pasha MA、Yerimani AS。油酰PC去饱和酶的新突变(gydF4y2BaahFAD2B型gydF4y2Ba)从γ射线诱导的花生高油分突变体中鉴定出来。作物科学。2017;57(5):2538 - 46。gydF4y2Ba
- 14.gydF4y2Ba
万丽,李乙,雷毅,严丽,任×,陈毅,戴×,江H,张J,郭W,陈甲,廖乙。突变转录组测序提供了花生荚果发育的信息(gydF4y2Baarachis hypogaea.gydF4y2BaL.)。前面。植物科学。2017;8:1900.gydF4y2Ba
- 15.gydF4y2Ba
万L,李B,Pandey Mk,Wu Y,Lei Y,Yan L,Dai X,江H,张J,Wei G,varshney rk,廖b.对生理调节机制新花生种子涂层突变体的转录组分析参与种子涂层裂纹和色素沉着。前植物SCI。2016; 7:1941。gydF4y2Ba
- 16。gydF4y2Ba
关键词:花生,半矮秆突变体,转录组分析,分子机制BMC基因组学。2020;21(1):1 - 16。gydF4y2Ba
- 17。gydF4y2Ba
Pierozzi NI,Galgaro M,L,Lopes CR.C-带在两种情况下的应用gydF4y2Ba花生gydF4y2Ba野生物种,gydF4y2Ba多年生花生gydF4y2BaKrapov。w。c。格雷戈里和gydF4y2BaA. Villosulicarpa.gydF4y2BaHoehne到有丝分裂染色体分析。鹅圈炎。2001; 54(4):377-84。gydF4y2Ba
- 18。gydF4y2Ba
Seijo G、Lavia GI、Fernández A、Krapovickas A、Ducasse DA、Bertioli DJ、Moscone EA。栽培花生的基因组关系(gydF4y2Baarachis hypogaea.gydF4y2Ba双GISH技术揭示的豆科植物及其近缘种。我是J Bot。2007;94(12):1963–71.gydF4y2Ba
- 19。gydF4y2Ba
Robledo G、Seijo G。花生野生B基因组的种间关系(第1节)gydF4y2Ba花生gydF4y2Ba)基于RDNA基因座和异铬胺检测的鱼类测绘:基因组排列的新提案。Al Appl Genet。2010; 121(6):1033-46。gydF4y2Ba
- 20。gydF4y2Ba
Bertioli DJ, Vidigal B, Nielen S, Ratnaparkhe MB, Lee TH, Leal-Bertioli SCM, Kim M, Guimarães PM, Seijo G, Schwarzacher T, Paterson AH, Heslop-Harrison P, Araujo ACG。花生基因组的重复成分(gydF4y2Baarachis hypogaea.gydF4y2Ba)以及它在重构基因间序列空间中的作用。安·博特。2013;112:545–59.gydF4y2Ba
- 21gydF4y2Ba
张璐,杨晓,田璐,陈璐,于伟花生的鉴定(gydF4y2Baarachis hypogaea.gydF4y2Ba)使用荧光的染色体gydF4y2Ba原位gydF4y2Ba杂交系统揭示了四倍体花生形成过程中发生的多重杂交事件。新植醇。2016;211(4):1424 - 39。gydF4y2Ba
- 22gydF4y2Ba
Do Nascimento EFDM、Dos Santos BV、Marques LOC、Guimarães PM、Brasileiro ACM、Leal Bertioli SCM、Bertioli DJ、Araujo ACG。人类的基因组结构gydF4y2Baarachis hypogaea.gydF4y2Ba(Linnaeus,1753)和分子细胞遗传学揭示的诱导的花生异源四倍体。复合细胞原。2018;12(1):111–40.gydF4y2Ba
- 23gydF4y2Ba
姜杰。荧光gydF4y2Ba原位gydF4y2Ba植物杂交:最新发展和未来的应用。Chromosom杂志2019;27(3):153 - 65。gydF4y2Ba
- 24.gydF4y2Ba
杜平,庄立,王勇,袁立,王Q,王D,达瓦东杜,沈杰,徐宏。快速准确鉴定小麦和小麦的寡核苷酸及复合探针的研制gydF4y2Ba北偃麦草gydF4y2Ba染色体。基因组。2017; 60(2):93-103。gydF4y2Ba
- 25.gydF4y2Ba
关键词:ND-FISH,黑麦,小麦,寡核苷酸探针Sci众议员2015;5(1):10552。gydF4y2Ba
- 26.gydF4y2Ba
唐S,邱L,肖Z,付S,唐Z。用于大麦染色体鉴定和小麦染色体多态性研究的ND-FISH寡核苷酸探针。基因巴塞尔。2016;7(12):118.gydF4y2Ba
- 27。gydF4y2Ba
朱明、杜平、庄丽、朱聪、赵浩、齐兹。利用单链寡核苷酸探针建立了一种简单有效的非变性FISH玉米染色体分化方法。基因组。2017;60(8):657–64.gydF4y2Ba
- 28。gydF4y2Ba
刘x,太阳,吴y,周y,顾s,yu h,yi c,ug m,姜j,刘b,张t,锣z.双色寡核鱼可以揭示染色体变化和进化gydF4y2Ba野生稻gydF4y2Ba物种。植物J。2019;101:112–21.gydF4y2Ba
- 29。gydF4y2Ba
Braz GT、He L、Zhao H、Zhang T、Semrau K、Rouillard J、Torres GA、Jiang J。比较寡核苷酸FISH作图:揭示核型和染色体进化的有效方法。遗传学。2018;208(2):513–23.gydF4y2Ba
- 30.gydF4y2Ba
韩义,张t,泰国译,翁y,姜j.染色体特定绘画gydF4y2Bacucumis.gydF4y2Ba使用大量寡核苷酸的物种。遗传学。2015;200(3):771–9.gydF4y2Ba
- 31。gydF4y2Ba
关键词:小麦,串联重复序列,物理定位,染色体鉴定足底。2018;249:663 - 75。gydF4y2Ba
- 32。gydF4y2Ba
唐S,唐Z,秋l,杨z,李g,郎t,朱武,张j,富科。开发新的寡核苷酸探针区分特定染色体段和a,b,d小麦的a,b,d基因组(gydF4y2Ba小麦gydF4y2BaL.)使用ND-Fish。正面。植物SCI。2018; 9:1104。gydF4y2Ba
- 33gydF4y2Ba
江J,吉尔理学学士。荧光技术的现状与展望gydF4y2Ba原位gydF4y2Ba杂交(FISH)在植物基因组研究中的应用。基因组。2006;49(9):1057 - 68。gydF4y2Ba
- 34gydF4y2Ba
杜平、李丽、刘H、傅丽、秦丽、张Z、崔C、孙Z、韩S、徐杰、戴X、黄B、董W、唐F、庄L、韩Y、齐Z、张X。利用重复和单拷贝寡核苷酸绘制花生高分辨率染色体,鉴定结构重排和基因组分化。BMC植物生物学。2018;18(1):240–53.gydF4y2Ba
- 35gydF4y2Ba
杜平,崔C,刘H,傅L,李L,戴X,秦L,王S,韩S,徐J,刘B,黄B,唐F,董W,齐Z,张X。寡核苷酸染色液的开发有助于花生染色体的高通量和经济高效的鉴定。植物方法。2019;15(1):69.gydF4y2Ba
- 36.gydF4y2Ba
Bertioli DJ,詹金斯J, Clevenger J, Dudchenko O,高D, Seijo G, SCM是,任L,农民广告,Pandey可,Samoluk党卫军,B,阿伯纳西Agarwal G, Ballen-Taborda C,卡梅伦C,坎贝尔J,查瓦罗C, Chitikineni,楚Y,破折号,El Baidouri M,郭B,黄W, Kim KD Korani W, Lanciano年代,他CG, Mirouze M, Moretzsohn MC,范教授M,胫骨JH, Shirasawa K,Sinharoy S, Sreedasyam A, Weeks NT, Zhang X, Zheng Z, Sun Z, Froenicke L, Aiden EL, Michelmore R, Varshney RK, Holbrook CC, EKS C, Scheffler BE, Grimwood J, Ozias-Akins P, Cannon SB, Jackson SA, Schmutz J.花生异源四倍体基因组序列gydF4y2Baarachis hypogaea.gydF4y2Ba.NAT Genet。2019; 51(5):877-84。gydF4y2Ba
- 37.gydF4y2Ba
壮族W,陈H,杨M,王J, Pandey可,张C, Chang W,张L,张X,唐R,加戈V,王X,唐H, Chow C,王J,邓Y,王D,汗啊,杨问,Cai T, Bajaj P,吴K,郭B,张X,李J,梁F,胡锦涛J,廖B,刘年代,Chitikineni,燕H,郑Y,山,刘问,谢D,王Z,汗撒,阿里N,赵C,李X, Z,张,壮族R,彭Z,王年代Mamadou G,壮族Y, Z, Yu W,熊F,关丽珍W元M,李Y,邹H,夏H,咋L,风扇J, Yu J,谢W元J, K,赵年代,楚W,陈Y,太阳P,孟F,卓T,赵Y,李C、G,赵Y,王C, Kavikishor铅、锅R,帕特森啊,王X,明R, Varshney家乡。栽培花生基因组为研究豆科植物核型、多倍体进化和作物驯化提供了新思路。51 Nat麝猫。2019;(5):865 - 76。gydF4y2Ba
- 38.gydF4y2Ba
陈X,陆Q,刘H,张J,洪Y,兰H,李H,王J,刘H,李S,潘迪MK,张Z,周G,于J,张G,袁J,李X,文S,孟F,于S,王X,锡迪克KHM,刘Z,帕特森AH,瓦尔什尼RK,梁X。栽培花生的测序,gydF4y2Baarachis hypogaea.gydF4y2Ba,揭示了基因组进化和石油改良。摩尔植物。2019;12(7):920–34.gydF4y2Ba
- 39。gydF4y2Ba
Bertioli DJ、Cannon SB、Froenicke L、Huang G、Farmer AD、EKS C、Liu X、Gao D、Clevenger J、Dash S、Ren L、Moretzohn MC、Shirasawa K、Huang W、Vidigal B、Abernathy B、Chu Y、Niederhuth CE、Umale P、ACG A、Kozik A、Do Kim K、Burow MD、Varshney RK、Wang X、Zhang X、Barkley N、Guimarães PM、Isobe S、Guo B、Liao B、Stalker HT、Schmitz RJ、,谢夫勒BE、SCM L-B、Xun X、Jackson SA、Michelmore R、Ozias Akins P。人类的基因组序列gydF4y2Ba落花生duranensisgydF4y2Ba和gydF4y2Baarachis ipaensis.gydF4y2Ba栽培花生的二倍体祖先。纳特·吉内特。2016;48(4):438.gydF4y2Ba
- 40.gydF4y2Ba
Benson G. Tandem重复发现:分析DNA序列的程序。核酸RES。1999年; 27(2):573-80。gydF4y2Ba
- 41.gydF4y2Ba
Li W,Godzik A. CD-his:用于聚类和比较大套蛋白质或核苷酸序列的快速计划。生物信息学。2006; 22(13):1658-9。gydF4y2Ba
- 42.gydF4y2Ba
赖奇利克W。oligo7引物分析软件。方法分子生物学。2007;402:35.gydF4y2Ba
- 43。gydF4y2Ba
奥格尔,克里克。自私的DNA:终极的寄生虫。自然。1980;284(5757):604–7.gydF4y2Ba
- 44。gydF4y2Ba
Gemayel R,Vinces Md,Legendre M,Verstrepen KJ。变量串联重复加速编码和调节序列的演变。Annu Rev Genet。2010; 44:445-77。gydF4y2Ba
- 45。gydF4y2Ba
Gemayel R, Cho J, Boeynaems S, Verstrepen KJ。除了垃圾可变串联重复序列作为调节和编码序列快速进化的促进因子。Genes-Basel。2012; 3(3): 461 - 80。gydF4y2Ba
- 46gydF4y2Ba
Kato A,Lamb Jc,Birchler Ja。使用重复DNA序列的染色体涂料作为玉米体细胞染色体鉴定的探针。Proc Natl Acad Sci U S A. 2004; 101(37):13554-9。gydF4y2Ba
- 47gydF4y2Ba
黄X,朱M,庄L,张S,王J,陈X,王D,陈J,包Y,郭J,张J,冯Y,楚C,杜P,齐Z,王H,陈P。中国小麦品种染色体结构重排和多态性的高分辨多重寡核苷酸FISH鉴定。理论应用基因。2018;131(9):1967–86.gydF4y2Ba
- 48.gydF4y2Ba
Nielen S、Campos Fonseca F、Leal Bertioli S、Guimarães P、Seijo G、Town C、Arrial R、Bertioli D。FIDEL-a类逆转录病毒转座子及其在栽培花生a、B基因组组分中的独特进化史。染色体研究,2010年;18(2):227–46.gydF4y2Ba
- 49.gydF4y2Ba
Robledo G,Lavia GI,Seijo G。从rDNA基因座的FISH定位和异染色质检测揭示具有基因组的野生花生种间的种间关系。理论应用基因。2009;118(7):1295–307.gydF4y2Ba
- 50gydF4y2Ba
Tiwari vk,Heesacker A,Riera-Lizarazu O,Gunn H,Wang S,Wang Y,Gu YQ,Paux E,Koo D,Kumar A,Luo M,Lazo G,Zemetra R,Akhunov E,Friebe B,Poland J,Gill BS,Kianian S,Leonard JM。一种全基因组,六倍体小麦的辐射杂交映射资源。工厂J. 2016; 86(2):195-207。gydF4y2Ba
- 51gydF4y2Ba
Masoudi Nejad A,Nasuda S,Bihoreau M,Waugh R,Endo TR.大麦大规模基因组分析的辐射杂交图谱的替代方法。分子基因组学。2005;274(6):589–94.gydF4y2Ba
- 52.gydF4y2Ba
杜平、李丽、张Z、刘H、秦L、黄B、董W、唐F、齐Z、张X。端粒重复序列的染色体绘制揭示了花生基因组进化的新证据。综合农业杂志。2016;15(11):2488–96.gydF4y2Ba
致谢gydF4y2Ba
感谢南京南京农业大学增俊琦教授;祖俊阳教授,中国电子科技大学成都;舒兰付教授和成都四川农业大学的舒耀堂博士,他们都在帮助设计寡核苷酸。gydF4y2Ba
基金gydF4y2Ba
本研究由河南省农业科学院杰出青年基金(2020JQ03)资助;河南省农业科学院科技发展专项资金(2020CY07);国家自然科学基金(31801397);中国农业研究系统(CARS-13)。资助机构在研究的设计、数据的收集、分析和解释以及手稿的撰写中没有扮演任何角色。gydF4y2Ba
作者信息gydF4y2Ba
隶属关系gydF4y2Ba
贡献gydF4y2Ba
XZ,PD,BH和WD设计了实验;LF,PD,QW,LL,TL,SW,JG,ZS和SH执行探头设计和鱼类,并分析了数据;XZ和PD写了稿件。所有作者都读过并批准了稿件。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
不适用。gydF4y2Ba
同意出版gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
提交人声明他们没有竞争利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
斯普林格自然保持中立,就管辖权的要求,在出版的地图和机构的联系。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:图S1。gydF4y2Ba
Oligo TIF-165-3(A),DP-1(B),TIF-439(C),TIF-185-1(D),TIF-134-3(E),DP-5(F.)的物理映射),使用B2DSC服务器中的绘图工具,IPA-1162(G)和IPA-1137(H)。染色体地块上的绿色,红色和蓝线表示寡核苷酸序列的拷贝数;线的长度对应于份数。gydF4y2Ba
附加文件2:图S2。gydF4y2Ba
使用染色体特异性寡核桃文库L1A-1(A,RIG)和L3A-1(D,RED),顺序荧光原位杂交/基因组原位杂交(鱼/ GIRH);SSON探针多路复用#3(绿色)和多路复用#4(红色)(B和E);和总基因组DNAgydF4y2BaA.硬脑膜gydF4y2Ba(绿色)和gydF4y2Ba答:ipaensisgydF4y2Ba(红色)(c和f)在品种中。(a)中的绿色和红色箭头分别表示染色体A1和B1;(d)中的绿色和红色箭头分别显示了染色体A3和B3。比例尺:10 μm。gydF4y2Ba
附加文件3:图S3。gydF4y2Ba
以TAMRA-Ipa-1162和TAMRA-Ipa-1137 (a,红色)和质粒克隆45S rDNA (b,红色)为探针进行序列FISH。比例尺:10 μm。gydF4y2Ba
附加文件4:图S4。gydF4y2Ba
使用B2DSC服务器中的绘图工具对oligo-TIF-89-3(a)、TIF-155-5(b)、TIF-198-1(c)、TIF-359-3(d)、TIF-76-1(e)和Ipa-1757(f)进行物理绘图。染色体图上红线的长度表示寡核苷酸序列的拷贝数。gydF4y2Ba
附加文件5:图S5。gydF4y2Ba
辐射诱发小鼠染色体畸变变异的研究gydF4y2Ba1.gydF4y2Ba以花生品种思里红(SLH) 161-1a为试材,进行FISH/GISH序列分析。FISH使用Multiplex #3 (a, c, e, g, i, k, m, o, q, s, u, w和y);吉斯”使用gydF4y2BaA.硬脑膜gydF4y2Ba基因组DNA(绿色)和gydF4y2Ba答:ipaensisgydF4y2Ba基因组DNA(红色)(b, d, f, h, j, h, j, l, n, p, r, t, v, x和z),比例尺:10 μm。gydF4y2Ba
附加文件6:表S1。gydF4y2Ba
来自品种TIFRUNNER的新oligo探针的细节gydF4y2Ba答:ipaensisgydF4y2Ba.gydF4y2Ba
附加文件7:表S2。gydF4y2Ba
寡探针分类和染色体信息。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.“创作共用公共领域”豁免书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条提供的数据,除非信用额度中另有规定。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
傅,L.,王,Q.,李,L。gydF4y2Baet al。gydF4y2Ba重复性寡核苷酸的物理作图有助于建立基于基因组图的核型,以鉴定花生的染色体变异。gydF4y2BaBMC植物BIOL.gydF4y2Ba21,gydF4y2Ba107 (2021). https://doi.org/10.1186/s12870-021-02875-0gydF4y2Ba
已收到gydF4y2Ba:gydF4y2Ba
认可的gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-021-02875-0gydF4y2Ba
关键字gydF4y2Ba
- 花生gydF4y2Ba
- TRS.gydF4y2Ba
- 寡头gydF4y2Ba
- 鱼gydF4y2Ba
- 核型gydF4y2Ba
- 染色体变体gydF4y2Ba
- 参考序列gydF4y2Ba