抽象的
背景
转录研究有一个良好注释的基因组联合已经为分子过程的新的理解奠定了基础。工具,可视化基因表达模式进一步添加到这些资源。的手动注释猕猴桃对(猕猴桃)的基因组产生了一组高质量的33044个基因。在这里,我们通过电子荧光象形文字(eFP)浏览器观察不同组织中的基因表达模式,利用网络分析研究转录因子(TF)表达的关系。
结果
我们选择了61个不同发育时间点的不同组织样本进行RNA-seq分析,并生成了一个eFP浏览器来可视化该数据集。代表57个不同类别的2839个助教被识别并命名。通过对TF表达模式的网络分析,将TF分为14个不同的模块。两个模块共237个转录因子与花芽和花的发育有关,另外两个模块共160个转录因子与果实的发育和成熟有关。480个TFs与乙烯诱导的果实成熟有关。与花和果实发育相关的3个“枢纽”基因包括雌蕊发育中心的一个HAF-like基因、一个ERF和一个DOF基因。成熟和成熟中心基因包括一个与种子成熟相关的KNOX基因和一个类grass的TF。
结论
本研究为了解花和果实发育的转录调控复杂性提供了新的思路,同时也为植物群落提供了新的资源。actindia eFP浏览器以一种可访问的格式提供,允许研究人员下载并在内部工作。
背景
全球转录组学方法是一个常用的工具,以获得更好的了解基因功能和调控。转录组的组成是染色质状态、转录因子(TFs)激活基因表达和转录降解速度之间动态平衡的结果。良好的基因组信息和健壮的基因模型的结合为系统和一致的基因和基因家族命名铺平了道路。它结合了其他基因组学工具,如电子荧光象形文字(eFP)浏览器[1以帮助观察基因在何处表达,从而更快地识别不同物种的基因功能。迄今为止,eFP浏览器已经在拟南芥等植物中成功开发出来[1,番茄2),草莓3.,菠萝[4].
TFS是基因组中最大的基因组之一;在拟南芥中,有超过1500个TFS描述的,属于许多不同类别,代表所有基因的5%[5].在其他物种中,转录因子占编码基因的3-5%,其功能通常跨物种保守[6].课程分为57个不同的类别[5有些类具有多种类型的DNA结合域。TF的每一类都由一个基因家族来代表。这些基因家族的大小因物种而异,这取决于个体基因和基因组复制等事件,导致某些或大多数家族的扩展[6].在高等植物中,MYB、bHLH和锌指类TF含有数百个成员[6].有大量的例子表明TF一级蛋白结构在物种间的强大进化维持,同源基因具有类似的基因功能。这使得研究人员可以通过同源性来预测功能[7].
包含TF的MADS-box可以说是TF理解最好的类别之一。MADS-box基因家族的成员,包括众所周知的花器官结构ABCE TFs,决定了植物发育的许多方面[8,9].尽管被子植物的子实体是同源的,肉质果实在许多植物科中进化了无数次,但这些基因的功能似乎是保守的[10].被子植物的花结构和子实体非常保守,有轮生的萼片、花瓣、雄蕊和心皮[8].MADS蛋白序列在植物中也很保守,在许多物种中显示了类似的控制机制[7,11].
猕猴桃是猕猴桃科它是埃里卡莱斯的一个基本家庭[12,并包含属猕猴桃包括许多经济上重要的水果品种,如猕猴桃对var。deliciosa(绿色的猕猴桃),答:对var。对(金色和红色的猕猴桃)和答:arguta(猕猴桃或猕猴桃)。绿色的“海沃德”猕猴桃是六倍体,而商业发行的黄色果肉品种答:对var。对“Hort16A”,红色的肉答:对var。对“红羊”是大果二倍体基因型,这使它们成为了解红羊的分子过程的理想材料猕猴桃科.最近,一个新的丁香假单胞菌光伏.actinidiae(PSA.)耐受性四倍体金品种‘Zesy002’已在市场上取代‘Hort16A’。这两个二倍体品种已被用于理解许多发育方面的分子控制,包括开花、果实成熟、颜色和风味发育[13,14,15,16].基因组学工具,如CRISPR基因编辑已经成功地用于编辑“Hort16A”中的花阻遏物,以创建一个小型的结果植物,可用于快速测试水果中的基因功能,进一步建立它们的效用[17].
第一个猕猴桃基因组草图是答:对《红阳》,2013年出版[18],为基因组学铺平道路猕猴桃科.最近答:对一个更近交相关基因型Red5的基因组,进一步完善了基因模型的构建和重要的人工注释[19].猕猴桃基因组的手动标注改善了出版计算机预测基因模型的质量,并为今后的基因挖掘优质资源。在这里,我们通过确定TF的基因,在若干组织中分析它们的表达并提供EFP浏览器工具来分析基因表达的基础上这些数据。
结果
转录因子基因家族的挖掘
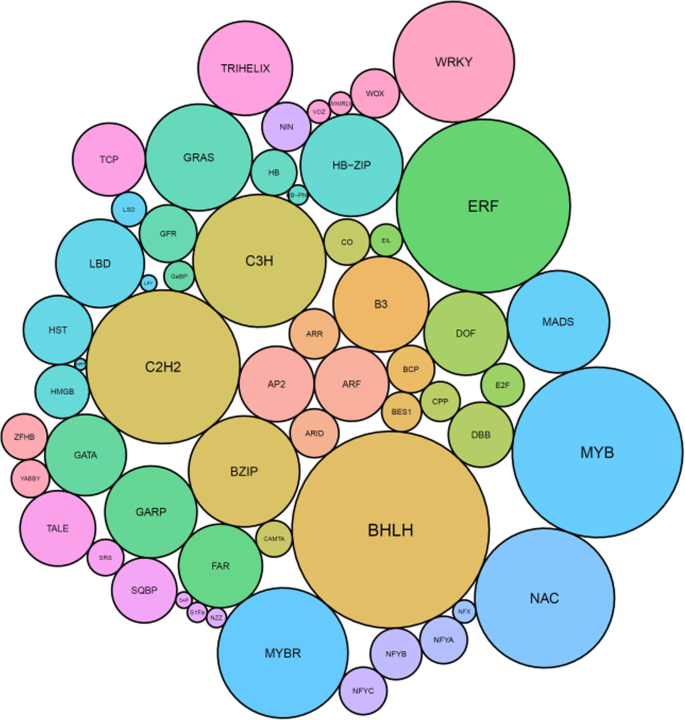
使用猕猴桃手动注释基因模型[19,含有InterPro DNA结合域的蛋白质翻译(http://www.ebi.ac.uk/Interpro.)被确定。经过人工检查,在32个全局类中的61个TF类中,有2839个基因模型至少有一个TF域(图)。1、表1,额外的数据1).全球猕猴桃TFs中最丰富的类群是锌指类群,有11个不同基因类群中的571个基因,其次是4个不同基因类群中的428个MYB结构域的基因,以及2个不同基因类群中的333个bHLH基因(见表)1,额外的数据1).总共2839个TFS在Kiwifruit基因组中表示8.6%的注释基因。
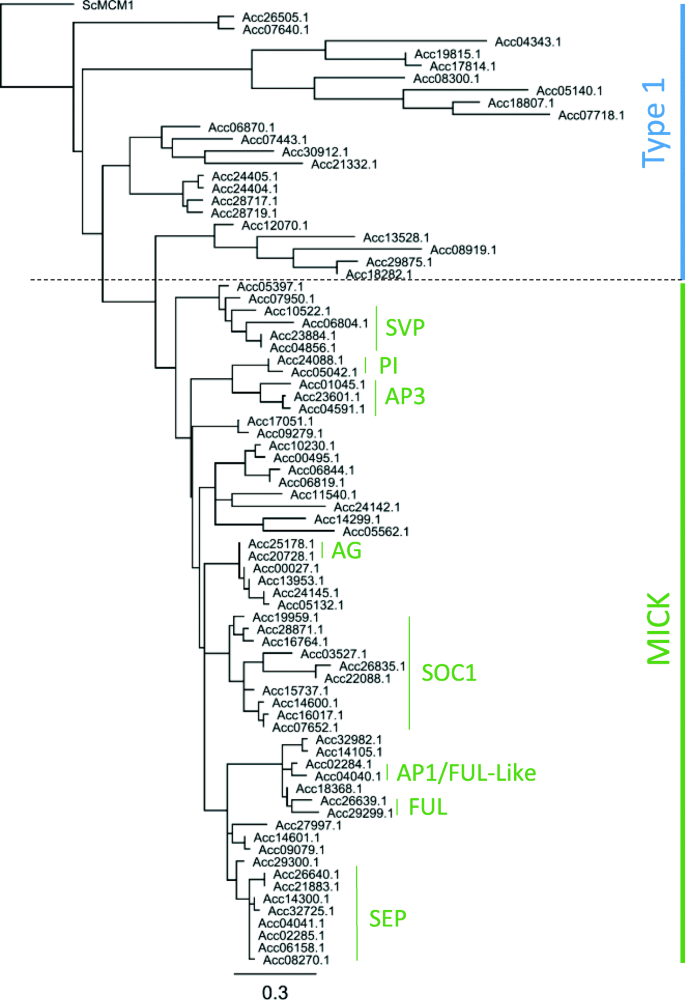
利用DNA结合域,每个TF类在系统发育树中对齐,并使用以下标准命名。首先,如果这个基因之前已经在文献中发表过,就用这个名字。对于以前未发表过的基因,使用的是按照最初的系统发育树的顺序命名。这种命名方法允许亚分支内的基因有相互接近的数量。ARF基因是一个例外,在许多物种中整个家族已被很好地描述[20.,21,22],因此一个与拟南芥和番茄同系物最近的命名约定[22]。MADS TF Type 1和MICK聚类的示例聚类如图所示。2完整的系统发育可以在其他数据中找到2.在所有的基因家族,还有通常在每个分支与一致的观察两个密切相关的基因报告基因组复制[19].
在猕猴桃中,已有四类转录因子的报道,其中两类(R2R3类MYBs [23]及wrkyy [24])是基于先前版本的猕猴桃基因组中,第三(米克型MADS-box基因)使用EST序列数据[出版25]第四(AP2 / ERF类别)基于手动注释的基因组[26].另外两种基因家族,R2R3 MYB类和NAC类TFS是单独研究的主题,并在更多的深度上报道(Rodrigues等人。提交,Nieuwenhuizen等。
96个已发表的WRKY转录因子以前是根据序列染色体位置命名的。其中5个没有Acc注释的基因模型,2个模型(WRKY95和WRKY96)似乎是剪接变体。这5个样本使用Web Apollo软件进行了注释,并分配了Acc编号。这项研究还发现了另外21个WRKY基因和这些新基因被顺序编号,使总数达到116个WRKY基因。
Varkonyi-Gasic等报道了9个MADS-box基因的EST序列和全长序列的综合分析[25].自从这项研究以来,有四个高级副总裁就像基因(27)和八个SOC1就像基因(28的报道。进一步挖掘发现了58个包含MADS-box DNA结合域的预测基因模型。MADS基因分为两个主要分支;Type 1和MICK类型。此前,MICK型MADS-box基因已被证明是植物发育的关键调控因子,特别是在花和果实发育中。系统发育比对鉴定出与特征良好的MICK-MADS基因高度相似的序列,并鉴定出可能的同源对:无性生殖的(AG))这样的基因,Acc25178.1(MADS28) 和ACC20728.1.(MADS29);PISTILLATA(π)这样的基因,Acc24088.1(MADS11) 和ACC05042.1.(MADS12);和APETALA1(AP1)这样的基因,Acc04040.1(MADS40) 和Acc02284.1(MADS41) (图。2).
表达分析
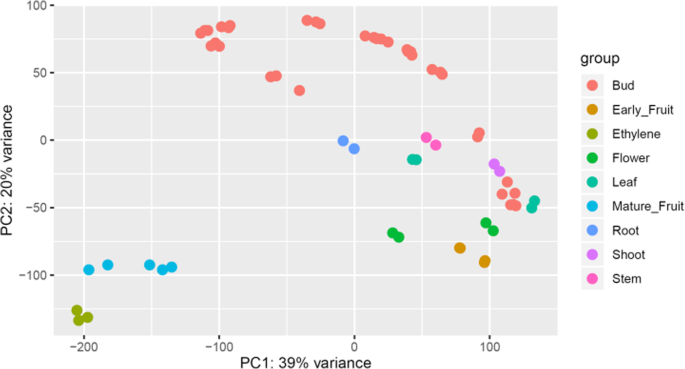
为了确定每个转录因子在何时何地表达,我们采用了转录组学方法。不同组织和不同植物发育阶段的基因表达答:对var。对,对金果‘Hort16A’和‘Zesy002’进行了测量。来自根、茎、芽、叶、花和早期果实发育的61组RNA-seq与来自果实发育的RNA-seq reads相结合[15]及采后[15和芽发育系列(vogd等人在准备中)(附加数据3.,4).主成分分析(PCA)显示成熟果实和乙烯处理的果实从其他组织中分离出来(图。3.).提取TFS的归一化表达模式。基于每百万(TPM)值的转录物,发现大多数TFS在其中一个数据集中具有至少一个RNA-SEQ,只有47(1.67%)在单个组织中没有读数。
猕猴桃eFP浏览器的开发
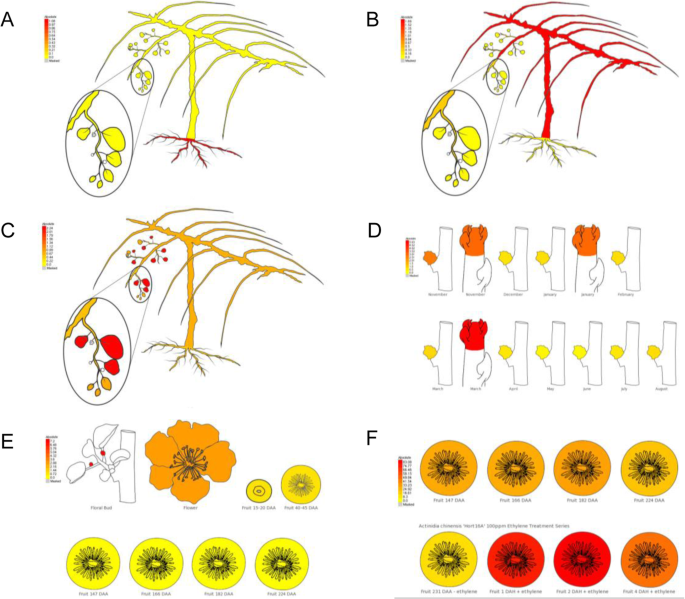
为了便于可视化每个基因的表达模式,我们开发了一个涵盖不同组织和发育阶段的所有基因模型的eFP浏览器。在MADS基因中,以根为主表达MADS12(Acc10230.1) 和MADS19(Acc00495.1) (图。4一种。)。在模式生物功能分析一致,MADS19显示出最接近的同源性AtAGL21(At4g37940.1),调控拟南芥侧根的发育。第二个MADS基因,MADS45(Acc27997.1)显示出主要是茎和甘蔗表达(图4B)和表现出最高的同源性AGL6,在拟南芥中负调控FLC/MAF分支基因并正调控FT的花启动子[29].MADS77(Acc08919.1)显示叶特异性表达(图。4c)。SVP1(MADS6.-Acc10522.1)在芽中表达良好(图。4D)和具有高度同源性高级副总裁拟南芥基因(At2g22540).花的主要表现无性生殖的喜欢基因MADS29(ACC20728.1.) (图。4E)被观察到,而RIN / SEP4[15)基因MADS52(Acc26640.1) (图。4F)表现为采后表达。
网络分析
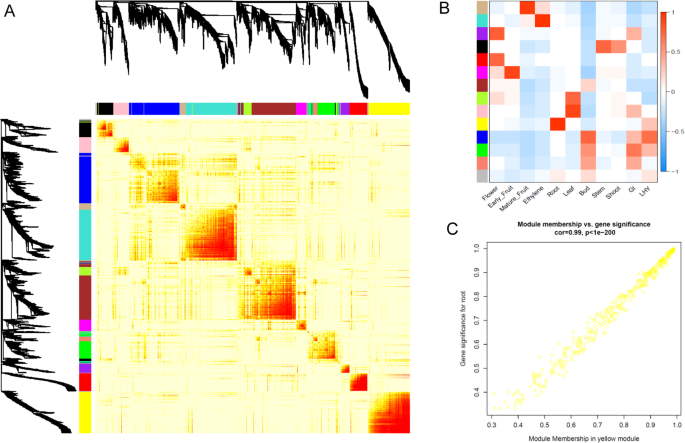
加权基因共表达网络分析[30.]将2773个表达的TFs分配到14个不同的模块颜色组(图。5a).当这些与不同组织类型进行比较时,某些模块与不同组织之间存在明显的相关性(图2)。5b).考虑到一些组织可能是在一天的不同时间采集的,两个昼夜节律基因(一个早晨MYB相关基因,LHY[31] -MYBR92 Acc24169.1,和下午基因,GIGANTEA[32]Acc12229.1)也与该组相关。这两个基因在TPM 2.7和28.1之间的表达模式不同(MYBR92)及TPM 2.8及46.7 (GIGANTEA),表明在收获时间有一些变化(补充数据5).然而,这似乎并不影响网络分析,因为这些基因与蓝色和绿色模块之间只有微弱的相关性(图。5b)。最强大的相关性之一是根组织和含有323个TFS的黄色模块(表2,额外的数据6)显示出96%的相关性(图。5b, c)。如预期的根主导MADS TF (ACC00495.1 MADS19)在这个黄色模块中被发现。
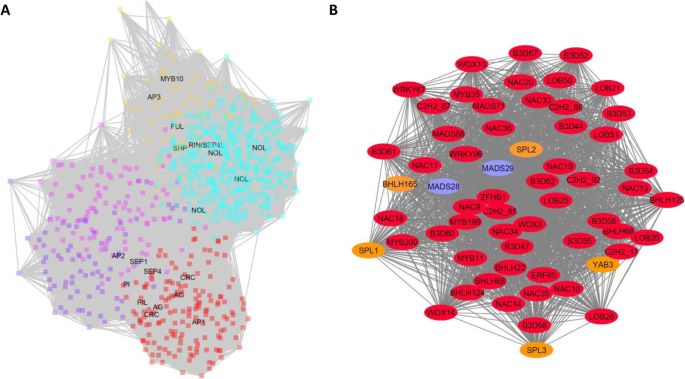
一些组织类型显示出与一个以上的网络组显著相关。花芽和开放花红色和紫色模块分别与159和78个基因相关。含有100个基因的品红模组与果实早期发育有关,而含有60个基因的棕黄模组与果实后期发育成熟有关。在绿松石模块中,457个TFs与成熟果实的采后乙烯处理相关。所有开花和结果基因生成了一个具有已知关键转录因子的网络图(图2)。6)显示出这种基因选择有很强的相关性。计算出红色、紫色、品红、棕褐色和绿松石簇的中心基因Acc17850.1 - ERF65,Acc28494.1 - bHLH281,Acc18135.1 - DOF43,Acc15461.1 - KNOX3,Acc20237.1 - GRAS13分别。最类似的拟南芥基因bHLH281是AT1G25330-半填满(HAF),指明拟南芥的生殖道发育[33].棕色中心基因KNOX3类似于AtKNAT7谁被建议与之合作AtPAP1(它的同系物,Acc00493.1-MYB10/75,也发现在黄褐色簇中)形成种皮[34].
讨论
通过对猕猴桃TF家族的基因挖掘和表达分析,我们构建了一个针对植物生命周期中不同发育阶段不同组织的基因网络。通过对与红色、紫色、品红、棕褐色和青绿色模块相关的花和果实网络的仔细检查(图。6a),发现了一些与其他物种特征相似的MADS-box tf。在红色模块中有10个MICK MADS-box基因,包括之前发表的无性生殖的(AG))基因MADS29(ACC20728.1.)及其同源物MADS28(Acc25178.1),一个PISTILLATA例如基因,MADS11(Acc24088.1),和两个9月类似基因,而紫色模块包含5个MICK MADS基因,包括第二个PISTILLATA例如基因,MADS12(ACC05042.1.).与绿松石模块相关的乙烯处理的果实包含三种MICK MADS样基因,包括此前发表的RIN / SEP4例如基因(Mads52 - ACC26640.1.) [15].
其他类别的特征良好的转录因子也进行了研究,包括在最近的一篇综述中描述的已知的花决定性基因[35].通过检查最接近的猕猴桃同源物和模块成员资格,可以识别潜在的关键基因,如APETALLA2样基因AP2L11(Acc06022.1),螃蟹爪子(儿童权利公约)YAB3(Acc19364.1) 和YAB4(Acc06415.1),内没有外(伊诺)基因YAB1(Acc08170.1和YAB2(Acc06179.1).三个猕猴桃喷嘴课程SPOREOCYTLESS与胚珠发育相关的基因都位于红色模块中:SPL1(Acc13721.1),SPL2(Acc19456.1) 和SPL3(Acc21678.1).B3类Lav基因也有思工扩张B3D47(ACC13067.1.),B3D48(Acc30137.1),B3D49(Acc30138.1),B3D50(Acc30139.1),B3D51(Acc13066.1),B3D52(Acc06689.1),B3D53(Acc21264.1),B3D55(Acc31957.1) 和B3D56(Acc11738.1)均在红色模块内找到。此外,闪亮的shn.和Shnl.ERF基因(Acc12549.1和Acc17850.1)与表皮蜡的形成有关。虽然这些关键基因在红色模块中被识别,但应该注意的是,并不是所有最好的同源基因在这篇综述中被识别,实际上在其他颜色模块中发现了大量的同源基因。
随着AG)其他生物的基因已经被证明是关键的心皮特性基因,连接AG)同系物MADS28和MADS29进一步检查。Fifty-nine genes with a high (> 0.50 weight) association with the AG genes were identified and mapped (Fig.6b).这个子集的网络图显示出这些基因高度的相互依赖。在这个子网络中,已经被证明是直接目标的基因AG)拟南芥(图。6b橙色)。这些包括上述的SPL,儿童权利公约和一个HEC2像bHLH(bHLH65)基因。
在果实成熟乙烯相关的绿松石模块中,鉴定了相当多的(29)NAC tf,包括前面描述的也不就像基因(母,NAC2,NAC3) [36,以及8个中的7个EIN3-like基因。这个模块还包括45小块土地基因[26,以及DOF4[37,38].果实成熟分析的先前的研究[37]描述了10个与成熟相关的TFs(补充数据1),其中大多数位于其他颜色模块,这表明本文提出的更广泛的研究提供了更好的组织特异性基因的分辨率。
结论
总之,我们证明了我们构建和定制以显示基因表达数据的EFP浏览器,与基因组的识别TFS和加权基因共表达网络分析组合提供了一个强大的控制和调控的强大平台在植物生命周期中的重要过程,这些工具可以容易地定制到其他结果。
方法
植物材料
本研究中出现的所有新组织(附加数据)3.)来自二倍体猕猴桃对var。对简历。“Hort16A”。这些数据与从此前公布的数据合并答:对var.对简历。' Hort16A '果实成熟和成熟研究[15,并对相关四倍体的芽系进行了新的综合研究猕猴桃对var。对简历。“Zesy002”(Voogd等人,在准备中)。采用标准藤架果园管理技术,从新西兰Kerikeri的植物与食品研究基地收获水果。每次复制的组织都是将至少五种不同藤蔓的组织结合在一起收获的。每种组织类型取2 - 3个重复,在液氮中快速冷冻,在−80°C下保存,直到需要时。
基因挖掘和比较
为了确定一套完整的转录因子,我们采取了多种方法,每种方法都生成、组合和过滤列表。简单地说,根据确定的阶级选择一个家庭进行采矿拟南芥[5].植物转录因子数据库(http://planttfdb.cbi.pku.edu.cn/),其中包含2296个猕猴桃模型,由2013年原始基因组生成[18也进行了评估。使用内部数据库(Bioview)平台,手动注释基因模型[19鉴定含有适当的PFAM结构域[39].最后,使用适当的多样化的TF匹配多个BLASTP搜索鉴定基因模型进行了比较,手动注释的基因组。这些都在geneious对准。40]使用肌肉排列[41].使用PFAM结构域在对准区域中鉴定DNA结合结构域,除去没有结合结构域的基因,从而创建完整的TFS列表。对于R2R3 Mybs和Wrky家庭,一些基因似乎没有ACC模型。发生这种情况时,Web Apollo [42用于手动注释另外的基因模型,并在附加数据1中用Achn型号或“无ACC匹配”指定。然后使用PHYM1对准DNA结合结构域[43],并依次命名。圈地块的图。1在R中使用“packcircles”库生成[44].
RNA测序和转录组学
使用Spectrum™植物总RNA试剂盒(Sigma-Aldrich, St. Louis, MO, USA)分离猕猴桃总RNA,使用RNA 6000 Nano试剂盒和Agilent 2100 Bioanalyzer (Agilent Technologies, Santa Clara, CA, USA)评估其完整性。每次采样3个RNA样本用于随后的文库构建和测序。测序文库根据TruSeq RNA样品制备指南(Illumina, San Diego, CA, USA)构建,随后使用HiSeq 2500测序系统(Illumina)进行测序,获得125 bp的配对reads。在Macrogen (www.macrogen.com).从Hort16a组织中获得至少16米的读数,百分比读取映射范围从69到92%的额外数据3..Zys002芽系列的最小读数为11.9 M,读映射百分率为51-79%。所有原始序列数据都可以在NCBI RNA-seq存储库中找到,详细信息请参见附加数据3..使用拼接转录物对齐对齐默认设置的参考(星形)(3.5.2a)对齐原始读取[45]到答:对Red 5 v1.69基因模型[19].PCA分析采用了R [46使用5000个差异表达最多的基因(ntop = 5000)。原始读取被归一化为每百万记录(TPM)(额外数据4).
eFP浏览器的开发
eFP浏览器[1]是使用存放在SourceForge (https://sourceforge.net/projects/efpbrowser/).模板图像是由植物与食品研究所的平面设计团队生成的。模板图像被着色和重新格式化为eFP浏览器使用GIMP(https://www.gimp.org/)版本2.8.22软件程序。eFP源代码中添加了几个额外的特性。修改后的源代码可以通过github(https://github.com/pfrnz/eFP-Browser).实验图像由XML文件描述的每个部分的组件图像构造而成。每个实验视图中可以包含多个部分。这允许对不同的部分和实验视图重用组件图像,大大减少了添加新实验视图所需的工作。使用JavaScript和PHP实现接口上基因ID输入的自动完成,引用现有ID查找SQL表。eFP浏览器是使用Docker容器化的。Dockerfile构建了一个Docker容器,其中包含了软件需求、eFP浏览器源代码和实验图像。第二个Docker容器用于读取数表达式数据的SQL数据库。包含一个Docker-Compose文件以运行eFP浏览器和SQL容器。在“植物生物学生物分析资源”服务器上建立了actindia eFP浏览器bar.utoronto.ca可在http://bar.utoronto.ca/efp_actinidia/cgi-bin/efpWeb.cgi
WGCNA网络分析
提取与转录因子相关的基因模型的RNA-SEQ数据,并从分析中除去任何样品中未表达(基于读取的比对的转录因子。加权基因共表达网络分析[30.v1.68是在R环境v3.5.1中进行的。在WGCNA环境中,计算软实力并设置为6。最小模块尺寸设置为30,合并切割高度设置为0.25。使用“chooseTopHubInEachModule”计算每种颜色环境的Hub基因。网络数据被导出到Cytoscape v3.7.1用于可视化。
数据和材料的可用性
所有RNA-seq数据均可在NCBI生物工程PRJNA691387研究SRP301347中找到,登录号SRR13413552: SRR13413581(详细信息见附加数据)3.).
在猕猴桃EFP浏览器可以在这里找到http://bar.utoronto.ca/efp_actinidia/cgi-bin/efpWeb.cgi
缩写
- eFP:
-
电子荧光象形文字
- TF:
-
转录因子
- WGCNA:
-
加权基因共表达网络分析
参考
- 1.
Winter D, Vinegar B, Nahal H, Ammar R, Wilson GV, Provart NJ。一个“电子荧光象形文字”浏览器,用于探索和分析大规模的生物数据集。《公共科学图书馆•综合》。2007;2 (8):e718。
- 2.
Fernandez-Pozo N, Zheng Y, Snyder SI, Nicolas P, Shinozaki Y, Fei Z, Catala C, Giovannoni JJ, Rose JKC, Mueller LA。番茄表达图谱。生物信息学。2017;33(15):2397 - 8。
- 3.
基于eFP浏览器的草莓果实和花卉转录组可视化研究。园艺杂志2017;4:17029。
- 4.
毛青,陈灿,谢涛,栾安,刘灿,何玉霞(菠萝comosus),并构建一个efp浏览器以供进一步研究。PeerJ。2018; 6: e6028。
- 5.
Riechmann JL, Heard J, Martin G, Reuber L, Jiang C-Z, Keddie J, Adam L, Pineda O, Ratcliffe OJ, Samaha RR,等。拟南芥转录因子:真核生物全基因组比较分析。科学。2000;290(5499):2105 - 10。
- 6.
Lehti-Shiu MD, Panchy N, Wang P, Uygun S, Shiu S- h。植物dna结合转录因子家族的多样性、扩展和进化新颖性。生物物理学报。2017;1860(1):3-20。
- 7.
Zhao T, Holmer R, de Bruijn S, Angenent GC, van den Burg HA, Schranz ME。系统基因组同位网络分析麦斯-box转录因子基因揭示了谱系特异性的转位、古老的串联复制和深度位置保护。植物细胞。2017;29(6):1278 - 92。
- 8.
王志强,王志强。花发育的遗传调控。植物学报。大自然。1991;353(6339):主场。
- 9.
Ditta G, Pinyopich A, Robles P, Pelaz S, Yanofsky MF。的SEP4基因的拟南芥拟南芥花器官和分生组织的功能。咕咕叫医学杂志。2004;14(21):1935 - 40。
- 10.
Knapp S.烟草到西红柿:一种关于水果多样性的系统发育视角茄科.中国农业大学学报(自然科学版)2002;53(377):2001-22。
- 11.
Becker A, The ßen G.的主要分支麦斯-box基因及其在开花植物发育和进化中的作用。中国生物医学工程学报。2003;29(3):464-89。
- 12.
弗格森AR。猕猴桃:植物学审查。Hortic修订版1984; 6:1-64。
- 13.
Richardson AC, Boldingh HL, McAtee PA, Gunaseelan K, Luo Z, Atkinson RG, David KM, Burdon JN, Schaffer RJ。二倍体猕猴桃的果实发育,猕猴桃对“Hort16A”.植物学报。2011;11(1):182。
- 14.
Atkinson Rg,Gunaseelan K,Wang My,Luo L,Wang T,Norling Cl,Johnston Sl,Maddumage R,Schröderr,Schaffer RJ。解剖血液乙烯在猕猴桃中的作用(猕猴桃对)使成熟1-AMINOCYCLYPROPANE-1-CARBOXYLIC酸氧化酶可拆卸的线。中国生物医学工程学报。2011;62(11):3821-35。
- 15.
McAtee PA, Richardson AC, Nieuwenhuizen NJ, Gunaseelan K, Hoong L, Chen X, Atkinson RG, Burdon JN, David KM, Schaffer RJ。猕猴桃杂交非乙烯和乙烯成熟反应(猕猴桃对)与差动调节有关麦斯框转录因子。植物学报。2015;15(1):304。
- 16.
Ampomah-Dwamena C, Thrimawithana AH, Dejnoprat S, Lewis D, Espley RV, Allan AC.猕猴桃(猕猴桃deliciosa) R2R3-MYB转录因子调节叶绿素和类胡萝卜素的积累。新植醇。2019;221(1):309 - 25所示。
- 17.
王涛。猕猴桃诱变的研究进展CENTRORADIALIS类基因将一种具有较长的幼体和腋生开花的攀援多年生木本植物转变为具有快速的顶开花的紧凑植物。植物生态学报;2019;17(5):869-80。
- 18.
黄S,Ding J,Deng D,Tang W,Sun H,Liu D,Zhang L,Niu X,张X,孟M等。Kiwifruit的基因组草案猕猴桃对.Nat Commun。2013;4(1):2640。
- 19.
Pilkington SM, Crowhurst R, hillary E, Nardozza S, Fraser L, Peng Y, Gunaseelan K, Simpson R, Tahir J, Deroles SC,等。一个手工注释猕猴桃对var。对(猕猴桃)基因组突出了与草图基因组和植物基因预测相关的挑战。BMC基因组学。2018;19(1):257。
- 20.
吴军,王峰,程磊,孔峰,彭震,刘松,于旭,卢刚生长素响应因子(东盟地区论坛)基因。植物学报2011;30(11):2059。
- 21.
吉尔福伊尔TJ,哈根G.生长素响应因子。CurrOp植物BIOL。2007; 10(5):453-60。
- 22.
王海涛,王海涛,王海涛,王海涛。番茄生长特性的研究进展东盟地区论坛基因家族揭示了包括可变剪接在内的多水平转录后调控。《公共科学图书馆•综合》。2014;9 (1):e84203。
- 23.
李W,丁Z,阮M,Yu X,Peng M,刘Y.Kiwifruit R2R3-MYB转录因子和新型ACMYB75对红猕猴桃的贡献,促进猕猴桃蛋白生物合成。SCI批准。2017; 7(1):16861。
- 24.
猕猴桃WRKY转录因子的全基因组鉴定猕猴桃spp.)和分析WRKY对生物和非生物胁迫的反应中的表达。基因基因组学。2018; 40(4):429-46。
- 25.
Varkonyi-Gasic E, Moss SM, Voogd C, Wu R, Lough RH, Wang Y-Y, Hellens RP。猕猴桃开花基因的鉴定和特性:序列保存和在猕猴桃花发育中的作用。植物学报。2011;11(1):72。
- 26.
Gunaseelan K, McAtee PA, Nardozza S, Pidakala P, Wang R, David K, Burdon J, Schaffer RJ。复制数量变异猕猴桃乙烯/ APETALA2响应因素(小块土地/AP2)类基因在果实成熟过程中表现出与低温和乙烯反应相关的差异C-REPEAT / DRE绑定的因素例如基因。《公共科学图书馆•综合》。2019;14 (5):e0216120。
- 27.
吴荣敏,沃尔顿EF,理查森AC, Wood M, Hellens RP, varkoni - gasic E.四种猕猴桃的保护和分化高级副总裁例如麦斯-box基因在猕猴桃芽休眠和开花过程中起着重要作用。中国农业科学。2011;63(2):797-807。
- 28。
王涛。猕猴桃的功能和表达分析SOC1类基因表明,它们可能在向开花的过渡中没有作用,但可能影响休眠的时间。J Exp Bot. 2015;66(15): 4699-710。
- 29。
yoo sk,wu x,李杰,ahn jh。AGAMOUS-LIKE 6花的启动子是负调控方法/加进化枝基因和正向调控英国《金融时报》在拟南芥。植物j . 2011; 65(1): 62 - 76。
- 30.
加权相关网络分析的R包。BMC生物信息学。2008;9(1):559。
- 31.
Schaffer R, Ramsay N, Samach A, Corden S, Putterill J, Carré IA, Coupland G. The晚细长的下胚轴突变的拟南芥扰乱昼夜节律和开花的光周期控制。细胞。1998;93(7):1219 - 29。
- 32.
Fowler S, Lee K, Onouchi H, Samach A, Richardson K, Morris B, Coupland G, Putterill J。GIGANTEA:一种生物钟控制的基因,调节植物的光周期开花拟南芥并编码了一种具有几个可能的跨膜结构域的蛋白质。EMBO j . 1999; 18(17): 4679 - 88。
- 33.
Crawford BCW,Yanofsky MF。半填满促进生殖道发育和受精效率拟南芥.发展。2011;138(14):2999 - 3009。
- 34.
Bhargava A, Ahad A, Wang S, Mansfield SD,豪恩GW, Douglas CJ, Ellis BE。MYB75和KNAT7的相互作用转录因子在茎和种皮中调节次生细胞壁沉积拟南芥.足底。2013;237(5):1199 - 211。
- 35.
Pfannebecker KC,Lange M,Rupp O,Becker A.种子植物特异性基因谱系参与Carpel发育。mol Biol Evol。2017; 34(4):925-42。
- 36.
Nieuwenhuizen NJ, Chen X, Wang MY, Matich AJ, Perez RL, Allan AC, Green SA, Atkinson RG。猕猴桃单萜合成的自然变异:萜类合成酶的转录调控南汽和ETHYLENE-INSENSITIVE3 -像转录因子。植物杂志。2015;167(4):1243 - 58。
- 37.
A-D张,王W-Q,Tong Y,Li M-J,Grierson D,Ferguson I,Chen K-S,Yin X-R。转录组分析鉴定了Kiwifruit中的锌指蛋白调节淀粉降解。植物理性。2018; 178(2):850-63。
- 38.
张安,张强,李军,龚辉,范旭,杨勇,刘旭,尹晓霞。猕猴桃酯生物合成关键基因及其调控因子的转录组共表达网络分析。植物学报。2020;20(1):103。
- 39.
Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A, Hetherington K, Holm L, Mistry J,等。蛋白质家族数据库。核酸Res. 2013;42(D1): D222-30。
- 40.
Kearse M, Moir R, Wilson A, Stones-Havas S,张M, Sturrock S, Buxton S, Cooper A, Markowitz S, Duran C, et al.;Geneious basic:一个集成的、可扩展的用于序列数据组织和分析的桌面软件平台。生物信息学。2012;28(12):1647 - 9。
- 41.
埃德加钢筋混凝土。MUSCLE:多序列比对,高精度,高通量。核酸学报2004;32(5):1792-7。
- 42.
Lee E, Helt GA, Reese JT, Munoz-Torres MC, Childers CP, Buels RM, Stein L, Holmes IH, Elsik CG, Lewis SE。Web Apollo:基于网络的基因组注释编辑平台。基因组医学杂志。2013;14 (8):R93。
- 43.
用PhyML估计最大似然系统发育。在:Posada D,编辑。用于DNA序列分析的生物信息学。风险:胡玛纳出版社;2009.113 - 37页。
- 44.
D, Menzel P. packcircles:圆形包装。Rpackages 2018。
- 45.
Dobin A,Davis Ca,Schlesinger F,Drenkow J,Zaleski C,JHA S,Batut P,Chaisson M,Gingeras TR。星:超快通用RNA-SEQ对齐器。生物信息学。2012; 29(1):15-21。
- 46.
利用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。基因组医学杂志。2014;15(12):550。
确认
我们要感谢Tony Corbett提供的与eFP浏览器相关的图形,Peggy Kashuba和Phil Martin帮助组织收获,野外研究网络维护猕猴桃植物,Richard Espley博士和Sarah Moss博士批判性地阅读了这份手稿。
资金
这份出版物的工作由MBIE: Turbo Breeding和猕猴桃专利使用费投资基金资助。资助机构在本手稿的设计、收集、分析或解释数据中没有发挥作用。
作者信息
从属关系
贡献
AR对实验组织进行采集和建议,LB负责RNA-seq的制备,并与PM和BW一起进行RNA-seq数据处理。BW创建了actindia eFP浏览器和NJP, AP将其安装到BAR服务器上。NN, KD, JR, AA和RS挖掘TF基因家族并进行系统发育比对。EV、AA和RS监督LB和BW,并对研究进行概念化。RS负责网络分析并撰写手稿。所有作者都阅读和编辑了手稿。作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
“附加文件1”:附加数据
.转录因子表,与相应的颜色模块分配和连接。
附加文件2:附加数据2
.的DNA结合转录因子类网站有超过9个基因进化路线。
附加文件3:附加信息3
.本研究中使用的RNA-seq数据的详细信息。
附加文件4:附加数据4
.RNA-seq数据的TPM表。
附加文件5:附加数据5
.昼夜节律基因表达分析。
附加文件6:附加数据
.每个颜色模块内的转录因子类的数量。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
布赖恩,L.,沃伦,B.,麦克蒂,P.。et al。猕猴桃基因表达图谱(猕猴桃对)和转录因子的网络分析。BMC植物杂志21,121(2021)。https://doi.org/10.1186/s12870-021-02894-x
收到了:
接受:
发表:
关键字
- 猕猴桃
- eFP浏览器
- 转录因子