摘要
背景
重要的工业作物大麻对光周期具有很高的敏感性。大麻的开花时间是其重要的农艺性状之一,对其产量和质量产生重大影响。这CONSTANS-like(上校)基因在植物开花调控中起着关键作用。但是,特定的角色上校大麻的基因家族仍然未知。
结果
在这项研究中,13CsCOL在大麻基因组中鉴定出了基因。系统发育分析表明,CsCOL蛋白可分为3个亚组,每个亚组包含保守的内含子/外显子结构和基序。染色体分布分析表明:13CsCOL基因在7条染色体上分布不均匀,以10号染色体最多CsCOL成员。共线性分析表明,两对共连基因对CsCOL4和cscol11.在大米和蔬菜中都发现了Gossypium raimondii就. 13人中CsCOL基因,CsCOL6和cscol12.是一对重复的串联基因吗CsCOL8和cscol11.可能是由于分段重复造成的。此外,组织特异性表达显示10CsCOL基因优先于叶子中表达1CsCOL在茎和2CsCOL在雌花里。大多数CsCOL在不同的光照处理下表现出日振荡模式。此外,序列分析表明CsCOL3和CsCOL7早花和晚花品种间氨基酸含量存在差异。
结论
这项研究提供了洞察的潜在功能CsCOL基因,并强调了在大麻开花时间调节中的作用。我们的结果为进一步阐明的功能奠定了基础上校大麻的基因。
背景
麻(大麻L.)是一种古老的经济作物,广泛用于纺织、食品、建筑材料等领域[1].近年来,以大麻素(cannabinoid, CBD)为代表的大麻二酚的使用不断扩大,大麻产业呈现出良好的发展前景[2].大麻是一种一年生短日照作物,对光周期很敏感[3.].大麻品种自然生长在高纬度地区。然而,大麻种质资源已被引进低纬度地区种植,导致开花时间较早。导致生长期缩短,严重降低了CBD和纤维的产量和含量[3.].因此,开发适应性广的大麻品种是当前大麻育种的主要目标之一。明确大麻开花的调控机制,可为大麻品种的培育提供理论依据。然而,目前对大麻开花调控机制的研究还很缺乏。
植物的开花时期是一种复杂的定量特征,其由许多内部和外部因素全面调节,包括光周期,温度,激素和自我发展[4.]. 在这些因素中,光周期是花转变的重要调控因素。在农业中,可以通过改变光照时间来调整栽培植物的开花时间,以满足消费者的需求。随着分子遗传学和分子生物学领域的迅速发展,许多与光周期相关的基因被发现和克隆[5.那6.].研究表明CONSTANS-like (COL)基因是对光周期的植物反应的重要调节因子,并且是植物开花调节中的核心元素[7.那8.那9.].上校属于锌指转录因子家族,其含有B盒式和CCT(CO,CO,CO,TOC1)结构域[10].根据b盒和CCT域的数量,上校家族基因可分为五组[11].在以前的报告中上校基因家族在许多植物中已经得到了广泛的研究拟南芥、大米(栽培稻l .)、玉米(Zea Mays.l .)杨属,萝卜(Raphanus sativusL.),莫斯竹子(撂荒),百合×formolongi[5.那6.那10那11那12那13那14]. 数量上校不同物种的基因不同。例如,在双子叶植物中上校萝卜家族有20个成员,萝卜家族有17个成员拟南芥单子叶中,水稻有16个成员,玉米有19个成员,玉米有14个成员杨属[12那13那14那15那16].
这上校基因作为转录因子参与多种生长发育途径,尤其是光周期介导的开花途径。这个家族的一些基因已经被发现在光反应介导的开花调节中发挥重要作用[5.那17那18那19],具有不同于短日(SD)和长日(LD)条件的功能。例如,OsCOL10 OsCOL13,和OsCOL16在米饭中为SD和LD条件下的开花负调节器的功能Hd1型的一员上校基因家族,促进SD开花,抑制LD开花[5.那17那20.那21].在拟南芥的过度表达ATCOL3.那AtCOL7,和ATCON8.可以延迟开花时间,而相比,相反,过度表达ATCON5.基因通过增强表达促进开花开花轨迹T(FT.)[18那19那22那23].类的成员之间的表达式模式也不同,与它们的功能相似上校基因家族。在香蕉(穆萨娜娜不悦之色),MaCOL基因在光照下比在黑暗中表现出更高的表达,在光照期间达到高峰[24].转录水平PaCOL1和PaCOL2, 2个成员上校基因家庭在挪威云杉(云杉L.),通过光诱导,从暗度过渡到光线[25].不像PaCOL1和PaCOL2那PttCO1和PttCO2表现出明显的表达模式,在傍晚早些时候表达增加[26].同时,这些基因在CDS区域的序列差异与它们在光周期介导的开花途径中的功能有关。例如,在第二个外显子中缺失2bpHd1型“Kasalath”导致水稻品种花期延迟[20.].类似地,核苷酸多态性在OsCOL16编码序列主要由3个等位基因(A1、A15、A22)组成,这些等位基因随花期而变化[5.].这些研究表明,由于在表达模式和CDS序列上的差异上校基因家族在SD和LD条件下的花期调控中具有多种功能。
虽然上校基因在许多植物的生长发育中起着重要的作用,本文对其进行了全面的分析上校大麻中的家庭基因目前缺乏。此外,由于大麻基因组组装的不可用,没有进行大麻中的任何其他基因家族的全身分析,缺乏关于染色体水平的基因位置的信息。最近大麻的基因组在大麻基因组数据库上进行测序并提供[27],以便全面分析上校大麻的基因家族。在本研究中上校利用生物信息学对大麻基因家族进行了分析,并对其时空表达模式进行了研究上校基因进行了研究。此外,氨基酸(aa)序列的差异CsCOL3和CsCOL7研究了早花和晚花品种间的差异。因此,本研究的结果为进一步分析其分子功能提供了生物学基础CsCOL大麻的基因家族。
结果
确定13CsCOL基因在大麻
共13CsCOL从大麻基因组数据库(CsCOL1通过CSCOL13).13.CsCOL基因同时包含B-box和CCT保守结构域。使用ProtParam (http://web.expasy.org/protparam/) (桌子1). 如表所示1CsCOL蛋白的长度范围为184 (CsCOL8) ~ 507 (CsCOL12) aa,分子量范围为26.02 ~ 56.24 kDa, pI范围为4.99 ~ 6.36。此外,亲水性的总体平均值在−1.088到−0.245之间,脂肪族指数在38.10到69.90之间1).
基因结构,系统发育关系,序列分析CsCOL基因
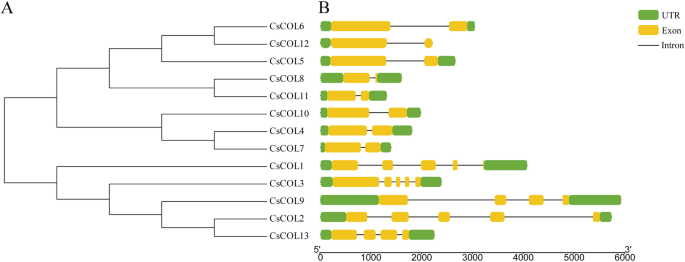
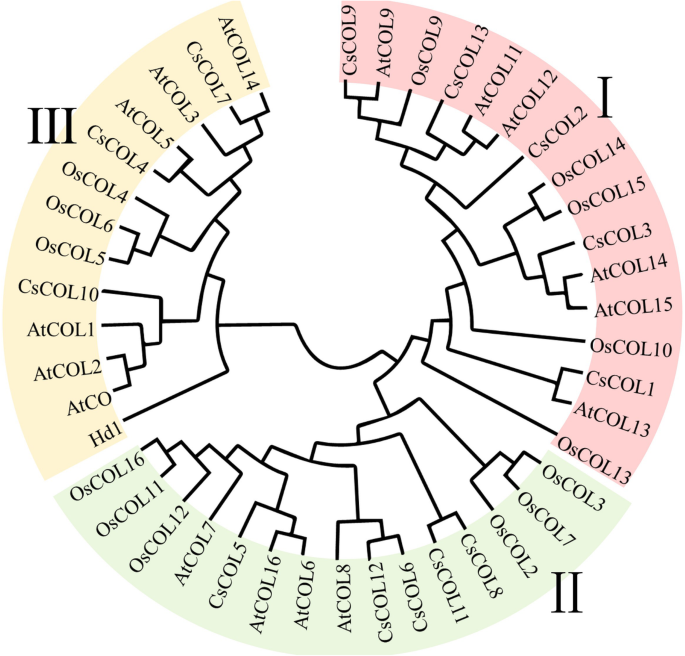
来估计各成员之间的进化关系CsCOL通过比较CsCOL蛋白的基因结构,研究了CsCOL蛋白的结构多样性。如图1所示。1, 所有CsCOL基因中分别含有2 ~ 5个外显子和1 ~ 4个内含子。所有的CsCOL基因包含3’和5’UTR区域(图1)。1).进一步,探讨两者之间的进化关系上校不同物种的基因,系统发育树分析与来自不同植物的Col蛋白进行,包括拟南芥,大麻和米饭。这些包括来自二坐子植物的30个基因(例如,拟南芥以及来自单子叶植物(如水稻)的14个基因。结果显示,这些COL蛋白可以聚为3大类,命名为I-III类(图1)。2).组III是最小的亚家族,由最少的COL蛋白组成(图3)。2).此外,我们还对晚花品种“Y7”和早花品种“Q1”的CsCOL基因的氨基酸序列进行了研究(图1)。S1还有无花果。S2).不幸的是,只有CsCOL3和CsCOL7被成功克隆。对于CsCOL3,“Y7”与“Q1”之间存在2个氨基酸差异,均不位于B-Box结构域,也不位于CCT结构域(图1)。S3A)。对于CsCOL7,“Y7”与“Q1”之间存在4个氨基酸差异,均位于B-BoxIdomain(图1)。S3b)。
染色体位置和同步分析
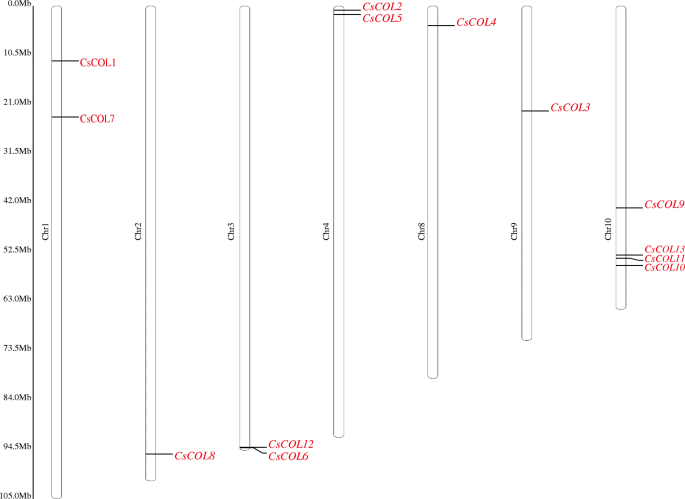
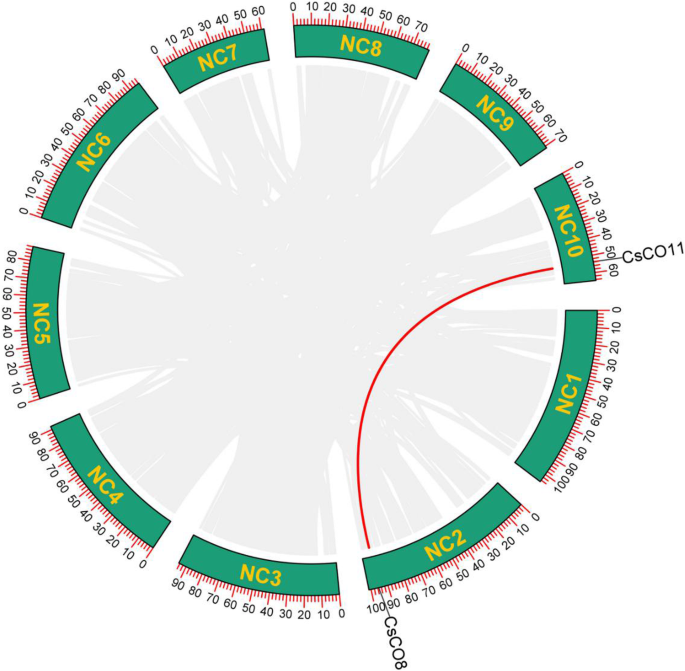
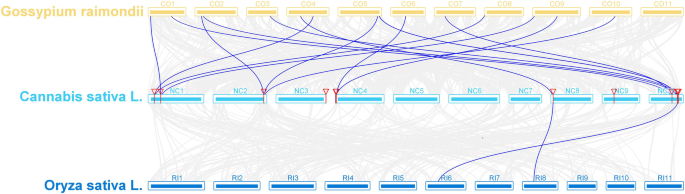
如图1所示。3., 13CsCOL基因成员在大麻基因组的7条染色体上分布不均匀,除了第5、6和7条染色体。其中,10号染色体的染色体数最高CsCOL基因(4),而染色体2,8,和9只包含1。有趣的是,一对串联复制基因被确定在染色体3(CsCOL6 / CsCOL12),表明串联重复事件参与了种群的扩张上校在大麻的家庭。因此,调查了重复事件CsCOL大麻基因组的基因。结果,只有一对重复的基因(CsCOL8 / CsCOL11)在大麻基因组内被鉴定,它们可能是由片段复制或全基因组复制(WGD)引起的(图。4.).为了进一步了解进化机制上校大麻的家庭,联合性图上校在2种双子叶植物中构建了该科(Gossypium raimondii就和大麻)和1株单子叶植物(栽培稻L).如图所示。5.,在大麻与cotton Raymond之间共鉴定出15对同源基因,远远大于在大麻与水稻之间的同源基因(2)。CsCOL4和cscol11.在大米和棉花中都被发现了雷蒙德CsCOL1那CsCOL7那CsCOL8那CsCOL5,CsCOL9仅在棉花雷蒙德发现,其余的在任何复制块中都不存在(图。5.).
13CsCOL基因
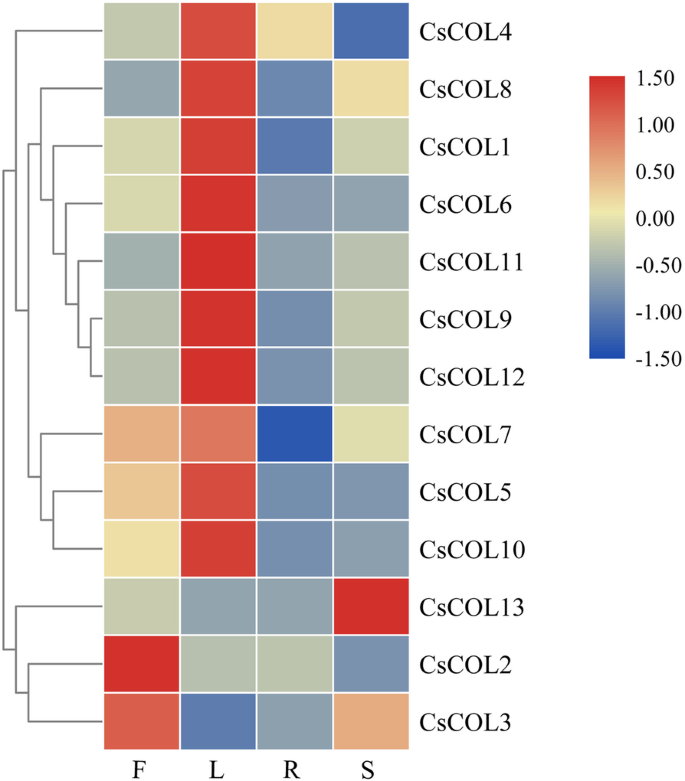
以便深入了解可能的角色CsCOL基因在大麻发育过程中的表达模式CsCOL对雌花、茎、叶和根4个植物组织进行了基因分析。结果显示,所有基因在不同组织中均有建设性表达,但表达模式不同(图1)。6.).在13中CsCOL有10个基因在叶片组织中高表达,CsCOL2和CsCOL3在雌花中均有高表达,且CSCOL13,其在茎中表达量最高,在其他组织中表达量较低(图。6.).
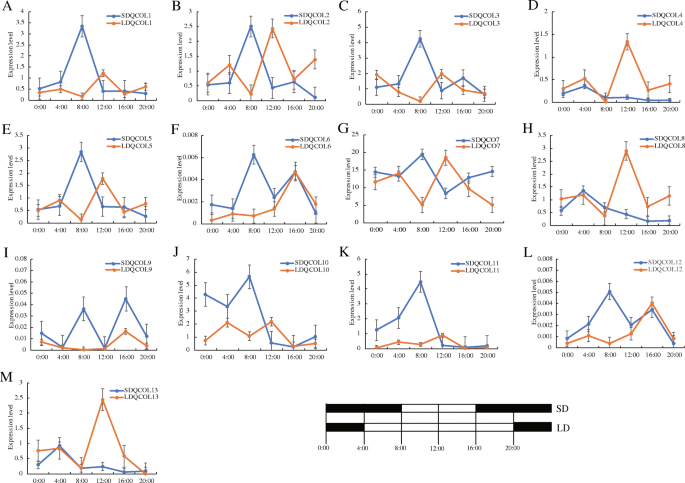
以前的研究发现了上校基因在开花时间的调节中发挥了重要作用。评估可能的功能CsCOL采用qRT-PCR方法分析基因的表达水平CsCOL不同光周期处理间隔4 h的基因(图。7.).SD条件下的日表达谱CsCOL基因各种各样。表达模式大致分为三种类型(图。7.). 第一类在黑暗结束时表现出高水平的表达,包括CsCOL1-3, CsCOL5-7和CsCOL10-12.第二种在夜晚(黑暗)04:00表达增加,包括CsCOL4那CsCOL8和CSCOL13.剩余的CsCOL基因在光末端的表达量最高。7.). 在LD条件下,尽管上校在光中诱导基因,观察到两种类型的昼夜表达模式(图。7.).第一种类型包括大多数的转录水平CsCOL,在白天(光)中午12点时达到峰值(CsCOL1-5那CsCOL7那CsCOL8那cscol10.那cscol11.,CSCOL13),而第二类在白天的16:00表达最高的表达水平(光)(CsCOL6那CsCOL9,cscol12.).总的来说,这些结果表明大多数CsCOL在SD和LD条件下,基因表现出日振荡的表达模式。
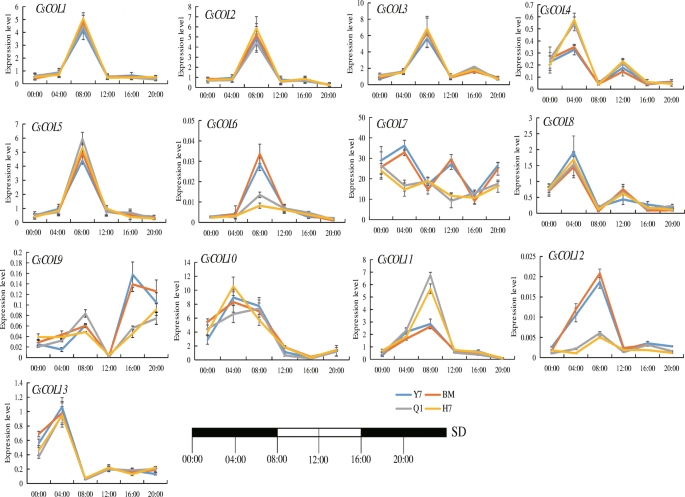
进一步探索的功能上校大麻的基因,2个早期开花品种,“青马1”(“Q1”)和“H7”,以及2个晚花品种,“Yunma 7”(“Yunma 7”(“Y7”)和“BM”,选自126种进入生长在短期内,用于调查13的表达模式CsCOL基因。在播种之后,“Q1”和“H7”的开花时间分别是29d和31d,而“Y7”和“Bm”是117d和113d,在该领域的SD条件下(图。S1b)。在SD条件下,在13中CsCOL基因,2个基因(CsCOL4,cscol11.)在转录高峰时,这2个早花品种的表达量高于这2个晚花品种,而4个基因(CsCOL6那CsCOL7那CsCOL9,cscol12.)显示了相对的模式。剩余的上校基因在这4个品种之间表现出类似的表达水平(图。8.).
讨论
这上校基因家族在植物开花时间的调控中起着关键作用,已在许多植物物种中得到报道,其中包括拟南芥,米饭,玉米,大豆(大豆), 番茄 (茄属植物lycopersicum),杨属那百合×formolongi[5.那10那12那13那14那27那28].然而,一个全基因组的调查上校大麻的家族基因尚未进行研究。由于缺乏高质量的大麻基因组序列,对全基因组进行鉴定工作上校大麻基因组中的基因一直缺乏。最近组装的大麻基因组包含染色体水平的基因定位信息[29],从而可以对上校大麻的基因家族。在本研究中,13上校成员在大麻基因组中被确认。将其分为3个亚组。1和2),这与水稻和拟南芥[16].同样数量的上校在其他植物中也发现了基因,包括杨属(14.上校基因),高粱(高粱L.)(15)和大米(16)[13那16那30].四种植物的基因组的大小不同的事实表明了数量上校基因在上校超家族是稳定的,不随基因组大小而变化。
串联复制事件与新功能的发生和基因扩增有关。在大麻中,复制事件已被发现发生在CBCAS那Thcas.,展览业基因(29那31].另一方面,没有观察到串联重复事件上校棉花基因[30].然而,在这项研究中,一个串联基因对(CsCOL6和cscol12.)在大麻染色体上发现,表明基因重复可能是大麻基因进化的重要驱动力。除了串联复制事件之外,还报告了分段重复作为基因扩张的主要驱动力COL基因家族Gossypium和玉米[12那30].与这些发现一致,在这项研究中,一个片段重复基因对(CsCOL8 / CsCOL11)在大麻基因组中被发现(图。4.).然而,在涉及重复的基因中,这对重复基因在SD和LD条件下表现出不同的表达模式(图)。1那7.,8.)表示这些基因在基因重复期间经历了功能性分歧。另外,鉴定了15对大麻和棉雷蒙德之间的正交基因,而大麻和水稻之间仅发现2对(图。5.).这个观察暗示了大麻上校基因与棉花雷蒙德的关系比米饭更接近,这可能与单胶质龙和双旋肌之间的进化关系一致。有趣的是,CsCOL4和cscol11.在大米和棉花里都发现了,雷蒙德,这表明这些上校基因以特异性的方式从常见的祖传基因膨胀,然后在Dicot-单子宫偶像发散之前。
尽管之前的研究已经证明了这一点上校基因在不同的植物组织中广泛表达,人们发现它们优先在叶片中表达[5.那14那17].叶片感知光周期信号并表达上校激活FT并促进开花[9.].在本研究中,我们调查了13CsCOL不同植物器官的基因,包括雌花、叶、根和茎。因此,10上校在大麻中发现基因在叶片中优先表达,其表达模式与在其他植物中观察到的相似,这表明了其潜在的功能(图。7.).
光周期被认为是决定植物开花时间的关键因素上校基因已被证明参与调控光周期介导的开花[5.那9.那17].因此,我们研究了日变化的转录水平CsCOLLD和SD条件下的基因。在SD条件下,所有上校基因表现出一种日振荡的表达模式,它们之间的差异很小。成绩单水平为9上校基因成员在黎明时达到高峰(图。7.),类似于OsCOL16那PtCOL1/2那ATCOL1.那ATCON2.,AtCO在其他工厂[5.那13].在LD条件下,表达模式各不相同CsCOL基因大致分为两类。第一种包括10个CsCOL在光照处理后表达更高的基因,在12小时达到峰值 h、 符合上校基因包括第14页在杨属[13].类似于第13–16页那oscol10.,OsCOL16,剩下的3个CsCOL基因在光照条件下的表达水平高于黑暗条件下,并在16小时达到高峰[5.那14那17].
不同表达水平的上校基因可能与不同品种的开花时间相关联。在该研究中,在场上的SD条件下,“Q1”和“H7”变化比“Y7”和“BM”呈现比“Y7”和“BM”(图。S1B).进一步确定…的潜在功能CsCOL在调控开花时间的基因中,我们评估了所有基因的转录水平CsCOL“Q1”、“H7”、“BM”和“Y7”品种的基因在SD条件下的表达。如图1所示。8.的表达水平CsCOL4和CsCOL8两个早花型品种(Q1和H7)的转录峰值水平高于两个晚花型品种(Y7和BM),而6个基因(CsCOL6那CsCOL7那CsCOL9,cscol12.)显示了一个对比模式(图。8.).有趣的是,除了类似的表达模式CsCOL6那cscol12.,OsCOL16在SD和LD条件下(图。7.)[13[基于它们的系统发育关系分析,这3个基因还属于相同的亚组(图。2). 在之前的研究中,CsCOL6被发现在大米中压制开花[5.].因此,CsCOL6 / CsCOL12可以起到类似的作用吗OsCOL16在大麻。然而,这需要进一步的研究。
以前的研究表明,氨基酸序列的差异上校基因可以解释它们在光周期介导的开花途径中的不同功能[5.那10那20.].在本研究中,氨基酸序列的差异CsCOL3和CsCOL7分别在早花品种“Q1”和晚花品种“Y7”之间观察到(图1)。S1还有无花果。S2). 此外,在氨基酸序列上也存在差异CsCOL7在b盒内,一个保守的区域,以其在蛋白质相互作用中的功能而闻名。然而,这些变化是否会影响这类功能还有待进一步研究。
结论
综上所述,本研究首次对这一问题进行了全面的分析上校大麻的基因家族。我们的目的是阐明这些基因在大麻开花调控中的进化,表达谱和潜在功能。虽然可能的功能CsCOL该基因家族有待进一步研究验证,本研究进行的系统分析为今后的生物学和分子功能研究提供了基础上校大麻的基因。
方法
理化性质鉴定与分析CsCOL大麻的基因家庭成员
17的序列拟南芥constans样蛋白从拟南芥信息资源(TAIR)(http://www.aabidopsis.org/). 大麻基因组文件和基因组注释文件(装配号:gca900626175.2)来自NCBI数据库(https://www.ncbi.nlm.nih.gov/)[29].软件TBTools用于比较拟南芥通过blast序列比对(e值<1E− 5.)并筛选CsCOL大麻基因组中的家族候选基因。接下来,将候选基因提交到Uniprot数据库(https://www.uniprot.org/)进行批比较,以验证它们是否同时包含CCT和b盒保守域。ProtParam (http://web.expasy.org/protparam/),分析了各种理化参数CsCOL基因。
基因克隆
引物对CsCOL根据CDS序列设计基因(补充表S1).“Y7”和“Q1”的cDNA用作每个基因的模板。PCR如下进行:在94℃下初始步骤5分钟,然后在98℃下在55℃下为30 s的30次,在68℃下2分钟,最终延伸10分钟在68°C。完成PCR程序后,纯化PCR产物,连接到PGEM-T易于载体,并转化为E大肠杆菌DH5a型。选择阳性克隆进行测序。用于基因克隆的引物列表见补充表S1.
CsCOL蛋白的多序列比对,系统发育分析,基因结构分析CsCOL
多序列比对分析拟南芥,大米和大麻COL蛋白使用Clustal X2.1与默认参数[32].采用MEGA7.0和邻域连接法(NJ)构建系统发育树。使用1000次重复估计引导值(> 50%)。用FigTree软件编辑系统发育树。利用NCBI-CDD在线软件预测CsCOL蛋白结构(https://www.ncbi.nlm.nih.gov/cdd/),使用默认参数(E-value < 0.01)。保守的母题(母题)CsCOL基因分析使用模因软件在线(http://meme-suite.org.),预测值设置为10。的编码序列(CDS)和非翻译区(UTR)CsCOL利用TBtools从大麻基因组注释文件中提取,并结合进化树、基因保守motif、CDS和UTR构建图谱,比较CsCOL.
水稻染色体分布与共线性分析CsCOL
关于染色体位置的信息CsCOL利用TBtools从大麻基因组文件和基因注释文件中提取基因。接下来,物理位置CsCOL利用TBtools构建染色体上的基因。利用TBtools、MCscanX和Circos计算并绘制了序列重复序列上校在染色体上,大麻基因组中的共线基因,以及不同物种。
开花时间和光周期处理的评价
“云马7号”(“Y7”)、“BM”、“H7”和“青马1号”(“Q1”)均采自中国农业科学院长沙麻类研究所。2个品种在长沙(112°58′E/28°11′N,营养期昼长< 12 h)自然短日照条件下随机种植。一旦每个品种的植株开花超过50%,就对开花时间进行评分。在LD (16 h光/8 h暗)和SD (8 h光/16 h暗)条件下,分别于光周期处理后的0:00、04:00、08:00、12:00、16:00和20:00采集幼苗叶片。得到的材料被迅速转移到液氮中进行RNA提取,独立重复三次。
RNA提取和QRT-PCR分析
利用RNAprep Pure Plant Kit(天根,北京)从不同光周期下的不同组织和叶片中提取总RNA。使用PrimeScript第一链cDNA合成试剂盒(TaKaRa, Japan)进行cDNA合成。根据制造商说明书,定量RT-PCR (qRT-PCR)在7500序列检测系统(Applied Biosystems, USA)上使用SYBR Premix Ex TaqTM试剂盒(TaKaRa)进行。扩增DHS2基因作为内对照。qRT-PCR分析所用引物见补充表S2.实验重复进行了三次。
可用性数据和材料
本文及其附加文件中列出了支持本文结论的数据集。
的所有编码序列CsCOL3和CsCOL7“青马1号”和“云马7号”已上传到NCBI SRA数据库。SRA加入:PRJNA704531。数据可以通过以下链接访问:"https://www.ncbi.nlm.nih.gov/sra/PRJNA704531”.
缩写
- 列:
-
恒定的
- 生物多样性公约:
-
大麻素
- LD:
-
漫长的一天
- SD:
-
短暂的一天
- 英国《金融时报》:
-
开花轨迹T
- WGD:
-
全基因组复制
- CBCAS:
-
CBCA合成酶
- THCAS:
-
THCA合成酶
- CBDAS公司:
-
展览业合成酶
- QRT-PCR:
-
定量实时聚合酶链反应
参考文献
- 1.
Andre CM, Hausman JF, Guerriero G.大麻:一千分子植物。植物科学,2016;7:19。
- 2.
腐蚀J,菲利普斯JA。(2018)。一项对大麻二酚使用者的横断面研究。3(1): 152-61。
- 3.
一种大麻的发展(大麻l)仿真模型两个大麻品种对光周期的开花响应。农业实验学报,2000;40(3):413-7。
- 4.
[11]张文华,张文华,张文华,等。水稻花期调控的研究进展[J]。植物生态学报;2013;
- 5.
吴伟,郑晓明,陈德,张勇,马伟,张浩,等。OsCOL16编码一种君士坦斯样蛋白,通过上调Ghd7在水稻中的表达来抑制开花。植物科学。2017;260:60–9.
- 6.
刘杰,程Z,李X,谢L,白Y,彭L,李杰,高J。毛竹类CONSTANS基因家族的表达分析及调控网络鉴定(植被类型)在光周期处理下。DNA细胞BIOL。2019; 38(7):607-26。
- 7.
iaizumi等。FKF1F-BOX蛋白介导拟南芥CONSTANS抑制因子的循环降解。科学。2005;309(5732):293 - 7。
- 8.
Kobayashi D. Weigel,继续,是时候改变了——移动信号控制依赖光周期的开花。基因Dev。2007;21:2371 - 84。
- 9.
成花素的调控与特性:开花位点T移至中心阶段。植物生物学杂志,2008;59:57 - 62。
- 10.
Robson F,Costa MM,Hepworth SR,等。开花时间基因中保守结构域的功能重要性君士坦斯通过突变等位基因和转基因植物的分析证实。植物j . 2001; 28(6): 619 - 31所示。
- 11.
等。拟南芥Bbox锌指家族。植物细胞。2009;21:3416-20。
- 12.
宋,徐Z,王继,秦q,姜h,si w,li x。玉米的基因组宽分析CONSTANS-LIKE基因家族和轻质/暗和脱落酸处理下的表达分析。基因。2018; 673:1-11。
- 13.
李军,高科,杨欣,吴可汗,郭斌,郭涛,安欣CONSTANS-like基因家族及其在光处理下的表达谱杨属.int j biol macromol。2020; 161:999-1010。
- 14
李艳,赵艳,张敏,贾刚,扎才敏CONSTANS-like害怕家庭Liliumi½formolongi。植物生理学杂志。2018;59(9):1874-88。
- 15
胡涛,魏青,王伟,胡辉,毛伟,朱青,包春。萝卜constans样基因家族的全基因组鉴定与鉴定(Raphanus sativus).Plos一个。2018; 13(9):E204137。
- 16
Griffiths S, Dunford RP, Coupland G, Laurie DA。的进化CONSTANS-like大麦、水稻和拟南芥的基因家族。植物生理学。2003;131:1855–67.
- 17
谭杰,金敏,王杰,吴芳,盛平,程智,王杰,郑鑫,陈龙,王敏,朱森,郭鑫,张鑫,刘鑫,王超,王浩,吴超,万杰CONSTANS-Like基因,作为开花时间抑制因子下游Ghd7在米饭中。植物细胞生理。2016; 57(4):798-812。
- 18.
Takase T,Kakikubo Y,Niskasone A,Nishiyama Y,Yasuhara M,Tokioka-Ono Y,Kiyosue T.表征和转基因研究君士坦斯式(COL8)拟南芥35S: COL8基因的表达延缓了长日照条件下的开花。生物科技植物》。2011;28:439 - 66。
- 19.
王H,张Z,李H,赵X,刘X,奥尔蒂斯M,林C,刘B。类君士坦斯7调控拟南芥的分枝和避荫反应。J实验机器人。2013;64(4):1017–24.
- 20.
Yano M、Katayose Y、Ashikari M、Yamanouchi U、Monna L、Fuse T、Baba T、Yamamoto K、Umehara Y、Nagamura Y等。Hd1是水稻主要光周期敏感数量性状位点,与拟南芥开花时间基因CONSTANS密切相关。植物细胞。2000;12(12):2473 - 84。
- 21.
盛平,吴飞,谭军,张辉,马伟,陈磊,王杰,王杰,朱松,郭鑫,等。一种类似constans的转录激活剂,oscol13.,在水稻中作为OsphyB下游和Ehd1上游开花的负调控因子。中国生物医学工程学报,2016;
- 22.
关键词:拟南芥;拟南芥CONSTANS-LIKE3是红光信号和根生长的正调节因子。植物细胞。2006; 18(1):70-84。
- 23.
Hassidim M,Harir Y,Yakir E,等。过度表达类君士坦斯5能诱导短日生长拟南芥开花。足底。2009;230(3):481 - 91。
- 24.
Chaurasia Ak,Patil Hb,Azeez A,Subramaniam VR,Krishna B,Sane Ap,等。分子表征CONSTANS-Like(COL)基因(小果野蕉L. AAA集团,cv。大拿)。植物生理学。2016;22:1-15。
- 25.
Holefors A, Opseth L, Ree RA, Ripel L, Snipen L, Fossdal CG, Olsen JE。的识别PaCOL1和PaCOL2,两个CONSTANS-like在挪威云杉中,显示短日照前转录水平下降的基因导致生长停止。植物生理学杂志。2009;47(2):105-15。
- 26
丁健,Böhlenius H, Rühl MG,陈鹏,Sane S, Zambrano JA,等。GIGANTEA-like基因控制季节性生长停止杨属. 新植物醇。2018;218:1491–503.
- 27
杨t,他,牛,n y s,张y。识别和表征君士坦斯(CO.)/CONSTANS-like(上校),番茄光周期信号和开花相关基因。植物科学。2020;301:110653。
- 28
大豆(Glycine max) CCT基因家族的克隆与表达分析。足底。2021, 253(1): 15。
- 29
Laverty KU, Stout JM, Sullivan MJ, Shah H, Gill N, Holbrook L, Deikus G, Sebra R, Hughes TR, Page JE等。一份大麻的物理和遗传图谱发现了THC/CBD酸合酶位点的广泛重排。基因组研究》2019;29(1):146 - 56。
- 30.
秦伟,于勇,金勇,王旭,刘军,席军,李忠,李辉,赵刚,胡伟,等。全基因组分析阐明的作用CONSTANS-like棉花逆境响应基因的研究。国际分子科学杂志。2018;19(9):2658。
- 31.
关键词:大麻,基因复制,基因分化,药物含量新植醇。2015;208(4):1241 - 50。
- 32.
Larkin M、Blackshields G、Brown N、Chenna R、McGettigan P、Larkin MA、Blackshields G、Brown NP、Chenna R、McGettigan PA、McWilliam H、Valentin F、Wallace IM、Wilm A、Lopez R等。Clustal W和Clustal X 2.0版。生物信息学。2007;23(21):2947–8.
致谢
我们感谢我们实验室的所有工作人员提供有用的建议和技术支持。非常感谢编辑和审稿人对稿件的改进提出的建设性意见。
资金
该研究得到了中国农业技术研究体系(Cars-16-E-02),中央公共利益科学机构基础研究基金(1610242020003,1610242019001)支持。资金用于执行实验,分析数据以及写作稿件。
作者信息
从属关系
贡献
G. P.和Z. L进行了实验;g。P写了这篇论文;M.Y., J.T.参与了样本采集和RNA提取;a.g.c、j.j.l、h.j.t、l.c和y.d进行了生物信息学分析。s - q.h修改了手稿;L.-N.Z, D.-F。L设计了实验,L - n。Z也提供了材料。所有作者阅读并批准了即将出版的版本。所有作者都已阅读并同意该手稿的出版版本。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者们宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
泛,G.,Li,Z.,Yin,M。等等。全基因组的鉴定、表达和序列分析CONSTANS-like大麻基因家族在植物开花时间调控中具有潜在的作用。BMC植物生物学21,142(2021)。https://doi.org/10.1186/s12870-021-02913-x.
收到了:
公认:
发表:
关键词
- 大麻
- 基因组 -
- CONSTANS-like基因(上校)
- 表达模式
- 开花时间