- 研究文章
- 开放访问
- 发布:
整合microRNA和转录组分析揭示苦荞种子发育相关的关键miRNA-mRNA相互作用对(Fagopyrum tataricum.)
BMC植物生物学体积21.文章编号:132.(2021.)
摘要
背景
Tartary荞麦种子发育是一种极其复杂的过程,涉及许多基因调节途径。MicroRNAS(miRNA)已被鉴定为基因表达的重要负调节因子,并在各种植物生物过程中进行了至关重要的调节作用。但是,MIRNA是否参与鞑靼荞麦种子发展仍未开发。
结果
在本研究中,我们首先在苦荞基因组中鉴定了26个miRNA生物合成基因,并描述了它们的系统发育和表达谱。然后,我们对苦荞种子发育的三个阶段进行小RNA (sRNA)测序,以确定与种子发育相关的mirna。在苦荞中首次鉴定出包括101个保守mirna和129个新mirna在内的230个mirna,并成功预测了3268个靶基因。在这些mirna中,76个在种子发育过程中表现出差异表达,共鉴定出1534个靶基因,与74个差异表达mirna (differential expressed mirna, dm)对应。在综合分析dem及其靶基因表达的基础上,鉴定出65对miRNA-mRNA互作对(25对对应于65个靶基因),在苦荞种子发育过程中表现出明显的相反表达。其中6对miRNA-mRNA对通过定量实时聚合酶链反应(qRT-PCR)和连接酶介导的5 ' cDNA末端快速扩增(5 ' -RLM-RACE)进一步验证。65个靶mrna的功能注释显示,56对miRNA-mRNA相互作用主要涉及细胞分化和增殖、细胞伸长、激素反应、器官发生、胚和胚乳发育、种子大小、矿质元素运输和类黄酮生物合成等。这表明它们是苦荞种子发育的关键miRNA-mRNA对。
结论
我们的研究首次揭示了苦荞种子发育中mirna介导的调控途径,表明mirna在苦荞种子发育中发挥着重要作用。这些发现将有助于研究mirna在苦荞种子发育中的作用和调控机制。
背景
microRNAS(miRNA),一类内源性非编码小RNA(SRNA)的长度为20-24nt,通过在转录或转录后的靶基因表达中抑制其靶基因表达,在动物和植物中起着至关重要的调节功能[1,2,3.,4].在植物中,miRNA的生物合成是涉及许多基因和酶的多体途径[5].植物miRNA基因最初通过核中的RNA聚合酶II转录成原发性miRNA(PRI-miRNA)[5,6].然后,通过DICER-LIKE1 (DCL1)、HYPONASTIC LEAVES1 (HYL1)和SERRATE (SE)蛋白复合物将pri-miRNA加工成具有茎环结构的miRNA前体(pre-miRNA) [5,6,7].接下来,这些pri-miRNA进一步切割成miRNA :: miRNA*在DCL1,Hyl1和Se蛋白质复合物的作用下的双工,然后通过甲基转移酶Hua Enhancer1(HEN1)甲基化双链体的3末端[5].甲基化后,通过仓场蛋白质将双链体出口到细胞质[5,8].最后,miRNA双链体由Argonaute1(前1)束缚,形成RNA诱导的沉默复合物(RISC),通过切割靶mRNA或压抑翻译过程来进行其功能[5,9].目前,MIRNA已在许多植物中鉴定出来,越来越多的证据表明它们在植物生长和发育,次生新陈代谢,生物和非生物胁迫耐受性和信号转导中起着至关重要的作用[4,10.,11.,12.,13.,14.,15.,16.].
种子是许多作物植物中的繁殖和原发性营养储存器官,其发育成功或失败直接决定了最终的作物产量和种子质量,以及遗传信息是否可以成功传送到下一代[4].作物种子的发育是一个非常复杂的生物学过程,涉及许多基因调控途径[17.,18.].越来越多的证据表明,mirna参与了作物植物种子发育的调控。迄今为止,包括水稻在内的多种作物的种子发育过程中已经发现了数千个mirna [19.,20.)、玉米(21.,22.], 小麦 [23.,24.],大麦[25.,26.,大豆27.,28.)、花生(18.],芸苔属植物显著[29.,30.狭叶扁豆[6普通荞麦[4]通过使用高吞吐量排序方法。这些研究发现,种子发育过程中各种miRNA的表达是极其动态的,并且一些miRNA在种子中特异性表达,意味着miRNA在种子发育中具有非常重要的调节作用。事实上,一些miRNA已经在功能上证明在模型作物水稻种子发育中起重要的调节作用,通过对其靶基因的表达产生负面调节的少量其他作物。例如,miR159,miR160,miR397,miR398和miR408积极调节米粒尺寸[31.,32.,33.,34.,35.],而miR1432、miR156、miR167、miR396c、miR396e、miR396f、miR530则在调节粒径方面发挥相反的作用[35.,36.,37.,38.,39.,40,41.].值得注意的是,miR408和miR160在调节种子大小方面也有保守作用拟南芥分别是烟草和棉花[42.,43.].除了种子大小的作用外,还证明了MiRNA来调节显影种子中的营养积累[4].例如,MiR160正常调节水稻种子中的淀粉积累[4,32.].EGMIR5179和CSA-MIR167A调节油棕中的油和亚麻酸生物合成Camelina Sativa种子,分别44.,45.].
鞑靼荞麦(Fagopyrum tataricum.)是一年生药物和食用的作物,属于犹太人家庭多元族科ee[46.].在亚洲和东欧广泛种植,特别是在中国西南部的山区[46.].苦荞籽是淀粉、蛋白质、膳食纤维、脂肪酸(亚油酸)和各种矿物质的良好来源[46.].重要的是,苦荞籽还含有丰富的黄酮类化合物,特别是芦丁,已被证明可以有效预防肝损伤,特别是炎症性肝损伤[43.,46.].因此,了解苦荞麦种子发育的分子机制,对苦荞麦高产优质育种具有重要意义[46.].到目前为止,已经有一些转录组分析报告研究苦荞种子发育的分子机制[4,43.,46.,47.,48.].然而,为了我们的知识,没有人在鞑靼荞麦中研究了MiRNA,而MiRNA是否以及如何调节季后赛荞麦种子的发展在很大程度上。在这项研究中,我们首先通过同源性鉴定和miRNA生物合成基因的同源性鉴定和系统发育分析,研究了与其他植物相比,在Tartary Buckfeeat中检测了MiRNA生物合成的养护演变。然后,我们鉴定了在甘油荞麦的显影种子中的已知和新的miRNA,并预测了它们的靶基因。最后,我们在MiRNA和mRNA表达之间进行了集成分析,以了解急性荞麦种子发育的miRNA介导的分子机制,并确定了季后血种子发育的关键miRNA-mRNA相互作用对。我们的结果提供了提高对急性荞麦种子发育的MiRNA介导的监管机制的有价值的信息,并援助Tartary荞麦种子改善。
结果
季后遇荞麦中miRNA生物合成基因的鉴定,系统发育和表达谱分析
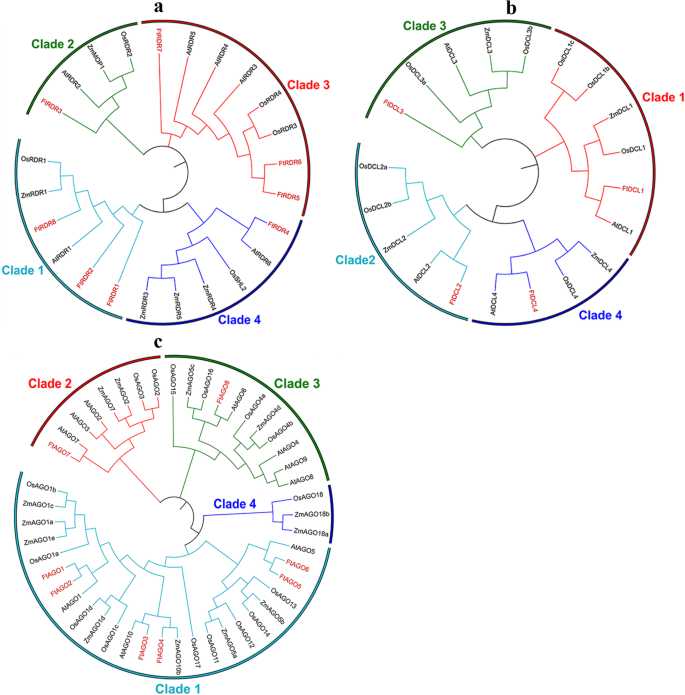
RDR,DCL,h,SE,母鸡,HST.和前基因已被证明在植物miRNA生物合成中发挥重要作用[5].作为在急性荞麦中研究miRNA的第一个也是最重要的一步,我们确定了在季后荞麦中这些基因的直脑。共8岁RDR,4DCL,1h,2SE,1母鸡,2HST., 8前在Tartary荞麦基因组中鉴定基因(附加文件1:表S1)分别。系统发育分析显示,植物RDR蛋白可以分为四个片状,如先前由Qian等人定义。[49.(图。1).在8个FtRDR蛋白中,3、1、3、1分别属于支1、支2、支3、支4。此外,FtRDR蛋白与答:芥RDR蛋白。DCl蛋白也可以归类为四组,4个FTDCL蛋白质显示一对一的对应物答:芥DCL蛋白(无花果。1).同样地,AGO蛋白也可以被分离为4个进化支,但是进化支4只包含Zhang等人定义的grass AGO蛋白[50(图。1).在苦荞AGO蛋白中,进化枝1是最大的进化枝,包含6个FtAGO蛋白。相比之下,两个进化支1和2只含有1个FtAGO蛋白(图。1).
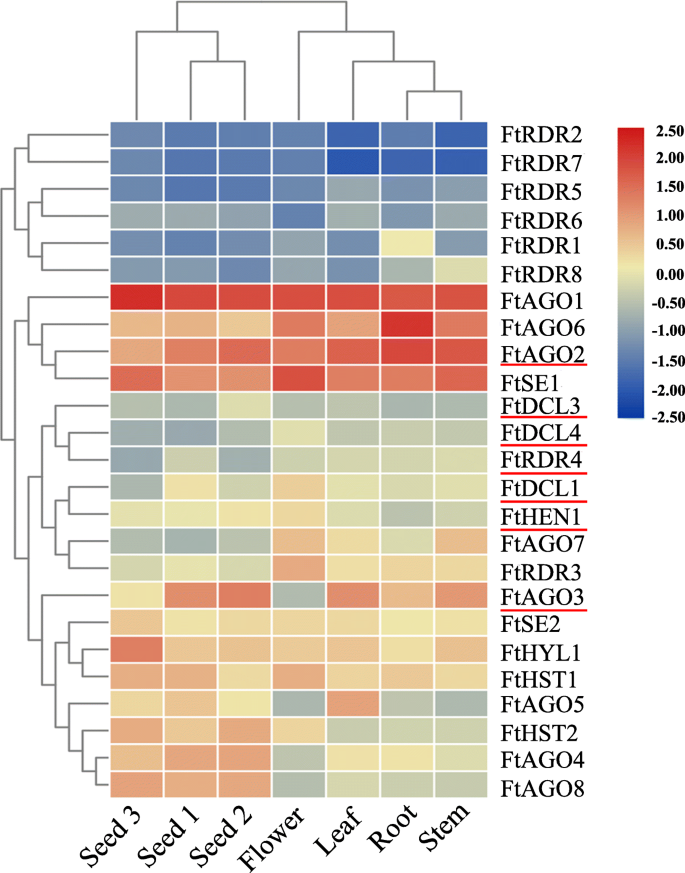
利用RNA-seq数据进一步研究了26个基因在根、茎、叶、花和种子发育过程中的表达谱。如图所示。2,最RDR基因(6)在所有组织中表达较低。相比之下,4个基因(FtAGO1,FtAGO2,FtAGO6,FtSE1)在所有组织中具有本构型高表达。剩余的16个基因以中等水平表达。其中,4个基因(Fthyl1.,FtHST2,FtAGO4,FtAGO8)在种子中表现出特别高的表达。此外,6个基因(ftdcl1.,FTDCL3.,FTDCL4.,FtAGO2,FtAGO3,和FtRDR4在种子发育过程中显示出显着的差异表达(| LOG2(折叠变化)| 1和FDR值<0.05)(图。2).6种差异表达的基因(DEGS)显示出在显影季耳荞麦种子(1)中的表达模式FtAGO2和FtAGO3仅在成熟期初期下调(S3), (2)ftdcl1.和FtRDR4在种子发育过程中呈持续下降趋势;FTDCL3.和FTDCL4.在峰填充阶段(S2)上调并在初始成熟度阶段下调(S3)(图。2).
鞑靼荞麦种子SRNA的测序
为了探讨MiRNA对季后荞麦种子发育的影响,我们在三种不同发展阶段的种子构建和测序六个SRNA文库(图。3.).在6个sRNA库中总共生成了1.2063亿原始读取(附加文件1:表S2)。过滤后,6个图书馆分别获得1389万、1709万、1719万、1454万、1364万、120万次clean reads(附加文件1:表S2)。显示大多数小RNA读数的长度分布为21-24 nt(附加文件2:图S1)。其中,24nt小RNA是最丰富的类型,在苦荞种子发育过程中表现出差异积累(附加文件)2:图S1)。为了进一步获得包含miRNAs的sRNA reads,将clean reads与Silv、GtRNAdb、Rfam和Repbase数据库进行比较,去除非编码rna(包括rrna、tRNAs、snrna和snoRNAs)和重复序列。剔除非编码rna和重复序列后,6个文库共获得1045万条、1416万条、1460万条、1305万条、680万条和563万条含有mirna的未注释sRNA reads(附加文件)1:表S2)。在这些未经发布的清洁读取中,共61.94,66.76,46.98,39.20,37.22和39.42%的读数分别映射到季后赛荞麦面参考基因组(附加文件1:表S2)。
已知和新的mirna的鉴定
为了鉴定正在发育中的苦荞种子中已知的mirna,将映射的sRNA reads用miRBase v21.0进行Blastn搜索。共鉴定出101个保守的miRNA,隶属于25个已知的miRNA家族1:表S3)。在这些家族中,17个家族包含2个以上成员,3个家族(MiR390、MiR399和MiR858)包含2个成员,其余5个家族(MiR159、MiR394、MiR397、MiR828和MiR845)仅包含1个成员。这些已知mirna的长度范围为19 ~ 22 nt,长度为19、20、21和22 nt的mirna分别为8、29、56和8(附加文件)1:表S3)。在这些被鉴定的保守mirna中,有29个mirna是高表达的,至少在一个文库中有超过1000个读计数(Additional file)1:表S3)。其余mirna以中等水平(36)或低水平(36)表达,每个库分别有< 10个读计数(附加文件1:表S3)。值得注意的是,最高表达的miRNA为ft - mir159,每个文库的读计数在14806 - 95,396之间。
为了进一步鉴定在显影荞麦种子中的新型miRNA,使用MIDDeep2程序分析未映射到季后荞麦基因组的剩余SRNA序列。结果,确定了总共129个新的miRNA(附加文件1:表S4)。这些新型mirna的长度范围为18 nt ~ 24 nt,最小自由能(AMFE)分布范围为−116.2 kcal moL- 1至−26.2千卡moL- 1(附加文件1:表S4)。在这些新的miRNA中,66个miRNA被分配到miRNA数据库中35个已知的miRNA家族中,其余63个miRNA与任何已知的miRNA家族都没有相似之处。与上述鉴定的保守mirna一样,超过三分之二的新mirna以中或高水平表达,最高表达为fta_novel_miR71 (Additional file)1:表S4)。
miRNA目标预测和功能分析
为了理解这些鉴定的miRNA的潜在功能,使用TargetFinder软件预测230 miRNA的推定靶基因。在213 miRNA中,共成功预测了3268个潜在的靶基因,其中包括来自101名已知的miRNA和1372个靶基因的2052个靶基因,来自112个新的miRNA(附加档案1:表S5)。这些预测的靶基因主要编码转录因子(TFS,285),蛋白激酶(254),磷酸酶,E3泛素 - 蛋白质连接酶,激素信号转导和其他细胞过程中的蛋白质,以及各种代谢中的酶(附加档案1:表S6)。这些miRNA的主要目标TFS在附加文件中显示1:表S7和前三个TFS分别为MYB(65),AP2 / ERF(18)和NAC(18)。与先前研究的结果一致,一些保守的miRNA靶向已知的TFS。例如,miR156,miR160,miR164,miR171,miR172,miR319,miR396和miR858分别靶向SPL,ARF,NAC,GRAS,AP2-ERF,TCP,GRF和MYB TFS(附加文件1:表S7)。另外,基于推定靶基因的同源搜索,发现61 miRNA的预测靶基因,包括39个保守和22个新的miRNA,是47个已知种子或器官尺寸基因的同源基因(附加文件1:表S8)。其中,39个保守miRNA来自14个已知miRNA家族,包括MiR156/157(10)、MiR159(1)、MiR162(1)、MiR164(1)、MiR166(2)、MiR167(1)、MiR168(4)、MiR169(1)、MiR172(5)、MiR319(2)、MiR390(2)、MiR395(2)、MiR396(4)和MiR530(3)。除了MiR156/157和MiR396家族成员分别靶向种子大小SPL和GRF转录因子外,大多数mirna最初被发现靶向已知的种子大小或器官大小基因。此外,基于同源搜索,鉴定了8个miRNAs(4个已知和4个新)靶向类黄酮生物合成的结构或调控基因(附加文件1:表S9)。其中,fta_novel_miR26靶向苯丙氨酸解氨酶(朋友),fta_novel_mir74靶向4-香豆素-CoA连接酶(4 cl),FTA-MiR394A-5P靶向黄酮合成酶(FLS的), fta_novel_miR1靶向的同源基因AtMYB123fta_novel_miR58靶向调控花青素生物合成的同源基因AtbHLH42/TT8fta-miR828-5p靶向的同源基因ATMYB75./90./113./114.调节花青素生物合成。此外,FTA-miR858a-5p和fta-mir858b-3p靶向13和14个Myb tfs,分别参与了黄酮酚生物合成和花青素生物合成的调节。
发展季后赛荞麦种子及其目标的DEM
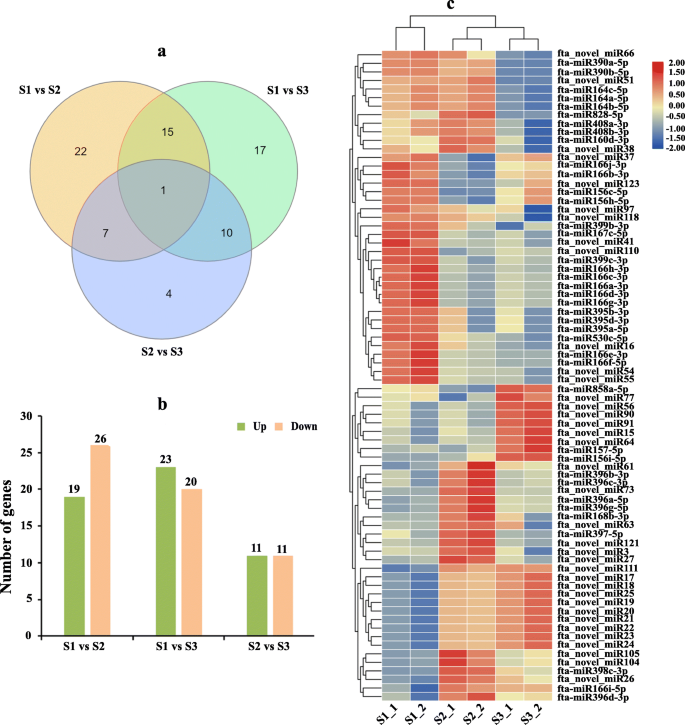
为了鉴定苦荞麦种子发育相关的mirna并了解其潜在的调控机制,通过比较TPM表达值鉴定了dem。共76个mirna,包括39个保守mirna和37个新mirna,表现出显著的差异表达(图2)。4).在这些45,43和22个miRNA中,在S1和S2,S1和S3和S2和S3之间的比较中被发现(图。4a).与S1期相比,S2期和S3期分别有1个miRNA (fta_novel_miR110)差异表达,S2期和S3期分别有22个和17个特异表达miRNA(图2)。4a).与S2期相比,S3期有4个特异差异表达的mirna(图2)。4a)。此外,S1期和S2期之间,分别有19个和26个mirna上调或下调(图2)。4b).在S1和S3比较中,有23个上调的mirna和20个下调的mirna。在S2和S3比较中,分别有11和11个mirna上调或下调(图2)。4b)。
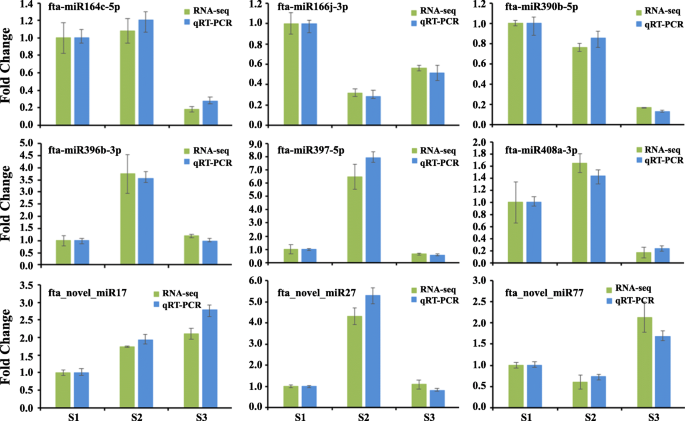
各dem的表达式热图如图所示。4C。在这些DEMS中,26,18和9个MIRNA分别在初始填充阶段,峰填充阶段和初始成熟阶段的特异性高表达。12个MiRNA在初始填充阶段和峰填充阶段表现出高表达。值得注意的是,在种子发育过程中,9种新的miRNA(FTA_Novel_mir17-25)的表达持续增长(图。4c).为验证miRNA测序结果,采用茎环定量RT-PCR技术检测苦荞麦种子发育过程中9个dem的表达水平。如图所示。5,结果与在miRNA测序中获得的结果广泛一致。
为了更好地理解这些DEM的功能,研究其预测的靶基因并进一步进行基因本体(GO)和基因组(KEGG)途径分析的基因本体论(GO)和京都百科全书。在74个DEM中确定了总共1534个靶基因(附加档案1:表S5)。其中,在S1与S2、S1与S3、S2与S3的比较中,分别有44、41和22个dem对850、765和735个基因进行了靶向。GO分析显示,在S1 vs. S2、S1 vs. S3和S2 vs. S3比较中,分别有514、448和419个目标被分配到生物过程、细胞成分和分子功能类别(附加文件)2:图S2)。每个比较的生物过程,细胞组分和分子函数类别中有18,14和14个术语。“代谢过程”,“细胞部分”和“催化活动”是这三类的最大丰富术语(附加档案2:图S2)。KEGG分析显示,在S1 vs. S2, S1 vs. S3, S2 vs. S3比较中,共有332,309和272个目标基因被分配到61、54和55个KEGG通路中(附加文件)1:表S10)。其中,“氨基酸生物合成”(ko01230)、“淀粉和蔗糖代谢”(ko00500)、“苯丙类生物合成”(ko00940)、“嘌呤代谢”(ko00230)和“氨基糖和核苷酸糖代谢”(ko00520)是主要的代谢途径。此外,“植物激素信号转导”(ko04075)中有大量的靶基因。每个比较的前50条路径显示在附加文件中2:图S3。
鉴定与急性荞麦种子发育相关的关键miRNA-mRNA对
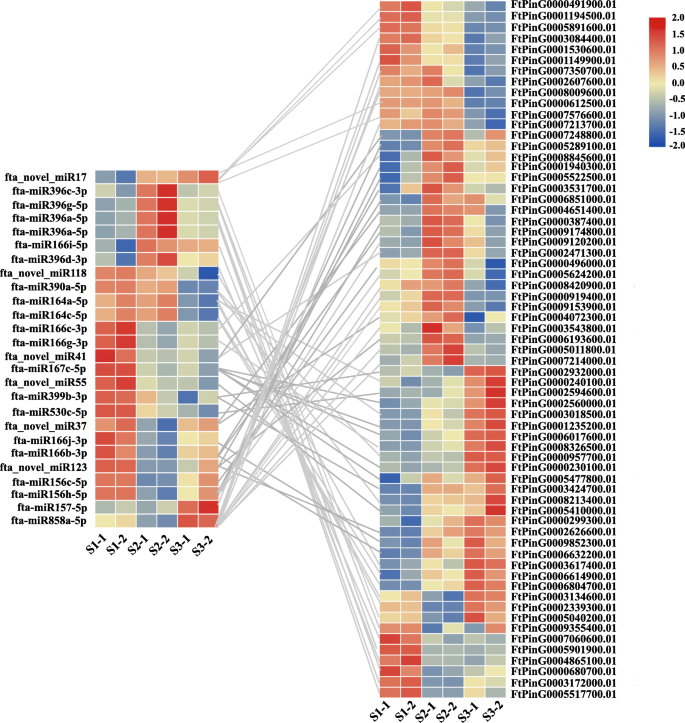
为了鉴定与苦荞麦种子发育相关的miRNA-mRNA对,我们对苦荞麦种子发育过程中差异表达的靶mrna和dnas进行了表达相关性分析。基于我们之前的转录组数据[46.,在苦荞种子发育过程中,74个dem的1534个靶基因中有439个表达差异显著。439个DEGs与相应的dem进行相关性分析,发现117对miRNA-mRNA呈负相关(附加文件)1:表S11)。其中,由25 miRNA和65个靶基因组成的65 miRNA-mRNA对显示出显着的表达负相关(R≥0.5,P < 0.05) (Table1和无花果。6).通过对这65个靶基因的同源注释,得到了56对miRNA-mRNA的同源功能注释(表1)1).发现这些56 miRNA-mRNA对有显着涉及细胞分化和增殖,细胞伸长,激素反应和平衡,组织组织,胚胎和胚乳的发育,矿物质元素(PI,Fe和Mn)和脂肪酸和黄酮类化合物的运输生物合成(表格1).此外,在这65个靶基因中鉴定出2个钙结合蛋白、1个受体样激酶、1个cbl相互作用蛋白激酶和1个SUMO蛋白酶,尽管在其他植物中未发现其同源物参与种子发育(见表)1).值得注意的是,在这些已鉴定的miRNA-mRNA对中,有5对miRNA-mRNA对,包括fa - mir156c -5p-FtPinG0000496000.01,fta-mir167c-5p-FtPinG0002560000.01fta-miR396a-5p -FtPinG0000680700.01fta-miR530c-5p -ftping000259-4600.01, fta_novel_miR17ftping0007576600.01,可能参与苦荞种子大小调控,因为目标mrna与已知的水稻种子大小相关基因同源(表)1).此外,FTA-MiR858A-5P与几种MYB TFS表现出显眼的负相关,这是植物类黄酮生物合成的公知稳压剂(表1).
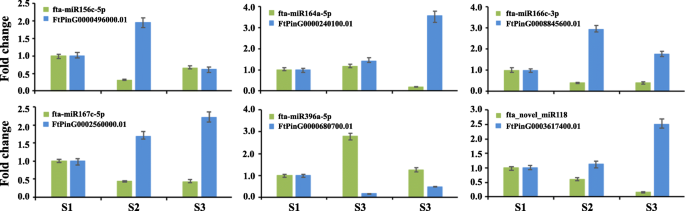
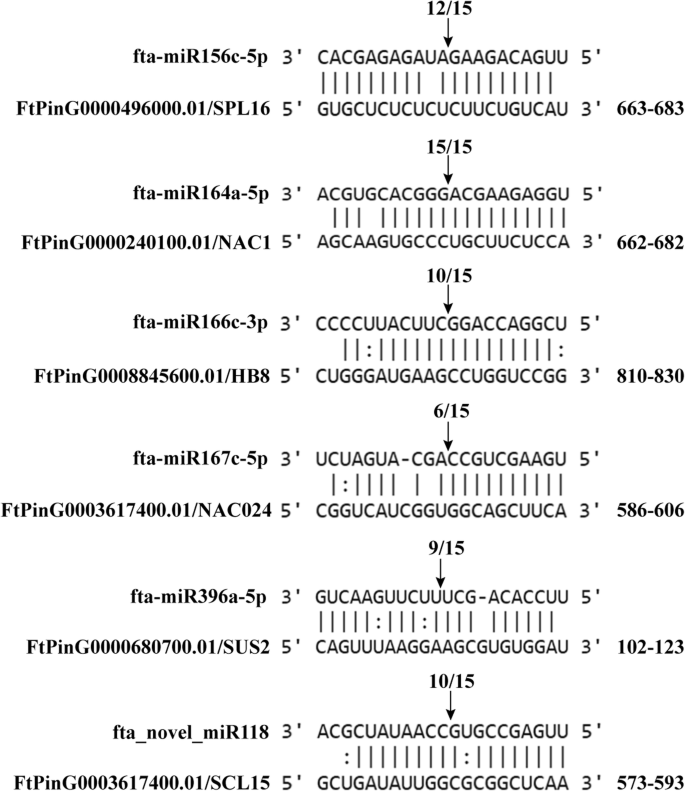
QRT-PCR和5'-RLM-RACE验证与季后赛荞麦种子发育相关的关键miRNA-mRNA对
为了进一步证实与季后荞麦种子发育相关的鉴定的关键miRNA-mRNA对,进行QRT-PCR和5'-RLM族进行分析FTA-MIR156C-5P-FtPinG0000496000.01fta-miR164a-5p -FtPinG0000240100.01,FTA-MIR166C-3P-FTPing0008845600.01,FTA-MIR167C-5P-FtPinG0002560000.01fta-miR396a-5p -FtPinG0000680700.01, fta_novel_miR118FtPinG0003617400.01对,分别参与种子大小、细胞分化和增殖、生长素反应。qRT-PCR结果显示,这些mirna的表达与相应的靶基因表达呈明显的负相关(图1)。7).5 ' -RLM-RACE结果表明,这些mirna能够切割其相应的靶基因(图2)。8).
讨论
MiRNA,基因表达的类负调节因子,在包括种子发展的许多植物发育过程中起关键作用[10.,51].然而,是否以及如何调控苦荞麦种子的发展,目前还没有定论。为了更好地理解mirnas调控苦荞种子发育的分子机制,我们系统地鉴定了苦荞基因组中的miRNA生物合成基因和苦荞种子发育过程中的miRNA。更重要的是,我们对苦荞种子发育过程中的miRNA和靶点mRNA表达谱进行了整合分析,确定了苦荞种子发育的关键miRNA-mRNA对。
鞑靼荞麦基因组中的miRNA生物合成灭绝
大量研究表明,几种不同的基因,包括RDR,DCL,h,SE,母鸡,HST.,前,在植物miRNA生物合成中发挥至关重要的作用[5].在这项研究中,我们确定了8RDR,4DCL,1h,2SE,1母鸡,2HST., 8前苦荞基因组中的基因系统发育分析表明,8个RDR、4个DCL和8个AGO分别可分为4、4和3个亚科。结果与之前对其他植物的研究结果一致[6,49.,50,52,53[并表明这些植物中这些基因家族的演变是保守的。表达分析显示1前例如FtAGO1这些基因在种子中的表达量最高,1前例如FtAGO4在种子中表现出特定的高表达和2前例如FtAGO2和FtAGO3在种子发展过程中表现出显着的差异表达。尤其,OsAGO17,它们与这四种植物属于同一亚科FTAGO.基因,已经在功能上被证明可以正向调节水稻的粒大小和粒重[54,55].这表明这四个FTAGO.基因也可能起到类似的作用OsAGO17在鞑靼荞麦种子发展中。另外,我们也发现了Fthyl1.,FtHST2,FtAGO8在种子中特别高表达,和ftdcl1.,FTDCL3.,FTDCL4.,FtRDR4在种子发育过程中有明显的差异表达。这表明这些miRNA生物合成基因在苦荞种子发育中也可能具有重要的调控作用。
苦荞种子发育过程中srna的特性
通过对3个不同发育阶段种子的6个sRNA文库的测序,我们获得了大量长度为18-30 nt的sRNA。其中,这24个nt srna在苦荞种子发育过程中最为丰富,这与之前在许多植物种子发育过程中观察到的情况相似[6,56].在苦荞种子发育过程中,24个ntsrna表现出不同程度的积累。这表明24个nt srna可能在苦荞等植物种子发育过程中发挥重要作用。据报道,许多24-nt sRNAs是异色sirna (hetsinas),它通过DNA甲基化(RdDM)介导转录基因沉默[56].一般来说,需要从细胞质中导入24nt srna才能将其甲基化,这一过程是由AGO4介导的[57].值得注意的是,我们的研究发现FtAGO8,这是AGO4在苦荞种子发育过程中表现出特殊的高表达。这些观察表明FtAGO8Might的作用类似于AGO4在苦荞种子发育过程中,这些24-nt srna可能通过RdDM途径介导转录基因沉默。
众所周知和新的miRNA及其靶基因在发育季荞麦种子中
在Tartary荞麦基因组中,通过基因组的生物信息学分析预测了278 miRNA [58].在苦荞种子发育过程中,共鉴定出230个mirna,其中101个为已知mirna, 129个为新mirna。在mirna中,超过三分之二的表达在高水平和中水平。这表明苦荞基因组中的大部分mirna参与了苦荞种子的发育,进一步说明苦荞种子的发育是一个极其复杂的生物学过程。值得注意的是,fta-miR159在发育中的苦荞种子中表达量最高。在草莓中,Fa-miR159b和Fa-miR159b在发育果实中表达较高,并在果实发育中起关键调控作用[4,59].在水稻中,osa-miR159在发育中的种子中也有更高的表达,并正调控粒长和粒宽[4,20.,33.,35.].因此,我们的研究结果表明,ft - mir159在苦荞种子发育过程中可能也具有重要的调控作用。
使用TargetFinder软件,我们确定了来自213 miRNA的3268个潜在的靶基因。在这些靶基因中,最大数量是编码的TFS。值得注意的是,在具有靶基因编码的TFS的这些miRNA中,一些miRNA,例如miR156,miR160,miR396和miR858分别靶向SBP,ARF,GRF和MYB TFS。有趣的是,这些miRNA已经通过实验验证以靶向其他植物中的这些相应的TFS [32.,36.,37.,60].这表明这些miRNA靶在不同的植物中受到高度保守,并进一步证实了我们研究中的目标鉴定的高可靠性。基于对预测的靶基因的同源搜索,61 miRNA具有靶基因是47个已知种子或器官尺寸基因的同源基因[61].值得注意的是,在这些miRNA中,除了靶向已知的种子尺寸SPL和GRF TFS的miR156和miR396之外,大多数鉴定靶向已知的种子或器官尺寸基因。22.,32.,36.].这表明这些mirna可能在苦荞种子大小中发挥重要的调控作用,这些已知的种子或器官大小调控基因中大多数可能存在转录后调控种子大小的机制。在植物中,有报道称mirna通过调控类黄酮生物合成的结构靶标或调控基因调控类黄酮生物合成[62].在我们的研究中,鉴定了3个miRNA(FTA_NOVEL_MIR26,FTA_NOVEL_MIR74和FTA-MIR394A-5P)以靶向黄酮类生物合成结构基因朋友,4 cl,FLS的,分别。此外,我们还发现了5个mirna (fta_novel_miR1, fta_novel_miR58, ft - mir858a -5p, ft - mir858a -5p,和ft - mir858b -3p)靶向已知类黄酮生物合成调控基因的同源基因拟南芥[63,64,65,66].这些暗示这些miRNA可以通过调节类黄酮化生物合成的结构或调节基因的表达来调节季节性荞麦面的黄酮类生物合成。
在季后赛荞麦种子发展期间的DEM
在许多植物中,在显影种子中鉴定了更多的DEM [4,6,18.,19.,20.,21.,22.,23.,24.,25.,26.,27.,28.,29.,30.].在苦荞种子发育过程中,我们鉴定了76个dem。表达谱分析表明,这些dem在苦荞种子发育过程中具有阶段特异性的高表达模式,提示这些基因型可能在苦荞麦种子发育的特定阶段发挥调控作用,相同表达模式的mirna可能在苦荞麦种子发育过程中发挥类似的调控作用。值得注意的是,在许多种子植物的种子发育过程中也发现miR156、miR160、miR166、miR167、miR168、miR395、miR396、miR397、miR398、miR399和miR408等mird存在差异表达[4,18.,19.,21.,22.,23.,24.,25.,26.,27.,28.,29.,30.].在水稻中,miR156、miR160、miR167、miR396、miR397、miR389和miR408已经被证实具有调节水稻种子发育的功能[31.,32.,33.,34.,35.,36.,37.,38.,40].这些表明,这些已知的miRNA是不同植物种子发育调节中的关键和保守的miRNA,也可以用作滋养荞麦种子发育的候选MiRNA。此外,在与其他种子植物相比,37种新的miRNA和几个保守的miRNA,包括miR530,miR828和miR858,在发展季后荞麦种子时,在开发季耳荞麦种子中的特异性表达。这表明这些miRNA可能在季后血种子发育中具有特定的监管功能。此外,所有这些信息表明,种子发育中的保守和多样的miRNA介导的调节机制可能存在于不同的种子植物中。
miRNA与靶mRNA的整合分析
整合miRNA及其靶mRNA表达分析有助于理解miRNA的功能,识别与种子发育相关的功能性miRNA-mRNA对[67].据我们所知,在植物种子发育以前研究的MiRNA和靶mRNA没有整合分析。在这项研究中,我们鉴定了65个显着负相关的miRNA-mRNA对(R≥0.5,P< 0.05),由25个mirna和65个相应的靶基因组成。通过对这些mirna对应的靶基因进行同源查询,获得了56对miRNA-mRNA的功能注释。这些miRNA-mRNA对参与不同方面,最显著的是细胞分化和增殖、细胞伸长、激素反应和平衡、器官发生、胚胎和胚乳发育、矿质元素运输和积累以及类黄酮生物合成。以上结果表明,这些miRNA-mRNA对是苦荞种子发育的关键miRNA-mRNA对。
在米饭,OsSPL16[68,69],OsNAC024[70],奥苏斯[71],OsGIF1[38.,72),而OsAGO17[54基因已经在功能上被证明可以调节种子的大小。在这项研究中,我们发现目标mrna从5 miRNA-mRNA双(fta-miR156c-5p-FtPinG0000496000.01、fta-miR167c-5p-FtPinG0002560000.01 fta-miR396a-5p-FtPinG0000680700.01, fta-miR530c-5p-FtPinG0002594600.01和fta_novel_miR17-FtPinG0007576600.01)这5个水稻种子大小是同源的基因,分别这说明这些基因可能对苦荞种子大小具有保守的调节作用,相应的mirna是控制苦荞种子大小的关键mirna。值得注意的是,到目前为止,在以往的研究中,这些种子大小的基因尚未被确定为任何mirna的靶基因。因此,我们的研究结果为这些种子大小基因的转录后调控提供了第一手资料。众所周知,Ca2+信号转导途径在许多发育过程中起重要作用,包括植物种子发育[73,74].在这项研究中,我们发现56对miRNA-mRNA中有3对参与了Ca2+这是因为靶mrna分别与钙结合蛋白和cbl相互作用蛋白激酶同源。这表明这些mirna可以通过Ca调控苦荞种子的发育2+信号转导通路。在前期研究中,我们发现苦荞麦种子发育过程中总黄酮含量是动态积累的,并鉴定出一个SG7亚群R2R3-MYB TF基因(ftping0009153900.01)是在季后润荞麦种子中黄酮类生物合成的关键调节基因[46.].有趣的是,在这项研究中,我们发现了ft - mir858a -5p-ftping0009153900.01对显示显着表达负相关。此外,FTA-MiR858A-5P还与orthoologs表现出显着的负相关Arabidopsis Thaliana MyB123,这是发展种子中的原霉素积累的着名调节剂[64].这些表明,Tarrary荞麦种子的类黄酮生物合成涉及MiRNA介导的转录后调节,FTA-MiR858A-5P是关键调节剂。
为了进一步验证与Tartary荞麦种子发育相关的这些鉴定的miRNA-mRNA对的可靠性,我们在参与种子大小,细胞分化和涉及种子大小的6 miRNA-mRNA对中进行QRT-PCR和5'-RLM分析。增殖和生长素反应。这些结果表明,这些miRNA的表达及其相应的靶基因明显呈负相关,并且可以切割它们相应的靶基因。上述结果表明,这些鉴定的miRNA-mRNA对可以被认为是用于调节季后荞麦种子的发育的候选miRNA-mRNA对,并且还表明,组合小的RNA和转录组分析是识别关键miRNA的有效方法参与植物种子发展。
结论
本研究首次尝试整合miRNA和mRNA的表达数据,以鉴定苦荞种子发育过程中关键的调控miRNA-mRNA对。在苦荞中首次鉴定出230个mirna,其中101个为已知mirna, 129个为新mirna。在苦荞种子发育过程中,有76个mirna表达差异显著,鉴定出1543个靶基因。此外,还鉴定出56个靶基因对应的25个miRNA-mRNA对,为苦荞种子发育的关键候选miRNA-mRNA对。本研究中mirna和靶mrna的综合分析不仅首次为苦荞种子发育中mirna介导的调控提供了新的见解,同时也为进一步研究这些候选mirna及其靶点在苦荞种子发育中的作用和苦荞种子改良提供了基础。
方法
植物材料和样品制备
Tarrary Buckwheat品种Jinqiao No.2,该研究是从贵州师范大学(贵州贵州贵阳)的荞麦工业技术研究中心获得的。用于SRNA测序的种子样本与我们之前的转录组分析相同[46.].简而言之,授粉(DAP)后12天(S1:初灌浆期)、15天(S2:灌浆高峰期)和21天(S3:初成熟期)采集种子[46.].
季后血基因组中miRNA生物合成基因的鉴定,系统发育和表达谱分析
鞑靼荞麦基因组信息从中下载了http://www.mbkbase.org/Pinku1[58].注释基因的所有蛋白质序列用于在BioEdit软件(7.0.9.0版)中构建局部蛋白质Blast数据库。蛋白质序列RDR1- - - - - -6,DCL1- - - - - -4,AGO1- - - - - -10.,亨1,HYL1,SE,HST.基因拟南芥用作查询以使用电子值执行本地BLASTP
RNA提取,SRNA文库构建和测序
如前所述,从三个样本中提取总RNA [46.],用于sRNA和qRT-PCR检测。分别用1.2%琼脂糖凝胶电泳和NanoDrop 2000c分光光度计(NanoDrop, Wilmington, DE, USA)检测RNA质量和浓度。
为了构建和测序sRNA文库,每个样本进行两个重复。首先,用聚丙烯酰胺凝胶电泳(PAGE)从总RNA中分离出srna。然后,利用T4 RNA连接酶(TaKaRa, Dalian, China)将分离的srna分别添加到5 ' RNA适配器和3 ' RNA适配器上。然后用RT-PCR将添加5 '和3 ' RNA适配器的srna逆转录为单链cDNA。然后利用适配引物进行PCR扩增,将单链cDNA合成为双链cDNA。最后,PCR产物得到纯化,并使用Biomarker Technologies Co., Ltd. (Beijing, China)的Illumina SE50系统进行高通量测序。
测序数据的生物信息学分析
通过去除低质量读、包含poly-N或poly-A序列的读、缺少3 '适配器序列的读、长度< 18 nt或> 30 nt的读和适配器序列,原始读被处理以生成干净读。然后将这些清晰的基因序列与苦荞基因组(http://www.mbkbase.org/Pinku1/)[58].利用Silva数据库、GtRNAdb数据库、Rfam数据库和Repbase数据库对rRNA、tRNA、snRNA和snoRNA进行过滤,生成包含miRNA的未注释reads [4].未经发布的读被抨击miRBase以搜索已知的miRNA [77].利用miRdeep2程序预测了潜在的新mirna [78].mifam.dat(http://www.miRbase.org/ftp.shtml)用于研究这些鉴定的miRNA的miRNA家族类别。通过每百万(TPM)的转录物计算和标准化MiRNA表达丰度,并且通过DDSeq R封装进行差异表达分析,具有FDR(假发现率)值<0.05和log2(折叠变化)| > 1 as the threshold for significant difference. The potential target genes of miRNA were predicted by using TargetFinder software [79].对预测的靶基因进行氧化石墨烯(http://geneontology.org/)[80]和kegg(www.kegg.jp/kegg/kegg1..html) [81.路径分析以预测和分类可能的功能。
鉴定Tarrary荞麦种子发育的关键miRNA-mRNA对
利用我们之前报告的转录组数据分析了所有dem靶基因的表达谱[46.].然后通过GO、KEGG和同源注释对差异显著的表达目的基因进行功能分析。最后,进行dem与差异表达靶基因的表达相关性分析。mirna和靶mRNA配对R≥0.5和P值< 0.05,通过靶mrna的同源注释获得苦荞麦种子发育的关键miRNA-mRNA对。
QRT-PCR对DEM的验证及其不同表达的靶基因
通过使用Easyspin Plus植物RNA试剂盒(AIDLAB,北京,中国)和DNASE I(大连,大连,中国)消化,从三种不同发育阶段中分离出总RNA分离出来的三种不同的发育阶段。然后,在制造商的指示之后,使用麦克金斯miRNA第一链CDNA合成套件(天根,北京)逆转2μg总RNA被逆转录成单链cDNA。使用Mircute Plus MiRNA QPCR套件(Sybr Green)(Tiangen,北京,中国)对CFX96实时系统(Bio-Rad,USA)进行QRT-PCR。使用了三个独立的生物重复,而且FtU6作为内参基因。用2 . 2计算各miRNA的相对表达量-ΔΔct方法。本实验中使用的所有引物列于附加文件中1:表S12。
对于不同表达的靶基因,通过使用Primescript™RT主混合物(Takara,北京,中国)和TBGreen®PremixExTaq™(TLI RNASEH Plus)进行逆转录转录和QRT-PCR反应进行逆转录和QRT-PCR反应(Takara,北京,中国)分别。FtActin作为内参基因。所有样品进行三个独立的生物学重复。利用2-ΔΔct方法。本实验使用的引物列在附加文件中1:表S12。
验证mirna对预测目标的定向切割
5'-RLM族方法用于研究其预测靶mRNA的miRNA定向切割体外。将总RNA连接到RNA适配器上,并根据制造商的说明,使用FirstChoice®RLM-ram-race(Ambion)逆转录。如前所述进行了5'-RLM比赛,由Deboer等人进行。[6].本实验使用的引物列在附加文件中1:表向。
数据和材料的可用性
支持本文结论的数据集包含在本文及其附加文件中。调查中提供的数据集和材料可由通讯作者要求获得。
缩写
- DEMS:
-
差异化的mirnas
- 存在:
-
定量实时聚合酶链反应
- 5 ' -RLM-RACE:
-
连接酶介导的5 ' cDNA末端快速扩增
- microrna:
-
小分子核糖核酸
- SRNA:
-
小rna
- DCL1:
-
像偶然的1
- Hyl1:
-
偏下性的叶1
- SE:
-
锯齿状的
- HEN1:
-
华源人1
- AGO1:
-
Argonaute 1
- RISC:
-
RNA-induced沉默复杂
- 度:
-
差异表达基因
- TFs:
-
转录因子
- hetsirnas:
-
异色谱系
- AMFE:
-
最小自由能量
- 朋友:
-
苯丙氨酸ammonia-lyase
- 4 cl:
-
4-香豆素 - COA连接酶
- 弗尔斯:
-
黄酮醇合成酶
- 走:
-
基因本体论
- KEGG:
-
Kyoto基因和基因组的百科全书
- DAP:
-
几天后授粉
参考文献
- 1。
Filipowicz W,Bhattacharyya Sn,Sonenberg N. MicroRNAS后转录后调节机制:是视线的答案吗?NAT Rev Genet。2008; 9(2):102-14。
- 2。
植物microrna的起源、生物起源和活性。细胞。2009;136(4):669 - 87。
- 3.
何昕,王伟,徐胜,等百合lancifolium研究。通过高通量测序。Sci众议员2018;8(1):2880。
- 4。
李H,Sun X,Ma C,Meng H,Chen Q.常见荞麦中微小RnA的鉴定与表征(Fagopyrum esculentum)种子发展。国际农业生物学杂志。2020;24:1115-24。
- 5.
余勇,贾涛,陈晓霞。植物microrna的“如何”和“在何处”。新植醇。2017;216(4):1002 - 17所示。
- 6.
DeBoer K, Melser S, Sperschneider J, Kamphuis LG, Garg G, Gao LL, Frick K, Singh KB。狭叶卢平的鉴定和摹写(卢比斯angustifolius) microrna在种子发育过程中的作用。BMC基因组学。2019;20(1):135。
- 7.
梁东,张志强,尹伟,夏晓霞。干旱胁迫下胡杨微rna的高通量测序鉴定及其降解组分析。BMC基因组学。2013;14:233。
- 8。
吴国平,吴国平。核加工与microrna出口技术研究拟南芥.Proc Natl Acad Sci U S A A. 2005; 102(10):3691-6。
- 9。
李斌,秦义,段H,尹W,x X.基因组 - 宽的新和干旱胁迫响应微大车Populus Euphratica..J Exp Bot。2011; 62(11):3765-79。
- 10。
D'Ario M,Griffiths-Jones S,Kim M.小RNA:对植物开发的影响大。趋势植物SCI。2017; 22(12):1056-68。
- 11.
盛杰,郑X,王继,刘某,庞米,周F等人。通过高通量测序和差异表达分析的微小RNA及其目标miscanthus.物种。ind crop prod。2018; 118:196-205。
- 12.
李杰,丁J,yu x,李h,阮cj。临界微小瘤转录因子调节模块鉴定及表达分析,与种子发育和油积累中的种子发育和溢油中的种子。ind crop prod。2019; 137:33-42。
- 13。
陈成,钟捷,玉孚,徐米序列识别麦芽瘤及其参与萜类化合物生物合成的靶基因Cinnamomum camphora..ind crop prod。2020; 145:111853。
- 14。
杨y,guo j,cheng j,姜z,xu n,x等。UV-B辐射响应性微大腹腔及其菊花靶基因的鉴定(菊花使用高通量测序。农业科学与技术。2020;151:112484
- 15.
叶宇军,王建伟,倪志新,孟X,冯宇辉,杨志强,等。小RNA和降解组测序揭示miRNAs在马尾松球果发育中的作用Pinus Massoniana.).ind crop prod。2020; 154:112724。
- 16.
李C,张班。植物开发控制中的微大稻草。J Cell Physiol。2016; 231:303-13。
- 17.
Savadi S.作物植物中种子发展的分子调节和工程种子大小的策略。植物生长调节。2018; 84:401-22。
- 18.
MA X,Zhang X,Zhao K,Li F,Li K,Ning L等。小RNA和降低深度测序揭示了MicroRNA在花生种子膨胀中的作用(arachis hypogaea.L.)。前植物SCI。2018; 9:349。
- 19。
朱庆华,张志强,张志强,等。一组不同的microrna和类似microrna的小rna在稻米发育中。基因组研究》2008;18(9):1456 - 65。
- 20。
彭T,太阳H,乔米,赵Y, Du Y,张J, et al。水稻种子发育过程中microRNA群的表达差异可能是导致弱穗籽粒灌浆不良的原因之一。BMC Plant Biol. 2014;14:196。
- 21。
金X,Fu Z,LV P,Peng Q,Ding D,Li W等人。玉米籽粒灌浆过程中微小RNA的鉴定与表征。Plos一个。2015; 10(5):E0125800。
- 22.
李东,刘震,高磊,王磊,高敏,焦震,等。小麦籽粒发育中microrna的全基因组鉴定和特性研究玉米acta optica sinica, 2016; 32(4): 457 - 461。
- 23.
刘洪辉,王凯,刘丽丽,王胜,赵艳,等。小麦粒中发育相关的microrna (Triticum aestivum.L.)。BMC植物BIOL。2013; 13:140。
- 24.
侯g,du c,gao h,liu s,sun w,lu h等。鉴定小麦籽粒在潜在参与调节晶粒特征和对氮水平响应的粒子籽粒中的鉴定。BMC植物BIOL。2020; 20(1):87。
- 25.
Curaba J,Spriggs A,Taylor J,Li Z. Helliwell C miRNA规定在大麦种子的早期发展中。BMC植物BIOL。2012; 12:120。
- 26.
白斌,石斌,侯宁,曹颖,孟Y,卞洪,等。大麦种子发育和萌发过程中microrna参与基因表达调控和激素交互作用。植物学报。2017;17(1):150。
- 27.
宋QX,刘毅,胡XY,张WK,MA B,Chen Sy,等。深度测序鉴定麦芽瘤及其靶基因的鉴定。BMC植物BIOL。2011; 11:5。
- 28.
yu l,guo r,jiang y,ye x,yang z,meng y等。大豆种子发育中新型MicroRNA的基因组鉴定与表征。Biosci Biotechnol Biochem。2019; 83(2):233-42。
- 29。
王建军,王涛,李军,李超,等。开发中积极参与脂肪酸生物合成的microrna的鉴定芸苔属植物显著使用高通量测序的种子。前植物SCI。2016; 7:1570。
- 30。
王魏W,李G,江X, Y, Z,妞妞Z,等。小RNA和降解谱涉及种子发育和油脂合成芸苔属植物显著.《公共科学图书馆•综合》。2018;13 (10):e0204998。
- 31.
张永成,于勇,王春英,李志勇,刘强,徐军,等。过表达microRNA OsmiR397通过增加水稻粒级和促进穗分枝来提高水稻产量。生物科技Nat》。2013;31(9):848 - 52。
- 32.
黄俊,李志,赵丹丹。OsmiR160靶基因的调控osarf18通过改变生长素信号导致水稻生长发育缺陷。Sci众议员2016;6:29938。
- 33.
张建平,于勇,冯永忠,周云飞,张飞,杨宇文,等。MiR408通过植物蓝蛋白调控粮食产量和光合作用。植物杂志。2017;175(3):1175 - 85。
- 34。
张浩,张建军,闫建军,苟飞,毛勇,唐国光,等。短串联目标模拟水稻系揭示mirna调控重要农艺性状的功能。中国科学(d辑:地球科学)2017;
- 35。
彭涛,乔明,刘海涛,张志强,赵永强。利用短串联目标模拟技术在模型和作物植物中灭活microrna的资源。摩尔。2018;11(11):1400 - 17所示。
- 36。
焦宇,王颖,薛东,王健,严明,刘国光,等。的监管OsSPL14由OsmiR156定义了水稻理想的植株结构。Nat麝猫。2010;42(6):541 - 4。
- 37。
段鹏,倪胜,王军,张斌,徐锐,王勇,等。的监管OsGRF4利用OsmiR396控制水稻籽粒大小和产量。Nat植物。2015;2:15203。
- 38。
李士,高f,谢克,曾x,cao y,zeng j等。OSMIR396C-OSGRF4-OSGIF1调节模块确定水稻中的晶粒尺寸和产量。植物Biotechnol J. 2016; 14(11):2134-46。
- 39.
赵云飞,彭涛,孙洪涛,Teotia S,温海林,杜玉祥,等。miR1432-OsACOT(酰基辅酶a硫酯酶)模块通过提高水稻灌浆速率来决定产量。植物生态学报;2019;17(4):712-23。
- 40。
缪灿,王东,何锐,刘胜,朱家强。MIR396e和MIR396f的突变增加水稻的粒级并调节水稻的茎结构。植物生态学报;2018;29(2):491 - 497。
- 41。
孙伟,徐晓华,李勇,谢林,何勇,李伟,等。OsmiR530作用于OsPIL15调节水稻的粮食产量。新植醇。2020;226(3):823 - 37。
- 42。
Pan J,Huang D,Guo Z,Kuang Z,Zhang H,Xie X等。MicroRNA408的过度表达增强了不同植物中的光合作用,生长和种子产量。J Intent Plant Biol。2018; 60(4):323-40。
- 43。
刘敏,马铮,郑涛,孙伟,张勇,金伟,等。苦荞麦生理变化与种子发育相关性研究(Fagopyrum tataricum.盖特。)。BMC基因组学。2018; 19(1):648。
- 44。
油棕中果皮Egmir5179Elaeis guineensisJACQ。)通过靶向NAD Transporter 1. Ind Crop Prod来调节油积累。2019; 137:126-36。
- 45。
Na g,mu x,grabowski p,schmutzj,lu c.增强种子中的microRna167a表达降低了α-亚麻酸含量并增加了种子尺寸Camelina Sativa.植物j . 2019; 98(2): 346 - 58。
- 46.
李华,吕强,马超,曲静,蔡峰,邓军,等。代谢产物分析和转录组分析为苦荞种子发育中的类黄酮生物合成提供了见解(Fagopyrum tataricum.).[J] .农业科学与技术。2019;67(40):11262-76。
- 47.
关键词:荞麦,灌浆期,转录组,芦丁积累《公共科学图书馆•综合》。2017;12 (12):e0189672。
- 48.
黄建军,邓军,石涛,陈强,梁超,等。水稻苦荞种子发育过程中营养物质积累相关基因的全球转录组分析与鉴定(Fagopyrum Tararicum).Sci众议员2017;7(1):11792。
- 49.
钱永强,程永华,程斌。玉米Dicer-like、Argonaute和RNA依赖RNA聚合酶基因家族的鉴定与鉴定。植物学报2011;30(7):1347-63。
- 50。
张海峰,夏锐,张海峰。植物ARGONAUTE蛋白的进化、功能和机理。植物学报2015;27:84-90。
- 51。
Ma J,赵P,刘S,杨Q,郭H。微稻草的发育阶段过渡控制及其在种子植物中的目标。int j mol sci。2020; 21(6):1971。
- 52。
Kapoor M,Arora R,Lama T,Nijhawan A,Khurana JP,Tyagi Ak等。基因组鉴定,组织和解性,嗜族和RNA依赖性RNA聚合酶基因家族的组织和系统发育分析及其在水稻中生殖发育和胁迫期间的表达分析。BMC基因组学。2008; 9:451。
- 53。
关键词:Dicer-like, Argonaute, RNA依赖RNA聚合酶,基因家族,表达谱谷子.植物学报。2015;33(1):43-55。
- 54。
Zhong J, He W, Peng Z, Zhang H, Li F, Yao J. A putative AGO protein, OsAGO17通过OsmiR397b正向调节水稻粒级和粒重。中国生物技术学报;2018;29(4):917 - 918。
- 55.
等。水稻特有的Argonaute 17控制生殖生长和产量相关的表型。acta botanica sinica(云南植物学报);2012;40(2):457 - 461。
- 56.
Rodrigues as,miguel cm。小非编码RNA在种子发育调控中的关键作用。植物细胞批准。2017; 36(5):653-67。
- 57.
谢敏,余斌。sirna介导的植物DNA甲基化。咕咕叫基因组学。2015;16(1):23-31。
- 58。
张l,李奇,ma b,gao q,du h,han y等。Tarrary Buckwheat基因组在芦丁生物合成和非生物胁迫耐受性方面提供了见解。莫尔植物。2017; 10(9):1224-37。
- 59。
张志强,张志强,张志强,等。两个草莓miR159家族成员在花托中表现出发育特异性的表达模式,并协同调控Fa-GAMYB。新植醇。2012;195(1):47-57。
- 60。
Sharma D,Tiwari M,Pandey A,Bhatia C,Sharma A,Trivedi PK。MicroRNA858是苯丙烷途径和植物发育的潜在调节剂。植物理性。2016; 171(2):944-59。
- 61。
李恩,徐河,李玉。植物种子规模控制的分子网络。Annu Rev植物Biol。2019; 70:435-63。
- 62。
基于mirna的植物次生代谢产物生物合成调控机制的研究。植物学报2017;8:374。
- 63。
Nesi N, Debeaujon I, Jond C, Pelletier G, Caboche M, Lepiniec L. TheTT8基因编码表达所需的基本螺旋环螺旋域蛋白DFR和禁止基因在拟南芥长角果。植物细胞。2000;12(10):1863 - 78。
- 64。
Nesi N, Jond C, Debeaujon I, Caboche M, Lepiniec L. The拟南芥TT2基因编码一个R2R3 MYB结构域蛋白,该蛋白在种子发育过程中对原花青素积累起关键决定作用。植物细胞。2001;13(9):2099 - 114。
- 65。
梅赫滕斯,克兰兹,贝德纳雷克,P拟南芥转录因子myb12.是苯丙烷化生物合成的一种类黄酮特异性调节剂。植物理性。2005; 138(2):1083-96。
- 66。
张志强,张志强,张志强,等。水稻花青素合成的蔗糖特异性诱导拟南芥要求myb75/PAP1基因。植物杂志。2005;139(4):1840 - 52。
- 67。
谭军,吴勇,郭军,李华,朱丽,陈锐,等。结合microRNA和转录组分析揭示了水稻对褐飞虱的抗性反应。BMC基因组学。2020;21(1):144。
- 68。
王胜,吴科,袁强,刘旭,刘震,林旭,等。控制晶粒尺寸、形状和质量OsSPL16在大米。Nat麝猫。2012;44(8):950 - 4。
- 69。
王胜,李胜,刘强,吴科,张军,王胜,等。的OsSPL16-GW7.调节模块决定籽粒形态,同时提高水稻产量和品质。Nat麝猫。2015;47(8):949 - 54。
- 70.
Dwivedi N, Maji S, Waseem M, Thakur P, Kumar V, Parida SK,等。中介亚基OsMED15a是水稻种子大小/重量调节基因的转录共调节因子。生物物理学报。2019;1862(10):194432。
- 71.
范超,王刚,王勇,张锐,王颖,冯胜,等。蔗糖合酶通过调节转基因水稻的细胞分裂和淀粉积累来提高稻壳大小和粒重。中华医学杂志。2019;20(20):4971。
- 72.
何震,曾军,任勇,陈东,李伟,高飞,等。OsGIF1积极调节水稻中茎,叶和谷物的尺寸。前植物SCI。2017; 8:1730。
- 73。
Jain M,Pathak BP,Harmon Ac,Tillman Bl,Gallo M.培养花生果实开发期间的钙依赖性蛋白激酶(CDPK)表达(arachis hypogaea.)在CA下2+-充足和不足的生长方案。植物生理学杂志。2011;168(18):2272-7。
- 74。
李华,吕强,邓军,黄军,蔡峰,梁超,等。转录组分析揭示普通荞麦种子发育关键基因(Fagopyrum esculentum).int j mol sci。2019; 20(17):4303。
- 75。
王志强,王志强,王志强,等。Pfam:部落,网络工具和服务。核酸Res. 2006;34(数据库版):D247-51。
- 76。
陈超,陈洪,张勇,何勇,等。TBtools:一个用于大生物数据交互分析的综合工具。摩尔。2020;13(8):1194 - 202。
- 77。
miRBase:从microRNA序列到功能。核酸Res. 2019;47(D1): D155-62。
- 78。
FriedländerMr,Mackowiak SD,Li N,Chen W,Rajewsky N.Mideepe2准确地识别七只动物的七分之一的新的MicroRNA基因。核酸RES。2012; 40(1):37-52。
- 79.
Allen E, Xie Z, Gustafson AM, Carrington JC。植物中反式作用siRNA生物发生中的microrna定向阶段。细胞。2005;121(2):207 - 21所示。
- 80.
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT,等。基因本体论:生物学统一的工具。中华医学杂志。2000;25(1):25 - 9。
- 81.
Okuda S,Yamada T,Hamajima M,Itoh M,Katayama T,Bork P,Goto S,Kanehisa M. Kegg地图集,用于全球分析代谢途径。核酸RES。2008; 36:W423-6。
确认
不适用。
资金
这项工作由中国国家重点研发计划(2019YFD1001300,2019YFD1001302),中国国家自然科学基金(31701494,31760430,31860408),贵州省的科技基础(Qiankehejichu [2019] 1235),国家自然科学基金中国 - 贵州省政府联合方案(U1812401),贵州省教育部青年科技人才增长项目(黔卓京泽[2018] 128),贵州师范大学培训计划(Qiankehepingtairencai [2017]5726),贵州师范大学医生研究初期(11904/0517051)。我们感谢所有经济支持的基础。资金组织为研究项目提供了财政支持,但没有参与研究,数据收集,数据分析或书面文章的设计。
作者信息
隶属关系
贡献
HYL和QFC设计了这项研究。HYL进行了实验并撰写了手稿。HYL、HLM、XQS、JD、LWZ进行数据分析。QYL, TXS和QFC对稿件进行了审核和修改。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准,指导方针和同意参与
植物材料(金桥2号)为我国苦荞麦品种,由我们实验室(贵州师范大学荞麦产业技术研究中心,贵州贵阳,中国)采集。所有作者都表示,这项研究符合中国的立法和《濒危野生动植物种贸易公约》。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
所有被识别的特征DCL,前,HYL1,SE,HST.,rdr在苦荞基因组中。表S2。苦荞麦种子发育过程中sRNA序列的长度分布。表S3。已鉴定的保守mirna信息。表S4。已识别的新mirna信息。表S5。苦荞mirna的靶基因预测。表S6。苦荞mirna靶基因注释。表S7。miRNAs靶向tf的信息。表S8。miRNA具有靶基因是已知种子或器官尺寸的正交。表S9。mirna的靶基因是类黄酮生物合成的已知结构或调控基因的同源基因。表S10。Dems的目标基因的Kegg途径。表S11。苦荞种子发育过程中miRNA-mRNA互作对的表达呈负相关。表S12。QRT-PCR分析序列引物。表向。RLM-5'RACE分析序列的引物。
附加文件2:图S1。
读取SRNA的长度分布。图S2。目的基因的GO分析。图S3。DEMS靶基因的KEGG分析。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
李,H.,孟,H.,Sun,X.等等。整合microRNA和转录组分析揭示苦荞种子发育相关的关键miRNA-mRNA相互作用对(Fagopyrum tataricum.).BMC植物杂志21,132(2021)。https://doi.org/10.1186/s12870-021-02914-021-02914
收到了:
接受:
发表:
关键词
- 苦荞麦
- 综合分析
- Mirnas.
- 目标基因
- 种子发展
- 种子大小
- 类黄酮
- DCL
- 前
- RDR