摘要
背景
玉米是世界上最重要的农作物之一。最关键的农艺性状,包括产量性状和工厂建筑特质,是定量的。基因/数量性状基因座(QTL)影响的关键特征的精细定位是玉米育种标记辅助选择(MAS)是必不可少的。然而,具有高密度和高多态性SNP标记缺乏,特别是kompetitive等位基因特异性,可被用于基因分型的自动PCR(KASP)SNP标记。迄今为止,大量的测序数据已经由新一代的测序技术,它提供了SNP位点为SNP标记的发展提供了良好池产生的。在这项研究中,我们进行了一个多步骤的筛选方法来确定基于所述RNA测序数据集368个玉米自交系kompetitive等位基因特异性PCR(KASP)SNP标记。
结果
高通量RNA-Seq数据集共鉴定出2,948,985个SNP,平均密度为1.4 SNP/kb。其中71311个KASP SNP标记(平均密度为34个KASP SNP/Mb)被定位于16161个基因上,均符合基因组独特区、双等位基因、多态信息含量(PIC)≥0.4和引物序列保守的要求。这些16161个基因被注释为52个基因本体论(GO)术语,包括大多数初级和次级代谢途径。随后,在368个RNA-Seq数据集中选择PIC值在0.14 ~ 0.5之间以及在硅分析中玉米自交系1212和B73之间存在多态性的50个KASP SNP标记,通过实验验证SNP的准确性和多态性。结果表明,玉米自交系1212与B73之间有46个snp位点出现多态性,占92.00%。利用这46个多态性SNP对其他20个玉米自交系进行基因型分析,20个玉米自交系中46个SNP均表现出多态性,每个SNP的PIC值为0.11 ~ 0.50,平均为0.35。结果表明,本研究开发的KASP SNP标记具有较高的准确性和多态性。
结论
这些高密度的KASP多态性SNP标记将为玉米QTL/基因的图位克隆和标记辅助选择提供有价值的资源。此外,用于开发玉米SNP标记的方法也可以应用于其他物种。
背景
玉米(玉蜀黍ssp。梅斯是世界上最重要的三种谷类作物之一,主要用作动物饲料和工业原料。一个多世纪以来,玉米一直被用作遗传研究的经典模型系统。面对快速增长的人口和恶化的环境条件,迫切需要通过育种迅速提高玉米产量。到目前为止,主要农艺性状的遗传克隆和标记辅助选择(MAS)已经被探索,以提高育种的效率。分子标记技术是生物学研究的革命性技术进步,已成为MAS的重要工具。在过去的30年里,各种类型的分子标记,包括AFLP、RFLP、RAPD、SSR和InDel标记被开发出来,并成功地在玉米中广泛应用,[1,2,3.,4,5].目前,遗传学家和分子育种家迫切需要具有高密度、高多态性、易于使用、性价比高、功能性强的分子标记。
SNPs是植物基因组中最丰富的遗传变异位点来源[6].单核苷酸多态性是目前公认的密度最高的分子标记,是遗传学研究的理想选择。高覆盖度和高分辨率标记的使用可以大大提高遗传研究和植物育种的效率。先进的SNP基因分型技术的应用,如下一代测序(NGS)和基于阵列的基因分型,不仅为高通量和中通量SNP基因分型提供了一种有效的方法,而且每基因型成本更低[7,8,9,10,11,12,13,14,15,16].因此,高密度SNP标记已广泛应用于育种和基因组研究,如全基因组关联研究(GWAS)、QTL定位和种质资源评价等。QTL/基因的精细定位和经典的MAS技术通常需要在几个位点上对大量样本(成百上千)进行基因分型。然而,高通量基因分型平台不适合这种类型的研究,因为每个样本成本较高。幸运的是,结合等位基因特异性oligo延伸和荧光共振能量转移(FRET)来产生信号,KASP检测已经成为一种准确、灵活和经济的SNP基因分型方法[7].此外,KASP技术可以在基础分子实验室进行。此外,由于反应量的减少和自动化平台的使用,基因型呼叫的吞吐量得到了提高,成本显著降低。
与全基因组测序(WGS)数据相比,RNA-Seq数据为潜在功能标记的开发提供了丰富的资源。以苏丹草S722和高粱Tx623B的转录组数据为例,共开发了5344个InDel标记[17],根据大黄鱼综合转录组数据,鉴定出1276个具有功能意义的多态SSRs和261000个具有功能意义的SNPs [18].与来自基因组随机区域的传统标记相比,基因标记具有更大的潜力与基因功能相关。基因标记在植物育种中的应用避免了标记与感兴趣性状基因编码的重组,提高了选择效率。
此外,除了缺乏高密度的KASP SNP标记外,KASP技术应用于基因图位克隆和MAS育种的另一个限制是PCR扩增的成功率,这会影响KASP SNP基因分型反应的表现。因此,迫切需要缩小大量测序数据与有效的KASP SNP标记之间的知识差距。在某些情况下,育种家和实验研究人员很难处理通过测序产生的大量数据,因为他们不熟悉用于从数据中提取有趣信息的生物信息学工具。此外,不同类型的分子标记和用于基因分型的方法具有不同的性质。因此,用于开发特异性分子标记的方法必须满足特定的标准。在对影响SNP基因分型效率的因素进行硅分析后,将提高SNP基因分型的成功率和转化率,从而得出准确的基因型。
近年来,KASP测定已广泛在玉米的研究遗传多样性,遗传纯度,定量轨迹映射应用,QTL /基因精细定位,标记辅助选择,和标记辅助育种[19,20.].例如,1057个KASP SNP标记用于基因型100个玉米自交系,揭示了大量的这些线路分为四组[之间的遗传变异的21].利用191个KASP SNP标记对16个玉米自交系的种子进行质量控制分析;191个KASP SNP标记的效率接近257,268个下一代测序(GBS)标记的效率,相关性为0.88,说明更小、更高质量的KASP SNP标记足以用于QC分析[22].利用玉米敏感株系(LM11)和耐高温株系(CML25)的RNA-seq数据,分别获得了129,804个和117,550个KASP单核苷酸多态性;其中,90 F基因型验证了100个KASP单核苷酸多态性2来自LM11 × CML25的分离群体[23].此外,KASP SNP可以作为功能性分子标记。KASP SNP (snpZM0015)位于crtRB1影响维生素原A (PVA)含量的基因在70个玉米自交系中有效区分有利等位基因和不利等位基因[24].两个snp位于GRMZM2G315401和GRMZM2G430362通过基因分型确定50株不育和50株部分挽救的植物与育性密切相关[25].虽然在多个玉米自交系中开发了许多KASP SNP标记,但全基因组高密度、高多态性的KASP SNP标记还很有限。
本研究基于368个玉米自交系籽粒的RNA-Seq数据,开发了KASP SNP标记,以满足在大量样本中基因分型较少的要求。共鉴定出71311个高密度、高多态性的用户友好型KASP SNP标记,选取50个KASP SNP标记,对其准确性和多态性进行实验验证。这些高多态性标记的应用将有助于提高玉米图谱克隆和标记辅助选择的效率。
结果
368 RNA-Seq数据集的snp特征
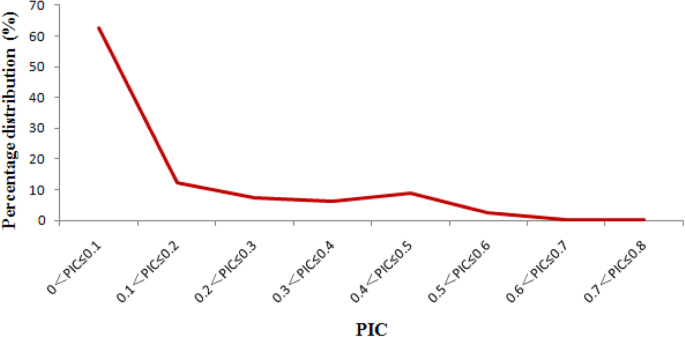
通过对368个玉米自交系发育籽粒的RNA-Seq数据集进行SNP挖掘,共鉴定出2948,985个SNP位点。单个染色体上的SNP数量为196,912 ~ 460,512个,玉米基因组中SNP的平均密度为1.4个/kb。其中,26820个基因的基因区共定位到2,545,815个SNPs(86.33%),平均snp /基因密度为94.92个。随后,根据368个玉米自交系的基因型,计算每个SNP位点的多态信息含量(PIC)值,PIC值范围为0.01 ~ 0.75,平均值为0.13(图1)。1).随着PIC值的增加,SNPs的数量急剧减少,PIC值≤0.1的SNPs占62.61%。值得注意的是,PIC值≥0.4的高多态性snp有339378个(11.51%)。此外,对每个SNP的等位基因进行了统计。多数单核苷酸多态性为2个等位基因(2,725321),占92.42%;190,694(6.47%)和32,970 (1.12%)snp分别具有3个和4个等位基因。因为双等位SNP标记在遗传研究中应用最为广泛;估计了双等位基因SNPs的转化/转化比值,转化/转化比值为0.91:1。最常见的转变类型为T-to-C(28.79%),其次为A-to-G(28.36%)、C-to-T(21.43%)和G-to-A (21.41%);最丰富的颠换是T-to-G(22.16%),其次是a到c(22.05%)、G-to-C(14.71%)、C-to-G(14.68%)、G-to-T(8.31%)、C-to-A(8.24%)、T-to-A(5.01%),和A-to-T(4.84%)。
高多态性KASP SNP标记的开发
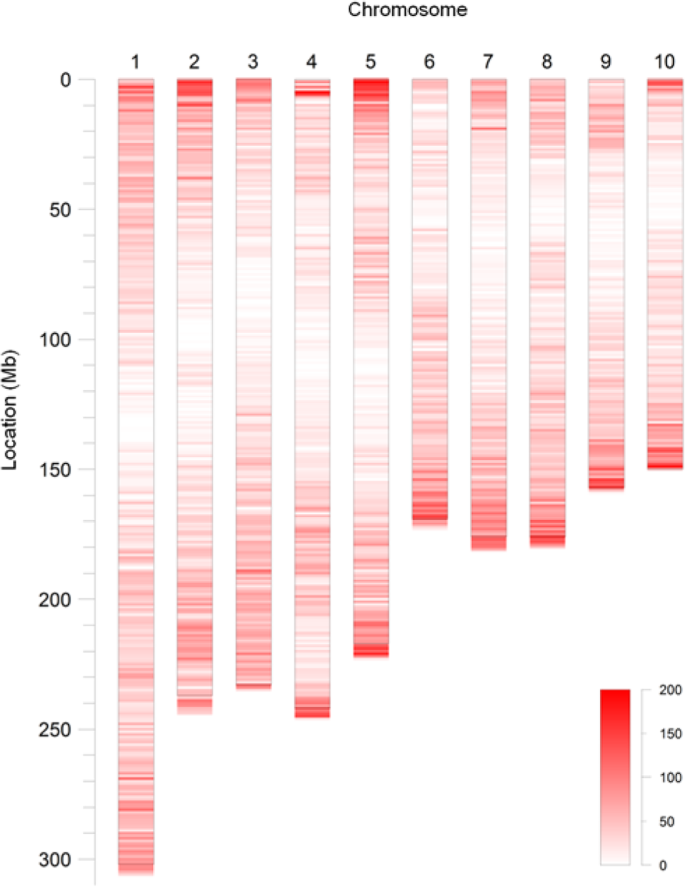
为了开发出高质量和多态Kasp标记,应用了相当严格的标准,包括具有PIC值≥0.4的SNP,基因型缺失率≤27.17%(≥100个基因型),存在双等性多态性,unique primer sequences unfiltered by mapping the primer sequence to the reference genome with 1 mismatch, and highly conserved primer sequences among maize individuals (the PIC value of the SNP located within the KASP primer sequence was < 0.2). As a result, 71,311 KASP SNP markers were identified in 2,948,985 SNPs, which accounted for 2.42%. These KASP SNP markers were distributed on 10 chromosomes, ranging from 4678 to 11,756 on each chromosome. The highest density of KASP SNP occurred on chromosome 5 (39.26 SNP/Mb) and the lowest on chromosome 4 (25.77 SNP/Mb), and the average density was 33.40 SNP/Mb. In addition, the density of KASP SNP located at the end of the chromosomes was high, whereas near centromere it was low (Fig.2).在71311个SNPs中,63,127个SNPs位于基因区,占89.36%。SNPs的详细信息,包括这些SNPs在玉米基因组上的物理位置(B73 RefGen v4.55和v5)、PIC值、基因ID、GO注释、SNP基因分型、等位基因数以及368个玉米自交系中具有基因分型的样本数量,均在附加文件中提供1.
此外,8275个标记一组核心是通过应用一组更严格的标准(附加文件中选择2).第一个标准是引物序列中snp位点的PIC值< 0.1;第二个标准是引物序列需要映射到唯一的基因组区域,允许≤2个不匹配。其中,每条染色体上的SNPs数量从561个到1374个不等。这些顶级KASP SNP标记将在未来的研究中更加友好。
基因相关withKASP SNP标记的GO注释
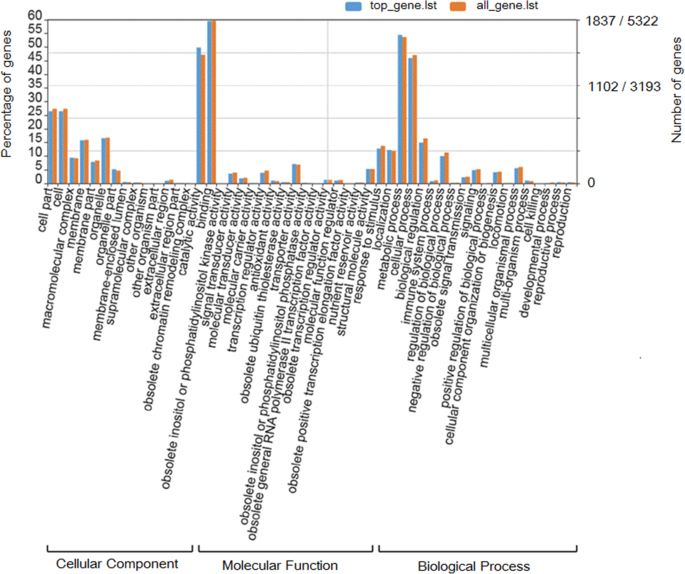
为探明这些KASP SNP标记的潜在生物学功能,对含有KASP SNP标记的基因进行GO富集分析。在包含至少一个KASP SNP标记的16161个基因中,8870个基因使用AgriGO在线单一富集分析工具(SEA)成功地分为生物过程、分子功能和细胞组分三大类(图1)。3.;额外的文件3.).总体而言,在第二层次识别出54个显著性氧化石墨烯术语,其中19个术语与“生物过程”有关,17个术语与“细胞成分”有关,18个术语与“分子功能”有关。其中,涉及生物过程的主要术语是“代谢过程”(GO:0008152)、“细胞过程”(GO:0009987)和“初级代谢过程”(GO:0044238)。排名前三的细胞成分术语是“cell”(GO:0005623)、“cell part”(GO:0044464)和“胞内”(GO:0005622)。在分子功能类别中,GO的主要术语为“催化活性”(GO:000382)、“核苷酸结合活性”(GO:0000166)和“水解酶活性”(GO:0016787)。这些结果表明,参与KASP SNP标记的基因可能影响大多数代谢途径;因此,KASP SNP标记可以广泛应用于不同性状和代谢途径的研究。
KASP SNP标记的准确性和多态性验证
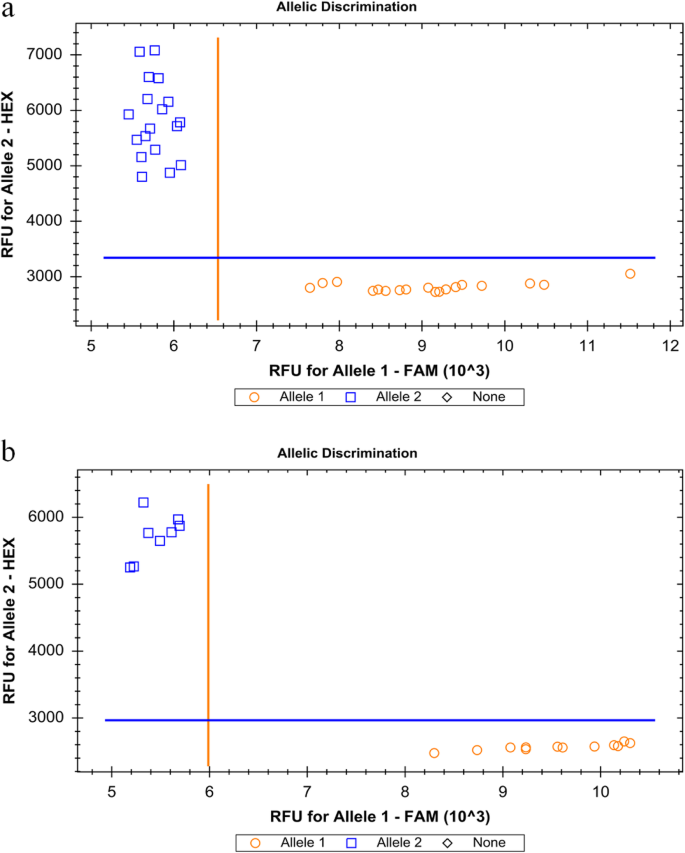
为验证KASP SNP标记的准确性和多态性,随机在每条染色体上选取50个KASP SNP标记,每条染色体上选取3 ~ 7个KASP SNP标记进行PCR扩增检测。在368个RNA-Seq数据集中,这50个KASP SNP标记的PIC值为0.14 ~ 0.50;玉米自交系1212和B73在硅质分析中均表现出多态性(附文件)4).通过PCR扩增,玉米自交系1212和B73之间的KASP SNP标记中有46个(92.00%)存在多态性,与硅分析结果基本一致。随后,选取46个多态性KASP SNP标记对20个玉米自交系进行基因分型。在20个玉米自交系中,46个KASP SNP标记均表现出多态性。对于每个KASP SNP标记,PIC值的范围为0.10 ~ 0.50,平均值为0.35(图3)。4;额外的文件4).
利用KASP平台进行SNP标记的基因型分析。一个SNP标记3个重复的基因型图(附加文件第41 - 47号)4玉米自交系1212和B73。b20个玉米自交系的SNP标记Chr1-4,122,559的基因型图
讨论
KASP基因分型技术的主要优点
与TaqMan、高分辨率熔解(HRM)和基质辅助激光解吸/电离飞行时间质谱(MALDI-TOF-MS)等其他SNP基因分型方法相比,KASP基因分型有几个优势[26,27].首先,KASP基因分型技术是SNP基因分型的具有成本效益的方法。对于KASP每个样品的成本小于最常用的低的通量SNP基因分型方法如上所述。SNP基因分型测定法的比较研究表明,每个样品的成本是0.11€为KASP相比0.29€为的TaqMan;此外,商业服务,SNP基因分型成本比的TaqMan [便宜五倍28,29].Yuan等人报道,大豆基因分型中等位基因特异性KASP试验的每个数据点成本约为TaqMan的1/50 [30.].此外,在自动化平台上使用KASP SNP基因分型的小型化反应,可进一步将成本降低10倍。此外,考虑到标记和样本的数量,KASP方法比现有的高通量SNP基因分型技术具有优势,目前的SNP基因分型技术在大量样本中对少数位点进行基因分型时,每个样本的成本更高[31,32,33].其次,KASP SNP基因分型是一种简单的无凝胶荧光PCR检测方法。一方面,不需要专门的设备,研究人员可以使用常规的qPCR仪器进行SNP基因分型。通过设计两对引物对等位基因特异性SNP位点进行不同的定向扩增,将SNP转化为长度多态性,直接利用DNA凝胶电泳检测和分离PCR产物。此外,可以选择几种简单的方法进行KASP测定;例如,研究人员可以直接向LGC基因组办公室或其他商业测试公司提供DNA样本和引物,或通过订购KASP主混合剂或购买自动化系统,在qRT-PCR仪器中进行KASP分析。
高多态性KASP SNP标记的鉴定
PIC值是判断分子标记是否有用的最重要指标;PIC值越高,两个任意个体之间发生多态性的概率就越大。PIC值≥0.5的标记通常被认为信息丰富[34,35].在分子标记的开发过程中,分子标记PIC值的准确性取决于群体大小及其遗传结构。在本研究中,我们使用PIC值来评估SNP标记的有效性,并进一步使用大规模RNA-Seq数据集来确保PIC值的准确性。本研究选择的368个玉米自交系作为KASP SNP标记,在全球温带、亚热带和热带玉米种质资源中具有相当代表性;该数据集适合于避免多态性偏差。与以往研究开发的SNP标记相比,本研究开发的KASP SNP标记具有更大的代表性和较高的多态性水平。例如,Panzea数据库中列出的SNPs主要来自一个相对狭窄的数据集,包括14个玉米品种和16个玉米野生相对大刍草自交系[36].在另一个例子,深入分析多态性水平的高密度maizeSNP50阵列基于相同的368个玉米自交系我们使用目前的研究发现,大约一半的snp图片值低于0.3,和877个snp non-polymorphic标记(12,37].另一方面,与其他物种相比,本研究基于RNA-Seq数据开发的SNPs的平均PIC值(0.13)低于芝麻(0.38)[38],萝卜(0.32)[39,葡萄藤(0.25)[40]、甜橙(0.2)[41].考虑多态性编码区的相对较低的水平,发达核不KASP SNP标记将是图位克隆和玉米分子标记辅助选择有用的。
优化KASP SNP标记的扩增效率
由于NGS技术的成本急剧下降,数以百万计的SNPs已经产生、发表或储存在公共数据库,如Panzea和MaizeGDB。到目前为止,对玉米SNP标记转化为KASP标记的研究还很少。毫无疑问,并不是所有的SNP都符合KASP SNP标记设计的要求[42].因此,选择基因型调用率高的SNP位点是KASP SNP标记检测的重要步骤。在影响基因分型成功率的因素中,在KASP基因分型试验中,引物序列对SNP基因分型能力的影响起着关键作用。在KASP试验引物设计中,引物序列的唯一性和保守性是两个最重要的参数。引物序列的独特性和保守程度越高,SNP基因分型成功率越高。这种效应在具有复杂基因组的物种中尤为明显。例如,由于85%的玉米基因组是由重复序列组成的,因此很难避免非特异性PCR扩增[43].因此,我们采用新的方法和标准来提高PCR扩增检测的效率和准确性。PCR特异性扩增是我们研究的主要因素。为了提高PCR特异性扩增,我们将KASP SNP标记引物序列与不匹配≤2个的参考基因组进行比对,以评估引物序列的位点特异性,只保留映射到唯一基因组区域的序列。KASP引物序列的保守性是我们在开发KASP SNP标记时考虑的第二个重要因素。保守的引物序列可提高不同玉米品系PCR扩增的成功率。本研究利用两个因子对保守引物序列进行筛选。其中一个因素是368个样本的基因分型数,阈值设为≥100。另一个因素是位于KASP引物序列内的SNP的PIC值,当PIC值为> 0.2时,KASP引物序列被删除。20个自交系中50个KASP SNP标记的PCR扩增成功率为98.5%,表明优化SNP发育参数可显著提高KASP SNP标记的扩增效率。
开发用户友好、高密度功能分子标记
与随机分布在染色体上的分子标记相比,本研究开发的基因KASP SNP标记在QTL/基因精细定位和MAS定位方面具有显著优势[44].作为一种基因SNP标记,来自编码区域的SNP变异可能会改变蛋白质的结构,从而更有可能在功能上与靶性状相关联。本研究开发的71311个KASP SNP标记被定位到16161个基因上,平均每个基因有4.4个SNP标记,大约是Mammadov等人2012年鉴定的基于基因的KSAP SNP标记的116倍[42],大约是Xu等人2017年开发的玉米55k SNP序列内含子区域19,425个SNPs的1.3倍[9].此外,值得注意的是,这些高性能的基因kasp SNP标记是从368型玉米自交系的面板开发的。其中,许多自交系,如B73,S37和18-599,是在大面积中栽培的母细胞母细胞系,其中一些线已成为玉米育种计划中的细菌的关键来源。总的来说,这些Kasp SNP标记不仅可以用于基于地图的基因克隆,而且可能是优化玉米产量的有力工具。
结论
面临几个位点pcr的高需求大量的样本在基础和应用研究,我们的研究提供了一个面板71311 KASP SNP标记的平均KASP SNP标记每29.94 kb被识别和严格的选择标准结合RNA-Seq 368玉米自交系和生物信息学方法的数据。在玉米自交系1212和B73中验证的KASP SNP标记的准确率达到92.00%,20个自交系的PIC值与硅分析一致。综上所述,这些KASP SNP标记将有助于玉米基因的图位克隆和MAS研究,标记开发和筛选参数的生物信息学管道可应用于其他物种。
方法
基因组数据和植物材料
玉米B73参考基因组(RefGen v4.55)和带有注释外显子-内含子结构的工作基因集均从玉米基因数据库(http://www.maizegdb.org/assemly/) [42,45].玉米籽粒368 RNA-Seq数据从NCBI网站(http://www.ncbi.nlm.nih.gov/sra/?term=SRP026161),并用于挖掘snp(附加文件5) [16].
选取糯玉米自交系1212和B73进行RNA-Seq分析。将1212和B73的未成熟果仁(20 DAP)顶部切开,立即在液氮中冷冻。使用Bioteke RNA提取试剂盒(Bioteke,北京,中国)根据制造商的方案从两个样品中分离总RNA,并根据制造商的建议构建RNA- seq库。两个库都使用HiSeq2500平台进行了测序,每个库产生了超过18 Gb的干净数据。1212和B73的RNA-Seq数据用于硅分析。
以20个玉米自交系(SCML203、SCML202、SAM3001、PH6WC、CA1108、2142、891、78599 - 211、WA-1、YA8201、CA211、YA3237、SCML103、21A、9953、08-641、LIAO6082、JH961、SN8-1-1、9614)为材料,评价KASP SNP标记的准确性和多态性。
SNP位点的SNP挖掘和多态性评价
使用NGSQC工具包v2.3.3过滤368 RNA-Seq数据集中< Q20的低质量读取后[46],通过BWA-ALN以默认参数对玉米B73参考基因组进行比对[47].然后,识别惟一映射的读,并用于进一步分析。随后,使用GATK(3.6-0版本)评估基因型呼叫,并满足以下过滤条件:qal≥20,覆盖深度≥5 [48].删除缺失数据≤5.43%(≤20个玉米样本)的基因型集。最后,利用多态信息含量(PIC)值评价各SNP位点的等位基因多态性,将其定义为PIC我= 1 -\({\总和}_ {j = 1} ^ n p {} _ {ij} ^ 2 \),其中pij频率是多少j第一个模式我th标记(3.].此外,还利用上述管道在1212和B73的RNA-Seq数据中鉴定了SNPs。
鉴定独特和保守的引物序列和底漆序列的设计
管道鉴定独特和保守的引物序列。首先,使用Python脚本提取位于相应SNP轨迹上游的20bp DNA序列。然后,使用多达1个错配的Bowtie软件对准20bp DNA序列与B73参考基因组对齐[49].最后,在进一步的分析中除去映射到多个基因组区域并含有具有PIC值> 0.2的SNP的序列。值得注意的是,所选择的SNP标记的两种等位基因特异性前底漆是在KASP技术(LGC基因组学,Beverly,MA)手册中设计的[50],常用反向引物采用primer 3软件设计[51].
去分析
选择含有KASP SNP标记的基因进行GO分析。GO注释和GO富集分析使用在线agriGO GO分析工具包和单一富集分析(SEA) (http://bioinfo.cau.edu.cn/agriGO/index.php) [52[玉米参考基因组作为背景参考。进行Hypejamini-Hochberg假发现率(FDR)的超距测试以调整P-value使用默认参数。
KASP SNP标记的准确性和多态性的实验验证
选择了一组50个双等位基因SNP以验证KASP SNP标记的多态性水平和精度。在CTAB(十六烷基三甲基溴化铵)DNA提取方案中,从3周龄幼苗中提取基因组DNA。The PCR amplification for the SNP genotyping analysis was performed in a BioRad CFX-96 RT-PCR thermal cycler in a reaction mixture of 10 μL containing 1× KASP reaction mix, 0.17 μM KASP assay mix (two forward allele-specific primers and a common reverse primer) and 30–40 ng of genomic DNA. The reactions were performed with the following cycling conditions: an initial denaturation at 95 °C for 5 min, 35 cycles of denaturation at 95 °C for 30 s, annealing at 55 °C for 90 s, extension at 72 °C for 90 s, and a final extension at 72 °C for 10 min. Fluorescent SNP genotyping was analyzed using the CFX96 manager software. The PIC value for each SNP marker was calculated using the formula described above.
声明的方法
作者声明,所有方法都符合相关机构、国家和国际准则和法律。
数据和材料的可用性
这项研究的所有数据都包含在手稿以及在补充文件。这项研究的所有shell脚本程序可以从应合理要求通讯作者。
缩写
- KASP:
-
等位基因特异性PCR Kompetitive
- SNP:
-
单核苷酸多态性
- InDel:
-
插入和删除
- 妊娠:
-
扩增片段长度多态性
- RFLP:
-
限制性片段长度多态性
- RAPD:
-
随机扩增多态性DNA
- 苏维埃社会主义共和国:
-
简单序列重复
- MAS:
-
分子标记辅助选择育种
- QTL:
-
数量性状位点
- 图片:
-
多态信息含量
- GWAS:
-
全基因组关联研究
- 走:
-
基因本体论
参考文献
- 1.
基于RFLP连锁图谱的数量性状孟德尔因子定位。遗传学。1989;121(1):185 - 99。https://doi.org/10.1093/genetics/121.1.185.
- 2.
曲静,刘建军。玉米全基因组简单重复序列分析及其多态性标记的开发。北京大学学报(自然科学版)。https://doi.org/10.1186/1756-0500-6-403.
- 3.
刘建军,曲建军,杨超,唐东,李建军,兰洪,等。基于下一代测序数据的玉米全基因组插入和缺失标记的开发。BMC基因组学。2015;16(1):601。https://doi.org/10.1186/s12864-015-1797-5.
- 4.
Vos P,Hogers R,Bleeker M,Reijans M,TVD L,Hornes M等。AFLP:DNA指纹识别的一种新技术。核酸RES。1995年; 23(21):4407-14。https://doi.org/10.1093/nar/23.21.4407.
- 5.
Lynch M, Milligan BG。利用RAPD标记分析群体遗传结构。摩尔生态。1994;3(2):91 - 9。https://doi.org/10.1111/j.1365-294X.1994.tb00109.x.
- 6.
李志强。单核苷酸多态性在作物遗传研究中的应用。植物生态学报。2002;5(2):94-100。https://doi.org/10.1016/s1369-5266(02)00240-6.
- 7.
彭inger S, Dander A, Fischer M, Snajder R, Sperk M, Efremova M,等。下一代基因组测序数据变异分析工具的调查。短暂的Bioinform。2014;15(2):256 - 78。https://doi.org/10.1093/bib/bbs086.
- 8.
严建军,杨晓霞,沙涛,Sánchez-Villeda H .,李军,Warburton M .,等。玉米GoldenGate高通量SNP基因分型摩尔繁殖。2010;25(3):441 - 51。https://doi.org/10.1007/s11032-009-9343-2.
- 9.
徐C,任y,jian y,guo z,zhang y,谢c等。玉米55K SNP阵列的发展,具有改进的分子育种基因组覆盖。分子育种。2017; 37(3):20。https://doi.org/10.1007/s11032-017-0622-z.
- 10.
田海龙,王福刚,赵建荣,易慧敏,王磊,王锐,等。用于中国玉米品种DNA指纹鉴定的高通量兼容SNP芯片maizeSNP3072的研制摩尔繁殖。2015;35(6):136。https://doi.org/10.1007/s11032-015-0335-0.
- 11.
等。玉米基因组分析的强大工具:高密度600k SNP基因分型阵列的开发和评价。BMC基因组学,2014;15(1):823。https://doi.org/10.1186/1471-2164-15-823.
- 12.
张志强,张志强,张志强,等。大玉米(玉米L.)SNP基因分型阵列:发展和种质基因分型和遗传作图以与B73参照基因组进行比较。Plos一个。2011; 6(12):e28334。
- 13.
赖建军,李锐,徐昕,金伟,徐敏,赵辉,等。玉米优良自交系遗传变异的全基因组模式。42 Nat麝猫。2010;(11):1027 - 30。https://doi.org/10.1038/ng.684.
- 14.
娇y,赵h,任l,歌,zeng b,guo j等。玉米现代育种过程中的基因组遗传变化。NAT Genet。2012; 44(7):812-5。https://doi.org/10.1038/ng.2312.
- 15.
王志强,王志强,王志强,等。玉米HapMap2从一个不断变化的基因组中识别出现有的变异。Nat麝猫。2012;44(7):803 - 7。https://doi.org/10.1038/ng.2313.
- 16.
李华,彭震,杨新宇,王伟,付军,王军,等。全基因组关联研究剖析了玉米籽粒油脂生物合成的遗传结构。Nat麝猫。2013;45(1):43-50。https://doi.org/10.1038/ng.2484.
- 17.
李军,王玲,詹强,刘艳,杨欣。高粱转录组特征及功能标记的开发。《公共科学图书馆•综合》。2016;11 (5):e0154947。https://doi.org/10.1371/journal.pone.0154947.
- 18.
肖胜,韩震,王鹏,韩飞,刘勇,李军,等。大黄鱼综合转录组功能标记检测及下一代测序分析《公共科学图书馆•综合》。2015;10 (4):e0124432。https://doi.org/10.1371/journal.pone.0124432.
- 19.
Semagn K,巴布R,赫恩S,使用Kompetitive等位基因特异性PCR(KASP)奥尔森M.单核苷酸多态性基因分型:该技术的概述及其在作物改良应用。分子育种。2014; 33(1):1-14。https://doi.org/10.1007/s11032-013-9917-x.
- 20。
Волкова Н, Соколов В。KASP TM基因分型技术及其在玉米遗传育种中的应用。植物种质资源保护。2017;13(2):131-40。https://doi.org/10.21498/2518-1017.13.2.2017.105394.
- 21.
王志强,王志强,王志强。INERA玉米自交系单核苷酸多态性(SNP)标记的遗传多样性及其与CIMMYT、IITA和温带系的关系BMC麝猫。2014;15(1):1 - 14。
- 22.
et al., et al., et al.等。比较竞争等位基因特异性PCR (KASP)和基因分型测序(GBS)在玉米品质控制分析中的应用。BMC基因组学。2015;16(1):1 - 12。
- 23.
Jagtap AB, Vikal Y, Johal GS。玉米耐热性遗传分析中性价比高的KASP标记的全基因组开发和验证。中国生物医学工程学报。2020;21(19):7386。https://doi.org/10.3390/ijms21197386.
- 24.
Badu-Apraku B,Elorhor Ifie B,Danquah A,Blay et,Dadzie Ma。肽的表型表征和验证早熟肽中的官能基因A - 质量蛋白玉米(玉米)自交系。植物品种。2020;139(3):575 - 88。https://doi.org/10.1111/pbr.12798.
- 25.
蔌啊,宋W,邢Ĵ,赵Y,张蓉,李C,等人。基因的鉴定潜在地与S型细胞质雄性不育的玉米经由分群RNA测序生育不稳定性相关。Plos一个。2016; 11(9):e0163489。https://doi.org/10.1371/journal.pone.0163489.
- 26.
陈晓东,陈晓东。单核苷酸多态性基因分型的研究进展。药物基因组学j . 2003; 3(2): 77 - 96。https://doi.org/10.1038/sj.tpj.6500167.
- 27.
李志刚,李志刚。SNP基因分型技术在生物医学分类中的应用。生物工程学报。2007;9(1):289-320。https://doi.org/10.1146/annurev.bioeng.9.060906.152037.
- 28.
基于pcr的植物SNP基因分型方法的比较。工厂方法。2018;14(1):28。https://doi.org/10.1186/s13007-018-0295-6.
- 29.
Landoulsi Z,Benromdhan S,Djebara MB,达马克男,Dallali H,凯菲R,等人。使用KASP技术屏幕LRRK2 G2019S突变的大突尼斯队列。BMC医学遗传学杂志。2017; 18(1):70。https://doi.org/10.1186/s12881-017-0432-5.
- 30.
关键词:大豆,SNP,基因分型,高通量,高性价比植物基因工程学报。2014;2(1):90-4。https://doi.org/10.5147/pggb.v2i1.155.
- 31。
Liu Q ., Liu Q ., Liu Q ., Liu Q ., Liu Q ., Liu Q ., Liu Q ., Liu Q ., Liu Q ., Liu Q ., Liu Q . .基因组研究》1997;7(4):389 - 98。https://doi.org/10.1101/gr.7.4.389.
- 32。
Ye S, Dhillon S, Ke X, Collins AR, Day IN。一种有效的单核苷酸多态性基因分型程序。核酸学报2001;29(17):e88。https://doi.org/10.1093/nar/29.17.e88.
- 33.
刘军,黄胜,孙敏,刘胜,刘勇,王伟,等。一种改良的等位基因特异性PCR引物设计方法及其应用。工厂方法。2012;8(1):34。https://doi.org/10.1186/1746-4811-8-34.
- 34.
关键词:多态DNA标记;洛斯阿拉莫斯科学1994;20:100-2。
- 35.
Botstein d,白色rl,skolnick m,davis rw。利用限制性片段长度多态性构建人类遗传联系地图。我是j嗡嗡声的遗传。1980; 32(3):314-31。
- 36.
赵伟,张志强,张志强,等。玉米基因组的分子和功能多样性数据库和资源。核酸Res. 2006;34(sup1): D752-7。
- 37.
付建军,程勇,令湖杰,杨晓明,康磊,张震,等。RNA测序揭示了玉米籽粒中复杂的调控网络。Nat Commun。2013;4(1):2832。https://doi.org/10.1038/ncomms3832.
- 38.
魏林,苗华,李超,段勇,牛军,张涛,等。acta physica sinica, 2014;34(4): 2205-17。https://doi.org/10.1007/s11032-014-0174-4.
- 39.
王y,刘开,徐l,王y,chen y,luo x等。基于转录组序列的SNP标记及其在萝卜种质鉴定中的应用(萝卜l .)。摩尔品种。2017;37(3):26。
- 40.
Lijavetzky D, Cabezas JA, Ibáñez A, Rodríguez V, Martínez-Zapater JM。葡萄高通量SNP发现和基因分型(葡萄通过结合重测序方法和SNPlex技术。BMC基因组学。2007;8(1):424。
- 41.
姜东,叶启龙,王福生,李超。柑橘EST-SNP基因的挖掘及其在品种鉴定中的应用。中国农业科学。2010;9(2):179-90。https://doi.org/10.1016/s1671 - 2927 (09) 60082 - 1.
- 42.
马马多夫,陈伟,明格斯,汤普生。玉米多基因SNP分析的研究进展(玉米l .)。mol繁殖。2012; 29(3):779-90。https://doi.org/10.1007/s11032-011-9589-3.
- 43。
王志强,王志强,王志强,等。B73玉米基因组:复杂性、多样性和动态。科学。2009;326(5956):1112 - 5。https://doi.org/10.1126/science.1178534.
- 44。
陈志强,陈志强,陈志强,等。植物基因标记的研究进展。应用生态学报。在:基因组学辅助作物改良:施普林格;2007.13页。
- 45.
焦勇,王博,梁涛,等。利用单分子技术改进玉米参考基因组。大自然。2017;546(7659):524 - 7。https://doi.org/10.1038/nature22971.
- 46.
Jain M. NGS QC工具包:下一代测序数据质量控制的平台。《公共科学图书馆•综合》。2012;7 (2):e30619。https://doi.org/10.1371/journal.pone.0030619.
- 47.
基于burrowwheeler变换的快速、准确的短读对齐。生物信息学,2009,25(14):1754 - 60。
- 48.
等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组研究》2010;20(9):1297 - 303。https://doi.org/10.1101/gr.107524.110.
- 49.
领结:一种超快内存高效短读对准器。基因组医学杂志。2009;10 (3):R25。https://doi.org/10.1186/gb-2009-10-3-R25..
- 50.
Graves H, Rayburn AL, Gonzalez-Hernandez JL, Nah G, Kim DS, Lee DK。利用KASP法验证草原网茅DNA多态性(摘要pectinata链接)《美国前线植物科学》2016;6:1271。
- 51.
Rozen S, Skaletsky H. Primer3在万维网上为一般用户和生物学程序员。在:生物信息学方法和协议:施普林格;2000.p . 365 - 86。
- 52.
杜志华,周旭,凌云,张志华,苏志华。一种面向农业社区的GO分析工具。核酸Res. 2010;38(suppl_2): W64-70。
确认
感谢华中农业大学植物科技学院刘磊博士在SNP基因分型方面提供的技术支持。感谢成都银艺科技有限公司杨泽光大师为NGS数据分析提供的生物信息学支持。
植物或种子标本集合的许可和/或许可证
作者声明,他们遵守国际自然保护联盟关于濒危物种研究的政策声明和濒危野生动植物物种贸易公约。
资金
国家重点基础研究发展计划(973)项目(no . 2014CB138203);草地农业生态系统国家重点实验室(no . SKLGAE201509);国家自然科学基金项目(no . 31101161)。
作者信息
隶属关系
贡献
JL构思并设计了实验。ZJC、DGT、JHZ、HL和LJL进行实验。JL开展了生物信息学工作。JXN, PL, LW提供输入,材料。ZJC和JL对稿件进行了撰写和修改。所有作者阅读并批准了最终的手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
陈智,唐东,倪娇。et al。从RNA-Seq数据中开发基因KASP SNP标记用于玉米的图位克隆和标记辅助选择。BMC植物杂志21,157(2021)。https://doi.org/10.1186/s12870-021-02932-8
已收到:
接受:
发表:
关键字
- 玉米
- KASP
- SNPS.
- 高多态性
- 分子标记
- RNA-Seq