摘要
背景
科学文学承载了丰富的研究至关重要,但只有一小部分作为数据库中的结构化信息存在,因此可以使用传统数据分析工具进行分析。自然语言处理(NLP)通常和成功地通过从自由文本的大型公司中蒸馏相关信息并以旨在提供进一步计算分析的方式构建它来支持人类。对于此导频,我们开发了一种在生物文献中使用NLP的管道来生产知识网络。我们专注于马铃薯的肉体,一个熟练的特质,具有已知的协会,我们调查了这些知识网络是否可以帮助我们在潜在的生物过程中制定新的假设。
结果
我们基于34篇马铃薯全文文章的人工标注语料库训练了一个NLP模型,以识别文本中相关的生物实体和它们之间的关系(基因、蛋白质、代谢物和性状)。该模型对训练集中的生物实体数量的检测精度为97.65%,召回率为88.91%。我们在4023年进行了时间序列分析PubMed抽象植物基因的文章关注4主要茄属的作物(番茄、土豆、茄子和辣椒),确定网络包含先前已知和未知同时导致随后发现肉颜色相关的生物现象。一项基于时间的对这些网络的新分析表明,我们的性状与一个候选基因(玉米黄质环氧化酶)之间的联系,早在文献中明确陈述这种联系的两年前就已经存在了。
结论
我们的基于时间的分析表明,网络辅助假设生成在科学研究中为知识发现、数据集成和假设生成提供了希望。
背景
科学出版物积累了任何研究领域的知识和发展。在研究人员的工作和职业生涯中,最重要的任务之一是跟上不断增加的科学文献的步伐,将新的产出放到上下文中,并调查它们在各自领域中的含义。然而,随着科学出版物的数量呈指数级增长,有必要使用人工智能使机器能够阅读、提取和分析文本来源中的信息。
土豆(茄属植物tuberosum L。)是最重要的粮食作物人体营养的一个。除了其烹饪的通用性,马铃薯是一种高性价比的产品,并在满足世界不断增长的粮食需求中起主要作用。其块茎淀粉,蛋白质和维生素[一个很好的来源1].不同的马铃薯基因型会产生不同性质的块茎,如形状、大小、颜色、淀粉含量和营养价值。
马铃薯块茎肉色是马铃薯研究最广泛的性状之一。土豆块茎有很多种颜色,从橙色到白色和紫色。类胡萝卜素被认为是块茎果肉颜色的主要决定因素[2].类胡萝卜素在光合作用中发挥着重要作用,而在非光合作用组织中,它们发挥着广泛的功能,作为色素、抗氧化剂和信号分子的前体,包括挥发物[3.].先前的研究表明玉米黄质及其前体-胡萝卜素是块茎果肉颜色的主要决定因素[4.那5.].近年来,人们发现β -胡萝卜素羟化酶(BCH/CHY2)和玉米黄质环氧化酶(ZEP)等候选基因与块茎肉色有关。BCH/CHY2是与产生-胡萝卜素相关的基因,而ZEP被认为是负责玉米黄质积累的基因[6.].尽管在四倍体马铃薯品种中没有观察到高水平的β -胡萝卜素在转基因管中积累[5.,在马铃薯品种中观察到的橙色果肉等位基因频率较低[4.].这表明育种选择了浅色的等位基因。
在科学文献或生物数据库中发现了与遗传和分子实体的块茎肉颜色协会的科学证据。例如,Acharjee等人。以前公布的实验发现与2011年和2016年有关块茎肉颜色的生物实体网络[6.那7.].在这项研究中,我们从科学出版物中自动提取影响块茎果肉颜色变化的分子实体(基因/蛋白质/代谢物)的知识。
与结构化信息(如数据库)相比,文本信息庞大、嘈杂且冗余。人工智能有助于自动化文本信息的处理和新知识的发现。自然语言处理(NLP)是人工智能的一个领域,专注于使机器能够理解和分析文本形式的(非结构化)数据[8.].尽管有各种各样的植物研究数据存储库,但大量的信息目前仍隐藏在科学文献中。因此,基于自然语言处理的信息提取越来越受到重视。NLP可以使科学文本在计算上可访问,支持信息提取、知识网络(KN)构建和假设生成。
在过去的几年里,许多基于NLP的研究已经在分子生物学的文献上进行了[9.那10].它们主要集中在基于规则的命名实体识别(NER),即识别和注释生物实体,如基因或蛋白质[11那12),代谢产物(13那14],特征[15),主要16],疾病[17和毒品[18在文学。一些NLP研究关注于利用NER系统提取这些生物实体之间的关联(关系和事件)[12那19那20.].自动接近挖掘知识就需要进一步推进精确育种领域的实体,它的表型的关联[21].基于规则的NLP从生物上下文中更广泛地用于挖掘知识,而不是基于机器学习的NLP [22那23].然而,在基于规则的自然语言处理中,规则的构建和形式化是一项复杂的任务。通常,基于规则的NLP用户倾向于将规则过度拟合到训练集中,这影响了测试集中的性能。在基于规则的NLP中,字典和本体被用作构建块。另一方面,在基于监督机器学习的NLP中,领域专家手工注释文档的训练集。这些由字典和本体支持的手工注释文档被算法用于生成上下文特定的规则。最后,这些规则用于对未注释的测试集执行NLP。
在本研究中,我们调查了科学文献中的潜在知识是否可以利用NLP,以及是否可以及时强调基因-性状关联的新线索以生成假设。然而,我们的贡献不是在NLP领域,而是在揭示其潜力。我们选择重点研究马铃薯块茎的肉色,这是一个具有已知关联的重要农艺性状。这使我们能够将从文献中提取的关系与已确定的事实进行比较,作为管道性能的度量。在关注这个问题的时间维度之前,我们需要验证更多的次要假设,即1)NLP模型是否能够从文献中的自由文本中提取预期关系;2)单独的摘要是否可以作为其相应文章的高确定性、信息密集的代理。
我们的产品线从NLP模型开始,该模型是基于领域相关文献定制的,用于寻找生物实体(基因、蛋白质、代谢物和性状)及其之间的一般关系。我们选择使用IBM(国际商业机器公司)的沃森软件套装,因为它以前曾成功地从大量的在线文本中挖掘知识。24那25].Watson Knowledge Studio是一个专有的基于云的应用程序,用于根据特定文献领域的上下文和语言细微差别训练NLP模型。除了注释给定文本中感兴趣的实体(命名实体识别),Watson还执行关系提取;也就是说,标记感兴趣的检测实体之间的连接。沃森提取的关系用于构建kn。经过规范化的步骤后,我们能够整合这些内容,并从一组文本中提炼出可视化的知识。
我们选择了34篇文章组成了一个初级语料库,主要是关于土豆肉色的,我们用它来训练我们的NLP模型。随后,我们将其部署在这34个摘要中的一个子集(仅为摘要)和一个包含4023个PubMed摘要的更广泛的语料库上,这些摘要发表于2000年至2016年。对于前者,我们比较了网络的节点和边缘来检验我们的次级假设。对于后者,我们也进行了基于时间的分析,跟踪我们的兴趣特征与其他相关实体的亲密程度,标记重大发展发生的时间点,以评估这种方法是否确实有助于研究。这次时间分析和由此得出的结果是我们的主要贡献。
概念的证明了这一点(尽管大小限制)是文献挖掘如何帮助植物科学家获得更清晰的“大画面”有关特定区域在其专业领域的例子。在文学的扩大体难以捉摸的研究结果可能被曝光,自动组织成KNS,最终帮助加快研究中很少人为干预的过程。
结果
首先,为了确认我们的领域特定NLP模型按照预期执行,并从科学文献中提取以茎肉颜色为重点的知识网络(KNs),我们将其部署在两个不同的语料库上,即包含全文文章的训练集和仅包含PubMed摘要的测试集。接下来是对测试集的时间分析,以调查这些kn中的知识是否真的可以按照我们预想的方式来使用,以产生新的假设。
案例1:培训语料库分析(全文文章)
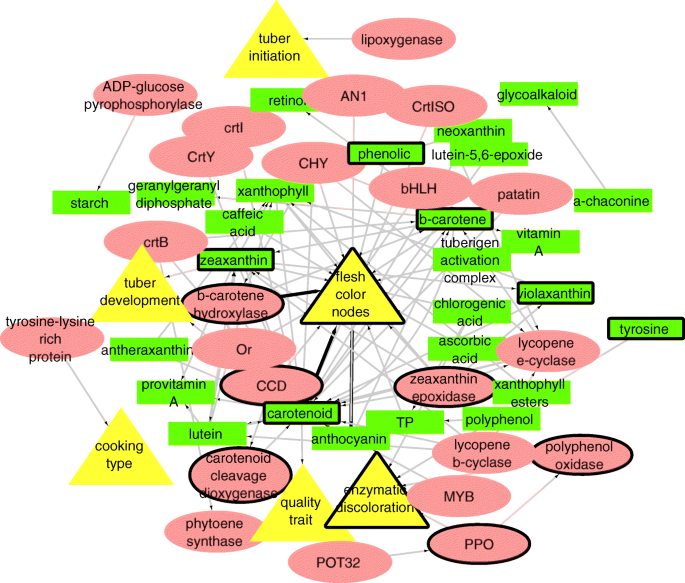
我们在培训套装34件文章上建立了一个kn,共有293个节点和551个独特的边缘。在这293个节点中,总共有159个基因/蛋白质,112代谢物和22个特征(图。1).类胡萝卜素(一类代谢物的实体)是这个网络的主要中心,有76个一级邻居。为了评估该KN的节点和连接,我们基于目前已知的块茎果肉颜色的实验知识,对其整体结构进行分析。我们的KN包含了科学上可靠的节点之间的联系和兴趣的特征,块茎肉的颜色。该网络中的大多数基因/蛋白质和代谢产物是类胡萝卜素生物合成途径的一部分,包括-胡萝卜素生物合成、叶黄素循环、脱落酸生物合成、叶黄素生物合成等。
表示34篇完整文章的训练集中的知识三元组的KN。黄色节点代表性状实体,红色节点代表基因/蛋白质实体,绿色节点代表代谢产物实体。此网的质心为块茎肉色。带有粗体轮廓的节点表明这些实体与块茎果肉颜色(兴趣特征)有实验证明的关联。这些具有块茎果肉颜色的实体的实验证据已在文章中报道[6.那7.].边缘的颜色反映了一个关系的文档频率(权重)。灰色边只出现在一个文档中,而黑色边出现在多个文档中。这些节点围绕感兴趣的特征以圆圈组织。最内层圆(圆1)和第二内层圆(圆2)内的节点为肉色节点的一级邻居;圆3,4和5中的节点是它的二阶邻居;圆6中的节点为三阶邻居;圆圈7中的节点(最外面的一个)是肉色节点的高阶邻居,或者根本没有连接到它
正在研究的特征,块茎肉色,有38个一级邻居,包括11个基因/蛋白质和27个代谢物(Cytoscape网络可在[26])。表中也列出了这些基因/蛋白质和代谢物1.此前进行的研究发现,ZEP和BCH/CHY与白色、黄色和橙色的肉色有关。AN1是一个负责产生花青素的基因,它与紫色的果肉颜色有关。在我们的网络中,所有这些基因都是块茎果肉颜色的直接邻居。
我们的NLP模型,模型训练集中的实体的97.65%的精度,88.91的%召回和93.07%的F1分数。补充文件1呈现一个混淆矩阵,显示每个文档的实体总数、真阳性数(TP)、假阴性数(FN)和假阳性数(FP)。计算精度和召回率分别为TP / (TP + FP)和TP / (TP + FN)。
此外,为了比较从文章摘要和全文版本中提取的信息在数量和质量上的差异,我们的NLP模型单独应用于训练语料库的摘要。
这突出了科学文章的这两种表现形式之间的数量差异。我们假设,摘要将具体和简明地呈现一份出版物的核心产出,而“介绍”部分将主要概述已建立的理论和相关的生物学联系,但没有贡献新的知识。最后,“结果”和“讨论”部分将更详细地结合本文的重要贡献,并为未来的实验提供进一步的建议。我们发现了支持这一假设的证据,因为纯抽象网络仍然包括实验证明对块茎果肉颜色最重要的实体。在集合A和B中,表1列出了全文表示的knn中块茎果肉颜色节点的直接邻居(图。1仅限)和摘要(图。2).
表示34篇文章的训练集中的知识三元组的KN,仅包含摘要。黄色节点代表性状实体,红色节点代表基因/蛋白质实体,绿色节点代表代谢产物实体。此网的质心为块茎肉色。带有粗体轮廓的节点表明这些实体与块茎果肉颜色(兴趣特征)有实验证明的关联。这些具有块茎果肉颜色的实体的实验证据已在文章中报道[6.那7.].边缘的颜色反映了一个关系的文档频率(权重)。灰色边只出现在一个文档中,而黑色边出现在多个文档中。这些节点围绕感兴趣的特征以圆圈组织。在最里面的圆(圆1)中的节点是肤色节点的第一顺序的邻居;在圆2中的节点是它的第二顺序的邻居;在圈3的节点(最外面的一个)是肤色节点的第三(或更高阶)的邻居,或者根本不连接到它
这两组之间的差异(表1;也显示了集合A -集合B)。这20个实体在全文KN中作为血肉颜色的直接邻居出现,但在抽象KN中不是这样——仅在抽象KN中。在这20个实体中,6个(AN1,叶黄素,叶黄素-5,6-环氧化物,多酚,八烯合酶,紫黄质)仍然存在于摘要的KN中(图。2),尽管他们不是直接的邻居,但块茎肉色的,而第二次的邻居和类胡萝卜素,BCH,或ZEP的一阶邻居。此外,隐性ZEP也被表示在抽象仅KN。由于ZEP的隐性等位变体是类似于占主导地位,这些节点不被表示为独立的实体。这同样适用于基因/蛋白的特性,如化学异构体和特质的措施,这是我们与主要的实体组合在一起,以减少我们的KNS碎片其他方面。剩余的12个实体(nonepoxide,甲基花青素,花色素,矮牵牛,天竺葵色素,花青素,PF,二甲花翠素,环氧化物,糖苷)在抽象仅KN中均未给出。这些实体与导致肉色变化关键代谢物有关。然而,他们不直接影响性状。因此,我们的研究结果表明,该全文网络中最重要的节点依然存在降低的抽象只有网络。
案例2:测试语料库分析(PubMed摘要)
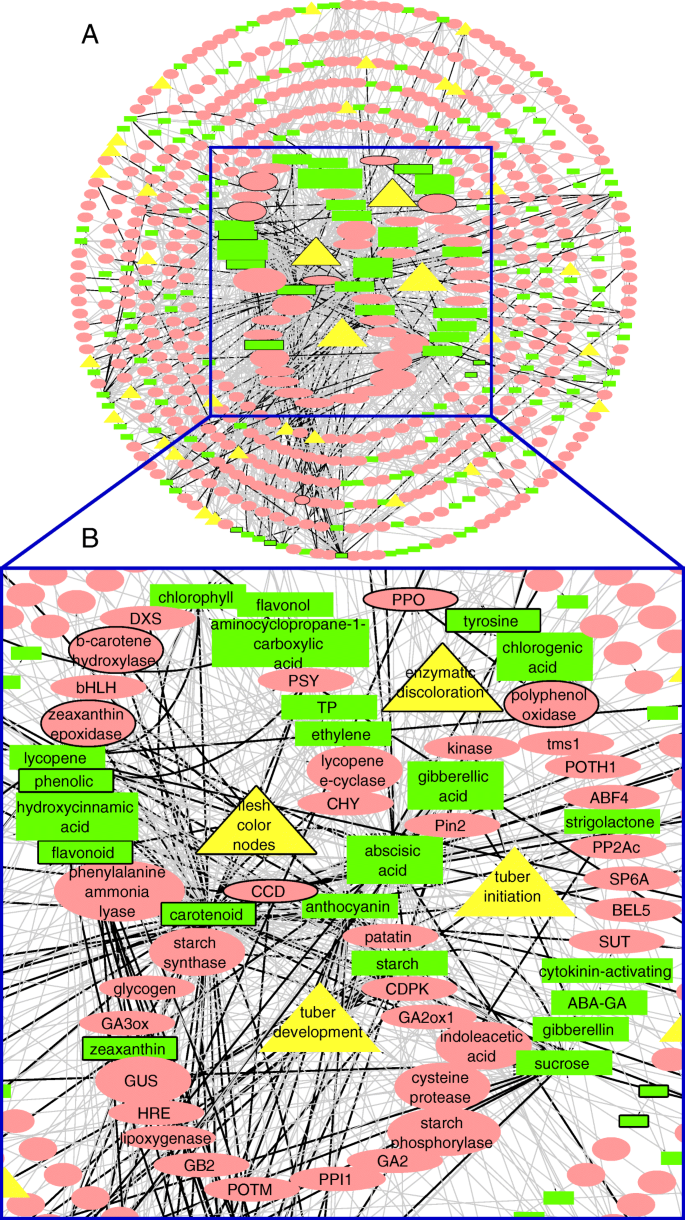
为了评估我们的NLP模型在一个未知语料库上的执行情况,我们将它部署在一个由来自PubMed文章的4023个摘要组成的测试语料库上。Watson检索到一个共有681个节点和976条独特边的KN(图)。3.a),超过一个(293 resp.551),这意味着我们的模型能够在该语料库中识别新节点和边缘。胡萝卜素再次是这个网络的主要质心,107个一阶邻居。我们正在研究的特质,块茎肉颜色,具有21个一阶邻居,包含9个基因/蛋白质和12个代谢物(参见[中的Cytoscape网络)26])。
代表在PubMed的4023篇文章的测试集中发现的知识三元组的KN。黄色节点表示性状实体,红色节点表示基因实体,绿色节点表示代谢实体。一种完整的缩放知识网络B.放大的知识网络的快照,集中在块茎肉的颜色和其他性状及其各自的生物学联系。边缘的颜色反映了一个关系的文档频率(权重)。灰色边只出现在一个文档中,而黑色边出现在多个文档中。这些节点围绕感兴趣的特征以圆圈形式组织起来。这些特征中的每一个都有一个围绕着它的一阶邻居(圆圈1)。其余的节点是根据兴趣的主要特征(肉色节点)组织的。圆圈2、3、4(从中心数起)中的节点为果肉颜色节点的二阶邻居;圆5和圆6中的节点是它的三阶邻居;圆圈7,8中的节点(最外面的)是肉色节点的第4(或更高)阶邻居,或者根本不连接它
虽然我们的模型是针对土豆块茎的肉色(介于白色和橙色之间)设计的,但我们也检测到了其他性状及其各自的生物学关联。例如,来自测试集的KN还检测到影响其他性状的基因/蛋白质和代谢物,如酶促变色、块茎起始、块茎发育、块茎成熟、蒸煮类型、匍匐茎膨大、花发育等(图。3.b).这说明了信息内容超出了特定的用例。此外,我们的NLP模型可以在比用例更广泛的环境中提取与块茎肉色相关的信息,而不需要进一步的具体训练。
通过时间分析确定潜在的候选人
为了随着时间的推移评估知识的积累,测试集的摘要在时间顺序排列的子集中组织(即,到其出版日期)。从2000年开始,每年递增(即2000年的所有出版物,所有出版物,高达2001年的出版物,......,所有出版物,最高可达2016年),成套组成。这些子集中的每一个用于构造单独的KN。给定年份的网络始终是焦糖的子集,从而实现了前几年的超级空间。
为了研究实体连接的发展与我们的兴趣特征(块茎肉的颜色),我们逆向工作。最近的收集是最完整的,因此选择了广泛涉及块茎肉色的节点(颜色、肉、肉色、肉性状、橘黄色肉色、块茎肉色、块茎肉色、块茎肉色、白色肉色、黄橙色肉色),从此统称为肉色节点。我们将注意力集中在那些最终直接连接到肉色结点的节点上。然后,我们跟踪这些选定的节点到每个单独的肉色节点的距离,以及随时间的变化。补充文件2显示了2009年到2010年间发生的变化的一个例子。最终编写了脚本来解析语料库中所有年份的集合。在这些逐年总结的基础上,我们做了一个总汇总表(table2).
表格2表明文献中已经包含了重要的迹象,表明了被发现对马铃薯肉色很重要的特定基因的相关性[6.].最显著的是,β -胡萝卜素羟化酶(BCH)和玉米黄质环氧化酶(ZEP)从2007年开始接近(二级邻居),并在2010年转变为肉色节点的直接邻居。在调查2006年至2010年期间促成ZEP转变的句子时,我们发现该基因被假设与肉色相关[4.那27直到2011年实验证据发表。提供这些联系的文献(出版物和确切的句子)的细节可以在补充文件中找到3..
类似地,假阳性如番茄红素(一种在马铃薯块茎中未发现的代谢物)在KN中作为一级邻体出现。虽然对大多数领域专家来说,番茄红素是导致番茄果肉颜色的化合物,因此从知识网络中消除番茄红素是微不足道的,但它确实加强了对领域专家将他们的知识应用于这些结果的要求。
讨论
这项工作担任试点,用于研究使用NLP平台,如Watson,以便在植物科学文献中表现知识发现。随着学术出版物数量的指数增加和可用生物文学的纯粹数量,研究人员正在寻找越来越困难,以了解与其领域相关的所有信息。在单个网络中组装来自可用文献的知识对于为其感兴趣领域的组件的更好整体图像提供更好的整体画面,可以为新的假设或援助研究人员提供有用。然而,与人类研究专家不同,机器更具挑战性,了解复杂句子的生物见解和科学文学的文本结构。
在汇编我们的训练语料库时所做的选择,特别是主题和技术文章的预选,可能会对我们的模式产生偏见。每个NLP模型都有一个它能解决的研究问题的有限范围,而这种特殊的偏差在我们的统计分数中表现得足够好。我们所建立的NLP模型的类型系统无法捕捉和反映知识网络中所有的生物复杂性。然而,我们开发的NLP模型旨在只挖掘基因型-表型信息和从科学文献到knn的潜在机制,使这些知识成为结构化数据,便于机器和人类阅读。由于我们对语料库的选择,这个模型必须学会在非常特定的环境中识别基因、蛋白质、代谢物和性状关联。其他具有完全不同功能和上下文的作物、基因、蛋白质和性状可能用不同的语言模式进行描述,这些在我们的文献集中没有出现。例如,像开花时间这样的特征通常用与组织颜色截然不同的方式描述,因此,为了成功地捕捉细节,应该给模型提供不同的训练。
而且,只捕获了这些实体之间关联的一般关系(“与……相关”)。在我们的模型中,忽略了两个实体(正的、负的、不明确的)之间的关联程度。然而,我们的模型的表现是令人满意的试点研究,并解决上述研究目标。为了优化人工标注训练集的效率,我们将自己限制在有限的34篇全文文章的训练语料库中。尽管训练是有限的,但它仍然足以使我们的模型从测试集中提取相似的知识,测试集中是指不同作物、性状和过程的文档集合。
在为我们的NLP模型制作测试语料库时,我们吸收了来自其他国家的文献茄科作物种类(番茄,辣椒,茄子)。从所有这些不同的文献资源中挖掘和收集信息到一个单一的KN中是有点争议的。许多基因和代谢物参与了这些作物物种的一个类似的生物机制。然而,在某些情况下,关于其他物种的文献可能会引入噪音,而在另一些情况下,它可能是思想的来源。这里有一定的权衡:处理过的文档的范围越广,噪音的幅度就越大,但潜力也越大。毕竟,这项试验的前提是,在一个广泛的科学领域,新发表的研究将不加区别地汇集到一个NLP模型中,以产生可以帮助人类的网络。
我们根据出现关系的文档数量应用的权重函数可以提供进一步的见解。很明显,在所有网络中,大多数关系只出现一次(灰色边缘)。然而,我们观察到经常出现的关系分布的差异(黑色边)。在无花果。1(完全训练集),在第五圆区域外部没有延伸的黑边(最多2nd邻居),而在图4中。3.(测试集),有跨越整个KN的黑色边缘(区域包括第四阶邻居和更高)。我们可以假设这种效果是测试集专题多样性的结果,它专注于整个茄科家庭,而不仅仅是土豆。有些关系可能会出现更好的成立,是因为某些相互作用可能更多的一个物种,而不是在所有其他被调查。然而,在种类及其在KNS以这样的方式可视化之间转移知识的潜力。因此,我们可以进一步推测,我们的范围的附加扩展到除属其他茄科(如。拟南芥)将产生更多的见解。
尽管将多个属集成到kn中可以提供许多好处,如上所述,但我们选择在这个用例中避免这样做。我们的目标是进行一项试点研究,以确定这些方法的潜力,鉴于此,我们的解释有必要选择一个相对较好映射的有限领域。通过这种方式,我们可以理清不同的关系,为研究问题收集信息,而不会因为大量的物种而使我们的调查复杂化。专家知识已经证实,有限范围的KNs将自由文本提取成实体之间的真实连接,并支持更广泛的多物种网络也有可能生成假设。
当涉及到用于文本分析的文档部分时,存在一种平衡。摘要是文章中重要信息的一种易于获取和总结的形式。然而,不同的期刊对他们发表的科学文章的摘要和其他部分规定了不同的格式。因此,在摘要中提到的可挖掘信息的质量取决于期刊以及文章的类型。诸如评论、科学方法或覆盖广泛主题的文章的摘要可能不能提供全面的可挖掘的科学线索。例如,在《自然》杂志上,投稿不一定总是在其摘要中正式描述所有的科学前沿,而结果更多地在主要文本中被提及。
值得一提的是,在某些情况下,民族lp的方法未能达到预期。在生物实体被缩写的情况下,或者两个实体之间的关联在一个以上的句子中被提到,我们的NLP模型不能预测这些实体和关系。沃森的打字系统包括将缩写条目或代词参照其原始形式的功能。然而,由于我们的训练语料库中实例数量相对较少,Watson的NLP模型无法捕获这些实体和关系。然而,在这方面,沃森并不是唯一的。事实上,大多数NLP工具都有同样的缺陷。生物缩写是随意的。通常,两个生物学概念有相同的缩写。例如,缩写MIC可能意味着最低抑制浓度,或指主要组织相容性复合体(MHC) I类链相关(MIC)基因。在更大的语料库上进行训练可能会提高预测正确实体的准确性。
总的来说,我们的工作产生了一个模型,在有限努力条件下为有意义的KNs的构建和时间分析提供了动力。我们得出的结论是,在这些链接被实验证实或发表之前,拥有我们上述描述的可用信息可以提供科学相关链接的关键迹象。鼓励和促进假设生成的主要因素是将知识整合到网络中,在网络中,没有直接连接的节点可以是紧密的(例如二级或三级邻居)。整合视角对于将多个物种的知识积累到同一个网络中也很重要,尽管这种方法有内在的风险。总而言之,我们相信,对于训练集大小和类型系统定义,更密集的努力将产生更好的结果,并可以在汇集来自大型文献语料库的不同信息和假设生成方面发挥重要作用。我们的kn中的边是根据每条边出现在的文档的数量来衡量的。
在未来,我们将尝试进一步的权重归因方法,可能基于从精选数据库中获得的实验性重要信息,或者特定关系在文本中出现的次数。交叉引用精选资源将很好地服务于这些网络,因为实验验证的关系可以在文本中帮助过滤不太可靠的(消极或环境)关系。因此,文本挖掘可以以不同的方式更有效地用于比较已建立和新出现的知识。这种方法与其他数据库在他们的记录和支持他们的出版物之间建立联系的方法不同,如pubmed2ensembl BioMart [11].我们的NLP模型的另一种应用方式是在线期刊文章的文献注释。它可以检测到基因与性状之间的关联,并突出显示以前见过的关联,甚至包括它们发生频率的迹象。这可以帮助读者理解和欣赏所提出的主张的新颖性。
结论
我们的工作强烈表明,计算机辅助提取来自植物科学文献的知识可以促进研究。我们时间分析的结果表明,可以公布制定新假设所需的各个组成部分,但仍然保持不关联。因此,将这些组件集成到综合知识网络中可以加速生成新假设。
方法
实验全集
要制定监督的NLP模型,我们将科学文章组装成2个语料库,包括培训集和测试集。培训集由开源全文文章组成,而测试集是由PubMed摘要构建的。
该培训语料库收集了34篇全文科学文章(见补充文件)4.)和已知的生物实体,如涉及类胡萝卜素途径的代谢物和蛋白质,例如-胡萝卜素羟化酶和玉米黄质环氧化酶[7.].此语料库是在各种学术平台上搜索的结果,不仅在那里审查了内容,也是呈现的方式。培训集用Watson知识工作室(WKS)手动注释。由于WKS仅在句子中检测实体关系的限制,我们消除了在同一个句子中没有或少数关系的例子的文章。WKS使用这些手动注释来生成可捕获表型块茎特征和相关基因,蛋白质和代谢物的监督NLP模型。稍后,我们评估了该监督NLP模型的能力,在此训练集上构建知识网络(KN)以及更大的测试集。
测试集由PubMed从2000年到2016年的4023个摘要组成(可以在[28])。这些摘要是基于植物遗传学的文章,重点关注4种主要的茄科作物(番茄,土豆,茄子和辣椒)。为了限制NLP模型的范围,以发现与块茎肉色相关的直接基因组关联,测试集中没有包含与病原体相关的文章。建立的NLP模型能够提取块茎肉色性状的KNs。然而,在测试集的文章处理植物遗传学的各种不同的主题,不仅限于块茎肉颜色性状。这个测试集对NLP模型提出了挑战,使之成为一个更真实的应用程序,而不是我们训练集中的受限用例。
此外,分析包含在摘要和文章的全文表示信息之间的差异,我们把训练集中到基于片段的子集。我们还分了测试集摘要基于其出版年份子集,研究知识的随时间的变化。
沃森知识工作室和沃森探险
IBM的沃森知识工作室(WKS)是一种专有的文本挖掘解决方案。它可用于构建执行命名实体识别(NER)和关系提取机器学习模型,采用的技术方法[状态29-32].该模型可以针对不同类型的文本(例如,营销,法律,科学),并定制对他们产生的注释类型。
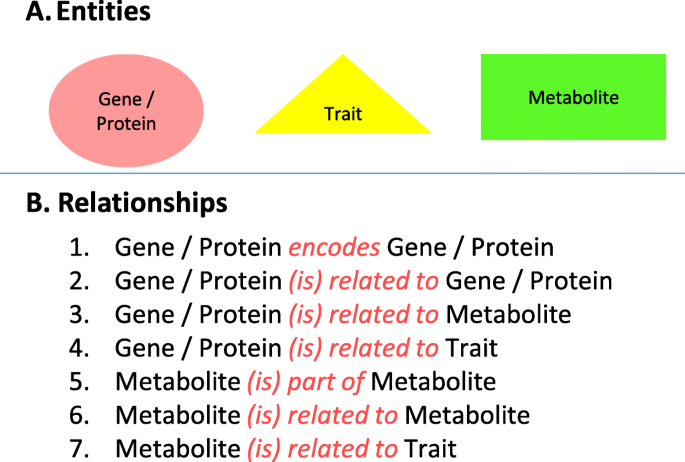
为了在WKS中构建机器学习注释器,用户必须首先定义一个类型系统来建立“实体”(即他们希望它捕获的事物的类别/类)以及它们之间的“关系”。在类型系统到位后,他们标记了这些实体的所有出现(“提及”)和具有代表性的文本集合中的关系,产生了一个基本事实。这些集合中的一部分,即训练集,然后由WKS分析特定领域的语言结构、模式和细微差别,从而生成机器学习模型。另一部分,测试集,只用于量化模型的性能(精度,召回率)。可以迭代地更改类型系统和注释,直到模型执行得令人满意为止。
我们使用WKS训练一个NLP模型,然后将其部署到同一个训练集和一个进一步的测试集上。我们模型的最终类型系统包括三个实体(Gene/Protein, Metabolite, Trait)以及它们之间的七种关系,如图所示。4..我们在简单而包罗万象的关系中取得了最好的结果,这就是为什么许多关系只被贴上了“相关”的标签。例外(“编码”,“部分”)被包括在内,因为语料库中的大量实例允许WKS生成能够在文本中成功识别它们的模型。
每个实体都可以由特定于实体的字典支持。在人工注释语料库之前,NER的预注释步骤中使用字典。为了减少噪音(实体和关系的不希望的注释),所有的字典都很小,并且仅限于已知与块茎果肉颜色或类胡萝卜素途径相关的分子实体。我们从已知的分子数据库或本体中选择我们喜欢的标签。基因/蛋白质和代谢物词典分别包含183个基因/蛋白质和85个代谢物。来自茄科表型本体论的56个马铃薯相关性状[33组成了Trait字典。
沃森资源管理器(WEX)可以使用该模型来注释新的文件。其管道的示意图可以在补充文件中可以看出5..它的输出是XML/CAS(可扩展标记语言/内容和结构)文件中的文本文档,包含对已提取的实体及其关系的注释,以及它们的原始文档(和文档位置)。我们使用这些XML/CAS文件来构建我们的kn。
决策建模
为了训练我们的NLP模型仅捕捉基因型-表型实体及其关系的kn,类型系统在迭代过程中经历了许多重大的变化和修订。通过试错优化,根据知识在KN中捕获和呈现的情况,引入或抛弃实体和关系。在我们的分析中,知识三元组被定义为一个数据结构,由两个实体和它们的底层关系的标签组成。
以下介绍了一些重要的模拟决策如下。
已测试但未包含在最终模型中的生物实体:
生化过程
代谢途径
特征值
生物体名称,物种名称和基因型
虽然这些生物实体出现在文本中,并包含理解表型所涉及的生物学机制的知识来源,但文本中提到的数量不足以让WKS充分训练一个模型。因此,我们选择不在NLP模型的类型系统中包含这些实体。此外,将这些实体纳入我们的模型将转移研究重点,从挖掘文本中的基因型-表型关系。
基因和蛋白质与单个实体的组合:
最初,我们把基因和蛋白质作为两个独立的实体。然而,在手工注释的过程中,在区分这两者时遇到了困难,因为它们经常在文本中互换使用。此外,对于主题专家来说,将它们结合在一起并没有丢失多少信息,而将它们分离会导致许多错误的分类。因此,在我们的类型系统中,基因和蛋白质是一个单一的实体。
代谢物的注释规则(特定代谢物提及vs通用提及):
代谢物以不同的形式出现在科学文献中。其中提到的可能包括特定的复合术语(例如petunidin-3-p-香豆素-芦丁苷-5-葡萄糖苷)或更多的通用术语(例如类胡萝卜素)。根据我们的类型系统,我们注释了所有形式的代谢物提到,这样我们既可以捕获具有特定实体的知识三元组,也可以捕获具有通用实体的知识三元组。
基因注释规则:
就像代谢产物的情况一样,基因可能以不同的形式引入。有时以全称(玉米黄质环氧化酶),有时以缩写形式(ZEP),有时以物种标志作为前缀(勒齐柏林飞艇(Lycopersicon esculentumzep])。我们选择注释所有这些案件以培训模型。
建设和知识网络的可视化
在构造KN时,只使用具有关系的实体。提及本身没有联系的实体不包括在KN中。在Python脚本的帮助下,我们从XML/CAS文件中过滤出实体数据和关系数据[34].此脚本捕获在容易解析的CSV作为知识三元关系(逗号分隔值)包含关系ID的文件,关系类型,每个实体的原始提,实体标签,实体类型,在其中发生这句话文件,句子位置和位置源和目标节点。
由于在语料库中不同的实体出现在不同的拼写中(例如:β-Carotene,B-胡萝卜素,β-胡萝卜素),我们还包括归一化步骤,将另外的首选标签归因于每个实体。这是在提取的个人实体列表中手动完成。在规范化过程中,我们首先将实体的所有拼写转换为美国英语大写字符。另外,从基因名称中除去与物种有关的前缀。例如,这个术语英石AN1,指花青素1 inSolanum Tuberosum.(马铃薯)转化为AN1。同样,表示基因家族个体成员的后缀也被移除,例如BCH1和BCH2(均指β -胡萝卜素羟化酶的形式)被转化为β -胡萝卜素羟化酶。
对于代谢产物,EC数参考被转换为酶的全名。此外,撇号和#符号被删除,如类黄酮-3 ',5 ' -羟化酶变成类黄酮-3,5-羟化酶,9#-顺-新黄质变成9-顺-新黄质。最后,所有缩写都扩展为长形式,如NCED2扩展为9-顺式环氧类胡萝卜素双加氧酶。这些首选标签基于Uniprot [35]为基因/蛋白质,KEGG [36为代谢物,茄科表型性状本体论[37]的性状。
虽然上述步骤降低了特定实体的特异性(例如,我们将BCH1和BCH2标记为BCH),但与token化的情况一样,这种简化增强了网络连通性,尽管会丢失信息。
最后,使用Cytoscape 3.7.1版本可视化这些kn [38].Cytoscape可以使用CSV文件作为输入来绘制kn。这些网络还包含一个基于每条边出现的文档数量的权重函数。我们选择用两种颜色来表示这个文档频率:灰色,当边缘只出现一次时,黑色,当它出现多次时。这种区分使我们能够区分潜在的新关联和更广泛的研究关联。
可用性数据和材料
本研究中产生或分析的所有数据均包含在本文及其补充文件中。此外,在Watson Knowledge Studio (WKS)上提取马铃薯科学文章中的基因型-表型关系的监督NLP模型被归档在这里[39].
声明
缩写
- BCH:
-
β-胡萝卜素羟化酶
- 中科院:
-
内容和结构
- CHY2:
-
β-胡萝卜素羟化酶2
- CSV:
-
逗号分隔值
- FN:
-
假阴性
- FP:
-
误报
- IBM:
-
国际商业机器公司
- KN:
-
知识网络
- 勒齐柏林飞艇:
-
Lycopersicon esculentumZEP.
- MHC:
-
主要的组织相容性复合物
- 麦克风:
-
最小抑制浓度
- 麦克风:
-
MHC类连锁相关
- 尼珥:
-
命名实体认可
- NLP:
-
自然语言处理
- 英石AN1:
-
Solanum Tuberosum.花青素1
- TP:
-
真正的阳性
- WEx:
-
Watson Explorer.
- WKS:
-
沃森知识工作室
- XML:
-
可扩展标记语言
- 齐柏林飞艇:
-
玉米黄质环氧酶
参考文献
- 1
Sulli M, Mandolino G, Sturaro M, Onofri C, Diretto G, Parisi B, Giuliano G。PloS ONE。2017;12(9): 0184143。
- 2
黄志强,王志强,王志强,等。马铃薯高品质种质类胡萝卜素的分离及其与β -胡萝卜素羟化酶多态性的关系。马铃薯学报2006;83(5): 365 - 72。
- 3.
Giuliano G.植物类胡萝卜素:基因组学符合多基因工程。CurrOp植物BIOL。2014;19:111-7。
- 4.
威科A-MA,Uitdewilligen JG,Kloosterman BA,胡腾RC,维瑟RG,凡埃克HJ。的类胡萝卜素合成途径基因马铃薯块茎中的玉米黄质的积累重要的等位基因的鉴定。植物mol biol。2010;73(6):659-71。
- 5.
Chitchumronchokchai C,Diretto G,Parisi B,Giuliano G,Failla ML。金色土豆的潜力改善了发展中国家的维生素A和维生素E状态。Plos一个。2017;12(11):0187102。
- 6.
Acharjee A, Kloosterman B, de Vos RC, Werij JS, Bachem CW, Visser RG, Maliepaard C.基于随机森林回归的马铃薯组学数据集成和网络重建。Acta agriculturae sinica . 2011;705(1 - 2): 56 - 63。
- 7.
Acharjee A,Kloosterman B,Visser RG,Maliepaard C.使用随机林预测表型性状的多OMIC数据的集成。BMC生物信息学。2016;17(5):180。
- 8.
Hirschberg J, Manning CD.自然语言处理进展。科学。2015;349(6245): 261 - 6。
- 9.
生物医学文本挖掘十年来面临的挑战:成功、失败与未来。短暂的Bioinform。2015;17(1): 132 - 144。
- 10
harston N, Filsell W, Stumpf MP。论文内容:基因组学和系统生物学的文本挖掘。哼基因组学,2010;5(1): 17。
- 11
引用本文:Baran J, Gerner M, Haeussler M, Nenadic G, Bergman CM。Pubmed2ensembl:基因生物学文献挖掘资源。PloS ONE。2011;6(9): 24716。
- 12
Ding R,Arighi CN,Lee J-Y,Wu Ch,Vijay-Shanker K.Penn,科学文献中的植物基因和蛋白质的基因标准化工具。Plos一个。2015;10(8):0135305。
- 13
生物医学领域植物化学关系的语料库。BMC生物信息学。2016;17(1): 386。
- 14
基于生物医学命名实体识别的多源数据挖掘和评估。生物信息学。2018;34(14): 2474 - 82。
- 15
崔华。利用自然语言处理从生命树的分类描述中提取表型特征。应用植物学报2018;6(3): 1035。
- 16
QTLTableMiner++:科学论文中QTL表的语义挖掘。BMC生物信息学。2018;19(1): 183。
- 17
赵洪波,李洪波。一种用于生物医学产品命名实体规范化的方法:应用于疾病和植物。BMC生物信息学。2017;18(1): 451。
- 18
使用主题建模和自然语言处理预测药物适应症和副作用。J Biomed Inform. 2018;87:96 - 107。
- 19
哈恩U,科恩KB,加藤Y,沙阿NH。挖掘药物基因组学的文献-对目前状况的调查。短暂的Bioinform。2012;13(4): 460 - 94。
- 20.
关键词:文本挖掘,数据集成,网络生物学,植物研究拟南芥.植物学报。2013;25(3): 794 - 807。
- 21
Sharma V, Law W, Balick MJ, Sarkar IN。利用生物医学自然语言处理工具从历史文本中识别药用植物知识。AMIA Annu Symp Proc. 2017;2017:1537。
- 22
库克HV,詹森LJ。指南字典基于文本挖掘在:拉尔森R,奥普雷亚T,编辑器。生物信息学和药物发现。在分子生物学方法,第1939年纽约:Humana公司出版社:2019。https://doi.org/10.1007/978-1-4939-9089-4_5.
- 23
基于自然语言处理和机器学习的脑MRI报告识别方法。PloS ONE。2019;14(2): 0212778。
- 24
费鲁奇哒。"这是华生"的介绍。IBM Journal of Research and Development. 2012;56(3.4): 1 - 1。
- 25
IBM Watson:认知计算如何应用于生命科学研究中的大数据挑战。其他。2016;38(4): 688 - 701。
- 26
辛格g,papoutsoglou ea。Cytoscape会议与IBM Watson的监督NLP模型提取的马铃薯知识图表。Zenodo》2019。https://doi.org/10.5281/zenodo.3275105.2020年9月14日。
- 27
β -胡萝卜素羟化酶的沉默增加了马铃薯块茎中总类胡萝卜素和β -胡萝卜素的水平。BMC Plant Biol. 2007;7(1): 11。
- 28
Papoutsoglou EA, Singh G. Test set - 4023 PubMed摘要(用于手稿:从植物科学文献中提取知识网络:马铃薯块茎果肉颜色作为一个示范性状)。Zenodo》2020。https://doi.org/10.5281/zenodo.3999597.2020年9月14日。
- 29
张涛。基于分类器组合的命名实体识别。见:第7届自然语言学习学术会议论文集。埃德蒙顿:计算语言学协会:2003。p . 168 - 71。https://doi.org/10.3115/1119176.1119201.
- 30.
Kambhatla N.将词汇,句法和语义特征结合起来,具有最大熵模型来提取关系。在:互动海报和示范会议上的ACL 2004的诉讼程序。巴塞罗那:计算语言学协会:2004年。22-es。https://doi.org/10.3115/1219044.1219066.
- 31
McCord MC, Murdock JW, Boguraev BK.沃森的深度解析。IBM Journal of Research and Development. 2012;56(3.4): 3 - 1。
- 32
王超,范建平,王志强,王志强。深度qa中关系提取与评分方法研究。IBM J Res Dev. 2012;56(3.4): 1胜9负。
- 33
Bioportal。《茄科表型本体论》,2018。http://bioportal.bioontology.org/ontologies/SPTO?p=classes&conceptid=root。2020年9月14日。
- 34
Papoutsoglou EA,辛格G. WatsonPotato。GitHub。2020。https://github.com/PBR/WatsonPotato.2020年9月14日。
- 35
pdir S, Martin MJ, O’donovan C. UniProt蛋白质知识库。方法:Mol Biol. 2017;1558:41-55。https://doi.org/10.1007/978-1-4939-6783-4_2.
- 36
Kanehisa M, Furumichi M, Tanabe M, Sato Y, Morishima K. KEGG:基因组、通路、疾病和药物的新视角。核酸Res. 2016;45 (D1): 353 - 61。
- 37
Shrestha R, Matteis L, Skofic M, Portugal A, McLaren G, Hyman G, Arnaud E.通过使用实践作物群落开发的作物本体的数据注释,架起对综合育种有用的表型和遗传数据。杂志。2012;3:326。https://doi.org/10.3389/fphys.2012.00326.
- 38
Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape:一种生物分子相互作用网络集成模型的软件环境。基因组研究》2003年;13(11): 2498 - 504。
- 39
IBM Watson注释马铃薯文献的NLP模型。Zenodo》2019。https://doi.org/10.5281/zenodo.3260364.2020年9月14日。
- 40
Singh G.基因组学使用分子数据库和科学文献的基因组注释的知识发现数据集成。博士文博士大学。2019年。https://doi.org/10.18174/505685.2020年9月13日。
致谢
我们要感谢Dick De Ridder(Bioinformatics,Wageningen大学和研究),Willem Jan Knibbe(数据能力中心,Wageningen大学和研究)和Matthijs Brouwer(植物育种,Wageningen大学和研究),用于批评手稿和他们的稿件宝贵的反馈。此外,我们还感谢IBM技术支持,继续支持。本文基于题为“采用分子数据库和科学文学的基因组数据集成的基因组数据集成的知识发现的基因组学数据集成的基因组学数据集成”和“科学文学”的第一个作者Gknoor Singh章节之一的结果[40].
资金
这项工作部分由荷兰埃斯特北京·诺维州授予(Candygene,Grant Eset.14.011)部分支持,部分是由Wageningen大学和研究植物育种的补助金。资金机构在研究和收集,分析和解释的设计中没有作用,或者在书面上的书面上。
作者信息
从属关系
贡献
构思设计实验:EP、GS、CWBB、RF、RGFV。方法论的发展:GS, EP, FKL, BV。进行实验:EP、GS、FKL、CWBB。分析数据:GS, EP。撰写手稿:GS, EP, CWBB, RGFV。编辑稿件:EP, GS, CWBB, RF, RGFV, MR, FKL, BV。所有作者都对稿件进行了修改,阅读并批准了提交的版本。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
额外的文件1
混淆矩阵。一个文本文件(.csv),其中包含一个混淆矩阵表,显示34篇完整训练集的每篇文章的实体检测。
额外的文件2
肉色和它的最终邻居之间的联系的单年差异汇总表。一个PDF文档,显示了每个肉色节点之间的分离程度,以及最终成为其直接邻居的节点。
额外的文件3
追踪zEP/BCH与肉色之间的关键联系。一个PDF文档文件,详细描述了2007年和2009年BCH/ZEP和肉色之间的关键联系,如结果部分所述。
额外的文件4
培训集文档清单。一个文本文件(.csv),包含在训练集中使用的34篇文章的列表。
额外的文件5
Watson Explorer管道的原理图。A.PDF文件具有图,示出了Watson使用的统计信息和关系提取(SIRE)管道。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
辛格、帕波索格卢、凯杰茨-拉勒曼。等等。从植物科学文献中提取知识网络:马铃薯块茎果肉颜色作为典型性状。BMC植物BIOL.21日,198(2021)。https://doi.org/10.1186/s12870-021-02943-5
收到了:
公认:
发表:
关键词
- NLP
- 植物科学文献
- IBM华生
- 文字矿业
- 关系抽取
- 知识网络