抽象的
背景
DNA序列组合物影响减数分裂重组。然而,串联重复组合物与普通小麦的重复组成和减数重组之间的相关性(小麦L.)还不清楚。
结果
非变性原位杂交(ND-FISH)与来自串联重复和单拷贝FISH衍生的寡核苷酸(低聚)探针的荧光,使用3种小麦5A染色体(5AL)的长臂的调查重组。5AL535-18 / 275.臂携带串联重复pTa-535, Oligo-18和pTa-275, 5AL119.2-18 / 275臂携带串联重复pSc119.2、Oligo-18和pTa-275和5AL119.2手臂携带串联重复pSc119.2。在5AL的后代中535-18 / 275. × 5AL119.2在pSc119.2和pTa-535簇之间(119-535区间)发生双重组,而pTa-535和Oligo-18/pTa-275簇之间(535-18区间)发生双重组。5AL的后代在119-535区间的重组率535-18 / 275. × 5AL119.2-18 / 275高于5AL535-18 / 275. × 5AL119.2.在119-535间隔中产生的重组5Al119 + 535.与PTA-535和pSc119.2串联重复和5AL段不没有这些重复的细分。该5AL119 + 535.和5al.不段进行了单拷贝探针SC5A-479和SC5A-527的信号点之间的局部。在中期5AL SC5A-479和SC5A-527之间的段不显着长于中期5al中的时间119 + 535..
结论
串联重复引起的结构变异可能是影响小麦减数分裂重组的因素之一。减数分裂重组将两种连续重复的簇聚集到同一染色体上,使中期染色体更加密集。总之,我们的研究为小麦减数分裂重组和亲本选择提供了一个强有力的工具。
背景
提高染色体重组率可以加速普通小麦新品种的发育(小麦l .)。研究人员对植物减数分裂重组的分子机制进行了广泛的研究,并报道了许多参与这一过程的基因[1],它提供了操纵作物改善机制的机会[2].逆转基因中的突变福建(Fanconi贫血互补组M)导致杂交水稻和豌豆减数分裂重组增加两倍[3.].然而,福建突变几乎没有对杂种中的重组产生影响拟南芥但导致近交系数量增加了三倍[3.,4,5].结合反杂交突变体recq4(RecQ样螺旋酶4)和图1(海豹状物 - 1)导致交叉频率增加7.8倍,而福建,recq4和图1三重突变体显示较少的重组[5].这些发现表明,基因以外的因素有助于控制减数分裂重组。两种基因和染色质结构控制重组率。在人类和动物中,重组率与通过G条带和序列组合物的染色强度显示的细胞遗传结构有关,包括重复元素,GC含量,CPG密度和多(a)/ poly(a)延伸[6,7].在拟南芥蒂利亚纳富含CCN重复和CTT-Repeat基序在交叉区域中富集[8].CG位点DNA甲基化的减少增加了中性染色质区域的重组率,但不在中心异染色质区域[9].异铬胺在减数分裂重组中起作用。它抑制裂变酵母中的浓缩物质重组[10.].在真核生物中广泛研究了仔细杂料蛋白在抑制减少体状重组中的作用[11.].通过基因突变的H3K9ME2和非CG DNA甲基化途径的破坏增加了杂种和近交的脑大分子交叉拟南芥(12.].同时,在小鼠中,mo-2小卫星串联阵列赋予了性染色体假常染色体区重组的高阶结构[13.].因此,串联重复序列对染色体重组的影响值得研究。串联重复序列在染色体重组中的作用可以作为参考模型。以往的研究表明,小麦染色体交叉与基因富集区之间存在相关性[14.,15.,16.,17.,18.].然而,由于异源多倍性和重复序列的存在,普通小麦的重组应考虑不同方面[19.].
从普通小麦中发现了一些新的串联重复[20.,21.].在我们之前的工作中,由这些串联重复序列衍生的寡核苷酸(oligo)探针在普通小麦的5AL臂上显示出很大的结构变化[22.,23.].然而,串联重复组成与小麦中重组率之间的相关性仍然不清楚。本研究研究了基因组结构变化对小麦染色体5A中的重组频率反射的基因组结构变化的影响。
结果
5A染色体核型,FISH
在小麦中,在图5A的染色体可以从基于寡-713,低聚pSc119.2-1,和寡聚pTa535-1 [的信号模式的其他染色体区分开22.,24.].在5BB的端粒区域(5B染色体的短臂)上的寡核糖-CAC119.2-1信号比5A(5A染色体的短臂)更强,以及Oligo-PTA535-1的信号模式在5D中染色体不同于5A染色体[22.,24.].然而,当Oligo-pSc119.2-1信号同时出现在5AS和5AL上时,很难区分5A和2B染色体。然后,根据亚稳心特征对5A染色体进行鉴定。pericentromeric地区7(7号染色体的短臂)和7 ds (7 d染色体的短臂)含有益生元- 713信号,然而,7和7 d染色体可以区别5染色体基于Oligo-pSc119.2-1的信号模式和Oligo-pTa535-1 [22.].
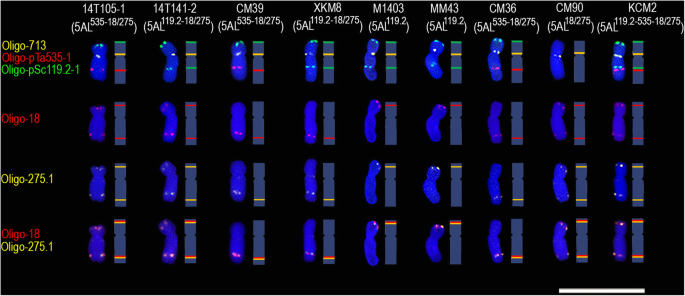
本研究原位杂交(Nd-Fish)的非变性荧光表明,Oligo-713的信号发生在所有九个小麦品种/线的5As臂的蠕虫熵区域上(图。1).探针Oligo-PSC119.2-1在除CM90外,除了CM90之外的八个小麦品种/线的5As臂,以及14℃,XKM8,M1403和MM43的5A臂(图。1).在14t105-1,cm36和cm39的5Al臂上发生oligo-pta535-1的信号(图。1).Oligo-pSc119.2-1和Oligo-pTa535-1探针都在KCM2的5AL臂上产生信号(图)。1).寡聚-275.1和寡聚-18的信号发生在14T105-1,141-2,CM90和KCM2的5As和5Al臂上(图。1)和CM39、XKM8和CM36的5AL臂(图。1).虽然在M1403和MM43的5AS臂上出现了Oligo-275.1和Oligo-18信号,但在两个品种的5AL臂上没有出现。1).因此,我们命名为5AL武器,如下所示:14 T105-1,CM39和CM36为5AL535-18 / 275.;14 T141-2和XKM8作为5AL119.2-18 / 275;M1403和MM43为5AL119.2;CM90为5al.18/275;和kcm2为5al119.2-535-18 / 275.(无花果。1).
鱼核型和f的重组率2一代
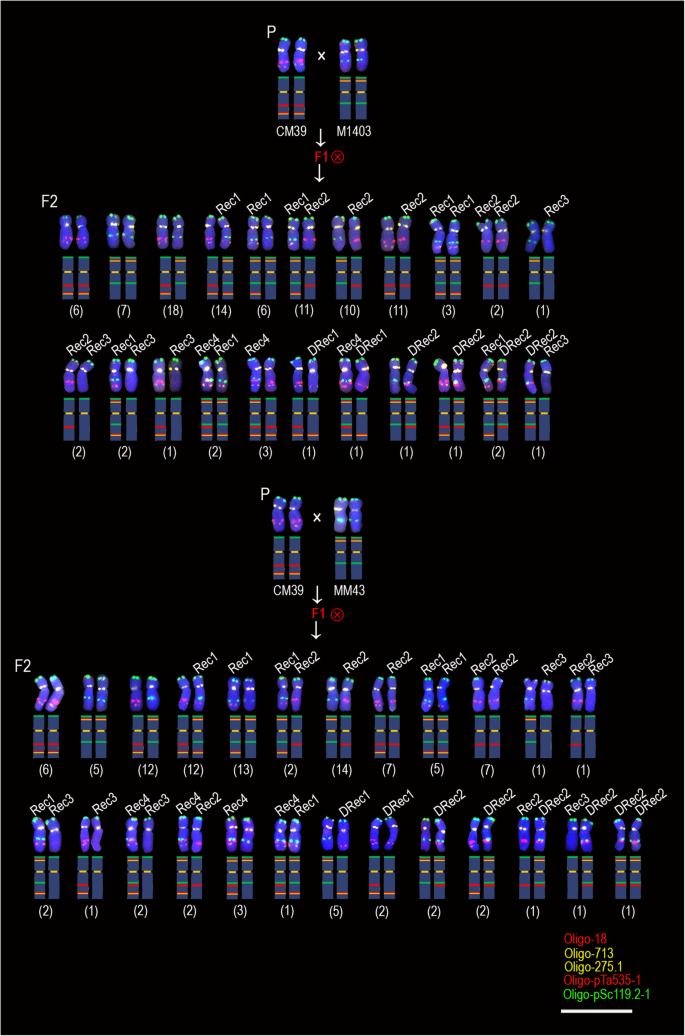
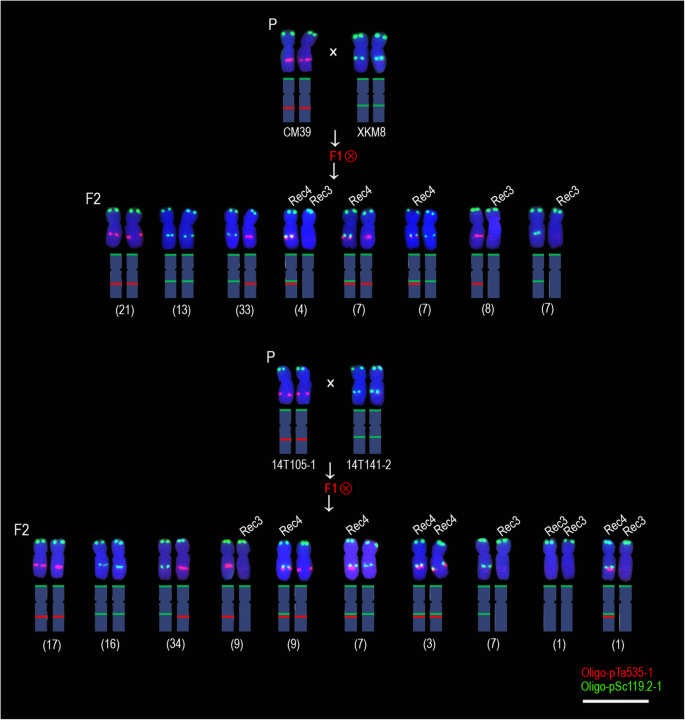
共420 f2以CM39 × M1403、CM39 × MM43、CM39 × XKM8和14个T105-1 × 14 T141-2杂交组合衍生的8405a染色体为材料,分析5AL臂的重组率。其中,CM39 × M1403 (5AL)衍生出106个种子(212条5A染色体)535-18 / 275. × 5AL119.2)、110条(220条5A染色体)来自CM39 × MM43 (5AL .535-18 / 275. × 5AL119.2),10.0 (200 5A chromosomes) from CM39 × XKM8 (5AL535-18 / 275. × 5AL119.2-18 / 27514 T105-1 × 14 T141-2 (5AL .535-18 / 275. × 5AL119.2-18 / 275). 对于源自CM39的后代 × M1403和CM39 × MM43,检测到两个重组间隔(图。2和其他文件1,2).Oligo-pSc119.2-1和Oligo-pTa535-1信号位点之间发生了一次重组,命名为119-535区间。另一个重组发生在Oligo-pTa535-1和Oligo-18/Oligo-275.1信号位点之间,该区域命名为535-18区间(图535-18区间)。2和其他文件1,2).对于CM39×XKM8和14 T105-1×14 T141-2的后代,观察到在寡核苷酸-275.1和寡核苷酸-18信号中仅观察到119-535间隔中发生的重组。535-18 / 275.和5al.119.2-18 / 275武器(图。3.和附加文件3.).CM39×M1403的106种子具有22种组合类型的5A染色体(图。2),重组分别发生在20条和88条5A染色体的119-535和535-18区间,其中7条染色体发生了双重组(图5)。2).CM39 × MM43的110个种子有25个5A染色体组合类型(图2)。2).在220 5A染色体中,分别在119-535和535-18间隔中发生重组,分别在31和96 5A染色体上,包括具有双重重组的15染色体(图。2).在CM39 × XKM8衍生的100粒种子(200条染色体)中,仅观察到8条5A染色体的组合类型,37条5A染色体在119-535区间内发生重组(图)。3.).在这些5A染色体上未显示oligo-713,oligo-275.1和oligo-18的信号,因为它们不能在535-18间隔中反映重组。类似地,在104种子(208 5A 5A染色体)中仅观察到衍生14t105-1×14 t141-2的10个组合类型的5a染色体,并且在42 5a染色体上发生119-535间隔中的重组(图。3.).
F的组合5A染色体类型2progeny derived from CM39 × XKM8 and 14 T105–1 × 14 T141–2. ‘P’ indicates parental plants. ‘F1’ indicates F1的一代。'Ⓧ”表明自体受精。“F2”表明F2的一代。' Rec1 '和' Rec2 '表示535-18区间重组形成的5AL臂。' Rec3 '和' Rec4 '表示119-535区间内重组形成的5AL臂。' DRec1 '和' DRec2 '表示535-18和119-535区间重组形成的5AL臂。括号中的数字表示含有对应的5A染色体组合类型的种子数。每条染色体的示意图如图所示。比例尺:50 μm
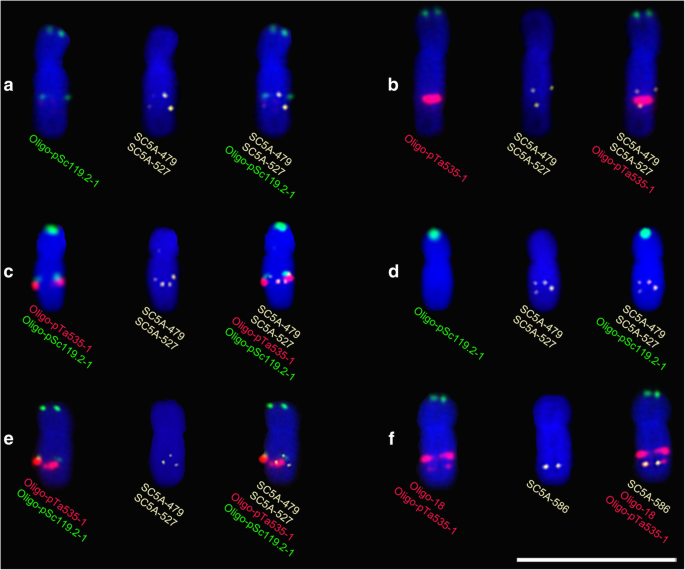
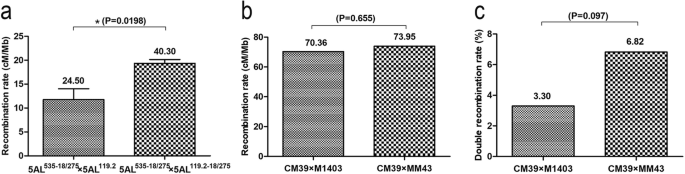
序列单拷贝FISH和ND-FISH检测表明,Oligo-pSc119.2-1和SC5A-479的信号位点非常接近(图2)。4A,C和附加文件4c, f)与Oligo-pTa535-1和SC5A-527的亲缘关系相近(图5)。4b,c和附加文件4c、 f)。SC5A-479和SC5A-527在5AL上的信号位置119.2-535-18 / 275.KCM2的武器证实了这种观察结果(图。4E和其他文件4i)。此外,Oligo-18和SC5A-586信号相近(图5)。4f和附加文件4 l) (The details of obtaining the three single copy probes are described in Methods section.). The 119–535 and 535–18 intervals were determined as 48 Mb and 59 Mb long, respectively, based on the physical distance of the single-copy probes SC5A-479, SC5A-527, and SC5A-586. The recombination rate of the 5AL arms in the four kinds of F2人口数量见表1.CM39 × XKM8 (5AL .)的后代在119-535区间的重组率535-18 / 275. × 5AL119.2-18 / 275)和14 t105-1×14 t141-2(5al535-18 / 275. × 5AL119.2-18 / 275)的表达量高于其他两种组合(5AL)的后代535-18 / 275. × 5AL119.2)(表1),在5al的后代之间观察到显着差异535-18 / 275. × 5AL119.2-18 / 275和5al.535-18 / 275. × 5AL119.2(无花果。5一种)。然而,在CM39×M1403的后代之间的535-18个间隔中没有观察到重组率没有显着差异(5Al535-18 / 275. × 5AL119.2)和CM39 × MM43 (5AL535-18 / 275. × 5AL119.2)(图。5b).我们的分析表明119 ~ 535和535 ~ 18区间存在交叉干扰(表1CM39 × M1403 (5AL .)子代的双重组率无显著差异535-18 / 275. × 5AL119.2)和CM39 × MM43 (5AL535-18 / 275. × 5AL119.2)(图。5c)(在方法部分中描述了重组和干扰的计算细节。)。)。)。
单拷贝探针和oligo探针在5AL臂上的信号位置。一个5A染色体来自MM43,显示5AL119.与SC5A-479和SC5A-527站点之间的119–535间隔相对应的段。b来自CM39的5A染色体显示5AL535与SC5A-479和SC5A-527站点之间的119–535间隔相对应的段。c在119-535区间重组形成的5A染色体显示5AL119 + 535.SC5A-479和SC5A-527位点之间的片段。d在119-535区间重组形成的5A染色体显示5AL不SC5A-479和SC5A-527位点之间的片段。e5A含有5AL的KCM2染色体119 + 535.SC5A-479和SC5A-527位点之间的片段。f5A CM39的染色体,显示SC5A-586和OLIGO-18的关闭信号位点。比例尺:50 μm
SC5A-479和SC5A-527之间的中期染色体区段的相对长度
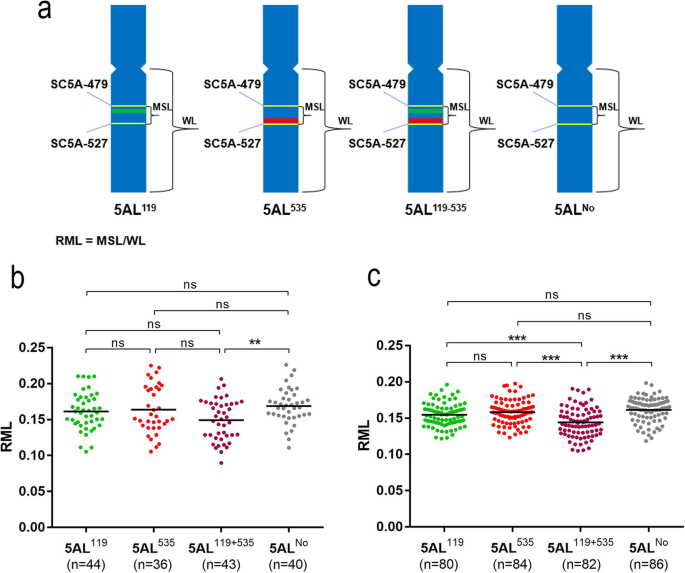
数字2和3.表明,在119-535区间的重组形成的重组类型REC3,REC4和DRec2。和Oligo-pSc119.2-1和寡聚pTa535-1的信号发生在类型REC4 5AL臂从型REC3 5AL臂消失(图2,3.和其他文件1,2,3.).在119-535间隔对应于SC5A-479和SC5A-527对中期5AL武器信号位点之间的片段(图4和附加文件4).我们将SC5A-479和SC5A-527之间的段命名为5AL119.2,5al.535-18 / 275., Rec3和Rec4的5AL臂为5AL119.,5al.535,5al.不,和5119 + 535.分别地四种中期5AL片段(5AL)的相对中期长度(RML)119.,5al.535,5al.119 + 535.,和5不)测量(图。6一种)。从四种类型的F2各代5AL共44、36、43和40个片段119.,5al.535,5al.119 + 535.和5al.不,分别成功测量(图。6b)。该5AL不节段明显长于5AL119 + 535.(无花果。6b)。此外,5al119.,5al.535,5al.119 + 535.,和5不段(图1)在小麦品种XKM8,CM36,KCM2和CM90中也测量以确认(图。6c)。总共80,84,82和86 5al119.,5al.535,5al.119 + 535.,和5不获得并且从4个小麦品种(图测量段。6c)。5119.,5al.535,和5不节段明显长于5AL119 + 535.(无花果。6c)。
5AL的比较119.,5al.535,5al.119 + 535.,和5不段。一个半臂臂测量段的模型。黄频带表示单拷贝探针SC5A-479和SC5A-527的信号。绿色频段表示寡核糖-PSC119.2-1的信号。红色频段表示寡聚PTA535-1的信号。'WL'意味着5AL手臂的整个长度。“MSL”是指SC5A-479和SC5A-527探针之间的中期5AL段的长度。b四种段中的相对中期长度(RML)的比较2几代人。c4个小麦品种4个片段的RML比较。每个点代表单个5AL段的RML。点之间的条形代表RML的平均值。括号中显示了每种线段测量的5AL线段的数量(n)。Ns:无显著性差异,**:p< 0.01, * * *:p < 0.001
讨论
串联重复序列与减数分裂重组的相关性
pSc119.2、pTa-535、pTa-275和Oligo-18 4种串联重复序列在小麦5AL臂上表现出明显的结构变异。本试验中,CM39 × XKM8、CM39 × M1403和CM39 × MM43的杂交组合均为同一亲本CM39。而CM39 × M1403 (5AL)的后代在119-535区间的重组率较低535-18 / 275. × 5AL119.2)和CM39 × MM43 (5AL535-18 / 275. × 5AL119.2CM39 × XKM8 (5AL .535-18 / 275. × 5AL119.2-18 / 275).M1403和MM43的5AL臂表现出与XKM8不同的ND-FISH信号模式。推测119-535区间的重组率变化与5AL臂上的ND-FISH信号模式一致,由14 T105-1 × 14 T141-2 (5AL)衍生的子代支持535-18 / 275. × 5AL119.2-18 / 275).因此,5AL臂串联重复序列的组成与减数分裂重组可能存在相关性。
研究人员以重组间隔观察到DNA转座的较高速率。在土豆,偷渡者微型倒置重复转换元素系列跨越交叉区域[25.].Darrier等。观察到的DNA基序特定的较高的频率到行动-水手普通小麦DNA转座子的重组16.].这些研究发现重组和非重组热点之间在DNA转座子成分的差异沿着相同的染色体中,这样的现象,不同人群观察到16.,25.].推测的序列差异或插入/缺失导致了两种不同F染色体沿3B染色体的交叉频率的显著差异2隔离人群(15.].同时,用于映射影响交叉分布和频率的QTL的嵌套关联映射(NAM)群体表明了具有更多单个核苷酸多态性(SNP)的区域中的重组率较低,而小麦中的SNP更少[17.].此外,在亲本亲缘关系密切的群体中也发现了类似的交叉模式[18.].即使在逆转基因的突变体中,通过SNP显示出最高序列分歧的区域,预防重组3.].在拟南芥,msh2(muts相关异二聚体)突变体与野生型相比,在杂交周围显示出显著减少的SNP富集[26.].这些研究表明,SNP多态性和内含子是影响减数分裂重组的重要因素。虽然在119-535区间内,5AL衍生后代的重组率存在显著差异535-18 / 275. × 5AL119.2和5al.535-18 / 275. × 5AL119.2-18 / 275(无花果。5a)中,5AL臂除其他序列差异,特别是在119-535和535-18的间隔,其导致不同的遗传距离是不容忽视的。除了串联重复,这些序列分量也可能影响不同5AL臂之间的重组率。同时,重组率在119-535区间在后代从5AL衍生535-18 / 275. × 5AL119.2-18 / 275高于5AL535-18 / 275. × 5AL119.2.因此,串联重复序列对减数分裂重组的影响有待进一步研究。
杂合区与纯合区并置对重组的影响
虽然不能得出单独串联重复序列构成导致CM39 × M1403 (5AL)子代119-535区间重组率较低的结论535-18 / 275. × 5AL119.2)和CM39 × MM43 (5AL535-18 / 275. × 5AL119.2),在该间隔中的复合率的变化与5AL臂中的结构差异一致。PSC119.2和PTA-535位点发生不同的ND鱼型在5al之间发生535-18 / 275.和5al.119.2-18 / 275.然而,在5al之间观察到不同的ND鱼型535-18 / 275.和5al.119.2在pSc119.2、pTa-535和Oligo-18/pTa-275现场。在里面拟南芥杂合子区增加了并列巨碱基纯合子区和杂合子区的交叉,杂合子性可增加交叉干扰[27.].因此,我们的研究发现,与这些以前的报告一起,表明需要探讨减数分裂重组杂合和纯合间隔并列的效果。
串联重复对中期染色体凝聚的影响
在这项研究中,重组形成了5AL119 + 535.和5al.不中期段。仅在5AL之间观察到长度的显著差异119 + 535.和5al.不源自F的段2人群;5AL119 + 535.KCM2明显短于XKM8(5Al)119.),cm36(5al535)和cm90(5al不)(图。6b、 c)。这些观察结果表明,5AL臂119–535间隔的其他序列差异影响5AL的长度119.,5al.535,5al.119 + 535.和5al.不段。但是,对于f2群体和小麦品种间的5AL不细分明显长于5al119 + 535.段。这些发现表明,在同一染色体中含有两个纵向重复簇的中期染色体段将更加稠凝。相反,将更加放松较少的串联重复的中期染色体段。此外,本研究中使用的基于细胞的基于软件的方法来测量长度是可靠的。Tandemly反复卫星DNA序列可以通过诱导异铬胺和/或Centromere的改变来驱动种群和物种差异[28.].卫星DNA序列是异染色质的主要组成部分,在异染色质的形成和调控中发挥重要作用[29.].在小鼠中,异染色质改变有丝分裂染色体染色质环的大小[30.].因此,5119 + 535.pSc119.2和pTa-535的聚集可能使本研究的片段高度异染色质化,使片段更加凝聚。
ND-FISH在小麦育种中的应用
了解影响重组率和重组点变化的因素在小麦育种方案中是重要的。通常,交叉频率从端粒降低到真核生物中的CENTROMERE [31.],包括小麦[15.,16.,32.].逆转基因中的突变不会增加靠近Centromere的区域中的重组[3.].然而,增加间隙重组率可以减少有害基因负荷,这将有利于作物改良[17.].因此,我们应该了解提高近端区域重组率的机制和方法[3.].本研究中检测到的重组率的差异反映了119-535和535-18衍生自不同十字的序列组合物的差异。ND-Fish测定显示小麦染色体中串联重复组合物的差异,可用于预测重组率。
早些时候,Derrier等人利用3B染色体伪分子和高通量SNP检测绘制了252个间隔< 26 kb的交叉事件,这是普通小麦中的一个高分辨率交叉位置[16.].虽然高通量测序和DNA标记在研究染色体重组方面有很大优势[15.,16.,17.,18.,32.]需要测序大量的单独重组剂。细胞学标志物具有在研究染色体重组中的可视化和直觉的优点[33.,34.,35.].基于寡核苷酸探针ND-FISH技术可以用于建立丰富的鱼类小麦染色体核型方便[21.,36.,由串联重复序列衍生的寡聚探针可以显示小麦染色体的结构变异[22.,23.,36.,37.].此外,鱼核型显示的染色体重组是视觉理解,可以在小麦育种计划中方便地使用[34.,35.]. 因此,应将高通量测序和DNA标记与鱼类核型相结合,研究小麦染色体重组规律。
结论
本研究证实了串联重复序列对小麦减数分裂重组的影响,但还需要进一步研究决定5个al组减数分裂重组率的其他因素。我们的研究提供了一个基于ND-FISH的健壮的可视化工具来测量小麦减数分裂重组和交叉干扰。
方法
植物材料
四个小麦品种川迈39(CM39),Xikemai 8(XKM8),MiAN 1403(M1403)和MMIANMAI 43(MM43)和两种小麦线14 T105-1和14 T141-2用作父母进行杂交。进行四个交叉组合,CM39×M1403,CM39×MM43,CM39×XKM8和14 T105-1×14 T141-2,四个F.2几代人通过自行获得。使用小麦品种川迈36(CM36),川麦90(CM90)和KECHENGMAI 2(KCM2)测量单拷贝探针SC5A-479和SC5A-527在中期的5Al臂上的鱼类信号之间的距离.
Non-denaturing鱼(ND-FISH)
oligo-psc119.2-1,oligo-pta535-1 [24.],oligo-713,oligo-275.1 [22.]和Oligo-18 [21.]用作ND-Fish的寡核苷酸探针。这些有关这些寡头探针的信息列于附加文件中5.这些寡聚探针可以取代其原始序列在识别小麦染色体中的作用[22.,24.].Oligo-18探针序列与原始探针序列相同,均为18 bp长[21.].根据HAN等人描述的方法,由根尖制备中期染色体。(38.].根据TANG等人描述的方法进行ND-FISH。(21.].使用表面荧光显微镜(BX51, Olympus Corporation, Tokyo, Japan)和cellSens Dimension软件(Olympus Corporation, Tokyo, Japan)捕捉图像。
单拷贝鱼
串联重复查找器(TRF,版本4.09)[39.]程序用于过滤5A染色体(IWGSC Refseq版本2.0)中的串联重复。然后使用内部R包来过滤其他重复序列,包括转渣和转座子元件。使用剩余序列用作使用内部R包装的面包小麦品种中国弹簧(IWGSC Refseq版本2.0)的全长染色体5A对准。将其自身对齐的查询序列作为预备探针保持。Oligo-psc119.2-1,oligo-pta535-1的序列[24.]和Oligo-18 [21.]也用作使用B2DSC Web服务器中使用BLAST的5A染色体的全长序列对齐的查询(http://mcgb.uestc.edu.cn/b2dsc.)[40].Oligo-pTa535-1和Oligo-pSc119.2-1序列分别在5AL臂上的431-432 Mbp和507-508 Mbp位点上以高拷贝数靶向。这些方法用于缩小单拷贝探针的选择范围。最后选择两个单拷贝序列SC5A-479 (479272790 - 479275822 bp)和SC5A-527 (527288389 - 527289202 bp)作为探针。Oligo-18序列在5AL臂的一个位点(584-585 Mbp)上以高拷贝数命中目标。然后选取单个拷贝序列SC5A-586 (586379936-586,380,903 bp)作为探针。利用引物SC5A-479 (5'TCGTTGACTAGAAAGACGTG TGT3 ', 5'ACGCCTGTGTTAAGTTAAGTGAC3 ')、SC5A-527 (5'TGCGTACATAGGGTGAGTGTATG3 ', 5'GGCCTCTGGAAGAACGTTTTAT3 ')和SC5A-586 (5'TTGCTCGTGTCCACCATTGA3 ', 5'TGTGGAATACTTACCGCGCA3 ')对3个单拷贝探针序列进行扩增。根据Han等人描述的方法,将目的序列克隆到TSINGKE pClone007载体(TSINGKE,成都,中国)中,并用Texas-Red-5-dUTP (PerkinElmer,美国)标记。38.].制备根末端中染色体染色体,并如HAN等人所述进行杂交。(38.略作修改。探针混合物中每个探针含有5-6 ng/μL和1 × ENZO缓冲液(ENZO Life Science Inc.)。美国)。载玻片在含0.1% NP-40洗涤剂(Solarbio Life Sciences Ltd.)的2 × SSC缓冲液中洗涤。(中国)在45-50°C下放置3-5分钟。使用表面荧光显微镜(BX51, Olympus Corporation, Tokyo, Japan)和cellSens Dimension软件(Olympus Corporation, Tokyo, Japan)捕捉图像。
重组率和交叉干扰的计算
基于探针寡核糖-PSC119.2-1,oligo-pta535-1,oligo-275.1和oligo-18的鱼类测定重组。重组率以Cm / Mb表示。在该研究中,以两种间隔观察到重组:寡聚-PSC119.2-1和寡核糖-PTA535-1的119-535间隔在寡核糖-PTA535-1和Oligo-18 / oligo之间的535-18间隔之间-275.1信号网站。根据Ziolkowski等人描述的方法计算重组率和交叉干扰。(27.]使用以下公式:119-535 cm =在119-535中重组的5AL臂的数量/总数的5Al臂×100,535-18cm = 535-18个间隔/总数中的535-18cm = 5次臂的数量。5Al臂×100,重组率119-535间隔= 119-535厘米/ 48 MB×100,重组率在535-18间隔= 535-18cm / 59 MB×100,观察到双交叉(OBS DCO)= number of 5AL arms with recombination in both 119–535 and 535–18 intervals/total number of 5AL arms × 100, Expected double crossover (Exp DCO) = (119–535 cM/100) × (535–18 cM/100) × total number of 5AL arms, Coefficient of Coincidence (CoC) = Obs DCO/Exp DCO, and Crossover interference = 1 − CoC. Further,t分别进行检验和卡方检验,以确定重组率和交叉干扰的显著差异,如Girard等[4].接近零的干扰值表示干扰,靠近1的值表示无干扰。
SC5A-479和SC5A-527探针在中期染色体上的长度测量
两个单拷贝探针SC5A-479和SC5A-527的信号之间的中期5AL段(MSL)的长度进行测量。测定四种中期5AL段:段与寡pSc119.2-1信号(名为5AL119.),具有寡聚PTA535-1信号的段(名为5Al535),与两个探针的信号段(名为5AL119 + 535.)和没有两个探针信号的段(命名为5al不).这四种片段是由四个F衍生而来的2小麦品种CM36、CM90、XKM8和KCM2。将SC5A-479和SC5A-527 (MSL)之间的中期5AL段长度确定为相对中期长度(RML),以避免染色体凝聚引起的误差。RML = MSL/WL,其中WL代表5AL臂从着丝粒中心点到远端的整个长度。MSL和WL采用cellSens Dimension软件(Olympus Corporation, Tokyo, Japan)进行测量。采用GraphPad Prism软件(Version 5)进行单因素方差分析,进行组间两两比较。图表也使用GraphPad Prism软件绘制。
数据和材料的可用性
在当前研究中使用和/或分析的材料可从通信作者在合理的要求。
缩写
- 益生元:
-
寡核苷酸
- ND-FISH:
-
非变性荧光原位杂交
- OBS DCO:
-
观察双交叉
- EXP DCO:
-
预期的双交叉
- COC:
-
并发系数
- MSL:
-
中期段的长度
- RML:
-
相对中期长度
- 王:
-
全长5AL臂
工具书类
- 1。
王Y,Copenhaver GP。减数分裂重组:将其混合在植物中。Annu Rev植物Biol。2018; 69(1):577-609。https://doi.org/10.1146/annurev-arplant-042817-040431.
- 2。
Blary A,Jenczewski。植物育种交叉频率和分布的操纵。Al Appl Genet。2019; 132(3):575-92。https://doi.org/10.1007/S00122-018-3240-1..
- 3.
作者简介:米尤莱特,男,四川人,博士,主要从事生物医学工程研究。作物减数分裂的释放。Nat植物。2018;4(12):1010 - 6。https://doi.org/10.1038/s41477-018-0311-x.
- 4。
Girard C,Chelysheva L,Choinard S,Froger N,Macaisne N,Lemhemdi A等。AAA-ATPase FIDGETIN 1和Helicase FANCM通过独特机制拮抗减数分裂的十字架。Plos Genet。2015; 11(7):E1005369。https://doi.org/10.1371/journal.pgen.1005369.
- 5。
Fernandes JB,Séguéla-Arnaud M,LarchevêqueC,Lloyd啊,Mercier R.在杂交植物中释放出释放的十手队。Proc Natl Acad Sci U S A. 2018; 115(10):2431-6。https://doi.org/10.1073/pnas.1713078114.
- 6。
Kong A,Gudbjartsson DF,Sainz J,Jonsdottir Gm,Gudjonsson SA,Richardsson B等人。人类基因组的高分辨率重组图。NAT Genet。2002; 31(3):241-7。https://doi.org/10.1038/ng917.
- 7。
陈春芳,陈春芳,陈春芳,等。比较大鼠、小鼠和人类基因组的重组率。基因组研究》2004;14(4):528 - 38。https://doi.org/10.1101/gr.1970304.
- 8。
Shilo S, Melamed-Bessudo C, Dorone Y, Barkai N, Levy AA。与表观遗传修饰相关的DNA交叉基序描绘了染色质的开放区域拟南芥.植物细胞。2015; 27(9):2427-36。https://doi.org/10.1105/tpc.15.00391.
- 9。
Melamed Bessudo C,Levy AA。DNA甲基化缺陷会增加细胞中常染色区而非异色区的减数分裂交叉率拟南芥.Proc Natl Acad Sci U S A. 2012; 109(16):E981-8。https://doi.org/10.1073/pnas.1120742109.
- 10。
Ellermeier C,Higuchi Ec,Phadnis N,Holm L,Geelhood JL,Thon G等。RNAi和异铬胺抑制了焦化的减数分裂重组。Proc Natl Acad Sci U S A. 2010; 107(19):8701-5。https://doi.org/10.1073/pnas.0914160107.
- 11.
Nambiar M,Smith Gr。浓度抵制焦化区中有害的减肥重组。Semin Cell Dev Biol。2016; 54:188-97。https://doi.org/10.1016/j.semcdb.2016.01.042.
- 12.
Conswwood CJ,Choi K,Lambing C,Zhao X,Serra H,Borges F等人。近距离减数分裂重组的表观遗传活化拟南芥蒂利亚纳着丝粒通过H3K9me2缺失和非cg DNA甲基化。基因组研究》2018年;28(4):519 - 31所示。https://doi.org/10.1101/gr227116.117.
- 13。
Acquaviva L,Boekhout M,Karasu Me,Brick K,Pratto F,Li T,等。确保小鼠假染色体区域的减数分裂DNA断裂形成。自然。2020; 582(7812):426-31。https://doi.org/10.1038/s41586-020-2327-4.
- 14。
Erayman M,Sandhu D,Sidhu D,Dilbirligi M,Baenziger PS,Gill Ks。划分小麦基因组的富含基因的区域。核酸RES。2004; 32(12):3546-65。https://doi.org/10.1093/nar/gkh639..
- 15.
作者简介:陈志强,男,四川人,博士,主要研究方向为光化学。3 mb面包小麦杂交率的变化(小麦)揭示了减肥重组热点的存在。染色体瘤。2011; 120(2):185-98。https://doi.org/10.1007/S00412-010-0302-9.
- 16。
张志强,张志强,张志强,等。对六倍体小麦基因组交叉事件的高分辨率定位表明了一种普遍的重组机制。遗传学。2017;206(3):1373 - 88。https://doi.org/10.1534/Genetics.116.196014.
- 17。
王胜,何飞,赵胜,李永华,等。巢式关联作图揭示异源多倍体小麦全基因组重组率变异的遗传结构。植物j . 2018; 95(6): 1039 - 54。https://doi.org/10.1111/tpj.14009.
- 18。
加德纳LJ,WINGEN LU,贝利P,Joynson R,Brabbs T,赖特J,等。六倍体普通小麦的重组景观的分析揭示基因控制重组和基因转换频率。基因组Biol。2019; 20(1):69。https://doi.org/10.1186/s13059-019-1675-6.
- 19。
罕有的C,富兰克林CH,王CJR。理解和操纵植物中的减数分裂重组。植物理性。2017; 173(3):1530-42。https://doi.org/10.1104/pp.16.01530.
- 20。
Komuro S,Endo R,Shikata k,Kato A.在原位杂交过程中荧光揭示的小麦中各种重复DNA序列的基因组和染色体分布模式。基因组。2013; 56(3):131-7。https://doi.org/10.1139/gen-2013-0003.
- 21。
唐S,Tang Z,秋L,杨Z,Li G,Lang T等人。开发新的寡核苷酸探针,以区分特定的染色体段和A,B,D小麦的D基因组(小麦L.)使用ND-Fish。正面。植物SCI。2018; 9:1104。
- 22。
关键词:大麦,染色体,ND-FISH,寡核苷酸,多态性基因(巴塞尔)。2016; 7(12): 118。https://doi.org/10.3390/genes7120118.
- 23。
蒋M,萧ZQ,傅SL,唐ZX。用寡核苷酸探针显示85个普通小麦品种/品系的FISH核型。谷物公社。2017;45(4):549–63.
- 24。
唐ZX,杨ZJ,FU SL。寡核苷酸取代重复序列PAS1,PSC119.2,PTA-535,PTA71,CCS1和PAWRC.1用于鱼类分析的作用。J Appl Genet。2014; 55(3):313-8。https://doi.org/10.1007/s13353-014-0215-z.
- 25。
赵海涛,朱旭,曾志,等。减数分裂杂交与开放染色质有关,并富含偷渡者马铃薯的转座子。基因组生物学。2017;18(1):203.https://doi.org/10.1186/s13059-017-1326-8.
- 26。
布莱克韦尔AR,DluzewskaĴ,Szymanska-Lejman男,德贾斯丁S,笃AJ,Kbiri N,等人。MSH2形状相对于减数分裂交叉景观在interhomolog多态性拟南芥.EMBO j . 2020; 39 (21): e104858。https://doi.org/10.15252/embj.2020104858.
- 27。
赵旭东,张志强,张志强,等。杂合子和纯合子区域的并置通过干扰导致交互交叉重构拟南芥减数分裂。Elife。2015; 4:E03708。https://doi.org/10.7554/eLife.03708.
- 28。
Feliciello I, Akrap I, Brajković J, Zlatar I, Ugarković Đ。卫星DNA是红面粉甲虫种群差异的驱动因素赤拟谷盗.基因组生物学进展。2014;71(1):228-39。
- 29。
Pezer Ž, Brajković J, Feliciello I, Ugarkovć Đ。卫星dna介导的基因组调控效应。基因组达因。2012;7:153 - 69。https://doi.org/10.1159/000337116.
- 30.
Fitz James MH,Tong P,Pidoux AL,Ozadam H,Yang L,White SA等。异染色质的大区域直接形成短的有丝分裂染色体环。埃利夫。2020;9:e57212。https://doi.org/10.7554/eLife.57212.
- 31。
陈志强,陈志强,陈志强,等。真核生物染色体规模交叉率变异的meta分析及其在进化基因组学中的意义。摩尔生态。2018;27(11):2477 - 97。https://doi.org/10.1111/mec.14699.
- 32。
染色体3B重组的详细研究为小麦的交叉分布提供了新的见解(小麦l .)。遗传学。2009;181(2):393 - 403。https://doi.org/10.1534/genetics.108.097469.
- 33。
Lukaszewski AJ,柯蒂斯CA.在四倍体小麦的B-组染色体重组的物理分布。Al Appl Genet。1993; 86(1):121-7。https://doi.org/10.1007/BF00223816.
- 34。
杜H,Li S,He Y,Tang Z,Fu S,Ren Y. Mianmai 37及其衍生物的6Vs / 6al易位染色体的结构变化。J Triticeae作物。2019; 39(6):659-65。
- 35。
席伟,蒋军,唐智,王胜。南麦品种染色体结构变异研究。麦类作物学报,2019;39(5):567-73。
- 36。
黄X,朱米,庄L,张某,王俊,陈X等。高分辨率多重寡核苷酸鱼类中小麦品种中鉴定的结构染色体重排和多态性。Al Appl Genet。2018; 131(9):1967-86。https://doi.org/10.1007/s00122-018-3126-2.
- 37。
郭军,高东,龚伟,李华,李军,李国光,等。荧光原位杂交显示普通小麦品系的遗传多样性。植物生态学报。2019;305(3):247-54。https://doi.org/10.1007/s00606-019-1567-y.
- 38。
韩FP,LAMB JC,Birchler A.高频率的Centromere失活,导致玉米的稳定的Dicentric染色体。Proc Natl Acad Sci U S A. 2006; 103(9):3238-43。https://doi.org/10.1073/pnas.0509650103.
- 39。
Benson G. Tandem重复发现:分析DNA序列的程序。核酸RES。1999年; 27(2):573-80。https://doi.org/10.1093/nar/27.2.573.
- 40.
朗T,李G,王H, Yu Z,陈问杨E, et al。小麦基因组串联重复序列的物理定位及其染色体鉴定的应用。足底。2019;249(3):663 - 75。https://doi.org/10.1007/s00425-018-3033-4.
致谢
我们感谢杨恩年(中国四川省农业科学院作物研究所)和任勇(中国绵阳市农业科学院国家小麦改良中心绵阳分院)为我们提供了CM39、CM36、CM90、KCM2、M1403、MM43和XKM8的种子。
资金
本手稿由中国四川科技项目提供(第2020亿号)。资助机构在研究设计,数据收集,分析和解释中没有作用,以及手稿写作。
作者信息
隶属关系
贡献
SF和ZT设计了这项研究并写了稿件。SF,ZT和JL开发了F.2几代人。YZ进行了ND-Fish和单拷贝鱼类测定并分析了数据。LW开发了单拷贝探针并进行了单拷贝鱼类测定。所有作者都读过并批准了稿件。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
F的重组类型的5AL臂2后代衍生自CM39×M1403。'5A-REC1'和'5A-REC2'表示通过在535-18间隔中重组形成的5AL臂。“5A-REC3”和“5A-REC4”表示通过重组在119-535间隔形成的5AL臂。'5A-DREC1'和'5A-DREC2'表示通过在535-18和119-535间隔中重组形成的5AL臂。染色体用DAPI(蓝色)复染。比例尺:10 μm。
附加文件2:图S2。
F的重组类型的5AL臂2后代来自CM39×MM43。'5A-REC1'和'5A-REC2'表示通过在535-18间隔中重组形成的5AL臂。“5A-REC3”和“5A-REC4”表示通过重组在119-535间隔形成的5AL臂。'5A-DREC1'和'5A-DREC2'表示通过在535-18和119-535间隔中重组形成的5AL臂。'5A'表示衍生自CM39的5A染色体。染色体用DAPI(蓝色)复染。比例尺:10 μm。
附加文件3:图S3。
F的重组类型的5AL臂2progeny derived from CM39 × XKM8 and 14 T105–1 × 14 T141–2. ‘5A-Rec3’ and ‘5A-Rec4’ indicate the 5AL arms formed by recombination in the 119–535 interval. ‘5A’ in (a) and (d) indicates the 5A chromosomes derived from XKM8 and 14 T141–2. ‘5A’ in (b) and (c) indicates the 5A chromosomes derived from CM39 and 14 T105–1. Chromosomes were counterstained with DAPI (blue). Scale bar: 10 μm.
附加文件4:图S4。
顺序单拷贝鱼和Nd-Fish测定以确定SC5A-479,SC5A-527,SC5A-586,Oligo-PTA119.2-1,oligo-Pta535-1和寡核糖-18探针的位置的位置。(a, b, c)Cells derived from CM39 × MM43 showing the signal sites of SC5A479 and SC5A527 close to those of Oligo-pSc119.2–1 and Oligo-pTa535–1, respectively, and 5AL119.和5al.535段对应于SC5A479和SC5A527信号网站之间的119-535间隔。(d, e, f)来自CM39×MM1403的细胞显示为5Al119 + 535.和5al.不段对应于SC5A479和SC5A527信号网站之间的119-535间隔。(g,h,我)表示KCM2 5AL的细胞119 + 535.SC5A479和SC5A527网站之间的段。(j,k,l) CM39 × MM43细胞显示Oligo-18和SC5A-586的信号位点相近。染色体用DAPI(蓝色)复染。比例尺:10 μm。
附加文件5:表S1。
本研究中用于ND-Fish测定的寡核苷酸探针。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条中提供的数据,除非数据信用额度中另有规定。
关于这篇文章
引用这篇文章
邹勇,万丽,罗娇。et al。FISH地标反映减数分裂重组和在小麦染色体的结构改变(小麦l .)。BMC植物生物学21,167(2021)。https://doi.org/10.1186/s12870-021-02947-1
已收到:
接受:
发表:
关键字
- 小麦
- 串联重复
- 减数分裂重组
- 染色体缩合