摘要
背景
的杜鹃sanguineum它是中国云南西北部和西藏东南部高山特有的。在这个复杂的品种显示独特的花朵颜色,甚至在花蕾阶段。然而,花色变化的分子调控机制还没有很好地阐明。在这里,我们通过测量花的反射率剖面和比较转录组分析,对三个共存的品种进行了研究r . sanguineum复杂的,花分别为黄红粉红色、亮深红色和深黑深红色。我们利用RNA-seq和qRT-PCR数据比较了花青素/类黄酮生物合成途径差异表达基因(DEGs)的表达水平。我们基于转录组衍生的单核苷酸多态性(SNPs)数据进行聚类分析,最后分析了DEGs的启动子结构。
结果
在400 ~ 700 nm范围内,三种颜色形态的反射光谱变化明显,在饱和度、亮度、色相和饱和度/色相比(花青素含量的间接测量指标)上存在明显差异。我们鉴定了三个品种共有的15164个正位类群。SNP聚类分析表明,这些品种并非单系。共有40个同源基因编码12种酶参与了花色多态性。这些花青素生物合成相关基因与花青素的合成、修饰和运输特性相关(RsCHS,RsCHI,RsF3H,RsF3′H,RsFLS,RsANS,RsAT,RsOMT,RsGST),以及参与分解代谢和降解的基因(RsBGLU,负责,RsCAD).序列和序列的变化独联体这些基因的作用元件可能与花青素的积累有关,从而导致了花青素的分化r . sanguineum复杂。
结论
结果表明,该品种亲缘关系非常密切,花色变异也非常明显r . sanguineum复合体与参与花青素合成和分解合成网络的基因的不同表达水平密切相关。我们的研究提供了一个复杂的场景,包括花的颜色遗传机制之间的关系,伴随基因流动的品种,这可能代表了传粉者介导的早期同域物种形成的一个早期案例。
背景
数百年来,植物学家、生态学家和园艺学家都为花卉颜色的显著多样性,尤其是野生植物的多样性而着迷。1,2,3.].花器官的着色是开花植物的一个显著特征,也是被子植物辐射的一个显著特征[4,5].花朵颜色多样性被认为是主要与传粉者(如昆虫、鸟类)和动物有关的种子传播的关键适应性特征之一[6,7].此外,花朵颜色表型是植物分类学家用来分类的一个重要特征。然而,花色在进化上似乎是最不稳定的性状之一,甚至在同一物种的种群中也是如此。7,8].
花朵的细胞化合物有助于颜色的轮廓和视觉上被人类感知,通常被称为“色素”。一类属于类黄酮的次生代谢物是植物着色色素的主要决定因素,其中花青素负责花、叶、果实、种子和其他组织中从红橙到红、从紫到紫的色素[9,10].花青素是花卉着色的主要化合物,存在于90%以上的被子植物中[11].导致花青素积累的类黄酮生物合成途径具有高度的保守性和良好的特征,在许多物种中得到了广泛的研究,其中大多数是模式植物或具有重要农业和园艺价值的植物[12,13,14,15].很少有研究探讨野生物种花青素形成和积累的分子基础[16,17].基于这些研究,提出了涉及花青素积累的三个主要相关因素,包括发生在细胞核中的转录调节基因(MYB-bHLH-WD40复合体)、结构基因(CHS,FLS的,DFR,答)作用于生物合成途径,以及转运基因(销售税)将花青素从胞浆转移到液泡[10,18,19].这些基因的表达也可能受到序列和自然变异的影响独联体-启动子区域的调控元件以及表观遗传修饰(如DNA甲基化)[18,20.].此外,黄酮醇对花青素的共色素作用可以稳定和增强花的颜色,这种作用被观察到为深色素效应,即花青素含量的强度得到加强[21].例如,DFR基因和FLS的基因可以通过两个主要分支竞争导致产生不同花青素和黄烷醇的底物[22,23],从而导致共色素沉着。与生物合成途径相比,植物中花青素分解代谢的知识有限。一些分解代谢基因BGLU而且每已经被证明是导致花青素降解的原因[24,25].然而,调节花青素合成的分子机制已被证明在不同的植物种类中存在差异,从而导致花青素的结构多样性,因为生物合成途径是通过调节网络受到多个因素的调节[26].
颜色是可见光谱范围内的一种电磁辐射形式。色素反射的波长决定了花的颜色[27].颜色可以根据亮度(信号的强度,B)、饱和度(颜色的纯度,S)和色相(颜色的光谱描述符,H)来定义和分类,这些特征通常用于区分颜色[27,28].亮度是指由花青素含量决定的颜色强度[29,30.],不同颜色成分组合如B/H、S/H也与花青素含量显著相关[31].刘等人。[32]提出颜色亮度随花青素含量的增加而降低。研究还表明,饱和度/色相比(S/H)与花青素含量之间存在相关性[31].利用这些参数,可以快速、无损地测定花青素含量。
常绿杜鹃花(杜鹃),花青素和黄烷醇是花色素的主要成分,尤其是花青素成分(即花青素、飞燕草苷、麦草苷、天竺葵苷、芍药苷、芍药苷)的组成,其数量决定了花的颜色从浅粉色到紫色不等[11,33].一些研究已经报道了这一点r . kiusianum紫色的花含有花青素和飞燕草素的衍生物,而红色的花r . kaempferi只含蓝靛素衍生物[34].勒迈特等人[35,36)研究艾丽卡种,属于同一科的鸢尾科杜鹃,利用qRT-PCR和UPLC-MS,揭示了红色或粉色花与白色或黄色花的花色转移的花青素遗传网络,发现单通路基因表达丢失,或通路基因中某一转录因子表达丢失或功能突变丢失导致整个通路的失效,从而导致显著的花色转移。
在此,我们利用高度的颜色多态性研究了花着色的遗传基础杜鹃sanguineum复杂。复杂的(R.subgen。Hymenanthes)包括花由黄至粉或由深红色至暗红色的植物,它们主要根据花的颜色差异被分为六个品种[37].该建筑群的成员基本上位于与积雪有关的高海拔地区(> 3000米)[37].它们是全球生物多样性热点之一的云南西北部和西藏东南部的特有物种[38].这一地区也被认为是多样化和差异化的中心之一杜鹃(37,39].本属的花朵颜色多态性传统上被视为一种生态适应性特征,对吸引特定的传粉者至关重要[40,41,42],亦可能是对环境变化的反应,例如不同海拔、温度和土壤条件下的紫外线辐射[32].虽然有研究发表了花青素的成分和含量杜鹃花,大部分专门用于用薄层色谱法(TLC)和高效液相色谱法(HPLC)鉴定一些野生和栽培杜鹃花花瓣中的色素成分[11,33].到目前为止,还没有研究集中在植物的种下颜色多态性的分子机制杜鹃.研究密切相关的实体,如物种复合体,具有相当同质的遗传背景的优势,花的颜色基因不同,同质性的情况是有限的。以前的研究主要集中在单一物种不同发育阶段的颜色变化[14,18],或涵盖若干相关物种[26,35].
在本研究中,我们将转录组测序(RNA-seq)和基因组重测序与反射光谱分析相结合,以阐明三种不同颜色的天然品种之间的分子和花青素含量的差异r . sanguineum繁花,黄红粉红至深黑深红色。本研究旨在研究花青素/类黄酮生物合成途径候选基因表达与花种下花色变异的相关性。我们的研究结果可能为花朵颜色变异表达的遗传机制提出假说,并为植物中传粉者介导的早期同域物种形成提供了一个代表性案例r . sanguineum复杂。此外,这是第一个比较非模式物种在自然系统中的转录组谱的研究杜鹃.
结果
反射光谱和颜色形态的差异
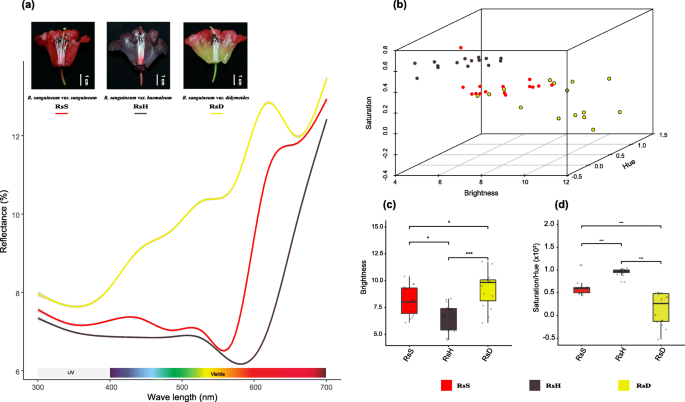
采样的个体可以分为三种颜色类别代表r . sanguineumvar。sanguineum(RsS鲜红的花朵。1d),R。var。haemaleum(RsH那里的花是深黑深红色的。1e),R。var。didymoides(标准偏差粉红色的花管和红色的花瓣。1f).三种颜色变体的反射率光谱在波长范围内变化明显,特别是在400 ~ 620 nm之间(图5)。2一个)。RsS红光谱(约620 nm)有一个离散峰RsH在更远的红色范围(> 700nm)有一个峰。的反射标准偏差在蓝到黄光谱范围内增加,并在橙红色范围内达到峰值(~ 600 nm)(图5)。2a)三个品种的花色在亮度、饱和度和色相上有显著差异。标准偏差亮度值最高的是RsS,RsH与最小值,而RsH饱和度最高,是吗RsS与标准偏差(图的最低水平。2b, c).色相值在样品中最高标准偏差,然后RsS而且RsH(无花果。2b).花青素的饱和度和色相的比值(花青素含量的间接测量)RsH是最高的与那些标准偏差而且RsS样品(图。2d).的比值值标准偏差样品的差异比其他两个品种要大得多,这可能与它的双色花的颜色组成的变化有关(图。1).
三个品种的反射率光谱的比较杜鹃sanguineum复杂。一个在3个品种的花冠管中点处测定了其反射光谱。b可见光谱的三维图,按亮度(x轴),色调(y轴)和饱和度(z轴)分类的三个品种。c三个品种的反射率光谱亮度值箱线图。d三个品种的反射光谱中饱和度与色相的比值的箱线图。箱形图表示中位数,盒边表示每个品种15个个体的值的第25和第75个百分位。统计显著性由双尾学生确定t测试。显著差异用星号(*)表示P< 0.05;**P< 0.01;***P< 0.001)。红色代表R.var。sanguineum(RsS);紫色代表R.var。haemaleum(RsH);黄色代表R.var。didymoides(标准偏差)
转录组从头组装和质量评估
我们从两种组织(花芽和叶芽)中为三个个体分别测序了共18个RNA-seq文库r . sanguineum使用Illumina配对端测序的复合物。经过质量控制后,大约有6.558亿M (M)的清洁读取(约96千兆对,Gbp),库之间的读取数量非常一致,在32,111,674和43,353,842之间S1).3个品种的CORSET的contig N50长度相似,碱基对(bp)在903 ~ 1125之间,转录本数在117,976 ~ 171,725之间(表2)S1).将18个库的reads与品种特异性参考文献进行比对,比对率为84.25 ~ 94.85%,平均比对率分别为92.82、90.30和93.77%R。var。sanguineum,R。var。haemaleum而且R。var。didymoides分别(表S2).基于1614个保守的BUSCO胚胎植物同源序列,对其转录组完整性进行评估,确定了1540个(95.4%)完整和片段化的BUSCOR。var。sanguineum, 1548 (95.9%)R。var。haemaleum1538人(95.3%)R。var。didymoides(图S1一、表S3).因此,我们的结果表明,三个转录组组装良好,相对完整。
正交鉴别与功能注释
ORF预测发现,在最终的非冗余转录本中,共有53,207个蛋白质编码转录本R。var。sanguineum46754年,R。var。haemaleum和38548年R。var。didymoides.对于三个品种,OrthoVenn2 [43]在总共138,509个蛋白质编码转录本中鉴定出31,525个簇,其中16,361个是同源基因(包含至少两个物种),15,164个是单拷贝基因。有54,192个单例未包含在任何集群中(表S4).基于15,164个一对一的单拷贝正交关系的注释(表S53个品种中,我们在国家生物技术信息中心非冗余(NR)蛋白数据库中找到14441个(95.23%)匹配项,UniProt/Swiss-Prot中找到10648个(70.22%)匹配项,COG/KOG中找到13089个(86.32%)匹配项,eggNOG中找到14109个(93.04%)匹配项,基因本体(GO)中找到7445个(49.1%)匹配项,京都基因与基因组百科(KEGG)中找到4302个(28.37%)匹配项(图)S1b,图S2).
SNP检测和聚类分析
以15164个同源单拷贝基因为参考序列集,构建了该9个个体的cDNA文库r . sanguineum复合物共产生50853个snp。基于这个SNP数据集,snphyo树和PCA分析都没有将样本聚类到特定品种的聚类中(图S3).只有RsH样本落在SNPhylo树的一个分支中,但也有一个样本落在标准偏差(废话= 100%)。标准偏差而且RsS样品混合在两个枝中。
整体差异表达基因和功能富集
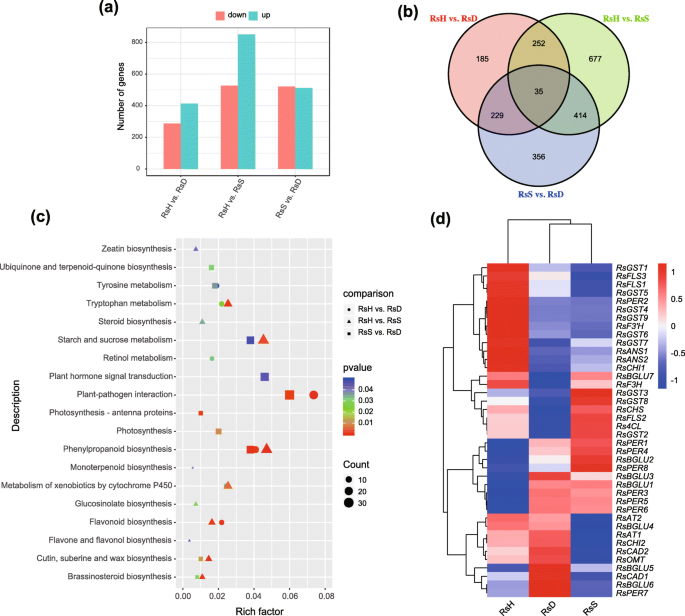
我们进行了三个成对的转录组比较,以确定在花的颜色变体中的DEGsr . sanguineum通过计算基因的FPKM值。在三向比较中共检测到2148个差异表达基因(DEGs)。特别是对比深黑深红色的变形RsH带着黄色的粉红色的变形标准偏差鉴定出701个DEGs(413个上调,288个下调)RsH);的比较RsH对比深黑深红色的变形RsS共发现1378个DEGs,其中851个表达上调,527个表达下调RsH);的比较RsS与标准偏差给出1034个DEGs(513个上调,521个下调)RsS)(图。3.a, b).差异表达基因最高RsS而且RsH.所有deg模式均在补充图中给出S4&S5.基于PCA和热图结果的基因表达谱(图S4;数字S6a),每个品种聚集在一起(显示种特异性模式),这表明RNA-seq文库是可靠的。
3个品种的RNA-seq分析结果杜鹃sanguineum复杂。富集和分层聚类显示之间的差异基因图谱RsHvs标准偏差,RsHvsRsS,RsSvs标准偏差.一个三个品种间两两比较差异表达基因的直方图。b差异表达基因的维恩图。每个圆大截面上的数字之和代表两两品种比较中差异表达的基因总数;圆圈的重叠部分代表了比较品种之间共享的差异表达基因。c差异表达基因的KEGG途径富集分析的散点图结果。着色的p-values表示富因子的显著性,取值范围为−1 ~ 1。丰富因子是在给定通路项中标注的DEG数与通路项中标注的所有基因数的比值,丰富因子值越大表示强度越大。KEGG数据库中丰富的三个比较的19个最常用路径术语列于图中。符号的大小表示deg的数量。d所有40个候选基因的归一化表达水平的层次聚类,显示不同的基因表达谱在配对比较r . sanguineum复杂。对花蕾样品进行了分析。红色代表高表达水平,蓝色代表低表达水平
所有的DEGs被划分为三个主要的基因本体类别:生物过程(BP)、细胞成分(CC)和分子功能(MF),其中GO术语大部分属于生物过程,其次是分子功能和细胞成分。三个两两比较结果显示,共有40个显著GO术语,包括27个BPs、8个CCs和5个mf(图5)S6b, c).来自不同比较的基因清楚地表明了相同的分子和细胞事件,如植物抗性(GO:0006952,防御反应)和代谢过程(GO:0009737,脱落酸反应;GO:0009753,茉莉酸反应)。KEGG通路富集分析结果显示,DEGs与多种代谢和生物合成通路相关:分别有20、17和29个通路富集,分别对应701、1378和1034个DEGsRsHvs。标准偏差,RsHvs。RsS,RsSvs。标准偏差,分别。特别是编码苯丙烷类生物合成酶的基因(ko00940)均显著富集。此外,与类黄酮生物合成(ko00941)和细胞色素P450 (ko00980)代谢异种生物相关的途径也被丰富RsHvs。标准偏差而且RsHvs。RsS.图中显示了一些代表性的最显著富集的KEGG通路。3.c.为了更深入地了解所有deg的表达模式,使用TMM归一化表达值生成了一个热图(图1)S6A),这里我们关注的是与色素相关的基因。
与颜色多态性相关的候选基因r . sanguineum复杂的
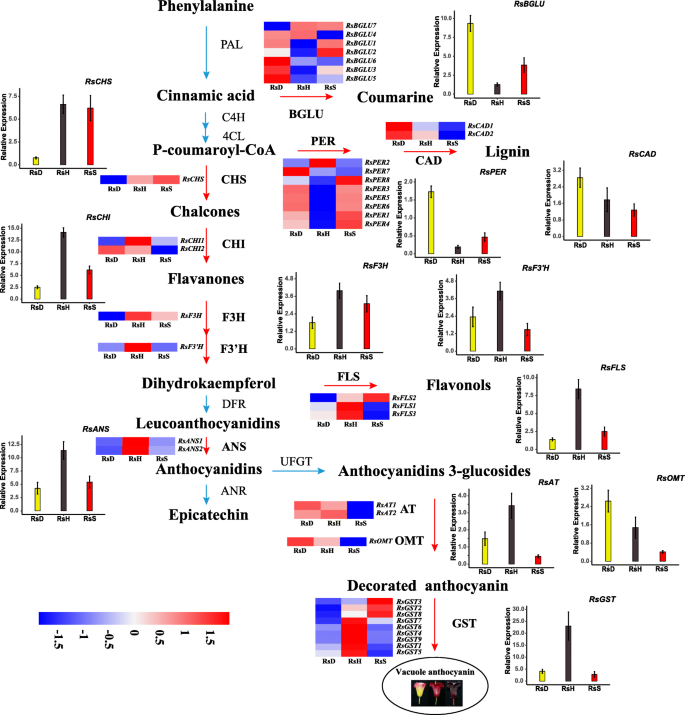
基于DEGs的KEGG途径富集的结果,并考虑去除极低表达的基因(FPKM < 1),我们从40个同源基因中确定了13个候选基因,这些基因可能与花的色素沉积相关,有助于该复合物中的花青素积累和/或共色素沉积(表1)S6).这些都是花青素相关基因,参与花青素生物合成、花青素修饰和花青素转运。其中包括花青素合酶(答)、酰基转移酶(在), beta-glucosidase (BGLU)、4-香豆酸辅酶a连接酶(4 cl)、肉桂醇脱氢酶(计算机辅助设计)、查尔酮异构酶(气)、查尔酮合酶(CHS)、黄酮3-羟化酶(F3H)、类黄酮3 ' -羟化酶(F3′H)、黄酮醇合酶(FLS的)、谷胱甘肽s -转移酶(销售税), O-methyltransferase (OMT)和过氧化物酶(每).在这其中,Rs4CL,RsCHS,RsF3H,RsF3'H而且RsOMT只有一本,其他的(RsANS,RsAT,RsBGLU,RsCAD,RsCHI,RsFLS,RsGST,负责)代表多基因家族有2个(例如:RsAT,RsCAD,RsCHI)至8或9份(例如:RsBGLU,RsGST,负责).对这些基因序列进行两两比较,鉴定出23个DEGs, 25个DEGs和12个DEGsRsHvs。标准偏差,RsHvs。RsS而且RsSvs。标准偏差分别(无花果。3.d;数据S7,S8,S9).获得了13个候选基因的代表性拷贝的qRT-PCR验证候选基因的可靠性表达谱,除Rs4CL未能放大(表S7).
如图所示。4,结果表明,绝大多数花青素合成、修饰和转移酶的基因在中表达较高RsH,在标准偏差而且RsS,分配给水解酶的基因下调RsH.两个类群共有6种酶共有9个基因(RsHvs。标准偏差而且RsHvs。RsS),包括RsBGLU1/3,RsPER1/3/4,RsCHI1,RsFLS1,RsANS2而且RsAT2(无花果。3.d;数字S7,S8).然而,只有少数的deg,包括Rs4CL,RsBGLU7而且RsPER1表现出更高的表达RsS在标准偏差.此外,在标准偏差,RsBGLU4/5,RsPER5/7/8,RsCAD1/2,RsAT1,RsOMT的表达高于RsS(无花果。3.d;无花果。4;数字S9).假设花青素生物合成途径与花的颜色有关的初步示意图r . sanguineum如图所示。4.
与花色素沉着相关的黄酮途径示意图杜鹃sanguineum复杂。酶首字母缩略词,表达模式和qRT-PCR结果显示在每一个代谢步骤和合成方向用红色箭头标记。每个基因的RNA-seq表达模式在热图中显示。色标表示log2转换的FPKM(每千碱基外显子每百万映射读取的片段)值。红色代表高表达,蓝色代表低表达。RsS,RsH,标准偏差代表r . sanguineumvar。sanguineum,R。var。haemaleum,R。var。didymoides,分别。三个品种花青素基因的qRT-PCR表达结果用柱状图表示,并以三次生物重复的平均值表示,误差为标准误差。对花蕾样品进行了分析。相对mRNA (y轴)表达量归一化为GAPDH(FN552706)
花青素通路相关基因启动子区序列多样性
每个基因的启动子序列(翻译起始位点上游2 kb)在序列相似性(98.6 - 99.7%)和GC含量上都表现出高度的保守性,尽管在某些位置上存在差异(表2)S8;数字S10).的详细分析顺式以MYB转录结合位点为靶点的这些启动子的作用元件显示,除了RsANS而且RsF3'H在美国,其他11个候选基因都至少在一个基因上有变异顺式图案(图S10).这些启动子在其他类型中也有变异顺式主题(表S8).因此,这些数据提供了启动子区域(序列的变异和独联体-作用元件)和各自花青素基因的差异表达。
讨论
类黄酮/花青素代谢被认为是促进花色素形成的最重要途径之一,由一种多酶复合物催化[10].花卉颜色的遗传基础最近在物种水平上得到了研究艾丽卡,与…密切相关的属杜鹃在杜鹃花科(35,36].这些研究表明,在颜色变化的基础上,单通路基因的表达丢失,或转录因子丢失或功能突变丢失导致的通路崩溃[35].虽然这些研究中的颜色变化涉及物种之间的变异,但在这项研究中,我们努力阐明更密切相关的进化实体之间的颜色变化的遗传学,在不同的物种之间r . sanguineum复杂。这些变化可能更加微妙,因为进化距离比物种之间的距离更小,可能更不受同形性的影响,反映了更准确的进化路径变化。之前的研究的一个缺点是缺乏转录组数据,无法检测到基因同源谬误[35,36].事实上,在目前的研究中,我们发现许多花青素基因是小基因家族的一部分(图。3.D),因此更精确地解开花青素途径。
通过比较转录组和确证qRT-PCR实验,我们发现在具有相似反射谱差异特征的类群中,差异表达基因的模式相似。RsHvs。标准偏差而且RsHvs。RsS.因此,当比较两个反射率光谱更相似的类群时,发现的deg数量更少,RsSvs。标准偏差.因此,密切相关的类群之间表型差异(即花青素含量差异)的大小可能是各自花组织中颜色基因表达水平变化的结果。在这里,我们确定了两个类群中共有的6种酶的9个基因,RsHvs。标准偏差而且RsHvs。RsS(图S7,S8).在我们的反射光谱结果中,亮度参数(B)(通常指强度)是最低的,饱和度(S)和色相(H)的比值是最高的RsH(无花果。2C, d),表明花青素含量高[29,31].同样,之前的研究表明,花的颜色强度在很大程度上是由花青素的数量决定的。44,45],即花青素含量越高,花色越深。
高水平的表达会使颜色更加鲜艳。46我们发现了不同的基因,可能促成了深黑深红色的花R。var.haemalum(RsH).我们的研究结果表明RsCHI1,RsFLS1,RsANS2而且RsAT2在类群比较中高度表达RsH在该品种中显著表达,对应其深黑深红色的花(图。3.d;无花果。4).这一发现与蓝莓果实发育的情况类似,在蓝莓果实发育过程中发现了通路基因表达与花青素产生之间的相关性[46].在类黄酮/花青素途径中,气高度表达的RsH,被证明可以将查尔酮转化为黄酮。的答基因,也只在RsH,在催化无色白花青素合成为彩色花青素的过程中起着关键作用。例如,张等人。[47)报道,气在生菜的红叶中表达量显著高于在浅色叶中表达量(摘要以).赵等人。[48的低表达答的芍药导致花青素积累减少,导致颜色变黄。在我们的研究中,RsANS可能是花青素积累较弱的原因标准偏差,而高积累在RsH在后者达到相对较高的花瓣色彩强度。一般来说,花青素的结构由色素积累而成答催化是不稳定的,通过花青素酰基转移酶(在)形成稳定的花青素[10].据报道,当在被抑制,花瓣变得苍白[49].这在我们的结果中得到了证明RsAT基因在RsH亮度低(花色强度高),其他两个花冠较浅的品种亮度较低。有趣的是,FLS的基因在RsH.FLS的竞争相同的衬底DFR,从而产生不同的类黄酮和花青素[10,49],表明共色素作用[50].由于共色素沉淀而引起的颜色变化增加了花的颜色强度[51].在我们的研究结果中,辅色素沉着,通常与黄酮醇有关RsFLS,可能导致强烈的颜色花青素,使颜色转变为深黑深红色。这与研究的红紫色花瓣是一致的r . simsii花(52].
在比较RsSvs。标准偏差对比转录组分析表明,与花色生物合成相关的6个基因中只有12个片段存在差异表达(图1)S9).这很可能是相对较小的光谱反射差(即花色差)之间的差异RsS而且标准偏差,这就是与花青素生物合成相关的DEGs数量较低的原因。高表达的六个基因中的三个标准偏差,即RsBGLU,RsCAD,负责它们属于分解代谢酶,因此可能是颜色较浅的原因。我们的发现与Yang等人之前的研究一致。[53他对两个牡丹品种的花色变化进行了研究,结果表明,负责花色从红色变为橙色和黄色的花青素含量急剧下降。同样的,朱等人。[54的差异表达NnOMTs可能与花瓣的颜色差异有关吧莲属椰子具有黄色和白色花的栽培品种。但这些可能与Le Maitre等人的发现不同。[35,36]艾丽卡开黄色花的种。对于这些,他们发现要么基因表达正常,要么基因表达缺失F3′HMYB识别元件的缺失。其他色素,如类胡萝卜素也会影响花的颜色[10].然而,这些在植物中很少被检测到杜鹃(11,33].其他基因,比如RsCHS,RsF3H,RsF3′H而且RsGST也被发现在RsH与两者相比标准偏差或RsS.这些是单拷贝基因,很可能解释了这个品种的深色花朵。
我们的结果进一步表明RsBGLU1/3,RsPER1/3/4与代谢酶相关的基因显著升高标准偏差和更高的RsS相比RsH.在类黄酮/花青素途径(图。4),这两个基因被划分为与苯丙类早期生物合成途径相关的“早期生物合成基因”(egs)。它们分别参与了肉桂酸和p -香豆素酰辅酶a水解为香豆素和木质素[13].β-glucosidases (BGLU)及过氧化物酶(每)已经被证明是许多植物花青素降解的原因[24,25].一般来说,花青素的积累是由生物合成和降解的平衡所决定的。在我们的研究中,品种标准偏差而且RsS可能由于早期底物的分解代谢导致花青素积累减少[24]和黄酮/花青素通路中成熟花青素的降解,从而导致花的颜色强度低。很可能这些品种的花的颜色与通路中的通量转移(花青素含量)有关,从而导致了颜色强度的差异。
然而,上述所有候选基因都被归类为结构基因。MBW (MYB-bHLH-WD40)蛋白复合体基因表达差异不显著。一种可能的解释可能指向这里的采样方法,只使用一个阶段(花蕾后期)。杨等人。[19]表明,MBW转录因子在转录水平上共同调节花青素的积累,特别是在花着色的最初阶段,而可能不存在于花蕾后期。此外,MBW复合体可以与花青素基因的启动子结合,启动子序列的MYB结合位点在花青素合成中起着至关重要的作用[55].在本研究中,三个品种各基因的启动子序列总体上是保守的,但在基因序列上有明显的变异独联体-基序如MYB结合位点在11个基因中,除RsANS而且RsF3′H(表S8;数字S10).我们的结果也可能在某种程度上支持今晚的假设[56在那里,表型差异,特别是对于密切相关的物种,更有可能由独联体-调控区域比基因的蛋白质编码区域更明显,但它们被认为相对不受适应度的负多效性影响,而转录因子基因的突变变化是最不可能的,因为其潜在的影响更广泛[35].此外,一些研究表明甲基化水平在启动子区域MdGST(18),OgCHS(57与基因表达水平和花青素多样性显著相关。然而,各基因启动子区域的GC含量在品种间具有相似的水平(图S10),这表明DNA甲基化不太可能是颜色多样性的原因r . sanguineum复杂。因此,我们认为花青素通路基因的调控和转录可能不是独立的过程,虽然它们分别表达,但它们共同促进了花青素的积累[55,58].尽管如此,我们在黄酮类/花青素通路方面有了初步的发现r . sanguineum复杂的,但需要更多的实验来进一步研究每个遗传成分在这种情况下的贡献。
有人提出,花朵颜色的进化转变通常归因于传粉者介导的选择,这可能有助于生态位的占领和随后的生殖隔离和多样化/物种形成[5].例如,蜜蜂有三种光感受器,在光谱的UV、蓝色和绿色范围内达到峰值,分别对应于344、438和560 nm [59],而鸟类是四色的,并有对600至620纳米红光敏感的受体[60].
在一些授粉研究中杜鹃研究发现,鸟类、蜜蜂、蝴蝶和鞘形蛾是主要的传粉者杜鹃物种(40,41,42].宋等人[61]提出太阳鸟和大黄蜂是潜在的传粉者R.成为而且R.edgeworthii分别有红花和白花,而Epps等[62]观察到蝴蝶在开橙黄色花的r . calendulaceum.这说明传粉者表现出对花朵颜色的偏好,颜色相近的花朵往往会吸引特定的传粉者,即使是在相同的种群之间或密切相关的物种之间[6,59].因此,传粉者组合的时空波动可能导致花朵颜色的变化,反之亦然[5].然而,在一些密切相关的同域类群中,不完全生殖障碍有助于杂交,并在基因流动中发挥关键作用[63].低繁殖隔离的杂交可以频繁地传递颜色基因,特别是在有限的基因库中,如这里所示的品种r . sanguineum因为它们不形成单系基团(图S3).我们的研究结果与一些报告一致,这些报告显示,相对简单的基因变化导致了花朵颜色的重要变化。64].我们已经在这里的密切相关的变种中推断了这些。这种情况可能会引起传粉者聚集的转变,繁殖隔离随着时间的推移而加强。因此,我们的研究可能证明了一种由花朵颜色分化诱导的早期同域物种形成模式,正如通过行为隔离观察到的鸟类[65],以及植物物种的地质-土壤分化[66],或地理差异[67].
结论
本研究采用Illumina转录组测序(RNA-seq)和基因组重测序技术,对田间采集的3个品种的花色基因表达变化进行分析r . sanguineum复杂。本研究初步揭示了黄花菜花色分化的遗传机制r . sanguineum复杂。我们的研究结果表明,花色的变异与表达水平的差异有关,并在一定程度上影响花色的表达顺式作用调节花青素生物合成基因和花青素降解基因,而不是功能突变丢失。深黑的深红色花开了R.var。haemalum表现出几乎所有花青素基因的高表达,而明亮的深红色开花R.var。sanguineum在花青素合成的最初步骤中,是否有高水平的基因表达和黄色的粉红色花朵R.var。didymoides大多数花青素基因有低或中表达水平。后者花青素降解基因表达量较高,可能增加了其花中花青素含量较低的原因。这一发现与之前的一项研究不同艾丽卡花青素基因和MYB识别元件的移码突变被发现是红黄颜色转移的原因。可以提出一些假设来解释复合体中的花色变化:这些品种关系太近,花青素基因或转录因子的编码区域没有获得性突变,它们的花色变化是由于可诱导的表达水平的差异独联体-作用调控,以及合成代谢和分解代谢花青素途径中基因之间的平衡。该复合体目前在品种间存在复杂的杂交和基因流动关系,但如果传粉者偏好强化进一步发展,可能发展为传粉者驱动的早期同域物种形成。
方法
样品收集
三个品种的样品r . sanguineum复杂的(图。1),即r . sanguineumvar。sanguineum(RsS,开着鲜红的花),R。var。haemaleum(RsH,开着深黑深红色的花),R。var。didymoides(标准偏差于2018年6月在云南西北部的高黎贡山(N 27°47′11.40″,E 98°27′35.28″)采集。花蕾后期的花组织。1b)和叶芽(图。1C)从每个品种的三个个体中取样,立即用液氮冷冻。来自相同个体的叶片组织被取样,同时用硅胶干燥,花也是如此,用于花冠反射光谱测量。这9个个体的采样范围很小(20米)2)在类似的气候条件和环境因素(例如土壤、温度、降水和阳光辐射)下。每个人的代金券被收集并存放在中国科学院昆明植物研究所植物标本室。
花反射光谱测量
植物色素对可见光谱中的光的吸收产生了独特的光谱反射特征。为了获得三个品种不同花色的反射率光谱,采用分光光度法定量花青素花冠色素[11,68].在田间测量了5个新鲜的、健康的、完全开放的花(每个品种15个,共45个样品)。两个独立的光谱读数从花的相同位置,花冠管的一半。在200-800 nm范围内使用USB2000+光谱仪测量漫反射光谱,采用氘/钨卤素光源(Ocean Optics,达尼丁,美国佛罗里达州),集成时间为3 s,箱座为12。在每次样品测量前扫描真黑和真白对照参考文献。颜色数据处理采用光学2009 SpectraSuite(海洋光学)软件。光谱被截断到300-700 nm,并使用孔雀座包中R(69,70].用饱和度与色相的比值(S/H)来测定花的花青素含量[31].
RNA和DNA提取和Illumina测序
根据制造商的协议,使用Spectrum TM植物总RNA Kit (STRN250, Sigma)从花芽和叶芽组织中分别提取和纯化总RNA。对每个品种的花芽和叶芽材料进行三次生物重复(来自三株植物)。用DNeasy Plant kit (QIAGEN)从叶片中提取基因组DNA。RNA和DNA的质量和数量用NanoDrop 2000分光光度计(Thermo Fisher Scientific, Waltham, MA, USA)进行评估。根据制造商的协议,使用NEBNext Ultra™RNA/DNA文库准备试剂盒(NEB, MA, USA)生成转录组和基因组文库,并在Illumina HiSeq X Ten测序平台(San Diego, CA, USA)上进行测序,分别为每个RNA和DNA文库生成约6gb和7gb (~ 10x)的对端reads (2 × 150bp)。文库制备和Illumina测序在诺健生物信息技术有限公司(北京)完成。
数据处理,从头组装和映射
首先对原始数据进行过滤,去除带有适配器序列的reads、含有poly-Ns的reads、含有歧义核苷酸的reads和低质量的reads,然后使用SOAPnuke计算phred评分(Q20, Q30) [71].使用FastQC评估剩余读取的质量,包括gc含量、序列长度分布和清洁数据的序列重复水平[72].所有后续的分析都是基于干净的数据。
为了获得参考水平的转录组组装,从每个品种(叶芽、花芽,在三个重复中)的清洁reads进行组合,并使用Trinity v. 2.6.5重新组装[73].除了最小kmer覆盖率(设置为2)和最小contig长度(设置为200)外,所有Trinity参数都设置为默认值。程序集统计信息是使用Trinity包中的TrinityStats.pl脚本获得的。我们还使用了HISAT2 v. 2.1.0 [74]来评估组装质量,通过将读取映射回组装的转录本来计算总体对齐率。
程序集筛选和完整性评估
为减少冗余和复杂性,我们对所有组装的转录本进行过滤,方法如下:首先,我们使用CD-HIT-EST v. 4.7.0 [75],将字长设置为10,序列标识阈值设置为0.95,以去除重复。然后,紧身胸衣v. 1.07 [76]用于聚类转录本序列,过滤多余的转录本,每个基因提取一个具有代表性的转录本。在这种情况下,我们只保留了每个基因最长的转录本。对最终筛选的转录本进行下游分析。为了确定每个组合的转录组完整性,Benchmarking Universal Single-Copy Orthologs工具(BUSCO, v. 4.0.6) [77]用于获取胚胎植物数据库中单拷贝同源的百分比,也用于评估转录本组装的完整性。
正交线预测和函数注释
使用TransDecoder v. 5.5.0从每个过滤后的汇编转录本中预测开放阅读框(orf) [73].这个管道主要包括两个步骤。在第一步中,预测每个转录本最长的ORF,截断最小长度为100个氨基酸。然后,对预测的orf进行扫描,使用BLASTP v. 2.5.0搜索,以截断e值1e-10对策划的蛋白质数据库查找同源谱r .成为与…密切相关的物种r . sanguineum,从存入GigaDB的全基因组测序项目下载[78].所有命中最好的编码肽被保留用于最终的氨基酸序列预测。当一个文本中有多个预测时,我们为每个文本选择得分最高的ORF。我们使用CD-HIT v. 4.7.0 [75]进一步减少最终预测氨基酸的冗余,序列一致性阈值设置为0.95。用OrthoVenn2对3个品种的蛋白质序列进行同源簇(正交群)鉴定[43,使用e-value阈值1e-10。用三个品种注释ORF数据集之间的一对一单拷贝正交关系进行后续分析。的同源蛋白序列R。var。didymoides使用NCBI非冗余(NR)和UniProtKB/Swiss-Prot with BLASTP v. 2.5.0作为蛋白质数据库的代理搜索,设置e-值截止值为1e-10。我们还使用DIAMOND[执行了额外的函数注释]79命中eggNOG数据库[80],总结了不同蛋白质数据库中可用的功能信息,包括GO、COGs/KOGs和KEGG。最好的结果被用作最终的注释。
阅读映射,SNP调用和集群
对于每个品种,使用HISAT2 v. 2.1.0将三个复制植物的清洁reads(花芽和叶芽的组合)分别与参考转录组(同源簇)对齐[74参数设置为默认值。SAMtools v. 1.9 (https://github.com/samtools/)和Picard tools v. 2.21.8 (http://broadinstitute.github.io/picard/)用于对重复读取进行排序、标记和删除,并对9个样本中的每个样本的bam对齐结果进行重新排序。我们使用GATK v. 4.1.5 [81]执行SNP呼叫和Plink v. 1.9 (http://pngu.mgh.harvard.edu/purcell/plink/)使用设置的参数筛选snp——基因族群0.1——加0.01——indep-pairwise10、0.2。最后,将符合条件的snp合并到单个VCF文件中,作为SNPhylo v. 20180901的输入[82],以最大似然重建样本之间的系统发育关系,使用1000个自举复制进行分支支持。为了进一步研究遗传变异的分布,本文采用主成分分析(PCA)方法对黄曲霉的SNP变异进行了分析R(69].
转录丰度和差异表达分析
基因表达水平(丰度估计)通过使用RSEM v. 1.3.1将每个生物复制的花芽样本的所有配对端reads分别映射回每个品种的参考转录组的一对一单拷贝正交图来计算[83],采用Bowtie2 v. 2.3.5进行对齐[84].因为每个品种集合都是独立从头生成的,没有参考,所以Trinity ID头被随机分配给每个品种。为了确保每个量化文件被分配相同的ID头,从而可以集成,我们替换了Trinity生成的抄本ID,用其各自的单一副本对应名称(对应的集群)。在获得每个生物复制花芽样本的基因表达丰度后,我们基于每千碱基的外显子每百万片段映射reads (FPKM)的片段生成了基因表达矩阵。用Trinity脚本生成的读取计数矩阵用于差异表达基因(DEGs)分析。用DESeq2包进行差异分析[85在R以两两比较的方法鉴定差异基因。deg被认为是那些调整了错误发现率(FDR)的p取值≤0.05,log2(倍数变化)的绝对值≥1。为了比较三个品种的基因表达值,我们使用了m值归一化的修剪平均值(TMM),如在R包磨边机(86].所有下游分析均基于归一化表达式数据矩阵(TMM归一化)实现。从各品种间的比较中筛选出deg进行进一步的功能富集分析。在对所有同源基因进行功能注释的基础上,通过内部Perl脚本提取所有注释项,并导入到一个R包AnnotationForge生成一个OrgDB生物体注释对象,该对象包含上述数据库中基因ID和其他标识符之间的对应映射。差异表达基因(DEGs)的GO(基因本体)和KEGG(京都基因与基因组百科全书)富集分析由clusterProfiler包实现[87在R.
表达谱的统计分析
所有的表达分析分别对每个品种的三个生物重复进行。构建了9列15164行的基因表达矩阵。每一列代表一个样本,每一行对应一个同源基因的表达。用数据矩阵计算Pearson相关系数(r)。对所有样品进行了对称热图和主成分分析RPheatmap v. 1.0.12 (https://CRAN.R-project.org/package=pheatmap)和PCAtools v. 1.1.0 (https://github.com/kevinblighe/PCAtools),分别。GO术语和代谢途径p≤0.05的细胞被DEGs显著富集。
实时定量PCR (qRT-PCR)验证
为了验证RNA-seq结果与花青素生物合成途径的可靠性,选取了13个基因进行qRT-PCR分析。使用BioRT Master HiSensi cDNA First Stand Synthesis Kit (Bioer, Hangzhou, China),稀释20倍作为模板合成第一链cDNA文库。在QuantStudio™7 Flex实时PCR系统(Applied Biosystems, CA, US)上,使用BioEasy主混合SYBR Green (Bioer,杭州,中国)对3个生物重复(植物)进行QRT-PCR。qRT-PCR扩增条件为:95°C变性1 min, 95°C变性15 s, 60°C退火延伸60 60s,循环40次。12个基因的引物列于表中S7.GLYCERALDEHYDE-3-PHOSPHATE脱氢酶(GAPDH)r . simsii(基因库acc。不。FN552706)作为规范化的内部控制[50],相对表达量用2−ΔΔCT方法(88].
启动子序列多样性的鉴定与表征
进一步了解表达水平是否受独联体-调控元件和启动子的DNA甲基化,根据已发表的cDNA序列进行了检索杜鹃基因组(78],以序列相似度最高的最佳命中基因作为相应参考。然后我们将每个基因组测序品种(三个个体组合)的干净读数映射到每个杜鹃引用序列,并通过sam2consensus.py脚本(https://github.com/edgardomortiz/sam2consensus).生成每个基因的每个品种的一致性序列(fasta文件)。翻译起始位点上游的每个2kb启动子序列从一致序列和中缩短独联体-作用调控元件由PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)数据库。TBtools v. 1.068 [89,然后用来可视化的分布独联体表演元素。利用在线平台mVISTA将三个品种中每个基因的启动子序列成对比对到每个参考序列[90],并在50 bp的滑动窗口内绘制出基对识别图,范围为75 ~ 100%。滑动窗口为50 bp,计算各品种各基因启动子的GC含量,并用ggplot2包进行可视化R(69].
数据和材料的可用性
原始数据可从NCBI序列读取档案(SRA)平台访问,登录号为PRJNA720238 (http://www.ncbi.nlm.nih.gov/bioproject/720238).
缩写
- 4 cl:
-
4-coumarate-CoA连接酶
- ANR:
-
花青素还原酶;
- 答:
-
花青素合成酶
- :
-
乙酰转移酶
- BGLU:
-
Beta-glucosidase
- C4H:
-
肉桂acid4-hydroxylase
- 计算机辅助设计:
-
肉桂醇脱氢酶
- 气:
-
查耳酮异构酶
- CHS:
-
查耳酮合酶
- DFR:
-
Dihydroflavonol-4-reductase
- F3'H:
-
黄烷酮3 ' -羟化酶
- F3H:
-
黄烷酮3-hydroxylase
- 读者:
-
黄酮醇合成酶
- 销售税:
-
谷胱甘肽s-transferase
- MBW:
-
MYB-bHLH-WD40复杂
- OMT:
-
O-methyltransferase
- 朋友:
-
苯丙氨酸ammonia-lyase
- /:
-
过氧化物酶
- GAPDH:
-
甘油醛3磷酸脱氢酶
- RNA-seq:
-
RNA序列
- 度:
-
差异表达基因
- 爆炸:
-
基本的本地对齐搜索工具
- 车身:
-
对标通用单拷贝矫形仪
- NR:
-
NCBI冗余
- UniProtKB:
-
通用的蛋白质知识库
- 齿轮:
-
同源基团的簇
- 蛋酒:
-
基因的进化谱系:非监督的同源组
- GATK:
-
基因组分析工具箱
- 走:
-
基因本体论
- KOG:
-
真核蛋白质的同源基团
- KEGG:
-
京都基因和基因组百科全书
- 罗斯福:
-
错误发现率
- FPKM:
-
每kb每百万片段
- 主成分分析:
-
主成分分析
- SNP:
-
单核苷酸多态性
- TMM:
-
m值的修剪平均值。
参考文献
- 1.
为什么花是不同的?自然植物种群中花的大小和形态变化的功能生态学。生物科学。1999;49(8):631 - 40。
- 2.
阿罗尔迪CA,费里亚J,格洛弗BJ。植物结构颜色的细胞和遗传基础。植物生物学杂志2019;47:81-7。
- 3.
单海辉,程杰,张锐,姚旭,孔兆喆。花多样化的发育机制。Nat植物。2019;5(9):917 - 23所示。
- 4.
被子植物生殖特性的适应性辐射I:授粉机制。环境科学与技术学报1970;1(1):307-26。
- 5.
穆奇哈拉N,约翰森S,史密斯SD。蜂鸟授粉竞争形成了安第斯茄科花的颜色变化。进化。2014;68(8):2275 - 86。
- 6.
Bradshaw HD, Schemske DW。在猴花中,花色位点上的等位基因替换产生传粉者转移。大自然。2003;426(6963):176 - 8。
- 7.
环境和传粉媒介介导的邻花小种的选择Mimulus aurantiacus.中国病原生物学杂志2007;20(1):122-32。
- 8.
库利AM,莫德利舍夫斯基JL,隆美尔ML,威利斯JH。基因复制Mimulus通过独立的跨调控变化来实现平行花的进化。咕咕叫医学杂志。2011;21(8):700 - 4。
- 9.
植物色素的遗传与生物化学。植物生物学杂志2006;57:761-80。
- 10.
田中勇,王晓燕,王晓燕。植物色素的生物合成:花青素,β -甜菜碱和类胡萝卜素。植物j . 2008; 54(4): 733 - 49。
- 11.
杜辉,赖林,王芳,孙伟,张磊,李旭,等。花的颜色特征在30杜鹃通过花青素和黄酮醇的鉴定和数量性状。植物医学杂志。2018;20(1):121 - 9。
- 12.
朴妮,徐浩,李旭,张爱海,朴硕,安GH,等。萝卜花青素生物合成基因的积累与表达(萝卜).农业食品化学学报,2011;59(11):6034-9。
- 13.
李伟峰,毛杰,杨世杰,郭志刚,马志华,达达MM,等。“红好吃”及其四代芽运动突变体果皮中花青素积累与激素的关系。植物生物学杂志,2018;18(1):363。
- 14.
李涛,张伟,杨海辉,董强,任杰,范海辉,等。比较转录组分析揭示了与玉米果皮和糊粉层花青素组织特异性积累相关的差异表达基因。Sci众议员2019;9(1):1 - 12。
- 15.
蒋涛,张敏,文超,谢曦,田伟,文硕,等。花青素调控网络的综合代谢组学和转录组学分析丹参知母。花。BMC Plant biology 2020;20(1):349。
- 16.
Gurung PD, Upadhyay AK, Bhardwaj PK, Sowdhamini R, Ramakrishnan u樱草属植物sikkimensis是一种高海拔植物。BMC基因组学。2019;20(1):989。
- 17.
张颖,周涛,戴智,戴旭,李伟,曹明,等。比较转录组学为研究植物花色多态性提供了新的思路Pleione limprichtii兰花人口。国际分子生物学杂志2020;21(1):247。
- 18.
梁东,徐凯。苹果转录组分析(马吕斯×有明显)黄果体细胞突变发现了一个与花青素和表观遗传调控高度相关的基因网络模块。中国生物医学工程学报2015;66(22):7359-76。
- 19.
杨福生,聂松,刘浩,史立林,田小春,周树生,等。广泛栽培的杜鹃花亲本的染色体水平基因组组装。Nat Commun。2020;11(1):1-13。
- 20.
Magris G, Di Gaspero G, Marroni F, Zenoni S, Tornielli GB, Celii M,等。葡萄品种间基因表达变异的遗传、表观遗传和基因组效应。工厂j . 2019, 99(5): 895 - 909。
- 21.
王晓燕,王晓燕。植物组织中花青素的共色素作用及其对颜色的影响。植物化学,1972;11(3):1139 - 44。
- 22.
Mol J, Grotewold E, Koes R.基因如何描绘花朵和种子。植物学报,1998;3(6):212-7。
- 23.
金霞,黄宏,王丽,孙艳,戴森。转录组学和代谢产物分析揭示了不同花青素生物合成分支途径的分子机制狗舌草cruentus品种。植物科学进展2016;7:1307。
- 24.
zzipor G, Duarte P, Carqueijeiro I, Shahar L, Ovadia R, Teper-Bamnolker P,等。在植物花青素降解空泡ⅲ类过氧化物酶Brunfelsia calycina花。新植醇。2015;205(2):653 - 65。
- 25.
刘颖,Tikunov Y, Schouten RE, Marcelis LF, Visser RG, Bovy A.植物中花青素的生物合成及降解机理茄属的蔬菜:审查。前化学2018;52。
- 26.
藤野N,天马N, Waki T, Ito K, Komatsuzaki Y, Sugiyama K,等。金鱼草和牛蒡类黄酮酶之间的物理相互作用揭示了不同植物类黄酮代谢组织的多样性。植物j . 2018; 94(2): 372 - 92。
- 27.
van der Kooi CJ, Dyer AG, Kevan PG, Lunau K.花的光学特性对视觉信号的功能意义。安机器人。2019;123(2):263 - 76。
- 28.
对抗色理论的一些定量方面。2正常视力和二色视力的亮度、饱和度和色相。环境科学学报。1955;45(8):602-16。
- 29.
薛文K, Venail J, Shang Y, Mackay S, Alm V, Butelli E,等。myb调控基因的一个小家族控制着该属的花色素沉着强度和模式金鱼草属植物.植物细胞。2006;18(4):831 - 51。
- 30.
王磊,Albert NW,张浩,Arathoon S, Boase MR, Ngo H,等。花青素生物合成的时空调控提供了不同的花色强度和花型兰花兰花。足底。2014;240(5):983 - 1002。
- 31.
杨旭,张建军,郭德东,熊旭,常丽丽,牛清良,等。基于颜色信息的生菜叶片花青素含量测定与评价。IFAC-PapersOnLine。2016; 49(16): 96 - 9。
- 32.
刘亮,张丽丽,王立林,牛晓燕。花青素和黄酮醇含量分析杜鹃产于西藏东南部锡格拉山脉的一种。植物生理与生物化学,2016;104:250-6。
- 33.
水田D,班田T,宫岛I,中冢A,小林N.常绿杜鹃花花色苷组成模式的比较。Sci Hortic。2009;122(4):594 - 602。
- 34.
米柴皮文,小林,中冢。平朵杜鹃花品种色素组成及花青素合成基因表达分析。Hortic j . 2020; 89(3): 284 - 91。
- 35.
Le Maitre NC, Pirie MD, Bellstedt DU。Cape植物花色、花青素合成基因表达与调控艾丽卡物种。植物科学进展2019;10:1565。
- 36.
Le Maitre NC, Pirie MD, Bellstedt DU。在进化背景下确定花青素合成酶基因表达的方法:一个例子艾丽卡plukenetii.安机器人。2019;124(1):121 - 30。
- 37.
方爱美、方仁仁、何爱美、胡丽丽、杨惠平、张伯伦。杜鹃见《中国植物志》第14卷。吴,ZY和PH Raven。出版地点:科学出版社,北京,圣路易斯密苏里植物园;2005.
- 38.
Myers N, Mittermeier RA, Mittermeier CG, Da Fonseca GA, Kent J.生物多样性保护重点热点。大自然。2000;403(6772):853 - 8。
- 39.
闫丽娟,刘娟,Moller M,张亮,张晓明,李德智,等。DNA条码技术的杜鹃(Ericaceae)是喜马拉雅-横断山区生物多样性热点地区最大的中国植物属。中国生物医学工程学报,2015;15(4):932-44。
- 40.
黄志华,宋永平,黄sq。雀形目鸟类传粉的证据杜鹃物种。Aob植物。2017;9 (6):plx062。
- 41.
Basnett S, Ganesan R, Devy SM。花的性状决定传粉者访花杜鹃跨越锡金喜马拉雅海拔梯度的物种。高山机器人。2019;129(2):81 - 94。
- 42.
闫丽娟,Burgess KS,郑伟,陶志斌,李德志,高林明。不完全的生殖隔离杜鹃类群使杂交的形成和持续。植物生物学杂志,2019;61(4):433-48。
- 43.
徐磊,董志斌,方磊,罗玉军,魏志英,郭海林,等。OrthoVenn2:用于跨多个物种的全基因组比较和同源集群注释的web服务器。核酸学报2019;47(W1): W52-W8。
- 44.
佐佐木N,西崎Y,内田Y,若松E,元本N,莫濑M,等。康乃馨花颜色强度相关谷胱甘肽s -转移酶基因的鉴定。生物科技植物》。2012;29(3):223 - 7。
- 45.
宫川N,宫原T,冈本M,广濑Y,坂口K,波野S,等。二氢黄酮醇4-还原酶活性与花的颜色强弱有关飞燕草.生物科技植物》。2015;32(3):249 - 55。
- 46.
雅可拉L, Määttä K, Pirttilä AM, Törrönen R, Kärenlampi S, Hohtola .越橘果实发育过程中花青素生物合成相关基因表达与花青素、原花青素和黄酮醇水平的关系。植物杂志。2002;130(2):729 - 39。
- 47.
张永忠,徐淑贞,程亚伟,彭志峰,韩建明。花青素相关基因的转录组分析揭示了光强对红叶莴苣花青素生物合成的影响。PeerJ。2018; 6: e4607。
- 48.
赵德强,蒋勇,宁春林,孟JS,林士生,丁伟,等。一个嵌合体的转录组测序揭示了介导牡丹黄色形成的花青素生物合成基因的协调表达(芍药棺罩)。BMC基因组学,2014;15(1):689。
- 49.
赵德强,陶杰。观赏植物花色发育与调控的研究进展。植物科学,2015;6:261。
- 50.
De Keyser E, Desmet L, Van Bockstaele E, De Riek J.如何在植物物种中准确地进行RT-qPCR ?杜鹃花花色基因表达的个案研究(杜鹃simsii混合动力车)映射。分子生物学杂志,2013;14(1):13。
- 51.
邱贺,Azlan A, Tang ST, Lim SM。花青素和花青素:有色色素作为食品、药品的成分,并有潜在的健康益处。2017;61(1):1361779。
- 52.
Huyen DTT, Ureshino K, Van DT, Miyajima I.花青素黄酮醇在斑区协同着色杜鹃simsii木板。花。Hortic j . 2016; 85(3): 232 - 7。
- 53.
杨强,袁涛,孙小波。两个牡丹品种花期花色变化的初步研究。植物学报,2015;42(5):930-8。
- 54.
朱海红,杨建新,肖春,毛泰,张军,张海燕。类黄酮途径代谢产物和转录物的差异影响水生植物黄色花瓣的颜色莲属椰子.植物生物学杂志2019;19(1):277。
- 55.
Sapir Y, Gallagher MK, Senden e,是什么维持了种群内花朵颜色的变异?2021年生态发展趋势。https://doi.org/10.1016/j.tree.2021.01.011.
- 56.
进化有两个层面:基因和形态。公共科学图书馆杂志。2005;3 (7):e245。
- 57.
刘新军,庄燕,邱春春,陈直东,沈丰强,叶坤华。甲基化对查尔酮合成酶基因表达的影响决定了两个文兰品种花组织中花青素的色素沉淀。足底。2012;236(2):401 - 9。
- 58.
蒋硕,陈明,何新宁,陈晓丽,王宁,孙庆国,等。MdGSTF6由MdMYB1激活,在苹果花青素积累中起重要作用。Hortic杂志2019;40。
- 59.
传粉者表现出花的颜色偏好,但颜色相近的花并不会吸引相似的传粉者。安机器人。2016;118(2):249 - 57。
- 60.
鸟类视觉色素:特征、光谱调谐和进化。Nat。2007;169 (S1): S7-S26。
- 61.
宋永平,黄志华,黄sq。花粉通过粘蛋白线聚集杜鹃随授粉。新植醇。2018;221(2):1150 - 9。
- 62.
艾普斯MJ,艾利森SE,沃尔夫LM。杜鹃花的繁殖(杜鹃calendulaceum凤梨科):一种罕见的昆虫翅传粉个案。Nat。2015;186(2):294 - 301。
- 63.
在开花植物和传粉者的生物伙伴关系中花朵颜色的进化和生态学(特别指兰花)。澳大利亚:莫纳什大学;2013.
- 64.
花卉颜色的进化过渡。植物生态学报2008;29(1):1 - 8。
- 65.
江江宇,杨文华。鸟类的行为隔离与早期物种形成。中国生物医学工程学报2018;49(1):1 - 24。
- 66.
李凯旭,任晓峰,宋晓燕,李晓军,周勇,Harlev E,等。地质-土壤差异引起的野生大麦早期同域物种形成。生命科学联盟。2020;3(12):e202000827。
- 67.
Streisfeld MA, Young WN, Sobel JM。分化选择驱动R2R3-MYB转录因子的遗传分化,该转录因子有助于植物的早期物种形成Mimulusaurantiacus。公共科学图书馆麝猫。2013;9 (3):e1003385。
- 68.
伊里尔·A,拉格里奥MG。Biospectroscopy的杜鹃indicum花。基于反射法的花瓣花青素无损评价。光化学与生物学报2009;8(3):337-44。
- 69.
Ihaka R, Gentleman R. R:用于数据分析和图形的语言。计算图统计1996;5(3):299-314。
- 70.
Maia R, Eliason CM, Bitton PP, Doucet SM, Shawkey MD. pavo:光谱数据分析、可视化和组织的R包。方法中国环境科学,2013;4(10):906-13。
- 71.
陈云云,陈云云,史长明,黄志斌,张勇,李斯克,等。SOAPnuke:一个支持MapReduce加速的软件,用于高通量测序数据的集成质量控制和预处理。GigaScience。2018; 7(1): 1 - 6。
- 72.
FastQC:用于高通量序列数据的质量控制工具。2010.http://www.bioinformatics.babraham.ac.uk/projects/fastqc.
- 73.
Haas BJ, Papanicolaou A, Yassour M, Grabherr M, Blood PD, Bowden J,等。新创利用trinity平台从RNA-seq中重建转录本序列进行参考生成和分析。Nat Protoc。2013;8(8):1494 - 512。
- 74.
Kim D, Paggi JM, Park C, Bennett C, Salzberg SL.基于图谱的HISAT2和hisat基因型基因组对齐和基因分型。生物科技Nat》。2019;37(8):907 - 15所示。
- 75.
傅林敏,牛BF,朱志伟,吴圣,李文忠。CD-HIT:加速聚类下一代测序数据。生物信息学,2012,28(23):3150 - 2。
- 76.
Davidson NM, Oshlack A.胸衣:启用差异基因表达分析新创转录组组装。基因组医学杂志。2014;15(7):410。
- 77.
Simão FA, Waterhouse RM, Ioannidis P, Kriventseva EV, Zdobnov EM. BUSCO:用单拷贝矫形仪评估基因组组装和注释完整性。生物信息学,2015,31(19):3210 - 2。
- 78.
张亮,徐普伟,蔡艳芳,马丽丽,李顺生,李顺生,等。基因组组装草图杜鹃成为法兰治(姓氏)var。成为.GigaScience。2017; 6(10): 1 - 11。
- 79.
布芬克B,谢C,胡森DH。使用DIAMOND快速和灵敏的蛋白质校准。Nat方法。2015;12(1):59-60。
- 80.
Huerta-Cepas J, Forslund K, Coelho LP, Szklarczyk D, Jensen LJ, Von Mering C,等。eggNOG-mapper通过矫形学赋值实现全基因组快速功能注释。中华分子生物学杂志2017;34(8):2115-22。
- 81.
麦肯纳A,汉娜M,班克斯E, Sivachenko A, Cibulskis K, Kernytsky A,等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组研究》2010;20(9):1297 - 303。
- 82.
李th,郭浩,王旭,Kim C, Paterson AH。SNPhylo:从海量SNP数据构建系统发育树的管道。BMC基因组学,2014;15(1):162。
- 83.
杜威中国出版社。RSEM:从有或没有参考基因组的RNA-Seq数据中精确的转录物定量。BMC生物信息学。2011;12(1):323。
- 84.
朗米德B,萨尔茨伯格SL,快速间隙读取对准蝴蝶结2。Nat方法。2012;9(4):357 - 9。
- 85.
Love MI, Huber W, Anders S.用DESeq2调节RNA-seq数据的折叠变化和色散估计。基因组医学杂志。2014;15(12):550。
- 86.
罗宾逊医学博士,麦卡锡DJ,史密斯GK。edgeR:用于数字基因表达数据的差异表达分析的Bioconductor包。生物信息学,2010,26(1):139 - 40。
- 87.
于春春,王乐林,韩欢聚,何庆勇。clusterProfiler:一个R包,用于比较基因簇之间的生物主题。中国生物医学工程学报2012;16(5):284-7。
- 88.
Schmittgen TD, Livak KJ。比较CT法分析实时PCR数据。Nat Protoc。2008;3(6):1101。
- 89.
陈cj,陈浩,张颖,Thomas HR, Frank MH,何永华,等。TBtools:为大型生物数据的交互式分析而开发的集成工具包。摩尔。2020;13(8):1194 - 202。
- 90.
Frazer KA, Pachter L, Poliakov A, Rubin EM, Dubchak I. VISTA:比较基因组学的计算工具。核酸Res. 2004;32(增刊2):W273-9。
确认
我们感谢博士们。钟美才、蒋晓东等同事(中国科学院昆明植物研究所)和Kanae Nishii博士(神奈川大学和爱丁堡皇家植物园,RBGE)对数据分析和早期版本的手稿提出了建设性的建议。实验室工作和数据分析在中国科学院昆明植物研究所西南野生物种种质库分子生物学实验室和iFlora高性能计算中心进行。RBGE由苏格兰政府的农村和环境科学和分析服务部支持。
资金
本研究得到国家自然科学基金项目(91631101,31670213)、中国科学院战略重点研究项目(XDB31000000)、中国科学院大型科学设施项目(2017-LSFGBOWS-02)、中国科学院国际合作计划项目(151853KYSB20190027)和云南省科技人才培养计划项目(2017HA014)资助。
作者信息
从属关系
贡献
LMG和DZL构思和设计了这项研究。LJY, YHL, JYZ和WZ进行了实地工作和样品采集。LJY和JYZ进行了实验。LJY分析了这些数据。YHW, JL, ADZ, JYH和LMG引导数据分析。LJY, MM和LMG在其他作者的输入下撰写了手稿。所有作者阅读并批准了最终稿件。
相应的作者
道德声明
伦理批准和同意参与
本研究所用植物均采自中国国家级自然保护区高黎贡山。对植物的实地研究符合国家指导方针和立法,不涉及任何濒危或受保护物种。
同意出版
不适用。
相互竞争的利益
作者声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1:表S1。
3个品种样品的转录组测序、组装和筛选的统计杜鹃sanguineum复杂。表S2。3个品种的2个组织的18个RNA文库的转录组测序和对齐率的基本信息杜鹃sanguineum复杂。表S3。BUSCO统计用于评价三个品种的转录组组装质量杜鹃sanguineum复杂。表S4。跨三个品种的同源聚类统计杜鹃sanguineum复杂。表S5。三种品种的同源簇的鉴定和注释杜鹃sanguineum复杂。表S6。40个预测花青素相关基因的列表杜鹃sanguineum复杂。表S7。引物信息用于qRT-PCR验证三个品种中涉及花青素合成的12个基因序列杜鹃sanguineum复杂。表S8。所有基因启动子的统计信息杜鹃sanguineum复杂。图S1。三个品种的转录组组合的BUSCO质量评价结果杜鹃sanguineum复合体(a)和针对6个不同数据库的15,164个同源簇命中的注释(b)r . sanguineumvar。sanguineum;RsH。r . sanguineumvar。haemaleum;相对标准偏差,r . sanguineumvar。didymoides.图S2。注释的15,164个一对一的单拷贝正交关系的三个品种杜鹃sanguineum通过三种不同的数据库进行审讯。图S3。3个品种的系统发育和遗传聚类结果杜鹃sanguineum复杂。图S4。3个品种的全部18个样本的基因表达谱杜鹃sanguineum复杂。图S5。基于3个品种的两两比较,建立了差异表达基因(DEGs)的火山图杜鹃sanguineum复杂。图S6。3个品种间差异表达基因(DEGs)功能富集的热图和结果杜鹃sanguineum复杂。图S7。23个候选基因的归一化表达水平的层次聚类显示出不同的基因表达谱RsH而且标准偏差.变种代码如图所示S1.红色表示高表达,蓝色表示低表达。图S8。25个候选基因的归一化表达水平的层次聚类显示出不同的基因表达谱RsH而且RsS.变种代码如图所示S1.红色代表高表达,蓝色代表低表达。图S9。12个候选基因归一化表达水平的层次聚类显示出不同的基因表达谱RsS而且标准偏差.变种代码如图所示S1.红色代表高表达,蓝色代表低表达。图S10。13个花青素基因启动子结构的共同布局。变种代码如图所示S1.
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
你们,LJ。,Mӧller, M., Luo, YH.et al。花青素合成基因的差异表达是异域花颜色差异的基础杜鹃sanguineum复杂。BMC植物杂志21,204(2021)。https://doi.org/10.1186/s12870-021-02977-9
收到了:
接受:
发表:
关键字
- 花青素的合成
- 比较转录组
- 花的颜色
- 基因表达
- 杜鹃sanguineum复杂的
- 同域物种形成