摘要
背景
Myrtales是罗西邦的富含物种,具有许多具有重要经济,药用和观赏价值的物种。目前,虽然有关于Myrtales的叶绿体结构的报道,但缺乏对Myrtales的叶绿体结构的综合分析。通过使用叶绿体基因片段,Myrtales的系统发育和分歧时间估计主要是由叶绿体基因片段构建的,并且对关系的载体是低的。一种更可靠的重建物种分歧时间和系统发育关系的方法是通过使用全叶绿体基因组。在这项研究中,我们综合分析了细胞质叶绿体的结构特征,比较了变异热点,并重建了多种化石和一个二级校准点的Myrtales的物种分化时间。
结果
获得了92个Myrtales的叶绿体序列,代表六个家族,16个亚属和78属,包括全基因组测序的九个新序列的叶绿体。结构分析表明,叶绿体的尺寸范围为152,214-171,315bp,并且表现出典型的四部分结构。IR地区在23,901-36,747 BP之间,具有大的单拷贝区域,跨越83,691-91,249 BP和跨越11,150-19,703 BP的小单拷贝区域。总共,在叶绿体中存在123-133个基因,其中包括77-81个蛋白质编码基因,四个rRNA基因和30-31个TRNA基因。
GC含量为36.9 ~ 38.9%,平均GC含量为37%。LSC区、SSC区和IR区GC含量分别为34.7 ~ 37.3%、30.6 ~ 36.8%和39.7 ~ 43.5%。通过对桃金娘叶绿体核苷酸多态性的分析,我们提出了21个高变区作为桃金娘潜在的DNA条形码区。系统发育分析表明,桃金娘及其相应的科属为单系,菊科(combretacaceae)和淫羊藿科(Onagraceae + Lythraceae) (BS = 100%, PP = 1)为姐妹类群。分子年代测定结果表明,桃叶属植物的树冠最可能为104.90 Ma (95% HPD = 87.88 ~ 114.18 Ma),与老叶属植物的树冠最可能为111.59 Ma (95% HPD = 95.50 ~ 118.62 Ma)。
结论
桃金娘的叶绿体基因组结构与其他被子植物相似,具有典型的四部分结构。由于IR区域的扩张和收缩,这组叶绿体基因组大小略有不同。叶绿体基因组非编码区变异大于编码区变异。系统发育分析结果表明,菊科和羊草科+羊草科为良好的姊妹类群。分子年代测定表明桃金娘冠极有可能起源于下白垩世的阿尔比安时代。这些叶绿体基因组对研究桃金娘的遗传多样性和物种进化具有重要意义,同时也为桃金娘的分类和系统发育研究提供了有用的信息。
背景
桃金娘属蔷薇科(Rosidae),是被子植物蔷薇科(Rosanae)分枝中最具物种的类群之一[1那2].根据APG IV [3.],桃金娘由9科380属约13000种组成。该目的9个科分别为Alzateaceae、combreaceae、Crypteroniaceae、Lythraceae、melastomata aceae、myrtacaceae、onagaceae、对虾科和Vochysiaceae。科间物种丰富度不均衡,Alzateaceae、Crypteroniaceae和对虾科的物种相对较少。物种广泛分布于热带地区,其中Vochysiaceae显示出一种两栖-大西洋间断分布[2].珊瑚科植物主要分布在热带和亚热带地区,特别是非洲大草原[4.].该命令是与草本植物,莲花,树木和红树林的形态多样化,以及各种各样的水果类型(浆果,胶囊,撒母羚羊和芽)[1] (图。1).Myrtales的两种主要木子解剖学特征:双侧血管束在初级茎和血管束中的次级抑郁症中的边缘凹陷,在其他开花植物中并不常见。这两个解剖学特征的组合非常罕见[5.那6.那7.].许多桃金娘的品种具有重要的经济价值[8.],观赏[9.]和药物价值[10.那11.].
随着第二代排序技术的快速发展,测序成本使系统孕组接近在大规模上可行,迎来了植物鉴定和分类的新探索。完全的塑料序列已成为强大的工具,以回答有关从推断的文学发育的植物演化的问题[12.那13.那14.那15.那16.那17.那18.].塑料是光合细胞中的必需细胞器,在维持生命中发挥着重要作用[19.],主要是被子植物的母系遗传。大多数质体DNA由长度为120-220 kb的双链组成[20.和高度保守的典型四部分基因组结构。近年来,研究人员致力于许多类群叶绿体的结构和系统发育分析,包括桃金娘[21.那22.那23.].叶绿体的结构特征对研究遗传多样性和物种进化具有重要意义,对制定种质资源保护政策具有重要意义[24.那25.那26.].
Reginato等人[21.首次报道了Melastomataceae的叶绿体基因组的比较。比较和分析16种Meltomataceae和八种公布的叶绿体基因组和八种公布的叶绿体基因组的结构,基因含量和一般特征。他们发现,MelaStomataceae的叶绿体基因组,如大多数血管植物,具有典型的锥形结构,其具有84个蛋白质编码基因(CDS),37个TRNA和8 rRNA的大单拷贝区域,总共129个基因[21.].古等人。[22.报道了塑料海莫米娅myrtifolia是一种重要的药用植物,具有多种药理生物碱。随后,结合紫薇科其他22个种的样本,对紫薇属植物叶绿体基因组结构进行了综合分析,并与紫薇属其他种的叶绿体基因组结构进行了比较。22种百合科植物的叶绿体基因组范围为152049 ~ 160769 bp,其中包括10个变异热点作为潜在的分子标记[23.].此外,最近还报道了桃金娘的其他叶绿体基因组。Rodrigues等人[27.]对桃金娘6个叶绿体基因组的结构、基因数量和基因组大小进行了比较,发现它们与其他桃金娘的叶绿体基因组相似。然而,以往对桃金娘叶绿体基因组的研究并不一致,有的基于科、属或种的研究。目前,对桃金娘叶绿体基因组结构的全面分析还很缺乏。
研究人员除了对桃金娘叶绿体基因组结构进行研究外,还对桃金娘的分化时间和系统发育进行了探讨,但大多是基于基因片段的研究。强有力的系统发育框架是研究物种形成的基础。在以往的分子系统发育研究中,一些叶绿体位点以及细胞核DNA的内部转录间隔区(ITS)和其他核糖体区域被用于桃金娘的系统发育分析[2那7.那28.].Conti等人[7.]使用50个征集(包括39种和11个小组)和叶绿体基因rbcl.重建myrtales的系统发育。结果表明,Onagraceae和睫状症与梳子科密切相关[7.].sytsma等。[28.]基于桃金娘叶绿体基因片段构建了桃金娘的系统发育分化时间rbcl.和ndhF从79种Myrtales和五种化石校准点,表明Myrtales在早期的白葡萄球菌(111 mA)中差异化,梳理科学是Myrtales的最早,支持。Berger等人。[2]扩增6个基因片段并测序(RBCL,NDHF,MATK,MIGR,并利用10个化石定标点估算了桃金娘的散度时间。结果表明,桃金娘科(combretacae)的冠层最可能为116 Ma (95% HPD = 113.7-118.8 Ma),系统发育也表明,combretacae是桃金娘科(Myrtales)的姐妹群[2].最近,李等人[18.]利用来自2881个质体的80个基因和62个化石标记重建了被子植物的广泛系统发育,结果表明桃金娘及其所有科系都是单系的。系统发育结果表明,桃金娘科(Myrtales)和天竺葵科(Geraniales)的树冠年龄为112.26 Ma,菊科(combretacaceae)和龙舌兰科(onagracaceae) +百合科(Lythraceae)为姐妹类群,具有较强的支持。基于叶绿体基因片段的研究大多推断出支持度较低的关系,因此利用叶绿体基因组来探索物种分化时间和重建系统发育关系具有可信度。
目前有很少有关于Myrtales的叶绿体基因组结构的研究。虽然使用分子方法研究了Myrtales的系统发育位置和关系,但由于缺乏系统发育信号和稀疏的分类抽样,对细胞的放置的载体通常弱。因此,我们首先通过使用全叶绿体基因组来扩展抽样,重建Myrtales的系统发育关系,并相对分析Myrtales的塑性结构,为未来的研究提供基础。在这项研究中,我们测序了九种新物种的叶绿体基因组(包括Myrtaceae,Myrastomataceae和Combretaceae),并将其与来自NCBI的Myrtales的现有塑料数据组合在一起,以获得95个叶绿体基因组,代表六个家庭,78个属,和三个小组。本研究的主要目标是1)分析叶绿体基因组结构并阐明Myrtales的遗传多样性,2)重建Myrtales的系统发育关系,以特异性确定梳理群岛的系统发育位置,3)推断细胞瘤的发散时间。
结果
叶绿体基因组特征
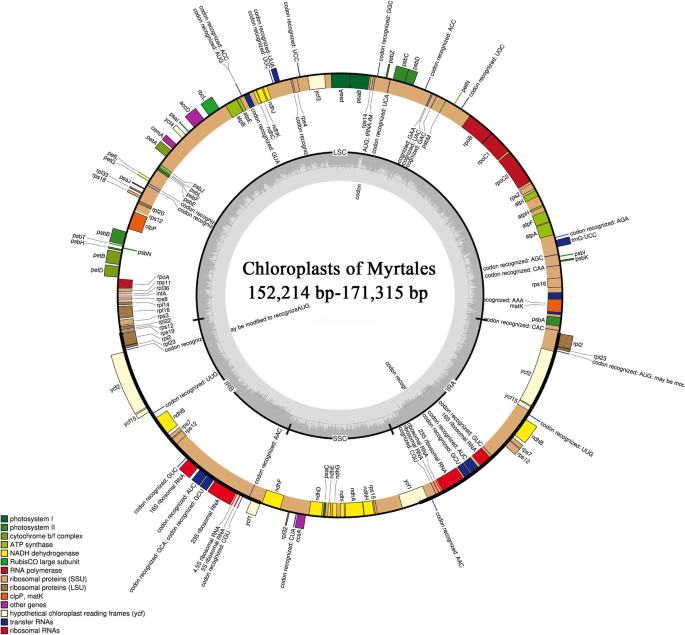
六个家庭用本研究中使用的92个Myrtales叶绿体基因组代表:Melastomataceae(五种亚毛利人中的42种),myrtaceae(包括五种亚属植物中的19种),vochysiaceae(七种),睫毛(三种亚属植物中的13种),Onagroceae(两个亚属的三种种类)和梳子族(一个亚家族中的八种)。所有叶绿体基因组都具有典型的四部分结构:大单拷贝区域(LSC),小单拷贝区域(SSC)和两个反相的重复区域(IRS)(图。2).42个牡丹科植物的叶绿体基因组长度范围为153,304 bp (Sarcopyramis Napalensis., MK994868.1)至157,991 bp (Astronia smilacifolia,MK994883.1),而19个Myrtaceae的样本范围从156,129 bp(Rhodomyrtus tomentosa,nc_043848.1)至160,459 bp(桉树祖母).7个Vochysiaceae样品的叶绿体基因组长度为160687 bp (erisma bracteosum,NC_043794.1)至171,315 BP(vochysia acuminata., NC_043811.1), 13个Lythraceae样品范围为152,214 bp (Lagerstroemia Excelsa.,nc_042896.1)至160,054 bp(Pemphis acidula., NC_041439.1); 3个羊草科样品的范围为159,396 bp (Ludwigia octovalvis, NC_031385.1)至165,779 bp (月见草villaricae,nc_030532.1)。最后,叶绿体基因组的八个样品中的叶绿体基因组的长度范围为159,750 bp(终端奥兰登斯,NC_043807.1)至161,773 BP(combretum littoreum.).横跨Myrtales的所有叶绿体基因组,家庭之间的塑料大小的差异为19,101bp,IR区域的差异为12,846bp,SSC区域的差异为8553 bp,LSC区域的差异为7558 bp。所有92个叶绿体基因组显示典型的四胞胎结构,包括由LSC(83,6691-91,249bp)分离的两个IR区(26,781-36,747bp)和SSC(11,150-19,703bp)区域(表1).此外,编码总共123-133个基因,其中106-116是单拷贝,IR区中重复17个基因。在独特的基因77-81中是蛋白质编码基因,29-31是TRNA基因,四个是RRNA基因。叶绿体基因组的总GC含量高度相似(36.9-38.9%),整个叶绿体基因组的平均GC含量为37%,而不同的区域具有稍可变的GC含量,具有LSC,SSC和IR的略微可变的GC含量34.7-37.3%,30.6-36.8%和39.7-43.5%(表1和2).
IR和SC地区之间的界限
总共分析并比较了SC和IR的边界区域在24个叶绿体基因组中的差异(来自NCBI的15个样品,九个新序列的叶绿体基因组覆盖了Myrtales内的16个亚壳/家族)。我们发现大多数叶绿体基因组具有相似的特性。LSC / IRB区域的23个叶绿体基因组的结位位于RPS19和rpl2.基因,而LSC / IRB区域的交界处Salpinga maranonensis(NC_031888.1)是唯一的,边界在rpl2.基因。除了月见草villaricae(NC_ 030532.1)IRB / SSC的边界是ccsA-ndhD。这ndhF在其他物种中均在IRb/SSC边界处检测到该基因。这ndhF11个物种的基因跨越了IRb/SSC的边界ndhF在SSC区域中完全发现12种,从边界到3和235bp之间。基因YCF1.在SSC / IRA边界处除了vochysia acuminata.(nc_043811.1)和月见草villaricae(NC_030532.1)。总共有20种YCF1.跨越了SSC/IRa之间的边界,这两个物种YCF1.完全在SSC中,远离边界的63至381磅,以及其中一个物种YCF1.完全在IRA 1063 BP远离边界。的基因rpl2.和trnH(rpl2.位于IRa区,距离边界53-139 bp,trnH位于LSC,距离边界0 - 216bp), 20个物种在IRa/LSC边界检测到。的基因RPS19和trnH(RPS19位于IRa区,距离边界0-3 bp,trnH位于LSC,距离边界1-41 bp), 3种物种在IRa/LSC边界检测到,rpl23和trnH在IRA / LSC边界中检测到Salpinga Maranensis.(NC_031888.1)(图。3.).
比较基因组分析和分化热点区
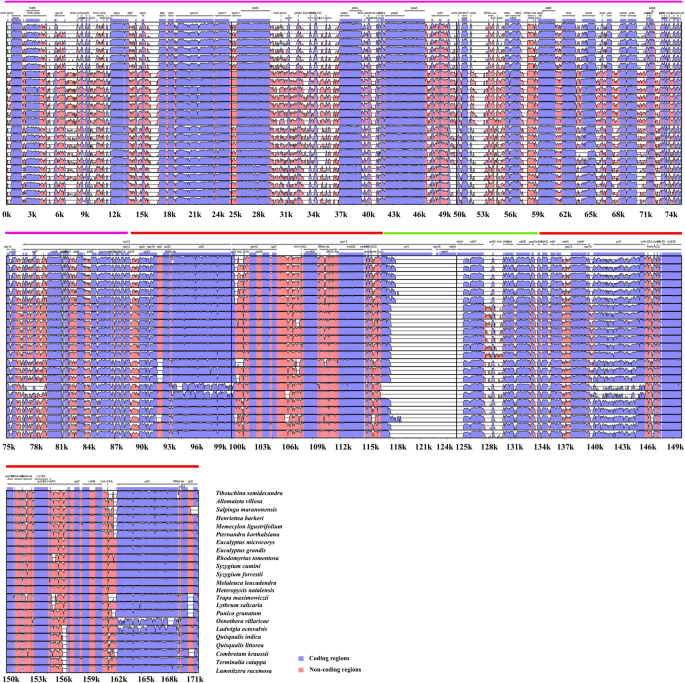
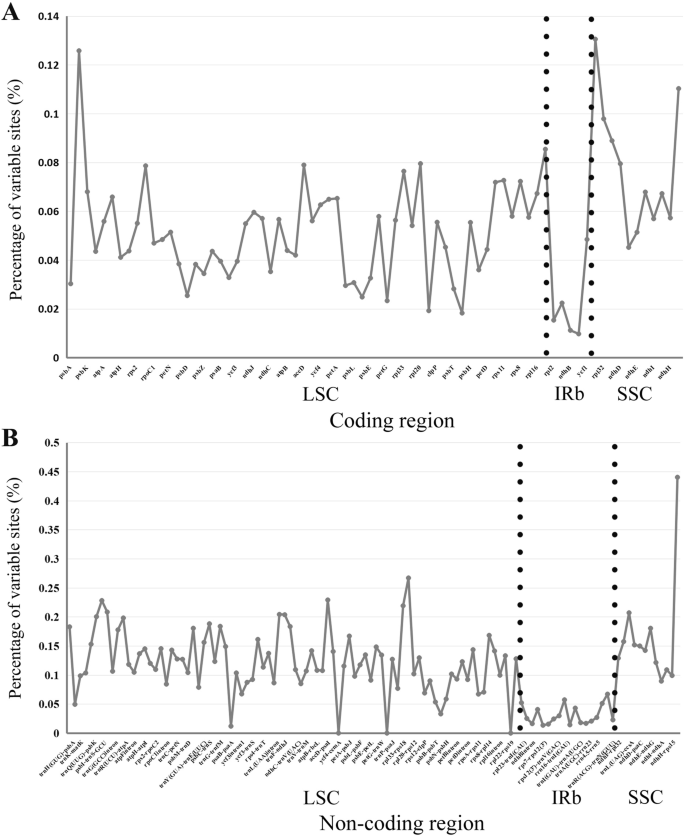
利用mVISTA软件对24个桃金娘叶绿体基因组(15个NCBI样本和9个新测序桃金娘叶绿体基因组的16个亚科/家系)的综合序列差异进行了分析诉渐尖用作参考。基因组宽对准揭示全局高序列相似性(> 90%同一性)(图。4.).LSC和SSC区域的序列发散程度高于反向重复区域。此外,我们还提取了188个区域来计算核苷酸变异性(TableS1).在编码区,变异最大的位点为MATK,RPOC2,ACCD,RPL20,NDHF,RPL32,CCSA那ndhD, 和RPS15;在非编码区域中,具有最大变化的基因位psbK-psbI, psbI-trnS (GCU), trnS (GCU)-trnG (GCC), trnR (UCU)-atpA, psbC-trnS (GCU), trnG-trnfM, trnF-ndhJ, ndhJ-ndhK, accD-psaI, rpl33-rps18, rps18-rpl20和rps15-ycf1.DNA条形码具有最大的核苷酸多样性,被认为是系统发育分析和植物鉴定的重点。5.).
系统发育的结果
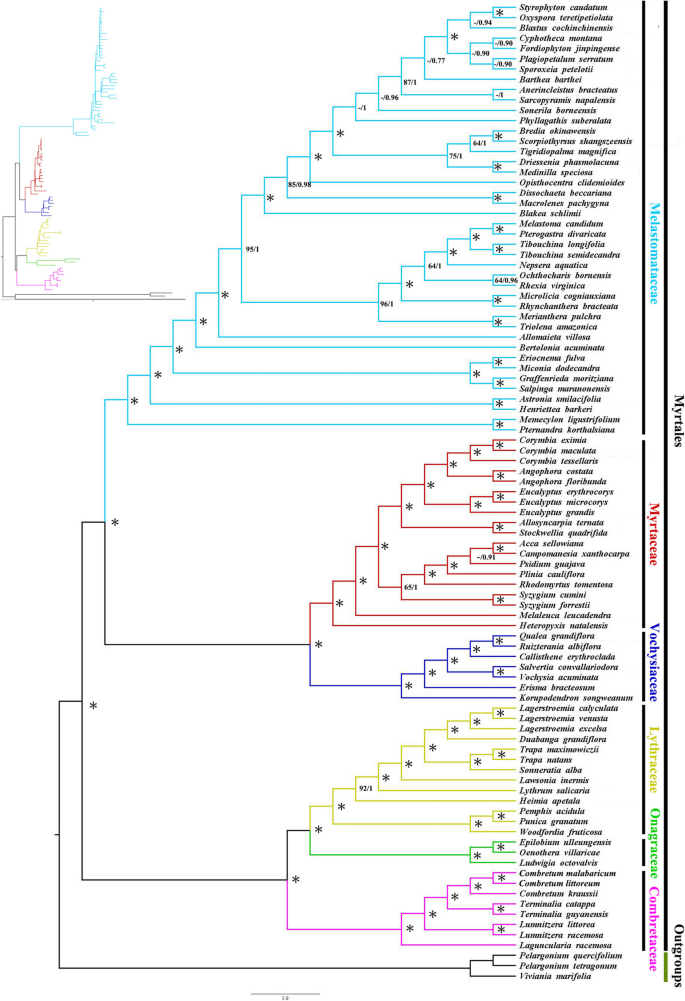
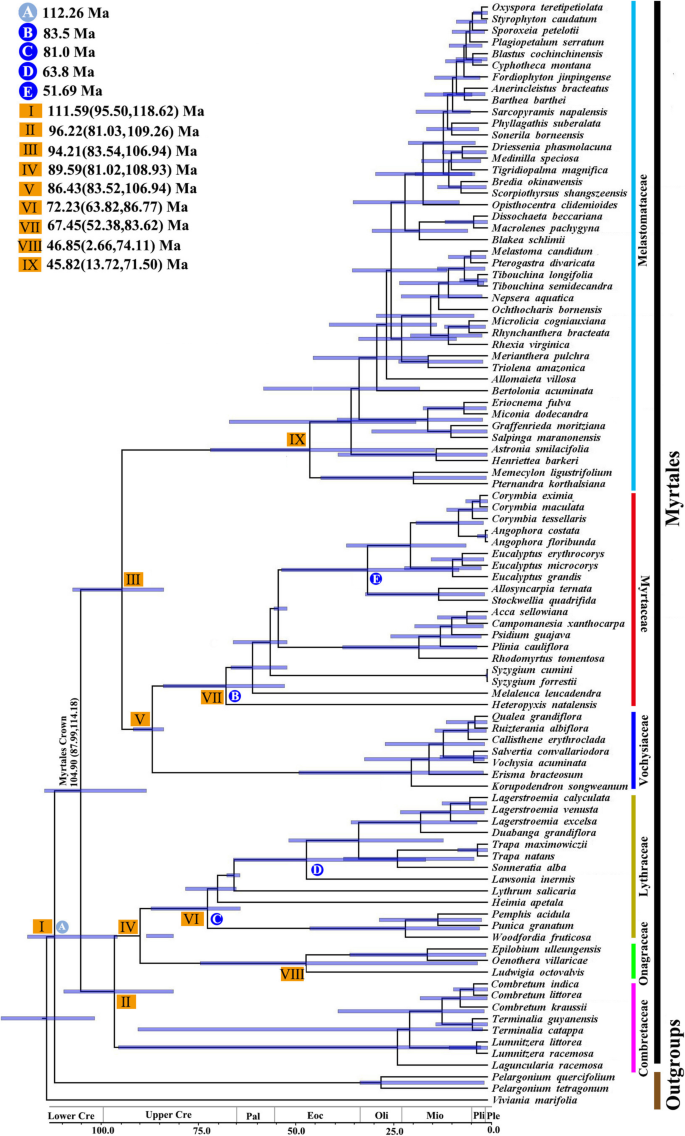
对完整叶绿体的ML和BI分析在每个节点上产生了几乎相同的强支持拓扑[ML bootstrap (BS) = 100%, Bayesian后验概率(PP) = 1](图。6.).山莨菪科、桃金娘科、野蔷薇科、淫羊藿科、蛇床子科等均支持单系分布,蛇床子科为淫羊藿科+蛇床子科的姊妹科(BS/PP = 100/1;(无花果。6.).野牡丹科(Melastomataceae)恢复为Myrtaceae + Vochysiaceae的姊妹科(BS/PP = 100/1)。在BS/PP = 100/1的支持下,野牡丹科(Melastomataceae) +桃金娘科(myrtaceaceae) +野蔷薇科(Vochysiaceae)的一个分支被恢复为菊科(combretacaceae) +羊草科(onagraceaceae) +羊草科(Lythraceae)的姊妹分支。此外,利用编码区(CR)、非编码区(NCR)、LSC、SSC和NO-IRa系统发育树(ML / BI)构建的系统发育树与全叶绿体推断的系统发育树具有相同的科级拓扑结构,具有较强的支持(图)S1那S2那S3.S4和S5).IRb区域构建的系统发育关系存在差异,Melastomataceae被解析为Myrtaceae + Vochysiaceae + Lythraceae + Combretaceae的姊妹科,Lythraceae被解析为Combretaceae的姊妹科,但支持度较低(图4)S6).此外,我们扩展了除孔的小组以构建麦芽腺的系统发育关系,并且强烈支持myrtales的系统发育关系(图S7).
桃金娘的分化时间估计
在细胞上的物种分析时间的野兽分析结果如图1所示。7..MYRTALES的皇冠年龄是104.90 mA(95%HPD = 87.88-114.18-114.18 mA),其中最近的常见祖先与大蕉为111.59 mA(95%HPD = 95.5.50-118.62 mA),在较低的白垩纪时期。基于野兽计时器,与onAgraceae +睫毛(Crown Group Age:89.59 mA,HPD = 81.02-108.93 mA)在上白垩纪的临曼诺曼年龄分叉96.22 mA(95%HPD = 81.03-109.26 mA)。MERTACEAE + vochysiaceae(皇冠组,95.82 mA,95%HPD = 13.72-71.50 mA)的皇冠组(冠组群)(冠组年龄:86.43 mA,95%HPD = 83.52-106.94 mA)在94.21 mA(95%HPD = 83.54-106.94 ma)在上层白垩纪的临曼诺曼。
讨论
质体结构比较和层序分歧热点
先前的研究表明,心血管中的叶绿体基因组的大小在120和180kb之间,IR区域的尺寸为20-30kb [29.].Myrtales的92个叶绿体基因组的尺寸范围为152,214-171,315bp,其中IR为26,781-36,747 bp。我们的研究结果表明,Myrtales的叶绿体基因组在被子植物中的细胞内基因组的较大末端。最大的塑料是在vochysiaceae中,最小的塑料在睫毛中。不同家庭之间的塑料长度的差异主要位于IR区域长度的差异。叶绿体基因组的总长度的变化通常与IR区域的膨胀和收缩有关[30.].所呈现的结果与发现的结果类似天竺葵属植物hortorum那Cryptomeria Fortunei.那天竺葵那Pisum一那蚕豆根尖, 和Erodium其中IR的大小增加,减少,甚至完全丢失[31.那32.那33.那34.].在心血管中,IR区域的高保守是常见的,对于稳定塑性基因结构非常重要[35.虽然报告了更改,但在一些早期发散的eddicots36.那37.].
虽然存在突变热点,但叶绿体的核苷酸含量相对稳定,基因结构高度保守。具有较高突变率的基因可以用作DNA条形码,以帮助区分特定分类单元内的可选物种[38.那39.]种质资源中的品种[40那41.].在这项研究中,我们使用MVISTA比较了24种Myrtales的整个叶绿体,并使用DNASP分析了74个编码基因和114个非编码区中可变基因座的百分比。类似于先前的结果,非编码区域的变化大于编码区域的变化[42.那43.].在菊科和田七, Myrtales的IR区域的变化小于SC区域的变化[44.那45.].以前的研究仅使用Myrtales的系统发育rbcl.未能确定该顺序的系统发育位置。我们的分析表明,核苷酸多样性rbcl.与其他基因座相比相对较低(PI <0.05)(图。5., 桌子S1),这有助于解释用该基因推断的系统发育中发现的低载体[7.].我们在编码区中检测到九个热点,在非分量区域中的12个热点,可用作未来研究的候选DNA条形码。这些可变区也可用于评估myRtales物种的系统发育关系和间隙差异。
Myrtales的系统发育关系
与以往基于少数叶绿体基因组片段的研究相比,我们基于桃金娘主要谱系(桃金娘共有6个科,其中桃金娘种类较多)的研究结果显示了桃金娘叶绿体全基因组的高度解析系统发育关系[2那6.那28.].代表主要家庭的六个主要赛车是完全解决的强势支持(图。7.).以往对桃金娘科的研究从形态学和分子生物学的角度对桃金娘科之间的系统发育关系有了更深入的了解,但对桃金娘科的定位尚未完全确定[2那6.那28.].combretacaceae的系统发育位置至关重要,因为它的位置直接影响Myrtales的年龄、扩散和变异情景的假设、物种多样化率和性状重建的特征[2].最近的系统发育研究使用有限数量的分类群和基因区域作为Combretaceae的占位符[7.那28.那46.那47.].桃金娘科(Myrtales)的质体基因组分析结果支持了桃金娘科(combretacaceae)与淫羊藿科(Onagraceae) +百合科(Lythraceae)的亲缘关系(BS = 100%, PP = 1;无花果。7.),其与我们以前的一些分子研究一致,+甘蓝的+ onAgraceae +睫毛+睫毛+ Melastomataceae + myrtaceae + vochysiaceae [18.那48.].我们的研究在科水平上取样不够全面,重建的系统发育关系包括9个科中的6个(缺少隐翅藓科、对虾科和阿尔扎藓科的样本)。但是,根据以往的研究,这并不影响我们对combretacaceae系统发育位置的确定。利用叶绿体全基因组构建系统发育关系,并利用多个叶绿体基因数据集(不包括IRa区、编码基因、非编码基因、LSC、SSC、IRb的叶绿体基因组)综合比较系统发育关系。我们还通过添加额外的分类群(Malvids分支内)来重建系统发育关系,为得到的系统发育树提供了更高的可信度[49.那50.]并确定梳子科的系统发育位置。进一步的研究应包括从野生群体中抽出更多个体,并获得更广泛的核数据,以确定我们的结果是否与来自核基因的结果一致。
分子的约会
生物地图估计通常表明,Myrtales起源于Gondwana [7.那28.那46.那51.那52.[所有主要茎谱的多样性在吉隆坡西部追溯到85-90 mA。分子约会的结果表明,MyRtales的皇冠组最有可能起源于下白垩纪的白蛋白(95%HPD = 87.88-114.18 mA)]。myrtales的估计发散时间(图。6.)与之前报道的数据接近(104.90 Ma,而111 Ma, Sytsma等人[28.];116.4 Ma, Berger等人[2];90.7 Ma, Thornhill等[53.])。但是,Gonçalves等人。[54.]使用来自122个叶绿体基因组的78个蛋白质编码基因,与四种Myrtales化石位点和二次校准点相结合,估计在上层白垩纪期间Myrtales的发散时间为125.5 mA(95%HPD = 130.9-120.3 mA)。在所有研究中,无能为力,不同的方法,分子数据和分类样品的规模,不同的方法,导致年龄估计差异导致差异的变化。我们的分析估计,Myrtales的主要谱系的多样性发生在60-90 mA [2那18.].在这一时期,Myrtales内的物种可能开始迅速分化,这与白垩纪-古近纪(K-Pg)边界之后,由于大规模灭绝和新栖息地的开放,许多物种经历了快速的多样化事件的普遍假设是一致的[55.那56.那57.].我们的研究结果表明,桃金猴主要茎系的物种多样性在坎帕纪末期有所增加,可能受到白垩纪冈瓦纳大陆分裂的影响[2].
结论
本研究对桃金娘的叶绿体基因组结构特征进行了分析比较,并推断了桃金娘的系统发育分化时间。桃金娘叶绿体基因组具有典型的四部分结构,包括77-81个蛋白编码基因、29-31个tRNA基因和4个rRNA基因,总长度为152,214-171,315 bp。我们发现了21个突变热点,这些热点可作为Myrtales未来系统发育研究的潜在DNA条形码。基于叶绿体全基因组和多个数据集的系统发育关系(Ml / BI)显示桃金娘及其科为单系,并支持combretacae和Onagraceae + Lythraceae为一个支系(BS = 100%, PP = 1)。重建Myrtales的分化时间表明,其冠为104.90 Ma (95% HPD = 87.88 ~ 114.18 Ma),与早白垩世阿尔比安(Albian)的Geraniales在111.59 Ma (95% HPD = 95.50 ~ 118.62 Ma)左右分化。桃金娘的种分化范围为60 ~ 90 Ma。这些叶绿体基因组对研究桃金娘的遗传多样性和物种进化具有重要意义,同时也为桃金娘的分类和系统发育研究提供了有用的信息。未来,我们将扩大包括核基因组在内的基因组取样,全面比较和讨论桃金娘物种的系统发育和进化。
方法
分类分类
采集了桃金娘3科7属9种植物的叶片材料,并将其保存在硅胶中。Combretum kraussiiHochst。,桉树祖母梅登前w·米尔,Melaleuca Leucadendra林恩。combretum littoreum.(英格兰)。Exell,syzygium forrestii.Merr。et erry,美国cumini(Linn。)梭子和Tibouchina Semidecandra.Cogn。从瑞丽植物园(云南省,中国; 23°52'至24°09'e,97°38'至98°05'n)收集。Combretum malabaricumLinn。和榄仁树属catappaLinn。从海南大学(中国海南省; 20°05'至20°06'e,110°33'至110°34'N)收集。九次新测序物种的采样由瑞丽植物园(云南省,中国)和海南大学(海南省)批准,并达到了地方政策要求。桌子3.表示新序列物种的详细凭证和局域信息。此外,83种,代表六个Myrtales和三个小组(viviania marifoliaNC_023259.1;Pelargonium tetragonum.,nc_031205.1;Pelargonium Quercifolium., NC_031203.1),详细信息如表所示1.我们还从NCBI下载了17个叶绿体基因组,包括6个不同的顺序作为外群构建Malvids的一个分支,以探索Myrtales的拓扑变化(Table .)S2).
DNA提取、测序和组装
我们使用改进的十六烷基三甲基溴化铵(CTAB)方法从干燥的叶子中提取高质量的DNA [58.].在加州大学戴维斯基因组中心(戴维斯,加利福尼亚州,美国戴维斯),在Agilent 2100生物分析仪上测定DNA质量。我们构建了与Illumina Truseq™纳米DNA样品准备试剂盒的插入尺寸的配对结束测序库,并在北京基因组学机构(BGI;深圳,中国)使用BGISEQ-500测序。用SOAPFilter_v2.2过滤原始读取的质量控制,下列参数:1)除以低质量读取(> 10%NS和/或> 40%低质量基础),2)删除PCR复制品和3)修剪适配器序列.我们选择了rbcl.基因拟南芥从NCBI(登录号:U91966)作为使用清洁读数与Novoplacty的每种物种的种子和组装叶绿体基因组[59.].将NOVOPlasty组装的最长的contig与NCBI数据库中保存的叶绿体进行比较,得到同源性最高的叶绿体基因组序列(最低要求:e-value < 10-7,同一性> 95%)作为我们的参考(Table .)3.),以便使用MITObim v1.8进行后续组装[60.].通过使用BWA MEM(HURROWS-WHAWER对准器)v0.7.17来评估组件的质量通过映射清洁读数。[61.]验证新组装的塑料的完整性[62.].
塑料诠释
首先使用geneous R11.0.4 (Biomatters Ltd., Auckland, New Zealand)对质体序列进行注释,然后使用Dual Organellar GenoMe Annotator (DOGMA)进一步注释[63.]改变基因边界。用Trnascan-SE1.21验证TRNA基因[64.].使用ConstellarGenomedraw v1.3.1绘制地图(在线提供:https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)[65.] (图。3.).所有质体序列均已上传到NCBI (Table .3.).
塑性对比分析和分子标记鉴定
在MVISTA程序上的Shuffle-Lagan模式下进行血管体积跨24种Myrtales物种(来自NCBI的15个样品和九个新序列的叶绿体基因组)在MVISTA程序上进行了Shuffle-Lagan模式(genome.lbl.gov / vista / index.shtml[66.;),使用的注释vochysia ascuminate.(NC_043811)作为参考。为了揭示未来物种鉴定研究中高度可变的区域,并评估可能显示不同进化模式的不同质体区域,我们在与MAFFT v7比对后,顺序提取了编码区和非编码区(包括基因间隔区和内含子)[67.]使用对准长度为200bp的标准,并且存在每个位点的至少一个突变。使用DNASP V5.10评估所选区域的核苷酸变异性[68.].使用Photoshop吸制了这24个Myrtales叶绿体的IR / SC边界地图。使用Unipro Ugene V1.32确认IR区域[69.].
系统发育分析
在95种物种中进行系统发育分析,使用viviania marifolia(NC_023259),Pelargonium tetragonum.(NC_031205),Pelargonium Quercifolium.(NC_031203)是基于之前的一项研究的外群体[2].使用Mafft V7对齐塑料序列[67.并在必要时进行人工检查。采用最大似然法(ML)和贝叶斯推理法(BI)构建叶绿体全基因组序列和叶绿体基因组负1个反向重复序列(No-IRa)的系统发育拓扑结构。为了评估其他假设,我们使用了最大似然(ML)和贝叶斯推理(BI)方法,利用完整的质体序列和整个质体减去一个反向重复序列(No-IRa)来推断系统发育拓扑。我们还纳入了其他数据集(即编码区、非编码区、LSC、SSC和IRb)进行分析。分子进化的最佳拟合模型(GTR + GAMMA+I)(表4.)使用JModeltest V2.1.7中的Akaike信息标准(AIC)确定[70].在RAXML-HPC V8.2.8中进行了最大似然分析[71],在CIPRES科学门户网站上有1000个引导复制[72].在MRBAYES v3.2中进行贝叶斯分析[73].同时进行两个独立的马尔科夫链蒙特卡罗链,每1000代采样一次。使用Tracer v1.7确定有效样本量(ESS > 200) [74[前25%的树木被丢弃,因为烧伤,从剩余的树木构成共识树以估计后验概率(PPS)。Figtree v1.4.4 [75用于可视化所得到的系统发育树。
发散时间估计
使用MrModelTest选择的GTR + GAMMA+I模型对完整的92个桃金娘质体数据集进行分析[76]在野兽v.1.8.4中[75]同时搜索最佳的树拓扑和估计节点年龄。利用Yule品种的速率估计谱系之间的分歧时间和利用轻松时钟的速率变化不相关的Lognormal模型。使用四个化石的校准点和一个二级校准点来限制Myrtales的冠节点年龄。(1)myrtaceidites(=略微植物)花粉[28.]在桃金娘科的花冠上放置一个优先权。这Myrtaceidites lisamae(83.5 ma)从加蓬,非洲在Santonian期间的化石[52.那77那78]被认为是Myrtaceae最古老的化石。因此,我们将Myrtaceae的茎与Lognormal平均值= 0,SD = 1.0和Offset = 83.5 mA。(2)在Chamelaucioideae的Myrtaceae中,我们放置了化石eucalyptus fenguelliana.(51.69 ma)在阿根廷楚鲁省拉古纳德·普尔公司的早期eocene日期为期[79那80].我们将Chamelaucioideae的茎设置为Lognormal平均值= 0,SD = 1.0和偏移= 51.69 mA。(3)基于花粉化石的Lythraceae的茎被设定为Lognormal平均值= 0,SD = 1.0和偏移= 81.0mALythrum elkensis美国怀俄明州晚白垩世(早Campanian, 82-81 Ma) Lythrum/Peplis的化石[80那81].(4)我们使用的是有记载的最早的木化石Sonneratioxylon preapetalumAwasthi [82](大年,67.3-63.8 Ma) [81]限制Trapoideae的节点。我们将阀杆设置为63.8 mA,逻辑平均值等于0,基于Li等人的结果,标准偏差为1.(5)。[18.[Myrtales和Carambycidales的冠长历时为112.26 mA,Myrtales + GerAniales的皇冠节点年龄受到约束为112.26 mA,正常的先前和SD = 5.九个跑为每代100百万代的持续时间总计900每1000代参数采样百万世代。使用示踪剂V1.6测定有效的样品尺寸(> 200)[75]并将前25%的样品被抛弃为燃烧。treeannotator v1.8.0 [75]用于产生最大的落叶思克可信度计时器,显示出具有95%最高后密度(HPD)间隔的平均发散时间估计。Figtree v1.4.4 [75]用来可视化产生的发散时间。
数据和材料的可用性
本研究中使用的所有序列可从国家生物技术信息中心(NCBI)(登录号:MT700492-MT700490;见其他表2)。
缩写
- 双:
-
贝叶斯推断
- CTAB:
-
十六烷基三甲基溴化物
- DnaSP:
-
DNA序列多态性
- 红外:
-
反向重复
- LSC:
-
大单副本
- GTR:
-
一般时间可逆
- ML:
-
最大似然
- PI:
-
系统发育信息
- 核糖体rna:
-
核糖体RNA
- SSC:
-
小单拷贝
- tRNA:
-
转移RNA.
参考
- 1。
桃金爱故事的顺序:界限、变异和关系。陈建平。2004;21(3):1 - 7。https://doi.org/10.2307/2399158.
- 2。
Berger BA, Kriebel R, Spalink D, Sytsma KJ。分化时代、历史生物地理学和桃金娘物种形成率的变化。分子系统进化。2016;95:116-36。https://doi.org/10.1016/j.ympev.2015.10.001.
- 3。
被子植物系统学。《植物分类学报》2016年第1期(1 - 6页)
- 4.
[10]谭峰,石松,杨忠,孙刚,王勇。基于质体、核基因和间隔序列的combretoidae (combretoidae)系统发育关系。植物学报,2002;https://doi.org/10.1007/s10265-002-0059-1.
- 5。
桃金娘属植物的解剖与分类。陈建平。2004;21(3):1 - 7。https://doi.org/10.2307/2399162.
- 6。
Conti E,Litt A,Sytsma KJ。Myrtales的界定及其与其他ROSID的关系:来自RBCL序列数据的证据。我是J机器人。1996年; 83(2):221-33。https://doi.org/10.1002/j.1537-2197.1996.tb12700.x.
- 7。
Conti E, Litt A, Wilson PG, Graham SA, Briggs BG, Johnson L等。桃金娘家系间关系:分子系统发育和形态进化模式。系统机器人。1997;22(4):629 - 47。https://doi.org/10.2307/2419432.
- 8.
Thornhill啊,Ho Sy,KülheimC,清脆MD。解释使用日期分子系统的Myrtaceae的现代分布。mol phylocyet evol。2015; 93:29-43。https://doi.org/10.1016/j.ympev.2015.07.007.
- 9.
彭东辉,张启新,黄廷堂。中国牡丹科观赏植物种质资源及福建省分布调查。中国景观设计师。2007;11:92-7。
- 10。
Granato D,Nunes DS,Barba FJ。食品化学,生物学,营养,药理学与统计统计功能食品的综合策略:提案。趋势食品掌握技术。2017; 62(填写):13-22。
- 11.
关键词:紫苔属植物,鞣花单宁,结构特征,生物学特性中国生物医学工程学报,2010;https://doi.org/10.3390/ijms11010079.
- 12.
Jansen RK,Cai Z,Raubeson La,Daniell H,De Pamphilis CW,Leebens-Mack J,等。81种塑性基因组的81个基因分析了Angiospers中的关系,并识别了基因组的进化模式。PROC NATL ACAD SCI。2007; 104:19369-74。
- 13。
Moore MJ,Dhingra A,Soltis PS,Shaw R,Farmerie Wg,Folta Km,等。高速准确的脱霉素血浆基因组织。BMC植物BIOL。2010; 6:1-13。
- 14。
杨y,周t,段d,杨j,冯l,赵g。五栎种类完全叶绿体基因组的比较分析。前植物SCI。2016; 7:573-5。
- 15。
吕荣生,李平,邱永祥。三种心莲(百合科)的叶绿体全基因组:比较基因组和系统发育分析。植物科学与技术,2017;
- 16。
Niu YT, Florian J, Barrett RL, Ye JF, Zhang ZZ, Lu KQ, et al. .将叶绿体全基因组序列与目标位点数据和形态相结合,解决三棱藓属(caprifolaceae)的物种限制。分子系统进化。2018;129:15-26。https://doi.org/10.1016/j.ympev.2018.07.013.
- 17。
巨桉的质体和线粒体基因组。BMC基因组学。2019;20:1471 - 2164。
- 18.
李HT,易Ts,高LM,MA PF,张T,杨杰,等。Agenperms的起源和侏罗纪差距的难题。Nat植物。2019; 5(5):461-70。https://doi.org/10.1038/s41477-019-0421-0.
- 19.
熊安思,彭瑞荣,庄健,高峰,朱斌,付晓燕,等。叶绿体基因组中的基因复制、转移和进化。Biotechnol放置2009;27(4):340 - 7。https://doi.org/10.1016/j.biotech.2009.01.012.
- 20.
Rogalski M,Do Nascimento Vieira L,Fraga HP,Guerra MP。园艺种类的塑性基因组学:植物群体遗传学,演化和生物技术的重要性和应用。前植物SCI。2015; 6:586。
- 21。
Reginato M, Neubig KM, Majure LC, Michelangeli FA。首个完整质体基因组在结构上高度保守。同行j . 2016; 4: e2715。https://doi.org/10.7717/peerj.2715.
- 22。
acta botanica botanica(云南植物研究),2011,34(5):564 - 564 .顾超,董斌,徐玲,Tembrock L,郑松,吴忠。桃金娘海星叶绿体全基因组及桃金娘属植物的比较分析。分子。2018;23(4):846。https://doi.org/10.3390/molecules23040846.
- 23。
GU C,MA L,Wu Z,Chen K,王Y.222溶质物种叶绿体基因组的比较分析:Myrtales中系统发育关系和基因组进化的推论。BMC植物BIOL。2019; 19(1):281。https://doi.org/10.1186/s12870-019-1870-3.
- 24。
林伟,黄杰,薛敏,等。月季叶绿体全基因组的鉴定,罗莎对(Rosaceae:Rosa)。线粒体DNA Part B Resour。2019; 4(2):2984-5。
- 25.
薛泽,薛杰,维多利托罗纳M,马吉。Trapa MaximoWiCzii Korsh(Trapaceae)的完整叶绿体DNA序列,以及与其他Myrtales物种的比较分析。aquat bot。2017; 143:54-62。https://doi.org/10.1016/j.aquabot.2017.09.003.
- 26.
1 . Yang JY, Pak JH, Kim SC. The complete plome sequence of tribus takesimensis (Rubus) in Ulleung Island, Korea: insights into angenetic derived species in Rubus (rosacae). acta botanica yunbotanica(云南植物研究),2011,32(5):574 - 584。基因。2018;668:221-8。https://doi.org/10.1016/j.gene.2018.05.071.
- 27.
Rodrigues NF, Balbinott N, Paim I等。桃金娘科六种新热带种叶绿体全基因组的比较分析。中国生物医学工程学报,2014;
- 28.
Sytsma Kj,Litt A,Zjhra Ml,Chris Pires J,Nepokroeff M,Conti E等人。片断,时钟和大陆:南半球历史和生物地图分析Myrtaceae,Vochyseae和亲属。int j植物sci。2004; 165(S4):S85-S105。https://doi.org/10.1086/421066.
- 29.
张涛,方勇,王鑫,邓鑫,张鑫,胡松,等。湿化虫的完整叶绿体和线粒体基因组序列:植物细胞器基因组进化的洞见。《公共科学图书馆•综合》。2012;7 (1):e30531。https://doi.org/10.1371/journal.pone.0030531.
- 30.
王伟,王文杰。3种lenoideae(浮萍)叶绿体基因组总DNA的高通量测序。《公共科学图书馆•综合》。2011;6 (9):e24670。https://doi.org/10.1371/journal.pone.0024670.
- 31.
Chumley TW,Palmer JD,Mower JP,Fourcade HM,Calie PJ,Boore JL等。颅骨×霍松松的完全叶绿体基因组序列:陆地植物最大和最高度重排叶绿体基因组的组织与演化。mol Biol Evol。2006; 23(11):2175-90。https://doi.org/10.1093/molbev/msl089.
- 32。
Guisinger MM, Kuehl JV, Boore JL, Jansen RK。被子植物天竺葵科叶绿体的极端重构:重排、重复和密码子使用。中国生物医学工程学报。2011;28(1):583-600。https://doi.org/10.1093/molbev/msq229.
- 33。
Hirao T,Watanabe A,Kurita M,Kondo T,Takata K.完全核苷酸序列的Cryptoeria japonica D. Don。叶绿体基因组和比较叶绿体基因组学:针叶种类的多样化基因组结构。BMC植物BIOL。2008; 8(1):1-20。
- 34。
胡锦涛YJ。质体基因组结构和质体基因。植物生理学通讯1985;2:65-71。
- 35。
MaréchalA,Brisson N.重组和植物细胞器基因组稳定性的维持。新植物。2010; 186(2):299-317。https://doi.org/10.1111/j.1469-8137.2010.03195.x.
- 36。
唐尼SR,詹森RK。从蜂群的整个质体的比较分析:反向重复序列的扩张和收缩,DNA从线粒体到质体的转移,以及高度分化的非编码区域的鉴定。系统机器人。2015;40(1):336 - 51。https://doi.org/10.1600/036364415X686620.
- 37。
关键词:Soltis PS, Soltis DE, Zhao T,等。几乎所有早分化真子属的18个完整叶绿体的系统基因组和结构分析,包括被子植物范围内的IR基因含量进化分析。分子系统进化。2016;96:93-101。https://doi.org/10.1016/j.ympev.2015.12.006.
- 38。
匡王,吴慧,王英,高LM,张Sz,Lu L.完全氯化物基因组碱磷酸盐基因组序列(MagnoliCeae):DNA条形码和群体遗传的含义。基因组。2011; 54(8):663-73。https://doi.org/10.1139/g11-026.
- 39。
关键词:木槿,叶绿体,基因组,突变区基因组学,2020;112(1):581 - 91。
- 40.
acta photonica sinica, 2011, 38(5): 689 - 693 .葛勇,董鑫,吴斌,王宁,陈丹,陈浩,等。美洲Persea生态小种6个叶绿体基因组的进化分析:序列差异和系统发育关系的洞见。《公共科学图书馆•综合》。2019;14 (9):e0221827。https://doi.org/10.1371/journal.pone.0221827.
- 41.
关键词:十字花科,龙胆,叶绿体基因组,比较分析国际分子科学杂志。2018;19(7):1962。https://doi.org/10.3390/ijms19071962.
- 42.
佩里,沃尔夫·哈哈。豆类叶绿体DNA中的核苷酸取代速率取决于倒置重复的存在。J Mol Evol。2002; 55(5):501-8。https://doi.org/10.1007/s00239-002-2333-y.
- 43.
黄鹤,施,刘义,毛苏,高lz。通过高通量测序测定的十三氯化胆血糖蛋白基因组序列:基因组结构和系统发育关系。BMC EVOL BIOL。2014; 14(1):151。
- 44.
风扇,吴y,阳j,shahzad k,李Zh。Dipsacales物种的比较叶绿体基因组学:静脉曲张变异,自适应演化和系统发育关系的见解。前植物SCI。2018; 9:689。https://doi.org/10.3389/fpls.2018.00689.
- 45。
董伟,刘慧,徐超,等。叶绿体基因组策略设计分类单元特异性DNA微条形码:以人参为例。BMC麝猫。2014;15(1):1 - 8。
- 46。
约翰逊拉斯,布里格斯BG。myrtales和myrtaceae-a phylo到期分析。安莫僵尸加德。1984; 71(3):700-56。https://doi.org/10.2307/2399159.
- 47。
MagAllóns。使用化石在分子约会中打破长分支:放松时钟应用于Angiospers的起源的比较。系统中的生物学。2010; 59(4):384-99。https://doi.org/10.1093/sysbio/syq027.
- 48。
王秀琴,宋伟伟,肖俊杰。基于叶绿体基因组的桃金娘及其相关类群的系统发育。Guihaia植物。2021;41:68 - 80。https://doi.org/10.11931/guihaia.gxzw201906024.
- 49.
Smith Sa,Beaulieu JM,Donoghue MJ。比较生物学的兆文学方法:超级和超大方法的替代方案。BMC EVOL BIOL。2009; 9(1):1-12。
- 50.
Sanderson MJ,McMahon MM,钢铁M.具有不完整的分类群的系统母组织:推理的限制。BMC EVOL BIOL。2010; 10(1):1-13。
- 51.
Rutschmann F, Eriksson T, Salim KA, Conti E.评估分子定年的校准不确定性:化石对替代校准点的分配。系统医学杂志。2007;56(4):591 - 608。https://doi.org/10.1080 / 1063515070/10635150701491156..
- 52.
中国现存被子植物花粉化石记录。机器人启1981;47(1):1 - 142。https://doi.org/10.1007/BF02860537.
- 53.
花粉化石对校准系统发育的分子钟年代有用吗?桃金娘科植物的比较研究。分子系统进化研究。2012;63(1):15-27。https://doi.org/10.1016/j.ympev.2011.12.003.
- 54.
Gonçalves DJP, Shimizu GH, Ortiz EM, Jansen RK, Simpson BB。历史生物地理学揭示了新热带地区植物进化的一个意想不到的视角。中国生物医学工程学报,2010;https://doi.org/10.1002/AJB2.1502.
- 55.
Jablonski D.大规模灭绝和宏观调查。古生物学。2005; 31(SP5):192-210。https://doi.org/10.1666/0094-8373(2005)031 [0192:meam]2.0.co;2.
- 56。
Schulte P,Alegret L,Arenillas I,Arz Ja,Barton PJ,Bown Pr等。肾小瓣突口突起和大规模古近缘边界的质量消光。科学。2010; 327(5970):1214-8。https://doi.org/10.1126/science.1177265.
- 57。
翟W,段x,张r,guo c,li l,xu g等。叶绿体基因组数据为毛茛科的系统发育和演化提供了新的和强大的见解。mol phylocyet evol。2019; 135:12-21。https://doi.org/10.1016/j.ympev.2019.02.024.
- 58。
多伊尔·JJ,多伊尔·JL。一种快速提取新鲜叶片组织DNA的方法[R],第19卷;1987.把页。
- 59。
基于全基因组数据的细胞器基因组重新组装。核酸研究。2017;45(4):e18。
- 60。
Hahn C,Bachmann L,Chevreux B.直接从基因组下一代测序重建线粒体基因组读取 - 一种诱饵和迭代映射方法。核酸RES。2013; 41(13):E129。https://doi.org/10.1093/nar/gkt371.
- 61。
Li H.用BWA-MEM对准序列读取,克隆序列和组装折叠。arxiv预印秋季。2013; 1303:3997。
- 62。
刘立,魏杰,杨特等。植物园的分子数字化:来自瑞利植物园689血管植物种类的高深综合测序。傻瓜。2019; 8(4):GIZ007。
- 63。
Wyman SK, Jansen RK, Boore JL。用DOGMA自动注释细胞器基因组。生物信息学,2004;20(17):3252 - 5。https://doi.org/10.1093/bioinformatics/bth352.
- 64.
Peter S,Angela NB,托德ML。用于检测TRNA和SNORNAS的TRNASCAN-SE,SNOSCAN和SNOGPS Web服务器。核酸RES。2005; 33:686-9。
- 65.
Lohse M,Drechsel O,Kahlau S,Bock R. Uniterellargenomedraw-一套用于生成塑体和线粒体基因组的物理图和可视化表达数据集的工具。核酸RES。2013; 41(W1):W575-81。https://doi.org/10.1093/nar/gkt289.
- 66.
Frazer Ka,Pachter L,Poliakov A,Rubin Em,Dubchak I. Vista:用于比较基因组学的计算工具。核酸RES。2004; 32(SUPPLE_2):W273-9。
- 67.
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30(4):772-80。https://doi.org/10.1093/molbev/mst010.
- 68.
Librado P, Rozas J. dnnasp v5:一个全面分析DNA多态性数据的软件。生物信息学。2009;25(11):1451 - 2。https://doi.org/10.1093/bioinformatics/btp187.
- 69。
玫瑰R,Golosova O,Sukhomlinov D,Tiunov A,Prosperi M.柔性设计多种偏心神经分类管道与UGENE。生物信息学。2019; 35(11):1963-5。https://doi.org/10.1093/bioinformatics/bty901.
- 70.
Santorum JM,Darriba D,Taboada GL,Posada D. JModeltest。ORG,在云中选择核苷酸替代模型。生物信息学。2014; 30(9):1310-1。https://doi.org/10.1093/bioinformatics/btu032.
- 71.
Stamatakis a . RAxML version 8:用于系统发育分析和大型系统发育后分析的工具。生物信息学。2014;30(9):1312 - 3。https://doi.org/10.1093/bioinformatics/btu033.
- 72.
Miller Ma,Pfeiffer W,Schwartz T.创建Cipres科学门户对大型系统发育树的推导。在:网关计算环境研讨会;2010. p。1-8。
- 73.
ronquist f,huelsenbeck jp。MRBAYES 3:混合模型下的贝叶斯系统发育推理。生物信息学。2003; 19(12):1572-4。https://doi.org/10.1093/bioinformatics/btg180.
- 74.
Rambaut A,Drummond AJ,谢D,Baele G,Suchar。跟踪器1.7贝叶斯文学性衰减后的后序列。系统中的生物学。2018; 67(5):901-4。https://doi.org/10.1093/sysbio/syy032.
- 75.
德拉蒙德AJ,Suchard Ma,谢D,Rambaut A. Beasesian Phylogenetics,Beauti和Beast 1.7。mol Biol Evol。2012; 29(8):1969-73。https://doi.org/10.1093/molbev/mss075.
- 76.
Posada D. jModelTest:系统发育模型平均。中国生物医学工程学报。2008;25(7):1236 - 1236。https://doi.org/10.1093/molbev/msn083.
- 77.
Boltenhagen E.花粉和孢子Senoniens du Gabon。《Micropaleontol。1976;3:1-21。
- 78.
Herngreen GFW。巴西阿拉戈斯的钻孔3-Pia-10-Al状态的上塞诺尼亚花粉组合。花粉孢子。1975; 17:93-140。
- 79.
MA, Hermsen EJ, Zamaloa MC, Nixon KC, González CC, Wilf P,等。已知最古老的桉树大化石来自南美洲。《公共科学图书馆•综合》。2011;6 (6):e21084。https://doi.org/10.1371/journal.pone.0021084.
- 80。
Grímssonf,zetter r,hofmann cc。Lythrum和Peplis来自北美晚期和新生代和欧亚亚洲:新的证据表明Lythraceae内早期多样化。我是J机器人。2011; 98(11):1801-15。https://doi.org/10.3732/ajb.1100204.
- 81。
格雷厄姆sa。化石记录在睫毛内。BOT Rev. 2013; 28:410-20。
- 82。
一种产于印度南部第三纪的海桑属的化石木材。古。1968;17:254-7。
确认
我们想要感谢匿名审稿人对我们的手稿提出的深思熟虑的意见和建设性的建议。
资金
本研究由海南大学启动基金(kyqd1633)资助。样本采集和测序分析的费用由该资金来源提供。
作者信息
从属关系
贡献
XFZ进行了所有实验,分析了数据并写了稿件。HXW和ZXZ协助实验。JBL帮助修改稿件。HFW计划和指导研究并修改了稿件。请提及所有作者阅读并批准稿件。
通讯作者
伦理宣言
伦理批准并同意参与
不适用。
同意出版
不适用。
利益争夺
作者们宣称他们没有相互竞争的利益。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1-S6。
分别是最大似然法和贝叶斯推理法基于:编码基因推断的系统发育关系;非编码基因座;LSC(大副本);SSC(小副本);NO-IRa数据集(数据集组成在方法中描述)和IRb(倒重复区域)。支持值为最大似然自助支持/贝叶斯后验概率。桃金娘的家族用不同的颜色表示。对于每个图,插图显示与系统发育图相同的树(除了IR数据集构建的系统发育关系有一些不一致)。分支上的支撑值为bootstrap值/贝叶斯后验概率:“*”表示100% /1.0支撑值,“-”表示bootstrap值/贝叶斯后验概率小于60 / 0.7。桃金娘的家族用不同的颜色来代表。 The small picture in the upper left corner is the ML phylogenetic tree (showing branch length).
附加文件2:图S7。
由Myrtales的92个完全叶绿体基因组分析和使用最大可能性(mL)的最佳系统发育树。支持值是最大似然引导支持后部概率。桃金娘的家族用不同的颜色表示。.分支上的支持值是引导值,“*”表示100%支持值,“ - ”表示引导值小于60. Myrtales的系列由不同的颜色表示。左上角的小图片是ML系统发育树(显示分支长度)。
附加文件3:表S1。
188个Myrtales同源位点的Eta、Pi值、H、Hd、PICs、长度和对齐长度。
附加文件4:表S2。
物种信息和叶绿体基因群Genbank登录本研究中的小组数。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
张,XF。那Landis, J.B., Wang, HX.等等。桃金娘叶绿体基因组结构与分子年代的比较分析。BMC植物BIOL.21,219(2021)。https://doi.org/10.1186/s12870-021-02985-9
已收到:
公认:
发表:
关键词
- myrtales.
- 塑料
- 基因组结构
- 种系发生
- 自适应演化