抽象的
背景
长终端重复转回转换(LTR-RES)是重复的DNA序列,其构成基因组的很大一部分。测序技术和序列组装策略的改善已经达到了与过去的那些更高的基因组序列,特别是与重复DNA序列相关的更大的可靠性。
结果
在这项研究中,我们分析了榕辣海参L.,利用第三代测序技术获得,最近发布,对全长LTR-REs的完整补体进行表征,研究其在无花果基因组进化过程中的动态。共鉴定全长元素1867个。
那些属于吉普赛人超级家族是最丰富的;在这些中,Chromovirus / Tekay血统是最代表的。为了Copia总科,啤酒是最丰富的血统。测量每个元素的估计插入时间表明,平均而言,伊凡娜和Chromovirus / Tekay最年轻的血统是Copia和吉普赛人Superfamilies分别。除了一些元素外,大多数元素在转录中均为无活性,两者在暴露于非生物应激的植物中,除了一些元素,主要是属于Copia /前者啤酒血统。建立了一个元件失活与邻近基因失活之间的关系。
结论
本研究提供了一套具有高度可靠基因组序列的植物中与LTR-REs相关的基因组动态信息。图LTR-REs在数量和插入时间上具有高度异质性,只有少数元件具有转录活性。总的来说,数据表明插入时间和一个元素的丰度之间有直接关系,插入时间(或丰度)和转录之间有反比关系,至少对于CopiaLTR-REs。
背景
无花果树(榕辣海参L.)属于桑科,是一种落叶果树,被认为是最古老的驯化树种之一[1].在过去的几十年里,由于无花果的营养价值和经济价值,人们对它的兴趣大大增加了[2,3.].F. Carica.种植遍及地中海地区、中东和亚洲。这些地区在夏季受到高温带气候的影响,由于水资源的减少,土壤盐分增加[4].与其他果树相比,无花果对盐水应激的抗性显微抗性[5,6].最近,F. Carica.基因组已经完全测序[1]显示总共〜333MBp,含有37,840个基因的13个伪染色体,主要由重复序列组成,重复序列的长终端重复转回转换(LTR-RES)构成绝大多数[1].然而,仍然缺乏对无花果LTR-RE的综合分析。
Retrotroansposons(RES)通常在所有真核生物中普遍存在,并构成基因组的大部分,特别是在植物物种中[7].它们可以在染色体之间移动,因此,它们的数量成倍增加到宿主植物的基因组中。事实上,REs通过“复制粘贴”机制进行复制,利用RNA中间体的表达转化为cDNA,然后再插入到染色体的不同位置。植物物种中最丰富的REs序列显示了位于多蛋白区两侧的两个长末端重复序列(LTRs),包含复制该序列所需的编码结构域,如蛋白酶、逆转录酶、RNAse H和一个整合酶,另外还有一个编码病毒样颗粒蛋白的GAG结构域[7,8].大多数植物的LTR-REs以两个主要超级科为代表,Copia和吉普赛人,其主要区别在于序列分歧和多蛋白结构域的组织。根据序列相似性,这些元素可以区分为几个谱系[7,9,10].
植物基因组的RE成分会发生更替[11,12];事实上,REs的数量可能会在相对较短的时间内增加。然而,REs也可以通过不平等的同源和不合理的重组从基因组中去除[13,14].
Retrotransposons在物种的演变中发挥着重要作用,因为它们的流动性产生遗传变异[15].它们可以通过非法重组促进染色体重排[13]在同一染色体上或在不同染色体上的位置之间[16].此外,RES可以在基因内或附近插入,改变其编码序列或其拼接图案[17].也许更重要的是,在基因附近插入或去除一个RE可能会改变其转录调控,改变其对不同刺激的表达率[18,19,20.,21.].
由于其迁移率的潜在有害影响,res通常由宿主灭活,例如,通过DNA甲基化[22.,23.].因此,在一个位点插入一个RE可能导致该插入位点的表观遗传设置的改变[17].事实上,已知REC调节基因组和染色质组织和结构的表观遗传环境[17].
然而,LTR-REs可以避开沉默机制,进入新的位点,导致潜在的基因变化。例如,LTR-REs的激活已经在许多植物物种中被报道;在许多情况下,RE表达的调控依赖于外部刺激,如那些与生物和非生物胁迫相关的刺激[24.,25.,26.,27.].
在使用高通量基因组序列获得第二代测序技术,用相对较短的DNA序列进行组装,组装LTR-REs可以如此复杂的REs是重复的(在许多情况下,高度重复)基因组中,迄今为止超过一个单一的阅读。此外,在某些情况下,组装的序列可能由多个重复元素的副本折叠在一起产生的序列读取组成,导致一致而不是真正的序列。
第三代测序(TGS)技术[28.,能够产生非常长的序列读取量,这使得高质量的基因组组装得以生产[29.,30.,31.]克服了许多与短读取测序组件相关的问题,例如重复序列的分辨率[22.,32.].
最近,一种高质量的基因组序列榕辣海参简历。Dottato [1]是利用太平洋生物科学公司(PacBio, Menlo Park, CA, USA)开发的单分子实时(SMRT)测序技术生产的,首次尝试通过长读装配对该杂合子物种的基因组进行测序,从而提供了优质的基因组资源。事实上,PacBio技术可以成功地解决大多数基因组的复杂重复部分,如那些植物,通过跨越完整的重复区域与独特的序列。获得这样一个连续的参考基因组组装是一个关键的先决条件,以正确的鉴定重复的从头方法。
在以前的工作中[1],对重复序列进行鉴定并进行初步鉴定。重复序列被用来掩盖组装,从而获得与假定重复区域相关的碱基的总数量,但没有对单个全长元件进行分析。在这里,我们提出了一个全面的研究无花果LTR-REs,与鉴定和完整的长度元素的特征。特别是,我们在鉴定和结构特征后,分析了每个分离元件在基因组中的丰度和插入时间F. Carica..此外,我们利用转录组学方法分析了LTR-REs在非生物胁迫中的表达,即同一品种(Dottato)的无花果叶片暴露于短时间和长时间的盐胁迫[33.].事实上,LTR-RE表达代表了逆转录转座子激活的第一步,可能对物种的遗传结构产生影响。最后,对所有LTR-REs的丰度、年龄和转录活性等数据进行了相关分析,为精确植物系统中REs的动态提供了一个全局的视角。
结果
全长LTR-RES隔离和结构特征
通过结构分析,我们鉴定出了1867个独特的全长长末端重复反转录转座子(LTR-REs)。
两个可用的图形基因组组件之间的比较,使用第三代排序技术产生的图中[1]和以前的基于ngs的组件[34.]的结果表明,使用长读测序,检索到的全长元素的数量要高得多。事实上,与1867相比,只有20个全长LTR-REs可以从基于ngs的组件中检索到。采用90%的序列相似性阈值对1887个LTR-REs进行聚类。在1867个LTR-REs中,只有16个LTR-REs与基于ngs装配的元素聚类,进一步显示了第三代测序技术在重复装配方面的巨大进步。
总的来说,1867个LTR-REs中有1010个显示了所有的功能域,而其余857个元素缺乏一个或多个功能域,因此可以认为它们是转位非自治的。
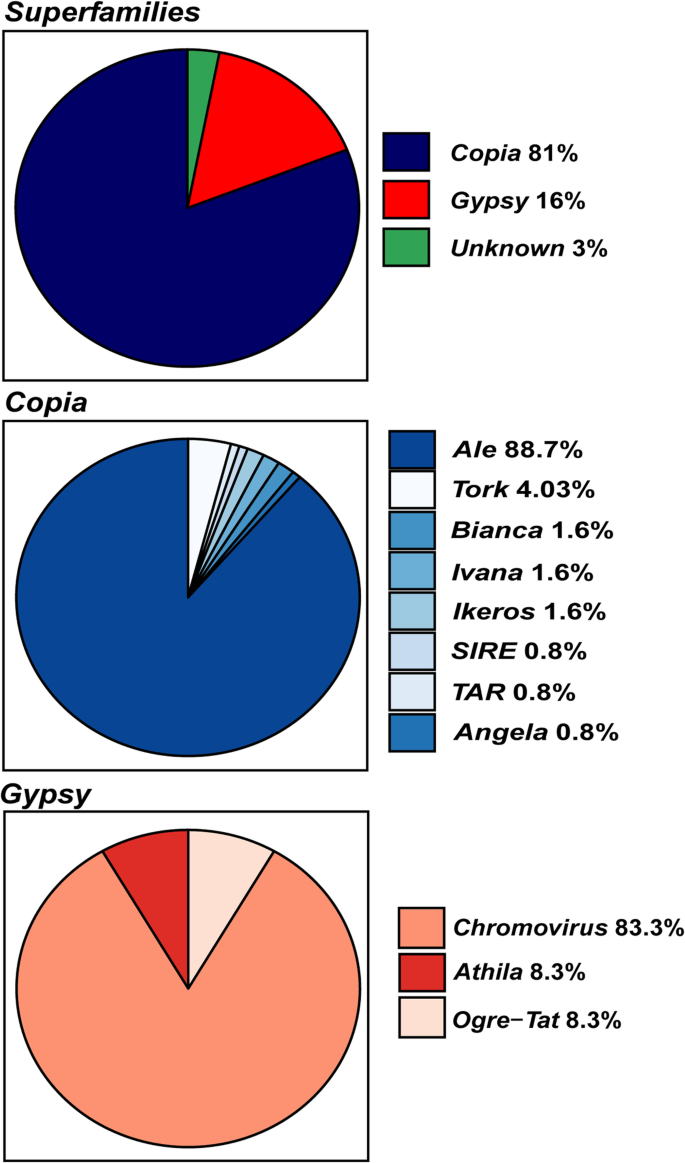
我们可以注释1163吉普赛人元素(62.52%)和623Copia元素(33.49%),即数量吉普赛人全长元素几乎高于2倍Copia的人。全长LTR-REs共74个(3.97%)为未知(图1)。1).
关于Copia总科,319啤酒(51.20%), 152年安琪拉(24.39%)、4Bianca.(0.64%)、34Ikeros.(5.45%),22伊凡娜(3.53%)、14陛下(2.24%), 41岁焦油(6.58%)和37托克(5.93%)元素的特征(图。1).的啤酒血统占主导地位,其次是安琪拉.其他识别的谱系被尊重。
至于吉普赛人总科,Chromovirus其中,1163种(71.77%)中有835种(71.77%),共108种(9.28%)凯兰崔尔女王(1.28%), 8雷纳(0.68%),和704年Tekay(60.53%)的元素。另一个吉普赛人谱系,Athila和Ogre-tat.分别有125(10.74%)和203(17.44%)个元素。1).
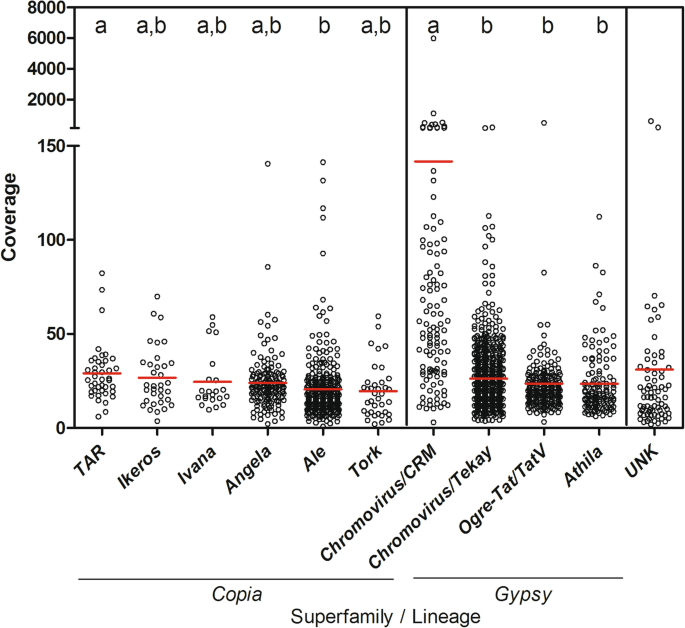
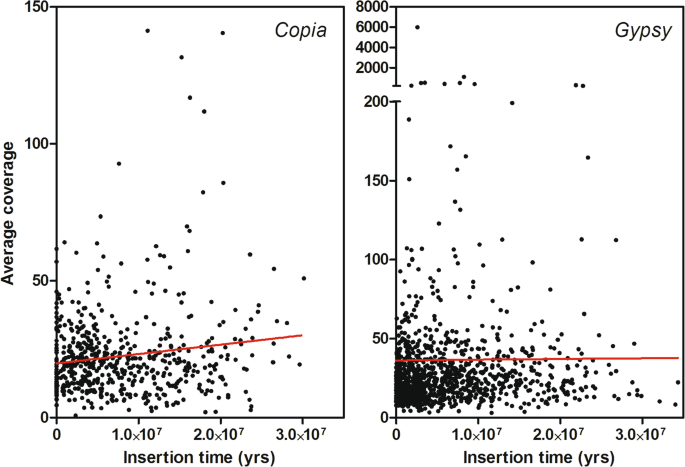
关于ltr -反转录转座子的基因组丰度F. Carica.基因组,映射illumina DNA读取到全长LTR-RODRORDRANSPOSONS(参见方法)表明吉普赛人是最丰富的超级家庭。特别是,最丰富的血统是Chromovirus / CRM,平均覆盖率为141.76Chromovirus / Tekay,Ogre-Tat / TatV,和Athila,平均覆盖率分别为26.23、23.58和23.47。2).属于的最具代表的谱系Copia超家庭是焦油,平均覆盖率为29.06,然后是Ikeros.(26.75平均覆盖率),伊凡娜(24.53),安琪拉(23.95),啤酒(20.49)托克(19.59)(图2).最后,未分类并命名为“未知”的ltr逆转录转座子的总平均覆盖率为31.04(图5)。2).
LTR-RODRONSPOSONS的插入时间F. Carica.基因组
一个LTR-RE的插入年龄可以通过计算同一元件的ltr之间的核苷酸替换来估计,这些替换应该在逆转录转座事件发生后立即相同,然后随着时间的推移积累突变[35.].在我们的测量中,我们应用Populus Trichocarpa.同义替代率2.36 × 10- 9(见方法)。分别对两个LTR-RE超家族和最丰富的两个超家族进行分析吉普赛人和Copia谱系。
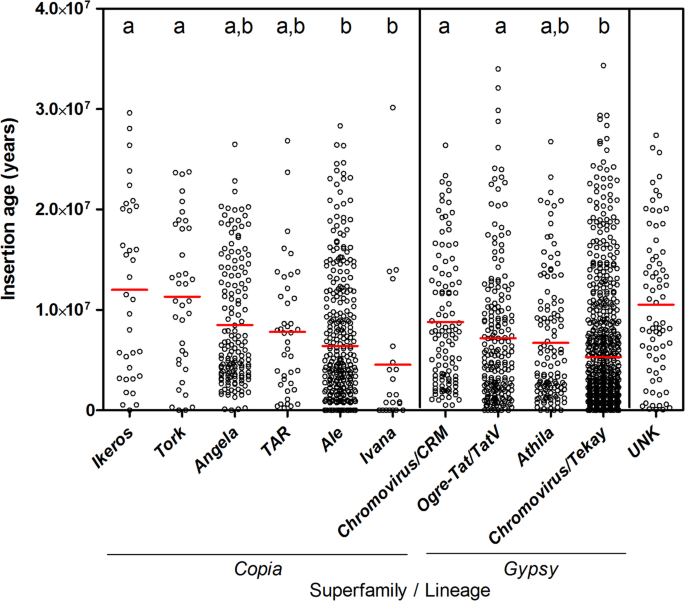
图中报告了每个图全长LTR-RE的插入时间估计。3..平均而言,较老的元素属于Copia(7,9我的平均)相比吉普赛人(6,1)。最古老的血统属于Copia超家庭是Ikeros.(12,0我的平均),而最年轻的LTR-RE属于伊凡娜(4,5我)和啤酒血统(6、4我的)。最古老的吉普赛人天堂是Chromovirus / CRM(8,8,我的平均),而最小的人是由Chromovirus / Tekay(5,3)。未知的LTR-REs显示平均估计插入时间约为12,5 MY(图。3.).
LTR-REDROTRANSPOSONS的表达分析F. Carica.
Illumina读取对照和盐胁迫植物叶片的cDNAF. Carica.在实验开始后的两个时间点,24和48天,于1867年全长LTR-RES映射。总的来说,总共250,464个读数在LTR-RE的参考组上对齐。平均而言,每个库的0.22%的读数映射在序列集上,范围为0.16到0.31%,具体取决于库(附加文件1).
在全球范围内,两者均检测到全长LTRs的低表达Copia和吉普赛人平均RPKM分别为0.64和0.06。4).高百分比的唯一读取(即,读取映射到一个LTR-RE)被观察到,从91.8到95.6%。如此高百分比的唯一读取应该确保对测量的LTR-REs表达值的高可靠性。
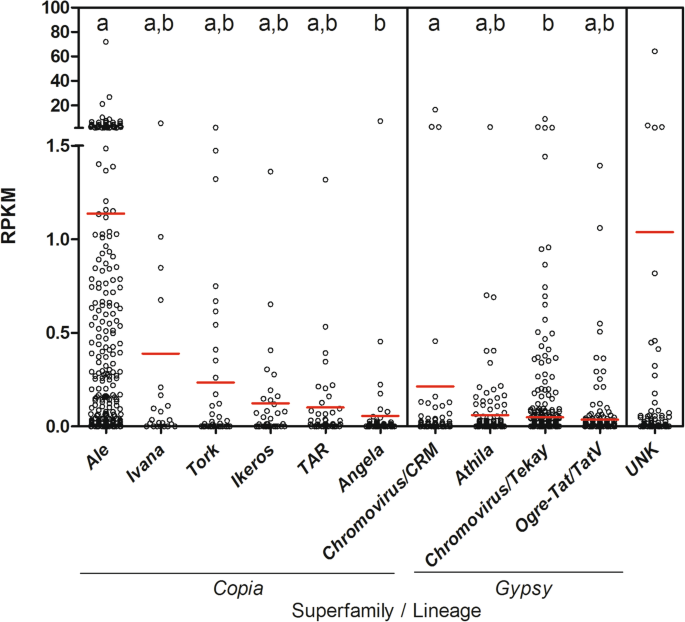
Copia与...相比,谱系表达略高吉普赛人的人。最表达的Copia天堂是啤酒(平均RPKM = 1.13)伊凡娜(0.38)和托克(0.23)(图。4).最表达的吉普赛人血统是Chromovirus / CRM(0.21),Athila(0.06)Chromovirus / Tekay(0.04)。
考虑未知全长LTR-REs,我们得到的平均RPKM为1.04(图。4).特别是一种UNK元素表现出很高的RPKM。对该元件的进一步分析显示,在该未知元件中存在两个推测的基因序列,这可能解释了该LTR-RE的高表达价值。
我们在至少一个库中设置RPKM> 1,作为考虑实验期间表达的全长元素的阈值;基于该阈值,1867年出现了153个表达的LTR-RE。考虑到153种表达的元素,97个编码的所有蛋白质结构域,因此可以被认为是完整和自主的。五十一个表达元素缺少一个或多个域,5个元素导致缺乏域。
表达的LTR-RE的主要部分属于Copia超级家族(81.0%),其次是吉普赛人(16.0%)和未知元素(3.0%)(图。5).关于血统,最具代表性的Copia血统是啤酒(88.7%)和托克(4.0%),而最多代表吉普赛人天堂是Chromovirus(88.3%),紧随其后的是Athila(8.3%),如图2所示。5.特别是表达Chromovirus元素属于四个次级谱系Tekay(60%),雷纳(20%),CRM(15%)和凯兰崔尔女王(5%)。
LTR-REDRANSPOSONS转录分析F. Carica.盐暴露植物的叶子
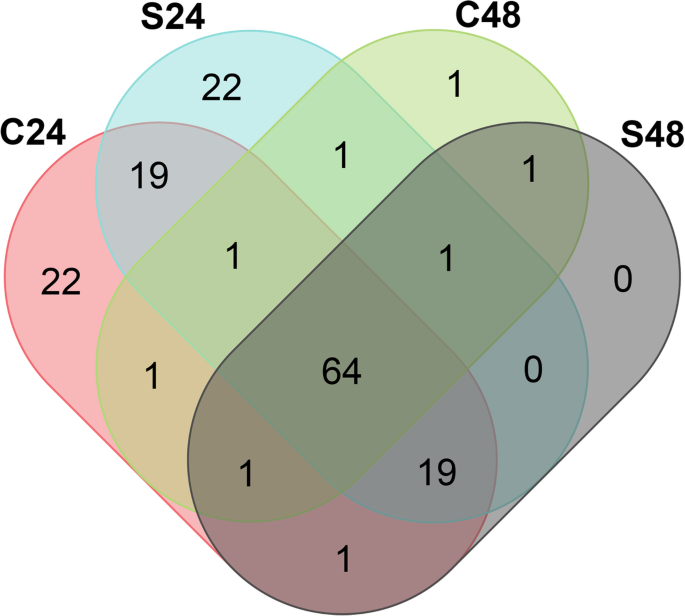
在生理盐水胁迫过程中,共表达了153个LTR-REs(即在至少一个rna序列库中显示RPKM > 1)F. Carica.对叶片进行进一步研究。这些REs(64)大部分在所有处理中表达,如图所示。6.然而,我们发现有22个LTR-REs在对照和盐胁迫下表达F. Carica.实验开始48天后,对照叶片中激活一个LTR-RE(图1)。6).我们没有发现任何在暴露于盐的植物叶子中特别表达的LTR-RES 48天。
关于LTR-REs的表达水平,我们检测到所有文库的RPKM平均值为2.74,最大RPKM为89.56,与低/中表达的内参基因相似,这些内参基因在对照和盐胁迫中均表现出稳定的表达(Additional File2).总体而言,在第一个时间点(试验开始后24天),对照和胁迫植株叶片的表达量均略高F. Carica.(附加文件3.),虽然这种差异被证明是Anova统计分析的重要性。
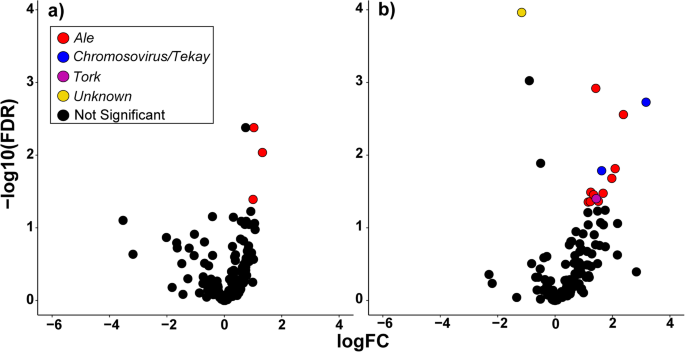
为了在盐胁迫期间找到差异表达的LTR-RES,我们在治疗的24和48天进行了对照和盐处理植物的叶子之间进行了成对试验。我们发现了三个啤酒实验开始后24天内过表达的元素(图。7a).在第二个时间点,我们提取了13个激活的LTR-REs(图。7b),属于啤酒(10个元素),Chromovirus / Tekay(2),和托克(1)血统.最后,在盐处理48天后不鉴定过鉴定的一个元素(图。7b)。
与表达全长ltr -逆转录转座子接近的基因的表达
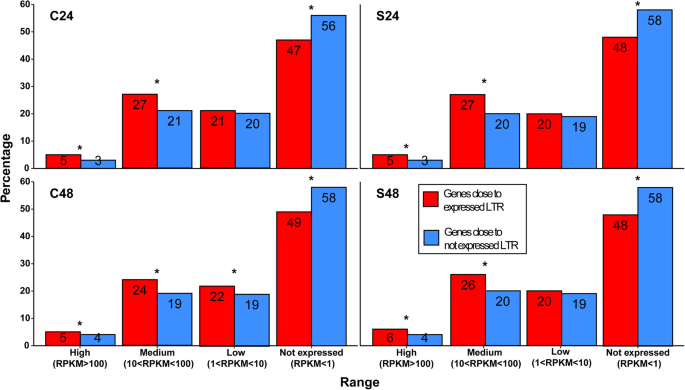
研究了无花果叶片在盐胁迫下LTR-REs上下50 kbp框架中表达(153个元件)和未表达(1714个元件)基因的表达和功能。为了分析表达模式,我们将基因分为四类:不表达(RPKM < 1)、低表达(1 < RPKM < 10)、中表达(10 < RPKM < 100)和高表达(RPKM > 100)。我们共检测到1816个与LTR-REs表达密切相关的基因。在这些基因中,未表达基因最多(平均48%),其次是中表达基因(平均26%),低表达基因(平均21%)和高表达基因(平均5%)(图1)。8).在表达或未表达的近距离接近的基因之间的表达水平的比较显示出在对照和盐的叶片上近似的高度和培养基表达基因百分比显着较高的高度表达基因百分比植物(图。8).
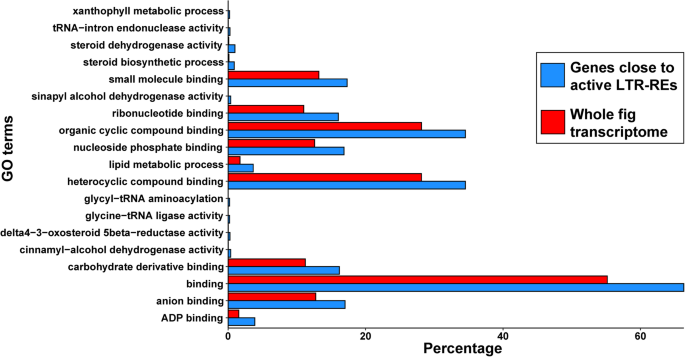
为了研究活性LTR-REs附近基因中特异的氧化石墨烯(go)项的发生,我们采用了富集分析。该分析将LTR-REs近端基因的go -term与整体的go -term进行了比较F. Carica.转录组,通过Fisher精确测试。只有显示罗斯福修正的GO条款P-值< 0.05被考虑。与整体相比,共有19个GO术语得到了特别丰富F. Carica.转录组(图。9).其中,我们发现参与脂质代谢过程,核糖核苷酸结合和类固醇代谢过程的术语。
LTR-retrotransposons动态F. Carica.基因组
分析了LTR-REs全长表达量、基因组丰度和预估插入时间的关系。这种相关性并不显著,可能是因为基因组中的LTR-REs是一个非常异构的群体,特别是由低重复和非活性元素组成。然而,测量的回归线可以突出LTR-RE丰度、插入年龄和表达之间的关系趋势。
例如,估计插入时间和基因组丰度(以平均覆盖率衡量)之间的相关性Copia和吉普赛人超家族不显著;然而,回归线表明更老CopiaLTR-REs含量高于年轻组F. Carica.基因组,而吉普赛人LTR-RE基因组丰度在年轻和旧元素之间没有差异(图。10).
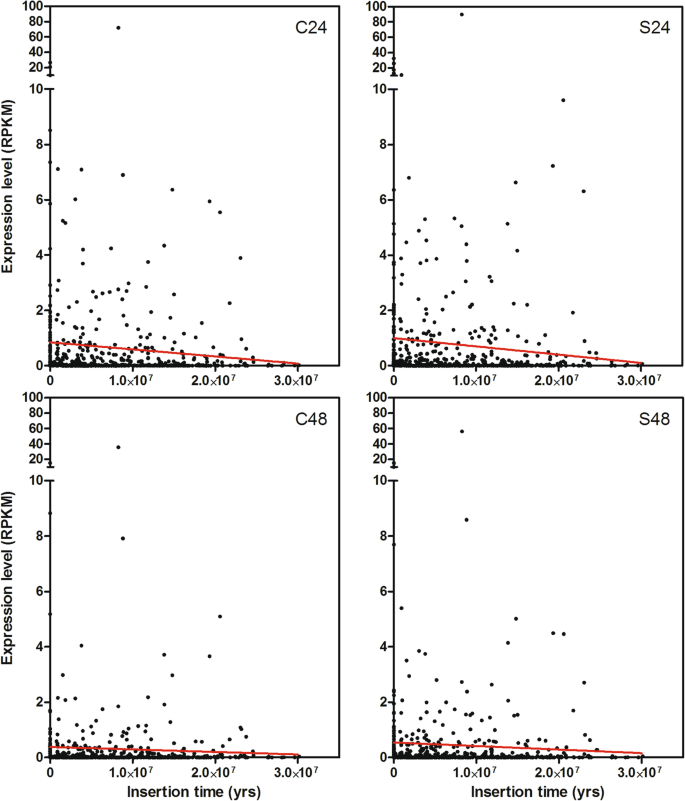
在插入时间与表达量的关系上,LTR-REs属于吉普赛人超家族未分析,因为它们都显示出非常低的RPKM值。属于全长LTR-RES的年龄和表达水平之间的回归线Copia在对照和盐处理植物的叶片中,超级科的表达量均随年龄的增加而降低(图5)。11),但相关性不显著。
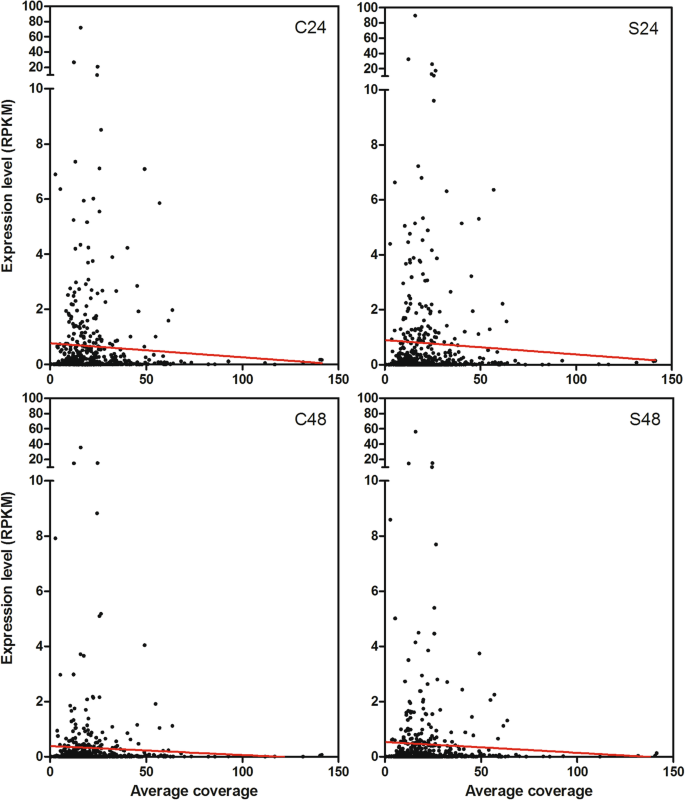
最后,我们分析了基因组丰度(平均覆盖率)和表达值(RPKM平均值)之间的相关性Copia全身LTR-REs (吉普赛人未分析元素,因为它们非常差别表示)。尽管在这种相关性也没有统计学意义,但回归线概述了根据其表达的LTR-RE的趋势,在所有治疗中,在基因组中越多的(平均覆盖率)越多(图。12).
此外,通过比较对照和盐处理样品间互作估计插入时间/表达水平的回归线斜率,我们发现在实验开始后的24天和48天内,对照和盐处理样品间的插入时间/表达水平的回归线斜率都有所增加(表1)1),提示在非生物胁迫中可能激活较老的LTR-REs。与对照叶片相比,盐处理24和48 d后,基因丰度的回归线在盐处理24和48 d后的斜率均有所增加。这些数据表明,最丰富的全长LTR-REs可能被激活F. Carica.基因组在盐处理过程中。
讨论
我们的作品的目的是提供物种LTR-RE的完整表征,F. Carica.,以及它们在基因组中的动态,通过对该物种基因组中可识别的所有全长元素的研究。对重复组的分析通常使用低覆盖率测序和聚类分析(例如,见Mascagni等[36.]和Zagorski等。[37.])很少考虑全长元素,例如,Populus Trichocarpa.[9),蕨麻micrantha[10].然而,DNA测序和序列组装策略的改进允许使用长序列读取,现在实现了比以前更完整的基因组序列,特别是相对于基因组的重复成分。因此,基因组测序使用这些新程序允许一个更精确和可靠的特征重复元素。
在本研究中描述的案例中,我们分析了F. Carica.,通过结合PACBIO和Illumina测序和最近发布的[1].该物种的基因组相对较小(356 Mbp [38.), TEs约占基因组的三分之一[1,这一比例与在其他小型基因组中发现的比例相似[39.]并且高于以前可用的植物组织序列测量的(16%[34.])。这种差异很可能是由于丢失或折叠重复序列造成的,这是通过NGS技术生成的程序集中广泛观察到的典型现象,特别是在程序集高度碎片化的情况下。其他研究已经强调了长读技术如何成功地提高重复基因组区域的分辨率。例如,牛津纳米孔技术(Oxford Nanopore Technology)对拟南芥(Arabidopsis)进行测序,可以解决一个此前无法通过BAC测序的数量性状位点(QTL),因为它的重复结构需要读取大于20kb的内容[40].小麦中也有类似的发现[41.]和玉米[42.].
在这项研究中,我们确定了1867个全长元素,这些元素被归类于他们的超家族和血统。绝大多数这些元素是特定于基于PACBIO的组件,证实了基于第三代测序技术的更高质量,以解决基因组的复杂重复区域。
全长元素吉普赛人超级小家族更频繁Copia,类似于其他树种,如杨树[9].在沿袭级别上,用于Copia总科,啤酒元素是最常见的,类似于其他物种,如Erythrostemon hughesii[43.].为了吉普赛人总科,Chromovirus血统(特别是TekaySublineage)显示出在其他物种中已经报道的最高丰富,如胡椒Hieracium[37.,44.].
对于每个全长元素,我们测量了对照植物的叶片和对盐胁迫的植物中的插入的基因组丰富,调用年龄和转录活性[33.].
的Chromovirus血统(sublineageCRM)的平均覆盖率最高。Chromovirus / CRM元件主要位于着丝粒和着丝粒间区域,它们可能在那里发挥结构作用[45.,46.,47.,48.,49.].值得注意的是,这种延期的全长要素约占这项工作中确定的所有全长元素的6%,而其平均平均覆盖率远高于所有其他谱系(从30.50到19.59的范围),表明存在的情况,在富有的基因组中Chromovirus / CRM残留和片段(即,不完整的元素)不能被软件识别,该软件只能预测全长的LTR-REs,并且可能与新元素插入其他元素(所谓的嵌套逆转录转座子)有关,这种结构已经在其他物种的着丝粒区域中描述过[50.].
对全长因子插入年龄的分析显示,在每个谱系内都有极端的变异性,表明插入时间跨度很宽,不同因子的活性有很大的重叠,如杨树、向日葵和桑树的报道[9,51.,52.].平均而言,在Copia元素,谱系Ikeros.和托克比血统更古老吗啤酒和伊凡娜.在吉普赛人LTR-RES,平均年龄Chromovirus / CRM和食人魔元素含量显著高于Chromovirus / Tekay元素。
通过将来自盐胁迫和对照植物的植物的叶片映射获得的illumina cDNA读取来研究全长元素的表达。在重复序列的情况下,这种类型的分析是偏置的,因为它不确定读取到全长LTR-RE的CDNA读取的实际不通过另一元素(或来自不完整元素)的RNA产生同一个家庭,因此与全长元素分享其序列,位于另一个轨迹处。然而,映射到全长LTR-RES(从91.8到95.6%映射的唯一读取百分比允许我们在每个元素的表达式模式中建立可靠的趋势。
我们的研究结果表明,控制和盐露的植物中,LTR-RE的表达通常非常低。这一发现是预期的,因为LTR-RES通常受到严格的控制,以防止它们通过不同机制的表达。转座子抑制的一个主要机制是RNA干扰,其由源自由宿主产生的双链RNA的小RNA介导的小RNA,其通过DNA甲基化,染色质重塑和Re转录物的后转录后降解来触发逆转录沉默和再转录的转录衰减[53.,54.,55.].
考虑到在至少一个库中的RPKM> 1作为用于定义给定表达的元素的阈值,在对照和盐处理的植物中仅表达了1867个元素的仅153个元素。虽然吉普赛人元素代表了无花果基因组中绝大多数的LTR-RE补体,大多数表达的元素属于Copia超家庭。邱和尚未接命者[56.]在三种野生物种中发现了类似的结果向日葵其中LTR-REs表达量最多的属Copia超级家族,尤其是很少重复的家族。事实上,吉普赛人已知元素会特别插入异染色质[57.,58.,59.],而Copia LTR-REs也倾向于插入常染色质区域[58.,60.],更常见的是转录。
在许多物种中描述了植物组织中的RE转录物的存在,没有表观诱导刺激或暴露于环境变化[61.].第一例是在类似素类,向日葵,水稻和杨树[62.,63.,64.,65.,66.].在第二组中,当模拟非生物和生物胁迫时,LTR-RE转录的诱导已被证明[67.,68.,69.,70,71.,72.,73.,74.].值得注意的是,LTR-RE序列的互补转录本的出现可以被认为是i) LTR-RE激活的第一步,这可能导致最终的逆转录转位或ii)它可能代表用于RE沉默的LTR-RE rna的产生。
关于不同的LTR-RE谱系的表达,绝大多数(88.7%)的表达Copia元素属于谱系啤酒,在吉普赛人超家族,一个血统(Chromovirus)占表达LTR-REs的83.3%。
通常,盐胁迫对LTR-RE的表达效果不大。在早期盐处理期间,只有三个元素(所有Copia /前者啤酒血统)调节。啤酒在盐胁迫48天后,13种元素含量均显著升高。值得注意的是,24天后上调的元素与48天后上调的元素并不相同,表明盐胁迫对同一谱系元素的诱导类型不同。
在48天后,使用盐暴露和对照植物的叶子的RNA-SEQ数据,我们分析了LTR-RE存在于邻近其邻近的基因表达的影响(在50kbp升高到下游每个全长LTR-RE)。我们观察到,没有表达靠近LTR-RES的大多数基因。然而,当LTR-RE表达时比未表达时,靠近LTR-RE的基因的表达更大。当它们在染色体上靠近时,该数据表明转座子和基因之间的互殖关系。这种影响可能与染色体基因座的表观遗传控制(例如,通过DNA甲基化)有关,其中基因和LTR-RE彼此接近。据报道,从植物转回到侧翼DNA区域的甲基化脱落[75.,76.].
逆转录转座子的插入可能意味着对其邻近区域的表达进行表观遗传控制,从而导致沿该染色体位点的转录抑制[75.].特别地,表明转换元件插入即使在上游成千上万的碱基对框架中,也可以改变基因表达模式,例如在情况下Teosinte分支机构1外源基因的插入改变了玉米基因的表达模式跳房子Retrotrasposon在Gene的距离超过60,000bp [77.].
然而,也可以假设,在转回的情况下,在其表达对于细胞所必需的基因附近并且应该保持并且应该保持,它可能使沉默是通过DNA甲基化沉默的。在这种情况下,其他LTR-RE沉默机制应该操作,可能在转录后水平上[78.].
通过分析位于表达LTR-REs附近的基因,我们发现一些GOs明显更频繁地出现在基因中存在表达元件的区域,这表明发现这些基因的染色体区域可能更难以被DNA甲基化沉默。显然,还需要其他研究,特别是在其他物种中,来评估反转录转座子和某些基因功能类别之间是否存在这种类型的关系。
然后,我们试图评估基因组丰富,插入年龄和LTR-RE的表达之间的可能相关性。由此产生的回归线并不重要,可能是由于LTR-RE之间关于插入年龄和丰度的极端变化。这种可变性与每个LTR-RE在宿主基因组中的复制中可以是自主的事实有关。关于表达,表达少数元素,表达水平通常非常低,使得难以建立显着的相关性。然而,我们的数据获得的回归线允许我们在LTR-RE动态中建立趋势。
关于基因组丰度与插入年龄之间的相关性,本文采用Copia在超家族中,最古老的元素也更丰富,这表明随着时间的推移,每个元素的新副本不断产生,或者古老的LTR-REs受到了一次复制爆发。相比之下,对于吉普赛人超级家族,可以假设对最古老的元素的复制有更强烈的控制,因此它们的丰度不大于最年轻的元素。据报道,甚至发现单一元素均具有复制活动的突发,例如在短时间内增加其基因组大量,包括一系列包括的物种野豌豆属pannonica[79.)、棉花(80],选用australiensis[81.),而向日葵agrestis)[66.].
对于表达和插入年龄之间的相关性,可以只评估Copia元素,作为表达的吉普赛人元素太小,不能进行相关性研究。这是另一个迹象,表明宿主对这个元素超家族的控制比对Copia超家庭。
考虑Copia基因的表达量随着插入年龄的增加而降低,这可能是由于宿主有更多的时间来建立对该基因的表观遗传控制。
观察到相同的趋势对于LTR-RES表达与其基因组丰度之间的相关性。事实上,在几项研究中确定了这种关系,其中一个元素越多,它被认可和遭受沉默的越多[15,65.,82.,83.].
结论
总之,我们的数据提供了完整的特征全长LTR-REsF. Carica.,提供了与这些元素相关的基因组动力学以及它们在基因调控中的作用的有趣信息。将这种分析扩展到使用第三代测序技术对其基因组进行排序的其他物种,将使人们能够通过比较分析,更好地了解这些因素在基因组水平上物种进化的重要性。
方法
全长LTR-RES隔离和表征
Usai等人在高度邻近的无花果基因组序列中鉴定出I类全长LTR-REs [1使用PacBio SMRT测序技术与Illumina测序相结合。
使用LTRharvest v1.5.10对这些元素进行了鉴定[84.]和注释使用LTRdigest v1.5.10 [85.]和基于RepeatExplorer的网站(https://repeatexplorer-elixir.cerit-sc.cz/galaxy/).LTRharvest在fig基因组装配上运行,参数如下:-minlenltr 100,−maxlenltr 6000,−mindistltr 1500,−maxdistltr 25,000,−mintsd 5,−maxtsd 5,−similar 85,−vic 10。
可用的基于ngs的无花果基因组序列[34.]也经受相同的基于结构的鉴定过程,并且通过使用CD-mit ent来进行序列比较。相似性阈值设定为90%[86.].
TRNA序列拟南芥,Populus Trichocarpa.,葡萄,和玉米从GtRNAdb (http://gtrnadb.ucsc.edu.),通过LTRdigest鉴定所鉴定元件的引物结合位点区域。
全文LTR-REs库提交给DANTE工具进行域注释。使用REXdb对转座元件蛋白域进行默认参数注释[48.]和Blosum80评分矩阵。通过默认参数的蛋白质匹配的意义来过滤结果。手动检查最终注释以删除任何嵌套案例。
在进一步表征全长元件时,通过对5’-和3’- ltrs的序列差异比较,估算每个LTR-RE的插入日期。LTR成对对齐和距离矩阵分别使用EMBOSS v6.6.0.0套件的stretcher和distmat工具计算[87],使用序列演进的Kimura双参数模型[88].以2.36 × 10的突变率估计元素插入时间- 9,即,在基因序列中计算同义替换的两倍Populus Trichocarpa.[89],因为与基因序列相比,随着时间的推移,LTR-REs会积累更多的突变。
在某些情况下,使用Augustus [90]和默认参数下的丁迪。
LTR-RES冗余分析
DNA库F. Carica.简历。Dottato从NCBI数据库下载(NCBI,华盛顿,美国,https://www.ncbi.nlm.nih.gov/sra.),使用注册号SRP109082。Solorzano Zambrano等人描述了图书馆的建设[91].用两种Illumina序列仪进行DNA测序:Miseq和Hiseq2000,产生125bp读数。使用Trimmomatic v0.33修剪读取[92]与以下标准:头:19,作物:100,滑动窗口:4:20,MINLEN:100。Illumina适配器也被移除。
通过将它们映射到内部序列来清除高质量的读取F. Carica.叶绿体和桑属notabilis线粒体基因组使用CLC基因组学工作台V9.5.3(CLC-BIO,丹麦)具有严格参数:不匹配成本= 1,删除成本= 1,插入成本= 1,相似性分数= 0.9,长度分数= 0.9。收集了所得未映射的读取。
LTR-REs的基因组丰度F. Carica.基因组的评估是通过将Illumina DNA序列与从组装中分离的全长LTR-REs进行比对[1].使用CLC基因组学工作台使用以下标准进行Illumina DNA的映射到全长LTR-RE:不匹配成本= 1,删除成本= 1,插入成本= 1,相似性分数= 0.9,长度分数= 0.9。
RNA孤立
两岁F. Carica.(简历。(Dottato),微型繁殖植物生长在5 L的塑料盆中,装满6.4%的粘土,8.6%的淤泥和85%的沙子,并受到盐度胁迫[33.].分别用0 mM NaCl和100 mM NaCl处理植株,分别选择试验开始后24天和48天的两个时间点。因此,在每个时间点,收集3个对照(0 mM NaCl)和3个盐胁迫(100 mM NaCl)植株的1片完全展开的叶片。
从叶片中提取总RNA,参考Giordani等[65.和基因组DNA污染清除DNAse I(罗氏)消化按照制造商的说明。然后,按照标准程序纯化RNA。对对照叶片(0 mM NaCl)和100 mM NaCl处理植株叶片进行RNA-seq分析。
LTR-REs和基因表达分析
对照叶(0mM NaCl)进行RNA-SEQ分析,并在实验开始后24和48天用100mM NaCl处理的植物的叶子。12个CDNA文库的控制和压力叶F. Carica.,如vangelisti等。[33.],从Bioproject加入代码Prjna508874下载了从NCBI SRA下载。
每个图书馆的质量由生物分析仪2100评估(安捷伦技术,Santa Clara,CA,USA)和使用Illumina Hiseq2000进行测序。使用FASTQC检查成对端(125bp长度)读取序列的质量(v。0.11.5 [93]并使用Trimmomatic进行修剪,裁剪前15个碱基和后10个碱基并移除适配器。
使用CLC基因组学工作台使用具有用于DNA对准的相同参数的CLC基因组学工作台映射到整个转录组和全长LTR-RE的cDNA读数。LTR-RE的表达水平被计算为每千克映射读数(RPKM,[94]);RPKM是一个规范化的表达值,它允许比较序列在文库内和文库之间的转录。RPKM的计算方法是将序列上对齐的读取数与库中的读取总数以及序列长度进行计算[94].仅考虑至少一个文库中具有RPKM> 1的LTR-RES。为了在两个时间点期间检测控制和盐的可能差异表达的LTR-RES,在两个时间点中,对编辑的成对似然测试[95应用于从对齐衍生的读数。虚假发现率(FDR)[96]用于结果p值。我们认为具有绝对Fold-Change > 2和FDR < 0.05的LTR-REs为差异表达。
为了评估LTR-REs附近基因的表达模式,我们对LTR-REs上、下游50kbp区域进行了研究。通过使用GFF文件提供的坐标来提取这些元素附近的基因F. Carica.基因组(1]并利用Bedtools v2.29.2的交叉功能[97].接近LTR-REs的基因的表达值用RPKM来测量。这些基因按RPKM的范围分类。然后,比较LTR-REs中接近表达和未表达的基因的表达情况。差异采用Fisher精确检验,并认为FDR < 0.05具有显著性。
基因功能分析是通过分析GO条款提供的F. Carica.基因注释表[1].GO术语的总体分布已在WEGO 2.0中可视化[98].使用Blast2GO对一组表达LTR-REs的相近基因与整个转录组之间进行富集分析,采用Fisher精确检验[99];在FDR校正中,GO术语被选为显著的p -值<0.05。使用Revigo进一步总结了丰富的GO术语[One hundred.通过微小参数选项。
可用性数据和材料
DNA测序数据已在Bioproject加入代码Prjna565858下沉积在NCBI存储库中。在加入VYVB00000000下,介于DDBJ / ENA / Genbank沉积了图。本文中描述的版本是版本VYVB01000000。TES的集合可在比萨大学农业,食品和环境部的存储库序列页面上获得(http://pgagl.agr.unipi.it/sequence-repository/).RNA测序数据已在BioProject加入码Prjna508874下的NCBI存储库中存放。中间文件和其他杂项信息可从合理的请求上获得相应的作者。
缩写
- 罗斯福:
-
错误发现率
- LTR:
-
长终端重复
- LTR-RE:
-
长末端Repeat-Retrotransposon
- 我:
-
万年
- PACBIO:
-
太平洋生物科学
- 再保险:
-
逆转录转座子
- rpkm:
-
每千克读数每千克映射读数
- SMRT:
-
单分子实时
- TGS:
-
Third-Generation-Sequencing
参考文献
- 1.
Usai G, Mascagni F, Giordani T, Vangelisti A, Bosi E, Zuccolo A,等。植物j . 2020; 102:600-14。
- 2.
Barolo MI, Mostacero NR, López SN。无花果(桑科):一种古老的食物和保健来源。食品化学。2014;164:119-27。https://doi.org/10.1016/j.foodchem.2014.04.112.
- 3.
Barghini E, Mascagni F, Giordani T, Solorzano Zambrano LJ, Natali L, Cavallini A.对无花果基因组结构和组成的洞察力。Acta Hortic。2017;1173:69 - 74。
- 4.
4种灌水量对6个无花果品种生长的影响。Acta Hortic。2003;605:113-8。
- 5。
Ayers SR, Westcost WD。农业水质,粮农组织灌溉和排水论文29。意大利罗马:联合国粮食及农业组织;1979.
- 6。
Caruso G, Gennai C, Ugolini F, Marchini F, Quartacci MF, Gucci R.对幼鱼的耐受性和生理反应榕辣海参L.植物用盐水灌溉。Acta Hortic。2017; 1173:137-42。
- 7。
Wicker T, Sabot F, Hua-Van A, Bennetzen JL, Capy P, Chalhoub B等。真核生物转座因子的统一分类系统。《自然》2007;8(12):973-82。https://do.org/10.1038/nrg2165.
- 8。
Kumar A, Bennetzen JL。植物反转位子活动。李建平。2003;3(1):1 - 7。https://doi.org/10.1146/annurev.genet.33.1.479.
- 9.
本文主要介绍了国内外学者的研究现状吉普赛人和Copialtr -反转录转座子超家族和谱系及其在Populus Trichocarpa.(L.)基因组。树遗传学基因组织。2015; 11:107。
- 10.
作者简介:张建平,男,硕士研究生,主要研究方向为神经网络、神经网络、神经网络等。的基因组序列和转录组蕨麻micrantha和他们的比较草莓属vesca(林地草莓)。GigaScience。2018; 7(4): 1 - 14。https://doi.org/10.1093/gigascience/giy010.
- 11.
Ma J, Devos KM, Bennetzen JL。ltr -反转录转座子结构分析揭示了最近和快速的水稻基因组DNA损失。基因组研究》2004;14(5):860 - 9。https://doi.org/10.1101/gr.1466204.
- 12.
王Q,Dooner HK。玉米基因组结构的显着变异从BZ基因座的单倍型多样性推断出来。Proc Natl Acad Sci U S A. 2006; 103(47):17644-9。https://doi.org/10.1073/pnas.0603080103.
- 13。
Devos KM, Brown JK, Bennetzen JL。在拟南芥中,通过不合理的重组导致的基因组大小减少抵消了基因组的扩张。基因组研究》2002;12(7):1075 - 9。https://doi.org/10.1101/gr.132102.
- 14。
Vitte C,Panaud O.通过不平等的同源重组抵抗LTR回收ansposons在水稻中的扩增形成Solo-LTR栽培稻L. mol Biol Evol。2003; 20(4):528-40。https://doi.org/10.1093/molbev/msg055.
- 15.
转座子在植物进化中有多重要?自然科学学报。2013;14(1):49-61。https://doi.org/10.1038/nrg3374.
- 16。
Pinosio S, Giacomello S, Faivre-Rampant P, Taylor G, Jorge V, Le Paslier MC等。杨树全基因组结构变异鉴定的泛基因组特征。《生物学报》2016;33(10):2706-19。https://doi.org/10.1093/molbev/msw161.
- 17。
Dubin MJ,Scheid Om,Becker C. rserposons:祝福诅咒。Curr意见植物Biol。2018; 42:23-9。https://doi.org/10.1016/j.pbi.2018.01.003.
- 18。
van driel r,fransz pf,verschure pj。真核基因组:在不同层次水平下调节的系统。J Cell SCI。2003; 116(20):4067-75。https://doi.org/10.1242/jcs.00779.
- 19.
歌曲JT,Lu H,McDowell JM,Greenberg JT。ALD1在拟南芥中激活当地和系统性防御的关键作用。工厂J. 2004; 40(2):200-12。https://doi.org/10.1111/j.1365-313X.2004.02200.x.
- 20.
Hollister JD,Gaut BS。转换元素的表观遗传沉默:降低转位与邻近基因表达的有害影响之间的权衡。Genome Res。2009; 19(8):1419-28。https://doi.org/10.1101/gr.091678.109.
- 21.
霍利斯特,史密斯,郭永利,奥特F,韦格尔D,高特BS。转座元件和小rna有助于基因表达之间的分歧拟南芥和rabidopsis lyrata..美国国家科学研究院学报2011;https://doi.org/10.1073/pnas.1018222108.
- 22.
刘家,塞内斯拉姆为,Chougule K,Ou S,Swentowsky KW,Gent Ji,等。使用长读技术的玉米染色体的无形组装。基因组Biol。2020; 21:1-17。
- 23.
Matzke Ma,Mosher Ra。RNA定向的DNA甲基化:一种增加复杂性的表观遗传途径。自然rev genet。2014; 15(6):394-408。https://doi.org/10.1038/nrg3683.
- 24.
拟南芥转座子转录和迁移的表观遗传控制。植物生态学报,2012;https://doi.org/10.1016/j.pbi.2012.08.006.
- 25.
关键词:环境适应,转座因子,环境效应,生物信息学摩尔生态。2013;22(6):1503 - 17所示。https://doi.org/10.1111/mec.12170.
- 26.
Vangelisti A,USAI G,Mascagni F,Natali L,Giordani T,Cavallini A.整个基因组分析在叶片中的长末端重复转回转录转录Populus Trichocarpa.L受到不同的应力。Caryologia。2019;72:69 - 79。
- 27.
Vangelisti A, Mascagni F, Usai G, Natali L, Giordani T, Cavallini A. Low long terminal repeat (LTR)-反转录转座子在海洋显色子中的表达伊大洋洲l .生活。2020;10(3):30。https://doi.org/10.3390/life10030030.
- 28.
Ansorge WJ。下一代DNA测序(II):技术,应用。Next generation sequence & Applic. 2016; 1:1-10。
- 29.
Daccord N,Celton JM,Linsmith G,Becker C,Choisne N,Schijlen E等人。高品质的德诺族组装苹果基因组和早期水果发育的甲虫动态。自然遗传。2017; 49(7):1099-106。https://doi.org/10.1038/ng.3886.
- 30.
研究结果表明,该方法具有较好的应用价值。寄生开花植物基因组中的寄生足迹五角定.自然通讯。2018;9:1-11。
- 31.
叶刚,张辉,陈斌,聂胜,刘辉,高伟,等。新创抗逆性森林物种的基因组组装木麻黄提供对次级增长的洞察力。植物j . 2019; 97(4): 779 - 94。https://doi.org/10.1111/tpj.14159.
- 32.
Veeckman E, Ruttink T, Van De Poele k,我们到了吗?植物基因组序列完整性的可靠估计。植物细胞。2016;28(8):1759 - 68。https://doi.org/10.1105/TPC.16.00349.
- 33.
Vangelisti A, Solorzano Zambrano L, Caruso G, Macheda D, Bernardi R, Usai G等。一种古老的耐盐水果作物榕辣海参L.,具有盐度的警察:转录组分析。SCI批准。2019; 9:1-13。
- 34.
Mori K,Shirasawa K,Nogata H,Hirata C,Tashiro K,Habu T等人。通过整体基因组测序分析鉴定与性别测定相关的RAN1正常序列(榕辣海参l .)。Sci众议员2017;7:41124。
- 35.
Sanmiguel P,Gaut BS,Tikhonov A,Nakajima Y,Bennetzen JL。玉米胚胎反射转移的古生物学。自然遗传。1998; 20(1):43-5。https://doi.org/10.1038/1695.
- 36.
Mascagni F ., Vangelisti A ., Giordani T ., Cavallini A ., Natali L.一项关于属中重复DNA的计算比较研究栎中国生物医学工程学报(英文版);2010;https://doi.org/10.1007/S11295-019-1401-2.
- 37。
Zagorski D,Hartmann M,Bertrand YJ,PaštováL,SlavíkováR,JosefiováJ,等。密切相关种类的重组物种特征和动态Hieracium(菊科)及其合成的和无融合生殖的杂种。植物科学与技术,2000,21(1):1 - 5。https://doi.org/10.3389/fpls.2020.591053.
- 38。
两种新的植物DNA流式细胞仪核分离缓冲液:一种37种植物的检测。安机器人。2007;100(4):875 - 88。https://doi.org/10.1093/aob/mcm152.
- 39。
Vitte C,Fustier Ma,Alix K,Tenaillon Mi。作物演化中的转座子的光亮侧。简短的Funct基因组学。2014; 13(4):276-95。https://doi.org/10.1093/bfgp/elu002.
- 40。
陈建平,陈建平,陈建平,等。高接触拟南芥基因组组装与一个单纳米孔流动细胞。自然通讯。2018;9:1-8。
- 41.
张伟,cicilira P, Messing J. PacBio基因家族测序——以小麦谷蛋白基因为例。基因。2014;533(2):541 - 6。https://doi.org/10.1016/j.gene.2013.10.009.
- 42.
Wang B, Tseng E, Regulski M, Clark TA, Hon T, Jiao Y,等。通过单分子长读测序揭示玉米转录组的复杂性。自然通讯。2016;7:1-13。
- 43.
Mata-Sucre Y, Sader M, Van-Lume B, Gagnon E, Pedrosa-Harand A, Leitch IJ等。异染色质在Caesalpinia组中有多不同?Cytogenomic表征Erythrostemon hughesiiGagnon & GP Lewis(豆科:Caesalpinioideae)。足底。2020;252:1-14。
- 44.
de Assis R, Baba VY, Cintra LA, Gonçalves LSA, Rodrigues R, Vanzela ALL。三种栽培植物的基因组关系和ltr -逆转录转座子多样性辣椒L.(solanaceae)物种。BMC基因组学。2020; 21:1-14。
- 45.
着丝粒反转录转座子谱系早于玉米/水稻的分化,在丰度和活性上存在差异。中国生物医学工程学报,2008;https://doi.org/10.1007/s00438-007-0302-5.
- 46.
龚志忠,吴玉芳,kobizkova A, Torres GA, Wang K, Iovene M,等。马铃薯中不重复和基于重复的着丝粒:对着丝粒进化的启示。植物细胞,2012;24(9):3559 - 74。https://doi.org/10.1105/tpc.112.100511.
- 47.
苏高清,刘yl,刘yx,lu zl,li hy,xie sj等。动态染色质变化与新创玉米常染色质中着丝粒的形成。工厂j . 2016, 88(5): 854 - 66。https://doi.org/10.1111/tpj.13305.
- 48.
Neumann P,NovákP,Hoštákován,Macas J.系统植物LTR-Retrotroansposons的系统调查阐明了它们的聚丙烯结构域的系统发育关系,并为元素分类提供了参考。暴毒DNA。2019; 10(1):1。https://doi.org/10.1186/s13100-018-0144-1.
- 49.
基洛夫(Kirov I, Odintsov S, Omarov M, Gvaramiya S, Merkulov P, Dudnikov M,等。功能葱属植物fistulosum着丝粒由长卫星重复序列、反转录转座子插入和叶绿体DNA组成。植物生态学报,2000,21(1):1 - 6。https://doi.org/10.3389/fpls.2020.562001.
- 50。
韦伯B、施密特T.嵌套Ty3-吉普赛单一的反转录转座子βprocumbensCentromere包含一个推定的染色体。斑驳。2009; 17(3):379-96。https://doi.org/10.1007/s10577-009-9029-y.
- 51.
Mascagni F, Vangelisti A, Giordani T, Cavallini A, Natali L.特定的ltr逆转录转座子显示野生向日葵和栽培向日葵之间的拷贝数差异。基因。2018;9(9):433。https://doi.org/10.3390/genes9090433..
- 52.
MA B,Kuang L,Xin Y,他N.新的见解进入Long终端重复转回桑椹物种的洞察。基因。2019; 10(4):285。https://doi.org/10.3390/genes10040285.
- 53.
转座因子与基因组的表观遗传调控。自然科学,2007;8(4):272-85。https://doi.org/10.1038/nrg2072.
- 54.
LISCH D.植物中转移元素的表观遗传调节。Ann Rev植物Biol。2009; 60(1):43-66。https://doi.org/10.1146/annurev.arplant.59.032607.092744.
- 55.
杨飞,王培军。哺乳动物种系中多个逆转录转座子沉默机制。见:细胞与发育生物学研讨会(第59卷):学术出版社;2016.118 - 25页。
- 56.
邱F,UNGERER MC。不同的基因组丰富和转录活动吉普赛和copia三种野生向日葵的长末端重复反转录转座子。植物生物学杂志。2018;18:1-8。
- 57.
高欣,侯毅,艾比娜,李文杰。反转录转座子与异染色质的直接整合。基因组研究》2008;18(3):359 - 69。https://doi.org/10.1101/gr.7146408.
- 58.
Cavallini A, Natali L, Zuccolo A, Giordani T, Jurman I, Ferrillo V等。向日葵的转座子和重复组成分析(Helianthus Annuus.l .)基因组。Al Appl Genet。2010; 120(3):491-508。https://doi.org/10.1007/s00122-009-1170-7.
- 59.
关键词:LTR,反转录转座子,染色体结构域,植物植物基因组学。植物基因组学。2012;https://doi.org/10.1155/2012/874743.
- 60。
Pearce SR, Harrison G, Li D, Heslop-Harrison JS, Kumar A, Flavell AJ。Ty1 -copia集团反转位子活动野豌豆属物种:拷贝数,序列异质性和染色体定位。Mol Gen Genet。1996年; 250(3):305-15。https://doi.org/10.1007/BF02174388.
- 61。
Grandbastien马。LTR逆转录转座子,植物调控和应激反应的方便的搭便车者。生物物理学报,1849;2015:403-16。
- 62。
Rico-Cabanas L,Martínez-Izquierdo JA。Cire1,一种新型转录活跃的Ty1-Copia Retrotransposon素类.中国生物医学工程学报,2007;https://doi.org/10.1007/S00438-006-0200-2..
- 63。
Vukich M, Schulman AH, Giordani T, Natali L, calendar R, Cavallini A.向日葵的遗传变异(Helianthus Annuus.L.)和在向日葵属评估的反转录转座子为基础的分子标记。理论应用,2009;119:1027-38。
- 64。
高丹,蒋宁,王永瑞,蒋俊,李国强。转座子在近缘物种间着丝粒的进化和多样化中起着丝粒的重要作用。植物科学与技术,2015;
- 65.
Giordani T,Cossu Rm,Mascagni F,Marroni F,Morgante M,Cavallini A等。基因组对叶片LTR-REDRANSPOSON表达分析杨树×黄花water-deprived植物。中国生物医学工程学报。2016;12(4):75。https://doi.org/10.1007/s11295-016-1036-5.
- 66.
Mascagni F, Giordani T, Ceccarelli M, Cavallini A, Natali L.全基因组分析ltr -逆转录转座子多样性及其对属进化的影响向日葵(l)。BMC基因组学。2017;18(1):634。https://doi.org/10.1186/s12864-017-4050-6.
- 67.
Kashkush K,Feldman M,Levy AA。回析衰退的转录激活改变了小麦中相邻基因的表达。自然遗传。2003; 33(1):102-6。https://doi.org/10.1038/ng1063.
- 68.
Hawkins JS, Hu G, Rapp RA, Grafenberg JL, Wendel JF。植物转座因子增殖速度的系统发育测定:copia和类线元素Gossypium.基因组。2008; 51(1):11-8。https://doi.org/10.1139/G07-099.
- 69.
何平,马勇,戴辉,李玲,刘勇,李辉,等。草莓中激素和胁迫诱导的FaRE1逆转录转座子启动子的表达特性植物学报,2012;55(1):1 - 7。https://doi.org/10.1007/s12374-011-9180-9.
- 70.
关键词:ONSEN,热胁迫,ONSEN,转座子,热胁迫基因。2013;518(2):256 - 61。https://doi.org/10.1016/j.gene.2013.01.034.
- 71。
马尾松ltr -逆转录转座子激活、IRAP标记开发及其在遗传多样性评估中的潜力(Pinus Massoniana.).中国生物医学工程学报,2014;https://doi.org/10.1007/S11295-013-0677-X..
- 72。
Voronova A,Belevich V,Jansons A,rungis D.压力诱导苏格兰松树中的转算响囊样序列的转录激活(抗旱性l .)基因组。中国生物医学工程学报(英文版)。
- 73。
Esposito S, Barteri F, Casacuberta J, Mirouze M, Carputo D, Aversano R. LTR-TEs丰度,时机和移动性茄属植物commersonii和美国tuberosum低温胁迫条件下的基因组。足底。2019;250(5):1781 - 7。https://doi.org/10.1007/s00425-019-03283-3.
- 74。
向日葵根长末端重复反转录转座子表达的全基因组分析(Helianthus Annuus.l .)。遗传。2020;148(1):13-23。https://doi.org/10.1007/S10709-020-00085-4..
- 75.
Arnaud P,Goubely C,Pelissier T,Deragon JM。正弦追回龙可用于体内作为成核中心新创甲基化。中国生物医学工程学报。2000;20(10):3434-41。https://doi.org/10.1128/MCB.20.10.3434-3441.2000.
- 76.
由于缺乏染色质 - 重塑因子DDM1,阳光H,Kakutani T.转座侧翼拟南芥基因的遗传表观突变。禁止J. 2007; 26(15):3641-52。https://doi.org/10.1038/sj.emboj.7601788.
- 77.
Studer A,Zhao Q,Ross-Ibarra J,Dobley J.鉴定玉米归铁基因中的功能转座子插入TB1..自然遗传。2011; 43(11):1160-3。https://doi.org/10.1038/ng.942.
- 78.
Sigman MJ, Slotkin RK。植物转座因子抑制的首要原则是:定位,定位,再定位。植物细胞。2016;28(2):304 - 13所示。https://doi.org/10.1105/tpc.15.00869.
- 79.
Neumann P, Koblížková A, Navrátilová A, Macas j野豌豆属pannonica基因组大小通过扩增单一类型的巨型逆阻介导。遗传学。2006; 173(2):1047-56。https://doi.org/10.1534/genetics.106.056259.
- 80.
Hawkins JS, Kim H, Nason JD, Wing RA, Wendel JF。转座因子的差异谱系特异性扩增是导致基因组大小变异的原因Gossypium.Genome Res。2006; 16(10):1252-61。https://doi.org/10.1101/gr.5282906.
- 81.
等。关键词:聚类;聚类;在没有多倍化的情况下使基因组大小加倍:逆转录转位驱动的基因组扩张的动力学选用australiensis是一种水稻的野生近亲。基因组研究》2006;16(10):1262 - 9。https://doi.org/10.1101/gr.5290206..
- 82.
Meyers BC,Tingey SV,Morgante M.在玉米基因组中重复元素的丰富,分布和转录活动。Genome Res。2001; 11(10):1660-76。https://doi.org/10.1101/gr.188201.
- 83.
(1)山崎(Yamazaki M, Tsugawa H, Miyao A, Yano M, Wu J, Yamamoto S,等。水稻反转录转座子Tos17偏好低拷贝数序列作为整合靶点。中国生物医学工程学报,2001;https://doi.org/10.1007/s004380000421.
- 84。
Ellinghaus D, Kurtz S, Willhoeft U. LTRharvest,一种高效灵活的LTR反转录转座子从头检测软件。BMC生物信息学。2008;9(1):18。https://doi.org/10.1186/1471-2105-9-18.
- 85。
Steinbiss, Willhoeft U, Gremme G, Kurtz S.细粒注释和分类de novo预测LTR逆转录转座子。中国核科学研究院。2009;https://doi.org/10.1093/nar/gkp759.
- 86。
付玲,牛波,张正伟,李伟忠。基于CD-HIT的新一代测序数据聚类算法。生物信息学,2012,28(23):3150 - 2。https://doi.org/10.1093/bioinformatics/bts565.
- 87。
EMBOSS:欧洲分子生物学开放软件套件。轻拍。2000; 16(6): 276 - 7。https://doi.org/10.1016/s0168 - 9525 (00) 02024 - 2.
- 88。
Kimura M.通过核苷酸序列的比较研究估算基取代的进化率的简单方法。J Mol Evol。1980; 16(2):111-20。https://doi.org/10.1007/BF01731581.
- 89.
Mascagni F, Usai G, Natali L, Cavallini A, Giordani T.比较ltr逆转录转座子插入时间分析方法Populus Trichocarpa.基因组。鹅圈炎。2018; 71(1):85-92。https://doi.org/10.1080/00087114.2018.1429749.
- 90.
Stanke M, Mongersterne B. AUGUSTUS:一个允许用户自定义约束的真核生物基因预测网络服务器。网络服务器:W465-7。https://doi.org/10.1093/nar/gki458.
- 91.
Solorzano Zambrano L, Usai G, Vangelisti A, Mascagni F, Giordani T, Bernardi R,等。品种特异性转录组预测与注释榕辣海参L.基因组学数据。2017; 13:64-6。https://doi.org/10.1016/j.gdata.2017.07.005.
- 92.
Bolger AM, Lohse M, Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014;30(15):2114 - 20。https://doi.org/10.1093/bioinformatics/btu170.
- 93.
FastQC:高通量序列数据的质量控制工具;2010.p。370。
- 94.
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B. Mapping and quantifying哺乳动物转录组by RNA-Seq. Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B. Mapping and quantifying哺乳动物转录组by RNA-Seq。自然冰毒。2008;5(7):621 - 8。https://doi.org/10.1038/nmeth.1226.
- 95。
罗宾逊MD,麦卡锡DJ,SMYTH GK。编辑:用于数字基因表达数据的差异表达分析的生物导体包。生物信息学。2010; 26(1):139-40。https://doi.org/10.1093/bioinformatics/btp616.
- 96。
Benjamini Y,Hochberg Y.控制虚假的发现率:多种测试的实用和强大的方法。J Royal Stat Soc Ser B.995; 57:289-300。
- 97。
昆兰AR,霍尔IM。BEDTools:一套用于比较基因组特性的灵活实用程序。生物信息学,2010,26(6):841 - 2。https://doi.org/10.1093/bioinformatics/btq033.
- 98。
叶静,张勇,崔辉,刘杰,吴勇,程勇,等。WEGO 2.0:一个用于分析和绘制GO注释的web工具,2018年更新。核酸研究进展;https://doi.org/10.1093/nar/gky400.
- 99.
coesa A, Götz S, García-Gómez JM, Terol J, Talón M, Robles M. Blast2GO:用于功能基因组学研究的注释、可视化和分析的通用工具。生物信息学。2005;21(18):3674 - 6。https://doi.org/10.1093/bioinformatics/bti610.
- One hundred.
Supek F,Bošnjakm,Škuncan,Šmuct.svigo总结并可视化了基因本体论术语的长名单。Plos一个。2011; 6(7):E21800。https://doi.org/10.1371/journal.pone.0021800.
确认
本文使用的部分计算资源由ELIXIR- cz项目(LM2015047)提供,该项目是国际ELIXIR基础设施的一部分。
资金
这项研究是在FIGGEN/PRIMA19_00197项目的框架下进行的,该项目是欧洲联盟通过国家投资和投资管理部(意大利)赠款支助的PRIMA方案的一部分。
作者信息
从属关系
贡献
TG,LN和AC构思了这项研究;AV,FM和GU设计实验;AV,SS,MV和GU进行实验并分析了数据;AC,FM,AV,SS,LN和TG写了稿件。所有作者都已经阅读并赞成最终的手稿。
相应的作者
伦理宣言
伦理批准并同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有利益冲突。
附加信息
出版商的注意
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充信息
附加文件1:表S1。
在24至48天后映射到对照(C)CDNA的每个文库的全长LTR-RES上的映射读数数量和盐处理的植物。
附加文件2:表S2。
在24和48天期间显示控制(c)和胁迫叶片中的稳定表达模式的基因。仅报道显示与LTR-RE类似的低/培养基表达的基因。
附加文件3:图S1。
LTR-RES表达式的RPKM值框F. Carica.叶子。4个盒子区分别代表对照(C)和盐处理(S)植株在试验开始后24天和48天内的表达水平。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
Vangelisti, A., Simoni, S., Usai, G。等等。ltr -反转录转座子动力学榕辣海参l .)基因组。BMC植物BIOL.21日,221(2021)。https://doi.org/10.1186/s12870-021-02991-x
收到了:
接受:
发表:
关键词
- 基因组结构和进化
- 长末端重复反转录转座子
- 植物反射转运动力学
- 逆转录转座子表达式
- Retrotransposon插入时间
- 榕辣海参l