摘要
背景
Setaria Italica.是世界上第二次种植的小米,是研究C4光合作用和非生物应激耐受性的重要模型谷物作物。通过三个基因组组件和注释努力,所有基因组基于下一代测序技术,限制了基因组连续性。
结果
本文利用单分子实时测序和高通量染色体构象捕获(Hi-C)作图技术,获得了高质量的华谷11号全基因组。花鼓11基因组总装配尺寸为408.37mb,支架N50大小为45.89mb。与其他三个报道的基于下一代测序技术的谷子基因组相比,花谷11基因组具有最高的基因组连续性。种内比较表明,约94.97%和94.66%的玉谷1号和花谷11号基因组能够以一对一的方式排列,并伴有四个染色体倒位。花谷11基因组含有约19.43mb的存在/缺失变异(PAV),编码627个蛋白质转录本;玉谷1基因组含有20.53mb的PAV序列,编码737个蛋白质。总的来说,在这两个基因组中发现了969596个单核苷酸多态性(SNPs)和156282个插入缺失(InDels)。花谷11号与豫谷1号的基因组比较应在一定程度上反映谷子品种间的遗传特性和变异。乙酰羟基酸合成酶中的Ser-626-Aln取代(啊哈)与华谷11号对咪唑乙啶的耐受性有关。
结论
一种新的改进的高质量参考基因组序列Setaria Italica.对谷子的种内基因组进行了比较,确定了谷子品种间的遗传特性和变异。根据基因组序列,推测ser626 - aln在啊哈是导致华谷11号耐吡唑菌的主要原因。新改良的参考基因组Setaria Italica.将促进该物种的基因和基因组研究,有利于品种改善。
背景
谷子(脚趾斜体)在中国北方8000年前,中国驯化的气候有弹性谷物作物[1]并且主要在干旱和半干旱地区培养。它还用作研究C4光合作用,胁迫耐受性和生物能量性状的模型作物,因为其小型基因组大小和短暂的生命周期[2那3.那4.那5.那6.]. 两个谷子品系“豫谷1号”和“张谷”的基因组组装于2012年发表,加速了谷子的生物学和遗传学研究[7.那8.那9.].基于Sanger测序的“豫谷1号”基因组组装比“张谷”短读基因组组装具有更高的同源性(contig N50: 126.3 kb vs 25.8 kb),目前用作谷子的参考基因组。虽然玉谷1号参比基因组注释良好,并已在许多研究中得到应用,但其总长度401mb仅占基于k-mer分析的估计基因组大小(~ 510 Mb)的80%,分布在6778个contigs中[9.].缺失的序列主要是长度的长度为5-10kb,这难以在当时使用Sanger或下一代测序策略组装。虽然重复序列的功能很少在Foxtail Millet中研究,但他们已经证明他们在其他谷物作物中的基因表达和基因组演变中起重要作用[9.]. 支架中难以测序的间隙位于基因间区域,也可能参与基因表达的调控。定量遗传分析表明,谷子表型变异的许多原因来自调控区,因此,一个完整、高质量的基因组组装对于理解谷子气候适应性特征、基因组进化和重要农艺性状的遗传机制至关重要。
抗除草剂性状是谷子的重要性状。谷子的谷粒相对较小,使得杂草问题更加严重。杂草种子与谷子相似,种子生产中杂草种子不易清除。而且,小种子在发芽期产生小植株。很容易被杂草覆盖。如果不除草,减产将非常严重[10].除草剂的使用是现代农业中有效控制杂草的主要措施。由于原谷子品种不具有抗除草剂的能力,通过与野生近缘种杂交或化学诱变,将若干抗除草剂基因导入现代谷子品种。不同抗除草剂基因对不同除草剂具有抗性,而对同一种除草剂的抗性不同[11].理解农业生产的除草剂的分子机制非常有帮助。在育种过程中,育种者通常使用几个父母获得一种含有除草剂抗性的一种品种,这导致少量追踪除草剂抗性基因的资源。品种的基因组组装将提供一种简单的方法来揭示除草剂抗性的机制。
在这里,我们通过三种技术提供高质量的Huagu11参考基因组的组装:单分子实时测序(SMRT)染色体构象捕获序列(HI-C)和下一代测序(NGS)。与以前的品种相比,华国11种在中国西部地区种植,应该出现一款特殊的福克斯小米生态型。这种新的参考基因组有助于进一步分析和比较粪小米中的内部内部基因组的基因组多样性。通过强烈对准yugu1和华润11基因组,我们鉴定了969,596个SNP,156,282个小插入/缺失(诱导,长度短于100bp)和超过19 MB的存在/缺失 - 变异(PAV,长度超过500bp)序列之间这两个小米基因组。有趣的是,我们的比较基因组学分析揭示了广泛的拆放基因阶变异:约7.54%的基因在这两个基因组之间是非同步。两种品种在灌浆天,植物高度,灌溉颜色,千粒重和对咪唑普罗斯的反应中有不同的表型(表S1级).考虑到Ahas中的氨基酸取代可以在几种植物中产生抗除草剂咪唑吡喃吡喃的抗性[12],序列啊哈被对齐以了解两种品种的功能多样性。
结果
基因组测序和组装
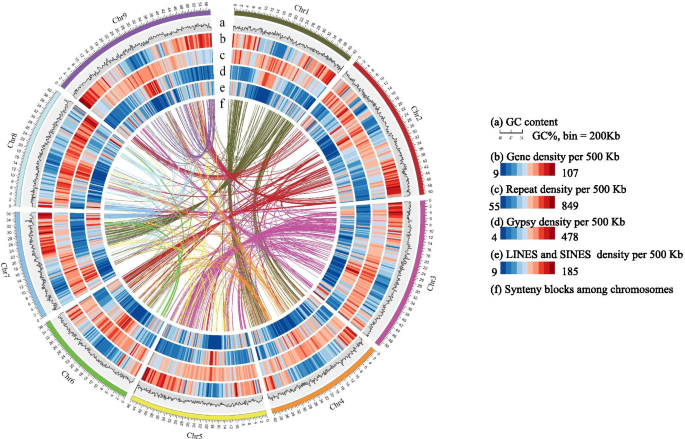
采用SMRT、Hi-C和NGS三种技术对花鼓11号基因组进行测序和组装。我们总共生成了64.43 Gb (~ 155 ×) SMRT序列,63.31 Gb高质量HiSeq干净配对reads (PE150)和34.34 Gb (~ 83 ×)有效Hi-C reads(表S2级).K-MER分析估计,华润11的基因组大小为456 MB(图S1级;桌子S3级).我们像之前报道的那样组装了基因组。首次组装仅使用PacBio SMRT数据,共产生379个N50长度为5.39 Mb的contigs(见下表)1). 用NGS读数校正重叠,然后用Hi-C数据构建支架(表1)S4级;数字S2级).Huagu11基因组总装配大小为408.37 Mb (Table .1),与最近更新的玉谷1号基因组(405.73 Mb)相似。Huagu11的组装基因组包含9个支架。支架数目与染色体数目相对应,即我们从Hi-C中得到9条假染色体。支架N50大小为45.89 Mb1),其构成了大约98%的全基因组。与其他三个报告的小米基因组相比,Huagu11基因组具有最小数量的折叠数和最长的折叠N50长度,这表明了最高的基因组连续性(表1;桌子S5级).
采用三种方法对组装的花鼓11基因组的质量和完整性进行了评价。首先,使用基准通用单拷贝同源序列(BUSCO)分析,在我们的装配中检测到约98.3%(1375个中的1351个)的胚胎植物基因,这一百分比与Yugu1基因组(97.8%)相似(表1)S6级).第二,虽然使用挖掘机轮式对齐(BWA)工具映射到组件的HiseQ短读数,但默认参数约为总读数的大约98.30%,可以很好地映射,其覆盖超过99.12%的组件(表S7级). 我们还发现约97.75%的装配被超过20倍的短NGS读数覆盖,这确保了在单核苷酸水平上装配的高准确性(表1)S7级).第三,来自NCBI的表达的序列标签(EST)序列通过BLAT软件对准Huagu11基因组组件[13使用默认参数。在超过500bp的19441条EST中,约98.61%的EST可以定位到组装好的基因组上,95.86%的EST被认为是完整的序列,因为至少90%的EST核苷酸可以定位到一个连续的支架上(Table .)S8级).这些结果表明,华润11的组装基因组具有高质量。
基因组注释
我们采用从头算结构分析和同源性比较的方法搜索基因组序列来分析重复序列。共有182 Mb(占组装基因组的44.63%)是重复序列(表1)第9部分).最丰富的亚型分别是吉普赛人和Copia样元素,分别代表组装基因组的21.55%和10.33%。剩余的转座元素是DNA转座(11.53%),长三个核心元素(线; 2.46%),短的间隙核心元素(均线,0.13%)和无表称重复(0.83%)(表第9部分).
结合蛋白同源性预测、RNA序列预测和从头预测结果,对花谷11基因组的蛋白编码基因进行了注释。花谷11基因组共预测了42,932个蛋白编码基因,其中36,652个基因被认为是高可信的,65.1%的基因可以被四种不同组织的转录组数据支持(表1)1).我们注释了基因组,发现82.5%的基因具有已知的功能(表S10).在九个假瘤组中发现了大约41,646(99.19%)的Huagu11预测基因。蛋白质编码基因主要位于染色体臂中并与转换元件的密度负相关(图。1).预测的非编码RNA基因包括91个核糖体RNA基因,976个tRNA基因,161个microRNA (miRNA)基因和503个小核RNA (snRNA)基因(表)S11).
豫谷1号和花谷11号的种内基因组比较
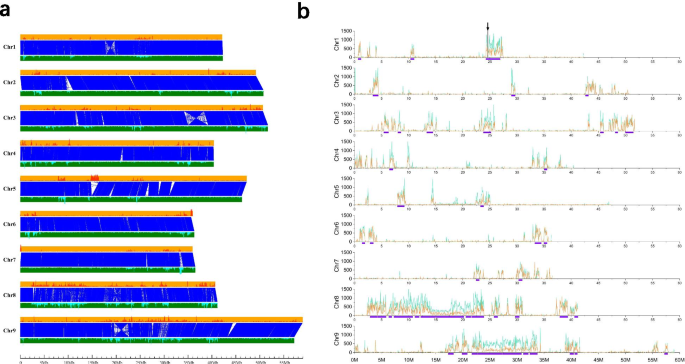
每个已发表的Foxtail Mill基因组都是与其他物种的间歇性比较。虽然,缺点比较仍然存在。考虑到yugu1基因组序列的质量非常靠近Huagu11,这将是执行梭菌比较的好机会。当我们将Huagu11 Genome与yugu1基因组在染色体尺寸的Mummer软件中对齐时[14),约94.66%的Huagu11基因组序列(381,136,172 bp)与94.97%的yugu1基因组序列(381,111,204 bp)在一对一的同位块模式中匹配。2a) 这表明这两个物种的基因组大部分区域是稳定的。我们鉴定了1858个倒位,462个内部易位和631个内部易位 ~ 基因组区域总数为17.61 Mb(表S12).在一对一共连块中观察到4个染色体倒位(图。2a) 是的。两个基因组之间的非共线序列主要是转座因子,其余为分散基因和种内特异性低拷贝序列。
通过比较yugu1和huagu11基因组,而仅计算大于500bp的DNA片段,我们还确定了18,584种Huagu11特异性基因组区段(总共19.43 MB)和18,437个yugu1特异性基因组段(总共20.53 MB)。大多数PAV碎片(99.04%的Huagu11特异性基因组段和yugu1特异性基因组段的98.43%)短于5 kB(图S3级).花鼓11号仅发现178条大于5kb的PAV序列,玉鼓1号发现294条大于5kb的PAV序列。这些PAV序列在全基因组中不均匀分布,部分序列位于集群中(图1)。2a).有趣的是,Yugu1特异性PAV序列仅1.12 Mb分布在聚类中,而Huagu11特异性PAV序列3.73 Mb分布在聚类中。我们在这些PAV区域发现了627个yugu1特异性基因组片段的蛋白编码转录本和737个huagu11特异性基因组片段的蛋白编码转录本,其中至少75%的编码序列与PAV序列重叠。特异基因注释和分类表明,花谷11号特异基因的富集网络包括:植物与病原互作、吲哚生物碱合成、Betalain生物合成、酪氨酸代谢、苯丙氨酸代谢、组氨酸代谢、异喹啉生物碱合成、花青素合成、α -亚麻酸代谢(图S4级;桌子向).Yugu1的富集网络包括氧化磷酸化、核糖体、硫胺素代谢、错配修复、泛酸和辅酶a生物合成、碱基切除修复、硫代谢、苯并嗪类生物合成、内吞作用和醚类脂质代谢(图)S5级;桌子S14系列). 有趣的是,花谷11的特异基因网络中含有花青素的生物合成,这可能是紫色颖片性状的原因。
orthofinder(v2.5.1)用于鉴定同源基因,并根据与默认参数的全节点所有BLASTP对齐进行Huagu11和Yugu1之间的基因重复事件[15那16]. 4583个基因聚集成917个花谷11特异性直系同源群,1781个基因聚集成418个玉谷11特异性直系同源群。花谷11的单拷贝基因137个在豫谷1号中重复,重复基因2798个(2.70个/基因);花谷11的单拷贝基因1785个,重复基因5750个(3.22个/基因)。花谷11号的8606个基因和玉谷1号的8829个基因聚集成3031个同源群,两个品种的多个基因聚集成一个同源群(表1)S15).
我们将花谷11基因组与玉谷1号假染色体基因组进行比对,以鉴定玉谷1号和花谷11号基因组之间的遗传变异,共鉴定出969596个SNPs和156282个indels(共617674bp),平均每kb鉴定出2.42个SNPs和0.39个indels(1.54bp)。SNPs分布与indels呈正相关(Pearson相关)R= 0.9859,P < 0.001; Fig.2b)。我们发现这些SNP没有均匀地分布在染色体上,而是聚集在一起(图S6级).SNP密度大于基因组平均密度1.5倍(每50 Kb≥180个SNP)的窗口被定义为SNP富集区。共鉴定出1593个窗口为snp富集区,占全基因组的19.78%,包含89.55%的snp。合并100 Kb内相邻的窗口,同时绘制超过450 Kb的区域(图1)。2b)。这是一种很高的可能性,这些富含Huagu11和yugu1的富集地区来自不同的遗传资源,并促进了遗传学中的表型多样性。
花谷11号抗除草剂吡虫啉的遗传机制
在Yugu1和Huagu11,对除草剂咪唑吡喃吡喃的抗性有明显差异(图。3.a)对除草剂噻嗪吡虫啉的抗性主要表现为相对抗性啊哈植物基因[17].啊哈位于华谷11号染色体1号染色体的24,623,024-24,624,955 bp处,与snp富集区相吻合(图5)。2b).包含的contig的长度啊哈基因是6,760,944bp,在翻译发起密码子之前,终止密码子后面的3,152,723bp是3,152,723bp,这意味着华宇11的基因组序列可以提供关于全方位的信息啊哈.的序列啊哈是从其它三个基因组中提取的Setaria Italica.[18].玉谷1号长207,997 bp,远短于花谷11号。的啊哈序列在张谷基因组中不完全,并且错过了TT8基因组的基因组。该现象清楚地表明,Huagu11基因组的质量更好,比其他基因组更有用。与yugu1相比,存在四个单一核苷酸突变啊哈花谷11基因(图S6级)仅引起编码蛋白的一个氨基酸变化(图。3.b).玉谷1号AHAS蛋白626位点Ser氨基酸在华谷11号转化为Asn(图)。3.b). s626n等位基因变异狗尾草那奥雅萨苜蓿那鸡脚棘球绦虫那大麦芽,Triticum aestivum.[19那20那21那22那23].携带ASN的所有品种都患上了这些物种的硫氮吡喃,而Ser的品种没有(图。3.c) 是的。为了验证单核苷酸突变,我们检测了啊哈被扩增并从四个品种和其他两种品种中进行测序。单核苷酸突变在Huagu11和Y6492中完全存在,具有除草剂抗性。剩余的品种与yugu1具有与yugu1相同的等位基因,对除草剂咪唑普罗斯(图)敏感S8级).AHA的整个编码和S626N的SNP可以在Huagu11叶片的转录组数据中找到。所有结果表明S626N都对Huagu11的耐药性抵抗力负责。
讨论
花谷11的装配基因组具有最高的连续性,这应归因于SMRT测序和Hi-C技术的应用[24].yugu1的基因组基于桑格测序技术[9.[Zhanggu和TT8基于全基因组霰弹枪 - 下一代测序策略。SMRT测序技术产生了高读数长度,这使得易于获得更长的Contig。HIC技术可以帮助生产较长的支架,并且容易锚固较长的支架和锚固到染色体。先进的技术使基因组组装经济,方便。更好的参考基因组将有助于Madding Foxtail Millet模型工厂用于C4光合作用和非生物应激耐受性的研究。
Huagu11和Yugu1的高质量组装基因组的可用性将为有关内部基因组的深入比较提供独特的机会。在一对一的同步块中,大约96%的Huagu11和Yugu1是相互同情的,远高于B73和Mo17玉米基因组的值。只有60%的B73和MO17基因组能够作为一对一的块对齐[25].花鼓11号和玉鼓1号的基因组序列具有较高的共长值,在种内比较中可以发现广泛的PVA、SNP和indel变异,以及结构变异。玉谷1号和花鼓11号基因组中PAV高强度基因1364个,其中玉谷1号627个,花鼓11号737个。两组基因组发生倒置1858次,内易位462次,间易位631次,共占约17.61 Mb基因组区域。在两个品种间共鉴定出969,596个SNPs和156,282个indels(总计617,674 bp)。这些变异导致了两个品种表型的差异。例如,一个单核苷酸多态性,导致一个氨基酸的变化啊哈花谷11对咪唑烟酸的抗性与基因有关。进一步评估这些差异对农艺性状表型变异的贡献将是一个有趣的未来研究方向。
结论
一种新的改进的高质量参考基因组序列Setaria Italica.基于SMRT测序和Hi-C作图技术组装。Huagu11基因组总组装大小为408.37 Mb,支架N50大小为45.89 Mb,是该基因组的最高连续性Setaria Italica.迄今为止。绞线物比较显示约96%的yugu1,Huagu11基因组可以被鉴定为具有四个表观染色体反转的一对一块。Huagu11基因组含有约19.43 MB的PAV,具有627个蛋白质编码转录物,而yugu1基因组在编码737蛋白的PAV序列中具有20.53 MB PAV序列。还有969,596个SNP和156,282个吉祥。基于Huagu11的基因组序列,发现AHAS中的Ser-626-AlN取代是对Huagu11的硫氮乙炔的抗性。新改良的参考基因组Setaria Italica.将促进该物种的基因和基因组研究,有利于品种改善。
方法
材料
华谷11是华大基因应用农业研究所(深圳)选育的谷子品种。华谷11号是豫谷18与Y6492的杂交后代,对除草剂咪草烟具有抗性。
采样和测序
采用CTAB法提取谷子品系‘花谷11’(NCBI Taxonomy ID: 4555)个体叶片的基因组DNA [26].所有测序库根据排序设备的质量要求构建。基于PACBIO平台制备DNA文库,SMRT时钟模板准备套件1.0(太平洋生物科公司,加利福尼亚州,美国,100-259-100)用于制备PACBIO图书馆。用PACBIO RSII测序仪P6-C4化学方法对长读DNA文库进行测序,电影长度为360分钟。根据标准程序制备HI-C库[27]. RNA是从不同的组织(根、茎和叶)中分离出来的。RNA-seq文库是使用Illumina平台(Illumina,Santiago,CA,USA)制备的。我们创造了 ~ 84.37gb配对终末数据,用SOAPfilter软件筛选原始基因组读数[28]和63.31 Gb的高质量reads用于K-mer分析和pilon分析。PacBio测序获得64.43 Gb数据,平均读长8657 bp (Table .S1级).约132.57 Gb的原始数据测序从Hi-C库和∼在质量控制后获得34.34 GB的有效读数。对于根,茎和叶,总共10.48gb,15.43 gb和10.65gb的RNA-SEQ原料数据(表S1级).然后使用SOAPFilter软件过滤原始读数,最后,10.25 GB,14.98 GB,10.37GB的高质量序列获得基因预测(表S1级).
基因组大小的估计
K-mer分析用于估计基因组大小。K-市场(K) = 17) 按水母数[29]使用14.5 Gb高质量短读。根据以下公式估算基因组大小:基因组大小 = K-mer_num/峰值深度。
基因组组装
去除长度小于1kb的PacBio reads和适配器后,用Falcon v0.3.0进行contig组装,得到64gb reads,代表Huagu11基因组的~ 155 ×测序覆盖率[30].然后,BLASR [31]被用来将PacBio SMRT获得的读数映射回contigs。SMRT用于修正部分测序错误。BWA mem用于将未使用Illumina PCR的对端reads映射到校正后的contig [32],并进一步使用高质量的读取进行组装[33].
利用Hi-C进行染色体组装
通过BWA对齐将干净的Hi-C序列定位到基因组组装。HiC-Pro用于重复阅读去除、分类和质量评估[34].Hi-C数据中25.11%为有效读数。Hi-C链接的原始计数使用Juicer [35]和3D-DNA[36].果汁盒(37]软件对离散染色质相互作用模式与玉谷1号遗传图谱相矛盾的位置和定位误差进行了调整。根据接触频率的阈值,将序列分为9组。共181个支架(占总长度的98.60%)被固定在染色体上美国italica(表S3级).
转位因子注释
从头预测和同源比对都被用来识别转座因子。结合LTR\u-Finder的结果建立了从头重复数据库[38]、堆垛机[39,及RepeatScout [40].这个De novo重复数据库连同Repbase [41]用RepeatMasker [42]并通过重复蛋白掩蔽鉴定重复相关蛋白质(http://www.repeatmasker.org/).RepeatMasker及TRF [43用来注释串联重复。根据其物理位置合并上述结果,通过爆炸进行转移元素,通过抗repbase进一步分类。
基因注释
基因预测分为同源预测、从头预测和转录组预测。基于同源性的预测,来自八个物种的蛋白质序列(Broachypodium distachyon.那大麦芽那奥雅萨苜蓿那高粱二色的那狗尾草那玉米那Setaria Italica.(张谷),和Setaria Italica.(Yugu1))(表S6级)以10的E值截距定位到花鼓11的基因组上−5,然后使用Genewise进行基因结构注释。奥古斯都(44](版本2.03)和FGENESH [45](1.3版)用于从头预测。用HISAT2处理三种组织的RNA序列数据[46]和系带[47]基于转录组的预测。证据建模器[48]利用软件对上述结果进行综合,得到最终的非冗余参考基因集。俄罗斯内部[49]、基因本体论(GO) [50],京都的基因和基因组(Kegg),Swissprot [51]、TrEMBL和Non-redundant protein NCBI数据库通过BLAST搜索(E-value cutoff 1 × 10)注释预测基因的功能−5).tRNAscan-SE [52]用于鉴定tRNA。Rfam数据库和Infernal[53]通过同源比对鉴定非编码rna(包括rRNA、miRNA和snRNA)。
基因组组装与注释的技术验证与评估
使用BWA将简短的插入尺寸读回组装,主要用于评估基因组组件的质量。为了评估基因组组件的完整性,使用具有默认参数的BLAT映射来自NCBI的EST序列。评估大会的完整性,Busco [54]使用默认参数执行。
PAV序列、聚类及基因的鉴定
我们用重叠滑动窗法鉴定了花谷11和玉谷1基因组中的PAV序列。为了鉴定花古11的特异性序列,将花古11的基因组分为500 bp的重叠窗口,步长为100 bp,产生了一系列500 bp的单端短读序列。用BWA将这些读到的片段定位到豫谷1号基因组,参数为“mem-W500-M”。如果不能与Yugu1对齐的序列,或者可以与Yugu1对齐但覆盖率小于20%的序列,则根据它们在华古11基因组中的位置合并这些序列,并定义为华古11特异性序列。以同样的方法鉴定了玉谷1号的特异序列。我们比较了花鼓11和玉鼓1基因组之间PAV序列的长度分布,发现大多数PAV序列小于2kb(图1)S3级). 相邻PAV序列(物理距离 < = 如果超过10%的合并区域被PAV序列覆盖,则定义PAV簇。PAV序列覆盖CDS区域75%以上的基因被定义为PAV基因。通过BLAST检测获得PAV基因的功能注释(E值1) × 10−5)针对InterproScan、基因本体(GO)、京都基因和基因组百科全书(KEGG)、SwissProt、TrEMBL和非冗余蛋白质NCBI数据库。使用超几何测试确定显著富集的网络[55].
花鼓11号与玉鼓1号同源基因及重复基因的研究
Huagu11和Yugu1的蛋白质序列用于鉴定同源基因,并通过Orthofinder V2.5.1研究基因重复事件,默认参数基于全部与所有BLASTP对齐方式。不能将其分配到原始基团中或聚集成特异性正原术组的基因被认为是特异性的。来自Huagu11的一个基因的正向组和来自yugu1的一个基因被认为是单拷贝同源基因对。虽然来自Huagu11的一个基因的正向群体和来自yugu1的多种基因,但我们认为该基因在yugu1中重复,反之亦然。如果来自一个物种的基因数量超过其他物种的两倍以两倍多,则在该正轨基团中存在这两个物种之间的基因重复事件。
染色体结构变异的鉴定
我们用的是木乃伊v3.23(http://mummer.sourceforge.net/(1)以花谷11号基因组为查询基因组,在参数-mum条件下,利用nucmer utility对玉谷1号基因组进行比对。(2) delta-filter工具用于过滤映射噪声,并使用参数−1 -r -q确定一对一对齐块,在参数-C -q -T下由show-snps工具报告snp,相邻的单碱基InDel合并为一个InDel。
通过通过两组参数设置过滤使用Delta-Filter实用程序来过滤Nucmer输出来完成反转和旋转性的检测分别:“-i 90 -1-q”和“-i 90-g -q”,其中用于获得允许重排的一对一对准块,并且用于获得作为全局对准的共线性区域,而不是允许重排。这些基因组重新排列区域最终定义为取决于其邻近块的位置和取向的反向或易位。
谷子品种中AHAS的分离
啊哈通过BLAST搜索从FOXTAIL MILLER GENOMES识别(E-Value Cutoff 1×10−5).氨基酸序列用clustalx软件进行比对[56].设计引物AHAS-F (ATGGCCACGACGACCGCCGC)和AHAS-R (TCAATACACGGTCCTGCCAT)扩增全长编码序列啊哈,扩增产物为1932年长度。引物由华大基因生物科技有限公司合成。50 μl反应体系为:10 μl 5 × GC Buffer, 4 μl 10 mmol dNTP, 0.5 μl 20 pmol引物,0.5 μl (100 ng) cdna (Thermo Fisher Science, Beijing, China)。在GeneAmp PCR系统9700中使用高保真Phushion DNA聚合酶(thermofisher Scientific)。实验条件如下:2分钟一个周期在98°C, 35周期在98°C 10 s, 65°C 30年代和72°C 2分钟,紧随其后的是一个周期在72°C 7 min.The PCR产品从1.0%琼脂糖凝胶中提取质粒和克隆到pGEM-T简单向量。将重组质粒转入大肠杆菌DH5α细胞,阳性克隆送上海圣工生物有限公司测序。
可用性数据和材料
支持本研究结果的数据已存入中国国家基因库数据库(CNGBdb)的CNGB序列库(CNSA),检索号为CNP0000993(https://db.cngb.org/search/project/CNP0000993/).
缩写
- 高c:
-
高通量染色体构象捕获
- 奶油水果蛋白饼:
-
存在/不存在变化
- SNP:
-
单核苷酸多态性
- InDels:
-
插入删除
- 啊哈:
-
Acetohydroxy酸合酶
- SMRT:
-
单分子实时测序
- Busco:
-
基准普遍单拷贝直脑道
- BWA公司:
-
Burrows-Wheeler-Alignment
- 美东时间:
-
表达序列标签
- 线条:
-
长散核元素
- 锡:
-
短途核心核心要素
- miRNA:
-
microRNA.
- 单核苷酸核糖核酸:
-
小核RNA
- 聚合酶链反应:
-
聚合酶链反应
- 走:
-
基因本体论
- 小桶:
-
京都基因和基因组百科全书
参考
- 1.
Barton L,Newsome SD,Chen F-H,Wang H,Guilderson TP,Bettinger RL。中国北方归国的农业起源与同位素认同。pnas。2009; 106(14):5523。
- 2.
Brutnell TP,Wang L,Swartwood K,Goldschmidt A,Jackson D,Zhu XG,Kellogg E,Van Eck J.Setaria Viridis:C4光合作用型号。植物细胞。2010; 22(8):2537-44。
- 3.
dust AN, Kellogg EA, Devos KM, Bennetzen JL。谷子:一种顺序驱动的草模型系统。植物杂志。2009;149(1):137 - 41。
- 4.
李博士,布鲁特内尔博士。狗尾草和意大利狗尾草,圆锥花序草的模式遗传系统。J实验机器人。2011;62(9):3031–7.
- 5.
Muthamilarasan M, Khan Y, Jaishankar J, Shweta S, Lata C, Prasad M.对C-4生物燃料模型狗尾草(Setaria italica)次生细胞壁基因的整合分析和表达谱揭示了木质纤维素生物工程的靶点。植物科学与技术,2015;
- 6.
作者:Muthamilarasan, Singh RK, Suresh BV, Rana S, Dulani P, Prasad M.解析了4种猫尾狗尾草(Setaria italica)和狗尾草(Setaria viridis)的胁迫应答基因。生物科技J》。2020;318:57 - 67。
- 7.
Lata C,Gupta S,Prasad M. Foxtail小米:生物能源草中遗传和基因组研究的模型作物。Crit Rev Biotechnol。2013; 33(3):328-43。
- 8.
张刚,刘旭,权哲,程森,徐鑫,潘森,谢敏,曾平,岳忠,王伟,等。谷子(狗尾草)基因组序列提供了深入了解草的进化和生物燃料潜力。生物科技Nat》。2012;30:549。
- 9
Bennetzen JL, Schmutz J, Wang H, Percifield R, Hawkins J, Pontaroli AC, Estep M, Feng L, Vaughn JN, Grimwood J, et al. .模式植物狗尾草的参考基因组序列。生物科技Nat》。2012;30(6):555 - 61。
- 10.
李泽,静X,李H,李,Xin L,MU T.福克斯米幼苗幼苗除草剂的安全和杂草控制效率。庄稼。2017; 1(5):155-9。
- 11.
枝江。谷子抗除草剂基因的发现与应用。基因组学应用生物学。2010;294:768–74.
- 12
夏W,潘L,李继,王Q,冯y,董L。脆赛狐尾(Alopecurus aequalis荞麦面癌的Als-and /或Accase抑制剂的分子基础。Perfist Biochem physiol。2015; 122:76-80。
- 13
Birney E, Clamp M, Durbin R. GeneWise和genomic wise。基因组研究》2004;14(5):988 - 95。
- 14
《京都基因与基因组百科全书》。中国生物医学工程学报2000;28(1):27-30。
- 15
EMMS DM,Kelly S. Orthofinder:对比较基因组学的系统发育外科学推断。基因组Biol。2019; 20(1):238。
- 16.
EMMS DM,Kelly S. Orthofinder:解决整个基因组比较的基本偏差显着提高了正交推理准确性。基因组Biol。2015; 16(157):157。
- 17.
余强,鲍斯SB.对AHAS抑制剂除草剂的抗性:当前了解。中国生物防治学报;2014;
- 18
蔡坤杰,卢明杰,杨坤杰,李美美,滕永昌,陈森,顾MSB,李文华。装配狗尾草意大利L. Beauv。基因组进入9条染色体,并深入了解影响生长和耐旱的区域。Sci Rep-Uk。2016; 6(1): 35076。
- 19
Laplante J,Rajcan I,Tardif FJ。乙酰羟基酸合成酶的多种等位基因形式与狗尾草的抗除草剂性有关。理论应用基因。2009;119(4):577–85.
- 20.
Rajguru SN、Burgos NR、Shivrain VK、Stewart JM。红米ALS基因突变与抗咪草烟有关。杂草科学。2005;53(5):567–77.
- 21.
Matzenbacher Fo,Bortoly Ed,Kalsing A,Merotto AJ。Barnyardgrass(Echinochloa Crus-Galli)抵抗机制的分布与分析咪唑啉酮和醋椒醋酸除草剂。j农业sci。2015; 153(6):1-15。
- 22.
Pozniak CJ, Hucl PJ。普通小麦突变衍生系咪唑啉酮抗性的遗传分析。作物科学。2004;(1):44 23-30。
- 23.
Lee H,Rustgi S,Kumar N,Burke I,Yenish JP。大麦丙基羟基酸合酶(AHAs)基因中的单核苷酸突变赋予咪唑啉酮除草剂的抗性。pnas。2011; 108(21):8909-13。
- 24
马军,向勇,熊勇,林智,薛勇,毛敏,孙玲,周勇,李学忠,黄晓明。SMRT测序分析揭示了Ananas comosus var. bracteatus的全长转录本和选择性剪接模式。同行j . 2019; 7: e7062。
- 25
Sun SL,周Ys,Chen J,Shi JP,Zhao HM,Zhao Hn,Song WB,Zhang M,Cui Y,Dong XM等。MO17与其他玉米基因组的广泛的拆放基因令和基因结构变化。NAT Genet。2018; 50(9):1289-95。
- 26
穆雷MG,汤普森WF。高分子量植物DNA的快速分离。《核酸研究》1980;8(19):4321-6。
- 27
Lieberman Aiden E,Van Berkum NL,Williams L,Imakaev M,Ragoczy T,Telling A,Amit I,Lajoie BR,Sabo PJ,Dorschner MO.长程相互作用的综合图谱揭示了人类基因组的折叠原理。科学。2009;326(5950):289–93.
- 28.
罗河,刘b,谢y,李z,黄某,元j,他g,chen y,pan q,liu y等。Soapdenovo2:经验化改进的记忆效率短读De Novo汇编器。傻瓜。2012; 1(1):18。
- 29.
Marçais G,Kingsford C。一种快速、无锁的k-mers并行计数方法。生物信息学。2011;27(6):764–70.
- 30.
Chin C- s, Peluso P, Sedlazeck FJ, Nattestad M, Concepcion GT, Clum A, Dunn C, O’malley R, Figueroa-Balderas R, Morales-Cruz A等。单分子实时测序的分阶段二倍体基因组组装。Nat方法。2016;13:1050。
- 31.
Chaisson MJ,Tesler G.使用基本局部对准与连续改进(BLASR)的基本局部对齐进行读取单分子测序读取的单分子测序读取:应用和理论。BMC生物信息学。2012; 13(1):238。
- 32.
李赫。将序列读取、克隆序列和组装重叠与BWA-MEM对齐。2013年ArXiv;1303
- 33.
Walker BJ,Abeel T,Shea T,Prist M,Abouelliel A,Sakthikumar S,Cuomo CA,Zeng Q,Wortman J,Young SK,et al.Pilon:综合微生物变异检测和基因组组装改进的综合工具。公共科学图书馆一号。2014;9(11):e112963。
- 34.
HiC-Pro:用于高温数据处理的优化和柔性管道。基因组医学杂志。2015;16(1):259。
- 35.
Durand NC, Shamim MS, Machol I, Rao SSP, Huntley MH, Lander ES, Aiden EL。榨汁机提供了一个一键式系统,用于分析环路分辨率的高温实验。细胞系统。2016;3(1):95 - 8。
- 36
Dudchenko O、Batra SS、Omer AD、Nyquist SK、Hoeger M、Durand NC、Shamim MS、Machol I、Lander ES、Aiden AP等。使用Hi-C对埃及伊蚊基因组进行从头组装产生染色体长度支架。科学。2017;356(6333):92.
- 37
Durand NC, Robinson JT, Shamim MS, Machol I, Mesirov JP, Lander ES, Aiden EL。果汁盒提供了一个可视化系统的Hi-C接触地图无限缩放。细胞系统。2016;3(1):99 - 101。
- 38
XU Z,Wang H.LTR_Finder:一种有效的工具,用于预测全长LTR回收输送。核酸RES。2007; 35(SUPPLE_2):W265-8。
- 39
埃德加·RC,迈尔斯EW。基因组重复序列的鉴定和分类。生物信息学。2005;21 (suppl_1): i152-8。
- 40.
价格Al,Jones NC,PEVZNER PA。大型基因组中重复家族的义目鉴定。生物信息学。2005; 21(SUPPLE_1):I351-8。
- 41.
应用RepeatMasker技术鉴定基因组序列中的重复元件。生物信息学学报。2009;25(1):4.10.11-14.10.14。
- 42.
jurka j,Kapitonov vv,Pavlicek A,Klonowski P,Kohany O,WalichiewiCz J. Repbase更新,一种真核性重复元素的数据库。细胞基因组。2005; 110(1-4):462-7。
- 43.
串联重复序列发现者:一个分析DNA序列的程序。微生物学杂志1999;27(2):583 - 589。
- 44.
Stanke M,Steinkamp R,Waack S,Morgenstern B.奥古斯斯图斯:用于基因在真核生物中发现的网页服务器。核酸RES。2004; 32(SUPPLE_2):W309-12。
- 45.
Salamov Aa,Solovyev VV。AB Initio基因在果蝇基因组DNA中发现。Genome Res。2000; 10(4):516-22。
- 46.
Kim D,Langmead B,Salzberg SL。Hisat:一种快速拼接对齐器,内存要求低。NAT方法。2015; 12:357。
- 47.
Pertea M,Pertea gm,Antonescu Cm,Chang T-C,Mendell JT,Salzberg SL。Stringtie使得能够从RNA-SEQ读取改进转录组的重建。NAT BIOTECHNOL。2015; 33:290。
- 48
Haas BJ,Salzberg SL,Zhu W,Pertea M,Allen JE,Orvis J,White O,Buell CR,Wortman JR.使用EVidenceModeler和程序组装拼接比对的自动化真核基因结构注释。基因组生物学。2008;9(1):R7。
- 49
Quevillon E,Silventoinen V,Pillai S,Harte N,Mulder N,Apweiler R,Lopez R.Ingroscan:蛋白质域标识符。核酸RES。2005; 33(Web服务器问题):W116-20。
- 50。
Ashburner M、Ball CA、Blake JA、Botstein D、Butler H、Cherry JM、Davis AP、Dolinski K、Dwight SS、Eppig JT等,《基因本体论:生物学统一的工具》。纳特·吉内特。2000;25(1):25–9.
- 51。
Uniprot C.通用蛋白质资源(Uniprot)。核酸RES。2006; 35(SUPPLE_1):D193-7。
- 52。
tRNAscan-SE:一种改进基因组序列中转移RNA基因检测的程序。核酸杂志1997;25(5):955-64。
- 53.
Nawrocki EP, Kolbe DL, Eddy SR. Infernal 1.0: RNA比对推理。生物信息学,2009,25(10):1335 - 7。
- 54.
Simão FA,Waterhouse RM,Ioannidis P,Kriventseva EV,Zdobnov EM.BUSCO:用单拷贝正射基因评估基因组组装和注释完整性。生物信息学。2015;31(19):3210–2.
- 55.
竞争对手我,personnaz l,taing l,potier mc。在一类基因中的富集或耗尽GO类别:哪种测试?生物信息学。2007; 23(4):401-7。
- 56.
Larkin Ma,Blackshields G,Brown NP,Chenna R,McGettigan Pa,McWilliam H,Valentin F,Wallace Im,Wilm A,Lopez R等人。Clustal W和Clustal X 2.0版。生物信息学。2007; 23(21):2947-8。
致谢
感谢《BMC植物生物学杂志》的两位审稿人和编辑对我们的稿件进行了认真的审稿,并提出了一些有价值的建议。感谢西北高原生物研究所的支持。
资金
该作品得到了中国深圳市科学,技术和创新委员会的支持。ZYC201105170390A和深圳大鹏新区科技,技术,创新和工业发展专项资金(肯定克吉港议定书号KJYF202001-11)。资金机构在设计和撰写稿件的研究和收集,分析和解释方面没有作用。
作者信息
从属关系
贡献
GP、BL和XN构思并设计了实验。JW, GH, MX, GD进行实验,JW, SL, LL, XG进行数据分析。JW和SL撰写稿件,BL和GP审阅并修改论文。所有作者均已阅读并批准稿件。
相应的作者
道德宣言
伦理批准并同意参与
不适用。
同意出版
不适用。
利益争夺
作者和华大基因深圳公司声明他们没有利益冲突。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1
.K-MER分析估算华宇11的基因组大小。
附加文件2:图S2
.集合contigs的high C连锁密度热图。
附加文件3:图S3
.花谷11号和玉谷1号基因组PAV序列长度分布。大部分片段小于5kb。
附加文件4:图S4
.花鼓11特异性基因的富集网络。
附加文件5:图S5
. 豫谷1号特异基因的富集网络。
附加文件6:图S6
. 染色体中遗传差异较大的染色体片段所占的比例。Chr8、Chr9和Chr3的百分率较高。
附加文件7:图S7
.花鼓11号和玉鼓1号基因组AHAS序列比对。
附加文件8:图S8
.氨基序列与栽培品种的氨基序列对齐与咪唑的不同抗性。
附加文件9:表S1
. 豫谷1号和华谷11号的主要不同表型。
附加文件10:表S2
.生成的测序数据列表。
附加文件11:表S3
. 17个月的统计分析。
附加文件12:表S4
.Hi-C结果的统计数据。
附加文件13:表S5
.Huagu11,Yugu,Zhanggu和TT8中的组装基因组比较。
附加文件14:表S6
.用BUSCO组进行基因组组装完整性评估。
附加文件15:表S7
.映射读取到基因组组装。
附加文件16:表S8
.使用29399 EST序列评估Huagu11基因组组件。
附加文件17:表S9
.谷子基因组中的重复元素。
附加文件18:表S10
.谷子基因组中的基因功能注释。
附加文件19:表S11
. 谷子基因组中的非编码RNA注释。
附加文件20:表S12
. 花谷11和玉谷1基因组重排细节。
附加文件21:表S13
.利用Nr(20170924)、KEGG (v89.1)、Interpro (interproscan-5.30-69.0)和GO (gene_ontology.1_2)数据库对花谷11 PAV特异基因进行功能注释。
附加文件22:表S14
. 利用Nr(20170924)、KEGG(v89.1)、Interpro(interproscan-5.30-69.0)和GO(gene\ u ontology.1\ u 2)数据库对Yugu1-PAV特异基因进行功能注释。
附加文件23:表S15
.不同同源组的基因数量统计。
权利和权限
开放存取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/. 知识共享公共领域放弃(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
王,J.,李,S.,兰,L。等等。De Novo基因组组装的福克尾部品种Huagu11对玉氮含量耐受的玉氮遗传学差异,遗传机制。BMC植物杂志21,271(2021)。https://doi.org/10.1186/s12870-021-03003-8
收到了:
公认:
发表:
关键词
- Setaria Italica.
- 基因组
- 比较基因组分析
- Acetohydroxy酸合酶
- Imazethapyr宽容