- 研究文章GyD.F4y2Ba
- 开放访问GyD.F4y2Ba
- 发表:GyD.F4y2Ba
基因组 - 范围的关联分析显示在棉花的多场环境下的产量部件中涉及的定量性状性状基因座和候选基因(GyD.F4y2Ba陆地棉GyD.F4y2Ba)GyD.F4y2Ba
BMC植物生物学GyD.F4y2Ba体积GyD.F4y2Ba21.GyD.F4y2Ba文章编号:GyD.F4y2Ba250.GyD.F4y2Ba(GyD.F4y2Ba2021.GyD.F4y2Ba)GyD.F4y2Ba
抽象的GyD.F4y2Ba
背景GyD.F4y2Ba
通过全基因组关联研究(GWAS)分析,在棉花中发现了大量与产量性状相关的数量性状位点(qtl)和候选基因。然而,表型数据大多来自单一或少数环境,稳定位点尚需在多个野外环境下进行验证。GyD.F4y2Ba
结果GyD.F4y2Ba
本研究对242份陆地棉种质资源进行了连续的表型分析,包括13种田间环境下的铃重和衣分,11种环境下的单株铃数和种子指数等4个主要产量组分的表型数据。相关分析显示BN与LP、BW、SI呈正相关,SI与LP、BN呈负相关。遗传分析表明,LP的遗传力最高,为94.97%,SI次之,为92.08%,BW为86.09%,BN为72.92%,表明LP和SI是更适合遗传改良的性状。基于56,010个高质量单核苷酸多态性(SNPs)和GWAS分析,共鉴定出95个非冗余qtl,其中BN 12个,BW 23个,LP 45个,SI 33个。其中,在A和D亚基因组间检测到10对同源qtl。在6个以上的环境中,共定位的2个以上性状的qtl分别为15个和12个高信度qtl。此外,两个NET基因(GyD.F4y2Bagh_a08g0716GyD.F4y2Ba和GyD.F4y2BaGH_A08G0783GyD.F4y2Ba),位于新型QTL热点(QTL24,QTL24,QTL25和QLT26)中主要在早期纤维发育阶段表达,与LP和Si显着相关。这GyD.F4y2Bagh_a07g1389GyD.F4y2Ba在稳定的QTL19区域中,编码了四肽重复(TPR) - 般的超家族蛋白,并且是涉及短纤维突变体Ligon无线-Y的同源基因(LiGyD.F4y2BayGyD.F4y2Ba),表明对棉花产量有重要影响。GyD.F4y2Ba
结论GyD.F4y2Ba
本研究为了解产量成分的调节机制,提供了通过棉花的分子育种来提高产量改善的基础。GyD.F4y2Ba
背景GyD.F4y2Ba
棉是一种重要的商业作物,可在全球范围内提供最具天然的纤维,也是食用油的重要来源。栽培棉质种类,高地棉花(GyD.F4y2Ba陆地棉GyD.F4y2Ba(L.)由于其高产和广泛的适应性,占棉花总产量的95%以上[GyD.F4y2Ba1GyD.F4y2Ba].开发高产品种是棉花育种中必不可少的目标之一。然而,通过常规育种计划的棉花产量的提高仍然低,因为旱地棉的狭窄遗传背景导致繁殖瓶颈[GyD.F4y2Ba2GyD.F4y2Ba].因此,通过分子育种方法探索和构建与产量构成因素相关的优质数量性状位点(qtl)/基因,对提高棉花产量具有重要意义。GyD.F4y2Ba
棉花产量组分主要包括每株植物(BN),棉铃重量(BW),棉绒百分比(LP),种子指数(SI)和棉绒指数(LI),其定量继承并容易受到的影响环境 [GyD.F4y2Ba3.GyD.F4y2Ba].利用分子标记和双亲本连锁作图分析已鉴定出几个与棉花产量相关性状的qtl [GyD.F4y2Ba4.GyD.F4y2Ba那GyD.F4y2Ba5.GyD.F4y2Ba].然而,由于标记数量有限和大型QTL区域,通过标记辅助育种来利用QTL挑战挑战。随着高通量测序技术和统计方法的快速发展,棉花产量相关性状的遗传基础已经预先揭示。近五年来,四倍体栽培棉麻基因组的组装和改善显着加速了基因的测绘,为棉花的重要特征[GyD.F4y2Ba6.GyD.F4y2Ba那GyD.F4y2Ba7.GyD.F4y2Ba那GyD.F4y2Ba8.GyD.F4y2Ba那GyD.F4y2Ba9.GyD.F4y2Ba那GyD.F4y2Ba10.GyD.F4y2Ba那GyD.F4y2Ba11.GyD.F4y2Ba].基于参考基因组序列,通过基因组 - 宽协会研究(GWAS)分析鉴定了与产量相关性状相关的大量QTL和候选基因[GyD.F4y2Ba12.GyD.F4y2Ba那GyD.F4y2Ba13.GyD.F4y2Ba那GyD.F4y2Ba14.GyD.F4y2Ba那GyD.F4y2Ba15.GyD.F4y2Ba].基因组关联分析的力量主要基于四个因素:遗传多样性的丰富性,特质采集,标记密度和统计方法的真实性[GyD.F4y2Ba16.GyD.F4y2Ba].在以往的研究中,由于单一环境的实验设计和单基因座GWAS方法,许多稳定的基因座仍有待检测。多环境、多位点GWAS结合改进的实验设计和相关方法,可提高对纤维产量性状相关qtl /基因的挖掘效率,这在棉花育种中仍是一个挑战。GyD.F4y2Ba
在本研究中,多年来,在多年来,在多年来,在多年来,在多年内为四个主要产量组分,BW和LP的表型调查,在13个自然环境下进行的表型调查,以及11个环境下的BN和Si,在多年内植入多元化的棉花。基于多基因座随机SNP效应混合线性模型进行GWAS分析,在多种环境中揭示了与产量组分相关的稳定QTL。结合转录组分析,研究了候选基因的表达模式,预测了促进棉花产量的关键基因。结果可能有助于了解产量特征的遗传架构更好,并提供用于通过分子育种设计高产棉线的分子标记和候选基因。GyD.F4y2Ba
结果GyD.F4y2Ba
四种产量相关性状的表型变异GyD.F4y2Ba
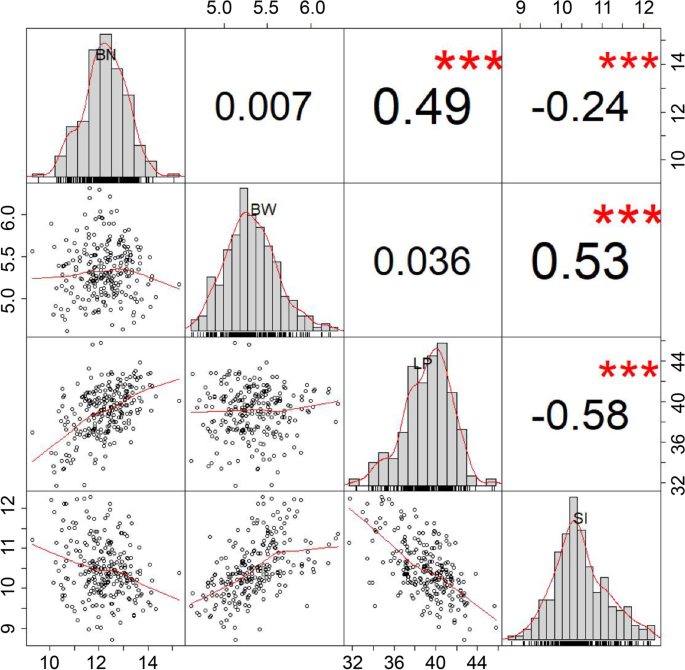
在多场环境中分析了四个产量相关的特征吹嘴(BN),棉铃重(BW),棉绒百分比(LP)和种子指数(SI)的表型数据,以评估天然人群的表型变异242Upland Cotton Rencess(附加文件GyD.F4y2Ba1GyD.F4y2Ba表S1)。BN、BW、LP和SI分别在1.7 ~ 35.5、2.5 g ~ 9.1 g、23.0% ~ 50.2%和6.7 g ~ 17.1 g之间差异显著GyD.F4y2Ba2GyD.F4y2Ba表S2)。BN变异系数最大,为13.13% ~ 24.26%,LP变异系数最小,为7.19% ~ 10.53%。在多个环境下的最佳线性无偏预测(BLUP)显示,BLUP的BN、BW、LP和SI的表型值分别为9.3 ~ 15.3、4.6 g ~ 6.3 g、31.7% ~ 45.8%和8.7 g ~ 12.3 gGyD.F4y2Ba2GyD.F4y2Ba表S2,附加文件GyD.F4y2Ba3.GyD.F4y2Ba图S1)。广义遗传性(GyD.F4y2BaHGyD.F4y2Ba2GyD.F4y2Ba)根据多环境表型数据估计每个特征(表GyD.F4y2Ba1GyD.F4y2Ba)探索特征的育种潜力。对LP(94.97%)和Si(92.08%)发现的高遗传性估计,其次是BW(86.09%)。然而,BN(72.92%)的可遗传性最低,表明BN倾向于受环境的影响。进行差异分析(ANOVA)以研究基因型(G),环境(E)和G×E相互作用的多环境中的四个特征的影响(表GyD.F4y2Ba1GyD.F4y2Ba)。结果表明,这些特征也显着进行了基因型和环境相互作用。相关分析显示BN和LP之间的正相关,而Si显示与BW的正相关和与BN和LP的负相关(图。GyD.F4y2Ba1GyD.F4y2Ba)。GyD.F4y2Ba
基因组关联分析GyD.F4y2Ba
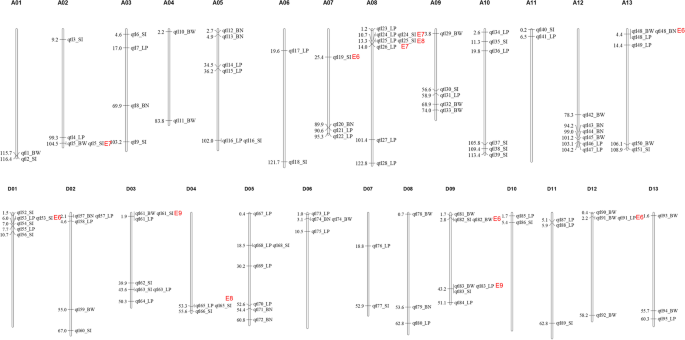
为了探讨与四种产量相关的特征相关的遗传因素,我们使用56,010高质量SNP的基因型数据进行了GWAS分析([GyD.F4y2Ba17.GyD.F4y2Ba),额外的文件GyD.F4y2Ba4.GyD.F4y2Ba表S3)和多种环境中的四个产量相关性状的表型数据和结合。总共有560个定量性状核苷酸(QTNS)被鉴定为与四个特征有显着相关的基因座。用200 kB作为将QTN合并到相同QTL中的链接不平衡(LD)阈值,从所有环境和BLUP中识别了总共360 QTL的四个特征(附加文件GyD.F4y2Ba5.GyD.F4y2Ba表S4)。此外,横跨至少两个环境一致的QTL被声明为稳定的QTL;因此,鉴定了95个候选QTL,其中BN,23的BW,45的LP和33的Si(图。GyD.F4y2Ba2GyD.F4y2Ba和附加文件GyD.F4y2Ba6.GyD.F4y2Ba表S5)。其中38名与以前的研究共同配合(附加文件GyD.F4y2Ba6.GyD.F4y2Ba表S5)和57是新颖的,并在目前的研究中首次报道。此外,在A和D子基因组之间检测到10对同源QTL(附加文件GyD.F4y2Ba7.GyD.F4y2Ba表S6),表明两种子基因组的常见选择,以改善Upland棉中的产量组分。GyD.F4y2Ba
同时检测到15个QTL,与两个或更多个特征相关联(附加文件GyD.F4y2Ba6.GyD.F4y2Ba表S5,附加文件GyD.F4y2Ba8.GyD.F4y2Ba图。S2)。例如,识别为LP和Si的九个常见QTL,与两个特征之间的高相关(R = -0.58)一致。这些候选QTL在与DT子基因组相比的次基因组(附加文件)时,在26个染色体上广泛分布在26个染色体上GyD.F4y2Ba9.GyD.F4y2Ba图S3)。GyD.F4y2Ba
在超过六个环境中始终检测到的QTL被定义为高频率QTL;因此,鉴定了12个高置信QTL(图。GyD.F4y2Ba2GyD.F4y2Ba,附加文件GyD.F4y2Ba6.GyD.F4y2Ba表S5)。例如,QTL24与七个环境中的LP和六个环境中的QTL19相关联。此外,发现三个QTL同时与三种性状相关联:QTL48被识别为在六种环境中与BW,BN和LP共定位;QTL61和QTL83与BW,Si和LP,九种环境,暗示了普通棉中这些QTL的遗传稳定性。GyD.F4y2Ba
鉴定产量相关QTL区域中候选基因GyD.F4y2Ba
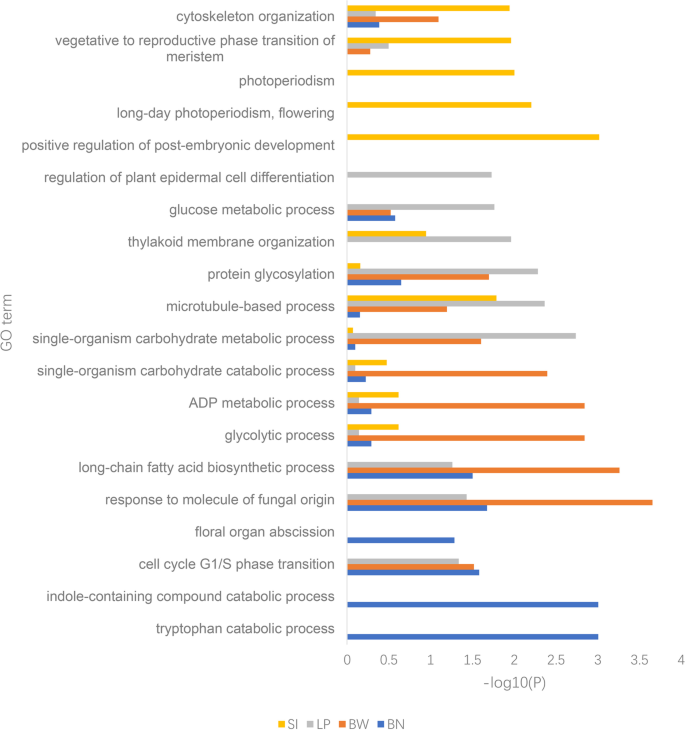
在释放的基础上提取棉花组织中定位于候选QTL区域且表达量大于3 transcripts per million (TPM)的基因GyD.F4y2BaG. HirsutumGyD.F4y2BaTM-1基因组[GyD.F4y2Ba11.GyD.F4y2Ba].共有4144个基因位于候选QTL区域中,547与BN,1096至BW,2236至LP和1430相关。通过过滤低表达的基因,鉴定了1490个候选基因,347次与BN,407至BW,606至LP和424相关的347(附加文件GyD.F4y2Ba10.GyD.F4y2Ba表S7)。单独进行每个性状的基因本体(GO)分析以研究候选基因的功能(图。GyD.F4y2Ba3.GyD.F4y2Ba,附加文件GyD.F4y2Ba11.GyD.F4y2Ba表S8)。结果表明,来自不同性状qtl区域的候选基因在不同的生物学过程中显著富集。LP基因的功能主要富集在与糖代谢相关的过程中,如单生物碳水化合物代谢过程、葡萄糖代谢过程、碳水化合物生物合成过程等。此外,还丰富了与纤维发育密切相关的其他过程,如基于微管的过程、类囊体膜组织、植物表皮细胞分化调控、植物型次生细胞壁生物发生等。SI优先富集胚胎发育和开花相关过程,如胚胎后期发育、长日光周期、开花和生殖期过渡分生组织的营养。BW候选基因涉及能量代谢过程,包括长链脂肪酸生物合成过程、糖酵解过程、ADP代谢过程和单生物碳水化合物分解过程。BN富集的过程包括色氨酸分解过程、细胞周期G1/S期转变和花器官脱落。总的来说,各性状QTL区富集的主要生物学过程与这些产量组分的发育过程高度相关。GyD.F4y2Ba
与产量相关性状相关的关键QTL和基因GyD.F4y2Ba
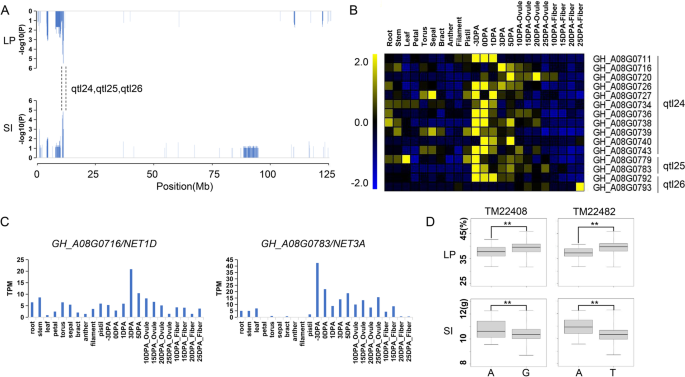
在12个高可信QTL中,我们发现了一个QTL热点(A08: 10.7-14.6 Mb),其中包含3个新的QTL,分别是LP、SI和LP的qtl24、qtl25和qlt26(图2)。GyD.F4y2Ba4.GyD.F4y2Baa).通过组织和器官转录组分析,我们发现15个位于稳定QTL热点的基因在纤维发育过程中主要表达于胚珠或纤维中(图2)。GyD.F4y2Ba4.GyD.F4y2Bab)。其中,两个编码激酶相互作用(KIP1样)家族蛋白基因(GyD.F4y2Bagh_a08g0716GyD.F4y2Ba和GyD.F4y2BaGH_A08G0783GyD.F4y2Ba),它的“编码植物特异性网络(净)超家族的肌动蛋白结合蛋白质的”正交基因“GyD.F4y2Ba拟南芥GyD.F4y2Ba,分别在qtl24和qtl25中鉴定。这两个基因在纤维发育过程中高度表达,特别是在纤维发育的早期阶段。GyD.F4y2Ba4.GyD.F4y2BaC)。此外,分别选择最接近两个净基因的两个QTNS(TM22408和TM2482)以研究基因与产率组分之间的相关性。通过比较两种基因型的表型差异(图。GyD.F4y2Ba4.GyD.F4y2Bad),具有G基因型的LP值显着高于QTN TM22408中的基因型,而G基因型较低。在QTN TM22482中,T基因型显示出比基因型更高的LP值和更低的Si值。结果表明,这两个基因在增加LP时具有潜在的功能。我们还发现了两个基因QTL24:GyD.F4y2Bagh_a08g0734GyD.F4y2Ba和QTL26:GyD.F4y2BaGH_A08G0793GyD.F4y2Ba,分别高于0dPa和25 dPa的纤维开发。GyD.F4y2Bagh_a08g0734GyD.F4y2Ba编码蛋白质磷酸酶2a(pp2a)调节b亚基家庭蛋白,以调节芸苔类固醇介导的信号通路[GyD.F4y2Ba18.GyD.F4y2Ba].GyD.F4y2BaGH_A08G0793GyD.F4y2Ba编码双向糖转运蛋白甜味剂,也在纤维发育中起着至关重要的作用[GyD.F4y2Ba19.GyD.F4y2Ba].GyD.F4y2Ba
由于衍生自种子外壳表皮的专用毛发状纤维,棉纤维和种子发育同样重要。稳定的QTL,大多数与LP和Si同时相关。我们发现,通过转录简谱分析,大多数候选基因在纤维发展早期阶段的胚珠中表达,表明Si在纤维产率中发挥着重要作用。鉴定QTL19与染色体A07的Si和17个候选基因相关联25.4-26.5 MB。在光纤开发期间,大多数基因大多数高于-3 dPA至1 dPa(附加文件GyD.F4y2Ba12.GyD.F4y2Ba图S4a)。GyD.F4y2Bagh_a07g1389GyD.F4y2Ba编码的四肽重复(TPR) - 般的超家族蛋白质,以前据报道与Ligon-无突变突变表型相关,并且在纤维发育期间从-3dPa至1dPa高度表达(附加文件GyD.F4y2Ba12.GyD.F4y2Ba图4B)。利用最近的QTN TM19889,具有G基因型的LP值显着高于与基因型的LP值,而Si值较低,G基因型(附加文件GyD.F4y2Ba12.GyD.F4y2Ba图。S4C)。我们还发现,在九种不同环境中检测到的QTL83(D09:43.2-44.0MB)同时与BW,LP和Si相关联。在QTL83区域内,鉴定了28个候选基因,其中大多数参与纤维发育,例如GyD.F4y2BaGH_D09G1670GyD.F4y2Ba(GyD.F4y2Baexpa4.GyD.F4y2Ba),GyD.F4y2Bagh_d09g1688GyD.F4y2Ba(GyD.F4y2Baagd7.GyD.F4y2Ba) 和GyD.F4y2BaGH_D09G1611GyD.F4y2Ba(GyD.F4y2BaARF16GyD.F4y2Ba),表明它们在产量组分形成中的重要作用。GyD.F4y2Ba
讨论GyD.F4y2Ba
提高产量是作物育种实践中的第一种育种目标。棉是一种重要的经济作物,其产量组分主要包括棉铃数,棉铃重量,棉绒百分比,种子指数,棉绒指数等。发现QTL和候选基因有关棉花遗传改善的产量性状有关的重要意义。发展高产品种。随着基因组范围的序列信息的释放,通过GWAS分析鉴定了许多与棉花产物特征相关的QTL和基因进行鉴定[GyD.F4y2Ba12.GyD.F4y2Ba那GyD.F4y2Ba20.GyD.F4y2Ba那GyD.F4y2Ba21.GyD.F4y2Ba那GyD.F4y2Ba22.GyD.F4y2Ba].然而,产量组分是由多种基因控制的复杂量性状,受环境影响。大多数GWAS方法都基于固定的SNP效应混合线性模型(MLM)和单标分析,需要严格校正GyD.F4y2BaP.GyD.F4y2Ba值并包含许多次要效果QTL。以前使用多基因座混合线性模型的研究还表明该模型可以提高关联分析的功率和稳健性[GyD.F4y2Ba23.GyD.F4y2Ba].本研究对242份不同产地的陆地棉花进行了多年的种植和调查。通过多位点混合线性模型GWAS分析,系统揭示了4个主要产量组分的稳定qtl和候选基因。GyD.F4y2Ba
BN是棉花单株产量的重要组成部分[GyD.F4y2Ba22.GyD.F4y2Ba].在先前的研究中,我们还发现BN是棉纤维产量最重要的因素[GyD.F4y2Ba20.GyD.F4y2Ba].然而,BN的高CV值和低遗传性(72.92%)表明该特征受到环境的大大影响,并且难以在遗传改进中控制。棉花的BW是受种子重量和纤维重量影响的复杂特性;因此,Si和LP是棉花产量改善的两个重要性状。在本研究中,LP和Si分别显示出高94.97%和92.08%的遗传性,类似于以前的报告[GyD.F4y2Ba12.GyD.F4y2Ba那GyD.F4y2Ba16.GyD.F4y2Ba].GyD.F4y2Ba
此外,在LP和Si之间鉴定了显着的负相关,并且鉴定了LP和Si的许多共同定位的QTL [GyD.F4y2Ba12.GyD.F4y2Ba那GyD.F4y2Ba22.GyD.F4y2Ba].目前,大多数研究都关注LP的遗传改善[GyD.F4y2Ba12.GyD.F4y2Ba那GyD.F4y2Ba21.GyD.F4y2Ba,而SI则相对较少。通过平衡棉花SI值和LP值的表型分析,发现了提高纤维产量的潜力。胚珠作为纤维的载体,在纤维发育中起着至关重要的作用[GyD.F4y2Ba24.GyD.F4y2Ba].操纵在纤维细胞中鉴定的一些候选基因没有产生对纤维产量或质量的效果或略微积极影响[GyD.F4y2Ba25.GyD.F4y2Ba],暗示卵子和纤维开发之间的协同效应促进棉花产量。GyD.F4y2Ba
我们发现许多纤维开发候选基因在胚珠中高度表达。在棉花的进化和驯化期间,棉纤维产量与种子尺寸的增加有强烈相关[GyD.F4y2Ba26.GyD.F4y2Ba那GyD.F4y2Ba27.GyD.F4y2Ba].然而,Si和LP之间的负相关表明,小种子有利于增加现代品种的棉纤维产量。因此,我们推测了合适的种子大小,有效地增加了棉纤维产量并保持种子活力。可以进一步探索对纤维发育对纤维发育的调节,以鉴定纤维产量改善的候选基因。GyD.F4y2Ba
先前的研究检测到通过GWAS分析检测到大量相关的纤维产量基因率。但是,产量特征是定量的继承和容易受环境因素的影响[GyD.F4y2Ba28.GyD.F4y2Ba].因此,大多数检测到的QTL难以确认和有限使用。在本研究中,确定了560 QTN。但是,几个QTN具有最小的QTN效果,几乎为零GyD.F4y2BaR.GyD.F4y2Ba2GyD.F4y2Ba,这可能是软件算法的假阳性,并且对繁殖无用。我们还发现这些QTNS低GyD.F4y2BaR.GyD.F4y2Ba2GyD.F4y2Ba值仅在单个环境中关联。因此,为了提高QTL的真实性和有效性,我们选择了在两个或多个环境中检测到的QTL,作为候选QTL。在95候选QTL中,在多个报告的研究中已经确定了38个共同定位的QTL [GyD.F4y2Ba12.GyD.F4y2Ba那GyD.F4y2Ba13.GyD.F4y2Ba那GyD.F4y2Ba15.GyD.F4y2Ba那GyD.F4y2Ba20.GyD.F4y2Ba那GyD.F4y2Ba21.GyD.F4y2Ba那GyD.F4y2Ba23.GyD.F4y2Ba那GyD.F4y2Ba29.GyD.F4y2Ba那GyD.F4y2Ba30.GyD.F4y2Ba].此外,在多种环境中检测到57个新型QTL;例如,QTL83在九种环境中与BW,LP和Si相关联,为棉花产物的遗传改善提供更多基因座和标记。我们还发现,大多数候选QTL是特异性的,暗示不同产量组分之间的独立调节机制。然而,在多种性状中同时鉴定了15个QTL,并通过标记辅助育种来改善棉花产量的滋润效果。GyD.F4y2Ba
为了识别适用于繁殖棉花使用的关键基因,我们选择了在多种环境中检测到的12个高置信QTL以进行进一步分析。在这些QTL中,检测到QTL热点包含三种新型QTL(QTL24,QTL25和QLT26),检测到A08上。QTL25解释了最高的表型变异,对于LP,26.49%,对于Si,20.25%。在QTL热点中,鉴定了15个候选基因和两个基因(GyD.F4y2Bagh_a08g0716GyD.F4y2Ba和GyD.F4y2BaGH_A08G0783GyD.F4y2Ba进一步分析了编码净超家族蛋白质,该蛋白质可能将不同的膜耦合到植物细胞中的肌动蛋白细胞骨架中。在GyD.F4y2Ba拟南芥GyD.F4y2Ba,Net1a以血浆膜固定在血浆膜上,在细胞连接处占主导地位,Plasmodesmata [GyD.F4y2Ba31.GyD.F4y2Ba].此外,还发现Net1A响应伴有脱钙酸(ABA),丝裂剂活化的蛋白激酶(MAPK)信号通路和UPLAND棉中的钙转导途径[GyD.F4y2Ba32.GyD.F4y2Ba].本研究中发现的两种净基因可能在光纤开发期间发挥重要作用。Si也是棉花产量的关键特征。QTL19仅在六个不同的环境中与SI相关联。在这个QTL区域内,GyD.F4y2Bagh_a07g1389GyD.F4y2Ba编码硫酸四肽重复(TPR),般的超家族蛋白,基因是报告的基因的同源基因,其负责LIGyD.F4y2BayGyD.F4y2Ba短纤维表型。沉默的沉默GyD.F4y2Ba李GyD.F4y2BayGyD.F4y2Ba基因可以显着降低高地棉中的纤维长度[GyD.F4y2Ba33.GyD.F4y2Ba].GyD.F4y2Bagh_a07g1389GyD.F4y2Ba在纤维开发中显示出高-3dPa至1 dPa的高表达,推断出胚珠中该基因的差异表达可能会影响棉花的纤维开发。还发现了稳定的QTL间隔中大量的纤维显影相关基因,例如GyD.F4y2BaGH_A08G0793GyD.F4y2Ba(GyD.F4y2BaSWEET12GyD.F4y2Ba)[GyD.F4y2Ba18.GyD.F4y2Ba] 和GyD.F4y2BaGH_D09G1670GyD.F4y2Ba(GyD.F4y2Baexpa4.GyD.F4y2Ba)[GyD.F4y2Ba34.GyD.F4y2Ba],这可能有助于育种实践的纤维产量提高。GyD.F4y2Ba
结论GyD.F4y2Ba
挖掘与产量组分相关的精英基因座和等位基因具有更实用的价值,可通过设计通过分子育种改善作物产量的遗传提高。这里,在多个现场环境下连续研究来自242个Upland棉签的四个主要产量组分,每株植物(BN),棉铃重量(BW),棉铃百分比(LP)和种子指数(Si)的表型数据。这些年。遗传分析表明,LP具有最高的遗传性,之后是SI,BW和BN。通过GWAS分析,我们识别出95个非冗余和稳定的QTL,分别为12,23,45,33,用于BN,BW,LP和Si。其中,15个QTL同时用两种或更多种以上特征相关联,促进磷酸棉产量组分,并且在超过六个环境中反复检测到12个QTL,如高置信QTL。此外,检测到10对A和D子基因组之间的同源QTL。此外,我们确定了两种净基因(GyD.F4y2Bagh_a08g0716GyD.F4y2Ba和GyD.F4y2BaGH_A08G0783GyD.F4y2Ba)定位于稳定的QTL热点(qtl24、qtl25和qlt26),在纤维发育早期主要表达。编码TPR样超家族蛋白的基因(GyD.F4y2Bagh_a07g1389GyD.F4y2Ba)在稳定的QTL19区域中,是改善棉花产量的重要候选基因。本研究可能有助于新颖的Elite基因座和基因资源,以提高棉育种实践。GyD.F4y2Ba
方法GyD.F4y2Ba
植物材料GyD.F4y2Ba
242份陆地棉花自然种群(附加文件)GyD.F4y2Ba1GyD.F4y2Ba按照中国当地农艺实践,在13个自然环境中种植,包括2011年、2012年和2013年的库尔勒、新乡和南阳,2016年和2017年的沙湾,2018年的盐城和当土(补充文件)GyD.F4y2Ba2GyD.F4y2Ba表S2)。所有材料采用随机完全区组设计(RCBD)种植,每个环境中有两个重复,每个重复中有两行。库尔勒和沙湾的行长为2 m,行距为66 + 10 cm(宽/窄),株距为10 cm。新乡市行长5 m,行距80 cm,株距25 cm。南阳、盐城和当土的行长为4.5 m,行距为80 cm,株距为33 cm。所有植物均由南京农业大学收集保存。所有必要的种植和调查自然种群的许可证都获得了中国南京农业大学。GyD.F4y2Ba
表型调查和数据分析GyD.F4y2Ba
在每种环境中测量四个产量相关的特征BN,BW,LP和Si,以探索棉花的表型变异。在棉花成熟阶段,每行中间随机选择每次加入的10厂。BN与每次加入的10个生物学复制计数。称重BW,用20种成熟的棉铃样品随机从中间枝上,每株植物2个铃。在轧辊之后,基于棉棉重量的胶质重量的分数计算LP,用100种通常开发的种子称重Si。GyD.F4y2Ba
在本研究中,来自13个自然环境的BW和LP和11个自然环境的BN和SI的数据用于分析。基于每个特征的混合线性模型的基因型效应的最佳线性非偏见预测因子(BLUP)由R软件的LME4包进行[GyD.F4y2Ba35.GyD.F4y2Ba].育种值由最佳线性无偏预测因子加种群均值(标记为“BLUP”)评估,并用于相关分析和GWAS。利用R软件中的“ggplot2”软件包进行表型值密度分布。使用SAS软件PROC GLM程序对不同环境下的表型数据进行方差分析[GyD.F4y2Ba36.GyD.F4y2Ba].不同来源的所有变化被视为随机效应,随机块线性模型。统计模型是yGyD.F4y2BaIJK.GyD.F4y2Ba=μ + α一世GyD.F4y2Ba + βjGyD.F4y2Ba + γkjGyD.F4y2Ba + (αβ)IJ.GyD.F4y2Ba + εIJK.GyD.F4y2Ba,μ表示整体平均值αGyD.F4y2Ba一世GyD.F4y2Ba表明我的遗传效果GyD.F4y2BaTH.GyD.F4y2Ba基因型,βGyD.F4y2BajGyD.F4y2Ba表示j的效果GyD.F4y2BaTH.GyD.F4y2Ba环境中,γGyD.F4y2BakjGyD.F4y2Ba表示k的随机效果GyD.F4y2BaTH.GyD.F4y2Baj复制GyD.F4y2BaTH.GyD.F4y2Ba环境,(αβ)GyD.F4y2BaIJ.GyD.F4y2Ba表示i之间的交互效应GyD.F4y2BaTH.GyD.F4y2Ba基因型和jGyD.F4y2BaTH.GyD.F4y2Ba环境中,εGyD.F4y2BaIJK.GyD.F4y2Ba剩余值[GyD.F4y2Ba37.GyD.F4y2Ba].该公式计算每个特征的广义遗传性:GyD.F4y2BaHGyD.F4y2Ba2GyD.F4y2Ba=GyD.F4y2BaσGyD.F4y2Ba2GyD.F4y2BaGGyD.F4y2Ba/(σ.GyD.F4y2Ba2GyD.F4y2BaGGyD.F4y2Ba + σGyD.F4y2Ba2GyD.F4y2BaGEGyD.F4y2Ba/ N.GyD.F4y2Ba + σGyD.F4y2Ba2GyD.F4y2BaεGyD.F4y2Ba/ rn)GyD.F4y2Ba, 那GyD.F4y2BaσGyD.F4y2Ba2GyD.F4y2BaGGyD.F4y2Ba基因型方差,GyD.F4y2BaσGyD.F4y2Ba2GyD.F4y2BaGEGyD.F4y2Ba表示环境互作方差的基因型,GyD.F4y2BaσGyD.F4y2Ba2GyD.F4y2BaεGyD.F4y2Ba表示错误方差,GyD.F4y2BaNGyD.F4y2Ba表示环境的数量和GyD.F4y2BaR.GyD.F4y2Ba复制次数。[GyD.F4y2Ba38.GyD.F4y2Ba].采用SAS软件中SAS VARCOMP程序的REML方法计算广义遗传力的方差值。采用SPSS 22.0软件进行性状间的相关系数和显著性分析。GyD.F4y2Ba
SNP基因分型GyD.F4y2Ba
每个加入的幼叶组织的基因组DNA是用Paterson等人(1993)描述的CTAB方法提取的[GyD.F4y2Ba39.GyD.F4y2Ba].242份材料采用CottonSNP80K序列进行基因分型,其中包含77774个SNPs [GyD.F4y2Ba16.GyD.F4y2Ba].在使用<0.9和MAF <0.05的呼叫速率过滤SNP后,获得高质量的SNP。高质量SNP的探针序列被映射到TM-1 V2.1基因组[GyD.F4y2Ba11.GyD.F4y2Ba]确定确切的物理位置。当探测序列映射到多个位置时,丢弃相应的SNP。最后,使用包含56,010个独特映射的高质量SNP的数据集用于GWAS分析。GyD.F4y2Ba
GWAS分析GyD.F4y2Ba
基于多基因座随机SNP效应混合线性模型(MRMLM)[GyD.F4y2Ba22.GyD.F4y2Ba[R包“MRMLM”中,六种方法(“MRMLM”,“FastMrMLM”,“FastMREMMMLM”,“FastMREMMA”,“PKWMEB”,“isis EM-Blasso”和“PLarmeB”)用于识别每个特征的QTN。程序的参数被设置为:至关重要GyD.F4y2BaP.GyD.F4y2Ba-Value = 0.001;搜索候选基因的半径(KB)= 100;临界LOD评分= 3.此外,我们选择了GWAS分析的Q + K统计模型。直接在“MRMLM”计划中获得亲属(K)矩阵。根据我们以前的研究[GyD.F4y2Ba40GyD.F4y2Ba选择,K = 3被选中,并且通过混合版本1.3计算群体结构(Q)矩阵。52个数据集,包括来自不同环境的表型数据和每个特征的弹簧,用于GWAS分析。GyD.F4y2Ba
qtl和候选基因的鉴定GyD.F4y2Ba
由于基因分型SNP的分布不均匀,并在上一份报告中引用[GyD.F4y2Ba19.GyD.F4y2Ba[我们选择了染色体的最低LD(200kb)作为阈值,以将相邻的QTN合并到相同的QTL中。根据自写壳脚本,从参考基因组上的QTL区域获得候选基因。为了确定哪种基因与棉花产量组分有关,TM-1组织的转录组FASTQ数据,包括根,茎,叶,花瓣,圆环,萼片,苞片,花药,长丝,雌蕊,胚轴和纤维组织在-3,在50,15,20和25dPa的50,15,20和25dPa下的0,1,3,5天,10,15,20和25dPa的胚珠,从NCBI序列读取归档集合Prjna490626[GyD.F4y2Ba11.GyD.F4y2Ba].使用映射软件hisat2 [GyD.F4y2Ba41.GyD.F4y2Ba],唯一映射的读数被映射到参考基因组,并使用HTSEQ计数计算所有棉花注释基因[GyD.F4y2Ba42.GyD.F4y2Ba].以下参数用于计数过程:-f bam,-r名称,-s no,-munion,-a 20.这些计数文件用于使用程序编辑器识别DEGS [GyD.F4y2Ba43.GyD.F4y2Ba].当TPM(每百万)大于3时,候选基因被认为是稳定的并且用于进一步分析。Agrigo v2.0是在线生物信息学工具,用于分析候选基因[GyD.F4y2Ba44.GyD.F4y2Ba].GyD.F4y2Ba
可用性数据和材料GyD.F4y2Ba
本研究中的RNA-SEQ数据已存入国家生物技术信息中心(NCBI,GyD.F4y2Bahttp://www.ncbi.nlm.nih.gov/GyD.F4y2Ba)在“加入”Prjna490626下。GyD.F4y2Ba
缩写GyD.F4y2Ba
- Anova:GyD.F4y2Ba
-
方差分析GyD.F4y2Ba
- BLUPs:GyD.F4y2Ba
-
最好的线性无偏见预测因子GyD.F4y2Ba
- BLUP:GyD.F4y2Ba
-
最好的线性无偏见预测GyD.F4y2Ba
- BN:GyD.F4y2Ba
-
棉子数量GyD.F4y2Ba
- 去:GyD.F4y2Ba
-
基因本体论GyD.F4y2Ba
- GWAS:GyD.F4y2Ba
-
全基因组关联研究GyD.F4y2Ba
- SI:GyD.F4y2Ba
-
种子指数GyD.F4y2Ba
- LD:GyD.F4y2Ba
-
连锁不平衡GyD.F4y2Ba
- LP:GyD.F4y2Ba
-
线头百分比GyD.F4y2Ba
- 加:GyD.F4y2Ba
-
轻微的等位基因频率GyD.F4y2Ba
- QTLS:GyD.F4y2Ba
-
定量特质基因座GyD.F4y2Ba
- 本考察团:GyD.F4y2Ba
-
定量性状核苷酸GyD.F4y2Ba
- BW:GyD.F4y2Ba
-
棉铃重GyD.F4y2Ba
- SNP:GyD.F4y2Ba
-
单核苷酸多态性GyD.F4y2Ba
参考文献GyD.F4y2Ba
- 1。GyD.F4y2Ba
Chen Zj,Scheffler,Dennis E,Triplett Ba,张T,Guo W,Chen X,Betelly DM,Rabinowicz PD,Town CD,等。朝向棉花(GyD.F4y2BaGossypiumGyD.F4y2Ba)的基因组。植物杂志。2007;145(4):1303 - 10。GyD.F4y2Ba
- 2。GyD.F4y2Ba
张赫伯,李义,王b,chee pw。棉基因组学的最新进展。int j植物基因组。2008; 2008:742304。GyD.F4y2Ba
- 3.GyD.F4y2Ba
秦辉,郭伟,张玉明,张涛。基于四杂交群体的产量和纤维性状QTL定位GyD.F4y2Ba陆地棉GyD.F4y2BaL. Al Appl Genet。2008; 117(6):883-94。GyD.F4y2Ba
- 4.GyD.F4y2Ba
沉XL,郭WZ,鲁QX,朱XF,元友,张TZ。旱地棉花纤维质量定量特质基因座的遗传映射。Euphytica。2007; 155(3):371-80。GyD.F4y2Ba
- 5。GyD.F4y2Ba
刘兰德,王立,郭禾,秦烨,王立,张扬,张tz。使用两种永生杂交种群的产量及其组分的定量特性基因座测绘GyD.F4y2Ba陆地棉GyD.F4y2BaL. mol品种。2012; 29(2):297-311。GyD.F4y2Ba
- 6。GyD.F4y2Ba
张涛,胡颖,姜伟,方磊,关旭,陈建军,张建军,Saski CA, Scheffler BE, Stelly DM,等。异源四倍体棉花的测序(GyD.F4y2Ba陆地棉GyD.F4y2BaL. ACC。TM-1)提供了一种用于纤维改进的资源。NAT BIOTECHNOL。2015; 33(5):531-7。GyD.F4y2Ba
- 7。GyD.F4y2Ba
李飞,粉丝,卢克,萧g,zou cs,kohel rj,ma zy,shang hh,ma xf,wujy等。栽培高地棉花的基因组序列(GyD.F4y2Ba陆地棉GyD.F4y2BaTM-1)提供了基因组进化的见解。生物科技本质》。2015;33(5):524 - 30。GyD.F4y2Ba
- 8。GyD.F4y2Ba
袁丹杰,唐振华,王明杰,高卫华,涂丽丽,金鑫,陈丽,何艳红,张丽,朱立峰,等。海岛棉(GyD.F4y2BaGossypium Barbadense.GyD.F4y2Ba)提供对各种可纺纱纤维的全多聚纤维化和开发的见解。SCI REP。2015; 5:17662。GyD.F4y2Ba
- 9。GyD.F4y2Ba
刘X,赵b,郑hj,胡y,lu g,杨cq,陈俊,陈俊,陈代,张l等。GyD.F4y2BaGossypium Barbadense.GyD.F4y2Ba基因组序列提供了对超长短纤维和专门代谢物的演变的洞察力。SCI REP。2015; 5:14139。GyD.F4y2Ba
- 10.GyD.F4y2Ba
王M,Tu L,Yuan D,Zhu D,Shen C,Li J,Liu F,Pei L,Wang P,Zhao G等。两倍培养的同种异体四倍体棉花的参考基因组序列,GyD.F4y2Ba陆地棉GyD.F4y2Ba和GyD.F4y2BaGossypium Barbadense.GyD.F4y2Ba.51 Nat麝猫。2019;(2):224 - 9。GyD.F4y2Ba
- 11.GyD.F4y2Ba
胡勇,陈建东,方磊,张志勇,马伟,牛永昌,鞠立忠,邓建强,赵涛,连建民,等。GyD.F4y2BaGossypium Barbadense.GyD.F4y2Ba和GyD.F4y2Ba陆地棉GyD.F4y2Ba基因组为异源四倍体棉花的起源和进化提供了深刻的见解。51 Nat麝猫。2019;(4):739 - 48。GyD.F4y2Ba
- 12.GyD.F4y2Ba
歌曲c,李w,pei x,liu y,ren z,他k,zhang f,sun k,周x,ma x等。旱地棉花基因组关联研究的粘液变异遗传变异和候选基因的解剖。Al Appl Genet。2019; 132(7):1991-2002。GyD.F4y2Ba
- 13。GyD.F4y2Ba
方l,王q,胡y,jia y,chen j,liu b,张z,guan x,陈某,周b等。棉质基因组分析识别与纤维质量和产量特征相关的选择和基因座的签名。NAT Genet。2017; 49(7):1089-98。GyD.F4y2Ba
- 14。GyD.F4y2Ba
王M,Tu L,Lin M,Lin Z,王P,杨Q,ye z,沉c,李j,zhang l等。棉花驯化期间的不对称亚基因选择和顺式调节性分歧。NAT Genet。2017; 49(4):579-87。GyD.F4y2Ba
- 15.GyD.F4y2Ba
Ma Z,他,王X,Sun J,张Y,张G,吴L,李Z,刘Z,Sun G等。重新安排普通棉的核心集合鉴定了影响纤维质量和产量的基因组变异和基因座。NAT Genet。2018; 50(6):803-13。GyD.F4y2Ba
- 16。GyD.F4y2Ba
黄C,NIE X,Shen C,You C,Li W,Zhao W,Zhang X,Lin Z.利用高密度的全基因组关联研究揭示了中国高地棉花农艺性状的人口结构和遗传基础snps。植物Biotechnol J. 2017; 15(11):1374-86。GyD.F4y2Ba
- 17。GyD.F4y2Ba
Cai C,Guozhong Z,Tianzhen Z,Wangzhen G.高密度80 K SNP阵列是基因分型的强大工具GyD.F4y2BaG. HirsutumGyD.F4y2Ba资源和基因组分析。BMC染色体组。2017;18(1):654。GyD.F4y2Ba
- 18。GyD.F4y2Ba
Wang R, Liu M, Yuan M, Oses-Prieto JA, Cai X, Sun Y, Burlingame AL, Wang ZY, Tang W. The brassinosteroid-activated BRI1 receptor kinase is switched off by dephosphorylation mediated by cytoplasm-localized PP2A B’ subunits. Mol Plant. 2016;9(1):148–57.
- 19。GyD.F4y2Ba
张泽,阮益,周,王某,关西,方l,尚xg,郭wz,zhu sj,张tz。抑制推定的甾醇载体基因降低了棉纤维伸长率期间的等离子体渗透性并在棉纤维伸长期间激活蔗糖转运蛋白基因。植物细胞。2017; 29(8):2027-46。GyD.F4y2Ba
- 20.GyD.F4y2Ba
朱克,高,宋X,太阳f,侯s,刘n,黄y,张d,ni z,chen q等。基因组 - 宽协会揭示了棉花咸田间条件下棉绒产量组分的遗传变异(GyD.F4y2Ba陆地棉GyD.F4y2BaL.)。BMC植物BIOL。2020; 20(1):23。GyD.F4y2Ba
- 21.GyD.F4y2Ba
苏军,范胜,李丽,王超,王辉,宋敏,张超,顾丽,赵胜,等。中国陆地棉衣分QTL优良等位基因和候选基因的检测。植物学报2016;7:1576。GyD.F4y2Ba
- 22.GyD.F4y2Ba
孙志军,王旭,刘震,顾强,张勇,李智,柯辉,杨杰,吴磊,等。一项全基因组关联研究揭示了陆地棉花产量相关性状的新基因组区域和候选基因。中国科学(d辑:地球科学)2018;GyD.F4y2Ba
- 23。GyD.F4y2Ba
王某某,冯·耶,仁WL,黄b,周l,温yj,张j,dunwell jm,xu s,张扬。通过多基因座混合线性模型方法提高基因组关联研究的功率和准确性。SCI REP。2016; 6:19444。GyD.F4y2Ba
- 24。GyD.F4y2Ba
阮yl。了解棉纤维和种子发展的最新进展。种子sci res。2005; 15(4):269-80。GyD.F4y2Ba
- 25。GyD.F4y2Ba
推进种子发育作为提高棉纤维产量和品质的新战略。acta botanica yunnanica(云南植物研究). 2013;55(7):572-5。GyD.F4y2Ba
- 26。GyD.F4y2Ba
Applequist WL,Cronn R,Wendel JF。纤维纤维纤维的比较发展。evol dev。2001; 3(1):3-17。GyD.F4y2Ba
- 27。GyD.F4y2Ba
张志强,张志强,张志强,等。棉花籽粒和纤维生物量的变化及其与转运细胞的关系。摩尔。2010;3(6):1075 - 86。GyD.F4y2Ba
- 28。GyD.F4y2Ba
帕特森啊,Saranga Y,Menz M,Jiang CX,Wright RJ。QTL基因型X环境相互作用影响棉纤维质量。Al Appl Genet。2003; 106(3):384-96。GyD.F4y2Ba
- 29。GyD.F4y2Ba
张泽,尚H,石y,黄l,李茹,葛q,龚j,刘安,陈t,王d等。通过特定基因座扩增片段测序(SLAF-SEQ)构建高密度遗传图谱及其在高地棉花中棉铃重量的定量性状基因座(QTL)分析的应用(GyD.F4y2BaGossypium hirsutum。GyD.F4y2Ba)。BMC植物BIOL。2016; 16:79。GyD.F4y2Ba
- 30.GyD.F4y2Ba
Nazir MF,贾Y,艾哈迈德H,HE S,IQBAL MS,Sarfraz Z,Ali M,Feng C,Raza I,Sun G等。基因组洞察分化和选择扫描旱地棉花的改善。植物。2020; 9(6):711。GyD.F4y2Ba
- 31.GyD.F4y2Ba
Deeks Mj,Calcutt Jr,Ingle Eks,Hawkins TJ,Chapman S,Richardson Ac,Mentlak da,Dixon Mr,Cartwright F,Smertenko Ap,等。在高等植物肌动蛋白膜Nexus的肌动蛋白结合蛋白的超家族。Curr Biol。2012; 22(17):1595-600。GyD.F4y2Ba
- 32.GyD.F4y2Ba
李·赫姆,刘SD,葛cw,张xm,张sp,陈继,沉q,ju fy,yang yf,li y等。幼苗阶段旱地棉花耐旱性及相关性状分析。int j mol sci。2019; 20(16):3888。GyD.F4y2Ba
- 33.GyD.F4y2Ba
芳迪德,Naoumkina M,Thyssen Gn,Bechere E,Li P,Flinane CB。在四氢肽重复的超家族蛋白基因中的EMS诱导突变(GyD.F4y2Baghir_a12g008870.GyD.F4y2Ba)在A12染色体上是引起棉花li(y)短纤维表型的主要原因。中国生物医学工程学报。2020;133(1):271-82。GyD.F4y2Ba
- 34.GyD.F4y2Ba
LV LM,Zuo Dy,Wang XF,Cheng HL,张YP,王QL,歌曲GL,MA ZY。扩大素基因的范围内鉴定揭示扩张素基因参与棉花纤维细胞生长。BMC植物BIOL。2020; 20(1):223。GyD.F4y2Ba
- 35。GyD.F4y2Ba
Bates D, Machler M, Bolker BM, Walker SC.使用lme4拟合线性混合效应模型。中国科学(d辑:信息科学),2015;GyD.F4y2Ba
- 36。GyD.F4y2Ba
SAS I. Base SAS 9.4程序指南:统计程序。Cary, NC, USA: SAS Institute Inc, 2013。GyD.F4y2Ba
- 37。GyD.F4y2Ba
李XN,张XL,朱LM,BU YP,Wang XF等。大豆R6阶段四种产量相关性状的基因组 - 宽协会研究。BMC Genet。2019; 20(1):39。GyD.F4y2Ba
- 38。GyD.F4y2Ba
Piepho HP,Mohring J.计算遗传性和选择性植物育种试验的遗传性和选择响应。遗传学。2007; 177:1881-8。GyD.F4y2Ba
- 39。GyD.F4y2Ba
帕特森啊,布鲁克Cl,Wendel JF。一种萃取棉花的快速方法(GyD.F4y2BaGossypiumGyD.F4y2BaSPP。)适用于RFLP或PCR分析的基因组DNA。植物mol Biol rep.1993; 11(2):122-7。GyD.F4y2Ba
- 40.GyD.F4y2Ba
朱G,李W,王G,Li L,Si Q,Cai C,Guo W.纤维改善的遗传基础,栽培培养与半驯化高地棉花胁迫降低。前植物SCI。2019; 10:1572。GyD.F4y2Ba
- 41.GyD.F4y2Ba
Pertea M,Kim D,Pertea Gm,韭菜JT,Salzberg SL。Hisat Stringtie和Ballgown的RNA-SEQ实验的转录级表达分析。NAT PROTOC。2016; 11(9):1650-67。GyD.F4y2Ba
- 42.GyD.F4y2Ba
Anders S,Pyl Pt,Huber W. Htseq-A Python框架,用于使用高吞吐量排序数据。生物信息学。2015; 31(2):166-9。GyD.F4y2Ba
- 43.GyD.F4y2Ba
罗宾逊医学博士,麦卡锡DJ,史密斯GK。edgeR: Bioconductor包,用于数字基因表达数据的差异表达分析。生物信息学,2010,26(1):139 - 40。GyD.F4y2Ba
- 44.GyD.F4y2Ba
田T,刘y,yan h,你q,yi x,du z,xu w,su z.农业多v2.0:2017年农业社区的GO分析工具包,2017年更新。核酸RES。2017; 45(W1):W122-9。GyD.F4y2Ba
致谢GyD.F4y2Ba
感谢南京农业大学棉花种质创新与应用工程研究中心(教育部)的成员和合作者多年来对自然群体和环境的持续表型调查。感谢南京农业大学农学院的Muhammad Waqas Amjid博士的帮助编辑和评论。南京农业大学生物信息学中心的高性能计算平台提供了数据分析设施,国家棉花中期基因库提供了一些地方品种种子。GyD.F4y2Ba
资金GyD.F4y2Ba
国家作物育种重点研发计划(2018YFD0100400)、中央高校基本科研业务费专项资金(KYYJ201901;KYZZ201904;作物遗传与种质创新国家重点实验室开放基金项目(ZW202007),江苏省现代作物生产协同创新中心项目(10号)。资助方没有参与研究的实验设计、数据收集、分析和解释,也没有参与手稿的撰写。GyD.F4y2Ba
作者信息GyD.F4y2Ba
从属关系GyD.F4y2Ba
贡献GyD.F4y2Ba
实验由WZG设计。通过GZZ,XHS,SH,XW,WW和QJC进行实验。GZZ和SH起草了稿件,WZG修改了手稿。所有作者阅读并认可的终稿。GyD.F4y2Ba
相应的作者GyD.F4y2Ba
伦理宣言GyD.F4y2Ba
伦理批准和同意参与GyD.F4y2Ba
不适用。GyD.F4y2Ba
同意出版物GyD.F4y2Ba
不适用。GyD.F4y2Ba
利益争夺GyD.F4y2Ba
作者宣布他们没有竞争利益。GyD.F4y2Ba
附加信息GyD.F4y2Ba
出版商的注意GyD.F4y2Ba
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。GyD.F4y2Ba
补充信息GyD.F4y2Ba
附加文件1:表S1。GyD.F4y2Ba
本研究中使用的242个高地棉花养护的信息。GyD.F4y2Ba
附加文件2:表S2。GyD.F4y2Ba
多场环境中四个产量相关性状的表型变化和结合。GyD.F4y2Ba
附加文件3:图S1。GyD.F4y2Ba
陆地棉自然群体中4个产量相关性状的密度分布GyD.F4y2Ba
附加文件4:表S3。GyD.F4y2Ba
利用CottonSNP80K序列对高品质snp进行基因分型。GyD.F4y2Ba
附加文件5:表S4。GyD.F4y2Ba
QTNS和QTLS每植物,棉铃重量,棉绒百分比和由多基因座MLM模型检测的种子指数。GyD.F4y2Ba
附加文件6:表S5。GyD.F4y2Ba
对4个产量相关性状的候选qtl、共定位qtl和高置信度qtl进行了鉴定。GyD.F4y2Ba
附加文件7:表S6。GyD.F4y2Ba
四个产量相关性状的同源QTL的信息。GyD.F4y2Ba
附加文件8:图S2。GyD.F4y2Ba
在At和Dt亚基因组上与产量相关性状相关的候选qtl数量。GyD.F4y2Ba
附加文件9:图S3。GyD.F4y2Ba
与四个产量相关的特征相关的QTL Venn图。GyD.F4y2Ba
附加文件10:表S7。GyD.F4y2Ba
4个产量相关性状QTL区域候选基因的表达谱。GyD.F4y2Ba
附加文件11:表S8。GyD.F4y2Ba
4个产量相关性状QTL区相关基因功能分析。GyD.F4y2Ba
附加文件12:图S4。GyD.F4y2Ba
与QLT19相关的候选基因与Si相关并位于染色体A07上。GyD.F4y2Ba一种GyD.F4y2Ba.Q119中候选基因的表达热图。GyD.F4y2BaB.GyD.F4y2Ba.表达模式GyD.F4y2Bagh_a07g1389GyD.F4y2Ba在不同的组织。GyD.F4y2BaCGyD.F4y2Ba.盒子图表最接近QTN的表型值GyD.F4y2Bagh_a07g1389GyD.F4y2Ba.GyD.F4y2Ba
权利和权限GyD.F4y2Ba
开放访问GyD.F4y2Ba本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问GyD.F4y2Bahttp://creativecommons.org/licenses/by/4.0/GyD.F4y2Ba.Creative Commons公共领域奉献豁免(GyD.F4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/GyD.F4y2Ba)适用于本文提供的数据,除非在数据的信贷额度中另有说明。GyD.F4y2Ba
关于这篇文章GyD.F4y2Ba
引用这篇文章GyD.F4y2Ba
朱,G.,侯,S.,歌,X.GyD.F4y2Ba等等。GyD.F4y2Ba基因组 - 范围的关联分析显示在棉花的多场环境下的产量部件中涉及的定量性状性状基因座和候选基因(GyD.F4y2Ba陆地棉GyD.F4y2Ba)。GyD.F4y2BaBMC植物BIOL.GyD.F4y2Ba21,GyD.F4y2Ba250(2021)。https://doi.org/10.1186/s12870-021-03009-2GyD.F4y2Ba
收到了GyD.F4y2Ba:GyD.F4y2Ba
公认GyD.F4y2Ba:GyD.F4y2Ba
发表GyD.F4y2Ba:GyD.F4y2Ba
迪伊GyD.F4y2Ba:GyD.F4y2Bahttps://doi.org/10.1186/s12870-021-03009-2GyD.F4y2Ba
关键词GyD.F4y2Ba
- 陆地棉GyD.F4y2Ba
- 产量组件GyD.F4y2Ba
- 全基因组关联研究GyD.F4y2Ba
- 定量特质基因座GyD.F4y2Ba
- 多个领域的环境GyD.F4y2Ba